生物信息学-蛋白质分析
生物信息学-课堂练习作业生物信息学蛋白质序列分析-课堂练习
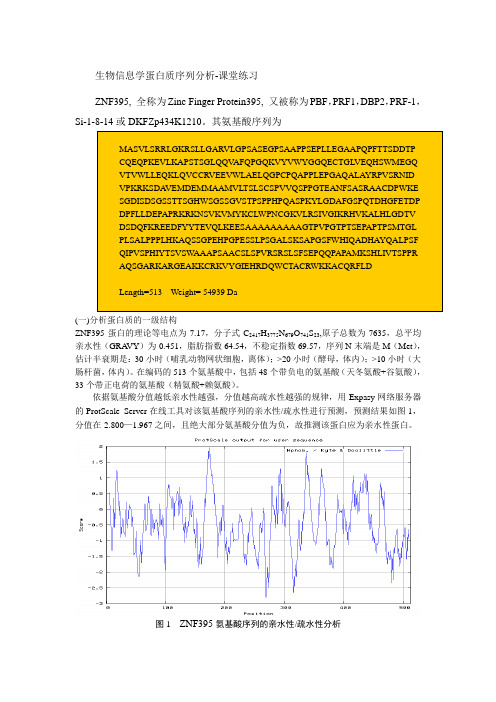
生物信息学蛋白质序列分析-课堂练习ZNF395, 全称为Zinc Finger Protein395, 又被称为PBF ,PRF1,DBP2,PRF-1,Si-1-8-14或DKFZp434K1210。
其氨基酸序列为(一)分析蛋白质的一级结构ZNF395蛋白的理论等电点为7.17,分子式C 2417H 3775N 679O 741S 23,原子总数为7635,总平均亲水性(GRA VY )为-0.451,脂肪指数64.54,不稳定指数69.57,序列N 末端是M (Met ),估计半衰期是:30小时(哺乳动物网状细胞,离体);>20小时(酵母,体内);>10小时(大肠杆菌,体内)。
在编码的513个氨基酸中,包括48个带负电的氨基酸(天冬氨酸+谷氨酸),33个带正电荷的氨基酸(精氨酸+赖氨酸)。
依据氨基酸分值越低亲水性越强,分值越高疏水性越强的规律,用Expasy 网络服务器的ProtScale Server 在线工具对该氨基酸序列的亲水性/疏水性进行预测,预测结果如图1,分值在-2.800—1.967之间,且绝大部分氨基酸分值为负,故推测该蛋白应为亲水性蛋白。
图1 ZNF395氨基酸序列的亲水性/疏水性分析(二)分析蛋白质的二级结构利用SOPMA在线工具对二级结构进行预测,如图2,α螺旋99个占19.30%,延伸链66个占12.87%,β-转角18个占3.51%,无规卷曲330个占64.33%,其二级结构主要由无规卷曲组成。
图2 ZNF395蛋白二级结构预测注:蓝色表示α螺旋;红色表示延伸链;紫色表示无规则卷曲(三)分析膜蛋白质利用在线分析工具TMHMM Server 2.0,对ZNF395氨基酸跨膜结构域进行在线预测和分析,结果表明,该序列编码的蛋白非跨膜蛋白(见图3)。
利用Signal P 3.0 Server在线预测工具对ZNF395蛋白质进行信号肽预测,无信号肽存在(图4)。
生物信息学实验报告3(三)蛋白质序列分析

⽣物信息学实验报告3(三)蛋⽩质序列分析(三)蛋⽩质序列分析实验⽬的:掌握蛋⽩质序列检索的操作⽅法,熟悉蛋⽩质基本性质分析,了解蛋⽩质结构分析和预测。
实验内容:1、检索SOX-21蛋⽩质序列,利⽤ProParam⼯具进⾏蛋⽩质的氨基酸组成、分⼦质量、等电点、氨基酸组成、原⼦总数及疏⽔性(ProtScale⼯具)等理化性质的分析。
2、利⽤PredictProtein、PROF、HNN等软件预测分析蛋⽩质的⼆级结构;利⽤Scan Prosite软件对蛋⽩质进⾏结构域分析。
3、利⽤TMHMM、TMPRED、SOSUI等⼯具对蛋⽩质进⾏跨膜分析;采⽤PredictNLS进⾏核定位信号分析;利⽤PSORT进⾏蛋⽩质的亚细胞定位预测;利⽤CBS(http://www.cbs.dtu.dk/services/ProtFun/)⽹站⼯具预测蛋⽩的功能,将序列⽤Blocks、SMART、InterProScan、PFSCAN等搜索其保守序列的特征,进⾏motif 的结构分析。
4、利⽤Swiss-Model数据库软件预测该蛋⽩的三级结构,结果⽤蛋⽩质三维图象软件Jmol查看。
CPHmodels 也是利⽤神经⽹络进⾏同源模建预测蛋⽩质结构的⽅法和⽹络服务器I-TASSER预测所选蛋⽩质的空间结构。
5、分析蛋⽩质的翻译后修饰:分析信号肽及其剪切位点: SignalIP http://www.cbs.dtu.dk/services/SignalP/;分析糖链连接点:分析O-连接糖蛋⽩,NetOGlyc,http://www.cbs.dtu.dk/services/NetOGlyc/;分析N-连接糖蛋⽩,NetNGlyc,http://www.cbs.dtu.dk/services/NetNGlyc/。
6、利⽤检索的序列,进⾏同源⽐对,获得并分析⽐对结果。
实验步骤(⼀)1、在NCBI 蛋⽩质数据库中查找SOX-21蛋⽩质序列分别选择⽖蟾(Xenopus laevis)、⼩家⿏[Mus musculus]、猕猴[Macaca mulatt a]的SOX-21蛋⽩质序列,并保存其FASTA格式。
2-蛋白质序列特征分析-生物信息学

TMPRED在线网页
生命科学学院
用TMPRED分析P51684序列所得到生的命可科能学学院 的7个跨膜螺旋区
生命科学学院
用TMPRED分析P51684序列所得到的7个可 能的跨膜螺旋区的相关性列表
含有卷曲螺旋结构最知名的蛋白质有原癌蛋白 (oncoprotein)c-fos和jun,以及原肌球蛋白 (tropomyosin)。
生命科学学院
利用COILS分析蛋白质的卷曲螺旋
COILS是由Swiss EMBNet维护的预测卷曲螺旋的在 线工具,该软件是基于Lupas算法,将查询序列在一个由 已知包含卷曲螺旋蛋白结构的数据库中进行搜索,同时也 将查询序列与包含球状蛋白序列的PDB次级库进行比较, 并根据两个库搜索得分决定查询序列形成卷曲螺旋的概率。 COILS也可以下载到本地进行运算。
生命科学学院
序列特征分析
Analysis of Sequence Characterristics
一、蛋白质结构 蛋白质的一级结构
生命科学学院
蛋白质的一级结构决定二级结构 蛋白质的二级结构决定三级结构
蛋白质的二级结构
生命科学学院
H表示螺旋 E表示折叠 B表示β桥 G表示3-螺旋 I表示π螺旋 T表示氢键转角 S代表转向
或者全部由碳原子和氢原子组成,因此这类氨基酸不太可 能与水分子形成氢键; 2. 极性氨基酸(polar amino acid),其测链通常由氧原子或 氮原子组成,它们比较容易与水分子形成氢键,因此也称 为亲水氨基酸; 3. 带电氨基酸(charged amino acids),这类氨基酸在生物 pH环境中带有正电或负电。
生命科学学院
第五章蛋白质分析及预测方法

第五章蛋白质分析及预测方法蛋白质是生物体内最基本的功能分子之一,其功能与结构密切相关。
蛋白质分析及预测方法是研究蛋白质结构和功能的重要手段之一、随着生物信息学和计算机技术的发展,越来越多的蛋白质分析及预测方法被提出和应用。
一、蛋白质分析方法1.序列分析蛋白质序列是理解和预测蛋白质功能和结构的重要基础。
序列分析可以通过比对已知蛋白质序列数据库,找出与待研究蛋白质相似的序列,从而预测其功能和结构。
常用的序列分析方法包括同源序列比对、Motif和Domain分析等。
2.结构分析蛋白质结构是蛋白质功能的基础,因此结构分析对于研究蛋白质功能至关重要。
通常通过实验方法如X射线晶体学、核磁共振等获得蛋白质结构。
此外,还可以利用计算方法预测蛋白质的二级结构和三级结构。
常用的结构分析方法包括蛋白质结构比对、分子模拟等。
3.功能分析蛋白质功能是指蛋白质所具有的生物学功能,如催化反应、运输物质、信息传递等。
功能分析通过研究蛋白质的序列和结构,以及模拟蛋白质与其他生物分子的相互作用,来理解和预测蛋白质的功能。
常用的功能分析方法包括结构-功能关系预测、生物分子对接等。
二、蛋白质预测方法1.序列预测蛋白质序列预测是指通过分析蛋白质的氨基酸序列,预测其结构和功能。
常见的序列预测方法包括序列比对、Motif和Domain预测、蛋白质家族预测等。
这些预测方法可以通过比对已知蛋白质序列数据库,找出与待研究蛋白质相似的序列,从而推测其结构和功能。
2.结构预测蛋白质的三级结构是指蛋白质的原子级结构,包括蛋白质中氨基酸残基的空间排列。
结构预测是通过计算方法来预测蛋白质的三级结构。
常用的结构预测方法包括亚氨基酸残基建模、蛋白质折叠模拟等。
这些方法通过计算蛋白质中氨基酸之间的相互作用力和空间约束,来预测蛋白质的三级结构。
3.功能预测蛋白质功能预测是通过研究蛋白质的结构和序列,来预测蛋白质所具有的生物学功能。
常用的功能预测方法包括结构-功能关系预测、蛋白质分子对接等。
生物信息学中的蛋白质结构预测与分析

生物信息学中的蛋白质结构预测与分析生物信息学是一个研究生物学中的信息处理和分析的交叉学科,在生物科学领域中扮演着重要的角色。
其中,蛋白质结构预测与分析是生物信息学中的一个重要领域。
蛋白质是生物体内最基本的功能分子,其结构与功能密切相关。
因此,了解蛋白质的结构信息对于理解其功能和启示药物设计具有重要意义。
蛋白质结构可以分为四个层次:一级结构、二级结构、三级结构和四级结构。
一级结构是指蛋白质的氨基酸序列,即由哪些氨基酸组成;二级结构是指蛋白质中氨基酸之间的空间关系,包括α-螺旋、β-折叠等;三级结构是指蛋白质整体的空间构型,由氨基酸之间的相互作用决定;四级结构是指由多个蛋白质组成的聚合体,例如蛋白质复合物。
了解蛋白质的结构有助于我们理解蛋白质的功能和机制。
蛋白质结构预测是指通过计算模型和算法,预测未知蛋白质的结构。
由于实验方法尚未能够确定所有蛋白质的结构,因此蛋白质结构预测具有重要的研究意义。
在蛋白质结构预测中,可以采用多种方法,如基于机器学习的方法、蒙特卡罗模拟等。
其中,基于机器学习的方法是目前较为常用的方法之一。
通过将已知蛋白质的结构信息输入机器学习算法中,对未知蛋白质进行结构预测。
这种方法能够通过学习已有的蛋白质结构信息,从而预测未知蛋白质的结构。
蛋白质结构预测对于生物学研究和药物设计有着重要的应用价值。
蛋白质结构分析是在蛋白质的结构已知的情况下,对其结构进行深入研究和分析。
蛋白质结构分析可以从多个角度进行,如结构功能关系、动力学研究等。
其中,结构功能关系是蛋白质结构分析中的重要方面。
通过研究蛋白质的结构信息,可以理解蛋白质的功能和作用机制。
这对于生物学的研究和药物设计具有重要意义。
此外,蛋白质的动力学研究也是蛋白质结构分析中的重要内容之一。
蛋白质在生物体内不断发生构象变化,了解蛋白质的动力学行为对于理解其功能和机制具有重要意义。
蛋白质结构预测与分析在生物信息学中扮演着重要的角色。
通过蛋白质结构预测和分析,我们可以了解蛋白质的结构和功能,为生物学研究和药物设计提供重要的启示。
BIOINF_蛋白质序列分析
蛋白质序列分析电子科技大学 生命科学与技术学院 生物信息学中心周鹏博士/副教授理化性质: 分子量、等电点、氨基酸组成等结构分析:一级结构、二级结构、三级结构功能预测:motif、domain、信号肽、跨膜区、亚细胞定位、GO等一、蛋白序列的获得1. 基因序列翻译推导得到2. 氨基酸测序(多肽)得到3. 双向电泳、质谱分析得到4. 数据库得到SRS(Sequence Retrieval System )序列检索系统胶质纤维酸性蛋白(glial fibrillary acidic protein, GFAP)二、蛋白质理化性质分析三、蛋白质结构分析(一)、二级结构预测二级结构。
– α螺旋,是蛋白质中最常见最典型含量最丰富的二级结构元件.在α螺旋中,每轮卷曲的螺旋包含3.6氨基酸残基,残基侧链伸向外侧,同一肽链上的每个残基的酰胺氢和位于它后面的第4个残基上的羰基氧彼此之间形成氢键。
这种氢键大致与螺旋轴平行。
在水环境中,肽键上的酰胺氢和羰基氧既能形成内部(α-螺旋内)的氢键,也能与水分子形成氢键。
– 不同的氨基酸对α螺旋形成的影响是不同的。
– β折叠是通过肽链间或肽段间的氢键维系。
可以把它们想象为由折叠的条状纸片侧向并排而成,每条纸片可看成是一条肽链, 称为β折叠股或β股(β-strand),肽主链沿纸条形成锯齿状。
需要注意的是在折叠片上的侧链都垂直于折叠片的平面,并交替的从平面上下二侧伸出。
-无规则卷曲(randon coil)无规则卷曲或称卷曲(coil),泛指那些不能被归入明确的二级结构如折叠片或螺旋的多肽区段。
实际上这些区段大多数既不是卷曲,也不是完全无规的,虽然也存在少数柔性的无序片段。
它们也像其他二级结构那样是明确而稳定的结构。
它们受侧链相互作用的影响很大,经常构成酶活性部位和其他蛋白质特异的功能部位如许多钙结合蛋白中结合钙离子的EF 手结构(E-F hand structure)的中央环二级结构预测面临的困难二级结构在不同的溶剂环境中构象可能会不同同一肽段在不同的蛋白质中的结构也不一样预测序列模体和结构域都是通过对相关蛋白质的多序列比对分析而获得的– 线性模体(Linear motif),较短的特定序列模式。
蛋白质序列分析及其应用

蛋白质序列分析及其应用蛋白质序列分析是生物信息学领域的一个重要研究方向,它通过计算和比较蛋白质的氨基酸序列,揭示蛋白质的结构、功能和进化的信息。
蛋白质序列分析的应用广泛,包括预测蛋白质结构、功能注释、蛋白质家族分类、药物设计等。
本文将简要介绍蛋白质序列分析的方法和应用。
一、蛋白质序列分析的方法1.氨基酸组成分析:通过计算蛋白质序列中各种氨基酸的相对数量,可以了解蛋白质的氨基酸组成,比较不同蛋白质之间的差异和相似性。
2.序列比对分析:序列比对是蛋白质序列分析的基础工具,可以找到序列之间的相似区域,并推测彼此之间的进化关系。
常用的序列比对方法有全局比对、局部比对和多序列比对等。
3.蛋白质结构预测:蛋白质结构预测是蛋白质序列分析的核心任务之一、常见的方法包括二级结构预测、三级结构预测和蛋白质折叠模拟等。
4.功能注释:根据蛋白质序列的特征和结构,可以预测蛋白质的功能。
常用的方法包括保守区域分析、功能域识别和模式等。
5.蛋白质家族分类:通过比较蛋白质序列的相似性,可以将蛋白质分为不同的家族或超家族,用于进一步研究蛋白质的结构和功能。
二、蛋白质序列分析的应用1.药物设计:蛋白质序列分析可以为药物设计提供重要的信息。
通过分析蛋白质序列的结构和功能,可以预测药物与目标蛋白质之间的相互作用,优化药物的设计。
2.疾病预测与诊断:蛋白质序列分析可以帮助预测蛋白质的功能异常和突变,从而预测患者的疾病风险和诊断结果。
3.进化研究:通过比较不同物种的蛋白质序列,可以推测它们之间的进化关系。
这有助于了解物种的进化历史和基因家族的起源。
4.蛋白质工程:通过分析蛋白质序列和结构,可以对蛋白质进行工程改造,使其具有更好的特性和功能,用于生物工艺和生物医药等领域。
5.新蛋白质发现:通过对未知蛋白质序列的分析,可以发现新的蛋白质,并探索其结构和功能,为新药物和生物材料的开发提供新思路。
三、现阶段的挑战和发展方向尽管蛋白质序列分析已经取得了很大的进展,但仍面临一些挑战。
生物信息学中的蛋白质分析技术

生物信息学中的蛋白质分析技术蛋白质是生物体中不可或缺的重要分子,其功能包括酶催化、信号传递、结构支持等多种生命活动。
蛋白质分析是生物信息学研究中的重要领域之一,目的是从生物样品中获取有关蛋白质的信息。
这项技术不仅可以揭示蛋白质的结构和功能,还可以为医学诊断和药物研发提供重要的参考。
一、蛋白质分析的基本流程蛋白质分析的基本流程包括蛋白质提取、分离纯化、分析鉴定等几个步骤。
蛋白质提取是将目标蛋白从生物样品中提取出来,一般采用机械破碎、化学分解、超声波等方法。
分离纯化是将目标蛋白与其他蛋白分离开来,可以采用电泳、层析、过滤等方法。
分析鉴定则是对分离得到的蛋白进行化学、物理和生物学的分析,如质谱分析、核酸测序、免疫学检测等方法。
二、质谱分析技术的应用质谱分析是一种可以同时检测多种蛋白质组成和结构的方法,其技术基础是将蛋白质分离并进行离子化后进行质量分析。
这种方法被广泛地应用于蛋白质组学和蛋白质互作等领域。
在蛋白质组学中,将样品中的所有蛋白质分离并进行质谱分析,可以获得大量的信息,如蛋白质的数量、种类、分布和修饰状态等。
质谱分析技术的应用还包括蛋白质互作的研究。
蛋白质互作通常是指两个或多个蛋白质之间的相互作用,这在生物活动中非常重要。
质谱分析可以用来鉴定已知的蛋白质互作或发现新的蛋白质互作,这对于深入理解生物活动机理具有重要意义。
三、结构生物学的应用结构生物学是研究蛋白质三维结构的一种技术,其目的是探究蛋白质结构与功能之间的关系。
现有的结构生物学技术主要包括X射线晶体学、核磁共振和电子显微镜。
通过这些技术,可以确定单个蛋白质的原子结构,也可以确定蛋白质的超分子结构,如蛋白质-DNA复合物和蛋白质-蛋白质复合物等。
在药物研发方面,结构生物学的应用也非常广泛。
通过了解蛋白质的结构,可以设计出针对特定靶标的药物,并对药物与靶标之间的相互作用进行优化和改良。
四、生物信息学的应用生物信息学是将计算机和数学等方法应用于生物学研究的一种学科。
生物信息学中的蛋白质序列分析

生物信息学中的蛋白质序列分析随着生物技术的不断发展,人们对于生物体内各种蛋白质的研究愈发深入。
而蛋白质序列分析则是生物信息学中重要的一环,可以用于蛋白质结构预测、功能分析、进化研究等方面。
在这篇文章中,我们将探讨蛋白质序列分析在生物信息学中的应用以及涉及到的技术和算法。
一、蛋白质序列的组成蛋白质由氨基酸组成,而蛋白质序列指的是氨基酸连接的线性序列。
氨基酸是构成蛋白质的基本单元,不同的氨基酸组合构成不同的蛋白质。
目前已知的氨基酸有20种,它们由不同的侧链和碳氮骨架组成,这种多样性导致了蛋白质具有丰富多样的结构和功能。
二、蛋白质序列分析的应用1、预测蛋白质结构蛋白质结构与其功能息息相关,因此对于蛋白质结构的预测一直是研究的热点问题。
蛋白质序列是进行蛋白质结构预测的重要依据之一。
一般来说,蛋白质结构预测可分为二级结构和三级结构预测。
二级结构指的是蛋白质中α-螺旋、β-折叠和无规则卷曲等局部的结构。
目前,常用的二级结构预测方法有Chou-Fasman算法、GOR算法等。
而三级结构预测指的是蛋白质整体的三维结构,其预测难度更大,目前还没有完全解决。
但是,针对蛋白质结构的许多研究都是基于蛋白质序列的分析和预测。
2、鉴定蛋白质功能蛋白质的功能与其序列和结构有关,因此通过分析蛋白质序列也可以预测蛋白质的功能。
一般来说,蛋白质的功能可以分为三类:催化、结构和调节。
催化作用指的是酶类蛋白质对化学反应的促进作用。
结构作用指的是蛋白质形成结构,对于细胞和组织的形态和机能具有重要作用。
调节作用指的是蛋白质对细胞、胚胎、发育和免疫系统等的调节作用。
对于蛋白质功能的鉴定,目前的方法主要有以下几种:1)基于序列的比对方法;2)结构基因学方法;3)基于基因组的方法。
三、蛋白质序列分析的技术和算法1、BLAST算法BLAST(Basic Local Alignment Search Tool)算法是常用的序列比对算法之一,它通过比对两条序列后,计算两个序列之间的相似性得分。
生物信息学-蛋白质性质和结构分析

(二) 分析蛋白质的二级结构 二级结构:主要是氢键维持的结构 -螺旋(-helix) -折叠(-sheet)
转角(turn) 环(loop)
无规则卷(random coil)
Chou-Fasman method
蛋白质的 pI、Mw、氨基酸组成等
2. 分析蛋白质的疏水性 打开/tools/ 在“Primary structure analysis”栏目选择 “ProtScale”分析软件 在ProtScale主页 (/protscale/) 粘贴序列、选择分析方法
(三) 分析蛋白质的三级结构 1. 根据已知蛋白质结构推测未知蛋白质结构
BLAST 检索 在蛋白质结构数据库(PDB) 中检索同源蛋白质的结构
2. 通过分子建模(molecular modeling)分析蛋白质结构
分析复杂 适用于专业人员
Phyre2 /phyre2/html/page.cgi?id=index
蛋白质性质和结构分析
ExPASy (Expert Protein Analysis System)
Nucleic Acids Research 2003, 31:3784-8
Swiss Institute of Bioinformatics (SIB) 的分析工具
蛋白质的亲水和疏水性分析结果,有文字 和图形两种显示方式
3. 分析蛋白质的保守结构域
在文本框“Scan a sequence against PROSITE patterns and profiles”粘贴序列
使用缺省参数( exclude patterns with a high probability of occurrence)
生物信息学在蛋白质研究中的应用

生物信息学在蛋白质研究中的应用
生物信息学在蛋白质研究中的应用非常广泛,涉及到蛋白质序列、结构、功能和相互作用等方面的研究。
以下是生物信息学在蛋白质研究中的一些常见应用:
1. 蛋白质序列分析:生物信息学可以用来分析蛋白质的氨基酸序列,包括寻找保守序列、构建进化树、预测蛋白质结构域、寻找信号肽和跨膜区域等。
这对于预测蛋白质的功能、结构和相互作用非常重要。
2. 蛋白质结构预测:基于生物信息学方法,可以预测蛋白质的三维结构,包括基于序列相似性的比对模拟、基于模板的结构比对和模型构建等方法。
这对于理解蛋白质的功能和相互作用机制非常重要。
3. 蛋白质功能注释:生物信息学可以用来预测蛋白质的功能,包括功能域的预测、功能家族的归类和功能通路的分析等。
这有助于理解蛋白质的生物学功能和参与的分子机制。
4. 蛋白质相互作用网络分析:利用生物信息学方法,可以构建蛋白质相互作用网络,并进行网络拓扑分析。
这有助于理解蛋白质相互作用的模式和机制,以及预测新的蛋白质相互作用。
5. 蛋白质结构与功能关联研究:利用生物信息学方法,可以分析蛋白质结构与功能之间的关联,通过预测结构域的位置和功能特征,进一步了解蛋白质的功能与结构之间的关系,例如酶活性位点的预测和功能蛋白质的筛选等。
总之,生物信息学在蛋白质研究中扮演着重要的角色,可以帮助揭示蛋白质的结构、功能和相互作用等方面的信息。
这些信息对于深入理解生物学过程以及药物研发和疾病研究具有重要意义。
2蛋白质序列特征分析~生物信息学全解

蛋白质的亲水性或疏水性
氨基酸的亲疏水性是构成蛋白质折叠的主要驱 动力,一般通过亲水性分布图(hydropathy profile)
反映蛋白质的折叠情况。蛋白质折叠时会形成疏水
内核和亲水表面,同时在潜在跨膜区出现高疏水值 区域,据此可以测定跨膜螺旋等二级结构和蛋白质
表面氨基酸分布。
利用PROTSCALE分析蛋白质的亲水性或疏水性
TMPRED在线网页
用TMPRED分析P51684序列所得到的可能的 7个跨膜螺旋区
用TMPRED分析P51684序列所得到的7个可 能的跨膜螺旋区的相关性列表
用TMPRED分析P51684序列所得到的7个可 能的跨膜螺旋区的建议的跨膜拓扑模型
用TMPRED分析P51684序列所得到的7个 可能的跨膜螺旋区的图形显示结果
工具,TMpred基于对TMbase数据库的统计分析来预测蛋白 质跨膜区和跨膜方向。TMbase来源于Swiss-Prot库,并包含了
每个序列的一些附加信息,如:跨膜结构区域的数量、跨
膜结构域的位置及其侧翼序列的情况。TMpred利用这些信息 并与若干加权矩阵结合来进行预测。 其网址为: /software/TMPRED_form.html
4、信号肽—SIGNAL PEPTIDE
信号肽是指新合成多肽链中用于指导蛋白质跨膜转移 的末端(通常为N末端)的氨基酸序列。信号肽中至少含
有一个带正电荷的氨基酸,中部有一个高度疏水区以通过
序列特征分析
Analysis of Sequence Characterristics
一、蛋白质结构
蛋白质的一级结构
蛋白质的一级结构决定二级结构 蛋白质的二级结构决定三级结构
蛋白质的二级结构Leabharlann H表示螺旋E表示折叠
生物信息学第七章蛋白质结构分析和预测

转角规则
➢ 四肽片段,若位置专一性转角形成几率 fi+1fi+2fi+3fi+4 >7.5*10-5,pt>1.0,并大于pα和pβ, 则预测为转角。
Chou-Fasman二级结构预测经验规则
重叠规则
➢ 螺旋和折叠的重叠区域,按pα和pβ的相对大小 进行预测,若pα>pβ,则预测为螺旋,反之为 折叠。
➢ 最后,将α螺旋两端各去掉3个残基,剩余部分 多于6个残基,且pα>1.3 ,则为α螺旋。
Chou-Fasman二级结构预测经验规则
β折叠规则 ➢ 如果相邻5个残基中若有3个倾向于形成β
折叠,则认为是β折叠核。 ➢ β折叠核向两端延伸直至4个残基的平均
折 叠 倾 向 因 子 pβ<1.0 。 若 延 伸 后 片 段 的 pβ>1.05,则预测为β折叠。
三、蛋白质二级结构预测
二级结构:主要是氢键维持的结构 -螺旋(-helix) -折叠(-sheet) 弯(turn) 襻(loop)
二级结构的预测是蛋白结 构预测的第一步。
蛋白质二级结构预测的方法
基于统计学的预测方法 1、 Chou-Fasman方法
➢ 直接以氨基酸序列来预测二级结构 统计各种氨基酸在不同二级结构中的各种
蛋白质二级结构预测的方法
基于实验数据的预测方法 3、混和方法
综合多种二级结构预测方法,通过调整不 同方法在预测时的权值做出综合判断以改善预 测准确率。
蛋白质的结构层次:
一级结构(氨基酸序列) 二级结构 三级结构 四级结构
采用ProtParam软件[1] (/tools/protpa ram.html)分析蛋白质的分子量、理论 等电点、氨基酸组成、带正负电荷的氨 基酸残基数目、消光系数、吸光系数、 疏水系数和半衰期等基本理化性质。
蛋白质的序列分析及结构预测

蛋白质的序列分析及结构预测蛋白质序列分析和结构预测是生物信息学中的一个重要研究领域。
蛋白质是生物体内具有功能的大分子,其结构决定了其功能。
了解蛋白质序列和结构可以帮助我们研究蛋白质的功能和生物过程。
蛋白质序列分析的第一步是进行蛋白质的序列比对。
蛋白质序列比对可以帮助我们找到相似的序列,从而进行进一步的研究。
常用的序列比对方法包括序列比对算法和数据库算法。
序列比对可以通过计算序列之间的相似性来研究蛋白质的进化关系和功能。
在序列比对的基础上,可以进行蛋白质的结构预测。
蛋白质结构预测是确定蛋白质在空间中的三维结构。
蛋白质的结构决定了其功能,所以了解蛋白质的结构对于研究蛋白质的功能和相互作用很重要。
蛋白质结构预测包括以下几种方法。
第一种方法是模板比对。
模板比对是基于已知蛋白质结构库的比对方法。
通过比对蛋白质序列和已知结构的序列,可以预测出蛋白质的结构。
这种方法利用已知结构和序列的对应关系,可以预测出蛋白质的结构。
第二种方法是基于物理性质的结构预测。
这种方法基于蛋白质的物理性质,通过计算蛋白质分子的力学能量和动力学来预测蛋白质的结构。
这种方法较为复杂,需要大量的计算资源。
第三种方法是基于机器学习的结构预测。
机器学习是一种利用算法和统计学方法来训练和预测的方法。
这种方法可以利用蛋白质的序列和结构的关系来预测蛋白质的结构。
利用机器学习方法,可以通过大量的样本和特征来训练模型,从而预测蛋白质的结构。
蛋白质序列分析和结构预测是生物信息学中的重要研究领域。
通过对蛋白质序列和结构的研究,可以揭示蛋白质的功能和生物过程。
这对于研究蛋白质的功能和疾病的机制有重要的意义,并为药物设计和治疗提供了理论基础。
生物信息学第七章蛋白质结构分析和预测

提交氨基酸序列
/~phyre/
五、蛋白质跨膜区预测
膜蛋白结构
脂双层
1
2
3
6 NH3
P
P
胞质
COOH
4
5
7
五、蛋白质跨膜区预测
跨膜区特点
➢ 膜蛋白跨膜区氨基酸具有极强疏水性 ➢ 跨膜区的二级结构一般为α螺旋和β筒状结构
20-30个连续高度疏水氨基酸可以α螺旋形式穿越 脂双层;β筒跨膜区的氨基酸只有20个左右。
构象分布概率、氨基酸在蛋白质中的相对出现 概率以及残基出现在结构中的频率,最后得到 构想参数,根据此参数得出氨基酸形成二级结 构的倾向性,从而预测二级结构。
Chou-Fasman二级结构预测经验规则
α螺旋规则
➢ 相邻的6个残基中如果有至少4个残基倾向于形 成α螺旋,则认为是螺旋核。
➢ 然后从螺旋核向两端延伸,直至四肽α螺旋倾 向性因子的平均值pα<1.0为止。此外,不容许 脯氨酸在螺旋内部出现,但可出现在C末端以 及N端的前三位。
蛋白质的结构层次:
一级结构(氨基酸序列) 二级结构 三级结构 四级结构
采用ProtParam软件[1] (/tools/protpa ram.html)分析蛋白质的分子量、理论 等电点、氨基酸组成、带正负电荷的氨 基酸残基数目、消光系数、吸光系数、 疏水系数和半衰期等基本理化性质。
信号肽预测
分泌蛋白新生肽链N端的一段20~30氨 基酸残基组成的肽段。将分泌蛋白引导 进入内质网,同时这个肽段被切除。现 这一概念已扩大到决定新生肽链在细胞 中的定位或决定某些氨基酸残基修饰的 一些肽段。
信号肽预测
预测给定的氨基酸序列中是否存在潜在 的信号肽剪切位点及其所在
2蛋白质序列特征分析~生物信息学总结

对DNA序列和蛋白质序列进行序列特征分析, 能够使我们从分子层次上了解基因的结构特点,
了解与基因表达调控相关的信息,了解 DNA序列与
蛋白质序列之间的编码,了解蛋白质序列与蛋白
质空间结构之间的关系和规律,为进一步研究了
解蛋白质功能与蛋白质结构之间的关系提供理论
依据。
二、蛋白质序列特征分析
基本假设:蛋白质的空间结构由蛋白质序列所决定。 即我们可以根据蛋白质序列预测蛋白质结构。
PROTPARAM在线页面
用PROTPARAM分析G00016序列理化性质的结果
2、蛋白质的亲水性或疏水性
蛋白质的基本组成单元是氨基酸。
氨基酸通常被分为三类:
1. 疏水氨基酸(hydrophobic amino acid),其侧链大部分 或者全部由碳原子和氢原子组成,因此这类氨基酸不太可 能与水分子形成氢键; 2. 极性氨基酸(polar amino acid),其测链通常由氧原子或 氮原子组成,它们比较容易与水分子形成氢键,因此也称 为亲水氨基酸; 3. 带电氨基酸(charged amino acids),这类氨基酸在生物 pH环境中带有正电或负电。
SignalP是丹麦技术大学的生物序列分析中心开发的信 号肽及其剪切位点检测的在线工具,该软件基于神经网络 方法,用已知信号序列的革兰氏阴性原核生物、革兰氏阳 性原核生物及真核生物的序列分别作为训练集。SignalP预 测的是分泌型信号肽,而不是那些参与细胞内信号传递的
蛋白。
其网址为:
http://genome.cbs.dtu.dk/services/SignalP/
SIGNALP在线网页
用SIGNALP(神经网络方法)分析P05019序 列前导肽的结果
用SIGNALP(隐马尔可夫方法)分析 P05019序列前导肽的结果
生物信息学在蛋白质分析中的应用

生物信息学在蛋白质分析中的应用生物信息学是一门涉及计算机科学、生物学和数学等多个学科的交叉学科,其主要研究生物系统中的大量生物分子如DNA、RNA和蛋白质等的序列、结构和功能等信息。
其中,在蛋白质分析中的应用越来越受到重视,因为蛋白质是生命活动的重要基础,对许多人类疾病的治疗提供着关键性的帮助。
在这篇文章中,我们将从蛋白质的结构、组成和功能等方面来探讨生物信息学在蛋白质分析中的应用。
蛋白质的结构蛋白质是一种复杂而多样化的分子,它们的结构包含了多个层次,从原始的氨基酸序列到最终的三维空间结构。
因此,理解蛋白质的结构是研究其表现和功能的重要基础。
在传统的实验方法中,蛋白质的结构可以通过X射线晶体学或NMR等技术来决定,但它们具有很高的成本和局限性。
现在,生物信息学提供了许多先进的计算方法来推测和预测蛋白质的结构,且这些预测方法通常会快捷、准确、成本低廉。
其中最常用的蛋白质结构预测方法是基于比对的方法。
这种方法基于已知的蛋白质结构,结合不同的算法和技术来预测新的蛋白质结构。
例如,如果已知的一部分蛋白质与某种新蛋白质有很高的相似性,那么这个新蛋白质的结构也可以通过计算的方法被预测出来。
此外,还可以利用分子动力学和蒙特卡罗模拟等方法来推测特定蛋白质在各种温度和压力下的结构。
这些方法为研究蛋白质的结构和功能提供了不同的思路和手段。
蛋白质的组成蛋白质分子由一系列氨基酸通过肽键链接而成,共有20种常用的氨基酸。
每种氨基酸具有独特的物化性质,如电荷、极性、亲水性和亲疏水性等,这些物化性质决定了蛋白质的功能和特征。
因此,了解蛋白质的氨基酸组成对于研究其功能和相互作用非常重要。
利用生物信息学的方法可以快速地分析和比较蛋白质的氨基酸序列。
例如,可以对不同物种中的同一蛋白质序列进行比较,从而了解它们之间的差异和相似性。
此外,还可以利用多序列比对的方法来比较不同蛋白质序列之间的相似性,从而进一步了解它们之间的生物学功能和结构特点。
蛋白质生物信息学

蛋白质生物信息学
蛋白质生物信息学是指应用计算机科学和数学方法,研究蛋白质的结构、功能和互作关系,并将这些信息应用于生物学研究中的一门学科。
蛋白质是生命体中最重要的分子之一,具有广泛的生物功能,在疾病诊断、药物研发、食品安全等领域都有着重要的应用价值。
蛋白质生物信息学主要包括蛋白质序列分析、蛋白质结构预测、蛋白质功能预测、蛋白质相互作用网络分析等内容。
其中,蛋白质序列分析是研究蛋白质基本构成和序列特征的方法;蛋白质结构预测则是通过计算方法来预测蛋白质的三维结构;蛋白质功能预测则是根据蛋白质的序列、结构和互作关系等信息预测其功能。
此外,蛋白质相互作用网络分析则是研究蛋白质之间相互作用的方法,可以揭示蛋白质在细胞内的相互作用关系和生物过程的调控机制。
蛋白质生物信息学是一门交叉学科,需要具备生物学、计算机科学和数学等多方面的知识。
随着科技的发展,蛋白质生物信息学在生命科学领域中的应用越来越广泛,为深入了解生命体系、开发新药物和治疗疾病提供了新的思路和方法。
- 1 -。
蛋白质谱生信分析

百泰派克生物科技
蛋白质谱生信分析
蛋白质谱生信分析就是对质谱数据进行生物信息学分析。
所谓质谱数据就是质谱仪通过检测肽段母离子的质荷比(m/z)而得到的图谱,如肽质量指纹图谱(PMF)、肽序列图谱(PST),一般利用各种软件、数据库将质谱获得的肽离子质量与理论肽质量进行比较和评价,从而实现该肽段乃至整个蛋白的鉴定和序列分析。
常用的检索工具主要包括PeptIdenet、MS-Fit、ProFound、PeptideSearch、MS-Taq、MS-Seq、PepFrag、Mascot等。
百泰派克生物科技采用Thermo Fisher的Orbitrap Fusion Lumos质谱平台结合nanoLC-MS/MS纳升色谱,提供蛋白质质谱分析技术服务,只需要将您的实验目的告诉我们并寄送样品,百泰派克提供包括蛋白提取、蛋白酶切、肽段富集、肽段分离、质谱分析、质谱原始数据分析、生物信息学分析一站式服务,欢迎免费咨询。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
二、功能注释
Uniprot中一个蛋白质的例子
/uniprot/P10962
三、序列特征
Uniprot中一个蛋白质的例子
/uniprot/P10962
四、蛋白质结构域组成和蛋白质家族
Uniprot中一个蛋白质的例子
有关注释内容的文献、蛋白质名称词典和其他有助于文献
挖掘的人文语言处理技术开发的信息、数据库校正、蛋白 质名称标记和功能注释标准体系(ontology)。使用
iProLINK可以获得描述蛋白质记录的文本文献资源,在
UniProtKB记录(生物词典)中加入蛋白质或基因命名的 图谱,获得用于开发文本挖掘算法的注释数据集、挖掘蛋
分类分布、分级和功能域结构,以及家族
成员,包括功能、结构、传导通路、功能
注释标准体系(ontology)和家族分类。
利用这些信息可以获得蛋白质的准确功能 或预测的功能和该蛋白质所属家族成员共 有的其他特征。
• 4. iProLINK-蛋白质文献、信息和知识整合数据库 iProLINK(/iprolink/)提供
通路、相互作用、家族分类、基因和基因组、功能
注释标准体系(ontology)、文献和分类学信息。
使用iProClass还可以检索ID图谱、蛋白质词典和相
关序列。
• 3. PIRSF-蛋白质家族分类系统 PIRSF
(/pirsf/)分类
系统概要论述家族的特征,如家族名称、
白质磷酸化(RLIMS-P)文献和获得蛋白质功能注释标准
体系(ontology)(PRO)信息。
二、MIPS数据库
• 慕尼黑蛋白质序列信息中心(MIPS) (http://www.helmholtz-muenchen.de/en/mips),它的 重点工作是基因组生物信息学,特别注重基因组信息系统分 析,包括应用生物信息学方法注释基因组、表达分析和蛋白 质组学方面研究。MIPS支持和维护一系列基因组数据库以及
系统,可以提供细菌、真菌和植物基因组比较分析服务。在
该站点提供基因组分析工具、数据库检索系统、表达分析、 蛋白相互作用等网络服务。
其他重要的蛋白质序列数据库
• PRINTS • Pfam
PRINTS
• PRINTS (/dbbrowser/PRI NTS/index.php)是蛋白基序指纹图综合数据库,每个指 纹图都是使用数据扫描程序ADSP或VISTAS序列分析软 件包反复优化后定义的。数据库中有两种类型指纹图,根 据指纹图的复杂性分为简单和复合指纹图:简单指纹图基 本上是单一的基序,而复合指纹图包含多个基序。
蛋白质二维凝胶电泳数据库
• WORLD-2DPAGE • Phoretix links
• • • • • • •
信号传导及蛋白质-蛋白质相互作 用相关数据库
DIP INTERACT ProNet KEGG CANSITE SPAD CSNDB等
• DNA和蛋白质相互作用数据库:DPInteract • 蛋白质翻译后修饰相关数据库: O-GlycBase、PhosphoBase、RES等
蛋白质序列数据库
• 随着分子生物学的发展,人们获得了越来越多关 于蛋白质序列、结构和功能的信息。世界各国的 生物学家和计算机科学家合作利用这些信息构建 了蛋白质序列数据库、蛋白质三维结构数据库、 蛋白质组数据库(二维凝胶电泳数据库)、信号 传导及蛋白质-蛋白质相互作用相关数据库、DNA 和蛋白质相互作用数据库等蛋白质相关数据库。
PIR信息库资源
PIR主要数据库:
• • • • 1. UniProt-通用蛋白质资源库 2. iProClass-蛋白质知识整合数据库 3. PIRSF-蛋白质家族分类系统 4. iProLINK-蛋白质文献、信息和知识整合 数据库
• 1.UniProt-通用蛋白质资源库 UniProt (/)是存储和链接其他 蛋白质数据库的资源库,并且是蛋白质序列和具 有综合功能注释目录的中心资源库。使用 UniprotKB可以检索准确、可靠的蛋白综合信息。 使用UniRef可以减少冗余,加速序列相似性搜索。 使用UniParc可以检索存档序列和它们来源的数 据库。
/uniprot/P10962
五、其他特征:结构、参与的蛋白质互作
常用的蛋白质序列数据库
• PIR • MIPS • Swiss-Prot
蛋白质功能、结构域和蛋白质家族 有关的数据库
• PROSITE • InterPro • Pfam
• ProDom
• SMART 等
蛋白质三维结构相关数据库
• • • • • • • • • • PDB BioMagResBank SWISS-MODEL RepositoriBase TOPS SWISS-3DIMAGE BioImage等
• 2. iProClass-蛋白质知识整合数据库 iProClass
(/iproclass/)提供来
自90多个生物学数据库的大量整合数据,包括蛋白 ID图谱服务、UniProtKB编注蛋白质摘要描述和筛 选UnParc数据库的蛋白质序列。使用iProClass可 以检索最新的蛋白质综合信息,包括:功能、转导
PIR数据库
• 蛋白质信息库(PIR) (/pirwww/)是一个支 持基因组学、蛋白质组学和系统生物学检索和科 学研究的综合公共生物信息学资源。
• PIR是由美国国家生物医学基金会(NBRF)于 1984年建立,帮助研究者确认和解释蛋白序列信 息的数据库。 • PIR免费为科学界提供包括蛋白序列数据库 (PSD)在内的蛋白数据库和分析工具。
Pfam
• 蛋白质一般是由一个或多个功能区域组成,这些 功能区域通常称作域(domain)。在不同的蛋白 质中不同的域以不同的组合出现,导致在自然界 发现多种多样组成成分的蛋白质。识别出现在蛋
白质中的域可以了解蛋白质的功能。
• Pfam数据库(/)是一个
大的蛋白质域家族集合,每个家族是用多序列比
对和隐马模型(HMMs)分析结果的代表。
Uniprot中一个蛋白质的例子
/uniprot/P10962
一、基本信息
Uniprot中一个蛋白质的例子
/uniprot/P10962
