地高辛标记与检测DNA试剂盒说明书
地高辛 (Digoxin)使用说明书

地高辛 (Digoxin)使用说明书地高辛 (Digoxin) 使用说明书地高辛是一种常用的心脏疾病治疗药物,主要用于治疗心力衰竭和心律失常。
本说明书将详细介绍地高辛的用法、剂量、注意事项和可能的副作用,帮助患者正确使用地高辛,从而达到最佳疗效。
一、药物名称和成分药物名称:地高辛(Digoxin)成分:每片含地高辛0.25毫克。
二、适应症地高辛适用于下列疾病的治疗:1. 心力衰竭:用于改善心脏收缩力,增加心脏排血量。
2. 心房颤动:用于控制心脏节律,减少心房颤动的发作并减轻症状。
三、用法和剂量1. 使用方法:口服。
最好与饭后服用,以减少不适感。
2. 常用剂量:成人一般剂量为每日0.125-0.25毫克,根据患者具体情况,可在医生指导下微调剂量。
3. 用量调整:用地高辛治疗时,应根据患者的病情和耐受性进行用量调整。
请遵循医生的指导,并定期检查药物浓度和心脏功能。
四、注意事项1. 专科医生指导:地高辛使用需要在专科医生的监护下进行,不得擅自调整剂量或停药。
2. 定期检查:使用地高辛期间,应定期检查心脏功能、药物浓度等指标,以确保疗效和安全性。
3. 过敏反应:如果出现过敏反应,如皮疹、荨麻疹、呼吸困难等症状,请立即就医。
4. 注意相互作用:地高辛有一些药物相互作用可能导致心脏毒性增加,如抗心律失常药物、某些抗生素等,请告知医生所用的所有药物。
5. 年老体弱者:对于年老体弱、肝肾功能减退者,应特别谨慎使用,避免出现中毒症状。
五、可能的副作用地高辛的使用可能会出现以下副作用:1. 胃肠道反应:如恶心、呕吐、腹泻等,特别是在剂量过高的情况下。
2. 心脏毒性:如心律失常、心搏骤停等,请密切关注心脏状况,若出现异常请立即就医。
3. 颜面潮红:部分患者使用地高辛后可能出现颜面潮红的不适感。
4. 视觉障碍:可能出现视觉模糊、视物有影等,若出现此类问题,请及时就医。
六、妊娠和哺乳期使用妊娠和哺乳期妇女慎用地高辛,应在医生的指导下使用。
地高辛标记及检测试剂盒I(POD)说明书

DIG Random Labeling and Detection KitⅠ(POD)(for color detection withDAB)DNA探针标记和检测试剂盒Ⅰ产品编号:MK1008保存期:一年-20℃冰冻保存。
用途:用于DNA探针的随机引物标记及各种膜上杂交和原位杂交。
工作量:可用于标记0.1—3μg的探针6次及1000张切片的原位杂交或12次10×10cm膜上杂交检测。
原理所谓DNA探针,实质上是一段已知的基因片段,应用这一基因片段即可与待测样品杂交。
如果靶基因和探针的核苷酸序列相同,就可按碱基配对原则进行核酸分子杂交,从而达到检查样品基因的目的。
在随机引物法标记反应液中,有随机合成的六聚体核昔酸(hexanucleotide)作为引物,dATP、dCTP、dGTP、dTTP和D1G-11-dUTP作为合成底物,以单链DNA作为模板,在Klenow 酶的作用下,合成掺入地高辛的DNA链。
以地高辛标记的探针与靶基因DNA链杂交后,再通过免疫反应来进行检测。
一般通过酶标抗地高辛抗体来检测,就可以肯定杂交反应的存在。
免疫检测采用过氧化物酶系统,DAB显色。
敏感性很高。
可用于膜上杂交和原位杂交。
试剂盒内容:1. DIG Random Labeling Mix(高效),25μl2.Biotin—MouseAnti—Digoxin200μl3. SABC—POD200μl4. DAB Stock Solution5ml5. Blocking Reagent,5g特点:(1)标记属高效标记反应。
以1μg DNA探针为例,1小时反应即可产生0.8μg标记探针,20小时可产生2μg左右的标记探针。
(2)试剂将标记反应所需的引物、核苷酸、DIG-11dUTP和Klenow酶等混合在一起。
一次标记只需吸取一次标记液,使用至为简便。
(3)标记反应适于下述对象:a.DNA从10ng—3μg。
b.不等的DNA长度100bp—10kb。
地高辛标记探针的Southern杂交
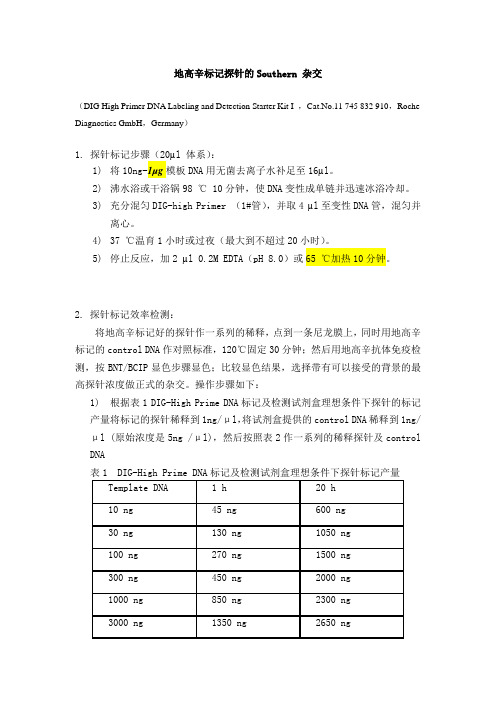
地高辛标记探针的Southern 杂交(DIG High Primer DNA Labeling and Detection Starter Kit I ,Cat.No.11 745 832 910,Roche Diagnostics GmbH,Germany)1. 探针标记步骤(20µl 体系):1)将10ng-1µg模板DNA用无菌去离子水补足至16µl。
2)沸水浴或干浴锅98 ℃ 10分钟,使DNA变性成单链并迅速冰浴冷却。
3)充分混匀DIG-high Primer (1#管),并取4 µl至变性DNA管,混匀并离心。
4)37 ℃温育1小时或过夜(最大到不超过20小时)。
5)停止反应,加2 µl 0.2M EDTA(pH 8.0)或65 ℃加热10分钟。
2. 探针标记效率检测:将地高辛标记好的探针作一系列的稀释,点到一条尼龙膜上,同时用地高辛标记的control DNA作对照标准,120℃固定30分钟;然后用地高辛抗体免疫检测,按BNT/BCIP显色步骤显色;比较显色结果,选择带有可以接受的背景的最高探针浓度做正式的杂交。
操作步骤如下:1)根据表1 DIG-High Prime DNA标记及检测试剂盒理想条件下探针的标记产量将标记的探针稀释到1ng/μl,将试剂盒提供的control DNA稀释到1ng/μl (原始浓度是5ng /μl),然后按照表2作一系列的稀释探针及control DNA表1 DIG-High Prime DNA标记及检测试剂盒理想条件下探针标记产量表2)将上述稀释的2-9 号管的control DNA 及探针DNA各取1μl点膜3)120℃固定30分钟或紫外交链3-5分钟4)将膜放入装有20ml Maleic acid buffer 的塑料器皿中,室温振荡2分钟5)将膜放入10ml Blocking solution中室温温育30分钟6)将膜放入10ml Antibody solution 中室温温育30分钟7)用10ml Washing buffer 洗2次,每次15分钟8)在10ml Detection buffer 平衡2-5分钟9)将膜放入2ml 新配制的Color substrate solution 中暗室条件下显色。
深圳依诺金生物地高辛杂交检测试剂盒 I 产品说明书

深圳依诺金生物科技有限公司 - 1 - 产品说明书地高辛杂交检测试剂盒 I目录号: DIGD-110装量: 10次显色检测反应保存期: 12个月.存储条件:NBT/BCIP显色底物–20℃抗DIG-AP酶结合物4℃其它组分室温试剂盒组成:阻断液母液(10x) 50 ml抗DIG-AP 酶结合物20 µl NBT/BCIP显色底物 2 ml检测缓冲液(5×) 50 ml顺丁烯二酸缓冲液母液(2x) 500 ml Tween-20 3 ml 10% SDS 20 ml 20×SSC 100 ml使用前先用DEPC/无RNase水将5×检测缓冲液及2×顺丁烯二酸缓冲液母液稀释成1×工作液.一.预杂交:1. 将膜从包装袋中取出,用干净的剪刀将膜溴酚蓝以下的部分剪去。
放入杂交袋中,在封口机上将杂交袋三面封口。
2. 加入65℃预热的Hyb高效杂交液5mL,排尽气泡,封口。
放65℃水浴振荡杂交1~2小时。
3. 如果使用杂交管杂交,则根据杂交管体积,加入适量的杂交液(5-10ml)。
如果膜的大小适中,也可以用无菌的50ml离心管(BD公司)进行,5ml杂交液已足够。
将杂交仪设定为65℃,8-15转/分钟预杂交1~2小时。
二.杂交:1. 探针的处理:将标记好的探针放100℃煮10分钟,立即放冰浴冷却10分钟。
2. 将杂交袋从水浴中取出,剪去一角并尽可能的排尽袋中的预杂交液,取5mLHyb高效杂交液,加入变性的探针(1-3µl/膜,5-20ng/mL Hyb杂交液),混匀。
加入到杂交袋中,小心排尽气泡,封口,65℃水浴振荡杂交过夜。
3. 如果使用杂交管杂交,则倒出预杂交液,另取适量的Hyb高效杂交液(5-10ml),加入变性过的探针(1-3µl/膜,或5-20ng/ml Hyb高效杂交液),混匀。
加入到杂交管中,杂交仪设定为65℃,8-15转/分钟,杂交过夜。
地高辛染色液说明书 一类

地高辛染色液说明书一类地高辛染色液是一种常用的染色试剂,常用于组织学研究中的免疫组化染色实验。
下面我将从多个角度全面介绍地高辛染色液的说明书内容。
1. 产品简介:地高辛染色液是一种含有地高辛标记的二抗试剂,用于检测目标蛋白在组织切片中的表达情况。
它可以与免疫组织切片中的靶蛋白发生特异性结合,并通过染色反应显示出目标蛋白的位置和分布。
2. 试剂成分:地高辛染色液主要由以下成分组成:地高辛标记的二抗,这是地高辛染色液的核心成分,通过特异性结合目标蛋白进行染色。
缓冲液,提供适宜的酸碱度和离子强度,维持染色反应的稳定性。
阻断剂,用于阻断非特异性结合,减少背景信号。
保存剂,保护试剂的稳定性和活性。
3. 使用方法:地高辛染色液的使用方法如下:准备组织切片,将待染色的组织切片固定在载玻片上,并进行适当的抗原修复和膜通透处理。
涂抹试剂,将地高辛染色液均匀涂抹在组织切片上,确保完全覆盖。
孵育反应,根据实验要求,在适当的温度和时间条件下孵育反应,使地高辛与目标蛋白发生特异性结合。
洗涤步骤,使用缓冲液或洗涤液进行适当的洗涤步骤,去除非特异性结合的试剂。
显色和显微观察,根据实验需要,使用适当的显色试剂进行染色反应,并在显微镜下观察和记录结果。
4. 注意事项:在使用地高辛染色液时,需要注意以下事项:试剂存储,请按照说明书中的建议存储试剂,避免温度过高或过低,避免阳光直射。
使用前检查,使用前请检查试剂的外观和标记,确保没有异常。
防止污染,使用过程中,请使用干净的工具和试剂盒,避免交叉污染。
实验条件优化,根据不同的样本和实验要求,可能需要优化反应条件,包括孵育温度、时间和洗涤步骤等。
总结:地高辛染色液是一种常用的免疫组织化学染色试剂,通过特异性结合目标蛋白进行染色反应。
在使用时,需要按照说明书中的方法和注意事项进行操作,以获得准确可靠的实验结果。
希望以上内容能够对你有所帮助。
罗氏地高辛标记说明书 英文版

For life science research only. Not for use in diagnostic procedures.FOR IN VITRO USE ONLY.DIG High Prime DNA Labeling and Detection Starter Kit IFor color detection with NBT/BCIP.Random primed DNA labeling with digoxigenin-dUTP,alkali-labile and detection of hybrids by enzyme immunoassayCat. No. 11 745 832 910Store at Ϫ15 to Ϫ25° C Kit for 12 labeling reactions of 10 ng-3 g DNAand detection of 24 blots of 100 cm²Instruction ManualVersion November 20091.Preface1.1Table of Contents1.Preface (2)1.1Table of Contents (2)1.2Kit contents (3)2.Introduction (5)2.1Product overview (5)3.Procedures and required materials (8)3.1Before you begin (8)3.2DIG-DNA Labeling (9)3.3Determination of labeling efficiency (12)3.4DNA transfer and fixation (15)3.5Hybridization (17)3.6Immunological detection (19)3.7Stripping and reprobing of DNA blots (21)4.Results (22)4.1Typical results (22)5.Appendix (23)5.1Trouble shooting (23)5.2References (24)5.3Ordering Information (25)1.2Kit contentsBottle/ Cap Label Contentincluding function1DIG-HighPrime •50 l DIG-High Prime• 5 × conc. labeling mixture containingoptimal concentrations of random primers, nucle-otides, DIG-dUTP (alkali-labile),Klenow enzyme and buffer components •ready-to-use•clear, viscous solution•for efficient random primed labeling of DNA2DIG-labeledControl DNA •20 l•[5 g/ml] pBR328 DNA(linearized with Bam HI)•clear solution•determination of labeling efficiency3DNA DilutionBuffer •3×1ml•[50 g/ml fish sperm DNA in 10 mM Tris-HCl, 1 mM EDTA; pH 8.0 at +25°C]•clear solution4Anti-Digoxigenin-AP Conjugate •100 l•[750 U/ml]•from sheep, Fab-fragments,conjugated to alkaline phosphatase •clear solution5NBT/BCIP• 6 × 1 ml•50x conc. stock solution [18.75 mg/ml nitrobluetetrazolium chloride and 9.4 mg/ml 5-bromo-4-chloro-3-indolyl-phosphate in67% (v/v) DMSO]•can vary between light yellow and brown, clearsolution•reacts with alkaline phosphatase.6Blockingsolution • 4 × 100 ml•10 × conc.•yellow, viscous solution7DIG Easy HybGranules •to give 4 × 100 ml (preparation page 17)•Hybridization solutionAdditional equipment and reagents requiredIn addition to the reagents listed above, you have to prepare several solutions.In the table you will find an overview about the equipment which is needed for the different procedures.Detailed information is given in front of each procedure.Procedure Equipment Reagents3.2 DIG-DNA labeling Water bath•H2O, sterile, doubledistilled water•EDTA, 0.2 M, pH 8.0,sterile3.3 Semi-quantitativedetermination oflabeling efficiencyNylon membranes positivelycharged*•DIG Wash andBlock Buffer Set*•TE-bufferor•Washing buffer•Maleic acid buffer•Detection buffer•TE-buffer3.4 DNA transferand fixation•UV-transilluminator•commercial availableUV-cross linker• 2 × SSCor•10 × SSC3.5 Hybridization•Nylon membranes, positivelycharged*•Hybridization bags*or•Temperature resistant,sealable plastic bags or rollerbottlesNote: Do not use open trays whenworking with DIG Easy Hyb buffer3.6 Immunologicaldetection•Container of appropriate size inrelation to filter size•Hybridization bags*•DIG Wash andBlock Buffer Set*•TE-bufferor•Washing buffer•Maleic acid buffer•Detection buffer•TE-buffer3.7 Stripping andreprobing of DNAblots•Large tray•Water bath•10 × SSC•10% SDS•0.2 M NaOH2.Introduction2.1Product overviewTest principle The DIG High Prime DNA Labeling and Detection Starter Kit I uses digoxigenin(DIG), a steroid hapten, to label DNA probes for hybridization and subsequentcolor detection by enzyme immunoassay [1].Stage DescriptionDNA labeling DIG-labeled DNA probes are generated with DIG-High Primeaccording to the random primed labeling technique. DIG-High Primeis a specially developed reaction mixture containing digoxigenin-dUTP, alkali-labile (Fig. 2) and all reagents, including enzymenecessary for random primed labeling, premixed in an optimized5 × concentrated reaction buffer.Hybridization DIG-labeled probes are used for hybridization to membraneblotted nucleic acids according to standard methods. The use of thealkali-labile form of DIG-11-dUTP enables easier and more efficientstripping of blots for rehybridization with a secondDIG-labeled probe.Immunological detection The hybridized probes are immunodetected with anti-digoxigenin-AP, Fab fragments and are then visualized with the colorimetric substrates NBT/BCIP.ApplicationDIG-labeled DNA probes can be used:•for all types of filter hybridization•for single copy gene detection in total genomic DNA, even from organisms with high complexity, e.g. human, barley, and wheat.Sample material•DNA fragments of at least 100 bp •linearized plasmid, cosmid or DNA •supercoiled DNAAssay timeThis table lists the reaction time of the single steps Number of tests1 kit is sufficient for•12 standard labeling reactions of up to 3 g template DNA and detection of•24 blots of 100 cm 2.Quality ControlUsing unlabeled control-DNA (pBR 328), labeled as described in the protocol, 0.1 pg of homologous DNA is detected in a dot blot after 16 h color develop-ment (1 pg of homologous DNA can be detected after 1h color development).Step Reaction time DNA labeling 1 h-O/N Hybridization6 h or O/N Immunological detection 1.5 h Color development0.5 - 16 hKit storage/ stabilityThe unopened kit is stable at -15 to -25° C until the expiration date printed on the label. Shipping conditions on dry ice.Once opened, please refer to the following table for proper storage.Sensitivity and specificityA single copy human gene is detected in a Southern blot of 1 g digested pla-centa DNA.Note: Sensitivity depends both on the concentration of labeled DNA in the hybridization and on the time of color reaction.AdvantagesThis table describes benefits and features of the kit.Kit componentStorageAnti-Digoxigenin-AP Conjugate cap 42-8° C, stableBlocking solution bottle 6•unopened, stable at 15-25° C•once opened, it should be aliquoted and stored at -15 to -25° C or at 2-8° C up to one month when keeping sterile•working solution should always be prepared fresh NBT/BCIP cap 6•2-8° C,stable•or at least 4 weeks at 15-25° CNote: During shipment of the kit on dry ice, a precipitate may occur which is easily dissolved by briefly warming to 37°CBenefitFeatureAccurate and fastThe use of premixed DIG-High Prime mini-mizes the hands-on-time required to label DNA probes and increases yields and reproducibility.Sensitive Single-copy genes can be detected in total human DNA complex and plant genomes.Time-savingDIG-labeled probes can be stored for at least one year. Hybridization solutions can be reused 3 – 5 times, depending on the amount of labeled probe used for signal generation in each hybridization.3.Procedures and required materials3.1Before you beginGeneral handling rec-ommendationsThis table describes general hints for DIG labeling and detection.OverviewRecommendation GuidelineWork under clean conditions•Autoclave DIG System solutions•Filter-sterilize solutions containing SDS•Tween 20 should be added to previouslysterilized solutionsUse clean incubation trays Rigorously clean and rinse laboratory traysbefore each use.Membrane handling requirements•Wear powder-free gloves•Handle membrane only on the edges andwith clean forcepsSection 3.2DIG-DNA labeling↓Section 3.3Quantification of labeling efficiency↓Section 3.4DNA fixation↓Section 3.5Hybridization↓Section 3.6Immunological detection↓Section 3.7Stripping and reprobing of DNA blots3.2DIG-DNA LabelingIntroduction DNA is random primed labeled with Digoxigenin-11-dUTP using DIG-HighPrime, a 5x concentrated labeling mixture of random hexamers, dNTP mix con-taining alkali-labile Digoxigenin-11-dUTP, labeling grade Klenow enzyme andan optimized reaction buffer.Additional equipment and reagents required •water bath•ice/waterThis table lists composition, storage and use of the required reagents in addi-tion to kit componentsTemplate DNA The following table lists the recommended features of the template DNALabeling of DNA isolated from agaroseIf you intend to perform genomic Southern blotting, you should separate thetemplate insert DNA from the vector by agarose gel electrophoresis.Excise the DNA fragment from an agarose gel. For best results use either our High Pure PCR Product Purification Kit * or our Agarose Gel DNA ExtractionKit* to separate the DNA from the agarose.Solution Composition Storage Use Water Autoclaved, double distilled water15-25° C,stableDilution of DNAEDTA0.2 M ethylenediamino- tetraacetic acid,pH 8.015-25° C,stableStopping thelabeling reactionFeature DetailPurity For plasmid DNA use the High Pure Plasmid Isolation Kit * for purification.When other commercially available purification kits are used, we recommendto do an additional phenol/chloroform extraction to remove residual protein.This step is also necessary when templates have been treated with restrictionor other modifying enzymes before labeling.Size To obtain optimal results, template DNA should be linearized andshould have a size of 100 or larger. Template DNA >5 kb should be restric-tion-digested using a 4 bp cutter (e.g. Hae III) prior to labeling.Amount With the procedure described below principally 10 ng – 3 g of template can be labeled, however, please check in the given table the necessaryamount of probe needed for your size of blot. By scaling up of all volumes andcomponents accordingly this procedure can be used for labeling of largeramounts. If single-copy gene detection in complex genomes is performed atleast 300 ng of template DNA (probe concentration: 25 ng/ml hybridizationsolution) should be labeled.Procedure This procedure is designed for 10 ng-3 g of DNA. Larger amounts (up to 10g) can be labeled by scaling up of all components and volumes.Yield of labeling reac-tion Table 1:This table shows you the yield of DIG-High Prime labeling under optimal condi-tions.In the standard reaction with 1 g DNA per assay approx. 15% of the nucle-otides are incorporated into about 0.8 g of newly synthesized DIG-labeled DNA within 1 h and approx. 38% of the nucleotides into about 2 g after 20 h. Using DIG-High Prime solution, reactions were performed with increasingamountsof different template DNAs for 1 h and 20 h. The yield of DIG-labeled DNA was determined by incorporation of a radioactive tracer and confirmed by a dot blot (Average of 10 independent labeling assays)Step Action1Add1g template DNA (linear or supercoiled) and autoclaved, double distilled water to a final volume of 16 l to a reaction vial.2Denature the DNA by heating in a boiling water bath for 10 min and quickly chilling in an ice/water bath.Note: Complete denaturation is essential for efficient labeling.3•Mix DIG-High Prime (vial 1) thoroughly and add 4 l to the denatured DNA, mix and centrifuge briefly.•Incubate for 1 h or O/N at 37° C.Note: Longer incubations (up to 20 h) will increase the yield of DIG-labeledDNA (see table below ).4Stop the reaction by adding 2 l 0.2 M EDTA (pH 8.0) and/or by heating to 65°C for 10 min.Note: The length of the DIG-labeled fragments obtained with DIG-High Prime range from 200 bp to 1000 bp or larger, depending on the length of theoriginal template.T emplate DNA 1 h20 h10 ng 45 ng 600 ng30 ng 130 ng1050 ng100 ng 270 ng1500 ng300 ng 450 ng2000 ng 1000 ng 850 ng2300 ng3000 ng1350 ng2650 ng.Fig. 2: Yield of DIG-labeled DNA from different amounts of template DNA after 1 and 20 h incu-bation of the DIG-High Prime reaction at 37°C.3.3Determination of labeling efficiencyIntroduction Determination of the yield of DIG-labeled DNA is most important for optimaland reproducible hybridization results. Too high of a probe concentration in thehybridization mix causes background, while too low of a concentration leads toweak signals.Test principle The preferred method for quantification of labeled probes is the direct detec-tion method.Preparation of additional solutions requiredPlease find in the following table composition and preparation of additionalreagents required. The following buffers are also available in the DIG Wash and Block Buffer Set, DNase and RNase free.*Please note: These solutions are also used in the detection procedure ofchapter 3.6. and can be prepared in larger quantities.Stage Description1• A series of dilutions of DIG-labeled DNA is applied to a small strip of nylon membrane positively charged*.•Part of the nylon membrane is preloaded with defined dilutions of DIG-labeled control DNA (vial 2) which are used as standards.2•The nylon membrane is subjected to immunological detection with anti-digoxigenin-AP conjugate (vial 4) and the premixed stock solutionof NBT/BCIP (vial 5).•The intensities of the dilution series of DIG-labeled DNA and control DNA are compared.Solution Composition / Preparation Storage andstabilityUseWashingbuffer0.1 M Maleic acid, 0.15 M NaCl;pH 7.5 (20° C); 0.3% (v/v) Tween 2015-25° C,stableRemovalof unboundantibody Maleic acidbuffer0.1 M Maleic acid, 0.15 M NaCl;adjust with NaOH (solid) to pH 7.5(20° C)15-25° C,stableDilution ofBlocking solutionDetectionbuffer0.1 M T ris-HCl, 0.1 M NaCl,pH 9.5 (20° C)15-25° C,stableAdjustment ofpH to 9.5TE-buffer10 mM T ris-HCl, 1 mM EDTA,pH 8.015-25° C,stableStopping colorreactionPreparation of kit working solutionsThe following table shows the preparation of kit working solutions ProbequantificationLabeled probes and the DIG-labeled control DNA (vial 2) must be diluted to 1 ng/l, according to the expected yield of synthesized nucleic acid to start the dilution series below. The expected yield of DIG-labeled DNA in your probe can best be estimated by using the table 1 in chapter 3.2. The yield depends on the starting amount of template and incubation time.Note: The yields given in table 1 were achieved under optimal conditions with highly purified template DNA.Dilution seriesPrepare a dilution series of your labeled probe and your control DNA as described in the table:Solution Composition/preparationStorage and stability Use Blocking solutionPrepare a 1 × working solution by diluting the 10x Blocking solution (vial 6) 1:10 in Maleic acid buffer.Always prepare fresh Blocking of unspecific binding sites on the membrane Antibody solutionCentrifuge Anti-Digoxigenin-AP (vial 4) for 5 min at 10 000 rpm in the original vial prior to each use, and pipet the necessary amount carefully from the surface. Dilute Anti-Digoxigenin-AP 1: 5 000 (150 mU/ml) in Blocking solution.12 h at 2-8° CBinding to the DIG-labeled probeColor-substrate solutionAdd 40 l of NBT/BCIP stock solution (vial 5) to 2 ml of Detection buffer.Note: Store protected from light!always prepare freshVisualization of antibody-binding TubeDNA (l)From tube #DNA Dilution Buffer (vial 3)(l)DilutionFinal concentration1diluted original1 ng/l 2211981:10010 pg/l 3152351:3.3 3 pg/l 452451:101 pg/l 553451:100.3 pg/l 654451:100.1 pg/l 755451:100.03 pg/l 856451:100.01 pg/l9-50-0Procedure The following procedure describes the direct detection.Note: Use sufficient buffer volumes to cover the membrane completely duringall steps.Analyzing the resultsCompare the intensity of the spots out of your labeling reaction to the control and calculate the amount of DIG-labeled DNA. If the 0.1 pg dilution spots of your probe and of the control are visible, then the labeled probe has reached the expected labeling efficiency (pls. see table 1 in 3.2.) and can be used in the recommended concentration in the hybridization.The following spots should be visibleStep Action1Apply a1l spot of tubes 2-9 from your labeled probes and the labeled control to the nylon membrane.2Fix the nucleic acid to the membrane by cross linking with UV-light or baking for 30 min at 120 ° C.3•Transfer the membrane into a plastic container with 20 mlMaleic acid buffer.•Incubate under shaking for 2 min at 15-25° C.4Incubate for 30 min in 10 ml Blocking solution.5Incubate for 30 min in 10 ml Antibody solution.6Wash with 10 ml Washing buffer, 2 × 15 min.7Equilibrate 2-5 min in 10 ml Detection buffer.8Incubate membrane in 2 ml freshly prepared color substrate solution in a appropriate container in the dark. Do not shake during color development.Note: The color precipitate starts to form within a few minutes. The membrane can be exposed to light for short time periods to monitor color development.9Stop the reaction, when desired spot or band intensities are achieved, by washing the membrane for 5 min with 50 ml of sterile double dist. wateror with TE-buffer.Results can be documented by photocopying the wet filter or by photography.Incubation time Appearance5-10 min30 pg spot30 min 3 pg spot3.4DNA transfer and fixationTransfer methods and membranes Standard protocols for gel electrophoresis, denaturation and neutralization of the gel are described in Sambrook et al. (2). Gels lacking ethidium bromide are preferred, because ethidium can cause uneven background problems. All com-mon types ofDNA transfer methods are suitable for subsequent DIG hybridization (4,5).In our experience, best results are obtained when gels are blotted by capillary transfer with 20 × SSC on nylon membranes*, positively charged.Note: Alkali transfer (e.g., in 0.4 M NaOH) is not suitable for the transfer of DIG-labeled molecular weight markers*.Fixation procedure Fix the DNA to the membrane by any of the following procedures:Storage of the membranePlease refer to the following table.IF you want to...THEN...UV-crosslinking(nylon membrane)•Place the membrane on Whatman 3MM-paper soakedwith 10 × SSC.•UV-crosslink the wet membrane without priorwashing.•After the UV-crosslinking, rinse the membrane brieflyin double distilled water and allow to air-dry.bake at 120° C(nylon membrane)•Wash the membrane briefly in 2 × SSC.•Bake the nylon membrane at 120° C for 30 min oraccording to the manufacturer`s instructions.bake at 80° C(nylon membrane)•Wash the membrane briefly in 2 × SSC.•Bake at 80° C for 2 h under vacuum.IF...THEN...you want to go e the membrane immediately for prehybridization. you want to work later on store the membrane dry at 2-8° C.3.5HybridizationAdditional equipment required •ice/water•shaking water-bath•or hybridization oven•temperature resistant plastic or glass boxes, petri dishes, roller bottles or sealable plastic bags.Note: Do not use open containers with DIG Easy Hyb buffer.Preparation ofDIG Easy Hyb working solution Add carefully 64 ml sterile double distilled water in two portions to the DIG Easy Hyb Granules (bottle 7), dissolve by stirring immediately for 5 min at 37° C.Hybridization temperature The appropriate hybridization temperature is calculated according to GC con-tent and percent homology of probe to target according to the following equa-tion:Tm= 49.82 + 0.41 (% G + C) - (600/l) [l = length of hybrid in base pairs]Topt.=Tm–20 to 25° C(The given numbers of the equation were calculated according to a standard equation for hybridization solutions containing formamide, 50%.) The actual hybridization temperature T opt. for hybridization with DIG Easy Hyb is 20-25° C below the calculated T m value. T opt. can be regarded as a stringent hybridization temperature allows up to 18% mismatches between probe and target. When the degree of homology of your probe to template is less than 80%, you should lower Topt. accordingly (approx. 1.4°C below Tmper 1 % mismatch) and also adjust the stringent washing steps accordingly (i.e. increase SSC concentration and lower washing temperature).Procedure Please refer to the following table.Storage of hybridization solution DIG Easy Hyb containing DIG-labeled probe can be stored at Ϫ15 to Ϫ25° C and be reused several times when freshly denatured at 68°C for 10 min before use.Note: Do not boil DIG Easy Hyb.Stringency washes For most DNA:DNA applications, a stringency wash with 0.5 × SSC is sufficient. The correct post washing conditions have to be determined empirically for each probe.•For human genomic DNA use 0.5 × SSC and 65° C.•Probes > 150 bp and with a high G/C content should be washed at 68° C.•For shorter probes around 100 bp or shorter, the wash temperature must belowered.This table describes how to perform post-hybridization washes.Step Action1•Pre-heat an appropriate volume of DIG Easy Hyb (10 ml/100 cm2 filter) to hybridization temperature (37 - 42° C).•Prehybridize filter for 30 min with gentle agitation in an appropri-ate container.Note: Membranes should move freely, especially if you use severalmembranes in the same prehybridization solution.2Denature DIG-labeled DNA probe (about 25 ng/ml) by boiling for5 min and rapidly cooling in ice/water.Note: As DIG-11-dUTP is alkali-labile, DNA probes cannot be dena-tured by alkali treatment (NaOH).3Add denatured DIG-labeled DNA probe to pre-heated DIG Easy Hyb(3.5 ml/100 cm2 membrane) and mix well but avoid foaming(bubbles may lead to background).4•Pour off prehybridization solution and add probe/hybridization mixture to membrane.•Incubate 4 hours - O/N with gentle agitation.Step Action1Wash 2 × 5 min in ample 2x SSC, 0.1% SDS at 15-25° C under con-stant agitation.2Wash 2 × 15 min in 0.5x SSC, 0.1% SDS (prewarmed to wash temper-ature) at 65-68° C under constant agitation.3.6Immunological detectionAdditional reagents required Please find in the following table composition and preparation of additional reagents required. The following buffers are also available in the DIG Wash and Block Buffer Set, DNase and RNase free*.Preparation of kit working solutions In the following table the preparation of kit working solutions is described.Solution Composition / Preparation Storage andstabilityUseWashingbuffer0.1 M Maleic acid, 0.15 M NaCl;pH 7.5 (20° C); 0.3% (v/v) Tween2015-25° C,stableWashing ofmembraneMaleic acidbuffer0.1 M Maleic acid, 0.15 M NaCl;adjust with NaOH (solid) to pH7.5 (20° C)15-25° C,stableDilution ofBlockingsolution Detectionbuffer0.1 M Tris-HCl, 0.1 M NaCl,pH 9.5 (20° C)15-25° C,stableAlkalinephosphatasebufferTE-buffer10 mM Tris-HCl, 1 mM EDTA, pH8.015-25° C,stableStoppingcolor reactionSolution Composition / Preparation Storage andstabilityUseBlockingsolutionPrepare a 1x working solution bydiluting 10 × Blocking solution(vial 6) 1:10 with Maleic acidbuffer.Alwaysprepare freshBlocking ofunspecificbinding sitesAntibodysolutionCentrifuge Anti-Digoxigenin-AP(vial 4) for 5 min at 10 000 rpm inthe original vial prior to each use,and pipet the necessary amountcarefully from the surface. DiluteAnti-Digoxigenin-AP 1:5000 (150mU/ml) in Blocking solution.12 h at 2-8° C Binding to theDIG-labeledprobeColor-substratesolutionAdd 200 l of NBT/BCIP stocksolution (vial 5) to 10 ml of Detec-tion buffer.Note: Store protected from light!Alwaysprepare freshVisualizationof antibody-bindingProcedure This table describes how to perform the immunological detection ona 100 cm2 membrane.Note: All incubations should be performed at 15-25° C with agitation. If themembrane is to be reprobed, do not allow the membrane to dry at any time.Storage of membrane Please refer to the following table.Step Action1After hybridization and stringency washes, rinse membrane briefly (1-5) min in Washing buffer.2Incubate for 30 min in 100 ml Blocking solution.3Incubate for 30 min in 20 ml Antibody solution.4Wash 2 x 15 min in 100 ml Washing buffer.5Equilibrate 2-5 min in 20 ml Detection buffer.6Incubate membrane in 10 ml freshly prepared color substrate solu-tion in a appropriate container in the dark. Do not shake duringcolor development.Note: The color precipitate starts to form within a few minutes andthe reaction is usually complete after 16 h. The membrane can beexposed to light for short time periods to monitor color development.7Stop the reaction, when desired spot or band intensities are achieved, by washing the membrane for 5 min with 50 ml of steriledouble dist. water or with TE-buffer.Results can be documented by photocopying the wet filter or by pho-tography.IF...THEN...you want to reprobe the membrane the membrane should not dry off at anytime, store in sealed plastic bag.Note: If you want to maintain the color,store membranes in TE buffer do notallow the membrane to dry.you don´t want to reprobe dry the membrane at 15 to 25°C for stor-age.Note: Color fades upon drying, to revita-lize the color, wet the membrane in TEbuffer.3.7Stripping and reprobing of DNA blotsGeneral The alkali-labile form of DIG-11-dUTP enables easier and more efficient strip-ping of blots for rehybridization experiment.Additional reagents required •Dimethylformamid (DMF)•0.2 N NaOH, 0.1% SDS (w/v)• 2 × SSCProtocolStorage of stripped membrane Please refer to the following table.Note: When stripping and rehybridization of blots is planned, the membrane should not dry off at any time.CAUTION: Work under a fume hoodStep Action1•Heat DMF in a large glass beaker in a water bath under a fume hood to 50-60° C.•Incubate the membranes in the heated DMF until the blue color precipitate is removed from the filter.CAUTION: DMF is volatile and can be ignited above 67° C.2Rinse membrane briefly in double distilled water.3Wash for 2 × 15 min in 0.2 N NaOH, SDS, 0.1% (w/v) at 37° C under constant agitation.4Equilibrate briefly in 2 × SSC.5Prehybridize and hybridize with a second probe.Once the membrane is stripped, it can be stored in Maleic acid buffer or2 × SSC until used again.4.Results4.1Typical resultsGenomic Southern blotFigure 3: This figure shows you the detection of a single-copy gene (ß-actin) in total human DNA using the standard protocol.Size (kb)1234215.1/5.04.33.52.01.91.61.40.950.830.562.551.0 。
地高辛标记及检测试剂盒说明书

仅仅研究于生命科学,不适合在诊断过程使用只能在体外使用地高辛DNA标记和检测试剂盒1和NBT/BCIP一起用于颜色检测通过酶联免疫法采用地高辛-dUTP进行随机引物DNA标记,碱性标记和检测货号:11 745 832 910此试剂盒储存条件-15o C—-25o C此试剂盒可对10ng-3ugDNA进行12次标记反应可检测100cm2面积的杂交膜24张指导手册2009.11版1前言1.1内容表1前言 (2)1.1内容表 (2)1.2试剂盒内容 (3)2简介 (5)2.1产品概述 (5)3步骤和所需材料 (8)3.1开始之前 (8)3.2地高辛DNA标记 (9)3.3 标记效率的测定 (11)3.4 DNA 的转移和固定 (14)3.5杂交 (16)3.6免疫检测 (18)3.7 DNA印记的洗脱和再杂交 (20)4结果 (21)4.1典型的结果 (21)5附录 (23)5.1故障排除 (23)5.2引用 (24)5.3订购信息 (25)1.2 试剂盒内容附加仪器与所需试剂除了上表中列出的试剂外,你必须准备一些溶液。
在下表中,你可以找到不同操作程序需要准备的设备概要。
*标记的产品可以从Roche Applied Science 获得2 介绍2.1 产品概况实验原则此试剂盒采用地高辛(DIG),一种甾类半抗原,去标记DNA探针从而通过酶免疫分析应用地高辛标记DNA探针可以用于:所有类型的滤膜杂交总基因组DNA中单拷贝基因的检测,甚至对于高度复杂的生物也可以进行检测,如人,大麦和小麦。
样品材料至少100bp的DNA片段线性的质粒,cos质粒或者λDNA超螺旋的DNA实验时间此表格列出了每一步实验所需的反应时间检测数量一个试剂盒足够用于:不超过3ug的模板DNA的12个标准的标记反应和100cm2有24个斑点的检测质量控制根据操作步骤中的描述,对未标记的对照品DNA(PBR328)进行标记,0.1pg同源DNA 在点杂交中通过16h的显色来检测(1pg同源DNA可以通过1小时的显色进行检测)。
地高辛(Digoxin )标准操作程序SOP文件

·Elecsys测量池清洗液(CleanCell)货号11662970
·Elecsys添加剂液(SysWash)货号11930346
·Elecsys系统清洗液支架(Adapter for SysClean))货号11933159
·Elecsys 2010反应杯(Assay Cup)货号11706802
概述:
地高辛被广泛应用于充血性心功能衰竭的治疗和各种心率失常。地高辛能改善心肌收缩的强度,增加心脏血液输出,缩小心脏,降低静脉压和血容量。地高辛治疗也
可稳定和抑制室性心率。由于地高辛使用的经常性和治疗过程中的不谨慎,容易引起地高辛中毒。更危险的是地高辛中毒症状常以心率失常形式表现出来,而病人正是由于这种症状而用地高辛治疗的。公认的血清或血浆地高辛治疗浓度是0.9-2.0ng/ml。人类地高辛中毒症状一般只出现在地高辛浓度超过2.0ng/ml,但有时低至1.4ng/ml也可出现中毒症。地高辛中毒可出现在以下几种情况a)药物疗效比率较低(如达到治疗效果的组织浓度与产生中毒的组织浓度之间差异很小;b)个体对地高辛的反应程度不相同;c)各种地高辛药片的吸收率差异可达二倍以上d)年龄增高,对地高辛中毒的易感性明显增强。与其它临床资料结合,监测地高辛浓度可以为医生及时调整用药剂量提供有用的信息,从而达到理想的治疗效果,避免药物不足或过量引起中毒。Elecsys地高辛测定方法采用竞争法原理和一种地高辛特异的单克隆抗体。标本中的地高辛与生物素化的地高辛衍生物竞争钌标记的单克隆抗体。
原理:
采用竞争法原理,整个过程18分钟完成。
·第1步:10µl标本与钌(Ru)标记的抗地高辛单克隆抗体混匀,形成免疫复合物,其数量取决于标本中分析物的浓度。
地高辛DNA标记试剂盒II

灵敏性: 在1µg已消化的人类胎盘DNA的
Southern Blot实验中,可检测到单拷贝的基 因。
DNA探针标记方法
步骤
操作方法
在反应管(例如1.5ml微量离心管)
中加入10ng-3µg的模板DNA,然后
1 加入无核酸酶的水至15µl。对于对
照 反 应 , 加 入 1µl 对 照 DNA 和 14µl
产品说明书
注意:孵育过程中应避光,避免摇动,可 短时观察显色进程。
7.用双蒸水或TE缓冲液洗涤5分钟终止反 应,照相记录结果。
图示:
标记的探针量:
模板DNA量 反应1小时
10 ng
15 ng
30 ng
30 ng
100 ng
60 ng
300 ng
Байду номын сангаас
120 ng
1000 ng
260 ng
3000 ng
530 ng
应。
二. 探针标记效率检测: 取1µl标记产物,用DNA稀释液稀释后点
样于试剂盒提供的标准膜条上,用紫外交联 仪或真空烘烤固定DNA探针,按下述方法进 行信号检测,然后与标准膜条上的信号强度 对比,推算出标记的探针浓度。
深圳莱伯克生物科技有限公司
稀释方法:
试管号
标记的 DNA(µl)
DNA 稀释液(µl)
应用领域: 地高辛标记的探针高度稳定,用途广泛。
可用于: z 非同位素的 Southern Blot z Northern Blot z 原位杂交(ISH) z 菌落杂交 z 斑点印迹杂交分析等。
质量控制: 用GAPDH对照DNA按照下述的操作
方法验证,在点杂交实验中,0.1 pg同源的 DNA在显色16小时后可检测到 (1 pg同源的 DNA,在显色1小时后可检测到)。
DNA 残留量检测试剂盒中文说明书

DNA-DIG标记:1、每个标记反应中加入10ng-3ug DNA,另加ddH2O补充至15ul体积。
(未标记的对照DNA加5ul,10ul ddH2O)2、将装有反应物的离心管放入沸水中变性10min,然后迅速冷却;3、然后依次加入以下溶液:Hexanucleotide Mix, 10× 2uldNTP Labeling Mix 2ulKlenow enzyme labeling grade 1ul轻微离心,37℃过夜。
4、加入2ul EDTA(0.2M Ph8.0)终止反应。
(或65℃,10min)标记效率检测:标记好的探针和对照稀释到1ng/ul。
然后依次稀释为10pg/ul、3pg/ul、1pg/ul、0.3pg/ul、0.1pg/ul、0.03pg/ul、0.01pg/ul,检测各浓度的显色情况。
1、分别取1ul各种浓度的探针点到一小块尼龙膜上;2、紫外交联固定(或120℃30min烘烤固定)3、将膜放在一个装有20ml Maleic acid buffer的塑料容器中,15℃-25℃摇2min;4、 10ml Blocking solution温浴30min;5、 10ml Antibody solution温浴30min;6、 10ml Washing buffer洗15min,换buffer后再洗15min;7、在10ml Detection buffer中平衡2-5min;8、黑暗条件下,将膜于2ml新鲜的colorsubstrate solution中显色(显色时不能摇动)9、待显色完全后TE-buffer或ddH2O中洗5min终止反应。
杂交:1、将适量体积(20ml/100cm2)的DIG Easy Hyb中预热到杂交温度,轻微摇动预杂交30min;2、 DIG-labeled 探针(约25ng/ml)于沸水中煮5min变性,迅速冰浴;3、变性的DIG-labeled探针加入适量(3.5ml/100cm2)预热的DIG Easy Hyb中,混合,避免气泡产生;4、倒掉预杂交液,加入探针和预热的DIG Easy Hyb的混合液于膜上,杂交过夜。
地高辛(Digoxin)使用说明书

地高辛(Digoxin)使用说明书地高辛(Digoxin)使用说明书一、药品名称地高辛(Digoxin)二、适应症地高辛主要用于心力衰竭和心律失常的治疗。
三、使用方法和剂量1. 成人用法:(1)心力衰竭:初始剂量:每日0.125-0.25毫克口服,分2-3次服用。
维持剂量:每日剂量根据患者的临床情况逐渐调整,但一般不超过0.5毫克。
(2)心律失常:房性心律失常:根据患者临床情况,起始剂量为0.25-0.5毫克每日1次。
随后根据患者反应和心电图监测结果逐渐调整剂量。
室性心律失常:根据患者具体情况,开始推荐剂量为1.0-1.5毫克每日1次。
剂量可根据患者反应和心电图监测结果逐渐调整。
2. 儿童用法:儿童剂量需根据患者的具体情况,结合体重和心功能状况进行调整。
首剂维持剂量通常为0.01-0.03毫克每千克,分剂2-3次口服。
四、禁忌症以下情况患者禁用地高辛:1. 对地高辛或其他洋地黄类药物过敏;2. 严重房室传导阻滞(第二度或二度以上);3. 窦房结功能低下,除非也内步或外步植入物并同时应用心脏起搏器;4. 快速性心律失常,如心房颤动或心室颤动;5. 严重的左心室功能受损;6. 妊娠期间禁用。
五、不良反应地高辛可能引起以下不良反应:1. 心律不齐:如房性和室性心律失常;2. 消化系统反应:如恶心、呕吐、腹泻和胃肠不适;3. 神经系统反应:如头痛、视觉异常和混乱;4. 毒性反应:如地高辛中毒,表现为恶心、呕吐、心律失常、心绞痛等。
地高辛中毒危险性增加主要是因为剂量过高或过敏,或者临床情况发生改变。
六、注意事项1. 在使用地高辛前,应对患者进行全面评估,包括心功能、肾功能、电解质水平等。
2. 患者需要定期监测心电图、心率和血药浓度,以确保地高辛在治疗范围内。
3. 地高辛具有窄治疗范围,必须严格控制剂量,注意患者年龄、体重、肾功能等因素的影响。
4. 患者必须告知医生正在使用或打算使用的其他药物,以避免与地高辛产生不良相互作用。
地高辛测定试剂盒(荧光免疫层析法)产品技术要求丹大

地高辛测定试剂盒(荧光免疫层析法)适用范围:该产品用于体外定量检测人血清、血浆或全血中的地高辛含量。
1.1规格1人份/盒;10人份/盒;25人份/盒;50人份/盒;100人份/盒质控品(选配,1平) :0.5mL×1 1mL×11.2组成试剂盒组成见表1表1地高辛测定试剂盒组成注:质控品赋值具有批特异性,每批次靶值详见标签。
2.1 外观试剂盒外观应整洁, 文字符号标识清晰,液体无渗漏,测试卡完整无破损,冻干品复溶后为无色至淡黄色液体。
2.2空白限应不高于0.2ng/mL。
2.3线性范围在[0.2,5] ng/mL内,相关系数R≥0.990。
2.4重复性重复测试(1.4±0.3)ng/mL和(2.8±0.6)ng/mL的样本,所得结果的变异系数(CV%)应不大于15%。
2.5批间差测试(1.4±0.3)ng/mL和(2.8±0.6)ng/mL的样本,批间相对极差(R)应不大于15%。
2.6准确度回收率为85~115%。
2.7质控品赋值有效性试剂盒内的质控品,检测结果均在质控范围内。
2.8质控品瓶间差瓶间差CV≤15%。
2.9稳定性2.9.1 效期稳定性试剂有效期为18个月取到效期后3个月内进行检测,测定结果应符合2.2-2.8、2.9.2(2.5项除外)项要求。
2.9.2质控品复溶稳定性质控品复溶后在2~8℃密封避光保存24h。
检测复溶后的质控品,检测结果均在质控范围内。
2.10溯源性根据GB/T21415-2008的要求,校准曲线溯源至工作校准品,工作校准品可溯源至药品标准物质100015。
地高辛标记与检测DNA试剂盒说明书

地高辛标记与检测D N A试剂盒DIGDNALabelingandDetectionKit采用地高辛-dUTP进行随机引物DNA标记,碱性标记,利用NBT/BCIP酶联免疫,随时即用。
此试剂盒可对10ng-3μgDNA进行25次标记反应,可检测10×10cm2面积的杂交膜50张指导手册1前言1.1内容表1前言1.1内容表1.2试剂盒成分2.介绍2.1产品简介3.操作步骤和所需材料3.1在你开始前3.2流程图3.3地高辛标记DNA3.4标记效率的确定3.5DNA的转移和固定3.6杂交3.7免疫检测3.8DNA印记的洗脱和再杂交1.2试剂盒的成分瓶号标记内容包括功能1 无标记对照DNA12 无标记对照DNA23 DNA稀释缓冲液2管1mL50μg/mL鱼精DNA,10mMTris-HCl,1mMEDTA,pH8.0在25℃澄清溶液4 DIG标记的对照DNA20μL5μg/mL质粒pBR328DNA(用BamHI线性化)澄清溶液用于确定标记效率5 六聚核苷酸混合物6 标记用混合物7 DNA聚合酶1大片段标记级8 抗地高辛的碱性磷酸酶结合物200μL750U/mL从羊中获取的,Fab段,结合碱性磷酸酶澄清溶液9 NBT/BCIP10 封阻试剂附加仪器与所需试剂除了上表中列出的试剂外,你必须准备一些溶液。
在下表中,你可以找到不同操作程序需要准备的设备概要。
在每个操作程序的前面提供了详细的信息。
操作程序仪器设备试剂3.3DIG-DNA标记水浴灭菌的双蒸水0.2MpH8.0灭菌的EDTA3.4标记效率的半定量带正电荷的尼龙膜* 地高辛洗涤和封阻缓冲组合*TE缓冲液或者洗涤缓冲液马来酸缓冲液检测缓冲液3.5DNA转移和固定紫外光盒或者商业化可用于紫外交联的其他设备2×SSC或者10×SSC3.6杂交尼龙膜,带正电荷*杂交袋*,或者耐热的塑料袋或者滚筒瓶(分子杂交炉)注意:当用地高辛杂交缓冲液工作时,不要用开口的托盘。
地高辛标记与检测DNA试剂盒说明书

地高辛标记与检测D N A试剂盒说明书集团企业公司编码:(LL3698-KKI1269-TM2483-LUI12689-ITT289-地高辛标记与检测D N A试剂盒DIGDNALabelingandDetectionKit采用地高辛-dUTP进行随机引物DNA标记,碱性标记,利用NBT/BCIP酶联免疫,随时即用。
此试剂盒可对10ng-3μgDNA进行25次标记反应,可检测10×10cm2面积的杂交膜50张指导手册1前言1.1内容表1前言1.1内容表1.2试剂盒成分2.介绍2.1产品简介3.操作步骤和所需材料3.1在你开始前3.2流程图3.3地高辛标记DNA3.4标记效率的确定3.5DNA的转移和固定3.6杂交3.7免疫检测3.8DNA印记的洗脱和再杂交1.2试剂盒的成分附加仪器与所需试剂除了上表中列出的试剂外,你必须准备一些溶液。
在下表中,你可以找到不同操作程序需要准备的设备概要。
在每个操作程序的前面提供了详细的信息。
*标记的产品可以从RocheAppliedScience获得。
2.介绍2.1产品概况实验原则此试剂盒采用DIG(地高辛),一种甾类半抗原,steroidhapten去标记DNA探针用于杂交和后续的免疫检测。
应用地高辛标记DNA探针可以用于:所有类型的滤膜杂交样品材料至少100bp的DNA片段线性的质粒,cos质粒或者DNA超螺旋的DNA实验时间检测数量一个试剂盒足够用于:25个标准的标记反应,每个反应的膜板DNA不超过3μg;并可以检测50张10×10cm2的杂交膜。
质量控制根据操作步骤中的说明对未标记的对照DNA【pBR328】进行标记,并按照下面的标准检测操作方法在点杂交中用0.1pg同源DNA点在膜上16h 后成色显影检测。
试剂盒的储存和稳定性未开封的试剂盒在保质期内可以稳定的存放在-15℃到-25℃(保质期印在标签上)。
在干冰中运输。
一旦开封,请根据下表选择适宜的储存条件。
地高辛(Dig)测定试剂盒(化学发光免疫分析法)产品技术要求yuande

地高辛(Dig)测定试剂盒(化学发光免疫分析法)适用范围:用于体外定量测定人血清或血浆中的地高辛(Dig)的含量。
1.1 产品规格试剂盒规格为48人份/盒、96人份/盒。
1.2 主要组成成分表1 地高辛(Dig)测定试剂盒(化学发光免疫分析法)主要组成成分a) 酶结合物以含1% BSA的0.9%Nacl缓冲液配制的联接HRP的Dig抗原,其中含0.5%ProClin300做为防腐剂。
b) 反应抗体以含1% BSA的0.9%Nacl缓冲液配制的兔抗Dig,其中含0.5%ProClin300做为防腐剂。
c) 校准品校准品以100%小牛血清为稀释液,其中含0.5%ProClin300做为防腐剂,校准品A~F中含Dig的目标浓度分别约为0、0.5µg/L、1.0µg/L、2.0µg/L、3.0µg/L、5.0 µg/L。
校准品具体浓度详见标签及试剂盒参数IC卡。
d) 发光液发光液A主要成份为鲁米诺,发光液B主要成份为过氧化脲,两者均以pH8.6的Tris-HCl缓冲液配制。
e) 包被微孔板包被有羊抗兔抗体的白色聚苯乙烯微孔板,用铝箔袋真空包装。
f) 质控品(备选)以100%正常人血清为基质制备的冻干品,其中含0.5%ProClin300做为防腐剂,靶值浓度范围为1.62 µg/L~2.31 µg/L。
质控品具体浓度详见质控品参数表。
不同批号试剂盒中的相同组分不能互换。
2.1 外观a)液体组分应澄清,无沉淀或絮状物,实际装量应不小于标示装量;b)冻干组分呈白色或淡黄色疏松体,加水后应在3分钟内完全溶解;c)所有组分均无包装破损,标示清楚。
2.2 准确度试剂盒内校准品与相应浓度的国家标准品(编号:100015)同时进行分析测定,用Logit-Ln模型拟合,要求两条剂量-反应曲线不显著偏离平行(t检验);以国家标准品为对照品,试剂盒内校准品的实测值与标示值的效价比应在0.900~1.100之间。
DIG试剂盒说明书(CSPD)中文翻译版讲解

只用于生命科学研究,不能用于诊断程序仅用于体外实验DIG High Prime DNA Labeling and Detection Starter KitⅡ采用地高辛-dUTP进行随机引物DNA标记,碱性标记,利用CSPD化学发光检测,随时即用。
货号:11 585 614 910此试剂盒储存条件:-15℃到-25℃。
此试剂盒可对10ng-3μgDNA进行12次标记反应,可检测10×10cm2面积的杂交膜24张。
指导手册2005.12 版1 前言1.1 内容表1 前言1.1 内容表1.2 试剂盒成分2.介绍2.1 产品简介3. 操作步骤和所需材料3.1 在你开始前3.2 流程图3.3 地高辛标记DNA3.4 标记效率的确定3.5 DNA的转移和固定3.6 杂交3.7 免疫检测3.8 DNA印记的洗脱和再杂交4. 附录4.1 故障排除4.2 参考文献4.3 订购信息1.2 试剂盒的成分附加仪器与所需试剂除了上表中列出的试剂外,你必须准备一些溶液。
在下表中,你可以找到不同操作程序需要准备的设备概要。
在每个操作程序的前面提供了详细的信息。
*标记的产品可以从Roche Applied Science 获得。
2.介绍2.1 产品概况实验原则此试剂盒采用DIG(地高辛),一种甾类半抗原,steroid hapten 去标记DNA探针用于杂交和后续的免疫检测[1,2,3]。
图1 DIG-dUTP ,碱性标记图2 CSPD反应应用地高辛标记DNA探针可以用于:所有类型的滤膜杂交总基因组DNA中单拷贝基因的检测,甚至对于高度复杂的生物体也可以进行检测,如人,大麦和小麦。
样品材料至少100bp的DNA片段线性的质粒,cos质粒或者DNA超螺旋的DNA实验时间此表格列出了每一步实验所需的反应时间。
检测数量一个试剂盒足够用于:12个标准的标记反应,每个反应的膜板DNA不超过3μg;并可以检测24张10×10cm2的杂交膜。
(中文) DIG High Prime DNA标记及杂交检测试剂盒 I

DIG High Prime DNA Labeling and Detection Starter Kit I DIG High Prime DNA标记及杂交检测试剂盒I说明书版本:Version11 (2010年8月)利用随机引物法及DIG-dUTP(碱不稳定)进行DNA标记反应,采用酶联免疫方法及发色底物NBT/BCIP进行杂交分子的显色。
本试剂盒可对10ng-3μg DNA进行12次标记反应,可进行24次杂交检测反应(100cm2杂交膜)。
产品目录号:11 745 832 910试剂盒于-15 ~ -25℃保存1. 概况1.1 说明书内容1. 概况 (2)1.1说明书内容 (2)1.2试剂盒组成 (3)2. 介绍 (4)2.1 产品概述 (4)3. 实验步骤和需要的材料 (7)3.1 实验开始前准备 (7)3.2 DIG-DNA标记 (7)3.3定量标记反应效率 (10)3.4 DNA转膜和固定 (12)3.5杂交 (13)3.6 免疫检测 (14)3.7探针洗脱和重探 (16)4. 结果 (17)4.1 典型结果 (17)5 附录 (18)5.1 疑难问题解决 (18)5.2参考文献 (18)1.2 试剂盒组成杂交反应所需的仪器和其他反应试剂请根据您的实验操作流程准备杂交反应所需的仪器和其他试剂,参见下表:带*的试剂为罗氏应用科学部提供的试剂。
2. 介绍2.1 产品概述反应原理DIG High Prime DNA标记检测试剂盒(I)采用地高辛(DIG,一种甾类半抗原)进行DNA 标记,所获得的探针用于后续的杂交反应,带有地高辛分子的杂交子通过酶联免疫反应进行显色检测。
应用DIG标记的DNA探针可用于:•所有形式的膜杂交反应•(微生物,人类,植物等)基因组DNA中的单拷贝基因检测样本•大小在100bp以上的DNA片段•线性化质粒,粘粒或λDNA•超螺旋DNA分析时间试剂盒反应次数本试剂盒包含•对10ng-3μg DNA进行12次标记反应• 24次杂交检测反应(100cm2杂交膜)质量控制对DNA对照品(pBR328,未标记)进行标准标记反应,将0.1pg同源DNA点膜后进行斑点杂交,显色16h呈阳性检测结果(1pg同源DNA的杂交反应显色1h后呈阳性检测结果)。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
地高辛标记与检测DNA试剂盒DIG DNA Labeling and Detection Kit 采用地高辛-dUTP进行随机引物DNA标记,碱性标记,利用NBT/BCIP 酶联免疫,随时即用。
此试剂盒可对10ng-3μgDNA进行25次标记反应,可检测10×10cm2面积的杂交膜50张指导手册1 前言1.1 内容表1 前言1.1 内容表1.2 试剂盒成分2.介绍2.1 产品简介3. 操作步骤和所需材料3.1 在你开始前3.2 流程图3.3 地高辛标记DNA3.4 标记效率的确定3.5 DNA的转移和固定3.6 杂交3.7 免疫检测3.8 DNA印记的洗脱和再杂交1.2 试剂盒的成分瓶号标记内容包括功能1 无标记对照DNA12 无标记对照DNA23 DNA稀释缓冲液2管1mL50μg/mL鱼精DNA,10mM Tris-HCl,1mM EDTA, pH8.0 在25℃澄清溶液4 DIG标记的对照DNA20μL5μg/mL 质粒pBR328 DNA (用Bam HI线性化)澄清溶液用于确定标记效率5 六聚核苷酸混合物6 标记用混合物7 DNA聚合酶1大片段标记级8 抗地高辛的碱性磷酸酶结合物200μL750U/mL从羊中获取的,Fab段,结合碱性磷酸酶澄清溶液9 NBT/BCIP10 封阻试剂附加仪器与所需试剂除了上表中列出的试剂外,你必须准备一些溶液。
在下表中,你可以找到不同操作程序需要准备的设备概要。
在每个操作程序的前面提供了详细的信息。
操作程序仪器设备试剂3.3 DIG-DNA标记水浴灭菌的双蒸水0.2M pH8.0 灭菌的EDTA3.4 标记效率的半定量带正电荷的尼龙膜* 地高辛洗涤和封阻缓冲组合*TE缓冲液或者洗涤缓冲液马来酸缓冲液检测缓冲液3.5 DNA转移和固定紫外光盒或者商业化可用于紫外交联的其他设备2×SSC或者10×SSC3.6 杂交尼龙膜,带正电荷*杂交袋*,或者耐热的塑料袋或者滚筒瓶(分子杂交炉)注意:当用地高辛杂交缓冲液工作时,不要用开口的托盘。
DIG Easy Hyb(杂交缓冲液,需单独购买)3.7 免疫检测耐热的塑料袋或者滚筒瓶杂交袋* 地高辛洗涤和封阻缓冲组合* TE缓冲液或者洗涤缓冲液马来酸缓冲液检测缓冲液3.8 DNA 斑点的脱色和大的托盘DMF重新标记探针水浴0.2M NaOH,0.1%SDS2×SSC*标记的产品可以从Roche Applied Science 获得。
2.介绍2.1 产品概况实验原则此试剂盒采用DIG(地高辛),一种甾类半抗原,steroid hapten去标记DNA 探针用于杂交和后续的免疫检测。
步骤描述DNA 标记根据随机引物标记技术采用地高辛标记引物获得了地高辛标记的DNA探针。
地高辛标记的高效引物是一种专门生产的反应混合物,其中含有地高辛dUTP,碱性标记和所有的试剂,包括随机引物标记所必须的酶,已预先混合优化的5×浓度反应缓冲液。
杂交根据标准方法,地高辛标记的探针可以与固定在膜上的核酸杂交。
采用碱标记形式的地高辛-11-dUTP能够更加容易和有效的被洗脱下来,使固定在膜上的核酸可以与其他的地高辛标记探针再次杂交。
免疫检测杂交探针可以用抗地高辛的碱性磷酸酶进行免疫检测,Fab 段,然后采用即时可用的化学发光底物NBT/BCIP可以看到杂交结果。
应用地高辛标记DNA探针可以用于:所有类型的滤膜杂交样品材料至少100bp的DNA片段线性的质粒,cos质粒或者DNA超螺旋的DNA实验时间步骤反应时间DNA标记1h-O/N杂交 6 h or O/N免疫检测 1.5h显色0.5-16h检测数量一个试剂盒足够用于:25个标准的标记反应,每个反应的膜板DNA不超过3μg;并可以检测50张10×10cm2的杂交膜。
质量控制根据操作步骤中的说明对未标记的对照DNA 【pBR328】进行标记,并按照下面的标准检测操作方法在点杂交中用0.1pg 同源DNA点在膜上16h后成色显影检测。
试剂盒的储存和稳定性未开封的试剂盒在保质期内可以稳定的存放在-15℃到-25℃(保质期印在标签上)。
在干冰中运输。
一旦开封,请根据下表选择适宜的储存条件。
试剂盒成分储存条件抗地高辛碱性磷酸酶结合物(8号管)2-8℃稳定。
NBT/BCIP (9号管) 2-8℃,避光保存或者15-25℃保存4周封阻液(10号管)未开封时,2-8℃或者15-25℃稳定;一旦开封,应分装并储存在-15℃到-25℃或者无菌条件下2-8℃间可以稳定保存1个月;工作溶液都应是新鲜配制的灵敏度和特异性采用southern杂交从1μg人胎盘DNA(经BglⅡ或EcoRⅠ酶切)中检测到单拷贝基因。
3.实验步骤和所需材料3.1 在你开始前主要的操作要求此表中描述了地高辛标记和检测中主要的提示:要求指引在清洁的条件下进行操作高压灭菌地高辛系统的试剂过滤灭菌的溶液包括:SDS,吐温20,并应该加入到已灭菌的溶液中。
用干净的孵育托盘每次使用前都要严格地清洗实验托盘处理膜的要求带无粉手套处理膜的时候,只能够用干净的镊子夹膜的边缘3.2 流程图3.3部分地高辛标记DNA↓3.4部分标记效率的检测↓3.5部分DNA固定↓3.6部分杂交↓3.7部分免疫检测↓3.8部分洗脱和DNA斑点与另一探针的杂交3.3 地高辛标记DNA介绍随机引物标记的DNA带有地高辛-11-dUTP,这一标记采用的是地高辛高效引物试剂,这是一种含有随机六碳糖,dNTP混合物的5×浓度标记混合物,其中dNTP混合物包括碱性标记的地高辛-11-dUTP,标记级的Klenow酶和适合的反应缓冲液。
附加设备和所需试剂水浴冰水混合物此表中列出了成分,储存条件和除了试剂盒成分外所需试剂的用途。
溶液成分储存条件/稳定性用途水高压灭菌的双蒸水15-25℃,稳定稀释DNAEDTA(乙二胺四乙酸)0.2M EDTA,pH 8.0 15-25℃,稳定终止标记反应模板DNA:模板DNA所需特征:特征详细信息纯度模板DNA应该采用the High Pure Plasmid Isolation Kit进行制备。
当采用其他商业化的纯化试剂盒进行纯化时,我们建议额外进行一次苯酚/氯仿抽提以去除残余的蛋白质。
在标记之前如果对模板进行限制酶切或者其他酶的修饰作用,那么此步骤也是需要进行的。
大小为了获得理想的结果,模板DNA应该线性化,且大小应该在100-10000bp以上。
如果模板DNA大于10kb,那么在标记之前应该采用限制性酶切序列为4个核苷酸的限制性酶(如Hae Ⅲ)对模板进行酶解。
数量上面步骤描述原则上10ng-3μg的模板均可以标记上,然而请仔细查看在给定的表中,用于你的杂交实验所需的探针。
可以根据提供的操作方法放大总体积和成分用量来获得更大量的标记探针。
如果要检测复杂基因组中的单拷贝基因,需标记的模板DNA用量至少300ng(探针浓度:25ng/mL 杂交液)。
标记从琼脂糖凝胶上分离的DNA如果你想要完成基因组的southern 杂交,你应该利用琼脂糖凝胶电泳将插入载体的模板DNA从载体上分离出来。
为了从凝胶中分离DNA,你可以用琼脂糖凝胶DNA提取试剂盒(the AgaroseGel DNA Extraction Kit)进行大小在400bp-5kbp范围内DNA片段的回收。
这一试剂盒既可以用于标准琼脂糖凝胶也可用于低熔点琼脂糖凝胶。
然后,不需要进一步纯化即可对DNA片段进行高效的地高辛标记。
然后标记好的探针应该用高效的PCR产物纯化试剂盒(High Pure PCR Product Purification Kit)进行纯化,以去除残留的琼脂糖颗粒。
步骤此步骤用于标记10ng-3μg DNA 。
可以通过扩大所有成分和体积标记更大量的DNA(最高达10μg)。
步骤操作1 向一个反应管仲中加入10ng-3μg模板DNA(线性或超螺旋)和高压灭菌的双蒸水,终体积达15μl;作为对照,向另一管中加入5μl无标记对照DNA (Vial 2)和高压灭菌水,终体积达15μl2 沸水浴10分钟以变性DNA,然后迅速插入冰水混合物中注意:完全变性对于标记的有效性是十分重要的3 加入以下试剂到变性探针或对照DNA中:试剂体积Vial 5 2μLVial 6 2μLVial 7 1μL充分混合并简单离心;37℃孵育1小时或者过夜注意:较长的孵育时间(最高达20小时)能够提高地高辛标记DNA的产量4 加入2μL 0.2M EDTA(pH8.0) 和/或65℃加热10分钟终止反应注意:采用地高辛高效引物获得的地高辛标记片段的长度范围可以从200bp到1000bp甚至更长,这主要取决于原始模板DNA的长度标记反应的产量表1:此表向您展示了在适宜条件下地高辛高效引物标记的产量。
在标准反应中每个实验采用1μg DNA 作为模板。
1小时内,大约有15%的核苷酸参与到了0.8μg 新合成的地高辛标记DNA中。
20小时后消耗了大约38%的核苷酸。
模板DNA 1h 20h10 ng 15 ng 50 ng30 ng 30 ng 120 ng100 ng 60 ng 260 ng300 ng 120 ng 500 ng1000 ng 260 ng 780 ng3000 ng 530 ng 890 ng使用DIG-High-Prime溶液,增加不同模板DNA的量进行反应,并检测反应1小时和反应20小时的差异。
地高辛标记DNA的产量由放射线示踪器进行测定,并通过斑点杂交进行证实(10个独立标记实验的平均值)。
3.4 标记效率的确定介绍地高辛标记DNA产量的确定对于获得最佳的,具有可重现性的杂交结果十分重要。
在杂交混合物中探针浓度过高容易产生背景,而太低浓度则容易导致信号弱。
实验原则检测标记探针质量的好方法是直接检测法。
步骤描述1 将地高辛标记DNA的一系列稀释梯度点到一小块带正电荷的尼龙膜上;尼龙膜部分区域已经点好一系列稀释梯度的地高辛标记的对照DNA(Vial 4)作为标准。
2 采用anti-DIG-AP结合物(Vial 8)和随时即用的NBT/BCIP对尼龙膜进行免疫检测。
比较地高辛标记DNA与对照DNA的一系列稀释点的强度确定标记效率。
所需附加溶液的制备请找出下表中的成分和所需制备的试剂。
下表中的缓冲液也可以在地高辛洗涤和封阻缓冲液无DNA酶和RNA酶系列产品中获得。
溶液成分/制备储存/稳定性用途洗涤缓冲液0.1M马来酸0.15M NaClpH 7.5(20℃)0.3% (v/v)Tween20 15-25℃,稳定去除未结合的抗体马来酸缓冲液0.1M马来酸0.15M NaCl用NaOH(固体)调节pH值到7.5(20℃)15-25℃,稳定封阻液的稀释检测缓冲液0.1M Tris-HCl0.1M NaClpH 9.5(20℃)15-25℃,稳定调整pH值到9.5TE缓冲液10mM Tris-HCl1 mM EDTApH 8.015-25℃,稳定终止颜色反应试剂盒工作溶液的制备下表中列出试剂盒工作溶液的制备方法:溶液成分/制备方法储存/稳定性用途封阻溶液用马来酸缓冲液按照1:10的比例将10×封阻液(10号瓶按体积比10%溶解)稀释成1×工作溶液一直新鲜制备封阻膜上非特异结合位点抗体溶液每次使用前将原始管中的anti-digoxigenin-AP(8号管)10000rpm离心5分钟。
