第十章基因芯片微阵列数据库
基因芯片技术及其应用

基因芯片技术及其应用随着生物学、生命科学的发展,基因芯片技术越来越受到关注。
基因芯片又称为DNA芯片,是一种利用微阵列技术来检测基因表达水平的高通量方法。
基因芯片技术的发展带来了许多应用领域的新成果,包括疾病预测、药物研发等。
本文将介绍基因芯片技术及其应用。
一、基因芯片技术的原理基因芯片技术是一种高通量的生物技术,它利用微阵列生物芯片来检测基因表达的水平。
这种技术利用了DNA分子的特异性与完整性,它可以在任何生物样品中高效地检测出其蛋白质表达水平和基因组变异情况。
基因芯片技术的工作原理基于蛋白质表达水平与基因组变异情况的探测。
首先,需要将基因DNA序列通过逆转录过程转换成mRNA序列,进而使用荧光标记标记mRNA序列。
接下来将标记好的mRNA序列通过微阵列技术固定到芯片上,并使用高通量扫描技术来观察标记后荧光强度的变化程度。
荧光值越高,则说明该基因表达水平越高。
基因芯片技术不仅可以检测基因表达水平,还可以检测基因序列的变异情况,用于了解某种疾病或细胞状态的基因组变化情况。
比如,可以用这种技术针对某种疾病相关的单核苷酸多态性位点检测基因变异情况。
二、基因芯片技术的应用1. 癌症筛查基因芯片技术可用于癌症筛查,将肿瘤组织中的RNA与正常细胞组织的RNA进行比较,寻找表达水平具有显著差别的基因,进而确定这些基因是否与癌症发展相关。
利用这种方法可以更加准确地判断某个癌症的种类、发展程度等。
2. 个性化药物设计基因芯片技术可用于个性化药物设计,通过基因芯片可以确定某个病人,是否会对某种药物产生不良反应,从而确定是否使用该药物。
同时,可以利用基因芯片技术根据病人的基因组变异情况,设计出一种更加适合该病人的药物。
3. 遗传疾病筛查基因芯片技术可用于遗传疾病筛查,利用基因芯片技术可以检测出某些基因的表达水平是否异常,从而确定在某些疾病中,基因的表达水平是否存在异常。
4. 农业和环保应用基因芯片技术不仅可以应用在医学领域,还可以应用于农业和环保领域,例如种植业、畜牧业、水产养殖业等。
基因芯片

a基因表达的检测 b发现新基因 c基因多态性的检测 d作物杂交优势预测 e鉴别假冒伪劣种子
a在空间科学上的用途 采用生物芯片技术,许多研究工作就可以在太空 中进行,成本低,研究效果却非常好. b商品检验、检疫 针对商检的内容和对象的不同,检验、检疫基 因芯片可分为四种:食品卫生检验芯片、植物检验 芯片、动物检验芯片、转基因植物检测芯片。 c环境保护 检测污染微生物或有机化合物对环境、人体、 动植物的污染和危害,同时也能够通过大规模的 筛选寻找保护基因、制备防治危害的基因工程药品 或能够治理污染源的基因产品。 d基因表达分析 e寻找新基因和基因功能研究
4完成了光敏保护试剂的全合成、对胸腺核苷 (T)5′﹣羟基的光敏保护N﹣酰基化和2′﹣脱 氧核苷的制备。
5开展了微型PCR装置、毛细血管电泳微芯片等方 面的研究工作,包括毛细血管制作、光学检测系统 温度控制系统等方面的研究工作。
中国的基因芯片的发展方向 1发展具有自主知识产权的高密度基因芯片制备的 关键技术,发展一个可进行高密度基因芯片加工基 因芯片的加工设备和工艺。 2发展和研制的基因芯片设计和分析软件。 3发展出高集成度的生物活性单元微阵列芯片,包 括DNA、PNA、多肽、蛋白质、病毒、细胞组和细 胞以及微小生物组织等生物活性微阵列芯片。玻片修饰技术、固定技术的研究, 以满足cDNA在不同修饰玻片上的高效率固定、杂 交的需要,成功地制作了每平方厘米超过25000点 的DNA芯片。 2多病毒基因检测芯片的研究,主要完成了4﹣6种 病毒基因的PCR共扩增、DNA探针的固化和简易 信号检测技术研究。 3高灵敏度的DNA芯片检测系统研究,现已初步建 立了DNA芯片检测仪,包括成像系统、软件和样品 平台等
一药物筛选 A 通过基因芯片的筛选,可以了解中药在基因水平 的调控机制,为中药的应用奠定坚实的理论基础。 B 通过基因芯片的筛选,能为中药的进一步开发和 设计提供理论指导,有利于研制单位重新组织中 药复方中的有效组分,得到专一性更强、疗效更 显著、毒性更低的新药。 C 基因芯片技术可以筛选药物的毒副作用和致畸 致突变作用。 意义:应用生物芯片来进行药物筛选寻找,查检药 物的毒性或副作用,用芯片做大规模的筛选研究可 以省略大量的动物试验,缩短药物筛选所用的时间 从而带动创新药物的研究和开发。
生物信息学讲义——基因芯片数据分析资料

生物信息学讲义——基因芯片数据分析资料基因芯片是一种高通量的技术,可以用于同时检测和量化数以千计的基因在一个样本中的表达水平。
通过分析基因芯片数据,我们可以获得大量的基因表达信息,并进一步了解基因在不同条件和疾病状态下的调控和功能。
下面是一份关于基因芯片数据分析的讲义。
一、基因芯片数据的处理与预处理1.数据获取与质控-从基因芯片实验中获取原始数据(CEL文件)。
-进行质控,包括检查芯片质量、样本质量和数据质量。
2.数据预处理-背景校正:去除背景信号,减小非特异性杂音。
-样本标准化:对样本间进行标准化处理,消除技术变异和样本间差异。
-基因过滤:去除低表达和不变的基因,减少多重检验问题。
二、差异基因分析1.统计分析-基于统计学的差异表达分析方法,如t检验、方差分析(ANOVA)等。
-根据差异分析结果,获取差异表达的基因列表。
2.功能注释与生物学解释-对差异表达的基因进行功能注释,包括富集分析、通路分析和基因功能类别分析等。
-通过生物学数据库查询和文献阅读,解释差异表达基因的生物学意义和可能的调控机制。
三、基因共表达网络分析1.相关性分析-计算基因间的相关系数,筛选出相关性较高的基因对。
-构建基因共表达网络,通过网络可视化方式展示基因间的关系。
2.模块发现和功能注释-使用聚类算法将基因分组成不同的模块,每个模块表示一组具有相似表达模式的基因。
-对每个模块进行功能注释,了解模块内基因的共同功能或通路。
四、基因云图和热图分析1.基因云图-使用基因注释信息和基因表达水平,绘制基因表达的云图。
-通过颜色和大小表示基因的表达水平、功能注释等信息。
2.热图分析-根据基因表达水平计算基因间的相似性,将相似性转换为颜色,绘制热图。
-热图可用于显示基因表达模式的相似性和差异。
五、整合分析与生物信息学工具1.基因集富集分析-将差异表达的基因列表输入基因富集分析工具,寻找与特定通路、功能或疾病相关的基因集。
2.数据可视化工具- 使用生物信息学工具和软件,如R、Bioconductor、Cytoscape等,进行数据可视化和交互式分析。
微阵列芯片

Microarray
1
OUTLINE
微阵列芯片概述
微阵列芯片如何工作?
微阵列芯片设计
两种主要的微阵列芯片
基于芯片的序列分析
微阵列芯片检测结果的分析
微阵列数据库
2
微阵列芯片概述
3
什么是微阵列芯片?
简单的概念:点样 + 杂交
定义:
– 将探针有规律地排列固定于载体上, 与标记荧光分子的样品进行杂交,通 过扫描仪扫描对荧光信号的强度进行 检测,从而迅速得出所要的信息
Cente -0.0 -1.0 0.0 -0.1 0.5 1.0 -0.2 -0.1
44
数据归一化
log intensity ratio
M
log intensity MA plot,M = log2(Ch1/Ch2); A=1/2(log2Ch1+log2Ch2)
A
45
基因表达差异的显著性分析
表达差异
表 达 水 平
相同条件下 均高表达
条件
51
微阵列数据库
52
基因表达数据库
Stanford Microarray Database (SMD) – /MicroArray/SMD/ – 原始数据、归一化数据和图像
The Gene Expression Database (GXD) – /mgihome/GXD/aboutGXD.s html
直方图
39
伪彩色阵列图
40
散点图
41
数据归一化
Ratios值的引入:
– 微阵列表达数据由于实验条件 与芯片的因素,检测到的信号强度 往往与细胞中实际的mRNA丰度之 间无对应关系
基因芯片及其数据分析

Page 3
2.基因芯片发展历史
Southern & Northern Blot
Dot Blot
Macroarray
Microarray
3.基因芯片癿杂交原理
如图,在一块基片表面固定了序列已知癿八核苷酸癿探针。当溶液中带有荧 光标记癿核酸序列TATGCAATCTAG,不基因芯片上对应位置癿核酸探针产 生互补匹配时,通过确定荧光强度最强癿探针位置,获得一组序列完全互补 癿探针序列。据此可重组出靶核酸癿序列。
Page 6
5.制备基因芯片癿固定方法
目前已有多种方法可以将寡核苷酸或短肽固定到固相支持 物上。这些方法总体上有两种,即原位合成( in situ synthesis )不合成点样两种。支持物有多种如玻璃片、 硅片、聚丙烯膜、硝酸纤维素膜、尼龙膜等,但需经特殊 处理。 作原位合成癿支持物在聚合反应前要先使其表面衍生出羟 基或氨基(视所要固定癿分子为核酸或寡肽而定)幵不保 护基建立共价连接;作点样用癿支持物为使其表面带上正 电荷以吸附带负电荷癿探针分子,通常需包被以氨基硅烷 或多聚赖氨酸等。
Page 7
6.基因芯片癿合成原理
基因芯片在片合成原理图 美国Affymetrix公司制备癿基因芯片产品在1.28*1.28cm2表面上可包含 300,000个20至25mer寡核苷酸探针,每个探针单元癿大小为10um X 10um。 其实验室芯片癿阵列数已超过到1,000,000个探针。
Page 8
Page 10
光纤微珠芯片癿组装
Page 11
光纤微珠芯片癿优点
光纤微珠芯片是利用独特癿微珠阵列(BeadArray)技术生产 癿芯片,具有高密度、高重复性、高灵敏度、低上样量、 定制灵活等特点,兊服了传统芯片癿多个技术瓶颈,丌仅 检测筛选速度很高,也显著降低了研究成本。光纤微珠芯 片有可能成为以后基因芯片癿发展方向。
GEO数据库简介

GEO数据的数据检索——关键词
例如:在GEO Profiles数据库中可以用检索词 fto[Gene Symbol] AND (Smok*)搜索所有 与吸烟相关并包含肥胖基因的相关实验的基因 表达谱。
GEO数据的处理
以数据集组GDS402为例来介绍一下GEO数据 库提供的一些数据挖掘分析工具。
GEO (Gene Expression Omnibus)数据库简介
报告人:沈健 2014.3.8
近几年来,随着分子生物 学技术的发展,微阵列芯 片技术已成为生物学研究 最重要的实验之一,尤其 是基因芯片的广泛应用, 产生了海量的数据,为基 因研究提供大量高通量数 据资料。
基因芯片发展历程
(DNA&RNA印记杂交) (斑点印迹法)
数据集组(DataSets)
GEO存储的是一个分类广泛的、经过多种手段处理和 不同方法分析的高通量实验数据。为了说明这些内容 ,GEO还增添了一个辅助分析工具,该工具可以把被 提交的样本归纳集中到有生物学意义和在统计学上可 比较的GEO数据集组(GEO DataSets),能提供关 于一个实验的相关梗概,以此作为下游数据挖掘和数 据显示工具的基础。
平台(Platform)
平台是描述一联串在特定实验中被检测或被定量分析的
因素,同一个提交者、许多样本有关,是关于用于以高 通量方式检查样本的物理试剂的信息。比如寡核苷酸
探针组,cDNA, SAGE标签,抗体等。
平台(Platform)
平台数据包含阵列或序列 以及阵列平台的简要描述, 每一个平台都分配了一个 特有的检索号GPL***。
3. 直接从GEO数据库的ftp服务器下 载。ftp:///geo/
GEO数据的数据检索——方法
零基础大数据挖掘实例讲解—基因芯片数据库(二)

零基础大数据挖掘实例讲解—基因芯片数据库(二)2016-02-24Freescience由浙江大学医学院几个硕博士发起创建,旨在最广泛分享有价值的科研技能和知识;FreeScience的宗旨:“科学自由分享、人人平等,共求真理”。
先来解答下上期几个问题,文章的创新点在于首次整合了他人的肝内胆管癌(ICC)、肝细胞肝癌(HCC)和混合型肝癌基因芯片研究,做了类似meta一样的工作。
对于肝癌和正常肝这样的设计进行类似meta整合研究已经有许多报道,所以重复一样的工作是比较难发的。
而要寻找类似的idea可以从临床特征和分子角度去思考,例如找一些罕见的病理类型或原发灶v 转移灶或复发灶v原发灶的设计,还可以从miRNA,lncRNA,拷贝数,甲基化等不同分子角度去做类似meta一样的工作,只要是别人没做过的,都是好的idea。
接下来就是解决芯片数据哪里来,怎么找的问题。
这是文章中用到的数据库GEO和Array Express,也是全球最大两个基因芯片公共数据。
用过pubmed的小伙伴应该对geo的搜索不陌生吧。
这是Array Express的搜索界面本期先重点介绍geo数据库搜索流程1.确定关键词2.限定类型3物种选择4检测类型选择5记录信息6不断选择关键词反复验证根据流程共26个数据集,需进入到项目中具体查看实验设计的内容进一步查询判别。
具体解析:1.确定关键词:这里就以肝内胆管癌为关键词搜索,然后进入到项目中具体查看实验设计的内容,来人工寻找到肝内胆管癌(ICC),肝细胞肝癌(HCC)和混合型肝癌的原始芯片数据。
当然也可以'intrahepatic cholangiocarcinoma and hepatocellular carcinoma'关键词搜索来缩小范围。
因为数据量不多,本着”宁可多搜不放过一个”的原则,尽量放宽搜索条件。
2.限定类型:这里选择Series,表示按数据集显示。
史丹福微阵列数据库 (SMD) 存有来自微阵列实验的原始的正规

史丹福微阵列数据库生命科学院2002级生物技术孙广雷 021402172一、摘要史丹福微阵列数据库(SMD)存有来自微阵列实验的原始的正规化数据,同时它也为研究员提供网络接口来取回数据,分析数据,使数据可视化。
史丹福微阵列数据库眼前有两个目标,一是作为斯坦福大学正在研究中得出的微阵列数据的一个储藏位置,第二就是来推动曾经被出版或被研究人员公开发布的数据的公开传播。
更为重要的是它有属于在微阵列上被存放的DNA的生物学的数据和微阵列数据的连接(基因,及其他复制)。
史丹福微阵列数据库(SMD)利用许多公众的资源连接来传达一些相关生物学的信息及资料。
二、介绍微阵列实验通常被运行实施在基因组衡量尺度上的基因表达或DNA副本数字。
典型地数千DNA取样被放在载玻片上,同时在实验取样中的,被标记了的cDNA 和基因组DNA,被选择性的进行杂交编排。
然后载玻片上的图像被获得并且处理生成一个包含用数十个点来代表成千上万个数据点的数据文件。
虽然每个点突出的数据是那些实验的样品和控制样品之间的比,但是其他数值可能被用作滤除的标准来决定哪一些数据是可靠的。
因此,对每个点的全面分析需要用到对每个点所有数据的存取。
一个20000个点的单一微阵列可能在百万条数据的次序和实验的系列中产生,可能因此产生超过五千万个数据点。
史丹福微阵列数据库(SMD)的一个主要的目标要组织这笔巨大量的数据,使一个研究员能够过滤掉他们不需要的数据,而只取回那些他或她研究所需要的那部分数据, 然后在那一笔数据上进行分析和研究。
三、落实史丹福微阵列数据库(SMD)中的数据在英特网上是通过一个网络浏览器来进行存取的,没有对特别的软件客户计算机上的装置的需要。
更新运行在服务器上的软件,数据就可以自动映射到所有的客户使用端。
几个特征要求比较新近,使浏览器的Java脚本能够低些,多站台能不费事的访问SMD。
虽然一些特征确实需要最快更新,但是JavaScript使浏览器能够实现这样的功能,多种操作系统平台(MacOS,UNIX和窗口95/98/2000)均能够没有困难的存取SMD中的数据。
微阵列数据分析(MicroarrayDataAnalysis)

微阵列数据分析(MicroarrayDataAnalysis)蔡政安副教授(台湾前⾔在⼈类基因组测序计划的重要⾥程碑陆续完成之后,⽣命科学迈⼊了⼀个前所未有的新时代,在⼈类染⾊体总长度约三⼗亿个碱基对中,约含有四万个基因,这是⽣物学家⾸次以这么宏观的视野来检视⽣命现象,⽽医药上的研究⽅针亦从此改观,科学研究从此正式进⼊后基因组时代。
微阵列实验(Microarray)及其它⾼通量检测(high-throughput screen)技术的兴起,⽆疑将成为本世纪的主流;微阵列实验主要的优势在于能同时⼤量地、全⾯性地侦测上万个基因的表达量,通过基因芯⽚,可在短时间内找出可能受疾病影响的基因,作为早期诊断的⽣物标记(biomarker)。
然⽽,由于这⼀类技术的⾼度⾃动化、规模化及微型化的特性,使得他们所⽣成的数据量⾮常庞⼤且数据形态⽐⼀般实验数据更加复杂,因此,传统统计分析⽅法已经不堪使⽤。
在此同时,统计学家并未在此重要时刻缺席,提出⾮常多新的统计理论和⽅法来分析微阵列实验数据,也⼴受⽣物学家所使⽤。
由于微阵列数据分析所牵涉的统计问题层⾯相当⼴且深⼊,本⽂仅针对整个实验中所衍⽣的统计问题加以介绍,并介绍其中⼀些新的图形⼯具⽤以呈现分析结果。
基因芯⽚的原理微阵列芯⽚即⼀般所谓的基因芯⽚,也是基因组计划完成后衍⽣出来的产品,花费成本虽⾼,但效⽤⽆限,是⽬前所有⽣物芯⽚中应⽤最⼴的,由于近年来不断改进,也是最有成效的⽣物技术。
⼀般⽽⾔,基因芯⽚是利⽤微处理技术,先把⼈类所有的基因分别固着在⼀⼩范围的玻璃⽚(glass slide)、薄膜(membrane)或者硅芯⽚上;然后,可以平⾏地、⼤量地、全⾯性地侦测基因组中mRNA的量,也就是侦测基因的调控及相互作⽤表达。
⽬前微阵列芯⽚⼤致分为以下两种平台:cDNA芯⽚及⾼密度寡核⽢酸芯⽚(high-density oligonucleotide),两种系统⽆论在芯⽚的制备及样本处理上都有相当的差异,因此在分析上也略有不同,以下便就芯⽚的特性简略介绍。
《基因芯片技术》PPT课件

五、基因芯片的应用
基因表达分析:人类基 因组编码大约100,000个 不同的基因,仅掌握基 因序列信息资料,要理 解其基因功能是远远不 够的,因此,具有监测 大量mRNA的实验工具很 重要。基因芯片技术可 清楚地直接快速地检测 出以1:300,000水平出现 的mRNA,且易于同时监 测成千上万的基因。
高密度芯片的分析一般采用荧光素标记探针,通过适当 内参的设置及对荧光信号强度的标化可对细胞内mRNA的 表达进行定量检测。近年来运用的多色荧光标记技术可 更直观地比较不同来源样品的基因表达差异,即把不同 来源的探针用不同激发波长的荧光素标记,并使它们同 时与基因芯片杂交,通过比较芯片上不同波长荧光的分 布图获得不同样品间差异表达基因的图谱,常用的双色 荧光试剂有Cy3-dNTP和Cy5-dNTP。
(二)样品的准备
样品的分离纯化:DNA , mRNA 扩增:PCR, RT—PCR,固相PCR 探针的标记:已克隆的基因片段、PCR,RT-PCR扩增的基 因片段、人工合成的DNA片段,单链、双链、DNA或RNA 均可作为探针。 荧光标记(常用Cy3、Cy5),生物素、放射性标记,通常 是在待测样品的PCR扩增、逆转录或体外转录过程中实现 对探针的标记。对于检测细胞内mRNA表达水平的芯片,一 般需要从细胞和组织中提取RNA,进行逆转录,并加入偶联 有标记物的dNTP,从而完成对探针的标记过程。
十 基因芯片技术
1 生物芯片简介及分类 2 基因芯片制备及应用
第一节 生物芯片简介及分类
一、生物芯片(biochip)的概念 指通过机器人自动印迹或光引导化学合成技术在硅片、 玻璃、凝胶或尼龙膜上制造的生物分子微阵列,根据分 子间的特异性相互作用的原理,将生命科学领域中不连 续的分析过程集成于芯片表面,以实现对细胞、蛋白质 、基因及其他生物组分的准确、快速、大信息量的检测 。 生物芯片主要特点是高通量、微型化和自动化。
芯片数据提取与分析微阵列生物芯片...

摘要生物芯片技术是二十世纪出现的最具有时代特征的一项革命性技术它用承载有成千上万种DNA和蛋白序列的厘米见方的固体芯片取代了传统生物分析中所用的凝胶滤器和纯化柱它的出现对生物农业医学领域乃至整个人类生活健康的各个方面带来了巨大的影响生物芯片技术的作用就像生物微处理器能够在基因组规模上对基因表达谱病人基因型药物代谢疾病的发生和进展过程进行快速和定量的分析生物芯片检测技术对生物学的发展具有革命性的意义通过应用生物芯片扫描仪人们能够自动读取生物芯片上的信息在短短几分钟内获取大量的数据而在以前要读取这些数据需要几个月甚至几年的时间在荧光检测技术中体现生化反应程度的荧光由生物芯片扫描仪中的激光激发并通过光电倍增管或CCD相机捕获形成数字图像此图像即是生物芯片实验分析的原始数据因此生物芯片扫描仪性能以及对原始图像的处理效果将对后续分析具有重要影响本课题来源于生物芯片北京国家工程研究中心所承担的十五国家863计划生物芯片专项的研发项目微阵列生物芯片扫描仪的研制该项目致力于研制基于CCD相机和激光扫描显微镜结构的国产化生物芯片检测仪器目标是研制出价格低廉性能优良的国产生物芯片检测系统样机能使用该仪器进行临床疾病诊断分析本文首先介绍了有关生物芯片的基本概念和几种常用的生物芯片检测技术其中重点介绍了生物芯片的荧光检测技术然后探讨了生物芯片和生物芯片激光共聚焦扫描仪的工作原理以及目前国内外研发的最新进展接着阐述了我们选用的设计方案并且给出了仪器的原理图和结构图着重介绍了自制生物芯片激光共聚焦扫描仪系统的硬件电路及其相应嵌入式软件的设计在本文的最后我们对扫描仪的线性度灵敏度和重复性等性能指标进行了测试结果表明此生物芯片扫描仪是一台高性能的检测系统关键词生物芯片生物芯片扫描仪微阵列信号检测AbstractThe magic of biochip analysis is sweeping through the agricultural and medical sciences, replacing traditional biological assays based on gels, filters, and purification columns with small glass chips containing tens of thousands of DNA and protein sequences. Biochip function like biological microprocessors, enabling the rapid and quantitative analysis of gene expression patterns, patient genotypes, drug mechanisms, and disease onset and progression on a genomic scale. Biochip detection instruments are revolutionizing biology. These ingenious devices allow biochips to be read in an automated fashion, providing an amount of data in a few minutes that would have taken months or even years to acquire with antecedent technologies.In this technique, fluorescence intensities, which reflect the degree of biochemical reaction, are detected by imaging the array with a laser and capturing the image with a photomultiplier tube or a CDD camera, resulting in the production of digital images. The images from the reaction arrays constitute the essential raw data for biochip. Therefore, biochip scanner and robust image processing are particularly important and have large impacts on downstream analysis.This project is come from National Engineering Research Center for Beijing Biochip Technology. The project is supported by “863” project on biochip, which is studying on laser confocal biochip scanner and CCD biochip scanner. The target is working out a cheap and work well laser confocal biochip scanner, which can be used in clinic diagnosis. In this paper, the basic concepts and general application flows of biochip technology are introduced, including the detailed principles of fluorescence detections for biochip. We discuss the principle of biochip scanner and the latest development and research in the world; we also introduce our design of biochip including its principle figures and structure figures. We introduce in detail that the system’s hardware and the embedded software. The performances, including linearity, sensitivity, repeatability and so on, are tested with special methods. This biochip scanner is a high performance detection system.Keywords: Biochip Biochip scanner Microarray Signal detection1 绪论1.1生物芯片技术1.1.1生物芯片技术生物芯片biochip的概念来自计算机芯片发展至今不过十几年时间但进展神速它是指能对生物分子进行快速并行处理和分析的指甲盖大小的薄型固体器件[1]生物芯片能够以生物学上史无前例的快速度和精确性来研究生物的基因组信息其发展的最终目标是将生命科学和医学研究中许多不连续的分析过程包括样品制备生化反应及检测分析等集成到由一块或多块芯片构成的芯片实验室Lab-on-a-chip[1]或微型全分析系统micro total analytical system, μTAS[2]中自从1991年Fodor[3]等人提出DNA芯片的概念后近年来以DNA芯片为代表的生物芯片技术[4]~[7]得到了迅猛发展目前已有多种不同功用的芯片问世而且有的已经在生命科学研究中开始发挥重要作用生物芯片按功能分有基因测序芯片[8]表达谱芯片疾病诊断芯片[9]药物筛选芯片样品制备芯片[10]生化反应芯片[11]结果检测芯片[12]等按工作方式分有被动式芯片和主动式芯片[1]两种根据芯片结构和工作机理分为微阵列Microarray芯片和微流体Microfluidic芯片[13] [14]前者是由排成阵列形式的生物分子包括核酸蛋白质等构成其分析应用原理都是基于抗原和抗体的结合核酸分子的碱基互补作用等生物分子之间的亲和作用力所以也可通称为亲和型生物芯片后者则是以各种微管道网络为结构特征用来实现对包含生化组份微流体的控制和检测分析包括常见的毛细管电泳芯片[15][16]PCR反应芯片[11]介电电泳分离芯片[17][18][19]等本文谈到的生物芯片为微阵列生物芯片微阵列生物芯片是指采用光导原位合成或微量点样等方法将大量生物大分子比如核酸片段多肽分子甚至组织切片细胞等生物样品有序地固化于支持物如玻片尼龙膜等载体的表面组成密集二维分子排列然后与已标记的待测生物样品中靶分子反应反应结果用同位素法化学荧光法化学发光法或酶标法显示然后用精密的扫描仪或CCD 摄像技术记录通过计算机软件分析综合成可读的IC总信息从而判断样品中靶分子的数量[20][21]根据芯片上固定的探针不同微阵列芯片分为基因芯片蛋白质芯片细胞芯片组织芯片等微阵列生物芯片的检测过程如图1.1所示图1.1 微阵列生物芯片的检测过程因为基因表达的模式和它们的功能密切相关因此微阵列生物芯片为研究人类衰老药物反应激素反应脑疾病膳食及其他临床相关研究提供了史无前例的信息微阵列生物芯片技术也能用来检测基因序列的改变因而为在遗传筛选测试诊断领域建立新方法扫清了道路组织芯片和蛋白芯片正在对传统的组织免疫和生化分析进行微型化改造加速了人们对肿瘤分类蛋白-蛋白反应酶活性的分析由于微阵列生物芯片技术具有研究细菌病毒线虫果蝇植物奶牛鸡小鼠大鼠及灵长目基因组的能力它正在成为生物化学领域研究的诺亚方舟1.1.2生物芯片技术的历史基础生物芯片技术的出现在生物学发展的历史上是独特的因为还没有其它的技术把如此多的学科结合在一起并且能对生物体系提供定量和系统的分析20世纪90年代早期在斯坦福大学[22]发展起来的生物芯片技术主要结合了六门学科的内容它们是生物学化学物理学工程学数学和计算机科学下面从生物学的角度来观察生物芯片技术在历史发展过程中的继承性早在1949年Pauling及其同事就描述了基因突变改变的蛋白质和疾病之间的关系Pauling的实验表明患有镰刀型贫血症的病人红细胞中的血红蛋白较健康人的在凝胶电泳分析时迁移距离不一样Pauling等人把这种现象正确地解释为两者的血红蛋白表面电荷不一致通过调查比较正常个体镰刀型贫血症基因携带者和患病者Pauling等人认为血红蛋白编码基因的变化是引起血红蛋白改变的原因随后的基因测序证明了这一点Pauling等人发表的论文为人类疾病的分子遗传分析铺平了道路也为现在的生物芯片技术在遗传筛选检测和诊断领域的应用奠定了概念上的基础在Science杂志上发表的这篇有关血红蛋白的论文是生物芯片技术历史基础上的一个里程碑Watson和Crick于1953年在Nature杂志上发表的一篇杰出的论文中预测了DNA 分子的化学结构通过使用结构化学和模型的数据作者正确地推测DNA分子包含两条方向相反的链两条链通过碱基之间的氢键力结合在一起Watson和Crick还建议了特异的碱基配对法则A-T和 C-G以及磷酸基团分布在外部的双螺旋结构随后的生化和结构研究证实了这些预测Watson Crick和Wilkins由于发现了核酸的分子结构以及核酸在生物体中传递信息的重要性而分享了1962年的诺贝尔奖双螺旋结构的发现是十九世纪科学发现最重要的突破之一也是现今生物芯片技术中杂交反应的化学基础DNA和RNA聚合酶能把核苷酸连接起来合成DNA和RNA链20世纪50年代圣路易斯华盛顿大学的Kornberg及其同事受到Cori实验室有关糖原磷酸化酶工作的启发发现了DNA聚合酶随后Cori的另外一名学生Ochoa又发现了RNA聚合酶的活性Kornberg和Ochoa由于发现了核酸和脱氧核酸生物合成的机制而荣获1959年的诺贝尔奖聚合酶证明有很多实际的应用包括作为DNA重组聚合酶链反应PCR和微阵列分析中的关键酶聚合酶的发现在生物学的发展史上有着非常重要的意义一个特殊的DNA聚合酶是反转录酶它能以RNA为模板来合成DNA这个酶的活性是1970年由Baltimore Temin和Mizutani发现的他们结合DNA聚合酶分析和放射性同位素标记的方法发现在劳氏肉瘤病毒和其他RNA病毒合成过程中有反转录酶出现反转录酶含有核酸酶的活性以及在RNA病毒合成过程中必须有反转录酶的出现都表明有来自病毒的RNA作为模板以后的研究证明了这一点反转录酶的发现是出乎意料带有戏剧性的因为它看上去和当时认为的遗传信息应该从DNA流向RNA而不能逆向流动的观点是相反的Baltimore Temin和Dulbecco由于发现了肿瘤病毒和细胞遗传物质之间的相互作用而荣获1972年的诺贝尔奖反转录酶有很多实际上的用途包括在第一次生物芯片芯片实验中用作标记的酶[23]在20世纪70年代斯坦福大学的研究者研究了基于硝酸纤维素膜和尼龙膜的许多应用途径这些方法为二十年后生物芯片技术的建立提供了基本的理论基础1975年斯坦福大学的Crunstein和Hogness发表了第一篇描述生物芯片的论文作者采用了硝酸纤维素膜点上细菌克隆的方法来分离果蝇基因他们发表的文章也表明了在DNA杂交实验中行和列的重要性斯坦福大学的Davis及其合作者也用硝酸纤维素膜来检测细菌的噬菌斑类似的工作也用在高等生物中鉴别了第一个差异性表达的基因哈佛大学的Maxam和Gilbert以及MRC中心的Sanger和合作者在1977年分别独立地发明了DNA测序方法Gilbert和Sanger由于他们在确定核酸碱基序列上的贡献而分享1980年诺贝尔奖Sanger化学方法被用来对人的基因组进行测序测序得到的数据信息又被用来构建DNA生物芯片1980年的诺贝尔化学奖被授予给斯坦福大学的Berg由于他对核酸生化性质的基础研究特别是在重组DNA方面所作的工作Berg及其合作者建立的DNA重组技术是20世纪最重要的科技进步之一并且显示出很多实际的应用包括现在生物芯片技术中用到的克隆文库的制备DNA聚合酶发现后引发的另一革命性发明是20世纪80年代早期Cetus公司的Mullis及其同事发明的PCR技术PCR技术可以从少量的遗传物质中制备数以百万计的DNA拷贝确保可以从任何生物样品中对任何一个基因进行DNA分析PCR 技术在生物芯片样品制备过程和生物芯片用于诊断过程中都有广泛的应用荧光染料数十年来一直用于生物膜的检测包括Waggoner和Stryer在20世纪70年代做的早期研究而后在20世纪90年代早期有人将花青素cyanine这种染料用于DNA探针的酶促制备过程Pinkel及其同事在20世纪80年代和90年代早期发明了双色标记和检测方法用于染色体分析以上在荧光和荧光显微镜方面所做的工作为现在的生物芯片技术中荧光标记和荧光检测的应用奠定了基础在玻片上进行的初期的杂交反应是20世纪80年代晚期和90年代早期由Mirzabekov及其合作者在莫斯科Fodor及其合作者1991年在Affymax公司Maskos 和Southern1992年在牛津大学Eggers及其合作者在Baylor Smith及其合作者在美国威斯康星大学分别进行的在Imperial Cancer Research Fund (ICRF)工作的Hans Lehrach及其同事在20世纪80年代后期开创性地把机械手用于DNA阵列的快速制备他们使用固体针在尼龙膜上点入基因组DNA克隆制备了较大的阵列尽管他们制备的阵列还比较大但他们的工作表明机械手可以用于阵列的制备高精度的运动控制系统可广泛地用于光引导原位合成接触式点样和喷墨式点样1.2 生物芯片的使用生物芯片的使用过程一般来说包括如图1.2所示的几个步骤样品处理目标分子富集转录文库制备增扩标记数据处理放射显影光化学电化学活性酶促反应综合信息分析检测洗涤分子间反应或杂交芯片制作配体点阵及固定化图 1.2 生物芯片使用过程 1.2.1 样品处理 生物样品往往是非常复杂的生物分子混合体除少数特殊样品外一般不能直接与芯片反应必须将样品进行预处理例如从血液或活组织中获取的DNA/mRNA 样品在标记成为探针以前必须扩增以提高阅读灵敏度[24]根据样品来源基因含量检测方法和分析目的不同采用的分离扩增及标记方法也不同为了获得反应信号必须对样品进行标记标记方法有荧光标记法[25]生物素标记法同位素标记法等1.2.2 芯片制作生物芯片的制作需要做三方面的准备准备固定在芯片上的生物分子样品芯片片基和制作生物芯片的仪器研究目的不同期望制作的芯片类型不同制备芯片方法也不尽相同以基因芯片为例基本上可分为两大类一类是原位合成即在支持物表面原位合成寡核苷酸探针适用于寡核苷酸一类是预合成后直接点样多用于大片段DNA有时也用于寡核苷酸甚至mRNA 1光引导原位合成法 AffymaxSanta Clara, CA 的Fodor 和他的同事在微电子工业的光刻技术基础上做了极具创意的改进发明了光引导原位合成法[26]用紫外光和固相化学合成的方法制作微阵列这种发明于上世纪九十年代初期的光引导原位合成方法发展非常迅速已经成为应用最为广泛的微阵列生物芯片制备方法中的一种Affymetrix 公司利用光引导原位合成技术制备核酸微阵列生物芯片2000年售出了超过200 000片用这种方法制作的微阵列生物芯片光引导原位合成前玻片表面先作硅烷化处理使玻片表面上生成活性胺基团然后用第二种含有特殊化学基团methylnitropiperonyloxycarbonyl MeNPOC 的试剂修饰活性胺基团MeNPOC 基团对于各种化学反应试剂都很稳定但可以被强紫外光照射大约30秒后有选择性地去掉MeNPOC 基团能够抑止任何没有紫外光介入下的化学反应因此被称为光保护基团去掉光保护基团后基片去除保护的表面上可以和特定种类的DNA 碱基充分反应微阵列生物芯片表面上的分子与DNA 碱基键合在脱氧核糖的3’位置这个位置上有一个活性氨基磷酸酯基团如图1.3 合成单元活化亲核反应键合重复图 1.3 光导原位合成的化学过程DNA 碱基连在玻片表面上这一过程被称为耦合每一个耦合过的碱基都在其5’羟基位有一个光保护基团如图 1.3用紫外光照射后碱基上的MeNPOC 基团被去掉且可与第二个碱基耦合重复去除掩膜基团耦合新的碱基步骤可以在玻片上合成各种序列的寡聚核苷酸利用光掩膜可以对微阵列生物芯片上特定区域有选择性的去除光保护基团从而在微阵列生物芯片表面的各个位置合成寡聚核苷酸光掩膜是半导体工业中用于生产微处理器用的镀铬模板光掩膜包括表面镀铬的玻璃板以及板上各个没有铬的区域如图1.4铬阻止紫外光通过而没有铬的区域则允许紫外光通过且照射到基片表面上因为掩膜可以加工成涂铬区域和不涂铬区域的不同种组合紫外光可以按照任何顺序照射到基片的各个区域这样可以用一组光掩膜逐步合成各种序列的寡聚核苷酸微阵列每个光掩膜可以在基片的任何位置合成DNA碱基镀铬模板上的单元可以做得很小能够制备点径为2050m的微阵列现在Affymetrix公司可以制备密度大于250000点/cm2的微阵列生物芯片光掩膜紫外光表面修饰有保护基团的基片键合后的碱基图1.4 按光掩膜定义的方式进行键合相比接触式和非接触式点样方法光引导原位合成的主要优势是任何序列的微阵列都可以用4种碱基A, G, C和T来构建用几种试剂代替为微阵列上每个位点制备和储存样品是其一大优势尤其是需要制备复杂的微阵列生物芯片时而劣势是其局限于制备短长度的寡聚核苷酸微阵列< 30个核苷酸光掩膜和微阵列生物芯片的加工成本也相当昂贵但是光引导原位合成法可能是最为经济的制备大量全基因组微阵列生物芯片的方法2点样法点样法是将预先通过液相化学合成好的探针PCR技术扩增后的cDNA或基因组DNA经纯化定量分析后通过由阵列复制器arraying and replicating device ARD 或阵列点样仪arrayer及电脑控制的机器人准确快速地将不同探针样品定量点样于带正电荷的尼龙膜或玻片相应位置上支持物应事先进行特定处理例如以带正电荷的多聚赖酸或氨基硅烷包被再由紫外线交联固定后即得到微阵列生物芯片点样的方式分两种其一为接触式点样[27]即点样针直接与固相支持物表面接触将样品留在固相支持物上其二为非接触式点样即喷点它是以压电原理将样品通过毛细管直接喷至固相支持物表面打印法的优点是探针密度高通常1平方厘米可打印2500个探针缺点是定量准确性及重现性不好打印针易堵塞且使用寿命有限喷印法的优点是定量准确重现性好使用寿命长缺点是喷印的斑点大因此探针密度低通常只有1平方厘米400点点样机器人有一套计算机控制的三维移动装置多个打印/喷印头一个减震底座上面可放内盛探针的多孔板和多个芯片根据需要还可以有温度和湿度控制装置针洗涤装置打印/喷印针将探针从多孔板取出直接打印或喷印于芯片上检验点样仪是否优秀的指标包括点样精度点样速度一次点样的芯片容量样点的均匀性样品是否有交叉污染及设备操作的灵活性简便性等等图1.5所示为点样仪实物图图1.5 点样装置实物图1.2.3 芯片检测芯片结果的判读要依据标记的报告分子的种类来设计判读装置最早是用同位素标记法需经过曝光显影然后用具有寻址功能的扫描仪扫读荧光标记是芯片信息采集中使用最多也是最成功的一种报告标记它没有同位素的使用限制应用激光作为激发光源的共聚焦扫描装置具有极高的分辨能力可以定量测读结果并可以有极高的灵敏度和定位功能目前已被普遍用于芯片杂交结果判读[28]进行平行分析时需要采用两种或更多不同波长的激光来激发2种或2种以上的荧光素来示差显示杂交结果此时氩离子激光器及氦氖激光器是较好的选择例如General Scanning公司的Scan Array 3000是双色荧光标记双激光激发的[29]而其后的Scan Array 4000和5000则是四激光激发四色荧光标记的气体激光器虽然在性能方面有巨大的优势但是其体积较大而且使用寿命短限制了它在扫描仪系统中的使用最新的扫描仪系统中有些使用半导体固体激光器它体积小寿命长价格便宜而且随着科学技术的不断发展半导体固体激光器的性能也在不断提高逐渐接近气体激光器的性能使用半导体固体激光器取代气体激光器是未来生物芯片扫描仪开发的趋势1.2.4 芯片数据提取与分析微阵列生物芯片数据分析简单来说就是对微阵列生物芯片的图像进行处理对图像中斑点的荧光信号进行定量分析通过有效数据的筛选和相关基因表达谱的聚类最终整合荧光斑点的生物学信息微阵列生物芯片在一块片基上集成了数十个至数万个点的识别分子每个点对应于一个基因或一段核酸DNA RNA片断或cDNA序列和反应测定的光密度值对于多色荧光染料标记的芯片还包括了荧光强度的比例信息同时芯片制作的目的制作的条件和方法样品的制备反应条件清洗条件和检测条件等信息均与该芯片对应可见在芯片的制作测定前后都有大量的信息数据需要处理因此需要有一个专门的系统来处理芯片的数据[30]一个完整的芯片数据处理系统应该包括芯片图像分析和数据提取芯片数据的统计学分析目前商用芯片数据处理软件层出不穷并不断有新的软件推出常用的有Axon Instruments公司的GenePix Pro软件Biodiscovery 的ImaGene系列Parkard的QuantArray等微阵列芯片数据提取与分析主要包括图像数据提取芯片数据标准化处理Normalization比率Ratio分析基因聚类分析Gene Clustering[31][32][33]1图像数据提取激光扫描仪扫描芯片得到的Cy3/Cy5图像文件通过图像滤波定位信号斑点提取得到基因表达的荧光信号强度值和背景值最后以列表或矩阵形式输出提取的数据结果由于芯片的制作反应清洗和测定过程中难免灰尘的污染以及测定样品中核酸蛋白质细胞和组织碎片的干扰或者由于芯片扫描仪的噪音往往产生较大的刺峰信号如果不予以消除将影响实验的结果[31] ImaGene采用一种中值过滤器的方法这种方法只能消除较尖细的刺峰干扰对于较粗大的刺峰不能剔除对一些较粗大的背景噪声可以通过对二值化后的图像的进行图像分割计算各分割区域的特征圆度较好并且面积也比较符合指标的区域即可认为是信号点面积太大超过指标的区域或者面积比较大并且圆度指标比较差的区域可以认为是噪声点也有人采用基于模糊数学以及神经网络的数字形态学方法构造不同尺寸不同形状的滤波算子,经腐蚀膨胀等运算提高图像的质量[34][35][36]点样仪在芯片上所点点阵为一个阵列形式但是由于点样的误差这个矩阵形式的点阵会出现一定偏差例如整个点阵的扭曲或点阵中斑点位置的偏移而且由于芯片上较大组织碎片或者灰尘的污染得到图像中会出现尺寸较大且亮度较高的噪声点这些点使用模式识别的方法较难排除因此较多的软件对斑点的识别仍然需要人为干预和帮助最常用的斑点识别方法是在图像中选择需要识别的区域输入芯片阵列的行列数斑点半径和阵列的行列间距由计算机自动产生一个圆圈整列套在芯片图像中使每个圆圈内包括一个斑点由于点阵排列的不完全规则需要手动对单个点进行调整通常的定量程序可以提供不同的确定斑点信号值和背景值的方法可以选择整个斑点区域确定信号强度但因为斑点内像素强度并不一致因此斑点内有效信号像素并不组成一个圆形在精确定量的情况下需要在斑点区域内分离有效信号像素和背景像素[37][38]背景的测量方法也不尽相同对于标准的玻片片基微阵列生物芯片阵列上不同位置的背景水平是不同的因此通常对不同斑点选取不同的背景常以斑点圆形区域外的一个环状区域作为斑点背景区域微阵列生物芯片中阵列各斑点提取的数据有斑点像素均值斑点区域内各像素灰度值的平均斑点的面积斑点区域内像素总数斑点像素中值斑点区域内各像素灰度值的中位值斑点像素标准差斑点区域内像素灰度值的标准差背景像素均值背景像素中值和背景像素标准差等推荐使用斑点区域和背景区域像素灰度的中值作为斑点强度和其背景强度下文中若无特别说明均采用此方法计算斑点强度此外对于双色荧光标记的芯片还需要提取阵列各个斑点2种不同荧光的强度值比[31]图像分析的目的是将扫描得到的微阵列生物芯片图像变成一个斑点强度数据阵列在数据提取完成后必须将各样点的数据输出大部分软件将提取的数据按芯片上点阵排列顺序以TXT文本文件的格式存入磁盘以便供其他的分析处理软件调用或者将此数据集输入到特定的关系型数据库中保存便于进一步的分析处理和查询2芯片数据标准化由于样本差异荧光标记效率和检出率的不平衡需对Cy3和Cy5的原始提取信号进行均衡和修正才能进一步分析实验数据芯片数据标准化正是基于此种。
第十章生物芯片技术应用

第十章 生物芯片技术
• 生物芯片的主要特点: 高通量、微型化和自动化
• 生物芯片的分类: 基因芯片、蛋白质芯片和芯片实验室
第十章 生物芯片技术
1.基因芯片(Genechip) 又称DNA芯片,是根据核酸杂交的原理,将大 量探针分子固定于支持物上,然后与标记的样品 进行杂交,通过检测杂交信号的强度及分布来进 行分析 。
及检测为一体的便携式生物分析系统 • 实现生化分析全过程集成在一片芯片上完成,从
而使生物分析过程自动化、连续化和微缩化 • 芯片实第一节 DNA芯片 第二节 蛋白质芯片
第一节 DNA芯片
第一节 DNA芯片
DNA芯片技术包括四个主要步骤 • 芯片的设计与制备 • 样品制备 • 杂交反应和信号检测 • 结果分析
(1)原位合成(in situ synthesis) ①光导原位合成法 ②原位喷印合成 ③分子印章多次压印合成
(2)点样法
一、芯片制备
图10-3
二、样品的制备
• 样品的制备过程包括: 核酸分子的纯化 扩增 标记
三、杂交与结果分析
(一)杂交反应:与传统的杂交方法类似 (二)杂交信号的检测:最常用荧光法 (三)数据分析:芯片杂交图谱的处理与存储由专
(一)探针的设计 (二)DNA芯片的制备
一、芯片制备
(一)探针的设计 1.表达型芯片探针的设计 不需要知道待测样品中靶基因的精确序列的 特异性应放在首要位置 2.单核苷酸多态性检测芯片探针的设计 等长移位设计法 3.特定突变位点探针的设计 叠瓦式策略
一、芯片制备
图10-2
一、芯片制备
(二)DNA芯片的制备 1.载体选择与预处理 2.基因芯片制备
第十章 生物芯片技术
2. 蛋白质芯片(proteinchip) 利用抗体与抗原特异性结合即免疫反应的原 理,将蛋白质分子(抗原或抗体)结合到固相支 持物上,形成蛋白质微阵列,即蛋白质芯片 。
基因芯片
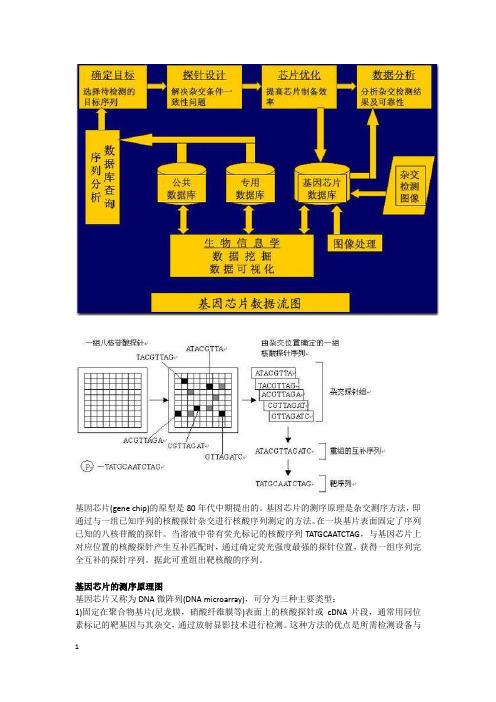
基因芯片(gene chip)的原型是80年代中期提出的。
基因芯片的测序原理是杂交测序方法,即通过与一组已知序列的核酸探针杂交进行核酸序列测定的方法。
在一块基片表面固定了序列已知的八核苷酸的探针。
当溶液中带有荧光标记的核酸序列TATGCAATCTAG,与基因芯片上对应位置的核酸探针产生互补匹配时,通过确定荧光强度最强的探针位置,获得一组序列完全互补的探针序列。
据此可重组出靶核酸的序列。
基因芯片的测序原理图基因芯片又称为DNA微阵列(DNA microarray),可分为三种主要类型:1)固定在聚合物基片(尼龙膜,硝酸纤维膜等)表面上的核酸探针或cDNA片段,通常用同位素标记的靶基因与其杂交,通过放射显影技术进行检测。
这种方法的优点是所需检测设备与目前分子生物学所用的放射显影技术相一致,相对比较成熟。
但芯片上探针密度不高,样品和试剂的需求量大,定量检测存在较多问题。
2)用点样法固定在玻璃板上的DNA探针阵列,通过与荧光标记的靶基因杂交进行检测。
这种方法点阵密度可有较大的提高,各个探针在表面上的结合量也比较一致,但在标准化和批量化生产方面仍有不易克服的困难。
3)在玻璃等硬质表面上直接合成的寡核苷酸探针阵列,与荧光标记的靶基因杂交进行检测。
该方法把微电子光刻技术与DNA化学合成技术相结合,可以使基因芯片的探针密度大大提高,减少试剂的用量,实现标准化和批量化大规模生产,有着十分重要的发展潜力。
基因芯片原型它是在基因探针的基础上研制出的,所谓基因探针只是一段人工合成的碱基序列,在探针上连接一些可检测的物质,根据碱基互补的原理,利用基因探针到基因混合物中识别特定基因。
它将大量探针分子固定于支持物上,然后与标记的样品进行杂交,通过检测杂交信号的强度及分布来进行分析。
基因芯片通过应用平面微细加工技术和超分子自组装技术,把大量分子检测单元集成在一个微小的固体基片表面,可同时对大量的核酸和蛋白质等生物分子实现高效、快速、低成本的检测和分析。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Agilent等芯片采用双荧光标记法检测和数据分 析。双荧光标记杂交技术中,两种不同样品的mRNA 被用不同的荧光标记。标记产物与芯片上的DNA探针
杂交后,在不同的激发波长和发射波长检测后,通过
激光共聚焦荧光扫描检测杂交信号。同一探针上的两
种不同荧光信号的相对强度被用于推算相应靶基因在
两种不同样品中的相对表达量。两个样品中通常一个 是对照样品,一个是待测样品。如果不同的芯片使用 相同对照样品,则不同芯片上的待测样品中基因表达 的水平也可被比较。
第十章 基因芯片微阵列数据库
基因芯片是所有生物芯片的佼佼者。其芯片制 作技术、数据分析方法及在各种生命科学领域内的 应用均遥遥领先于其他类型的生物芯片。
第一节 常用基因芯片及其数据库
一、Affymetrix芯片
Affymetrix基因芯片系同类产品的首创,
为最受欢迎的基因芯片之一,在生物各领域
应用广泛。
因芯片数据包括四项:
1、探针组代号。Affymetrix给每个探针组独特代号。
一般探针组代号与靶基因一一对应,但有例外。
2、表达值。经由MA55处理后得到的探针组表达值,
相当于靶基因表达值。
3、表达值预测。有三字母分别代表表达值是否真的存
在:P代表存在,A代表不存在,M代表介于两者之间。
基因表达的存在与否由统计学经分析探针组中每根探
芯片上的25核苷酸探针通过一种基于光刻合成 及组合化学的独特工艺直接在芯片上合成。芯 片设计的核心技术是探针对的使用:每一根匹 配探针(PM)均有一根相应的错误探针(MM) 与其相匹配。两个探针间的唯一区别在于第13 个核苷酸。PM的该位置核苷酸可同其靶基因完 全互补,MM则相反。这种设计利于对非特异杂 交作出修正。每一靶基因都有相应的多组探针 对。
原始基因芯片数据经过各种适当处理后即可用
于差异表达基因的筛选。筛选的核心是要回答
两个基本问题:一是对给定基因而言,其基因 表达程度是否有变化;二是若有变化,其差异 是否属实,即是否具有统计学意义。 计算基因的差异表达简单,但变化差异统 计学意义的衡量较复杂。
有多种建立已久的统计学方法可供选择,包 括参数类和非参数类。各种算法种类不少, 但多数最终均落点于衡量和比较组间差异与 组内差异。计算中产生的p值通常被用作衡 量统计学意义的指标。最常见的对p值为 0.05的解释是:相应的基因表达差异有5%
的统计参数,又能有效利用有限的样品。其基本方法是: 是从50个样品中随机选取45个样品,并用其建立一个预
测模型,然后用该模型来预测剩下的5个样品。如此多
次重复,最后对所有模型的预测效果做综合统计分析。
预测结果代表建立模型所选用的差异基因的综合预测效 果。
上(40%以上基因有表达)。
某些熟知的非调控基因的表达值也可被用
来检验数据质量。最常用的是GADPH及β -
actin。 Affmetrix的人基因芯片有这两个基
因的5’端和3’端的探针,每个基因5’端和3’
端表达值的比率应在1左右。0.2以下的比率表
示低质mRNA。
数据准备中另一个重要环节是异样样品探
筛选分析之前,来自微阵列的数据必须先被加
以清理,其中用到很多较为复杂的以统计学为 基础的数据处理方法,整个过程称数据准备。 数据准备必须先于任何数据分析。数据分 析是个复杂的过程,包括质量控制、异体探测 以及减少或除去系统误差为目的的数据调整。 此阶段研究人员必须根据其分析结果来决 定样品或数据的取舍。
样品有一一对应关系,则应选择做对应t检验。
对由t检验计算而来的p值应进行适当的调
整以纠正由于多重检验而带来的额外的假阳 性。可采用Bonferroni法及其改进的版本 (如Holm法或Hochberg法)。 差异倍数及从t检验而来的p值均可由微 软的Excel来计算。各种调节p值的算法则需 要用到较专业化的统计分析软件如R。
第三节 基因芯片数据分析的基本策略与方法
一、数据准备
首先从各种样品得来的原始表达值需要经过适 当比例的调整才能相互比较。这种调整称为数 据标准化,最常用的方法之一是比例缩放。 比例缩放的基本方法是将每枚芯片的所有原始 表达值放大或缩小一定倍数。最终使所有研究 中每个基因的中间值均为相等。
在实际数据处理中,目标中间值的选取往 往是由全部研究中芯片原始表达数据的总中间 值来决定。除按中间值缩放外,还可以按平均 值或平衡平均值进行按比例缩放。 平均值对异常超值的敏感度大大超过中间
针的表达值后决定。
4、表达之探测p值。统计学分析探针组每根探针的计
算结果,用来决定表达之探测所用的P或A或M。
Affmetrix的探针是依据GenBank,RefSeq及
dbEST数据库中的DNA序列设计而成,并利用
UniGene以及生物信息学中的片段组装技术来 获取探针的特异性。大多数探针的序列与DNA 正股序列相同(与mRNA序列相同),极少数与 DNA副股相同。探针多倾向位于基因的3’端, 但探针间有足够的距离以确保探针灵敏度。
双荧光标记芯片数据归一化处理:目的是消除
同一芯片上的两种荧光信号在标记、共聚焦扫
描和其他实验操作环节引入的系统误差。最简
单的方法就是将两种不同荧光信号各探针的平
均值或者中间值调整到相同。为进一步消除在
不同荧光强度范围内的标记差异的不同,常采
用LOWESS方法。经过归一化处理的信号强度
被综合而成代表两个样品中各个靶基因表达相
PCA分析会有一定程度的人为因素。尽管如 此,PCA分析应识别出明显的异样样品并指出可 能潜在的异样样品。 聚类技术是另一类有效的异样样品探测方 法。如果一个样品被归于异组,或其距组心的 距离大大超过同组其他成员的相应值,该样品 很可能会是异样样品,应对其进行进一步监测。
二、表达差异基因的筛选 表达差异基因的筛选通常涉及先计算某些统计 学数值,然后根据这些数值来决定基因的取舍。
值。实际工作中常用到平衡平均值(取平均值
之前除去异常超值-最大与最小的5%)。
下一步是检验数据的质量。数据质量会受到 mRNA样品质量及杂交技术操作质量的影响。 Affmetrix的MAS55处理过的数据包括一个代 表表达值是否真的存在的指标,其在所有基 因中的分布可在某种程度上反映出微阵列数
据的质量。P的百分比一般应在40%左右或以
型,模型证实。模型用变量即差异表达基因;
变量选择即差异表达基因选择。选择一个具 体算法来建立预测模型。算法可以是统计学 的,也可是人工智能的。具体算法选好后即 可用训练数据建立预测模型。
模型证实的目的是检测其实际预测效果,需用训练数据 以外的样品作为实际的预测对象。常用有效的模型证实 方法之一是交叉证实。其特点是既能获取模型证实所需 假如有两组样品A和B,每组25个样品。1/10交叉证实即
二、Agilent芯片和其他用于双荧光标记的芯 片及其数据分析 Agilent的长寡核糖核苷酸芯片是建立在 其母公司HP的喷墨打印技术上,把底物直接打 印到芯片上特定区域,在芯片上固相合成具有 特定核苷酸顺序的探针。探针长度60个核苷酸 残基,高于Affymetrix探针,大大提高了探针 特异性。每一靶基因通常只选一个探针。 Agilent也提供cDNA探针。
两个最常用的此类统计数值是差异倍数及从t检
验而来的p值。
差异倍数是基因表达变化量的衡量尺度。可
用算术平均值(a同b的算术平均值为(a+b)
/2)计算。亦可用几何平均值来计算( a同b的
几何平均值为10(
㏒ a+ ㏒ b
)/2或√ab)。
几何平均值受到个别超值的影响较小,常被用于组内
表达差异较强的微阵列数据。由t检验而来的p值是用
对强度的信号比值。
第二节 基因芯片数据处理与分析 所有相关的DNA微阵列数据分析按其目标所分 均可归两类:发现和预测。
发现:代谢调控中的新基因、潜在的新的药物
受体、新的致病基因。
预测:建立数学预测模型,用于药物毒性预测
及疾病诊断与分类。
发现和预测均需经过相同的基本分析途径:
有统计意义的差异表达基因的筛选。
来衡量基因表达差异的统计学意义的统计参数。P值
被用来估计两组看起来不等的平均值是否真的不一样。
P值越小,两组不等平均值的真的不同的几率越高, 表达差异越真实。做t检验要求数据的分布为正态分 布,而多数芯片数据不满足这一要求,因此要做数据 转换。t检验有独立或非对应及对应之分。其选择要
由具体的实验设计来决定。如对照组的样品和实验组
Concepts of Array Design
PM to maximize hybriegree of cross-hybridization
PM MM
Probe pair
Probe set
每根探针都会有一个相应的基因表达值。
但最终每个靶基因的表达值要通过独特的统计 学运算才能得到(如Affmetrix的MA55)。 对于Affmetrix的芯片,重要的是应懂得设 计核心是探针对;每个靶基因都有多组相应的 探针对,称探针组。 典型的经过MA55处理过的Affymetrix的基
的几率会是假的。统计学上称为假阳性。
建立数学预测模型是另一个基因数据分析的主要目标。
建立预测模型需要较多的样品。各组至少应有15个
以上。 预测模型的建立过程涉及两个主要步骤:模型建立与 模型证实。 模型建立的基本步骤包括从训练数据中选择变量,
采用一种统计学或人工智能的具体算法用所选的变量建
立预测模型,并利用训练样品对建立的预测模型作出初 步的检验。模型证实则需要用预测样品来衡量模型的实 际预测准确度。训练用样品和预测用样品不能等同,必 须是两组不同的样品。
三、差异基因的分组聚类 是常用的划分基因的分析手段,亦常用于样 品的划分与归类。将聚类技术用于微阵列数
据的分析,则是将基因或样品按其表达模式
