酶切位点序列
酶切位点识别序列

上一段碱基的特定序列,DNAcutting site):Restriction 酶切位点(Enzyme 序列切成两段。
限制性内切酶能够识别出这个序列并在此将DNA可能存在同尾酶,不同酶的识别序列不同,
WW 有的可能能识别多个酶切位点比如STY1识别序列有
PCR引物设计时酶切位点的保护碱基表不同内切酶对识别位点以外最少保护碱
基数目的要求(在本表中没有列出的)酶,则通常需在识别位点两端至少加上
6个保护碱基,以确保酶切反应的进行。
简DNA的尾端时,限制性内切酶经常不能成功切断(保护碱基:当酶切位点在双链所以经常在引物设计=_=|||)单的想象为酶遇到识别位点之后从旁边掉下去了……
个碱基以确保成功酶切。
比32时,在末端的限制性内切酶识别位点之后
再加上、改3'
成可以的引物是5‘
GCTAGCNNNNN……如你5‘CAGGCTAGCNNNNN……3' 呃……CAG是我随便加的,你可以考虑一下CG/AT含量
注释
1.如果要加在序列的5'端,就在酶切位点识别碱基序列(红色)的5'端加上相应的碱基(黑色),如果要在序列的3'端加上保护碱基,就在酶切位点识别碱基序列(红色)的3'端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
加保护碱基时最好选用切割率高时加的相应碱基。
3.。
takara快切酶酶切位点

takara快切酶酶切位点
Takara快切酶是一种用于分子生物学实验的酶,它能够识别特定的DNA序列并在该序列上进行切割。
酶切位点是指酶在DNA分子上识别并切割的特定序列。
Takara快切酶的切位点取决于具体使用的酶种类,不同的酶有不同的识别序列和切割方式。
从分子生物学角度来看,Takara快切酶的切位点是DNA双链上的特定序列,这些序列通常是4至8个碱基对长,具有特定的碱基配对规律。
酶切位点的选择对于DNA分子的切割和连接至关重要,因为它直接影响着DNA重组、连接和修复的效率和准确性。
在实验操作中,研究人员需要根据所使用的Takara快切酶的特性来选择合适的切位点,以确保实验能够顺利进行。
通常情况下,研究人员会根据酶的说明书或相关文献来确定切位点,然后设计合适的引物或寡核苷酸序列进行实验操作。
除此之外,Takara快切酶的切位点也与基因工程、基因编辑等领域密切相关。
在基因编辑技术中,研究人员经常利用
CRISPR/Cas9等系统来指导Takara快切酶在特定的DNA序列上进行切割,从而实现对基因组的精准编辑。
总的来说,Takara快切酶的切位点是分子生物学领域中非常重要的概念,它涉及到DNA序列的识别和切割,对于基因工程、基因编辑和分子生物学研究具有重要意义。
在实验操作中,科研人员需要根据具体的实验目的和所用酶的特性来选择合适的切位点,并进行相关的实验设计和操作。
酶切位点识别序列

酶切位点(Restriction Enzyme cutting site):DNA上一段碱基的特定序列,能够识别出这个序列并在此将序列切成两段。
可能存在同尾酶,不同酶的识别序列不同,
有的可能能识别多个酶切位点比如STY1识别序列有WW
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
)
保护碱基:当酶切位点在双链DNA的尾端时,限制性内切酶经常不能成功切断(简单的想象为酶遇到识别位点之后从旁边掉下去了…… =_=|||)所以经常在引物设计时,在末端的限制性内切酶识别位点之后再加上2、3个碱基以确保成功酶切。
比如你的引物是5‘ GCTAGCNNNNN……3’ 可以改成5‘ CAGGCTAGCNNNNN……3’ 呃……CAG是我随便加的,你可以考虑一下CG/AT含量
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相应的
碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。
酶切位点识别序列

酶切位点识别序列 Document serial number【KKGB-LBS98YT-BS8CB-BSUT-BST108】
酶切位点(Restriction Enzyme cutting site):DNA上一段碱基的特定序列,能够识别出这个序列并在此将序列切成两段。
可能存在同尾酶,不同酶的识别序列不同,
有的可能能识别多个酶切位点比如STY1识别序列有WW
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相应的
碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。
酶切位点平末端

酶切位点平末端
酶切位点平末端指的是一种特定的DNA或RNA序列结构,其中两个不同的序列在同一个平面上相互平行且相对。
这种结构通常出现在DNA或RNA的限制性内切核酸酶的切割位点处,这些酶能够识别特定的序列并切割DNA或RNA分子。
在DNA或RNA分子中,酶切位点平末端的具体示例包括:
1.EcoRI酶切位点:EcoRI是一种限制性内切核酸酶,能够识别并切割DNA
分子中的GAATTC序列。
切割后产生的片段具有平末端。
2.BamHI酶切位点:BamHI是一种限制性内切核酸酶,能够识别并切割
DNA分子中的GGATCC序列。
切割后产生的片段同样具有平末端。
在生物实验中,酶切位点平末端的应用非常广泛。
例如,在构建基因克隆载体时,需要将目的基因与载体进行连接,通常需要在目的基因和载体之间进行限制性内切核酸酶的切割。
当使用平末端的限制性内切核酸酶进行切割时,可以获得两个平末端的DNA片段,通过连接这些片段可以实现目的基因与载体的连接。
总结来说,酶切位点平末端指的是一种特定的DNA或RNA序列结构,其中两个不同的序列在同一个平面上相互平行且相对。
这种结构通常出现在限制性内切核酸酶的切割位点处,这些酶能够识别特定的序列并切割DNA或RNA 分子。
在生物实验中,酶切位点平末端的应用非常广泛,例如在构建基因克隆载体时需要进行限制性内切核酸酶的切割和连接。
酶切位点回文结构

酶切位点回文结构酶切位点回文结构是指DNA双链的某一位置有着对称的核苷酸序列,这个序列可以被特定的酶识别并切割。
这种对称结构通常具有二分结构,并在DNA序列中产生反向和正向两条不同的切割位点。
回文结构的命名来源于它的两边对称,就像是前后读取这个序列相同。
因此,回文结构也称为“自旋结构”,相对而言,在其他结构中,DNA序列中的每个核苷酸都需要与另一个核苷酸进行配对才能形成结构。
而在回文结构中,只需要序列的前一部分与后一部分相互配对,就能够形成结构。
回文结构的重要作用在于启动子、转录调控区和基因的切割,这些结构是基因的控制和调节关键点。
这些结构还可以在DNA修复中发挥重要作用,在DNA重组中起着非常重要的作用。
此外,在注册和DNA复制的过程中,回文结构也经常出现。
常见的酶切位点回文结构包括EcoRV酶、BamHI酶、EcoRI酶和HindIII酶。
这些酶都有特定的序列和结构,和它们适用的DNA酶切位点。
以BamHI酶为例,BamHI酶的切割位点为GGATCC,这个序列中心对称。
DNA序列的基本单位是核苷酸,它们包括脱氧核糖和配基。
在DNA的单链上,一个核苷酸会与它的相邻核苷酸连接在一起。
而在DNA的双链上,两个核苷酸会通过氢键相互作用连接在一起,形成一个碱基对。
回文结构通常由四种碱基构成:腺嘌呤、鸟嘌呤、胸腺嘧啶和鳞状细胞脱氧核糖核苷酸。
在回文结构上,这些碱基会按照逆序和对称的方式连接在一起。
这种对称结构使许多切割酶能够精准地切割DNA序列,有助于DNA的合成、修复和重组。
例如,EcoRI酶在寻找ATG|CTA(斜杆表示切割部位)序列时,只会在两个ATG|CTA之间找到,并且只在这些位点上进行切割。
回文结构也可以用于分子生物学技术中的分子克隆和DNA分析。
回文结构的两侧是相同的,一侧的序列可以用于构建DNA的克隆。
例如,一个DNA序列可以通过PCR扩增,然后在回文结构两侧插入适当的限制酶切位点。
这种方法可以使PCR扩增后的DNA序列在回文结构两侧具有相同的序列,并且可以通过对这两个酶切位点的切割进行定向克隆和DNA 分析。
酶切位点识别序列

酶切位点识别序列 Last revised by LE LE in 2021
酶切位点(Restriction Enzyme cutting site):DNA上一段碱基的特定序列,能够识别出这个序列并在此将序列切成两段。
可能存在同尾酶,不同酶的识别序列不同,
有的可能能识别多个酶切位点比如STY1识别序列有WW
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相应的
碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。
各种酶切位点的保护碱基引物设计必看

各种酶切位点的保护碱基引物设计必看酶切位点是指特定的序列,酶可以识别并在该位置切割DNA分子。
这些位点的特异性使得酶在分子生物学中广泛应用于DNA片段的定位和切割。
然而,在一些实验中,我们可能需要保护酶切位点周围的碱基,以免酶切,并且只在特定的位置引导酶切。
因此,保护碱基引物的设计对于实验的成功非常重要。
以下是保护碱基引物设计的一些建议。
首先,保护碱基引物的设计需要考虑引物的长度。
引物的长度通常为18到30个碱基,具体的长度需要根据实验的需求和酶切位点周围的序列特征来确定。
引物的长度应足够长,以确保引物和靶序列的特异性,但不应过长,以免引物形成二级结构或与非特异性位点结合。
其次,保护碱基引物的设计需要考虑引物的碱基组成。
在设计引物时,建议尽量避免引物中出现酶切位点周围的碱基序列,以防止酶的误切。
例如,如果我们希望保护酶切位点周围的AATTC序列,可以设计一个引物,其中没有AATTC序列。
同时,引物的碱基组成应尽量避免多聚核苷酸或含有GC碱基的片段,以防止引物之间的结合或引物与非特异靶序列的结合。
此外,保护碱基引物的设计需要考虑引物的特异性。
在设计引物时,建议使用特异性的引物序列,以确保引物只与目标酶切位点结合。
可以通过使用生物信息学工具,如BLAST,来验证引物的特异性。
引物的特异性还可以通过调整引物的长度和碱基组成来进一步提高。
最后,保护碱基引物的设计需要考虑引物的热力学性质。
引物的热力学性质包括引物的熔解温度(Tm值)和引物之间的配对。
引物的Tm值与引物的碱基组成、长度和引物与靶序列之间的碱基配对相关。
可以使用在线工具,如NEB的Tm计算器,来计算引物的Tm值,并对不同的引物进行比较。
此外,引物之间的配对可以通过设计引物的末端序列来调整,例如末端的碱基配对或非配对等。
总结起来,保护碱基引物的设计需要考虑引物的长度、碱基组成、特异性和热力学性质。
通过合理设计引物,可以保护酶切位点周围的碱基,并在特定位置引导酶切,为实验的成功提供有力的保障。
常见限制性内切酶识别序列

常见限制性内切酶识别序列(酶切位点)(BamHI、EcoRI、HindIII、NdeI、XhoI等)Time:2009-10-22 PM 15:38Author:bioer Hits: 7681 times在分子克隆实验中,限制性内切酶是必不可少的工具酶。
无论是构建克隆载体还是表达载体,要根据载体选择合适的内切酶(当然,使用T 载就不必考虑了)。
先将引物设计好,然后添加酶切识别序列到引物5' 端。
常用的内切酶比如BamHI、EcoRI、HindIII、NdeI、XhoI等可能你都已经记住了它们的识别序列,不过为了保险起见,还是得查证一下。
下面是一些常用的II型内切酶的识别序列,仅供参考。
先介绍一下什么是II型内切酶吧。
The Type II restriction systems typically contain individual restriction enzymes and modification enzymes encoded by separate genes. The Type II restriction enzymes typically recognize specific DNA sequences and cleave at constant positions at or close to that sequence to produce 5-phosphates and 3-hydroxyls. Usually they require Mg 2+ ions as a cofactor, although some have more exotic requirements. The methyltransferases usually recognize the same sequence although some are more promiscuous. Three types of DNA methyltransferases have been found as part of Type II R-M systems forming either C5-methylcytosine, N4-methylcytosine or N6-methyladenine.酶类型识别序列ApaIType II restrictionenzyme5'GGGCC^C 3'BamHIType II restrictionenzyme5' G^GATCC 3'BglIIType II restrictionenzyme5' A^GATCT 3'EcoRIType II restrictionenzyme5' G^AATTC 3'HindIIIType II restrictionenzyme5' A^AGCTT 3'KpnIType II restrictionenzyme5' GGTAC^C 3'NcoIType II restrictionenzyme5' C^CATGG 3'NdeIType II restrictionenzyme5' CA^TATG 3'NheIType II restrictionenzyme5' G^CTAGC 3'NotIType II restrictionenzyme5' GC^GGCCGC 3'SacIType II restrictionenzyme5' GAGCT^C 3'SalIType II restrictionenzyme5' G^TCGAC 3'SphIType II restrictionenzyme5' GCATG^C 3'XbaIType II restrictionenzyme5' T^CTAGA 3'XhoIType II restrictionenzyme5' C^TCGAG 3'要查找更多内切酶的识别序列,你还可以选择下面几种方法:1. 查你所使用的内切酶的公司的目录或者网站;2. 用软件如:Primer Premier5.0或Bioedit等,这些软件均提供了内切酶识别序列的信息;3. 推荐到NEB的REBASE数据库去查(网址:/rebase/rebase.html)当你设计好引物,添加上了内切酶识别序列,下一步或许是添加保护碱基了,可以参考:NEB公司网站提供的关于设计PCR引物保护碱基参考表下载(也可见图片)双酶切buffer的选择(MBI、罗氏、NEB、Promega、Takara)再给大家推荐一种新的不需要连接反应的分子克隆方法,优点包括:①设计引物不必考虑选择什么酶切位点;②不必考虑保护碱基的问题;③不必每次都选择合适的酶来酶切质粒制备载体;④而且不需要DNA连接酶;⑤假阳性几率低(因为没有连接反应这一步,载体自连的问题没有了)。
限制酶酶切位点

限制酶酶切位点限制酶酶切位点是一种常见的分子生物学技术,用于在DNA或RNA分子中特定的序列上进行切割。
酶切位点是酶识别的特定序列,一旦找到该序列,酶便会在该位置切割DNA或RNA分子。
限制酶酶切位点在基因工程和分子生物学研究中起着至关重要的作用,下面将详细介绍限制酶酶切位点的相关知识。
酶切位点是由限制酶识别的特定DNA或RNA序列决定的。
限制酶是一类能够识别并切割DNA或RNA特定序列的酶,也被称为内切酶。
限制酶通常识别的切割位点是对称的,可以是4-8个碱基对长。
常见的限制酶有EcoRI、HindIII、BamHI等。
限制酶酶切位点的限制酶识别序列通常具有一定的保守性,也就是说,相同的限制酶通常会识别相似的序列。
例如,EcoRI识别的酶切位点序列为5'-GAATTC-3',其中GAATTC为限制酶识别序列,5'和3'分别表示DNA的两个末端。
限制酶酶切位点的限制酶识别序列通常具有一定的特异性,也就是说,相同的限制酶通常只会识别特定的序列。
例如,EcoRI只会识别5'-GAATTC-3'序列,而不会识别其他序列。
限制酶酶切位点的选择对于实验的设计非常重要。
在分子生物学实验中,科研人员常常需要在特定的DNA或RNA序列上进行切割,以便进一步进行下游实验,如限制酶切割后的DNA片段的连接、测序等。
因此,选择适当的限制酶和相应的酶切位点非常关键。
限制酶酶切位点的选择需要考虑多个因素。
首先,需要考虑实验所需的切割位点是否存在于DNA或RNA序列中。
其次,需要考虑限制酶的切割效率和特异性。
一些限制酶可能对特定的序列有更高的切割效率,而对其他序列的切割效率较低。
此外,还需要考虑限制酶的切割位点的位置是否适合实验的要求。
在实际应用中,科研人员常常需要将限制酶切割后的DNA片段进行连接,以构建重组DNA分子。
在这种情况下,选择具有兼容的酶切位点的限制酶非常重要。
兼容的酶切位点是指两个限制酶切割产生的DNA片段的末端具有相同的序列。
酶切位点
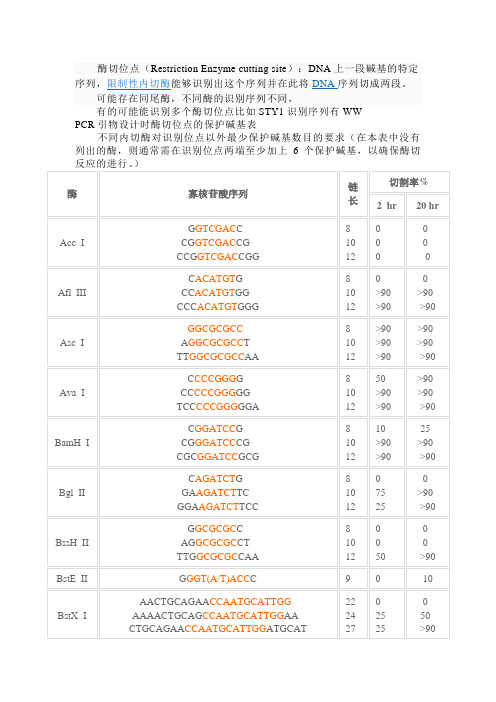
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
)
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相
应的碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。
各种酶切位点的保护碱基引物设计必看

各种酶切位点的保护碱基引物设计必看酶切位点的保护碱基引物设计在分子生物学领域中起着至关重要的作用。
它们是研究者在酶切实验中必不可少的工具,用于保护酶切位点周围的碱基,以避免酶的切割作用。
本文将介绍保护碱基引物设计的一般原则和具体步骤,并探讨一些常见的问题和注意事项。
保护碱基引物设计的一般原则如下:1.引物长度:保护碱基引物的长度通常为15-25个碱基对。
2.引物序列:引物应根据酶切位点的序列设计。
为了确保引物的特异性,通常将酶切位点和其周围的碱基考虑在内。
3.引物组成:引物的核苷酸组成应考虑碱基的GC含量,以保持引物的稳定性。
通常,GC含量高于50%的引物更稳定。
4.引物末端修饰:引物的末端修饰可以提高引物与目标DNA的亲和性,并增加引物的稳定性。
常用的末端修饰包括磷酸化和胺基修饰等。
保护碱基引物设计的步骤如下:1.获取酶切位点序列:首先,需要获取目标DNA序列中待保护的酶切位点的序列。
2.引物设计:根据酶切位点的序列设计引物。
引物的长度通常为15-25个碱基对。
为了提高特异性,可以考虑在引物序列中加入一些限制性内切酶无法识别的碱基。
3.引物末端修饰:根据需要选择引物的末端修饰方式,例如磷酸化和胺基修饰等。
4.引物的合成:完成引物设计后,可以委托专业的生物科技公司进行引物的合成。
确保引物的纯度和质量。
在进行保护碱基引物设计时,还需注意一些常见的问题和注意事项:1.引物特异性:在设计引物时,要确保引物与目标DNA的序列具有高度特异性,以避免引物与非目标区域的杂交。
2.引物的稳定性:引物的稳定性对于酶切实验的成功至关重要。
在设计引物时,要尽量选择稳定的引物序列,例如具有较高GC含量的引物。
3.引物纯度和质量:为了保证引物的质量和稳定性,引物的合成必须由专业的生物科技公司进行。
确保引物的纯度高,无杂质。
4.引物的浓度和稀释:在使用引物进行酶切实验时,要合理确定引物的浓度和稀释倍数,以保证实验的成功。
总之,保护碱基引物设计是分子生物学研究中不可或缺的一部分。
载体结构设计酶切位点和引物原理

载体结构设计酶切位点和引物原理一、概述随着生物技术的发展,载体结构设计在基因工程研究中扮演着至关重要的角色。
其中,酶切位点和引物的设计原理对于基因克隆、表达及遗传转化等方面均有重要意义。
本文将从酶切位点和引物的原理出发,探讨载体结构设计的相关内容。
二、酶切位点的设计原理1. 基本概念酶切位点是指DNA序列上特定的核苷酸序列,能够被特定的限制性内切酶识别并切割。
通过合理设计酶切位点,可以实现对DNA序列的特异性切割,为后续的基因工程操作提供必要的前提条件。
2. 设计原则(1)选择适当的限制性内切酶酶切位点的选择应基于限制性内切酶的特异性,并考虑到后续的克隆和操作需求。
通常选择常用的限制性内切酶,如EcoRI、BamHI等。
(2)避免酶切位点的相互重叠在设计酶切位点时,应当避免酶切位点之间的相互重叠,以免出现无法正常切割的情况。
还应考虑酶切位点的相对位置,以保证后续的克隆操作能够顺利进行。
(3)考虑DNA序列的完整性在设计酶切位点时,应当考虑到DNA序列的完整性,避免对基因序列造成不必要的破坏。
还需注意酶切位点的分布情况,以减少对DNA序列的干扰。
3. 应用示例以人类胰岛素基因为例,其序列包含多个酶切位点,如EcoRI和BamHI。
通过合理设计酶切位点,可以实现对人类胰岛素基因的特异性切割,为后续的基因工程操作奠定了基础。
三、引物的设计原理1. 基本概念引物是用于PCR扩增、基因克隆等实验操作中的一种寡核苷酸片段。
合理设计的引物能够与目标DNA序列特异性结合,从而实现对目标序列的扩增和克隆。
2. 设计原则(1)选择合适的引物长度引物的长度应控制在15-30个核苷酸之间,过长或过短的引物容易导致PCR扩增效率低下。
(2)避免引物间的相互作用在引物的设计中,需要避免引物之间的相互作用,以免出现不必要的二聚体或引物假扩增。
(3)考虑引物的特异性引物的设计应基于目标DNA序列的特异性,避免引物与非目标序列结合,从而影响后续实验结果的准确性。
引物加酶切位点的原理

引物加酶切位点的原理引物加酶切位点是分子生物学中常用的实验技术,它在DNA重组、PCR扩增、基因克隆等领域起着至关重要的作用。
引物是DNA 序列中的一小段,它能够特异性地与目标DNA序列结合,而酶切位点则是DNA分子上特定的酶切酶能够识别并切割的序列。
本文将介绍引物加酶切位点的原理及其在实验中的应用。
引物的设计是引物加酶切位点实验的第一步。
引物通常由20-30个碱基组成,它们的序列应该与目标DNA序列互补,以确保引物能够特异性地结合到目标DNA上。
在引物的设计中,需要考虑引物的长度、GC含量、Tm值等因素,以确保引物在实验条件下能够有效地结合到目标DNA上。
引物的加入使得酶切位点的选择变得更加灵活。
酶切位点是DNA序列中特定的核苷酸序列,它是酶切酶识别并切割的靶点。
在实验中,引物的引入可以使得酶切位点的选择更加精准,从而实现对目标DNA的特异性切割。
通过合理设计引物,可以在目标DNA上引入新的酶切位点,从而为后续的实验操作提供便利。
引物加酶切位点的原理是通过引物的特异性结合和酶切位点的特异性切割,实现对目标DNA的定点切割。
在实验中,首先将引物与目标DNA序列结合,形成引物-目标DNA复合物。
随后,加入酶切酶,酶切酶能够识别并切割酶切位点,从而在目标DNA上引入切割。
通过这一原理,可以实现对目标DNA的特异性切割,为后续的实验操作提供基础。
引物加酶切位点在分子生物学实验中有着广泛的应用。
它可以用于PCR扩增中的引物设计、基因克隆中的目标DNA切割等实验操作。
通过合理设计引物和选择合适的酶切位点,可以实现对目标DNA的精准操作,为分子生物学研究提供了重要的技术支持。
总之,引物加酶切位点的原理是通过引物的特异性结合和酶切位点的特异性切割,实现对目标DNA的定点切割。
它在分子生物学实验中有着重要的应用,为研究人员提供了强大的实验工具。
通过合理设计引物和选择合适的酶切位点,可以实现对目标DNA的精准操作,为分子生物学研究提供了重要的技术支持。
furin蛋白酶切位点

furin蛋白酶切位点
Furin蛋白酶切位点是指Furin蛋白酶在水解底物时识别和剪切的特定氨基酸序列。
Furin蛋白酶是一种负责剪切和激活其他蛋白质的酶,特别是那些在内质网和高尔基体合成的蛋白质前体。
Furin蛋白酶切位点通常由Arg-Xaa-(Lys/Arg)-Arg或Arg-Xaa-Xaa-Arg的序列组成,其中“Xaa”表示任意氨基酸。
Furin蛋白酶切割的底物包括许多生物活性物质、细胞因子、生长因子、肽类激素、病毒蛋白等。
由于Furin蛋白酶切割后的蛋白质具有更强的生物活性,因此Furin蛋白酶在许多生理和病理过程中发挥着重要作用。
Furin蛋白酶切位点的研究对于深入理解其生物学功能和探索其在疾病治疗中的应用具有重要意义。
近年来,基于Furin蛋白酶切位点的治疗策略已成为一种新兴的治疗手段,值得进一步研究和探索。
限制性内切酶消化技术的使用中常见问题

限制性内切酶消化技术的使用中常见问题引言:限制性内切酶消化技术是生物学研究和分子生物学实验中常用的一项技术。
通过切割DNA分子,限制性内切酶消化技术可以实现DNA的分离、测序、重组和修饰等多种用途。
然而,在使用限制性内切酶消化技术的过程中,也会遇到一些常见问题。
本文将针对这些问题进行详细阐述,并提供相应的解决方法。
问题一:限制性内切酶未能有效切割目标DNA在进行限制性内切酶消化反应时,有时会出现限制性内切酶未能完全切割目标DNA的情况。
这可能是由于以下原因造成的:1. 酶活性损失:限制性内切酶的活性受多种因素影响,如pH值、温度、离子浓度等。
在使用限制性内切酶之前,应确保其活性没有受到损害。
可以通过进行阳性对照实验,或者使用已被证实具有良好活性的酶来验证。
2. 酶切位点序列变异:限制性内切酶只能识别并切割特定的DNA序列。
若目标DNA序列中的限制性内切酶切割位点发生变异或缺失,将导致酶无法正常切割。
可以通过测序目标DNA来验证切割位点是否存在变异。
解决方法:1. 确认酶的活性:使用阳性对照实验或者使用已被证实具有良好活性的限制性内切酶来验证其活性。
2. 验证切割位点序列:通过测序目标DNA来验证切割位点的序列,确保其与限制性内切酶的切割位点一致。
问题二:限制性内切酶反应产物异常在进行限制性内切酶消化反应后,有时反应产物会出现异常的情况,如模板DNA完全被切割或完全未切割。
这可能是由于以下原因造成的:1. 反应条件不合适:反应缓冲液的pH值、离子浓度和温度等因素都会影响限制性内切酶的活性。
若反应条件不合适,将导致限制性内切酶无法正常切割目标DNA。
2. 酶过多或过少:限制性内切酶的用量应根据目标DNA的长度和预测切割位点来确定。
若酶用量过多或过少,会导致反应产物异常。
解决方法:1. 优化反应条件:通过调整反应缓冲液的pH值、离子浓度和温度等因素,优化反应条件,确保限制性内切酶能够正常切割目标DNA。
酪蛋白水解酶酶切位点

酪蛋白水解酶酶切位点
酪蛋白水解酶是一种能够水解酪蛋白的酶,其酶切位点通常是一段特定的氨基酸序列。
以下是一些常见的酪蛋白水解酶酶切位点:
1. 丝氨酸(Ser)酶切位点:丝氨酸是一个常见的酶切位点氨
基酸,酪蛋白水解酶通常在序列中的丝氨酸附近切割。
例如,在酪蛋白中,酶切位点可能是S/T-X-E,其中X表示任意氨基酸,T表示丝氨酸,E表示谷氨酸。
2. 脯氨酸(Pro)酶切位点:脯氨酸也是一个常见的酶切位点
氨基酸,酪蛋白水解酶可以在序列中的脯氨酸旁边进行切割。
例如,在酪蛋白中,酶切位点可能是P-X,其中P表示脯氨酸,X表示任意氨基酸。
3. 脯氨酸-丝氨酸(Pro-Ser)酶切位点:脯氨酸和丝氨酸组成
的序列也是一个常见的酶切位点,酪蛋白水解酶可以在脯氨酸和丝氨酸之间进行切割。
例如,在酪蛋白中,酶切位点可能是
P-X-S/T,其中P表示脯氨酸,X表示任意氨基酸,S/T表示丝氨酸或苏氨酸。
需要注意的是,不同的酪蛋白水解酶对酶切位点的选择有所不同,因此具体的酶切位点会根据酶的来源和特性而有所差异。
酶切位点——精选推荐
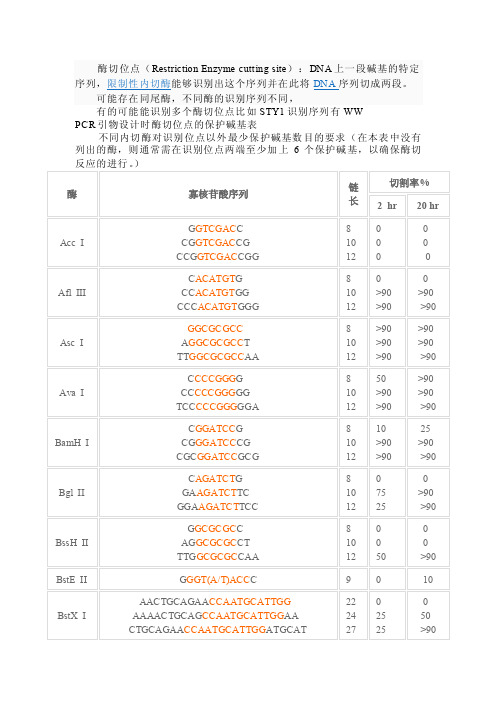
PCR引物设计时酶切位点的保护碱基表
不同内切酶对识别位点以外最少保护碱基数目的要求(在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
)
注释
1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相
应的碱基(黑色),如果要在序列的3’端加上保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
2.切割率:正确识别并切割的效率。
3.加保护碱基时最好选用切割率高时加的相应碱基。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
ApaI (类型:Type II restriction enzyme )识别序列:5'GGGCC^C 3' BamHI(类型:Type II restriction enzyme )识别序列: 5' G^GATCC 3' BglII (类型:Type II restriction enzyme )识别序列:5' A^GATCT 3' EcoRI (类型:Type II restriction enzyme )识别序列:5' G^AATTC 3' HindIII (类型:Type II restriction enzyme )识别序列:5' A^AGCTT 3' KpnI (类型:Type II restriction enzyme )识别序列:5' GGTAC^C 3' NcoI (类型:Type II restriction enzyme )识别序列:5' C^CATGG 3' NdeI (类型:Type II restriction enzyme )识别序列:5' CA^TATG 3' NheI (类型:Type II restriction enzyme )识别序列:5' G^CTAGC 3' NotI (类型:Type II restriction enzyme )识别序列:5' GC^GGCCGC 3' SacI (类型:Type II restriction enzyme )识别序列:5' GAGCT^C 3' SalI (类型:Type II restriction enzyme )识别序列:5' G^TCGAC 3' SphI (类型:Type II restriction enzyme )识别序列:5' GCATG^C 3' XbaI (类型:Type II restriction enzyme )识别序列:5' T^CTAGA 3' XhoI (类型:Type II restriction enzyme )识别序列:5' C^TCGAG 3'。
