NEB常用酶介绍
NEB保护碱基-各种酶切位点保护碱基
0
0
0
0
0
0
0
0
75
>90
75
>90
Nhe I
GGCTAGCC CGGCTAGCCG CTAGCTAGCTAG
0
0
10
25
10
50
Not I
TTGCGGCCGCAA
0
0
ATTTGCGGCCGCTTTA
10
10
AAATATGCGGCCGCTATAAA
10
10
ATAAGAATGCGGCCGCTAAACTAT
酶 Acc I Afl III Asc I Ava I BamH I Bgl II BssH II BstE II BstX I Cla I
EcoR I
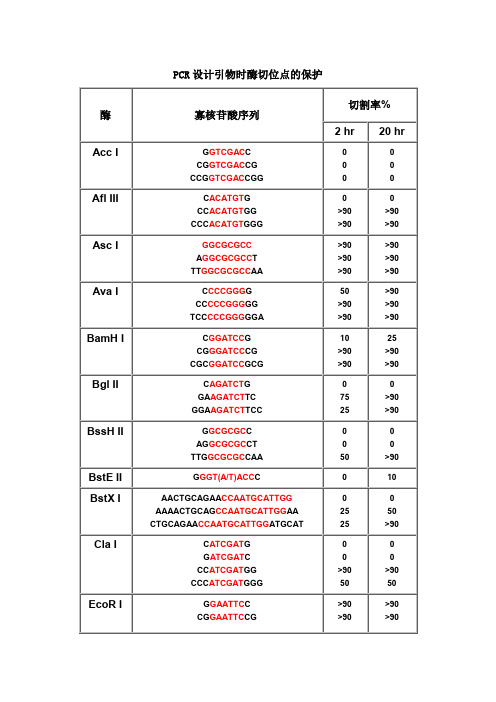
PCR 设计引物时酶切位点的保护
寡核苷酸序列
GGTCGACC CGGTCGACCG CCGGTCGACCGG
CACATGTG CCACATGTGG CCCACATGTGGG
0
0
0
25
0
50
75
>90
Pst I
GCTGCAGC
0
0
TGCACTGCAGTGCA
10
10
AACTGCAGAACCAATGCATTGG
>90
>90
AAAACTGCAGCCAATGCATTGGAA
>90
>90
CTGCAGAACCAATGCATTGGATGCAT
0
0
Pvu I
CCGATCGG ATCGATCGAT TCGCGATCGCGA
25
90
AAGGAAAAAAGCGGCCGCAAAAGGAAAA
NEB酶清单

使用
库存 BmgBI BmrI BmtI BmtI-HF™ BpmI Bpu10I BpuEI BsaAI BsaBI BsaHI BsaI BsaI-HF™ BsaJI BsaWI BsaXI BseRI BseYI BsgI BsiEI BsiHKAI BsiWI BslI BsmAI BsmBI BsmFI BsmI BsoBI Bsp1286I BspCNI BspDI BspEI BspHI BspMI BspQI BsrBI BsrDI BsrFI BsrGI BsrI BssHII BssK BssS BstAPI BstBI BstEII BstEII-HF™
库存
使用NcoI-HF™ScIINEB 库存 AatII Acc65I AccI AciI AclI AcuI AfeI AflII AflIII AgeI AgeI-HF™ AhdI AleI AluI AlwI AlwNI ApaI ApaLI ApeKI ApoI AscI AseI AsiSI AvaI AvaII AvrII BaeGI BaeI BamHI BamHI-HF™ BanI BanII BbsI BbvCI BbvI BccI BceAI BcgI BciVI BclI BcoDI BfaI BfuAI BfuCI BglI BglII
使用
Blp
BstNI
HhaI
库存 HincII HindIII HindIII-HF™ HinfI HinP1I HpaI HpaII HphI Hpy166II Hpy188I Hpy188III Hpy99I HpyAV HpyCH4III HpyCH4IV HpyCH4V I-CeuI I-SceI KasI KpnI KpnI-HF™ LpnPI MboI MboII MfeI MfeI-HF™ MluCI MluI MlyI MmeI MnlI MscI MseI MslI MspA1I MspI MspJI MwoI NaeI NarI Nb.BbvCI Nb.BsmI Nb.BsrDI Nb.BtsI NciI NcoI
NEB T4DNA连接酶

T4 DNA 连接酶货号规格价格#M0202L 100,000 units 3,029.00元#M0202M (高浓度5x)100,000 units 3,029.00元#M0202S 20,000 units 699.00元#M0202T (高浓度5x)20,000 units 699.00元#M0202V 10,000 units 349.00元■ 提高反应效率■粘性末端和平齐末端均可连接■限制性酶切片段的克隆(3)■将linker 或adapter 连接到DNA 片段的平齐末端■室温或16 ℃均有活性概述该酶催化契合的双链DNA或RNA的5´-磷酸末端和3´-羟基末端形成磷酸二酯键。
该酶不仅能够催化平滑末端或粘性末端DNA之间的连接,还可以修复双链DNA、RNA或DNA/ RNA杂交双链中的单链切口(1) 。
来源纯化自E. coli C600 pcl857 pPLc28 lig8(2) 。
反应条件1 X T4 DNA 连接酶缓冲液[ 50 mM Tris-HCl(pH 7.5 @ 25 ℃),10 mM MgCl2,10 mM DTT,1 mM ATP]。
推荐DNA 5' 末端浓度为0.1-1.0 μM。
质保声明无核酸内切酶和外切酶污染。
每批T4 DNA 连接酶均通过模拟克隆实验进行检测,该实验可以检测出连接后DNA 末端的任何损伤。
结果表明超过99.9% 的DNA 末端保持完好。
单位定义(粘性末端活性单位)1 单位指在20 μl 1X T4 DNA 连接酶反应缓冲液中,16 ℃反应条件下,30 分钟能使50% 的经HindIII 消化的λDNA 片段[5' 端浓度为0.12 μM (300 μg/ml)]连接所需的酶量。
浓度400,000 units/ml 和2,000,000 units/ml。
贮存条件50 mM KCl, 10 mM Tris-HCl (pH 7.4), 0.1 mM EDTA, 1 mM DTT, 200 μg/ml BSA 和50% 甘油,-20°C 贮存。
neb内切酶bamhi失活温度

neb内切酶bamhi失活温度(原创版)目录1.概述2.内切酶的作用和分类3.NEB 内切酶 BamHI 的特点4.NEB 内切酶 BamHI 的失活温度5.结论正文1.概述内切酶是一种可以切割 DNA 分子的酶,它在分子生物学和基因工程领域具有广泛的应用。
根据来源和特性的不同,内切酶可以分为多种类型,如 E.coli 内切酶、BamHI 内切酶等。
本文主要介绍 NEB 内切酶 BamHI 的失活温度。
2.内切酶的作用和分类内切酶可以识别特定的核苷酸序列,并在这些位点上切割 DNA 分子。
根据切割方式的不同,内切酶可分为 I 型和 II 型。
I 型内切酶切割后生成平末端,而 II 型内切酶切割后生成粘性末端。
NEB 内切酶 BamHI 属于 II 型内切酶,可以在 DNA 分子上切割出粘性末端。
3.NEB 内切酶 BamHI 的特点EB 内切酶 BamHI 是一种从 Bacillus amyloliquefaciens 中分离得到的内切酶,可以识别并切割 G/AATTC 序列。
这种酶具有较高的特异性和切割效率,因此在分子生物学实验中得到了广泛应用。
4.NEB 内切酶 BamHI 的失活温度EB 内切酶 BamHI 的失活温度是指酶在高温下失去活性的温度。
对于NEB 内切酶 BamHI 而言,其失活温度为 95℃。
当温度达到 95℃时,NEB 内切酶 BamHI 的空间结构会发生改变,从而使其失去活性。
需要注意的是,失活并不意味着酶完全失去功能,当温度降低后,部分失活的酶仍可能恢复活性。
5.结论EB 内切酶 BamHI 是一种具有高特异性和高效率的内切酶,广泛应用于分子生物学实验中。
该酶的失活温度为 95℃,当温度达到这一数值时,酶会失去活性。
NEB常用酶介绍

NEB常用酶介绍NEB(New England Biolabs)是一个位于美国的生命科学公司,致力于研究和开发分子生物学工具。
NEB是全球领先的酶制造商之一,其产品被广泛应用于分子生物学、基因工程和遗传学等领域。
下面将介绍几种NEB常用的酶。
1. EcoRI:EcoRI是一种限制性内切酶,它能够识别GGATCC序列并将DNA切割成两段。
这种酶广泛应用于DNA的定量分析、基因克隆和DNA序列分析等领域。
2. Taq DNA聚合酶:Taq DNA聚合酶是一种热稳定的DNA聚合酶,能够在高温(最高可达95℃)下工作。
由于其特殊的酶活性和热稳定性,Taq DNA聚合酶广泛应用于PCR(聚合酶链反应)技术,用于DNA扩增和基因分析。
3.T4DNA连接酶:T4DNA连接酶可以将DNA分子连接起来,形成DNA链的连续片段。
这种酶在基因克隆、DNA重组和DNA构建等实验中被广泛应用。
4. RNase H:RNase H是一种核酸内切酶,能够特异性地降解RNA/DNA杂合体分子中的RNA链。
RNase H广泛用于亲和纯化、DNA杂交、基因表达调控和RNAi技术等领域。
5.T4DNA解旋酶:T4DNA解旋酶是一种能够解旋双链DNA的酶,可以将DNA解开成两条单链的DNA。
这种酶可以用于DNA复制、转录、重组和修复等过程的研究。
6. DNase I:DNase I是一种降解DNA的核酸酶,能够将DNA分解成较小的片段。
DNase I广泛应用于DNA纯化、DNase footprinting等实验中。
7.克隆充填酶:NEB提供多种克隆充填酶,用于将DNA片段克隆到克隆载体中。
这些酶通常具有3'→5'外切内切酶活性,可以将3'末端的不互补碱基去除,并将DNA片段插入到克隆载体中。
8.T4DNA聚合酶:T4DNA聚合酶是一种特殊的DNA聚合酶,具有内切酶活性和3'→5'外切酶活性。
这种酶可以在DNA合成的同时修复DNA末端的缺口,用于DNA修复和DNA重组等研究。
NEB 同尾酶表

连接互相匹配的粘性末端,产生新的酶切位点
通常连接互相匹配的粘性末端可以产生新的酶切位点。
以下组合是通过计算机程序计算出来的,尽管我们希望能尽量保证其准确性,但有些未经实验证实,因此不能保证100%的可行性。
如果有同裂酶存在,则只列出其中的一个酶。
本表中列出的切酶均为已商品化的切酶。
识别序列不够严谨的切酶(例如,识别多个序列的酶)只在括号标出相关的序列,但注意这些酶不只切割该序列。
"-"表示连接产物不能被再切割。
NEB公司Q5聚合酶说明书

PCR Using Q5® High-Fidelity DNA Polymerase (M0491)https:///protocols/2012/09/27/pcr-using-q5-high-fidelity-dna-polymerase-m0491 Protocol1. Please note that protocols with Q5® High-Fidelity DNA Polymerase may differ from protocols with otherpolymerases. Conditions recommended below should be used for optimal performance.Reaction Setup:We recommend assembling all reaction components on ice and quickly transferring the reactions to a thermocycler preheated to the denaturation temperature (98°C). All components should be mixed prior to use. Q5 High-Fidelity DNA Polymerase may be diluted in 1X Q5 Reaction Buffer just prior to use in order to reduce pipetting errors.Notes: Gently mix the reaction. Collect all liquid to the bottom of the tube by a quick spin if necessary. Overlay the sample with mineral oil if using a PCR machine without a heated lid. Transfer PCR tubes to a PCR machine and begin thermocycling.Thermocycling Conditions for a Routine PCR:General Guidelines:Template:Use of high quality, purified DNA templates greatly enhances the success of PCR. Recommended amounts of DNA template for a 50 µl reaction are as follows:1. Primers:Oligonucleotide primers are generally 20–40 nucleotides in length and ideally have a GC content of 40–60%.Computer programs such as Primer3 can be used to design or analyze primers. The best results are typically seen when using each primer at a final concentration of 0.5 µM in the reaction.2. Mg++ and additives:Mg++ concentration of 2.0 mM is optimal for most PCR products generated with Q5 High-Fidelity DNA Polymerase.When used at a final concentration of 1X, the Q5 Reaction Buffer provides the optimal Mg++concentration.Amplification of some difficult targets, like GC-rich sequences, may be improved by the addition of 1X Q5 High GC Enhancer. The Q5 High GC Enhancer is not a buffer and should not be used alone. It should be added only to reactions with the Q5 Reaction Buffer when other conditions have failed.3. Deoxynucleotides:Th e final concentration of dNTPs is typically 200 μM of each deoxynucleotide. Q5 High-Fidelity DNA Polymerase cannot incorporate dUTP and is not recommended for use with uracil-containing primers or templates.4. Q5 High-Fidelity DNA Polymerase concentration:We generally recommend using Q5 High-Fidelity DNA Polymerase at a final concentration of 20 units/ml (1.0 unit/50 μl reaction). However, the optimal concentration of Q5 High-Fidelity DNA Polymerase may vary from 10–40 units/ml (0.5–2 units/50 μl reaction) depending on amplicon length and difficulty. Do not exceed 2 units/50 μl reaction, especially for amplicons longer than 5 kb.5. Buffers:The 5X Q5 Reaction Buffer provided with the enzyme is recommended as the first-choice buffer for robust,high-fidelity amplification. For difficult amplicons, such as GC-rich templates or those with secondary structure, the addition of the Q5 High GC Enhancer can improve reaction performance. The 5X Q5 Reaction Buffer isdetergent-free and contains 2.0 mM MgCl2 at the final (1X) concentration.6. Denaturation:An initial denaturation of 30 seconds at 98°C is sufficient for most amplicons from pure DNA templates. Longer denaturation times can be used (up to 3 minutes) for templates that require it.During thermocycling, thedenaturation step should be kept to a minimum. Typically, a 5–10 second denaturation at 98°C is recommended for most templates.7. Annealing:Optimal annealing temperatures for Q5 High-Fidelity DNA Polymerase tend to be higher than for other PCR polymerases. The NEB T m Calculator should be used to determine the annealing temperature when using this enzyme. Typically, use a 10–30 second annealing step at 3°C above the T m of the lower T m primer. A temperature gradient can also be used to optimize the annealing temperature for each primer pair.For high T m primer pairs, two-step cycling without a separate annealing step can be used (see note 12).8. Extension:The recommended extension temperature is 72°C. Extension times are generally 20–30 seconds per kb for complex, genomic samples, but can be reduced to 10 seconds per kb for simple templates (plasmid, E. coli, etc.) or complex templates < 1 kb. Extension time can be increased to 40 seconds per kb for cDNA or long, complex templates, if necessary.A final extension of 2 minutes at 72°C is recommended.9. Cycle number:Generally, 25–35 cycles yield sufficient product. For genomic amplicons, 30-35 cycles are recommended.10. 2-step PCR:When primers with annealing temperatures ≥ 72°C are used, a 2-step thermocycling protocol (combiningannealing and extension into one step) is possible.11. Amplification of long products:When amplifying products > 6 kb, it is often helpful to increase the extension time to40–50 seconds/kb.12. PCR product:The PCR products generated using Q5 High-Fidelity DNA Polymerase have blunt ends. If cloning isthe next step, then blunt-end cloning is recommended. If T/A-cloning is preferred, the DNA should be purified prior to A-addition, as Q5 High-Fidelity DNA Polymerase will degrade any overhangs generated.Addition of an untemplated -dA can be done with Taq DNA Polymerase (NEB #M0267 ) or Klenow exo– (NEB #M0212 ).。
NEB-T4 DNA Lagase注意事项

一、关于NEB M0202 T4 DNA 连接酶的使用方法和常见问题及解答1、产品特性和使用方法产品说明:该酶催化契合的双链DNA或RNA的5'-磷酸末端和3'-羟基末端形成磷酸二酯键。
该酶不仅能够催化平滑末端或粘性末端DNA之间的连接,还可以修复双链DNA、RNA或DNA/RNA杂交双链中的单链切口(1)。
--------------------------------------------------------------------------------来源:源于E.coli C600 pcl857 pPLc28 lig8 (2)。
应用:限制性酶切片段的克隆(3)。
将linker或adapters 连接到DNA片段的平滑末端。
反应缓冲液:1X T4 DNA Ligase Buffer:[50 mM Tris-HCl, 10 mM MgCl2, 10 mM DTT, 1 mM ATP, 25 µg/ml BSA, (pH 7.5 @ 25°C)]。
推荐DNA浓度(0.1 - 1 µM 的5′末端)。
使用注意:ATP是反应必须的辅因子。
这一点与要求NAD作辅因子的大肠杆菌DNA连接酶不同。
如在反应体系中补加1 mM的ATP,连接反应还可以在NEB提供的四种限制性内切酶缓冲液(NEBuffers)或在T4 DNA 多聚核苷酸激酶缓冲液中进行。
质量保证:无核酸内、外切酶污染。
每批T4 DNA连接酶均通过模拟克隆实验进行检测,该实验可以检测出待连接DNA 末端的任何破坏。
结果表明>99.9%的DNA末端未受任何降解。
单位定义(粘性末端活性单位):在20µl连接反应体系、0.12µM (300µg/ml)的5'-末端浓度条件下,16°C反应30分钟,能使50% 的经Hind Ⅲ消化的λDNA片段连接所需的酶量定义为一个NEB单位。
NEB常用酶介绍

注意事项
与 E. coli DNA 连接酶需要 NAD+ 作辅因子不同,T4 DNA 连接酶的辅因子是 ATP。
稀释后的 T4 DNA 连接酶应于 -20℃、50% 甘油贮存缓冲液(稀释缓冲液 A,NEB #B8001)中保 存。如稀释后马上使用,也可用 1X T4 DNA 连接酶反应缓冲液稀释。
NcoI
识别位点
在不同反应缓冲液的活性 NEBuffer 1NEBuffer 3.1: 100% CutSmart Buffer: 100%
特性 重组酶、省时酶。
反应条件 NEBuffer 3.1,37℃。 热失活:80℃ 20 分钟。
浓度 10,000 和 50,000 units/ml。
甲基化敏感性 对 dam、dcm 和哺乳动物 CpG 甲基化均不敏感。
NotI
识别位点
在不同反应缓冲液的活性 NEBuffer 1.1: <10% NEBuffer 2.1: 50% NEBuffer 3.1: 100% CutSmart Buffer: 25% 特性 重组酶、省时酶。 反应条件 NEBuffer 3.1,37℃。 热失活:65℃ 20 分钟。 浓度 10,000 和 50,000 units/ml。 甲基化敏感性 对哺乳动物基因组 DNA CpG 甲基化敏感。 注意事项 彻底消化超螺旋质粒需要的 NotI 酶量为消化线性 DNA 的 5 倍。
neb内切酶bamhi失活温度

Neb内切酶BamHI失活温度引言内切酶(endonuclease)是一类能够在DNA双链的特定序列中切割磷酸二酯键的酶。
内切酶在分子生物学和基因工程中起着至关重要的作用,被广泛应用于DNA的切割、克隆以及DNA测序等实验中。
其中,Neb内切酶BamHI是一种常用的内切酶,能够识别并切割DNA序列GGATCC。
然而,BamHI的活性受到温度的影响,本文将探讨BamHI的失活温度及其相关影响因素。
BamHI的特性BamHI是一种来自于Bacillus amyloliquefaciens H的内切酶,属于限制性内切酶(restriction endonuclease)家族。
它能够识别并切割DNA序列GGATCC,切割后的产物为5’ G^GATCC 3’和3’ CCTAG^G 5’,其中^表示切割位点。
BamHI是一种双链DNA内切酶,它在酶-底物结合后通过切割磷酸二酯键将DNA分子切割成两段。
BamHI在切割位点的5’和3’两侧均有特异性识别序列,这种特异性使得BamHI能够精确地切割DNA序列。
BamHI的活性与失活温度BamHI的活性受到温度的影响,高温能够使其失活。
一般来说,限制性内切酶的活性与温度呈反相关关系,即随着温度的升高,酶的活性逐渐降低。
针对BamHI的失活温度,研究表明其失活温度约为65摄氏度。
当温度超过65摄氏度时,BamHI的活性会迅速下降,甚至完全失活。
因此,在使用BamHI进行DNA切割实验时,需要控制反应体系的温度在65摄氏度以下,以确保BamHI的活性。
影响BamHI失活温度的因素BamHI的失活温度受到多种因素的影响,下面将介绍其中的几个重要因素。
pH值pH值是影响酶活性的重要因素之一。
研究表明,BamHI的最适pH为7.5-8.0,而在酸性或碱性条件下,BamHI的活性会降低。
因此,在进行BamHI酶切实验时,需要调节反应体系的pH值,以维持酶的最佳活性。
离子浓度离子浓度也是影响BamHI活性的重要因素之一。
neb内切酶bamhi失活温度

neb内切酶bamhi失活温度摘要:1.内切酶Neb的作用及应用领域2.BamHI失活温度的实验研究与应用3.内切酶Neb与BamHI在生物技术中的联合应用4.总结:内切酶Neb与BamHI在生物技术研究中的重要性正文:一、内切酶Neb的作用及应用领域内切酶Neb,全名为Neuraminidase from Bacillus amyloliquefaciens,是一种来源于枯草杆菌(Bacillus amyloliquefaciens)的酸性糖苷酶。
内切酶Neb具有广泛的应用领域,尤其在生物技术研究和基因工程中具有重要价值。
它能够水解糖苷键,将糖链从糖蛋白或其他糖基化分子上切割下来,从而为糖基化修饰和生物活性研究提供便利。
二、BamHI失活温度的实验研究与应用BamHI是一种常用的限制性内切酶,来源于芽孢杆菌(Bacillus amyloliquefaciens)。
在实验过程中,研究人员发现BamHI具有一定的失活温度,这意味着在过高温度下,BamHI的酶活性会受到影响。
这一发现对于实验室操作和实际应用中酶切反应条件的选择具有重要意义。
三、内切酶Neb与BamHI在生物技术中的联合应用内切酶Neb和BamHI在生物技术研究中具有密切的联系。
例如,在基因工程中,研究人员需要将目的基因与载体DNA连接,这时就需要使用限制性内切酶(如BamHI)来切割载体DNA,以产生适当的粘性末端。
随后,通过连接目的基因与载体DNA的粘性末端,实现基因的插入和表达。
在这个过程中,内切酶Neb可以用于切割糖链,从而简化糖基化分析。
四、总结内切酶Neb和BamHI在生物技术研究中具有重要作用。
内切酶Neb作为酸性糖苷酶,为糖基化修饰和生物活性研究提供便利;而BamHI作为一种限制性内切酶,则在基因工程中发挥着关键作用。
了解它们的失活温度等特性,有助于科研人员在实验过程中优化操作条件,提高实验成功率。
neb fs片段化酶原理

neb fs片段化酶原理
NEB FS片段化酶是New England Biolabs公司生产的一种酶,
它的全称是NEBNext® FFPE DNA Repair Mix。
它的主要作用是修
复和片段化来自于固定、石蜡包埋组织标本中的DNA。
这种酶的原
理涉及到几个方面:
1. DNA修复,NEB FS片段化酶中包含具有修复DNA损伤能力的
酶和辅因子。
在固定、石蜡包埋组织标本中,DNA常常受到化学修
饰和损伤,这会影响后续的DNA测序或PCR扩增实验。
NEB FS片段
化酶中的成分可以修复这些损伤,使DNA恢复到适合进行后续实验
的状态。
2. DNA片段化,NEB FS片段化酶还包含具有特定片段化酶活性
的成分。
在修复DNA的同时,这些成分可以将DNA分解成较短的片段,这有助于提高DNA的可测序性和扩增性。
通过对DNA进行片段化,可以更好地适应后续的实验操作。
NEB FS片段化酶的原理基于对固定、石蜡包埋组织标本中DNA
特殊性质的理解,通过修复和片段化来提高这些DNA样本的适用性。
在实际操作中,研究人员可以根据NEB FS片段化酶的原理,合理选
择实验条件和操作步骤,以达到最佳的实验效果。
这种酶的原理和应用为固定、石蜡包埋组织标本中DNA的后续分子生物学研究提供了重要的技术支持。
NEB T4DNA连接酶

T4 DNA 连接酶货号规格价格#M0202L 100,000 units 3,029.00元#M0202M (高浓度5x)100,000 units 3,029.00元#M0202S 20,000 units 699.00元#M0202T (高浓度5x)20,000 units 699.00元#M0202V 10,000 units 349.00元■ 提高反应效率■粘性末端和平齐末端均可连接■限制性酶切片段的克隆(3)■将linker 或adapter 连接到DNA 片段的平齐末端■室温或16 ℃均有活性概述该酶催化契合的双链DNA或RNA的5´-磷酸末端和3´-羟基末端形成磷酸二酯键。
该酶不仅能够催化平滑末端或粘性末端DNA之间的连接,还可以修复双链DNA、RNA或DNA/ RNA杂交双链中的单链切口(1) 。
来源纯化自E. coli C600 pcl857 pPLc28 lig8(2) 。
反应条件1 X T4 DNA 连接酶缓冲液[ 50 mM Tris-HCl(pH 7.5 @ 25 ℃),10 mM MgCl2,10 mM DTT,1 mM ATP]。
推荐DNA 5' 末端浓度为0.1-1.0 μM。
质保声明无核酸内切酶和外切酶污染。
每批T4 DNA 连接酶均通过模拟克隆实验进行检测,该实验可以检测出连接后DNA 末端的任何损伤。
结果表明超过99.9% 的DNA 末端保持完好。
单位定义(粘性末端活性单位)1 单位指在20 μl 1X T4 DNA 连接酶反应缓冲液中,16 ℃反应条件下,30 分钟能使50% 的经HindIII 消化的λDNA 片段[5' 端浓度为0.12 μM (300 μg/ml)]连接所需的酶量。
浓度400,000 units/ml 和2,000,000 units/ml。
贮存条件50 mM KCl, 10 mM Tris-HCl (pH 7.4), 0.1 mM EDTA, 1 mM DTT, 200 μg/ml BSA 和50% 甘油,-20°C 贮存。
NEB常用酶介绍

NotI
识别位点
在不同反应缓冲液的活性 NEBuffer 1.1: <10% NEBuffer 2.1: 50% NEBuffer 3.1: 100% CutSmart Buffer: 25% 特性 重组酶、省时酶。 反应条件 NEBuffer 3.1,37℃。 热失活:65℃ 20 分钟。 浓度 10,000 和 50,000 units/ml。 甲基化敏感性 对哺乳动物基因组 DNA CpG 甲基化敏感。 注意事项 彻底消化超螺旋质粒需要的 NotI 酶量为消化线性 DNA 的 5 倍。
NcoI
识别位点
在不同反应缓冲液的活性 NEBuffer 1.1: 100% NEBuffer 2.1: 100% NEBuffer 3.1: 100% CutSmart Buffer: 100%
特性 重组酶、省时酶。
反应条件 NEBuffer 3.1,37℃。 热失活:80℃ 20 分钟。
浓度 10,000 和 50,000 units/ml。
室温连接反应 为方便起见,连接反应可以在室温(20-25 ℃)条件下进行。粘性末端连接:在 20 μl 反应体系中加入 1 μl T4 DNA 连接酶,反应 10 分钟。平齐末端连接:在 20 μl 反应体系中加入 1 μl T4 DNA 连接酶反应 2 小时,或者加入 1 μl 高浓度 T4 DNA 连接酶反应 10 分钟。 另外,NEB 快速连接试剂盒 [NEB #M2200S(30 次反应),或 NEB #M2200L(150 次反应)] 可在 室温下 5 分钟内完成平齐和粘性末端的连接。
单位定义(粘性末端活性单位) 1 单位指在 20 μl 1X T4 DNA 连接酶反应缓冲液中,16℃ 反应条件下,30 分钟能使 50% 的经 Hin dⅢ 消化的 λDNA 片段 [ 5´ 端浓度为 0.12 μM(300 μg/ml)] 连接所需的酶量。
neb内切酶bamhi失活温度

neb内切酶bamhi失活温度摘要:I.引言- 介绍neb 内切酶和bamhi 失活温度II.neb 内切酶的概述- 解释neb 内切酶的功能和作用III.bamhi 失活温度的介绍- 描述bamhi 失活温度的定义和重要性IV.neb 内切酶bamhi 失活温度的关系- 分析neb 内切酶和bamhi 失活温度之间的联系V.实际应用中的考虑因素- 讨论在实际应用中如何考虑neb 内切酶bamhi 失活温度的影响VI.结论- 总结并强调neb 内切酶bamhi 失活温度的重要性正文:I.引言eb 内切酶是一种在分子生物学实验室中广泛使用的工具,能够识别特定的DNA 序列并在此处切割双链DNA。
而bamhi 失活温度则是指在特定的条件下,neb 内切酶bamhi 无法正常工作的温度。
理解这两者之间的关系对于实验的顺利进行至关重要。
II.neb 内切酶的概述eb 内切酶,全称为“N 末端缺失的内切酶”,来源于大肠杆菌的λ噬菌体。
它具有高度的特异性,只切割具有特定序列的DNA,从而在分子生物学实验中发挥着重要的作用。
neb 内切酶能够帮助研究人员在特定的位置剪切DNA,进而实现基因编辑、克隆等操作。
III.bamhi 失活温度的介绍bamhi 失活温度是指在特定的条件下,neb 内切酶bamhi 无法正常工作的温度。
这是由于酶的活性受温度的影响,当温度过高或过低时,酶的构象发生变化,导致其无法正常发挥作用。
因此,了解bamhi 失活温度对于实验条件的设定至关重要。
IV.neb 内切酶bamhi 失活温度的关系eb 内切酶bamhi 失活温度与酶的活性密切相关。
在一定的温度范围内,酶活性随着温度的升高而增加,这个范围通常被称为酶的最适工作温度。
然而,当温度超过最适工作温度的上限时,酶的活性会急剧下降,直至失活。
因此,掌握neb 内切酶bamhi 失活温度,有助于确保实验过程中酶的活性,从而保证实验结果的准确性。
neb fs片段化酶原理

neb fs片段化酶原理全文共四篇示例,供读者参考第一篇示例:NEB FS片段化酶是一种高效的DNA切割酶,常用于分子生物学实验中的DNA片段化处理。
它具有特定的工作原理,能够将DNA链切割为特定长度的片段,从而方便后续的分析和操作。
本文将详细介绍NEB FS片段化酶的原理及应用。
NEB FS片段化酶是一种双链DNA特异性酶,通过在DNA双螺旋结构中寻找特定的识别序列来实现切割作用。
NEB FS片段化酶的原理基于其结构特点和作用机制:该酶是一种核酸内切酶,能够识别特定的DNA序列,并在该序列的特定位点上进行切割,将DNA链断裂为两个片段。
NEB FS片段化酶的活性依赖于其特定的结构域和酶活性中心。
在实验条件下,NEB FS片段化酶可以被活化或抑制,从而调控其在DNA双链上的切割活性。
由于其高度特异性的识别能力,NEB FS片段化酶可以精确地切割目标DNA链,不会影响非特定序列。
NEB FS片段化酶的应用非常广泛,特别适用于DNA片段化处理。
在实验中,研究人员可以通过NEB FS片段化酶将DNA链切割为特定长度的片段,用于构建基因工程载体、DNA电泳分析、DNA测序等多种实验操作。
NEB FS片段化酶还可以配合其他酶类在PCR、限制性内切酶切割、DNA连接等实验中发挥作用。
第二篇示例:neb fs片段化酶,全称为New England Biolabs Fragmentation Sequence (NEB FS) Enzymes,是一种常用于DNA分子切割和片段化的酶,具有高度特异性和高效率。
这种酶的原理基于其结构和功能特点,通过特定的作用方式,可以将DNA分子切割成不同大小的片段,是分子生物学研究中不可或缺的工具之一。
NEB FS片段化酶的原理主要包括以下几个方面:作用机制、酶的结构和功能、酶的应用及优势。
在这篇文章中,我们将从这几个方面详细介绍NEB FS片段化酶的原理,并探讨其在科研和生物工程领域的重要性和应用价值。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
NEB常用酶介绍
NEB(New England Biolabs)是一家生物技术公司,专注于酶和相关
试剂的研发和生产。
该公司开发了许多常用酶,其中一些被广泛应用于分
子生物学和生物技术领域。
下面是一些常见的NEB酶的介绍。
1. 限制性内切酶(Restriction Enzymes):限制性内切酶是一类能
够识别特定DNA序列并在特定位点切割DNA的酶。
NEB生产了许多常见的
限制性内切酶,如EcoRI、HindIII、BamHI等。
这些酶在DNA分析、克隆
以及基因组工程等领域中广泛应用。
2. DNA连接酶(DNA Ligases):DNA连接酶能够将两条DNA分子连
接起来,形成一个连续的DNA链。
NEB提供了多种DNA连接酶,如T4 DNA
连接酶、Quick DNA连接酶等。
这些酶在DNA克隆和结构修复等实验中起
到至关重要的作用。
3. 反转录酶(Reverse Transcriptases):反转录酶能够将RNA模
板逆转录合成DNA,从而产生相应的cDNA。
NEB开发了多种反转录酶,如
M-MuLV反转录酶、AMV反转录酶等。
这些酶广泛应用于转录组学、基因表
达研究以及RT-PCR等实验中。
4. 核酸聚合酶(DNA Polymerases):核酸聚合酶是一类能够在DNA
合成过程中将新的核苷酸单元加入到已存在的DNA链上的酶。
NEB提供了
多种高质量的核酸聚合酶,如Phusion DNA聚合酶、Taq DNA聚合酶等。
这些酶在PCR、DNA扩增和DNA测序等实验中被广泛使用。
5. 电泳酶(Nucleases):电泳酶能够在DNA或RNA的特定位置切割
核酸链。
NEB生产的电泳酶包括RNase A等。
这些酶广泛应用于核酸纯化、RNA降解实验以及电泳分析中。
6. 磷酸二酯酶(Phosphatases):磷酸二酯酶能够催化磷酸二酯键
的水解反应,从而使DNA或RNA失去3'末端的磷酸基团。
NEB提供了多种
磷酸二酯酶,如CIP碱性磷酸酯酶等。
这些酶在DNA修饰和连接实验中起
到重要作用。
7. 酶标记酶(DNA and RNA Labeling):NEB提供了多种酶标记酶,如RNA多聚核苷酸连接酶、DNA 3'末端标记酶等。
这些酶广泛应用于DNA
和RNA的标记、探针制备以及核酸杂交技术中。
8.等电聚焦酶(Isoenzymes):NEB生产了许多等电聚焦酶,用于蛋
白质的等电聚焦分离与纯化实验中。
以上介绍了NEB常见的一些酶及其应用。
这些酶的优点包括高纯度、
高效率和广泛的适用性。
通过使用NEB酶,研究人员可以在实验中获得可
靠的结果,并加速他们的研究进程。
NEB酶的质量保证,使其成为科研人
员和生物技术公司们首选的酶供应商。
