用NCBI查找基因序列教程
NCBI 良心教程,寻找基因转录本序列及相关编码蛋白

NCBI 良心教程,寻找基因转录本序列及相
3.点击search 进入该界面关编码蛋白
提及NCBI 我们大家都不陌生,研究物种基因功能可都靠它呢。
在NCBI 的快速搜索下,基因转录本序列以及相关编码蛋白信息那也是手到擒来。
然而当我们查找某个基因的相关信息时,就会发现该基因有很多个转录本。
所以尽管基因查询很简单,但要精确定位到自己所需的转录本核酸序列及蛋白信息仍是一个问题,今天小编就手把手教大家如何通过经NCBI 搞定此难题,以栗子为主,包学包会。
步骤
1.打开pubmed:https:///pubmed
2.选择基因并输入你要查找的基因名称,这里以TP53 基因为例:
4.点击第一个TP53 进入以下界面
5.下拉到NCBI Reference Sequences (RefSeq)区域,mRNA and Protein(s)下显示的就是该基因的转录本及其编码的蛋白:
6.一直下拉会发现该基因共有15 个转录本
案例
以第一个转录本为例来告诉大家怎样查找转录本的核苷酸序列及其编码的蛋白质的氨基酸序列。
7.点击NM_000546.5 后会出现以下界面
8.下拉到ORIGIN 区域即为该转录本的核苷酸序列
9.返回到以下界面
10.点击NP_000537.3 后会看到以下界面
11.下拉到ORIGIN 区域即为该转录本编码的蛋白质的氨基酸序列
获得该转录本的ORF 序列12.返回到以下界面
13. 点击CCDS11118.1 后会出
现
14.下拉到Nucleotide Sequence 区域即为该转录本的ORF 序列,Translation 区域是该转录本编码的蛋白质的氨基酸序列。
ncbi使用指导

ncbi使用指导导言:NCBI(National Center for Biotechnology Information)是美国国家生物技术信息中心,是全球最大的生物信息学数据库。
NCBI提供了丰富的生物学资源,包括基因序列、蛋白质序列、科学文献等。
本文将为您介绍如何使用NCBI来获取和利用生物学信息。
一、注册NCBI账号在使用NCBI之前,首先需要注册一个账号。
在NCBI官方网站()的主页上,点击右上角的“Sign In”按钮,并选择“Register for an NCBI account”。
根据提示提供相关信息,完成注册流程。
二、搜索和获取基因序列1. 打开NCBI网站后,点击主页上的“Search”按钮,进入搜索页面。
2. 在搜索框中输入您感兴趣的基因名称或者序列标识符,如“BRCA1”。
3. 点击“Search”按钮进行搜索。
4. 在搜索结果中,选择您需要的基因序列,点击链接进入该基因的详细信息页面。
5. 在详细信息页面中,您可以获取该基因的序列信息,可以下载或拷贝该序列以供后续分析使用。
三、检索科学文献1. 在NCBI主页上方的搜索框中,选择“PubMed”为搜索目标。
2. 输入您感兴趣的科学文献关键词,如“cancer treatment”。
3. 点击搜索按钮进行检索。
4. 在搜索结果中,选择您需要的文献,点击链接进入该文献的详细页面。
5. 在详细页面中,您可以阅读文章摘要,并根据需要下载全文或进行其他操作。
四、利用NCBI工具进行序列分析1. 在NCBI主页上方的菜单栏中,选择“Tools”进行工具选择。
2. 根据您的需求选择相应的工具,如“BLAST”进行序列比对分析。
3. 进入选择的工具页面后,按照提示上传或输入您的序列数据。
4. 设定参数并运行分析。
5. 分析完成后,您可以查看比对结果、序列保守性等信息,并进行进一步的分析和解释。
五、订阅NCBI数据库更新信息1. 在NCBI主页上方菜单栏中选择“NCBI Account”。
如何在NCBI查找基因序列

如何查找基因序列?——在Genbank中寻找目的基因的实例1. 根据文献搞reasearch肯定要读文献的,如果你曾经在文献中看到过你感兴趣的基因,而且文中还提到了该基因在Genbank中的ID号,那就好办了,直接打开,在Search后的下拉框中选择Nucleotide,把Genbank ID号输入GO前面的文本框中,点“GO”,就可以找到他了。
举例说明,例如:在2003年JBC的文章(Conditional Knock-out of Integrin-linked Kinase Demonstrates an Essential Role in Protein Kinase B/Akt Activation)中出现了“calreticulin (GenBank accession number gi 16151096)”,那么把“16151096”输入GO前面的文本框中,点“GO”,就可以找到该基因了(当然包括基因序列等相关信息)。
见附图在出现了检索结果界面(下图)后,直接点击红箭头所指的AY047586就可以看到基因的相关信息了...这里需要指出一下,在显示基因的页面右侧有一个Link,点击后出现一个小菜单,里面是与该基因相关的链接,很有用的,值得一个一个地去看看,这里我就不多说了。
点击 AY047586后出现的界面如下:下面还有......如果你只想获得序列(例如去设计PCR引物的时候),那就可以选择FASTA,这样就得到了FASTA格式的序列文件,没有其他数字和格式的干扰。
这就是FASTA格式的序列:2. 根据已经获得的基因的相关信息进行查找(待续......)如果只是知道基因的名字,怎么查序列呢?还是举例说明,比如我想做的基因名称是人的VEGF基因,那么怎么在Genbank中找到它呢?还是一步一步来...打开/在search后面的下拉框中选择Gene,然后在中间的文本框中输入基因名称“VEGF”,点击GO...搜索结果出来了,let me see... 啊,怎么这么多?689条,哪一条是我想要的基因呢?(作者注:这也许是大多数人对Genbank的第一印象,即东西太多了,不知道是哪个。
ncbi使用方法

ncbi使用方法(原创版4篇)《ncbi使用方法》篇1CBI(National Center for Biotechnology Information)是美国国家生物技术信息中心的缩写,它提供了许多生物学和生命科学相关的数据库和工具。
以下是使用NCBI 的一些基本方法:1. 核酸序列数据库(Nucleotide Sequence Database):在NCBI 主页上,可以选择核酸序列数据库,输入序列名称或序列号,然后点击“Search”按钮即可查询序列信息。
2. 蛋白质序列数据库(Protein Sequence Database):在NCBI 主页上,可以选择蛋白质序列数据库,输入蛋白质名称或蛋白质号,然后点击“Search”按钮即可查询蛋白质信息。
3. 基因组数据库(Genome Database):在NCBI 主页上,可以选择基因组数据库,输入基因组名称或基因组号,然后点击“Search”按钮即可查询基因组信息。
4. 代谢通路数据库(Metabolic Pathway Database):在NCBI 主页上,可以选择代谢通路数据库,输入代谢通路名称或代谢通路号,然后点击“Search”按钮即可查询代谢通路信息。
5. 生物投影数据库(BioProject Database):在NCBI 主页上,可以选择生物投影数据库,输入生物投影名称或生物投影号,然后点击“Search”按钮即可查询生物投影信息。
6. 序列比对工具(Sequence Alignment Tool):NCBI 提供了一款名为“Clustal Omega”的序列比对工具,可以在NCBI 主页上使用该工具进行序列比对。
7. 基因表达数据库(Gene Expression Database):NCBI 提供了一款名为“GEO”的基因表达数据库,可以在NCBI 主页上查询基因表达数据。
8. 蛋白质结构数据库(Protein Structure Database):NCBI 提供了一款名为“RCSB PDB”的蛋白质结构数据库,可以在NCBI 主页上查询蛋白质结构信息。
NCBI良心教程寻找基因转录本序列及相关编码蛋白

NCBI良心教程寻找基因转录本序列及相关编码蛋白NCBI (National Center for Biotechnology Information) 是一个全球顶级的生物医学数据库,提供了大量的基因序列和相关的生物信息。
在这个教程里,我们将介绍如何在NCBI中基因转录本序列以及相关编码蛋白。
第一步:访问NCBI网站首先,打开你的浏览器,并输入 "NCBI",然后点击按钮。
在结果中,选择并点击进入 "NCBI - National Center for Biotechnology Information" 的官方网站。
第二步:选择数据库在NCBI的官方网站中,你可以看到很多不同的数据库。
对于本教程,我们将使用 "GenBank" 数据库来基因转录本序列以及相关编码蛋白。
点击页面左上角的 "Databases" 菜单,然后选择 "Nucleotide" 数据库,它是用于基因序列的数据库。
第三步:基因转录本序列在 "Nucleotide" 页面中,你会看到一个框,可以输入你感兴趣的基因的名称或相关关键词。
在框中输入基因的名称或相关关键词,然后点击按钮。
此时,你将看到与该基因相关的转录本序列的结果列表。
除了查找基因转录本序列,你还可以使用NCBI相关的编码蛋白质序列。
在 "Nucleotide" 页面中,选择 "Protein" 或 "Protein database" 选项卡。
在框中输入与你感兴趣的蛋白质相关的关键词,并点击按钮。
此时,你将看到与该蛋白质相关的转录本或基因的结果列表。
点击结果列表中感兴趣的蛋白质,你将进入蛋白质的详细页面。
在这个页面上,你可以找到该蛋白质的序列以及其他相关信息。
总结:在NCBI中基因转录本序列及相关编码蛋白可以通过以下步骤完成:1.访问NCBI官方网站;2.选择 "GenBank" 数据库;3.你感兴趣的基因的名称或相关关键词;4.点击结果中的转录本,查看序列和其他详细信息;5.如果你想相关编码蛋白质,选择 "Protein" 选项卡,并输入相关关键词;6.点击结果中的蛋白质,查看蛋白质序列和其他相关信息。
NCBI如何查找序列

1下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:/mapview/index.html 在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码、序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
现在普遍采用的是最上面的那个序列,这一条是世界范围的生物科学家用计算机合成的一个序列。
我也推荐大家使用这个序列。
4.点击上述三条序列第一条序列(即 reference)对应的"Genes seq",出现新的页面,页面下方为:5.点击上图出现的“Download/View Sequence/Evidence ”,即下载查看序列等功能,结果如图所示:先对上面这张图做点简要的说明,在 Sequence Format(序列输出格式)后面是一个下拉式选择菜单,默认的为 FASTA 格式,还有一个是 GenBank 格式。
我推荐大家选择 GenBnak格式,因为这个格式提供了很多该基因的信息,而 FASTA格式只有基因序列。
6.在 Sequence Format 后选择 GenBank,然后点击下面的 Display,目的基因的相关信息和序列就出现在眼前了。
点击后如图所示(网页较大,只抓取一小部分以作示范):在上述打开的网页中,你可以看到基因长度,基因序列,以及这个基因是如何被报道出来的等各种信息。
你会看到: mRNAjoin(3598..3678,3841..4031,5090..5203,5911..6057, 7803..8394) 这代表了从基因的 3598位开始就是转录区了,即我们常说的 mRNA 片断,由于内含子的存在,所以 mRNA 在DNA 序列上分成了几段。
教你使用 NCBI 查找DNA、mRNA、cDNA、Protein、promoter、引物设计、BLAST 序列比对

一步一步教你使用NCBI 查找DNA、mRNA、cDNA、Protein、promoter、引物设计、BLAST 序列比对等最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在NCBI 上都可以方便的找到答案。
现在我就结合我自己使用NCBI的一些经历(经验)跟大家交流一下BCBI 的使用。
希望大家都能发表自己的使用心得,让我们共同进步!我分以下几个部分说一下NCBI 的使用:Part one 如何查找基因序列、mRNA、PromoterPart two 如何查找连续的mRNA、cDNA、蛋白序列Part three 运用STS 查找已经公布的引物序列Part four 如何运用BLAST 进行序列比对、检验引物特异性特别感谢本版版主,将这个帖子置顶!从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友!请大家对以下我发表的内容提出自己的意见。
关于NCBI 其他方面的使用也请水平较高的战友给予补充First of all,还是让我们从查找基因序列开始。
第一部分利用Map viewer 查找基因序列、mRNA 序列、启动子(Promoter)下面以人的IL6(白细胞介素6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:/mapview/index.html在search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene 前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
NCBI如何查找序列

1下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:/mapview/index.html 在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码、序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
现在普遍采用的是最上面的那个序列,这一条是世界范围的生物科学家用计算机合成的一个序列。
我也推荐大家使用这个序列。
4.点击上述三条序列第一条序列(即 reference)对应的"Genes seq",出现新的页面,页面下方为:5.点击上图出现的“Download/View Sequence/Evidence ”,即下载查看序列等功能,结果如图所示:先对上面这张图做点简要的说明,在 Sequence Format(序列输出格式)后面是一个下拉式选择菜单,默认的为 FASTA 格式,还有一个是 GenBank 格式。
我推荐大家选择 GenBnak格式,因为这个格式提供了很多该基因的信息,而 FASTA格式只有基因序列。
6.在 Sequence Format 后选择 GenBank,然后点击下面的 Display,目的基因的相关信息和序列就出现在眼前了。
点击后如图所示(网页较大,只抓取一小部分以作示范):在上述打开的网页中,你可以看到基因长度,基因序列,以及这个基因是如何被报道出来的等各种信息。
你会看到: mRNAjoin(3598..3678,3841..4031,5090..5203,5911..6057, 7803..8394) 这代表了从基因的 3598位开始就是转录区了,即我们常说的 mRNA 片断,由于内含子的存在,所以 mRNA 在DNA 序列上分成了几段。
一步一步教你使用NCBI查找DNA、mRNA、cDNA

一步一步教你使用 NCBI 查找DNA、mRNA、cDNA、...最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在 NCBI 上都可以方便的找到答案。
现在我就结合我自己使用 NCBI的一些经历(经验)跟大家交流一下 BCBI 的使用。
希望大家都能发表自己的使用心得,让我们共同进步!我分以下几个部分说一下 NCBI 的使用:Part one 如何查找基因序列、mRNA、PromoterPart two 如何查找连续的 mRNA、cDNA、蛋白序列Part three 运用 STS 查找已经公布的引物序列Part four 如何运用 BLAST 进行序列比对、检验引物特异性特别感谢本版版主,将这个帖子置顶!从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友!请大家对以下我发表的内容提出自己的意见。
关于NCBI 其他方面的使用也请水平较高的战友给予补充First of all,还是让我们从查找基因序列开始。
第一部分利用Map viewer 查找基因序列、mRNA 序列、启动子(Promoter)下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:/mapview/index.html在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码、序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
ncbi中查找基因序列的方法和三个号码
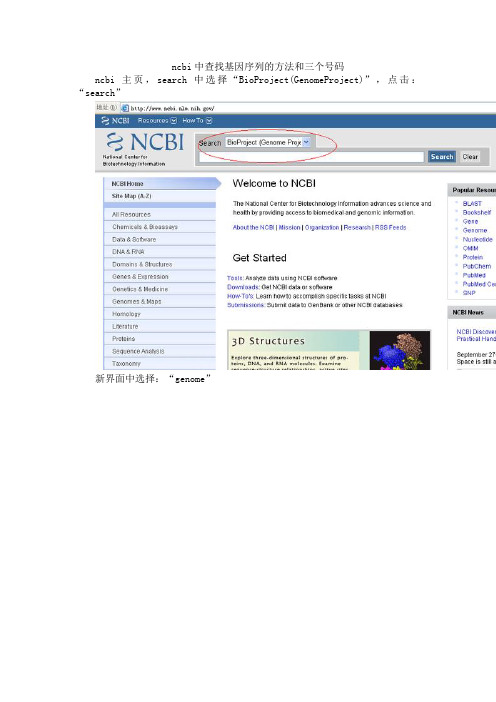
ncbi中查找基因序列的方法和三个号码
ncbi主页,search中选择“BioProject(GenomeProject)”,点击:“search”
新界面中选择:“genome”
新窗口中选择:“Prokaryotes”:
出现“genomeproject”选项,选择下拉框内的你要查找的菌的域名:例如:“Euryarchaeota(广古菌域)”
下拉滚动条,选择要下载基因组的菌株名,例如:MethanococcusvoltaeA3
点击对应的基因组号:
选择:NC_014222.1 进入MethanococcusvoltaeA3chromosome,completegenome
【注:若选择CP002057.1 可直接进入“MethanococcusvoltaeA3,completegenome”。
】
1.查找蛋白序列:
选择:Proteincoding: 1717 ,此界面上可以看到蛋白功能等方面的信息:
display选项中选择:“ProtienFASTA”,如果想下载序列,选择sendto 中的文件格式,保存即可。
2.查找核酸序列:
选择:GenBank: CP002057
然后选择页面右上角的sendto可以进行下载。
初步总结如上,NCBI的强大功能还要不断摸索!。
ncbi中查找基因序列的方法和三个登录号

ncbi 中查找基因序列的方法和三个号码中查找基因序列的方法和三个号码一.例子:查找酿酒酵母(Saccharomyces cerevisiae )里的海藻糖合成酶基因(tps1) 即可出现很多条目,找到Saccharomyces cerevisiae 的就是NC_001134了,点击后就进入该基因所在染色体的界面了,再在“编辑”中“查找” tps1就可以看该基因所在的位置,再点击CDS 或者GeneID:GeneID:852*********都可以出现相关链接!都可以出现相关链接!当然,如果你在文献查到目的蛋白的序列号如NP_009684.1或者GeneID:GeneID:852423852423,那分别在Search 后选择Protein 或者Gene 也可以出现相关链接!也可以出现相关链接!二.基因CDS 区界面的3个号码/entrez/viewer.fcgi?val=50593115&from=488899&to=490386&view=gbwithparts找到后,我发现该界面有3个标记,一个是NC_001134 ,其次是gi:50593115,最后是FEA TURES 中的gene 中的中的 /db_xref= “GeneID:GeneID:852423852423”,他们分别是什么号码,用在什么地方呢?尝试中,终于发现,地方呢?尝试中,终于发现,在Search “Nucleotide ”或者“Core Core NucleotideNucleotide ”时,for 后面是NC_001134,最终go 到该基因所在染色体全长序列的信息,所以NC_001134应该是该染色体的登录号吧?应该是该染色体的登录号吧?在Search “Nucleotide ”或者“Core Core NucleotideNucleotide ”时,for 后面是50593115,最终go 到该基因所在染色体全长序列的信息,所以50593115应该是该染色体的号吧?应该是该染色体的号吧?在Search “Gene ”时,for 后面是852423,最终go 到该基因的信息,所以852423应该是该基因的登录号吧?所以我们如果要记住目的基因在ncbi 中的位置就记住这个GeneID !其他像NP_009684当然是基因编码的蛋白质的登录号啦,不说了。
一步一步教你使用NCBI查找DNA、mRNA、cDNA

一步一步教你使用 NCBI 查找DNA、mRNA、cDNA、...最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在 NCBI 上都可以方便的找到答案。
现在我就结合我自己使用 NCBI的一些经历(经验)跟大家交流一下 BCBI 的使用。
希望大家都能发表自己的使用心得,让我们共同进步!我分以下几个部分说一下 NCBI 的使用:Part one 如何查找基因序列、mRNA、PromoterPart two 如何查找连续的 mRNA、cDNA、蛋白序列Part three 运用 STS 查找已经公布的引物序列Part four 如何运用 BLAST 进行序列比对、检验引物特异性特别感谢本版版主,将这个帖子置顶!从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以与想给我投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友!请大家对以下我发表的内容提出自己的意见。
关于NCBI 其他方面的使用也请水平较高的战友给予补充First of all,还是让我们从查找基因序列开始。
第一部分利用Map viewer 查找基因序列、mRNA 序列、启动子(Promoter)下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码、序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
教你使用NCBI查找DNA、mRNA、cDNA

一步一步教你使用 NCBI 查找DNA、mRNA、cDNA、...最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在 NCBI 上都可以方便的找到答案。
现在我就结合我自己使用 NCBI的一些经历(经验)跟大家交流一下 BCBI 的使用。
希望大家都能发表自己的使用心得,让我们共同进步!我分以下几个部分说一下 NCBI 的使用:Part one 如何查找基因序列、mRNA、PromoterPart two 如何查找连续的 mRNA、cDNA、蛋白序列Part three 运用 STS 查找已经公布的引物序列Part four 如何运用 BLAST 进行序列比对、检验引物特异性特别感谢本版版主,将这个帖子置顶!从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友!请大家对以下我发表的内容提出自己的意见。
关于NCBI 其他方面的使用也请水平较高的战友给予补充First of all,还是让我们从查找基因序列开始。
第一部分利用Map viewer 查找基因序列、mRNA 序列、启动子(Promoter)下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:/mapview/index.html在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码、序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
NCBI在线BLAST使用方法与结果详解

NCBI在线BLAST使用方法与结果详解NCBI(National Center for Biotechnology Information)是一个包含大量基因组学、生物信息学等相关数据和工具的数据库。
其中,BLAST (Basic Local Alignment Search Tool)是一种常用的序列比对工具,可用于在数据库中搜索相似序列。
一、BLAST简介BLAST是一种基于序列比对的方法,可用于确定一给定序列与数据库中序列的相似性。
其工作原理是将查询序列与数据库中的序列进行比对,并生成一个比对得分来衡量它们之间的相似程度。
通过BLAST的结果,可以获得序列的匹配位置、长度、相似性等信息,从而帮助研究人员进行更深入的生物学研究。
二、使用方法1. 打开NCBI网站首先,打开浏览器,输入NCBI的网址(https:///),进入NCBI的官方网站。
2. 进入BLAST页面在NCBI的主页上,找到“BLAST”或“BLAST and Alignments”选项,并点击进入BLAST页面。
3. 输入查询序列在BLAST页面上,找到“Enter Query Sequence”或“Enter accession number, gi, or FASTA sequence”等文本框,将需要查询的序列输入其中。
可以直接复制粘贴序列,或选择上传文件的方式输入。
4. 选择数据库在BLAST页面上,找到“Choose Search Set”或“Database”等选项,选择需要比对的数据库。
NCBI提供了多个数据库,如“nr”(非冗余蛋白数据库)、“nt”(非冗余核酸数据库)等,根据研究需要选择合适的数据库。
5. 设置参数根据需要,可以通过“Algorithm parameters”等选项来设置比对参数,如设置匹配的阈值、比对的方式等。
6. 运行BLAST设置完成后,点击“BLAST”或“Run BLAST”等按钮运行BLAST。
如何在NCBI查找基因序列

如何在NCBI查找基因序列在NCBI(美国国家生物技术信息中心)网站上查找基因序列是生物学和生物医学研究中常见的任务之一、NCBI提供了各种数据库和工具,可以轻松地和检索各种基因序列。
以下是一些可能有助于您查找基因序列的指南:1.登录NCBI网站:2.选择合适的数据库:- GenBank: GenBank是一个基因组、核酸序列和蛋白质序列的公共数据库。
您可以在GenBank上找到来自各个物种的多个基因序列。
- RefSeq: RefSeq是一个包含参考序列信息的数据库,包括基因组、转录本和蛋白质序列。
-GEO:GEO是一个基因表达谱数据库,可以提供基因序列在不同组织和条件下的表达情况。
- dbSNP: dbSNP是一个单核苷酸多态性(SNP)数据库,可以提供不同个体之间的基因序列差异信息。
3.使用工具:NCBI提供了多种工具和选项,可以帮助您查找特定基因序列。
-基础:在NCBI的主页上,您会看到一个栏。
在栏内输入基因名称、基因ID、关键词、物种等信息,然后按下按钮,系统会返回与您条件相匹配的结果。
-BLAST:BLAST(基本局部序列比对工具)是一个广泛使用的比对工具,可以用来查找特定序列。
您可以在主页上找到BLAST栏,将您的序列输入到栏内,选择相应的数据库,然后点击按钮,系统将返回与您的序列相似的其他序列。
4.进一步筛选和过滤结果:在结果页面,您可以使用不同的过滤选项来进一步筛选和过滤结果。
您可以根据物种、序列长度、相似性等进行筛选。
6.使用NCBIAPI和高级选项:如果您需要进一步定制化的和分析,您可以使用NCBI提供的API (应用程序编程接口)来自动化和批量处理。
此外,NCBI还提供了高级选项,可以通过高级菜单来设置更复杂的条件。
NCBI良心教程寻找基因转录本序列及相关编码蛋白

NCBI良心教程寻找基因转录本序列及相关编码蛋白NCBI(National Center for Biotechnology Information)是一个提供生物医学信息的国际性资源库,其中包含了大量的生物学数据库和工具,包括基因序列数据库(如GenBank)、蛋白质序列数据库(如UniProt)、文献数据库(如PubMed)等。
在NCBI上寻找基因转录本序列及其相关编码蛋白可以通过以下步骤实现。
第二步:使用NCBI的功能,输入你感兴趣的基因的名称或编号,点击。
第三步:在结果页面中选择合适的结果。
通常,结果中会列出与你内容相关的不同数据库的条目。
第四步:选择GenBank数据库的条目,该数据库包含了基因、转录本和蛋白质序列的信息。
第五步:在GenBank条目页面中,你可以找到关于该基因的详细信息,包括不同的转录本和它们的序列。
第六步:点击转录本序列链接,你将进入转录本信息页面。
在该页面上,你将看到与该转录本相关的信息,包括其序列。
第七步:如果你对转录本的编码蛋白感兴趣,在转录本信息页面上找到蛋白质序列的链接。
点击该链接,你将进入蛋白质信息页面,其中包含关于该蛋白质的详细信息,包括其序列。
第八步:通过阅读转录本和蛋白质的信息,你可以获取更多关于基因及其编码蛋白的详细了解,并进行进一步的研究。
除了以上的步骤,你还可以利用NCBI的一些工具和功能来辅助你寻找基因转录本序列及其相关编码蛋白。
比如,你可以使用NCBI的BLAST (Basic Local Alignment Search Tool)工具来比对你感兴趣的序列与已知的序列进行比对,从而找到与之相似的序列,这有助于寻找与你研究主题相关的基因转录本和蛋白质。
综上所述,NCBI是一个非常有用的资源,可以帮助你寻找基因转录本序列及其相关编码蛋白。
通过利用NCBI的功能、浏览GenBank数据库条目以及使用工具和功能,你可以找到你感兴趣的基因转录本和蛋白质的详细信息,并进行进一步的研究。
