常见酶切位点与保护碱基
NEB保护碱基-各种酶切位点保护碱基
0
0
0
0
0
0
0
0
75
>90
75
>90
Nhe I
GGCTAGCC CGGCTAGCCG CTAGCTAGCTAG
0
0
10
25
10
50
Not I
TTGCGGCCGCAA
0
0
ATTTGCGGCCGCTTTA
10
10
AAATATGCGGCCGCTATAAA
10
10
ATAAGAATGCGGCCGCTAAACTAT
酶 Acc I Afl III Asc I Ava I BamH I Bgl II BssH II BstE II BstX I Cla I
EcoR I
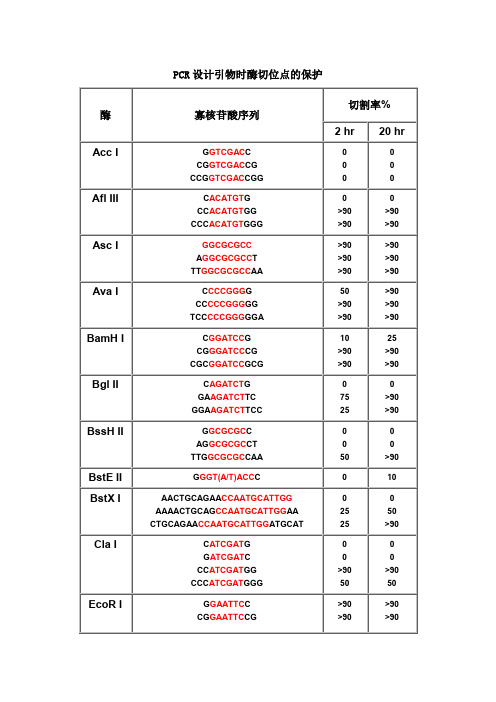
PCR 设计引物时酶切位点的保护
寡核苷酸序列
GGTCGACC CGGTCGACCG CCGGTCGACCGG
CACATGTG CCACATGTGG CCCACATGTGGG
0
0
0
25
0
50
75
>90
Pst I
GCTGCAGC
0
0
TGCACTGCAGTGCA
10
10
AACTGCAGAACCAATGCATTGG
>90
>90
AAAACTGCAGCCAATGCATTGGAA
>90
>90
CTGCAGAACCAATGCATTGGATGCAT
0
0
Pvu I
CCGATCGG ATCGATCGAT TCGCGATCGCGA
25
90
AAGGAAAAAAGCGGCCGCAAAAGGAAAA
各种酶切位点的保护碱基引物设计必看

各种酶切位点的保护碱基酶不同,所需要的酶切位点的保护碱基的数量也不同。
一般情况下,在酶切位点以外多出3个碱基即可满足几乎所有限制酶的酶切要求。
在资料上查不到的,我们一般都随便加3个碱基做保护。
寡核苷酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments(oligonucleotides)为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。
取1 μg 已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。
反应缓冲液含70 mM Tris-HCl (pH , 10 mM MgCl2 , 5 mM DTT及适量的NaCl或KCl(视酶的具体要求而定)。
20%的PAGE(7 M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核苷酸作为对照。
若底物有较长的回文结构,切割效率则可能因为出现发夹结构而降低。
2.双酶切的问题参看目录,选择共同的buffer。
其实,双酶切选哪种buffer是实验的结果,takara公司从1979年开始生产限制酶以来,做了大量的基础实验,也积累了很多经验,目录中所推荐的双酶切buffer 完全是依据具体实验结果得到的。
有共同buffer的,通常按照常规的酶切体系,在37℃进行同步酶切。
但BamH I在37℃下有时表现出star活性,常用30℃单切。
两个酶切位点相邻或没有共同buffer的,通常单切,即先做一种酶切,乙醇沉淀,再做另一种酶切。
各种酶切位点的保护碱基 引物设计必看
各种酶切位点的保护碱基酶不同,所需要的酶切位点的保护碱基的数量也不同。
一般情况下,在酶切位点以外多出3个碱基即可满足几乎所有限制酶的酶切要求。
在资料上查不到的,我们一般都随便加3个碱基做保护。
寡核苷酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments(oligonucleotides)为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。
取1 μg 已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。
反应缓冲液含70 mM Tris-HCl (pH 7.6), 10 mM MgCl2 , 5 mM DTT及适量的NaCl或KCl(视酶的具体要求而定)。
20%的PAGE(7 M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核苷酸作为对照。
若底物有较长的回文结构,切割效率则可能因为出现发夹结构而降低。
2.双酶切的问题参看目录,选择共同的buffer。
其实,双酶切选哪种buffer是实验的结果,takara公司从1979年开始生产限制酶以来,做了大量的基础实验,也积累了很多经验,目录中所推荐的双酶切buffer完全是依据具体实验结果得到的。
有共同buffer的,通常按照常规的酶切体系,在37℃进行同步酶切。
但BamH I在37℃下有时表现出star活性,常用30℃单切。
两个酶切位点相邻或没有共同buffer的,通常单切,即先做一种酶切,乙醇沉淀,再做另一种酶切。
PCR设计引物时酶切位点的保护碱基

PCR设计引物时酶切位点的保护碱基引物设计是PCR实验的关键步骤之一,引物的好坏会直接影响到PCR反应的成功与否。
而在引物设计过程中,酶切位点的保护碱基是需要考虑的重要因素之一在PCR实验中,引物的作用是指定PCR反应的放大区域,并提供启动位点供聚合酶结合。
一般情况下,引物至少需要包含一段特定的DNA序列,以便与目标序列互补配对。
在引物设计过程中,选择合适的酶切位点是十分必要的。
酶切位点是指位于特定DNA序列上的限制酶可以识别并切割的区域。
酶切位点的选择通常需要考虑如下几个方面:1.切割效果:选择切割效果好的酶切位点可以提高PCR反应的特异性和灵敏度。
经典的选择是选择一种具有4-6个碱基的酶切位点,并且该位点在引物中间的位置。
这可以有效防止酶切位点的保护碱基对PCR反应的影响。
2.特异性:引物需要选择适合的酶切位点,以确保只有目标序列被放大,而不包括其他与之相关的非特异性序列。
因此,在选择酶切位点时应尽量避免与其他非特异性序列存在相似性。
3.引物长度:引物长度的选择也与酶切位点相关。
如果引物长度过短,可能会导致酶切位点过于靠近PCR反应产物的端点,从而使切割效果不佳。
因此,在引物设计时,应选择适当的引物长度,以保证酶切位点的保护碱基不会对PCR反应产物的生成产生不利影响。
酶切位点的保护碱基是指在特定的DNA序列上,通过选择相应的碱基来避免受到酶切的影响。
常见的保护碱基有甲基化碱基、磷酸化碱基以及接上阻断扩增的非互补碱基等。
1.甲基化碱基:将酶切位点中的一些碱基进行甲基化处理,可以有效地阻止特定酶的切割作用。
甲基化碱基可以通过DNA甲基转移酶进行甲基化修饰。
2.磷酸化碱基:磷酸化碱基是在引物设计过程中添加磷酸基团的方法,通过给酶切位点添加一个磷酸基团来阻断酶的切割作用。
3.非互补碱基:为了阻断酶切位点的切割作用,可以在酶切位点的周围引入一个与其不互补的碱基序列。
这样可以阻断酶的结合和切割。
总的来说,选择合适的酶切位点和保护碱基对PCR实验的成功至关重要。
各种酶切位点的保护碱基引物设计必看

各种酶切位点的保护碱基引物设计必看Document serial number【KK89K-LLS98YT-SS8CB-SSUT-SST108】各种酶切位点的保护碱基酶不同,所需要的酶切位点的保护碱基的数量也不同。
一般情况下,在酶切位点以外多出3个碱基即可满足几乎所有限制酶的酶切要求。
在资料上查不到的,我们一般都随便加3个碱基做保护。
寡核苷酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments(oligonucleotides)为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。
取1 μg 已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。
反应缓冲液含70 mM Tris-HCl (pH , 10 mM MgCl2 , 5 mM DTT及适量的NaCl或KCl(视酶的具体要求而定)。
20%的PAGE(7 M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核苷酸作为对照。
若底物有较长的回文结构,切割效率则可能因为出现发夹结构而降低。
2.双酶切的问题参看目录,选择共同的buffer。
其实,双酶切选哪种buffer是实验的结果,takara公司从1979年开始生产限制酶以来,做了大量的基础实验,也积累了很多经验,目录中所推荐的双酶切buffer完全是依据具体实验结果得到的。
有共同buffer的,通常按照常规的酶切体系,在37℃进行同步酶切。
各种酶切位点的保护碱基
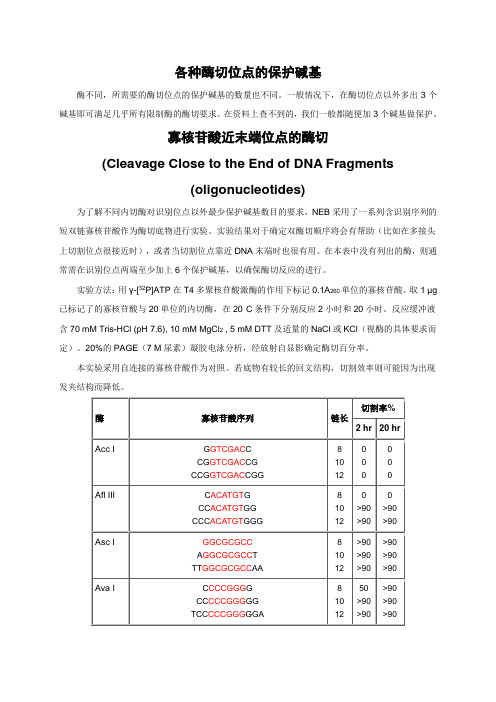
各种酶切位点的保护碱基酶不同,所需要的酶切位点的保护碱基的数量也不同。
一般情况下,在酶切位点以外多出3个碱基即可满足几乎所有限制酶的酶切要求。
在资料上查不到的,我们一般都随便加3个碱基做保护。
寡核苷酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments(oligonucleotides)为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。
取1 µg 已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。
反应缓冲液含70 mM Tris-HCl (pH 7.6), 10 mM MgCl2 , 5 mM DTT及适量的NaCl或KCl(视酶的具体要求而定)。
20%的PAGE(7 M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核苷酸作为对照。
若底物有较长的回文结构,切割效率则可能因为出现发夹结构而降低。
2.双酶切的问题参看目录,选择共同的buffer。
其实,双酶切选哪种buffer是实验的结果,takara公司从1979年开始生产限制酶以来,做了大量的基础实验,也积累了很多经验,目录中所推荐的双酶切buffer 完全是依据具体实验结果得到的。
有共同buffer的,通常按照常规的酶切体系,在37℃进行同步酶切。
但BamH I在37℃下有时表现出star活性,常用30℃单切。
两个酶切位点相邻或没有共同buffer的,通常单切,即先做一种酶切,乙醇沉淀,再做另一种酶切。
常用酶切位点表(含保护碱基)课件.doc
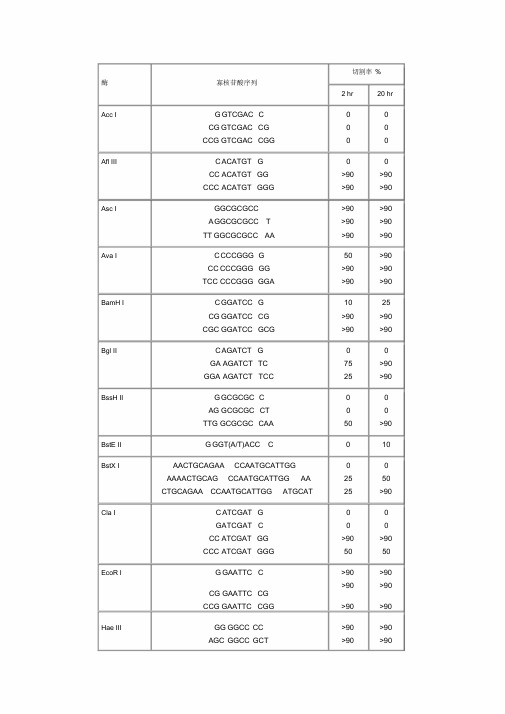
切割率%酶寡核苷酸序列2 hr 20 hrAcc I G GTCGAC C 0 0CG GTCGAC CG 0 0CCG GTCGAC CGG 0 0Afl III C ACATGT G 0 0CC ACATGT GG >90 >90CCC ACATGT GGG >90 >90Asc I GGCGCGCC >90 >90A GGCGCGCC T >90 >90TT GGCGCGCC AA >90 >90Ava I C CCCGGG G 50 >90CC CCCGGG GG >90 >90TCC CCCGGG GGA >90 >90BamH I C GGATCC G 10 25CG GGATCC CG >90 >90CGC GGATCC GCG >90 >90Bgl II C AGATCT G 0 0GA AGATCT TC 75 >90GGA AGATCT TCC 25 >90BssH II G GCGCGC C 0 0AG GCGCGC CT 0 0TTG GCGCGC CAA 50 >90BstE II G GGT(A/T)ACC C 0 10BstX I AACTGCAGAA CCAATGCATTGG 0 0 AAAACTGCAG CCAATGCATTGG AA 25 50CTGCAGAA CCAATGCATTGG ATGCAT 25 >90Cla I C ATCGAT G 0 0GATCGAT C 0 0CC ATCGAT GG >90 >90CCC ATCGAT GGG 50 50EcoR I G GAATTC C >90 >90>90 >90CG GAATTC CGCCG GAATTC CGG >90 >90Hae III GG GGCC CC >90 >90AGC GGCC GCT >90 >90TTGC GGCC GCAA >90 >90Hind III C AAGCTT G 0 0CC AAGCTT GG 0 0CCC AAGCTT GGG 10 75Kpn I G GGTACC C 0 0GG GGTACC CC >90 >90CGG GGTACC CCG >90 >90Mlu I G ACGCGT C 0 0CG ACGCGT CG 25 50Nco I C CCATGG G 0 0CATG CCATGG CATG 50 75Nde I C CATATG G 0 0CC CATATG GG 0 0CGC CATATG GCG 0 0GGGTTT CATATG AAACCC 0 0GGAATTC CATATG GAATTCC 75 >90GGGAATTC CATATG GAATTCCC 75 >90Nhe I G GCTAGC C 0 0CG GCTAGC CG 10 25CTA GCTAGC TAG 10 50切割率%酶寡核苷酸序列2 hr 20 hrNot I TT GCGGCCGC AA 0 0ATTT GCGGCCGC TTTA 10 10AAATAT GCGGCCGC TATAAA 10 10 ATAAGAAT GCGGCCGC TAAACTAT 25 90 AAGGAAAAAA GCGGCCGC AAAAGGAAAA 25 >90Nsi I TGC ATGCAT GCA 10 >90 CCA ATGCAT TGGTTCTGCAGTT >90 >90Pac I TTAATTAA 0 0G TTAATTAA C 0 25CC TTAATTAA GG 0 >90Pme I GTTTAAAC 0 0G GTTTAAAC C 0 25GG GTTTAAAC CC 0 50AGCTTT GTTTAAAC GGCGCGCCGG 75 >90Pst I G CTGCAG C 0 0TGCA CTGCAG TGCA 10 10 AA CTGCAG AACCAATGCATTGG >90 >90AAAA CTGCAG CCAATGCATTGGAA >90 >90CTGCAG AACCAATGCATTGGATGCAT 0 0Pvu I C CGATCG G 0 0AT CGATCG AT 10 25TCG CGATCG CGA 0 10 Sac I C GAGCTC G 10 10Sac II GCCGCGG C 0 0TCC CCGCGG GGA 50 >90Sal I GTCGAC GTCAAAAGGCCATAGCGGCCGC 0 0 GC GTCGAC GTCTTGGCCATAGCGGCCGCG 10 50G 10 75ACGC GTCGAC GTCGGCCATAGCGGCCGCGGAASca I GAGTACT C 10 25AAA AGTACT TTT 75 75Sma I CCCGGG 0 10CCCCGGG G 0 10CC CCCGGG GG 10 50TCC CCCGGG GGA >90 >90Spe I GACTAGT C 10 >90GG ACTAGT CC 10 >90CGG ACTAGT CCG 0 50CTAG ACTAGT CTAG 0 50Sph I G GCATGC C 0 0CAT GCATGC ATG 0 25ACAT GCATGC ATGT 10 50Stu I AAGGCCT T >90 >90GA AGGCCT TC >90 >90AAA AGGCCT TTT >90 >90Xba I CTCTAGA G 0 0GC TCTAGA GC >90 >90TGC TCTAGA GCA 75 >90CTAG TCTAGA CTAG 75 >90Xho I C CTCGAG G 0 0CC CTCGAG GG 10 25CCG CTCGAG CGG 10 75Xma I CCCCGGG G 0 0CC CCCGGG GG 25 75CCC CCCGGG GGG 50 >90TCCC CCCGGG GGGA >90 >90。
酶切位点保护碱基

本文给出了分子克隆中常用限制性内切酶的保护碱基序列,如AccI,AflIII,AscI,AvaI,BamHI,BglII,BssHII,BstEII,BstXI,ClaI,EcoRI,HaeIII,HindIII,KpnI,MluI,NcoI,NdeI,NheI,NotI,NsiI,PacI,PmeI,PstI,PvuI,SacI,SacII,SalI,ScaI,SmaI,SpeI,SphI,StuI,XbaI,XhoI,XmaI,为什么要添加保护碱基?在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
该如何添加保护碱基?添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A26单位的寡核苷酸。
取1µg已标记了的寡核苷酸与20单位的内切酶,在2 00°C条件下分别反应2小时和20小时。
各种酶切位点的保护碱基引物设计必看

酶不同,所需要的酶切位点的保护碱基的数量也不同。
一般情况下,在 酶切位点以外多出3个碱基即可满足几乎所有限制酶的酶切要求。
在资料上 查不到的,我们一般都随便加3个碱基做保护。
寡核昔酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments(oligonucleotides )为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB 采用了一系列含识别序列的 短双链寡核甘酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上 切割位点很接近时),或者当切割位点靠近DNA 末端时也很有用。
在本表中没有列出的酶,则通常 需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用Y -[32P]ATP 在T4多聚核甘酸激酶的作用下标记0.1A 260单位的寡核甘酸。
取1 pg 已标记了的寡核甘酸与20单位的内切酶,在20°C 条件下分别反应2小时和20小时。
反应缓冲液含70 mM Tris-HCl (pH 7.6), 10 mM MgCJ , 5 mM DTT 及适量的 NaCI 或 KCl (视酶的具体要求而定)。
20%的PAGE (7 M 尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核甘酸作为对照。
若底物有较长的回文结构,切割效率则可能因为出现发 夹结构而降低。
切位点 的保护碱基2.双酶切的问题参看目录,选择共同的buffer。
其实,双酶切选哪种buffer是实验的结果,takara公司从1979年开始生产限制酶以来,做了大量的基础实验,也积累了很多经验,目录中所推荐的双酶切buffer完全是依据具体实验结果得到的。
有共同buffer的,通常按照常规的酶切体系,在37℃进行同步酶切。
但BamH I在37℃下有时表现出star活性,常用30℃单切。
两个酶切位点相邻或没有共同buffer的,通常单切,即先做一种酶切,乙醇沉淀,再做另一种酶切。
常用限制性内切酶酶切位点保护残基

酶切位点保护碱基-PCR引物设计用于限制性内切酶发布: 2010-05-24 20:19| 来源:生物吧| 编辑:刘浩| 查看: 161 次本文给出了分子克隆中常用限制性内切酶的保护碱基序列,如AccI,AflIII,AscI,AvaI,BamHI,BglII,BssHII,BstEII,BstXI,ClaI,EcoRI,HaeIII,HindIII,KpnI,MluI,NcoI,NdeI,NheI,NotI,NsiI,PacI,PmeI,PstI,PvuI,SacI,SacII,SalI,ScaI,SmaI,SpeI,SphI,StuI,XbaI,XhoI,XmaI,为什么要添加保护碱基?在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
该如何添加保护碱基?添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。
详细酶切位点及保护序列

酶切位点保护碱基-PCR引物设计用于限制性内切酶酶切反应本文给出了分子克隆中常用限制性内切酶的保护碱基序列,如AccI,AflIII,AscI,AvaI,BamHI,BglII,BssHII,BstEII,BstXI,ClaI,EcoRI,HaeIII,HindIII,K pnI,MluI,NcoI,NdeI,NheI,NotI,NsiI,PacI,PmeI,PstI,PvuI,SacI,S acII,SalI,ScaI,SmaI,SpeI,SphI,StuI,XbaI,XhoI,XmaI,为什么要添加保护碱基?在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
该如何添加保护碱基?添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。
常用酶切位点的保护性碱基
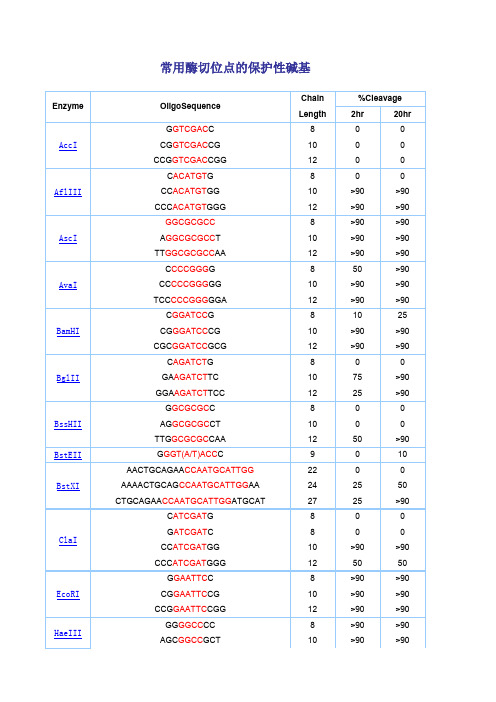
常用酶切位点的保护性碱基Enzyme OligoSequenceChain %Cleavage Length 2hr 20hrAccIG GTCGAC C 8 0 0 CG GTCGAC CG 10 0 0 CCG GTCGAC CGG 12 0 0AflIIIC ACATGT G 8 0 0 CC ACATGT GG 10 >90 >90 CCC ACATGT GGG 12 >90 >90AscIGGCGCGCC 8 >90 >90 A GGCGCGCC T 10 >90 >90 TT GGCGCGCC AA 12 >90 >90AvaIC CCCGGG G 8 50 >90 CC CCCGGG GG 10 >90 >90 TCC CCCGGG GGA 12 >90 >90BamHIC GGATCC G 8 10 25 CG GGATCC CG 10 >90 >90 CGC GGATCC GCG 12 >90 >90BglIIC AGATCT G 8 0 0 GA AGATCT TC 10 75 >90 GGA AGATCT TCC 12 25 >90BssHIIG GCGCGC C 8 0 0 AG GCGCGC CT 10 0 0 TTG GCGCGC CAA 12 50 >90BstEII G GGT(A/T)ACC C 9 0 10BstXIAACTGCAGAA CCAATGCATTGG22 0 0 AAAACTGCAG CCAATGCATTGG AA 24 25 50 CTGCAGAA CCAATGCATTGG ATGCAT 27 25 >90ClaIC ATCGAT G 8 0 0G ATCGAT C 8 0 0 CC ATCGAT GG 10 >90 >90 CCC ATCGAT GGG 12 50 50EcoRIG GAATTC C 8 >90 >90 CG GAATTC CG 10 >90 >90 CCG GAATTC CGG 12 >90 >90HaeIIIGG GGCC CC 8 >90 >90 AGC GGCC GCT 10 >90 >90TTGC GGCC GCAA 12 >90 >90HindIIIC AAGCTT G 8 0 0 CC AAGCTT GG 10 0 0 CCC AAGCTT GGG 12 10 75KpnIG GGTACC C 8 0 0 GG GGTACC CC 10 >90 >90 CGG GGTACC CCG 12 >90 >90MluIG ACGCGT C 8 0 0 CG ACGCGT CG 10 25 50NcoIC CCATGG G 8 0 0 CATG CCATGG CATG 14 50 75NdeIC CATATG G 8 0 0CC CATATG GG 10 0 0CGC CATATG GCG 12 0 0 GGGTTT CATATG AAACCC 18 0 0 GGAATTC CATATG GAATTCC 20 75 >90 GGGAATTC CATATG GAATTCCC 22 75 >90NheIG GCTAGC C 8 0 0 CG GCTAGC CG 10 10 25 CTA GCTAGC TAG 12 10 50NotITT GCGGCCGC AA 12 0 0 ATTT GCGGCCGC TTTA 16 10 10 AAATAT GCGGCCGC TATAAA 20 10 10 ATAAGAAT GCGGCCGC TAAACTAT 24 25 90 AAGGAAAAAA GCGGCCGC AAAAGGAAAA 28 25 >90NsiITGC ATGCAT GCA 12 10 >90 CCA ATGCAT TGGTTCTGCAGTT 22 >90 >90PacITTAATTAA 8 0 0 G TTAATTAA C 10 0 25 CC TTAATTAA GG 12 0 >90PmeIGTTTAAAC 8 0 0G GTTTAAAC C 10 0 25GG GTTTAAAC CC 12 0 50 AGCTTT GTTTAAAC GGCGCGCCGG 24 75 >90PstIG CTGCAG C 8 0 0TGCA CTGCAG TGCA 14 10 10 AA CTGCAG AACCAATGCATTGG 22 >90 >90 AAAA CTGCAG CCAATGCATTGGAA 24 >90 >90 CTGCAG AACCAATGCATTGGATGCAT26 0 0PvuIC CGATCG G 8 0 0 AT CGATCG AT 10 10 25 TCG CGATCG CGA 12 0 10SacI C GAGCTC G 8 10 10SacIIG CCGCGG C 8 0 0 TCC CCGCGG GGA 12 50 >90SalIGTCGAC GTCAAAAGGCCATAGCGGCCGC28 0 0 GC GTCGAC GTCTTGGCCATAGCGGCCGCGG 30 10 50 ACGC GTCGAC GTCGGCCATAGCGGCCGCGGAA 32 10 75ScaIG AGTACT C 8 10 25 AAA AGTACT TTT 12 75 75SmaICCCGGG 6 0 10C CCCGGG G 8 0 10 CC CCCGGG GG 10 10 50 TCC CCCGGG GGA 12 >90 >90SpeIG ACTAGT C 8 10 >90GG ACTAGT CC 10 10 >90 CGG ACTAGT CCG 12 0 50 CTAG ACTAGT CTAG 14 0 50SphIG GCATGC C 8 0 0 CAT GCATGC ATG 12 0 25 ACAT GCATGC ATGT 14 10 50StuIA AGGCCT T 8 >90 >90 GA AGGCCT TC 10 >90 >90 AAA AGGCCT TTT 12 >90 >90XbaIC TCTAGA G 8 0 0GC TCTAGA GC 10 >90 >90 TGC TCTAGA GCA 12 75 >90 CTAG TCTAGA CTAG 14 75 >90XhoIC CTCGAG G 8 0 0 CC CTCGAG GG 10 10 25 CCG CTCGAG CGG 12 10 75XmaIC CCCGGG G 8 0 0CC CCCGGG GG 10 25 75 CCC CCCGGG GGG 12 50 >90 TCCC CCCGGG GGGA 14 >90 >90。
常用酶切位点的保护性碱基

常⽤酶切位点的保护性碱基常⽤酶切位点的保护性碱基Enzyme Oligo SequenceChain % Cleavage Length 2 hr 20 hrAccIG GTCGAC C 8 0 0 CG GTCGAC CG 10 0 0 CCG GTCGAC CGG 12 0 0AflIIIC ACATGT G 8 0 0 CC ACATGT GG 10 >90 >90 CCC ACATGT GGG 12 >90 >90AscIGGCGCGCC 8 >90 >90 A GGCGCGCC T 10 >90 >90 TT GGCGCGCC AA 12 >90 >90AvaIC CCCGGG G 8 50 >90 CC CCCGGG GG 10 >90 >90 TCC CCCGGG GGA 12 >90 >90BamHIC GGATCC G 8 10 25 CG GGATCC CG 10 >90 >90 CGC GGATCC GCG 12 >90 >90BglIIC AGATCT G 8 0 0 GA AGATCT TC 10 75 >90 GGA AGATCT TCC 12 25 >90BssHIIG GCGCGC C 8 0 0 AG GCGCGC CT 10 0 0 TTG GCGCGC CAA 12 50 >90BstEII G GGT(A/T)ACC C 9 0 10BstXIAACTGCAGAA CCAATGCATTGG22 0 0 AAAACTGCAG CCAATGCATTGG AA 24 25 50 CTGCAGAA CCAATGCATTGG ATGCAT 27 25 >90ClaIC ATCGAT G 8 0 0G ATCGAT C 8 0 0 CC ATCGAT GG 10 >90 >90 CCC ATCGAT GGG 12 50 50EcoRIG GAATTC C 8 >90 >90 CG GAATTC CG 10 >90 >90 CCG GAATTC CGG 12 >90 >90HaeIII GG GGCC CC 8 >90 >90AGC GGCC GCT 10 >90 >90 TTGC GGCC GCAA 12 >90 >90HindIIIC AAGCTT G 8 0 0 CC AAGCTT GG 10 0 0 CCC AAGCTT GGG 12 10 75KpnIG GGTACC C 8 0 0 GG GGTACC CC 10 >90 >90 CGG GGTACC CCG 12 >90 >90MluIG ACGCGT C 8 0 0 CG ACGCGT CG 10 25 50C CCATGG G 8 0 0 CATG CCATGG CATG 14 50 75NdeIC CATATG G 8 0 0CC CATATG GG 10 0 0CGC CATATG GCG 12 0 0 GGGTTT CATATG AAACCC 18 0 0 GGAATTC CATATG GAATTCC 20 75 >90 GGGAATTC CATATG GAATTCCC 22 75 >90NheIG GCTAGC C 8 0 0 CG GCTAGC CG 10 10 25 CTA GCTAGC TAG 12 10 50NotITT GCGGCCGC AA 12 0 0 ATTT GCGGCCGC TTTA 16 10 10 AAATAT GCGGCCGC TATAAA 20 10 10 ATAAGAAT GCGGCCGC TAAACTAT 24 25 90 AAGGAAAAAA GCGGCCGC AAAAGGAAAA 28 25 >90NsiITGC ATGCAT GCA 12 10 >90 CCA ATGCAT TGGTTCTGCAGTT 22 >90 >90PacITTAATTAA 8 0 0 G TTAATTAA C 10 0 25 CC TTAATTAA GG 12 0 >90PmeIGTTTAAAC 8 0 0G GTTTAAAC C 10 0 25GG GTTTAAAC CC 12 0 50 AGCTTT GTTTAAAC GGCGCGCCGG 24 75 >90PstIG CTGCAG C 8 0 0 TGCA CTGCAG TGCA 14 10 10AA CTGCAG AACCAATGCATTGG 22 >90 >90 AAAA CTGCAG CCAATGCATTGGAA 24 >90 >90 CTGCAG AACCAATGCATTGGATGCAT26 0 0PvuIC CGATCG G 8 0 0 AT CGATCG AT 10 10 25 TCG CGATCG CGA 12 0 10SacI C GAGCTC G 8 10 10SacIIG CCGCGG C 8 0 0 TCC CCGCGG GGA 12 50 >90SalIGTCGAC GTCAAAAGGCCATAGCGGCCGC28 0 0 GC GTCGAC GTCTTGGCCATAGCGGCCGCGG 30 10 50 ACGC GTCGAC GTCGGCCATAGCGGCCGCGGAA 32 10 75ScaIG AGTACT C 8 10 25 AAA AGTACT TTT 12 75 75SmaICCCGGG 6 0 10C CCCGGG G 8 0 10 CC CCCGGG GG 10 10 50 TCC CCCGGG GGA 12 >90 >90G ACTAGT C 8 10 >90GG ACTAGT CC 10 10 >90 CGG ACTAGT CCG 12 0 50 CTAG ACTAGT CTAG 14 0 50 SphIG GCATGC C 8 0 0 CAT GCATGC ATG 12 0 25 ACAT GCATGC ATGT 14 10 50StuIA AGGCCT T 8 >90 >90 GA AGGCCT TC 10 >90 >90 AAA AGGCCT TTT 12 >90 >90XbaIC TCTAGA G 8 0 0GC TCTAGA GC 10 >90 >90 TGC TCTAGA GCA 12 75 >90 CTAG TCTAGA CTAG 14 75 >90 XhoIC CTCGAG G 8 0 0 CC CTCGAG GG 10 10 25 CCG CTCGAG CGG 12 10 75XmaIC CCCGGG G 8 0 0 CC CCCGGG GG 10 25 75CCC CCCGGG GGG 12 50 >90 TCCC CCCGGG GGGA 14 >90 >90。
