Miseq上机操作指南
QIAGEN--miScript PCR系统中文说明书(初次翻译,如有错误,敬请包涵)

miScript PCR 系统操作手册试剂盒成分:miScript Reverse Transcription KitmiScript Primer AssaysmiScript SYBR® Green PCR Kit10x miScript Primer Assay 和10x miScript Precursor Assay是干粉状的,需要稀释。
首先短暂离心小管,然后加入550μl TE,pH 8.0,最后在漩涡震荡仪上4-6次。
为了保持引物的活性,在冷冻条件下将稀释的引物分装成小管。
实验步骤:反转录反应1、在冰上融化RNA,室温下(15–25ºC)融化5x miScript RT Buffer 和RNasefree water 。
先轻弹每个小管使其充分混匀,然后短暂离心以收集管壁和管盖上的液体,最后放置在冰上。
2、在冰上配制reverse-transcription master mix。
配好后混合并放置在冰上。
如果miScript Reverse Transcriptase Mix是从-20°C 冰箱拿出来的话,那么在使用完毕后立刻放入冰箱。
如果要配制多次反应,先在一个大管中准备Mix,并且多配制10%。
3、将miScript Reverse Transcriptase Mix分装到每个小管中,然后加入RNA。
4、在37°C下孵育60分钟5、在95°C下孵育5分钟以灭活miScript Reverse TranscriptaseMix6、如果马上进行PCR,讲产物放置在冰上;如果不立刻做PCR,那么将反转录产物放置在-20°C实验步骤:Real-Time PCR检测miRNA或Noncoding RNA1、融化2x QuantiTect SYBR Green PCR Master Mix,10x miScriptUniversal Primer, 10x miScript Primer Assay, template cDNA, 和RNase-free water。
MIQE指南

Bulletin CN 5859
ԛິࠅݴǖ ںǖԛ࡛ۏ൶ኪؾୟ 113 ࡽ
ᆀྪዐ႐ 702 ࣆۉǖ010 - 8267 5748 دኈǖ010 - 6252 9800 ᆰՊǖ100086
Fleige S and Pfaffl MW (2006). RNA integrity and the effect on the real-time qRTPCR performance. Mol Aspects Med 27, 126-139.
Garson JA et al. (2009). Unreliable real-time PCR analysis of human endogenous retrovirus-w (HERV-W) RNA expression and DNA copy number in multiple sclerosis. AIDS Res Hum Retroviruses 25, 377-378; author reply 379-381.
Bio-Rad’s real-time thermal cyclers are licensed real-time thermal cyclers under Applera’s United States Patent 6,814,934 B1 for use in research, human in vitro diagnostics, and all other fields except veterinary diagnostics. This product is covered by one or more of the following U.S. patents or their foreign counterparts owned by Eppendorf AG: U.S. Patent Nos. 6,767,512 and 7,074,367.
基因组测序仪MiSeqDx使用手册说明书

MiSeq TM Dx使用手册MiSeqDx 使用手册文件号1000000039320 v04 | 1 2020年12月Material # 20030132引言本文档及其内容是Illumina,Inc.及其附属公司(“Illumina”)所有,并且仅供与所述产品相关的合同约定的客户使用,无其他用途。
未经Illumina事先书面同意,本文件及其内容不得以其他方式传播、披露或转载和/或用于任何其他目的。
Illumina不会通过本文件对专利、商标、版权或普通法权利或任何第三方类似的权利传达任何许可。
本文档中的说明必须由合格且经过适当培训的人员严格明确地遵循,从而确保本文所述产品的正确和安全使用。
在使用此类产品前,必须全面阅读并理解本文档的所有内容。
未能完全阅读并明确地遵守本文包含的所有说明可能会对产品造成损害,对人员造成伤害,包括用户或其他人员的损害以及对其他财产的损害,并失去该产品的保修。
由于本文所述产品(包括其中的任何部分或软件)的不当使用,ILLUMINA不承担任何责任。
© 2017 Illumina, Inc. 版权所有。
Illumina, MiSeq TM Dx,南瓜橙颜色和流动底纹设计是Illumina, Inc.和/或其在美国和/或其他国家/地区附属公司的商标。
所有其他名称、徽标和其他商标均为其各自所有者的财产。
MiSeqDx 使用手册文件号1000000039320 v04 | 2 2020年12月Material # 20030132修订历史MiSeqDx 使用手册文件号1000000039320 v04 | 3 2020年12月Material # 20030132目录引言 (2)修订历史 (3)目录 (4)第一章概述 (6)产品名称 (6)预期用途 (6)产品结构及组成 (6)第二章设备安装及环境要求 (14)运输和储存 (14)运输和安装 (14)实验室要求 (15)电气要求 (17)环境要求 (18)网络要求 (18)用户自备耗材和设备 (19)第三章工作原理 (21)工作原理 (21)需要但不提供的设备和材料 (21)第四章性能指标 (22)仪器技术指标 (22)仪器使用期限 (23)产品合规性和监管声明 (23)第五章运行操作 (26)P ART וL OCAL R UN M ANAGER(本地运行管理) (26)本地运行管理 (26)登陆信息管理 (26)操作界面概述 (27)管理设置和任务 (31)工作流程概述 (37)P ART װM I S EQ O PERATING S OFTWARE (M I S EQ操作软件) (41)启动和开机 (41)MiSeqDx 使用手册文件号1000000039320 v04 | 42020年12月Material # 20030132测序运行 (43)结果分析 (54)质量控制 (55)文件夹管理 (55)MOS软件界面图标 (56)第六章局限性和注意事项 (58)使用局限性 (58)警告和注意事项 (58)第七章危害及标志 (60)安全考虑和标志 (60)通用标志 (62)第八章设备维护 (63)维护频率 (63)维护清洗 (63)待机清洗 (65)关机步骤 (67)重启步骤 (68)所需磁盘空间 (68)杀毒软件 (68)第九章故障排除 (69)附件 (78)MiSeqDx 使用手册文件号1000000039320 v04 | 52020年12月Material # 20030132第一章概述产品名称中文名称:基因测序仪英文名称:MiSeq TM Dx Instrument型号:MiSeq TM Dx预期用途用于体外诊断。
illumina lrm 软件补丁说明指南说明书

简介Illumina ®注意到Local Run Manager 软件存在安全漏洞,并提供了软件补丁来防止他人远程利用此漏洞。
Local Run Manager 是一款独立的软件应用程序,隶属于以下系统的默认配置:•MiSeq •MiSeqDx *•NextSeq 500•NextSeq 550•NextSeq 550Dx *•MiniSeq •iSeq*供体外诊断使用。
本指南适用于上面列出的Illumina 仪器,以及安装了独立版Local Run Manager 的非仪器计算机。
该漏洞属于未经身份验证的远程命令执行(RCE ),未经缓解的CVSS 评分为10.0高风险(CVSS:3.1/AV:N/AC:L/PR:N/UI:N/S:C/C:H/I:H/A:H )。
上面列出的仪器需要执行以下缓解步骤,以防止未经授权的用户访问一台或多台仪器并执行远程访问攻击。
如果安装程序因为某些原因无法运行,请参见本文档结尾的其他缓解措施部分,或联系寻求更多协助。
请参见获取Local Run Manager 更新,了解可以通过哪几种方式下载或请求此补丁的副本。
•v1.0.0补丁—将更新Local Run Manager Web 配置并禁用远程Internet Information Services (IIS )访问。
获取Local Run Manager 安全补丁获取Local Run Manager 安全补丁的方式有四(4)种。
选项1—直接下载到您的仪器获取Local Run Manager 安全更新的最快方式是直接从托管网站将其下载到仪器。
1.通过安全电子邮件提供的链接,将补丁安装程序下载到您的仪器。
2.将文件传输到仪器上的C:\Illumina 文件夹。
3.按照应用Local Run Manager 安全补丁(第3页)中的说明操作。
LRM 软件补丁1.0说明指南选项2—将补丁安装程序下载到计算机,并通过U盘/共享文件夹传输到仪器如果无法将补丁安装程序下载到仪器,建议将其下载到单独的计算机,然后再传输到仪器。
Miseq操作流程及报价须知

一、最简单快速到建库方法Illumina为研究者提供了最简单快速到建库方法,使用Illumina Nextera转座子酶技术,在试管内即可完成DNA片段化,不需要物理剪切或者酶切步骤,一步完成DNA片段化,末端补平和加接头步骤,整个操作流程只需要90分钟,手动操作步骤不超过20分钟,起始DNA只需要50ng。
整合的高度自动化的系统,点击触摸屏即可开展测序Miseq提供了最简单的新一代测序流程。
通过直观的触摸屏界面开展简单的仪器操作,即插即用的试剂带有RFID 追踪,具有自动化的便利。
小巧、一体化的Miseq平台整合了簇生成,边和成边测序和完整的数据分析,不再需要其他辅助性设备。
整个簇生成和测序过程手动操作不超过20分钟。
经验证的数据质量以Illumina久经考验的边合成边测序技术为基础,通过专利的可逆终止法对数百万个片段进行大规模并行测序,在单个碱基掺入增长DNA链时检测它们。
由于每个检测循环中四种可逆终止子结合的dNTP都存在,所以自然竞争让掺入偏差最小化,与其他技术相比大大降低了原始错误率。
最终结果是高度准确的每个碱基的测序信息,即使是重复序列区和同聚物都确保了可靠的碱基检出。
文库构建:剪切,加接头二、整合的高度自动化的系统,点击触摸屏即可开展测序Miseq 提供了最简单的新一代测序流程。
通过直观的触摸屏界面开展简单的仪器操作,即插即用的试剂带有RFID 追踪,具有自动化的便利。
小巧、一体化的Miseq 平台整合了簇生成,边和成边测序和完整的数据分析,不再需要其他辅助性设备。
整个簇生成和测序过程手动操作不超过20 分钟。
三、经验证的数据质量以Illumina 久经考验的边合成边测序技术为基础,通过专利的可逆终止法对数百万个片段进行大规模并行测序,在单个碱基掺入增长DNA 链时检测它们。
由于每个检测循环中四种可逆终止子结合的dNTP 都存在,所以自然竞争让掺入偏差最小化,与其他技术相比大大降低了原始错误率。
MIQE Guidelines

/pcr
AMPLIFICATION
Nucleic Acid Extraction (核酸提 取)
/pcr
RNA 纯度和完整性
AMPLIFICATION
Experion Virtual Gel
L C 3’ 5’ 10’ 15’ 1h 2h 4h
Primer B
Forward Primer
Reverse Primer A
1
110
/pcr
Amplicon Secondary Structures
AMPLIFICATION
/mfold/applications
编辑们犯难了“定量PCR数据可信吗?”
/pcr
What are the MIQE guidelines?
AMPLIFICATION
qPCR的国际标准:就评价qPCR实验和发表文章时所必需的实验 信息提出了最低限度的标准。
/pcr
MIQE 指南好处
AMPLIFICATION
/pcr
Reverse Transcription 反转录
AMPLIFICATION
/pcr
Reverse Transcription
AMPLIFICATION
RNA
cDNA
Reality Ideal ?
Reproducible Data Not Reproducible
/pcr
qPCR Target Information
AMPLIFICATION
/pcr
序列同源性分析(BLAST)
AMPLIFICATION
http://www.ncbi.nlm.nih. gov/BLAST/Blast.cgi
miseq实验流程

2.1 MiSeq 测序实验流程2.1.1 基因组DNA 抽提默认用E.Z.N.A.® Soil DNA Kit (D5625-01)进行样品提取。
完成基因组DNA 抽提后,利用1%琼脂糖凝胶电泳检测抽提的基因组DNA ,用NanoDrop2000进行浓度的检测。
(具体参考多样性样品质检报告)一、 项目基本信息二、 样品检测方法● D NA 纯度检测方 NanoDrop2000 ● D N A 完整性、总量检三、 样品检测结果 N0. 美吉编号样品名称浓度(ng/μl ) OD 260/280OD 260/230项目名称 多样性 项目跟进销售 项目联系人 合同编号 收样员 收样日期 质检员 质检日期 报告撰写人 报告审核员 报告终审员报告日期胶图(取2μl样品跑胶):2.1.2 PCR扩增按指定测序区域,合成带有barcode的特异引物。
为保证后续数据分析的准确性及可靠性,需满足两个条件,1) 尽可能使用低循环数扩增;2) 保证每个样本扩增的循环数一致。
随机选取具有代表性的样本进行预实验,确保在最低循环数中使绝大多数样本能够扩增出浓度合适的产物。
PCR 采用TransGen AP221-02:TransStart Fastpfu DNA Polymerase;PCR仪:ABI GeneAmp® 9700型;全部样本按照正式实验条件进行,每个样本3个重复,将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳检测,使用AxyPrepDNA凝胶回收试剂盒(AXYGEN公司)切胶回收PCR产物,Tris_HCl洗脱;2%琼脂糖电泳检测。
(具体参照PCR实验报告)PCR扩增报告1.项目基本信息2. PCR预实验2.1引物设计2.2 PCR扩增条件为了保证后续数据分析的准确性及可靠性,需要两个条件,首先是尽可能使用低循环数扩增;其次保证每个样品扩增的循环数统一。
为了达到以上目的,我方首先随机选取10个样品进行预实验,以确保在最低的循环数中绝大多数的样品能够扩增出浓度合适的产物,为所有样品的正式试验做充分准备。
Miseq应用手册

Miseq应用手册MiSeq system applicationsChoose your application. Load, and go.MiSeq is the most accurate and easiest to use benchtop sequencer in the world.Optimized sample preparation kits, push-button sequencing, and automated data analysis, create the first end-to-end sequencing solution. Delivering the easiest workflow, and the most accurate results for the widest breadth of applications. And no need for extra hardware—store, analyze, and share data with BaseSpace?. From sample to data in a single day. All at the touch of a button.Amplicon SequencingCustom Ampliconde novo SequencingResequencingRNA SequencingAmplicon SequencingRNA-SeqChIP-SeqTargeted ResequencingCustom EnrichmentSmall GenomempliconSequencingSequencingSequencingMetagenomicsResequencingRNA SequencingSmall RNA Sequencing16S MetagenomicsLibrary QCRNA-SeqhIP-SeqTargeted ResequencingCustom EnrichmentSmall GenomePlasmidClone CheckingCustom AmpliconCustom Enrichmentde novo SequencingSmall PlasmidAmplicon Sequencing ResequencingCustom Ampliconde novo Sequencing ResequencingRNA Sequencing RNA Sequencing gCustom Genome Ampliconde novo SequencingClone Resequencing Checking RegulationRNA-SeqChIP-SeqTargeted ResequencingCustom Enrichmentde novo equencingResequencing ChIP-Seq Resequencing Custom ChIP-Seq AmpliconRNA TargetedSequencing Resequencing RNA de novo Resequencing Targeted Sequencing Sequencing Resequencing Small RNA Custom Sequencing Enrichment Small RNA RNA Custom Sequencing Sequencing EnrichmentSmall RNA Sequencing16 Small Metagen Genomee novo quencingAmplicon Sequencing16S 16S Small Metagen Metageno GenomeAmplicon SequencingCustom Ampliconde novo SequencingResequencingRNA-SeqChIP-SeqTargeted ResequencingCustom EnrichmentSmall GenomePlasmidClone CheckiTargeted uencing sequencing Library QCCustom RNA Enrichment Sequencing Custom EnrichmentRNA-SeqSmall Small RNA Genome Sequencing Small GenomeChIP-SeqPlasmid 16S Metagenomics PlasmidTargeted ResequencingClon Library Q Checargeted equencingClon CheckCustom EnrichmentThe fastest and easiest sample prep.Nextera XT DNA Sample Prep KitNextera technology combines with the MiSeq system to provide the fastest and easiest solution for amplicon and small-genome sequencing. In one quick step, DNA is simultaneously fragmented and tagged with sequencing adapters to deliver libraries that can be rapidly sequenced with the MiSeq system. Rapid sample preparation Complete sample prep in as little as 90 minutes with only 15 minutes of hands-on time Fastest time to results Go from DNA to data in only 8 hours Optimized for small genomes, PCR amplicons, and plasmids One sample prep kit for many applications Innovative sample normalization Eliminates the need for library quantification prior to sample pooling and sequencingPrepare Input DNANextera TagmentationSequencing and AnalysisSmall Genomes, PCR Amplicons, PlasmidsNextera XT Sample PrepAutomated Sequencing and Variant CallingNextera XT and rapid sequencing with the MiSeq system provides a complete DNA to data workflow in only 8 hours.Amplicon sequencing transformed.TruSeq Custom Amplicon KitOptimized for the MiSeq system, TruSeq Custom Amplicon (TSCA) offers a highly multiplexed amplicon and sample assay for powerful, fast, and cost-effective variant identification. Requiring just 150 ng of starting gDNA, TSCA is amenable to a wide range of samples, including non-human samples and samples available in limited quantity. Data analysis is performed directly on the MiSeq system, providing quick, easy-to-interpret results for both germline and somatic variation. Simplest assay customization Easily create projects online with DesignStudio? for a range of amplicon sizes and target genomes Fastest sample processing Prepare and enrich up to 1,536 amplicons per reaction and 96 samples per plate simultaneously using standard lab equipment Shortest time to project completion Go from genomic DNA to fully analyzed data in less than 2 days Easiest analysis Access pre-configured, on-instrument software for automated variant calling and data analysisStreamlined, intuitive analysis toolbars in Amplicon Viewer. Top pane: visualize coverage levels of each amplicon or across entire genome regions (shown) and view quality scores, as well as variant calls in the middle and lower panes, respectively. Left scroll bars allow users to navigate through samples and targets, and track variants.The widest breadth of sequencing applications.The only benchtop sequencing system capable of automated paired-end reads and up to 8.5 Gb per run, delivering over 500 bases of sequence information. Sample prep kits optimized for a variety of applications. Empower your research with the MiSeq system.Application Targeted ResequencingAmplicon Sequencing (tens of targets) Amplicon Sequencing (hundreds of targets) Hybrid Capture (thousands of targets) 16S Metagenomics Clone CheckingRecommended Read LengthIllumina Solution1 × 2502 × 250 2 × 75 2 × 150 1 × 36Small-Genome SequencingDe novo Resequencing Plasmids 1 × 36, 2 × 250 1 × 36, 2 × 250 1 × 36, 2 × 250RNA SequencingSmall RNA Sequencing RNA-Seq (microbial)? 1 × 36 2 × 50–250Quality ControlLibrary QC 2 × 25RegulationChIP-Seq1 × 36Use with Ribo-Zero?.DNATruSeq DNA Sample Prep Kit TruSeq Custom Enrichment Kit TruSeq Custom Amplicon Kit Nextera XT DNA Sample Prep Kit Nextera Custom Enrichment KitRNATruSeq RNA Sample Prep Kit TruSeq Small RNA Sample Prep KitMiSeq system applicationsMiSeq is the most accurate and easiest to use benchtop sequencer in the world. With the largest applications portfolio at your fingertips. And, access to an entire community of users andtools with BaseSpace. All designed to move your research forward. The pocket inserts showcase just a few of the applications we’ve explored with the MiSeq system. And this is just the beginning.Learn more at /doc/136204105.html,/miseq/doc/136204105.html,FOR RESEARCH USE ONLY2011-2012 Illumina, Inc. All rights reserved. Illumina, IlluminaDx, BaseSpace, BeadArray, BeadXpress, cBot, CSPro, DASL, DesignStudio, Eco, GAIIx, Genetic Energy, Genome Analyzer, GenomeStudio, GoldenGate, HiScan, HiSeq, Infinium, iSelect, MiSeq, Nextera, NuPCR, SeqMonitor, Solexa, TruSeq, VeraCode, the pumpkin orange color, and the Genetic Energy streaming bases design are trademarks or registered trademarks of Illumina, Inc. All other brands and names contained herein are the property of their respective owners. Pub. No. 770-2012-023 Current as of 24 October 2012。
Miseq上机操作指南

Miseq上机操作指南Miseq建库定量上机2016-7-25⼀、⽂库的定量、标准化和混合基于Qubit定量结果进⾏换算如:5ng/ul的Nana或Qubit定量结果,预估建库长度为500bp则,换算浓度为5/(660*500)*106 = 15.3nM根据计算结果,⽤10mM的Tris-HCl(pH 8.5)把⽂库稀释到终浓度为4nM。
⼆、⽂库稀释测序前,混合⽂库会以NaOH进⾏变性,再⽤杂交buffer进稀释,当MiSeq 测序前,再进⾏热变性。
对于低多态性的⽂库来说,每⼀个run中需要加⼊⾄5%的PhiX⽤作测序质控。
准备⼯作:1、800ul的⽔和200ul1N的NaOH。
颠倒数次混匀。
2、把Miseq的试剂盒从冻存冰箱⾥取出,在室温下解冻。
3、准备⼀个冻桶,其中冰和⽔的⽐例为3:1。
DNA变性:1、将最终的⽂库和新配的NaOH混合:5ul的4nM混合⽂库,5ul的0.2N 的NaOH2、在12⼩时以内,0.2N的NaOH与PhiX质控相混合,并保存。
3、旋涡振荡后,短暂离⼼。
4、室温静置5min,等DNA变性。
5、向⽂库中加⼊预冷的HT1(杂交液):10ul的变性DNA,990ul的预冷HT1结果是在1mM的NaOH中为20pM浓度的变性DNA⽂库,⽂库总量为0.02pmol。
6、所得稀释液置于冰上。
稀释变性DNA1、变性DNA⽂库稀释到上机的终浓度。
当使⽤MiSeq v3时,原始簇(raw cluster)的密度是800-1000K/mm2。
当第⼀次上机时,建议使⽤4pM的上机浓度,但可以随之调整。
⽬前采⽤8pM。
2、短暂离⼼后,把反应体系置于冰上。
三、⽂库上机1、取出测序试剂盒,放置于常温⽔中(注意不要漠过上线)。
静置60~90分钟,直⾄完全溶解。
2、从⽔浴中取出试剂盒,上下颠倒⼗次混匀。
擦⼲表⾯的⽔分。
3、创建Sample Sheet。
注:特别注意设置barcode4、开机,运⾏pre-Wash。
Miseq标准化操作指南中文翻译版
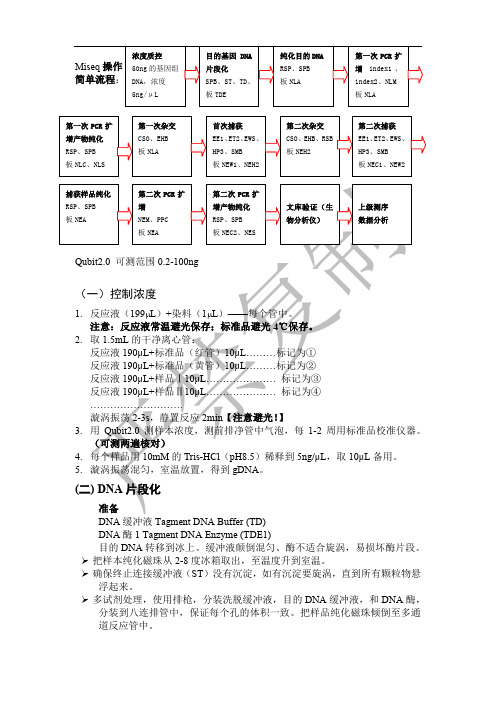
DNA缓冲液Tagment DNA Buffer (TD)DNA酶1 Tagment DNA Enzyme (TDE1)目的DNA转移到冰上。
缓冲液颠倒混匀、酶不适合旋涡,易损坏酶片段。
把样本纯化磁珠从2-8度冰箱取出,至温度升到室温。
确保终止连接缓冲液(ST)没有沉淀,如有沉淀要旋涡,直到所有颗粒物悬浮起来。
多试剂处理,使用排枪,分装洗脱缓冲液,目的DNA缓冲液,和DNA酶,分装到八连排管中,保证每个孔的体积一致。
把样品纯化磁珠倾倒至多通道反应管中。
将深孔板插入到加热体模块中,预加热垂直混匀仪至58度。
取一个深孔板,用记号笔签字。
操作确保反应试剂分装时,为了最好的描述试剂盒信息,反应液分装不需要再冰上操作。
1. 按照以下步骤标准化a)检测目的DNA样本的质量,使用紫外分光光度计或者Qubit,b)每个样品用10mM的Tris-HCl(pH8.5)稀释到10ng/μL。
c)再次检测5ng/μL的目的DNA样本质量,使用紫外分光光度计或者Qubit。
d)在此基础上,每个样品用10mM的Tris-HCl(pH8.5)稀释到5ng/μL,取10μL备用。
配置方法:原DNA浓度为18 ng/μL,配置成体积为20 μL,浓度5ng/μL 的DNA,取原液的体积是20*5/18,取Tris-HCl体积为20-(20*5/18)。
2. 取一0.2mL的干净离心管,加入以下成分:10μL(5ng/μL)gDNA + 25μL Tagment DNA Buffer + 15μL Tagment DNA Enzyme涡旋振荡混匀,1800转1min,避免有气泡,58℃,10min。
注意:用普通PCR仪,先让机器预热到58℃,再放样品。
大样本可用深孔板操作。
在垂直混匀仪上操作。
3. 结束后,向管中加15μL Stop Tagment Buffer,涡旋振荡,短暂离心。
室温放置反应4min。
【此时体系共65μL】(三)纯化:因为转座酶与DNA末端紧密结合,会干扰下游反应过程准备样品重悬洗脱液RSB,样品纯化磁珠SPB,80%乙醇取出,降温至室温。
MiSeqFGx仪器场地准备指引-Illumina

《MiSeq FGx 仪器场地准备指南》
3
交付和安装
Illumina 授权的服务器提供商将负责交付 MiSeq FGx、打开包装并将其放置到实验台上。 交付前必须先准备好空间和实验台。
4
部件号 15050525 修订版 A CHS
实验室要求
实验室要求
此部分提供合理设置 MiSeq FGx 实验室空间的要求和准则。有关详细信息,请参见环境限 制( 第 10 页) 。
仪器尺寸
图 1 MiSeq FGx 仪器
MiSeq FGx 具有以下安装尺寸:
度量 宽度 高度 深度 重量
仪器尺寸 68.6 厘米( 27 英寸) 52.3 厘米( 20.6 英寸) 56.5 厘米( 22.2 英寸) 57.2 公斤( 126 磅)
度量 宽度 高度 深度 重量
带板条箱的尺寸 72.4 厘米( 28.5 英寸) 76.8 厘米( 30.25 英寸) 83.8 厘米( 33 英寸) 90.7 公斤( 200 磅)
板条箱内装 MiSeq FGx 仪器及以下组件: } 废水瓶、承滴盘以及两个有关装运限制位置的标签。 } MiSeq FGx 配件包,含有以下组件: • 洗涤盘 • 洗涤瓶,500 毫升 • 废水瓶塞( 红色) • T 型手柄六方扳手工具,6 毫米 • T 型手柄六方扳手工具,5/64 英寸 • 网线,屏蔽 CAT6 } 电源线
简介
简介
本指南提供准备安装和操作 MiSeq FGx 的场地所需要的信息: } 实验室空间要求 } 电气要求 } 环境限制 } 计算要求 } 用户umina 网站下载以下文档: } 《MiSeq FGx 仪器安全性和合规性指南》( 部件号 15050819_CHS) — 提供有关仪器 标签、合规认证以及安全注意事项的信息。 } 《MiSeq FGx 仪器参考指南》( 部件号 15050524_CHS) — 提供仪器组件和软件的概 述、有关执行测序运行的说明,以及正确仪器维护和故障诊断的过程。 } 《MiSeq ForenSeq 测序试剂盒试剂制备指南》( 部件号 15055483) — 提供试剂盒 内容的描述,以及有关在开始测序运行之前制备试剂盒的说明。 } 《ForenSeq 通用分析软件指南》( 部件号 15053874) — 提供分析过程和计算要求的 全面概述。
sequenom-iPLEX实验操作

iPLEX实验操作一、引物设计用mysequenom网上工具设计(步骤略)二、扩增引物的稀释和UEP引物质量检测1.合成回来的扩增引物加去离子水,稀释为100uM的储液。
之后等体积混合使得终浓度为0.5uM。
如做14个Plex的反应,共28条扩增引物。
每条引物取1ul混合共28ul,再加入172ul的去离子水,总体积为200ul,即为引物Mix(0.5μM)。
2.合成回来的UEP引物按照“UEP引物稀释表”中的黄色高亮部分输入:ID号,Assay的命名,UEP引物的Mass,合成的管中的总OD量,总共有n 个Plex信息。
根据表格计算出每个管中需要加的去离子水量,进行引物重溶。
之后每个管中取等体积引物充分混合即为UEP Mix。
3.将前述UEP Mix 2ul加入40ul去离子水中混合。
加入384孔板中,可用于直接点芯片,打质谱。
这一步检测UEP的质量是必须的,如果质量不好必须重新设计。
三、DNA样品准备基因组DNA样品应电泳检测完整性,并测浓度。
用去离子水或Tris-Cl稀释为浓度10-20ng/ul。
由于样品量大,必须做好样品标记。
四、PCR反应1.在384孔板上一定要做好标记:横6,纵4的24个孔为一组。
以一组为将来点样时候的一针。
2.按多出来10-15%的量来配制Mix,如需检测40个样,则配50-55个样的量反应体系:均按ul计算模板DNA(10ng/ul) 1引物Mix(0.5μM)0.1µM 110*Buffer(含Mg2+)2mM MgCl2 0.5MgCl2(25mM) 2mM 0.4dNTP(25mM) 500µM 0.1Hotstar(5U/μl) 1 Unit 0.1*水 1.9总量5ul*如果做27个Plex以上的PCR,应用0.2 ul3.在384孔板中每孔加入4ul Mix,最后在加入1ul模板DNA。
4.用ABI的PCR封口膜封紧,防止样品蒸发。
Illumina iSeq 100 定序系统安装、维护和操作手册说明书

本指南提供Illumina ®iSeq™100定序系統有關安裝、維修和操作的重要安全資訊。
本手冊包含產品合規與法規聲明。
在系統上執行任何程序之前,請先閱讀此資訊。
系統的原產國和製造日期皆印製在儀器標籤上。
安全注意事項及標識本節旨在提出與安裝、維護和操作儀器有關的潛在危害。
請勿以任何會讓您曝露在這些危險的方式操作儀器或與儀器互動。
一般安全警告確認所有人員均接受訓練,了解如何正確操作儀器以及任何潛在的安全性考量。
在標有此標籤的區域工作時,請遵守所有操作指示,以將人員或儀器所受的風險降到最低。
電氣安全警告請勿取下任何儀器上的外部面板。
儀器內部並無可由使用者維修的零組件。
在移除任一面板的情況下操作本儀器,可能會接觸到線路電壓以及直流電壓而導致觸電。
本儀器電源要求為100至240伏特,50或60赫的交流電。
危險的電壓來源位於背板和側板後面,但如果其他面板移除仍然可能接觸到。
即使關閉儀器電源,儀器上仍會存在些許電壓。
請在所有面板均完好的情況下操作儀器,以免觸電。
電源規格100至240伏特、50或60赫交流電峰值耗電量80 瓦特必須進行電氣接地。
如果電壓波動超過10%,則需要電力線路調節器。
電源線取用請調整儀器的放置方式,以便從插座快速地拔除電源線。
保護性接地線本儀器有通過外殼連接至保護性接地線的線路。
電源線的安全接地使保護性接地線回到安全基準。
使用本裝置時,電源線的保護性接地線連接狀況務必處於良好的工作狀態。
保險絲電源輸入模組包含兩條位於高電壓輸入線路的輸入保險絲。
保險絲尺寸為5公釐×20公釐,額定為10安培,250VAC ,緩熔。
高溫表面安全警告請勿在移除任何面板的情況下操作儀器。
環境注意事項將實驗室溫度保持在15°C 至30°C 之間(22.5°C ±7.5°C )。
運行期間,不可讓周圍溫差大於±2°C 。
濕度將非冷凝相對濕度保持在20至80%之間。
电信行业技术学习-华为seq使用指导书2021

SEQ使用指导书(XX 移动)目录1 SEQ Analyst平台简介 (1)1.1 SEQ Analyst概述 (1)1.2 SEQ Analyst优势 (1)1.3 解决方案的定位 (3)1.4 解决方案组网 (7)2 平台登陆 (9)2.1 SEQ平台登陆地址 (9)2.2 使用的浏览器及插件 (9)2.3 注意事项 (10)3 VOLTE百日会战关键指标提取和分析 (11)3.1 VOLTE七项考核业务指标概览 (11)3.2 各项业务专题分析 (12)3.2.1 注册专题分析 (12)3.2.2 接入专题分析 (15)3.2.3 切换专题分析 (16)3.2.4 掉话专题分析 (17)3.3 多维数据查询的使用 (18)4 VIP保障及投诉处理 (19)4.1 投诉分析处理模块 (19)4.1.1 投诉之辅助分析定位 (19)4.1.2 投诉之CS/PS精准定位分析 (23)4.2 客户体验模块 (30)4.2.1 VVIP用户/VVIP用户群洞察 (30)4.2.2 VVIP用户/VVIP用户群分析 (33)5 ATU指标预测 (38)5.1 ATU语音预测 (38)5.1.1 ATU巡检测试实时监控 (38)5.1.2 掉话率、接通率指标预测 (40)5.1.3 IMS注册成功率指标预测 (42)5.1.4 呼叫建立时延指标预测 (43)5.1.5 eSRVCC切换成功率及时延指标预测 (44)5.1.6 单次通话详单查询 (44)5.2 ATU数据预测 (45)5.2.1 上传速率 (46)5.2.2 下载速率 (47)6 数据业务分析 (49)6.1 重点区域定义 (49)6.2 全网及重点区域KQI监控 (49)6.3 流量经营情况分析 (52)7 基于SEQ的问题分析方法 (54)8 总结 (57)1SEQ Analyst平台简介1.1SEQ Analyst概述SEQ Analyst(全称为Service & Experience Quality Analyst)作为客户体验管理使能平台,以数据分析存储平台和NetProbe被动探针为核心,可集成华为和第三方多种数据源,关联运营商网络中从无线、传输、核心网到应用等端到端数据,运用大数据分析方法进行高效业务质量与网络性能管理,快速处理客户投诉,支撑基于客户体验的精准营销和实时营销。
宏基因组上机操作手册

目录0. 准备工作 (2)1. 宏基因组比对 (2)2. 宏基因组组装 (3)2.1 组装软件:SOAPdenovo (3)2.2 组装软件:Meta-Velvet (5)3 基因预测 (6)4 构建基因集 (7)0. 准备工作上机步骤如下:mkdir ~/Metagenome #新建工作目录cd ~/Metagenome #进入工作目录cp -R /RealBio_Train/Metagenome/01_clean_reads ./ #拷贝数据1.宏基因组比对宏基因组的序列可以通过SOAPaligner比对软件,比对上目标基因组,从而进行物种注释或计算物种丰度。
SOAPaligner需要先对目标基因组进行建库,建库命令如下:2bwt-builder <sequence file>SOAPaligner用法:soap –a <reads_a> -b <reads_b> -D <index.files> -o <PE_output> -2 <SE_output> -m <min_insert_size> -x <max_insert_size>其他重要参数:Option Type Content匹配到多处时的策略:-r INT0:不显示;1:随机显示一个;2:全部匹配模式:0:只允许完全匹配;1:允许一个错配;-M INT2:允许两个错配;4:最佳匹配-p INT 程序运行的线程个数上机内容为:将拷贝得到的reads比对上微生物的基因组。
上机步骤如下:cd ~/Metagenome #先进入个人目录下的工作目录mkdir 02_alignment #新建02_alignment目录cd 02_alignment #进入比对目录cp /RealBio_Train/Metagenome/02_aligner/soapaligner.sh ./ #拷贝比对脚本less test01.pm #查看比对结果less test01.sm #查看比对结果2. 宏基因组组装基因组组装是指将测序仪产出的大量的DNA 片段(Reads )拼接成原始的待测物种的染色体序列,可以类比为拼图游戏。
The MIQE Guidelines-MIQE指南

PCR实验所需提供的最低信息量
钟艺 2014.12.15
背景
• 缺乏共识 • 细节不够
目的
• 使作者能够设计和报告更有价值的qPCR实验。 • 使评阅人员和编辑按照相应的标准来评价所提交的文稿的技术质 量。 • 使读者更容易重复按照该指南发表的实验研究。
学术术语
• qPCR——实时定量PCR(Quantitative Real-time PCR) • RT – PCR——反转录PCR(Reverse Reaction PCR) • 标准化基因——参照基因(reference genes) • TaqMan探针——水解探针(Hydrolysis Probes) • FRET探针——荧光共振能量转移探针 • LightCycler型探针——双杂交探针 • 定量——quantification • 循环数——Cq(quantification cycle)
核酸的质量控制——RNA样本
• RNA样本:保证各样本中含有大致相同量的RNA 检测和报告全基因组DNA的污染程度 RNA样品是否已经过RNA酶处理(酶类型及反应条件) 核酸目标在阳性质控品和NTC下Cq值的比较 RNA模板的质量评价——数量、污染情况、完整度 A260/280——中性PH的缓冲盐 测定mRNA浓度微小(<10X)差异:凝胶电泳 • DNA样本:阳性质控——检测抑制作用
qPCR验证
优化证据(梯度)
数据分析
生物学重复的数量和一致性 重现性(批间差异,CV) 将原始数据提交给RDML
谢 谢
研究和诊断
• 研究样本:来源广泛、低通量、样品类型不同 • 诊断样本:数量有限、高通量、样品类型少
样本的采集、处理和制备
SEQUENOM-iPLEX标准操作流程
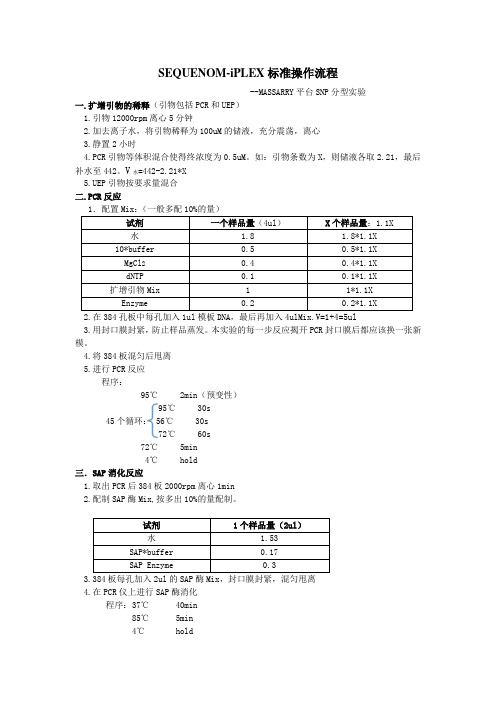
SEQUENOM-iPLEX标准操作流程--MASSARRY平台SNP分型实验一.扩增引物的稀释(引物包括PCR和UEP)1.引物12000rpm离心5分钟2.加去离子水,将引物稀释为100uM的储液,充分震荡,离心3.静置2小时4.PCR引物等体积混合使得终浓度为0.5uM。
如:引物条数为X,则储液各取2.21,最后补水至442。
V水=442-2.21*X5.UEP引物按要求量混合二.PCR反应1.配置Mix:(一般多配10%的量)试剂一个样品量(4ul)X个样品量:1.1X水 1.8 1.8*1.1X 10*buffer 0.5 0.5*1.1XMgCl20.4 0.4*1.1XdNTP 0.1 0.1*1.1X 扩增引物Mix 1 1*1.1XEnzyme 0.2 0.2*1.1X2.在384孔板中每孔加入1ul模板DNA,最后再加入4ulMix.V=1+4=5ul3.用封口膜封紧,防止样品蒸发。
本实验的每一步反应揭开PCR封口膜后都应该换一张新模。
4.将384板混匀后甩离5.进行PCR反应程序:95℃ 2min(预变性)95℃ 30s45个循环: 56℃ 30s72℃ 60s72℃ 5min4℃ hold三.SAP消化反应1.取出PCR后384板2000rpm离心1min2.配制SAP酶Mix,按多出10%的量配制。
试剂1个样品量(2ul)水 1.53SAP*buffer 0.17SAP Enzyme 0.33.384板每孔加入2ul的SAP酶Mix,封口膜封紧,混匀甩离4.在PCR仪上进行SAP酶消化程序:37℃ 40min85℃ 5min4℃ hold四.UEP延伸1. 取出SAP反应板2000rpm离心1min2. 配制延伸Mix,按多出10%的量配制。
试剂1个样本量(2ul)水0.619Gold*buffer 0.2Termination mix 0.2UEP引物 Mix 0.94Enzyme 0.0413.384板每孔加入2ul的延伸Mix,封口膜封紧,混匀甩离4.在PCR仪上进行下列反应程序:94℃ 30s94℃ 5s52℃ 5s 40个外部循环80℃ 5 5个内部循环72℃ 3min4℃ hold五.树脂纯化1.将384板2000rpm离心2min,每孔加入16ul水。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Miseq建库定量上机
2016-7-25
一、文库的定量、标准化和混合
基于Qubit定量结果进行换算
如:5ng/ul的Nana或Qubit定量结果,预估建库长度为500bp则,换算浓度为5/(660*500)*106 = 15.3nM
根据计算结果,用10mM的Tris-HCl(pH 8.5)把文库稀释到终浓度为4nM。
二、文库稀释
测序前,混合文库会以NaOH进行变性,再用杂交buffer进稀释,当MiSeq 测序前,再进行热变性。
对于低多态性的文库来说,每一个run中需要加入至5%的PhiX用作测序质控。
准备工作:
1、800ul的水和200ul1N的NaOH。
颠倒数次混匀。
2、把Miseq的试剂盒从冻存冰箱里取出,在室温下解冻。
3、准备一个冻桶,其中冰和水的比例为3:1。
DNA变性:
1、将最终的文库和新配的NaOH混合:5ul的4nM混合文库,5ul的0.2N 的NaOH
2、在12小时以内,0.2N的NaOH与PhiX质控相混合,并保存。
3、旋涡振荡后,短暂离心。
4、室温静置5min,等DNA变性。
5、向文库中加入预冷的HT1(杂交液):10ul的变性DNA,990ul的预冷HT1
结果是在1mM的NaOH中为20pM浓度的变性DNA文库,文库总量为0.02pmol。
6、所得稀释液置于冰上。
稀释变性DNA
1、变性DNA文库稀释到上机的终浓度。
当使用MiSeq v3时,原始簇(raw cluster)的密度是800-1000K/mm2。
当第一次上机时,建议使用4pM的上机浓度,但可以随之调整。
目前采
用8pM。
2、短暂离心后,把反应体系置于冰上。
三、文库上机
1、取出测序试剂盒,放置于常温水中(注意不要漠过上线)。
静置60~90分钟,直至完全溶解。
2、从水浴中取出试剂盒,上下颠倒十次混匀。
擦干表面的水分。
3、创建Sample Sheet。
注:特别注意设置barcode
4、开机,运行pre-Wash。
约1小时。
3、点击“Sequence”,按页面指示进行。
