文件标签(样本)
DNA数据库正文、DDEM文件、有效XML结构、常染色体、YX染色体 STR 检测基因座、样本分类

附录A(规范性)正文中附表表A.1DDEM文件中特殊字符的替代表示方式表A.2DDEM文件头的各部分细节注释表A.3DDEM文件中样本(Specimen)部分细节注释表A.4DDEM文件中各样本(Specimen)包含的基因座(locus)部分细节注释表A.5DDEM文件中各基因座(locus)中等位基因(allele)部分细节注释附录B(资料性)DDEM文件示例示例:DDEM 0.9版文件实例<?xml version="0.9" encoding="UTF-8"?><NDNADImportFile xmlns="urn:NDNADImportFile-schema"><HEADERVERSION>0.9</HEADERVERSION><MESSAGETYPE>Import</MESSAGETYPE><DESTINATIONLAB>BEIJING</DESTINATIONLAB><NDNADLABORATORYID></NDNADLABORATORYID><SOURCELAB>BEIJING</SOURCELAB><SOURCELABORTORYID></SOURCELABORTORYID><SUBMITBYUSERID>GNIBUIL</SUBMITBYUSERID><SUBMITDATETIME>2020-02-02T21:51:44</SUBMITDATETIME><BATCHID>GEL2012_01_04_101</BATCHID ><KITNAME>Sample KIT</KITNAME><KITID>2020-01-01.1</KITID><KITBRACODENUMBER></KITBRACODENUMBER><SEQUENCERMANUFACTURER>Sample SEQ 001</SEQUENCERMANUFACTURER><SEQUENCERMANUFACTURERID ></SEQUENCERMANUFACTURERID><SERIALNUMBER ></SERIALNUMBER><SPECIMEN SOURCEID="Yes" CASEID="FL2020_02_02_ABC" PARTIAL="true"><SPECIMENID>IMP_0001A</SPECIMENID><SPECIMENCATEGORY>Forensic, Unknown</SPECIMENCATEGORY><SPECIMENCOMMENT>Off-ladder allele value observed for FGA.</SPECIMENCOMMENT> <LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D5S818</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>11</ALLELEVALUE><ALLELESEQS>[ATCT]11</ALLELESEQS><ALLELESEQL>ATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCT</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>11</ALLELEVALUE><ALLELESEQS>[ATCT]11</ALLELESEQS><ALLELESEQL>ATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCT</ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D21S11</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>30</ALLELEVALUE><ALLELESEQS>[TCTA]6[TCTG]5[TCTA]3TA[TCTA]3TCA[TCTA]2TCCATA[TCTA]11</ALLELESEQS><ALLELESEQL>TCTATCTATCTATCTATCTATCTATCTGTCTGTCTGTCTGTCTGTCTATCTATCTATATCTATCTATCTATCATCTAT CTATCCATATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTA</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>30</ALLELEVALUE><ALLELESEQS>[TCTA]5[TCTG]6[TCTA]3TA[TCTA]3TCA[TCTA]2TCCATA[TCTA]11</ALLELESEQS><ALLELESEQL>TCTATCTATCTATCTATCTATCTGTCTGTCTGTCTGTCTGTCTGTCTATCTATCTATATCTATCTATCTATCATCTAT CTATCCATATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTA</ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D7S820</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>10</ALLELEVALUE><ALLELESEQS>[TATC]10</ALLELESEQS><ALLELESEQL>TATCTATCTATCTATCTATCTATCTATCTATCTATCTATC</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>11</ALLELEVALUE><ALLELESEQS>[TATC]11</ALLELESEQS><ALLELESEQL>TATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATC</ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>CSF1PO</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>10</ALLELEVALUE><ALLELESEQS>[ATCT]10</ALLELESEQS><ALLELESEQL>ATCTATCTATCTATCTATCTATCTATCTATCTATCTATCT</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>12</ALLELEVALUE><ALLELESEQS>[ATCT]12</ALLELESEQS><ALLELESEQL>ATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCT</ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D2S1338</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>19</ALLELEVALUE><ALLELESEQS>[GGAA]12[GGCA]7</ALLELESEQS><ALLELESEQL>GGAAGGAAGGAAGGAAGGAAGGAAGGAAGGAAGGAAGGAAGGAAGGAAGGCAGGCAGGCAGGCAGGCAGGCAGGCA</ ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>23</ALLELEVALUE><ALLELESEQS>[GGAA]2[GGAC]1[GGAA]13[GGCA]7</ALLELESEQS><ALLELESEQL>GGAAGGAAGGACGGAAGGAAGGAAGGAAGGAAGGAAGGAAGGAAGGAAGGAAGGAAGGAAGGAAGGCAGGCAGGCAGG CAGGCAGGCAGGCA</ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D3S1358</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>14</ALLELEVALUE><ALLELESEQS>[TCTA]1[TCTG]2[TCTA]11</ALLELESEQS><ALLELESEQL>TCTATCTGTCTGTCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTA</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>15</ALLELEVALUE><ALLELESEQS>[TCTA]1[TCTG]2[TCTA]12</ALLELESEQS><ALLELESEQL>TCTATCTGTCTGTCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTA</ALLELESEQL> </ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>vWA</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>17</ALLELEVALUE><ALLELESEQS>[TAGA]11[CAGA]4[TAGA]1</ALLELESEQS><ALLELESEQL>TAGATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGACAGACAGACAGACAGATAGA</ALLELESE QL></ALLELE><ALLELE><ALLELEVALUE>18</ALLELEVALUE><ALLELESEQS>[TAGA]12[CAGA]4[TAGA]1</ALLELESEQS><ALLELESEQL>TAGATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGACAGACAGACAGACAGATAGA</ALLE LESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D8S1179</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>13</ALLELEVALUE><ALLELESEQS>[TCTA]13</ALLELESEQS><ALLELESEQL>TCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTA</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>13</ALLELEVALUE><ALLELESEQS>[TCTA]1[TCTG]1[TCTA]11</ALLELESEQS><ALLELESEQL>TCTATCTGTCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTA</ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D6S539</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>11</ALLELEVALUE><ALLELESEQS>[GATA]11</ALLELESEQS><ALLELESEQL>GATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGATA</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>12</ALLELEVALUE><ALLELESEQS>[GATA]12</ALLELESEQS><ALLELESEQL>GATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGATA</ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>Penta E</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>12</ALLELEVALUE><ALLELESEQS>[TCTTT]12</ALLELESEQS><ALLELESEQL>TCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTT</ALLELESEQL> </ALLELE><ALLELE><ALLELEVALUE>13</ALLELEVALUE><ALLELESEQS>[TCTTT]13</ALLELESEQS><ALLELESEQL>TCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTTTCTTT</ALLELESEQL> </ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>TPOX</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>8</ALLELEVALUE><ALLELESEQS>[AATG]8</ALLELESEQS><ALLELESEQL>AATGAATGAATGAATGAATGAATGAATGAATG</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>8</ALLELEVALUE><ALLELESEQS>[AATG]8</ALLELESEQS><ALLELESEQL>AATGAATGAATGAATGAATGAATGAATGAATG</ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>TH01</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>8</ALLELEVALUE><ALLELESEQS>[AATG]8</ALLELESEQS><ALLELESEQL>AATGAATGAATGAATGAATGAATGAATGAATG</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>9.3</ALLELEVALUE><ALLELESEQS>[AATG]6ATG[AATG]3</ALLELESEQS><ALLELESEQL>AATGAATGAATGAATGAATGAATGATGAATGAATGAATG</ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D19S433</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>14</ALLELEVALUE><ALLELESEQS>[CCTT]12CCTA[CCTT]1CTTT[CCTT]1</ALLELESEQS><ALLELESEQL>CCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTACCTTCTTTCCTT</ALLELESEQL> </ALLELE><ALLELE><ALLELEVALUE>15</ALLELEVALUE><ALLELESEQS>[CCTT]13CCTA[CCTT]1CTTT[CCTT]1</ALLELESEQS><ALLELESEQL>CCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTTCCTACCTTCTTTCCTT</ALLELESE QL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D18S51</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>15</ALLELEVALUE><ALLELESEQS>[AGAA]15</ALLELESEQS><ALLELESEQL>AGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAA</ALLELESEQL> </ALLELE><ALLELE><ALLELEVALUE>19</ALLELEVALUE><ALLELESEQS>[AGAA]19</ALLELESEQS><ALLELESEQL>AGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAA</ ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>FGA</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>23</ALLELEVALUE><ALLELESEQS>[GGAA]2GGAG[AAAG]15AGAAAAAA[GAAA]3</ALLELESEQS><ALLELESEQL>GGAAGGAAGGAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAGAAAA AAGAAAGAAAGAAA</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>24</ALLELEVALUE><ALLELESEQS>[GGAA]2GGAG[AAAG]16AGAAAAAA[GAAA]3</ALLELESEQS><ALLELESEQL>GGAAGGAAGGAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAAAGAG AAAAAAGAAAGAAAGAAA</ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D6S043</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>12</ALLELEVALUE><ALLELESEQS>[ATCT]12</ALLELESEQS><ALLELESEQL>ATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCT</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>18</ALLELEVALUE><ALLELESEQS>[ATCT]5[ATGT]1[ATCT]12</ALLELESEQS><ALLELESEQL>ATCTATCTATCTATCTATCTATGTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCT</ALLE LESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D13S317</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>11</ALLELEVALUE><ALLELESEQS>[TATC]11</ALLELESEQS><ALLELESEQL>TATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATC</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>11</ALLELEVALUE><ALLELESEQS>[TATC]11</ALLELESEQS><ALLELESEQL>TATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATC</ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D12S391</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>18</ALLELEVALUE><ALLELESEQS>[AGAT]11[AGAC]6[AGAT]1</ALLELESEQS><ALLELESEQL>AGATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGACAGACAGACAGACAGACAGACAGAT</ALLE LESEQL></ALLELE><ALLELE><ALLELEVALUE>20</ALLELEVALUE><ALLELESEQS>[AGAT]12[AGAC]7[AGAT]1</ALLELESEQS><ALLELESEQL>AGATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGATAGACAGACAGACAGACAGACAGACAGACAG AT</ALLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>D1S1656</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>14</ALLELEVALUE><ALLELESEQS>CCTA[TCTA]13</ALLELESEQS><ALLELESEQL>CCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTA</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>18.3</ALLELEVALUE><ALLELESEQS>CCTA[TCTA]13TCA[TCTA]4</ALLELESEQS><ALLELESEQL>CCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCTATCATCTATCTATCTATCTA</A LLELESEQL></ALLELE></LOCUS><LOCUS BATCHID="GEL2020_02_02_100"><LOCUSNAME>Penta D</LOCUSNAME><READINGBY>GNIBUIL</READINGBY><READINGDATETIME>2020-02-02T21:50:42</READINGDATETIME><ALLELE ALLELEREQUIRED="true"><ALLELE><ALLELEVALUE>12</ALLELEVALUE><ALLELESEQS>[AAAGA]12</ALLELESEQS><ALLELESEQL>AAAGAAAAGAAAAGAAAAGAAAAGAAAAGAAAAGAAAAGAAAAGAAAAGAAAAGAAAAGA</ALLELESEQL></ALLELE><ALLELE><ALLELEVALUE>12</ALLELEVALUE><ALLELESEQS>[AAAGA]12</ALLELESEQS><ALLELESEQL>AAAGAAAAGAAAAGAAAAGAAAAGAAAAGAAAAGAAAAGAAAAGAAAAGAAAAGAAAAGA</ALLELESEQL></ALLELE></LOCUS></SPECIMEN></NDNADImportFile>附录C(规范性)DDEM文件使用的有效XML结构示例:DDEM文件的模式定义文件(XML Schema Definition,XSD),用于校验:<xsd:schema xmlns:xsd="/2001/XMLSchema" xmlns="urn:NDNADImportFile-schema"xmlns:sql="urn:schemas-microsoft-com:mapping-schema" elementFormDefault="qualified" targetNamespace="urn:NDNADImportFile-schema"xmlns:r="urn:NDNADImportFile-schema"><xsd:element name="NDNADImportFile" sql:is-constant="true"><xsd:complexType><xsd:sequence maxOccurs="1" id="SeqImportFile" minOccurs="1"><xsd:element name="HEADERVERSION" type="DDEMHeaderVersionType" minOccurs="0" maxOccurs="1" sql:mapped="false" /><xsd:element name="MESSAGETYPE" type="DDEMMessageType" minOccurs="1" maxOccurs="1" sql:mapped="false" /><xsd:element name="DESTINATIONLAB" type="DDEMLabType" minOccurs="1" maxOccurs="1" sql:mapped="false" /><xsd:element name="NDNADLABORATORYID" type="NDNADLABORATORYIDType" maxOccurs="1" minOccurs="0" sql:mapped="false" /><xsd:element name="SOURCELAB" type="DDEMLabType" minOccurs="1" maxOccurs="1" sql:mapped="false" /> <xsd:element name="SOURCELABORTORYID" type="SOURCELABORTORYIDType" maxOccurs="1" minOccurs="0" sql:mapped="false" /><xsd:element name="SUBMITBYUSERID" type="DDEMUserType" minOccurs="1" maxOccurs="1" sql:mapped="false" /><xsd:element name="SUBMITDATETIME" type="NDNADImportDate" minOccurs="1" maxOccurs="1" sql:mapped="false" /><xsd:element name="BATCHID" type="BatchIDType" maxOccurs="1" minOccurs="0" sql:mapped="false" /> <xsd:element name="KITNAME" type="KitNAMEType" maxOccurs="1" minOccurs="0" sql:mapped="false" /> <xsd:element name="KITID" type="KitIDType" maxOccurs="1" minOccurs="0" sql:mapped="false" /><xsd:element name="KITBRACODENUMBER" type="KITBRACODENUMBERType" maxOccurs="1" minOccurs="0" sql:mapped="false" /><xsd:element name="SEQUENCERMANUFACTURER" type="SEQUENCERMANUFACTURERType" maxOccurs="1" minOccurs="0" sql:mapped="false" /><xsd:element name="SEQUENCERMANUFACTURERID" type="SEQUENCERMANUFACTURERType" maxOccurs="1" minOccurs="0" sql:mapped="false" /><xsd:element name="SERIALNUMBER" type="SERIALNUMBERType" maxOccurs="1" minOccurs="0" sql:mapped="false" /><xsd:element name="SPECIMEN" type="SpecimenType" minOccurs="1" maxOccurs="unbounded" sql:relation="Import_Specimen"sql:key-fields="SPEC_ID"><xsd:unique name="UNIQUE_LOCI"><xsd:selector xpath="r:LOCUS" /><xsd:field xpath="r:LOCUSNAME" /></xsd:unique></xsd:element></xsd:sequence></xsd:complexType><xsd:unique name="UNIQUE_SPEC"><xsd:selector xpath="r:SPECIMEN" /><xsd:field xpath="r:SPECIMENID" /></xsd:unique></xsd:element><xsd:simpleType name="DDEMHeaderVersionType"><xsd:restriction base="xsd:decimal"><xsd:totalDigits value="2" /><xsd:fractionDigits value="1" /></xsd:restriction></xsd:simpleType><xsd:simpleType name="DDEMMessageType"><xsd:restriction base="xsd:string"><xsd:length value="6" /><xsd:enumeration value="Import" /></xsd:restriction></xsd:simpleType><xsd:simpleType name="DDEMLabType"><xsd:restriction base="xsd:string"><xsd:maxLength value="10" /><xsd:minLength value="1" /></xsd:restriction></xsd:simpleType><xsd:simpleType name="DDEMUserType"><xsd:restriction base="xsd:string"><xsd:maxLength value="20" /><xsd:minLength value="1" /></xsd:restriction></xsd:simpleType><xsd:simpleType name="DDEMImportDate"><xsd:restriction base="xsd:dateTime"><xsd:minExclusive value="1900-01-01T00:00:00" /> <xsd:maxExclusive value="2099-12-31T12:59:59" /> </xsd:restriction></xsd:simpleType><xsd:simpleType name="SpecimenIDType"><xsd:restriction base="xsd:string"><xsd:maxLength value="24" /><xsd:minLength value="1" /></xsd:restriction></xsd:simpleType><xsd:simpleType name="SpecimenCategoryType"> <xsd:restriction base="xsd:string"><xsd:maxLength value="21" /><xsd:minLength value="1" /><xsd:enumeration value="疑父" /><xsd:enumeration value="疑母" /><xsd:enumeration value="子女" /><xsd:enumeration value="生父" /><xsd:enumeration value="生母" /><xsd:enumeration value="同胞" /><xsd:enumeration value="罪犯" /><xsd:enumeration value="已死亡个体" /><xsd:enumeration value="嫌疑人" /><xsd:enumeration value="受害人" /><xsd:enumeration value="排除嫌疑个体" /><xsd:enumeration value="混合样本" /><xsd:enumeration value="未知样本" /><xsd:enumeration value="配偶" /><xsd:enumeration value="失踪人员" /><xsd:enumeration value="母系样本" /><xsd:enumeration value="父系样本" /><xsd:enumeration value="质控样本" /><xsd:enumeration value="能力测试样本" /><xsd:enumeration value="群体遗传学样本" /> <xsd:enumeration value="其他" /></xsd:restriction></xsd:simpleType><xsd:simpleType name="SpecimenCommentType"> <xsd:restriction base="xsd:string"><xsd:maxLength value="255" /><xsd:minLength value="0" /></xsd:restriction></xsd:simpleType><xsd:simpleType name="SourceIDType"><xsd:restriction base="xsd:string"><xsd:maxLength value="10" /><xsd:minLength value="0" /><xsd:enumeration value="Yes" /><xsd:enumeration value="No" /><xsd:enumeration value="N/A" /></xsd:restriction></xsd:simpleType><xsd:simpleType name="CaseIDType"><xsd:restriction base="xsd:string"><xsd:maxLength value="32" /><xsd:minLength value="0" /></xsd:restriction></xsd:simpleType><xsd:simpleType name="LocusNameType"><xsd:restriction base="xsd:string"><xsd:maxLength value="10" /><xsd:minLength value="1" /><xsd:enumeration value="AMEL" /><xsd:enumeration value="Amelogenin" /><xsd:enumeration value="CSF1PO" /><xsd:enumeration value="D13S317" /><xsd:enumeration value="D16S539" /><xsd:enumeration value="D18S51" /><xsd:enumeration value="D19S433" /><xsd:enumeration value="D21S11" /><xsd:enumeration value="D2S1338" /><xsd:enumeration value="D3S1358" /><xsd:enumeration value="D5S818" /><xsd:enumeration value="D7S820" /><xsd:enumeration value="D8S1179" /><xsd:enumeration value="FGA" /><xsd:enumeration value="Penta D" /><xsd:enumeration value="Penta E" /><xsd:enumeration value="TH01" /><xsd:enumeration value="TPOX" /><xsd:enumeration value="vWA" /><xsd:enumeration value=" GoldenEye 20A " /> </xsd:restriction></xsd:simpleType><xsd:simpleType name="AlleleValueType"><xsd:restriction base="xsd:string"><xsd:maxLength value="10" /><xsd:minLength value="1" /></xsd:restriction></xsd:simpleType><xsd:simpleType name="AlleleSeqSType"><xsd:restriction base="xsd:string"></xsd:restriction></xsd:simpleType><xsd:simpleType name="AlleleSeqLType"><xsd:restriction base="xsd:string"></xsd:restriction></xsd:simpleType><xsd:complexType name="SpecimenType"><xsd:sequence id="SeqSpecimen" minOccurs="1"><xsd:element name="SPECIMENID" type="SpecimenIDType" minOccurs="1" maxOccurs="1" sql:field="SPEC_ID" /><xsd:element name="SPECIMENCATEGORY" type="SpecimenCategoryType" minOccurs="1" maxOccurs="1"sql:field="SPEC_CATEGORY" /><xsd:element name="SPECIMENCOMMENT" type="SpecimenCommentType" maxOccurs="1" minOccurs="0" sql:field="COMMENT" /><xsd:element name="LOCUS" minOccurs="1" maxOccurs="40" sql:relation="Import_Locus" sql:relationship="SpecimenLocus"sql:key-fields="SPEC_ID LOCUS_NAME"><xsd:complexType><xsd:element name="LOCUSNAME" type="LocusNameType" minOccurs="1" maxOccurs="1" sql:field="LOCUS_NAME" /><xsd:element name="READINGBY" type="DDEMUserType" minOccurs="1" maxOccurs="1" sql:field="READING_BY" /><xsd:element name="READINGDATETIME" type="NDNADImportDate" minOccurs="1" maxOccurs="1" sql:field="READING_DT"sql:datatype="datetime" /><xsd:element name="ALLELE" type="AlleleType" minOccurs="1" maxOccurs="8" sql:relation="Import_Allele"sql:relationship="LocusAllele" sql:key-fields="SPEC_ID LOCUS_NAME PCR_VALUE" /><xsd:attribute name="BATCHID" type="BatchIDType" sql:field="BATCH_ID" /><xsd:attribute name="KIT" type="KitType" sql:field="KIT_NAME" /></xsd:complexType></xsd:element><xsd:attribute name="SOURCEID" type="SourceIDType" sql:field="SOURCE_ID" /><xsd:attribute name="CASEID" type="CaseIDType" sql:field="CASE_ID" /><xsd:attribute name="PARTIAL" type="xsd:boolean" sql:field="PARTIAL" /></xsd:complexType><xsd:element name="SPECIMENCATEGORY" type="SpecimenCategoryType" minOccurs="1" maxOccurs="1"sql:field="SPEC_CATEGORY" /><xsd:element name="SPECIMENCOMMENT" type="SpecimenCommentType" maxOccurs="1" minOccurs="0"sql:field="COMMENT" />。
【MyBatis系列3】最全MyBatis中XML映射文件(Mapper)标签分析及示例

【MyBatis系列3】最全MyBatis中XML映射⽂件(Mapper)标签分析及⽰例前⾔MyBatis的强⼤之处就在于它的映射器⽂件,⽽这也正是MyBatis的魔⼒所在,对于任何MyBatis的使⽤者来说,MyBatis的映射⽂件是必须要掌握的。
Mapper⽂件标签Mapper中⼀个提供了9个顶层标签,除了1个已经过期的我们不需要去了解,另外8个都是必须要掌握的,只要熟练掌握了标签的使⽤,使⽤MyBatis才能如鱼得⽔。
接下来我们就⼀个个来分析⼀下这些标签的使⽤。
<cache> – 该命名空间的缓存配置。
<cache-ref> – 引⽤其它命名空间的缓存配置。
<resultMap> – 描述如何从数据库结果集中加载对象,是最复杂也是最强⼤的元素。
<parameterMap> – ⽼式风格的参数映射。
此元素已被废弃,并可能在将来被移除!请使⽤⾏内参数映射。
⽂档中不会介绍此元素。
<sql> – 可被其它语句引⽤的可重⽤语句块。
<insert> – 映射插⼊语句。
<update> – 映射更新语句。
<delete> – 映射删除语句。
<select> – 映射查询语句。
selectselect⽤来映射查询语句,是我们使⽤最多的⼀种标签,也是最复杂的⼀种标签。
⽐如下⾯就是⼀个简单的select标签的使⽤<select id="listUserByUserName" parameterType="String" resultType="lwUser">select user_id,user_name from lw_user where user_name=#{userName}</select>select标签内,提供了⼀些⼆级标签,下⾯就列举出了全部的⼆级标签:<selectid="selectPerson"parameterType="int"parameterMap="deprecated"resultType="hashmap"resultMap="personResultMap"flushCache="false"useCache="true"timeout="10000"fetchSize="256"statementType="PREPARED"resultSetType="FORWARD_ONLY"databaseId="mysql"resultOrdered="false"resultSets="xxx,xxx"lang=""></select>id必选标签。
党建达标创建文件盒标签(竖排)

中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本组织建设组织设置组织设置()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本组织建设组织设置网上党支部()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本活动建设组织关系党组织关系隶属()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本组织建设班子选配班子职数()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本制度建设班子选配书记选配()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本保障建设班子选配委员配置()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本组织建设换届工作班子任期()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本组织建设换届工作按期换届()14141414中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本组织建设动态管理设置调整()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设党员发展规范计划管理()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设党员发展规范发展程序()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设党员发展规范工作监测()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设教育培训方式内容()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设教育培训培训()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设日常管理积分管理()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设日常管理组织关系()1414141414141414中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设日常管理党费缴纳()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设日常管理流动党员()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设激励关怀帮扶人文关怀()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设激励关怀帮扶走访慰问()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设激励关怀帮扶结对帮扶()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本队伍建设激励关怀帮扶表彰激励()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本活动建设支部活动支部主题党日()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本制度建设组织生活制度三会一课()1414141414141414中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本制度建设组织生活制度组织生活会()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本制度建设组织生活制度谈心谈话()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本制度建设组织生活制度民主评议党员()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本制度建设民主议事制度民主议事()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本制度建设党务公开公开内容()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本制度建设党务公开公开程序()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本制度建设党务公开公开方式()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本制度建设党务公开公开时限()14141414中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本保障建设党务工作者配备人员保障()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本保障建设经费保障落实工作经费()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本保障建设活动阵地建管场所保障()中共云南省石油化工锅炉压力容器检测中心站第一党支部规范化建设基本保障建设基础台账党务台账()。
客户标签规范

2013-04-20
受 控
A/1
状 态
WI-PE-07-12-011 附录B
--- 适用范围: AAC内包装 --- 规 格:UN16010001 210*148mm(16小张/大张) --- 颜 色:白底黑字 --- 标签内所有内容以BOM为准。
表示流水号,6位数,黑体8号 表示条形码基数,5位数,,黑体8号,基数由客户提供 表示生产批次(即生产日期),6位数,黑体8号 表示产品编号,11位数,黑体8号 表示供应商代码,4位数,黑体8号
xxxx有限公司
AAC客户标签规范
实施日期 版本 文件编号
一、标签种类:
- 内标签:指每箱内包装中每袋装多件产品的标签。
- 外箱标签:指外包装箱上粘贴的标签。
- 托盘标签:指外包装箱码放在托盘上,整个托盘打包后粘贴的标签。
二、胶箱、纸箱、托盘方向说明:
2013-04-20 A/1
WI-PE-07-12-011 附录B
制订日期:2013.04.15 审批日期:2013.04.18 页数: 第 3 页 共 5 页
xxxx有限公司
AAC客户标签规范
四、AAC不干胶条形码规则
实施日期 版本 文件编号
(1) 内条形码 码制:Code 128
□□□□ □□□□□□□□□□□ □□□□□□ - □□□□□ □□□□□□
内条形码样本:
外条形码样本:
2013-04-20
受 控
A/1
状 态
WI-PE-07-12-011 附录B
--- 适用范围: AAC外箱标签包装 --- 规 格:UN16010001 210*148mm(16小张/大张) --- 颜 色:白底黑字 --- 标签内打印内容以BOM为准,条形码字迹必须清晰。
常用文件格式手册-常用文件格式

标签:文件,文件格式,格式,位图,微软,程序,矢量,字模,是由,波形,表格,公司,源程序,图形,音频,数字,流媒体,标准,超星,机上简介:2常用文件格式手册ISO:标准光盘镜像文件;JAR:JAR压缩文件格式;JPG:JPG原是Apple Mac机器上使用的一种图像格式,现在在PC机上大行其道,其压缩比可以调节,而且失真又很小。
LBL:FOXBASE中的标签文件;LIB:程序库文件;LOG:正文:2常用文件格式手册ISO:标准光盘镜像文件;JAR:JAR压缩文件格式;JPG:JPG原是Apple Mac机器上使用的一种图像格式,现在在PC机上大行其道,其压缩比可以调节,而且失真又很小。
LBL:FOXBASE中的标签文件;LIB:程序库文件;LOG:日记文件;LRC:MP3歌词;LZH:压缩文件格式;M3U:文本文件,存放mp3、rm等多媒体文件列表;MAK:C语言中的工程文件;MEM:FOXBASE中的内存变量文件;MID:数字音频文件(乐器数字接口);MLI:3DS中的材质库;MMM:动画文件,是MacroMind公司著名多媒体写作软件Director天生的;MPG:视频文件,PC机上的全屏幕活动视频的标准文件;MOD、ST3、XT、S3M、FAR、669:该格式的文件里存放乐谱和乐曲使用的各种音色样本,。
现在已经逐渐淘汰,目前只有MOD迷及一些游戏程序中尚在使用。
MOV:QUICKTIME的视频影像格式,同样采用有损压缩方法,在Windows下必须安装QuickTime程序才能播放;MP2、音乐文件;MP3:音频格式,MPEG-1 Audio Layer-3 I格式;NO:一般是文本文件,如:Serial.no,通常还有一种格式为sn.txt,它说是你安装软件的CD-KEY、产品序列号、注册登记号;OBJ:目标文件,源程序编译输出的目标代码;OLD:备份文件,一般是一些程序对系统配置修改后将原文件复制一份存储为该文件格式;PAS:PASCAL语言源程序文件;PCD:是PhotoCD专用存储格式,由于其文件特别大,不得不存在CD-ROM上,但应用特别广;PCM:模拟音频信号经模数转换(A/D变换)直接形成的二进制序列;PCS:为PowerPoint中的图象文件,为矢量图形文件;PCX:是微机上使用最广泛的图像格式之一,能表现真彩图像;PDF:Aodbe acrobat的电子图书格式;PDG:超星电子图书格式;PDV:UCDOS中的自由表格UCTAB天生的表格文件,它天生的文件可以被WPS调用,也挺方便的;PFB:Adobe TYPE1字体Post Script字模文件,必须使用Adobe Type Manager治理安装使用,它的最大优点是放大时比TTF字幕可靠得多;PIC:同上,Lotus 1-2-3的图表文件也叫PIC文件;PIF:Windows中的运行非Windows应用程序建立的一个文件;PJX:Visual FoxPro下的项目文件与辅助文件PJT结合成项目治理文件;PNG:Portable Network Graphics可移植的网络图形,一种新兴的图片格式;PPT、PPS、POT:微软的PowerPoint电子演示文档格式;PRG:FOXBASE过程程序文件;PSD:PDD位图文件,是Adobe Photoshop直接天生的图象文件;PS1:为WPS下扩展字库文件,PS2一般为繁体;PYR:格式全称Pyramid Images。
HTML标签以及各个标签属性大全(网页制作必备)

<NOBR> 取消换行和<br>相反总类(所有HTML文件都有的)--------------------------------------------------------------------------------文件类型<HTML></HTML>(放在档案的开头与结尾)文件主题<TITLE></TITLE>(必须放在「文头」区块内)文头<HEAD></HEAD>(描述性资料,像是「主题」)文体<BODY></BODY>(文件本体)结构性定义(由浏览器控制的显示风格)--------------------------------------------------------------------------------标题<H?></H?>(?表示从1到6,有六层选择)数值越高字越小标题对齐<H? ALIGN=LEFT|CENTER|RIGHT></H?>左对齐、居中对齐、右对齐区分<DIV></DIV>区分对齐<DIV ALIGN=LEFT|RIGHT|CENTER|JUSTIFY></DIV>左对齐、右对齐、居中对齐、两端对齐引文区块<BLOCKQUOTE></BLOCKQUOTE>(通常会内缩)斜体<EM></EM>加粗<STRONG></STRONG>引文<CITE></CITE>(通常会以斜体显示)码<CODE></CODE>(显示原始码之用)样本<SAMP></SAMP>表示一段用户应该对其没有什么其他解释的文本。
aspcms入门教程(标签,文件)

Aspcms各个文件功能介绍全文模版目录说明模版目录位于templates下,默认的是cn模版html目录为模版文件存放的目录ASPCMS系统支持不同栏目采用不同的模板,并且可以自己新建栏目,无需修改ASP文件。
1.头部固定模板为 head.html ,固定调用标签 {aspcms:top}2.底部固定模板为 foot.html ,固定调用标签 {aspcms:foot}3.辅助模版名称可以自己定义,如 left.html ,调用辅助模版的标签为:{aspcms:template src=left.html}网站通用标签单篇文章调用1、首页单篇内容调用{aspcms:about sort=8 }[about:info len=80][about:title][about:link]{/aspcms:about}说明:sort=8单篇文章的栏目id[about:info len=80] 单篇文章内容,len表示截取的长度[about:title]单篇文章的标题[about:link] 单篇文章的链接2、单篇文章页调用(在about.html页标签的调用){aspcms:top} 调用头部{aspcms:template src=left.html} 调用辅助模版{aspcms:sortname} 单篇文章的栏目名称您的位置:首页{aspcms:position} > [position:link]{/aspcms:position} [about:title] 单篇文章标题[about:info] 单篇文章详细内容列表页的调用A.列表页调用标签{aspcms:sorttitle}标题在添加分类时设置{aspcms:sortkeyword}关键词在添加分类时设置{aspcms:sortdesc}描述在添加分类时设置{aspcms:sortname}栏目名称{aspcms:list size=10 order=id}[list:link]链接[list:i]计数[list:titlecolor]标题颜色[list:sortname]分类名称[list:sortlink]分类链接[list:date]日期[list:visits]浏览次数[list:author]作者[list:source]来源[list:tag]tag标签[list:istop]置顶[list:isrecommend]推荐[list:isimage]图片新闻[list:isfeatured]特别推荐[list:isheadline]头条[list:pic]图片{/aspcms:list}[list:pagenumber len=5] 分页条调用标签B.当前位置首页{aspcms:position} >>[position:link]{/aspcms:position}首页 >>新闻发布 >>公司新闻详细页调用1、首页调用{aspcms:content sort=2 num=4 order=order star=1}[content:title]标题[content:link]链接[content:i]计数[content:titlecolor]标题颜色[content:sortname]分类名称[content:sortlink]分类链接[content:date]日期[content:visits]浏览次数[content:author]作者[content:source]来源[content:tag]tag标签[content:istop]置顶[content:isrecommend]推荐[content:isimage]图片新闻[content:isfeatured]特别推荐[content:isheadline]头条[content:desc]描述[content:pic]图片{/aspcms:content}sort 分类IDnum 显示数量order 排序规则 ispic是否是图片order id visits time star 星级(1,2,3,4,5)2、详细页调用[content:title]标题[content:link]链接[content:i]计数[content:titlecolor]标题颜色[content:sortname]分类名称[content:sortlink]分类链接[content:date]日期[content:visits]浏览次数[content:author]作者[content:source]来源[content:tag]tag标签[content:istop]置顶[content:isrecommend]推荐[content:isimage]图片新闻[content:isfeatured]特别推荐[content:isheadline]头条[content:desc]描述[content:pic]图片{aspcms:comment} 评论标签上一篇:{aspcms:prev}下一篇:{aspcms:next}C、多图片的处理count 输出数量,小于1或者不为数字的作为0处理,不输出contentid 输出的内容id,一般在内容页调用,只需填写contentid=[content:id]即可,错误的输入被赋值为-1[cimages:src] 将被替换为/upLoad/product/month_1107/201107011448421775.png等数据循环输出Head部分栏目调用:{aspcms:navlist}∙[navlist:name]{if:[navlist:subcount]>0}{aspcms:subnavlist type=[navlist:sortid]}∙[subnavlist:name]{/aspcms:subnavlist}{end if}{/aspcms:navlist}辅助模版栏目调用:{aspcms:navlist type={aspcms:topsortid}}∙[navlist:name]{/aspcms:navlist}if标签的使用IF标签使用1、满足条件则显示{if:条件语句}显示内容{end if}2、满足条件则显示内容1,否则显示内容2{if:条件语句}显示内容1{else}显示内容2{end if}实例:1,给满足条件的添加不同的样式{aspcms:content}<A&NBSP;{IF:[CONTENT:I]=1} if} link="[content:link]"{end>[content:title]{/aspcms:content}2,显示当前栏目的样式{if:[navlist:sortid]=[navlist:cursortid]}class="nav_off"{else}cla ss="nav_on"{end if}统计标签统计标签今日{visits:today}昨日{visits:yesterday}本月{visits:month}全部{visits:all}友情链接标签调用文字链接{aspcms:linklist type=font}∙[linklist:name]{/aspcms:linklist}调用图片链接{aspcms:linklist type=pic}∙{/aspcms:linklist}type=all 或去掉type属性调用全部的链接tag标签TAG标签内容调用(和内容调用一样,只需要加一个tag属性)标签相关内容调用{aspcms:content sort=2 num=4 order=order tag=aspcms,asp,php star=1} [content:title]标题{/aspcms:content}标签列表调用{aspcms:taglist size=50 order=visits}[taglist:link][taglist:title]{/aspcms:taglist}[taglist:pagenumber len=5] 分页条调用标签order可选值visits,time,id会员功能调用首页调用可以参照模版login.html会员登录后显示用户在线可以用js实现loginyes.asp可以自己建立,位于member目录下代码内容为:document.write("/member/login.asp\" style=\"color:#1c97c0\">[请登录]")document.write("/member/reg.asp\" style=\" color:#1c97c0\">[注册]")document.write("欢迎您, ")document.write(" ")document.write("! | ");document.write("/member/editPass.asp\" style=\"color:#1c97c0\">修改资料");document.write(" | ");document.write("/member/login.asp?action=logout\"style=\"color:#1c97c0\">退出登录");document.write(" |");。
Prometheus学习系列(五)之Prometheus规则(rule)、模板配置说明

Prometheus学习系列(五)之Prometheus规则(rule)、模板配置说明前⾔本⽂来⾃、、、和、、、记录规则⼀、配置规则Prometheus⽀持两种类型的规则,这些规则可以定期配置,然后定期评估:记录规则和。
要在Prometheus中包含规则,请创建包含必要规则语句的⽂件,并让Prometheus通过Prometheus配置中的rule_files字段加载⽂件,规则⽂件使⽤YAML。
通过将SIGHUP发送到Prometheus进程,可以在运⾏时重新加载规则⽂件。
仅当所有规则⽂件格式正确时才会应⽤更改。
⼆、语法检查规则要在不启动Prometheus服务器的情况下快速检查规则⽂件在语法上是否正确,请安装并运⾏Prometheus的promtool命令⾏实⽤⼯具:⼀般下载来整个Prometheus已经包含了promtool⼯具。
go get /prometheus/prometheus/cmd/promtoolpromtool check rules /path/to/example.rules.yml当⽂件在语法上有效时,检查器将已解析规则的⽂本表⽰打印到标准输出,然后以0返回状态退出。
如果存在任何语法错误或⽆效的输⼊参数,则会向标准错误输出错误消息,并以1返回状态退出。
三、录制规则录制规则允许预先计算经常需要或计算上昂贵的表达式,并将其结果保存为⼀组新的时间序列。
因此,查询预先计算的结果通常⽐每次需要时执⾏原始表达式快得多。
这对于仪表板尤其有⽤,仪表板需要在每次刷新时重复查询相同的表达式。
记录和警报规则存在于规则组中,组内的规则以固定间隔顺序运⾏。
规则⽂件的语法是:groups:[ - <rule_group> ]⼀个简单的⽰例规则⽂件将是:groups:- name: examplerules:- record: job:http_inprogress_requests:sumexpr: sum(http_inprogress_requests) by (job)3.1 <rule_group># 组的名称。
合格证及袋签样图
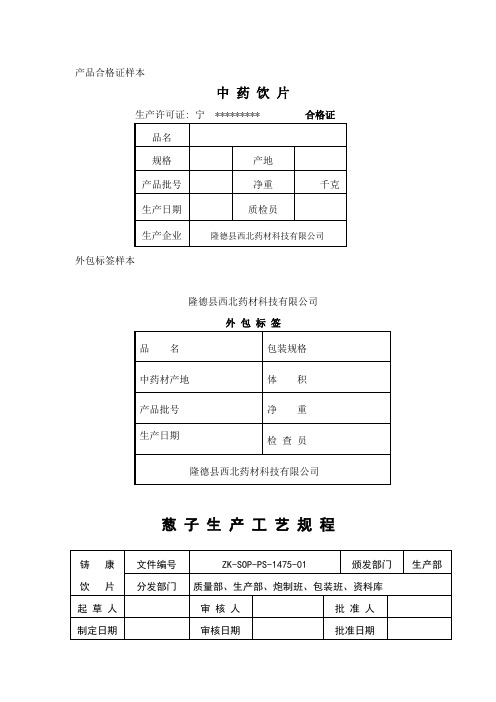
产品合格证样本中药饮片生产许可证: 宁********* 合格证品名规格产地产品批号净重千克生产日期质检员生产企业隆德县西北药材科技有限公司外包标签样本隆德县西北药材科技有限公司外包标签品名包装规格中药材产地体积产品批号净重生产日期检查员隆德县西北药材科技有限公司葱子生产工艺规程铸康饮片文件编号ZK-SOP-PS-1475-01 颁发部门生产部分发部门质量部、生产部、炮制班、包装班、资料库起草人审核人批准人制定日期审核日期批准日期颁发份数 5 版本状态 01 执行日期[品 名]:葱子 [别 名]:[来 源]:为百合科植物葱的干燥成熟种子。
全国各地大部分均产。
[剂 型]:饮片[执行标准]:部颁标准1992版/中药材/第一册/P91 [炮制规格]:净制 生产工艺流程图:[炮制方法]:净 制:除去杂质。
漂洗洗净,沥干水。
80℃以下温度干燥。
盛于洁净容器内,称量,检验合格后随批生产记录交接下一道工序。
[成品性状]:呈类三角状卵形,长3—4mm,宽2--3mm 。
表面黑色,一面微凹,一面隆起,隆起绵有棱线1—2条,光滑或有疏皱纹。
进步有两个小突起,较短的突起为种脐,顶端灰棕色或灰白色;较长的突起顶端为珠孔。
质坚硬,种皮较薄,p 破开后可见灰白色胚芽,富油性。
气特异,味如葱。
领 料入 库包 装包装材料炮 炙方法与程度原药材净 选烘 干 温度、时间洗 药流动水筛 选去除药屑整[炮制作用]:使药洁净,便于调剂和包装。
[用法与用量]:10--30g 。
[成品质量标准]:水 分:不得过8.0% 性状符合炮制规定 [包装材料、规格、质量要求]:按包装岗位操作SOP 执行。
核对品名、炮制规格、产品批号、数量、件数是否检验合格。
并做好交接记录。
核对领用的包装材料、标签、合格证的规格、数量是否相符。
确认无误后,方可包装。
包装过程中,QA 检查员应随机抽查操作程序及包装差异是否符合规定。
包装结束后,应准确统计实际产量,填写批包装记录,QA 检查签字后上报生产部。
第10章__数据库Access2010

打开数据库
10.2.1 数据库的创建与使用
②保存数据库 操作步骤如下: 单击屏幕左上方的“文件”标签,在打开的 Backstage视图中选择“保存”命令,即可保存输入 的信息。 弹出Microsoft Access对话框,提示保存数据库 前必须关闭所有打开对象,单击“是”按钮即可。 或者选择“数据库另存为”命令,可更改数据库 的保存位置和文件名。
10.1.2 Access2010的界面
状态栏
“状态栏”位于窗口底部,用于显示状态信息, 还包含用于切换视图的按钮。
微型工具栏
在Access2010中,可以使用微型工具栏轻松 设置文本格式。用户选择要设置格式的文本后, 微型工具栏会自动出现在所选文本的上方。如果 将鼠标指针靠近微型工具栏,则微型工具栏会渐 渐淡入,用户可以用它来加粗、倾斜、选择字号、 颜色等。如果将鼠标指针移开,则微型工具栏会 渐渐淡出。如果不想使用微型工具栏设置格式, 只需将指针移开一段距离,微型工具栏即会自动 消失。
“外部数据”选项卡
10.1.2 Access2010的界面
“数据库工具”选项卡 利用“数据库工具”选项卡可以完成以下功 能:启动VB编辑器或运行宏、创建或查看表关系、 显示隐藏对象相关性或属性工作表、运行数据库 文档或分析性能、将数据移至数据库、运行链接 表管理器、管理Access加载项、创建或编辑VBA模 块等。
10.2.2 表的建立
创建“联系人”表
表操作
10.2.2 表的建立
使用表设计创建表操作步骤: ①启动Access2010,新建数据库“表示例” ② 切换到“创建”选项卡,单击“表格”组中的 “表设计”按钮,进入表的设计视图。
10.2.2 表的建立
③ 在“字段名称”栏中输入字段的名称“学号”; 在“数据类型”选择该字段的数据类型,这里选择 “数字”选项;“说明”栏可以输入也可以不输入 ④ 用同样的方法,输入其它字段名称,并设置相应 的数据类型。 ⑤ 选择要设为主键(能唯一标识一条记录的字段) 的字段, 在“设计” 选项卡的“工具”组中,单 击 “主键”按钮,即可将其设为主键。
监理单位档案盒标签(全套)

速
第七册
工程变更资料类
目录
1、施工图纸审查报告
2、设计交底和图纸会审
纪要
3、设计变更、洽商文件
4、建设单位变更文件
5、承包单位变更文件
I
第十九册
质量控制类
地基与基础
技术资料
目录
1、工程定位测量记录地基验槽记录
2、检验批质量验收记录
3、隐蔽工程验收记录技术复核记录
4、地下室防水效果检查记录
5、中间结构验收记录
6、地基与基础分部(子分部)质量验收记录
第二十五册质量控制类
消防工程资料
目录
1、消防工程资料
2、消防材料出厂合格证、试验报告
3、消防隐蔽工程验收记录
4、施工记录
5、试验记录
6、运行记录
7、测试记录。
酒标签模板——精选推荐

酒标签模板篇一:标签规定葡萄酒标签的标准葡萄酒的标签主要是要向消费者提供正确准确的信息,是葡萄酒整体质量的一部分。
按标签在瓶子上的部位,分为:顶标、颈标、肩标、前(身)标和后(背)标,还有挂在瓶颈上的飞标。
葡萄酒的标签标准是为了方便国际交流并确保消费者获得公正的信息,它与各国葡萄酒法规密切相关,是葡萄酒质量等级法中不可分割的一部分,国内外都把它列入法律法规管理范畴,在国际上的格式通用且基本一致。
国际上葡萄酒的标签主要内容葡萄酒的称谓:它密切和葡萄酒的等级联系在一起。
在欧共体内酿制的葡萄酒,必须要表明其分类情况,如是佐餐酒、优质酒还是有关规定系列中的一个,比如法国的VIN DE PAYS (地方名酒)或德国的LAND WEIN(乡村酒)。
在各国有关葡萄酒的分类根据下,要描述具体是哪一个。
欧共体之外酿造的葡萄酒在欧共体内通行时,必须要执行说明“葡萄酒”这个辞的规定。
在美国,通常各种葡萄酒表明是“佐餐葡萄酒”。
地理范围:这里先要表明一个国家的名字(如希腊的佐餐葡萄酒),或是美国的一个美国种植区(A V A)、或任何一个州或更小的区域,或者可以是一个小控制命名区域的名称(如意大利西北部的BOCA)。
在法国,常常写作APPELLATION X CONTROLEE,或者写作APPELLATION D’ORIGINE X CONTROLEE,这里的X 就是地名参考。
在欧洲,如果在标签上引用一个地名区域,那么所有的葡萄酒就应该来自这个区域。
在美国,如果在标签上指明一个美国种植区(A V A),那么至少85%的酒应该产自这个区。
澳大利亚也是这个比例。
出口的葡萄酒必须执行国家产区名称的规定。
葡萄酒的容量:标签上表明的有750ml、75 cl、0.75 l或27 fl oz,欧共体国家用75 cl的多,还可以看到“75 cl e”或“e 75 cl”,这里的“e”,是欧共体国家葡萄酒的统一容量标志。
我国的法定计量单位是mL、ml或毫升,L或升。
档案标签(简化)

档案标签(简化)
档案标签(简化)
介绍
档案标签是一种用于标识和分类文件和档案的工具。
它们通常包括有关文件内容、归属、保存日期等重要信息。
本文将介绍档案标签的作用和简化策略。
作用
档案标签的作用主要包括:
1. 标识和区分文件:通过档案标签,可以轻松辨识不同文件和档案,避免混淆和错放。
2. 快速检索:标签中的关键信息可以帮助快速定位所需文件,提高工作效率。
3. 组织和分类:合理使用档案标签可以使文件归类有序,方便管理和维护。
简化策略
为了确保档案标签的简单易用,以下是一些简化策略:
1. 标签字数精简:尽量使用简短、明了的词语或简写,并避免重复信息。
2. 标签颜色分类:通过不同颜色的标签,可以快速识别不同类别的文件。
3. 使用图标或符号:可以在标签上加入适当的图标或符号,以增加可视化效果。
4. 利用条码或二维码:使用条码或二维码可以方便快速扫描和识别标签上的信息。
5. 集中管理标签信息:将标签信息统一保存在一个文档或数据库中,方便管理和查询。
通过采用上述简化策略,可以使档案标签更加简单易用,提高工作效率和组织能力。
总结:
档案标签是一种重要的文件管理工具,通过合理的标识和分类,可以提高工作效率和文件管理能力。
通过简化策略,使标签更加易用,进一步提升工作效率。
llama指令微调的数据集格式样例

标题:llama指令微调的数据集格式样例一、概述在进行深度学习模型的训练和调整过程中,数据集的格式和内容对模型的性能影响重大。
llama指令微调是一种常用的数据集处理工具,为了更好地利用这一工具,本文将介绍llama指令微调的数据集格式样例,并提供详细的格式要求和示例。
二、数据集格式样例要求1. 数据集的标签格式:标签文件是一个以“图片路径标签”为格式的文件,图片路径和标签之间以空格分隔,例如:```/path/to/image1.jpg label1/path/to/image2.jpg label2```2. 数据集的文件夹结构:数据集应该是按类别划分的,每个类别对应一个文件夹,文件夹内包含该类别的所有图片。
3. 数据集的格式要求:数据集应该是可读的,标签应该与图片相对应,数据集应该包含训练集、验证集和测试集。
三、数据集格式样例示例1. 训练集示例:训练集的目录结构如下:```/tr本人n/class1image1.jpgimage2.jpg.../class2image1.jpgimage2.jpg......```训练集的标签文件(tr本人n.txt)内容如下:```/tr本人n/class1/image1.jpg label1/tr本人n/class1/image2.jpg label1 .../tr本人n/class2/image1.jpg label2/tr本人n/class2/image2.jpg label2 ...```2. 验证集示例:验证集的目录结构和标签文件格式与训练集相似,这里不再赘述。
3. 测试集示例:测试集同样需要按照类别划分,并且需要有对应的标签文件。
四、总结llama指令微调的数据集格式样例要求数据集的标签格式正确,并且要求数据集的文件夹结构符合规范。
本文提供了训练集、验证集和测试集的数据集格式样例示例,希望对使用llama指令微调进行数据集处理的开发者提供一定的帮助。
tiff文件结构

tiff文件结构1. 什么是TIFF文件?TIFF(Tagged Image File Format)是一种常见的图像文件格式,它被广泛应用于数字摄影、印刷、扫描和档案存储等领域。
TIFF文件具有灵活的结构,能够存储多种类型的图像数据,以及附加的元数据和标签信息。
2. TIFF文件的基本结构TIFF文件由一系列的标签(tags)组成,每个标签都包含了一些元数据和图像信息。
这些标签按照特定的规则进行组织,以形成一个层次结构。
TIFF文件的基本结构如下所示:---------------------------------------------------| File Header | Image File Directory | Image Data |---------------------------------------------------•文件头(File Header):位于文件的开头,包含了一些基本信息,如文件版本号、字节顺序(大端或小端)等。
•图像文件目录(Image File Directory,IFD):包含了一系列的标签,每个标签都描述了一个特定的图像属性,如图像的宽度、高度、颜色模式等。
•图像数据(Image Data):存储了实际的图像像素数据。
3. 标签(Tags)的组织方式TIFF文件中的标签采用了一种类似于字典的结构,每个标签都由一个标签号(Tag ID)和一个值(Value)组成。
标签号用于标识标签的类型,而值则存储了标签的具体内容。
标签的组织方式如下所示:------------------------------------------| Tag ID | Type | Count | Value/Offset |------------------------------------------•标签号(Tag ID):用于标识标签的类型,每个标签都有一个唯一的标签号。
