uniprot 蛋白库下载及添加到PD流程
Uniprot数据库介绍及信息检索下载指南

UniProt数据库一、UniProt数据库简介蛋白质组常用数据库——UniProt数据库,是信息最丰富、资源最广的蛋白质数据库。
它由Swiss-Prot、TrEMBL 和PIR-PSD三大数据库的数据整合而成,数据主要来自于基因组测序项目完成后,后续获得的蛋白质序列,并包含了大量来自文献的蛋白质生物功能的信息。
一般蛋白质组搜库首选数据库也是UniProt,所以对于通过UniProt库搜库的组学数据,可以在此网站中进行蛋白功能查询。
UniProt数据库可以提供的信息包括蛋白功能描述、GO条目、细胞定位、组织特异性表达情况、生理病理情况描述、互作蛋白、Domain、翻译后修饰位点等信息。
蛋白的信息描述段落均会标出引用文章,并且可以跳转到PubMed界面进行浏览。
UniProt 数据库由UniProt 知识库(UniProtKB )、UniProt 档案(UniParc )、UniProt 参考资料库(UniRef)以及UniProt元基因组学与环境微生物序列数据库(UniMES)构成。
UniProtKB全称 UniProt Knowledgebase(UniProt知识库)它是经过专家校验的数据集,主要由两部分组成:UniProtKB/Swiss-Prot (包含检查过的、手工注释的条目) 和 UniProtKB/TrEMBL (包含未校验的、自动注释的条目)。
Swiss-Prot 数据库特点高质量的、手工注释的、非冗余的数据集;主要来自文献中的研究成果和E-value校验过计算分析结果。
有质量保证的数据才被加入该数据库!TrEMBL数据集包含高质量的计算分析结果,一般都在自动注释中富集,主要应对基因组项目获得的大量数据流以人工校验在时间上和人力上的不足。
它能注释所有可用的蛋白序列。
在三大核酸数据库(EMBL-Bank/GenBank/DDBJ)中注释的编码序列都被自动翻译并加入该数据库中。
它也有来自PDB数据库的序列,以及Ensembl、Refeq和CCDS基因预测的序列。
uniprot蛋白分区

uniprot蛋白分区摘要:1.引言2.什么是uniprot3.uniprot 蛋白分区的概述4.uniprot 蛋白分区的具体分类5.uniprot 蛋白分区的应用6.结论正文:【引言】蛋白质是生命活动中不可或缺的重要组成部分,它们在细胞中承担着各种生物学功能。
对蛋白质的研究,有助于我们深入理解生命活动的本质。
uniprot 是一个关于蛋白质的数据库,它包含了全球科学家们在蛋白质研究领域所取得的成果。
今天我们将重点介绍uniprot 蛋白分区。
【什么是uniprot】uniprot(Universal Protein Resource)是一个蛋白质信息数据库,它旨在为全球科学家提供一个免费、开放、全面的蛋白质数据资源。
uniprot 收录了大量蛋白质的序列、结构、功能等方面的信息,是蛋白质研究领域最为重要的数据库之一。
【uniprot 蛋白分区的概述】uniprot 蛋白分区是对蛋白质进行系统性分类的一种方法。
通过对蛋白质的分区,有助于我们更好地了解蛋白质的结构、功能和调控机制,从而为相关领域的研究提供便利。
【uniprot 蛋白分区的具体分类】uniprot 蛋白分区主要根据蛋白质的功能、结构、调控机制等方面进行分类。
常见的分区包括:1.酶类:催化生物体内化学反应的蛋白质。
2.结构蛋白:构成细胞和组织结构的蛋白质。
3.调节蛋白:参与细胞内信号传导和调控的蛋白质。
4.转运蛋白:负责物质跨膜运输的蛋白质。
5.免疫相关蛋白:参与免疫应答的蛋白质。
6.核酸结合蛋白:与核酸结合并参与相关生物学过程的蛋白质。
7.代谢相关蛋白:参与生物体内代谢反应的蛋白质。
等等。
【uniprot 蛋白分区的应用】uniprot 蛋白分区在蛋白质研究领域具有广泛的应用,包括:1.蛋白质功能预测:通过分析蛋白质的分区信息,可以推测蛋白质可能具有的功能。
2.蛋白质互作网络构建:根据蛋白质的分区信息,可以推测蛋白质之间可能存在的相互作用。
蛋白质数据库使用说明

引言:蛋白质数据是生物信息学领域中非常重要的资源之一,它提供了大量关于蛋白质序列、结构、功能以及相互作用等方面的信息。
本文旨在介绍如何使用蛋白质数据库,帮助用户更好地利用这一资源进行研究。
概述:蛋白质数据库是一个集成了许多蛋白质信息的在线资源,用户可以通过搜索、浏览、等方式获取所需的信息。
其中,常用的蛋白质数据库包括NCBI、UniProt、PDB等。
这些数据库提供了丰富的蛋白质数据,并且不断更新以满足用户需求。
正文内容:1.数据库搜索功能1.1.关键词搜索1.1.1.输入蛋白质名称1.1.2.输入序列片段1.1.3.输入关键词1.2.高级搜索选项1.2.1.提供更精确的搜索结果1.2.2.支持过滤和排序功能1.2.3.可以根据相关字段进行搜索2.数据库浏览功能2.1.蛋白质分类2.1.1.按物种分类2.1.2.按功能分类2.1.3.按家族分类2.2.数据表格浏览2.2.1.查看蛋白质基本信息2.2.2.查看蛋白质序列2.2.3.查看蛋白质结构2.3.数据图谱浏览2.3.1.查看蛋白质相互作用网络2.3.2.查看蛋白质结构域分布2.3.3.查看蛋白质功能注释3.数据库功能3.1.蛋白质序列数据3.1.1.全部序列3.1.2.特定物种的序列3.2.蛋白质结构数据3.2.1.已解析的蛋白质结构3.2.2.蛋白质结构预测结果3.3.蛋白质相互作用数据3.3.1.已验证的相互作用数据3.3.2.预测的相互作用数据4.数据库工具与资源4.1.序列比对工具4.1.1.BLAST4.1.2.PSIBLAST4.2.结构预测工具4.2.1.SWISSMODEL4.2.2.Phyre24.3.功能注释资源4.3.1.GeneOntology4.3.2.InterPro4.4.数据库交互接口4.4.1.提供API接口4.4.2.支持数据提交与5.数据库更新与维护5.1.数据更新频率5.2.数据质量保证5.3.用户反馈与支持5.4.数据库版本与历史记录总结:蛋白质数据库为研究人员提供了丰富的蛋白质信息资源,通过搜索、浏览、等功能,用户可以轻松地获取需要的数据。
第三讲:Uniprot蛋白数据库及其他蛋白质分析工具

第三讲Uniprot蛋白数据库及其他蛋白质分析工具2013/03/19Uniprot数据库•Uniprot(Universal protein resource)是蛋白质序列的联合数据库。
–SIB: Swiss Institute of Bioinformatics–EBI: European Bioinformatics Institute–PIR: Protein Information Resource–2002年三家联合形成了UniprotSwiss‐Prot•1986年建立•低冗余度•功能导向•由Swiss Institute of Bioinformatics 和EBI共同建立并维护TrEMBL •TrEMBL=Translation from EMBL •EBI建立并维护•是一个自动数据库•冗余度高,可信度低UniprotKB•部分经过专家注释的数据库•具有很高的可信度•包括两部分UniprotKB/Swiss‐Prot和UniprotKB/TrEMBL•UniprotKB/Swiss‐Prot包括539,165条序列•UniprotKB/TrEMBL包括29,769,971 条序列•具有非冗余性Uniparc•非冗余性•给予序列的特异性,非同一物种的相同序列被认为是同一个蛋白质•每一条序列被給予一个特异的编号Uniparc•INSDC EMBL‐Bank/DDBJ/GenBank nucleotide sequence databases•Ensembl•European Patent Office (EPO)•FlyBase•H‐Invitational Database (H‐Inv)•International Protein Index (IPI)•Japan Patent Office (JPO)•Protein Information Resource (PIR‐PSD)•Protein Data Bank (PDB)•Protein Research Foundation (PRF) RefSeq•Saccharomyces Genome Database (SGD)•The Arabidopsis Information Resource (TAIR)•TROME•US Patent Office (USPTO)•UniProtKB/Swiss‐Prot, UniProtKB/Swiss‐Prot protein isoforms, UniProtKB/TrEMBL •Vertebrate and Genome Annotation Database (VEGA)•WormBaseUniRef•包括UniRef100,UniRef90和UniRef50•分别包括了相似度为100%,90%和50%的序列的总和UniMES•UniMES是metagenomics和环境生物学的序列数据库•其中的数据可能是未知的•UniMES提供UniRef类似的聚类功能Uniprot的应用•在质谱领域有广泛的应用–因为其序列的非冗余性–举例:质谱分析–举例:Pyruvate: ferredoxin oxidoreductasesubunit alpha from Pyrococcus furiosus蛋白质的结构域‐‐二级库• 根据序列比对的策略不同存在较多的蛋白质序 列二级库,比如ProSite,PRINT, ProDom, Pfam, Gene3D,PANTHER, PIRSF,Tigrfams等等 • 目前诸多蛋白质序列二级库已经被整合到 Interpro数据库中 • 利用Interpro可以查找并鉴定蛋白质的结构 域,可能的功能基团以及预测其生理功能等 • 举例:查询actin‐like protein,找到其三维结构 和功能 • 举例:查询4Fe‐4S cluster binding site蛋白质序列分析‐interproscan蛋白质的保守结构域• 举例:利用interpro分析gene symbol为 MA0658的蛋白质,并预测它可能结合什么 cofactorpI和分子量的预测• /compute_pi/• 举例:预测大肠杆菌中WrbA的pI和分子量对信号肽的预测• SignalP 4.0 • http://www.cbs.dtu.dk/services/SignalP/ • 利用神经网络和HMM模型预测信号肽 • VKLIMFLLMVPLFSYLAAASLRVLSPNPASCDSPEL GYQCNSETTHTWGQYSPFFSVPSEISPSVPEGCR对膜蛋白和跨膜区域的预测• 一般来说是一个20AA长的alpha helix • TMpred • /software/TMPRED_f orm.html • TMHMM • http://www.cbs.dtu.dk/services/TMHMM/ • msyntslgls enivaalcyp vgwlsglffl llerknkfvr fhamqsvllf mpialfiflv awiptigwfi adgagmtaml lilipmymaf rgskfkipii gniaynfayg eExPASy• SIB运作的一个蛋白质专业网站蛋白质结构和功能的分析与预测Blast寻找相似 蛋白功能 利用Uniprot 分析结构域 分析蛋白质 的位置 利用Interpro 分析结构域 分析蛋白质 的MW和pI 已知序列 阅读相似蛋 白的文献提出蛋白质 功能的假说已知名称寻找序列。
uniprot实际应用

进入UNIPROT页面,搜索所需要的数据库
下载数据库
选取对应的格式进行下 载
此为下载完成后的UNIPROTMOUSE数据库
PD软件界面
添加数据库到软件
点击add进行添加
选择UNIPROT-HUMAN数据库
新建一个搜索
添加要搜索的数据
在设置参数中 选择数据库
打开搜索完成后的文件
稍后对 P10809在数 据库网页中 进行搜索
得到蛋白得分 ,双击可查看 蛋白序列
蛋白序列
蛋白在数据库 中对应的序列
对P10809进行数据库中搜索, 得到其具体信息
并且我们可以得到其数据库中的序列, 可与前面分析得到的数据比较进行评价
从软件分析中我们还可以得知在该来自据库 中符合该蛋白的肽段将所得数据导入EXCEL表,方便数据的查 询与筛选
谢谢大家!
UNIPROT数据库应用实例
医学生物化学与分子生物学
2014602619
郭振昌
实例:提取HELA细胞全细胞裂解液中的蛋 白,通过质谱分析得到相应肽段,再利用 PROTEOME DISCOVERER软件搜索HELA细胞 中蛋白的种类 意义:通过比较HELA细胞与正常细胞的蛋 白种类,进一步确定细胞癌变后蛋白种类 的变化,为研究治疗药物奠定基础
uniprot蛋白分区

uniprot蛋白分区摘要:一、前言二、UniProt蛋白数据库介绍三、UniProt蛋白分区概述1.蛋白质结构域2.功能域3.蛋白质家族域四、UniProt蛋白分区的应用1.蛋白质结构预测2.蛋白质功能预测3.蛋白质保守性分析五、结论正文:一、前言蛋白质是生命体系中功能最为多样的大分子,对于生物体的生长、发育、繁殖等过程起着至关重要的作用。
UniProt数据库作为目前最为全面的蛋白质信息资源库,提供了大量关于蛋白质的注释信息。
在这些注释信息中,蛋白分区是一个重要的组成部分,对于研究蛋白质的结构与功能有着重要的意义。
本文将对UniProt蛋白分区进行概述和分析,并探讨其在蛋白质结构预测、功能预测以及保守性分析等方面的应用。
二、UniProt蛋白数据库介绍UniProt是一个综合性的蛋白质信息数据库,它包含了来自多个物种的蛋白质序列、功能注释、保守性等信息。
UniProt数据库的建立旨在为生物学家、研究人员提供一个全面、准确、易于使用的蛋白质信息平台,以促进蛋白质科学的发展。
三、UniProt蛋白分区概述蛋白分区是根据蛋白质序列特征将蛋白质划分为不同结构域和功能域的过程。
UniProt蛋白分区主要包括以下三个方面:1.蛋白质结构域蛋白质结构域是指在蛋白质序列中具有一定功能的连续氨基酸残基。
结构域是蛋白质的三维结构中相对独立的部分,通常具有特定的功能和结构特征。
UniProt蛋白分区通过将蛋白质序列划分为结构域,有助于研究蛋白质的结构与功能关系。
2.功能域功能域是指在蛋白质序列中具有一定功能的连续氨基酸残基,通常与蛋白质的结构域不重叠。
功能域主要关注蛋白质的功能,而不关注其结构。
UniProt蛋白分区通过将蛋白质序列划分为功能域,有助于研究蛋白质的功能和结构域之间的关系。
3.蛋白质家族域蛋白质家族域是指在蛋白质序列中具有一定相似性和功能的连续氨基酸残基,通常来源于蛋白质家族成员之间的共享序列。
蛋白质家族域有助于研究蛋白质序列的保守性和进化关系,从而揭示蛋白质功能的起源和进化过程。
uniprot使用方法

uniprot使用方法一、什么是UniProt?UniProt(Universal Protein Resource)是一个全球性的蛋白质数据库,致力于提供蛋白质序列、结构、功能和概述相关信息的公共资源。
UniProt 由三个组件组成:UniProtKB、UniRef和UniParc。
其中,UniProtKB是最主要的组件,它包含了三个子数据库:Swiss-Prot、TrEMBL和PROSITE。
1. Swiss-Prot:Swiss-Prot是一个经过人工注释和校正的蛋白质序列数据库,提供了详细的蛋白质功能和注释信息。
2. TrEMBL:TrEMBL是一个基于计算的蛋白质序列数据库,它包含了从未经过详细注释的Swiss-Prot数据集中的序列。
这些序列待进一步注释和校正后会被转移到Swiss-Prot数据库中。
3. PROSITE:PROSITE是一个用于识别蛋白质序列中保守结构域和模体的数据库。
它提供了一系列的蛋白质域和模体的特征模式和描述。
UniRef是一个聚类蛋白质序列数据库,用于提高蛋白质注释效率,减少重复注释。
UniParc是一个蛋白质数据库,用于记录已知和未知蛋白质序列的标识符。
二、使用UniProt的步骤使用UniProt数据库可以帮助研究者快速获取蛋白质信息,查找已知蛋白质、发现新的蛋白质序列和结构等。
以下是使用UniProt的步骤:1. 访问UniProt官方网站,地址为2. 在搜索框中输入要查询的蛋白质名称、序列或标识符等关键词,并选择搜索类型。
3. 点击“搜索”按钮进行搜索。
4. UniProt将会显示与搜索关键词相关的蛋白质信息列表。
用户可以根据需求筛选蛋白质数据库(如Swiss-Prot或TrEMBL)或其他过滤条件,以缩小搜索范围。
5. 点击感兴趣的蛋白质条目,将显示该蛋白质的详细信息页面。
用户可以阅读蛋白质的注释信息、功能描述、序列特征、结构域、文献引用等内容。
6. 若需要进一步了解蛋白质的结构、亚细胞定位等信息,用户可以点击相关链接或标签,以跳转到其他相关数据库或工具。
UniProt:蛋白质的全信息数据库

Nucleic Acids Research, 2004, Vol. 32, Database issue D115-D119© 2004 Oxford University PressUniProt:蛋白质的全信息数据库摘要为了给科学界提供一个专门,集中,权威的蛋白质序列和功能的信息资源,瑞士-Prot,TrEMBL 和PIR蛋白质数据库已经合作组成了蛋白质的全信息数据库 (UniProt)。
我们的目的是用广泛的对照和询问接口来提供一个全面的,分类完全的,丰富并且准确的蛋白质序列信息。
中心数据库将有两个部分:符合熟悉的瑞士-Prot(完全手工操作入口)和TrEMBL(使用丰富的自动化的分类,注释和广泛的对照)。
为方便序列查寻,UniProt也提供几个无冗余的序列数据库。
UniProt NREF(UniRef)数据库为高效率的搜寻提供适当的蛋白质的全信息数据库的代表性的子集。
全面的UniProt 档案(UniParc)每天从很多公共来源数据库更新。
数据库那些UniProt接口可在线访问()或者以几个形式下载(ftp:///pub)。
我们鼓励科学界人士向UniProt 提供数据。
介绍近来,瑞士-Prot + TrEMBL和PIR-PSD如同蛋白质数据库不同的序列信息覆盖面和注释优势共存。
2002年,在生物信息科学(SIB)的瑞士研究所和欧洲生物信息科学研究所的瑞士-Prot + TrEMBL 组 (EBI)和蛋白质信息资源(PIR)组织在乔治敦大学医学中心和国家生物医学的研究基金会联合协作。
新联合的组织的主要任务是通过建立一个综合,详细分类,丰富并且准确注释蛋白质序列的优质的数据库和广泛序列对比和询问服务的到科学团体免费接口—knowledgebase来支持生物学的研究。
UniProt 将在组织成员多年合作的坚实基础上建立起来。
UniProt 数据库包括3 个数据库层:1、UniProt 档案(UniParc),通过储存全部可公开得到的蛋白质序列数据供一个稳定,综合,无冗余的序列收集。
UniProt:蛋白质的全信息数据库

Nucleic Acids Research, 2004, Vol. 32, Database issue D115-D119© 2004 Oxford University PressUniProt:蛋白质的全信息数据库摘要为了给科学界提供一个专门,集中,权威的蛋白质序列和功能的信息资源,瑞士-Prot,TrEMBL 和PIR蛋白质数据库已经合作组成了蛋白质的全信息数据库 (UniProt)。
我们的目的是用广泛的对照和询问接口来提供一个全面的,分类完全的,丰富并且准确的蛋白质序列信息。
中心数据库将有两个部分:符合熟悉的瑞士-Prot(完全手工操作入口)和TrEMBL(使用丰富的自动化的分类,注释和广泛的对照)。
为方便序列查寻,UniProt也提供几个无冗余的序列数据库。
UniProt NREF(UniRef)数据库为高效率的搜寻提供适当的蛋白质的全信息数据库的代表性的子集。
全面的UniProt 档案(UniParc)每天从很多公共来源数据库更新。
数据库那些UniProt接口可在线访问()或者以几个形式下载(ftp:///pub)。
我们鼓励科学界人士向UniProt 提供数据。
介绍近来,瑞士-Prot + TrEMBL和PIR-PSD如同蛋白质数据库不同的序列信息覆盖面和注释优势共存。
2002年,在生物信息科学(SIB)的瑞士研究所和欧洲生物信息科学研究所的瑞士-Prot + TrEMBL 组 (EBI)和蛋白质信息资源(PIR)组织在乔治敦大学医学中心和国家生物医学的研究基金会联合协作。
新联合的组织的主要任务是通过建立一个综合,详细分类,丰富并且准确注释蛋白质序列的优质的数据库和广泛序列对比和询问服务的到科学团体免费接口—knowledgebase来支持生物学的研究。
UniProt 将在组织成员多年合作的坚实基础上建立起来。
UniProt 数据库包括3 个数据库层:1、UniProt 档案(UniParc),通过储存全部可公开得到的蛋白质序列数据供一个稳定,综合,无冗余的序列收集。
实验四 蛋白质数据库uniprotkb的使用

生命科学学院生物技术091 罗信旭 0907040118实验四蛋白质数据库—uniprotkb/swiss-prot的使用一、实验目的:掌握蛋白质数据库uniprotkb/swiss-prot的蛋白质序列的查询方法,了解uniprotkb/swiss-prot数据库提供的有关蛋白质的信息资源。
二、实验原理:uniprotkb/swiss-prot蛋白质序列数据库是由SIB和EBI共同维护和管理,并随EMBL 数据库一起发行。
目前,该数据库是最为常用、注释最全、包含独立项最多的数据库,它包括其他蛋白质序列库中经过验证的全部序列、其注释及蛋白质的功能、结构域和活性位点、二级结构、四级结构、翻译后修饰、与其他蛋白质的相似性等内容。
三、实验器材:计算机,uniprotkb/swiss-prot蛋白质序列数据库。
四、实验内容:应用我们需要查找的物种的拉丁学名或蛋白质的名称,在uniprotkb/swiss-prot蛋白质序列数据库中进行查询,由此获得我们所需要查找的蛋白质序列。
五、实验步骤:1、输入域名/,打开EBI主界面,找到主界面下方的database一栏,点击下拉菜单,点击protein目录下uniprotkb/swiss-prot一栏,进入蛋白质序列数据库的Search界面。
2、找到uniprotkb/swiss-prot蛋白质序列数据库界面上端的Find一栏,在方框中输入需要查询的关键词,即物种的拉丁文学名或蛋白质的名称,然后开始搜索。
3、在进入查询结果界面后,选择界面下方的protein sequence/uniprotkb中的序列,点击in uniprot或in SRS,可以看到与查询要求相关的各种结果,根据实验要求将其拷贝下来即可。
六、实验要求:每个组每个同学至少查找四个蛋白质的序列。
将查询结果中的Sequence、Accession、similarity、Organism source等结果拷贝下来作为实验结果。
uniprot蛋白批量转换基因 -回复

uniprot蛋白批量转换基因-回复如何将Uniprot蛋白批量转换为基因序列?Uniprot是一个国际性的蛋白质序列数据库,它提供了大量已知蛋白质的序列和相关信息。
有时候我们可能需要将Uniprot中的蛋白质序列转换为相应的基因序列。
在本文中,我们将一步一步介绍如何使用常见的工具和技术来完成批量转换过程。
第一步,获取Uniprot蛋白ID列表。
在Uniprot网站( ID复制到一个文本文件中。
您可以使用Excel或任何文本编辑器将这些ID整理成一列。
第二步,批量下载蛋白质序列。
在ncbi网站上,有一个小工具叫Seqret,它可以帮助我们快速下载蛋白质序列。
首先,将您保存有Uniprot ID的文本文件上传到ncbi的Sequence Retrieval System(SRS)服务器。
然后,将SRS的搜索结果导出为FASTA格式的文件,该文件将包含与Uniprot ID对应的蛋白质序列。
第三步,使用BioMart进行基因转换。
BioMart是一个功能强大的生物信息学工具,可以进行生物序列和注释信息的转换和查询。
打开BioMart网站(Accession(s)"并将下载的FASTA文件中的Uniprot ID 复制到相应的文本框中。
然后在"Attributes"选项卡中选择您希望查询的基因属性,例如基因名或基因序列。
第四步,导出基因序列。
完成查询后,您可以导出结果并选择将查询结果导出为文本文件或其他格式文件。
在导出文件中,您将获得与Uniprot ID对应的基因序列。
第五步,数据整理和分析。
一旦您获取了转换后的基因序列,您可以使用Excel或任何其他工具来整理和分析这些数据。
您可以根据需要对基因序列进行进一步的分析,例如基因同源性比对或进化分析。
总结:将Uniprot蛋白批量转换为基因序列可以通过几个简单的步骤完成。
首先,获取您需要转换的Uniprot蛋白ID列表。
然后,使用Seqret 工具下载蛋白质序列。
蛋白质组学数据库的下载
蛋白质组学数据库的下载
在做蛋白组学的实验过程中,数据库的下载是一个不能忽视的问题,数据库不匹配的话会对整个实验的结果造成很大的影响,那么如何下载数据库呢?
我们通常用到的公共数据库有2个,一个是uniprot,应用十分广泛,另一个是NCBI。
UniProt是Uni versal Prot ein的英文缩写,是信息最丰富、资源最广的蛋白质数据库,Swiss-Prot、TrEMBL 和PIR-PSD 三大数据库的数据而成。
它的数据主要来自于基因组测序项目完成后,后续获得的蛋白质序列。
它包含了大量来自文献的蛋白质的生物功能的信息,也是目前蛋白组学文章中使用最广泛的公共数据库。
以人(Homo sapiens)为例进行数据库下载。
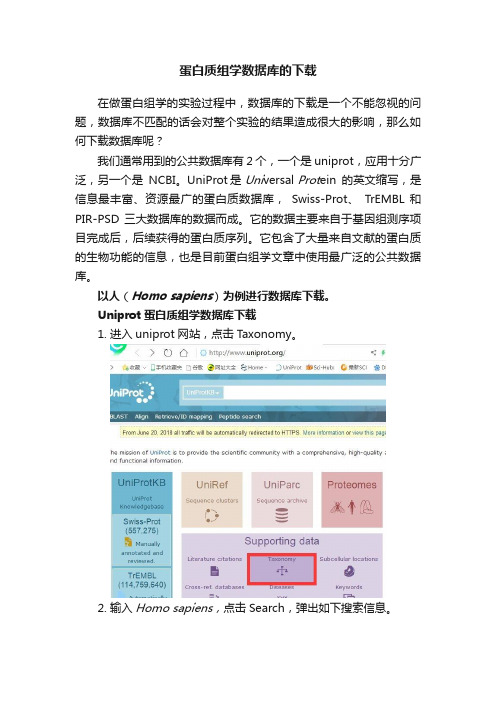
Uniprot蛋白质组学数据库下载
1.进入uniprot网站,点击Taxonomy。
2.输入Homo sapiens,点击Search,弹出如下搜索信息。
3.点击上图红框UniprotKB,弹出如下对话框,点击download,然后点击GO,即可下载该物种的数据库。
NCBI蛋白质组学数据库的下载
1.进入NCBI首页,选择Taxonomy。
2.输入Homo sapiens,点击Search,弹出如下搜索信息。
3.点击Protein,弹出如下界面,点击RefSeq。
4.点击Send to,send to下拉菜单选file,fasta文件,TaxonomyID,然后create file,保存文件。
好了,今天就介绍到这里了。
蛋白质数据库使用说明

蛋白质数据库使用说明1.高级查询 (1)2.限定词说明 (1)3.显示格式说明 (2)3.1.Summary显示格式 (2)3.2.FASTA显示格式 (3)3.3.SwissProt显示格式 (4)4.数据下载流程 (5)5.数据提交 (6)6.附录 (6)6.1.蛋白质研究的历史 (6)6.2.蛋白质组学与生物信息学 (7)2009年10月16日1.高级查询在首页上点击“数据库”按钮,选择“蛋白数据库”进入蛋白质数据库主页。
在蛋白质主页的左侧栏点击“高级检索”,进入如下图的高级检索页面:蛋白质数据库的高级检索可以最多使用三个限定词来进行更精确的检索,三个限定词之间可以用“AND”和“OR”相连接,其中“AND”表示查询的结果中必须包含它所连接的两个关键词,“OR”表示查询的结果中至少包含它所连接的关键词中的一个。
在左侧的限定词框中可以选择的限定词包括:CAC、AC、Entry Name、Description、Tax ID、Organism、Keywords、Gene Name、Organelle、Length以及Molecular Weight等十一个限定词。
其中Length和Molecular Weight可以进行范围查询。
2.限定词说明蛋白质数据库中相关的限定词说明如下:限定词描述CAC国内用户提交的数据编号AC SwissProt的序列或记录唯一的接收编号Entry Name录入名Description描述Tax ID物种分类号Organism与蛋白质有关的物种的学名和通用名Keywords与其它数据库专用词汇有关的索引名词Gene Name基因的标准名和通用名Organelle细胞器官Length序列的总长度Molecular Weight蛋白质的分子量,单位为道尔顿(Da)3.显示格式说明蛋白质数据库的查询结果有三种显示结果:Summary、FASTA和SwissProt。
3.1.Summary显示格式Summary格式显示了蛋白质条目的摘要信息,主要有两部分组成:1)蛋白质的名称,AC号以及来源物种2)对蛋白质的简要描述信息,如该组成该蛋白质的亚基和生物功能等3.2.FASTA显示格式FASTA格式第一行显示信息包括蛋白质录入名、AC号以及来源物种。
蛋白质序列PIR和PDB使用方法

随着核酸数据库不断发展以及数据库的建立,蛋白质序列、结构、功能不断引起人们的重视,生命科学的研究中蛋白质的研究显得尤为重要,一系列的蛋白质序列数据随之产生,数据库也在研究蛋白质的过程中有着不可或缺的地位。
本文主要通过实验说明蛋白质序列数据库PIR及蛋白质结构数据库PDB的使用方法,返回结果的含义,以及如何下载数据和批量下载数据。
由于蛋白质序列测定技术先于DNA序列测定技术问世,蛋白质序列的搜集也早于DNA序列。
蛋白质序列数据库的雏形可以追溯到60年代。
60年代中期到80年代初,美国国家生物医学研究基金会(National Biomedical Research Foundation,简称NBRF)Dayhoff领导的研究组将搜集到的蛋白质序列和结构信息以“蛋白质序列和结构地图集”(Atlas of Protein Sequence and Structure)的形式发表,主要用来研究蛋白质的进化关系。
时至今日,国际上已建立了许多关于生物分子的数据库,主要包括基因组图谱数据库、核酸序列数据库、蛋白质序列数据库、蛋白质结构数据库、生物大分子结构数据库等。
这些数据库均为公共数据库,由特定的组织维护、以及发布相关序列信息,供生物研究学者使用,称为生物研究中的必要工具之一,随着科学技术的发展,这些数据库不断壮大,也为研究人员提供了大量有用的数据。
本文主要通过课程实验,展示蛋白质序列数据库PIR及蛋白质结构数据库PDB的相关使用方法。
本论蛋白质序列数据库PIR介绍1984年,“蛋白质信息资源”(Protein Information Resource,简称PIR)计划正式启动,蛋白质序列数据库PIR也因此而诞生。
与核酸序列数据库的国际合作相呼应,1988年,美国的NBRF、日本的国际蛋白质信息数据库(Japanese International Protein Information Database,简称JIPID)和德国的慕尼黑蛋白质序列信息中心(Munich Information Center for Protein Sequences,简称MIPS)合作成立了国际蛋白质信息中心(PIR-International),共同收集和维护蛋白质序列数据库PIR。
用uniprot检索蛋白序列

用uniprot检索蛋白序列
Uniprot是世界上最大的蛋白质数据库之一,包括了大量的蛋白质序列、功能、结构和相互作用等信息。
在进行生物信息学研究的过程中,常常需要从Uniprot中检索蛋白质序列,以便进行后续的分析和研究。
在使用Uniprot检索蛋白序列时,首先需要进入Uniprot网站并进行
注册,注册完成后,即可开始使用Uniprot提供的各种功能。
在检索
蛋白序列之前,可以先根据需要设置相关的检索条件,例如蛋白名称、序列长度、生物学功能、亚细胞定位和物种等信息,以提高检索效率
和准确性。
在输入检索条件后,Uniprot会返回符合条件的蛋白质列表,用户可
以根据需要选择感兴趣的蛋白质进行查看和下载。
同时,Uniprot还
提供了详细的蛋白质信息,包括基因名称、功能注释、结构域、同源
序列、文献引用和序列特性等,这些信息可以帮助研究人员更深入地
了解蛋白质的生物学特性和功能。
Uniprot不仅提供了蛋白质序列的检索和下载服务,还提供了多种工
具和数据库,例如序列比对、序列注释、蛋白结构、基因本体论、互
作网络和化学生物学等,这些工具和数据库可以帮助研究人员深入地
探究蛋白质的结构和功能。
同时,Uniprot还定期更新数据库,保持
其信息的完整性和准确性。
综上所述,Uniprot是一个非常有价值的蛋白质数据库,在生物信息学研究中发挥着重要的作用,可以帮助研究人员更加深入地了解蛋白质的生物学特性和功能。
同时,通过Uniprot检索蛋白序列,研究人员可以为后续的生物信息学研究提供有力的支持和帮助。
uniprot 蛋白库下载及添加到PD流程

uniprot 蛋白库下载及添加到PD流程
Uniprot网址
/
1、打开网址
2、如果选择单个蛋白的数据库,则选择UniProtKB
3、输入蛋白名称,如breast milk,回车搜索。
结果显示有10, 395个蛋白。
4、点击download,在对话框中选择download all,FASTA文件可以选择正式蛋白,或者正式加同分异构体;选择不压缩文件。
点击GO,save。
5、如果选择下载蛋白质组,则选择Proteomes,在随后的搜索框中输入比如“Homo Spiens”(human 智人的学名),其他同上方法下载。
注意:下载过程中可能会下载不完全而显示下载完成的情况,这可能是由于网速不好造成的,可以多下载几次,比对数据库大小的差异,选择较为完整的数据库。
二、
1、打开PD软件,在任务栏点击Administration下拉的Maintain FASTA Files
2、在打开的窗口任务栏点击Add,找到所下载的数据库文件即可。
3、等待状态显示为“Completed”即为添加完成。
4、打开原有Workflow平台,在Sequest HT下更改Protein Database文件即可。
5、在菜单栏中Workflow下选择Save As Template一个新名称即可。
【技术分享】 UniProt 数据库使用指南
【技术分享】UniProt 数据库使用指南1753 人阅读发布时间:2020-07-01 13:42在这个信息大爆炸的年代,浩瀚的信息流如同汪洋大海一般,将每个人都紧紧环绕。
如何利用已有的工具从中筛选有用信息,对我们每个人,尤其是科研人而言,尤为重要。
在免疫学,医学,药学科研和工作中,我们常常会用到蛋白数据库,关于蛋白质的结构,蛋白质质谱等数据库均较多,今天给大家分享讲讲使用频率最高且冗余度最低的uniprot数据库。
希望能对你们有所帮助~下面,菲小恩以研究得比较多的IL-6为例,分享如何有机结合数据库和产品信息。
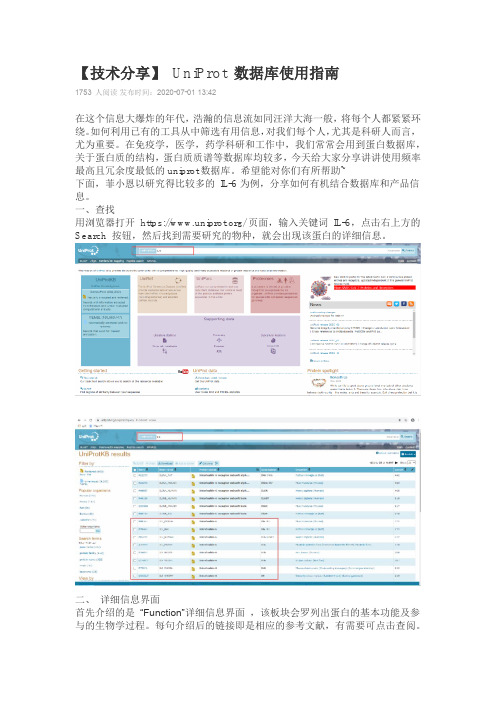
一、查找用浏览器打开https:/// 页面,输入关键词IL-6,点击右上方的Search 按钮,然后找到需要研究的物种,就会出现该蛋白的详细信息。
二、详细信息界面首先介绍的是“Function”详细信息界面,该板块会罗列出蛋白的基本功能及参与的生物学过程。
每句介绍后的链接即是相应的参考文献,有需要可点击查阅。
三、“Names & Taxonomy” 板块随后的“Names & Taxonomy” 板块展示的是命名和来源种属信息、NCBI 和Enzembl 的基因数据库链接,可直接点击查阅。
NCBI 和Enzembl 数据库也可查询。
四、表达谱和定位之后便是与实验密切相关的重要信息:蛋白的组织细胞表达谱和亚细胞定位。
我们在进行IHC、IF 实验时,常常会遇到一个重要而棘手的问题:我看到的阳性信号是不是特异的?UniProt 就可以给予一定的支持信息,在“Expression” 板块,阐述多细胞生物中,基因(mRNA 水平/蛋白水平)在细胞/组织中的表达情况;在“Subcellular location” 板块,包含成熟蛋白在细胞中的定位和拓扑结构信息。
综合以上信息,我们不难发现,IL-6在体内是普遍存在的,在脂肪和淋巴组织中会更高。
它的亚细胞定位是内质网,细胞膜和分泌表达,那么IHC 的阳性信号主要是细胞间隙,以及细胞外围一圈和细胞内的核周围。
uniport使用方法

uniport使用方法
好的,以下是重新生成的UniProt使用方法:
1. 打开UniProt官方网站,您可以使用搜索引擎输入UniProt进行搜索,或者直接在网址栏输入UniProt官方网址。
2. 进入UniProt首页后,您可以选择浏览数据库中的内容,也可以使用搜索功能查找特定蛋白质或基因相关信息。
3. 在搜索框中输入您要查询的蛋白质名称、序列或标识符等关键词,选择搜索类型,然后点击“搜索”按钮。
4. UniProt将显示与搜索关键词相关的蛋白质信息列表。
您可以根据需要筛选结果,例如选择特定的蛋白质数据库或应用其他过滤条件。
5. 点击感兴趣的蛋白质条目,将进入该蛋白质的详细信息页面。
这里您可以查看该蛋白质的描述、结构域、修饰、相互作用等信息。
6. UniProt还提供了多种工具和资源,例如UniProtKB、UniRef、UniGene等,您可以根据自己的需求选择使用。
需要注意的是,为了能够使用UniProt的所有功能,您需要在UniProt上注册并登录。
注册时需要填写常用的邮箱地址,并设置一个强密码,包括数字、字母和字符,且长度达到6-8位以上,以保护账户安全。
蛋白质相关下载方法及PyMOL简单应用

大目录一、使用pymol前的准备小目录1.确定你的蛋白质(以草酸氧化酶Oxalate oxidase为例)2.下载蛋白质.PDB3.下载相关文献4.通过浏览网页了解蛋白质(unipro,PDBsum)5.阅读中文文献(万方、知网等)二、用Pymol进行简单的作图本文采用从PDB数据库下载的文件1FI2与2ET1(1FI2为天然蛋白,2ET1为与配体结合的蛋白)做操作例子。
小目录1.导入pdb文件。
2.蛋白质的简单处理(显示二级结构条带模型)。
3.显示活性中心(1)寻找活性中心金属离子:(2)寻找与活性部位金属离子结合氨基酸残基(3)标记氨基酸残基(4)寻找与金属离子结合的水分子(5)最后的修改。
4.配体的处理。
(1FI2是没有配体的,我们打开有配体的2ET1)5.让模式图更加好看(以活性部位为例)一、使用pymol前的准备小目录1.确定你的蛋白质(以草酸氧化酶Oxalate oxidase为例)2.下载蛋白质.PDB3.下载相关文献4.通过浏览网页了解蛋白质5.阅读中文文献(万方、知网等)1.确定你的蛋白质(以草酸氧化酶Oxalate oxidase为例)在中搜索Oxalate oxidase。
下拉网页,一共出现5个结果,找到你需要的。
从下图很明显可以看出第一个是重组蛋白,所以我选择了第二个。
2.下载蛋白质.PDB3.下载相关文献:点击PubMed右侧编码,如下得到新网页,即NCBI中相关文献,如下图,点击右侧会弹出新窗口下拉新网页,找到真正的下载按钮,下载并了解它。
一般对应的文献会对相关蛋白的结构功能有较清楚的描述。
本例中,我们会发现最初的5个结果中有4个都是大麦中分离的草酸氧化酶的不同三维结构,这四个蛋白,分别为天然草酸氧化酶蛋白,与配体结合的草酸氧化酶蛋白,重组草酸氧化酶蛋白,突变草酸氧化酶蛋白。
这些会在文献中告之,我们也可以在中发现:而另一种蛋白应该是来源于其他生物体,比如细菌。
它的序列与大麦的序列有一定差别。
Uniprot蛋白数据库

1. Experimental evidence at protein level 蛋白质水平实验证据
2. Experimental evidence at tran rotein inferred from homology
从同源蛋白质推断
4. Protein predicted
基本局部比对搜索工具
i Align: t BLAST是对单个蛋白序列与数据库数据进行比对,i Align可以让你对多个蛋白质的序列之间进行相似性比对,这 种分析可以让你找到这些蛋白之间的结构保守区域,还可以根据蛋白质的相似性,分析这些蛋白之间的亲缘关系,进化 的先后顺序等。
= Download:这个容易理解,您可以下载蛋白的序列数据,可以下载选中的蛋白,也可以下载全部蛋白(做蛋白质组学 分析的人经常用,否则没有必要全部下载,不过有时候最好定期下载更新一下,还是很有必要的)下载的格式也多种多 样,比如FASTA,Text,Excel,XML,List等,如果数据量大,还可以选择压缩以后下载。 b Add to basket: 这个按钮的功能是可以随时将你感兴趣的蛋白质条目加入购物篮以备后期使用,最多可以加400条数据 ,呵呵,这个不是超市的购物篮,是不收费的。
检索区主要是为了让有经验同学快速找到自己感兴趣的蛋白质,可以按照蛋白质的名称、序列,ID号等方式检索。 红框1区是对这个主题区域的简要介绍
红框2区可以让游客根据自己的喜好来显示蛋白质数据,比如可以只显示经过人工校验的条目;可以只看专属于某个物 种的蛋白质;还可以检索物种分类;还有直接快速进入UniRef的通道以及视频化的帮助,演示文件
进入方式多种多样,1、主页默认的入口就是UniProt;2、可以直接点击红框1区域进入;3、也可以通过点击红框6,系 统会弹出下拉菜单如图2所示,选择UniProt红框1即可进入。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
3、输入蛋白名称,如breast milk,回车搜索。
结果显示有10, 395个蛋白。
4、点击download,在对话框中选择download all,FASTA文件可以选择正式蛋白,或者正式加同分异构体;选择不压缩文件。点击GO,save。
5、如果选择下载蛋白质组,则选择Proteomes,在随后的搜索框中输入比如“Homo Spiens”(human智人的学名),其他同上方法下载。
注意:下载过程中可能会下载不完全而显示下载完成的情况,这可能是由于网速不好造成的,可以多下载几次,比对数据库大小的差异,选择较为完整的数据库。
二、
1、打开PD软件,在任务栏点击Administration下拉的Maintain FASTA Files
2、在打开的窗口任务栏点击Add,找到所下载的数据库文件即可。
3、等待状态显示为low平台,在Sequest HT下更改Protein Database文件即可。
5、在菜单栏中Workflow下选择Save As Template一个新名称即可
