甲基化测序word版
亚硫酸氢盐修饰后测序法

甲基化检测方法(亚硫酸氢盐修饰后测序法)甲基化是目前的研究热点,就我所做的一点工作并其中一点心得,与大家分享。
希望能够对大家有所帮助。
第一部分基因组DNA的提取。
这一步没有悬念,完全可以购买供细胞或组织使用的DNA提取试剂盒,如果实验室条件成熟,自己配试剂提取完全可以。
DNA比较稳定,只要在操作中不要使用暴力,提出的基因组DNA应该是完整的。
此步重点在于DNA的纯度,即减少或避免RNA、蛋白的污染很重要。
因此在提取过程中需使用蛋白酶K及RNA酶以去除两者。
使用两者的细节:1:蛋白酶K可以使用灭菌双蒸水配制成20mg/ml;2:RNA酶必须要配制成不含DNA酶的RNA酶,即在购买市售RNA酶后进行再处理,配制成10mg/ml。
否则可能的后果是不仅没有RNA,连DNA也被消化了。
两者均于-20度保存。
验证提取DNA的纯度的方法有二:1:紫外分光光度计计算OD比值;2:1%-1.5%的琼脂糖凝胶电泳。
我倾向于第二种方法,这种方法完全可以明确所提基因组DNA的纯度,并根据Marker的上样量估计其浓度,以用于下一步的修饰第二部分亚硫酸氢钠修饰基因组DNA如不特别指出,所用双蒸水(DDW)均经高压蒸汽灭菌。
1:将约2ugDNA于1.5mlEP管中使用DDW稀释至50ul;2:加5.5ul新鲜配制的3M NaOH;3:42℃水浴30min;水浴期间配制:4:10mM对苯二酚(氢醌),加30ul至上述水浴后混合液中;(溶液变成淡黄色)5: 3.6M亚硫酸氢钠(Sigma,S9000),配制方法:1.88g亚硫酸氢钠使用DDW稀释,并以3M NaOH滴定溶液至PH 5.0,最终体积为5ml。
这么大浓度的亚硫酸氢钠很难溶,但加入NaOH后会慢慢溶解,需要有耐心。
PH一定要准确为5.0。
加520ul至上述水浴后溶液中。
6:EP管外裹以铝箔纸,避光,轻柔颠倒混匀溶液。
7:加200 ul 石蜡油,防止水分蒸发,限制氧化。
dna甲基化测序原理

dna甲基化测序原理DNA甲基化测序原理DNA甲基化测序是一种用于检测DNA分子上甲基化修饰形式的方法。
甲基化是一种常见的DNA化学修饰形式,它可以影响基因的表达和细胞分化过程。
通过测序和分析DNA中的甲基化位点,科学家能够深入研究这种修饰对基因组功能的影响。
甲基化测序的原理基于DNA甲基化位点与未甲基化位点之间的区别。
在DNA分子中,甲基化通常发生在CpG位点(即DNA的Cytosine和Guanine碱基之间的连接点)。
未甲基化的CpG位点在测序中会被处理成TpG位点,而甲基化的CpG位点则保持不变。
甲基化测序通常通过两种方法进行:1. 亚硫酸转换法(Bisulfite Conversion):该方法通过使用亚硫酸盐将未甲基化的CpG位点转换为尿嘧啶(T),而甲基化的CpG位点则保持不变。
经过亚硫酸转换后的DNA样本可以通过测序技术直接检测出甲基化位点和未甲基化位点的差异。
2. 甲基化特异性切割(Methylation-specific cleavage):该方法使用甲基化特异性的限制性内切酶来识别甲基化的CpG位点,并在CpG位点附近切割DNA。
未甲基化的CpG位点由于没有甲基化修饰而不被切割。
通过测序这些切割的DNA片段,可以确定DNA分子上的甲基化位点的位置。
甲基化测序技术的发展为研究DNA甲基化在基因组中的分布和功能提供了重要的工具。
科学家可以利用这些技术研究不同细胞类型、病理状态以及环境因素对甲基化模式的影响,以及通过甲基化修饰调控基因表达的机制。
这些研究对于揭示基因组调控和疾病发生机制具有重要意义。
甲基化测序原理

甲基化测序原理甲基化测序是一种用于检测DNA甲基化的方法。
DNA甲基化是一种重要的表观遗传修饰方式,在多种生物过程中发挥重要作用,如基因表达调控、细胞分化、基因组稳定性维持等。
甲基化测序已经成为研究癌症、神经退行性疾病等疾病的重要手段之一。
甲基化测序原理是基于DNA甲基化后所形成的5-甲基脱氧胞嘧啶(5-mC)与未甲基化的脱氧胞嘧啶(dC)在化学和物理性质上的不同之处。
甲基化测序主要分为两种类型:全基因组甲基化测序和目标区域甲基化测序。
全基因组甲基化测序是指将整个基因组的所有区域进行甲基化测序,而目标区域甲基化测序是指仅测序已知某些基因的甲基化状态。
在全基因组甲基化测序中,首先需要将DNA样品进行加样性处理,以消除非特异性结果。
接着通过酶切将整个基因组的DNA进行裂解,然后使用甲基化特异性的抗体来富集甲基化的DNA片段。
然后,将富集后的DNA片段进行二代测序,以获得所有DNA片段的甲基化情况。
在目标区域甲基化测序中,采用PCR扩增或捕获技术,只测序目标区域的甲基化状态。
在PCR扩增的情况下,可以设计特定的引物,使PCR仅扩增目标区域的DNA片段。
在捕获技术中,通常使用探针来捕获目标区域的DNA,然后进行二代测序,以获得目标区域的甲基化情况。
通过这些甲基化测序技术,可以获得DNA甲基化的高分辨率图谱,并进一步了解甲基化在基因表达调控和基因组可塑性等方面的功能和作用。
甲基化测序技术还可以用于诊断和治疗风险评估,以及个体化医疗。
DNA甲基化是一种非常重要的表观遗传修饰方式,甲基化测序技术可以帮助我们更深入地理解甲基化在基因表达调控和基因组可塑性等方面的功能和作用。
随着技术的不断更新和完善,相信甲基化测序技术将会在疾病预防、基因治疗和精准医疗等领域发挥越来越重要的作用。
甲基化测序技术的应用癌症:DNA甲基化调节基因活性,已经成为许多癌症的发病机制之一。
甲基化测序技术已被广泛应用于癌症研究中,以识别癌症相关的甲基化变化,并作为新型癌症治疗的候选靶点。
甲基化测序

DNA甲基化检测实验一、重亚硫酸盐的测序法实验流程(BSP)(Bisulfite Genomic Sequence)原理:结合重亚硫酸盐的测序法是一种灵敏的能直接检测分析基因组DNA甲基化模式的方法。
重亚硫酸盐处理后,用针对改变后的DNA序列设计特异性引物并进行聚合酶链式反应(PCR)。
PCR产物中原先非甲基化的胞嘧啶位点被胸腺嘧啶所替代,而甲基化的胞嘧啶位点保持不变。
PCR产物克隆后进行测序。
通过这个方法能得到特定位点在各个基因组DNA 分子中的甲基化状态。
该方法特点是:•特异性高,它能够提供特异性很高的分析结果,这是所有其他研究甲基化的分析方法所不能比拟的;•灵敏度高,可以用于分析少于100个细胞的检测样品。
用微量的基因组DNA进行分析就能得到各个DNA分子精确的甲基化位点分布图。
重亚硫酸盐测序法技术实验流程A.DNA制备用DNA抽提试剂盒(Promega, cat. no. A1125)抽提组织,细胞培养物,石蜡包埋组织切片样品中的基因组DNA。
B.重亚硫酸盐处理C.DNA纯化用Wizard DNA clean-up kit (Promega, cat. no. A7280)纯化重亚硫酸盐处理后的DNA样品。
D.PCR扩增E.PCR产物琼脂糖电泳后回收纯化F.PCR产物连接到pMD19-T (Takara) 载体中克隆及测序。
G.用分析软件对各样本测序结果进行甲基化程度分析二、甲基化特异性的PCR实验流程(methylation-specific PCR, MSP)原理:甲基化特异性的PCR是一种灵敏度高且操作相对简单的甲基化研究方法。
重亚硫酸盐处理DNA后,基因组DNA发生的由甲基化状态决定的序列改变。
随后进行引物特异性的PCR。
该方法引物设计是关键。
MSP中设计两对引物,即一对结合处理后的甲基化DNA链(引物对M),另一对结合处理后的非甲基化DNA链(引物对U)。
检测MSP扩增产物,如果用引物M能扩增出片段,则说明检测位点存在甲基化;若用引物U扩增出片段,则说明被检测的位点不存在甲基化(图3)。
(完整版)甲基化检测方法(亚硫酸氢盐修饰后测序法)

甲基化检测方法(亚硫酸氢盐修饰后测序法)第一部分基因组DNA的提取。
这一步没有悬念,完全可以购买供细胞或组织使用的DNA提取试剂盒,如果实验室条件成熟,自己配试剂提取完全可以。
DNA比较稳定,只要在操作中不要使用暴力,提出的基因组DNA应该是完整的。
此步重点在于DNA的纯度,即减少或避免RNA、蛋白的污染很重要。
因此在提取过程中需使用蛋白酶K及RNA酶以去除两者。
使用两者的细节:1:蛋白酶K可以使用灭菌双蒸水配制成20mg/ml;2:RNA酶必须要配制成不含DNA酶的RNA酶,即在购买市售RNA酶后进行再处理,配制成10mg/ml。
否则可能的后果是不仅没有RNA,连DNA也被消化了。
两者均于-20度保存。
验证提取DNA的纯度的方法有二:1:紫外分光光度计计算OD比值;2:1%-1.5%的琼脂糖凝胶电泳。
我倾向于第二种方法,这种方法完全可以明确所提基因组DNA的纯度,并根据Marker的上样量估计其浓度,以用于下一步的修饰。
第二部分亚硫酸氢钠修饰基因组DNA如不特别指出,所用双蒸水(DDW)均经高压蒸汽灭菌。
1:将约2ugDNA于1.5mlEP管中使用DDW稀释至50ul;2:加5.5ul新鲜配制的3M NaOH;3:42℃水浴30min;水浴期间配制:4:10mM对苯二酚(氢醌),加30ul至上述水浴后混合液中;(溶液变成淡黄色)5:3.6M亚硫酸氢钠(Sigma,S9000),配制方法:1.88g亚硫酸氢钠使用DDW稀释,并以3M NaOH滴定溶液至PH 5.0,最终体积为5ml。
这么大浓度的亚硫酸氢钠很难溶,但加入NaOH后会慢慢溶解,需要有耐心。
PH一定要准确为5.0。
加520ul至上述水浴后溶液中。
6:EP管外裹以铝箔纸,避光,轻柔颠倒混匀溶液。
7:加200 ul 石蜡油,防止水分蒸发,限制氧化。
8:50℃避光水浴16h。
一般此步在4pm开始做,熟练的话不到5pm即可完成,水浴16h正好至次日8am 以后收,时间上很合适。
甲基化-亚硫酸氢盐修饰后测序法

DNA 甲基化(英语:DNA methylation )为DNA 化学修饰的一种形式,能在不改变DNA 序列的前提下,改变遗传表现。
为序列的前提下,改变遗传表现。
为外遗传编码外遗传编码(epigenetic code )的一部分,是一种)的一部分,是一种外遗传外遗传机制。
DNA 甲基化过程会使甲基化过程会使甲基甲基添加到DNA 分子上,例如在分子上,例如在胞嘧啶胞嘧啶环的5'碳上:这种5'方向的DNA 甲基化方式可见于所有脊椎动物。
甲基化方式可见于所有脊椎动物。
在人类细胞内,大约有1%的DNA 碱基受到了受到了甲基化甲基化。
在成熟在成熟体细胞组织体细胞组织中,DNA 甲基化一般发生于CpG 双核苷酸(CpG dinucleotide )部位;而非CpG 甲基化则于甲基化则于胚胎干细胞胚胎干细胞中较为常见[1] [2]。
植物体内胞嘧啶的甲基化则可分为对称的CpG (或CpNpG ),或是不对称的CpNpNp 形式(C 与G 是碱基;p 是磷酸根;N 指的是任意的核苷酸)。
指的是任意的核苷酸)。
特定胞嘧碇受甲基化的情形,可利用特定胞嘧碇受甲基化的情形,可利用亚硫酸盐定序亚硫酸盐定序(bisulfite sequencing )方式测定。
DNA 甲基化可能使基因沉默化,进而使其失去功能。
此外,也有一些生物体内不存在DNA 甲基化作用。
甲基化作用。
参考文献1.Dodge, Jonathan E.; Bernard H. Ramsahoyeb, Z. Galen Woa, Masaki Okanoa, En Li. 1.Dodge, Jonathan E.; Bernard H. Ramsahoyeb, Z. Galen Woa, Masaki Okanoa, En Li. De novo methylation of MMLV provirus in embryonic stem cells: De novo methylation of MMLV provirus in embryonic stem cells: CpG versus non-CpG methylation . Science Direct. 2002.May.2.Haines, Thomas R.; Rodenhiser, David I.; Ainsworth, Peter J. J. Allele-Specific Allele-Specific Non-CpG Methylation of the Nf1 Gene during Early Mouse Development . Science Direct. 2001.Dec.DNA 甲基化是最早发现的基因表观修饰方式之一,真核生物中的甲基化仅发生于胞嘧啶,即在DNA 甲基化转移酶(DNMTs )的作用下使CpG 二核苷酸5’5’--端的胞嘧啶转变为5’5’--甲基胞嘧啶。
甲基化测序

甲基化测序
甲基化测序是当今生命技术发展的一个重要方向,也是当前热门的研究领域之一。
甲基化测序(Methylation Sequencing,MS)技术被用于研究 DNA 甲基化的
结构和功能,旨在理解一个物种或个体的基因组动态变化过程。
甲基化是基因组中一种重要的组成部分,它会影响基因转录过程和蛋白质活性,甲基化水平也会影响器官发育、病理变化和凋亡等重要的生物功能。
因此,甲基化测序能够为发现相关基因调控提供紧密、精准的信息,为临床治疗提供有效的策略。
甲基化测序技术比传统的基因组测序及转录组测序更精确,因为甲基化的位点
不易随着环境和药物的变化而发生变化,从而获得更为准确可靠的信息。
此外,由于甲基化测序不仅具有高通量性,还可以获得深度信息,因此被广泛应用于医学研究、分子生物学研究和发育生物学研究等。
甲基化测序技术的应用越来越广泛,从原始的DNA抽提,到实验研究,分析结
果的呈现,互联网技术都发挥了重要作用。
单个实验室把所有的实验流程和工具整合在一个地方,实现一站式的实验,大大降低了实验技术的施工难度,同时互联网技术让交叉比较、数据解读和结果分析变得更加便捷高效。
甲基化测序作为一种重要的生命技术,已经得到了田野中的广泛应用,在科学
研究和临床应用中扮演者重要角色,为科学研究提供新的思路,为临床治疗提供新的技术支持。
未来,由于互联网技术的不断成熟,甲基化测序技术将进一步发展及完善,以提供更加有效的实验、分析和解读服务。
甲基化检测方法精编WORD版

甲基化检测方法精编W O R D版IBM system office room 【A0816H-A0912AAAHH-GX8Q8-GNTHHJ8】甲基化检测(d e t e c t i o n o f m e t h y l a t i o n)概念:DNA甲基化是最早发现的基因表观修饰方式之一,真核生物中的甲基化仅发生于胞嘧啶,即在DNA甲基化转移酶(DNMTs)的作用下使CpG二核苷酸5'-端的胞嘧啶转变为5'-甲基胞嘧啶。
DNA甲基化通常抑制基因表达,去甲基化则诱导了基因的重新活化和表达。
这种DNA修饰方式在不改变基因序列前提下实现对基因表达的调控。
脊椎动物DNA的甲基化状态与生长发育调控密切相关,比如在肿瘤发生时,抑癌基因CpG岛以外的CpG序列非甲基化程度增加,CpG岛中的CpG则呈高度甲基化状态,导致抑癌基因表达的下降。
现有检测方法1.甲基化特异性的PCR(Methylation-specific PCR,MSP)用亚硫酸氢盐处理基因组DNA,所有未发生甲基化的胞嘧啶被转化为尿嘧啶,而甲基化的胞嘧啶不变;随后设计针对甲基化和非甲基化序列的引物进行PCR。
通过电泳检测MSP扩增产物,如果用针对处理后甲基化DNA链的引物能得到扩增片段,则说明该位点存在甲基化; 反之,说明被检测的位点不存在甲基化。
2.亚硫酸氢盐测序法(Bisulfite sequencing PCR,BSP)用亚硫酸氢盐处理基因组DNA,则未发生甲基化的胞嘧啶被转化为尿嘧啶,而甲基化的胞嘧啶不变。
随后设计BSP引物进行PCR,在扩增过程中尿嘧啶全部转化为胸腺嘧啶,最后对PCR产物进行测序就可以判断CpG位点是否发生甲基化称为BSP-直接测序方法。
将PCR 产物克隆至载体后进行测序,可以提高测序成功率,这种方法称为BSP-克隆测序法。
3.高分辨率熔解曲线法(High Resolution Melting,HRM)在非CpG岛位置设计一对针对亚硫酸氢盐修饰后的DNA双链的引物,这对引物中间的片段包含感兴趣的CpG岛。
艾德科技m6A RNA甲基化文库构建试剂盒说明书

*仅用于科学研究,不用于诊断与治疗。
请随时跟我们索取并使用最新版本的说明书。
m 6A RNA 甲基化文库构建试剂盒(测序版)此品非常适合从广泛的物种比如从哺乳类动物,植物,真菌和细菌,不仅如此,还包括培养细胞与新鲜和冷冻的组织等感兴趣的样本中提取总RNA 来富集片段化的含有m6A 的RNA 。
整个实验时间不到6小时。
手动操作不到1小时。
目录号:A-P-9016(24次、12次)操作手册在您收到定购的产品时,请确认操作手册是配套的!同时,有翻译不妥的地方还请各位老师批评指正!反馈信箱:2021年11月,第1版,对应英文第20210915版艾德科技(北京)有限公司A&D TechnologyCorporation扫我收藏分享,还有机会拿红包哦!目录表目录表 (2)试剂盒组成 (3)运输和保存 (4)配套器材(自备) (4)重点提示 (4)说明 (5)一般特性 (5)产品简介 (6)原理/步骤 (7)用法 (8)操作手册为了得到最好的实验结果,在实验开始前,请通读这个操作手册。
(8)附录 (13)凝难解答 (13)定购信息 (14)推荐产品 (15)相关产品 (15)如何下单 (15)试剂盒组成内容A-P-9016-12(12次)A-P-9016-24(24次)保存条件WB(Wash Buffer)15ml30ml4°C CB(Capture Buffer)2ml4ml常温NDE(Nuclear Digestion Enhancer)150ul300ul常温CEM(Cleavage Enzyme Mix)*30ul60ul–20°C m6A Antibody(1mg/ml)*25ul50ul–20°C Non-Immune IgG(1mg/ml)*10ul20ul4°C PDB(Protein Digestion Buffer) 2.5ml5ml常温Proteinase K(10mg/ml)*50ul100ul4°C Affinity Beads*50ul100ul4°C RPS(RNA Puritication Solution)300ul600ul常温NA Binding Beads*30ul60ul4°C Elution Buffer1ml2ml常温内容A-P-9016-12(12次)A-P-9016-24(24次)保存条件5X Reaction Bufffer*100ul200ul–20°C RT Enzyme*13ul26ul–20°C Adaptor-A(10uM)*28ul56ul–20°C Reaction Enzyme Mix*25ul50ul–20°C Adaptor-B(10uM)*28ul56ul–20°C MQ Binding Beads* 1.8ml 3.6ml4°C 2X HiFi PCR Master Mix*160ul320ul–20°C Primer U(10uM)*15ul30ul–20°C Primer I(10uM)*15ul30ul–20°C 使用手册11常温运输和保存该试剂盒分二部分运输,第一部分是在室温环境下;第二部分需在4°C加冰袋运输。
MGMT甲基化-SOP
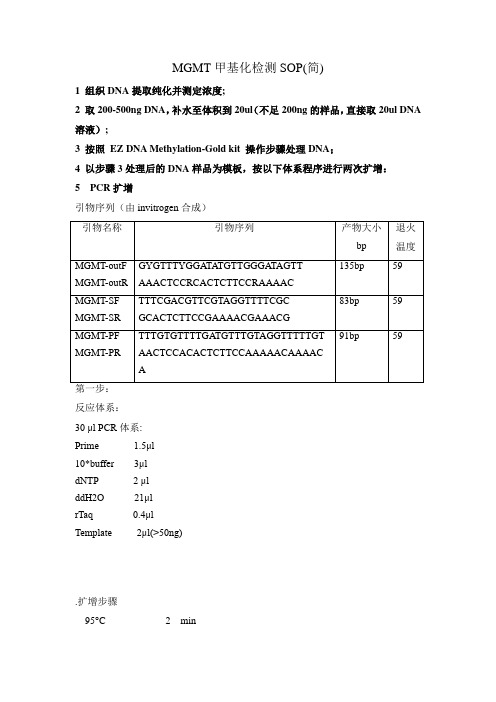
MGMT甲基化检测SOP(简)1 组织DNA提取纯化并测定浓度;2 取200-500ng DNA,补水至体积到20ul(不足200ng的样品,直接取20ul DNA 溶液);3 按照EZ DNA Methylation-Gold kit 操作步骤处理DNA;4 以步骤3处理后的DNA样品为模板,按以下体系程序进行两次扩增:5 PCR扩增引物序列(由invitrogen合成)反应体系:30 μl PCR体系:Prime 1.5μl10*buffer 3μldNTP 2 μlddH2O 21μlrTaq 0.4μlTemplate 2μl(>50ng).扩增步骤95°C 2 min94°C 30 s40* 59°C 30 s72°C 30 s72°C 5 min16°C ∞第二步:反应体系30 μl PCR体系:Prime 1.5μl10*buffer 3μldNTP 2 μlddH2O 22μlrTaq 0.4μlTemplate 1μl.扩增步骤95°C 2 min94°C 30 s40* 59°C 30 s72°C 30 s72°C 5 min16°C ∞6 在3%的琼脂糖凝胶电泳上检测7结果判定情况1:MGMT-SFR(甲基化引物)有扩增MGMT-PFR(非甲基化引物)无扩增该样品为完全甲基化情况2 :MGMT-SFR(甲基化引物)无扩增MGMT-PFR(非甲基化引物)有扩增该样品无甲基化情况3:MGMT-SFR(甲基化引物)有扩增MGMT-PFR(非甲基化引物)有扩增该样品为半甲基化。
(完整版)甲基化检测方法(亚硫酸氢盐修饰后测序法)

(完整版)甲基化检测方法(亚硫酸氢盐修饰后测序法)甲基化检测方法(亚硫酸氢盐修饰后测序法)第一部分基因组DNA的提取。
这一步没有悬念,完全可以购买供细胞或组织使用的DNA提取试剂盒,如果实验室条件成熟,自己配试剂提取完全可以。
DNA比较稳定,只要在操作中不要使用暴力,提出的基因组DNA应该是完整的。
此步重点在于DNA的纯度,即减少或避免RNA、蛋白的污染很重要。
因此在提取过程中需使用蛋白酶K及RNA酶以去除两者。
使用两者的细节:1:蛋白酶K可以使用灭菌双蒸水配制成20mg/ml;2:RNA酶必须要配制成不含DNA酶的RNA酶,即在购买市售RNA酶后进行再处理,配制成10mg/ml。
否则可能的后果是不仅没有RNA,连DNA也被消化了。
两者均于-20度保存。
验证提取DNA的纯度的方法有二:1:紫外分光光度计计算OD比值;2:1%-1.5%的琼脂糖凝胶电泳。
我倾向于第二种方法,这种方法完全可以明确所提基因组DNA的纯度,并根据Marker的上样量估计其浓度,以用于下一步的修饰。
第二部分亚硫酸氢钠修饰基因组DNA如不特别指出,所用双蒸水(DDW)均经高压蒸汽灭菌。
1:将约2ugDNA于1.5mlEP管中使用DDW稀释至50ul;2:加5.5ul新鲜配制的3M NaOH;3:42℃水浴30min;水浴期间配制:4:10mM对苯二酚(氢醌),加30ul至上述水浴后混合液中;(溶液变成淡黄色)5:3.6M亚硫酸氢钠(Sigma,S9000),配制方法:1.88g亚硫酸氢钠使用DDW稀释,并以3M NaOH滴定溶液至PH 5.0,最终体积为5ml。
这么大浓度的亚硫酸氢钠很难溶,但加入NaOH后会慢慢溶解,需要有耐心。
PH一定要准确为5.0。
加520ul至上述水浴后溶液中。
6:EP管外裹以铝箔纸,避光,轻柔颠倒混匀溶液。
7:加200 ul 石蜡油,防止水分蒸发,限制氧化。
8:50℃避光水浴16h。
一般此步在4pm开始做,熟练的话不到5pm即可完成,水浴16h 正好至次日8am 以后收,时间上很合适。
单细胞DNA甲基化测序数据处理流程与分析方法

单细胞DNA甲基化测序数据处理流程与分析方法1. 内容简述单细胞DNA甲基化测序是一种高分辨率的基因表达和表观遗传学研究方法,它允许研究者检测单个细胞的DNA甲基化状态。
这种技术为理解细胞异质性、基因调控机制以及疾病发展中的表观遗传变化提供了有力工具。
样本制备:首先,从生物体中提取单细胞,然后利用亚硫酸盐转化技术将DNA中的甲基化修饰转换为羟基化修饰,以供后续测序。
文库构建:转化后的DNA被随机打断成小片段,并加上特定的接头序列,以便进行PCR扩增和测序。
测序:构建好的文库被加载到测序芯片上,通过高通量测序技术进行测序。
数据分析:获得的原始数据需要经过一系列清洗、比对、标准化等处理步骤,以获得高质量的甲基化数据集。
甲基化状态分析:识别每个细胞中的甲基化位点,并比较不同细胞之间的甲基化差异。
差异甲基化分析:识别在不同实验条件下(如疾病状态、环境压力等)甲基化模式的差异。
生物信息学分析:使用统计软件和算法对数据进行深度挖掘,发现与特定生物学过程或疾病相关的甲基化模式。
通过对这些数据的综合分析,研究者可以揭示细胞功能的动态变化、基因表达的调控机制以及表观遗传学在疾病发生中的作用。
1.1 单细胞DNA甲基化测序技术简介简称SCDBS)是一种高通量、高分辨率的分析方法,用于研究单个细胞中基因组水平的DNA甲基化状态。
该技术通过测序和分析单细胞中的甲基化位点序列,揭示了基因表达差异、发育过程、疾病发生机制等方面的信息。
随着高通量测序技术的快速发展,SCDBS已经成为生物学研究的重要工具之一。
SCDBS的主要流程包括:样品准备、文库构建、测序、数据处理和分析等步骤。
需要将单细胞样本进行处理,如去除血浆等杂质,保证测序结果的准确性。
通过构建文库来存储待测的DNA片段,通常采用Illumina测序平台进行高通量测序。
对测序数据进行质量控制和过滤,以去除低质量序列和伪迹。
利用生物信息学工具对数据进行处理和分析,包括聚类分析、差异基因表达分析、甲基化模式比较等。
甲基化测序

DNA甲基化检测实验一、重亚硫酸盐的测序法实验流程(BSP)(Bisulfite Genomic Sequence)原理:结合重亚硫酸盐的测序法是一种灵敏的能直接检测分析基因组DNA甲基化模式的方法。
重亚硫酸盐处理后,用针对改变后的DNA序列设计特异性引物并进行聚合酶链式反应(PCR)。
PCR产物中原先非甲基化的胞嘧啶位点被胸腺嘧啶所替代,而甲基化的胞嘧啶位点保持不变。
PCR产物克隆后进行测序。
通过这个方法能得到特定位点在各个基因组DNA 分子中的甲基化状态。
该方法特点是:•特异性高,它能够提供特异性很高的分析结果,这是所有其他研究甲基化的分析方法所不能比拟的;•灵敏度高,可以用于分析少于100个细胞的检测样品。
用微量的基因组DNA进行分析就能得到各个DNA分子精确的甲基化位点分布图。
重亚硫酸盐测序法技术实验流程A.DNA制备用DNA抽提试剂盒(Promega, cat. no. A1125)抽提组织,细胞培养物,石蜡包埋组织切片样品中的基因组DNA。
B.重亚硫酸盐处理C.DNA纯化用Wizard DNA clean-up kit (Promega, cat. no. A7280)纯化重亚硫酸盐处理后的DNA样品。
D.PCR扩增E.PCR产物琼脂糖电泳后回收纯化F.PCR产物连接到pMD19-T (Takara) 载体中克隆及测序。
G.用分析软件对各样本测序结果进行甲基化程度分析二、甲基化特异性的PCR实验流程(methylation-specific PCR, MSP)原理:甲基化特异性的PCR是一种灵敏度高且操作相对简单的甲基化研究方法。
重亚硫酸盐处理DNA后,基因组DNA发生的由甲基化状态决定的序列改变。
随后进行引物特异性的PCR。
该方法引物设计是关键。
MSP中设计两对引物,即一对结合处理后的甲基化DNA链(引物对M),另一对结合处理后的非甲基化DNA链(引物对U)。
检测MSP扩增产物,如果用引物M能扩增出片段,则说明检测位点存在甲基化;若用引物U扩增出片段,则说明被检测的位点不存在甲基化(图3)。
(完整版)基因甲基化检测

基因甲基化检测甲基化是在DNA甲基转移酶(DNA Methyltransferase, DNMT)催化作用下, 利用S-腺苷甲硫氨酸(S-adenosylmethionine, SAM)提供甲基,在CpG二核苷酸中胞嘧啶嘧啶环的五号碳原子上加上甲基的共价修饰过程.DNA甲基化是表观遗传修饰的主要方式,它不改变DNA的一级结构,却在细胞的发育、基因的表达、及基因组的稳定性中起着重要的作用。
CpG岛的高甲基化是肿瘤中存在的普遍现象,而启动子CpG 岛的高甲基化是除突变和缺失外肿瘤中抑癌基因失活的第三种机制。
因此,基因启动子甲基化检测在临床诊断(如甲基化检测与低剂量CT相结合)、药物敏感性检测等方面具有很高的应用价值。
目前甲基化特异性PCR(Methylmion Specific PCR,MSP)及其改进方法是检测基因甲基化的经典方法.MSP法的原理是首先用亚硫酸氢钠修饰处理基因组DNA,所有未发生甲基化的胞嘧啶都被转化为尿嘧啶,而甲基化的胞嘧啶则不变。
然后设计针对甲基化和非甲基化序列的引物并进行聚合酶链反应(PCR)扩增,最后通过琼脂糖凝胶电泳分析,确定与引物互补的DNA 序列的甲基化状态。
MSP法灵敏度较高,应用范围广。
一. MSP实验流程:1.基因组抽提;2.基因组DNA定量;3.重亚硫酸盐转化(C转化为U),本步实验至关重要,转化效率高低直接影响实验结果,所以,需要实验者在实验过程中不断摸索合适的实验条件以确保较高的转化效率;4.引物设计(每个基因设计两对引物,分别为:甲基化引物M,非基因化引物U,对应扩增甲基化和非甲基化的目的片段),有时需设计巢式引物;5.PCR扩增:对甲基化和非甲基化的目的片段分别扩增,此时尽可能使用梯度PCR仪,可以同时使用不同的退火温度,以筛选合适的退火温度;6.PCR产物电泳;7.电泳结果分析(定性即有无甲基化,半定量即甲基化程度高低)。
图1:MSP PCR产物电泳图二. qMSP实验流程:1.基因组抽提;2.基因组DNA定量;3.重亚硫酸盐转化(C转化为U),本步实验至关重要,转化效率高低直接影响实验结果,所以,需要实验者在实验过程中不断摸索合适的实验条件以确保较高的转化效率;4.引物和探针设计(每个基因设计一对引物和两条条探针),定量引物和探针的设计难度是比较大的,设计好的引物和探针既需要理论上的分析验证(软件),又需要通过实验验证和筛选,所以一个目的基因有时需设计几对不同引物和探针以供筛选;5.标准品制备:1.完全非甲基化DNA的制备:筛选合适的基因组DNA,确保其完全没有甲基化,作为阴性对照;2.完全甲基化DNA的制备:用甲基转移酶对完全非甲基化的DNA进行甲基化修饰,目的是把DNA中所有的CG中的C甲基化,修饰后的DNA是完全甲基化的,作为阳性对照;3.把A作为0%甲基化,B作为100%甲基化,对A和B进行不同比例的混合,形成梯度甲基化的标准品;4.对C步的系列标准品进行Realtime PCR扩增,制备多个不同的标准曲线;5.根据标准曲线的相关系数,对D步的标准曲线进行筛选,选择合适的标准曲线用于后续的实验.6. PCR扩增:对实验样本进行PCR扩增,此时尽可能使用梯度PCR仪,可以同时使用不同的退火温度,以筛选合适的退火温度.7. Realtime PCR扩增:对实验样品进行定量PCR扩增,同时扩增标准曲线;8. 结果分析:通过PCR结果可以直观的看出每个样本的甲基化程度(百分比)。
甲基化 焦磷酸测序

甲基化焦磷酸测序
甲基化是指DNA中的嘌呤和胸腺嘧啶在碳5位置上的甲基化修饰。
在DNA序列中,甲基化可以影响基因的表达和基因组稳定性。
焦磷酸测序(Bisulfite Sequencing)是一种常用的用于检测DNA甲基化的方法。
该方法利用二硫酸盐处理DNA样本,将未甲基化的胞嘧啶转化为尿嘧啶,但甲基化的胞嘧啶不会被转化。
然后再通过测序分析 DNA样本中的胞嘧啶和尿嘧啶的比例,从而确定DNA序列中的甲基化位点。
甲基化和焦磷酸测序在研究中常用于探究基因组中的表观遗传修饰和调控以及基因的表达和调控机制。
甲基化测序结果分析

甲基化测序结果分析甲基化测序(MethylationSequencing)技术在研究基因表达状态及基因功能上具有重要的意义。
现今,在生物医学以及其他基础科学领域中其应用正越来越广泛。
由此需要研究者对甲基化的测序结果分析进行深入的研究。
本文以近几年主流的甲基化测序结果分析方法为基础,结合开源甲基化测序结果分析软件,详细介绍甲基化测序结果分析的主要步骤。
首先,分析准备:甲基化测序数据应当经过完整的质控,以确保质量较高的序列,比如,需要去除宿主背景及不需要的序列,以保证高品质的数据。
在此阶段,研究者需要根据自己的实验策略,筛选出符合条件的实验样品,并对样品做相应的质控处理。
其次,特征表示:特征表示是甲基化测序结果分析中最重要的一步。
研究者需要使用一些软件将原始数据进行编码或标记,以便更好的显示出这些数据的复杂性和差异性。
常用的特征表示方法有公共伪计数(CPC),基因表达聚类(GECluster),甲基化数据库(MethylDB)等。
接下来,数据分析:通过数据分析,可以根据生物学数据探索表型数据的分布情况,从而发现与状态变化相关的特定基因。
此外,可以使用统计分析,如假设检验、相关分析等,来检验特定基因与表型变化之间的关系。
最后,结果可视化:可视化是进行结果展示以及验证分析结果的重要方法。
常用的可视化技术包括热图(heatmap)、统计曲线(statistical curves)、直方图(histograms)等,它们可以帮助研究者更清楚地了解甲基化测序结果,并且更好地发现重要的特征。
以上便是甲基化测序结果分析的主要步骤,本文综合介绍了甲基化测序的结果分析过程,希望能对研究者提供一些帮助。
未来,人们将会有更多的进步,更好地发掘甲基化测序结果的运用价值,充分挖掘基因表达的信息及功能,从而为未来的研究提供实质性的支持。
人类全基因组甲基化测序

建库测序 测序策略:Illumina HiSeq 平台;PE125 测序深度:≥ 30X 数据量:90 Gb data 项目周期 90 天(样本数小于 50 个)
案例解析
案例一 北京大学和诺禾致源联合构建人脑全基因组DNA甲基化图谱
DNA甲基化是最早发现的修饰方式之一,能引起染色质结构、DNA构象与稳定性、DNA与蛋白质相互作用方式的 改变,从而影响基因表达。北京大学和诺禾致源合作构建了人类脑组织单碱基分辨率的全基因组DNA甲基化图谱,揭 示了5-甲基胞嘧啶及其去甲基化过程的中间产物在调控可变剪切和基因表达中的作用。
全基因组 DNA 甲基化测序
全基因组 Bisulfite 测序(Whole Genome Bisulfite Sequencing,WGBS)是将重亚硫酸盐处理方法和 Illumina HiSeq 高通量测序平台相结合,进行全基因组范围内的精确甲基化研究。WGBS 可以达到单碱基分 辨率,精确分析每一个胞嘧啶的甲基化状态,从而构建精细的全基因组 DNA 甲基化图谱。
图1 人脑基因外显子-内含子边界区域甲基化修饰水平
参考文献
Wen L, Li X, Yan L, et al. Whole-genome analysis of 5-hydroxymethylcytosine and 5-methylcytosine at base resolution in the human brain [J]. Genome biology, 2014, 15(3): R49.
组比对 甲基化位点检测
甲基化水平分析 甲基化密相关基因功能注释 DMR相关基因富集分析
技术参数
样本要求 样品类型:基因组 DNA 样品总量:≥ 6 μg 样品浓度:≥ 100 ng/ul
DNA甲基化检测技术.

Part 2 ofour afternoondoublefeature is the instrumentationaspect ofthis ampliconmeltingapplication.
We currentlyhave 2 instruments designedspecificallyfor Hi-Res melting, …the LightScannerand the HR-1.
用HRM方法检测MGMT启动子区域内的甲基化状态,可检测 低达0.1%的甲基化程度;并且可以根据已知甲基化程度的标准 曲线对未知样品的甲基化百分比进行测定。
MS-HRM检测技术原理
采用重亚硫酸盐处理DNA模板后,在非CpG岛位置设计 一对针对经亚硫酸氢钠处理后DNA链的引物,这对引物中 间包含有意义的甲基化CpG岛,一旦这些CpG岛发生甲基化, 胞嘧啶不发生变化,而未甲基化的胞嘧啶转变成胸腺嘧啶, 样品中的GC含量发生了变化,最终被转化成了熔解曲线Tm 值之间的差异。CpG位点在小的扩增片段内的相对位置也 会对熔解峰的形状产生影响,因此,根据Tm值和熔解峰的 形状可以检测出样本的甲基化位点和甲基化程度.
Melttime is approximately5 minutesper plate whichgivesmeltingcurveresultsonallofthe samples inthe plate. Just to be clear, 5 minutesof meltinggivesyou96 or 384 results, whichis approximately4 seconds per sampleina 96 wellplate, and 1 second per sample ina 384 wellplate.
RRBS甲基化测序步骤

RRBS甲基化测序步骤概述RRBS(Reduced Representation Bisulfite Sequencing)是一种准确且高性价比的DNA甲基化研究方法,在大规模临床样本的研究中有着广泛的应用。
RRBS通过限制性内切酶对基因组进行酶切,富集启动子及CpG岛区域,再进行Bisulfite转化,用较小的数据量得到包含最多CpG位点甲基化信息的单碱基精度的甲基化图谱。
测序步骤·基因组DNA提取:根据不同的样品类型采用不同的提取方案,获取高质量的基因组DNA。
· DNA质量检测:Qubit检测DNA样品浓度,琼脂糖凝胶电泳检测DNA样品完整性。
· MspⅠ酶切:通过酶切富集CpG片段。
·末端修复,加A,甲基化接头连接:在DNA分子两端引入接头序列及index序列。
·选择CpG富集的DNA片段· Bisulfite转化:通过Bisulfite作用将非甲基化碱基C转化成U。
· PCR富集:PCR扩增富集文库片段。
·文库质检:Agilent 2100 Bioanalyzer检测文库大小分布,Qubit 3.0或荧光定量PCR测定文库浓度。
·上机测序:根据数据量要求将文库pooling上机测序。
·数据分析:下机数据由专业生物信息分析团队进行数据分析,提供全面数据分析报告。
样品要求·样品类型:细胞、新鲜组织或DNA样品。
·样品量:细胞样品请提供至少5×106个细胞,组织样品请提供至少2-3 g的组织块或切片,DNA样品请提供6 μg以上的总DNA。
·样品质量:DNA无明显降解,无蛋白污染,OD260/280值≥1.5,OD260/230值≥1.0,浓度≥50 ng/μL,·样品保存:细胞样品:收集细胞至RNase Free 1.5 mL EP管,弃去培养基,用PBS洗一次,弃去PBS,把细胞沉淀保存在-80℃。
全基因组甲基化测序(WGBS)

全基因组甲基化测序(WGBS)
技术简介:
全基因组重亚硫酸盐测序(whole genome bisulfite Sequencing)是基于重亚硫酸盐的甲基化分析方法,首先通过重亚硫酸盐对样本DNA进行处理,将未甲基化的C碱基转化为U碱基,而甲基化的C碱基则不会改变,进行PCR扩增后U碱基会变成T,与原本甲基化的C碱基区分开,再结合高通量测序技术,可绘制单碱基分辨率的全基因组DNA甲基化图谱。
应用领域:
•基因表达调控
•发育表观组学
•细胞分化、组织发育
技术优势:
•可精确分析每一个C碱基的甲基化状态
•可在全基因组水平上最大限度的获取完整的甲基化信息,精确绘制全基因组甲基化图谱•适用于所有具有参考基因组的物种
•性价比高,相对于传统BSP或MSP方法,费用少
实验流程
A.建库测序流程
.
B.数据分析流程
;.。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
DNA甲基化检测实验
一、重亚硫酸盐的测序法实验流程(BSP)
(Bisulfite Genomic Sequence)
原理:结合重亚硫酸盐的测序法是一种灵敏的能直接检测分析基因组DNA甲基化模式的方法。
重亚硫酸盐处理后,用针对改变后的DNA序列设计特异性引物并进行聚合酶链式反应(PCR)。
PCR产物中原先非甲基化的胞嘧啶位点被胸腺嘧啶所替代,而甲基化的胞嘧啶位点保持不变。
PCR产物克隆后进行测序。
通过这个方法能得到特定位点在各个基因组DNA分子中的甲基化状态。
该方法特点是:
•特异性高,它能够提供特异性很高的分析结果,这是所有其他研究甲基化的分析方法所不能比拟的;
•灵敏度高,可以用于分析少于100个细胞的检测样品。
用微量的基因组DNA进行分析就能得到各个DNA分子精确的甲基化位点分布图。
重亚硫酸盐测序法技术实验流程
A. DNA制备
用DNA抽提试剂盒(Promega, cat. no. A1125)抽提组织,细胞培养物,石蜡包埋组织切片样品中的基因组DNA。
B.重亚硫酸盐处理
C. DNA纯化
用Wizard DNA clean-up kit (Promega, cat. no. A7280)纯化重亚硫酸盐处理后的DNA样品。
D. PCR扩增
E. PCR产物琼脂糖电泳后回收纯化
F. PCR产物连接到pMD19-T (Takara) 载体中克隆及测序。
G.用分析软件对各样本测序结果进行甲基化程度分析
二、甲基化特异性的PCR实验流程
(methylation-specific PCR, MSP)
原理:甲基化特异性的PCR是一种灵敏度高且操作相对简单的甲基化研究方法。
重亚硫酸盐
处理
DNA后,基因组DNA发生的由甲基化状态决定的序列改变。
随后进行引物特异性的PCR。
该方法引物设计是关键。
MSP中设计两对引物,即一对结合处理后的甲基化DNA链(引物对M),另一对结合处理后的非甲基化DNA链(引物对U)。
检测MSP扩增产物,如果用引物M能扩增出片段,则说明检测位点存在甲基化;若用引物U扩增出片段,则说明被检测的位点不存在甲基化(图3)。
该方法优点是:
1、检测灵敏度高,样品消耗少,仅需1μg的基因组DNA,可用于石蜡包埋样本;
2、快速、简单,省时;
3、如果结合Real-time定量PCR技术(Taqman 探针)则能对样品中检测位点的甲基化水平进行定量检测(Methylight)。
MSP技术实验流程
A.DNA抽提
用DNA抽提试剂盒(Promega, cat. no. A1125)抽提组织,细胞培养物,石蜡包埋组织切片样品中的基因组DNA。
B. 重亚硫酸盐处理
C.DNA纯化
用Wizard DNA clean-up kit (Promega, cat. no. A7280)纯化重亚硫酸盐处理后的DNA样品。
D.DNA脱磺基反应
E.甲基化特异性的PCR反应
针对改变后的DNA序列设计两对引物(M,U),进行PCR反应。
为了避免非特异性扩增用TaKaRa 的hotstart酶。
随后对PCR产物的凝胶电泳图进行分析。
实例:
检测肝癌细胞株Bel7405,Bel7404中SFRP1基因启动子甲基化。
重亚硫酸盐处理后,针对改变的DAN序列设计两对引物
M: (forward: AGTTAGTGTCGCGCGTTC; reversal: CCGATACCCATACCGACTC) 299 bp
U:(forward:GGAGTTGGGGTGTATTTAGTTTG; reversal: CCAATACCCATACCAACTCTACA) 247 bp PCR扩增结果图
三、高分辨率熔解曲线(HRM)法
高分辨率熔解曲线分析技术(High Resolution Melting ),简称HRM,是近年来兴起的一种检测基因突变、进行基因分型而及甲基化检测的新方法。
基本原理是基于核酸分子由于片段长短、GC含量、GC分布及单个碱基差异等物理性质不同而造成DNA分子在加热变性时都会有不同的熔解曲线的形状和位置,并配合运用高浓度的饱和荧光染料,可以迅速的检测出核酸片段中GC含量和单碱基的突变。
近年来,公司引进了RocheLightCycler® 480 II 和ABI VIIA7 全自动荧光定量PCR系统,为客户提供专业化、个性化的甲基化HRM检测服务。
操作流程:
﹡待测基因序列片段或位点甲基化引物设计(300bp以内)。
﹡利用甲基化转移酶修饰的DNA和非甲基化修饰的DNA制备标准品(0%,1%,5%,10%,20%,50%,100%梯度HRM标准曲线)
﹡亚硫酸氢盐处理样品DNA
﹡上机检测
﹡数据处理,形成报告(包括实验过程、试剂耗材、荧光PCR仪原始文件、测序文件等)。
实验实例:
对2个病例的某基因20个CG位点进行检测,分析出不同的甲基化程度。
四、焦磷酸测序法
焦磷酸测序(Pyrosequencing)技术是由4种酶(DNA聚合酶(DNA polymerase)、ATP硫酸化酶(ATP sulfurytase)、荧光素酶(luciferase)和三磷酸腺苷双磷酸酶(Apyrase))催化的同一反应体系中的酶级联化学发光反应。
每一轮测序反应体系中只加入一种脱氧核苷酸三磷酸(dNTP)。
如果该dNTP与模板配对,则会在DNA聚合酶的作用下,添加到测序引物的3'末端,同时释放出一个分子的焦磷酸(PPi)。
掺入的dNTP和释放的PPi是等量的。
CpG 甲基化焦磷酸测序检测法基于引物延伸的Pyrosequencing技术,通过将链合成过程中释放出的焦磷酸(PPi)转化成光信号来监测链的合成过程。
通过检测CpG对应位点上C/T渗入的比例对目标位点的甲基化程度进行定量分析方法。
操作流程:
﹡DNA亚硫酸盐处理
﹡用焦磷酸测序专门引物设计软件设计甲基化引物﹡进行PCR扩增预实验及正式实验
﹡PCR产物上焦磷酸测序仪检测
实验实例:
各种方案比较
检测方法 检测基因位置 定量情况 位点信息
亚硫酸氢盐修饰后测序法(BSP ) ≤350bp 精确,但要大量克隆测序。
精确了解每一位点甲基化程度 甲基化特异性的PCR 法(MSP ) 引物设计位置 只能定性、定量不精确
待测位点甲基化程度不能了解 结合重亚硫酸盐的限制性内切酶法(COBRA ) 单CG 位点 只能定性、定量不精确
待测位点甲基化程度大致了解 高分辨率熔解曲线法(HRM ) ≤200bp 精确检测待测片段甲基化程度
待测每一位点甲基化程度不能了解 焦磷酸测序法(Pyrosequencing ) ≤60bp 精确检测待测片段甲基化程度
精确了解每一位点甲基化程度
服务说明
①样品要求:
﹡血液样品:样品为EDTA 抗凝或枸椽酸钠抗凝,样品量大于0.5 ml ,样品采集后于-20℃或-80℃保存,低温(≤-4℃)运输。
﹡组织样品:样品可以为新鲜组织(最好-80℃保存)或石蜡包埋的组织,也可以是95%乙醇中固定的组织,组织量大于100 mg 。
﹡DNA 样品:纯度OD260/280 比值为1.8-1.9,浓度≥50ng/ul,体积≥50ul。
②提供详细的基因信息
BSP 方法要求待测序列≤300bp,HRM 待测序列≤200bp,HRM 待测序列≤200bp,焦磷酸测序待测序列≤60bp,且上下游保留200bp 用于引物设计;MS 方法要求提供待测序列的CG 侯选位点。
我们会根据客户的基因序列信息选择不同的检测方法,并在方案实验过程中随时与客户沟通。
③提供结果
实验报告(实验步骤,包括BSP 法的5个克隆统计结果等)、PCR 电泳照片、测序彩色波形图、HRM 标准曲线图、焦磷酸测序原始文件、基因芯片的原始文件等。
关键词:DNA 甲基化检 测实验
(本资料素材和资料部分来自网络,仅供参考。
请预览后才下载,期待您的好评与关注!)。
