pMD18-T
pMD18-T Simple说明书

TaKaRa Code:D103A pMD®18-T Simple Vector目录内 容 Page●制品说明 1●制品内容 1●保 存 1●纯 度 1●用 途 1●pMD®18-T Simple Vector的结构 1 ●实验操作 2■Control DNA片段的克隆实验2■一般DNA片段的克隆实验2●相关说明 3 ●使用注意 4 ●Q&A4●制品说明pMD ®18-T Simple Vector是一种高效克隆PCR产物(TA Cloning)的专用载体。
本载体由pUC18载体改建而成,它消除了pUC18载体上的多克隆酶切位点,再在pUC18载体的多克隆酶切位点处导入了Eco R V酶切位点,使用Eco R V进行酶切反应后,再在两侧的3'端添加“T”而成。
因大部分耐热性DNA聚合酶进行PCR反应时都有在PCR 产物的3'末端添加一个“A”的特性,所以使用本制品可以大大提高PCR产物的连接、克隆效率。
本载体尽管消除了LacZ 基因上的多克隆酶切位点,但不影响β-半乳糖苷酶的正常表达,因此,PCR 产物克隆后仍可以利用α-互补性进行蓝白菌落的筛选,挑选阳性克隆。
由于本载体上消除了多克隆酶切位点,克隆后的PCR 产物将无法使用载体上的限制酶切下,需要在PCR 扩增引物上导入合适的酶切位点。
此时如果使用PCR 扩增引物导入的酶切位点进行DNA 酶切时,酶切反应将不会受到T 载体上其它多克隆酶切位点上的限制酶影响,可以大大提高酶切效率,增加亚克隆成功率。
由于本载体以pUC18载体为基础构建而成,所以它具有同pUC18载体相同的功能。
此外,本制品中的高效连接液Solution I 可以在短时间内(约30分钟)完成连接反应,其连接液可以直接用于细菌转化,大大方便了实验操作。
本制品中的Control Insert(500 bp)还可以用于Control 反应。
●制品内容pMD ®18-T Simple Vector(50 ng/μl) 20 μl×1支 Control Insert(50 ng/μl) 10 μl×1支 Solution I*75 μl×2支** 使用时请于冰中融解。
Motorola 3.5 kHz 产品说明书

RVN4126 3.59100-386-9100-386/T DEVICERVN41772-CD2-3.5MCS/MTSRVN41821-CD2-3.5XTS3000/SABER PORTABLE YES RKN4046KHVN9085 3.51-20 R NO HLN9359 PROG. STAND RVN4057 3.532 X 8 CODEPLUG NO3080385B23 & 5880385B30 MDVN4965 3.59100-WS/T CONFIG KITRVN4053 3.5ASTRO DIGITAL INTERFACE NO3080385B23RVN41842-CD RKN4046A (Portable) 2-3.5ASTRO PORTABLE /MOBILE YES3080369B73 or0180300B10 (Mobile) RVN41831-CD3080369B732-3.5ASTRO SPECTRA MOBILE YES(Low / Mid Power)0180300B10 (High Power) RVN4185CD ASTRO SPECTRA PLUS MOBILE NO MANY OPTIONS; SEESERVICE BRIEF#SB-MO-0101RVN4186CD ASTRO SPECTRA PLUS MANY OPTIONS;MOBILE/PORTABLE COMB SEE SERVICE BRIEF#SB-MO-0101RVN4154 3.5ASTROTAC 3000 COMPAR.3080385B23RVN5003 3.5ASTROTAC COMPARATORS NO3080399E31 Adpt.5880385B34RVN4083 3.5BSC II NO FKN5836ARVN4171 3.5C200RVN4029 3.5CENTRACOM SERIES II NO VARIOUS-SEE MANUAL6881121E49RVN4112 3.5COMMAND PLUS NORVN4149 3.5COMTEGRA YES3082056X02HVN6053CD CT250, 450, 450LS YES AAPMKN4004RVN4079 3.5DESKTRAC CONVENTIONAL YES3080070N01RVN4093 3.5DESKTRAC TRUNKED YES3080070N01RVN4091 3.5DGT 9000 DESKSET YES0180358A22RVN4114 3.5GLOBAL POSITIONING SYS.NO RKN4021AHVN8177 3.5GM/GR300/GR500/GR400M10/M120/130YES3080070N01RVN4159 3.5GP60 SERIES YES PMLN4074AHVN9128 3.5GP300 & GP350RVN4152 3.5GP350 AVSRVN4150 3.5GTX YES HKN9857 (Portable)3080070N01(Mobile) HVN9025CD HT CDM/MTX/EX SERIES YES AARKN4083/AARKN4081RiblessAARKN4075RIBLESS NON-USA RKN4074RVN4098H 3.5HT1000/JT1000-VISAR YES3080371E46(VISAR CONV)RVN4151 3.5HT1000 AVSRVN4098 3.5HT1000/ VISAR CONV’L.YES RKN4035B (HT1000) HVN9084 3.5i750YES HLN-9102ARVN4156 3.5LCS/LTS 2000YES HKN9857(Portable)3080070N01(Mobile) RVN4087 3.5LORAN C LOC. RECV’R.NO RKN4021ARVN4135 3.5M100/M200,M110,M400,R100 includesHVN9173,9177,9646,9774YES3080070N01RVN4023 3.5MARATRAC YES3080070N01RVN4019 3.5MAXTRAC CONVENTIONAL YES3080070N01RVN4139 3.5MAXTRAC LS YES3080070N01RVN4043 3.5MAXTRAC TRK DUPLEX YES3080070N01RVN4178CD MC SERIES, MC2000/2500DDN6124AW/DB25 CONNECTORDDN6367AW/DB9 CONNECTOR RVN41751-CD Rib to MIC connector 1-3.5MCS2000 RKN4062BRVN41131-3.5MCS2000RVN4011 3.5MCX1000YES3000056M01RVN4063 3.5MCX1000 MARINE YES3000056M01RVN4117 3.5MDC/RDLAP DEVICESRVN4105 3.5MOBILE PROG. TOOLRVN4119 3.5MOBITEX DEVICESRVN4128 3.5MPT1327-1200 SERIES YES SEE MANUALRVN4025 3.5MSF5000/PURC/ANALOG YES0180355A30RVN4077 3.5MSF5000/10000FLD YES0180355A30RVN4017K 3.5MT 1000YES RTK4205CRVN4148 3.5MTR 2000YES3082056X02RVN4140 3.5MTRI 2000NORVN41761-CD MTS2000, MT2000*, MTX8000, MTX90001-3.5*programmed by DOS which is included in the RVN4176RVN4131 3.5MTVA CODE PLUG FIXRVN4142 3.5MTVA DOCTOR YES3080070N01RVN4131 3.5MTVA3.EXERVN4013 3.5MTX800 & MTX800S YES RTK4205CRVN4097 1-CD MTX8000/MTX9000,MTS2000,MT2000*,* programmed by DOS which is included in the RVN4176HVN9067CD MTX850/MTX8250MTX950,MTX925RVN4138 3.5MTX-LS YES RKN4035DRVN4035 3.5MX 1000YES RTK4203CRVN4073 3.5MX 800YES RKN4006BHVN9395 P100, P200 LB, P50+, P210, P500, PR3000RVN4134 3.5P100 (HVN9175)P200 LB (HVN9794)P50+ (HVN9395)P210 (HVN9763)P500 (HVN9941)PR3000 (HVN9586)YES RTK4205HVN9852 3.5P110YES HKN9755A/REX1143 HVN9262 3.5P200 UHF/VHF YES RTK4205RVN4129 3.5PDT220YVN4051 3.5PORTABLE REPEATER Portable rptr.P1820/P1821AXRVN4061C 3.5PP 1000/500NO3080385B23 & 5880385B30 RVN5002 3.5QUANTAR/QUANTRO NO3O80369E31RVN4135 3.5R100 (HVN9177)M100/M200/M110/M400YES0180358A52RVN4146 3.5RPM500/660RVN4002 3.5SABER YES RTK4203CRVN4131 3.5SETTLET.EXEHVN9007 3.5SM50 & SM120YESRVN4039 3.5SMART STATUS YES FKN5825AHVN9054 3.5SOFTWARE R03.2 P1225YES3080070N01HVN9001 3.5SOFTWARE R05.00.00 1225LS YES HLN9359AHVN9012 3.5SP50RVN4001N 3.5SPECTRA YES3080369B73 (STANDARD)0180300B10 (HIGH POWER) RVN4099 3.5SPECTRA RAILROAD YES3080369B73RVN4110 3.5STATION ACCESS MODULE NO3080369E31RVN4089A 3.5STX TRANSIT YES0180357A54RVN4051 3.5SYSTEMS SABER YES RTK4203BRVN4075 3.5T5600/T5620 SERIES NO3080385B23HVN9060CD TC3000, TS3000, TR3000RVN4123 3.5VISAR PRIVACY PLUS YES3080371E46FVN4333 3.5VRM 100 TOOLBOX FKN4486A CABLE &ADAPTORRVN4133 3.5VRM 500/600/650/850NORVN4181CD XTS 2500/5000 PORTABLES RKN4105A/RKN4106A RVN41002- 3.5XTS3000 ASTRO PORTABLE/MOBILERVN4170 3.5XTS3500YES RKN4035DRIB SET UPRLN4008E RADIO INTERFACE BOX (RIB)0180357A57RIB AC POWER PACK 120V0180358A56RIB AC POWER PACK 220V3080369B71IBM TO RIB CABLE (25 PIN) (USE WITH XT & PS2)3080369B72IBM TO RIB CABLE (9 PIN)RLN443825 PIN (F) TO 9 PIN (M) ADAPTOR (USE W/3080369B72 FOR AT APPLICATION) 5880385B308 PIN MODULAR TO 25 PIN ”D” ADAPTOR (FOR T5600 ONLY)0180359A29DUPLEX ADAPTOR (MOSTAR/TRAXAR TRNK’D ONLY)Item Disk Radio RIB Cable Number Size Product Required Number Item Disk Radio RIB Cable Number Size Product Required NumberUtilizing your personal computer, Radio Service Software (RSS)/Customer Programming Software (CPS)/CustomerConfiguration Software (CCS) enables you to add or reprogram features/parameters as your requirements change. RSS/CPS/CCS is compatible with IBM XT, AT, PS/2 models 30, 50, 60 and 80.Requires 640K RAM. DOS 3.1 or later. Consult the RSS users guide for the computer configuration and DOS requirements. (ForHT1000, MT/MTS2000, MTX838/8000/9000, Visar and some newer products —IBM model 386, 4 MEG RAM and DOS 5.0 or higher are recommended.) A Radio Interface Box (RIB) may be required as well as the appropriate cables. The RIB and cables must be ordered separately.Licensing:A license is required before a software (RVN) order is placed. The software license is site specific (customer number and ultimate destination tag). All sites/locations must purchase their own software.Be sure to place subsequent orders using the original customer number and ship-to-tag or other licensed sites; ordering software without a licensed customer number and ultimate tag may result in unnecessary delays. To obtain a no charge license agreement kit, order RPX4719. To place an order in the U.S. call 1-800-422-4210. Outside the U.S., FAX 847-576-3023.Subscription Program:The purchase of Radio ServiceSoftware/Customer Programming/Customer ConfigurationSoftware (RVN & HVN kits) entitles the buyer/subscriber to three years of free upgrades. At the end of these three years, the sub-scriber must purchase the same Radio Service Software kit to receive an additional three years of free upgrades. If the sub-scriber does not elect to purchase the same Radio Service Software kit, no upgrades will be sent. Annually a subscription status report is mailed to inform subscribers of the RSS/CPS/CCS items on our database and their expiration dates.Notes:1)A subscription service is offered on “RVN”-Radio Service Software/Customer Programming/Customer Configuration Software kits only.2)“RVN” software must only be procured through Radio Products and Services Division (RPSD). Software not procured through the RPSD will not be recorded on the subscription database; upgrades will not be mailed.3)Upgrades are mailed to the original buyer (customer number & ultimate tag).4)SP software is available through the radio product groups.The Motorola General Radio Service Software Agreement is now available on Motorola Online. If you need assistance please feel free to submit a “Contact Us” or call 800-422-4210.SMART RIB SET UPRLN1015D SMART RIB0180302E27 AC POWER PACK 120V 2580373E86 AC POWER PACK 220V3080390B49SMARTRIB CABLE (9 PIN (F) TO 9 PIN (M) (USE WITH AT)3080390B48SMARTRIB CABLE (25 PIN (F) TO 9 PIN (M) (USE WITH XT)RLN4488ASMART RIB BATTERY PACKWIRELESS DATA GROUP PRODUTS SOFTWARERVN4126 3.59100-386/9100T DEVICES MDVN4965 3.59100-WS/T CONFIG’TN RVN41173.5MDC/RDLAP DEVICESPAGING PRODUCTS MANUALS6881011B54 3.5ADVISOR6881029B90 3.5ADVISOR ELITE 6881023B20 3.5ADVISOR GOLD 6881020B35 3.5ADVISOR PRO FLX 6881032B30 3.5BR8506881032B30 3.5LS3506881032B30 3.5LS5506881032B30 3.5LS7506881033B10 3.5LS9506881035B20 3.5MINITOR III8262947A15 3.5PAGEWRITER 20008262947A15 3.5PAGEWRITER 2000X 6881028B10 3.5TALKABOUT T3406881029B35 3.5TIMEPORT P7308262947A15 3.5TIMEPORT P930NLN3548BUNIVERSAL INTERFACE KITItem Disk Radio NumberSize Product。
pMD18-T载体说明书

TaKaRa Code:D101ApMD®18-T Vector宝生物工程(大连)有限公司目录内 容 Page●制品说明 1●制品内容 1●保 存 1●纯 度 1●用 途 1●pMD®18-T Vector的结构 1 ●实验操作 2■Control DNA片段的克隆实验2■一般DNA片段的克隆实验2●相关说明 3 ●使用注意 4 ●Q&A4●制品说明pMD®18-T Vector是一种高效克隆PCR产物(TA Cloning)的专用载体。
本载体由pUC18载体改建而成,在pUC18载体的多克隆位点处的Xba I和Sal I识别位点之间插入了Eco R V识别位点,用Eco R V进行酶切反应后,再在两侧的3'端添加“T”而成。
因大部分耐热性DNA聚合酶进行PCR反应时都有在PCR 产物的3'末端添加一个“A”的特性,所以使用本制品可以大大提高PCR产物的连接、克隆效率。
由于本载体以pUC18载体为基础构建而成,所以它具有同pUC18载体相同的功能。
此外,本制品中的高效连接液Solution I可以在短时间内(约30分钟)完成连接反应,其连接液可以直接用于细菌转化,大大方便了实验操作。
本制品中的Control Insert(500 bp)还可以用于Control反应。
●制品内容pMD®18-T Vector(50 ng/μl) 20 μl×1支Control Insert(50 ng/μl) 10 μl×1支Solution I* 75 μl×2支* 使用时请于冰中融解。
●保存:-20℃●纯度■ Control Insert经克隆后的白色菌落中,有90%以上含有Insert DNA片段。
●用途■ 进行TA克隆,克隆PCR产物。
■ 对克隆后的PCR产物使用Bca BEST TM Sequencing Primers、M13 Primers进行DNA测序。
pMD19T

BcaBESTTM Sequencing Primer RV-M
GAGCGGATAACAATTTCACACAGG lacZ
Hind III
Hinc II Sse8387 I Acc I
Sph I Pst I Sal I EcoR V
Xba I
BamH I
Xma I Sma I Kpn I
Sac I
EcoR I
PCR产物克隆系列载体
Amp r
pMD18-T Vector pMD19-T Vector
D101A D102A
1 µg
500 元
1 µg
500 元
pMD18-T Vector、pMD19-T Vector是一种高效克隆PCR产物 (TA Cloning) 的专用载体。这两种载体分别由pUC18、pUC19 载体改建而成,在pUC18、pUC19载体的多克隆位点处的Xba I和Sal I识别位点之间插入了EcoR V识别位点,用EcoR V 进行酶切反应后,再在两侧的3'端添加 "T" 而成。因大部分耐热性DNA聚合酶进行PCR反应时都有在PCR产物的3'末端添加一个 "A" 的特性,所以使用这两种制品可以大大提高PCR产物的连接、克隆效率。 由于这两种载体是分别以pUC18、pUC19载体为基础构建而成,所以它具有同pUC18、pUC19载体相同的功能。此外,这 两种制品中的高效连接液Ligation Mix可以在极短时间内 (约5分钟) 完成连接反应,且此连接液可以直接用于细菌转化,大 大方便了实验操作。制品中的Control Insert (500 bp) 还可以用于Control反应。 pMD19-T Vector与pMD18-T Vector相比,pMD19-T Vector的β-半乳糖苷酶的表达活性更高,菌落显示蓝色的时间缩短,菌 落显示的蓝色更深。因此,克隆后更容易进行克隆体的蓝白筛选。
【精选】基于重叠延伸PCR法的定点突变技术 doc资料

基于重叠延伸PCR法的定点突变技术E-mail:daican@live △通讯作者:卢光琇, :mutant location Primers Sequences for primers (listed 5'to 3'-189~-193bp (relative to ATGF ggggtaccCTCGCTGTCGCACTCAGGCTRm CAGTCAACCGCCACAAAATT Fm AATTTTGTGGCGGTTGACTG RcggctagcAACTGGGTAGGGACGAGGAG基于重叠延伸PCR 法的定点突变技术*戴灿苗聪秀卢光琇△(中南大学生殖与干细胞工程研究所,人类干细胞工程研究中心湖南长沙410078摘要:目的:建立一种高效而经济的定点突变方法。
方法:采用重叠延伸PCR 定点突变技术,引物设计时引入目的突变,以前两次PCR 产物为模板,进行第三次PCR ,即可获得突变后的目的DNA 片段。
将此片段连入pMD TM 18-T 载体后测序验证突变结果。
结果:DNA 测序表明,待突变位点已由ATTGG 突变为ATTTT 。
结论:成功实现了目的位点的定点突变,重叠延伸PCR 法是一种高效且经济的定点突变方法。
关键词:重叠延伸PCR ;定点突变中图分类号:Q75,Q78,R392文献标识码:A 文章编号:1673-6273(202103-411-02Site-directed Mutagenesis Based on Overlap Extension PCR *DAI Can,MIAO Cong-xiu,LU Guang-xiu △(Institute of Reproductive and Stem Cell Engineering,Central SouthUniversity,National Engineering and Research Center of HumanStem Cell,Changsha,410078,ChinaABSTRACT Objective:To establish a fast,saving method for site-directed mutagenesis.Methods:Overlap extension PCR was used.Briefly,target mutation was introduced into primers,and the two previous PCR products were used as template for the third PCR.The final PCR segment with target mutant was then cloned into pMD?18-T vector for sequencing.Results:DNA sequencing showed that the target site ATTGG had been changed into ATTTT.Conclusion:Site-directed mutagenesis was successfully implemented based on the overlap extension PCR which is a fast and saving method.Key words:Overlap extension PCR;Site-directed mutagenesis Chinese Library Classification:Q75,78,R392Document code:Article ID:1673-6273(202103-411-02前言定点突变(Site-directed mutagenesis,SDM 是指通过聚合酶链式反应(PCR 等方法在目的DNA 片段的特定位点中引入碱基改变,如插入、缺失、点突变等。
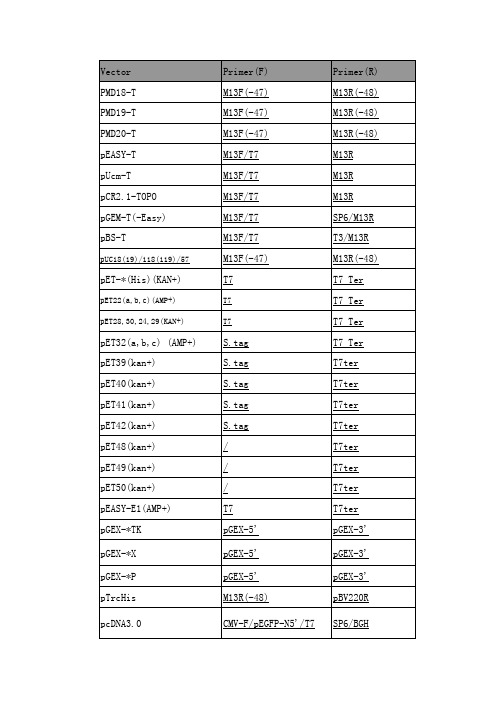
Vector Primer(F) Primer(R) PMD18-T M13F(-47) M13R(
CMV-F/pEGFP-N5'/T7 SP6/BGH
Vector pcDNA3.1 pcDNA4 pcDNA6 pcDNAII
pBluescript SK/KS(+/-) pGCSilencer-U6/Neo/DsRed
PCANTB5E PCR2.1-TOPO pDonar pDrive pENTR/D-TOPO(KAN+) pGBKT7(Kana+) PinpointTM pGL3 pPROEXHTA pSilencer3.1-H1 pSUPER pCMV-Sport pCMV-Tag(KAN+) pFlag-cmv pEG/Y/CFP-N(KAN+) pEG/Y/CFP-C(KAN+) pIRES2-EGFP(KAN+) pEG/Y/CFP-1(KAN+) pEG/Y/CFP(5'MCS) pEG/Y/CFP(3'MCS) pAcGFP1-N(KAN+)
vectorprimerfprimerrpmd18tm13f47m13r48载体引物f引物rpmd18tm13f4748m13r论文总结英语资料ppt文档免费阅读免费分享如需请下载
Vector PMD18-T PMD19-T PMD20-T pEASY-T pUcm-T pCR2.1-TOPO pGEM-T(-Easy) pBS-T
pEGFP-N-5'
pDsRED1-C1
pDsRED-ex-C1-F
pDsRED2-C1(KAN+)
pDsRED-ex-C1-F
PCR3.1
T7
pDonar
M13F
PEGFP-N1
PEGFP-N5
PEGFP-C1
pMD18-T载体说明

Hale Waihona Puke ●制品说明pMD18-T Vector 是一种高效克隆 PCR 产物(TA Cloning)的专用载体。本载体由 pUC18 载体改建 而成,在 pUC18 载体的多克隆位点处的 Xba I 和 Sal I 识别位点之间插入了 EcoR V 识别位点,用 EcoR V 进行酶切反应后,再在两侧的 3'端添加“T”而成。因大部分耐热性 DNA 聚合酶进行 PCR 反应时都 有在 PCR 产物的 3'末端添加一个“A”的特性,所以使用本制品可以大大提高 PCR 产物的连接、克 隆效率。 由于本载体以 pUC18 载体为基础构建而成,所以它具有同 pUC18 载体相同的功能。此外,本制品中的 高效连接液 Ligation Mix 可以在极短时间内(约 5 分钟)完成连接反应,且此连接液可以直接用于细菌 转化,大大方便了实验操作。本制品中的 Control Insert(500 bp)还可以用于 Control 反应。
419 347~370 478~500 146~469 852~1466 1626~2486
●实验操作
■ Control DNA片段的克隆实验
A)操作方法
1)在微量离心管中配制下列 DNA 溶液,全量为 5 µl。
pMD18-T Vector*1
1 µl
Control Insert*2
1 µl
dH2O
3 µl
2)加入 5 µl(等量)的 Ligation Mix。 3)16℃反应 30 分钟。
注) ① 室温(25℃)也能正常进行连接反应,但反应效率稍微降低。 ② 5 分钟也能正常进行连接反应,但反应效率稍微降低。
4)全量(10 µl)加入至 100 µl JM109 感受态细胞中,冰中放置 30 分钟。 5)42℃加热 45 秒钟后,再在冰中放置 1 分钟。 6)加入 890 µl SOC 培养基,37℃振荡培养 60 分钟。 7)在含有 X-Gal、IPTG、Amp 的 L-琼脂平板培养基上培养,形成单菌落。计数白色、蓝色菌落。 8)挑选白色菌落,使用 PCR 法确认载体中插入片段的长度大小。
GFP实验报告

绿色荧光蛋白的克隆摘要:目的:研究绿色荧光蛋白基因的基因克隆。
方法:分别提取DH-5α(pEGFP-N1)和DH-5α(pMD-18T)质粒,将两个质粒酶切并连接形成重组质粒pMD-18T-GFP,将重组质粒导入DH-5α克隆菌中进行转化,用抗生素抗性筛选后,通过限制性核酸内切酶EcoR I 和 Hind III对所建质粒进行分析鉴定。
关键词:绿色荧光蛋白 DNA重组The cloning of green fluorescent proteinAbstract:Objective: Studies indicated the cloning of the GFP gene.Methods: Extract the plasmid of the DH-5α(pEGFP-N1) and DH-5α (pMD-18T). Then cutting by enzyme and connecting the two plasmids to form pMD-18T-GFP recombined plasmid. The recombinant plasmid confirmed by restriction enzyme and PCR was transferred into E.coli DH-5αto ensure the expression of green fluorescent protein. After resistant screening with antibiotics, analyze and identify the recombined plasmid.Keywords: Green Fluorescent Protein DNA recombination随着分子生物学和基因工程技术的迅速发展和广泛应用,重组DNA技术在发展蛋白质、多肽类药物与疫苗、转基因和基因敲除动物、HGP、基因诊断和基因治疗等方面得到广泛应用。
pMD18-T载体说明

●用 途
■ 进行 TA 克隆,克隆 PCR 产物。 ■ 对克隆后的PCR产物使用BcaBESTTM Sequencing Primers、M13 Primers进行DNA测序。
●pMD18-T Vector 的结构
-1-
【pMD18-T Vector 相关位点说明】
Cloning site BcaBEST Sequencing Primer M13-47 binding site BcaBEST Sequencing Primer RV-M binding site LacZ operator ColE1 ori Ampr
●使用注意
1. Ligation Mix 请于冰中融解。 2. 克隆时使用的 Insert DNA 片段(PCR 产物)建议进行切胶回收纯化,否则 PCR 产物中的短片段
DNA(甚至是电泳也无法确认的非特异性小片段)、残存引物等杂质都会影响 TA 克隆效率。 3. 按照本实验操作进行连接后,直接进行转化时的连接液不要超过 20 µl。当要转化的 DNA 量较大或
40.4
0
* 效率是指白色菌落中的目的 DNA Insert 片段的连入效率。
■一般DNA片段的克隆实验
1)在微量离心管中配制下列 DNA 溶液,全量为 5 µl。
基因工程操作注意事项

基因工程实验注意事项1.上实验课不能迟到。
2.一定要提前预习实验内容,弄懂实验原理,理清实验顺序。
根据实验内容和步骤制定一个详细的试验计划(没有计划或计划不合理者不能进入实验操作)。
3.实验指导中所写的实验一、实验二等并非严格的实验顺序,只是提供一种相关的实验操作技术。
各小组学生需要根据整体的实验策略制定自己的实验顺序和计划。
4.由于实验内容多,时间短,多数实验需要同时或穿插进行,一定要做好统筹安排。
5.本实验课中的所有实验都属于从克隆到表达的整体流程,时间上没有上下午晚上休息等严格的作息安排,一切服从实验进度,但必须在10天之内完成。
6.实验的每一步都要详细地记录操作内容、时间、步骤、结果等,以备查询!7.对任何自己不熟悉的实验仪器都不要随意操作(尤其是微量移液器!)。
在操作的过程中发现任何意外的现象都要及时向任课教师汇报。
8.写作实验报告一定要文字整洁,观察记录详细(包括操作的错误),分析现象透彻。
9.在实验室内不能大声喧哗。
10.在实验的过程中制造的任何垃圾都要丢到垃圾筐里(或先放在自己的桌面一角),严禁随地投弃!11.实验结束后要把用过的器皿清洗后归放整齐并清点数目,向教师汇报征得同意后方可离开实验实。
12.值日组的同学最后离开,等待清扫实验室的卫生,关闭门窗水电。
13.实验时损坏的任何物品都要按规定赔偿。
实验流程参考图12入pH4.8的乙酸钾高盐缓冲液使pH恢复中性时,共价闭合环状质粒DNA复性快速,而线性的染色体DNA复性缓慢,经过离心与蛋白质和大分子RNA一起沉淀下去。
3.器材超净工作台,接种环,酒精灯,台式离心机,旋涡混合器,微量移液取样器,1.5ml微量离心管,恒温摇床,摇菌试管,双面离心管架,试管架,标签纸等,磁力搅拌机。
4.试剂pET-his质粒载体菌,pMD18-T-NK载体菌,LB培养基1000ml(含100ug/ml氨苄青霉素),葡萄糖/Tris/EDTA(GTE)溶液(solution I),NaOH/SDS溶液(solution II),KAc溶液(pH4.8)(solution III),RNase A,95%乙醇,70%乙醇,TE buffer(pH8.0)。
pMD18-T simple vector图谱序列

pMD18-T simple Vector1 TCGCGCGTTT CGGTGATGAC GGTGAAAACC TCTGACACAT GCAGCTCCCG51 GAGACGGTCA CAGCTTGTCT GTAAGCGGAT GCCGGGAGCA GACAAGCCCG 101 TCAGGGCGCG TCAGCGGGTG TTGGCGGGTG TCGGGGCTGG CTTAACTATG 151 CGGCATCAGA GCAGATTGTA CTGAGAGTGC ACCATATGCG GTGTGAAATA 201 CCGCACAGAT GCGTAAGGAG AAAATACCGC ATCAGGCGCC ATTCGCCATT 251 CAGGCTGCGC AACTGTTGGG AAGGGCGATC GGTGCGGGCC TCTTCGCTAT 301 TACGCCAGCT GGCGAAAGGG GGATGTGCTG CAAGGCGATT AAGTTGGGTA 351 ACGCCAGGGT TTTCCCAGTC ACGACGTTGT AAAACGACGG CCAGTGCCAA 401 AGAAGCATGA CGGCAAGTGG ACGAT t------ATCTC CAGAGGATCG CCGGGAACCG 451 AGGACGAGTT CGTAATCATG GTCATAGCTG TTTCCTGTGT GAAATTGTTA501 TCCGCTCACA ATTCCACACA ACATACGAGC CGGAAGCATA AAGTGTAAAG 551 CCTGGGGTGC CTAATGAGTG AGCTAACTCA CATTAATTGC GTTGCGCTCA601 CTGCCCGCTT TCCAGTCGGG AAACCTGTCG TGCCAGCTGC ATTAATGAAT651 CGGCCAACGC GCGGGGAGAG GCGGTTTGCG TATTGGGCGC TCTTCCGCTT 701 CCTCGCTCAC TGACTCGCTG CGCTCGGTCG TTCGGCTGCG GCGAGCGGTA 751 TCAGCTCACT CAAAGGCGGT AATACGGTTA TCCACAGAAT CAGGGGATAA801 CGCAGGAAAG AACATGTGAG CAAAAGGCCA GCAAAAGGCC AGGAACCGTA 851 AAAAGGCCGC GTTGCTGGCG TTTTTCCATA GGCTCCGCCC CCCTGACGAG901 CATCACAAAA ATCGACGCTC AAGTCAGAGG TGGCGAAACC CGACAGGACT 951 ATAAAGATAC CAGGCGTTTC CCCCTGGAAG CTCCCTCGTG CGCTCTCCTG 1001 TTCCGACCCT GCCGCTTACC GGATACCTGT CCGCCTTTCT CCCTTCGGGA 1051 AGCGTGGCGC TTTCTCATAG CTCACGCTGT AGGTATCTCA GTTCGGTGTA 1101 GGTCGTTCGC TCCAAGCTGG GCTGTGTGCA CGAACCCCCC GTTCAGCCCG 1151 ACCGCTGCGC CTTATCCGGT AACTATCGTC TTGAGTCCAA CCCGGTAAGA1201 CACGACTTAT CGCCACTGGC AGCAGCCACT GGTAACAGGA TTAGCAGAGC 1251 GAGGTATGTA GGCGGTGCTA CAGAGTTCTT GAAGTGGTGG CCTAACTACG 1301 GCTACACTAG AAGAACAGTA TTTGGTATCT GCGCTCTGCT GAAGCCAGTT 1351 ACCTTCGGAA AAAGAGTTGG TAGCTCTTGA TCCGGCAAAC AAACCACCGC 1401 TGGTAGCGGT GGTTTTTTTG TTTGCAAGCA GCAGATTACG CGCAGAAAAA 1451 AAGGATCTCA AGAAGATCCT TTGATCTTTT CTACGGGGTC TGACGCTCAG 1501 TGGAACGAAA ACTCACGTTA AGGGATTTTG GTCATGAGAT TATCAAAAAG 1551 GATCTTCACC TAGATCCTTT TAAATTAAAA ATGAAGTTTT AAATCAATCT1601 AAAGTATATA TGAGTAAACT TGGTCTGACA GTTACCAATG CTTAATCAGT 1651 GAGGCACCTA TCTCAGCGAT CTGTCTATTT CGTTCATCCA TAGTTGCCTG1701 ACTCCCCGTC GTGTAGATAA CTACGATACG GGAGGGCTTA CCATCTGGCC1751 CCAGTGCTGC AATGATACCG CGAGACCCAC GCTCACCGGC TCCAGATTTA1801 TCAGCAATAA ACCAGCCAGC CGGAAGGGCC GAGCGCAGAA GTGGTCCTGC1851 AACTTTATCC GCCTCCATCC AGTCTATTAA TTGTTGCCGG GAAGCTAGAG 1901 TAAGTAGTTC GCCAGTTAAT AGTTTGCGCA ACGTTGTTGC CATTGCTACA 1951 GGCATCGTGG TGTCACGCTC GTCGTTTGGT ATGGCTTCAT TCAGCTCCGG 2001 TTCCCAACGA TCAAGGCGAG TTACATGATC CCCCATGTTG TGCAAAAAAG2051 CGGTTAGCTC CTTCGGTCCT CCGATCGTTG TCAGAAGTAA GTTGGCCGCA 2101 GTGTTATCAC TCATGGTTAT GGCAGCACTG CATAATTCTC TTACTGTCAT 2151 GCCATCCGTA AGATGCTTTT CTGTGACTGG TGAGTACTCA ACCAAGTCAT 2201 TCTGAGAATA GTGTATGCGG CGACCGAGTT GCTCTTGCCC GGCGTCAATA 2251 CGGGATAATA CCGCGCCACA TAGCAGAACT TTAAAAGTGC TCATCATTGG2301 AAAACGTTCT TCGGGGCGAA AACTCTCAAG GATCTTACCG CTGTTGAGAT 2351 CCAGTTCGAT GTAACCCACT CGTGCACCCA ACTGATCTTC AGCATCTTTT 2401 ACTTTCACCA GCGTTTCTGG GTGAGCAAAA ACAGGAAGGC AAAATGCCGC2451 AAAAAAGGGA ATAAGGGCGA CACGGAAATG TTGAATACTC ATACTCTTCC2501 TTTTTCAATA TTATTGAAGC ATTTATCAGG GTTATTGTCT CATGAGCGGA 2551 TACATATTTG AATGTATTTA GAAAAATAAA CAAATAGGGG TTCCGCGCAC 2601 ATTTCCCCGA AAAGTGCCAC CTGACGTCTA AGAAACCATT ATTATCATGA 2651 CATTAACCTA TAAAAATAGG CGTATCACGA GGCCCTTTCG TC。
TA克隆

PCR产物克隆大致分为两类,即平头连接和粘头连接。
平头连接是将制备好的平头载体和补平或削平的PCR产物直接进行连接。
载体可用EcoR V 或Sma I切成平头;PCR产物纯化后,可以在22℃用DNA聚合酶I作用30min(利用该酶所具有的3’→5’外切酶活性和5’→3’的聚合酶活性)。
如果要求不高,PCR产物也可不加处理。
如果使用Stratagene公司的pfu DNA聚合酶或New England Biolabs公司的Vent DNA聚合酶,这两种酶有5’→3’校对能力,扩增出来的PCR产物已经是平头,可以不作平端处理。
平端连接的一个显而易见的缺陷是连接效率低下,即使使用很高单位的连接酶,或在反应体系中加入PEG 8000,也只能很有限地提高效率。
粘头连接也可以大致分为两类,一类粘头连接是用某种方法,在载体和PCR产物上产生长的可互补的粘性末端。
最普遍的方法是在引物的5’端加入一段某种限制酶的识别序列。
如果两个引物选用不同的限制性内切酶识别序列,就可以做到定向连接。
另一类利用部分PCR产物3’端带有一个凸出的dAMP的特性,构建3’端带有凸出的dTMP的载体。
一般采用的方法是先把载体用某种限制性内切酶消化成平头,在70℃或72℃下在只加入一种dNTP、即dTTP的反应体系中用Taq DNA聚合酶处理半小时(也有人报道处理1~2小时能提高克隆效率,这样加T反应会更彻底)。
也可以用末端转移酶来完成加T反应。
载体自连、PCR产物串连可以忽略。
如果使用ddTTP,效果会更好。
这种方法一般称为加T/A法克隆,比平头连接效率高50~100倍。
一、PCR产物的TA克隆1. TA克隆构建原理:TA克隆系统由Invitrogen公司(San Diego,CA)发展而来的商业性试剂盒,它用于PCR产物的克隆和测序。
其原理是利用Taq酶能够在PCR产物的3’末端加上一个非模板依赖的A,而T载体是一种带有3’T突出端的载体,在连接酶作用下,可以快速地、一步到位地把PCR产物直接插入到质粒载体的多克隆位点(MCS)中。
pmd18-19-T 质粒图谱

GAGCGGATAACAATTTCACACAGGAAACAGCTATGACCATGATTACGCCAAGCTTGCATGCCTGCAGGTCGACGATATCTCTAGAGGATCCCCGGGTACCGAGCTCGAATTC
ACTGGCCGTCGTTTTACAACGTCGTGACTGGGAAAACCCTGGCG CAGCACTGACCCTTTTGGGACCGC BcaBESTTM Sequencing Primer M13-47
LacZ operator
146 ~ 469
ColE1 ori
852 ~ 1466
Ampr
1626 ~ 2486
■ 实验操作 Control DNA片段的克隆实验 A.操作方法
1)在微量离心管中配制下列DNA溶液,全量为5 µl。
pMD18-T or pMD19-T Vector*1
1 µl
Control Insert*2
■ 相关说明 1.感受态细胞的选择。
PCR产物克隆系列载体
PCR产物克隆系列载体
转化时请使用高效的热转化感受态细胞(转化效率≥1
×108 cfu/µg pUC19),这样才可能得到比较理想的阳
性克隆。 如果需要进行蓝白筛选时,宿主细胞必须具
有正确的基因型(F'编码的[ △M15])产生 ω
Fragment,才可能和载体DNA产生的
ng DNA/ml),将上述培养液稀释10倍后(0.01 ng
DNA/ml)取100 µl涂布平板(0.001 ng DNA/100
µl),记录菌落数。以得到200个克隆体为例计算转化效 率,此时的转化效率=200 cfu/0.001 ng=2×105
cfu/ng=2×108 cfu/µg pUC19 DNA。
PMD永磁t调速产品介绍

GB/T14549-93中规定,公用电网谐波电压 (相电压)限值为380V(220V)电网电压总THDv为 5%,各次谐波电压含有率奇次为4%,偶次为2%。
如图所示,永磁调速器没有被测试到它对其它与之相连 的设备会产生谐波。永磁调速器只是一个简单的机械装 置,当它对负载进行调速时,电机始终处于额定转速运 行,故不会降低电机的功率因数,因此它对周围系统的 功效均没有影响。
永磁调速器安装于电机和负载之间,在电机转 速不变的情况下,通过调节气隙或耦合面积,改变 负载转速,从而实现对流量或压力的连续控制。
永磁传动技术主要是传递扭矩
电机输出的扭矩=负载设备所需扭矩
离心式负载符合流体机械相似定律
Q1/Q2 = (n1/n2) (流量变化与转速变化成正比) H1/H2 = (n1/n2)2 (压力变化与转速变化的平方成正比) T1/T2 = (n1/n2)2 (负载扭矩变化与转速变化的平方成正比) 电机输出功率P1=T*n (功率=扭矩*转速)
2
永磁调速器显著优点
减少 振动
减缓负 载冲击
高效 节能
无级 调速
零负载 启动
容易 安装
高可 靠性
延长设 备寿命
堵转 保护
无谐波
恶劣环 境使用
1 高效节能
(可无级0~98.5%调整转速)
2 简单
(构造简单、容易安装、本身无需电源)
3 可靠
(不怕恶劣环境,MTBF长达30年)
4 软启动
(电机可在空载状态启动,降低启动电流, 减少启动时间)
KW
5.5 7.5 11 15 18.5 22 30 37 45 55 75 90 110 132 160 200 250 315 355 400 450 500 500~800 800~1300 1300~1800 1800~2500 2500~3200 3200~4000
pMD18-T酶切位点及序列

pMD18-T酶切位点及序列Restriction analysis on DNAMAN1Methylation: dam-No dcm-NoScreened with 1364 enzymes, 809 sites found AacI 1 G/GATCC436AaeI 1 G/GATCC436AaqI 3 G/TGCAC177 **** ****AatII 1 GACGT/C2628AcaII 1 G/GATCC436AcaIII 2 TGC/GCA258 1928Acc113I 1 AGT/ACT2186Acc16I 2 TGC/GCA258 1928Acc65I 1 G/GTACC445AccBSI 3 GAG/CGG746 2547 505AccEBI 1 G/GATCC436AceIII 3 CAGCTCNNNNNNN/54 765 2005AclI 2 AA/CGTT1932 2305Acs1371I 1 G/TCGAC 417Acs1372I 1 G/TCGAC 417Acs1373I 1 G/TCGAC 417Acs1421I 1 G/TCGAC 417Acs1422I 1 G/TCGAC 417Afa16RI 2 CGAT/CG279 2076Afa22MI 2 CGAT/CG 279 2076AflIV 1 AGT/ACT2186AhaB8I 1 G/GTACC445AhaIII 3 TTT/AAA1572 1591 2283AhdI 1 GACNNN/NNGTC 1706AhyI 1 C/CCGGG441AinI 1 CTGCA/G415AinII 1 G/GATCC436AjoI 1 CTGCA/GAli12257I 1 G/GATCC 436Ali12258I 1 G/GATCC 436Ali2882I 1 CTGCA/G415AliAJI 1 CTGCA/G415AliI 1 G/GATCC436Alw44I 3 G/TGCAC177 **** ****AlwNI 1 CAGNNN/CTG 1229AmeI 3 G/TGCAC177 **** ****AosI 2 TGC/GCA258 1928ApaBI 1 GCANNNNN/TGC 186ApaCI 1 G/GATCC436ApaLI 3 G/TGCAC177 **** ****ApiI 1 CTGCA/G415AseI 3 AT/TAAT584 643 1878AsiI 1 G/GATCCAsnI 3 AT/TAAT584 643 1878Asp3065I 1 A/AGCTT399Asp36I 1 CTGCA/G415Asp52I 1 A/AGCTT399Asp5HI 1 GCATG/C409Asp700I 1 GAANN/NNTTC 2305Asp708I 1 CTGCA/G415Asp713I 1 CTGCA/G415Asp718I 1 G/GTACC445Asp763I 1 AGT/ACT 2186AspEI 1 GACNNN/NNGTC 1706AspJI 1 GACGT/C2628AspTI 1 CTGCA/G415AspTII 1 G/GATCC436Atu1II 1 G/GATCCAviII 2 TGC/GCA258 1928BamFI 1 G/GATCC436BamHI 1 G/GATCC436BamKI 1 G/GATCC436BamNI 1 G/GATCC436BavAI 2 CAG/CTG308 637BavBI 2 CAG/CTG308 637BavI 2 CAG/CTG308 637BbeAI 1 GG/CGCC236BbeI 1 GGCGC/C239BbiI 1 CTGCA/G415BbrAI 1 A/AGCTT399BbrI 1 A/AGCTT399BbuI 1 GCATG/C409BbvAI 1 GAANN/NNTTCBca1259I 1 G/GATCC436Bce170I 1 CTGCA/G415Bce751I 1 G/GATCC436Bce83I 4 CTTGAGNNNNNNNNNNNNNNNN/ 1202 904 1443 2311BcgI 1 GCANNNNNNTCGNNNNNNNNNNNN/ 2211Bco10278I 1 G/GATCC436Bco116I 3 CTCTTCN/296 697 2501Bco35I 1 CTGGAGNNNNNNNNNNNNNNNN/ 1776Bco5I 3 CTCTTCN/296 697 2501Bco6I 2 TGC/GCA258 1928BcoKI 3 CTCTTCN/296 697 2501BglI 2 GCCNNNN/NGGC251 1826BinSII 1 GG/CGCC236Bli49I 1 GGTCTCN/1767Bli736I 1 GGTCTCN/BmaAI 2 CGAT/CG279 2076BmaBI 2 CGAT/CG279 2076BmaCI 2 CGAT/CG279 2076BmaDI 2 CGAT/CG279 2076BmaI 2 CGAT/CG279 2076BmeBI 1 CTGCA/G415BnaI 1 G/GATCC436BpeI 1 A/AGCTT399BpmI 1 CTGGAGNNNNNNNNNNNNNNNN/ 1776 BpoAI 3 AT/TAAT584 643 1878BsaDI 1 G/GATCC436BsaI 1 GGTCTCN/1767BsaNII 1 CTGCA/G415BsaQI 1 CTGCA/G415BsaTI 2 TGC/GCA258 1928BscDI 1 CTGCA/G415BseHI 1 A/AGCTT399BseZI 3 CTCTTCN/296 697 2501BshHI 1 AGT/ACT2186BsiI 3 C/TCGTG986 2370 2677BsmBI 1 CGTCTCN/45BsmGII 1 A/AGCTT399BsoJI 2 GCCNNNN/NGGC 251 1826BsoSI 1 AGT/ACT2186Bsp107I 1 CTGCA/G415Bsp108I 1 CTGCA/G415Bsp121I 1 GCATG/C409Bsp130I 1 G/GATCC436Bsp131I 1 G/GATCC436Bsp144I 1 G/GATCC436Bsp146I 3 G/TGCAC177 **** ****Bsp153AI 2 CAG/CTG308 637Bsp17I 1 CTGCA/G415Bsp22I 1 CTGGAGNNNNNNNNNNNNNNNN/1776Bsp24I 2 GACNNNNNNTGGNNNNNNNNNNNN/ 1516 2610 Bsp268I 1 CTGCA/G415Bsp28I 1 CTGGAGNNNNNNNNNNNNNNNN/1776Bsp30I 1 G/GATCC436Bsp4009I 1 G/GATCC436Bsp43I 1 CTGCA/G415Bsp46I 1 G/GATCC436Bsp63I 1 CTGCA/G415Bsp6II 2 CTGAAGNNNNNNNNNNNNNNNN/1361 2373Bsp78I 1 CTGCA/G415Bsp81I 1 CTGCA/G415Bsp93I 1 CTGCA/G415Bsp98I 1 G/GATCC436BspBI 1 CTGCA/G415BspCI 2 CGAT/CG279 2076BspHI 3 T/CATGA1533 2541 2646BspJ106I 1 GGTAC/C449BspJ74I 1 CTGGAGNNNNNNNNNNNNNNNN/ 1776 BspKT5I 2 CTGAAGNNNNNNNNNNNNNNNN/ 1361 2373 BspKT8I 1 A/AGCTT399BspLU11I 1 A/CATGT813BspLU11II 1 T/CTAGA430BspM39I 2 CAG/CTG308 637BspMI 1 ACCTGCNNNN/404BspO4I 2 CAG/CTG308 637BsrBI 3 GAG/CGG746 2547 505BsrDI 2 GCAATGNN/1767 1941BsrEI 3 CTCTTCN/296 697 2501BsrXI 1 T/CTAGA430BssSI 3 C/TCGTG986 2370 2677Bst1126I 1 G/GATCC436Bst158I 3 CTCTTCN/296 697 2501Bst170II 1 A/AGCTT399Bst2464I 1 G/GATCC436Bst2902I 1 G/GATCC436Bst2BI 3 C/TCGTG986 2370 2677BstD102I 3 GAG/CGG746 2547 505BstFI 1 A/AGCTT399BstI 1 G/GATCC436BstMI 1 AGT/ACT2186BstQI 1 G/GATCC436BstZ16I 1 G/TCGAC417BstZ2I 1 GACNNN/NNGTC 1706279 2076Bsu8565I 1 G/GATCC 436Bsu8646I 1 G/GATCC 436Bsu90I 1 G/GATCC 436BsuBI 1 CTGCA/G 415BtgAI 1 G/TCGAC 417BtgAII 1 GCATG/C 409Cas2I 2 CGAT/CG 279 2076CauIII 1 CTGCA/G 415CelI 1 G/GATCC 436CflI 1 CTGCA/G415Cfr32I 1 A/AGCTT 399Cfr51I 2 CGAT/CG 279 2076Cfr56I 1 GGTCTCN/ 1767Cfr6I 2 CAG/CTG 308 637441CfrA4I 1 CTGCA/G415CfrJ4I 1 CCC/GGG443CfuII 1 CTGCA/G415CglAI 1 GCATG/C409CglAII 1 G/TCGAC417ChuI 1 A/AGCTT399ClcI 1 CTGCA/G415ClcII 2 TGC/GCA258 1928 CliII 2 TGC/GCA 258 1928CstI 1 CTGCA/G415DdsI 1 G/GATCC436DmaI 2 CAG/CTG308 637DpaI 1 AGT/ACT2186DraI 3 TTT/AAA1572 1591 2283DrdI 2 GACNNNN/NNGTC97 921DrdIII 2 CGAT/CG279 2076EaeAI 1 C/CCGGG441EaePI 1 CTGCA/G415EagBI 2 CGAT/CG279 2076Eam1104I 3 CTCTTCN/296 697 2501Eam1105I 1 GACNNN/NNGTC 1706EarI 3 CTCTTCN/296 697 2501Ecl133I 1 CTGCA/G415Ecl136II 1 GAG/CTC453Ecl137I 1 GAGCT/C455Ecl1zI 1 CTGCA/G415Ecl2zI 1 CTGCA/G415Ecl37kI 1 CTGCA/G415Ecl593I 1 CTGCA/G415Ecl699kI 1 CTGCA/GEcl77I 1 CTGCA/G415EclHKI 1 GACNNN/NNGTC1706EclI 2 CAG/CTG308 637EclJI 2 CGAT/CG279 2076EclRI 1 C/CCGGG441Eco101I 1 GGTCTCN/1767Eco112I 2 CTGAAGNNNNNNNNNNNNNNNN/ 1361 2373 Eco120I 1 GGTCTCN/1767Eco125I 2 CTGAAGNNNNNNNNNNNNNNNN/ 1361 2373 Eco127I 1 GGTCTCN/1767Eco129I 1 GGTCTCN/1767Eco149I 1 GGTAC/C449Eco155I 1 GGTCTCN/1767Eco156I 1 GGTCTCN/1767Eco157I 1 GGTCTCN/1767Eco159I 1 G/AATTCEco161I 1 CTGCA/G415Eco162I 1 GGTCTCN/ 1767Eco167I 1 CTGCA/G415Eco185I 1 GGTCTCN/ 1767Eco188I 1 A/AGCTT399Eco191I 1 GGTCTCN/ 1767Eco203I 1 GGTCTCN/ 1767Eco204I 1 GGTCTCN/ 1767 Eco205I 1 GGTCTCN/ 1767 Eco217I 1 GGTCTCN/ 1767 Eco225I 1 GGTCTCN/ 1767 Eco228I 1 G/AATTC 457 Eco231I 1 A/AGCTT 399 Eco233I 1 GGTCTCN/ 1767 Eco237I 1 G/AATTC 457 Eco239I 1 GGTCTCN/ 1767 Eco240I 1 GGTCTCN/ 1767 Eco241I 1 GGTCTCN/ 1767 Eco246I 1 GGTCTCN/ 1767 Eco247I 1 GGTCTCN/ 1767 Eco252I 1 G/AATTC 457 Eco255I 1 AGT/ACTEco260I 1 CTGCA/G415Eco261I 1 CTGCA/G415Eco263I 1 GGTCTCN/1767Eco31I 1 GGTCTCN/1767Eco42I 1 GGTCTCN/1767Eco48I 1 CTGCA/G415Eco49I 1 CTGCA/G415Eco51I 1 GGTCTCN/1767Eco57I 2 CTGAAGNNNNNNNNNNNNNNNN/ 1361 2373 Eco65I 1 A/AGCTT399Eco78I 1 GGC/GCC237Eco82I 1 G/AATTC457Eco83I 1 CTGCA/G415Eco95I 1 GGTCTCN/1767Eco97I 1 GGTCTCN/1767Eco98I 1 A/AGCTT399EcoA4I 1 GGTCTCN/ 1767EcoICRI 1 GAG/CTC453EcoO44I 1 GGTCTCN/ 1767 EcoRI 1 G/AATTC457EcoVIII 1 A/AGCTT399EheI 1 GGC/GCC237ErhB9I 2 CGAT/CG279 2076Esp141I 1 CTGCA/G415Esp16I 1 CGTCTCN/45Esp19I 1 GGTAC/C449Esp23I 1 CGTCTCN/45Esp3I 1 CGTCTCN/45Esp5II 1 CTGCA/G。
pMD18-T

上培养,形成单菌落。计数白色、蓝色菌落。 8)挑选白色菌落,使用PCR法确认载体中插入片段的
长度大小。 B. 结 果
使用Control Insert时的连接/转化结果如下,使用的感受 态细胞的转化效率为:1.5×108 cfu/µg pUC19 DNA。
上培养,形成单菌落。计数白色、蓝色菌落。 8)挑选白色菌落,使用PCR法确认载体中插入片段的
长度大小。
*1 pMD18-T Simple Vector or pMD19-T Simple Vector的使用量 取0.5 µl实验也可得到满意的结果。实际操作时,可按实验需要确 定T Vector 的使用量。T Vector 1 µl(50 ng)的摩尔数约为0.03 pmol。
852 ~ 1466
Ampr
1626 ~ 2486
【pMD19-T Simple Vector相关位点说明】
Cloning site
431
BcaBEST Sequencing Primer M13-47 binding site 347 ~ 370
BcaBEST Sequencing Primer RV-M binding site 478 ~ 500
化感受态细胞中后,再加入900 µl的SOC培养基(0.1
ng DNA/ml),将上述培养液稀释10倍后(0.01 ng
DNA/ml)取100 µl涂布平板(0.001 ng DNA/100
µl),记录菌落数。以得到200个克隆体为例计算转化效 率,此时的转化效率=200 cfu/0.001 ng=2×105
T-A基因克隆的技术要点.ppt

Page ▪ 14
外源DNA的重组与转化
▪DNA的体外连接重组
▪ 其本质是一个酶促反应过程,即在一定的条件下,DNA连接酶 催化两个双链DNA片段的5’端磷酸和3’端羟基之间相互作用, 形成磷酸二酯键的过程。
▪ DNA连接酶有两种:T4噬菌体DNA连接酶 大肠杆菌DNA连接酶。
▪ lacZ 编码半乳糖苷酶的N端,它可以和宿主菌株产生的半乳糖苷酶C端互
补,形成有活性的β-半乳糖苷酶。
▪ Xgal 5-溴-4-氯-3-吲哚-β-D-半乳糖苷 ,β–半乳糖苷酶的底物,水解后呈
蓝色
▪ IPTG 异丙基硫代半21
蓝白斑
Page ▪ 22
T-A基因克隆的技术要点.ppt
Page ▪ 2
Page ▪ 3
3’-A突出的PCR产物
▪ 使用不同的Taq酶,在PCR扩增循环结束后,加上72℃10分钟 一个过程,Taq酶可以在扩增产物的3末端加上A 。
▪ 使用高保真的DNA聚合酶,如pfu酶,其不能在扩增产物的3 末端加上A,得到的DNA序列为钝端,因此,在回收纯化后进 行加A的过程,通常是以PCR回收产物为模板,加上一定量的 普通Taq酶和反应液,加入dATP(或dNTP), 72℃ 10分钟 。
混合物中的DNA形成抗DNA酶的羟基-钙磷酸复合物黏附于细 胞表面,经42˚C短时间热击处理,促进细胞吸收DNA复合物 。将细菌放置在非选择性培养基中保温一段时间,促使在转化 过程中获得新的表型(如Amp+等)得到表达,然后将此细菌 培养物涂在含有氨苄青霉素的选择性培养基上进行培养获得阳 性克隆。
Page ▪ 17
▪ 如果使用ddTTP,效果会更好。
工艺库说明——精选推荐

⼯艺库说明⼯艺库说明⼯艺库是采⽤0.18um的⼯艺,下⾯是调⽤⼯艺库名称及尺⼨范围,⼯艺库的调⽤模型的名称可以⾃⼰进⼯艺库⾥⾯改,⽐如说我这⾥⽤的是nmos18表⽰nmos管,供给电压为1.8V。
⼯艺⾥⾯的数值可以⾃⼰修订,但是如果要流⽚的话必须要和⼯⼚的⼯艺库⼀样。
具体尺⼨说明如下,调⽤名称调⽤类型基本的宽长范围nmos18TT L:240nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8Vpmos18TT L:240nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8Vnmos18SS L:240nm-50um,W:240nm-100um,tox=4.45nm,vdd=1.8Vpmos18SS L:240nm-50um,W:240nm-100um,tox=4.45nm,vdd=1.8Vnmos33TT L:500nm-50um,W:800nm-100um,tox=7nm,vdd=3.3Vpmos33TT L:500nm-50um,W:800nm-100um,tox=7nm,vdd=3.3Vnmos18FF L:240nm-50um,W:240nm-100um,tox=3.5nm,vdd=1.8Vpmos18FF L:240nm-50um,W:240nm-100um,tox=3.5nm,vdd=1.8Vnmos18SNFP L:240nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8Vpmos18SNFP L:240nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8Vnmos18FNSP L:240nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8Vpmos18FNSP L:240nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8Vnmos33MM L:500nm-50um,W:800nm-100um,tox=7nm,vdd=3.3V pmos33MM L:500nm-50um,W:800nm-100um,tox=7nm,vdd=3.3V nmos33NN L:500nm-50um,W:800nm-100um,tox=7.5nm,vdd=3.3V pmos33NN L:500nm-50um,W:800nm-100um,tox=7.5nm,vdd=3.3V nmos33OO L:500nm-50um,W:800nm-100um,tox=6.5nm,vdd=3.3V pmos33OO L:500nm-50um,W:800nm-100um,tox=6.5nm,vdd=3.3V nmos33PP L:500nm-50um,W:800nm-100um,tox=7nm,vdd=3.3V pmos33PP L:500nm-50um,W:800nm-100um,tox=7nm,vdd=3.3V nmos33QQ L:500nm-50um,W:800nm-100um,tox=7nm,vdd=3.3V pmos33QQ L:500nm-50um,W:800nm-100um,tox=7nm,vdd=3.3V nmos018RR L:300nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8V nmos033RR L:500nm-50um,W:800nm-100um,tox=7nm,vdd=3.3V nmos033UU L:500nm-50um,W:800nm-100um,tox=7.5nm,vdd=3.3V nmos033VV L:500nm-50um,W:800nm-100um,tox=6.5nm,vdd=3.3V nmos033WW L:500nm-50um,W:800nm-100um,tox=7.25nm,vdd=3.3V nmos033XX L:500nm-50um,W:800nm-100um,tox=6.75nm,vdd=3.3V nch YY L:180nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8V pch YY L:180nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8V nch ZZ L:180nm-50um,W:240nm-100um,tox=3.95nm,vdd=1.8V pch ZZ L:180nm-50um,W:240nm-100um,tox=3.95nm,vdd=1.8V nch AA L:180nm-50um,W:240nm-100um,tox=4.45nm,vdd=1.8V pch AA L:180nm-50um,W:240nm-100um,tox=4.45nm,vdd=1.8V nch BB L:180nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8V pch BB L:180nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8V nch CC L:180nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8V pch CC L:180nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8V nch EE L:180nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8V pch EE L:180nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8V nch GG L:180nm-50um,W:240nm-100um,tox=3.95nm,vdd=1.8V pch GG L:180nm-50um,W:240nm-100um,tox=3.95nm,vdd=1.8V nch HH L:180nm-50um,W:240nm-100um,tox=4.45nm,vdd=1.8Vpch HH L:180nm-50um,W:240nm-100um,tox=4.45nm,vdd=1.8Vnch II L:180nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8Vpch II L:180nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8Vnch JJ L:180nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8Vpch JJ L:180nm-50um,W:240nm-100um,tox=4.2nm,vdd=1.8Vbjtp KK饱和电流is=5.6*10-18,放⼤倍数bf=1.38,pnp,5*5um2bjtp1KK饱和电流is=2.8*10-17,放⼤倍数bf=1.36,pnp,10*10um2np KK⾯积=8e-9,饱和电流=2.82e-7np1KK⾯积=8e-9,饱和电流=1.17e-7np2KK⾯积=9.72e-8,饱和电流=4.204e-6np1FM⾯积=8e-9,饱和电流=2.82e-7pn1FM⾯积=8e-9,饱和电流=1.17e-7Psub/np FM⾯积=9.72e-8,饱和电流=4.204e-6注:还有很多其他的类型如FF,SNFP你都可以通过打开⼯艺库⾃⼰根据需要选择。
