分子进化树的构建
分子进化树构建方法

MP法建树流程
Sequence1 Sequence2 Sequence3
Sequence4
Position 1
Position 1 2 3 T G C T A C A G G A A G
If 1 and 2 are grouped a total of four changes are needed.
5
genetic change
系统发生树术语
Rooted tree vs. Unrooted tree
无 A 有 根 根 树 B 树 two major ways to root trees:
A
10 3 2 5
C D
By midpoint or distance
d (A,D) = 10 + 3 + 5 = 18 Midpoint = 18 / 2 = 9
Distance Uses only pairwise distances Minimizes distance between nearest neighbors Very fast Easily trapped in local optima Good for generating tentative tree, or choosing among multiple trees Maximum parsimony Uses only shared derived characters Minimizes total distance Maximum likelihood Uses all data Maximizes tree likelihood given specific parameter values Very slow Highly dependent on assumed evolution model Good for very small data sets and for testing trees built using other methods
分子进化树构建方法

C B
2
D
outgroup
外群、外围支
Rooted tree vs. Unrooted tree
plant animal
plant
plant animal
Unrooted tree
fungus
animal
bacterium
plant plant plant
animal
Rooted tree
Monophyletic group
Cat Dog Rat Cow 3 4 6 5 7 6 Dog Dog Rat Cat
1
2 2 1 4
计算序 列的距 离,建 立距离 矩阵
Rat
通过距 离矩阵 建进化 树
Cow
Step1. 计算序列的距离,建立距离矩阵
对位排列, 去除空格 (选择替代模型)
Uncorrected “p” distance (=observed percent sequence difference) Kimura 2-parameter distance (estimate of the true number of substitutions between taxa)
A
节点 Node
祖先节点/树 根
Root
内部节点/分歧点
该分支可能的祖先 HTU
系统发生树术语
A clade(进化支) is a group of organisms that includes an ancestor and all descendents of that ancestor. 分支树
Step2. 通过矩阵建树 由进化距离构建进化树的方法有很多,常见有:
1. Unweighted Pair Group Method with Arithmetic mean (UPGMA)
分子进化:系统树的构建

98
祖先序列
A C T G A A C G T A A C G C
A C T G A→C→T A C→G G T→A A A→C→* T C G C
A C→A T G A A C→A G T→A A A * →T C G C→ +T→C
单一置换 (single substitution) 多重置换 (multiple substitutions) 同义置换 (coincidental substitutions) 平行置换 (parallel substitutions) 趋同置换 (convergent substitution) 反转置换 (back substitution)
101
(应用算术平均数的非加权成组配对法, unweighted pair-group method using an arithmetic average)。该法将类间距离定义为两个类的成员所列的线粒体 DNA 序列的资料。每对序列间的 Jukes-Cantor 距离取决于每对序列间差异核苷酸的观察数。如果在两条序列中 相同碱基的比例为 q,则距离 K 可估计为
第五章
分子进化:系统树的构建
自 20 世纪中叶,随着分子生物学的不断发展,进化研究也进入了分子进化 (molecular evolution)研究水平,并建立了一套依赖于核酸、蛋白质序列信息 的理论和方法。随着基因组测序计划的实施,基因组的巨量信息对若干生物领域 重大问题的研究提供了有力的帮助, 分子进化研究再次成为生命科学中最引人注 目的领域之一。 这些重大问题包括: 遗传密码的起源、 基因组结构的形成与演化、 进化的动力、生物进化等等。分子进化研究目前更多地是集中在分子序列上,但 随着越来越多生物基因组的测序完成,从基因组水平上探索进化奥秘,将开创进 化研究的新天地。人与老鼠的基因组大小相似,都含有约 30 亿碱基对,基因的 数量也相近, 可人与老鼠为何差异如此之大?从进化的角度如此解释?是否可以 在浩如烟海的基因组密码中获得答案?
分子进化总结分析—系统发生树的构建要求

系统发育树构建的基本方法
Distance-based methods 基于距离的方法
Unweightedpair group method using arithmetic average (UPGMA) 非加权分组平均法 Minimum evolution(ME)最小进化方法 Neighbor joining(NJ)邻位归并法
打开软件clustalx
• CLUSTALX-是CLUSTAL多重序列比对程序的 Windows版本。Clustal X为进行多重序列和轮廓比 对和分析结果提供一个整体的环境。 序列将显示屏幕的窗口中。采用多色彩的模式可 以在比对中加亮保守区的特征。窗口上面的下拉 菜单可让你选择传统多重比对和轮廓比对需要的 所有选项。
分子进化分析—— 系统发生发育分析是研究物种进化和系统分类的一种 方法,研究对象为携带遗传信息的生物大分子序 列,采用特定的数理统计算法来计算生物间的生 物系统发生的关系。并用系统进化树来概括生物 间的这种亲缘关系。
2
分子系统发育分析
• 系统发育进化树( Phylogenetic tree) 用一种类似树状分支的图形来概括各种生物之间的亲缘关系。
• 名 称: Uncultured bacterium clone YU201H10 • 序列号: FJ694683 /FJ694514 • 文 献: TITLE Circumpolar synchrony in big river
bacterioplankton • 序列长度:353 • 相 似 比: 99% • 核酸序列 • 分类地位
• Clustalx比对结果是构建系统发育树的前提
具体步骤
• 根据需要,选定要比对的菌株及相应的序 列。将序列COPY至记事本
分子进化与系统进化树的构建

分子进化与系统进化树的构建分子进化与系统进化树的构建分子进化与系统进化树的构建主要内容:1、分子进化的研究方法2、系统进化树的构建方法3、系统进化树构建常用软件汇集4、系统进化树构建方法及软件的选择5、Phylip分子进化分析软件包简介及使用6、如何利用MEGA3.1构建进化树声明:1、本篇涉及的资源主要源于网络及相关书籍,由酷友搜集、分析、整理、审改,供大家学习参考用,如有转载、传播请注明源于基因酷及本篇的工作人员;若本篇侵犯了您的版权或有任何不妥,请Email genecool@告知。
2、由于我们的学识、经验有限,本篇难免会存在一些错误及缺陷,敬请不吝赐教:请到基因酷论坛(/bbs)本篇对应的专题跟贴指出或Email genecool@。
致谢:整编者:flashhyh主要参考资料:《生物信息学札记》樊龙江;《分子进化分析与相关软件的应用》作者不详;《进化树构建》ZHAO Yangguo;《如何用MEGA 3.1构建进化树》作者不详;《MEGA3指南》作者不详;分子进化的研究方法分子进化的研究方法分子进化的研究方法分子进化研究的意义自20世纪中叶,随着分子生物学的不断发展,进化研究也进入了分子进化(molecularevolution)研究水平,并建立了一套依赖于核酸、蛋白质序列信息的理论和方法。
随着基因组测序计划的实施,基因组的巨量信息对若干生物领域重大问题的研究提供了有力的帮助,分子进化研究再次成为生命科学中最引人注目的领域之一。
这些重大问题包括:遗传密码的起源、基因组结构的形成与演化、进化的动力、生物进化等等。
分子进化研究目前更多地是集中在分子序列上,但随着越来越多生物基因组的测序完成,从基因组水平上探索进化奥秘,将开创进化研究的新天地。
分子进化研究最根本的目的就是从物种的一些分子特性出发,从而了解物种之间的生物系统发生的关系。
通过核酸、蛋白质序列同源性的比较进而了解基因的进化以及生物系统发生的内在规律。
进化树构建方法

P(B)=0.001*0.99+0.999*0.02=0.02097=> 人群中任取一人被检测为阳性的概率
贝叶斯-例子
临床检测: 初检为阳性的结果并不可怕,因此确诊需要复检 假设二次检查,再次检出为阳性 问: 患病的概率有多大 初检为阳性:P(B) 复检为阳性:P(C)
则两次都为阳性的情况下该人患病的概率为
给定核苷酸 i 在时间t之后变成j 的概率。矩阵P(t)= {pijt)} 时间*速率=距离=>概率
距离计算-JC69
横坐标d=3 *t
此公式的推导,考虑了所有的路径,因此可以矫正回复突变或平行突变 进化速率 和进化时间 t 以乘积形式出现 =>
AAAAAAAA => AATTGGCC
距离计算-JC69
贝叶斯定理
贝叶斯-例子
临床检测: 假设一个人被感染HIV,医院检测其为阳性的概率为99%。 真阳性 假设一个人未被感染,医院检测其为阳性的概率为2%。假阳性 假设HIV的人群发病率0.1% 问:若一个人被查出阳性,那么此人患病的概率为多少?
A: 感染, B: 阳性, B|A: 染病情况下查出阳性,A|B, 查出阳性情况下染病
进化树构建方法
邢鹏伟
2018.11
内节点(灭绝物种) 外节点(现存物种)
分子钟置根法:如果在所有时间内进化速率是恒定的,即假定存在分子钟 产生有根树的条件: 外类群置根法:在树重建中引入关系较远的物种,同时在对所有物种重建的无根树中, 将树根置于连接外类群的枝,使得内类群的子树有根
邻接法 Neighbour joining 基于距离 distance-based 最小二乘法 Least squares 非加权算数平均组对(UPGMA )法
分子系统发育树构建的简易方法

分子系统发育树构建的简易方法
分子系统发育树的构建是根据分子序列的差异来推断不同物种之间的进化关系。
下面是一个简易的分子系统发育树构建方法:
1. 选择目标基因序列:选择与所研究物种相关的基因序列(如核糖体RNA或蛋白质编码基因)作为目标序列。
2. 数据收集:收集各个相关物种的目标基因序列数据。
可以通过公共数据库(如NCBI)或研究文献中的已有数据进行获取。
3. 序列比对:使用序列比对软件将收集到的序列进行比对,找出相同和不同的碱基或氨基酸位置。
常用的比对软件有CLUSTALW和MAFFT。
4. 构建进化树:根据序列比对结果,使用进化树构建软件(如MEGA)进行系统发育树的构建。
常用的进化树构建方法包括最大简约法(UPGMA)和最大似然法(ML)。
5. 进化树评估:对构建的系统发育树进行评估,可以使用Bootstrap方法进行支持值分析,提高树的可靠性。
6. 结果解读:根据构建的系统发育树,可以解读不同物种之间的进化关系和群体间的分化程度。
需要注意的是,分子系统发育树是基于目标基因序列的进化关系推断,仅仅代表目标基因的进化历史,并不一定能完全反映
整个物种的进化历史。
因此,在研究中还需要综合考虑其他重要因素,如形态特征和生态行为等。
分子进化树的构建方法

分子进化树的构建方法分子进化树的构建方法分类:实验探索|标签:|字号大2011-05-21 09:33:32|中小订阅分子进化树的构建方法自夕岚一瞥的博客一、引言开始动笔写这篇短文之前,我问自己,为什么要写这样的文章?写这样的文章有实际的意义吗?我希望能够解决什么样的问题?带着这样的疑惑,我随手在丁香园(DXY)上以关键字“进化分析求助”进行了搜索,居然有289篇相关的帖子(2006年9月12日)。
而以关键字“进化分析”和“进化”为关键字搜索,分别找到2,733和7,724篇相关的帖子。
考虑到有些帖子的内容与分子进化无关,这里我保守的估计,大约有3,000~4,000篇帖子的内容,是关于分子进化的。
粗略地归纳一下,我大致将提出的问题分为下述的几类:1.涉及基本概念。
例如,“分子进化与生物进化是不是一个概念”,“关于微卫星进化模型有没有什么新的进展”以及“关于Kruglyak的模型有没有改进的出现”,等等。
2.关于构建进化树的方法的选择。
例如,“用boostrap NJ 得到XX图,请问该怎样理解?能否应用于文章?用boostrap test中的ME法得到的是XXX树,请问与上个树比,哪个更好”,等等。
3.关于软件的选择。
例如,“想做一个进化树,不知道什么软件能更好的使用且可以说明问题,并且有没有说明如何做”,“拿到了16sr RNA数据,打算做一个系统进化树分析,可是原来没有做过这方面的工作啊,都要什么软件”,“请问各位高手用clustalx做出来的进化树与phylip做的有什么区别”,“请问有做过进化树分析的朋友,能不能提供一下,做树的时候参数的设置,以及代表的意思。
还有各个分支等数值的意思,说明的问题等”,等等。
4.蛋白家族的分类问题。
例如,“搜集所有的关于一个特定domain 的序列,共141条,做的进化树不知具体怎么分析”,等等。
5.新基因功能的推断。
例如,“根据一个新基因A 氨基酸序列构建的系统发生树,这个进化树能否说明这个新基因A 和B同源,属于同一基因家族”,等等。
分子进化树算法

分子进化树算法分子进化树算法是一种用于研究生物进化关系的计算方法。
通过分析DNA、RNA或蛋白质序列的差异和相似性,可以构建出生物物种的进化树。
本文将介绍分子进化树算法的原理、应用和局限性。
一、原理分子进化树算法的原理基于遗传变异和进化。
生物个体的遗传信息通过DNA、RNA或蛋白质序列传递给后代,而在这个过程中会出现突变和重组等变异事件。
这些变异事件积累起来,形成了不同物种之间的差异。
分子进化树算法通过比较不同物种之间的序列差异和相似性,来推断它们之间的进化关系。
具体而言,分子进化树算法首先收集不同物种的DNA、RNA或蛋白质序列数据,然后利用计算方法计算它们之间的差异和相似性。
常用的计算方法包括序列比对、距离计算和进化模型推断。
通过这些计算,可得到一个差异矩阵或距离矩阵,它描述了不同物种之间的关系。
接下来,算法会利用这个矩阵来构建进化树,常见的构建方法有最小进化树、最大似然法和贝叶斯推断等。
二、应用分子进化树算法在生物学研究中有着广泛的应用。
首先,它可以帮助研究者揭示不同物种之间的进化关系。
通过构建进化树,可以了解物种的亲缘关系、起源时间和地理分布等信息。
这对于研究物种的进化历史和生态演化具有重要意义。
分子进化树算法可以用于物种鉴定和系统学研究。
在分类学中,鉴定物种是一个基础性任务。
通过分析物种的分子序列,可以判断它们是否属于同一物种,进而指导分类学的研究和实践。
分子进化树算法还可以用于研究基因功能和基因家族的进化。
通过比较不同物种中的基因序列,可以推断基因的功能和进化过程。
这对于深入理解基因的演化和功能具有重要意义。
三、局限性尽管分子进化树算法在生物学研究中有广泛应用,但也存在一些局限性。
首先,算法的结果受到数据质量和选择的进化模型的影响。
如果数据质量不高或选择的进化模型不合适,可能会导致结果的不准确性。
分子进化树算法无法解决样本不完整或有限的情况。
如果物种样本有限或者存在缺失数据,算法可能无法准确地构建进化树。
分子进化学中的进化树构建方法

分子进化学中的进化树构建方法随着科技的进步和生物技术的广泛应用,分子生物学的研究逐渐深入,成为生物学、生物技术和医药学等领域的重要研究方向。
而分子进化学作为分子生物学中的一个重要分支,研究物种间的分子差异和进化关系。
其中,构建进化树是分子进化学研究中的重要工作,下面我们来了解一下进化树构建的方法。
一、进化树的基本概念进化树是描述不同物种、不同基因或不同蛋白质之间进化关系的图形化表示。
在进化树中,每一个分支代表了一个物种、一个基因或一个蛋白质序列,分支的长度表示了物种、基因或序列的进化距离,而进化距离则是衡量不同物种或不同序列之间关系的基本参数。
而构建进化树的过程则是根据分子序列数据的重构得到物种或基因的进化树。
二、进化树的构建方法构建进化树有多种方法,主要有距离矩阵法、系统发育学法、最大似然法和贝叶斯法等。
下面我们逐一介绍这些方法的基本原理。
1.距离矩阵法距离矩阵法是最早采用的一种构建进化树的方法,它基于序列之间的距离矩阵计算和聚类方法来得到进化树。
该方法首先计算所有分子序列之间的距离(距离可由序列相似性计算得出),然后根据聚类方法构建进化树。
聚类方法包括单链接聚类、均链接聚类和最大链接聚类等。
距离矩阵法的优点是构建速度快、适用性广,但是对于高变异的序列来说,该方法可能会产生误导性的结果。
2.系统发育学法系统发育学法是基于系统学原理,采用系统发生学的理论和方法来构建进化树。
该方法主要是通过分子序列的相似性构建系统发育分析矩阵,然后利用不同的计算方法(如UPGMA、NJ和ML等)推断进化树。
系统发育学法的优点是能够更准确地反映分子序列的演化,并且可以通过不同的方法比较结果,但是该方法需要大量的计算资源和长时间的计算。
3.最大似然法最大似然法是一种统计学上的方法,通过最大化序列数据与观测数据的相似度,来推断出最可能的进化树。
该方法需要整合进化模型和数据,然后计算不同进化模型下数据的似然函数,最终选择似然度最大的进化树。
利用mega构建树原理

利用mega构建树原理
Mega构建树的原理主要基于系统发育树(又称分子进化树)的概念。
这是一种描述一群有机体发生或进化顺序的拓扑结构,用于在生物信息学中描述不同生物之间的相关关系。
拓扑结构将讨论范围内的事物之间的相互关系表示出来,将这些事物之间的关系通过图表示出来。
Mega软件可用于序列比对、进化树的推断、估计分子进化速度、验证进化假说等。
在构建系统发育树时,它采用了一系列的算法和模型,如邻接法(NJ)、最大似然法(ML)、最大简约法(MP)和贝叶斯法(Bayes)等。
这些方法和模型的选择取决于具体的数据和研究目标。
构建系统发育树的一般过程包括以下几个步骤:
1. 数据准备:收集需要研究的物种的基因或蛋白序列,并进行比对,以确保它们的同源性。
比对的结果可以保存为特定的格式,如FASTA。
2. 模型选择:根据数据的特性,选择一个合适的进化模型。
例如,对于DNA序列,可以选择GTR、TN93、HKY等模型;对于蛋白序列,可以选择JTT、WAG、LG等模型。
3. 树的构建:使用选择的模型和方法,构建系统发育树。
这个过程可能包括搜索最优的树结构、计算分支长度等。
4. 树的评估和优化:通过一些统计方法,如自展值(Bootstrap)等,对构建的树进行评估和优化,以提高其可靠性。
需要注意的是,构建系统发育树是一个复杂的过程,需要一定的专业知识和经验。
同时,由于生物进化的复杂性,构建的树可能并不完全准确,需要结合其他证据进行解释和验证。
建立进化树的方法

建立进化树的一般步骤:MEGA 的全称是Molecular Evolutionary Genetics Analysis 分子进化遗传分析。
MEGA 可用于序列比对、进化树的推断、估计分子进化速度、验证进化假说等。
MEGA 还可以通过网络(NCBI)进行序列的比对和数据的搜索。
打开软件选择Alignment ---- Alignment Explorer/CLUSTAL,出现一个对话框:根据提示内容,进行选择,在此我选择第一个“Create a new alignment”,出现:根据自己的序列是核酸还是氨基酸序列进行选择,在此我选择“Yes”,出现:Date --- Open --- Retrieve Sequences from File ,选择已在Clustal X中已对齐的格式文件[CLUSTAL文件(.aln)],如下图:选择之后,得到:双击文件名可以进行修改(某些Clustal X版本无法识别原FASTA文件名的,在这里就可以修改了,就像我用汉化版的Clustal X 1.81不可以识别某些序列文件名) ,修改后如下:右键菜单点击删除Clustal X中附带的“※”号行,修改文件名后可以保存“当前比对结果”,以便下次再用。
然后再补充一下,此软件整合了Clustal X程序,菜单Alignment中选择“Align by ClustalX”即可。
选择所要比对的序列,单击后出现下面这个对话框:选择默认设置,点击OK就进行比对了。
此后会出现一个过渡对话框,显示的是两两比对和多序列比对的过程:等待其运行完成后,可以保存,也可以直接删除,出现对话框:选择Yes,出现:输入一个名称,如SIV-N2,接下来几步类似,保存后点YES出现:当这个序列数据界面出来后,注意软件的主界面发生了一定的变化,多出了几个功能菜单:选择主界面中的Phylogeny菜单,Bootstrap Test of Phylogeny --- Neighbor-joining…Bootstrap选择1000次重复,模型选择核酸---p-distance。
利用MEGA4构建分子系统进化树
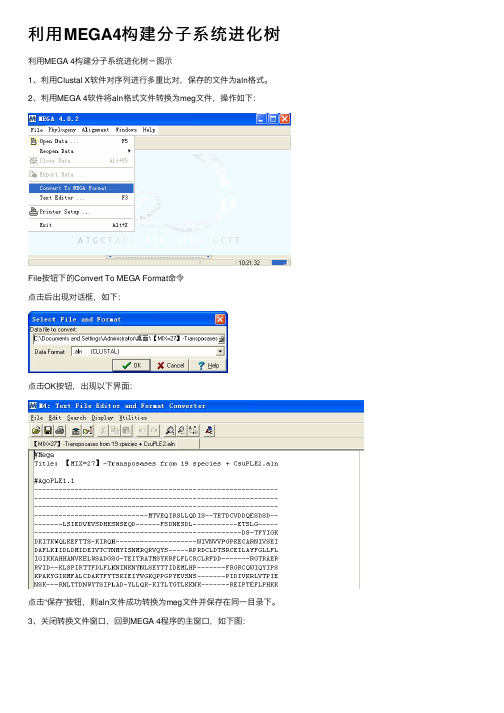
利⽤MEGA4构建分⼦系统进化树利⽤MEGA 4构建分⼦系统进化树-图⽰
1、利⽤Clustal X软件对序列进⾏多重⽐对,保存的⽂件为aln格式。
2、利⽤MEGA 4软件将aln格式⽂件转换为meg⽂件,操作如下:
File按钮下的Convert To MEGA Format命令
点击后出现对话框,如下:
点击OK按钮,出现以下界⾯:
点击“保存”按钮,则aln⽂件成功转换为meg⽂件并保存在同⼀⽬录下。
3、关闭转换⽂件窗⼝,回到MEGA 4程序的主窗⼝,如下图:
点击“Click me to activate a data file”按钮,选择之前转换好的meg⽂件并打开,如下图:
选择所输⼊的数据类型(核酸or蛋⽩),之后点击OK即可。
此时,在MEGA4主程序窗⼝的底部出现了我们所输⼊的⽂件名(如下图),之后就可以构建分⼦系统进化树了。
4、通常选择邻接法(neighbor-joining,NJ)构建分⼦系统进化树。
分子进化树构建的简要步骤(以蛋白为例)

分⼦进化树构建的简要步骤(以蛋⽩为例)PhyML利⽤氨基酸序列建树步骤(核酸建树也可以作为参考)前⾔:本⽂阅读对象适合建树新⼿,⽣物信息学⾼⼿请勿嘲笑,其中有什么错误还恳请指点。
为什么要建树及其你要解决什么问题这⾥不做讨论,只是⼀个纯粹的建树过程,前期的序列收集过程⾃⼰费⼼,根据⾃⼰的需要来做。
这⾥主要是最⼤似然法来建树,NJ法像mega这些软件中都有集成,最新的mega7也集成ML法,不过模型及各种参数不⼀定适合你,所以学习多种多种⽅法也是有⽤的,PhyML速度较慢,如果数列数量较多、步长检验次数多,等待时间会很长,有可能达到⼏⼗⼩时,也与电脑配置有关,⼀般时间都是以⼩时计数,所以要有⼼理准备,如果数据量⼤,推荐⽤RaxML或其他⽅法建树,它处理速度要⽐PhyML 快,不过RaxML是纯命令操作,对不熟悉命令及参数意义的⼈有⼀定难度,我只在linux 下操作过,在win下没有使⽤过。
本⽂是⽤氨基酸建树过程,如果你是⽤核酸序列建树,也可以参考这个过程,核酸替代模型请⽤jmodeltest或其他同功软件计算。
由于PhyML计算过程⽐较长,做⼀遍⽐较耗时,推荐你⽤其他软件⽤NJ法先⾏试验建树,看看你选择的序列是否有效及符合你的预期结果,调整好序列后再⽤PhyML跑⼀遍看结果是否符合⾃⼰的要求。
PhyML有线上版本,只需要提交序列⽐对结果,设置模型参数,留下邮箱等待就会给你返回结果,不过时间不可控,根据⾃⾝情况选择线上还是本地⾃⼰建树。
⽔平有限,如有错误遗漏恳请各位指点。
如果在⽂库不能下载,可以去⽹盘下载,见⽂末。
●建树过程:序列准备-模型选择-建树及树的验证。
●环境准备:电脑^-^Windows或者Linux都可以(没试过mac,如果是mac环境,请参考具体的操作⼿册)、ProtTest、PhyMl及序列⽐对的软件,线上或本地都可以。
1.序列准备:在⾃⼰熟悉的数据库中(我⾃⼰⽐较熟悉Ncbi)上做blast,选取跟要建树蛋⽩同源的各物种序列,下载到本地,整合到⼀个fasta⽂件中,注意修改物种名称,字数最好不要太长,序列⽐对后.phy格式⽂件对⽂件名长度有限制(这个可能跟软件有关系,只要⾃⼰知道是什么物种,不⾄于混淆就⾏),注意规范性,fasta⽂件中最好除了>头标,字母及下划线不要有其他不相关的字符,因为如果后⾯你要⽤软件分析.phy⽂件的时候这些软件对.phy的格式要求⽐较变态,有其他多余字符它都会报错的(你如果在dos 下⽤命令合并⽂件请注意⽂件中最后⼀⾏的字符,请删除)。
分子进化:系统树的构建_图文(精)

计:ˆ1 + v ˆ 2 = K 12 v ˆ1 + v ˆ3 = K 13 v ˆ2 + v ˆ3 = K 23 v 估值为 1 ( K 12 + K 13 − K 23 2 1 ˆ2 = ( K 12 + K 23 − K 13 v 2 1 ˆ3 = (K 13 + K 23 − K 12 v 2 实际序列并非具有相等的碱基频率,因而 Jukes-Cantor 距离不会使似然值最大,但它们的确为迭代法提供了很好的初始值。
Newton-Raphson 迭代法为找 -vi 到最大似然值的数值解提供了直接的方法,且从寻求 pi=1-e 的估值来看,这一方法在描述上是最为简单的。
表 5.7 给出了图 5.4 中人类(1、大猩猩(2、长臂猿(3线粒体序列收敛过程的例子。
三个序列间的平均碱基频率用作模型中的概率项πi。
ˆ1 = v 表 5.7 图 5.4 中人类、大猩猩和长臂猿线粒体序列非约束型最大似然树分枝长度的连续迭代 v2 v3 迭代 v1 初始值 0.0423 0.0174 0.2215 1 0.0420 0.0196 0.2230 2 0.0420 0.01990.2299 3 0.0420 0.0199 0.2299 标准差 0.0297 0.0218 0.0600 用几个序列作为树端来构建系统树时,可采用以上所述的一般方法。
先指定一种系统树,然后对来自该系统树似然函数的方程进行 Newton-Raphson 迭代来估计分枝长度。
在理论上,应研究所有可能的系统树来寻找具有最大似然值的系统树。
Fukami 和 Tateno(1989证实至多存在一组对于 L 给出平稳值的分枝长度,且这组分枝长度提供了所需的最大似然估计。
将这一方法应用于图 5.4 所列的 5 种线粒体序列,获得了图 5.16 所示的无根树状图。
117人类 0.015 0.030 1 0.000 黑猩猩大猩猩 0.000 0.051 0.045 2 3 0.138 猩猩长臂猿图 5.16 利用 Felsenstein 的 PHYLIP 软件构建的图 5.4 线粒体序列资料的最大似然树四.对系统树 Bootstrap 抽样在任一特定的树状拓扑结构内,已知最大似然值提供了分枝长度的一致估计值,这意味着随着资料量的增加,估计值逐渐接近真值。
分子进化的推导与系统发育树构建研究

分子进化的推导与系统发育树构建研究分子进化的推导和系统发育树构建研究是现代生物学领域中一项重要的研究课题。
它通过分析生物体内的分子遗传信息,来推导物种间的进化关系,并进一步构建系统发育树。
本文将介绍分子进化的推导过程以及系统发育树的构建方法。
在分子进化的推导过程中,研究者通常会选择一段具有较高变异性的DNA、RNA或蛋白质序列作为研究对象。
这些序列在不同物种之间的差异反映了它们的进化关系。
首先,研究者需要对所选序列进行测序,并通过生物信息学方法对序列进行比对和分析。
比对可以揭示序列中的共有特征与差异,而分析则可以计算序列之间的相似性和进化距离。
为了推导物种之间的进化关系,研究者可以利用不同的进化模型进行分析,例如Jukes-Cantor模型、Kimura两参数模型和最大似然法等。
这些模型基于一系列假设和统计方法,可以估计序列的演化速率和进化关系。
通过计算进化距离矩阵,研究者可以建立物种之间的相似性网络图,并利用聚类算法将物种进行分类和分组。
系统发育树是推导物种间进化关系的重要工具。
它是一种图形化的表示方式,用树状结构展示不同物种之间的演化关系。
构建系统发育树的方法有多种,例如最简原则、最大拟然法和贝叶斯推断等。
最简原则是一种直观且简单的构建方法,它假设进化关系中的分支数目最少。
最大拟然法则基于最大似然估计原理,通过计算相似性矩阵的概率分布来确定最优的拓扑结构。
贝叶斯推断则是一种统计推断方法,它通过考虑先验概率和后验概率来推测系统发育树的结构。
在构建系统发育树的过程中,研究者还需要对结果进行评估和验证。
常用的评估指标包括支持率和置信度。
支持率可以评估进化树的可靠性,它通过重复计算获得统计学意义上的支持度。
而置信度则通过随机重抽样验证树的一致性和稳定性。
综上所述,分子进化的推导和系统发育树构建是研究生物进化关系的重要方法。
通过分析分子遗传信息和构建系统发育树,我们可以更好地了解不同物种之间的进化历程和亲缘关系。
进化树构建方法-MEGA

利用MEGA 来构建进化树(molecular evolutionary genetics analysis 分子进化遗传分析)打开mega5,选择Align----edit/built alignment----create a new alignment—OK选择DNA/protein出现新的对话框Open------选择已经保存好的用clustalx 经过比对保存的以.aln格式的文件打开之后,出现下面的页面双击文件名可以进行修改的。
我的就是从这里开始修改把A,B,C 都去掉,只留号码就好右键菜单点击delete 删除带※的那一行。
得到下面的图示,点击保存,重新起名字。
之后点击此图内的Alignment 选择Align by clustalW即可。
默认设置即可,点击OK就进行比对了,此后会出现一个过渡对话框,显示的是两两比对和多序列比对的过程之后回到初始页面,就是这个页面之后点File---点开,把刚才保留的文件点开然后出现下面的页面多了几个内容,点击TA的那个框框。
之后出现这样的框框图片然后在主程序中选择phylogeny---construct/test neighbor-joining tree,然后出现下面的页面黄色框框处的的参数是可以改变的,该图为我已经改变好的,把Bootstrap 的值改为1000 Methods根据文献上的参考改为了Kimura2-parameter model.之后点击compute,就出现了,而且还带有必需的支持率即自展值,是用来检验你所计算的进化树分支可信度的。
简单地讲就是把序列的位点都重排,重排后的序列再用相同的办法构树,如果原来树的分枝在重排后构的树中也出现了,就给这个分枝打上一分,如果没出现就给0分,这样经过你给定的repetitions 次(至少1000次)重排构树打分后,每个分枝就都得出分值,计算机会给你换算成bootstrap值。
重排的序列有很多组合,值越小说明分枝的可信度越低,最好根据数据的情况选用不同的构树方法和模型。
