NEB保护碱基各种酶切位点保护碱基
酶切位点所加保护碱基
99 (2)
LITMUS 29
LITMUS 29
pNEB193
SpeI
SacI
SacI
MluI
2
SacI
BamHI
1
97 (2)
LITMUS 29
HindIII
BglII
3
100 (2)
LITMUS 29
NsiI
BsiWI
2
100 (2)
LITMUS 29
BssHII
BspEI
2
1
100 (1)
8 (2)
LITMUS 39
LITMUS 38
BsrGI
BsrGI
BsrGI
2
1
99 (2)
88 (2)
8
10
12
0
10
10
0
25
50
Not I
TTGCGGCCGCAA
ATTTGCGGCCGCTTTA
AAATATGCGGCCGCTATAAA
ATAAGAATGCGGCCGCTAAACTAT
AAGGAAAAAAGCGGCCGCAAAAGGAAAA
12
16
20
24
28
0
10
10
25
25
0
10
10
90
>90
Nsi I
Enzyme
Base pairs
from End
%Cleavage
Efficiency
Vector
Initial Cut
AatII
3
2
1
88 (2)
100 (2)
95 (2)
NEB保护碱基-各种酶切位点保护碱基
0
0
0
0
0
0
0
0
75
>90
75
>90
Nhe I
GGCTAGCC CGGCTAGCCG CTAGCTAGCTAG
0
0
10
25
10
50
Not I
TTGCGGCCGCAA
0
0
ATTTGCGGCCGCTTTA
10
10
AAATATGCGGCCGCTATAAA
10
10
ATAAGAATGCGGCCGCTAAACTAT
酶 Acc I Afl III Asc I Ava I BamH I Bgl II BssH II BstE II BstX I Cla I
EcoR I
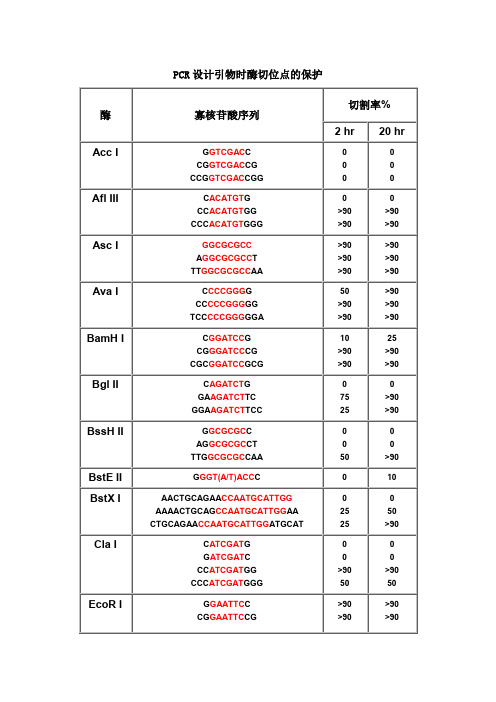
PCR 设计引物时酶切位点的保护
寡核苷酸序列
GGTCGACC CGGTCGACCG CCGGTCGACCGG
CACATGTG CCACATGTGG CCCACATGTGGG
0
0
0
25
0
50
75
>90
Pst I
GCTGCAGC
0
0
TGCACTGCAGTGCA
10
10
AACTGCAGAACCAATGCATTGG
>90
>90
AAAACTGCAGCCAATGCATTGGAA
>90
>90
CTGCAGAACCAATGCATTGGATGCAT
0
0
Pvu I
CCGATCGG ATCGATCGAT TCGCGATCGCGA
25
90
AAGGAAAAAAGCGGCCGCAAAAGGAAAA
酶切位点所加保护碱基

BamHI
1
97 (2)
LITMUS 29
HindIII
BglII
3
100 (2)
LITMUS 29
NsiI
BsiWI
2
100 (2)
LITMUS 29
BssHII
BspEI
2
1
100 (1)
8 (2)
LITMUS 39
LITMUS 38
BsrGI
BsrGI
BsrGI
2
1
99 (2)
88 (2)
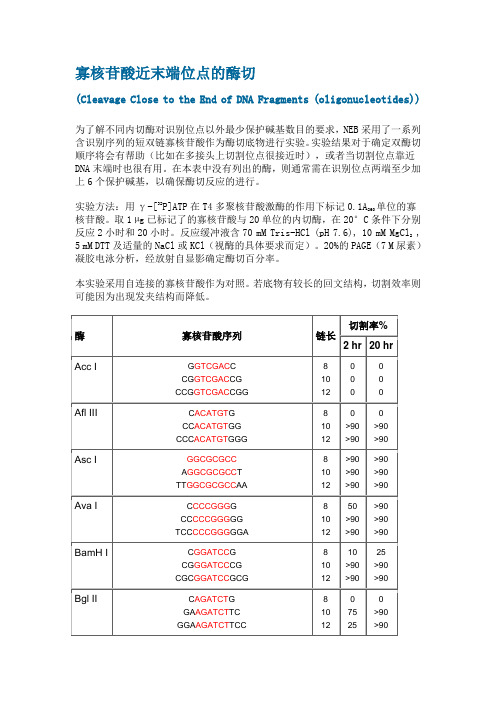
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。取1 µg已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。反应缓冲液含70 mMTris-HCl (pH 7.6),10 mMMgCl2,5 mMDTT及适量的NaCl或KCl(视酶的具体要求而定)。20%的PAGE(7 M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
>90
50
0
0
>90
50
EcoR I
GGAATTCC
CGGAATTCCG
CCGGAATTCCGG
8
10
12
>90
>90
>90
>90
>90
>90
Hae III
GGGGCCCC
AGCGGCCGCT
TTGCGGCCGCAA
8
10
12
>90
>90
>90
>90
>90
>90
Hind III
限制性内切酶酶切位点及保护碱基

寡核苷酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments (oligonucleotides))为什么要添加保护碱基?在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
该如何添加保护碱基?添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
单位的寡实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260核苷酸。
取1 µg已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。
反应缓冲液含70 mM Tris-HCl (pH 7.6), 10 mM MgCl,25 mM DTT及适量的NaCl或KCl(视酶的具体要求而定)。
20%的PAGE(7 M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核苷酸作为对照。
各种酶切位点的保护碱基引物设计必看

各种酶切位点的保护碱基引物设计必看酶切位点是指特定的序列,酶可以识别并在该位置切割DNA分子。
这些位点的特异性使得酶在分子生物学中广泛应用于DNA片段的定位和切割。
然而,在一些实验中,我们可能需要保护酶切位点周围的碱基,以免酶切,并且只在特定的位置引导酶切。
因此,保护碱基引物的设计对于实验的成功非常重要。
以下是保护碱基引物设计的一些建议。
首先,保护碱基引物的设计需要考虑引物的长度。
引物的长度通常为18到30个碱基,具体的长度需要根据实验的需求和酶切位点周围的序列特征来确定。
引物的长度应足够长,以确保引物和靶序列的特异性,但不应过长,以免引物形成二级结构或与非特异性位点结合。
其次,保护碱基引物的设计需要考虑引物的碱基组成。
在设计引物时,建议尽量避免引物中出现酶切位点周围的碱基序列,以防止酶的误切。
例如,如果我们希望保护酶切位点周围的AATTC序列,可以设计一个引物,其中没有AATTC序列。
同时,引物的碱基组成应尽量避免多聚核苷酸或含有GC碱基的片段,以防止引物之间的结合或引物与非特异靶序列的结合。
此外,保护碱基引物的设计需要考虑引物的特异性。
在设计引物时,建议使用特异性的引物序列,以确保引物只与目标酶切位点结合。
可以通过使用生物信息学工具,如BLAST,来验证引物的特异性。
引物的特异性还可以通过调整引物的长度和碱基组成来进一步提高。
最后,保护碱基引物的设计需要考虑引物的热力学性质。
引物的热力学性质包括引物的熔解温度(Tm值)和引物之间的配对。
引物的Tm值与引物的碱基组成、长度和引物与靶序列之间的碱基配对相关。
可以使用在线工具,如NEB的Tm计算器,来计算引物的Tm值,并对不同的引物进行比较。
此外,引物之间的配对可以通过设计引物的末端序列来调整,例如末端的碱基配对或非配对等。
总结起来,保护碱基引物的设计需要考虑引物的长度、碱基组成、特异性和热力学性质。
通过合理设计引物,可以保护酶切位点周围的碱基,并在特定位置引导酶切,为实验的成功提供有力的保障。
各种酶切位点的保护碱基引物设计必看)

各种酶切位点的保护碱基酶不同,所需要的酶切位点的保护碱基的数量也不同。
一般情况下,在酶切位点以外多出3个碱基即可满足几乎所有限制酶的酶切要求。
在资料上查不到的,我们一般都随便加3个碱基做保护。
寡核苷酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments(oligonucleotides)为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。
取1 μg 已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。
反应缓冲液含70 mM Tris-HCl (pH 7.6), 10 mM MgCl2 , 5 mM DTT及适量的NaCl或KCl(视酶的具体要求而定)。
20%的PAGE(7 M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核苷酸作为对照。
若底物有较长的回文结构,切割效率则可能因为出现发夹结构而降低。
2.双酶切的问题参看目录,选择共同的buffer。
其实,双酶切选哪种buffer是实验的结果,takara公司从1979年开始生产限制酶以来,做了大量的基础实验,也积累了很多经验,目录中所推荐的双酶切buffer完全是依据具体实验结果得到的。
有共同buffer的,通常按照常规的酶切体系,在37℃进行同步酶切。
但BamH I在37℃下有时表现出star活性,常用30℃单切。
两个酶切位点相邻或没有共同buffer的,通常单切,即先做一种酶切,乙醇沉淀,再做另一种酶切。
酶切位点所加保护碱基

28
30
32
0
10
10
0
50
75
Sca I
GAGTACTC
AAAAGTACTTTT
8
12
10
75
25
75
Sma I
CCCGGG
CCCCGGGG
CCCCCGGGGG
TCCCCCGGGGGA
6
8
10
12
0
0
10
>90
10
10
50
>90
Spe I
CCGATCGG
ATCGATCGAT
TCGCGATCGCGA
8
10
12
0
10
0
0
25
10
Sac I
CGAGCTCG
8
10
10
Sac II
GCCGCGGC
TCCCCGCGGGGA
8
12
0
50
0
>90
Sal I
GTCGACGTCAAAAGGCCATAGCGGCCGC
GCGTCGACGTCTTGGCCATAGCGGCCGCGG
CCATATGG
CCCATATGGG
CGCCATATGGCG
GGGTTTCATATGAAACCC
GGAATTCCATATGGAATTCC
GGGAATTCCATATGGAATTCCC
8
10
12
18
20
22
0
0
0
0
75
75
0
0
0
0
限制性内切酶酶切位点及保护碱基

寡核苷酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments (oligonucleotides))为什么要添加保护碱基?在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
该如何添加保护碱基?添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
单位的寡实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260核苷酸。
取1 µg已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。
反应缓冲液含70 mM Tris-HCl (pH 7.6), 10 mM MgCl,25 mM DTT及适量的NaCl或KCl(视酶的具体要求而定)。
20%的PAGE(7 M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核苷酸作为对照。
常用酶切位点序列和保护碱基

常用酶切位点序列和保护碱基引言在分子生物学和遗传工程领域,酶切位点序列和保护碱基是非常重要的概念。
酶切位点序列指的是DNA或RNA上特定的核苷酸序列,这些序列可以被特定的酶识别并切割。
保护碱基则是指在实验过程中采取措施来保护DNA或RNA上特定的核苷酸,使其不被酶切割。
本文将对常用的酶切位点序列和保护碱基进行详细介绍,包括其定义、常见的酶切位点序列、如何选择合适的保护碱基等内容。
酶切位点序列定义酶切位点序列是指DNA或RNA分子上具有一定规律性、可以被特定的限制性内切酶识别并结合从而发挥催化作用的核苷酸序列。
这些限制性内切酶通常能够识别4-8个核苷酸,并在识别到相应的位点后将DNA或RNA分子切割成片段。
常见的酶切位点序列1.EcoRI: 5’-GAATTC-3’,3’-CTTAAG-5’2.HindIII: 5’-AAGCTT-3’,3’-TTCGAA-5’3.BamHI: 5’-GGATCC-3’,3’-CCTAGG-5’4.XhoI: 5’-CTCGAG-3’,3’-GAGCTC-5’5.NotI: 5’-GCGGCCGC-3’,3’-CGCCGGCG-5’这些酶切位点序列是常用的限制性内切酶的识别序列,它们在分子生物学实验中被广泛应用。
通过将DNA或RNA与特定的限制性内切酶一起反应,可以实现DNA或RNA的特定部位切割。
保护碱基定义保护碱基是指在实验过程中采取措施来保护DNA或RNA上特定的核苷酸,使其不被酶切割。
这种保护通常通过对特定的碱基进行修饰或使用化学试剂来实现。
如何选择合适的保护碱基选择合适的保护碱基需要考虑以下几个因素: 1. 酶切位点序列:首先要了解所使用的限制性内切酶的酶切位点序列,以确定需要保护的碱基。
2. 保护方法:根据实验需求和实验条件选择合适的保护方法。
常见的保护方法包括使用化学修饰剂修饰碱基、使用特殊的核苷酸引物或引入特定的修饰基团等。
3. 保护效果:选择的保护碱基应能够有效地阻止限制性内切酶与目标位点结合并发挥催化作用。
各种酶切位点的保护碱基

各种酶切位点的保护碱基酶切位点是指酶在DNA或RNA分子中特定的位置识别并切割的区域。
在分子生物学研究中,酶切位点的保护碱基是进行引物设计的重要参考依据之一、本文将介绍各种酶切位点的保护碱基以及在引物设计中的应用。
1.核酸酶A切割位点保护碱基核酸酶A(RNase A)是一种特定的核酸酶,能够将单链RNA切割为5'-磷酸核酸和3'-核磷酸。
核酸酶A识别和切割位点的保护碱基主要为对尿嘧啶核苷酸(Uridine,U)和鸟嘌呤核苷酸(Adenine,A)。
具体来说,核酸酶A主要作用于RNA链上U和A的周围碱基,特别是位于U或A的下一个碱基。
在引物设计中,需要考虑核酸酶A切割位点的保护碱基,以避免产生无法预测的酶切割产物。
特别是在设计RNA引物时,需要避免在酶切位点附近出现U和A的保护碱基。
2.核酸酶T1切割位点保护碱基核酸酶T1(RNase T1)是一种特定的核酸酶,能够识别并切割由磷酸鸟苷(Guanosine,G)形成的RNA链。
核酸酶T1在RNA链上识别和切割位点的保护碱基为G。
具体来说,核酸酶T1主要作用于G的下一个碱基。
在引物设计中,需要考虑核酸酶T1切割位点的保护碱基,以避免产生无法预测的酶切割产物。
特别是在设计RNA引物时,需要避免在酶切位点附近出现G的保护碱基。
3.限制性内切酶切割位点保护碱基限制性内切酶是一类广泛应用于DNA分子生物学研究的酶,其识别和切割DNA分子中的特定序列。
限制性内切酶识别和切割位点的保护碱基由酶自身的特异性决定。
每一种限制性内切酶都有其特定的酶切位点保护碱基要求。
一般来说,酶切位点的保护碱基主要存在于酶切位点的上下游碱基中。
在引物设计中,需要考虑限制性内切酶切割位点的保护碱基,以避免引物和酶切位点之间存在相互作用而导致切割不完全或无法切割的情况。
总而言之,在引物设计中,需要考虑各种酶切位点的保护碱基,以提高引物的特异性和稳定性。
根据不同酶的切割特点和要求,设计合适的引物序列可以避免酶切位点的保护碱基产生干扰,保证实验结果的准确性。
限制性内切酶酶切位点保护碱基.doc
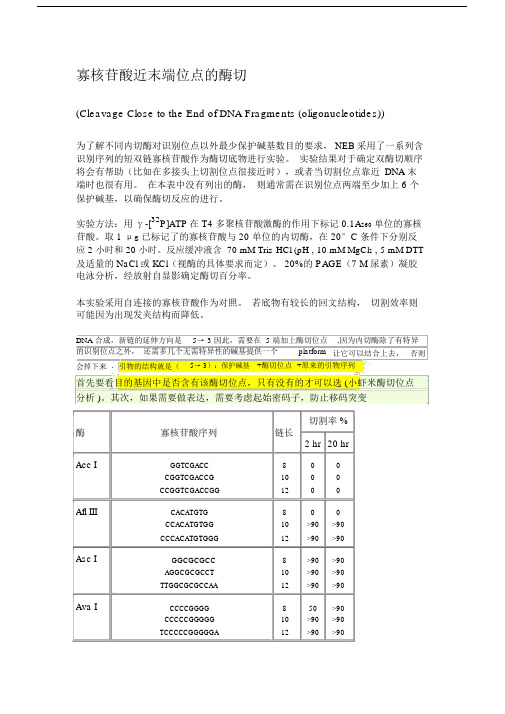
寡核苷酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments (oligonucleotides))为了解不同内切酶对识别位点以外最少保护碱基数目的要求, NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA 末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上 6 个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在 T4 多聚核苷酸激酶的作用下标记 0.1A260单位的寡核苷酸。
取 1 μg已标记了的寡核苷酸与 20 单位的内切酶,在 20°C 条件下分别反应 2 小时和 20 小时。
反应缓冲液含 70 mM Tris-HCl (pH , 10 mM MgCl2 , 5 mM DTT 及适量的 NaCl 或 KCl(视酶的具体要求而定)。
20%的 PAGE(7 M 尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核苷酸作为对照。
若底物有较长的回文结构,切割效率则可能因为出现发夹结构而降低。
DNA 合成,新链的延伸方向是5→ 3 因此,需要在 5 端加上酶切位点的识别位点之外,还需多几个无需特异性的碱基提供一个platform,因为内切酶除了有特异让它可以结合上去,否则会掉下来. 引物的结构就是(5→ 3):保护碱基+酶切位点+原来的引物序列首先要看目的基因中是否含有该酶切位点,只有没有的才可以选 (小虾米酶切位点分析 )。
其次,如果需要做表达,需要考虑起始密码子,防止移码突变切割率 %酶寡核苷酸序列链长2 hr 20 hrAcc I GGTCGACC 8 0 0CGGTCGACCG 10 0 0CCGGTCGACCGG 12 0 0Afl III CACATGTG 8 0 0CCACATGTGG 10 >90 >90CCCACATGTGGG 12 >90 >90Asc I GGCGCGCC 8 >90 >90AGGCGCGCCT 10 >90 >90TTGGCGCGCCAA 12 >90 >90Ava I CCCCGGGG 8 50 >90CCCCCGGGGG 10 >90 >90TCCCCCGGGGGA 12 >90 >90BamH IBgl II BssH IIBstE II BstX ICla I EcoR I Hae III Hind III Kpn I Mlu I Nco ICGGATCCG 8 10 25CGGGATCCCG 10 >90 >90CGCGGATCCGCG 12 >90 >90CAGATCTG 8 0 0GAAGATCTTC 10 75 >90GGAAGATCTTCC 12 25 >90GGCGCGC 8 0 0AGGCGCGCCT 10 0 0TTGGCGCGCCAA 12 50 >90GGGT(A/T)ACCC 9 0 10AACTGCAGAACCAATGCATTGG 22 0 0 AAAACTGCAGCCAATGCATTGGAA 24 25 50 CTGCAGAACCAATGCATTGGATGCAT 27 25 >90CATCGATG 8 0 0GATCGATC 8 0 0CCATCGATGG 10 >90 >90CCCATCGATGGG 12 50 50GGAATTCC 8 >90 >90CGGAATTCCG 10 >90 >90CCGGAATTCCGG 12 >90 >90GGGGCCCC 8 >90 >90AGCGGCCGCT 10 >90 >90TTGCGGCCGCAA 12 >90 >90CAAGCTTG 8 0 0CCAAGCTTGG 10 0 0CCCAAGCTTGGG 12 10 75GGGTACCC 8 0 0GGGGTACCCC 10 >90 >90CGGGGTACCCCG 12 >90 >90GACGCGTC 8 0 0CGACGCGTCG 10 25 50CCCATGGG 8 0 0 CATGCCATGGCATG 14 50 75Nde INhe I Not INsi I Pac I Pme I Pst I Pvu ISac I Sac IISal ICCATATGG 8 0 0CCCATATGGG 10 0 0CGCCATATGGCG 12 0 0 GGGTTTCATATGAAACCC 18 0 0GGAATTCCATATGGAATTCC 20 75 >90GGGAATTCCATATGGAATTCCC 22 75 >90GGCTAGCC 8 0 0CGGCTAGCCG 10 10 25CTAGCTAGCTAG 12 10 50TTGCGGCCGCAA 12 0 0ATTTGCGGCCGCTTTA 16 10 10 AAATATGCGGCCGCTATAAA 20 10 10 ATAAGAATGCGGCCGCTAAACTAT 24 25 90 AAGGAAAAAAGCGGCCGCAAAAGGAAAA 28 25 >90TGCATGCATGCA 12 10 >90 CCAATGCATTGGTTCTGCAGTT 22 >90 >90TTAATTAA 8 0 0GTTAATTAAC 10 0 25CCTTAATTAAGG 12 0 >90GTTTAAAC 8 0 0GGTTTAAACC 10 0 25GGGTTTAAACCC 12 0 50 AGCTTTGTTTAAACGGCGCGCCGG 24 75 >90GCTGCAGC 8 0 0 TGCACTGCAGTGCA 14 10 10 AACTGCAGAACCAATGCATTGG 22 >90 >90AAAACTGCAGCCAATGCATTGGAA 24 >90 >90 CTGCAGAACCAATGCATTGGATGCAT 26 0 0CCGATCGG 8 0 0ATCGATCGAT 10 10 25TCGCGATCGCGA 12 0 10CGAGCTCG 8 10 10GCCGCGG 8 0 0 TCCCCGCGGGGA 12 50 >90GTCGACGTCAAAAGGCCATAGCGGCCGC 28 0 0 GCGTCGACGTCTTGGCCATAGCGGCCGCGG 30 10 50 ACGCGTCGACGTCGGCCATAGCGGCCGCGGAA 32 10 75Sca I Sma ISpe ISph I Stu I Xba I Xho I Xma IGAGTACTC 8 10 25 AAAAGTACTTTT 12 75 75CCCGGG 6 0 10 CCCCGGGG 8 0 10 CCCCCGGGGG 10 10 50 TCCCCCGGGGGA 12 >90 >90GACTAGTC 8 10 >90 GGACTAGTCC 10 10 >90 CGGACTAGTCCG 12 0 50 CTAGACTAGTCTAG 14 0 50GGCATGCC 8 0 0 CATGCATGCATG 12 0 25 ACATGCATGCATGT 14 10 50AAGGCCTT 8 >90 >90 GAAGGCCTTC 10 >90 >90 AAAAGGCCTTTT 12 >90 >90CTCTAGAG 8 0 0 GCTCTAGAGC 10 >90 >90 TGCTCTAGAGCA 12 75 >90 CTAGTCTAGACTAG 14 75 >90CCTCGAGG 8 0 0 CCCTCGAGGG 10 10 25 CCGCTCGAGCGG 12 10 75CCCCGGGG 8 0 0 CCCCCGGGGG 10 25 75 CCCCCCGGGGGG 12 50 >90 TCCCCCCGGGGGGA 14 >90 >90DNA 合成,新链的延伸方向是5→ 3 因此,需要在 5 端加上酶切位点,因为内切酶除了有特异的识别位点之外,还需多几个无需特异性的碱基提供一个platform 让它可以结合上去,否则会掉下来 . 引物的结构就是(5→ 3):保护碱基 +酶切位点 +原来的引物序列。
限制性内切酶酶切位点及保护碱基

寡核苷酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments (oligonucleotides))为什么要添加保护碱基?在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
该如何添加保护碱基?添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
单位的寡实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260核苷酸。
取1 µg已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。
反应缓冲液含70 mM Tris-HCl (pH 7.6), 10 mM MgCl,25 mM DTT及适量的NaCl或KCl(视酶的具体要求而定)。
20%的PAGE(7 M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核苷酸作为对照。
各种酶切位点的保护碱基

各种酶切位点的保护碱基酶不同,所需要的酶切位点的保护碱基的数量也不同。
一般情况下,在酶切位点以外多出3个碱基即可满足几乎所有限制酶的酶切要求。
在资料上查不到的,我们一般都随便加3个碱基做保护。
寡核昔酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments(oligonucleotides)为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核昔酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用Y[32P]ATP在T4多聚核甘酸激酶的作用下标记0.1 A 260单位的寡核昔酸。
取1 Q已标记了的寡核昔酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。
反应缓冲液含70 mM Tris-HCI (pH 7.6) , 10 mM MgCI 2,5 mM DTT S适量的NaCI 或KCI (视酶的具体要求而定)。
20%的PAGE(7M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核昔酸作为对照。
若底物有较长的回文结构,切割效率则可能因为出现发夹结构而降低。
TCC CCCGGG GGA 12 >90 >90参看目录,选择共同的buffero其实,双酶切选哪种buffer是实验的结果,takara公司从1979年开始生产限制酶以来,做了大量的基础实验,也积累了很多经验,目录中所推荐的双酶切buffer完全是依据具体实验结果得到的。
有共同buffer的,通常按照常规的酶切体系,在37 C进行同步酶切。
但BamH I在37 C下有时表现出star活性,常用30 C单切。
切。
3. 酶切底物DNA ,切不开1 )底物DNA±没有相应的限制酶识别位点,或酶切位点被甲基化。
限制性内切酶酶切位点及保护碱基(DOC)

寡核苷酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments (oligonucleotides))为什么要添加保护碱基?在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
该如何添加保护碱基?添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260单位的寡核苷酸。
取1 µg已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。
反应缓冲液含70 mM Tris-HCl (pH 7.6), 10 mM MgCl2, 5 mM DTT及适量的NaCl或KCl(视酶的具体要求而定)。
20%的PAGE(7 M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核苷酸作为对照。
【免费下载】NEB保护碱基 各种酶切位点保护碱基

PCR设计引物时酶切位点的保护切割率%酶寡核苷酸序列2 hr20 hrAcc I G GTCGAC CCG GTCGAC CGCCG GTCGAC CGG 0Afl III C ACATGT GCC ACATGT GGCCC ACATGT GGG>90>90>90>90Asc I GGCGCGCCA GGCGCGCC TTT GGCGCGCC AA >90>90>90>90>90>90Ava I C CCCGGG GCC CCCGGG GGTCC CCCGGG GGA50>90>90>90>90>90BamH I C GGATCC GCG GGATCC CGCGC GGATCC GCG10>90>9025>90>90Bgl II C AGATCT GGA AGATCT TCGGA AGATCT TCC7525>90>90BssH II G GCGCGC CAG GCGCGC CTTTG GCGCGC CAA50>90BstE II G GGT(A/T)ACC C010BstX I AACTGCAGAA CCAATGCATTGGAAAACTGCAG CCAATGCATTGG AACTGCAGAA CCAATGCATTGG ATGCAT252550>90Cla I C ATCGAT GG ATCGAT CCC ATCGAT GGCCC ATCGAT GGG>9050>9050EcoR I G GAATTC CCG GAATTC CG >90>90>90>90CCG GAATTC CGG>90>90Hae III GG GGCC CCAGC GGCC GCTTTGC GGCC GCAA >90>90>90>90>90>90Hind III C AAGCTT GCC AAGCTT GGCCC AAGCTT GGG1075Kpn I G GGTACC CGG GGTACC CCCGG GGTACC CCG>90>90>90>90Mlu I G ACGCGT CCG ACGCGT CG2550Nco I C CCATGG GCATG CCATGG CATG5075Nde I C CATATG GCC CATATG GGCGC CATATG GCGGGGTTT CATATG AAACCCGGAATTC CATATG GAATTCCGGGAATTC CATATG GAATTCCC7575>90>90Nhe I G GCTAGC CCG GCTAGC CGCTA GCTAGC TAG10102550Not I TT GCGGCCGC AAATTT GCGGCCGC TTTAAAATAT GCGGCCGC TATAAAATAAGAAT GCGGCCGC TAAACTATAAGGAAAAAA GCGGCCGC AAAAGGAAAA10102525101090>90Nsi I TGC ATGCAT GCACCA ATGCAT TGGTTCTGCAGTT10>90>90>90Pac I TTAATTAAG TTAATTAA CCC TTAATTAA GG 025>90Pme I GTTTAAACG GTTTAAAC CGG GTTTAAAC CC 02550AGCTTT GTTTAAAC GGCGCGCCGG75>90Pst I G CTGCAG CTGCA CTGCAG TGCAAA CTGCAG AACCAATGCATTGGAAAA CTGCAG CCAATGCATTGGAACTGCAG AACCAATGCATTGGATGCAT10>90>9010>90>90Pvu I C CGATCG GAT CGATCG ATTCG CGATCG CGA102510Sac I C GAGCTC G1010Sac II G CCGCGG CTCC CCGCGG GGA50>90Sal I GTCGAC GTCAAAAGGCCATAGCGGCCGC GC GTCGAC GTCTTGGCCATAGCGGCCGCGGACGC GTCGAC GTCGGCCATAGCGGCCGCGGAA10105075Sca I G AGTACT CAAA AGTACT TTT 10752575Sma I CCCGGGC CCCGGG GCC CCCGGG GGTCC CCCGGG GGA10>90101050>90Spe I G ACTAGT CGG ACTAGT CCCGG ACTAGT CCGCTAG ACTAGT CTAG 1010>90>905050Sph I G GCATGC CCAT GCATGC ATGACAT GCATGC ATGT102550Stu I A AGGCCT TGA AGGCCT TCAAA AGGCCT TTT >90>90>90>90>90>90Xba I C TCTAGA GGC TCTAGA GCTGC TCTAGA GCACTAG TCTAGA CTAG>907575>90>90>90Xho I C CTCGAG GCC CTCGAG GGCCG CTCGAG CGG10102575Xma I C CCCGGG GCC CCCGGG GGCCC CCCGGG GGGTCCC CCCGGG GGGA2550>9075>90>90注释:1.如果要加在序列的5’端,就在酶切位点识别碱基序列(红色)的5’端加上相应的碱基(黑色),相同如果要在3’端加保护碱基,就在酶切位点识别碱基序列(红色)的3’端加上相应的碱基(黑色)。
常见限制性内切酶识别序列(酶切位点),保护碱基,缓冲液

常见限制性内切酶识别序列(酶切位点),保护碱基,缓冲液常见限制性内切酶识别序列(酶切位点),保护碱基,缓冲液在分子克隆实验中,限制性内切酶是必不可少的工具酶。
无论是构建克隆载体还是表达载体,要根据载体选择合适的内切酶(当然,使用T载就不必考虑了)。
先将引物设计好,然后添加酶切识别序列到引物5’端。
常用的内切酶比如BamHI、EcoRI、HindIII、NdeI、XhoI等可能你都已经记住了它们的识别序列,不过为了保险起见,还是得查证一下。
下面,我就总结了一些常用的内切酶的识别序列,仅供各位参考。
下面这些内切酶都属于II型内切酶。
先介绍一下什么是II型内切酶吧。
The Type II restriction systems typically contain individual restriction enzymes and modification enzymes encoded by separate genes. The Type II restriction enzymes typically recognize specific DNA sequences and cleave at constant positions at or close to that sequence to produce 5-phosphates and 3-hydroxyls. Usually they require Mg 2+ ions as a cofactor, although some have more exotic requirements. The methyltransferases usually recognize the same sequence although some are more promiscuous. Three types of DNA methyltransferases have been found as part of Type II R-M systems forming either C5-methylcytosine, N4-methylcytosine or N6-methyladenine.ApaI (类型:Type II restriction enzyme )识别序列:5'GGGCC^C 3'BamHI(类型:Type II restriction enzyme )识别序列:5' G^GATCC 3'BglII (类型:Type II restriction enzyme )识别序列:5'A^GATCT 3'EcoRI (类型:Type II restriction enzyme )识别序列:5' G^AATTC 3'HindIII (类型:Type II restriction enzyme )识别序列:5' A^AGCTT 3'KpnI (类型:Type II restriction enzyme )识别序列:5' GGTAC^C 3'NcoI (类型:Type II restriction enzyme )识别序列:5' C^CATGG 3'NdeI (类型:Type II restriction enzyme )识别序列:5' CA^TATG 3'NheI (类型:Type II restriction enzyme )识别序列:5' G^CTAGC 3'NotI (类型:Type II restriction enzyme )识别序列:5' GC^GGCCGC 3'SacI (类型:Type II restriction enzyme )识别序列:5' GAGCT^C 3'SalI (类型:Type II restriction enzyme )识别序列:5' G^TCGAC 3'SphI (类型:Type II restriction enzyme )识别序列:5' GCATG^C 3'XbaI (类型:Type II restriction enzyme )识别序列:5' T^CTAGA 3'XhoI (类型:Type II restriction enzyme )识别序列:5' C^TCGAG 3'当然,上面总结的这些肯定不全,要查找更多内切酶的识别序列,你还可以选择下面几种方法:1. 查你所使用的内切酶的公司的目录或者网站;NEB网站上提供的识别序列图表下载2. 用软件如:Primer Premier5.0或Bioedit等,这些软件均提供了内切酶识别序列的信息;3. 推荐到NEB的REBASE数据库去查(网址:/rebase/rebase.html)当你设计好引物,添加上了内切酶识别序列,下一步或许是添加保护碱基了,可以参考:/html/86.htmlNEB公司网站提供的保护碱基参考表下载/user1/2081/archives/2009/230768.shtml NEB公司网站上关于设计PCR引物保护碱基的参考/userfiles/file/protect.doc双酶切buffer的选择MBI/doubledigest/index.html罗氏http://www.roche-applied-/benchmate/refinder.htmNEB/nebecomm/DoubleDigestCalculator.as pPromega/guides/re_guide/research.asp?se arch=bufferTakara http://catalog.takara-bio.co.jp/en/product/basic_info.asp?unitid=U100005593 这里再给大家推荐一种新的不需要连接反应的分子克隆方法,优点包括:①设计引物不必考虑选择什么酶切位点;②不必考虑保护碱基的问题;③不必每次都选择合适的酶来酶切质粒制备载体;④而且不需要DNA连接酶;⑤假阳性几率低(因为没有连接反应这一步,载体自连的问题没有了)。
限制性内切酶酶切位点保护碱基
12 0
0
16 10 10
20 10 10
24 25 90
28 25 >90
Nsi I
TGCATGCATGCA CCAATGCATTGGTTCTGCAGTT
12 10 >90 22 >90 >90
Pac I
TTAATTAA GTTAATTAAC CCTTAATTAAGG
8
0
0
10 0
25
12 0 >90
8
0
0
10 0
0
12 10 75
8
0
0
10 >90 >90
12 >90 >90
8
0
0
10 25 50
8
0
0
14 50 75
Nde I
CCATATGG CCCATATGGG CGCCATATGGCG GGGTTTCATATGAAACCC GGAATTCCATATGGAATTCC GGGAATTCCATATGGAATTCCC
GTCGACGTCAAAAGGCCATAGCGGCCGC
28 0
0
GCGTCGACGTCTTGGCCATAGCGGCCGCGG 30 10 50
ACGCGTCGACGTCGGCCATAGCGGCCGCGGAA 32 10 75
Sca I
GAGTACTC AAAAGTACTTTT
8 10 25 12 75 75
GGCGCGCC AGGCGCGCCT TTGGCGCGCCAA
CCCCGGGG CCCCCGGGGG TCCCCCGGGGGA
切割率% 链长
2 hr 20 hr
8
限制性内切酶酶切位点及保护碱基

寡核苷酸近末端位点的酶切(Cleavage Close to the End of DNA Fragments (oligonucleotides))为什么要添加保护碱基?在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为的在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
该如何添加保护碱基?添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
单位的寡实验方法:用γ-[32P]ATP在T4多聚核苷酸激酶的作用下标记0.1A260核苷酸。
取1 µg已标记了的寡核苷酸与20单位的内切酶,在20°C条件下分别反应2小时和20小时。
反应缓冲液含70 mM Tris-HCl (pH 7.6), 10 mM MgCl,25 mM DTT及适量的NaCl或KCl(视酶的具体要求而定)。
20%的PAGE(7 M尿素)凝胶电泳分析,经放射自显影确定酶切百分率。
本实验采用自连接的寡核苷酸作为对照。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
>90
>90
>90
0
0
0
25
0
>90
Pme I
GTTTAAAC GGTTTAAACC GGGTTTAAACCC AGCTTTGTTTAAACGGCGCGCCGG
0
0
0
25
0
50
75
>90
Pst I
GCTGCAGC TGCACTGCAGTGCA AACTGCAGAACCAATGCATTGG AAAACTGCAGCCAATGCATTGGAA CTGCAGAACCAATGCATTGGATGCAT
0
0
>90
>90
75
>90
75
>90
0
0
10
25
10
75
0
0
25
75
50
>90
>90
>90
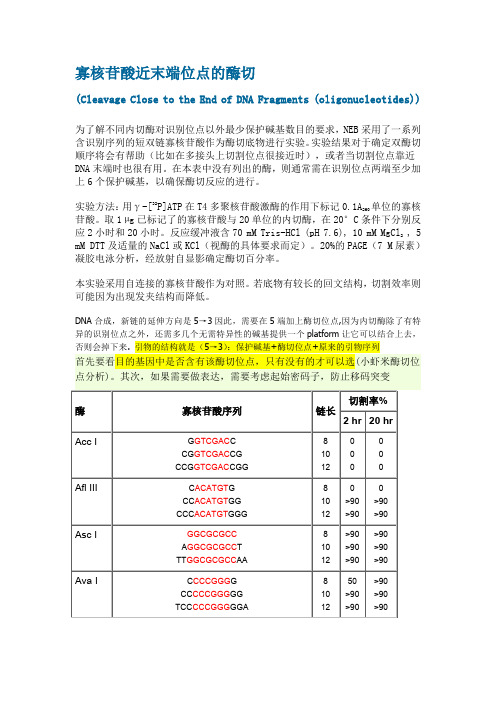
注释:
1.如果要加在序列的 5’端,就在酶切位点识别碱基序列(红色)的
5’端加上相应的碱基(黑色),相同如果要在 3’端加保护碱基,就
在酶切位点识别碱基序列(红色)的 3’端加上相应的碱基(黑
色)。
2.切割率:正确识别并酶切的效率
CAGATCTG GAAGATCTTC GGAAGATCTTCC
GGCGCGCC AGGCGCGCCT TTGGCGCGCCAA
GGGT(A/T)ACCC
AACTGCAGAACCAATGCATTGG AAAACTGCAGCCAATGCATTGGAA CTGCAGAACCAATGCATTGGATGCAT
0
0
25
50
25
>90
0
0
0
0
>90
>90
50
50
EcoR I Hae III Hind III Kpn I Mlu I Nco I Nde I
Nhe I Not I
Nsi I Pac I
GGAATTCC CGGAATTCCG CCGGAATTCCGG
GGGGCCCC AGCGGCCGCT TTGCGGCCGCAA
CATCGATG GATCGATC CCATCGATGG CCCATCGATGGG
切割率%
2 hr 20 hr
0
0
0
0
0
0
0
0
>90
>90
>90
>90
>90
>90
>90
>90
>90
>90
50
>90
>90
>90
>90
>90
10
25
>90
>90
>90
>90
0
0
75
>90
25
>90
0
0
0
0
50
>90
0
10
寡核苷酸序列
GGTCGACC CGGTCGACCG CCGGTCGACCGG
CACATGTG CCACATGTGG CCCACATGTGGG
GGCGCGCC AGGCGCGCCT TTGGCGCGCCAA
CCCCGGGG CCCCCGGGGG TCCCCCGGGGGA
CGGATCCG CGGGATCCCG CGCGGATCCGCG
>90
>90
>90
>90
>90
>90
Xba I Xho I Xma I
CTCTAGAG GCTCTAGAGC TGCTCTAGAGCA CTAGTCTAGACTAG
CCTCGAGG CCCTCGAGGG CCGCTCGAGCGG
CCCCGGGG CCCCCGGGGG CCCCCCGGGGGG TCCCCCCGGGGGGA
0
0
10
10
>90
>90
>90
>90
0
0
Pvu I
CCGATCGG ATCGATCGAT TCGCGATCGCGA
0
0
10
25
0
10
Sac I
CGAGCTCG
10
10
Sac II
GCCGCGGC TCCCCGCGGGGA
0
0
50
>90
Sal I
GTCGACGTCAAAAGGCCATAGCGGCCGC
0
10
0
10
10
50
>90
>90
Spe I
GACTAGTC GGACTAGTCC CGGACTAGTCCG CTAGACTAGTCTAG
10
>90
10
>90
0
50
0
50
Sph I
GGCATGCC CATGCATGCATG ACATGCATGCATGT
0
0
0
25
10
50
Stu I
AAGGCCTT GAAGGCCTTC AAAAGGCCTTTT
TTAATTAA GTTAATTAAC CCTTAATTAAGG
>90
>90
>90
>90
>90
>90
>90
>90
>90
>90
>90
>90
0
0
0
0
10
75
0
0
>90
>90
>90
>90
0
0
25
50
0
0
50
75
0
0
0
0
0
0
0
075>源自075>90
0
0
10
25
10
50
0
0
10
10
10
10
25
90
25
>90
10
CAAGCTTG CCAAGCTTGG CCCAAGCTTGGG
GGGTACCC GGGGTACCCC CGGGGTACCCCG
GACGCGTC CGACGCGTCG
CCCATGGG CATGCCATGGCATG
CCATATGG CCCATATGGG CGCCATATGGCG GGGTTTCATATGAAACCC GGAATTCCATATGGAATTCC GGGAATTCCATATGGAATTCCC
NEB 保护碱基各种酶切 位点保护碱基
19882)
集团文件发布号:(9816-UATWW-MWUB-WUNN-INNUL-DQQTY-
酶 Acc I Afl III Asc I Ava I BamH I Bgl II BssH II BstE II BstX I Cla I
PCR 设计引物时酶切位点的保护
0
0
GCGTCGACGTCTTGGCCATAGCGGCCGCGG
10
50
ACGCGTCGACGTCGGCCATAGCGGCCGCGGAA 10
75
Sca I
GAGTACTC AAAAGTACTTTT
10
25
75
75
Sma I
CCCGGG CCCCGGGG CCCCCGGGGG TCCCCCGGGGGA
GGCTAGCC CGGCTAGCCG CTAGCTAGCTAG
TTGCGGCCGCAA ATTTGCGGCCGCTTTA AAATATGCGGCCGCTATAAA ATAAGAATGCGGCCGCTAAACTAT AAGGAAAAAAGCGGCCGCAAAAGGAAAA
TGCATGCATGCA CCAATGCATTGGTTCTGCAGTT
3。加保护碱基时最好选用切割率高时加的相应碱基。
