ChIPSeq概述及技术路线
chip—seq原理

chip—seq原理Chip-seq(Chromatin Immunoprecipitation Sequencing)是一种能够识别染色质结构和转录因子结合位点的技术。
它结合了染色质免疫共沉淀和高通量测序技术,可以高效地鉴定蛋白质与DNA相互作用的位点。
Chip-seq的原理主要包括以下几个步骤:样品处理、免疫沉淀、DNA 提取、测序和数据分析。
首先,样品处理是为了获得高质量的细胞或组织样品。
样品要经过交联处理,将蛋白质与DNA交联在一起,然后通过细胞裂解和核蛋白提取等步骤,将染色质释放出来。
接下来,免疫沉淀是将特定的抗体与目标蛋白质结合,形成抗体-蛋白质复合物。
这些抗体可以是特定的转录因子抗体,也可以是与染色质结构相关的抗体。
将这些抗体与染色质样品混合,使抗体与靶标结合,形成免疫沉淀复合物。
然后,通过蛋白质A/G磁珠等亲和剂,将抗体-蛋白质复合物与磁珠结合。
这些磁珠可以有效地分离免疫沉淀复合物,并去除非特异性结合的蛋白质。
接下来,DNA提取是为了从免疫沉淀复合物中纯化出与目标蛋白质结合的DNA。
通过裂解免疫沉淀复合物,释放出DNA,并经过一些处理(如蛋白酶消化)去除非特异性结合的DNA。
然后,通过DNA纯化方法,如酚/氯仿提取或商业化的DNA纯化试剂盒,从混合物中纯化出DNA。
接下来,测序是将纯化的DNA进行高通量测序。
Chip-Seq通常采用Illumina测序技术,将DNA片段连接到测序芯片上,通过测序仪进行测序。
这样就可以获得大量的短序列片段,每个片段都对应着原来染色质上的一个特定区域。
最后,数据分析是将测序得到的片段与参考基因组进行比对,确定它们的起始位置和覆盖范围。
通过对片段的分布模式进行统计分析,可以鉴定出与目标蛋白质结合的DNA序列区域,也就是转录因子结合位点或染色质修饰位点。
总结起来,Chip-seq技术通过将特定的抗体与目标蛋白质结合,形成免疫沉淀复合物,然后纯化出与目标蛋白质结合的DNA,并进行高通量测序和数据分析,从而鉴定出染色质结构和转录因子结合位点。
chipseq实验原理和callpeak原理

chipseq实验原理和callpeak原理全文共四篇示例,供读者参考第一篇示例:Chip-seq实验原理是一种用于研究染色质上蛋白质与DNA相互作用的技术。
ChIP-seq(Chromatin Immunoprecipitation with Sequencing)技术结合了染色质免疫沉淀(ChIP)和高通量测序(seq),可以帮助科研人员探究基因的调控机制、染色质的结构与功能等重要生物学问题。
ChIP-seq实验的步骤分为:杀细胞、交联DNA和结合蛋白、细胞裂解、柱层纯化DNA-蛋白质复合物、解交联、DNA纯化、建立图书馆、测序等多个环节。
通过这个实验,可以获得与特定蛋白结合的DNA片段,并使用高通量测序技术对这些片段进行快速测序。
通过对测序数据的分析,可以识别出蛋白与DNA结合的位点、研究基因的表达调控等。
在ChIP-seq数据的分析中,一个重要的步骤是Callpeak。
Callpeak是一个用于识别ChIP-seq数据中蛋白与DNA结合位点的算法。
其主要目的是从测序数据中识别出富集的区域,即可能与特定蛋白结合的DNA序列。
Callpeak的原理是通过对ChIP-seq数据进行统计学分析,识别在基因组中具有高富集性的区域。
这种高富集性可能是由于特定蛋白在该区域与DNA结合,或者其他生物学过程所导致的。
Callpeak算法采用了一系列统计指标,如reads数量,reads的空间分布情况等,来确定哪些区域是与特定蛋白结合的位点。
Callpeak算法的核心是建立一个背景模型,用来描述在没有结合事件发生时的随机测序数据的分布。
通过比较实验组和对照组的测序数据,Callpeak可以识别出真正富集的区域,并给出统计学显著性的评估。
Chip-seq实验原理和Callpeak原理是ChIP-seq技术中非常重要的两个部分。
通过利用这些原理,科研人员可以更好地理解基因的调控机制,揭示染色质的结构与功能等生物学问题。
chip-seq过程

chip-seq过程Chip-seq(染色质免疫沉淀测序)是一种常用的基因组学技术,它可以用来研究蛋白质与DNA之间的相互作用。
本文将详细介绍chip-seq的过程及其应用。
一、引言Chip-seq是一种结合了染色质免疫沉淀(ChIP)和高通量测序(sequencing)的技术,用于研究特定蛋白质与DNA之间的相互作用。
通过该技术,我们可以确定蛋白质与DNA的结合位点,并进一步了解这些结合位点在基因调控中的作用。
二、实验步骤1. 交联:首先,将细胞或组织交联,使DNA与蛋白质相互交联形成复合物。
这一步骤可以使用甲醛等交联剂进行。
2. 染色质免疫沉淀:将交联后的细胞或组织进行裂解,使DNA与蛋白质分离。
然后,使用特异性抗体与目标蛋白质结合,形成抗原抗体复合物。
接着,使用磁珠或琼脂糖柱等材料,将抗原抗体复合物与其他非特异性结合的蛋白质和DNA分离。
3. 反交联:将抗原抗体复合物中的DNA与蛋白质进行反交联,使其分离。
这一步骤可以通过高温或酶切等方法进行。
4. DNA纯化:将反交联后的DNA进行纯化,去除杂质。
可以使用酚/氯仿等方法进行DNA的提取和纯化。
5. DNA测序:将纯化后的DNA进行高通量测序。
通过测序,可以得到大量的DNA片段序列。
6. 数据分析:对测序得到的数据进行分析,包括数据过滤、比对和富集分析等。
通过对数据的分析,可以确定蛋白质与DNA的结合位点,并推测这些结合位点在基因调控中的功能。
三、应用1. 确定转录因子结合位点:转录因子是调控基因表达的关键蛋白质,chip-seq可以用来确定转录因子与DNA的结合位点。
通过分析转录因子的结合位点,我们可以了解基因调控网络的组成和功能。
2. 研究组蛋白修饰:组蛋白修饰是一种重要的基因调控机制,chip-seq可以用来研究组蛋白修饰与DNA的相互作用。
通过分析组蛋白修饰的分布情况,我们可以了解基因的激活或抑制状态。
3. 鉴定染色体可及性:染色体的可及性是指染色体上的DNA片段是否容易被蛋白质结合。
chipseq分析流程

chipseq分析流程CHIPseq(chromatinimmunoprecipitationsequencing,抗原沉淀测序)是目前最常用的一种基因调控技术,也是一种全面、高效的技术,可用于研究特定组蛋白在特定细胞中的结合情况,同时也能够查明组蛋白在特定基因应激下的基因表达调控。
在分析CHIP-seq数据时,需要遵循一定的步骤和流程,内容主要包括数据准备、质粒提取、测序、碱基质量核查、序列对齐、拼接、建立peaks、转录因子结合位点的鉴定等,下文将详细介绍每一步的操作流程。
首先,CHIP-seq分析的数据准备工作是实验的第一步,准备的内容主要是两类:一类是含有DNA信号的样品和无DNA信号的对照样品,另一类是抗原质粒的提取及其他相关的实验准备,如抗原结合条件的调节、抗原结合及其他信号的检测。
其次,是质粒提取,一般采用抗原质粒提取技术,该技术可以提取抗原结合位点上的DNA片段,以实现转录因子与底物DNA结合的研究。
抗原质粒提取的基本流程包括多种操作:蛋白质的纯化、质粒的捕获、DNA的提取、质粒的洗脱及其他后续处理步骤。
第三步是测序,即通过测序仪进行序列测定,一般采用高通量测序技术,而该步骤是CHIP-seq研究中最重要也是最耗时的步骤,因为要用到大量的DNA片段,测序结果往往会产生大量数据。
接下来,针对测序得到的数据,需要对碱基质量进行核查,一般采用碱基质量检测软件,以评估序列质量并去除低质量数据,以最大程度的保证序列的准确性,提高数据的可用性。
接着,进行序列对齐,一般使用alignment软件,比如Bowtie、BWA等,将reads数据与参考基因组序列进行比对,进行reads的对齐,对比结果存储为SAM文件,该文件包括序列的物理位置等重要信息。
随后,是reads拼接,一般使用Picard软件,将reads拼接成更长的片段,大大提高了后续研究的效率。
接下来,是建立peaks,即根据碱基质量和序列对齐结果,识别抗原结合位点,从而获取各个基因调控位点的信息,建立peaks需要使用有效的软件,常用的有F-seq、 MACS和HTSeq等。
CHIPSEQ技术在转录因子结合位点分析的应用

CHIPSEQ技术在转录因子结合位点分析的应用CHIP SEQ(Chromatin Immunoprecipitation Sequencing)是一种高通量测定转录因子、组蛋白和DNA互作的方法。
它结合了染色质免疫沉淀(ChIP)和高通量测序技术,可以有效地鉴定转录因子在基因组上的结合位点,从而揭示基因表达调控的分子机制。
在本篇文章中,我们将探索CHIP SEQ技术在转录因子结合位点分析的应用。
CHIPSEQ技术的基本原理是将细胞或组织中的染色质进行交联固定,并利用特异性抗体对目标蛋白进行免疫沉淀。
然后,通过DNA片段的解链、末端修复和连接测序适配体等处理后,进行高通量测序。
最后,通过比对整个基因组的测序结果,可以确定转录因子结合位点的位置。
利用CHIPSEQ技术,可以鉴定和研究转录因子的结合位点,对于揭示基因调控网络、再表达调控、启动子选择以及逆转录及病理性过程中等尤为重要。
以下是CHIPSEQ技术在转录因子结合位点分析中的几个应用方面:1.定位转录因子结合位点:通过CHIPSEQ可以确定转录因子在基因组上的结合位点,并标记转录因子结合位点的丰度。
这有助于了解转录因子与基因调控网络之间的关系,以及转录因子在基因调控过程中所扮演的角色。
2.揭示转录因子的作用目标:CHIPSEQ技术可以鉴定转录因子结合位点附近的启动子和增强子等调控区域。
通过分析转录因子结合位点周围的DNA序列,可以预测经过转录因子调控的潜在靶基因,并进一步揭示转录因子对基因表达的调控机制。
3.研究转录因子的功能:通过CHIPSEQ技术可以鉴定转录因子结合位点的重叠情况,即多个转录因子共同结合的位点。
这有助于了解转录因子之间的相互作用关系,以及它们在调控基因表达中的合作作用和竞争作用。
4.鉴定转录因子与疾病的关联:通过CHIPSEQ技术可以鉴定在一些疾病状态下,转录因子结合位点的改变情况。
这有助于我们理解转录因子在疾病发生和发展中的角色,并为疾病的诊断和治疗提供新的靶点和策略。
ChIP-Seq综述
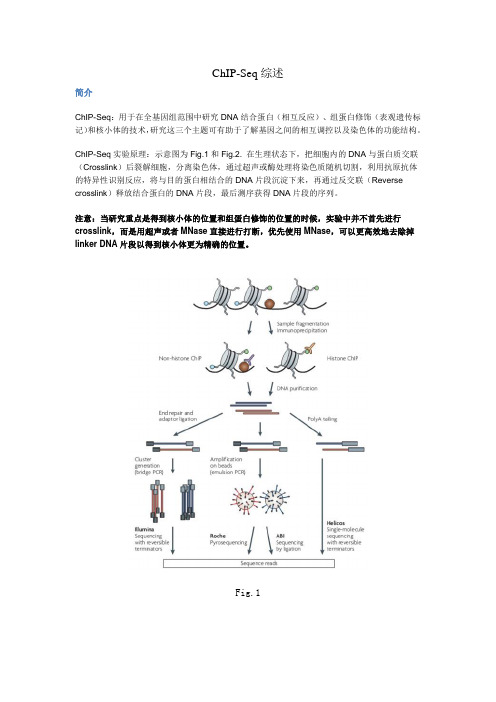
ChIP-Seq综述简介ChIP-Seq:用于在全基因组范围中研究DNA结合蛋白(相互反应)、组蛋白修饰(表观遗传标记)和核小体的技术,研究这三个主题可有助于了解基因之间的相互调控以及染色体的功能结构。
ChIP-Seq实验原理:示意图为Fig.1和Fig.2. 在生理状态下,把细胞内的DNA与蛋白质交联(Crosslink)后裂解细胞,分离染色体,通过超声或酶处理将染色质随机切割,利用抗原抗体的特异性识别反应,将与目的蛋白相结合的DNA片段沉淀下来,再通过反交联(Reverse crosslink)释放结合蛋白的DNA片段,最后测序获得DNA片段的序列。
注意:当研究重点是得到核小体的位置和组蛋白修饰的位置的时候,实验中并不首先进行crosslink,而是用超声或者MNase直接进行打断,优先使用MNase,可以更高效地去除掉linker DNA片段以得到核小体更为精确的位置。
Fig.1Fig.2ChIP-Seq的优势:1. 具有碱基层面的分辨率;2.不会有ChIP-chip中由DNA片段杂交导致的噪音,GC含量、片段长度、片段浓度以及耳机结构都会对杂交造成影响;3.ChIP-chip中的微阵列信号不是线性增长的,其所测量的范围有限。
4. 由于在设计array时,探针的数量、种类有限,当coverage比较高的时候无法准确测量,也无法发现新的序列。
ChIP-Seq与ChIP-chip的比较见Fig.3.Fig.3实验设计的关键抗体质量:一个灵敏度高和特异性高的抗体可以得到富集的DNA片段,这有利于探测结合位点。
样本量:Illumina,10-50ng DNA,需要用PCR进行扩增的轮数也比较少,因而由PCR导致的偏差比较小空白对照:空白对照是必要的,存在很多假阳性情况,举例:1. 开放的染色体区域更容易被打断成片段,这样导致tag数在基因组上的分布是不均匀的;2.很多重复序列会使做map的时候得到结果难以解释。
hichip-seq原理

hichip-seq原理hichipseq原理Hichipseq(也称为Hi-C ChIP-Seq)是一种结合了高通量染色体构象测序(Hi-C)和染色质免疫沉淀测序(ChIP-Seq)的方法。
Hi-C是一种能够揭示染色体3D结构的技术,可以探测基因组中的染色体互作和染色质细微结构的相对距离。
而ChIP-Seq则是一种用来寻找与某个特定蛋白质结合的DNA序列的技术。
将这两种技术结合起来,hichipseq能够研究特定蛋白质与基因组中其他区域的3D相互作用。
下面我们来一步一步探讨hichipseq的原理。
第一步:Hi-C技术Hi-C技术的第一步是对细胞进行交联。
这是为了固定细胞内染色质的空间构象,并保留染色体之间的相互作用。
一般会使用一个交联剂,如甲醛,对细胞进行交联。
交联后,细胞核膜被较强的荧光素去除剂处理,以消除核膜对Hi-C测序的影响。
第二步:染色质的两端连接在Hi-C测序中,细胞核被溶解,细胞核内的DNA被酶切成诸多碎片。
切碎的DNA片段会在内部结合,连接成一个环。
通过连接的方式,可以揭示染色质的三维结构。
第三步:免疫沉淀过程在hichipseq中,我们将ChIP过程引入到Hi-C中。
具体来说,我们将维持DNA的物质性结构和提交的抗体应用于DNA连接的过程中。
这样,我们就可以通过ChIP过程来寻找染色质上结合了我们感兴趣的特定蛋白质的DNA序列。
第四步:将交联的DNA连接蛋白质与DNA相互作用后,将进行反交联,以将蛋白质与DNA分离。
此过程中,将使用蛋白酶等方法,切割掉蛋白质,使得DNA被释放出来。
第五步:DNA测序DNA在连接后被测序,得到测序的读数。
这些读数将揭示DNA之间的相互作用及其3D结构。
同时,ChIP-Seq的原理也能够帮助我们了解蛋白质与DNA的结合情况。
通过上述步骤,hichipseq可以实现对特定蛋白质与基因组中其他区域的3D相互作用的研究。
这种结合了Hi-C和ChIP-Seq技术的方法,为我们揭示基因组的结构与功能之间的关系提供了一个有效的手段。
chip—seq原理

chip—seq原理Chip-seq(Chromatin Immunoprecipitation Sequencing,染色质免疫沉淀测序)是一种用于研究染色质上的转录因子结合位点和组蛋白修饰的方法。
该方法首先对细胞进行染色质交联,使得DNA与染色质蛋白交联在一起。
然后使用适当的抗体选择性地免疫沉淀目标转录因子或组蛋白修饰。
接下来,将沉淀物中的DNA进行解交联,并通过DNA纯化获取目标DNA片段。
之后,对目标DNA片段进行测序。
通过高通量测序技术,可以得到很多短序列片段,这些片段对应于染色体上的不同位置。
这些测序片段可以与参考基因组进行比对,从而确定它们在基因组上的位置。
最后,通过对比实验组和对照组的测序数据,可以鉴定转录因子结合位点或组蛋白修饰位点的位置和富集情况,进而研究染色质的功能和调控机制。
总结起来,Chip-seq的原理可以简化为以下几个步骤:1. 染色质交联:将DNA与染色质蛋白交联在一起。
2. 免疫沉淀:使用抗体选择性地沉淀目标转录因子或组蛋白修饰。
3. DNA解交联:将沉淀物中的DNA解除交联,并进行纯化。
4. DNA测序:对纯化后的DNA片段进行高通量测序。
5. 数据分析:通过比对测序数据,确定DNA片段在基因组上的位置,并进行ChIP峰检测和差异分析等。
当使用Chip-seq技术进行染色质免疫沉淀测序时,还可以进一步进行数据分析来获得更多的信息。
1. Peak calling(峰检测):通过对测序数据进行分析和统计,可以识别出在特定条件下与目标蛋白结合的区域,称为峰。
峰通常表示转录因子结合位点或组蛋白修饰位点。
2. Motif analysis(基序分析):对峰区域进行进一步分析,可以识别出其中的共有序列模式,称为基序。
这些基序可能与特定转录因子的结合相关,从而可以推断特定转录因子在染色质上的结合位点。
3. Differential binding analysis(差异结合分析):比较不同实验条件下的Chip-seq数据,可以发现转录因子的结合差异。
chipseq原理及流程

chipseq原理及流程ChIP-seq 流程包括样品处理、染色质免疫沉淀、DNA提取和准备、测序和数据分析阶段。
首先,样品处理包括准备细胞、核和染色质。
通过细胞生长、核提取和染色质交联等步骤,得到可以进行后续实验的样品。
接下来,进行染色质免疫沉淀。
这一步骤使用抗体特异性地选择性地沉淀与特定转录因子或组蛋白修饰结合的DNA片段。
一般而言,可以使用针对所研究蛋白的特异性抗体,将染色质中和蛋白相互作用的DNA序列捕获到特定的抗体上,从而实现染色质免疫沉淀。
然后,进行DNA提取和准备。
这一步骤涉及到将沉淀下来的染色质中的DNA片段进行纯化、修复和连接,以准备进行下一步的高通量测序。
通过这一过程,可以得到具有特定DNA序列的文库。
接下来,进行高通量测序。
这一步骤使用二代测序技术,对文库中的DNA序列进行测序。
通常可以选择使用Illumina技术进行测序,该技术能够高效地产生大量的短序列。
最后,进行数据分析。
这一步骤包括测序数据的处理和详细的生物信息学分析。
首先,测序数据需要进行质量控制和去除低质量序列。
然后,利用比对算法,将测序序列与参考基因组进行比对,以确定它们的起源和位置。
最后,通过统计分析和计算模型,可以鉴定特定转录因子或组蛋白在基因组上的结合位点和其附近的调控元件。
ChIP-seq技术的优点在于其高分辨率、高灵敏度和高通量测序的能力。
它可以帮助我们更好地了解蛋白与DNA相互作用的机制,识别转录因子、组蛋白修饰和表观遗传学调控在基因调控中的重要性。
总而言之,ChIP-seq技术为我们揭示染色质结构与功能之间的关系提供了有力的工具,为基因组学和生物医学研究提供了深入而广阔的透视。
chipseq流程 简书

chipseq流程简书Chip-seq(Chromatin Immunoprecipitation followed by sequencing)是一种广泛应用于研究基因组中蛋白质与DNA相互作用的方法。
本文将介绍Chip-seq实验的基本流程和分析方法,以及其在生物学研究中的应用。
第一部分:Chip-seq实验流程1. 细胞或组织样品的处理:首先,需要从感兴趣的细胞或组织中提取染色质。
常用的方法包括细胞裂解、核裂解和DNA剪切等步骤。
这些步骤的目的是获得高质量的染色质样品。
2. 免疫沉淀:接下来,将特定抗体与染色质样品一起孵育,使抗体与目标蛋白质结合。
这一步骤的目的是选择性地富集与特定蛋白质相结合的DNA片段。
3. DNA纯化:通过洗涤和离心等步骤,将非特异性结合的DNA片段去除,从而得到与特定蛋白质结合的DNA片段。
4. DNA片段的测序:将纯化的DNA片段进行测序,通常采用高通量测序技术,如Illumina测序。
这样可以得到大量的短序列读数,用于后续的分析。
第二部分:Chip-seq数据分析方法1. 数据预处理:首先,对测序得到的短序列读数进行质量控制和去除低质量reads。
然后,根据测序引物的序列去除引物序列。
2. 读数比对:将预处理后的短序列读数与参考基因组进行比对,常用的比对工具有Bowtie、BWA等。
比对的目的是将读数与其在基因组中的位置关联起来。
3. 峰识别:通过统计每个位置上的测序读数数量,可以得到染色质上的富集区域。
常用的峰识别算法有MACS、SICER等。
峰识别的目的是找出与特定蛋白质结合的DNA片段的富集区域。
4. 峰注释:对识别出的峰进行注释,可以了解峰所在的基因、转录因子结合位点等信息。
常用的注释工具有HOMER、ChIPseeker等。
5. 富集分析:通过对峰进行富集分析,可以了解特定蛋白质在基因组中的结合模式和功能。
常用的富集分析方法有Motif分析、GO 分析等。
ChIP-seq实践(非转录因子,非组蛋白)

ChIP-seq实践(非转录因子,非组蛋白)转自简书生信start_site在实战之前,首先对CHIP-seq分析做一些了解,以下是从各个地方copy过来综合起来的,有些散乱,是我认为重要以及应该了解的知识点。
chip-seq 实战就跟在转录组和外显子上的实战是没有本质区别的,只是它多了一些分析,转录组只找表达量做差异分析,外显子只找变异做变异注释,那chip-seq 找的是peaks和motif 。
这里biobabble公众号有比较完整的chip-seq的介绍:CS1: ChIPseq简介CS2: BED文件CS3: peak注释CS4:关于ChIPseq注释的几个问题CS5: 吃着火锅,唱着歌,还把分析给做了CS6: ChIPseeker的可视化方法CS7:Genomic coordination的富集性分析(1)CS8:Genomic coordination的富集性分析(2)CS9: GEO数据挖掘CS番外1:如何定义下游的区间?ChIP-seq 数据分析流程1.得到原始序列文件后,第一步是执行标准的高通量数据质量控制。
2.原始的 reads 回比到研究物种的参考基因组上3.特异性 ChIP-seq 的质量控制,检查 ChIP-seq 样品中的富集情况,并排除过度碎片4.【核心】鉴定全基因上感兴趣的因子富集的区域,也叫Peak calling5.一旦确定了峰值区域,还可以评估需要峰值位置的特定ChIP-seq 的质量控制措施6.在 Peak calling 之后,要确保预测的峰值准确地捕捉到binding pattern。
7.Peak calling后的分析取决于实验设计与生物学问题。
比如,如果在实验中有重复和/或多个条件,那么下一步可能是样本之间的比较(见第 8 章)。
生物学重复将提供有关数据(噪声)中的重现性和内在生物和技术变异性的信息。
差异结合分析解决了哪一Peak区域在两种情况下显示出明显不同的丰度(例如:与观察到的相同条件的重复之间的变化相比,差异比预期的要大得多)。
chipseq流程 简书

chipseq流程简书Chip-seq是一种高通量测序技术,用于研究蛋白质与DNA相互作用的位置和数量。
Chip-seq技术的流程包括以下步骤:1. 样品制备样品制备是Chip-seq实验的第一步。
样品可以是细胞、组织或DNA-protein 复合物。
样品制备的关键是保持样品的完整性和质量。
对于细胞和组织样品,需要进行细胞裂解和核提取;对于DNA-protein复合物,需要进行交联和裂解。
2. 免疫共沉淀免疫共沉淀是Chip-seq实验的核心步骤。
在这个步骤中,使用特定的抗体识别和捕获特定的蛋白质-DNA复合物。
抗体可以是单克隆抗体或多克隆抗体。
免疫共沉淀的关键是选择合适的抗体和优化实验条件,以确保高效的共沉淀。
3. DNA片段化在免疫共沉淀后,需要将DNA片段化为适当的长度。
这个步骤可以通过酶切、超声波或化学方法完成。
片段化的目的是使DNA片段长度适合测序,并且可以减少背景噪音。
4. DNA净化和富集在DNA片段化后,需要对DNA进行净化和富集。
这个步骤可以通过凝胶电泳、柱层析或磁珠富集等方法完成。
净化和富集的目的是去除杂质和富集目标DNA 片段,以提高Chip-seq实验的灵敏度和特异性。
5. DNA测序在DNA净化和富集后,需要进行高通量测序。
Chip-seq实验通常使用Illumina 测序平台进行测序。
测序的目的是获得大量的DNA序列数据,以便分析蛋白质-DNA相互作用的位置和数量。
6. 数据分析在DNA测序后,需要对数据进行分析。
数据分析的目的是确定蛋白质-DNA相互作用的位置和数量,并且确定这些相互作用是否具有生物学意义。
数据分析的方法包括序列比对、峰检测、注释和差异分析等。
总结:Chip-seq技术的流程包括样品制备、免疫共沉淀、DNA片段化、DNA净化和富集、DNA测序和数据分析。
这个流程需要仔细的操作和优化,以确保高质量的实验结果。
ChIP-seq技术

ChIP-seq技术简介:染色质免疫共沉淀技术(Chromatin Immunoprecipitation,ChIP),是研究体内蛋白质与DNA 相互作用的有力方法,通常用于转录因子结合位点或组蛋白特异修饰位点的研究。
将ChIP与第二代测序技术相结合的ChIP-Seq技术,能够高效地在全基因组范围内检测与组蛋白、转录因子等互作的DNA区段。
ChIP-seq的原理是首先通过染色质免疫共沉淀技术(ChIP)特异性地富集目的蛋白结合的DNA片段,并对其进行纯化与文库构建;然后对富集得到的DNA片段进行高通量测序。
研究人员通过将获得的数百万条序列标签精确定位到基因组上,从而获得全基因组范围内与组蛋白、转录因子等互作的DNA区段信息。
应用领域:1. 筛选调控蛋白在基因组上的结合靶点:针对转录因子、DNA/RNA聚合酶、转录复合物等调控因子蛋白进行特异性的富集,分离纯化与之结合的靶DNA片段并测序,分析筛选到准确有效的靶位点、靶基因数据2. 揭示差异化表观遗传变异的原理:通过对不同表型组织内组蛋白、转录因子蛋白及其他结合蛋白进行染色质免疫共沉淀测序并定量分析靶基因表达调控水平或针对同种组蛋白进行甲基化、磷酸化、泛素化修饰所导致的结合位点变异进行分析,即可准确揭示差异化表观变异原理;3. 研究表观遗传变异疾病:借助染色质免疫共沉淀测序技术结合生物信息分析揭示由蛋白与DNA相互作用变异而引起的疾病的原理,明确表观遗传修饰在癌症等表观遗传变异疾病的发生发展过程中的具体作用机制。
技术优势:•全基因组覆盖:ChIP-Seq可在全基因组范围单碱基水平对蛋白结合位点进行筛选与鉴定。
•高灵敏度:每个样本可获得数百万条的序列标签,可发现研究基因组上稀有的蛋白结合位点。
•高精确率:可获得高水平的信噪比数据,准确区分真实事件与噪音,精确定位蛋白结合位点。
•高性价比:仅需较少的数据量即可鉴定全基因组范围内的结合位点。
实验流程:A.ChIP-seq建库测序流程B.数据分析流程。
ChIPSeq概述及技术路线

ChIP-Seq概述及技术路线概述染色质免疫共沉淀技术 Chromatin Immunoprecipitation , ChIP 也称结合位点分析法,是研究体内蛋白质与 DNA 相互作用的有力工具,通常用于转录因子结合位点或组蛋白特异性修饰位点的研究.将 ChIP 与第二代测序技术相结合的 ChIP-Seq 技术,能够高效地在全基因组范围内检测与组蛋白、转录因子等互作的 DNA 区段.ChIP-Seq 的原理是:首先通过染色质免疫共沉淀技术 ChIP 特异性地富集目的蛋白结合的 DNA 片段,并对其进行纯化与文库构建;然后对富集得到的 DNA 片段进行高通量测序.研究人员通过将获得的数百万条序列标签精确定位到基因组上,从而获得全基因组范围内与组蛋白、转录因子等互作的 DNA 区段信息.技术路线1 .实验流程 Solexa2. 生物信息分析流程示意图研究内容1 .测序对客户提供的 ChIP 样品如果有阴阳参启动子区域或 DNA 序列的进行定量检测,检测合格后进行测序文库构建、 DNA 成簇 Cluster generation 扩增、高通量测序.2 .基本数据分析数据产出统计:对测序结果进行图像识别 Base calling ,去除污染及接头序列;统计结果包括:测定的序列 Reads 长度、 Reads 数量、数据产量.3. 高级数据分析标准高级数据分析内容包括:1 ChIP-Seq 序列与参考序列比对;2 Peak calling :统计样品 Peak 信息峰检测及计数、平均峰长度、峰长中位数;3 统计样品 Uniquely mapped reads 在基因上、基因间区的分布情况及覆盖深度;4 给出每个样品 Peak 关联基因列表及 GO 功能注释;5 在多个样品间,对与 Peak 关联基因做差异分析.技术特点应用领域由于 ChIP-Seq 的数据是 DNA 测序的结果,为研究者提供了进一步深度挖掘生物信息的资源,研究者可以在以下几方面展开研究:1判断 DNA 链的某一特定位置会出现何种组蛋白修饰;2检测 RNA polymerase II 及其它反式因子在基因组上结合位点的精确定位;3研究组蛋白共价修饰与基因表达的关系;4 CTCF 转录因子研究.。
ChIP-seq

• 染色质免疫共沉淀测序(ChIP-Seq)是指对染色质免疫 共沉淀(ChIP)获得的DNA片段进行大规模测序,并能 把所研究蛋白的DNA结合位点精确定位到基因组上。 • Roche GS FLX Titanium 、Illumina Solexa GA IIx和AB SOLID 4 这3种测序技术均可以用于ChIP-seq,其中采用 Illumina Solexa GA IIx进行ChIP-Seq已有较多文献报道。 • ChIP-Seq技术高质量、高通量、低成本的数据产出,为 表观遗传组学研究奠定了技术基础。研究者可以在以下几 方面展开研究:(1)判断DNA链的某一特定位置会出现 何种组蛋白修饰;(2)检测RNA polymerase II及其它反 式因子在基因组上结合位点的精确定位;(3)研究组蛋 白共价修饰与基因表达的关系;(4)CTCF转录因子研究。
ChIP-Seq有什么样品要求?
• (1) 请提供浓度≥10 ng/ul、总量≥200 ng、OD260/280为 1.8~2.2的DNA样品;若单次ChIP后DNA量不够,建议将2~3次 ChIP的DNA合并在一起。 • (2) 请提供DNA打断时检测胶图,要求打断后DNA电泳主带 在200-500 bp范围内;请对于ChIP获得 DNA设计引物进行 QPCR验证和定量,能够提供检测位点的检 ml管中,管上注明样品名称、浓度以 及制备时间,管口使用Parafilm封口。在运输前将所有样品管 固定于50 ml带盖离心管中,再将50 ml管放在封口袋中。为了 防止低浓度样品黏附在离心管壁上,请使用non-stick tube运输 DNA ;建议使用冰袋运输,并且尽量选用较快的邮递方式,以 降低运输过程中样品降解的可能性。
rip-seq实验原理

rip-seq实验原理
ChIP-seq实验原理ChIP-seq是一种用于研究DNA-蛋白相互作用的实验技术,由“Chromatin Immunoprecipitation-sequencing”(ChIP-seq)组成。
它利用特异性的免疫沉淀来捕获蛋白质与DNA的相互作用,可用于确定蛋白质结合的基因组位点。
ChIP-seq技术结合了免疫沉淀和高通量测序技术,使得它可以实现对整个基因组范围内特定转录因子结合位点的全面研究。
ChIP-seq实验的基本流程包括四个步骤:(1)获取原始样本;(2)免疫沉淀,通过免疫技术将特定蛋白与DNA结合;(3)限制性内切酶切割,利用限制性内切酶将结合DNA 分解成若干片段;(4)高通量测序,通过测序产生的序列数据,研究特定蛋白质与DNA的相互作用。
ChIP-seq实验能够精准定位特定蛋白与基因组DNA的结合位点,可以用来研究转录因子与基因表达的关系,解析基因调控机制,以及研究表观遗传学机制。
在自然系统中,它也可以用来揭示DNA-蛋白相互作用的变化规律,如揭示细菌的耐药性机制。
ChIP-seq实验的准确性取决于许多因素,包括所使用的免疫技术、样本量、DNA片段的长度、高通量测序的深度等。
此外,为了获得较为准确的结果,还需要对实验数据进行严格
的质控和分析,如采用聚类分析或位点检测方法来检测特定位点的蛋白质-DNA结合情况。
总之,ChIP-seq是一种高效、高灵敏度的技术,可以用来研究特定转录因子与基因组DNA的结合情况,是表观遗传学研究的重要手段。
它的成功应用为研究转录调控机制,解析基因功能和疾病机制提供了重要线索。
chipseq建库方法

chipseq建库方法
ChIP-seq是一种用于研究细胞内特定DNA位点和蛋白质结合的技术,具有广泛的应用,包括解析转录因子靶基因信息和分子调控机制,研究表观遗传修饰的特征和生物学功能等。
以下是ChIP-seq的建库方法:
1. 样本准备:首先,需要准备待检测的细胞样本,并将其进行处理,以使蛋白质与DNA分离。
2. 染色质免疫沉淀:使用特定的抗体对目标蛋白质进行免疫沉淀,同时将DNA与蛋白质分离。
3. 文库构建:将纯化的DNA片段进行末端修复、加尾和连接接头等操作,构建成可用于测序的文库。
4. 测序:将构建好的文库进行测序,获得ChIP-seq数据。
5. 数据分析:对测序得到的ChIP-seq数据进行质量控制、峰值分析和差异表达基因筛选等分析,以获取实验所需的信息。
需要注意的是,ChIP-seq技术需要使用高质量的抗体和优化的实验条件,以保证结果的准确性和可靠性。
同时,数据分析也是关键的一步,需要选择适合的方法和软件进行数据处理和分析,以获得可靠的实验结果。
chipseq基本流程

chipseq基本流程下载温馨提示:该文档是我店铺精心编制而成,希望大家下载以后,能够帮助大家解决实际的问题。
文档下载后可定制随意修改,请根据实际需要进行相应的调整和使用,谢谢!并且,本店铺为大家提供各种各样类型的实用资料,如教育随笔、日记赏析、句子摘抄、古诗大全、经典美文、话题作文、工作总结、词语解析、文案摘录、其他资料等等,如想了解不同资料格式和写法,敬请关注!Download tips: This document is carefully compiled by theeditor. I hope that after you download them,they can help yousolve practical problems. The document can be customized andmodified after downloading,please adjust and use it according toactual needs, thank you!In addition, our shop provides you with various types ofpractical materials,such as educational essays, diaryappreciation,sentence excerpts,ancient poems,classic articles,topic composition,work summary,word parsing,copy excerpts,other materials and so on,want to know different data formats andwriting methods,please pay attention!ChIP-seq 是一种用于研究蛋白质与 DNA 相互作用的技术,其基本流程包括以下步骤:1. 细胞固定与染色质断裂细胞培养:在适当的条件下培养细胞,使其处于所需的生理状态。
3D基因组—实验技术
3D基因组—实验技术一、3D基因组之ChIP-seq基因组的三维立体结构的主要组成是DNA和蛋白质。
研究基因组的三维立体结构就是研究DNA与蛋白之间的互作。
ChIP一直是研究蛋白与DNA互作的重要方法。
他可以显示众多调节蛋白在基因组上的分布。
下表是生物体内常见的几种DNA调节蛋白。
Histone是核小体的基本组成蛋白,核小体是染色质的基本组成单位。
组蛋白的修饰(如:甲基化,乙酰化,磷酸化等)使核小体在DNA上发生移动,改变核小体的位置。
这使得特定的组蛋白修饰代表特定的生物学特征。
例如H3K27ac代表激活型启动子区域,H3K27me3代表抑制型启动子区域,等等。
除了组蛋白外,还有一类重要的调控元件-转录因子来调节染色质的结构和功能,然而,转录因子要与RNA聚合酶II互作,才可以调控基因表达。
CTCF是一类重要的绝缘子蛋白,与RAD21蛋白形成稳定的3维立体结构区域,维持染色质的正常形态。
另外,您需要根据目的基因的表达情况选择相应的ChIP组合方式。
1.ChIP-seq数据分析ChIP-seq的数据呈现形式是多种多样的。
但主要以3种形式出现在文章中。
①最常见的peak图②研究转录起始位点的TSS距离分布图,可以清楚的观察到TSS 位点处,多种蛋白的分布情况。
③比较直观的热图,可以清楚的看出同一DNA区域,不同蛋白的分布情况,以及同一蛋白在不同DNA区域的分布情况。
二、3D基因组之Hi-C技术Hi-C(High-throughput/resolutionchromosome conformation capture)技术,以整个细胞核为研究对象,利用高通量测序技术,结合生物信息学方法,研究全基因组范围内整个染色质DNA在空间位置上的关系;通过对染色质内全部DNA相互作用模式进行捕获,获得高分辨率的染色质三维结构信息。
1.Hi-C技术流程Hi-C,Hi-C的基本流程是首先将染色质,用交联HindIII限制性核酸内切酶将DNA片段切开,形成粘性末端,将带有dCTP-biotion补齐粘性末端,用T4连接酶连接,纯化链接产物,用链霉亲和磁珠捕获带有biotion的DNA互作片段,测序比对DNA互作区域。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
ChIP-Seq概述及技术路线
概述
染色质免疫共沉淀技术( Chromatin Immunoprecipitation , ChIP )也称结合位点分析法,是研究体内蛋白质与 DNA 相互作用的有力工具,通常用于转录因子结合位点或组蛋白特异性修饰位点的研究。
将 ChIP 与第二代测序技术相结合的 ChIP-Seq 技术,能够高效地在全基因组范围内检测与组蛋白、转录因子等互作的 DNA 区段。
ChIP-Seq 的原理是:首先通过染色质免疫共沉淀技术( ChIP )特异性地富集目的蛋白结合的DNA 片段,并对其进行纯化与文库构建;然后对富集得到的 DNA 片段进行高通量测序。
研究人员通过将获得的数百万条序列标签精确定位到基因组上,从而获得全基因组范围内与组蛋白、转录因子等互作的 DNA 区段信息。
技术路线
1 .实验流程( Solexa )
2. 生物信息分析流程示意图
研究内容
1 .测序
对客户提供的 ChIP 样品(如果有阴阳参启动子区域或 DNA 序列的)进行定量检测,检测合格后进行测序文库构建、 DNA 成簇( Cluster generation )扩增、高通量测序。
2 .基本数据分析数据产出统计:对测序结果进行图像识别( Base calling ),去除污染及接头序列;统计结果包括:测定的序列( Reads )长度、 Reads 数量、数据产量。
3. 高级数据分析
标准高级数据分析内容包括:
( 1 ) ChIP-Seq 序列与参考序列比对;
( 2 ) Peak calling :统计样品 Peak 信息(峰检测及计数、平均峰长度、峰长中位数);( 3 )统计样品 Uniquely mapped reads 在基因上、基因间区的分布情况及覆盖深度;
( 4 )给出每个样品 Peak 关联基因列表及 GO 功能注释;
( 5 )在多个样品间,对与 Peak 关联基因做差异分析。
技术特点
应用领域
由于 ChIP-Seq 的数据是 DNA 测序的结果,为研究者提供了进一步深度挖掘生物信息的资源,研究者可以在以下几方面展开研究:
(1)判断 DNA 链的某一特定位置会出现何种组蛋白修饰;
(2)检测 RNA polymerase II 及其它反式因子在基因组上结合位点的精确定位;
(3)研究组蛋白共价修饰与基因表达的关系;
(4) CTCF 转录因子研究。
