不同的酶切位点
酶切位点汇总

酶切位点汇总
酶切位点,又称为限制性内切酶位点,是指DNA分子上特定的序列,这些序列是限制
性内切酶可以识别和切割的地方。
限制性内切酶是一种在细菌和其它生物中广泛存在的酶,能够切割或切除一个或多个DNA碱基对。
这些限制性内切酶在生物技术领域广泛应用,用
于DNA序列分析、DNA重组、基因工程等方面。
以下是常见的几种酶切位点:
1. EcoRI切割位点是5′-GAATTC-3′,这是一种广泛应用的限制性内切酶,通常用于DNA纯化、制备DNA载体等。
2. BamHI切割位点是5′-GGATCC-3′,BamHI能够切割链间,产生具有黏性末端的DNA 序列。
常被用于制备双链DNA的黏性末端。
4. PstI切割位点是5′-CTGCAG-3′,PstI是一种双切酶,可以切割成不同长度的DNA 序列,适用于构建多种不同长度的DNA分子。
总之,酶切位点及其对应的限制性内切酶在现代生物领域有着广泛的应用和重要的作用。
了解不同的酶切位点是有很大帮助的,它可以为实验设计和分子生物学研究提供基础。
同时,也让我们更好地理解限制性内切酶在DNA分子上的作用,帮助我们在生物技术领域
更加熟练地掌握其应用。
tev蛋白酶切位点的dna序列

tev蛋白酶切位点的dna序列
Tev蛋白酶切位点的DNA序列是指Tev蛋白酶可以识别并切割的DNA序列,也被称为Tev切割位点。
Tev蛋白酶是一种常用的内切酶,具有高度特异性和高效性。
Tev蛋白酶的切割位点为5'-G|TATAC-3',其中“|”所示的位置为切割位点。
因此,Tev蛋白酶切割位点的DNA序列为5'-GTTAAC-3'。
Tev蛋白酶的切割位点的序列具有较高的特异性和保守性,因此在进行基因克隆和重组DNA技术时,Tev蛋白酶切割位点的选择非常重要。
在进行DNA片段连接时,通过在DNA的末端引入Tev蛋白酶切割位点,可以使用Tev蛋白酶将DNA 片段剪切开,并通过该切口将不同的DNA片段连接起来。
需要注意的是,Tev蛋白酶切割位点的DNA序列只是其中一种常用的切割位点序列,不同种类的内切酶所识别的切割位点序列也是不同的。
因此,在进行基因克隆和重组DNA技术时,需要选择适合特定目的的内切酶切割位点序列。
基因工程中常用的三种工具酶

一、限制性核酸内切酶(restriction endonuclease)1.定义:凡能识别和切割双链DNA分子内特定核苷酸序列的酶,也称为限制酶(restriction enzyme,RE)。
2.类型:来自原核生物,有三种类型。
Ⅰ型:兼具甲基化修饰和ATP参与的核酸内切酶活性,随机切割。
Ⅱ型:大多能特异识别4~6个核苷酸序列(回文结构),最大识别序列为8个核苷酸,如SfiI、NotI;但有近10种Ⅱ型限制酶的识别序列为非回文结构,如SfaNI、MnlI等,Ⅱ型限制酶均可作为基因工程的工具酶。
另有一些来源不同的限制酶的识别位点是相同的核苷酸序列,将这类酶特称为同工异源酶(isoschizomers)或同裂酶。
同工异源酶切割产生相同的末端;有一些同工异源酶对于切割位点上的甲基化碱基的敏感性有所差别,故可用来研究DNA 甲基化作用,如SmaI和XmaI;HpaII和MspI;MboI和Sau3AI是成对的同工异源酶;其中HpaII和MspI是一对同工异源酶,其识别位点是CCGG。
与同工异源酶对应的一类限制酶,它们虽然来源各异,识别序列也各不相同,但都产生出相同的粘性末端,称为同尾酶(isocaudamers)。
常用的限制酶BamHI、BclI、BglII、Sau3AI和XhoII就是一组同尾酶,它们切割DNA之后都形成由GATC4个核苷酸组成的粘性末端。
显而易见,由同尾酶所产生的DNA片段,是能够通过其粘性末端之间的互补作用而彼此连接起来的,因此在基因克隆实验中很有用处。
但必须指出,由两种同尾酶消化产生的粘性末端,重组之后所形成的序列结构再不能被原来的任何一种同尾酶所识别。
Ⅲ型:功能基本同Ⅰ型,但为特定位点切割。
三种限制酶的区别如下表所示:Ⅰ型Ⅱ型Ⅲ型DNA底物dsDNA dsDNA dsDNA辅助因子Mg2+,A TP,SAM Mg2+ Mg2+,A TP识别序列特异特异特异切割位点非特定(于识别序列前后100~1000bp范围之内)特定(切割于识别序列之中或近处,固定位点)特定(切割点在识别序列后25~75bp处)与甲基化作用的关系内切酶蛋白同时具有甲基化酶的作用酶蛋白不具有甲基化作用内切酶蛋白同时具有甲基化酶的作用3.命名:第一个字母取自产生该酶的细菌属名,用大写;第二、第三个字母是该细菌的种名,用小写;第四个字母代表株。
多克隆位点mcs名词解释

多克隆位点mcs名词解释MCS (Multiple Cloning Site)是用于分子生物学实验中插入DNA片段的特定DNA序列。
MCS通常用于载体(如质粒)中,其序列包含了多个不同限制酶切位点,使得可以方便地将目标DNA插入到载体中。
MCS也被称为多克隆位点。
Multiple Cloning Site (MCS) is a specific DNA sequence used for inserting DNA fragments in molecular biology experiments. MCS is usually located in vectors, such as plasmids, and contains multiple different restriction enzyme recognition sites, allowing convenient insertion of target DNA into the vector. MCS is also referred to as a multiple cloning site.1. The MCS in the plasmid vector contains different restriction enzyme recognition sites.质粒载体中的MCS包含不同的限制酶切位点。
2. We used the MCS to insert our gene of interest into the vector.我们使用了MCS将我们感兴趣的基因插入到质粒中。
3. The MCS provides a convenient way to clone DNA fragments into a vector.MCS提供了一个方便的方式将DNA片段克隆到质粒中。
4. The MCS allows for efficient cloning of multiple DNA fragments in the vector.MCS允许在质粒中高效克隆多个DNA片段。
载体双酶切位点选择

载体双酶切位点选择全文共四篇示例,供读者参考第一篇示例:载体双酶切位点选择是分子生物学研究中的重要步骤之一,它决定了在载体DNA中插入外源DNA片段的位置,进而影响到重组DNA 的构建和表达。
在构建重组载体时,选择合适的酶切位点可以提高重组效率,减少不必要的工作量,并使实验更加方便高效。
酶切位点是DNA分子上的一些特定区域,通过特定的酶切割可以将DNA分子切割成特定长度的片段。
一般来说,酶切位点具有以下一些特点:1. 酶切位点长度较短,一般为4-8个碱基对;2. 酶切位点具有对称性,即在该位点的两侧序列是互补的;3. 酶切位点不应与外源DNA片段中的位点相同,以避免反复切割和插入。
在选择载体双酶切位点时,首先需要考虑载体本身的结构和特点。
常见的载体包括质粒、病毒基因组和人工染色体等,它们具有不同的大小、复制方式和表达能力,因此需要根据实际需要选择合适的载体。
需要考虑插入外源DNA片段的大小和结构。
外源DNA片段一般较长,因此需要选择足够大的酶切位点,以确保插入片段可以被正确连接和表达。
在实际操作中,常用的双酶切位点包括EcoRI/BamHI、XhoI/HindIII、SalI/XbaI等。
这些切位点具有一定的选择性和灵活性,可以满足不同长度和结构外源DNA片段的插入需求。
这些切位点在实验中的使用也相对方便,不仅酶切效率高,而且可以通过PCR扩增等方法在外源DNA片段两端引入相关的酶切位点。
除了常用的双酶切位点外,还有一些特殊的切位点可以用于特定实验需求。
有些酶切位点可以在酶切的同时引入特定的序列标记,用于检测和筛选重组载体;有些切位点具有较高的酶切效率,适合于插入较长的外源DNA片段等。
在选择载体双酶切位点时,需要综合考虑实验需求和操作方便性,选择最合适的切位点进行操作。
在实际操作中,选择适当的载体双酶切位点可以提高重组效率,减少不必要的工作量和时间,使实验更加高效和方便。
研究人员应该根据实验需求和操作经验选择合适的双酶切位点,确保重组实验的顺利进行,为分子生物学研究和应用提供更大的便利性和效率。
双酶切实验原理

双酶切实验原理
双酶切实验是一种用于确定DNA序列的特定区域的酶切位点
的实验方法。
它基于两个限制性内切酶共同作用于DNA分子,产生不同的酶切位点的原理。
在双酶切实验中,首先选择两个具有不同的限制性内切酶,这两个酶分别具有不同的酶切位点序列。
然后,将待检测的
DNA样品与这两个限制性内切酶一起反应,使其在特定的条
件下与DNA发生切割。
由于每个限制性内切酶在特定的酶切位点周围识别并切割DNA,因此在双酶切实验中,如果DNA序列中同时存在这两
个酶切位点,那么DNA分子将在两个酶切位点处被切割成三
个部分。
如果DNA序列中只存在其中一个酶切位点,DNA分子将在该位点处被切割成两个部分。
如果DNA序列中不存在
这两个酶切位点,DNA分子将不被切割。
实验结束后,通过电泳将不同长度的DNA片段分离出来,并
通过染色或荧光等方法可视化这些DNA片段。
通过观察
DNA片段的迁移距离和相对大小,可以确定DNA序列的特定区域是否存在以上述的两个酶切位点。
从而对DNA序列进行
分析和确认。
值得注意的是,双酶切实验的成功与否取决于待检测的DNA
序列中是否存在与所选择的限制性内切酶的酶切位点匹配的序列。
如果DNA序列中不存在所选酶切位点,DNA将不会被切割,从而无法进行相关分析和确认。
因此,在进行双酶切实验
之前,需要进行相关的DNA序列分析和预测,以确定所选酶切位点是否适用于目标DNA序列的研究。
酶切原理及步骤

酶切原理及步骤一、酶切原理1.酶切反应酶切反应是指使用酶作为催化剂,对底物进行切割或降解的反应。
在酶切反应中,酶的活性中心与底物特异性结合,通过催化作用将底物分解成小分子片段。
2.酶切位点酶切位点是指酶与底物特异性结合的部位。
不同的酶具有不同的酶切位点,通常由特定的氨基酸序列组成。
3.酶切动力学酶切动力学描述了酶切反应的速度和底物浓度之间的关系。
在一定条件下,当底物浓度高于某一阈值时,反应速度将达到最大值。
二、酶切步骤1.酶液准备在进行酶切实验前,需要准备适量的酶液。
根据实验需求选择合适的酶种类和浓度。
通常,酶液需要在冰箱中冷藏保存。
2.样品准备将待测样品进行预处理,以便与酶液混合后进行反应。
样品处理方法因实验而异,常见的处理方法包括细胞破碎、蛋白质提取等。
3.酶切反应设置将准备好的酶液和样品混合,加入适量的缓冲液(如pH 7.4的Tris-HCl缓冲液),设置反应温度和时间。
4.酶切反应温度和时间设置根据所选酶的活性要求和实验条件,设置适宜的反应温度和时间。
通常,适宜的反应温度为37℃,反应时间因底物种类和浓度而异。
5.反应终止和产物检测在反应结束后,需要终止反应并检测产物。
常用的终止方法包括加入酚/氯仿抽提、加热或加入抑蛋白剂。
产物检测方法因实验而异,常见的检测方法包括蛋白质印迹、电泳、光谱分析等。
三、酶切实验设计1.酶种选择2.根据实验需求选择合适的酶种类。
不同的酶具有不同的特异性,需要根据目标蛋白质序列选择具有相应酶切位点的酶。
同时还需要考虑所选酶的活性、稳定性和安全性。
3.实验条件优化在进行酶切实验前,需要对实验条件进行优化。
主要包括底物浓度、缓冲液pH值、离子强度、反应温度和时间等方面的优化。
通过调整实验条件可以提高产物的产量和质量。
4.产物检测方法选择根据实验需求选择合适的产物检测方法。
常用的检测方法包括蛋白质印迹、电泳、光谱分析等。
需要根据目标产物性质选择适宜的检测方法以便于后续分析。
酶切位点平末端

酶切位点平末端
酶切位点平末端指的是一种特定的DNA或RNA序列结构,其中两个不同的序列在同一个平面上相互平行且相对。
这种结构通常出现在DNA或RNA的限制性内切核酸酶的切割位点处,这些酶能够识别特定的序列并切割DNA或RNA分子。
在DNA或RNA分子中,酶切位点平末端的具体示例包括:
1.EcoRI酶切位点:EcoRI是一种限制性内切核酸酶,能够识别并切割DNA
分子中的GAATTC序列。
切割后产生的片段具有平末端。
2.BamHI酶切位点:BamHI是一种限制性内切核酸酶,能够识别并切割
DNA分子中的GGATCC序列。
切割后产生的片段同样具有平末端。
在生物实验中,酶切位点平末端的应用非常广泛。
例如,在构建基因克隆载体时,需要将目的基因与载体进行连接,通常需要在目的基因和载体之间进行限制性内切核酸酶的切割。
当使用平末端的限制性内切核酸酶进行切割时,可以获得两个平末端的DNA片段,通过连接这些片段可以实现目的基因与载体的连接。
总结来说,酶切位点平末端指的是一种特定的DNA或RNA序列结构,其中两个不同的序列在同一个平面上相互平行且相对。
这种结构通常出现在限制性内切核酸酶的切割位点处,这些酶能够识别特定的序列并切割DNA或RNA 分子。
在生物实验中,酶切位点平末端的应用非常广泛,例如在构建基因克隆载体时需要进行限制性内切核酸酶的切割和连接。
简述dna指纹的原理及其应用

简述DNA指纹的原理及其应用1. DNA指纹的原理DNA指纹是一种通过比较DNA序列的方法,将个体的DNA样本与其他个体的DNA样本进行区分和识别的技术。
其原理基于下面几个关键步骤:1.1 DNA提取首先,从样本中提取出DNA。
常用的样本包括血液、口腔拭子、头发、唾液等。
提取DNA的方法通常采用蛋白酶、溶剂等物质将细胞壁破坏,并使用离心等技术将DNA从细胞中提取出来。
1.2 PCR扩增接下来,使用聚合酶链反应(PCR)对DNA进行扩增。
PCR是一种可以在体外迅速复制DNA片段的技术,通过多次循环反应使得少量的DNA片段迅速增加至足够数量用于后续分析。
1.3 酶切然后,使用限制性内切酶切割扩增得到的DNA片段。
限制性内切酶是一种可以识别并切割特定DNA序列的酶,不同的酶具有不同的酶切位点。
1.4 凝胶电泳将经过酶切的DNA片段通过凝胶电泳分离。
凝胶电泳是一种基于DNA片段的长度差异将其分离的技术。
在电场的作用下,DNA片段会在凝胶上进行迁移,根据片段大小的不同,迁移速度也不同。
1.5 探针杂交与检测最后,使用适当的DNA探针对分离得到的DNA片段进行杂交检测。
DNA探针是与目标DNA序列互补的DNA片段,通过与目标DNA序列发生特异性杂交,来检测目标DNA序列的存在与否。
2. DNA指纹的应用DNA指纹技术具有高度的准确性和唯一性,因此广泛应用于以下领域:2.1 刑事司法在刑事司法领域,DNA指纹技术被广泛应用于犯罪侦破和司法鉴定。
通过对犯罪现场、嫌疑犯或受害者的DNA进行比对,可以确定嫌疑犯的身份,确认罪犯或无辜者,为法庭提供有力的证据。
2.2 亲子鉴定DNA指纹技术可以用于亲子鉴定。
通过比对父母和子女之间的DNA序列,可以确定亲子关系的真实性。
亲子鉴定在民事案件、遗产继承和收养等方面有重要的应用。
2.3 遗传疾病诊断与预防DNA指纹技术可用于遗传疾病的诊断和预防。
通过分析个体的DNA序列,可以检测到一些遗传性的疾病风险,提前采取适当的预防措施,减少疾病的发生。
酶切常见问题

酶切常见问题限制性内切酶酶切的常见问题及解决⽅法,个⼈觉得总结得很好,特转贴供本供⼤家分享。
酶切出现问题,先看内切酶说明书,相应试剂公司⽬录。
不同公司出产的内切酶,菌株来源、制备⼯艺、纯度活⼒、酶切活性优化可能不同,酶切效果也有差别。
可在上⾯找到酶单位定义、保存条件、酶切体系[buffer及与其它酶双切的buffer等]、酶切反应温度[有些酶是在50、55或30等温度下反应的]、酶是否受甲基化影响、是否有星号活性及出现星号活可能因素、保护性碱基,同尾酶、同裂酶等等。
1. 酶切不开或不完全1.1 质粒问题纯度差或残留酶切抑制物最为常见。
杂蛋⽩存在会影响酶切,表现为A260/A280低于1.8;抑制物常见酚、盐、⼄醇等。
[重新提DNA,使⽤可靠试剂盒或可靠⼿⼯提取试剂]1.2 酶的问题:确认内切酶有效 [很多内切酶虽然有过期时间,但过期后只要能够有效酶切,可⽤。
确认酶切效果不好,做标记,更换] 。
【题外话:⽤内切酶注意】a. 内切酶如⽆特殊要求,保存-20~-30度。
并⾮越低越好,酶通常是保存在50%⽢油缓冲液中,温度过低时,酶会发⽣冻结(运输过程例外,由于酶需要低温运输,所以⽅便的情况下是使⽤⼲冰,酶会冻结,但冻结次数有限,相对是⼀种⽐较好的选择),如果是经常使⽤的话,酶会被反复冻融,从⽽降低了活性。
当然如果温度过⾼,呵呵,你就⾃⼰想去吧。
b. 酶在使⽤时应置于冰盒中取酶。
这点⼤家都清楚,不过多强调也没坏处。
因为实验室有些同学,酶取出后是放在冰盒上的,但有两点忽略了:拿酶的时候,⼿不是抓着管⼦上端,⽽握在管⼦的底部,相当于⽤⼿在给酶进⾏加热;有些酶是放在冰盒中,但吸酶的时候,还是将酶拿出来。
偶尔⼀次没有⼤碍,反复如此,可能会影响酶活。
c. 酶使⽤完后应尽快旋紧盖⼦。
有时我们可以发现,即使是-20~-30度放置,酶仍然会冻结。
个⼈认为的可能原因是由于在配置酶切反应时,酶管的盖⼦在较长时间的开着,⽢油会吸取空⽓中的⽔分,对于常⽤的⼤包装的酶有时就会出现冻结现象。
不同限制酶切割目的基因和质粒

不同限制酶切割目的基因和质粒在分子生物学中,限制酶起到了至关重要的作用。
限制酶是一类具有特异性识别和切割DNA序列的酶,能够定位并切割DNA分子中的特定碱基序列。
通过限制酶的切割,科学家们能够实现对基因和质粒的精确操作,进而实现对生物体的遗传信息的研究和调控。
1. 切割目的基因基因是生物体内负责遗传信息传递和功能表达的基本单位。
切割目的基因是通过限制酶切割来实现对该基因的操作,包括分离、提取、重组等。
限制酶的选择取决于目的基因的特定序列。
例如,对于目的基因中含有特定酶切位点的情况,我们可以选择限制酶来切割该位点,实现对目的基因的精确操作。
以限制酶EcoRI为例,EcoRI是一种常用的限制酶,它能够识别并切割具有序列5'-GAATTC-3'的DNA。
如果我们要切割一个目的基因,而该基因具有GAATTC序列,我们可以选择EcoRI来切割该序列,从而将目的基因切割出来。
这样一来,我们就可以对目的基因进行进一步的操作,如PCR扩增、测序等。
2. 切割质粒质粒是一种存在于细菌细胞中的环状DNA分子,具有自主复制的能力。
质粒在基因工程和分子生物学研究中起着重要的作用,可用于携带外源基因、表达蛋白等。
限制酶的切割可以使质粒在特定位置断裂,从而实现质粒的分离、提取和重组。
以限制酶BamHI为例,BamHI是一种常用的限制酶,它能够识别并切割具有序列5'-GGATCC-3'的DNA。
如果我们要切割一个质粒,而该质粒具有GGATCC序列,我们可以选择BamHI来切割该序列,从而将质粒切割成两个片段。
这样一来,我们就可以对质粒进行进一步的操作,如插入外源基因、构建重组质粒等。
通过限制酶的切割,科学家们能够实现对目的基因和质粒的精确操作。
这为基因工程、分子生物学研究和生物医学领域的发展提供了重要的工具和技术支持。
同时,限制酶的应用也需要科学家们对酶的特性和操作技术有深入的了解和掌握,以确保实验结果的准确性和可靠性。
基因工程名词解释

基因工程:按照预先设计好的蓝图,利用现代分子生物学技术,特别是酶学技术,对遗传物质DNA直接进行体外重组操作与改造,将一种生物(供体)的基因转移到另外一种生物(受体)中去,从而实现受体生物的定向改造与改良。
遗传工程:广义:指以改变生物有机体性状为目标,采用类似工程技术手段而进行的对遗传物质的操作,以改良品质或创造新品种。
包括细胞工程、染色体工程、细胞器工程和基因工程等不同的技术层次。
狭义:基因工程。
限制性核酸内切酶:是可以识别DNA的特异序列,并在识别位点或其周围切割双链DNA的一类内切酶,简称限制酶回文结构:每条单链以任一方向阅读时都与另一条链以相同方向阅读时的序列是一致的,例如5'GGTACC3' 3'CCATGG5'.同裂酶(isoschizomer)或异源同工酶:不同来源的限制酶可切割同一靶序列(BamH I 和Bst I具有相同的识别序列G↓GATGC)同尾酶(isocaudiners):来源不同、识别序列不同,但产生相同粘性末端的酶。
两个同尾酶形成的黏性末端连接之后,一般情况下连接处不能够再被其任何一种同尾酶识别。
BamH I 识别序列: G↓GATCCBgl II 识别序列: A↓GATCT黏性末端 (cohesive terminus/sticky ends):DNA末端一条链突出的几个核苷酸能与另一个具有突出单链的DNA末端通过互补配对粘合,这样的DNA末端,称为黏性末端。
平末端(blunt ends): DNA片段的末端是平齐的。
星活性(star activity):指限制性内切酶在非标准条件下,对与识别序列相似的其它序列也进行切割反应,导致出现非特异性的DNA片段的现象。
易产生星活性的内切酶用*标记。
如:EcoR I*底物位点优势效应:酶对同一个DNA底物上的不同酶切位点的切割速率不同。
连杆/衔接物(linker):化学合成的8~12个核苷酸组成的寡核苷酸片段。
不同的酶切位点
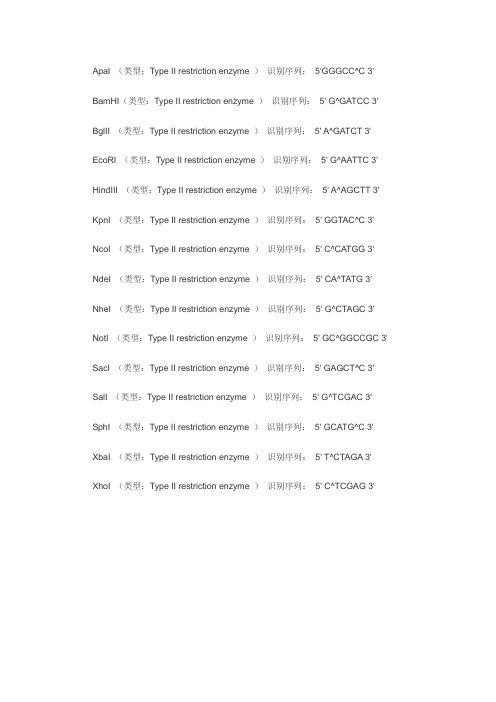
ApaI (类型:Type II restriction enzyme )识别序列:5'GGGCC^C 3' BamHI(类型:Type II restriction enzyme )识别序列:5' G^GATCC 3' BglII (类型:Type II restriction enzyme )识别序列:5' A^GATCT 3' EcoRI (类型:Type II restriction enzyme )识别序列:5' G^AATTC 3' HindIII (类型:Type II restriction enzyme )识别序列:5' A^AGCTT 3' KpnI (类型:Type II restriction enzyme )识别序列:5' GGTAC^C 3' NcoI (类型:Type II restriction enzyme )识别序列:5' C^CATGG 3' NdeI (类型:Type II restriction enzyme )识别序列:5' CA^TATG 3' NheI (类型:Type II restriction enzyme )识别序列:5' G^CTAGC 3' NotI (类型:Type II restriction enzyme )识别序列:5' GC^GGCCGC 3' SacI (类型:Type II restriction enzyme )识别序列:5' GAGCT^C 3' SalI (类型:Type II restriction enzyme )识别序列:5' G^TCGAC 3' SphI (类型:Type II restriction enzyme )识别序列:5' GCATG^C 3' XbaI (类型:Type II restriction enzyme )识别序列:5' T^CTAGA 3' XhoI (类型:Type II restriction enzyme )识别序列:5' C^TCGAG 3'Trx-tag硫氧还蛋白标签是个融合标签,有利于蛋白可溶性表达。
基因工程名词解释

基因工程:按照预先设计好的蓝图,利用现代分子生物学技术,特别是酶学技术,对遗传物质DNA直接进行体外重组操作与改造,将一种生物(供体)的基因转移到另外一种生物(受体)中去,从而实现受体生物的定向改造与改良。
遗传工程:广义:指以改变生物有机体性状为目标,采用类似工程技术手段而进行的对遗传物质的操作,以改良品质或创造新品种。
包括细胞工程、染色体工程、细胞器工程和基因工程等不同的技术层次。
狭义:基因工程。
限制性核酸内切酶:是可以识别DNA的特异序列,并在识别位点或其周围切割双链DNA的一类内切酶,简称限制酶回文结构:每条单链以任一方向阅读时都与另一条链以相同方向阅读时的序列是一致的,例如5'GGTACC3' 3'CCATGG5'.同裂酶(isoschizomer)或异源同工酶:不同来源的限制酶可切割同一靶序列(BamH I 和Bst I具有相同的识别序列G↓GATGC)同尾酶(isocaudiners):来源不同、识别序列不同,但产生相同粘性末端的酶。
两个同尾酶形成的黏性末端连接之后,一般情况下连接处不能够再被其任何一种同尾酶识别。
BamH I 识别序列: G↓GATCCBgl II 识别序列: A↓GATCT黏性末端 (cohesive terminus/sticky ends):DNA末端一条链突出的几个核苷酸能与另一个具有突出单链的DNA末端通过互补配对粘合,这样的DNA末端,称为黏性末端。
平末端(blunt ends): DNA片段的末端是平齐的。
星活性(star activity):指限制性内切酶在非标准条件下,对与识别序列相似的其它序列也进行切割反应,导致出现非特异性的DNA片段的现象。
易产生星活性的内切酶用*标记。
如:EcoR I*底物位点优势效应:酶对同一个DNA底物上的不同酶切位点的切割速率不同。
连杆/衔接物(linker):化学合成的8~12个核苷酸组成的寡核苷酸片段。
酶切位点保护碱基表

酶切位点保护碱基表:PRC引物保护碱基的设计首先要明确什么是保护碱基限制性内切酶识别特定的DNA序列,除此之外,酶蛋白还要占据识别位点两边的若干个碱基,这些碱基对内切酶稳定的结合到DNA双链并发挥切割DNA作用是有很大影响的,被称为保护碱基。
添加保护碱基的目的在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。
但实验证明,大多数限制酶对裸露的酶切位点不能切断。
必须在酶切位点旁边加上一个至几个保护碱基,才能使所定的限制酶对其识别位点进行有效切断。
因此在设计PCR引物时,为保护5` 端外加的内切酶识别位点,人为地在酶切位点序列的5‘端外侧添加额外的碱基序列,即保护碱基,用来提高酶切时的活性,使酶切完全。
其次,在分子克隆实验中选择载体的酶切位点时,相临的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。
添加保护碱基的原则添加保护碱基,需要考虑两个因素:一是碱基数目,一是碱基种类。
添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。
什么样的酶切位点,添加几个保护碱基,是有数据可以参考的。
一般情况下,普通的内切酶只加入两个保护碱基,其内切反应就可以正常进行;而有一类,仅仅只加入两个保护碱基,其内切反应就不能正常进行,这是因为内切酶不能正常结合DNA段上。
如NdeI就属这类,需要加入至少6个保护碱基,常用的HindIII也要三个。
添加什么保护碱基,如果严格点,是根据两条引物的Tm值和各引物的碱基分布及GC含量。
如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
为了解不同内切酶对识别位点以外最少保护碱基数目的要求,NEB采用了一系列含识别序列的短双链寡核苷酸作为酶切底物进行实验。
实验结果对于确定双酶切顺序将会有帮助(比如在多接头上切割位点很接近时),或者当切割位点靠近DNA末端时也很有用。
在本表中没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基,以确保酶切反应的进行。
酶切分析实验报告

一、实验目的1. 理解限制性核酸内切酶的原理及其在基因工程中的应用。
2. 掌握质粒DNA的提取方法。
3. 学习使用限制性核酸内切酶进行DNA片段的切割。
4. 通过琼脂糖凝胶电泳分析酶切结果,鉴定目的DNA片段。
二、实验原理限制性核酸内切酶(Restriction Endonucleases)是一类能够识别特定DNA序列并在该序列的特定位置切割双链DNA的酶。
在基因工程中,限制性核酸内切酶用于切割DNA分子,以便进行进一步的克隆、测序或分子标记等操作。
本实验中,我们使用限制性核酸内切酶切割质粒DNA,并通过琼脂糖凝胶电泳分析酶切产物。
根据酶切位点的不同,质粒DNA会被切割成不同长度的片段。
通过比较酶切前后的DNA片段,可以鉴定目的DNA片段。
三、实验材料1. 质粒DNA2. 限制性核酸内切酶3. 琼脂糖4. 电泳缓冲液5. 标准DNA分子量标记6. 琼脂糖凝胶电泳仪7. 凝胶成像系统四、实验步骤1. 质粒DNA提取:根据试剂盒说明书提取质粒DNA。
2. 酶切反应:将提取的质粒DNA与限制性核酸内切酶混合,加入缓冲液,进行酶切反应。
3. 琼脂糖凝胶电泳:- 准备琼脂糖凝胶,加入电泳缓冲液。
- 在凝胶孔中加入标准DNA分子量标记和酶切后的质粒DNA。
- 连接电源,进行电泳。
- 电泳完成后,关闭电源,取出凝胶。
4. 凝胶成像与分析:- 使用凝胶成像系统观察电泳结果。
- 根据标准DNA分子量标记,分析酶切产物的长度。
- 比较酶切前后的质粒DNA片段,鉴定目的DNA片段。
五、实验结果与分析1. 质粒DNA提取:成功提取出质粒DNA,通过紫外分光光度计检测,A260/A280比值在1.8-2.0之间。
2. 酶切反应:限制性核酸内切酶成功切割质粒DNA,产生不同长度的片段。
3. 琼脂糖凝胶电泳:- 电泳结果显示,酶切后的质粒DNA片段在凝胶上呈现清晰的条带。
- 通过比较标准DNA分子量标记和酶切产物,可以确定酶切位点的位置。
tev酶切盐浓度-概述说明以及解释

tev酶切盐浓度-概述说明以及解释1.引言1.1 概述酶切是分子生物学中常用的实验技术之一,是将DNA或RNA的特定序列切割成较小的片段的过程。
为了进行酶切反应,常常需要使用特定的酶来识别和切割目标序列。
其中,Tev酶被广泛应用于DNA分子的酶切。
Tev酶(tev endonuclease)是Tobacco Etch Virus (TEV) 的内切酶,能够识别特定的DNA序列并在特定位置切割。
这使得Tev酶在分子生物学实验中广受欢迎。
然而,在使用Tev酶进行酶切反应时,酶切盐浓度的调节是一个非常重要的因素。
盐浓度是指反应体系中所含盐类的浓度。
在Tev酶的酶切反应中,盐浓度的调节可以影响酶的活性和特异性。
过高或过低的盐浓度都可能影响到酶切反应的效果。
较高的盐浓度可能干扰酶与底物之间的相互作用,限制了酶的结合和切割效率。
而较低的盐浓度可能使得酶分子间的相互作用减弱,影响到酶的稳定性和活性。
因此,为了获得较好的酶切效果,需要适当调节酶切反应中的盐浓度。
目前,关于盐浓度对Tev酶切反应的影响还存在一些争议。
一些研究表明,适当的盐浓度可以提高Tev酶的活性和特异性,从而增强酶切反应的效果。
然而,也有研究发现,过高的盐浓度可能抑制酶活性,而过低的盐浓度则会降低酶的稳定性。
基于对盐浓度对Tev酶切反应的影响尚存在争议的情况,本文旨在系统地综述盐浓度对Tev酶切反应的影响,并探讨不同盐浓度下酶切效果的差异。
通过研究和总结现有的文献,我们希望可以为使用Tev酶进行酶切反应提供一定的指导,同时也为进一步的研究提供新的思路和方向。
1.2文章结构文章结构部分是对整篇文章的组织和内容进行概述,帮助读者了解文章的章节和内容安排。
在这篇长文中,文章结构如下:1. 引言1.1 概述1.2 文章结构1.3 目的2. 正文2.1 酶切反应简介2.2 盐浓度对酶切反应的影响3. 结论3.1 盐浓度对tev酶切的影响总结3.2 进一步研究的方向在引言部分,我们将提供关于tev酶切盐浓度研究的背景和重要性。
多克隆位点名词解释

多克隆位点名词解释多克隆位点(Multiple Cloning Site,MCS)是在遗传工程中常用的一个术语,指的是一个DNA分子中含有多个不同的酶切位点,使得该分子能够被多种限制性内切酶切割。
多克隆位点通常用于构建重组DNA或载体,方便插入外源DNA片段并进行转化、复制和表达。
多克隆位点通常位于载体的靶标基因的上游或下游,主要功能是提供多个不同的酶切位点,使得在插入外源DNA片段时能够选择合适的酶切位点进行剪切和连接。
多克隆位点的设计要求满足以下几个条件:1. 含有多个不同的酶切位点:多克隆位点通常具有3-10个不同的酶切位点,使得可以选择性地在不同的位点进行剪切和连接。
这样可以方便地构建不同的重组DNA。
2. 位点之间无重叠:不同酶切位点之间应该没有重叠,以避免在连接过程中发生自身连接而形成侧重组。
3. 位点序列简短:多克隆位点的序列应该尽可能简短,以节省载体的长度,并且减少潜在的序列重复导致重组发生的风险。
多克隆位点的常见酶切位点包括但不限于以下几种:1. 入站启动子(Promoter):用于启动表达载体上游的外源基因的转录活性。
2. 5'末端:常见的酶切位点包括NdeI、EcoRI、BamHI等,用于插入外源DNA时创建外源基因的5'末端。
3. 3'末端:常见的酶切位点包括XhoI、HindIII、SalI等,用于插入外源DNA时创建外源基因的3'末端。
4. 多聚链终止序列(Polylinker Site):是多克隆位点最常见的组成部分,其上含有多个不同的酶切位点,如EcoRI、NotI、XbaI等,方便构建重组DNA。
5. 稳定性序列:用于维持载体在细胞中的稳定复制。
常见的酶切位点包括AmpR(抗生素阻抗基因)和ColE1(能源复制基因)等。
总之,多克隆位点在遗传工程中扮演着重要的角色,提供了方便快捷的方式构建重组DNA和表达外源基因。
通过选择合适的酶切位点,在插入外源DNA片段时能够灵活地进行剪切和连接,从而实现特定基因的导入、表达和研究。
质粒双酶切

质粒双酶切
质粒双酶切是分子生物学领域中常用的技术之一。
质粒是一种环状的DNA分子,常用于重组DNA和基因工程研究中。
质粒双酶切是利用限制性内切酶对质粒DNA进行切割,使得质粒分子上产生单一或多个切口。
通过不同的酶切位点选取,可以得到不同的切割模式和切割产物。
这种技术可以用于构建重组DNA、基因克隆、DNA序列分析等实验中。
在实验过程中,需要注意质粒DNA的纯度和酶切条件的选择。
质粒双酶切技术的广泛应用,推动了分子生物学和基因工程领域的发展。
- 1 -。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
ApaI (类型:Type II restriction enzyme )识别序列:5'GGGCC^C 3' BamHI(类型:Type II restriction enzyme )识别序列:5' G^GATCC 3' BglII (类型:Type II restriction enzyme )识别序列:5' A^GATCT 3' EcoRI (类型:Type II restriction enzyme )识别序列:5' G^AATTC 3' HindIII (类型:Type II restriction enzyme )识别序列:5' A^AGCTT 3' KpnI (类型:Type II restriction enzyme )识别序列:5' GGTAC^C 3' NcoI (类型:Type II restriction enzyme )识别序列:5' C^CATGG 3' NdeI (类型:Type II restriction enzyme )识别序列:5' CA^TATG 3' NheI (类型:Type II restriction enzyme )识别序列:5' G^CTAGC 3' NotI (类型:Type II restriction enzyme )识别序列:5' GC^GGCCGC 3' SacI (类型:Type II restriction enzyme )识别序列:5' GAGCT^C 3' SalI (类型:Type II restriction enzyme )识别序列:5' G^TCGAC 3' SphI (类型:Type II restriction enzyme )识别序列:5' GCATG^C 3' XbaI (类型:Type II restriction enzyme )识别序列:5' T^CTAGA 3' XhoI (类型:Type II restriction enzyme )识别序列:5' C^TCGAG 3'
Trx-tag硫氧还蛋白标签是个融合标签,有利于蛋白可溶性表达。
His-tag是纯化标签,能与Ni柱亲和层析,便于下游蛋白纯化,2个His-tag分别设计在N,C 端、自己通过选择酶切位点选择其中的一个就可以。
S-tag是用于做western blotting 分子杂交鉴定的,能与Anti-S-tag产生抗原抗体反应。
thrombin 不是凝血酶,是凝血酶酶切位点,enterokinase也不是肠激酶,而是肠激酶酶切位点,因为pET32是融合表达载体,为了让你可以得到非融合的目标蛋白,设计了这两个蛋白酶酶切位点,用于表达后的融合标签去除。
