作系统进化树的方法
upgma系统发育构建原理

upgma系统发育构建原理UPGMA(Unweighted Pair Group Method with Arithmetic Mean)是一种常用的系统发育构建原理,它通过计算物种之间的遗传距离来构建进化树。
UPGMA方法在遗传距离不准确或存在误差的情况下,可以得到一棵近似的进化树。
UPGMA方法的基本思想是将物种按照遗传距离的大小进行聚类,每次将距离最近的两个物种合并成一个新的聚类,直到所有物种都聚类为一棵进化树为止。
这种方法假设进化速率是恒定的,并且所有物种是等距离分布的。
我们需要计算物种之间的遗传距离。
遗传距离是通过测量不同物种之间的遗传差异来计算的,可以使用分子标记数据(如DNA序列或蛋白质序列)来进行计算。
然后,根据遗传距离构建一个距离矩阵,矩阵中的每个元素表示两个物种之间的遗传距离。
接下来,我们需要选择距离最近的两个物种进行合并。
合并的原则是选择距离最近的两个物种,计算它们的平均距离,并将它们合并成一个新的聚类。
新的聚类将代替原来的两个物种,并更新距离矩阵中的相应值。
然后,重复上述步骤,直到所有物种都被聚类为止。
每次合并两个物种后,都会更新距离矩阵中的值。
最终,我们将得到一棵进化树,树的叶子节点代表每个物种,内部节点代表聚类。
UPGMA方法的优点是简单易用,特别适用于物种较少且进化速率基本恒定的情况。
然而,UPGMA方法对遗传距离的准确性要求较高,如果遗传距离存在误差或不准确,可能会导致构建的进化树出现偏差。
UPGMA方法是一种常用的系统发育构建原理,通过计算物种之间的遗传距离来构建进化树。
它的基本思想是将距离最近的两个物种合并成一个新的聚类,直到所有物种都聚类为一棵进化树。
UPGMA方法简单易用,适用于物种较少且进化速率恒定的情况。
然而,它对遗传距离的准确性要求较高,可能会受到误差的影响。
菌株系统进化树的构建-概述说明以及解释

菌株系统进化树的构建-概述说明以及解释1.引言1.1 概述概述菌株系统进化树的构建是一项重要的研究工作,它能够帮助我们了解不同菌株之间的进化关系和演化历史。
菌株系统进化树可以被看作是一种表示不同菌株间亲缘关系的有向无环图,它能够揭示这些菌株之间的共同祖先和演化路径。
菌株系统进化树是基于菌株间的遗传差异来构建的。
通过对不同菌株的基因组、基因序列和遗传标记进行比较分析,我们可以获得它们之间的遗传距离或相异度。
这些数据可以用来构建菌株系统进化树,从而揭示菌株间的进化关系。
构建菌株系统进化树的过程通常包括以下几个步骤:首先收集不同菌株的样本,提取其基因组或基因序列;然后对这些样本进行测序并得到相应的遗传数据;接着利用生物信息学方法对这些数据进行分析和比较,计算出菌株间的遗传距离;最后利用分子进化模型和统计方法构建进化树,并对其进行进一步的验证和分析。
菌株系统进化树的构建具有重要的应用价值。
首先,它可以帮助我们确定不同菌株之间的亲缘关系,进一步理解它们之间的演化过程和机制。
其次,菌株系统进化树可以为微生物分类学和菌群动态变化研究提供重要的参考和指导。
此外,对于研究菌株的致病性、抗药性和生物学特性等方面,菌株系统进化树也具有重要意义。
综上所述,构建菌株系统进化树是一个重要而复杂的研究课题。
通过比较和分析菌株间的遗传数据,我们可以揭示菌株间的亲缘关系和进化历史,进一步推动微生物学和生物进化学的发展。
在接下来的内容中,我们将详细介绍构建菌株系统进化树的方法和应用,以及对未来研究的展望。
1.2 文章结构文章结构是指文章的组织框架和各个部分的排列顺序。
一个良好的文章结构能够帮助读者更好地理解和掌握文章的内容,并且能够使文章的逻辑关系更加清晰和流畅。
本文的结构分为引言、正文和结论三个部分,具体如下:引言部分(Introduction):在引言部分,首先要对菌株系统进化树的概念进行介绍,解释其所涉及的基本概念和理论背景。
构建系统发生树的方法

构建系统发生树的方法构建系统发生树是一种对于系统进行分析和优化的有效方法,在实践中有许多种方法可以构建系统发生树。
以下是10条关于构建系统发生树的方法,并对每条方法进行详细描述。
1. 系统流程图系统流程图是一种常见的构建系统发生树的方法。
通过对系统的主要流程进行图形化的描述,可以更好地了解系统的组成部分以及它们之间的关系。
系统流程图往往是由开始和结束节点、处理节点和决策节点组成的。
前者用来表示系统的输入和输出,后者则用来表示系统的核心过程和逻辑判断。
2. 系统分层结构图系统分层结构图是将系统按照层次进行组织和描述的一种方法。
通过将系统分解为多个层次,并描述这些层次之间的关系,可以更好地了解系统的组成和结构。
这种方法通常用于处理大型和复杂的系统,能够帮助开发人员更好地管理和优化系统。
3. 系统模块图系统模块图是一种用于展示系统模块和它们之间关系的图形化表示方法。
系统模块图通常由多个模块和模块之间的输入和输出组成。
每个模块通常都对应一个特定的功能或业务逻辑。
通过了解系统中各个模块之间的关系和作用,可以更好地理解系统的架构和逻辑。
4. 系统数据流图系统数据流图是一种用来描述系统数据传输流程的图形化表示方法。
该方法通常由多个数据流和与这些数据流相关的处理过程组成。
每个数据流都对应一个特定的数据,而每个处理过程通常都包含两个或多个数据流。
通过了解系统中各个数据流之间的关系和流动过程,可以更好地理解系统的功能和性能。
5. 系统性能图系统性能图是一种用于展示系统性能指标和变化趋势的图形化表示方法。
该方法通常包括多个参数和变量,比如系统响应时间、吞吐量、并发数等。
通过了解系统性能参数的表现和变化趋势,可以更好地理解系统的性能瓶颈和瓶颈优化的方向。
6. 事件序列图事件序列图是一种用于展示系统中事件和处理过程之间关系的图形化表示方法。
该方法通常由一个或多个故障事件和与之相关的处理过程组成。
通过了解系统中各个事件和处理过程之间的关系,可以更好地了解系统的运行过程和故障排查过程。
分子系统发育树构建的简易方法

分子系统发育树构建的简易方法
分子系统发育树的构建是根据分子序列的差异来推断不同物种之间的进化关系。
下面是一个简易的分子系统发育树构建方法:
1. 选择目标基因序列:选择与所研究物种相关的基因序列(如核糖体RNA或蛋白质编码基因)作为目标序列。
2. 数据收集:收集各个相关物种的目标基因序列数据。
可以通过公共数据库(如NCBI)或研究文献中的已有数据进行获取。
3. 序列比对:使用序列比对软件将收集到的序列进行比对,找出相同和不同的碱基或氨基酸位置。
常用的比对软件有CLUSTALW和MAFFT。
4. 构建进化树:根据序列比对结果,使用进化树构建软件(如MEGA)进行系统发育树的构建。
常用的进化树构建方法包括最大简约法(UPGMA)和最大似然法(ML)。
5. 进化树评估:对构建的系统发育树进行评估,可以使用Bootstrap方法进行支持值分析,提高树的可靠性。
6. 结果解读:根据构建的系统发育树,可以解读不同物种之间的进化关系和群体间的分化程度。
需要注意的是,分子系统发育树是基于目标基因序列的进化关系推断,仅仅代表目标基因的进化历史,并不一定能完全反映
整个物种的进化历史。
因此,在研究中还需要综合考虑其他重要因素,如形态特征和生态行为等。
如何用MEGA5.0和Clustalx1.83构建进化树

如何用MEGA5.0和Clustalx1.83构建进化树MEGA是一个关于序列分析以及比较统计的工具包,从3.1版本到后来的4.0版本一直都广为大家熟悉,现在推出了Mega5.0版本。
功能比以前多有改进。
现主要介绍使用Mega 5.0构建系统进化树的方法。
供大家参考。
用MEGA构建进化树有以下步骤:1、测序:将克隆扩增测序得到的16S rDNA序列进行测序。
2、NCBI上做Blast/blast/Blast.cgi找到相似度最高的几个序列,确定一下你分离的细菌大约属于哪个科哪个属,如果相似度达到百分之百那基本可以确定你分离得到的就是Blast到的那个,然后寻找相似性最高的细菌,通常把该属的序列(Fasta格式文件)下载下来,或点击GenBank登录号,复制FSA TA 格式,整合在一个*.txt文档中(单独建立一个文件夹存放,后面的很多文件会自动装入该文件夹),如>XXXXAGGCTTAACACA TGCAAGTCGAGCGGAGCGAGGGTGCTTGCACCTTAGCTTAGCGGCG GACGGGTGAGTAA TGCTTAGGAA TCTGCCTA TTAGTGGGGGACAACA TTCCGAAAGGA A TGCTAA TACCGCA TACGCCCTACGGGGGAAAGCAGGGGA TCTTCGGACCTTGCGCTAA TAGA TGAGCCTAAGTCGGA TTAGCTAGTTGGTGGG>gi|289469964|gb|GU388381.1| Acinetobacter tandoii strain DSM 14970 16S ribosomal RNA gene, partial sequenceACTTAGCGGCGGACGGGTGAGTAA TGCTTAGGAA TCTGCCTA TTAGTGGGGGACAACA TTCCGAAAGGGA TGCTAA TACCGCA TACGCCCTACGGGGGAAAGCAGGGGA TCTTCGG ACCTTGCGCTAA TAGA TGAGCCTAAGTCGGA TTAGCTAGTTGGTGGGGTAAAGGCCTAC CAAGGCGACGA TCTGTAGCGGGTCTGAGAGGA TGA………………………….参考序列选择注意事项:1、不选非培养(unclutured)微生物为参比;2、不选未定分类地位的微生物,最相近的仅作参考;c,在保证同属的前提下,优先选择16S rDNA全长测序或全基因组测序的种;d,每个种属选择一个参考序列,如果自己的序列中同一属的较多,可适当选择两个参考序列。
分子进化学中的进化树构建方法

分子进化学中的进化树构建方法随着科技的进步和生物技术的广泛应用,分子生物学的研究逐渐深入,成为生物学、生物技术和医药学等领域的重要研究方向。
而分子进化学作为分子生物学中的一个重要分支,研究物种间的分子差异和进化关系。
其中,构建进化树是分子进化学研究中的重要工作,下面我们来了解一下进化树构建的方法。
一、进化树的基本概念进化树是描述不同物种、不同基因或不同蛋白质之间进化关系的图形化表示。
在进化树中,每一个分支代表了一个物种、一个基因或一个蛋白质序列,分支的长度表示了物种、基因或序列的进化距离,而进化距离则是衡量不同物种或不同序列之间关系的基本参数。
而构建进化树的过程则是根据分子序列数据的重构得到物种或基因的进化树。
二、进化树的构建方法构建进化树有多种方法,主要有距离矩阵法、系统发育学法、最大似然法和贝叶斯法等。
下面我们逐一介绍这些方法的基本原理。
1.距离矩阵法距离矩阵法是最早采用的一种构建进化树的方法,它基于序列之间的距离矩阵计算和聚类方法来得到进化树。
该方法首先计算所有分子序列之间的距离(距离可由序列相似性计算得出),然后根据聚类方法构建进化树。
聚类方法包括单链接聚类、均链接聚类和最大链接聚类等。
距离矩阵法的优点是构建速度快、适用性广,但是对于高变异的序列来说,该方法可能会产生误导性的结果。
2.系统发育学法系统发育学法是基于系统学原理,采用系统发生学的理论和方法来构建进化树。
该方法主要是通过分子序列的相似性构建系统发育分析矩阵,然后利用不同的计算方法(如UPGMA、NJ和ML等)推断进化树。
系统发育学法的优点是能够更准确地反映分子序列的演化,并且可以通过不同的方法比较结果,但是该方法需要大量的计算资源和长时间的计算。
3.最大似然法最大似然法是一种统计学上的方法,通过最大化序列数据与观测数据的相似度,来推断出最可能的进化树。
该方法需要整合进化模型和数据,然后计算不同进化模型下数据的似然函数,最终选择似然度最大的进化树。
构建系统发育树的三大方法

构建系统发育树的三大方法
1、距离法:基于距离的方法,首先通过各个物种之间的比较,根
据一定的假设(进化距离模型)推导得出分类群之间的进化距离,构建一个进化距离矩阵。
进化树的构建则是基于这个矩阵中的进化距离关系。
2、特征法:基于特征的方法,不计算序列间的距离,而是将序列
中有差异的位点作为单独的特征,并根据这些特征来建树。
3、简约法:基于简约的方法,通过构建一棵由所有可能的子树组
成的树,然后从这个树中选择一个最优的子树作为进化树。
系统发育树构建的三种方法

系统发育树构建的三种方法
系统发育树(Systems 发育 Tree,简称Stree)是一种用于描述生物系统进化的图形化工具,通常用于模拟生物系统行为的演化过程。
以下是三种构建系统发育树的方法:
1. 基于规则的方法:这种方法使用预定义的规则和偏好来构建
系统发育树。
例如,可以使用遗传算法或人工神经网络等机器学习方法,来预测一个物种的遗传特征或行为演化轨迹。
这种方法需要大量
的人工工作,但可以生成较为准确的演化树。
2. 基于统计方法的方法:这种方法使用统计学方法来推断物种
之间的演化关系。
例如,可以使用最大似然估计或贝叶斯推断等方法,来预测一个物种的遗传特征或行为演化轨迹。
这种方法不需要人工工作,但需要更多的计算资源和时间,才能得到比较准确的演化树。
3. 基于模型的方法:这种方法使用已经建立的模型和数据来构
建系统发育树。
例如,可以使用层次结构模型(如生物进化树、社会网络模型等)来预测一个物种的遗传特征或行为演化轨迹。
这种方法可
以快速构建系统发育树,但需要更多的人工工作来验证模型的准确性。
系统发育树构建方法优劣

1.邻接法邻接法(neighbor-joiningmethod,NJ)由Saitou和Nei(1987)提出,NJ法是基于最小进化原理经常被使用的一种算法,它不检验所有可能的拓扑结构,能同时给出拓扑结构和分支长度。
在重建系统发生树时,它取消了UPGMA法所做的假定,认为在进化分支上,发生趋异的次数可以不同。
最近的计算机模拟已表明它是最有效的基于距离数据重建系统树的方法之一。
该方法通过确定距离最近(或相邻)的成对分类单位来使系统树的总距离达到最小。
它的特点是重建的树相对准确,假设少,计算速度快,只得一棵树。
其缺点主要表现在将序列上的所有位点等同对待,且所分析序列的进化距离不能太大。
故NJ法适用于进化距离不大,信息位点少的短序列。
邻接法在距离建树中经常会用到,而不用理会使用什么样的优化标准。
完全解析出的进化树是通过对完全没有解析出的“星型”进化树进行“分解”得到的,分解的步骤是连续不断地在最接近(实际上是最孤立的)的序列对中插入树枝,而保留进化树的终端。
于是,最接近的序列对被巩固了,而“星型”进化树被改善了,这个过程将不断重复。
这个方法相对而言很快,也就是说,对于一个50个序列的进化树,只需要若干秒甚至更少。
2.最大简约法最大简约法(maximum parsimony method,MP)最早是基于形态特征分类的需要发展起来的,具体的算法有许多不同版本,其中有些已被广泛地应用于分子进化研究中。
利用MP方法重建系统发生树,实际上是一个对给定OTUs其所有可能的树进行比较的过程。
对某一个可能的树,首先对每个位点祖先序列的核苷酸组成做出推断,然后统计每个位点用来阐明差异的核苷酸最小替换数目。
在整个树中,所有信息简约位点最小核苷酸替换数的总和称为树的长度(常青和周开亚,1998)。
MP法是一种优化标准,这种标准遵循“奥卡姆剃刀原则(Occam’S Razor principle)”:对数据最好的解释也是最简单的,而最简单的所需要的特别假定也最少。
MEGA构建系统进化树的步骤(以MEGA7为例)

MEGA构建系统进化树的步骤(以MEGA7为例)本文是看中国慕课山东大学生物信息学课程总结出来的分子进化的研究对象是核酸和蛋白质序列。
研究某个基因的进化,是用它的DNA序列,还是翻译后的蛋白质序列呢?序列的选取要遵循以下原则:1)如果DNA序列的两两间的一致度≥70%,选用DNA 序列。
因为,如果DNA序列都如此相似,它的蛋白质会相似到看不出区别,这对构建系统发生树是不利的。
所以这种情况下应该选用DNA序列,而不选蛋白质序列。
2)如果DNA序列的两两间的一致度≤70%,DNA序列和蛋白质序列都可以选用。
1. 将要用于构建系统进化树的所有序列合并到同一个fasta格式文件,注意:所有序列的方向都要保持一致( 5’-3’)。
想要做系统发生树先要做多序列比对,然后把多序列比对的结果提交给建树软件进行建树,所以在用MEGA建树时可以输入一个已经比对好的多序列比对,也可以输入一条原始序列,让MEGA先来做多序列比对,再建树(一般我们都是原始序列)。
所以我们以后者为例。
2.打开MEGA软件,选择主窗口的”File”→“Open A File”→找到并打开fasta文件,这时会询问以何种方式打开,我们是原始序列,需要先进行多序列比对,所以选择“Align”。
如果是比对好的多序列比对可以直接选择“Analyze”。
3.在打开的Alignment Explorer窗口中选择”Alignment”-“Align by ClustalW”进行多序列比对(MEGA提供了ClustalW和Muscle两种多序列比对方法,这里选择熟悉的ClustalW),弹出窗口询问“Nothing selected for alignment,Select all?”选择“OK”。
4. 之后,弹出多序列比对参数设置窗口。
这个窗口和EMBL在线多序列比对一样,可以设置替换记分矩阵、不同的空位罚分(罚分填写的是正数,计算时按负数计算)等参数。
手把手教你构建系统进化树

生物多样性的研究
生物多样性起源
系统进化树有助于研究生物多样性的起源和演化,了解不同物种的起源和演化历程。
生物多样性分布
通过分析不同地区或生态系统中的系统进化树,可以研究生物多样性的地理分布和生态分布。
生物地理学的研究
物种分布
系统进化树揭示了物种的分布特征和演化历程,有助于研究物种分布的规律和机制。
蛋白质结构预测
结合蛋白质结构预测技术, 从蛋白质结构层面揭示物 种间的进化关系。
新的解读方法的研究
树的可视化
研究如何将进化树以更直观、易懂的方式呈现, 帮助用户更好地理解物种间的进化关系。
树的意义
探索进化树在生物多样性保护、生物进化研究等 方面的实际应用价值。
树的可解释性
研究如何将进化树的构建过程和结果以可解释的 方式呈现,提高用户对进化树的理解和信任。
03 常用的构建系统进化树的 方法
Neighbor-Joining方法
总结词
基于距离矩阵的构建方法
详细描述
Neighbor-Joining方法是一种基于距离矩阵的进化树构建方法,通过比较不同物种之 间的进化距离,将距离最近的两个物种先聚类在一起,然后逐步添加其他物种,直到所
有物种都被包含在进化树中。
树的构建
树构建
详细描述:在完成序列比对后,可以使用各 种算法和软件工具来构建系统进化树。常用 的方法有距离矩阵法和最大似然法等。这些 方法基于不同的原理和假设,可以根据具体 情况选择适合的方法。构建系统进化树的过 程通常需要多次迭代和优化,以确保树的准
确性和可靠性。
树的优化
优化调整
VS
详细描述:在初步构建出系统进化树 后,需要进行优化调整。这一步骤包 括对树的布局、分支长度和节点标注 等进行调整,以提高树的易读性和可 解释性。此外,还可以使用各种软件 工具和可视化技术来增强树的可视化 效果和交互性。
MEGA构建系统进化树的步骤(以MEGA7为例)

MEGA构建系统进化树的步骤(以MEGA7为例)本文是看中国慕课山东大学生物信息学课程总结出来的分子进化的研究对象是核酸和蛋白质序列。
研究某个基因的进化,是用它的DNA序列,还是翻译后的蛋白质序列呢?序列的选取要遵循以下原则:1)如果DNA序列的两两间的一致度≥70%,选用DNA 序列。
因为,如果DNA序列都如此相似,它的蛋白质会相似到看不出区别,这对构建系统发生树是不利的。
所以这种情况下应该选用DNA序列,而不选蛋白质序列。
2)如果DNA序列的两两间的一致度≤70%,DNA序列和蛋白质序列都可以选用。
1. 将要用于构建系统进化树的所有序列合并到同一个fasta格式文件,注意:所有序列的方向都要保持一致( 5’-3’)。
想要做系统发生树先要做多序列比对,然后把多序列比对的结果提交给建树软件进行建树,所以在用MEGA建树时可以输入一个已经比对好的多序列比对,也可以输入一条原始序列,让MEGA先来做多序列比对,再建树(一般我们都是原始序列)。
所以我们以后者为例。
2.打开MEGA软件,选择主窗口的”File”→“Open A File”→找到并打开fasta文件,这时会询问以何种方式打开,我们是原始序列,需要先进行多序列比对,所以选择“Align”。
如果是比对好的多序列比对可以直接选择“Analyze”。
3.在打开的Alignment Explorer窗口中选择”Alignment”-“Align by ClustalW”进行多序列比对(MEGA提供了ClustalW和Muscle两种多序列比对方法,这里选择熟悉的ClustalW),弹出窗口询问“Nothing selected for alignment,Select all?”选择“OK”。
4. 之后,弹出多序列比对参数设置窗口。
这个窗口和EMBL在线多序列比对一样,可以设置替换记分矩阵、不同的空位罚分(罚分填写的是正数,计算时按负数计算)等参数。
生物大数据技术的进化树构建方法与工具

生物大数据技术的进化树构建方法与工具随着现代生物学研究范式不断发展,生物大数据成为生物学研究的重要资源。
在生物大数据中,进化树构建是解决物种分类和亲缘关系的关键环节之一。
进化树提供了生物物种之间的演化关系,帮助我们理解生物多样性的起源和演化过程。
在本文中,我将介绍生物大数据技术中用于构建进化树的方法与工具。
进化树构建的方法包括距离法、最大简约法和贝叶斯法等。
距离法是一种基于物种间差异的测量方法,常用的距离指标有进化距离、遗传距离和相似性距离等。
最大简约法则基于进化过程中最简单的演化树,寻找一棵树,使得所有的观察数据与这棵树的解释最为一致。
贝叶斯法是一种基于概率统计的方法,利用贝叶斯统计推断物种之间的关系,它可以通过蒙特卡罗马尔科夫链蒙特卡罗(MCMC)方法来求解。
生物大数据技术的进化树构建方法中有许多重要的工具。
其中,最广泛使用的方法之一是分子系统学。
分子系统学利用生物大数据中的遗传序列信息来构建进化树,最常用的序列包括基因组序列和蛋白质序列。
常见的分子系统学工具有MEGA、PHYLIP、RAxML和MrBayes等。
MEGA是一个综合的分子进化分析软件,集成了多种进化模型和构建方法。
PHYLIP是最早的公开可用的构建进化树的软件包,其中包含了多种构建方法和分析工具。
RAxML是一种用于大规模物种分类研究的软件,它具有高效的计算性能和准确的模型选择。
MrBayes是一种基于贝叶斯统计学的软件,能够估计单个和多个基因的进化树。
此外,还有一些新兴的工具用于生物大数据中进化树的构建。
一种常见的方法是使用基于物种演化树的软件包,例如ASTRAL和PhyloNet。
ASTRAL利用结合物种组织树关系和基因树关系的联合推断来构建物种进化树,它能够处理物种树混淆或基因树不完整的情况。
PhyloNet是一种基于网络理论和统计学的方法,可以推断出复杂的物种进化网络,包括基因水平的基因转移和混合。
除了这些方法和工具外,还有一些改进的技术被用于生物大数据中的进化树构建。
回顾系统进化树构建的常见方法

回顾系统进化树构建的常见方法构建进化树的方法主要分为1.距离矩阵法(含 UPGMA、ME、NJ等)2.最大简约法(MP)3.极大似然法(ML)4.贝叶斯法(Bayesian)基于距离的进化树构建方法常用的距离法构建系统树:1.不加权算术平均对方法(Unweighted Pair-Group Method using Arithmetic average, UPGMA)2.最小进化距离法(Mnimal Evolution Method)3.邻接法(Neighbor-Joining Method, NJ)这一系列方法主要考量参数是:1.如何计算距离,节点间的距离;cluster之间的距离;校正的距离2.如何聚类?UPGMA 法本质上是“自下而上”或者说“聚合”的层次聚类(hclust)法,且距离的计算采用“平均距离法”。
一般绘制热图,常见的表达模式聚类方法也是 hclust,往往默认“最长距离法”。
两者只是cluster之间距离的计算方式不同。
当所有分支的突变率相同,UPGMA效果较好。
最小进化法(ME)寻找某一进化树的拓扑结构,使得全树枝长总和最短。
逻辑上需要对每一个拓扑结构进行评估,当序列增加时,计算量暴增。
这个与后续提到的最大简约法(MP)的最大区别是:(1)ME 法直接基于一个距离矩阵,计算的是最终进化树枝长总和最小;(2)MP法直接基于序列,计算的最终是在当前拓扑结构下,所有序列需要发生突变的位点的总和最少。
邻接法(NJ)与UPGMA几乎相反,UPGMA约等于层次聚类的聚合法;而NJ 法从拓扑结构的变化上来看,与层次聚类的分离法比较像。
当然还是有比较大的区别。
层次聚类的分离实现考量的是分离后两个cluster的内部距离总和最小。
NJ法考量的是分离出来的两个leaf node的校正距离最小。
这一校正距离综合考量了每个leaf node以及cluster的距离。
在距离计算上的实现,逻辑上比层次聚类的分离法要简单一些。
MEGA软件——系统发育树构建方法
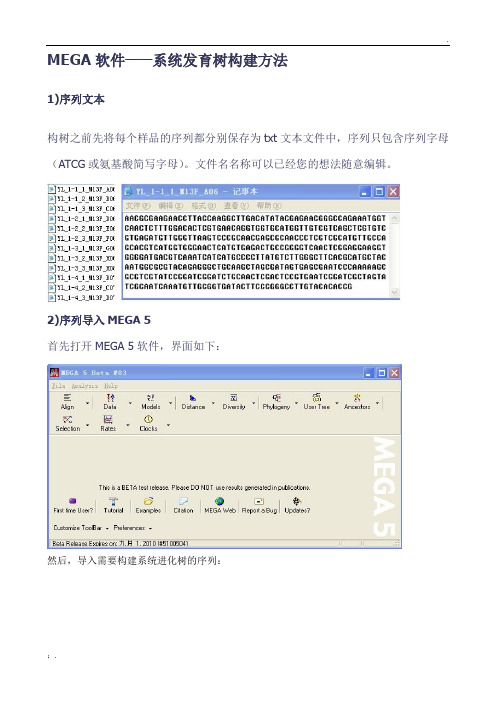
MEGA软件——系统发育树构建方法1)序列文本构树之前先将每个样品的序列都分别保存为txt文本文件中,序列只包含序列字母(ATCG或氨基酸简写字母)。
文件名名称可以已经您的想法随意编辑。
2)序列导入MEGA 5首先打开MEGA 5软件,界面如下:然后,导入需要构建系统进化树的序列:点击OK出现新的对话框,创建新的数据文件导入成功3)序列比对分析点击W,开始比对。
比对完成后删除序列两端不能完全对其的碱基。
系统分析然后,关闭该窗口,在弹出的对话框中选择保存文件,文件名随便去,比如保存为1。
4)系统发育树构建以NJ为例Bootstrap选择1000,点Computer,开始计算计算完毕后,生成系统发育树。
以下“系统发育树树的修饰”方法沿用斑竹brightfuture01的方法5)树的修饰建好树之后,往往需要对树做一些美化。
这个工作完全可以在word中完成,达到发表文章的要求。
点击image,copy to clipboard。
新建一个word文档,选择粘贴。
见下图:在图上点击右键-编辑图片,就可以对文字的字体大小,倾斜等做出修饰。
见下图:这个时候可以通过Adobe professional 对其进行图像导出:先将此word文档打印成PDF,见下图:将打印出来的PDF保存在桌面上,打开,如下图:此时,点击工具,高级编辑工具,裁剪工具,如下图所示:选择需要的区域以删除周围的空白区,双击发育树,会出现下图:点击确定,出现下图(把空边切掉了):点击文件,另存为,在保存类型一栏中选择TIFF格式,点击确定后会生成下面这个图片,所生成图片绝对可以满足文章的发表:OK,结束了,自己玩一把吧。
利用MEGA4构建分子系统进化树
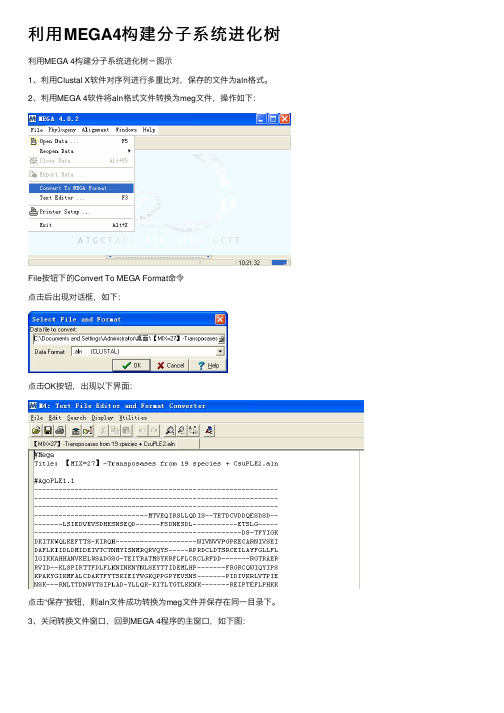
利⽤MEGA4构建分⼦系统进化树利⽤MEGA 4构建分⼦系统进化树-图⽰
1、利⽤Clustal X软件对序列进⾏多重⽐对,保存的⽂件为aln格式。
2、利⽤MEGA 4软件将aln格式⽂件转换为meg⽂件,操作如下:
File按钮下的Convert To MEGA Format命令
点击后出现对话框,如下:
点击OK按钮,出现以下界⾯:
点击“保存”按钮,则aln⽂件成功转换为meg⽂件并保存在同⼀⽬录下。
3、关闭转换⽂件窗⼝,回到MEGA 4程序的主窗⼝,如下图:
点击“Click me to activate a data file”按钮,选择之前转换好的meg⽂件并打开,如下图:
选择所输⼊的数据类型(核酸or蛋⽩),之后点击OK即可。
此时,在MEGA4主程序窗⼝的底部出现了我们所输⼊的⽂件名(如下图),之后就可以构建分⼦系统进化树了。
4、通常选择邻接法(neighbor-joining,NJ)构建分⼦系统进化树。
系统进化树的构建

运算结束后按任意键会保存运算后的数据结果。
3. 再用MEGA打开运算后保存的数据,数据的格 式是phyml_tree。
接着选择所保存好的文件
(4)一般通过NJ构建进化树,并且进行Bootstrap 分析所得到的结果已足够。如果序列近缘,可以 再使用MP构建进化树,进行比较。如果序列较远 源,则可以做ML树比较。使用两种方法得到的树, 如果差别不大,并且Bootstrap总体较高,则得到 的进化树较为可靠。 (5)如果使用MEGA进行分析,选项中有一项是 “Gaps/Missing Data”,一般选择“Pairwise Deletion”。其他多数的选项保持缺省的参数。
2.序列分析:(1)启动.
单击后,会出现如下界面:
这里有三个选项分别对应三种不同的情况:以下分 别予介绍: Create a new alignment :是在你没有任何比对 的时候使用,比如你只有一个fasta 格式的序列 就可以选择这个选项。 Open a saved alignment session:使用它可以打 开一个我们已经比对好的序列文件. Retreve a sequence from a file :这种情况同 第一种情况相似,只是不用选择是DNA 还是蛋白 质序列比对,选择的也是fasta 格式的文件,打 开后的界面都是一样的。
(5)对于进化树的构建,如果对理论的了解并不 深入,需要选择模型的时候用NJ或者ML建树。 (6)一般推荐用两种不同的方法构建进化树,如 果所得到的进化树类似,则结果较为可靠。 (7)Bootstrap几乎是一个必须的选项。一般 Bootstrap的值>70,则认为构建的进化树较为可 靠。如果Bootstrap的值太低,则有可能进化树的 拓扑结构有错误,进化树是不可靠的。
手把手教你构建系统进化树

3、比对序列,比对结果转化为*.meg格式
用 Mega 6.0 的 ClustalW 做多序列联配,比对结果用 *.meg格式保存。或者用Clustal X软件进行比对,比对结果 保存为*.aln,再用Mega 6.0转化为*.meg格式。
4、构建系统进化树
打开保存的*.meg格式文件,选择邻接法构建系统发育 进化树。
以外米缀蛾的cds为例,点击cds,出现下图。
点击FASTA,出现下图。
该图为外米缀蛾的 FASTA格式,如何保 存见下图
一般情况下点 击该页的右上 角有send 图标, 选择后点击 create file 即 可下载。Txt可 以打开。 该图显示的是 序列全长的 FASTA格式下 载。
因为我采取基于氨 基酸序列比对,所 以选择coding sequences和fasta protein,下载编码 区氨基酸序列。
文件名未下载时不要更改,下下来之后再更改
MEGA6可以识别fasta格式文件。如图,将全 部-基因.txt重命名为全部-基因.fasta
•选择打开方式为MEGA6,打开全部-基因.fasta,自动跳出序列窗口 •用ClustalW做多序列联配
如何构建系统进化树
YZU.TRY
系统发生树(英文: Phylogenetic tree ) 又称为演化树( evolutionary tree ),是 表明被认为具有共同祖先的各物种间演化关 系的树。是一种亲缘分支分类方法 ( cladogram )。在树中,每个节点代表其 各分支的最近共同祖先,而节点间的线段长 度对应演化距离(如估计的演名称要么全部 斜体,要么全部不斜体,无法只让拉丁文斜体
系统进化树的构建

进化支
结点
猩 猩
根
一个单位
分支 长度
狒 狒
距离标尺
外 群
系统发育进化树示例
系统发育树重建分析步骤
多序列比对(自动比对,手工校正)
选择建树方法
建立进化树
进化树评估
系统发育树重建的基本方法
• 1. 距离法 (distance)
适用序列有较高相似性时
• 2. 最大简约法 (maximum parsimony, MP)
•实例讲解
文件下载完之后,这里我们采用事先准备好的序列。 将Fasta 文件直接用 ClustalX 1.83打开
•实例讲解
在进行多序列比对之前我们需要对软件进行一些设置 1.选择Alignment标签 2.选择Output format options PHYLIP软件:PHYLIP MEGA软件:FASTA
99 BANNAch 68 100 88
BJ9575 YN6 YN0556 LN0684
100
LN0688
81 LN0689
JKT6969
100 94
JKT6423 JKT7043 LNVNE9712
0.05
如果结点的Bootstrap Value >70我们认为 这个分支是可靠的
优化图标
优化选项栏
适用序列有很高相似性时
• 3. 最大似然法 (maximum likelihood, ML)
– 可用于任何相关序列集合
1. 基于序列距离特征 2+3基于序列离散特征
• 计算速度:
– 距离法 >最大简约法 >最大似然法
系统发育树重建分析过程
直系同源序列 合理的外群
点阵法
构建系统进化树的详细步骤

构建系统进化树的详细步骤1. 建树前的准备工作相似序列的获得——BLASTBLAST是目前常用的数据库搜索程序,它是Basic Local Alignment Search Tool 的缩写,意为“基本局部相似性比对搜索工具”Altschul et al.,199062;199763;国际著名生物信息中心都提供基于Web的BLAST服务器;BLAST算法的基本思路是首先找出检测序列和目标序列之间相似性程度最高的片段,并作为内核向两端延伸,以找出尽可能长的相似序列片段;首先登录到提供BLAST服务的常用网站,比如国内的CBI、美国的NCBI、欧洲的EBI和日本的DDBJ;这些网站提供的BLAST服务在界面上差不多,但所用的程序有所差异;它们都有一个大的文本框,用于粘贴需要搜索的序列;把序列以FASTA格式即第一行为说明行,以“>”符号开始,后面是序列的名称、说明等,其中“>”是必需的,名称及说明等可以是任意形式,换行之后是序列粘贴到那个大的文本框,选择合适的BLAST程序和数据库,就可以开始搜索了;如果是DNA序列,一般选择BLASTN搜索DNA数据库;这里以NCBI为例;登录NCBI主页-点击BLAST-点击Nucleotide-nucleotide BLAST blastn-在Search文本框中粘贴检测序列-点击BLAST-点击Format-得到result of BLAST;BLASTN结果如何分析参数意义:>gi||gb|| Nocardia sp. ATCC 49872 16S ribosomal RNA gene, completesequenceScore = 2020 bits 1019, Expect =Identities = 1382/1497 92%, Gaps = 8/1497 0% Strand = Plus / PlusQuery: 1 gacgaacgctggcggcgtgcttaacacatgcaagtcgagcggaaaggccctttcgggggt 60|||||||||||||||||||||||||||||||||||||||||| ||||||||| ||||| Sbjct: 1 gacgaacgctggcggcgtgcttaacacatgcaagtcgagcggtaaggcccttc--ggggt 58Query: 61 actcgagcggcgaacgggtgagtaacacgtgggtaacctgccttcagctctgggataagc 120|| ||||||||||||||||||||||||||||||| | |||||| ||||||||||||| Sbjct: 59 acacgagcggcgaacgggtgagtaacacgtgggtgatctgcctcgtactctgggataagc 118Score :指的是提交的序列和搜索出的序列之间的分值,越高说明越相似; Expect:比对的期望值;比对越好,expect越小,一般在核酸层次的比对,expect小于1e-10,就比对很好了,多数情况下为0;Identities:提交的序列和参比序列的相似性,如上所指为1497个核苷酸中二者有1382个相同;Gaps:一般翻译成空位,指的是对不上的碱基数目;Strand:链的方向,Plus / Minus意味着提交的序列和参比序列是反向互补的,如果是Plus /Plus则二者皆为正向;序列格式:FASTA格式由于EMBL和GenBank数据格式较为复杂,所以为了分析方便也出现了十分简单的FASTA数据格式;FASTA格式又称为Pearson格式,该种序列格式要求序列的标题行以大于号“>”开头,下一行起为具体的序列;一般建议每行的字符数不超过60或80个,以方便程序处理;多条核酸和蛋白质序列格式即将该格式连续列出即可,如下所示:>1 aaattgaaga gtttgatcat ggctcagatt gaacgctggc ggcaggccta acacatgcaa61 gtcgaacggt aacaggaaga agcttgcttc tttgctgacg agtggcggac ……>AY631071 Jiangella gansuensis YIM 002 1 gacgaacgct ggcggcgtgc ttaacacatg caagtcgagc ggaaaggccc tttcgggggt61 actcgagcgg cgaacgggtg agtaacacgt gggtaacctg ccttcagctc tgggataagc……其中的…>‟为Clustal X默认的序列输入格式,必不可少;其后可以是种属名称,也可以是序列在Genbank中的登录号Accession No.,自编号也可以,不过需要注意名字不能太长,一般由英文字母和数字组成,开首几个字母最好不要相同,因为有时Clustal X程序只默认前几位为该序列名称;回车换行后是序列;将检测序列和搜索到的同源序列以FASTA格式编辑成为一个文本文件例:C:\temp\,即可导入Clustal X 等程序进行比对建树; 2. 构建系统树的相关软件和操作步骤构建进化树的主要步骤是比对,建立取代模型,建立进化树以及进化树评估;鉴于以上对于构建系统树的评价,结合本实验室实际情况,以下主要介绍N-J Tree构建的相关软件和操作步骤;用Clustal X构建N-J系统树的过程1 打开Clustal X程序,载入源文件.File-Load sequences- C:\temp\. 2 序列比对Alignment - Output format options - Clustal format; CLUSTALW sequence numbers: ONAlignment - Do complete alignment Output Guide Tree file,C:\temp\;Output Alignment file, C:\temp\; Alignwaiting……等待时间与序列长度、数量以及计算机配置有关;3 掐头去尾File-Save Sequence as…Format: CLUSTALGDE output case: LowerCLUSTALW sequence numbers: ONSave from residue: 39 to 1504 以前后最短序列为准Save sequence as: C:\temp\ OK将开始和末尾处长短不同的序列剪切整齐;这里,因为测序引物不尽相同,所以比对后序列参差不齐;一般来说,要“掐头去尾”,以避免因序列前后参差不齐而增加序列间的差异;剪切后的文件存为ALN格式;4 File-Load sequences-Replace existing sequences-Yes- C:\temp\重新载入剪切后的序列;5 Trees-Output Format Options Output Files : CLUSTAL format tree Phylip format tree Phylip distance matrix Bootstrap labels on: NODECLOSETrees-Exclude positions with gaps Trees-Bootstrap N-J Tree :Random number generator seed1-1000 : 111 Number of bootstrap trails1-1000: 1000 SAVE CLUSTAL TREE AS: C:\temp\ SAVE PHYLIP TREE AS: C:\temp\ OKwaiting……等待时间与序列长度、数量以及计算机配置有关;在此过程中,生成进化树文件.njbphb,可以用TreeView打开查看;6 Trees-Draw N-J TreesSAVE CLUSTAL TREE AS: C:\temp\ SAVE PHYLIP TREE AS: C:\temp\ SAVE DISTANCE MATRIX AS: C:\temp\ OK此过程中生成的报告文件.nj比较有用,里面列出了比对序列两两之间的相似度,以及转换和颠换分别各占多少;7 TreeViewFile-Open-C:\temp\Tree- phylogramunrooted, slanted cladogram,Rectangular cladogram多种树型 Tree- Show internal edge labels Bootstrap value显示数值Tree- Define outgroup… ingroup >> outgroup OK定义外群Tree- Root with outgroup通常需要对进化树进行编辑,这时首先要Edit-Copy至PowerPoint上,然后Copy 至Word上,再进行图片编辑;如果直接Copy至Word则显示乱码,而进化树不能正确显示; Mega建树虽然Clustal X可以构建系统树,但是结果比较粗放,现在一般很少用它构树,Mega因为操作简单,结果美观,很多研究者选择用它来建树;1 首先用Clustal X进行序列比对,剪切后生成C:\temp\文件;同上2 打开BioEdit 程序,将目标文件格式转化为FASTA格式,File-Open- C:\temp\,File-Save As- C:\temp\ ;3 打开Mega程序,转化为mega格式并激活目标文件,File-Convert To MEGA Format- C:\temp\C:\temp\ ,关闭Text Editor窗口-Do you want to save your changes before closing-Yes; Click me to activate a data file- C:\temp\Protein-coding nucleotide sequence data-No;Phylogeny-Neighbor-JoiningNJDistance Options-Models-Nucleotide: Kimura 2-parameter;d: Transitions+Transversions;Include Sites-Pairwise DeletionTest of Phylogeny-Bootstrap; Replications 1000; Random Seed 64238OK;开始计算,得到结果;4 Image-Copy to Clipboard-粘贴至Word文档进行编辑;此外,Subtree中提供了多个命令可以对生成的进化树进行编辑,Mega窗口左侧提供了很多快捷键方便使用;View中则给出了多个树型的模式;下面只介绍几种最常用的: Subtree-Swap:任意相邻两个分支互换位置;-Flip:所选分支翻转180度;-Compress/Expand:合并/展开多个分支;-Root:定义外群;View-Topology:只显示树的拓扑结构;-Tree/Branch Style:多种树型转换;-Options:关于树的诸多方面的改动;TREECON打开Clustal X,File-Load ,File-Save Sequence as…Format-PHYLIP;Save from residue-1 to 末尾;Save sequence as : C:\temp\;打开TREECON程序,1 Distance estimation点击Distance estimation-Start distance estimation,打开上面保存的文件,Sequence Type-Nuleic Acid Sequence,Sequence format-PHYLIP interleaved,Select ALL,OK; Distance Estimation-Jukes&Cantoror Kimura,Alignment positions-All,Bootstrap analysis-Yes,Insertions&Deletions-Not taken into account,OK;Bootstrap samples-1000,OK;运算,等待……Finished-OK;2 Infer tree topology点击Infer tree topology-Start inferring tree topology,Method-Neighbor-joining, Bootstrapanalysis-Yes,OK.;运算,等待……Finished-OK;3 Root unrooted trees点击Root unrooted trees-Start rooting unrooted trees,Outgroup opition-single sequenceforced,Bootstrap analysis-Yes,OK;Select Root-X89947,OK;运算,等待……Finished-OK;4 Draw phylogenetic tree点击Draw phylogenetic tree,File-Open-new tree,Show-Bootstrap values/ Distance scale; File-Copy,粘贴至Word文档,编辑;TREECON的操作过程看起来似乎较MEGA烦琐,且运算速度明显不及MEGA,如果参数选择一样,用它构建出来的系统树几乎和MEGA构建的完全一样,只在细节上,比如Bootstrap值二者在某些分支稍有不同;在参数选择方面,TREECON和MEGA 也有些不同,但总体上相差不大;PHYLIPPHYLIP是多个软件的压缩包,下载后双击则自动解压;当你解压后就会发现PHYLIP 的功能极其强大,主要包括五个方面的功能软件:i,DNA和蛋白质序列数据的分析软件;ii,序列数据转变成距离数据后,对距离数据分析的软件; iii,对基因频率和连续的元素分析的软件;iv,把序列的每个碱基/氨基酸独立看待碱基/氨基酸只有0和1的状态时,对序列进行分析的软件;v,按照DOLLO简约性算法对序列进行分析的软件;vi,绘制和修改进化树的软件;在此,主要对DNA序列分析和构建系统树的功能软件进行说明; 1 生成PHY格式文件首先用Clustal X等软件打开剪切后的序列文件C:\temp\另存为C:\temp\使用File-Save Sequences As命令,Format项选“PHY”;用BioEdit或记事本打开2 打开Phylip软件包里的SEQBOOT: can't find input file "infile" Please enter a new file name> C:\temp\ 按路径输入刚才生成的 .PHY文件,显示如下:Bootstrapping algorithm, versionSettings for this run:D Sequence, Morph, Rest., Gene Freqs Molecular sequences J Bootstrap, Jackknife, Permute, Rewrite Bootstrap B Block size for block-bootstrapping 1 R How many replicates 100W Read weights of characters NoC Read categories of sites NoF Write out data sets or just weights Data sets I Input sequences interleaved Yes0 Terminal type none1 Print out the data at start of run No2 Print indications of progress of run YesY to accept these of type the letter for one to changeRNumber of replicates1000Settings for this run:D Sequence, Morph, Rest., Gene Freqs Molecular sequences J Bootstrap, Jackknife, Permute, Rewrite Bootstrap B Block size for block-bootstrapping 1 R How many replicates 1000W Read weights of characters NoC Read categories of sites NoF Write out data sets or just weights Data sets I Input sequences interleaved Yes0 Terminal type IBM PC 1 Print out the data at start of run No2 Print indications of progress of run YesY to accept these of type the letter for one to changeYRandom number seed must be odd5any odd numbercompleted replicate number 100completed replicate number 200completed replicate number 300completed replicate number 400completed replicate number 500completed replicate number 600completed replicate number 700completed replicate number 800completed replicate number 900completed replicate number 1000上面的D、J、R、I、O、1、2代表可选择的选项,键入这些字母后敲回车键,程序的条件就会发生改变;D选项无须改变;J选项有三种条件可以选择,分别是Bootstrap、Jackknife和Permute;R选项让使用者输入republicate的数目;所谓republicate就是用Bootstrap法生成的一个多序列组;根据多序列中所含的序列的数目的不同可以选取不同的republicate;当我们设置好条件后,键入Y按回车;得到一个文件outfile:C:\Program Files\Phylip\exe\ outfile.重命名outfile infile;3 打开Nucleic acid sequence Distance Matrix program, versionSettings for this run:D Distance F84 G Gamma distributed rates across sites No T Transition/transversion ratio C One category of substitution rates Yes W Use weights for sites NoF Use emperical base frequencies Yes L Form of distance matrix SquareM Analyze multiple data sets NoI Input sequences interleaved Yes0 Terminal type 1 Print out the data at start of run No 2 Print indications of progress of run YesY to accept these of type the letter for one to changedD Distance Kimura 2-parametermMultiple data sets or multiple weighs type D or W dHow many data sets1000Settings for this run:D Distance Kimura 2-parameterG Gamma distributed rates across sites No T Transition/transversion ratio C One category of substitution rates Yes W Use weights for sites NoF Use emperical base frequencies YesL Form of distance matrix SquareM Analyze multiple data sets Yes, 1000 data sets I Input sequences interleaved Yes0 Terminal type IBM PC 1 Print out the data at start of run No 2 Print indications of progress of run YesY to accept these of type the letter for one to changeY选项D有四种距离模式可以选择,分别是Kimura 2-parameter、Jin/Nei、Maximum-likelihood和Jukes-Cantor;选项T一般键入一个之间的数字;选项M 键入1000;运行后生成文件C:\Program Files\Phylip\exe\ outfile;重命名outfile infile;4 打开Neighbor-Joining/UPGMA method versionSettings for this run:N Neighbor-Joining or UPGMA tree Neighbor-Joining O Outgroup root No, Use as outgroup species 1 L Lower-triangular data metrix NoR Upper-triangular data metrix NoS Subreplication NoJ Randomize input order of species No, Use input order M Analyze multiple data sets No0 Terminal type 1 Print out the data at start of run No 2 Print indications of progress of run Yes 3 Print out tree Yes4 Write out trees onto tree file YesY to accept these of type the letter for one to changemHow many data sets1000Random number seed must be odd5Settings for this run:N Neighbor-Joining or UPGMA tree Neighbor-Joining O Outgroup root No, Use as outgroup species 1 L Lower-triangular data metrix NoR Upper-triangular data metrix NoS Subreplication NoJ Randomize input order of species YesM Analyze multiple data sets Yes, 1000 sets 0 Terminal type IBM PC 1 Print out the data at start of run No 2 Print indications of progress of run Yes 3 Print out tree Yes4 Write out trees onto tree file YesY to accept these of type the letter for one to changeY生成文件C:\Program Files\Phylip\exe\ outtree&outfile;重命名outtreeintree;outfileinfile;打开Consensus tree program, versionSettings for this run:C Consensus type Majority rule extendedO Outgroop root No, use as outgroup species 1R Trees to be treated as Rooted NoT Terminal type 1 Print out the sets of the species Yes 2 Print indications of progress of run Yes 3 Print out tree Yes4 Write out trees onto tree file YesAre these settings correctRTSettings for this run:C Consensus type Majority rule extendedR Trees to be treated as Rooted YesT Terminal type IBM PC 1 Print out the sets of the species Yes 2 Print indications of progress of run Yes3 Print out tree Yes4 Write out trees onto tree file Yes Y生成文件C:\Program Files\Phylip\exe\ outtree;重命名outtree ;打开TreeView打开C:\Program Files\Phylip\exe\ ;以下操作参照前述详细说明即可;。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
作系统进化树的方法
系统进化树(Phylogenetic tree)是一种表示生物物种之间进化关系的图
形结构。
它基于生物的遗传物质或形态特征等数据,通过一定的算法和模型来构建,以揭示物种之间的亲缘关系和进化历程。
以下是构建系统进化树的一般步骤:
1. 数据收集:首先需要收集用于构建进化树的基因或形态特征数据。
这通常涉及从各种来源获取DNA、蛋白质或其他分子序列数据,或者从博物馆和
标本馆获取生物形态特征数据。
2. 序列比对:对于DNA或蛋白质序列数据,需要将这些序列进行比对,以确保它们可以一起进行比较和分析。
3. 选择适当的距离度量:在构建系统进化树时,需要计算物种之间的“距离”。
这些距离是基于序列或形态特征的差异来计算的。
有多种方法可以计算这些距离,例如基于遗传物质的p距离(代表两个序列之间的差异比例)或形态特征的欧几里得距离。
4. 选择合适的建树算法:系统进化树可以通过多种算法来构建,包括但不限于UPGMA(Unweighted Pair Group Method with Arithmetic Mean)、WPGMA(Weighted Pair Group Method with Arithmetic Mean)、WPGMC(Weighted Pair Group Method with Centroid Linkage)、Neighbor Joining、Fitch-Margoliash、Maximum Parsimony、
Maximum Likelihood等。
选择哪种算法取决于你的具体需求和所处理数
据的性质。
5. 构建系统进化树:使用选择的算法和距离度量,将物种按照它们的亲缘关系分组。
这一步通常涉及到一个迭代过程,其中算法会尝试不同的分组方案,直到找到一个最优解。
6. 评估和验证树:一旦构建了系统进化树,就需要对其进行评估和验证,以确保其合理性和可靠性。
这通常涉及使用多种统计测试和可视化工具,例如Bootstrapping、P-distance、Tree-bisection-reconnection (TBR) 操作等。
7. 解释和解读树:最后,需要解释和解读系统进化树的结果,以理解物种之间的进化关系和亲缘关系。
这可能涉及对特定区域或分支的深入分析,以及将其与已知的生物地理学、生物多样性和物种分布等信息结合起来。
请注意,这只是构建系统进化树的一般步骤,具体的实践可能会因研究问题和可用数据的性质而有所不同。
此外,随着新的技术和方法的出现,这些步骤可能会进一步发展和改进。
