转录因子ERF家族
ERF转录因子研究进展

ERF转录因子研究进展高浩;竺锡武【摘要】ERF(Ethylene-responsive factor)转录因子是AP2/ERF大家族中的一个大的亚家族,仅含1个AP2/ERF结构域,每个成员都含有1个由大约60个氨基酸组成的非常保守的DNA结合域.有研究表明,每种植物有100种以上ERF转录因子,其功能各不相同,分别具有调节植物生长发育、抗生物胁迫和非生物胁迫的作用等.本文就ERF转录因子的研究现状及发展趋势进行分析,以期为ERF转录因子的应用提供参考.【期刊名称】《现代农业科技》【年(卷),期】2017(000)008【总页数】3页(P130-131,134)【关键词】ERF转录因子;功能;作用机理【作者】高浩;竺锡武【作者单位】湖南人文科技学院,湖南娄底 417000;湖南人文科技学院,湖南娄底417000【正文语种】中文【中图分类】Q943随着环境条件的恶化,植物在生长发育过程中受到的非生物因素和生物因素影响会更多,如高温、低温、干旱、盐碱、病虫害等。
在不断适应环境和进化过程中,植物形成了复杂有效的逆境胁迫应答体系,可以调节植物使其能够适应新的生长环境。
其中,在转录水平的调控过程中转录因子发挥了非常重要的作用[1]。
转录因子又称反式作用因子,是一群能与真核基因启动子区域中的顺式作用元件发生特异性结合,从而保证目的基因以特定的强度、在特定的时间与空间表达的蛋白质分子[2]。
AP2/ERF家族转录因子对植物非常重要,可以调控植物整个生命周期的生长发育和逆境[3-7]。
根据AP2结构域的数目和结构特点,AP2/ERF家族转录因子可分为4个亚族(ERF、DREB、AP2、RAV)和单独成员(Soloist)[4-5,8]。
ERF(Ethylene-responsive factor)转录因子是 AP2/ERF大家族中的一个大的亚家族,仅含1个AP2/ERF结构域,在结构域序列的第14位和第19位分别是丙氨酸和天冬氨酸。
《黄花苜蓿AP2-ERF家族MfERF014基因调控植物响应非生物胁迫的功能研究》范文

《黄花苜蓿AP2-ERF家族MfERF014基因调控植物响应非生物胁迫的功能研究》篇一黄花苜蓿AP2-ERF家族MfERF014基因调控植物响应非生物胁迫的功能研究摘要:本研究针对黄花苜蓿AP2/ERF家族的MfERF014基因进行了深入的功能研究,探讨了该基因在植物响应非生物胁迫过程中的作用机制。
通过基因克隆、表达模式分析、转基因技术及胁迫处理等手段,揭示了MfERF014基因在提高植物抗逆性方面的潜在应用价值。
一、引言黄花苜蓿作为一种重要的豆科作物,在农业生态系统中具有较高的经济价值和生态价值。
然而,植物在生长过程中常常面临各种非生物胁迫,如干旱、盐渍、低温等,这些环境因素严重影响了作物的产量和品质。
因此,研究植物如何响应非生物胁迫,尤其是从分子层面解析相关基因的功能,对于提高作物的抗逆性具有重要意义。
AP2/ERF家族是一类重要的植物转录因子家族,参与植物对多种生物和非生物胁迫的响应。
其中,MfERF014基因作为黄花苜蓿AP2/ERF家族的一员,其功能尚不明确。
本研究旨在探讨MfERF014基因在植物响应非生物胁迫过程中的功能及作用机制。
二、材料与方法1. 材料选取黄花苜蓿作为实验材料,通过基因克隆技术获取MfERF014基因的序列信息。
2. 方法(1)基因克隆:利用PCR技术扩增MfERF014基因的编码区序列。
(2)表达模式分析:通过实时荧光定量PCR(qRT-PCR)技术,分析MfERF014基因在不同非生物胁迫条件下的表达模式。
(3)转基因技术:构建MfERF014基因的过表达和沉默载体,通过遗传转化获得转基因黄花苜蓿植株。
(4)胁迫处理:对转基因植株进行干旱、盐渍、低温等非生物胁迫处理,观察并记录植株的生长状况及生理指标变化。
三、结果与分析1. 基因克隆与序列分析成功克隆了MfERF014基因的编码区序列,该基因编码一个含有AP2结构域的转录因子。
2. 表达模式分析qRT-PCR结果显示,MfERF014基因在干旱、盐渍和低温等非生物胁迫条件下表达量显著上调,表明该基因可能参与植物对非生物胁迫的响应。
《花椰菜乙烯受体转录因子(ERF)的克隆及其在逆境应答中的功能研究》

《花椰菜乙烯受体转录因子(ERF)的克隆及其在逆境应答中的功能研究》摘要:本文旨在研究花椰菜乙烯受体转录因子(ERF)的克隆过程,并探讨其在逆境应答中的功能。
通过分子生物学技术,成功克隆了花椰菜ERF基因,并对其表达模式进行了分析。
研究结果表明,该基因在逆境条件下具有重要调控作用,对植物抗逆性具有潜在的应用价值。
一、引言乙烯是植物生长和发育过程中的重要激素之一,其信号转导途径在植物逆境应答中发挥着关键作用。
乙烯受体转录因子(ERF)作为乙烯信号转导途径的关键组成部分,在植物应对环境胁迫时起着重要的调控作用。
因此,研究花椰菜乙烯受体转录因子(ERF)的克隆及其在逆境应答中的功能具有重要意义。
二、材料与方法1. 材料准备实验所用材料为花椰菜组织,实验中所用的引物、试剂等均为市售高品质产品。
2. 方法(1)基因克隆:采用PCR技术,以花椰菜基因组DNA为模板,扩增ERF基因。
(2)序列分析:对克隆得到的ERF基因进行序列分析,确定其编码序列及结构特征。
(3)表达分析:采用实时荧光定量PCR技术,分析ERF基因在花椰菜不同组织及逆境条件下的表达模式。
(4)功能研究:通过转基因技术,研究ERF基因在植物逆境应答中的功能。
三、实验结果1. 基因克隆与序列分析通过PCR技术成功克隆了花椰菜ERF基因,序列分析表明该基因编码一个含有AP2结构域的转录因子。
该基因具有典型的ERF家族特征,表明其可能参与乙烯信号转导途径。
2. 表达分析实时荧光定量PCR结果显示,ERF基因在花椰菜不同组织中均有表达,且在逆境条件下表达量明显上升。
其中,在受到病原菌侵染、干旱、高温等逆境条件时,ERF基因的表达量显著增加。
3. 功能研究通过转基因技术,研究了ERF基因在植物逆境应答中的功能。
结果表明,过表达ERF基因的植物在逆境条件下的抗性明显增强,表现出更强的生长恢复能力和抗病能力。
这表明ERF基因在植物逆境应答中具有重要调控作用。
ERF植物转录因子与植物抗逆研究

酵母单杂交方法—体内分析转录因子与DNA结合特性或转录激活活性
(3)同源克隆 同源克隆的优点是在植物材料的基因信息未知的情况下,通过已知
(3)泛素介导的蛋白降解途径是翻译后调控转录因子蛋 白水平的另一个机制。
4、转录因子研究方法
4.1 克隆转录因子的方法
基于转录因子具有表达丰度低、与顺式元件特异性结合且DNA结合序 列保守等特点。目前,分离克隆转录因子常用的方法主要有以下几种:
(1)转座子标签法与RNA差异显示法 相结合的克隆方法
1、转录因子的结构特征
转录因子的三维结构中,N端通常含有与顺式作用元件 相结合的关键结构域。在转录调控过程中,转录因子通过 其 DNA结合域与靶序列特异结合,来实现对靶基因的精确 调控。
转录因子一般由4个功能区域组成: DNA结合区(DNA binding domain) 转录调控区(Transcription regulation domain)
(5)规模化分离、鉴定转录因子超家族成员 随着基因组测序技术的迅猛发展,多种植物的基因组已经测序完毕。
因此,利用生物信息学方法从全基因内分离、鉴定家族基因已成为研究 热点。这有助于全面系统深入解析基因的功能,在拟南芥、水稻、大豆 及其他一些作物的基因组中系统分析AP2/ERF家族基因已有报道。
总之,上述五种克隆转录因子的方法并不是孤立的, 需要结合实 验材料、实验目的等实际情况,综合采用上述一种或多种方法克隆目 的基因。
ERF植物转录因子研究与植物抗逆
转录因子在植物防卫反应和逆境胁迫应答过程中扮演着非 常重要的角色。在植物抗逆反应过程中,当植物受到外界环境 胁迫后,植物通过其信号传导途径有效地调控体内相关功能基 因的表达,进而引发一系列生理、生化反应,形成高效有序的 信号调控网络,以降低或消除给植株带来的危害。
ERF转录因子

一、乙烯信号转导通路乙烯是一种非常重要的植物激素。
乙烯在植物生长发育和适应生物和非生物胁迫反应中起到了非常重要的作用。
种子萌发、开花、叶片衰老、果实成熟、根瘤、细胞程序性死亡以及对非生物胁迫和病原体入侵的反应等生理过程都与乙烯密切相关。
乙烯信号转导通路的最上游是位于内质网膜上的5个乙烯受体,分别被称为:ETR1、ETR2、ERS1、ERS2和EIN4。
位于乙烯受体下游的是一个负调节因子,蛋白激酶CTR1。
CTR1蛋白激酶通过与乙烯受体相结合定位在内质网上。
在没有乙烯存在的条件下,CTR1和受体的结合会协同抑制下游乙烯信号途径。
在CTR1负调控因子下游是一个正调控因子EIN2。
EIN2基因发生功能缺失突变会产生乙烯不敏感表型,显示出EIN2在乙烯信号通路中起到了核心作用。
EIN2的半衰期很短,两个F-Box蛋白ETP1和ETP2负责调控EIN2的泛素化降解。
位于EIN2下游的是正调控的转录因子家族EIN3及5个EILs。
研究发现,他们同样是受泛素化途径降解的,负责调控EIN3及EILs泛素化降解的F-Box蛋白是EBF1和EBF2。
EBF5是一种外切核酸酶它能够通过促进EBF1和EBF2的mRNA的降解来拮抗这两个蛋白对EIN3的负反馈调控。
EIN3和EIL1通过启动乙烯信号转导途径示意图转录级联反应来激活下游乙烯响应基因的表达。
二、乙烯响应因子(ethylene response factor、ERF)的结构特点及生物信息学分析ERF基因家族是一个很大的转录因子家族,属于AP2/ERF转录因子超家族。
Ohme-Takagi和Shinshi研究发现,GCC box是植物乙烯响应的DNA序列元件;同时他们在烟草(Nicotiana tabacuum)中发现了能特异性结合GCC box元件的数个乙烯响应元件结合蛋白(EREBPs),并发现,EREBPs同GCC元件相结合的结构域是59个保守的氨基酸残基。
转录因子在植物抗逆性中的调控机制

转录因子在植物抗逆性中的调控机制转录因子在植物抗逆性中的调控机制是一个复杂而精细的生物学过程。
以下是根据您提供的文档结构,撰写的关于该主题的文章。
一、转录因子概述转录因子是一类能够结合到DNA上的蛋白质,调控基因的转录过程。
在植物中,转录因子对抗逆性基因的表达起着至关重要的作用。
植物在面临逆境如干旱、盐碱、低温、高温、病原菌侵染等环境压力时,转录因子能够通过调节下游基因的表达,增强植物的适应性和生存能力。
1.1 转录因子的功能转录因子通过识别特定的DNA序列,与基因的启动子区域结合,从而激活或抑制基因的转录。
它们可以是激活因子,促进基因表达;也可以是抑制因子,抑制基因表达。
转录因子的活性受到多种信号通路的调控,包括植物激素信号、环境信号和内部代谢信号等。
1.2 转录因子的分类转录因子可以根据其结构域和功能进行分类。
常见的转录因子家族包括AP2/ERF家族、bZIP家族、WRKY家族、MYB 家族等。
每个家族的转录因子都有其特定的DNA结合模式和调控特性。
二、转录因子在植物抗逆性中的调控机制植物在逆境条件下,转录因子通过多种机制调控基因表达,以应对不同的环境压力。
2.1 逆境信号的识别与响应植物首先需要识别逆境信号,如干旱、盐分、低温等。
这些信号通过植物的感知系统被识别后,会激活一系列的信号传导途径,最终导致转录因子的激活或抑制。
2.2 转录因子的激活与功能逆境信号激活的转录因子会进入细胞核,结合到特定基因的启动子区域,调控这些基因的表达。
这些基因通常编码与抗逆性相关的蛋白质,如渗透调节蛋白、抗氧化酶、抗冻蛋白等。
2.3 转录因子的相互作用转录因子之间也存在相互作用,它们可以通过形成同源或异源二聚体,或者通过相互竞争DNA结合位点,来协同调控基因表达。
这种相互作用增加了调控网络的复杂性,使得植物能够精细调控其抗逆性反应。
2.4 转录因子的后转录调控除了直接调控基因的转录,转录因子还可以通过影响mRNA的加工、稳定性和翻译等后转录过程,进一步调节基因表达。
ERF类转录因子的结构与功能

Ab ta t T e E r n c p in fco s ae k o o b n q e i ln s T e me e so i fmi a e aCo s r c : h RF t s r t a tr r n wn t e u i u n p a t h mb r ft s a l h v mmo e — a i o h y n la
t r n s u tr u e i t cu e:alt e me es c n an o e c n ev d DN —b n i g d man ,w ih C n ito b u 0 a n cd . r l h mb r o t i n o s r e A i d n o i s h c o s f a o t6 mio a i s s E rn c i t n f cos e itc mmo l n p a t ,a d t e r n ov d i r wt ,d v lp n n in lta s u t n RF ta s r i a t r x s o p o n y i l ns n h y we e i v le n go h e e o me t d s a r n d ci a g o i n h soo ia n ic e c r c s e . I h sr v e w il n r d c e p o e si e sr c u e a d f n — n ma y p y i lgc a d b o h mia p o e s s n t i e iw e man y i t u e t rg s n t t t r n u c l l o h r h u t n o h F t n c i t n f co . i f e ER a s rp i a tr o t r o s K e r s E y wo d : RF; r n c i t n f co T a s r i a t r p o s
珙桐AP2

2024 年 3月第 61 卷第 2 期Mar. 2024Vol. 61 No. 2四川大学学报(自然科学版)Journal of Sichuan University (Natural Science Edition)珙桐AP2/ERF家族相关基因的克隆及功能鉴定曾沥,孙曼丽,刘晋芸,胡小盼,魏炜(四川大学生命科学学院生物资源与生态环境教育部重点实验室,成都 610065)摘要: 为探究AP2/ERF类转录因子在珙桐开花调控及花器官发育中的作用,本研究通过从实验室珙桐不同发育时期苞片和叶片的转录组数据中筛选出调控珙桐苞片发育相关AP2/ERF家族基因DiAP2-1、DiDREB-1和DiRAV-1,经过生物信息学分析、基因克隆及功能鉴定初步研究了目的基因在珙桐苞片发育过程中的调控机制.亚细胞定位实验显示DiAP2-1主要定位于细胞质,DiDREB-1和DiRAV-1主要定位于细胞核.对目的基因在珙桐中的表达模式分析发现DiAP2-1和DiRAV-1在苞片发育过程中表达量逐渐降低,DiDREB-1只在苞片第三时期高表达,推测DiDREB-1、DiRAV-1属于A类基因,DiAP2-1属于B类基因.表型实验显示DiDREB-1和DiRAV-1转基因拟南芥株系较野生型表现为明显的早花,DiAP2-1同源基因对应的拟南芥突变体与同一时期野生型拟南芥对比表现为花瓣减少且萼片大,角果呈短粗状且败育.对关键开花调控基因表达模式分析发现DiDREB-1和DiRAV-1异源表达后使其他促花因子表达量上调进而使拟南芥花期提前.以上结果表明,AP2/ERF类转录因子DiAP2-1、DiDREB-1和DiRAV-1在珙桐苞片及花器官发育和珙桐开花调控中发挥了重要作用.关键词: 珙桐;花器官;苞片; AP2/ERF中图分类号: Q37 文献标志码: A DOI:10.19907/j.0490-6756.2024.026002Cloning and functional identification of AP2/ERF family-relatedgenes in Davidia involucrata Baill.ZENG Li, SUN Man-Li, LIU Jin-Yun, HU Xiao-Pan, WEI Wei(Key Laboratory of Bio-Resource and Eco-Environment of Ministry of Education, College of Life Sciences,Sichuan University, Chengdu 610065, China)Abstract: To investigate the role of AP2/ERF transcription factors in the regulation of Dove tree flowering and floral organ development, this study was conducted to screen the AP2/ERF family genes DiAP2-1, Di⁃DREB-1and DiRAV-1from the transcriptomic data of dove bracts and leaves at different developmental stages in the laboratory.A preliminary study of the regulatory mechanisms of these genes in the development of dove bracts was conducted by bioinformatics analysis, gene cloning and functional characterization.Subcel‑lular localization experiments showed that DiDREB-1and DiRAV-1 were expressed in the nucleus, except for DiAP2-1, which was mainly expressed in the cytoplasm.Analysis of the expression pattern of the target genes in dove showed that the expression levels of DiAP2-1 and DiRAV-1 gradually decreased during bract development,while DiDREB-1was only highly expressed during the third bract period,presumably Di⁃DREB-1 and DiRAV-1belonged to class A genes and DiAP2-1 belonged to class B genes.Phenotypic ex‑收稿日期: 2023-03-24基金项目: 四川省科技厅育种攻关项目(2021YJ0296)作者简介: 曾沥(1997-),男,四川南充人,硕士研究生,主要从事珙桐基因的功能研究.E-mail: 310953866@通讯作者: 魏炜.E-mail: weiwscu@第 61 卷四川大学学报(自然科学版)第 2 期periments showed that DiDREB-1 and DiRAV-1 transgenic Arabidopsis strains exhibited early flowering compared to the wild type, and the Arabidopsis mutant corresponding to the DiAP2-1 homologue showed re‑duced petals and larger sepals,and short,stubby and failure to breed hornbeam compared to the wild type Arabidopsis at the same time.These results suggest that the AP2/ERF-like transcription factors DiAP2-1,DiDREB-1 and DiRAV-1 play key roles in the development of dove bracts and flowering organs and in the regulation of Dove tree flowering.Keywords: Dove tree; Flowering organ; Bract; AP2/ERF1 引言珙桐(Davidia involucrata Baill.)第三纪古热带孑遗树种,为中国特有的单属植物[1],1999年被列为国家首批一级重点保护野生植物,有植物大熊猫和活化石之称[2, 3].而珙桐独特的形似飞翔鸽子的一对苞片(实质是一种变态叶)是珙桐最引人入胜的地方[4],也是其成为重要观赏植株并得名鸽子树的原因.花器官是植物生长繁殖过程中重要的组织器官之一,对植物种群规模的维持及扩大尤为重要[5].花器官的发育可大致分为以下几个阶段:首先是开花诱导阶段,它是花发育过程中尤为重要的关键步骤,决定着植物是否开花以及何时开花;其次是花的发端阶段,植物通过整合不同途径通路信号激活花分生组织特征基因,进而实现对花器官特征基因的激活;最后是花器官发育阶段,主要由部分花器官特征基因对负责花器官形态发育的部分特征基因进行激活,进而实现花器官正常发育过程[6, 7].植物AP2/ERF是一个具有众多成员的庞大转录因子基因家族,此类转录因子都含此家族特殊的结构域,如AP2结构域或B3结构域.根据结构域的数量和识别序列的不同可以将其分为5个亚家族,存在于几乎所有的植物中且参与多种生物学过程[8].已有研究表明AP2类基因在拟南芥花器官特性的形成和花同源异型基因表达的调控中发挥着核心作用,并发现其在花的四种器官萼片、花瓣、雄蕊和心皮中都有表达[9].也有研究表明,属于拟南芥RAV亚家族成员的TEM1和TEM2受多个开花通路的基因调控[10].AP2/ERF 家族不仅参与调节植物生长发育尤其是花器官的发育,而且在响应外界胁迫中也发挥重要作用.现阶段AP2/ERF转录因子作为重要的抗性调控基因在基因工程或作物育种中越来越受到重视[11],也为将来创造出兼具高产、抗病及富含营养等优良性状农作物品种提供资料[12].在对植物开花途径的研究中发现,植物正常开花主要受到以下开花诱导途径的影响,其中主要包括光周期途径、春化途径、赤霉素途径和自主促进途径[13].各种开花诱导途径在独立发挥作用的同时,也共同构成了一个复杂而精密的调控网络,共同调控着整个开花过程.而花器官的发育也有着极为复杂的调控过程,在对花器官发育调控网络研究过程中逐步确立了花发育“ABCDE”模型[14-16].并在该模型基础上又提出四因子发育模型[17].但是随着对植物花器官发育研究的深入,发现除花发育“ABCDE”模型包含的相关功能基因外,还存在很多模型外基因在花发育过程中也发挥着重要作用,对花器官形态构建有着重要影响.例如,在矮牵牛中AGL6亚家族基因PhAGL6具有着与E类功能基因类似的功能,对矮牵牛花药及花瓣的发育产生影响,与其内部E类功能基因存在部分程度上的功能冗余.这类基因不仅存在于MADS-box基因家族中,其他基因家族中的部分基因同样在植物花发育过程中发挥着独特作用,并参与到植物花器官正常发育过程当中[18].本研究在珙桐叶片以及苞片不同发育时期的转录组数据中筛选出部分在珙桐叶片和苞片差异表达的AP2/ERF家族基因,进行生物信息学分析、相关基因的克隆、鉴定、亚细胞定位及目的基因在珙桐中的时空表达模式分析,结合目的基因异源表达进行拟南芥遗传转化,对筛选出的基因的功能进行初步鉴定及探究.初步阐明此类转录因子在珙桐花发育过程中的作用,以期为进一步解析珙桐苞片发育的分子调控机制奠定基础.2 材料与方法2.1 材料在四川省都江堰市虹口乡龙溪虹口国家级自然保护区内采集野生珙桐不同发育时期苞片、叶片、叶芽、根、枝条等组织;本氏烟草(Nicotiana第 2 期曾沥,等:珙桐AP2/ERF家族相关基因的克隆及功能鉴定第 61 卷benthamiana)种子由本课题组保存并提供,于课题组现有温室内种植培养,培养条件:室温23 ℃,空气湿度55%,光周期为光照16 h黑暗8 h交替,光照强度为120 μmol∙m-2∙s-1;哥伦比亚型拟南芥(Arabidopsis thaliana)种子由本课题组保存并提供,突变体(拟南芥突变体(CS148、SALK_ 083090、CS313884)采购于ABRC;过表达拟南芥植株:构建过表达重组载体,通过农杆菌介导法获得过表达目的基因拟南芥植株,所用转基因植株均为筛选至 T3 代的纯合系.2.2 方法2.2.1 目的基因的确定及克隆 利用实验室自建的珙桐全转录组数据,通过结构域分析、表达模式分析及差异聚类分析等,最终从中筛选出了可能与珙桐苞片发育相关的分别来自三个不同亚家族的三个基因.以珙桐总cDNA为模板通过常规PCR扩增获得DiAP2-1、DiDREB-1和DiRAV-1的目的基因片段.2.2.2 载体构建 为构建亚细胞定位载体,将目的基因与pBI221-EGFP表达载体构建融合载体;为构建过表达载体,将目的基因与pBI121-EGFP 表达载体构建融合载体.2.2.3 目的基因在珙桐中的时空表达分析 以珙桐β-Diactin为内参基因,采用qRT-PCR实验方法,对DiAP2-1、DiDREB-1和DiRAV-1在珙桐不同发育时期不同组织内的表达情况进行检测,数据结果由三次独立重复实验提供,使用的引物见表1.2.2.4 亚细胞定位分析 将构建的亚细胞定位载体通过PEG-Calcium介导转入烟草原生质体中表达,在正置荧光显微镜下观察DiAP2-1、Di⁃DREB-1和DiRAV-1的亚细胞定位.2.2.5 纯合转基因株系表型分析 将野生型、ap2突变体、DiAP2-1、DiDREB-1、DiRAV-1转基因株系(每个株系均种植10株)正常培养,对各拟南芥株系的表型进行观察,测定相关生理指标并统计相关数据.2.2.6 转基因植株中开花调控关键基因表达分析 为探究异源表达DiAP2‑1、DiDREB-1和DiRAV‑1是否影响到开花的关键基因表达量,我们挑选6个参与拟南芥开花调控的关键基因:AtSPL、AtAP1、AtAGL24、AtLFC、AtFT和AtLFY,利用qRT-PCR进行基因相对表达量检测,野生型作为对照组,β-actin为内参基因,使用的引物见表2.3 结果与分析3.1 目的基因的确定及克隆从自建的珙桐全转录组数据筛选出三个AP2/ERF家族目的基因,分别为AP2亚家族的CL18617.Contig3_All、DREB亚家族的CL16625. Contig4_All以及RAV亚家族的CL4058.Con‑tig1_All,并命名为DiAP2‑1、DiDREB‑1和Di⁃RAV‑1.PCR扩增获得目的基因片段与转录组数据比对见图 1.3.2 DiAP1、DiTFL1s在珙桐中的时空表达分析利用qRT-RCR技术,分析珙桐DiAP2‑1、Di⁃DREB‑1、DiRAV‑1三个基因在叶片苞片不同时期和不同组织中相对表达量,以叶片第一时期L1的表达量为对照,以珙桐β-actin为内参基因.表1 珙桐荧光定量所用引物Tab.1 Primer sequence used in qRT-PCR of D.involu⁃crata Baill.引物名称q-DiAP2-1q-DiDREB-1q-DiRAV-1 DiActin引物序列(5′→3′)F:GACATGGCAGCTATAGAGTACCR:GTCAACATTAGGGTTAGGGF:CGAGATAGTAGAGTTGCCGAGTCTGR:CTGCTCGAAATCGCACTCTCGGF:CCGGTTCAGATGGTTAGACR:GTTACAAAGCTCCAATGGCCF:GGTCGTACAACTGGTATTR:GAGCATAGCCTTCATAGAT表2 拟南芥荧光定量所用引物Tab.2 Primer sequence used in qRT-PCR of A.thaliana引物名称q-AtAP1q-AtAGL24q-AtLFCq-AtFTq-AtSPL-q-AtLFYβ-AtActin引物序列(5′→3′)F:AAGAGGATAGAGAACAAGR:AAGAATCAGTGGAGTATTF:ATGGCGAGAGAGAAGATAAGGR:CCTTCCCAATATGTCTCTCF:CGATAACCTGGTCAAGATR:CTATCCACAAGTTCAAGTAGF:CTACAACTGGAACAACCTTR:AACACGACACGATGAATTF:CCAATAATCGCTGTGACACR:AACCTTGGCTCCTCTGATF:TTATCTGTTCCACTTGTAR:TTTAGGCTTGTTTATGTAAF:CTAATCGTGAGAAGATGACTR:AAGAACAGCCTGAATAGC第 61 卷四川大学学报(自然科学版)第 2 期qRT -RCR 结果如图2,结果发现DiAP 2-1和DiRAV -1这两个基因荧光定量结果大概一致,都在叶片和苞片中表达模式差异大,在叶片三个发育时期先下降后升高,而苞片三个发育时期中则表达量逐渐降低,且苞片中的表达量相比叶片较低.DiDREB -1表达模式却不同,DiDREB -1主要在叶片第一时期高表达,在苞片第三时期高表达,DiDREB -1基因在珙桐叶片和苞片发育过程中表达模式也具有差异性.DiAP 2-1和DiDREB -1主要与苞片第二时期到第三时期发育或苞片由淡绿色转变为乳白色变化有关.在同一时期珙桐不同组织包括第三时期叶片、苞片、根、茎、雄蕊、雌蕊、萼片、芽八个组织中的表达模式分析.如图3可知,DiAP 2-1在苞片、雄雌蕊中表达较高,在根、茎和萼片中几乎不表达;DiDREB -1在雌蕊、萼片和芽中高表达,根、茎和雄蕊几乎不表达;DiRAV -1在萼片和芽中表达较高,只有茎和雄蕊几乎不表达.图1 珙桐目的基因克隆序列比对图Fig.1 Sequence alignment of D.involucrataBaill.图2 珙桐目的基因在叶片和苞片中不同时期的表达情况L1-L3分别指叶片第一时期至叶片第三时期;B1-B3分别指苞片第一时期至苞片第三时期Fig.2 Expression of genes in leaves and bracts of D.involucrata Baill.at different stagesL1-L3 refer to leaf first period to leaf third period ; B1-B3 refer to bract first period to bract third period第 2 期曾沥,等: 珙桐AP2/ERF 家族相关基因的克隆及功能鉴定第 61 卷3.3 亚细胞定位试验将表达载体(由已去除终止密码子的DiAP 2-1、DiDREB -1、DiRAV -1核苷酸序列与pBI 221-EGFP 载体共同构建)转化至当天制备的高质量烟草原生质体中,暗培养后的烟草原生质体通过正置荧光显微镜观察拍照,结果如图 4所示.烟草瞬时转化亚细胞定位实验结果发现,绿色荧光信号显示除了DiAP2-1主要在细胞质中表达以外,Di⁃DREB -1、DiRAV -1都在细胞核中表达.3.4 纯合转基因株系表型分析将野生型、ap 2突变体、DiAP 2-1、DiDREB -1和DiRAV -1转基因株系(每个株系均种植10株)正常培养,对各拟南芥株系的表型进行观察,测定相关生理指标并统计相关数据.对比同一时期的转基因植株、野生型与对应功能突变体的表型见图5和图6.结果表明:DiAP 2-1转基因植株和野生型比图3 珙桐目的基因在不同组织的表达情况L3指叶片第三时期;B3指苞片第三时期Fig.3 Expression of genes in different tissues of D.involucrate Baill at different stagesL3 refer to leaf third period ;B3 refer to bract third period图4 珙桐目标基因亚细胞定位结果GFP ,GFP 荧光;BF ,明场;Merged ,合并图像Fig.4 Subcellular localization of target genes of D.involucrata Baill.GFP , GFP fluorescence ; BF , bright -field ; Merged , merged image第 61 卷四川大学学报(自然科学版)第 2 期较,不论在花期、株高还是微观的花器官中都无明显的区别,如图 6a.而AP2-1对应功能缺失突变体与野生型相比发现有明显的表型差异如图6a,具体表现为花期提前,花瓣减少且花瓣变小,萼片变大,花的形态发生在第1轮和第4轮受到影响,在第2轮没有器官形成,第3轮大部分空置,角果呈短粗状且败育如图6b.DiDREB-1和DiRAV-1两个基因的转基因植株开花时间明显早于野生型植株如图5,而后期株高无明显差异.DiDREB-1和DiRAV-1这两个基因对应的同源拟南芥突变体,只有后期表现为株高比野生型高,其他表型无明显区别如图6c和6d.莲座叶统计实验所用株系为WT、DiAP2-1、DiDREB-1和DiRAV-1异源表达纯合植株.当各株系拟南芥主花序约生长到4~5 cm时,统计拟南芥的莲座叶数目如图7,各株系均统计10株.WT植株开花时的莲座叶平均数为11.9;DiAP2-1异源表达株系开花时的莲座叶平均数为11.6;而DiDREB-1和DiRAV-1异源表达拟南芥株系开花时的莲座叶统计平均数为9.1和9.4,显著少于野生型植株,进一步表明在拟南芥中异源表达DiDREB-1和DiRAV-1可以使植株提前开花,而DiAP2-1异源表达植株与WT植株没有显著差异.图5 目的基因异源表达拟南芥植株早花现象Fig.5 Early flowering of A.thaliana plants with heterolo‑gous expression of target genes图6 突变体、野生型及转基因拟南芥表型Fig.6 Mutant,wild type and transgenic Arabidopsis phe‑notype图7 各拟南芥株系莲座叶数目“*”代表显著性差异(P<0.05),代表目的基因与野生型具有显著性差异Fig.7 The number of rosette leaves“*” represents significantly different(P<0.05), represents thatthe target genes was significantly different from WT图8 转基因拟南芥中关键调控开花基因的表达情况“*”代表显著性差异(P<0.05),代表目的基因与野生型具有显著性差异Fig.8 Expression of key regulatory flowering genes intransgenic Arabidopsis thaliana“*” represents significantly different(P<0.05), represents thatthe target genes was significantly different from WT第 2 期曾沥,等:珙桐AP2/ERF家族相关基因的克隆及功能鉴定第 61 卷3.5 转基因植株中开花调控关键基因表达分析转基因植株中开花调控关键基因表达分析实验结果发现每个株系中6个调控拟南芥开花关键基因的表达模式相似,如图8所示,除了AtAGL24这一个基因以外,其他促花因子的表达量相比野生型都上调.其中异源表达DiAP2-1后,拟南芥AtSPL基因表达水平显著上调并且表达量都高于其他株系,根据开花年龄途径预测该转基因植株表型为晚花,实际却和野生型无区别,所以推测异源AP2类基因可能引起了某一正调控途径使得拟南芥AtSPL基因的表达量上调而表现出晚花现象不明显.这也进一步证明了异源表达DiEDRB-1和DiRAV-1基因可能会影响其他促花因子的表达量上调使得拟南芥表现为花期提前.4 讨论珙桐作为第三纪孑遗植物,同时也是世界著名观赏植物,因其独特的花器官形态而闻名.对于这样一个古老的物种,研究其独特花器官的形成原因,及其花器官对物种生长繁殖的影响是非常有必要的.AP2/ERF家族转录因子可以广泛参与植物次生代谢、花器官发育、植物激素应答和胁迫响应等多种生命活动.对所筛选出的三个来自三个不同亚家族的基因:DiAP2-1、DiDREB-1和DiRAV-1.克隆后三个目的基因的序列与转录组数据序列比对正确.为了探究各个目的基因在细胞中的表达位置.为了探究DiAP2-1、DiDREB-1、DiRAV-1的蛋白在细胞中的亚细胞定位的情况,我们构建了pBI221-DiAP2/ERFs-EGFP表达载体,然后通过PEG-Calcium介导转入烟草原生质体中观察荧光.结果发现除了DiAP2-1的蛋白主要定位在细胞质以外,其余两个基因DiDREB-1、DiRAV-1均定位于细胞核,这与多数其他转录因子亚细胞定位研究中的结果一致[19],另外此结果也表明了不是所有AP2/ERF转录因子家族都定位在细胞核,也有研究中发现拟南芥其他家族的某些转录因子定位于细胞质[20].将可能参与珙桐苞片发育的三个目的基因DiAP2-1、DiDREB-1和DiRAV-1通过花序浸染法获取了拟南芥稳定转化植株.通过对这三个基因在珙桐中的表达模式分析,结合各目的基因的稳定转化植株、对应的功能缺失突变体和野生型植株三者进行表型对比观察,再结合表型现象分析异源表达目的基因对拟南芥调控开花关键基因表达量的变化,验证各目的基因的功能.结果表明:DiAP2-1、DiDREB-1和DiRAV-1三个基因在苞片和叶片中的时空表达和组织特异性表达的相关水平差异都很大;首先是组织特异性表达,在不同组织中,各目的基因主要在花器官中表达而根和茎等其他组织中几乎不表达;表型观察中发现异源表达DiDREB-1和DiRAV-1使拟南芥出现早花现像;对应功能缺失突变体表型观察发现拟南芥缺失AP2基因严重影响了花瓣、萼片以及角果的形态发育;转基因株系中拟南芥花发育相关调控基因的表达水平变化显示,异源表达DiAP2-1、DiDREB-1和DiRAV-1会引起一部分拟南芥促花因子的表达量上调,但在DiAP2-1异源表达植株中AtSPL的表达量显著上调.三个基因主要在花器官中表达,存在组织器官特异性,根据植物花发育ABCDE模型推测出DiDREB-1、DiRAV-1可能属于A类基因,DiAP2-1属于B类基因,因为植物花发育的ABCDE模型还不能解释所有种类植物的花发育类型,内容仍需进一步完善和不断修正,而且珙桐具有独特的花器官且无花瓣,所以本结果只是在原本植物花发育ABCDE模型基础上的一种推测,后续需要进一步证实.结合表型再通过对拟南芥其他已知关键调控基因表达模式的分析,发现DiDREB-1和DiRAV2-1这两个基因异源表达后确实使其他促花因子表达量有一定的上调进而使拟南芥花期提前.SPL和AP2基因都是参与植物花发育年龄途径的重要成员[21],本来预测DiAP2-1的异源表达有一定程度的抑制开花[22],但分析结果后推测可能存在某一正调控途径使得开花促进因子AtSPL表达量上调,最终表型和野生型拟南芥没有区别.研究表明在拟南芥过表达AP2亚家族的ANT和AIL6基因时,拟南芥花的发育受到严重影响,过表达植株的花丝和萼片相较于野生型明显膨大,深入研究发现ANT和AIL6基因可以参与调控生长素的运输[23].在本章结果中也观察到AP2基因对应功能缺失突变体花瓣减少且花瓣变小,萼片变大,并结合表达模式分析DiAP2-1主要在珙桐花器官中表达且在苞片第二时期到第三时期发育过程中表达量差异最大,所以推测该基因在珙桐苞片发育过程中也有与ANT和AIL6基因类第 61 卷四川大学学报(自然科学版)第 2 期似的调控模式并参与珙桐的花期调控和苞片发育等.最近的研究表明异源表达非AP2/ERF家族基因,如异源表达文心兰OnGI基因促进拟南芥开花[24],异源表达牡丹PsSPL基因通过促进LFY基因的表达使转基因拟南芥花期提前,且早期的根长和后期株高都有改变[25].本研究中DiAP2-1转基因拟南芥中暂未观察到花器官的改变但能使AtSPL基因显著上调从而调控花期,这可能与异源表达的基因同源性等问题有关,后续还需要借助其他模式植物进一步探究.本研究发现异源表达珙桐DiAP2-1、DiDREB-1和DiRAV-1引起其他拟南芥关键调控开花基因表达的变化从而调控花期,结合珙桐中的表达模式分析等推测这三个基因都与珙桐苞片发育密切相关,后续还需要更深入的研究进一步证明或阐明在苞片发育中的具体途径.综上所述,本研究认为AP2/ERF基因家族的目的基因在珙桐开花时间调控、花器官的发育过程中具有重要作用,丰富了现有的关于珙桐花发育的理论知识,为未来珙桐的保护和研究提供了理论依据参考文献:[1]Tang X J.Characteristics and value of Dove trees [J].Southwest Horticult, 2002(3): 54.[唐晓军.珙桐的特性与价值[J].西南园艺, 2002(3): 54.][2]He J S, Lin J, Chen W L.The current status of endemic and endangered species Davidia involucrata and thepreserving strategies [J].Biodiv Sci, 1995(4): 213.[贺金生,林洁,陈伟烈.我国珍稀特有植物珙桐的现状及其保护[J].生物多样性, 1995(4): 213.][3]Yu Y T,Xu G B,Wang X P.Literature review of researches on Davidia involucrate Baill [J].Non-WoodForest Res, 2006(4): 92.[禹玉婷,徐刚标,汪晓萍.珙桐研究进展[J].经济林研究, 2006(4): 92.][4]Feng C M,Liu X,Yu Y,et al.Evolution of bract development and B-class MADS box gene expressionin petaloid bracts of Cornus s.l.(Cornaceae)[J].NewPhytol, 2012, 196:631.[5]Causier B,Schwarz-Sommer Z,Davieb B.Floral or‑gan identity: 20 years of ABCs [J].Semin Cell DevBiol, 2010, 21: 73.[6]Shan H,Cheng J,Zhang R,et al.Developmental mechanisms involved in the diversification of flow‑ers [J].Nat Plants, 2019, 5: 917.[7]Komeda Y.Genetic regulation of time to flower inArabidopsis thaliana[J].Annu Rev Plant Biol,2004, 55: 521.[8]Zhang J Y, Wang Q J, Guo Z R.Progresses on plant AP2/ERF transcription factors [J].Hereditas,2012,34:44.[张计育,王庆菊,郭忠仁.植物AP2/ERF类转录因子研究进展[J].遗传, 2012,34: 44.][9]Jofuku K D, Den Boer B G, Van Montagu M,et al.Control of Arabidopsis flower and seed developmentby the homeotic gene APETALA2 [J].Plant Cell,1994, 6: 1211.[10]Matias-Hernandez L, Aguilar-Jaramillo A E, Marin-Gonzalez E,et al.RAV genes:Regulation of floralinduction and beyond [J].Ann Bot-London,2014,114: 1459.[11]Gao C Y,Wu R,Yuan Y,et al.Plant AP2/ERF transcription factors and their role in abiotic stress re‑sponse [J].J Jianghan Univer(Nat Sci Ed),2017,45: 236.[高春艳,吴芮,袁玉,等.植物AP2/ERF转录因子及其在非生物胁迫应答中的作用[J].江汉大学学报(自然科学版), 2017, 45: 236.][12]Zhang Q, Chen J, Li L,et al.Progress in the study of the AP2/ERF family of transcription factors inplants [J].aBiotech, 2018, 34: 1.[张麒,陈静,李俐,等.植物AP2/ERF转录因子家族的研究进展[J].生物技术通报, 2018,34: 1.][13]Xu H.Preliminary studies on the involvement of Ara‑bidopsis thaliana IQM3 in the regulation of photoperi‑odic flower formation [D].Guangzhou:GuangzhouUniversity, 2019.[徐浩.拟南芥IQM3参与光周期成花调控的初步研究[D].广州:广州大学, 2019.][14]Ji H,Zhu Y,Tian S,et al.Downregulation of leaf flavin content induces early flowering and photope‑riod gene expression in Arabidopsis[J].BMC PlantBiol, 2014, 14: 237.[15]Rijpkema A S,Vandenbussche M,Koes R,et al.Variations on a theme: changes in the floral ABCs inangiosperms[J].Semin Cell Dev Biol,2010,21: 100.[16]Soltis D E, Chanderbali A S, Kim S,et al.The ABC model and its applicability to basal angiosperms [J].Ann Bot, 2007, 100: 155.[17]Theissen G, Melzer R, RumUMPLER F.MADS-domain transcription factors and the floral quartet modelof flower development: linking plant development andevolution [J].Development, 2016, 143: 3259.[18]Qu G Z, Zheng T, Liu G,et al.Overexpression of a MADS⁃box gene from birch (Betula platyphylla)第 2 期曾沥,等:珙桐AP2/ERF家族相关基因的克隆及功能鉴定第 61 卷promotes flowering and enhances chloroplast develop‑ment in transgenic tobacco [J].PLoS One, 2013, 8:e63398.[19]Dai Y L.Cloning and functional study of AP2-like transcriptional regulators related to isoprene metabolicpathway in the Taxus chinensis (Pilger)Rehd [D].Shanghai: Fudan University, 2008.[戴怡龄.红豆杉中与异戊二烯代谢途径相关的AP2类转录调控因子的克隆与功能研究[D].上海:复旦大学, 2008.][20]Liang M,Li H,Zhou F,et al.Subcellular distribu‑tion of NTL transcription factors in Arabidopsis thali⁃ana[J].Traffic, 2015, 16: 1062.[21]Yu S, Wang J W.Advances in miR156-mediated age pathways in higher plants [J].Sci Bull,2014,59:1398.[虞莎,王佳伟. miR156介导的高等植物年龄途径研究进展[J].科学通报, 2014, 59: 1398.][22]Jung J H, Seo P J, Kang S K,et al.miR172 signals are incorporated into the miR156 signaling pathwayat the SPL3/4/5 genes in Arabidopsis developmentaltransitions [J]. Plant Mol Biol, 2011, 76: 35.[23]Krizek B.AINTEGUMENTA and AINTEGUME NTA-LIKE6 act redundantly to regulate Arabidopsisfloral growth and patterning [J].Plant Physiol,2009,150: 1916.[24]Fang N Y, Fan R H, Luo Y H,et al.The heterolo‑gous expression of OnGI in Arabidopsis thaliana topromote flowering [J].Acta Horticult Sini,2022,49:841.[方能炎,樊荣辉,罗远华,等.文心兰OnGI在拟南芥中异源表达促进开花[J].园艺学报, 2022, 49: 841.][25]Wang Y Y, Guan S M, Gai S P,et al.Heterologous expression of peony PsSPL gene affects nutrient growthand flowering time in Arabidopsis thaliana[J]. J PlantPhysiol, 2016, 52:1207.[王艳艳,管世铭,盖树鹏,等.异源表达牡丹PsSPL基因影响拟南芥营养生长与开花时间[J].植物生理学报, 2016, 52: 1207.]。
ERF转录因子
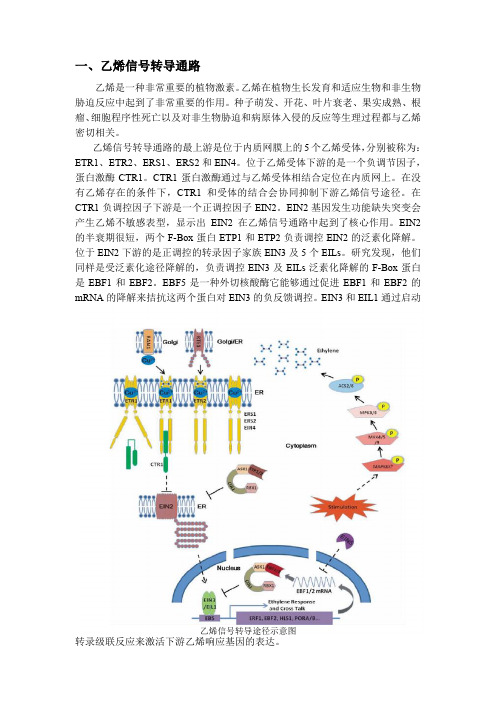
一、乙烯信号转导通路乙烯是一种非常重要的植物激素。
乙烯在植物生长发育和适应生物和非生物胁迫反应中起到了非常重要的作用。
种子萌发、开花、叶片衰老、果实成熟、根瘤、细胞程序性死亡以及对非生物胁迫和病原体入侵的反应等生理过程都与乙烯密切相关。
乙烯信号转导通路的最上游是位于内质网膜上的5个乙烯受体,分别被称为:ETR1、ETR2、ERS1、ERS2和EIN4。
位于乙烯受体下游的是一个负调节因子,蛋白激酶CTR1。
CTR1蛋白激酶通过与乙烯受体相结合定位在内质网上。
在没有乙烯存在的条件下,CTR1和受体的结合会协同抑制下游乙烯信号途径。
在CTR1负调控因子下游是一个正调控因子EIN2。
EIN2基因发生功能缺失突变会产生乙烯不敏感表型,显示出EIN2在乙烯信号通路中起到了核心作用。
EIN2的半衰期很短,两个F-Box蛋白ETP1和ETP2负责调控EIN2的泛素化降解。
位于EIN2下游的是正调控的转录因子家族EIN3及5个EILs。
研究发现,他们同样是受泛素化途径降解的,负责调控EIN3及EILs泛素化降解的F-Box蛋白是EBF1和EBF2。
EBF5是一种外切核酸酶它能够通过促进EBF1和EBF2的mRNA的降解来拮抗这两个蛋白对EIN3的负反馈调控。
EIN3和EIL1通过启动乙烯信号转导途径示意图转录级联反应来激活下游乙烯响应基因的表达。
二、乙烯响应因子(ethylene response factor、ERF)的结构特点及生物信息学分析ERF基因家族是一个很大的转录因子家族,属于AP2/ERF转录因子超家族。
Ohme-Takagi和Shinshi研究发现,GCC box是植物乙烯响应的DNA序列元件;同时他们在烟草(Nicotiana tabacuum)中发现了能特异性结合GCC box元件的数个乙烯响应元件结合蛋白(EREBPs),并发现,EREBPs同GCC元件相结合的结构域是59个保守的氨基酸残基。
甜橙ERF B3亚组转录因子的鉴定和表达分析

引文格式:李玲,刘新军,张保莲,等.甜橙E R F B 3亚组转录因子的鉴定和表达分析[J ].赣南师范大学学报,2022,43(3):77-84.甜橙E R F B 3亚组转录因子的鉴定和表达分析*李 玲1,2,刘新军1,2,张保莲1,张斯羽1,欧阳智刚1,2,†(1.赣南师范大学生命科学学院;2.国家脐橙工程技术研究中心,江西赣州 341000)摘 要:E R F 转录因子是A P 2/E R F 转录因子大家族的一个主要亚家族,E R F 转录因子进一步分为包含B 3(I X )在内的6~7个亚组.研究表明E R F B 3亚组转录因子在植物抗病反应中发挥重要作用.文章在甜橙基因组数据库中筛选了21个C s E R F B 3亚组转录因子㊂通过分析感染黄龙病的柑橘转录组数据发现,有9个E R F B 3亚组转录因子在道县野橘㊁宜昌橙和/或甜橙中有差异表达㊂在外源激素诱导的表达分析中发现6个甜橙E R F B 3亚组转录因子有差异表达.进一步研究发现甜橙E R F B 3亚组转录因子为核定位蛋白,且具有转录激活活性.这为研究甜橙E R F B 3亚组转录因子在植物抗病反应中的功能分析提供理论基础.关键词:甜橙;E R F 转录因子;黄龙病;表达分析中图分类号:S 666 文献标志码:A 文章编号:2096-7659(2022)03-0077-08植物在生长发育过程中一直遭受干旱㊁高盐㊁低温及病虫害等各种生物和非生物胁迫的影响[1].在长期的协同进化中,植物形成了一个复杂㊁有效的信号传导网络,以抵抗外界不利环境[2].转录因子作为反式作用元件,通过与靶标基因上游的顺式作用元件特异性结合来调节基因表达,以转录水平的调控方式来精确地应答胁迫反应[3].近年来研究显示A P 2/E R F (A P E T A L A 2/e t h y l e n e -r e s p o n s i v e e l e m e n t b i n d i n g f a c t o r )㊁b Z -I P (B a s i c -d o m a i n l e u c i n e -z i p pe r )㊁WR K Y ㊁MY B ㊁N A C (N AM ㊁A T A F 1/2和C U C 2)㊁b H L H (B a s i c h e l i x -l o o p -h e l i x )㊁N F -Y (N u c l e a r F a c t o r Y )和C AMT A (C a M -b i n d i n g t r a n s c r i p t i o n a c t i v a t o r )等转录因子参与了调控植物多种逆境胁迫反应[4-11].其中A P 2/E R F 转录因子是最大的家族之一.A P 2/E R F 转录因子编码的蛋白含有高度保守的A P 2结构域.A P 2结构域包含58个或59个氨基酸,与靶标D N A 序列具有高亲和力.根据A P 2结构域的数量和特征,A P 2/E R F 家族可分为四个亚家族,包括A P 2(A P E T A L A 2)㊁R A V (r e l a t e d t o A B I 3/V P 1)㊁R E B (脱水反应性元素结合蛋白,d e h y d r a t i o n -r e s po n s i v e e l e m e n t b i n d i n g p r o t e i n )和E R F (乙烯反应因子,e t h y l e n e -r e s po n s i v e f a c t o r )[12].A P 2家族含2个A P 2/E R E B P 结构域,E R F 和D R E B 家族仅含1个A P 2/E R E B P 结构域,这两个家族均又进一步细分为6或7个亚组(A 1-A 6;B 1-B 6或-B 7).R A V 家族除含有一个A P 2/E R E B P 结构域外,还包含1个B 3结构域[13-14].E R F 家族是A P 2/E R F 转录因子大家族中的一个主要亚家族,E R F 蛋白可以与许多乙烯调节的防御基因启动子区域G C C 框(A G C C G C C )的顺式元件特异性结合,起转录激活因子或阻遏物的作用[15].E R F转录因子广泛分布于植物中,N a k a n o 等(2006)在拟南芥(A r a b i d o p s i s t h a l i a n a )和水稻(O r yz a s a t i v a )基因组中分别鉴定了65和77个E R F 转录因子[16-17].最近几年随着多种植物基因组测序完成,相继在凤梨(A n a n a s c o m o s u s )㊁芹菜(A p i u m g r a v e o l e n s )㊁二穗短柄草(B r a c h y p o d i u m d i s t a c h yo n )㊁甘蓝型油菜(B r a s -s i c a n a p u s )㊁棉花(G o s s s y p i u m r a i m o n d i i )㊁苹果(M a l u s ˑd o m e s t i c a )㊁甘蔗(S a c c h a r u m s po n t a n e u m )㊁番茄(S o l a n u m l y c o p e r s i c u m )㊁绿豆(V i g n a r a d i a t a )㊁葡萄(V i t i s v i n i f e r a )和玉米(Z e a m a ys )等中筛选和鉴定了E R F 转录因子[18-28].E RF 亚家族不同亚组中的成员具有不同的功能.研究表明E R F B 3亚组转录因子在植物防御病菌的反2022年 赣南师范大学学报 ɴ.3第三期 J o u r n a l o f G a n n a n N o r m a l U n i v e r s i t y M a y.2022*收稿日期:2021-03-24 D O I :10.13698/j.c n k i .c n 36-1346/c .2022.03.017 基金项目:江西省重点研发计划项目(20171A C F 60022) 作者简介:李玲(1997-),女,江西新余人,赣南师范大学生命科学学院硕士研究生,研究方向:园艺植物病理. †通讯作者:欧阳智刚(1984-),男,江西新余人,赣南师范大学生命科学学院讲师,博士,研究方向:植物病理学.87赣南师范大学学报2022年应中发挥重要作用.本文根据E R F B3亚组转录因子的结构特点,在甜橙基因组数据库中筛选和鉴定了21个E R F B3亚组转录因子,进一步分析了甜橙E R F B3亚组转录因子受黄龙病菌和外源激素的诱导表达情况.同时研究了甜橙E R F B3亚组转录因子的亚细胞定位及转录激活活性.本文的研究可以为甜橙E R F B3亚组转录因子的功能分析提供理论参考.1材料与方法1.1供试材料和前处理本试验所用柑橘材料为赣南早(C i t r u s s i n e n s i s O s b e c k c v.G a n n a n z a o)幼苗,均由赣南师范大学国家脐橙工程与技术研究中心提供.激素诱导处理:分别用1mM S A,100μM J A,100μM A B A喷雾处理赣南早幼苗,对照为等量喷无菌水,恒温保湿培养[29].分别于0h㊁24h和48h后采集样品叶片,-80ħ下保存. 1.2植物叶片总R N A提取将植物叶片置于液氮中磨碎,每0.5g叶片加入1m L T r i z o l,震荡混匀,并室温放置5m i n,4ħ10000g 离心10m i n,取上清,每使用1m L T r i z o l加入0.2m L氯仿,剧烈振荡15s,于室温放置5m i n.4ħ10000g 离心10m i n,取上清,重复加入0.2m L和0.5m L氯仿萃取2次.离心后把水相转移到新管中,等体积加入异丙醇,于室温放置10m i n.4ħ10000g离心10m i n,弃上清.用75%乙醇洗涤R N A沉淀.每使用1m L T r i z o l至少加1m L75%乙醇.4ħ不超过5000g离心5m i n,弃上清.室温放置干燥5m i n,再加入20μL~ 30μL无R N a s e的水溶解R N A,置于-80ħ下保存.1.3柑橘E R F B3亚组转录因子的系统筛选从柑橘基因组数据库(h t t p s://w w w.c i t r u s g e n o m e d b.o r g/)下载甜橙全基因组1.0(f t p://f t p.b i o i n f o. w s u.e d u/w w w.c i t r u s g e n o m e d b.o r g/C i t r u s_s i n e n s i s/C.s i n e n s i s_H z a u_v1.0_g e n o m e/a n n o t a t i o n/)蛋白组数据.利用HMM E R3.1b2软件包中的h mm b u i l d程序对E R F结构域种子序列(P F04404)建立隐马尔可夫模型,利用h mm s e a r c h程序在甜橙蛋白组数据中进行搜索(E-v a l u e<10-3),获得含E R F结构域蛋白.将挖掘出的所有含E R F结构域蛋白和已报道的893条真核及原核生物中的含E R F结构域蛋白序列一起利用h mm a l i g n程序基于P F04404的隐马尔可夫模型进行多重序列比对,得到比对序列.利用M E G A7软件打开比对文件,并人工调整.利用S m a r t m o d e l s e l e c t i o n软件对比对序列最适的最大似然法建树模型进行检测.根据最适建树模型,利用P h y M L3.0对比对文件建立系统发生关系,利用外群法确定树根.系统发生树上,与植物含E R F结构域序列形成单系群的甜橙含E R F结构域序列为甜橙的E R F B3亚组转录因子.1.4甜橙E R F B3亚组转录因子亚细胞定位以甜橙c D N A为模板,根据数据库序列设计引物,特异性扩增目的基因全长序列(引物见附表1),将P C R产物插入瞬时表达载体p G R106中,分别命名为p G R106:C s E R F B3-9/10/13/20/21并转入农杆菌G V3101中.将携带p G R106:C s E R F B3-9/10/13/20/21的农杆菌在含有50m g/L卡那霉素(K a n)㊁50m g/L氯霉素(C h o l)和25m g/L庆大霉素(G e n)的Y E P平板上划线,放置于28ħ恒温培养箱内培养2d,并采用菌落P C R验证转化结果.按1:50~1:100的比例接种于10m L含相同抗生素的Y E P中,28ħ㊁220r p m培养8h~12h扩大培养至O D600=0.8~1.2,4000r p m,4ħ离心10m i n收集菌体,弃上清,用含有乙酰丁香酮(50mm o l/L,A S)的侵染液(1mm o l/L M g C l2,1mm o l/L M E S,p H5.6)悬浮菌体,使其O D600=1.0~1.5,室温放置3h后,以携带p G R106:G F P载体的G V3101为阴性对照,用不带针头的1m L 针管抵住烟草叶片的背面,注入叶片,48h后在激光共聚焦显微镜下观察蛋白定位.1.5甜橙E R F B3亚组转录因子的转录活性分析以甜橙c D N A为模板,根据数据库序列设计C s E R F B3-9/10/13/20/21引物特异性P C R扩增其全长序列(引物见附表1),将P C R产物酶切插入载体p G B K T7中.将重组质粒和p G B K T7空白载体分别转入酵母菌株Y R G-2中.酵母转化方法:用Y P D A固体培养基(10g/L Y e a s t e x t r a c t,20g/L T p y p t o n e,20g/L A g a r,2%d e x t r o s e g l u c o s e)培养酵母菌株2d~3d后,刮取25μL酵母菌落悬于1m L d d H2O中,高速离心5s后,用1m L100mM L i A c悬浮沉淀;30ħ水浴中温育5m i n,高速离心5s,弃上清;在沉淀物中按顺序分别加入以下液体:240μL P E G(50%w/v,重量/体积),36μL L i A c(1.0M)㊁50μL s s D N A(2.0m g/m L)㊁5μL p l a s m i d D N A(100n g至5μg)㊁20μL d d H2O;涡旋1m i n,42ħ温育40m i n后;12000r p m离心10s,弃去上清,用200μL~400μL d d H2O重悬沉淀,取适量涂于S D/-T r p培养基上,30ħ培养2d.挑单菌落分别划线于S D/-T r p/-H i s培养基上,2d后观察转录因子活性结果.1.6 R T -qP C R 按S u p e r S c r i p t I I I K i t (I n v i t r o ge n ,中国上海)方法对提取的R N A 进行反转录合成c D N A.根据数据库中的E R F B 3亚组转录因子的序列设计R T -qP C R 特异性引物(引物见附表1),以基因C i t E F 1作为内参.每个R T -q P C R 反应体系包含12.5μL S Y B R P r e m i x E x T a qT M ㊁1μL 正反向引物(10μm o l /L )㊁1μL c D -N A 模板和9.5μL R N A s e -f r e e 水.反应程序为94ħ5m i n ;94ħ45s ,58ħ45s ,72ħ45s ,34个循环.图1 甜橙(C .s i n e n s i s )㊁拟南芥(A .t h a l i a n a )㊁番茄(S .l y c o p e r s i c u m )㊁甘蔗(S .s p o n t a n e u m )㊁玉米(Z .m a y s s s p )和葡萄(V .v i n i fe r a )E R F B 3亚组转录因子氨基酸序列系统进化树2 试验结果2.1 甜橙E R F B 3亚组转录因子系统进化分析本文在柑橘数据库中筛选和鉴定了21个甜橙E RF B 3亚组转录因子.根据染色体上的位置,我们将其分别命名为C s E R F B 3-1~21(对应的G e n eI D 分别为:C s 1g 03250_1㊁C s 1g03260_1㊁C s 1g 03270_1㊁C s 1g 03280_1㊁C s 1g03290_1㊁C s 1g 03300_1㊁C s 1g 03310_1㊁C s 2g 05270_1㊁C s 2g 05280_1㊁C s 5g 08360_1㊁C s 5g 23980_1㊁C s 5g 24010_1㊁C s 5g 29870_1㊁C s 5g 29880_1㊁C s 5g 29890_1㊁C s 5g 29900_1㊁C s 8g 12780_1㊁C s 9g 10680_1㊁C s 9g 13610_1㊁C s 9g13620_1㊁C s 9g13630_1).将C s E R F B 3亚组转录因子蛋白序列与拟南芥㊁甘蔗㊁番茄㊁玉米和葡萄共149个E R FB 3亚组转录因子蛋白序列构建亲缘关系进化树(如图1所示).E R F B 3亚组转录因子被分为4组,其中第1组中包括12个甜橙㊁4个拟南芥㊁5个甘蔗㊁12个番茄㊁5个玉米和6个葡萄E R F B 3亚组转录因子;第2组包括1个甜橙㊁1个甘蔗㊁1个番茄㊁1个玉米和1个葡萄E R F B 3亚组转录因子;第3组分别包括4个甜橙㊁10个拟南芥㊁5个甘蔗㊁11个番茄㊁2个玉米和20个葡萄E R F B 3亚组转录因子;第4组4个甜橙㊁3个拟南芥㊁5个番茄㊁12个甘蔗㊁10个玉米和14个葡萄E R F B 3亚组转录因子.此外我们发现几个甜橙E R F B 3亚组转录因子与番茄已报道的具有抗病功能的转录因子聚类关系比较近,比如C s E R F B 3-10和番茄S l E R F .A 1㊁C s E R F B 3-17和番茄S l E R F .C 3㊁C s E R F B 3-19与C s E R F B 3-20和S l E R F .B 4㊁C s E R F B 3-21和S l E R F .A 4以及C s E R F B 3-13和拟南芥A t E R F 93/94等.图2 甜橙E R F B 3亚组转录因子在不同组织中的表达分析2.2 甜橙E R F B 3亚组转录因子在不同组织中的表达特征我们在柑橘基因组数据库(h t t p://c i t r u s .h z a u .e d u .c n /o r a n ge /)中下载甜橙E R F B 3亚组转录因子在愈伤组织㊁花㊁叶和果实中的表达数据进行分析,发现甜橙E R F B 3亚组转录因子在不同组织中的表达模式不一样(如图2所示.C a l -l u s :愈伤组织;F l o w e r :花;L e af :叶;F r u i t :果实).C s E R F B 3-3/7/8/11/12/14/15/16/17/18在愈伤组织㊁花㊁叶和果实这四个组织中表达均很低.在其余的C s E R F B 3亚组转录因子中,除了C s E R F B 3-19之外,其他C s E R F B 3亚组转录因子均在果实中表达最高,叶片中的表达量次之.2.3 甜橙E R F B 3亚组转录因子在感染黄龙病不同柑橘品种的表达特征甜橙E R F B 3亚组转录因子对黄龙病菌有着不同的响应,相同基因在不同的品种中也存在差异表达.我们根据不同品种感染黄龙病的转录组数据[30],分析了柑橘E R F B 3亚组转录因子在道县野橘(C .d a o x i a n e n s i s )㊁宜昌橙(C .97第3期 李 玲,刘新军,张保莲,等 甜橙E R F B 3亚组转录因子的鉴定和表达分析i c h a n g e n s i s )和甜橙中应答黄龙病侵染的表达特征(如图3所示.F P KM.C D-H L B :道县野橘黄龙病患病植株F P KM 值;F P KM.C D-MO C K :健康道县野橘F P KM 值;F P KM.C I -H L B :宜昌橙黄龙病患病植株F P -KM 值;F P KM.C D-MO C K :健康宜昌橙F P KM 值;F P KM.C S _H L B :甜橙黄龙病患病植株F P KM 值;F P -KM.C D _MO C K :健康甜橙F P KM 值).图3 甜橙E R F B 3亚组转录因子受H L B 病原菌诱导的表达分析图4 甜橙E R F B 3亚组转录因子受激素诱导的表达分析该转录组数据显示,C s E R F B 3-1/2/3/7/8/11/12/14/15/16/17/18这12个基因的表达丰度在3个品种中表达都非常低,F P KM (F r a g m e n t s P e r K i l o b a s e M i l l i o n )值大部分为0或接近于0,这与柑橘基因组数据库中的表达情况相似(图2).在道县野橘中,C s E R F B 3-2/5/6/21这四个基因在感染黄龙病后上调表达显著,相反C s E R F B 3-10和C s E R F B 3-13两个基因下调表达显著;在宜昌橙中,C s E R F B 3-5和B 3-6两个基因是上调表达,C s E R FB 3-19/20/21为下调表达;在甜橙中,C s E R F B 3-5/6/9/19/20/21六个基因显著上调表达,只有C s E R F B 3-1一个基因下调表达.同时还发现C s E R F B 3-5和B 3-6这两个基因在3个品种中均呈上调表达,但C s E R F B 3-21在道县野橘和甜橙中上调表达显著,而在宜昌橙中则为下调表达显著.2.4 激素诱导E R F B 3亚组转录因子的表达特征根据柑橘感染黄龙病菌的转录组数据,我们挑选了表达相对较高的C s E R F B 3亚组转录因子(C s E R F B 3-4/5/6/9/10/13/19/20/21)进行外源激素诱导表达分析(如图4所示).通过荧光定量P C R 技术分析激素处理后甜橙E R F B 3亚组转录因子表达情况,结果显示C s E R F B 3-9/10/13/20/21基因在12h 和24h 两个时间点受S A 诱导上调表达显著,而C s E R F B 3-4显著下调表达.受J A 诱导12h 或/和24h 后C s E R F B 3-10/13/20/21四个基因显著上调表达,同样C s E R FB 3-4基因下调表达显著.A B A 诱导对C s E R F B 3亚组转录因子的表达影响较小,只有在24h 时,C s E R F B 3-4和C s E R F B 3-21分别下调和上调表达显著.另外C s E R F B 3-5/6/19这三个基因在3种激素诱导下表达均无差异.2.5 甜橙E R F B 3亚组转录因子亚细胞定位为了明确甜橙E R F B 3亚组转录因子的亚细胞定位,根据E R F B 3亚组转录因子应答黄龙病病原菌的表达情况,我们挑选了C s E R F B 3-9/10/13/20/21五个转录因子进行亚细胞定位研究.结果显示:在对照组中,本氏烟细胞核和细胞膜上均可观察到G F P 信号,而C s E R F B 3-9/10/13/20/21-G F P 融合蛋白则只08赣南师范大学学报 2022年定位在细胞核上(如图5所示),表明C s E R F B 3-9/10/13/20/21-G F P 为核定位蛋白.图5 甜橙E R F B 3亚组转录因子亚细胞定位图6 甜橙E R F B 3亚组转录因子转录激活活性检测2.6 甜橙E R F B 3亚组转录因子转录活性分析在携带对照载体p G B K T 7的酵母细胞中,载体上酵母转录激活因子G A L 4D N A 结合域(D N A b i n d i n g do m a i n ,B D )可以和上游激活序列结合,但不能引起报告基因L a c Z转录.如将一段具有转录激活活性的转录因子构建到pG B K T 7载体上,其表达产生的B D 与上游激活序列结合,同时引起下游报告基因L a c Z 的转录,进而水解培养基上的x -α-G a l,使菌落产生蓝斑.利用这一原理,我们对C s E R F B 3-9/10/13/20/21进行了转录活性验证.结果表明(如图6所示),携带p G B K T 7:C s E R F B 3-9/10/13/20/21的酵母均能在双缺培养基S D /-T r p /-H i s 上正常生长,并且双缺培养基中加入x -α-ga l 后显示蓝色反应,说明C s E R F B 3-9/10/13/20/21均具有转录激活活性.3 讨论E RF 转录因子是植物A P 2/E R F 转录因子家族中最大的亚家族之一,根据其结构特点又进一步分为6~7个亚组(B 1~B 7)不等,比如在拟南芥㊁水稻㊁番茄㊁葡萄和玉米等植物中分为6个亚组[16-17,25,27-28],而在二穗短柄草和棉花等植物中则分为7个亚组[20,22].在E R F 亚家族中,E R FB 3亚组成员相对较多.比如在小麦中有22个,在拟南芥中有18个,葡萄有37个[16,27-28].本文在柑橘基因组数据库中筛选鉴定了21个E R F B 3亚组成员.我们进一步对植物E R F B 3亚组转录因子进行了聚类分析,发现甜橙E R F B 3亚组转录因子与番茄和拟南芥中已报道的具有抗病功能的转录因子聚类关系比较近,比如C s E R F B 3-10和番茄S l E R F .A 1㊁C s E R F B 3-17和番茄S l E R F .C 3㊁C s E R F B 3-19与C s E R F B 3-20和S l E R F .B 4㊁C s E R F B 3-21和S l E R F .A 4以及C s E R F B 3-13和拟南芥A t E R F 93/94等.聚类关系较近的基因在功能上具有相似性,我们推测与报道的具有抗病功能的转录因子聚类关系较近的甜橙C s E R F B 3亚组转录因子可能在抗病反应中也发挥作用.植物E R F 蛋白可以作为转录激活物或抑制因子发挥作用[31].顺式作用元件G C C 盒是E R F 转录因子的主要靶位点,E R F 转录因子能结合启动子中G C C 盒调控靶标基因的表达.研究表明拟南芥A t E R F 15(A t E R F 93)㊁T E R F 1㊁番茄S l E R F .A 1㊁S l E R F .B 4和S l E R F .C 3在酵母细胞中均具有转录激活活性[29,32-33].此外拟南芥E R F 1和O R A 59可以和J A /E T 响应的病原相关基因P D F 1.2和b -C H I 上的C G G b o x 结合来诱导其表达[34-35].本文中,C s E R F B 3-9/10/13/20/21等5个甜橙E R F B 3亚组转录因子也均有转录激活作用,并且位于细胞核内,因此我们推测该5个甜橙E R F B 3亚组转录因子也具有诱导含有C G G b o x 基因表达的能力.植物激素水杨酸㊁茉莉酸和乙烯在抗生物胁迫信号转导途径的调节中起主要作用.这种调节是通过连接不同途径的复杂调节网络来实现的,从而使每个途径根据需要协助或拮抗其他途径来微调对单个病原体的防御反应.一般认为S A 在激活对活体营养性病原菌的防御中起主要作用,而J A 和E T 则对坏死性病原菌攻击的防御相关,此外S A 抗病信号途径和J A /E T 抗病信号途径间存在相互拮抗的关系.比如S A 可以通过下调O R A 59的表达拟制J A 应答相关基因的表达.E F R B 3亚组转录因子能通过依赖S A 和/或J A 途径18第3期 李 玲,刘新军,张保莲,等 甜橙E R F B 3亚组转录因子的鉴定和表达分析28赣南师范大学学报2022年激活植物对病原菌的抵抗力.研究表明G b E R F b㊁G m E R F113㊁拟南芥E R F1(A t E R F92)㊁A t E R F15 (A t E R F93)㊁O R A59(A t E R F94)㊁A T E R F14(A t E R F97)㊁N b E R F173㊁番茄S l E R F.A1㊁S l E R F.B4㊁S l E R F.C3㊁Z m E R F105㊁小麦T a E R F3和T a A P2-155等E F R B3亚组转录因子参与了植物对病原菌的防御反应[29,33,35-36].反过来E R F B3亚组转录因子受病原菌诱导存在差异表达,例如S l E R F.A1㊁S l E R F. B4和S l E R F.C3受病原菌的诱导上调表达显著[29].本文C s E R F B3-9/10/13/20/21被S A和/或J A诱导表达,同时也受黄龙病菌的诱导上调表达显著.我们推测这5个E R F B3亚组转录因子可能通过依赖S A 和J A信号传导途径正调控甜橙对黄龙病菌的抗性反应.然而要证明这些推测,还需要通过进一步的实验验证,比如通过过表达或基因沉默来验证基因功能.参考文献:[1] S A I J O Y,L O O E P I.P l a n t i mm u n i t y i n s i g n a l i n t e g r a t i o n b e t w e e n b i o t i c a n d a b i o t i c s t r e s s r e s p o n s e s[J].N e w P h y t o l o g i s t,2020,225(18):87-104.[2]S T A V R I D O U E,V O U L G A R I G,B O S MA L I I,e t a l.P l a n t a d a p t a t i o n t o s t r e s s c o n d i t i o n s:t h e c a s e o f g l u t a t h i o n e s t r a n s f e r a s e s(G S T s)[J].B i o t i c a n d A b i o t i c S t r e s s T o l e r a n c e i n P l a n t s,2018,52:173-202.[3] L A T A C,P R A S A D M.R o l e o f D R E B s i n r e g u l a t i o n o f a b i o t i c s t r e s s r e s p o n s e s i n p l a n t s[J].J o u r n a l o f E x p e r i m e n t a l B o t a n y,2011,62(14):4731-4748.[4] MY E R S Z A,HO L T B F.N U C L E A R F A C T O R-Y:s t i l l c o m p l e x a f t e r a l l t h e s e y e a r s[J].C u r r e n t O p i n i o n i n P l a n t B i o l o g y,2018,45:96-102.[5] S H K O L N I K D,F I N K L E R A,P A S MA N I K-C HO R M,e t a l.C A L MO D U L I N-B I N D I N G T R A N S C R I P T I O N A C T I V A T O R6:A k e yr e g u l a t o r o f N a+h o m e o s t a s i s d u r i n g g e r m i n a t i o n[J].P l a n t P h y s i o l o g y,2019,180(2):1101-1118.[6] T H I R UMA L A I K UMA R V P,D E V K A R V,M E H T E R O V N,e t a l.N A C t r a n s c r i p t i o n f a c t o r J U N G B R U N N E N1e n h a n c e s d r o u g h t t o l-e r a n c e i n t o m a t o[J].P l a n t B i o t e c h n o l o g y J o u r n a l,2018,16(2):354-366.[7] Y U J J,D O N G H K,L E E H,e t a l.K n o c k o u t o f S l M S10g e n e(S o l y c02g079810)e n c o d i n g b H L H t r a n s c r i p t i o n f a c t o r u s i n g C R I S P R/C a s9s y s t e m c o n f e r s m a l e s t e r i l i t y p h e n o t y p e i n t o m a t o[J].P l a n t s,2020,9(9):1189.[8] Z H A O Q,HU R,L I U D,e t a l.T h e A P2t r a n s c r i p t i o n f a c t o r N t E R F172c o n f e r s d r o u g h t r e s i s t a n c e b y m o d i f y i n g N t C A T[J].P l a n t B i o-t e c h n o l o g y J o u r n a l,2020,18(12):2444-2455.[9] Z H E N G X W,C H E N G L,L I N G Q,e t a l.T h e MY B t r a n s c r i p t i o n f a c t o r T a PH R3-A1i s i n v o l v e d i n p h o s p h a t e s i g n a l i n g a n d g o v e r n sy i e l d-r e l a t e d t r a i t s i n b r e a d w h e a t[J].J o u r n a l o f E x p e r i m e n t a l B o t a n y,2020,71(19):5808-5822.[10] L I Z Y.B a s i c l e u c i n e z i p p e r t r a n s c r i p t i o n f a c t o r S l b Z I P1m e d i a t e s s a l t a n d d r o u g h t s t r e s s t o l e r a n c e i n t o m a t o[J].B M C P l a n t B i o l o g y,2018,18(1):83.[11] Z HU D,HO U L,X I A O P,e t a l.V v WR K Y30,a g r a p e WR K Y t r a n s c r i p t i o n f a c t o r,p l a y s a p o s i t i v e r e g u l a t o r y r o l e u n d e r s a l i n i t ys t r e s s[J].P l a n t S c i e n c e,2019,280:132-142.[12]S A K UMA Y,L I U Q,D U B O U Z E T J G,e t a l.D N A-b i n d i n g s p e c i f i c i t y o f t h e E R F/A P2d o m a i n o f A r a b i d o p s i s D R E B s,t r a n s c r i p t i o nf a c t o r s i n v o l v e d i n d e h y d r a t i o n-a n d c o l d-i n d u c i b l eg e n e e x p r e s s i o n[J].B i o ch e mi c a l a n d B i o p h y s i c a l R e s e a r c h C o mm u n i c a t i o n s,2002,290(3):998-1009.[13]S H I G Y O M,I T O M.A n a l y s i s o f g y m n o s p e r m t w o-A P2-d o m a i n-c o n t a i n i n g g e n e s[J].D e v e l o p m e n t G e n e s a n d E v o l u t i o n,2004,214:105-114.[14]S WAM I N A T H A N K,P E T E R S O N K,J A C K T.T h e p l a n t B3s u p e r f a m i l y[J].T r e n d s i n P l a n t S c i e n c e,2008,13(12):647-655.[15] BüT T N E R M,S I N G H K B.A r a b i d o p s i s t h a l i a n a e t h y l e n e-r e s p o n s i v e e l e m e n t b i n d i n g p r o t e i n(A t E B P),a n e t h y l e n e-i n d u c i b l e,G C Cb o x D N A-b i n d i n g p r o t e i n i n t e r ac t s w i t h a n o c s e l e m e n t b i nd i n g p r o te i n[J].P r o c e e d i n g s of t h e N a t i o n a l A c a d e m y o f S c i e n c e s o f t h e U n i t-e d S t a t e s of A m e r i c a,1997,11(11):5961-5966.[16] N A K A N O T,S U Z U K I K,F U J I MU R A T,e t a l.G e n o m e-w i d e a n a l y s i s o f t h e E R F g e n e f a m i l y i n A r a b i d o p s i s a n d r i c e[J].P l a n t P h y s i-o l o g y,2006,140(2):411-432.[17] MUHAMMA D R,H E G Y,Y A N G G X,e t a l.A P2/E R F t r a n s c r i p t i o n f a c t o r i n r i c e:g e n o m e-w i d e c a n v a s a n d s y n t e n i c r e l a t i o n s h i p sb e t w e e n m o n oc o t s a nde u d i c o t s[J].E v o l u t i o n a r y B i o i nf o r m a t i c s,2012,8:321-355.[18] HU A N G Y M,L I U Y H,Z H A N G M,e t a l.G e n o m e-w i d e i d e n t i f i c a t i o n a n d e x p r e s s i o n a n a l y s i s o f t h e E R F t r a n s c r i p t i o n f a c t o r f a m i l yi n p i n e a p p l e(A n a n a s c o m o s u s(L.)M e r r.)[J].P e e r J,2020,8(4):e10014.[19] L I M Y,L I U J X,H A O J N,e t a l.G e n o m i c i d e n t i f i c a t i o n o f A P2/E R F t r a n s c r i p t i o n f a c t o r s a n d f u n c t i o n a l c h a r a c t e r i z a t i o n o f t w o c o l dr e s i s t a n c e-r e l a t e d A P2/E R F g e n e s i n c e l e r y(A p i u m g r a v e o l e n s L.)[J].P l a n t a,2019,250(4):1265-1280.[20] C U I L,F E N G K,WA N G M,e t a l.G e n o m e-w i d e i d e n t i f i c a t i o n,p h y l o g e n y a n d e x p r e s s i o n a n a l y s i s o f A P2/E R F t r a n s c r i p t i o n f a c t o r sf a m i l y i n B r a c h y p o d i u m d i s t a c h y o n[J].B m c G e n o m i c s,2016,17(1):636-645.[21] G H O R B A N I R,Z A K I P O U R Z,A L E MA Z A D E H A,e t a l.G e n o m e-w i d e a n a l y s i s o f A P2/E R F t r a n s c r i p t i o n f a c t o r s f a m i l y i n B r a s s i c an a p u s[J].P h y s i o l o g y a n d M o l e c u l a r B i o l o g y o f P l a n t s,2020,26:1463-1476.[22] L I U C,Z HA N G T.E x p a n s i o n a n d s t r e s s r e s p o n s e s o f t h e A P2/E R E B P s u p e r f a m i l y i n c o t t o n[J].B m c G e n o m i c s,2017,18(1):118.[23] L I H ,D O N G Q ,Z H A O Q ,e t a l .I s o l a t i o n ,s e q u e n c i n g ,a n d e x p r e s s i o n a n a l y s i s o f 30A P 2/E R F t r a n s c r i p t i o n f a c t o r s i n a p p l e [J ].P e e r J ,2020,8(9):e 8391.[24] L I P T ,C HA I Z ,L I N P D ,e t a l .G e n o m e -w i d e a n a l y s i s o f A P 2/E R F t r a n s c r i p t i o n f a c t o r s i n s u g a r c a n e S a c c h a r u m s po n t a n e u m L .R e -v e a l s f u n c t i o n a l d i v e r g e n c e d u r i n g d r o u gh t ,s a l t s t r e s s a n d p l a n t h o r m o n e s t r e a t m e n t [J ].B m c G e n o m i c s ,2017,21:685.[25] S H A R MA M K ,K UMA R R ,S O L A N K E A U ,e t a l .I d e n t i f i c a t i o n ,p h y l o g e n y ,a n d t r a n s c r i p t p r o f i l i n g o f E R F f a m i l y g e n e s d u r i n g de -v e l o pm e n t a n d a b i o t i c s t r e s s t r e a t m e n t s i n t o m a t o [J ].M o l e c u l a r G e n e t i c s a n d G e n o m i c s ,2010,284(6):455-475.[26] L A B B O A M ,M E HMO O D M ,A K H T A R M N ,e t a l .G e n o m e -w i d e i d e n t i f i c a t i o n o f A P 2/E R F t r a n s c r i p t i o n f a c t o r s i n m u n gb e a n (V i g n a r a d i a t a )a n d e x p r e s s i o n p r o f i l i n g o f t h e V r D R E B s u b f a m i l y u n d e r d r o u g h t s t r e s s [J ].C r o p an d P a s t u r e S c i e n c e ,2018,69:1009-1019.[27] Z HU A N G J ,P E N G R H ,C H E N G Z M ,e t a l .G e n o m e -w i d e a n a l y s i s o f t h e p u t a t i v e A P 2/E R F f a m i l y ge n e s i n V i t i s v i n if e r a [J ].S c i e n -t i a H o r t i c u l t u r a e ,2009,123(1):73-81.[28] D U H ,HU A N G M ,Z HA N G Z ,e t a l .G e n o m e -w i d e a n a l y s i s o f t h e A P 2/E R F g e n e f a m i l y i n m a i z e w a t e r l o g g i n g s t r e s s r e s po n s e [J ].E u p h yt i c a ,2014,198(1):115-126.[29] O U Y A N G Z ,L I U S X ,HU A N G L H ,e t a l .T o m a t o S l E R F .A 1,S l E R F .B 4,S l E R F .C 3a n d S l E R F .A 3,m e m b e r s o f B 3g r o u p of E R F f a m i l y ,a r e r e q u i r e d f o r r e s i s t a n c e t o B o t r yt i s c i n e r e a [J ].F r o n t i e r s i n P l a n t S c i e n c e ,2016,142(7):1964.[30] WU H ,HU Y ,F U S ,e t a l .C o o r d i n a t i o n o f m u l t i p l e r e g u l a t i o n p a t h w a y s c o n t r i b u t e s t o t h e t o l e r a n c e o f a w i l d c i t r u s s pe c i e s (C i t r u s i c h a n g e n s i s '2586')a g a i n s t H u a n g l o n g b i n g [J ].P h y s i o l o g i c a l a n d M o l e c u l a r P l a n t P a t h o l o g y ,2020,109:101457.[31] HU A N G P Y ,Z HA N G J S ,J I A N G B ,e t a l .N I N J A -a s s o c i a t e d E R F 19n e g a t i v e l y r e g u l a t e s A r a b i d o p s i s p a t t e r n -t r i g g e r e d i mm u n i t y[J ].J o u r n a l o f E x p e r i m e n t a l B o t a n y,2019,70(3):1033-1047.[32] Z HA N G H ,L I A ,Z H A N G Z ,e t a l .E t h y l e n e r e s p o n s e f a c t o r T E R F 1,r e g u l a t e d b y ET H Y L E N E -I N S E N S I T I V E 3-l i k e f a c t o r s ,f u n c -t i o n s i n r e a c t i v e o x y g e n s p e c i e s (R O S )s c a v e n g i n g i n t o b a c c o (N i c o t i a n a t a b a c u m L .)[J ].S c i e n t i f i c R e po r t s ,2016,6:29948.[33] Z HA N G H J ,HU A N G L ,D A I Y ,e t a l .A r a b i d o p s i s A t E R F 15p o s i t i v e l y r e g u l a t e s i mm u n i t y a g a i n s t P s e u d o m o n a s s y r i n ga e p v .t o -m a t o D C 3000a n d B o t r yt i s c i n e r e a [J ].F r o n t i e r s i n P l a n t S c i e n c e ,2015,6:686.[34] L O R E N Z O O ,P I Q U E R A S R ,S A N C H E Z -S E R R A N O J J ,e t a l .E t h y l e n e r e s p o n s e f a c t o r 1i n t e g r a t e s s i g n a l s f r o m e t h yl e n e a n d j a s -m o n a t e p a t h w a ys i n p l a n t d e f e n s e [J ].T h e P l a n t C e l l ,2003,15(1):165-178.[35] P R éM ,A T A L L A H M ,C H AM P I O N A ,e t a l .T h e A P 2/E R F d o m a i n t r a n s c r i p t i o n f a c t o r O R A 59i n t e g r a t e s j a s m o n i c a c i d a n d e t h yl -e n e s i g n a l s i n p l a n t d e f e n s e [J ].P l a n t P h y s i o l o g y,2008,147(3):1347-1357.[36] L I U J G ,WA N G Y Q ,Z H A O G Y ,e t a l .A n o v e l G o s s y pi u m b a r b a d e n s e E R F t r a n s c r i p t i o n f a c t o r ,G b E R F b ,r e g u l a t i o n h o s t r e s p o n s e a n d r e s i s t a n c e t o V e r t i c i l l i u m d a h l i a e i n t o b a c c o [J ].P h y s i o l o g y a n d M o l e c u l a r B i o l o g y of P l a n t s ,2017,23(1):125-134.I d d e n t i f i c a t i o n a n d E x p r e s s i o n A n a l y s i s o f E R F B 3S u bg r o u pT r a n s c r i p t i o n F a c t o r s i n S w e e t O r a n ge (C i t r u s s i n e n s i s )L I L i n g 1,2,L I U X i n j u n 1,2,Z H A N G B a o l i a n 1,Z H A N G S i y u 1,O U Y A N G Z h i g a n g1,2(1.S c h o o l o f L i f e S c i e n c e ,G a n n a n N o r m a l U n i v e r s i t y;2.N a t i o n a l N a v e l O r a n g e E n g i n e e r i n g a n d T e c h n o l o g y Re s e a r c h C e n t e r ,G a n z h o u 341000,C h i n a )A b s t r a c t :A s t h e s i g n if i c a n t s u b f a m i l y o f t h e A P 2/E R F t r a n s c r i p t i o n f a c t o r f a m i l y ,E R F t r a n s c r i pt i o n f a c t o r s a r e d i v i d e d i n t o s e v e n s u b g r o u p s ,i n c l u d i n g B 3(I X ).E R F B 3t r a n s c r i p t i o n f a c t o r s a r e s u s p e c t e d o f p l a y i n g an e s s e n t i a l r o l e i n p l a n t d i s -e a s e r e s i s t a n c e .I n t h i s s t u d y ,w e i d e n t i f i e d 21E R F B 3s u b g r o u p t r a n s c r i pt i o n f a c t o r s i n C .s i n e n s i s .9o u t o f 21E R F B 3s u b -g r o u p t r a n s c r i p t i o n f a c t o r m e m b e r s s i g n i f i c a n t l y e x p r e s s e d i n C .d a o x i a n e n s i s ,C .i c h a n ge n s i s a n d C .s i n e n s i s b y a n a l y z i n g t h e t r a n s c r i p t o m e d a t a of c i t r u s i n f e c t e d w i t h h u a ng l o n g b i n g .A d d i t i o n a l l y ,th e e x p r e s si o n s o f 6C s E R F B 3s u b g r o u p t r a n s c r i pt i o n f a c t o r s w e r e i n d u c e d o r d o w n -r e g u l a t e d b y a p p l y i n g e x o g e n o u s h o r m o n e s .F u r t h e r m o r e ,t h e E R F B 3t r a n s c r i pt i o n f a c t o r s f u s e d w i t h g r e e n f l u o r e s c e n t p r o t e i n w e r e e x p r e s s e d e x p l i c i t l y i n t h e n u c l e u s o f N i c o t i a n a b e n t h a m i a n a c e l l s .T h e t r a n s c r i pt i o n a l a c -t i v a t i o n a c t i v i t y o f E R F B 3t r a n s c r i p t i o n f a c t o r s h a s b e e n a p p r o v e d b y u s i n g t h e y e a s t t w o -h y b r i d a s s a y .T h e r e f o r e ,t h i s s t u d y w i l l p r o v i d e a t h e o r e t i c a l b a s i s f o r s t u d y i n g t h e f u n c t i o n o f E R F B 3t r a n s c r i pt i o n f a c t o r s i n p l a n t d i s e a s e r e s i s t a n c e .K e y wo r d s :C i t r u s s i n e n s i s ;E R F t r a n s c r i p t i o n f a c t o r s ;H u a n g l o n g b i n g ;e x p r e s s i o n a n a l y s i s 38第3期 李 玲,刘新军,张保莲,等 甜橙E R F B 3亚组转录因子的鉴定和表达分析48赣南师范大学学报2022年附表1 P C R所用特异性引物引物名称序列扩增长度(b p) q R T-P C RC s E R F B3-4-1F A T G G A G G A T T G C G A A G C T T T A C A C173C s E R F B3-4-1R C C T C G A T A A T T T A C A C G C A C G T G A AC s E R F B3-5-1F A T G A A C A G A G A A A A T A T G T C C G A G T C A G A151C s E R F B3-5-1R C G A A A C A G G G G A A T T T A A T G T T T C A T T A TC s E R F B3-6-1F A T G T A T T C G G A A A G T A T T T C T C A A A C G G A154C s E R F B3-6-1R G T G C T G G T G C T G T T G C A A T A T T G AC s E R F B3-9-1F A T G T T T C C G G C C T C C C A A A T T149C s E R F B3-9-1R G T T C A A G C A T G C T T G A T G T G T G A A CC s E R F B3-10-1F A T G T A T G G A C A G A G T A G G T G T G G T G143C s E R F B3-10-1R C G T T G G A C T T G G A A C T C G C T G A TC s E R F B3-13-1F A T G G A A T C A T C T T G T C T T C A A A T C T C G G C130C s E R F B3-13-1R A G T C G T T G A T G T T A A A A G G C A G C G A AC s E R F B3-19-1F A T G G C A G C T G G T C A T C A T C A T C A C122C s E R F B3-19-1R T G C G G G A G G T T G G A G A T G A A GC s E R F B3-20-1F A T G C C A A G G T T C C A A G A A G C C T160C s E R F B3-20-1R C A C C A A A C C A A T T T C C A T T T A C A T T T C A T CC s E R F B3-21-1F C T C A T C T T C A C C A T C A T C A A T A T C A T G G A180C s E R F B3-21-1R C G T C A A C T C A G G C T G G T C TT r a n s c r i p t i o n a c t i v a t i o nC s E R F B3-9-2F C G G A A T T C A T G T T T C C G G C C T C C C A A A T T(E c o R I)501C s E R F B3-9-2R C G G G A T C C C T C C G A T T C C G A T C T C T G A G T C G(B a m H I)C s E R F B3-10-2F C G G A A T T C A T G T A T G G A C A G A G T A G G T G T G G T G(E c o R I)813C s E R F B3-10-2R C G G G A T C C G C T T A C C A A T G G C T G G T C A C C A(B a m H I)C s E R F B3-13-2F C G G A A T T C A T G G A A T C A T C T T G T C T T C A A A T C T C G G(E c o R I)822C s E R F B3-13-2R C G G G A T C C C C A A A T A G T A G C A G T A G C A G C T C C A(B a m H I)C s E R F B3-20-2F C G G A A T T C A T G C C A A G G T T C C A A G A A G C C T(E c o R I)1026C s E R F B3-20-2R C G G G A T C C G C A A A C A C G C A G C G G G C T(B a m H I)C s E R F B3-21-2F C G G A A T T C A T G A T T T C T C A T C T T C A C C A T C A T C A A T A(E c o R I)621C s E R F B3-21-2R C G G G A T C C A T T T A C C A A T A A G T G C T C G C C A G C(B a m H I)S u b c e l l u a r l o c a l i z a t i o nC s E R F B3-9-3F G C T C T A G A A T G T T T C C G G C C T C C C A A A T T(X b a I)501C s E R F B3-9-3R C G G G A T C C C T C C G A T T C C G A T C T C T G A G T C G(B a m H I)C s E R F B3-10-3F G C T C T A G A G C T T A C C A A T G G C T G G T C A C C A(X b a I)813C s E R F B3-10-3R C G G G A T C C A T G G A A T C A T C T T G T C T T C A A A T C T C G G(B a m H I)C s E R F B3-13-3F G G G G T A C C A T G G A A T C A T C T T G T C T T C A A A T C T C G G C(K p n I)822C s E R F B3-13-3R C G G G A T C C C C A A A T A G T A G C A G T A G C A G C T C C A(B a m H I)C s E R F B3-20-3F G C T C T A G A A T G C C A A G G T T C C A A G A A G C C T(X b a I)1026C s E R F B3-20-3R C G G G A T C C G C A A A C A C G C A G C G G G C T(B a m H I)C s E R F B3-21-3F G C T C T A G A A T G A T T T C T C A T C T T C A C C A T C A T C A A T A T(X b a I)621C s E R F B3-21-3R C G G G A T C C A T T T A C C A A T A A G T G C T C G C C A G C(B a m H I)。
ERF转录因子

一、乙烯信号转导通路乙烯是一种非常重要的植物激素。
乙烯在植物生长发育和适应生物和非生物胁迫反应中起到了非常重要的作用。
种子萌发、开花、叶片衰老、果实成熟、根瘤、细胞程序性死亡以及对非生物胁迫和病原体入侵的反应等生理过程都与乙烯密切相关。
乙烯信号转导通路的最上游是位于内质网膜上的5个乙烯受体,分别被称为:ETR1、ETR2、ERS1、ERS2和EIN4。
位于乙烯受体下游的是一个负调节因子,蛋白激酶CTR1。
CTR1蛋白激酶通过与乙烯受体相结合定位在内质网上。
在没有乙烯存在的条件下,CTR1和受体的结合会协同抑制下游乙烯信号途径。
在CTR1负调控因子下游是一个正调控因子EIN2。
EIN2基因发生功能缺失突变会产生乙烯不敏感表型,显示出EIN2在乙烯信号通路中起到了核心作用。
EIN2的半衰期很短,两个F-Box蛋白ETP1和ETP2负责调控EIN2的泛素化降解。
位于EIN2下游的是正调控的转录因子家族EIN3及5个EILs。
研究发现,他们同样是受泛素化途径降解的,负责调控EIN3及EILs泛素化降解的F-Box蛋白是EBF1和EBF2。
EBF5是一种外切核酸酶它能够通过促进EBF1和EBF2的mRNA的降解来拮抗这两个蛋白对EIN3的负反馈调控。
EIN3和EIL1通过启动乙烯信号转导途径示意图转录级联反应来激活下游乙烯响应基因的表达。
二、乙烯响应因子(ethylene response factor、ERF)的结构特点及生物信息学分析ERF基因家族是一个很大的转录因子家族,属于AP2/ERF转录因子超家族。
Ohme-Takagi和Shinshi研究发现,GCC box是植物乙烯响应的DNA序列元件;同时他们在烟草(Nicotiana tabacuum)中发现了能特异性结合GCC box元件的数个乙烯响应元件结合蛋白(EREBPs),并发现,EREBPs同GCC元件相结合的结构域是59个保守的氨基酸残基。
普通烟草ERF转录因子亚家族成员鉴定及表达模式分析

2017-02,38(1)中国烟草科学 Chinese Tobacco Science 15 普通烟草ERF转录因子亚家族成员鉴定及表达模式分析任昂彦1,2,孔英珍1*(1.中国农业科学院烟草研究所,青岛266101;2.中国农业科学院研究生院,北京100081)摘要:乙烯结合响应因子(Ethylene-responsive element binding factors, ERF)家族广泛参与植物的生长发育、逆境调节及激素信号应答等过程。
利用生物信息方法,鉴定普通烟草基因组ERF转录因子亚家族成员,并对该亚家族成员的染色体定位、理化性质、系统发生、保守结构域、亚细胞定位和组织表达模式进行分析。
结果显示,目前在普通烟草中得到121个ERF基因,在烟草24条染色体分布位置具有不均匀性。
通过构建进化树,根据拟南芥ERF亚家族的分类方式将烟草ERF 亚家族分为6组,同一分组成员的理化性质及保守域呈现高度一致性。
亚细胞定位分析结果显示,绝大部分成员定位在细胞核,少数定位在线粒体和叶绿体上。
组织表达模式分析结果显示,烟草ERF基因在幼根、成熟根、离体叶和茎中表达量较高,不同分组成员之间表达存在差异。
关键词:普通烟草;ERF基因家族;生物信息;表达模式;基因家族中图分类号:S572.03 文章编号:1007-5119(2017)01-0015-08 DOI:10.13496/j.issn.1007-5119.2017.01.003Genome-wide Identification and Expression Pattern Analysis of the ERFGene Subfamily in Nicotiana tabacumREN Angyan1,2, KONG Yingzhen1*(1. Tobacco Research Institute, Chinese Academy of Agricultural Sciences, Qingdao 266101, China; 2. Graduate School of ChineseAcademy of Agricultural Sciences, Beijing 100081, China)Abstract: Ethylene-responsive element binding factors (ERF transcription factors) family is widely involved in plant development, stress resistance and hormone response. Using bioinformatics methods, we have identified 121 ERF transcription factors that are located on 24 chromosomes irregularly in Nicotiana tabacum. This was followed by analyzing their chromosomal location, protein properties, phylogenetic relation, conserved domain structure, subcellular localization as well as tissue expression profile. Based on phylogenetic analysis these 121 members of the ERF family were classified into six groups, according to the classification of Arabidopsis thaliana. Genes from the same group have similar properties and the conserved domains show a high degree of conservation. The results of subcellular localization analysis indicate that most of the genes are located in the nucleus, while a small number of them are localized on the mitochondria and chloroplasts. Some genes of ERF transcript factors have high level expression in young roots, mature roots, leaves and stems. Also, genes from different groups have different expression profiles.Keywords:nicotiana tabacum; ERF transcript factors; bioinformatics; expression pattern; gene family乙烯结合响应因子(Ethylene-responsive element binding factors, ERF)也可称为EREBP (Ethylene-responsive element binding protein)是AP2/ERF转录因子超家族中最大的转录因子亚家族分支,是植物所特有的参与乙烯响应途径调节的转录因子家族,主要参与植物的逆境调节,比如干旱、低温、虫害等。
ERF转录因子在农作物中的研究进展

农业资讯NONGYEZIXUN农业信息ERF转录因子在农作物中的研究进展林珊珊(福建省种子管理总站,福建福州 350000)摘 要 ERF转录因子是调控乙烯信号转导途径的转录因子,参与植物对生物和非生物胁迫的响应机制,在植物的生长发育过程中起到不可或缺的作用。
因此,阐述ERF转录因子的结构特征以及在抗逆性方面上的研究进展,能够为培育农作物抗逆新品种提供重要的参考依据,对改进农作物抗逆性具有指导意义。
关键词 ERF转录因子;转基因;抗逆性植物的萌芽、生长发育及其繁殖极易受到外界环境的影响,因此,植物通过在生理和生化水平上进化出各种响应环境机制来适应环境。
ERF蛋白最初是在与逆境响应基因的启动区相结合的转录因子中鉴定出来的,诸多研究表明ERF基因能够响应生物胁迫及非生物胁迫。
ERF转录因子是参与乙烯信号转导途径的乙烯响应因子,可通过对乙烯响应基因进行正负调控影响乙烯水平,从而应对外界环境的刺激。
此外,与胁迫相关的激素如茉莉酸和脱落酸等也能够诱导ERF基因表达[1]。
因此接下来介绍ERF转录因子在抗逆基因工程方面的研究进展,为农作物抗逆基因的筛选范围提供了一个大方向。
1 ERF转录因子的结构特征ERF转录因子是AP2/ERF转录因子超家族中最主要的家族成员,仅包含一个非常保守的AP2/ERF结构域,普遍分布在所有农作物中[2],目前已经在水稻、小麦、番茄等农作物中得到功能上的验证。
ERF转录因子根据其激活或阻遏特定靶基因的转录功能分成激活因子和抑制因子两大类。
大多ERF抑制因子都含有EAR结构域,这些包含EAR结构域的转录因子都具有转录抑制活性。
但是去除EAR结构域的ERF转录因子则表现出转录激活活性,例如ERF3基因去除EAR结构域编码区后在番茄的过量表达能够激活抗病基因表达,从而提高植物的抗病性[3]。
然而植物转录因子的激活域并没有特定的保守序列,往往是集中在富含酸性氨基酸的区域。
2 ERF转录因子对生物抗性的作用诸多研究表明,ERF转录因子可通过对农作物中的抗病基因表达的正负调控来提高作物对病菌和病毒的抗性[3-4]。
ERF转录因子亚家族基因在甜瓜果实发育中的功能

ERF转录因子亚家族基因在甜瓜果实发育中的功能甜瓜(Cucumis melo L.)是全世界广泛栽培的一种重要的经济类瓜果作物,贮藏性是甜瓜经济价值最重要的影响因素之一。
乙烯在甜瓜果实成熟过程中发挥重要作用,而ERF又是乙烯信号转导途径中重要的转录因子,然而ERF在甜瓜果实发育过程中的功能仍然未知。
本研究利用生物信息学方法分析了甜瓜ERF转录因子亚家族64个成员蛋白序列特性及其基因的启动子元件。
以甜瓜品种河套蜜瓜为试材,使用实时定量PCR的方法构建了该亚家族成员在不同组织和不同发育时期果实中的表达谱,获得了 8个在果实发育中高表达的候选基因。
利用瞬时表达的方法分析了候选基因的功能,进一步用候选基因的过表达、RNAi和基因编辑载体进行稳定遗传转化甜瓜,分析其在果实发育过程中的功能。
主要结果如下:1.生物信息学分析发现甜瓜64个ERF蛋白的分子量介于14.00 kDa~46.62 kDa;40个蛋白的理论等电点小于7;预测所有蛋白均为亲水性蛋白;除CmERFIV-5,其他蛋白都是不稳定蛋白;58个蛋白定位于细胞核内、1个蛋白位于细胞质、5个蛋白定位于叶绿体;28个蛋白含有信号肽;64个基因获得83种启动子元件,其中包含赤霉素、茉莉酸、生长素、水杨酸、脱落酸和乙烯的不同应答元件,还包括厌氧、低温、热、干旱、创伤和真菌胁迫的不同应答元件;进化分析发现ERF在不同物种间相对保守;64个基因在12条染色上呈不均匀分布。
2.实时定量PCR检测64个CmERFs在根、茎、叶,9 d、18 d、27 d、36 d、呼吸跃变期和跃变后期果实中的表达量,发现8个基因在呼吸跃变期高表达,且在跃变期和跃变后期的表达量存在显著差异,其中(CmERFⅠ-15、CmERⅡ-9、CmERF Ⅳ-2、CmERFⅣ-3在呼吸跃变后期的表达量显著高于根、茎、叶中的表达量。
3.利用瞬时表达分析8个基因在果实发育中的功能,发现过表达CmERFⅠ-15、CmERF Ⅱ-9、CmERFⅢ-1、CmERFⅣ-2、CmERFⅣ-3、CmERFⅤ-4促进成熟,RNAi抑制其表达则延迟成熟;过表达CmERFI-18和CmERFⅢ-13延迟成熟,RNAi抑制其表达则促进成熟。
水稻erf 结构域

水稻erf 结构域
水稻中的ERF(Ethylene Responsive Factor)结构域是一个重要的转录因子结构域,通常与植物的乙烯响应有关。
乙烯是一种植物激素,对植物的生长发育、逆境响应等多个方面发挥重要作用。
ERF结构域是一个AP2/ERF超家族的一部分,包含一个或多个AP2/ERF域,这些域通常由约60个氨基酸组成,包含一个由18个氨基酸组成的β-折叠和一个由α-螺旋组成的C端。
在水稻中,ERF结构域通常由多个基因编码,这些基因被划分为不同的亚族。
这些基因可以通过与乙烯响应元件(ERE)结合来调控植物对乙烯的响应。
ERF转录因子可以通过与ERE结合来激活或抑制乙烯响应基因的表达,从而调控植物的生长发育、逆境响应等方面。
除了乙烯响应外,ERF结构域还可能参与其他生物过程的调控,例如植物对病原菌的响应、对干旱和盐碱等逆境的适应等。
因此,对水稻中ERF结构域的研究不仅有助于深入了解植物对乙烯响应的分子机制,还有助于揭示植物适应环境的复杂机制。
erf基因簇

erf基因簇
ERF(ethylene response factor)基因簇是一类植物转录因子家族,属于AP2/ERF超家族。
ERF基因簇在植物的生长发育、逆境胁迫应答以及植物激素信号传导中起重要作用。
ERF基因簇中的转录因子含有一个或多个AP2/ERF结构域,这个结构域可以与DNA结合并调控靶基因的转录。
ERF基因簇成员的特点是在N端含有一个AP2/ERF结构域,而在C端则含有一个独特的B3结构域。
ERF基因簇在植物的逆境胁迫应答中具有重要的功能。
例如,在干旱、高盐和低温等逆境条件下,ERF基因簇转录因子可以调控与逆境应答相关的基因的表达,从而帮助植物增强逆境耐受性。
此外,ERF基因簇还参与植物的生长发育调控以及植物激素信号传导调节。
ERF基因簇是植物中重要的转录因子家族,对植物的生长发育和逆境胁迫应答具有重要作用。
小麦ERF亚族转录因子参与逆境胁迫的研究进展

小麦ERF亚族转录因子参与逆境胁迫的研究进展
崔德周;王丽丽;陈祥龙;李永波;黄琛;隋新霞;楚秀生;樊庆琦
【期刊名称】《山东农业科学》
【年(卷),期】2024(56)2
【摘要】小麦是中国三大粮食作物之一,其生长发育过程中会受到多种逆境胁迫的影响。
AP2/EREBP是植物特有的一个庞大的转录因子超家族,普遍参与生长发育和逆境胁迫应答等生物学进程。
ERF类转录因子是AP2/EREBP转录因子超家族的一个亚族。
本研究结合国内外相关研究进展,简要综述了小麦ERF亚族转录因子的结构特征与分布,重点阐述近年来小麦ERF亚族转录因子响应高盐、干旱、低温、重金属、病原菌侵染等逆境胁迫的功能和机制研究进展,最后展望了ERF亚族转录因子的研究方向和应用前景。
【总页数】5页(P176-180)
【作者】崔德周;王丽丽;陈祥龙;李永波;黄琛;隋新霞;楚秀生;樊庆琦
【作者单位】山东省农业科学院作物研究所/小麦玉米国家工程研究中心/农业农村部黄淮北部小麦生物学与遗传育种重点实验室/山东省小麦技术创新中心/济南市小麦遗传改良重点实验室;山东省林草种质资源中心;山东鲁研农业良种有限公司【正文语种】中文
【中图分类】S512.1
【相关文献】
1.ERF转录因子在植物抗逆境胁迫的研究进展
2.一个小麦AP2/ERF转录因子家族单独亚族基因的克隆及分析
3.棉花中ERF亚族转录因子抗逆育种潜力的研究进展
4.番茄2个ERF-B1亚族转录因子基因的克隆及其对生物和非生物胁迫响应
5.水稻AP2/ERF转录因子参与逆境胁迫应答的分子机制研究进展
因版权原因,仅展示原文概要,查看原文内容请购买。
根据 ERF 转录因子的结构特征可以将其分为四个亚类

根据ERF 转录因子的结构特征可以将其分为四个亚类。
第I 亚类:AP2/ERF结合域由59 个保守的氨基酸组成,位于蛋白的中部,转录调控域位于蛋白N 端;第II 亚类:AP2/ERF 结合域由58 个保守的氨基酸组成,位于蛋白的N 端,蛋白的C 末端存在一个EAR 基序;第III 亚类:AP2/ERF 结合域也由58 个氨基酸组成,其N 和C 两端各有一个酸性激活域,蛋白C 末端还有一个MAPK 作用位点。
第IV 类:AP2/ERF 结合域同样由58 个保守的氨基酸组成,位于蛋白的N 端,转录调控域位于C 端,此外N 末端还具有一个保守的MCGGAI(I/L)元件[68]ERFs类转录因子可以受到多种胁迫的诱导表达,同时参与不同胁迫信号转导途径的交叉应答,所以它在非生物胁迫中有十分重要的调控作用番前的?STE/JF/基因可以提高番莉根系发育过程中的耐盐性,还可以在转基因植株中表现较高的相对含水量和较低的丙二酸含量及电解质外渗,同时可以积累游离脯氨酸和可溶性糖含量,此外还调节抗逆相关基因、P5CS、DREB3】、的激活表达(Lu et al.,2011)。
Gao等(Gao et al.,2008)将番燕基因转入水稻,经过干旱和高盐处理后,同对照相比转基因植株的抗旱以及耐盐能力增强,并积累了脯氨酸含量,降低了叶片的水分流失。
另外,基因不仅可以影响含有GCC-box或DRE/CRT顺式元件的Z//>5、Wcor413L OsPrx和等胁迫相关的功能基因表达,还影响Os-CDP欠7、OsCDPKlS和OsCDP欠/9等调节基因的表达。
ERF类基因WXPl和WXP2可以使叶蜡积累并提高拟南芥的抗旱性(Zhangetal.,2007)。
己有许多研宄表明胁迫相关基因可以受外源的诱导表达,也有-?些基因虽然ABA对其表达无影响,但是确可以受到干早和低温等的诱导,这就又涉及到ABA依赖和ABA非依赖的非生物胁迫的信号转导途径,Zhu等(Zhu ct al.,2010)通过EMSA试验证明RAP2.6可以与CE丨顺式元件结合,进而可以调控AtABI4等ABA相关基因的特异表达。
《ERF转录因子亚家族基因在甜瓜果实发育中的功能》范文

《ERF转录因子亚家族基因在甜瓜果实发育中的功能》篇一摘要:本文通过对ERF(乙烯响应因子)转录因子亚家族基因在甜瓜果实发育过程中的表达与功能进行深入研究,探讨了其在果实发育、成熟及品质形成过程中的作用机制。
本研究不仅为甜瓜分子育种提供了新的理论依据,也为果实发育的分子调控机制提供了新的认识。
一、引言甜瓜作为重要的经济作物,其果实发育过程中的品质形成与调控机制一直是研究的热点。
近年来,随着分子生物学技术的发展,转录因子在植物生长发育中的重要作用逐渐被揭示。
其中,ERF转录因子亚家族作为植物特有的转录因子,在果实发育、成熟及抗逆过程中发挥着重要作用。
因此,研究ERF转录因子亚家族基因在甜瓜果实发育中的功能具有重要意义。
二、材料与方法1. 材料选择选择不同发育阶段的甜瓜果实作为实验材料,包括幼果期、膨大期、白熟期、成熟期等。
2. 实验方法(1)利用生物信息学方法,分析ERF转录因子亚家族基因在甜瓜基因组中的分布及结构特点;(2)通过实时荧光定量PCR技术,检测ERF基因在不同发育阶段甜瓜果实中的表达模式;(3)利用基因克隆技术,获得ERF基因的过表达及沉默载体;(4)通过遗传转化技术,获得转基因甜瓜植株,并分析其果实发育过程中的表型变化。
三、结果与分析1. ERF转录因子亚家族基因的分布与结构特点通过生物信息学分析,发现甜瓜基因组中存在多个ERF转录因子亚家族基因,它们在染色体上的分布具有一定的规律性,且具有典型的ERF结构域。
2. ERF基因在甜瓜果实发育中的表达模式实时荧光定量PCR结果显示,ERF基因在甜瓜果实发育的不同阶段表达量存在显著差异。
在果实成熟期,ERF基因的表达量达到高峰,表明其在果实成熟过程中发挥重要作用。
3. 过表达及沉默ERF基因对甜瓜果实发育的影响过表达ERF基因的转基因甜瓜植株,其果实发育速度加快,成熟期提前,果实体积增大,品质提高;而沉默ERF基因的转基因甜瓜植株则表现出相反的表型,果实发育受阻,成熟期延迟,品质下降。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Arabidopsis thaliana ERF FamilylERF Family Introduction lDownload Sequences lMultiple Sequences Alignment lPhylogenetic TreePlant Transcription Factor Databasev3.0Center for Bioinformatics , Peking University , ChinaPrevious versions:v1.0v2.0Home | Blast | Search | Download | Prediction | Help | About |LinksLFY)Species TF IDDescription AT1G01250.1Integrase-type DNA-binding superfamily proteinAT1G03800.1ERF domain protein 10AT1G04370.1Ethylene-responsive element binding factor 14AT1G06160.1octadecanoid-responsive Arabidopsis AP2/ERF 59AT1G12610.1Integrase-type DNA-binding superfamily protein AT1G12630.1Integrase-type DNA-binding superfamily protein AT1G12890.1Integrase-type DNA-binding superfamily protein AT1G12980.1Integrase-type DNA-binding superfamily protein AT1G15360.1Integrase-type DNA-binding superfamily protein AT1G19210.1Integrase-type DNA-binding superfamily protein AT1G21910.1Integrase-type DNA-binding superfamily protein AT1G22190.1Integrase-type DNA-binding superfamily protein AT1G22810.1Integrase-type DNA-binding superfamily protein AT1G22985.1Integrase-type DNA-binding superfamily protein AT1G24590.1DORNROSCHEN-like AT1G25470.1AP2 domain-containing transcription factor family protein AT1G25470.2AP2 domain-containing transcription factor family protein AT1G28160.1Integrase-type DNA-binding superfamily protein AT1G28360.1ERF domain protein 12AT1G28370.1ERF domain protein 11AT1G33760.1Integrase-type DNA-binding superfamily protein AT1G36060.1Integrase-type DNA-binding superfamily protein AT1G43160.1related to AP2 6AT1G44830.1Integrase-type DNA-binding superfamily protein AT1G46768.1related to AP2 1AT1G49120.1Integrase-type DNA-binding superfamily protein AT1G50640.1ethylene responsive element binding factor 3AT1G53170.1ethylene response factor 8AT1G53910.1related to AP2 12AT1G53910.2related to AP2 12AT1G53910.3related to AP2 12AT1G63030.1Integrase-type DNA-binding superfamily protein AT1G63030.2Integrase-type DNA-binding superfamily protein AT1G64380.1Integrase-type DNA-binding superfamily protein AT1G68550.1Integrase-type DNA-binding superfamily protein AT1G68550.2Integrase-type DNA-binding superfamily protein AT1G71130.1Integrase-type DNA-binding superfamily protein AT1G71450.1Integrase-type DNA-binding superfamily protein AT1G71520.1Integrase-type DNA-binding superfamily protein AT1G72360.1Integrase-type DNA-binding superfamily protein AT1G72360.2Integrase-type DNA-binding superfamily protein AT1G72360.3Integrase-type DNA-binding superfamily protein AT1G74930.1Integrase-type DNA-binding superfamily protein AT1G75490.1Integrase-type DNA-binding superfamily protein AT1G77200.1Integrase-type DNA-binding superfamily proteinAT1G78080.1related to AP2 4AT1G80580.1Integrase-type DNA-binding superfamily proteinAT2G20350.1Integrase-type DNA-binding superfamily proteinAT2G20880.1Integrase-type DNA-binding superfamily proteinAT2G22200.1Integrase-type DNA-binding superfamily proteinAT2G23340.1DREB and EAR motif protein 3AT2G25820.1Integrase-type DNA-binding superfamily proteinAT2G31230.1ethylene-responsive element binding factor 15AT2G33710.1Integrase-type DNA-binding superfamily proteinAT2G33710.2Integrase-type DNA-binding superfamily proteinAT2G35700.1ERF family protein 38AT2G36450.1Integrase-type DNA-binding superfamily proteinAT2G38340.1Integrase-type DNA-binding superfamily proteinAT2G40220.1Integrase-type DNA-binding superfamily proteinAT2G40340.1Integrase-type DNA-binding superfamily proteinAT2G40350.1Integrase-type DNA-binding superfamily proteinAT2G44840.1ethylene-responsive element binding factor 13AT2G44940.1Integrase-type DNA-binding superfamily proteinAT2G46310.1cytokinin response factor 5AT2G47520.1Integrase-type DNA-binding superfamily proteinAT3G11020.1DRE/CRT-binding protein 2BAT3G14230.1related to AP2 2AT3G14230.2related to AP2 2AT3G14230.3related to AP2 2AT3G15210.1ethylene responsive element binding factor 4AT3G16280.1Integrase-type DNA-binding superfamily proteinAT3G16770.1ethylene-responsive element binding proteinAT3G20310.1ethylene response factor 7AT3G23220.1Integrase-type DNA-binding superfamily proteinAT3G23230.1Integrase-type DNA-binding superfamily proteinAT3G23240.1ethylene response factor 1AT3G25890.1Integrase-type DNA-binding superfamily proteinAT3G25890.2Integrase-type DNA-binding superfamily proteinAT3G50260.1cooperatively regulated by ethylene and jasmonate 1AT3G54320.2Integrase-type DNA-binding superfamily proteinAT3G57600.1Integrase-type DNA-binding superfamily proteinAT3G60490.1Integrase-type DNA-binding superfamily proteinAT3G61630.1cytokinin response factor 6AT4G06746.1related to AP2 9AT4G11140.1cytokinin response factor 1AT4G13040.1Integrase-type DNA-binding superfamily proteinAT4G13040.2Integrase-type DNA-binding superfamily proteinAT4G13040.3Integrase-type DNA-binding superfamily proteinAT4G13620.1Integrase-type DNA-binding superfamily proteinAT4G16750.1Integrase-type DNA-binding superfamily proteinAT4G17490.1ethylene responsive element binding factor 6AT4G17500.1ethylene responsive element binding factor 1AT4G18450.1Integrase-type DNA-binding superfamily proteinAT4G23750.1cytokinin response factor 2AT4G23750.2cytokinin response factor 2AT4G25470.1C-repeat/DRE binding factor 2AT4G25480.1dehydration response element B1AAT4G25490.1C-repeat/DRE binding factor 1AT4G27950.1cytokinin response factor 4AT4G28140.1Integrase-type DNA-binding superfamily proteinAT4G31060.1Integrase-type DNA-binding superfamily proteinAT4G32800.1Integrase-type DNA-binding superfamily proteinArabidopsis thaliana(139)AT4G34410.1redox responsive transcription factor 1AT4G36900.1related to AP2 10AT4G39780.1Integrase-type DNA-binding superfamily proteinAT5G05410.1DRE-binding protein 2AAT5G05410.2DRE-binding protein 2AAT5G07310.1Integrase-type DNA-binding superfamily proteinAT5G07580.1Integrase-type DNA-binding superfamily proteinAT5G11190.1Integrase-type DNA-binding superfamily proteinAT5G11590.1Integrase-type DNA-binding superfamily proteinAT5G13330.1related to AP2 6lAT5G13910.1Integrase-type DNA-binding superfamily proteinAT5G18450.1Integrase-type DNA-binding superfamily proteinAT5G18560.1Integrase-type DNA-binding superfamily proteinAT5G19790.1related to AP2 11AT5G21960.1Integrase-type DNA-binding superfamily proteinAT5G25190.1Integrase-type DNA-binding superfamily proteinAT5G25390.1Integrase-type DNA-binding superfamily proteinAT5G25390.2Integrase-type DNA-binding superfamily proteinAT5G25810.1Integrase-type DNA-binding superfamily proteinAT5G43410.1Integrase-type DNA-binding superfamily proteinAT5G44210.1erf domain protein 9AT5G47220.1ethylene responsive element binding factor 2AT5G47230.1ethylene responsive element binding factor 5AT5G50080.1ethylene response factor 110AT5G51190.1Integrase-type DNA-binding superfamily proteinAT5G51990.1C-repeat-binding factor 4AT5G52020.1Integrase-type DNA-binding superfamily proteinAT5G53290.1cytokinin response factor 3AT5G61590.1Integrase-type DNA-binding superfamily proteinAT5G61600.1ethylene response factor 104AT5G61890.1Integrase-type DNA-binding superfamily proteinAT5G64750.1Integrase-type DNA-binding superfamily proteinAT5G65130.1Integrase-type DNA-binding superfamily proteinAT5G67000.1Integrase-type DNA-binding superfamily proteinAT5G67010.1Integrase-type DNA-binding superfamily proteinAT5G67190.1DREB and EAR motif protein 2ERF Family IntroductionThe AP2/ERF superfamily is defined by the AP2/ERF domain, which consists of about 60 to 70 amino acids and is involved in DNA binding. These three families have been defined as follows. The AP2 family proteins contain two repeated AP2/ERF domains, the ERF family proteins contain a single AP2/ERF domain, and the RAV family proteins contain a B3 domain, which is a DNA-binding domain conserved in other plant-specific transcription factors, in addition to the single AP2/ERF domain.It has been demonstrated that the AP2/ERF proteins have important functions in the transcriptional regulation of a variety of biological processes related to growth and development, as well as various responses to environmental stimuli.After finding the tobacco ERFs, many proteins in the ERF family were identified and implicated in many diverse functions in cellular processes, such as hormonal signal transduction, response to biotic and abiotic stresses, regulation of metabolism, and in developmental processes in various plant species.Toshitsugu Nakano, Kaoru Suzuki, Tatsuhito Fujimura, and Hideaki Shinshi.Genome-wide analysis of the ERF gene family in Arabidopsis and rice.Plant Physiol, 2006. 140(2): p. 411-32.PMID: 16407444The first class are proteins which bind to ethylene response elements (ERE) or GCC boxes (tobacco EREBPs, Arabidopsis AtEBP and AtERF1-5, and tomato Pti4-6) found in the promoters of ethylene-inducible pathogenesis related genes. The GCC box is an 11 bp sequence (TAAGAGCCGCC) with a core GCCGCC sequence that is required for binding.The second class includes proteins that bind to the C-repeat or dehydration response element (DRE) in the promoters of genes that are turned on in response to low temperatures and/or water deficit (CBF1, CBF2, CBF3/DREB1A andDREB2A). The C-repeat/DREs contain the core sequence CCGAC.Nole-Wilson S, Krizek BA.DNA binding properties of the Arabidopsis floral development protein AINTEGUMENTA.Nucleic Acids Res. 2000 Nov 1;28(21):4076-82. Erratum in: Nucleic Acids Res 2001 Mar 1;29(5):1261.PMID: 11058102©2010-2013, Center for Bioinformatics, Peking UniversitySupported ByLast Modified: 2013-8-23。
