参与茉莉酸甲酯反应的顺式作用元件
MYB转录因子在水稻抗逆基因工程中的研究进展

MYB转录因子在水稻抗逆基因工程中的研究进展董勤勇张圆圆魏景芳朱昀摘要:干旱、寒冷、高盐以及病虫害胁迫是造成水稻减产的重要因素。
近年来,植物特异性转录因子在水稻抗旱、抗寒、抗盐以及抗病虫害胁迫机制上扮演着重要角色。
MYB转录因子是植物最大的转录因子家族之一,其结构高度保守,常见1R-MYB/MYB-related、R2R3-MYB、3R-MYB以及4R-MYB4种结构类型。
MYB转录因子主要参与植物生长发育、生物以及非生物胁迫的应答过程。
本文就MYB转录因子的结构特征、分类以及在水稻(Oryzaativa)生物及非生物胁迫中的应答进行综述,为MYB转录因子的研究及植物抗逆新品种培育提供参考。
关键词:MYB转录因子;生物胁迫;非生物胁迫Keyword:MYBtrancriptionfactor;biotictre;abiotictre植物在田間会遭受干旱、寒冷、高盐等非生物胁迫以及包括害虫和病原体在内的生物胁迫。
植物自身具备应对复杂胁迫反应的机制与策略,转录因子(Trancriptionfactor)是逆境响应的主要调控因子,其编码基因是作物改良的最佳候选基因[1]。
转录因子是一类调节基因表达水平的重要调控蛋白,通过与靶标基因启动子区的顺式作用元件结合,激活或抑制靶标基因的转录表达[1]。
据报道在已发现的80个转录因子家族中,只有MYB、NAC、bZIP、锌指蛋白等少量转录因子在逆境胁迫响应中起到重要作用。
其中MYB转录因子是最大的植物转录因子家族之一,它在植物生长发育、激素信号转导以及植物对生物及非生物应答中起到十分重要的作用[2-5]。
目前从水稻中已鉴定出185个MYB转录因子[6],研究发现这些转录因子的功能不仅体现在调节植物生长发育上,在植物应对复杂的生物和非生物胁迫反应方面上也具有显著的作用。
这些MYB转录因子基因提高了水稻的综合抗逆能力,是实现水稻抗逆遗传改良的重要资源。
1 MYB转录因子的结构与分类MYB转录因子结构上具有1~4个重复单元构成的MYB结构域,每个重复单元由50~53个氨基酸构成[7]。
栀子TPS_基因家族鉴定及与萜类物质代谢的相关性分析

沈阳农业大学学报,2024,55(1):66-78Journal of Shenyang Agricultural Universityhttp ://DOI:10.3969/j.issn.1000-1700.2024.01.008收稿日期:2023-10-11基金项目:福建农林大学科技计划项目(闽林科便函[2020]29号);福建省林业科技项目(闽林科便函[2022]9号);福建省林业科技推广项目(2022TG09)第一作者:李金燃(1995-),女,硕士,助理工程师,从事药用植物栽培与利用研究,E-mail :****************通信作者:邹双全(1963-),男,博士,研究员,从事药用植物栽培与利用研究,E-mail:****************栀子TPS 基因家族鉴定及与萜类物质代谢的相关性分析李金燃1,2,张麒功1,3,陈丝雨1,3,陈淑颖1,3,陈清海4,邹双全1,3(1.福建农林大学林学院,福州350002;2.南平市延平区东坑林业站,福建南平353000;3.自然生物资源保育利用福建省高校工程技术研究中心,福州350002;4.泉州市城市森林公园发展中心,福建泉州362000)摘要:TPS 基因家族是萜类化合物合成的末端关键酶,在栀子花香的形成中起重要作用。
为了明确栀子TPS 基因家族成员的基本特征,利用生物信息学方法对栀子TPS 基因进行家族成员鉴定;通过外源激素喷施试验进行转录组测序,结合顶空固相微萃取和气相色谱-质谱联用技术,分析不同外源激素浓度下栀子花朵TPS 基因家族成员表达水平和代谢物含量变化关系。
结果表明:从栀子基因组中共鉴定获得GjTPS 家族成员41个,编码氨基酸380~849个,含有外显子5~15个不等,GjTPS 家族成员定位在叶绿体中,并且不均匀地分布在10条染色体上;共线性分析表明栀子与其同科植物中粒咖啡的TPS 基因有更近的亲缘关系。
系统发育分析结果显示GjTPS 基因分为5个亚家族,TPS-a 、TPS-b 亚家族包含了大多数GjTPS 家族成员。
茉莉酸甲酯综述Word文档

茉莉酸甲酯的研究进展摘要:茉莉酸及其衍生物茉莉酸甲酯统称为茉莉酸盐,是广泛存在于植物中的一种生长调节物质,在植物体内起着重要的作用.本文主要就茉莉酸和茉莉酸甲酯的生物合成、茉莉酸甲酯的生理生化作用、茉莉酸类物质在植物抗性方面的研究及其在植物生长代谢方面的作用进行了综述.关键词:茉莉酸类物质;茉莉酸甲酯;抗逆性茉莉酸(JA)和茉莉酸甲酯(MeJA)作为与损伤相关的植物激素和信号分子, 广泛存在于自然界,是许多植物体内产生的天然化合物[1].外源应用类化合物能够激发防御植物基因的表达,诱导植物的化学防御,产生与机械损伤和昆虫取食相似的效果。
大量研究表明,用茉莉酸类化合物处理植物可系统诱导蛋白酶抑制剂(PI)和多酚氧化酶(PPO),从而影响植食动物对营养物质的吸收,还能增加过氧化物酶、壳聚糖酶和脂氧合酶等防御蛋白的活性水平,导致生物碱和酚酸类次生物质的积累,增加并改变挥发性信号化合物的释放,甚至形成防御结构,如毛状体和树脂导管。
茉莉酸甲酯可以从植物的气孔进入植物体内,在细胞质中被酯酶水解为茉莉酸,实现长距离的信号传导和植物间的交流,诱导邻近植物产生诱导防御反应。
大量的研究表明,茉莉酸类化合物具有广谱的生理效应,它不仅调节植物的生长和发育,如萌发、衰老、果实成熟、根的生长、花粉发育和球茎的形成、卷须的缠绕等[ 1 ,2 ],而且还参与植物对机械伤害、病害、虫害等环境的胁迫做出防御响应,和经典植物激素或植物生长调节剂相似[3 ] .因此,近年来茉莉酸类化合物引起了植物学家广泛的关注。
1 茉莉酸及其茉莉酸甲酯的生物合成茉莉酸(Jasmonic acid)及其挥发性茉莉酸甲酯(MeJA)是通过硬脂酸途径(octadecanoid pathway)产生的脂肪酸衍生物,是环戊酮衍生物(cyclopentanone derivatives)之一.植物受到创伤、昆虫咬食或病源菌感染后引发的局部及系统性的伤害信号,如寡糖激发子、多肽、脱落酸及甲壳素等,与细胞膜上的受体结合进入细胞,或不经与受体结合直接穿过细胞膜进入细胞。
一种顺式-二氢茉莉酮酸甲酯的合成方法[发明专利]
![一种顺式-二氢茉莉酮酸甲酯的合成方法[发明专利]](https://img.taocdn.com/s3/m/fce278e227fff705cc1755270722192e453658a2.png)
[19]中华人民共和国国家知识产权局[12]发明专利申请公布说明书[11]公开号CN 101475480A [43]公开日2009年7月8日[21]申请号200810244580.3[22]申请日2008.12.11[21]申请号200810244580.3[71]申请人淮安万邦香料工业有限公司地址223003江苏省淮安市淮阴区长江东路216号[72]发明人林祥 陈文昌 陈兆刚 [74]专利代理机构淮安市科翔专利商标事务所代理人韩晓斌[51]Int.CI.C07C 69/716 (2006.01)C07C 67/30 (2006.01)权利要求书 3 页 说明书 8 页[54]发明名称一种顺式-二氢茉莉酮酸甲酯的合成方法[57]摘要本发明公开了一种顺式-二氢茉莉酮酸甲酯的合成方法,第一步庚酰氯生成反应,庚酸与氯化亚砜反应生成庚酰氯;第二步Fridel-Crafts酰基化反应,庚酰氯与丁二酸反应得2-戊基-1,3-环戊二酮;第三步醚化反应,2-戊基-1,3-环戊二酮与甲醇反应得3-甲氧基-2-戊基-环戊-2-烯酮;第四步加成反应,3-甲氧基-2-戊基-环戊-2-烯酮与丙二酸二甲酸酯反应得反式二氢茉莉酮酸甲酯;第五步氢化反应,反应二氢茉莉酮酸甲酯氢气置换得主含量为顺式-二氢茉莉酮酸甲酯的二氢茉莉酮酸甲酯。
本发明是以丁二酸与庚酰氯进行酰基化反应,再经醚化、加成、氢化反应合成制得顺式-二氢茉莉酮酸甲酯,合成步骤短,反应条件温和,操作简便,生产成本低,安全环保,易于工业化生产。
200810244580.3权 利 要 求 书第1/3页 1.一种顺式-二氢茉莉酮酸甲酯的合成方法,其特征在于该合成方法如下:第一频庚酰氯生成反应,庚酸与氯化亚砜反应生成庚酰氯;第二步Fridel-Crafts酰基化反应,庚酰氯与丁二酸反应得2-戊基-1,3-环戊二酮;第三步醚化反应,2-戊基-1,3-环戊二酮与甲醇反应得3-甲氧基-2-戊基-环戊-2-烯酮;第四步加成反应,3-甲氧基-2-戊基-环戊-2-烯酮与丙二酸二甲酸酯反应得反式-二氢茉莉酮酸甲酯;第五步氢化反应,反式-二氢茉莉酮酸甲酯氢气置换得主含量为顺式-二氢茉莉酮酸甲酯的二氢茉莉酮酸甲。
枳漆酶基因家族鉴定及其响应盐胁迫的表达分析

徐小勇,顾铭洲,梁梦鸽,等.枳漆酶基因家族鉴定及其响应盐胁迫的表达分析[J].江苏农业科学,2023,51(9):52-59.doi:10.15889/j.issn.1002-1302.2023.09.007枳漆酶基因家族鉴定及其响应盐胁迫的表达分析徐小勇,顾铭洲,梁梦鸽,姜丽娟(扬州大学园艺园林学院,江苏扬州225009) 摘要:漆酶(LAC)是植物木质素生物合成中催化木质素单体聚合的关键酶,在调控植物生长发育和胁迫响应中发挥着重要作用。
对枳漆酶基因家族进行鉴定和分析,并探究其在盐胁迫下的表达模式,可为进一步研究枳漆酶基因功能提供重要的参考信息。
采用生物信息学手段鉴定枳漆酶基因家族成员,对其家族成员的理化性质、基因结构、系统进化关系、启动子顺式作用元件等进行分析,并通过qRT-PCR方法分析其盐胁迫表达模式。
结果表明,枳基因组中共有20个LAC基因家族成员,其中18个分布在5条已知染色体上,2个分布在未知染色体上,被预测定位到细胞膜和细胞核中;共有6~14个外显子,5~13个内含子,9~11个motif;系统进化树分析结果显示,20个枳LAC基因家族成员与17个拟南芥LAC基因家族成员共分成7个亚组,20个枳LAC基因家族成员分布在其中的6组;20个枳LAC基因家族成员与拟南芥LAC基因间存在19对共线性关系;启动子区域含有24种顺式作用元件,其中厌氧诱导元件、干旱响应元件和茉莉酸甲酯响应元件数量最多;16个枳LAC基因在盐胁迫下显著上调表达,推测枳漆酶基因参与了盐胁迫响应。
关键词:枳;漆酶基因;进化树;顺式作用元件;盐胁迫 中图分类号:S188+.3 文献标志码:A 文章编号:1002-1302(2023)09-0052-08收稿日期:2022-09-26基金项目:国家重点研发计划(编号:2018YFD1000107);江苏省农业科技自主创新资金[编号:CX(21)3024]。
作者简介:徐小勇(1979—),男,江西奉新人,博士,副教授,主要从事果树生物技术研究。
植物激素应答元件研究进展

植物激素应答元件研究进展何访;梅文莉;郭冬;李辉亮;彭世清;戴好富【摘要】植物激素在植物生长发育的过程中发挥了重要作用,对激素应答元件的研究将有助于对植物激素作用机制的研究.对常用植物激素生长素、赤霉素、脱落酸、乙烯、水杨酸和茉莉酸的应答元件研究进行了全面的综述,可为植物激素基因表达调控方面的研究提供有益的资料.【期刊名称】《热带作物学报》【年(卷),期】2015(036)001【总页数】8页(P211-218)【关键词】植物激素;激素响应基因;启动子;应答元件【作者】何访;梅文莉;郭冬;李辉亮;彭世清;戴好富【作者单位】中国热带农业科学院热带生物技术研究所农业部热带作物生物学与遗传资源利用重点实验室海南海口571101;海南大学农学院,海南海口 570228;中国热带农业科学院热带生物技术研究所农业部热带作物生物学与遗传资源利用重点实验室海南海口571101;中国热带农业科学院热带生物技术研究所农业部热带作物生物学与遗传资源利用重点实验室海南海口571101;中国热带农业科学院热带生物技术研究所农业部热带作物生物学与遗传资源利用重点实验室海南海口571101;中国热带农业科学院热带生物技术研究所农业部热带作物生物学与遗传资源利用重点实验室海南海口571101;中国热带农业科学院热带生物技术研究所农业部热带作物生物学与遗传资源利用重点实验室海南海口571101【正文语种】中文【中图分类】Q78Abstarct Phytohormones play an important role in plant growth and development, and the study of phytohormone response cis-elements will contribute to the understanding of the mechanisms of phytohormones. The paper makes a comprehensive review on the response cis-elements of several phytohormones, such as auxin, gibberellin, abscisic acid, ethylene, jasmonic acid and salicylic acid.Key words Phytohormones; Gene respone phytohormone; Promoter; cis-element.doi 10.3969/j.issn.1000-2561.2015.01.035植物激素对植物的生长发育以及在植物应对逆境方面具有重要的调节作用。
过氧化物酶基因PagPRX19对银腺杨‘84K’耐盐性的影响

过氧化物酶基因PagPRX 19对银腺杨‘84K ’耐盐性的影响黄清晨1,2,赖建新1,2,黄李超1,2,卢孟柱1,2(1. 浙江农林大学 林业与生物技术学院,浙江 杭州 311300;2. 浙江农林大学 省部共建亚热带森林培育国家重点实验室,浙江 杭州 311300)摘要:【目的】盐害作为影响植物生长发育的非生物胁迫因子,严重威胁林木生长。
在受到盐胁迫时,植物内源活性氧(ROS)水平增加,造成氧化胁迫,影响植株正常生长发育。
因此,可通过增强过氧化物酶PRX 家族成员表达水平,改变ROS 水平,以增强杨树Populus 耐盐能力,揭示PRX 成员参与调控杨树盐胁迫响应的机制。
【方法】以银腺杨‘84K ’Populus alba × P. glandulosa ‘84K ’为材料,生物信息学分析选取PRX 家族成员PagPRX 19进行克隆并构建过表达载体,农杆菌Agrobacterium tumefaciens 介导叶盘转化法获得过表达植株。
以银腺杨‘84K ’ PagPRX 19过表达植株生长45 d 的组培苗和生长2个月的土培苗为实验材料,进行盐胁迫处理,以非转基因植株为对照。
观察植株表型,检测脯氨酸、丙二醛、电解质渗透率等生理指标并进行分析。
【结果】①克隆了PagPRX 19基因,构建过表达载体,获得转基因阳性植株。
经分子鉴定选取2个过表达株系OE#1和OE#2为实验材料做后续分析。
②与对照相比,过表达植株株高下降,地径增加。
③盐胁迫处理下,过表达植株相较于对照表现为叶片皱缩以及植株生长受到抑制程度低,组培苗的盐胁迫处理表现为相似结果。
④转基因植株的ROS 水平降低,而且在盐胁迫下过表达植株叶片和根的ROS 仍保持较对照低的水平。
盐胁迫下过表达植株较对照脯氨酸增加,叶片持水能力增强,丙二醛和电解质渗透率降低。
从生理方面显示转基因植株具有较高的耐盐能力。
【结论】过表达PagPRX 19可降低盐胁迫下杨树转基因植株的ROS 水平,缓解氧化胁迫,增强了植株耐盐性。
211017066_茶树ANR基因家族的全基因组鉴定与分析

第40卷第2期贵州大学学报(自然科学版)Vol.40No.22023年 3月JournalofGuizhouUniversity(NaturalSciences)Mar.2023文章编号 1000 5269(2023)02 0047 08DOI:10.15958/j.cnki.gdxbzrb.2023.02.08茶树ANR基因家族的全基因组鉴定与分析段丽丽1,2,李魁印 3(1.贵州大学农学院,贵州贵阳550025;2.贵州大学学报编辑部,贵州贵阳550025;3.安顺学院农学院,贵州安顺561000)摘 要:花青素还原酶(anthocyanidinreductase,ANR)是原花青素生物合成途径中的关键酶之一,同时,ANR通过调节植物不同组织中花青素的积累影响植物对光的吸收程度和叶面温度,提高茶树的防御力和对环境的适应性。
利用生物信息学分析方法,从‘舒茶早’基因组数据中鉴定出茶树ANR基因家族成员,并分析ANR家族成员的基因结构、蛋白理化性质、保守基序及顺势作用元件。
结果表明,CsANR家族有21个成员,分为7类,含有多个保守基序,编码287~627个氨基酸,其中,有15个蛋白为亲水性蛋白,6个为疏水性蛋白。
对CsANR家族基因的顺势作用元件分析表明CsANR家族含有光响应、防御和胁迫响应、植物激素响应(茉莉酸甲酯、生长素、脱落酸及赤霉素)和低温响应元件,说明该基因家族可能在参与植物生长发育及应对胁迫中发挥作用,本研究结果可为茶树ANR基因功能研究提供依据。
关键词:茶树;花青素还原酶;基因家族;鉴定;分析中图分类号:S571.1 文献标志码:A 茶树(Camelliasinensis)是山茶科、山茶属灌木或小乔木。
茶树起源于中国,是一种非常重要的叶型经济作物,且在我国已经有2000多年的种植历史[1 2]。
同时,茶树作为经济作物在世界上30多个国家广泛种植[3 4]。
茶是一种不含酒精的饮料,因其具有多种健康功效而得到广泛关注[5]。
假单胞菌亮氨酸氨肽酶基因克隆及生物信息学分析

第39卷㊀第1期2021年1月㊀㊀㊀㊀广西师范大学学报(自然科学版)Journal of Guangxi Normal University(Natural Science Edition)㊀㊀㊀㊀㊀㊀Vol.39㊀No.1Jan.2021DOI:10.16088/j.issn.1001-6600.2020083102 郭辰,周飞,韩彪,等.假单胞菌亮氨酸氨肽酶基因克隆及生物信息学分析[J].广西师范大学学报(自然科学版),2021,39(1):156-164. GUO C,ZHOU F,HAN B,et al.Cloning and bioinformatics analysis of LAP gene from Pseudomona[J].Journal of Guangxi Normal University (Natural Science Edition),2021,39(1):156-164.假单胞菌亮氨酸氨肽酶基因克隆及生物信息学分析郭㊀辰1,周㊀飞2,3,韩㊀彪1,潘㊀翠1,吴洁敏1,杨㊀婷2,3,尚常花2,3∗(1.广西壮族自治区环境保护科学研究院,广西南宁530022;2.广西师范大学生命科学学院,广西桂林541006;3.珍稀濒危动植物生态与环境保护教育部重点实验室(广西师范大学),广西桂林541006)摘㊀要:为探索假单胞菌亮氨酸氨肽酶(leucine aminopeptidase,LAP)编码基因LAP的结构和功能特征及其在生长发育中的作用,克隆假单胞菌Cr13菌株的LAP基因,并对其进行生物信息学分析㊂首先,克隆假单胞菌亮氨酸氨肽酶的一个DNA片段;其次,以此为基础,通过2次染色体步移(genome walking)对假单胞菌LAP基因全长序列进行克隆;最后,采用生物信息学软件对LAP进行分析㊂结果表明:Cr13LAP基因全长为1491bp㊂推测LAP基因定位于线粒体,具有保守的细胞质氨肽酶模体NTDAEGRL,在其启动子区域(426bp)发现了基本元件TATA-Box和CAAT-box及一系列潜在的顺式作用元件㊂LAP全长序列的克隆为将来深入研究该基因的功能奠定了基础,LAP启动子序列的克隆与分析为进一步研究假单胞菌Cr13LAP的表达调控提供了数据依据㊂关键词:假单胞菌;亮氨酸氨肽酶;染色体步移;生物信息学中图分类号:Q933㊀㊀㊀文献标志码:A㊀㊀㊀文章编号:1001-6600(2021)01-0156-09亮氨酸氨肽酶(leucine aminopeptidase,LAP)是一种肽链外切酶,催化多肽链的N端氨基酸残基的水解,在生物的新陈代谢中发挥着重要的作用㊂作为一种外肽酶,LAP可以切除多肽链或蛋白质N末端的疏水性氨基酸,降低蛋白质水解液苦味,促进蛋白质的水解[1]㊂LAP还可用于制备生物活性肽及测定分析蛋白序列[1]㊂植物LAP在响应干旱㊁高盐㊁创伤伤害系统㊁茉莉酸甲酯和脱落酸等,调控生长素转运㊁减数分裂发育及植物对昆虫食草动物的防御具有重要作用[2-4]㊂前人对不同生物的LAP基因和LAP蛋白进行了详细研究㊂Zhang等[5]从豆状带绦虫Taenia pisiformis中克隆了LAP基因TpLAP,发现它编码的蛋白是M17LAP家族的一个新成员,可以被认为是一种阶段性差异表达蛋白,这些发现为研究豆状带绦虫在最终宿主中的生长㊁发育和存活机制提供了新观点㊂Wu等[6]首次报道了寄生虫细粒棘球蚴Echinococcus granulosus的LAP蛋白,发现其属于M17肽酶家族,参与细粒棘球蚴的重要生物学功能㊂此外,重组的该寄生虫的LAP蛋白适用于绵羊囊性棘球蚴病的现场诊断和监测㊂Huang等[7]开发了一种具有聚集诱导发光特性的荧光发光系统DPA-TPE-Leu,其中识别单元l-亮氨酸酰胺基为亲水部分,该系统是一种高选择性㊁高灵敏度的LAP检测系统㊂Jia等[8]在大肠埃希氏菌Escherichia coli中表达了重组的刚地弓形虫Toxoplasma gondii的LAP㊂Xi等[9]克隆了枯草芽孢杆菌Bacillus subtilis的LAP基因,将该基因在毕赤酵母Pichia pastoris中进行异源表达,结果发现可产生具有较高的热稳定性和催化效率的N端糖基化的LAP蛋白㊂Singh等[10]首次纯化金黄色葡萄球菌Staphylococcus aureus的LAP蛋白,发现其属于分泌型蛋白,而且有助于生物膜的形成,认为其在金黄色葡萄球菌的生存和发病机制中非常重要㊂Jarocki等[11]的研究表明重组MHJ_0461(rMHJ_0461)是一种对亮氨酸㊁丙氨酸㊁苯丙氨酸㊁甲硫氨酸和精氨酸具有广泛底物特异性的LAP蛋白,MHJ_0461存在于猪肺炎支原体Mycoplasma hyopneumoniae表面㊂收稿日期:2020-08-31㊀修回日期:2020-09-22基金项目:广西地方标准制定项目(2018-0103);广西自然科学基金(2018GXNSFAA138008)通信作者:尚常花(1980 ),男,山西泽州人,广西师范大学副教授,博士㊂E-mail:**********************.cn751本课题组前期从广西河池市南丹县大厂镇矿区的重金属污染土壤中筛选出一株细菌,它具有一定的去除六价铬能力,可在含高达250mg/L六价铬的培养基中生长繁殖㊂经过16S rDNA测序,鉴定该菌为假单胞菌属,将其命名为假单胞菌Cr13(Pseudomonas sp.Cr13)㊂假单胞菌为革兰氏阴性菌,在泥土㊁水体及植物体内均可以找到[12]㊂前人对假单胞菌在生物防治㊁生物转化㊁基因克隆㊁基因表达调控㊁蛋白质工程等方面做了大量的研究工作[13-14]㊂但是,在假单胞菌亮氨酸氨肽酶的基因克隆方面研究较少,而亮氨酸氨肽酶与微生物对氮源的利用密切相关㊂因此,本文克隆了假单胞菌Cr13菌株的亮氨酸氨肽酶基因,并进行相应的生物信息学分析,拟为将来对该基因的深入研究奠定良好的基础㊂1㊀材料与方法1.1㊀试验材料假单胞菌Cr13(Pseudomonas sp.Cr13)保存于广西师范大学生命科学学院,过夜培养后用于基因组DNA提取㊂细菌基因组DNA快速抽提试剂盒㊁SanPrep柱式DNA胶回收试剂盒㊁DH5α感受态细胞及氨苄青霉素均购买自生工生物工程(上海)股份有限公司㊂DL2000DNA Marker㊁r Taq㊁T4DNA Ligase㊁pMD19-T Vector及Genome Walking Kit均购买自宝生物工程(大连)有限公司㊂LB液体培养基:胰蛋白胨10g,酵母提取物5g,NaCl10g,加去离子水定容至1000mL,调pH至7.0㊂LB固体培养基则加入2%琼脂粉,LB液体和固体培养基均在121ħ高压灭菌20min㊂如需加入氨苄青霉素(Amp),需等待培养基冷却至约60ħ后,加入Amp母液(100g/L)摇匀,其在培养基中终质量浓度为100mg/L㊂假单胞菌Cr13在LB液体培养基中于30ħ㊁150r/min条件下进行培养㊂1.2㊀试验方法1.2.1㊀基因组DNA的提取取1mL过夜培养的假单胞菌Cr13菌液,12000r/min离心2min弃上清,对细菌沉淀使用细菌基因组DNA快速抽提试剂盒提取总DNA㊂提取的DNA通过1%琼脂糖凝胶电泳检测㊂1.2.2㊀LAP基因核心片段的扩增在NCBI网站上(https:///)查找不同物种LAP基因的保守核苷酸序列,设计一对引物LAP-F/LAP-R(表1),以Cr13菌株的基因组DNA为模板,进行PCR扩增㊂查找的LAP基因序列包括如下物种:Pseudomonas sp.B14-6(登录号:CP053929.1)㊁Pseudomonas mandelii strain LMG21607(登录号:LT629796.1)㊁Pseudomonas mandelii JR-1(登录号:CP005960.1)㊁Pseudomonas sp.ACM7(登录号: CP024866.1)和Pseudomonas sp.URMO17WK12:I11(登录号:LN854573.1)㊂将这些序列用Clustal Omega 软件进行比对,找出保守的核苷酸序列㊂50μL PCR反应体系为:10XPCR Buffer5μL㊁dNTP Mixture (2.5mmol/L)4μL㊁引物LAP-F(20μmol/L)1μL㊁引物LAP-R(20μmol/L)1μL㊁Cr13基因组DNA 2μL㊁r Taq(5U/μL)0.2μL㊁ddH2O36.8μL㊂PCR扩增参数为:94ħ3min;35个循环(94ħ30s, 49.7ħ30s,72ħ50s);72ħ5min㊂PCR产物经2%琼脂糖凝胶电泳检测后,进行回收纯化㊁连接㊁转化㊁测序㊂测序结果在NCBI网站进行Blastx比对分析㊂1.2.2.1㊀第1次genome walking扩增LAP基因5ᶄ端序列根据1.2.2节获得的Cr13LAP基因的核心片段,设计3条基因特异引物yt5-sp1㊁yt5-sp2㊁yt5-sp3(表1)㊂第1轮以Cr13基因组DNA为模板,分别使用Genome Walking Kit中的兼并引物AP1㊁AP2与3条特异引物yt5-sp1㊁yt5-sp2㊁yt5-sp3配对,进行3轮PCR(1st PCR㊁2nd PCR㊁3rd PCR)㊂PCR体系及反应条件均按照试剂盒说明书进行㊂第2轮和第3轮PCR产物经2%琼脂糖凝胶电泳确定目的片段后,对目的片段进行切胶回收纯化㊁连接㊁转化㊁测序及序列分析㊂851广西师范大学学报(自然科学版),2021,39(1)表1㊀本研究使用的引物Tab.1㊀Primers used in this study引物序列(5ᶄ-3ᶄ)LAP-F ATGATCGGCACCAGCATGTGCLAP-R TGTGCATGATCGGTTTCAGyt5-sp1AGCAGGTACTGGGTCAGCAATGGAAyt5-sp2GGAAGCAGGCAGCGGTGATGGTyt5-sp3CTTCGGCGTCGGTGTTGAGGATyt(5)-sp11CGCCCATGTCATACTTCATCTCATCCyt(5)-sp22GGATTTCTTGCCACCGTTGTATTGCyt(5)-sp33GAGGTTCGGCTTTCTGGCTCTTGM13(-40)Forward GTTTTCCCAGTCACGACM13Reverse CAGGAAACAGCTATGAC1.2.2.2㊀第2次genome walking扩增LAP基因5ᶄ端序列对使用AP1引物进行的第1次genome walking第3轮PCR获得的目的条带测序,发现尚未扩增到起始密码子ATG㊂因此,按照第1次genome walking克隆及测序的序列(AP1引物3rd PCR产物的序列),再次设计3条基因特异引物yt(5)-sp11㊁yt(5)-sp22㊁yt(5)-sp33(表1),进行第2次genome walking㊂第1轮以Cr13基因组DNA为模板,分别使用Genome Walking Kit中的兼并引物AP1㊁AP2与3条基因特异引物yt(5)-sp11㊁yt(5)-sp22㊁yt(5)-sp33配对,进行3轮PCR㊂PCR反应体系及程序均按照试剂盒说明书进行,对目的片段进行切胶回收纯化㊁连接㊁转化㊁测序及序列分析㊂1.2.3㊀PCR片段的回收纯化㊁连接㊁转化㊁测序首先,对获得的PCR目的片段切胶后通过SanPrep柱式DNA胶回收试剂盒进行回收纯化㊂然后, PCR纯化产物与pMD19-T Vector进行连接反应,连接体系为PCR纯化产物16μL㊁pMD19-T Vector1μL㊁10xT4DNA Ligase Buffer2μL㊁T4DNA Ligase1μL,总体积20μL㊂连接条件为16ħ反应1h㊂取20μL 连接产物按照说明书采用热激法转化DH5α感受态细胞㊂最后,吸取100μL菌液均匀涂布到含100mg/L Amp的LB固体培养基中㊂倒置后37ħ过夜培养12~16h,使用无菌牙签挑取转化子的单菌落到2mL含100mg/L Amp的LB液体培养基中进行摇菌㊂以转化子菌液为模板,M13(-40)Forward和M13Reverse为引物,使用r Taq进行PCR扩增以验证阳性转化子㊂PCR体系参照说明书,退火温度为41.5ħ㊂挑选鉴定的阳性转化子菌液送赛默飞世尔广州办事处,使用载体引物M13F和M13R进行双向测序㊂1.2.4㊀3次测序结果的序列拼接及生物信息学分析对上述3次测序结果,根据重叠序列进行拼接,获得假单胞菌Cr13亮氨酸氨肽酶基因LAP全长DNA 序列㊂将全长DNA在NCBI上进行Blastx比对,检测与其他同源蛋白的相似性㊂利用Signal P-5.0㊁ExPASy㊁CD-search㊁TMPred㊁ScanProsite㊁InterProscan㊁Psort等软件对Cr13LAP进行生物信息学分析㊂2㊀结果与分析2.1㊀克隆LAP全长DNA序列以Cr13基因组DNA为模板,使用一对引物LAP-F㊁LAP-R进行PCR扩增后,获得约500bp条带(图1(A))㊂测序后获得513bp序列,将序列进行Blastx比对,与其他物种的LAP表现出较高的同源性,表明获得的513bp核心片段确实是Cr13LAP的核心片段㊂根据获得的513bp核心片段设计1组引物yt5-sp1㊁yt5-sp2㊁yt5-sp3,分别与Genome Walking Kit中的AP1/AP2primer配对进行3轮PCR㊂其中,与试剂盒中AP1Primer配对时,条带较大,因此选用AP1 primer的3rd PCR产物进行纯化㊁连接㊁转化和测序㊂LAP基因5ᶄ端第1次genome walking扩增序列为867bp(图1(B))㊂ 对第1次genome walking 扩增获得的序列进行Blastx 比对分析,发现没有扩增到起始密码子㊂因此,根据867bp 序列设计一组引物yt(5)-sp11㊁yt(5)-sp22㊁yt(5)-sp33,分别与Genome Walking Kit 中的AP1/AP2primer 配对进行3轮PCR㊂其中,与试剂盒中的AP1Primer 配对时,条带清晰,因此选用AP1primer 的3rd PCR 产物进行纯化㊁连接㊁转化和测序㊂LAP 基因5ᶄ端第2次genome walking 扩增序列为810bp (图1(C))㊂M:DL2000DNA Marker;A 中1~4为4个平行样品;B 中1㊁2为AP1引物的2nd 和3rd PCR 产物,3㊁4为AP2引物的2nd 和3rd PCR 产物,送去测序片段长867bp;C 中1㊁2为AP1引物的2nd 和3rd PCR 产物,3㊁4为AP2引物的2nd 和3rd PCR 产物,送去测序片段长810bp㊂图1㊀假单胞菌Cr13LAP 基因全长DNA 的克隆Fig.1㊀Cloning of full-length DNA sequence of LAP from Pseudomonas sp.Cr13根据获得的核心片段513bp㊁第1次和第2次genome walking 扩增获得的5ᶄ端序列867bp 和810bp,去除重叠序列进行拼接,获得LAP 全长DNA 序列为1491bp,编码496aa(见图2,起始密码子ATG 到终止密码子TGA)㊂图2㊀假单胞菌Cr13LAP 基因全长DNAFig.2㊀Full-length DNA sequence of LAP from Pseudomonas sp.Cr13951广西师范大学学报(自然科学版),2021,39(1)2.2㊀分析Cr13菌株LAP 编码蛋白质序列的特征与亲缘关系Cr13菌株LAP 基因编码496aa 的蛋白质,使用Signal P-5.0预测其LAP 蛋白的信号肽序列,结果表明该蛋白质没有信号肽,为非分泌蛋白㊂在ExPASy 网站(https:// /)预测Cr13菌株LAP 蛋白的一系列理化性质,预测结果如下:相对分子质量为52.45649kD,分子式为C 2327H 3796N 638O 704S 15,等电点pI 为8.06,半衰期大于10h,不稳定系数为22.17,属于稳定蛋白㊂该蛋白中含量较高的氨基酸包括Leu (12.7%)㊁Gly (11.1%)㊁Ala (10.9%)㊁Lys (7.5%)㊂带负电荷氨基酸残基(Asp +Glu)总数为54个,带正电荷氨基酸残基(Arg +Lys)总数为56个㊂Grand average of hydropathicity (GRAVY)为-0.017,预测该蛋白质为水溶性蛋白㊂使用NCBI 网站的CD-search 预测LAP 蛋白存在保守的PRK00913superfamily domain㊂利用TMPred 软件预测Cr13LAP 蛋白的跨膜区(图3)㊂预测结果表明:Cr13LAP 蛋白含5个由内至外的跨膜区和4个由外至内的跨膜区,共计9个跨膜区㊂Cr13菌株LAP 蛋白具有较高的序列保守性㊂利用ScanProsite 软件查找Cr13菌株LAP 蛋白的模体(motif),发现该蛋白具有1个保守的细胞质氨肽酶模体(从第346个氨基酸残基到第353个氨基酸残基),D 和E 氨基酸是Zn 2+/Mn 2+结合位点㊂利用InterProscan 软件分析Cr13LAP 蛋白,结果表明:C 端含有1个M17肽酶保守结构域(第184~486位氨基酸残基),N 端含有1个M17肽酶保守结构域(第19~147位氨基酸残基),推测菌株Cr13LAP 属于M17肽酶家族成员㊂利用Psort 预测LAP 蛋白的亚细胞定位,结果表明:定位于线粒体和内质网的可能性分别为39.1%和30.4%,明显高于其他部位的可能性㊂由此推测,Cr13菌株的LAP 蛋白极有可能定位于线粒体中㊂图3㊀预测的假单胞菌Cr13的跨膜结构域Fig.3㊀Transmembrane regions prediction of LAP from Pseudomonas sp.Cr13061http:// 图4㊀不同物种LAP 蛋白质序列比对结果Fig.4㊀Alignment result of LAP sequence from different species㊀㊀Cr13菌株的LAP 与假单胞菌属的其他物种的LAP 蛋白有很高的氨基酸序列同源性,它与Pseudomonas fluorescens (WP _150631252.1)㊁Pseudomonas frederiksbergensis (WP _074879959.1)㊁Pseudomonas brassicacearum (WP _123365725.1)㊁Pseudomonas arsenicoxydans (WP _140666549.1)及Pseudomonas mandelii (WP_140677786.1)亮氨酸氨肽酶的氨基酸序列同源性分别为99%㊁98%㊁99%㊁98%和98%㊂Cr13菌株的161广西师范大学学报(自然科学版),2021,39(1)LAP 与假单胞菌属的其他物种LAP 的蛋白质序列比对结果见图4㊂由此可见,亮氨酸氨肽酶的氨基酸序列在假单胞菌属内具有很强的保守性㊂作为一类水解蛋白质的外肽酶,亮氨酸氨肽酶属于M1或M17肽酶家族成员,对植物的生长发育和逆境响应具有重要作用,与微生物对氮源的利用密切相关[1,4,15]㊂M1和M17肽酶家族成员的C 端均含有1个催化结构域㊂但是,金属肽酶模体HEXXH 仅存在于M1肽酶的催化结构域[16]㊂目前,已经报道的植物和微生物的LAP 多属于M17肽酶家族成员㊂本文报道的Pseudomonas sp.Cr13LAP 也属于M17肽酶家族成员㊂本文研究还获得了Cr13LAP 基因的426bp 启动子序列(图5)㊂通过PlantCARE 在启动子序列中预测了一系列潜在的顺式作用元件,包括ARE (厌氧诱导)㊁LTR (低温响应)㊁MYB㊁MYB-like sequence㊁TGA-element (生长素响应)㊁TGACG-motif (茉莉酸甲酯响应)㊁Unnamed_4㊁W box 及chs-Unit 1m1(光响应)㊂此外,启动子中还发现了典型的启动子元件TATA-box 和CAAT-box,这些结果为将来深入研究环境因素对Cr13菌株LAP 基因的表达调控奠定了基础㊂图5㊀假单胞菌Cr13LAP 基因的启动子Fig.5㊀Promoter sequence of LAP from Pseudomonas sp.Cr133㊀讨论假单胞菌Cr13LAP 基因全长为1491bp,编码496个氨基酸,与假单胞菌属其他细菌编码的氨基酸个数基本一致㊂但是,在不同种类的生物中,LAP 基因编码区的长度差别较大㊂在皱纹盘鲍Haliotis discus hannai ㊁文昌鱼Branchiostoma belcheri ㊁太平洋牡蛎Crassostrea gigas ㊁舌形贝Lingula anatina 和囊舌虫Saccoglossus kowalevskii 等5种动物中LAP 基因编码的氨基酸个数为847~870[17]㊂在橡胶树Hevea brasiliensis ㊁拟南芥Arabidopsis thaliana ㊁番茄Lycopersicon esculentum ㊁葡萄Vitis vinifera ㊁水稻Oryza sativa 和欧芹Petroselinum crispum 等6种植物中LAP 基因编码的氨基酸个数为520~528[18]㊂由此可见,不同种类的生物中LAP 基因编码的氨基酸个数差别较大,其中植物和微生物的较为接近㊂本文所用Cr13菌株与假单胞菌属的其他细菌亲缘关系最近,符合传统的分类标准㊂对菌株Cr13LAP 蛋白进行了预测,结果表明:1)Cr13LAP 含9个跨膜区,与文蛤Meretrix meretrix 中LAP 的结果[19]不一致㊂2)Cr13的LAP 为稳定蛋白,定位于线粒体的可能性最大,为39.1%,与橡胶树中LAP 的定位部位(细胞质)[18]不一致,表明不同生物中LAP 的亚细胞定位不同㊂3)Cr13LAP 具有1个保守的细胞质氨肽酶模体NTDAEGRL(从第346个氨基酸残基到第353个氨基酸残基),D 和E 氨基酸是Zn 2+/Mn 2+结合位点,这与前人对其他物种LAP 的研究结果[18-19]一致,说明了保守模体功能的重要性㊂4)Cr13LAP 属于M17肽酶家族成员,因此没有金属肽酶模体HEXXH,这与前人关于其他生物M17肽酶家族成员的结果[18-19]一致㊂综上所述,Cr13菌株LAP 的相关生物信息学预测为将来进一步研究该基因的功能奠定了良好的基础㊂染色体步移技术是一种重要的分子生物学技术,使用该技术能够高效地获取与已知序列相邻的未知序列㊂染色体步移技术可以通过Thermal asymmetric interlaced PCR㊁SiteFinding-PCR 及Self-Formed261361Adaptor PCR等方式实现[20-21]㊂已有不少研究人员通过该技术成功获取到未知序列㊂韩琪等[22]利用染色体步移技术克隆了斜纹夜蛾Spodoptera litura GOBP2基因的5ᶄ侧翼区㊂郭丽娜等[23]利用该技术成功地克隆了中华蜜蜂Apis cerana气味受体AcerOr1基因启动子㊂周惠强等[24]利用同源克隆和染色体步移技术克隆了光倒刺鲃Spinibarbus hollandi的黑素皮质素受体-5基因㊂本文使用Genome Walking Kit成功克隆了假单胞菌Cr13菌株LAP基因的未知序列㊂此外,本研究还获得了Cr13菌株LAP基因的426bp启动子序列,预测了一系列潜在的顺式作用元件㊂细胞内外的信号物质可以通过调控蛋白质结合于顺式作用元件上影响靶标基因的表达[25-26]㊂本研究的启动子预测为将来深入研究环境因素对Cr13菌株LAP基因的表达调控机制奠定了良好的基础㊂4㊀结论本研究通过染色体步移对假单胞菌LAP基因全长序列进行克隆,并对该基因进行了一系列生物信息学分析㊂假单胞菌Cr13菌株LAP基因全长为1491bp,在其启动子区域发现了一系列潜在的顺式作用元件㊂上述结果为深入研究该基因的功能和表达调控提供了基础㊂参㊀考㊀文㊀献[1]㊀CHAO W S,GU Y Q,PAUTOT V V,et al.Leucine aminopeptidase RNAs,proteins,and activities increase in response towater deficit,salinity,and the wound signals systemin,methyl jasmonate,and abscisic acid[J].Plant Physiology,1999, 120(4):979-992.DOI:10.1104/pp.120.4.979.[2]㊀MURPHY A,PEER W A,TAIZ L.Regulation of auxin transport by aminopeptidases and endogenous flavonoids[J].Planta,2000,211(3):315-324.DOI:10.1007/s004250000300.[3]㊀ISHIZAKI T,TOSAKA A,NARA T,et al.Leucine aminopeptidase during meiotic development[J].European Journal ofBiochemistry,2002,269(3):826-832.DOI:10.1046/j.0014-2956.2001.02713.x.[4]㊀MATSUI M,FOWLER J H,WALLING L L.Leucine aminopeptidases:diversity in structure and function[J].BiologicalChemistry,2006,387(12):1535-1544.DOI:10.1515/BC.2006.191.[5]㊀ZHANG S,CAI X,LUO X,et al.Molecular cloning and characterization of leucine aminopeptidase gene from Taeniapisiformis[J].Experimental Parasitology,2018,186:1-9.DOI:10.1016/j.exppara.2018.01.012.[6]㊀WU M,YAN M,XU J,et al.Expression,tissue localization and serodiagnostic potential of Echinococcus granulosus leucineaminopeptidase[J].International Journal of Molecular Sciences,2018,19(4):1063.DOI:10.3390/ijms19041063.[7]㊀HUANG S,WU Y,ZENG F,et al.A turn-on fluorescence probe based on aggregation-induced emission for leucineaminopeptidase in living cells and tumor tissue[J].Analytica Chimica Acta,2018,1031:169-177.DOI:10.1016/j.aca.2018.05.032.[8]㊀JIA H,NISHIKAWA Y,LUO Y,et al.Characterization of a leucine aminopeptidase from Toxoplasma gondii[J].Molecularand Biochemical Parasitology,2010,170(1):1-6.DOI:10.1016/j.molbiopara.2009.11.005.[9]㊀XI H,TIAN Y,ZHOU N,et al.Characterization of an N-glycosylated Bacillus subtilis leucine aminopeptidase expressed inPichia pastoris[J].Journal of Basic Microbiology,2015,55(2):236-246.DOI:10.1002/jobm.201400368. [10]SINGH A K,SINGH R,TOMAR D,et al.The leucine aminopeptidase of Staphylococcus aureus is secreted and contributesto biofilm formation[J].International Journal of Infectious Diseases,2012,16(5):e375-e381.DOI:10.1016/j.ijid.2012.01.009.[11]JAROCKI V M,SANTOS J,TACCHI J L,et al.MHJ_0461is a multifunctional leucine aminopeptidase on the surface ofMycoplasma hyopneumoniae[J].Open Biology,2015,5(1):140175.DOI:10.1098/rsob.140175.[12]向照举,李小璐,李治.假单胞菌属合成环脂肽分子的研究进展[J].中国生物制品学杂志,2014,27(7):974-980.DOI:10.13200/ki.cjb.000356.[13]杨光富,魏云林.假单胞菌研究现状及应用前景[J].生物技术通报,2011(1):37-39,49.DOI:10.13560/ki.biotech.bull.1985.2011.01.005.[14]查代明,张炳火,李汉全,等.假单胞菌属脂肪酶的分子生物学研究进展[J].中国生物工程杂志,2015,35(9):461广西师范大学学报(自然科学版),2021,39(1)114-121.DOI:10.13523/j.cb.20150916.[15]MOORE B D.Bifunctional and moonlighting enzymes:lighting the way to regulatory,control[J].Trends in Plant Science,2004,9(5):221-228.DOI:10.1016/j.tplants.2004.03.005.[16]RAWLINGS N D,BARRETT A J,BATEMAN A.MEROPS:the peptidase database[J].Nucleic Acids Research,2010,38:D227-D233.DOI:10.1093/nar/gkp971.[17]胡健健,陈玉磊,段雪昆,等.皱纹盘鲍亮氨酸氨肽酶基因cDNA克隆及表达分析[J].集美大学学报(自然科学版),2017,22(5):31-39.DOI:10.19715/j.jmuzr.2017.05.005.[18]刘登冉,赵丽,陈月异,等.巴西橡胶树胶乳亮氨酸氨肽酶基因克隆及表达[J].热带作物学报,2008,29(4):403-409.DOI:10.3969/j.issn.1000-2561.2008.04.001.[19]阮文斌,董迎辉,高晓艳,等.文蛤亮氨酸氨肽酶LAP3的基因克隆及其在幼体不同发育时期㊁成体不同组织的表达分析[J].海洋学报,2017,39(2):96-104.DOI:10.3969/j.issn.0253-4193.2017.02.009.[20]SHAPTER F M,WATERS D L.Genome walking[J].Methods in Molecular Biology,2014,1099:133-146.DOI:10.1007/978-1-62703-715-0_12.[21]LEONI C,VOLPICELLA M,DE LEO F,et al.Genome walking in eukaryotes[J].The FEBS Journal,2011,278(21):3953-3977.DOI:10.1111/j.1742-4658.2011.08307.x.[22]韩琪,胡波,李升云,等.斜纹夜蛾气味结合蛋白基因GOBP2转录调控区的克隆及活性分析[J].南京农业大学学报,2019,42(6):1059-1066.DOI:10.7685/jnau.201904004.[23]郭丽娜,赵慧婷,任有蛇,等.中华蜜蜂气味受体AcerOr1基因启动子克隆及序列分析[J].山西农业科学,2020,48(4):623-626.DOI:10.3969/j.issn.1002-2481.2020.04.32.[24]周惠强,陈凯棱,舒琥,等.光倒刺鲃mc5r基因的克隆及对饥饿的响应[J].南方水产科学,2019,15(4):99-106.DOI:10.12131/20190027.[25]梁明丽,刘波,张伟.巴斯德毕赤酵母鼠李糖代谢基因LRA3功能及其启动子顺式作用元件的研究[J].生物技术进展,2019,9(5):518-526.DOI:10.19586/j.2095-2341.2019.0031.[26]冯靖,杨灿,卢娟芳,等.杏CCD1和CCD4启动子克隆及顺式作用元件分析[J].园艺学报,2020,47(5):939-952.DOI:10.16420/j.issn.0513-353x.2019-0610.Cloning and Bioinformatics Analysis of LAP Gene from Pseudomonas GUO Chen1,ZHOU Fei2,3,HAN Biao1,PAN Cui1,WU Jiemin1,YANG Ting2,3,SHANG Changhua2,3∗(1.Scientific Research Academy of Guangxi Environmental Protection,Nanning Guangxi530022,China;2.College of Life Science,Guangxi Normal University,Guilin Guangxi541006,China;3.Key Laboratory of Ecology of Rare and Endangered Species and Environmental Protection(Guangxi Normal University),Ministry of Education,Guilin Guangxi541006,China) Abstract:To explore the molecular structure and functional characteristics of LAP gene encoding Leucine aminopeptidases(LAPs)and its role in the process of growth and development,the Pseudomonas Cr13LAP gene was cloned and characterized by bioinformatics in this study.Firstly,the core fragment of LAP was cloned. Secondly,the full-length DNA of LAP gene was cloned by two Genome stly,LAP gene was analyzed by bioinformatics software.The result showed that the full-length DNA of LAP gene was1491bp in Pseudomonas Cr13.Bioinformatics analysis indicated that the LAP gene was located in the mitochondrion.The LAP gene had a conserved cytoplasmic aminopeptidase motif NTDAEGRL.The Pseudomonas Cr13LAP promoter(426bp) contained the basic elements such as TATA-Box and CAAT-box and some potential cis-acting element.Cloning of full-length DNA laid a good foundation for further research on LAP gene function.Cloning and characterization of the promoter region of Pseudomonas sp.Cr13LAP gene provided substantial basis for further research on the mechanism of regulation and expression of LAP gene.Keywords:Pseudomonas;leucine aminopeptidase;genome walking;bioinformatics(责任编辑㊀王龙杰)。
MYB转录因子在植物抗逆胁迫中的作用及其分子机理_刘蕾

已在拟南芥、玉米等真核生物中发现存在着大量的 MYB 转录因子。例如, 拟南芥中已发现大约 130 个 成员, 玉米中大约有 100 个成员[7~9]等。它们广泛参 与植物次生代谢的调控、对激素和环境因子的应 答[10~13], 并对细胞分化、器官的形成、植物叶片的 形态建成及抗病具有重要的调节作用[14~17]。
据统计, 在世界范围内适于耕种的土地不足 10%, 大部分土地处于干旱、盐渍、沼泽等逆境中。 随着环境恶化和人口不断增长, 迫切需要培育出能 在各种逆境下生长的经济作物。利用转录因子改良 和提高植物的综合抗逆性成为一种很有潜力的方 法。文章简要介绍了 MYB 转录因子的结构特征和 功能特性, 着重讨论了其在植物抗逆反应中的作用 及其分子机理。
摘要: 在植物抗逆反应体系中, 通过转录因子调控功能基因的表达, 是植物逆境应答反应的关键环节。作为植
物体内最大的转录因子家族之一, MYB(v-myb avian myeloblastosis viral oncogene homolog)类转录因子在
植物抗逆胁迫过程中起着重要的作用。文章论述了 MYB 转录因子的结构特征、功能特性及其分子机理, 并结 合研究进展着重讨论了它们在植物抗逆胁迫中的作用。 关键词: MYB; 转录因子; 抗逆胁迫; 转录调控
transcriptionalregulation转录因子transcriptionfactortf又称反式作用因子是指能与真核基因启动子区域中的顺式作用元件发生特异性结合通过它们之间以及与其他相关蛋白之间的相互作用激活或抑制转录从而保证目的基因以特定的强度在特定的时间与空间表达的蛋白质分子
HEREDITAS (Beijing) 2008 年 10 月, 30(10): 1265―1271 ISSN 0253-9772
番茄CKX基因家族生物信息学分析

番茄CKX基因家族生物信息学分析李伟范胜男王星贾兵国王丽萍*(河北工程大学园林与生态工程学院,河北邯郸056038)摘要植物细胞内的细胞分裂素受到细胞分裂素脱氢酶/氧化酶(CKX)的调节,维持植物体内细胞分裂素动态平衡。
为探究番茄基因组中CKX基因(SlCKX)家族成员的信息,本研究通过现代生物信息学分析,对番茄中CKX基因家族进行鉴定和分析。
结果表明,在番茄全基因组中鉴定出9个CKX基因家族成员,蛋白长度在453~553个氨基酸之间,编码蛋白分子量在51660.72~52493.64kD之间,为亲水性蛋白;番茄CKX基因分在4个亚族内,且SlCKX家族成员中含有3~5个内含子以及4~6个外显子;9个番茄CKX家族基因不均匀地分布在5条染色体上,番茄CKX基因家族包含11种顺式作用元件,其中脱落酸响应元件分布最广。
本研究可为番茄CKX基因家族的功能和应用研究提供一定参考。
关键词番茄;生长素氧化酶/脱氢酶;基因家族;生物信息学中图分类号S641.2文献标识码A文章编号1007-5739(2023)12-0056-04DOI:10.3969/j.issn.1007-5739.2023.12.012开放科学(资源服务)标识码(OSID):Bioinformatics Analysis of CKX Gene Family in TomatoLI Wei FAN Shengnan WANG Xing JIA Bingguo WANG Liping*(School of Landscape and Ecological Engineering,Hebei University of Engineering,Handan Hebei056038) Abstract Cytokinin in plant cells is regulated by cytokinin oxidase/dehydrogenase(CKX)to maintain cytokinin homeostasis in plants.In order to explore the information of CKX gene family members(SlCKX)in the tomato genome, this paper identified and analyzed the CKX gene family in tomato through modern bioinformatics analysis.The results showed that9members of the CKX gene family were identified in the whole tomato genome,the protein length was between453and553amino acids,and the encoded protein molecular weight was between51660.72and52493.64kD, which were hydrophilic proteins.Tomato CKX gene was divided into four subfamilies,and members of the SlCKX family contain3-5introns and4-6exons;9tomato CKX family genes were unevenly distributed on5chromosomes,and the tomato CKX gene family contained11cis-acting elements,among which,abscisic acid-responsive elements were the most widely distributed.This study can provide some references for the function and application of tomato CKX gene family.Keywords tomato;auxin oxidase/dehydrogenase;gene family;bioinformatics细胞分裂素(cytokinin,CK)是一种植物激素,通过介导内源性细胞分裂素的代谢,影响植物的生长、发育和产量,其还是化学N6取代嘌呤衍生物[1-2]。
【国家自然科学基金】_茉莉酸甲酯(meja)_基金支持热词逐年推荐_【万方软件创新助手】_20140802
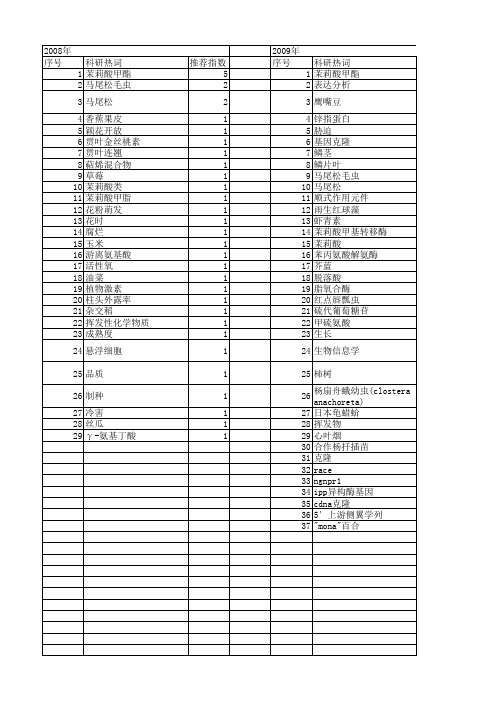
推荐指数 4 3 2 2 2 2 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
2010年 序号 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45
2012年 序号 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52
科研热词 茉莉酸甲酯 测定 昆虫取食 黑色软蚧蚜小蜂 高效液相色谱法 马尾松苗木 顶空-固相微萃取 防御酶 金橘 酚酸 超氧化物歧化酶 褐软蚧 茉莉酸类物质 茉莉酸 色质联用 色谱 胰凝乳蛋白酶抑制剂 稻瘟病 疏水相互作用 电化学行为 电化学反应 甲酯化 玻碳电极 水稻 水杨酸 气相色谱 气体防御信号 毛细管电泳 植物样品 机械损伤 提取纯化 多酚氧化酶 同时提取 叶绿素荧光 十二烷基硫酸钠 分离与检测 内源水杨酸 免疫分析 光谱分析法 光合作用 信号分子 串联质谱法 sds胶束 meja hplc
沙冬青 1 水分 1 氮 1 毛葡萄 1 毛白杨 1 植保素 1 桃 1 柿树 1 枣树 1 果实品质 1 果实 1 枇杷 1 日本龟蜡蚧 1 总酚含量 1 序列分析 1 实时荧光定量pcr 1 品质控制 1 冷害 1 低温预贮 1 乙烯பைடு நூலகம்1 中国野生葡萄 1 一氧化氮 1 xa21基因启动子 1 vq基因家族 1 vqsap基因 1 vqcoi1基因 1 seed germination 1 pppgip1启动子 1 perilla frutescens 1 meja 1 jasmonic acid 1 indolic glucosinolate 1 high temperature and air humidity 1 h2o2 1 gus活性 1 coil 1 carknif2 1 ca2 离子流 1 arabidopsis thaliana 1 antioxidant 1
两种不同化感潜力水稻CYP73A38基因及其启动子的克隆分析

基因组学与应用生物学,2020年,第39卷,第9期,第4073-4081页研究报告Research Report两种不同化感潜力水稻CYP73A38基因及其启动子的克隆分析杨奕*孙一丁*马继琼许明辉**云南省农业科学院生物技术与种质资源研究所,云南省农业生物技术重点实验室,农业部西南作物基因资源与种质创制重点实验室,昆明, 650223*同等贡献作者**通信作者,*****************摘要水稻化感作用指水稻通过向环境中释放次生代谢产物来影响周围植物的生长。
本研究以国际公认的化感水稻PI312777和非化感水稻Lemont为材料,克隆肉桂酸-4-羟化酶(C4H)编码基因CYP73A38的cDNA序列进行对比分析,发现两种水稻在该基因的序列差异极小,存在6个位点差异。
为了进一步揭示两种水稻化感潜力不同的原因,对CYP73A38启动子进行了克隆分析,获得CYP73A38基因启动子序列,该启动子序列包含CAAT-box、TATA-box、光响应元件、赤霉素响应元件、生长素响应元件、低温响应元件等多个顺式作用元件及MYB类转录因子结合位点,且差异位点均不在顺式作用元件上,由此推测两种水稻化感潜力的差异与基因结构没有相关性。
不同化感潜力水稻CYP73A38基因及其启动子的克隆与序列分析,将为进一步研究CYP73A38基因的表达调控及其启动子功能分析提供参考,为加快水稻化感抗性品种的选育进程提供依据。
关键词化感水稻,CYP73A38,启动子Cloning Analysis of CYP73A38Gene and Its Promoter in Two Different Allelopathic Potential of RiceYang Yi*Sun Yiding*Ma Jiqiong Xu Minghui**The Key Laboratory of Biotechnology Research of Yunnan Province,Key Lab of Southwestern Crop Gene Resources and Germplasm Innovation of Ministry of Agriculture,Institute of Biotechnology and Genetic Resources,YAAS,Kunming,650223*The authors contributed equally to this work**Corresponding author,*****************DOI:10.13417/j.gab.039.004073Abstract Rice alle lopathy is mainly responsible for the growth of around plant by releasing secondary metabolites into the environment.In this study,allelopathic rice PI312777and non-allelopathic rice Lemont were used as materials,cloned the cinnamic acid-4-hydroxylase(C4H)coding gene CYP73A38full length of sequence cDNA and comparatived difference.The differences in the sequence of this gene in allelopathic and non-allelic rice were slight.A total of6nucleotide variations were found in the ORFs.In order to research the reason of two rice with different allelopathic potential,the study cloned the promoter sequence of CYP73A38.The promoter sequence contained multiple cis-acting elements,such as CAAT-box,TATA-box,light responsive element,gibberellins responsive element,auxin responsive element,low-temperature responsive element,MYB recognition site,and others.And all variation sites are not in the cis-acting elements.Thus speculate that the differences of two allelopathy potential rice do es not correlate with the genetic structure.Cloning and characterization of the基金项目:本研究由云南省科技计划青年项目(2017FD204)资助引用格式:Yang Y.,Sun Y.D.,Ma J.Q.,and Xu M.H.,2020,Cloning analysis of CYP73A38gene and its promoter in two different allelopathic potential of rice,Jiyinzuxue Yu Yingyong Shengwuxue(Genomics and Applied Biology),39(9):4073-4081(杨奕,孙一丁,马继琼,许明辉,2020,两种不同化感潜力水稻CYP73A38基因及其启动子的克隆分析,基因组学与应用生物学,39(9): 4073-4081)基因组学与应用生物学水稻是全世界最主要的粮食作物之一,据联合国粮农组织(FAO)调查,每年因病虫草害等生物因素造成的粮食损失约占全球粮食总产量的1/3,其中草害导致的农作物产量损失约为11%。
高粱SBP_基因家族鉴定及干旱胁迫下的表达模式分析

㊀山东农业科学㊀2024ꎬ56(3):1~10ShandongAgriculturalSciences㊀DOI:10.14083/j.issn.1001-4942.2024.03.001收稿日期:2024-01-12基金项目:贵州省科技计划项目(黔科合基础-ZK 2023 一般169)ꎻ贵州省基层农技推广项目(仁怀基地示范 2301 01号)ꎻ贵州省科技成果应用及产业计划项目(黔科中引地 2022 4045)作者简介:姜昱雯(1996 )ꎬ女ꎬ贵州遵义人ꎬ硕士ꎬ研究实习员ꎬ主要从事分子植物育种研究ꎮE-mail:450141704@qq.com通信作者:邵明波(1970 )ꎬ男ꎬ贵州石阡人ꎬ研究员ꎬ从事高粱育种等工作ꎮE-mail:563189433@qq.com高粱SBP基因家族鉴定及干旱胁迫下的表达模式分析姜昱雯1ꎬ陈满静1ꎬ赵应2ꎬ任艳1ꎬ周棱波1ꎬ沈佳奇1ꎬ邵明波1(1.贵州省旱粮研究所ꎬ贵州贵阳㊀550006ꎻ2.仁怀市农业农村局ꎬ贵州仁怀㊀564500)㊀㊀摘要:在高等植物中ꎬSQUAMOSAPromoterBindingProtein-Box(SBP)普遍参与植物的生长㊁开花调控及细胞程序性死亡等过程ꎮ本研究对高粱(SorghumbicolorL.)SBP转录因子家族进行鉴定并进行生物信息学分析ꎬ同时利用qRT-PCR对高粱SBP基因在PEG-6000模拟干旱胁迫下的表达进行分析ꎬ以验证其功能ꎮ结果表明ꎬ从高粱中共鉴定出19个SBP成员ꎬ除第8条染色体外ꎬ其他9条染色体上均分布有SBP基因ꎻ绝大部分SBP蛋白在亚细胞水平定位于细胞核ꎬ且均为亲水性蛋白质ꎮ系统发育分析结果表明ꎬ与水稻类似ꎬ这19个高粱SBP基因也可分为3个亚类ꎬ分别含有7㊁5㊁7个SBP基因ꎻ相比于拟南芥ꎬ高粱SBP与水稻㊁玉米的SBP关系更近ꎮ启动子顺式作用元件预测结果显示ꎬ高粱SBP可能参与了脱落酸㊁茉莉酸甲酯信号通路ꎬ部分成员可能与植物的低温响应㊁昼夜节律调控有关ꎮqRT-PCR分析结果显示ꎬ高粱SBP基因的表达具有组织特异性ꎬ有17个基因受干旱胁迫诱导上调表达ꎬ且随胁迫时间的延长存在差异化表达模式ꎮ本研究结果可为后续研究高粱SBP基因的功能提供理论依据ꎮ关键词:高粱ꎻSBP基因家族ꎻ生物信息学分析ꎻ基因表达ꎻ干旱胁迫中图分类号:S514:Q78㊀㊀文献标识号:A㊀㊀文章编号:1001-4942(2024)03-0001-10IdentificationofSorghumSBPGeneFamilyandTheirExpressionPatternsunderDroughtStressJiangYuwen1ꎬChenManjing1ꎬZhaoYing2ꎬRenYan1ꎬZhouLengbo1ꎬShenJiaqi1ꎬShaoMingbo1(1.GuizhouInstituteofUplandCropsꎬGuiyang550006ꎬChinaꎻ2.RenhuaiBureauofAgricultureandRuralAffairsꎬRenhuai564500ꎬChina)Abstract㊀InthehigherplantsꎬSQUAMOSApromoterbindingprotein ̄box(SBP)iswidelyinvolvedinplantgrowthꎬfloweringregulationandprogrammedcelldeath.InthisstudyꎬthesorghumSBPgeneswerei ̄dentifiedꎬandthentheirbioinformaticsanalysiswasconductedandtheirexpressionunderPEG ̄6000simula ̄teddroughtstresswasanalyzedusingqRT ̄PCR.Theresultsshowedthat19membersofsorghumSBPfamilywereidentified.Exceptthe8thchromosomeꎬtheSBPgenesweredistributedonalltheother9chromosomes.TheSBPproteinswerehydrophilicproteinsꎬandmostofthemwerelocatedinnucleusatsubcellularlevel.SimilartothoseofOryzasativaꎬthesorghumSBPmembersalsocouldbedividedintothreegroupsthroughphylogeneticanalysisꎬcontaining7ꎬ5and7membersrespectivelyꎻsorghumSBPshadcloserrelationshipswiththoseofOryzasativaandZeamays.Thepredictionresultsofcis ̄actingelementsindicatedthatsorghumSBPsmightparticipateinabscisicacidandmethyljasmonatesignalingpathwaysꎬandsomemembersmightberelatedtoplantlowtemperatureresponseandcircadianrhythmregulation.qRT ̄PCRanalysisresultsshowedthattheexpressionofsorghumSBPgenesweretissue ̄specificꎬand17geneswereinducedtoup ̄regulateex ̄pressionundersimulateddroughtconditionsandshoweddifferentiatedexpressionpatternswiththeextensionofstresstime.TheseresultscouldprovidetheoreticalbasesforsubsequentresearchonSBPgenesfunction.Keywords㊀SorghumbicolorL.ꎻSBPgenefamilyꎻBioinformaticsanalysisꎻGeneexpressionꎻDroughtstress㊀㊀转录因子是生物体中分子调控网络的重要组成部分ꎬ控制着植物生长发育㊁胁迫响应等一系列生物学过程ꎬ不同的转录因子家族结合的下游基因启动子基序存在显著差别[1-2]ꎮSQUAMOSAPromoterBindingProtein-Box(SBP)是一类植物特异性的转录因子家族ꎬ因其成员含有高度保守的DNA结合结构域SBP而得名[3]ꎮSBP保守结构域由约76个氨基酸残基组成ꎬ参与下游基因启动子DNA的GTAC核心基序结合及细胞核定位ꎬ并有两个典型的锌指结构[4]ꎮSBP家族成员于1996年首次在金鱼草(Antirrhinummajus)中鉴定获得ꎬ即AmSBP1与AmSBP2ꎬ且研究发现这两个SBP成员可以直接结合到花分生相关基因SQUA ̄MOSA的启动子上调控基因表达[3]ꎮ其后在多个物种中鉴定到SBP家族成员ꎬ如蓝莓(Vacciniumspp.)㊁甜根子草(SaccharumspontaneumL.)㊁黑胡椒(PipernigrumL.)㊁拟南芥(Arabidopsisthali ̄ana)及水稻(Oryzasativa)等ꎬ其功能涉及到植物信号转导㊁防御响应㊁花和果实发育及相变过程等[4-8]ꎮ如在模式植物拟南芥中共鉴定到16个SBP家族成员ꎬ其中ꎬSPL3㊁SPL4和SPL5是开花调控基因LEAFY㊁FRUITFUL和APETALA1的上游调控子ꎬ三者功能冗余ꎬ共同调控拟南芥的花发育过程[9]ꎻSPL9和SPL15是植物间隔期和分枝的调控子[10]ꎮ在黑胡椒中共鉴定到34个SBP成员ꎬ其中17个成员上发现有MiR156的结合位点[7]ꎮ水稻中共鉴定到19个SBP成员ꎬ其中SPL14控制着水稻分蘖ꎬSPL16可以调控水稻籽粒性状和质量[5ꎬ11-12]ꎮ高粱(SorghumbicolorL.)是禾本科高粱属一年生草本植物ꎬ是世界第五大禾谷类作物ꎬ同时也是极为重要的旱粮作物[13]ꎬ被广泛应用于酿酒㊁食用㊁饲料㊁制糖及淀粉制造等方面ꎬ具有很高的应用价值[14]ꎮ2009年高粱全基因组测序完成[15]ꎬ推动了其生长发育调控关键基因家族鉴定工作的开展[16-20]ꎮ另外ꎬ全球气候的极端变化也使得生物基因家族的鉴定及功能研究变得尤为重要ꎮ高粱具有较强的抗旱性ꎬ挖掘其抗旱基因对加快其抗旱育种进程㊁提高产量具有重要意义[21]ꎮ本研究对高粱SBP基因家族进行鉴定ꎬ并基于基本理化性质分析㊁系统发育分析㊁蛋白一级结构及启动子基序预测等对其进行生物信息学分析ꎬ同时利用qRT-PCR分析其在高粱不同组织中及在干旱胁迫不同时间下的表达模式ꎬ以期为后续深入研究SBP转录因子家族在高粱生长发育过程中的功能并筛选出优良基因奠定基础ꎮ1㊀材料与方法1.1㊀样品处理受试高粱种子购自红缨子农业科技发展有限公司ꎮ2023年10月将种子播于营养丰富的土壤基质中进行光照恒温培养ꎬ温度为23ħꎮ待幼苗长出4~5片叶时进行干旱胁迫处理ꎬ即将幼苗根部直接浸在10%的PEG-6000溶液中ꎬ分别于处理0㊁4㊁12㊁18㊁24㊁36h采集样品ꎬ并置于液氮中速冻后保存于-80ħ冰箱ꎮ1.2㊀数据来源拟南芥的SBP基因下载于TAIR(https://www.arabidopsis.org/)ꎬ水稻和高粱的SBP家族成员从PlantTFDB(http://planttfdb.cbi.pku.edu.cn/)网站获得ꎮ高粱基因组㊁mRNA㊁蛋白组及相应的GFF文件来自于Phytozome(http://phyto ̄zome.jgi.doe.gov/pz/portal.html)数据库ꎮ1.3㊀试验方法1.3.1㊀高粱SBP家族成员鉴定㊀通过NCBI(ht ̄2山东农业科学㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀第56卷㊀tps://www.ncbi.nlm.nih.gov/)的Blast功能ꎬ利用拟南芥16个SBP成员的氨基酸序列在高粱蛋白组中进行序列预测(Evalueɤ10-5)ꎬ获得SBP候选蛋白序列ꎬ再通过NCBI的保守结构域分析功能及pfam(http://pfam.xfam.org/)网站检测序列中是否存在SBP结构域ꎮ通过TBtools软件提取高粱SBP成员的CDS㊁启动子序列ꎬ方便后续分析ꎮ1.3.2㊀SBP家族系统发育分析㊀获得拟南芥㊁水稻㊁玉米及高粱的SBP氨基酸序列后ꎬ利用MEGA软件使用邻接法(Neighbor-Joining)构建系统发育树ꎬBootstrap设置为1000ꎬ构建好的系统发育树使用Figtree软件进行优化ꎮ另用高粱的19个SBP家族成员构建系统发育树ꎬBootstrap设置为1000ꎮ1.3.3㊀高粱SBP基因染色体定位㊀使用TBtools工具箱中的GeneLocationVisualizefromGFT/GFF工具ꎬ按照操作指示ꎬ分别导入高粱基因组的GFF文件和SBP基因的ID信息ꎬ获得染色体定位图片ꎬ并用Photoshop软件进行美化调整ꎮ1.3.4㊀高粱SBP成员的理化性质分析㊀在Prot ̄Param(http://us.expasy.org/tools/protparam.html)网站上对高粱SBP成员的氨基酸序列长度㊁等电点㊁带负电荷残基数㊁带正电荷残基数㊁不稳定指数㊁脂肪族氨基酸指数和平均疏水性进行预测ꎬ并利用PSORT(http://psort.hgc.jp/)对其进行亚细胞定位预测ꎮ1.3.5㊀高粱SBP基因启动子顺式作用元件分析㊀利用TBtools提取SBP基因的2000bp启动子序列ꎬ然后以fasta格式导入Plantcare(http://bioinformatics.psb.ugent.be/webtools/plantcare/ht ̄ml/)网站分析潜在的顺式作用元件ꎬ并统计各基序在SBP启动子上的分布ꎮ1.3.6㊀SBP基因家族共线性分析㊀利用TBtools软件分析高粱与拟南芥SBP基因在染色体上的分布和共线性关系并作图ꎬ使用AdobeIllustratorCS5软件对图片进行优化ꎮ1.3.7㊀总RNA的提取和实时荧光定量PCR(qRT-PCR)分析㊀用TIANGENRNAEasyFast植物组织RNA快速提取试剂盒提取总RNAꎬ用TIAN ̄GENFastKingcDNA第一链合成试剂盒反转录为cDNAꎬ-20ħ保存备用ꎮ用Premier5.0设计qRT-PCR引物(表1)ꎬ以SbACTIN为内参基因ꎮPCR反应体系10μL:2ˑSYBRGreen5μLꎬ上㊁下游引物各0.2μLꎬcDNA0.5μLꎬddH2O4.1μLꎮqRT-PCR程序:95ħ30sꎻ95ħ5sꎬ58ħ25sꎬ72ħ18sꎬ循环40次ꎻ95ħ5sꎬ65ħ1minꎮ用2-ΔΔCt法进行基因表达量计算[22]ꎮ表1㊀qRT-PCR引物基因上游引物(5ᶄ-3ᶄ)下游引物(5ᶄ-3ᶄ)SbSBP1TTGATAAAGTTGGCTAAAGACGCAGTGGGGAGATGATAGATGGGSbSBP2GCCTATGTTCCAGTTGTAAGACGGTGCTGCAAGATTCGATGTGSbSBP3GCGTTGCGGAGGTGGTATACGTCAAAATGGACTCGGGATCSbSBP4AAGGGCTGATTTATCCGTTCGCCACTATGTTTGGTTTTGTTTGGSbSBP5AATGCTGTCGCAGCTCCTTCAACCAAATCCTCCGCCTASbSBP6CAAAGACACCCCGCTACCCGCAACCTTTCCATCCCTGACSbSBP7TCAAAAGCGAGGGAGACGAAATAAACAGACAAGTGGGGAGGSbSBP8CGTAATCGGTTATGGTTAAGACAGCCAGCAAAGTCCGTTGTGSbSBP9ATAAACCGGATAAAAGGAAGCAGGAACTTGGAAACCCGATGASbSBP10TCGGCTGATACCATTGAAAGACAACGACGGTCGAAACCTTACSbSBP12CTGGAAGAAACCAATCCTCAACGCAGCAAAGAACATCATCAATCSbSBP13TCTGCATTGTGAGTGCCTTTAGCAACCATTATGACTTCGTTTGGSbSBP14GCCAAAGAAAAGCAAAGCACAACACCAGCATGGAACCCTSbSBP15GGCGGCAAAATGAGACTGGGCATGGACAATAGGCTGGAACTSbSBP17CAGGAGCGTGGTACTTGATGCGGTTGCGGTTGTGGTGGATSbSBP18TCGACTACTCCGTCCCAAACAAGCCGTGCTCGTATCCTGSbSBP19CTGTCCGATACTCTGAGCGATTTCCACCCCGTGACAACCAAASbACTINAAGTGCGACGTGGATATTAGGATCTTGGGCGGAAAGAATTAGA3㊀第3期㊀㊀㊀㊀㊀姜昱雯ꎬ等:高粱SBP基因家族鉴定及干旱胁迫下的表达模式分析2㊀结果与分析2.1㊀高粱SBP家族成员的鉴定利用已发表的16个拟南芥SBP成员的氨基酸序列在Phytozome网站进行BLASTP序列比对ꎬ共获得21个高粱候选SBP基因ꎮ进一步通过Pfam和NCBI在线网站分析获得基因的蛋白结构域ꎬ去除不含SBP结构域的序列ꎬ最终得到19个高粱SBP家族成员(表2㊁图1)ꎮ理化性质分析(表2)发现ꎬ高粱SBP家族成员的氨基酸序列长度差异较大ꎬ最短的仅218aaꎬ最长的为SORBI_3007G207000ꎬ达1119aaꎮ预测等电点差异也较为明显ꎬ其中酸性蛋白较少ꎬ仅有3条ꎬ碱性蛋白有16条ꎬ表明高粱SBP蛋白多为碱性蛋白ꎮ不稳定指数小于40的蛋白仅有SOR ̄BI_3010G254200ꎬ稳定性较高ꎬ其余蛋白的稳定性均较低ꎮ所有高粱SBP成员的平均疏水性均为负数ꎬ表明SBP蛋白都是亲水蛋白ꎮ亚细胞定位预测结果显示ꎬ除SORBI_3007G207000外ꎬ其余蛋白均定位在细胞核中ꎬ这可能与SBP转录因子行使功能有关ꎮ表2㊀高粱SBP基因家族成员信息基因名序列号氨基酸数等电点带负电荷残基总数带正电荷残基总数不稳定指数脂肪族氨基酸指数平均疏水性亚细胞定位SbSBP1SORBI_3001G0265009695.291259849.8479.29-0.301细胞核SbSBP2SORBI_3003G1399008845.951059257.0979.88-0.359细胞核SbSBP3SORBI_3007G20700011197.5311912056.6876.80-0.450叶绿体SbSBP4SORBI_3002G3123002189.97182860.9854.95-0.519细胞核SbSBP5SORBI_3010G2542003589.74264437.3861.48-0.561细胞核SbSBP6SORBI_3007G1935004568.21404364.6254.69-0.478细胞核SbSBP7SORBI_3004G0589003379.08364456.7356.50-0.713细胞核SbSBP8SORBI_3004G0621004518.01373958.3264.35-0.343细胞核SbSBP9SORBI_3005G1206003258.97232957.4156.95-0.375细胞核SbSBP10SORBI_3006G2477004007.50353549.6156.30-0.699细胞核SbSBP11SORBI_3002G31220024510.48183364.1256.94-0.454细胞核SbSBP12SORBI_3003G4066004059.05263458.1957.23-0.462细胞核SbSBP13SORBI_3010G2157004449.18283867.9353.24-0.666细胞核SbSBP14SORBI_3002G2579004566.80403865.3555.81-0.365细胞核SbSBP15SORBI_3009G1350008645.561179353.5985.93-0.289细胞核SbSBP16SORBI_3004G0369004808.93435150.9455.60-0.598细胞核SbSBP17SORBI_3002G2478003888.93273451.5152.42-0.511细胞核SbSBP18SORBI_3006G1710004159.44284254.7960.29-0.379细胞核SbSBP19SORBI_3007G2102002809.00222952.4652.89-0.741细胞核㊀㊀蛋白结构域分析结果(图1)表明ꎬ所有高粱SBP成员的氨基酸序列上均含有典型的SBP结构域ꎬ包括两个锌指结构和核定位信号ꎮ此外ꎬSORBI_3001G026500㊁SORBI_3003G139900㊁SOR ̄BI_3007G207000及SORBI_3009G135000的氨基酸序列羧基端包含一个Ank_2superfamily结构域ꎬ暗示着他们可能有一些共同的特殊功能ꎮ2.2㊀高粱SBP家族系统发育分析用拟南芥(16个)㊁水稻(19个)㊁玉米(28个)及高粱(19个)的SBP成员氨基酸序列ꎬ通过MEGA进行系统发育分析ꎬ结果将所有SBP大致分为3个亚类(图2)ꎬ这与在水稻中的研究结果类似ꎮ每个亚类均包含水稻㊁拟南芥㊁玉米和高粱的SBP成员ꎬ表明SBP家族的分化在物种分化前即已形成ꎮ从进化枝上看ꎬ相对于拟南芥ꎬ高粱与水稻㊁玉米的SBP家族进化关系更为接近ꎮ高粱的SBP成员也分为3类ꎬ分别含有7㊁5㊁7个SBP成员(图3)ꎮDNA结构分析发现ꎬ在系统发育关系上比较靠近的基因ꎬ其外显子和内含子分布结构更为相似ꎬ如SORBI_3006G247700㊁4山东农业科学㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀第56卷㊀SORBI_3004G062100和SORBI_3010G215700均包含3个外显子和2个内含子ꎻSORBI_3001G026500㊁SORBI_3003G139900和SORBI_3007G207000分别含有10㊁10个和9个内含子ꎮ图1㊀高粱SBP家族成员蛋白结构分析2.3㊀高粱SBP家族成员的染色体定位分析对高粱SBP基因的染色体定位分析发现ꎬ除第8条染色体外ꎬ其余9条染色体上均分布有SBP基因(图4)ꎮ其中ꎬ2号染色体上分布有4个SBP基因ꎬ数量最多ꎬ且表现出聚类分布的情况ꎬ两两聚在一起ꎻ4号和7号染色体上均分布有3个SBP基因ꎬ3㊁6㊁10号染色体上均有2个SBP基因ꎬ1㊁5㊁9号染色体上均仅有一个SBP基因ꎮ对高粱染色体的基因密度及其与拟南芥的共线性分析结果(图5)显示ꎬ高粱与拟南芥间存在61对共线性关系ꎬ并且高粱的SBP基因在染色体上主要位于基因高密度区域ꎬ表明SBP基因在高粱染色体上呈热点区域分布ꎮ2.4㊀高粱SBP家族基因的启动子基序分析为了解高粱SBP基因可能参与的生物学过程ꎬ对19个成员转录起始位点上游2000bp的启动子区域进行顺式作用元件预测ꎬ结果(表3)显示ꎬ高粱SBP基因启动子区拥有丰富的顺式作用元件ꎬ主要包含有脱落酸响应元件㊁厌氧诱导响应元件㊁茉莉酸甲酯响应元件㊁光响应元件㊁低温响应元件㊁MYB转录因子结合位点及昼夜节律调控位点等ꎮ这些顺式作用元件的存在暗示着SBP基因广泛参与了高粱的生物学过程ꎮ如除SORBI_3004G036900和SORBI_3002G312200外ꎬ其余17个成员启动子上均包含有脱落酸响应的ABRE结合基序ꎬ暗示着高粱SBP家族可能普遍参与了其干旱响应过程ꎻ茉莉酸甲酯响应元件TGACG在SORBI_3001G026500等15个成员启动子上均有分布ꎬ表明这些基因可能受到茉莉酸甲酯的诱导ꎻ此外ꎬ光响应元件G-box㊁MyB转录因子结合位点等在高粱SBP基因启动子上的分布也较为普遍ꎮ推测高粱SBP家族可能参与了植物响应干旱㊁光和低温等多种生物学过程ꎮ5㊀第3期㊀㊀㊀㊀㊀姜昱雯ꎬ等:高粱SBP基因家族鉴定及干旱胁迫下的表达模式分析序列号前端为 GRMZM2G 代表玉米ꎬ LOC_Os 代表水稻ꎬ AT 代表拟南芥ꎬ SORBI 代表高粱ꎮ图2㊀拟南芥㊁水稻㊁玉米和高粱SBP家族成员系统发育分析图3㊀高粱SBP家族系统发育和基因结构分析6山东农业科学㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀第56卷㊀图4㊀高粱SBP基因的染色体定位红色标记的Chr.1 Chr.5是拟南芥的5条染色体ꎬ黑色的Chr.1 Chr.10是高粱的10条染色体ꎬ0~80的数字表示基因长度ꎻ黑色的线表示拟南芥和高粱SBP间的共线性关系ꎮ图5㊀高粱与拟南芥SBP基因共线性Circos图2.5㊀高粱SBP家族基因的表达模式分析为进一步研究高粱SBP基因的潜在功能ꎬ对19个SBP基因在高粱幼苗全株㊁叶㊁根㊁茎和花/种子中的表达模式进行了分析ꎮ通过Phytozome7㊀第3期㊀㊀㊀㊀㊀姜昱雯ꎬ等:高粱SBP基因家族鉴定及干旱胁迫下的表达模式分析表3㊀高粱SBP家族基因启动子顺式作用元件顺式作用元件序列功能MYBTAACCAMYB转录因子结合位点G-BoxCACGTT光响应TATA-boxTATA转录起始位点附近-30bp的核心启动子CAAT-boxCAAAT启动子与增强子共有ABREACGTGꎬGCCGCGTGGC脱落酸响应AREAAACCA厌氧诱导必需CGTCA-motifCGTCAꎬCAAT茉莉酸甲酯响应circadianCAAAGATATC昼夜节律调控RY-elementCATGCATG种子特异性调控Sp1GGGCGG光响应LTRCCGAAA低温响应O2-siteGATGATGTGG玉米蛋白代谢调控MBSCAACTG干旱诱导的MYB结合位点网站获得各个成员的组织表达量ꎬ并利用TBtools软件构建组织表达热图ꎮ结果(图6A)发现ꎬ各基因在高粱幼苗不同组织中的表达量存在明显差异ꎬ其中SORBI_3003G139900㊁SORBI_3007G207000㊁SORBI_3001G026500和SORBI_3009G135000在各组织中的表达量均最高ꎬ而SORBI_3006G171000㊁SORBI_3004G062100和SORBI_3010G215700在各组织中的表达丰度均较低ꎮ其中ꎬSORBI_3003G139900㊁SORBI_3007G207000㊁SORBI_3001G026500等在茎中的表达量最高ꎬSORBI_3007G193500在花/种子中的表达量最高ꎬSORBI_3002G312200和SORBI_3002G312300在根中的表达量最低ꎮ表明这些SBP基因在高粱生长发育过程中发挥的功能具有差异性ꎮ此外ꎬ本研究选取启动子区含有脱落酸响应元件ABRE的17个SBP基因检测其在干旱胁迫下的表达模式ꎬ结果(图6B)发现ꎬ这17个基因在干旱处理一段时间后ꎬ均呈现出表达量上调的趋势ꎬ暗示SBP家族基因参与高粱响应干旱胁迫的积极作用ꎮ其中SORBI_3001G026500㊁SORBI_3003G139900㊁SORBI_3004G058900㊁SORBI_3004G062100㊁SORBI_3005G120600㊁SORBI_3006G24770和SORBI_3009G135000均在干旱处理24h表达量最高ꎬ而SORBI_3010G254200㊁SORBI_3010G215700㊁SORBI_3002G247800㊁SOR ̄BI_3006G171000和SORBI_3007G210200在干旱处理36h表达量才达到最高ꎮ此外ꎬSORBI_3010G254200和SORBI_3007G193500在干旱诱导初期先表现出抑制下调ꎬ随后才逐渐上调表达ꎮ可见ꎬ高粱的SBP家族成员随干旱胁迫时间的延长呈现出差异化的表达模式ꎬ暗示着其在响应干旱胁迫时可能有着不同的功能ꎮA.高粱SBP家族成员组织表达模式ꎻB.模拟干旱处理下17个SBP基因的表达模式ꎮ图6㊀高粱SBP家族基因的组织表达特异性及干旱胁迫下的表达模式8山东农业科学㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀第56卷㊀3㊀讨论与结论作为植物特有的转录因子家族ꎬ当前的研究认为SBP家族参与了植物体内多种生物学过程ꎬ不仅参与了植物的生长发育调控ꎬ而且在植物响应逆境胁迫过程中发挥着重要作用[3ꎬ5ꎬ9ꎬ12ꎬ23]ꎮ因此ꎬ开发植物SBP家族基因并进行功能研究具有重要意义ꎮ不同物种的SBP基因数量存在显著差异ꎬ如矮牵牛(Petuniaaxillaris)中有21个SBP基因[24]ꎬ梅花(Prunusmume)中有15个SBP基因[25]ꎬ白梨(Pyrusbretschneideri)中有32个SBP基因[26]ꎮ本研究从高粱中鉴定出19个SBP家族基因ꎬ与水稻的SBP基因数量相当ꎬ多于拟南芥ꎬ少于玉米[5]ꎮSBP基因数量的变化可能与物种的进化历史及环境选择相关ꎮ本研究结果表明ꎬ高粱SBP基因的氨基酸序列均包含有典型且高度保守的SBP结构域ꎬ与在其他物种中的结果一致[5ꎬ25]ꎻ部分基因的氨基酸序列羧基端包含一个Ank_2superfamily结构域ꎬ该结构域为锚蛋白重复序列ꎬ但其在SBP行使功能过程中发挥的作用还未知ꎮ系统发育分析发现ꎬ高粱㊁拟南芥㊁水稻㊁玉米的SBP家族成员可分为3个亚枝ꎬ并且每个亚枝中均含有这4个物种的SBP成员ꎬ表明SBP家族在单子叶和双子叶植物分化前即已扩张为3个亚枝ꎬ这与之前的研究结果[5]一致ꎮ此外ꎬ高粱与水稻㊁玉米的SBP基因具有更近的进化关系ꎬ且同一进化枝的SBP直系同源基因可能具有类似功能ꎮ从系统发育树还可看出ꎬ拟南芥的SBP家族拥有更多的旁系同源基因对ꎬ如AT1G20980.1与AT2G47070.1ꎬAT1G53160.1与AT3G15270.1等ꎬ表明在单双子叶物种分化后ꎬ高粱与拟南芥SBP基因经历了不同的物种特异性基因复制或丢失事件ꎮ有报道表明ꎬSBP基因在植物响应非生物胁迫如干旱㊁盐胁迫等过程中发挥重要功能ꎮ如白桦(BetulaplatyphyllaSuk.)SBP家族成员SPL9会受到盐和PEG-6000的诱导表达ꎬ拟南芥异源转基因实验发现SPL9通过清除活性氧提高植物对盐旱的耐受性[27]ꎻ中国野生葡萄(Vitis)的SBP16在拟南芥中表达后通过调控SOS和ROS信号通路提高植株的干旱和盐抗性[28]ꎻ水稻miR156k通过降低靶基因SPL3㊁SPL14和SPL17的表达减弱其抗冷性[29]ꎻ玉米的部分SBP家族基因会受到干旱㊁冷㊁盐等的诱导表达[30]ꎮ本研究发现ꎬ高粱19个SBP成员中有17个含有ABRE脱落酸响应元件ꎬ进一步的qRT-PCR检测显示ꎬ这17个基因受到干旱胁迫的诱导表达ꎬ但随胁迫时间延长的表达模式存在差异ꎬ结合这些基因在高粱不同组织中的差异化表达ꎬ推测它们可能在高粱响应干旱过程中发挥着不同的功能ꎬ这为后续探究SBP基因在高粱生长发育及非生物胁迫响应方面的作用机制奠定了一定的理论基础ꎮ参㊀考㊀文㊀献:[1]㊀Franco ̄ZorrillaJMꎬSolanoR.Identificationofplanttranscrip ̄tionfactortargetsequences[J].BiochimicaetBiophysicaActa-GeneRegulatoryMechnismsꎬ2017ꎬ1860(1):21-30. [2]㊀StraderLꎬWeijersDꎬWagnerD.Planttranscriptionfactors ̄beingintherightplacewiththerightcompany[J].CurrentO ̄pinioninPlantBiologyꎬ2022ꎬ65:102136.[3]㊀KleinJꎬSaedlerHꎬHuijserP.AnewfamilyofDNAbindingproteinsincludesputativetranscriptionalregulatorsoftheAntir ̄rhinummajusfloralmeristemidentitygeneSQUAMOSA[J].MolecularGeneralandGeneticsꎬ1996ꎬ250(1):7-16. [4]㊀YamasakiKꎬKigawaTꎬInoueMꎬetal.Anovelzinc ̄bindingmotifrevealedbysolutionstructuresofDNA ̄bindingdomainsofArabidopsisSBP ̄familytranscriptionfactors[J].JournalofMo ̄lecularBiologyꎬ2004ꎬ337(1):49-63.[5]㊀YangZFꎬWangXFꎬGuSLꎬetal.ComparativestudyofSBP ̄boxgenefamilyinArabidopsisandrice[J].Geneꎬ2008ꎬ407(1/2):1-11.[6]㊀XieXꎬYueSKꎬShiBSꎬetal.ComprehensiveanalysisoftheSBPfamilyinblueberryandtheirregulatorymechanismcon ̄trollingchlorophyllaccumulation[J].FrontiersinPlantSci ̄enceꎬ2021ꎬ12:703994.[7]㊀LiJꎬFanRꎬWuBDꎬetal.Genome ̄wideidentificationandfunctionalexplorationofSBP ̄Boxgenefamilyinblackpepper(PipernigrumL.)[J].Genesꎬ2021ꎬ12(11):1740. [8]㊀LiuYHꎬAslamMꎬYaoLAꎬetal.GenomicanalysisofSBPgenefamilyinSaccharumspontaneumrevealstheirassociationwithvegetativeandreproductivedevelopment[J].BMCGe ̄nomicsꎬ2021ꎬ22(1):767.[9]㊀YamaguchiAꎬWuMFꎬYangLꎬetal.ThemicroRNA ̄regula ̄9㊀第3期㊀㊀㊀㊀㊀姜昱雯ꎬ等:高粱SBP基因家族鉴定及干旱胁迫下的表达模式分析tedSBP ̄BoxtranscriptionfactorSPL3isadirectupstreamacti ̄vatorofLEAFYꎬFRUITFULLꎬandAPETALA1[J].Develop ̄mentalCellꎬ2009ꎬ17(2):268-278.[10]SchwarzSꎬGrandeAVꎬBujdosoNꎬetal.ThemicroRNAreg ̄ulatedSBP ̄boxgenesSPL9andSPL15controlshootmaturationinArabidopsis[J].PlantMolecularBiologyꎬ2008ꎬ67(1/2):183-195.[11]LuoLꎬLiWQꎬMiuraKꎬetal.ControloftillergrowthofricebyOsSPL14andStrigolactonesꎬwhichworkintwoindependentpathways[J].PlantandCellPhysiologyꎬ2012ꎬ53(10):1793-1801.[12]WangSKꎬWuKꎬYuanQBꎬetal.ControlofgrainsizeꎬshapeandqualitybyOsSPL16inrice[J].NatureGeneticsꎬ2012ꎬ44(8):950-954.[13]姚茂星ꎬ周光怡ꎬ丁延庆ꎬ等.高粱GATA转录因子家族的鉴定和表达模式分析[J].分子植物育种ꎬ2022ꎬ20(10):3178-3187.[14]马建华ꎬ杨艳君ꎬ赵红梅ꎬ等.高粱低氮胁迫相关novel ̄miRNA的鉴定及其靶基因预测[J/OL].应用与环境生物学报ꎬ2023(2023-09-22).https://doi.org/10.19675/j.cnki.1006 ̄687x.2023.05010.[15]宋迎辉ꎬ朱灿灿ꎬ代书桃ꎬ等.高粱GATA基因家族的全基因组鉴定及表达分析[J].山东农业科学ꎬ2022ꎬ54(8):14-23. [16]TaoYFꎬLuoHꎬXuJBꎬetal.Extensivevariationwithinthepan ̄genomeofcultivatedandwildSorghum[J].NaturePlantsꎬ2021ꎬ7(6):766-773.[17]常建忠ꎬ闫凤霞ꎬ乔麟轶ꎬ等.高粱SBP ̄box基因家族全基因组鉴定及表达分析[J].遗传ꎬ2016ꎬ38(6):569-580. [18]郑玲ꎬ白雪婷ꎬ李会云.高粱TCP基因家族全基因组鉴定及表达分析[J].河南农业科学ꎬ2019ꎬ48(10):30-36. [19]赵兴奎ꎬ范昕琦ꎬ聂萌恩ꎬ等.高粱WRYK家族成员鉴定及生物信息学分析[J].分子植物育种ꎬ2020ꎬ18(13):4170-4181.[20]刘梦ꎬ魏玉磊ꎬ丁冬ꎬ等.高粱LOX基因家族全基因组鉴定及表达模式分析[J].河南农业科学ꎬ2020ꎬ49(11):37-44.[21]王晓东ꎬ李俊志ꎬ窦爽ꎬ等.高粱抗旱性研究进展[J].山东农业科学ꎬ2024ꎬ56(1):164-173.[22]LivakKJꎬSchmittgenTD.Analysisofrelativegeneexpressiondatausingreal ̄timequantitativePCRandthe2-ΔΔCTmethod[J].Methodsꎬ2001ꎬ25(4):402-408.[23]王春昱ꎬ范付华.马尾松SPL基因家族鉴定及其响应低磷胁迫的表达分析[J].农业生物技术学报ꎬ2023ꎬ31(3):509-517.[24]ZhouQꎬZhangSSꎬChenFꎬetal.Genome ̄wideidentificationandcharacterizationoftheSBP ̄boxgenefamilyinPetunia[J].BMCGenomicsꎬ2018ꎬ19(1):193.[25]XuZDꎬSunLDꎬZhouYZꎬetal.Identificationandexpres ̄sionanalysisoftheSQUAMOSApromoter ̄bindingprotein(SBP) ̄boxgenefamilyinPrunusmume[J].MolecularGenet ̄icsandGenomicsꎬ2015ꎬ290:1701-1715.[26]AbdullahMꎬCaoYPꎬChengXꎬetal.Genome ̄wideanalysischaracterizationandevolutionofSBPgenesinFragariavescaꎬPyrusbretschneideriꎬPrunuspersicaandPrunusmume[J].FrontiersinGeneticsꎬ2018ꎬ9:64.[27]NingKꎬChenSꎬHuangHJꎬetal.MolecularcharacterizationandexpressionanalysisoftheSPLgenefamilywithBpSPL9transgeniclinesfoundtoconfertolerancetoabioticstressinBetulaplatyphyllaSuk[J].PlantCellTissueandOrganCul ̄tureꎬ2017ꎬ130:469-481.[28]HouHMꎬJiaHꎬYanQꎬetal.OverexpressionofaSBP ̄boxgene(VpSBP16)fromChinesewildVitisspeciesinArabidopsisimprovessalinityanddroughtstresstolerance[J].InternationalJournalofMolecularSciencesꎬ2018ꎬ19(4):940.[29]CuiNꎬSunXLꎬSunMZꎬetal.OverexpressionofOs ̄miR156kleadstoreducedtolerancetocoldstressinrice(Ory ̄zasativa)[J].MolecularBreedingꎬ2015ꎬ35:214.[30]MaoHDꎬYuLJꎬLiZJꎬetal.Genome ̄wideanalysisoftheSPLfamilytranscriptionfactorsandtheirresponsestoabioticstressesinmaize[J].PlantGeneꎬ2016ꎬ6:1-12.01山东农业科学㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀㊀第56卷㊀。
毛竹IDD基因家族启动子分析

毛竹IDD基因家族启动子分析陈虹君;高臻毅;郭小勤【摘要】IDD gene family, which encode hybrid transcription factor, are mostly involved in regulating plant growth and develop-ment. To elucidate the function of upstream cis-elements of PhIDD gene family, upstream sequences of start codons of 8 IDD genes ( PhIDD1-2, PhIDD4-8 and PhID1) at the length of 2 000 bp in Phyllostachys edulis were obtained and analyzed by bioinformatics methods. Results indicated that there were TATA box and CAAT box in all promoter sequences. Various conservative elements exis-ted in the promoter sequence, including light responsive element, phytohormone responsive element, stress responsive element, etc. Some cis-elements were specific to certain genes, suggesting that each gene in this family may show distinctive expression pattern and play different roles in plant growth and development.%IDD基因家族编码一种混合型的转录因子,多数参与植物的生长发育。
大白菜两个APXl基因的鉴定

大白菜两个APXl基因的鉴定作者:李利斌徐寿春刘立峰李化银陈伟高建伟来源:《山东农业科学》2011年第01期摘要:利用比较基因组学的方法,从大白菜基因组中鉴定出两个与拟南芥的耐热关键基因APXl同源的基因,并对这两个基因的结构、遗传进化、顺式元件进行了分析,为下一步研究这两个基因的功能和利用这两个基因进行大白菜耐热分子育种奠定了基础。
关键词:大白菜;APXl;基因结构;遗传进化;顺式元件中图分类号:Q781文献标识号:A文章编号:1001-4942(2011)01-0009-03大白菜性喜冷凉,主要栽培于温带地区,是我国的重要蔬菜之一。
生产上随着春夏季节大白菜种植面积的日益扩大,对品种的耐热性提出了较高的要求。
目前,国内外学者对植物耐热遗传机制的研究取得了很大进展,克隆了一大批与植物耐热相关的基因。
有关白菜类蔬菜耐热的生理机制、耐热性指标的筛选、耐热QTL定位和耐热品种的选育研究得也比较多,但是有关耐热分子机理的研究目前报道很少。
苏小俊、杨妍妍等对白菜类蔬菜的耐热鉴定方法、耐热生理指标进行了探讨;于拴仓等鉴定出5个大白菜耐热QTL,并对它们的遗传效应进行了分析;Yang等克隆了与大白菜耐热相关的细胞壁基因,郑佳秋和朱波分别克隆了一个不结球白菜的热激蛋白BcHSP基因及NAC基因。
鉴于大白菜耐热遗传研究的现状,为了更好地为大白菜耐热育种服务,有必要加强大白菜耐热分子遗传机制的研究,鉴定和克隆大白菜的耐热响应关键基因。
现有研究表明,植物热胁迫过程中会产生大量的活性氧物质,对植物细胞产生毒害作用。
植物清除这些有害物质的能力与耐热性存在密切的关系。
拟南芥的抗坏血酸还原酶基因APXl 在植物抗氧化和热、干旱等多种逆境胁迫响应过程中具有重要功能。
但是有关大白菜APXl基因的研究还未见报道。
本文通过比较基因组学的方法,从大白菜的基因组中鉴定出两个与拟南芥的APXl同源的基因,并对它们的结构特征、进化关系以及启动子上游序列中的顺式元件进行了分析,为进一步研究大白菜APXl基因的功能和利用此类基因进行大白菜耐热分子育种提供借鉴。
茉莉酸的生物合成

Disease development and jasmonate-induced resistance in mutant plants inoculated with Pythium mastophorum: (A) Wild-type and fad3–2 fad7– 2 fad8 plants were grown on pasteurized soil for 15 days and then transferred to pots containing soil infested with P.mastophorum. Starting 2 days before transfer, the roots of each plant were drenched each day with 1 ml of methyl jasmonate solution or an equal volume of water.
O
COOH
dnOPDA
十六碳脂肪酸的JA合成 途径与十八碳脂肪酸 的途径平行进行,只 是dnOPDA经过2次b-氧 化形成JA。
(过氧化物酶体)
OPDA还原酶
3×β-氧化
OPC-8:0辅 酶A连接酶
·叶绿体中的OPDA经转运蛋白PXA1转运到过氧化物酶体中。
·OPDA在OPDA还原酶(OPR)催化下生成OPC-8:0。
infection by Pythium mastophorum. Wild-type (Left) and fad3–2 fad7–2
fad8 plants (Right) were grown axenically on agar medium for 20 days
before being inoculated with P. mastophorum. (A) Mock inoculation
延胡索去甲乌药碱合成酶基因CyNCS1克隆及其表达模式分析

延胡索去甲乌药碱合成酶基因CyNCS1克隆及其表达模式分析冯飞雪;吕瑞华;李依民;高静;王凯;周嘉迪;颜永刚;张岗【期刊名称】《西北农林科技大学学报(自然科学版)》【年(卷),期】2024(52)3【摘要】【目的】克隆延胡索去甲乌药碱合成酶(norcoclaurine synthase, NCS)基因,分析其在延胡索异喹啉类生物碱合成中的关键作用。
【方法】基于转录组数据库,利用逆转录PCR克隆延胡索CyNCS1基因及其启动子区域序列,采用生物信息学方法分析其编码蛋白的理化性质、结构特征及其启动子区域顺式作用元件,同时进行多序列比对和系统进化树分析,确定其进化关系;利用实时荧光定量PCR对其根、茎、叶及发育早、中、晚期块茎的组织表达模式进行分析;对茉莉酸甲酯(MeJA)、脱落酸(ABA)处理0,4,8,12 h的叶片进行表达谱分析,以体积分数75%酒精溶剂处理为对照(CK),确定该基因的表达特征。
【结果】CyNCS1基因开放读码框长873 bp,编码291个氨基酸,编码蛋白分子量为33.09 ku,等电点为6.90,具有去甲乌药碱合成酶保守的催化结构域(GDGGVGTV/IL、YKEKF和MIEGGHLDMG),且不含信号肽,预测定位于细胞核中。
多序列比对结果显示,CyNCS1与石生黄堇、罂粟的NCS蛋白同源性较高,分别为83.6%和82.1%;系统进化树结果显示,CyNCS1与石生黄堇CsNCS蛋白聚为一支,说明二者亲缘关系较近。
该基因启动子区长2 238 bp,包含多种植物激素及低温、热胁迫等环境因子的顺式作用元件。
实时荧光定量PCR结果显示,随着发育推进块茎中CyNCS1的表达量显著增高,且受到MeJA和ABA的诱导,MeJA处理8 h时表达量为0 h的3.10倍,ABA处理8 h为0 h的3.16倍;而对照CyNCS1表达量随时间推移无明显变化。
【结论】克隆并鉴定了延胡索NCS酶基因CyNCS1,明确了其在发育进程中的表达模式,并确定该基因表达受MeJA和ABA的诱导。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
参与茉莉酸甲酯反应的顺式作用元件化学反应是物理世界中最神奇的过程之一,而各种反应中,茉莉酸
甲酯反应也是一个备受关注的研究方向。
这种反应能够产生医学上非
常重要的分子——环氧胆固醇,因此在化学领域中,关于此反应的研
究也相当深入。
而在这个反应中,顺式作用元件则具有至关重要的作用。
顺式作用原件是一类拥有特定功能、结构完美、信号传导高效的分子,它们一般来自生物大分子如蛋白质、DNA/RNA等分子,起着完成生物学功能的重要作用。
对于化学反应中茉莉酸甲酯反应而言,顺式作用
元件也是必不可少的。
顺式作用元件可以指导反应的定向和速率,从而产生效果更好的化学
反应。
在茉莉酸甲酯反应中,顺式作用元件往往是通过结构上的空间
匹配来诱导反应发生的。
在这种反应过程中,具备非常重要的两个特性:一是能够控制反应的位置,二是能够控制作用力度。
这两点特性
对于茉莉酸甲酯反应来说,尤其是关键作用的地方。
同时,茉莉酸甲酯反应出现了反应选择性问题,通过引入顺式作用元
件可以调控反应的方向性,提高反应的选择性,从而对反应中的混杂
物进行有效控制。
因此,在茉莉酸甲酯反应中,通过含有顺式作用元
件的催化剂来控制反应是非常重要的一步。
在茉莉酸甲酯反应中,许多化学家研究了各种不同的顺式作用元件,
并发现在这个过程中它们具有不同的作用。
例如,如果带有大环结构
的顺式作用元件,通常会促进茉莉酸甲酯反应中的立体控制。
而许多
重要的寡聚葡萄糖酶也被发现可以与甲酯结合,进一步促进该反应的
发生。
值得注意的是,虽然使用顺式作用元件可以提高化学反应的效率和选
择性,但它们的工作原理是非常复杂的。
其中包括各种化学和物理特
性的协作,例如极性、疏水特性等等。
因此,为了能够深入了解茉莉
酸甲酯反应中的顺式作用元件,研究人员必须在这些分子内部的结构
和分子间相互作用方面进行深入的研究。
总而言之,参与茉莉酸甲酯反应的顺式作用元件对化学反应的定向和
速率起着至关重要的作用。
通过引入这些元件,可以调控茉莉酸甲酯
反应中的反应选择性和方向性,从而有效地控制该反应的发生。
因此,对于研究人员来说,理解和掌握顺式作用元件的工作原理,对于提高
反应效率和选择性,有着非常重要的意义。
