celref软件基本操作
直读光谱仪软件操作规程(3篇)

第1篇直读光谱仪软件操作规程是针对直读光谱仪配套软件的操作指南,旨在指导用户正确、高效地使用软件进行光谱分析。
本规程适用于所有类型的直读光谱仪软件,包括数据采集、处理、分析及报告等功能。
二、软件安装与启动1. 确保计算机操作系统符合软件要求,并安装必要的驱动程序。
2. 将软件安装光盘放入光驱,按照提示完成安装。
3. 双击桌面上的软件图标,或通过开始菜单找到软件并启动。
4. 在软件启动过程中,如需进行系统设置,请按照提示进行操作。
三、软件界面介绍1. 菜单栏:包含文件、编辑、视图、工具、帮助等菜单项,用于执行各种操作。
2. 工具栏:提供常用功能的快捷按钮,如新建、打开、保存、打印等。
3. 工作区:显示当前操作的数据、图表等信息。
4. 状态栏:显示软件状态、当前操作等信息。
四、数据采集与处理1. 连接光谱仪:确保光谱仪与计算机连接正常,打开光谱仪电源。
2. 采集数据:在软件中选择合适的分析方法,设置相关参数,开始采集数据。
3. 数据处理:采集完成后,对数据进行平滑、滤波、提取等处理。
五、光谱分析1. 选择分析方法:根据样品成分和含量要求,选择合适的光谱分析方法。
2. 设置分析参数:根据样品特性,设置分析参数,如积分时间、扫描速度等。
3. 进行分析:点击“分析”按钮,软件将自动进行光谱分析。
4. 查看分析结果:分析完成后,在工作区显示分析结果,包括含量、标准偏差等。
六、报告生成与打印1. 生成报告:点击“报告”菜单,选择报告模板,将分析结果生成报告。
2. 打印报告:将生成的报告导出为PDF或Word格式,进行打印。
七、软件关闭1. 关闭报告:完成报告打印后,关闭报告窗口。
2. 退出软件:点击菜单栏中的“文件”菜单,选择“退出”或点击软件右上角的关闭按钮。
3. 断开光谱仪连接:关闭光谱仪电源,确保光谱仪与计算机连接正常断开。
八、注意事项1. 确保光谱仪与计算机连接正常,避免数据采集失败。
2. 在进行光谱分析时,请仔细设置分析参数,确保分析结果的准确性。
徕卡全站仪数据上传详细介绍

数据上传
1,把所要上传到仪器的数据从Excel上复制到一个新的文本文档(.txt)里面。
可以用逗号隔开。
也可以用制表符等隔开。
2,打开LEICA Flexoffice 软件。
到文件菜单里面新建一项目。
输入项目名。
点击确定。
3,输入ASCII数据
找到要上传的数据,点击输入。
用户自定义如下图操作。
点击下一步,通过选择栏分隔符把点号,x坐标,y坐标,高程分隔开。
点击下一步,右键在0,1,2,3上分别命名。
点击下一步,点击完成。
点击分配,然后点击关闭。
项目数据显示在软件桌面上。
4,输出LandXML文件,并保存到我的电脑盘下面。
5,数据上传。
到“工具”---“数据交换管理器”。
直接把D盘下面的0822.xml文件拉到仪器的“文件-作业”里面,命名,就可以了。
数据上传完毕。
可以在仪器 3 管理里面查看上传的已知点数据。
高效液相软件操作方法

高效液相软件操作方法
以下是高效液相软件操作方法:
1. 创建项目:打开高效液相软件后,选择“新建项目”,输入项目名称和相关信息,创建新的项目。
2. 输入试样信息:在新建项目后,可输入试样信息,包括样品名称、样品编号、分析日期等。
输入完毕后,保存信息。
3. 创建实验方法:在新建项目中,可创建实验方法。
点击“实验方法”选项卡,选择“新建方法”,输入方法名称和相关信息,设置实验条件和参数。
4. 导入样品:在“样品”选项卡中,选择“导入样品”,选择需要分析的样品列表,将样品导入到项目中。
5. 运行实验:选择实验方法后,点击“运行实验”,系统将开始执行实验方法。
实验结果将自动保存。
6. 数据分析:在“分析结果”选项卡中,可查看和分析实验结果。
根据需要选择分析方法,进行数据分析和结果呈现。
7. 数据输出:在数据分析后,可将分析结果输出为PDF、Excel等格式。
选择
“输出数据”选项卡,设置输出格式和参数,将分析结果输出到指定位置。
以上是高效液相软件的基本操作方法,具体操作方法可能因不同厂家软件版本略有不同。
直读光谱仪软件操作规程

直读光谱仪软件操作规程
《直读光谱仪软件操作规程》
一、启动软件
1. 点击光谱仪软件的图标,启动软件。
2. 在软件界面上点击“新建测量”,开始新的测量任务。
二、样品设置
1. 在样品设置界面上,选择待测样品的信息,如样品名称、测量温度等。
2. 确认样品信息无误后,点击“确定”按钮。
三、参数设置
1. 在参数设置界面上,选择光谱测量的相关参数,如波长范围、积分时间等。
2. 设定参数后,点击“确定”按钮。
四、测量样品
1. 将待测样品放置到光谱仪测量台上,并确保样品位置准确。
2. 点击软件界面上的“开始测量”按钮,触发光谱仪进行测量。
五、数据处理
1. 测量完成后,软件将自动生成测量数据并显示在界面上。
2. 可根据需求进行数据处理,如波峰识别、数据平滑等操作。
六、保存数据
1. 确保数据处理完成后,点击“保存”按钮,将测量数据保存到
指定位置。
2. 可选择数据格式并填写相关信息后保存数据。
七、关闭软件
1. 测量完成后,点击软件界面上的“关闭软件”按钮,关闭光谱仪软件。
2. 确认是否保存数据并进行相关操作。
以上就是关于直读光谱仪软件操作规程的简要介绍,希望能对使用者有所帮助。
安捷伦 BioConfirm 软件 6.0 用户手册说明书

两个批次蛋白质色谱图、质谱图和 MS/MS 质谱图的可视化对比BIOCONFIRM 软件 为生物学家实现全面的蛋白质表征安捷伦 MassHunter BioConfirm 的优势BioConfirm 软件结合精确质量 LC/MS 和 LC/MS/MS 数据,可为单克隆抗体等生物药物的表征提供更全面的解决方案。
但 BioConfirm 的最大优势在于其易学易用,使您能够专注于工作而无需在学习软件使用方法上耗费过多时间。
更简便、更快速和更高效的数据处理使用出色的工具进行完整蛋白质以及蛋白质酶解产物的表征。
• 对多肽 MS/MS 质谱图中的子离子进行直接分配,从而完成多肽序列的快速确认• 对比分析模块生成的镜像图有助于对两个批次生物工程制造的蛋白质之间的差异进行鉴定和可视化,从而实现轻松检查• 通过安捷伦数据库的集成轻松鉴定翻译后修饰 (PTM),使您可在蛋白质序列中搜索更多不同的 PTM• 简便易用的质量数差异测量工具 (Delta Mass Ruler) 通过显示质谱峰之间的质量数差异、氨基酸或修饰,实现多肽或蛋白质的即刻表征新型 BioConfirm B.07.00 的特性• 化学数据词典编辑器在您的掌握之下,因此您可以轻松创建反映生物分子特有修饰的谱图• 使用混合匹配修饰 (Mixed Match Modification) 可发现更多完整蛋白质异型体(以及更少假阴性结果)• 通过集成紫外色谱和用户控制的时间设置的最大熵解卷积方法来节省时间并提高效率• 通过免费的序列编辑器及导入 FASTA 和 TXT 序列格式的能力提高分析速度和灵活性安捷伦产品仅用于研究。
不可用于诊断目的。
本文中的信息、说明和性能指标如有变更,恕不另行通知。
© 安捷伦科技(中国)有限公司,20152015 年 2 月 27 日,中国出版5990-7926CHCN新型化学数据词典编辑器使您能快速添加修饰并建立蛋白质(如 IgG mAb )的典型修饰谱图。
实时定量PCR引物和探针设计操作步骤Primer Express软件

实时定量PCR引物和探针设计操作步骤Primer Express软件Primer Express 是实时定量PCR引物和探针设计的专用软件。
遵守以下三个原则有助于快速建立定量PCR反应体系:1.所有扩增按照同样的原则设计 (Primer Express);2.所有PCR反应在ABI PRISM ?7000/7900上使用同样的热循环条件;3.所有反应使用相同的PCR试剂。
引物和探针的设计原则下述原则的重要程度由上往下越来越低,请尽量满足编号靠前的条件。
它们中有的已经在Primer Expre软件中设置成缺省值,有的则需要在选择引物和探针时由设计者加以运用。
如果是设计SYBRGreen 引物,也要选择TaqMan Primer and Probe design并遵守这些规则,但是只需要合成引物就可以了。
TaqMan 探针:1. 保持G-C含量在30-80%之间。
2. 避免同一碱基重复过多。
特别是G,不可超过4个及以上。
3. 5' end不能是G。
4. 尽量使探针中的Cs多于Gs。
如果不能满足,则使用互补链上的探针。
5. 对于单探针反应,用Primer Express?软件计算出来的Tm值应当在68-70 °C 之间。
引物:1. 在探针确定以后再选择引物。
2. 引物要尽可能地接近探针,但是不要重叠。
3. 保持G-C含量在30-80%之间。
4. 避免同一碱基重复过多。
特别是G,不可超过4个及以上。
5. 用Primer Express?软件计算出来的Tm值应当在58-60 °C之间。
6. 3' end 的5个碱基中G and/or C碱基的总数不能超过2个。
实时TaqMan 引物和探针设计Begin by opening Primer Express and selecting "File", "New", and "TaqMan? Primer & Probe Design". The following screen will appear. You can close the TaqMan? Primer & Probe Data box as shown.输入或插入序列Import or paste a sequence into the window (Import shown). To paste a sequence from a Word or text file, first copy it to the clipboard. Be sure to only select the sequence (including numbers or annotations is OK); do not include extraneous information such as accession numbers etc. Next, select "Edit" and "Paste". The sequence will appear in the Sequence screen of Primer Express. Or, to Import a Sequence, click the "Import DNA File" button as shown. The software will then ask you to locate the sequence file. Select it from a folder, hard drive, disk, or desktop. Again, no annotations should be present in this sequence.A file is then imported after selecting the file location.保存输入的序列Select "File" and "Save" to give the sequence a name. This will be displayed in the File Name Box and will save the sequence in the Archive Folder.引物和探针设计参数Click the "Parameters" tab. This displays the Universal default parameters used to search for suitable TaqMan? primer & probe sets for real-time assays. It is strongly recommended that you do not adjust any of the parameters.引物和探针的排序及选择Primer Express is now ready to find Primers and Probes. Click the "Primers" tab, select "Options" and "Find Primers/Probes Now". The software will display the progress in the small window below the sequence.** Please disregard the "Optimal Primer Pairs Only" checkbox and the "Penalty" heading. By checking the Optimal Primer Pairs Only box, you will be severely limiting the range of your search, since the parameters it employs are not based on TaqMan? design guidelines. The Penalty score assigned to your Primer & Probe set is based on factors such as amplicon length. Since the default TaqMan? design parameters keep amplicons under 150 bp, this can be disregarded as well.Primer/probe sets will be listed when the search is complete. Scroll to the right to view the Probes. Click on the "Start" heading under probes to sort probes by sequence. This will group similar probes, simplifying the search.探针的选择Select a probe that is less than 30 bp in length and contains more C's than G's. The probes displayed are on the sense strand only. If the probes displayed do not have more C's than G's, then you will need to use the complement probe (as illustrated in this example). If you need to use the complement, make sure that the probe selected here does not have a C at the 3' end of the probe (otherwise, the complement will have a G at the 5' end ? whichis not allowed).The probe selected meets the first criteria above, but not the second (9 G's, 5 C's). Highlight this probe.Return to the sequence by clicking the "Sequence" tab.Lock in the probe sequence by clicking the Probe Button on the Tool Bar and highlight the probe sequence. The probe will turn green and be displayed in lower case when it is locked.引物选择Find compatible primers by returning to the "Primers" tab, selecting "Options" and "Find Primers & Probes Now". This will find new primer sets that will work with the probe you have selected. You can click on "Start" under Forward Primer to sort the displayed sequences.Search for a primer from the list displayed the meets the following criteria:1.No more than 2 G's and/or C's within the last 5 bases on the 3' end of the primer; and2.No runs of identical nucleotides, especially 4 or more G's.From the list of forward primers displayed, select a primer that has no more than 2 G's and/or C's within the last 5 bases on the 3' end of the primer. Highlight one of the primers that matches this criteria. If no forward primer matches this criteria then select a primer with 3 G's and/or C's. The example shown below matches the criteria and will serve as a suitable forward primer. Once you have selected the appropriate primer click on the "Sequence" tab to return to the Sequence window.Lock the forward primer by clicking the "Forward Primer" button on the toolbar, then highlighting the forward primer sequence. A blue arrow will be displayed under the forward primer showing that it is locked.Click on the "Primers" tab and perform a new search. Scroll to the Reverse Primers displayed and select a reverse primer following the same criteria for forward primer selection (G/C rule on the 3' end of primer).Return to the Sequence page and lock in on the Reverse Primer using the Reverse Primer Tool.This now displays the primers and probe you have selected. Return to the Primers tab and perform one final search to display your results.保存搜索结果Click on "Save List" at the bottom of the screen to save your selection in a tab delimitedformat. Click "Order" to generate an editable/printable text file of your sequences:互补探针的选择In the example above, you must use the complementary probe so as to insure that the probe has more C's than G's. Remember, the probe you use cannot have a G at the 5' end, thus the sense probe used for this search cannot have a C at the 3' end.In order to generate the probe complement, return to the Sequence screen. Highlight the probe sequence, select "Edit", and "Copy Complement". You will not see the complementary sequence at this point; it is copied to the clipboard:Return to the Order window and "Paste" the complement in this window, overwriting the probe displayed. You have the option of editing the primer/probe names, and adding the reporter/quencher dyes to the probe sequence.This document can now be saved and put into a Word document or attached to an e-mail message.在Results Archive中保存搜索结果Your search can also be saved in the Results Archive Folder. Click on the "Results" tab. The forward and reverse primers are displayed in their respective boxes, and the probe sequence is displayed in the "Cycle Params" box The probe sequence displayed is the original strand. To view/save the complementary strand, highlight the probe from the Sequence and select "Copy Complement". "Paste" the complement probe into the "Cycle Params". The complementary probe strand is now displayed. It is important to note that if you leave the Results page, the probe sequence will default back to the original. Each time you returnto the Results page you will need to re-paste the complementary probe strand. Note: The information displayed below the selected primer and probe sequences should be ignored when performing TaqMan Assays. The Universal TaqMan? Guidelines do not require you to perform optimizations, thus, the cycling/concentration, etc. information displayed here can be ignored. Save the Results by selecting "Save Results". A message will display showing the results were saved.打印结果 To print the Results, select "Open Results" from the "File" menu. The last (newest) results file will be the last one in the list (at the bottom of the list): Highlight and click "Open".This is the relevant information needed to order your primer/probe set. To print, click and drag, highlighting the information you want and selecting "Copy" from the "Edit" menu, placing it on the clipboard. This should be everything from the Sequence name through the TaqMan? probe annealing information.This is the relevant information needed to order your primer/probe set. To print, click and drag, highlighting the information you want and selecting "Copy" from the "Edit" menu, placing it on the clipboard. This should be everything from the Sequence name through the TaqMan? probe annealing information.You can then paste your sequence information in to a Word document; from here you can print a copy for your records.订购信息Be sure to include information on your needed synthesis scale and the corresponding part number, your reporter dye(s), your quencher (TAMRA), and your personal information (name, institution, address, phone fax etc.).。
生物信息学软件的基本使用方法介绍

生物信息学软件的基本使用方法介绍生物信息学是研究生物学中大规模数据的获取、存储、管理、分析和解释的学科。
为了能够有效地处理这些复杂的生物数据,生物信息学研究者使用了许多专门设计的软件工具。
本文将介绍几种常见的生物信息学软件,并提供基本的使用方法。
1. BLAST(Basic Local Alignment Search Tool):BLAST是一种用于基因序列比对和相似性搜索的软件工具。
它能够找到在数据库中与输入序列相似的序列,并计算它们之间的相似度分数。
使用BLAST时,首先需要选择要比对的数据库,如NCBI的nr数据库。
然后,将待比对的序列输入到BLAST中,并选择合适的算法和参数,最后点击运行按钮即可得到比对结果。
2. ClustalW:ClustalW是一种常用的多序列比对软件。
它能够将多个序列对齐,并生成比对结果。
使用ClustalW 时,首先需要输入要比对的序列。
可以通过手动输入、从文件中导入或从数据库中获取序列。
然后,选择合适的比对算法和参数,并点击运行按钮。
在比对结果中,会显示相似性分数矩阵和序列的对齐信息。
3. FASTA:FASTA是一种用于快速比对和搜索序列相似性的工具。
它使用一种快速的搜索算法,能够在大型数据库中快速找到与输入序列相似的序列。
使用FASTA时,需要将待比对的序列输入到软件中,并选择匹配的算法和搜索参数。
运行后,软件会生成相似序列的列表和相似性评分。
4. R:R是一种统计分析软件,也被广泛用于生物信息学领域。
它提供了丰富的函数和库供生物信息学研究者使用,用于数据处理、统计分析和可视化。
使用R时,可以通过命令行或脚本编写代码来执行各种操作。
例如,可以使用R中的Bioconductor库进行基因表达数据的分析和可视化。
5. IGV(Integrative Genomics Viewer):IGV是一种用于基因组数据可视化的软件工具。
它能够显示基因组位置上的测序深度、SNP、CNV等信息,并支持交互式操作和注释查看。
XRD分析软件jade5.0应用

相对于其它XRD分析软件,JADE系列软件应该说是做的最好的,它囊括了从定性,指标化,峰型拟合(JADE5.0)到无标定量,晶体结构分析(Jade6.0)等一系列的功能。
我个人以为,只要有Jade5.0就够了,至于后面两项功能,最好是用其它免费软件执行(在上应有尽有),傻瓜相机虽然好用,但要拍出好照片,还是专业相机好。
NO1.Jade5.0的安装和设置Jade5.0都是自动安装的,这不成问题。
要把PDF卡片引入,先将ICDD的光盘插入,然后pdf/setup/select all/,其它按提示进行。
可以对优选项进行设置:EDIT/preference/,里面包括了对显示窗口的设置,仪器参数的设置,打印输出的设置等,一般来说按默认就行,我本人则喜欢将MISC栏里的“Materials Data, Inc.”改为我自己的大名No2 数据的输入Jade软件可以直接读取Rigaku、Bruker、Philips、Scintag等很多衍射仪的原始数据。
打开File\patterns,将出现如附件中所示画面,先(I)找到你文件位置,从(III)的下拉框中选择你的数据格式,按(II)选择。
很多仪器输出文件的格式都是*.raw,实际上都是不一样的,但格式选错了也没关系,软件会给你自动转到合适的格式中去的。
高级一点的:有一些数据格式在(III)的下拉框中没有,比如最常见的txt,xy等,此时你可以自己动手设置,在以上的数据输入面板中,点击工具栏上的“import",进入格式设置画面,如附件所示,a区为注释区,b区为数据格式区,对于最简单的一列角度,一列强度的数据格式,a区不用填写,b区在” angle column“前打上勾,数据从第1行开始读,每行1列数据,强度数据从第8行开始(角度不算),角度从1至6列,所得数据格式即为附件中所示的数据格式。
你也可以按照自己的数据格式进行自由改动,如果a区中表明第1行有说明文字,则数据从第2行读入,相应在b区就将data starts改成2。
主成分分析软件操作说明

打开示 1.xls,找到sheet allele fqs
新建一个EXCEL文件,命名为示 2.xls,打开示 2.xls
把sheet allele fqs中BF到TAN选中,复制
复制选择性粘贴,到示 2.xls sheet1中,注意选择性粘贴转置到A2
将sheet allele fqs中22 23行、34 35行等等这样的行删掉,
选取B4到H190的区域,复制
选择性粘贴,转置到示 2.xls sheet1B2处
打开示 2.xls sheet2 鼠标到B2处键入=sheet1!b2/100 回车为0.047619
往下拉6行到B8,选取B2到B8,往右拉到GF
查找替换查找容空替换为0,减少小数点到小数点后4位
B2到GF8,复制选择性粘贴数值到sheet3 B2处,减少小数点到小数点后4位
将sheet1中A2到A8复制到sheet3A2处
Sheet3 B1处键入 1 C1处键入2,直到GF187
将SHEET1SHEET2删掉sheet3改成sheet1
OK 保存为示 3.xls
打开MVSP,FILE-IMPORT 示3.xls
Options 中选names at row import as strings -OK
Analyses-principle components options 中选center data ok
之后出来MVSP results ,注意axis 1、axis2、axis3中percentage 和一定要大于60%才可以做聚类分析
Graphs-scatter plot ok。
celescope用法

Celescope是一个开源的虚拟天文台软件,主要用于观测和分析天文数据。
它提供了一系列功能,包括数据查询、图像显示、数据处理和分析等。
以下是Celescope的基本用法:
1. 安装Celescope:首先,你需要从Celescope的官方网站下载并安装Celescope软件。
2. 打开Celescope:安装完成后,你可以通过启动Celescope软件来打开它。
3. 查询数据:在Celescope中,你可以通过输入关键字或选择目录来查询天文数据。
你可以查询各种类型的数据,如星表、恒星数据、行星数据等。
4. 浏览数据:查询到的数据会在Celescope的界面中显示出来,你可以浏览这些数据,并进行进一步的分析。
5. 数据处理:Celescope提供了一系列数据处理工具,如数据过滤、数据排序、数据统计等。
你可以使用这些工具对数据进行处理。
6. 分析数据:Celescope还提供了一系列数据分析工具,如图表生成、数据模拟等。
你可以使用这些工具对数据进行分析。
7. 保存和导出数据:在Celescope中,你可以将查询到的数据保存到本地计算机,或者导出为其他格式的数据。
以上就是Celescope的基本用法。
需要注意的是,Celescope是一个功能丰富的软件,具体使用方法可能会因人而异。
如果你想了解更多关于Celescope的使用方法,可以参考Celescope的官方文档或在线教程。
cell search操作流程

cell search操作流程摘要:1.cell search 操作流程概述2.cell search 操作流程的具体步骤3.cell search 操作流程的注意事项正文:一、cell search 操作流程概述cell search 操作流程,即细胞搜索操作流程,是一种用于检测和分析细胞结构的生物学实验技术。
该技术通过显微镜观察细胞,借助计算机分析系统对细胞结构进行定量和定位分析。
cell search 操作流程广泛应用于细胞生物学、生物医学和药物研发等领域,有助于科学家深入了解细胞结构和功能,为相关研究提供有力支持。
二、cell search 操作流程的具体步骤cell search 操作流程主要包括以下几个步骤:1.样本制备:首先,需要从生物体中提取细胞样本,并将其固定在载玻片上。
固定液可以防止细胞在实验过程中发生形态改变。
2.染色:将固定后的细胞样本进行染色,以便在显微镜下观察到细胞结构。
常用的染色方法包括荧光染色、免疫染色等。
3.扫描:使用激光共聚焦显微镜对染色后的细胞进行扫描,获取细胞结构的二维或三维图像。
4.图像处理:将扫描得到的图像输入计算机分析系统,进行图像处理和分析。
这一步通常包括去噪、分割、定位等操作,目的是提取出细胞结构信息。
5.分析:根据分析目的,对提取到的细胞结构信息进行统计和分析。
例如,可以计算细胞内各个结构的数量、大小、位置等参数,或者分析不同结构之间的相互关系。
6.结果输出:将分析结果以可视化的方式展示,便于科学家进行观察和分析。
常用的结果输出形式包括图像、图表等。
三、cell search 操作流程的注意事项在进行cell search 操作流程时,需要注意以下几点:1.样本处理:在制备细胞样本时,要确保细胞形态不变,以免影响实验结果。
2.染色方法:选择合适的染色方法,使细胞结构对比鲜明,便于观察和分析。
3.显微镜选择:使用合适的显微镜,确保获取的图像质量高,有利于后续图像处理和分析。
celref软件基本的操作如次

celref软件基本的操作如次:1. 开界面2. 点击diagram按钮3. file---open 打开相应的文件4. peaksearch 选择合适的寻峰条件5. 查看寻峰的结果,根据具体情况手动添加或删除一些衍射峰。
6. 切换到主窗口7. 在initial cell parameters窗口里填写相关的信息a. 选择正确的晶系和空间群b. 根据已经获得的信息,给出晶包参数的近似值。
c. 计算8. 切换到selection of reflections窗口,选择合适的衍射峰进行计算。
可以手动选择,也可以自动选择。
如果你在上一步的晶包参数给的初值和真实值之间差得很大,可能无法自动选择。
9. 切换到cell refinement窗口10.输入相应的波长和零点,然后refine。
建议(1).最好在进行计算之前,把衍射峰的ka2剔除,波长就不需要校正了,一般都是用的Cu的波长,1.54056埃。
建议(2).关于0点的校正,一般是用内标法,对于标样的衍射峰最好多取几个,加权平均,这样会更准确一些。
11.观察Sigmas值越小越好,一般要求3/10000以下;initial和refine的值,最好一致。
如果差得比较多,建议把refine得到的值当作晶包参数的初始值,回到initial cell parameters 窗口,按照以上步骤迭代运算。
直到得到满意的结果。
12.在计算中,主要是在选择衍射峰的过程中,要时刻注意选择的衍射峰是否正确,通过diagram窗口观测,一般可以避免错选。
13.结果一般可以 .txt的格式予以保存。
celref2.0就可以满足一般的计算要求;celref3.0还提供了更多的小工具,在每一步的计算都有很多微调的按钮,等你熟悉了,自然就能使用它,那时候,你计算的结果就更加可靠了。
OrcaFlex软件操作指引说课材料
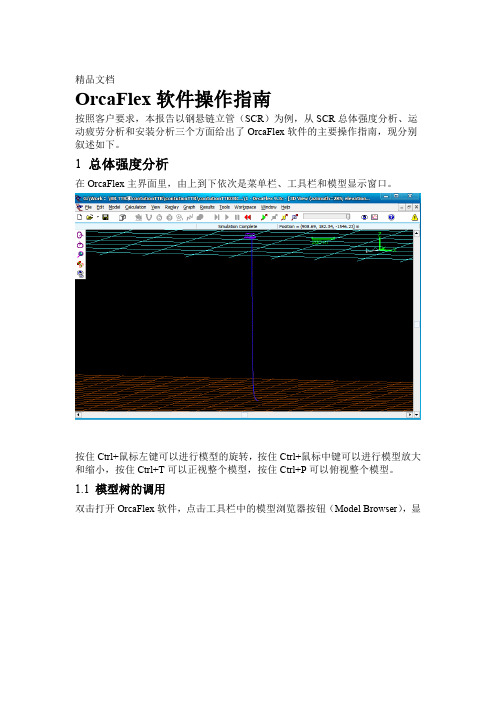
精品文档OrcaFlex软件操作指南按照客户要求,本报告以钢悬链立管(SCR)为例,从SCR总体强度分析、运动疲劳分析和安装分析三个方面给出了OrcaFlex软件的主要操作指南,现分别叙述如下。
1总体强度分析在OrcaFlex主界面里,由上到下依次是菜单栏、工具栏和模型显示窗口。
按住Ctrl+鼠标左键可以进行模型的旋转,按住Ctrl+鼠标中键可以进行模型放大和缩小,按住Ctrl+T可以正视整个模型,按住Ctrl+P可以俯视整个模型。
1.1模型树的调用双击打开OrcaFlex软件,点击工具栏中的模型浏览器按钮(Model Browser),显示模型树。
精品文档.精品文档环境参数设置1.2按钮打开环境参数设置界面。
双击EnvironmentSea1.2.1雷诺数计算方海水温度,由上到下可依次设置海平面位置,运动粘性系数其中海平面位置数值是相对于总体坐标系而言;温度为摄氏温度,它的大小直接影响到运动粘性系数。
而雷诺数的计算方法,主要取决于流速和结构特征长度的计算。
软件中三种方法雷诺数最终的计算公式分别为Re = |Vr|D/ν,Re crossnom= |Vr|Dcos(α)/ν,Re = |Vr|D/νcos(α),其中Vr径向速度。
OrcaFlex calculates flow Reynolds number in order to calculate drag and lift coefficients1.2.2Sea Density设置海水密度,可以是变化的,也可以是恒定不变的。
如该海域的海水密度为1025Kg/m3,具体如下图所示。
精品文档.精品文档1.2.3Sea Bed设置海底形状,海水的深度、斜度以及海底土壤的刚度系数,其中海底斜度和海底方向都是相对于总体坐标系而言,具体参数应在立管总体设计参数中给出。
具1.2.4Waves设置波浪的参数,主要包括波浪方向、波高、周期、起始时间,波浪类型等,其中波浪方向是相对于总体坐标系而言,波浪类型的选取取决于分析类型和实际海况,波高和周期根据海况资料给出,具体参数设置如下面表格和图所示。
酶标仪软件使用说明

酶标仪软件使用说明引言
概述
1.软件安装和启动
2.样本准备和加载
3.实验参数设置
4.数据分析和结果呈现
5.软件的高级功能
正文内容
1.软件安装和启动
1.1软件安装包
1.2安装软件
1.3启动软件
2.样本准备和加载
2.1样本准备
2.2样本加载
2.3样本容器设置
3.实验参数设置
3.1实验类型选择
3.2实验板设置
3.3样本控制设置
3.4标准曲线设置
3.5实验参数保存和加载
4.数据分析和结果呈现
4.1数据导入和预处理
4.2数据标准化
4.3曲线拟合和计算
4.4数据分析和结果计算
4.5结果呈现和导出
5.软件的高级功能
5.1质量控制设置
5.2实验过程监控
5.3多通道分析
5.4自动化实验
5.5数据备份和恢复
总结
酶标仪软件的使用方法应根据具体的软件版本和型号进行适配。
用户在使用过程中应仔细阅读软件的用户手册,并按照要求进
行操作。
如有任何问题或困惑,用户可随时联系厂商或寻求技术支持。
通过合理使用酶标仪软件,用户能够更好地完成实验并获得准确的结果,进而推动科学研究的进展。
cellquest和cellquest pro软件使用

CellQuest 和CellQuest Pro 软件使用CellQuest 和CellQuest Pro 软件是目前运用最为广泛的流式细胞仪采集及分析软件。
它运行于苹果电脑上,优质的矢量图显示使得操作界面特别优美。
现最新的微软公司的Vista 软件也是仿苹果操作系统界面的,可想而知在使用CellQuest 和CellQuest Pro 时的强大功能及视觉质感。
CellQuest 和CellQuest Pro 软件主安装在BD FACSCalibur 型流式细胞仪上。
1.软件数据流程在CellQuest 和CellQuest Pro 软件的数据流分为二个部分:方案和数据包。
方案是指用户可以建立采集或分析所需的直方图或散点图、设门并定义门与门、门与图之间的逻辑关系。
可以使用方案进行数据采集或分析以往已有的数据。
每个采集方案分析样本后会生成数据包,该数据包中包括了样本中每个颗粒在各个检测参数上的数值,格式为FCS2.0或FCS3.0。
数据包必须由方案文件打开,无法单独显示,或者可由第三方软件进行分析,如WinMDI 。
2.软件与仪器的连接流式细胞仪需要加电开启后会向计算机发出信息,所以如要进行实验需要先开启流式细胞仪,然后再开启计算机。
1、 先开仪器后开计算机,以确保仪器和计算机之间的正常通讯。
2、 在苹果菜单下点击CellQuest 和CellQuest Pro 软件。
3、 从Aquire 菜单下选择connet to cytometer 。
3.方案与数据之间的关系CellQuest 和CellQuest Pro 软件的方案与数据相互独立保存,数据包可以由任一方案文件进行分析,分析后不改变数据包中的任何数据。
CellQuest 和CellQuest Pro 软件中方案内的直方图或散点图有三种状态:Acquire ,Analysis 和Acquire-Analysis 。
当图的属性为Acquire 时,此图只限于采集下用,即只当进行检测时它才能显示颗CellQuest 和CellQuest Pro 方案文件的图标 CellQuest 和CellQuest Pro 数据文件的图标 CellQuest 和CellQuest Pro 软件中还会有关于仪器设置的文件,保存所有的实验条件。
流式细胞仪软件CELL Quest Pro 的简要操作步骤

Acquisition获取数据1.打开获取模板(ACQ文件)制作新模板:∙选择点图或直方图工具,在窗口拖出大小合适的方框,点击图形边缘∙Windows-Show Inspector,Inspector窗口选择:Acquisition获取X Parameter横轴参数:如FSCY Parameter纵轴参数:如SSCGate设门:如No Gate或G1=R1∙图形设置完毕,选择SAVE AS,保存为ACQ文件2.联机Acquire-Connect to cytometer,出现Acquisition Control窗口3.打开获取条件窗口,调出实验获取条件(Instrument Setting)∙Cytometer-Detectors/Voltage,Threshold,Compensation∙Cytometer- Instrument Setting-Open打开实验获取条件-Set-Done实验获取条件的调整: Acquisition Control- Setup-上样-Acquire-进行实验获取条件的调整1)FSC、SSC电压。
2)FSC阈值(Threshold):减少碎片。
3)设门:FSC/SSC点图设门(R1);荧光(FL)点图(如FL1/FL2)或直方图(如FL1)取门G1=R1(点击图形边缘-Inspector-Gate:G1=R1)。
4)阴性对照管,调整荧光电压(FL1、FL2、FL3、FL4 Voltage),阴性群体在左下角,确定十字位置。
5)多色实验的补偿管(单阳性),调整荧光间补偿(Compensation)Cytometer- Instrument Setting-Save保存实验获取条件(InstrSet)-Done;File--Save保存模板ACQ文件(新作模板)。
4.数据获取前的设定∙Acquire-Acquisition & Storage-设定获取细胞数(10000-All)-OK∙Windows-Browser对话框选择:—Directory设定存储数据文件的文件夹;—File设定数据文件的名字:文件前缀(Prefix)-自定义;文件后缀(Surfix)-管号;—检测或设置panel,确定试验组合;∙Acquire—Counter 打开计数器。
iCycler iQ 荧光定量PCR仪软件操作指南

iCycler iQ 荧光定量PCR仪软件操作指南V3.0版本Bio-Rad(中国)公司温度程序的编辑与修改查看一个实验的温度程序(View Protocol)1. 在Library模块中点击View Protocol标签可以查看已保存过的温度程序;2. 在Protocol Files下面点击想查看的温度程序名称可显示该程序内容;3. 如需运行这个程序,可点击Run with selected plate setup,这样可使用这个温度程序和样品盘设置来运行实验。
4. 如需打印这个温度程序,可点击Print this protocol。
修改和编辑一个实验的温度程序(Editing and Creating a Protocol)1. 开始修改和编辑一个实验的温度程序可以从以下任何一种途径进入Workshop模块来进行:• 在Library模块中的View Protocol 标签下点击Edit this protocol;• 在Library模块中的View Protocol 标签下点击Create a new protocol;• 点击Workshop 模块,然后选择Edit Protocol标签;2. 通过点击Insert Cycle, Delete Cycle, Insert Step, 和Delete Step来修改默认的或所选择的温度程序;3. This option stays active until you indicate that you’re done inserting/deleting. Deselect the option when completed;4. 点击电子数据表格中Cycle或Step单元格,然后输入相应的Dwell Time(温度维持时间)和Setpoint (所要求的温度);5. 如有需要,可在Show Options方格内选择程序功能选项可在电子数据表格中显示相应的功能列,比如Gradient (梯度)或Melt Curve(熔点曲线), 然后输入有关数据。
ELISACAL软件使用举例说明

ELISACAL软件使用举例说明。
该软件是长春陈万革先生编写的免费软件,目前仍在进一步完善中。
软件只能计算,目前不能打印。
ELISA回归软件使用举例:一、LH 试剂盒----双对数模型该类试剂盒属于夹心法,浓度与吸光值正相关。
包括FSH,PRL等。
大部分蛋白、1)标准系列有S0,S1,S2,S3,S4五个浓度。
可以做单孔或双孔。
然后带血样与质控。
2)做完实验,需要用酶标仪测出标准与血样的吸光值。
3)运行软件,X代表浓度,Y代表吸光值。
在软件中部的X、Y列中,第1行添入S1的浓度与对应的吸光值,第2行添入S2的浓度与对应的吸光值,第3行添入S3的浓度与对应的吸光值,第4行添入S4的浓度与对应的吸光值。
如果是做了平行孔,则可以添两行。
注意这里不需要添S0的吸光值。
4)S0的吸光值添在左侧“扣除本底”后面的空格中,并将前面的小方框选中。
5)在左侧“X数据列”的下拉框选“对数(10为底)”,“Y数据列”的下拉框选“对数(10为底)”。
6)此时左侧的“回归/拟合模型”自动设置为“直线回归”。
点击“回归/拟合”进行计算。
7)在此介面点“数据输入”可以回到前一屏幕对标准系列进行修改。
点“回归方程”可以察看拟合的参数。
8)r^2 = 0.99594 代表拟合的好坏。
如果这一数值低于0.9时表示拟合不好。
9)如果从前面的曲线图上能明显看出S4代表的点在直线下方,各个标准点不呈直线关系,则必须用曲线回归。
此时需要点“曲线”再点“数据输入”返回到最初的屏幕选其它回归方式如“Hill曲线”或四参数。
参考后面PRL例子。
10)没有问题的话,点“由Y计算X”。
在空格中输入样品或质控的吸光值,回车或点击“计算”。
即能计算对应的浓度。
使用ELISAcalc软件 绘制标准曲线以及批量计算样品浓度的教程

如何使用ELISAcalc软件绘制标准曲线以及批量得出样品浓度
此处选用的是“Logistic曲线拟合2(四参数)”模型。
1)输入数据。
这里中间的数据框中要输入标准品STA A至STA F的浓度(后面测出的结果的单位和此标准品单位一致)与吸光值。
在左侧“扣除本底”处可以输入空白孔(只加显色液及终止液,)的吸光值,如果没有做,则就不用填了。
2)“回/拟合模型”选择“Logistic曲线拟合2(四参数)”方式。
3)点击“回归/拟合”显示标准曲线。
4)点“回归方程”显示参数(内容可以直接复制)
5)点“由Y计算X”计算样品浓度。
在空格中输入样品的吸光值,回车或点击“计算”。
即能计算对应的浓度。
(技巧:可以在表格
中复制所有样品的Y值,在软件中点击“粘贴”按钮,就可以得出所有的X值哦!全部计算完可以点击“复制”按钮将结果复制下来,粘贴到EXCEL表格里面),另:若数值超出曲线范围,则有可能得不到结果,如下图的Y值所示。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
celref软件基本的操作如次:
1. 开界面
2. 点击diagram按钮
3. file---open 打开相应的文件
4. peaksearch 选择合适的寻峰条件
5. 查看寻峰的结果,根据具体情况手动添加或删除一些衍射峰。
6. 切换到主窗口
7. 在initial cell parameters窗口里填写相关的信息
a. 选择正确的晶系和空间群
b. 根据已经获得的信息,给出晶包参数的近似值。
c. 计算
8. 切换到selection of reflections窗口,选择合适的衍射峰进行计算。
可以手动选择,也可以自动选择。
如果你在上一步的晶包参数给的初值和真实值之间差得很大,可能无法自动选择。
9. 切换到cell refinement窗口
10.输入相应的波长和零点,然后refine。
建议(1).最好在进行计算之前,把衍射峰的ka2剔除,波长就不需要校正了,一般都是用的Cu的波长,1.54056埃。
建议(2).关于0点的校正,一般是用内标法,对于标样的衍射峰最好多取几个,加权平均,这样会更准确一些。
11.观察Sigmas值越小越好,一般要求3/10000以下;initial和refine的值,最好一致。
如果差得比较多,建议把refine得到的值当作晶包参数的初始值,回到initial cell parameters 窗口,按照以上步骤迭代运算。
直到得到满意的结果。
12.在计算中,主要是在选择衍射峰的过程中,要时刻注意选择的衍射峰是否正确,通过diagram窗口观测,一般可以避免错选。
13.结果一般可以 .txt的格式予以保存。
celref2.0就可以满足一般的计算要求;celref3.0还提供了更多的小工具,在每一步的计算都有很多微调的按钮,等你熟悉了,自然就能使用它,那时候,你计算的结果就更加可靠了。
