利用NCBI进行同源性一级结构分析
一步一步教你使用NCBI数据库资源解读

一步一步教你使用NCBI数据库资源随着ncbi数据库各种资源的涌现,NCBI已经成为科研工作者必不可少的资料查找,数据分析的工具。
那么NCBI 数据如何使用,新手入门一步一步教你认识和使用NCBI数据库。
一综合数据库NCBI数据库集美国国立生物技术信息中心(National Center for Biotechnology Information),即我们所熟知的NCBI 是由美国国立卫生研究院(NIH)于1988年创办。
创办NCBI 的初衷是为了给分子生物学家提供一个信息储存和处理的系统。
除了建有GenBank核酸序列数据库(该数据库的数据资源来自全球几大DNA数据库,其中包括日本DNA数据库DDBJ、欧洲分子生物学实验室数据库EMBL以及其它几个知名科研机构)之外,NCBI还可以提供众多功能强大的数据检索与分析工具。
目前,NCBI提供的资源有Entrez、Entrez Programming Utilities、My NCBI、PubMed、PubMed Central、Entrez Gene、NCBI Taxonomy Browser、BLAST、BLAST Link (BLink)、Electronic PCR等共计36种功能,而且都可以在NCBI的主页上找到相应链接,其中多半是由BLAST功能发展而来的。
1 NCBI最新进展1.1 PubMed搜索功能的增强去年,NCBI对PubMed进行了几项改进工作,改动最大的是搜索界面和摘要浏览界面。
其中,搜索界面中新增了“Advanced Search”选项(这实际上是对以往“Limits”和“Preview/Index”功能的整合),并且增加了一个新的窗口,用户可以在此窗口下通过“论文作者名”、“论文所属杂志名称”、“论文出版日期”等限定条件进行搜索。
而且,“论文作者名”和“论文所属杂志名称”还设有文本框自动填充功能。
现在,在PubMed数据库中进行文本搜索的同时还可以立即通过两个“内容传感器(content sensors)”进行分析。
NCBI中Blast种类及利用简介

NCBI中Blast种类及利用简介NCBI中Blast种类简介1. Blast Assembled Genomes在一个选择的物种基因组序列中去搜索。
2.Basic Blast2.1 nucleotide blast--- 用核酸序列到核酸数据库中进行搜索,包括3个程序2.1.1 Blastn----核酸序列(n)到核酸序列数据库中搜索,是一种标准的搜索。
2.1.2 megablast----该程序利用“模糊算法”加速了比较速度,能够用于快速比较两大系列序列。
能够用来搜索一匹ESTs序列和大的cDNA或基因组序列, 适用于由于测序或其他缘故形成的轻微的不同的序列之间的比较2.1.3 discontiguous megablast----与megablast不同的是要紧用来比较来自不同物种之间的相似性较低的不合序列。
2.2 Protein Blast2.2.1 Blastp ---蛋白质序列到蛋白质序列数据库中搜索,是一种标准的搜索。
2.2.2 psi-blast---位点特异迭代BLAST —用蛋白查询来搜索蛋白资料库的一个程式。
所有被BLAST发觉的统计有效的对齐被总和起来形成一个多次对齐,从那个对齐,一个位置特异的分值矩阵成立起来。
那个矩阵被用来搜索资料库,以找到额外的显著对齐,那个进程可能被反复迭代一直到没有新的对齐能够被发觉。
2.2.3 PHI-BLAST---以常规的表达模型为专门位置进行PSI - BLAST检索,找出和待查询序列具有一样的表达模型且具有同源性的蛋白质序列。
2.3 Translating BLAST2.3.1 blastx----先将待查询的核酸序列按6 种读框翻译成蛋白质序列,然后将翻译出的蛋白质序列与NCBI 蛋白质序列数据库比较。
2.3.2 tblastn-----先将核酸序列数据库中的核酸序列按6 种读框翻译成蛋白质序列,然后将待查询的蛋白质序列与翻译结果进行比较。
ncbi使用指导

ncbi使用指导摘要:一、NCBI简介1.NCBI的定义与作用2.NCBI的主要数据库二、NCBI数据库的使用1.基因数据库1.1 基因序列数据库1.2 基因表达数据库1.3 基因调控数据库2.蛋白质数据库2.1 蛋白质序列数据库2.2 蛋白质结构数据库3.核酸序列数据库3.1 核酸序列数据库概述3.2 核酸序列数据库的使用方法4.文献数据库4.1 PubMed简介4.2 如何利用PubMed进行文献检索三、NCBI工具的使用1.基因芯片数据分析工具2.基因序列比对工具3.蛋白质结构预测工具四、NCBI的进阶使用技巧1.如何利用NCBI进行基因注释2.如何利用NCBI进行基因家族分析3.如何利用NCBI进行共表达网络分析正文:一、NCBI简介CBI(National Center for Biotechnology Information,美国国家生物技术信息中心)是一个提供生物信息学资源的网站,它为全球科研工作者提供了大量的生物学数据和工具。
NCBI的主要数据库包括基因数据库、蛋白质数据库、核酸序列数据库和文献数据库等。
二、NCBI数据库的使用1.基因数据库基因数据库包括基因序列数据库、基因表达数据库和基因调控数据库。
基因序列数据库提供了大量的基因序列信息,用户可以通过关键词搜索、序列相似性搜索等方式找到需要的基因序列。
基因表达数据库则提供了基因在不同生物体、不同组织、不同发育阶段的表达信息。
基因调控数据库则包含了基因调控相关的信息,如启动子、转录因子结合位点等。
2.蛋白质数据库蛋白质数据库包括蛋白质序列数据库和蛋白质结构数据库。
蛋白质序列数据库提供了蛋白质的氨基酸序列信息,用户可以通过序列相似性搜索找到相似的蛋白质序列。
蛋白质结构数据库则提供了蛋白质的三维结构信息,用户可以通过结构域、功能域等关键词搜索需要的蛋白质结构。
3.核酸序列数据库核酸序列数据库包括DNA序列数据库和RNA序列数据库。
ncbi使用方法

ncbi使用方法(原创版4篇)《ncbi使用方法》篇1CBI(National Center for Biotechnology Information)是美国国家生物技术信息中心的缩写,它提供了许多生物学和生命科学相关的数据库和工具。
以下是使用NCBI 的一些基本方法:1. 核酸序列数据库(Nucleotide Sequence Database):在NCBI 主页上,可以选择核酸序列数据库,输入序列名称或序列号,然后点击“Search”按钮即可查询序列信息。
2. 蛋白质序列数据库(Protein Sequence Database):在NCBI 主页上,可以选择蛋白质序列数据库,输入蛋白质名称或蛋白质号,然后点击“Search”按钮即可查询蛋白质信息。
3. 基因组数据库(Genome Database):在NCBI 主页上,可以选择基因组数据库,输入基因组名称或基因组号,然后点击“Search”按钮即可查询基因组信息。
4. 代谢通路数据库(Metabolic Pathway Database):在NCBI 主页上,可以选择代谢通路数据库,输入代谢通路名称或代谢通路号,然后点击“Search”按钮即可查询代谢通路信息。
5. 生物投影数据库(BioProject Database):在NCBI 主页上,可以选择生物投影数据库,输入生物投影名称或生物投影号,然后点击“Search”按钮即可查询生物投影信息。
6. 序列比对工具(Sequence Alignment Tool):NCBI 提供了一款名为“Clustal Omega”的序列比对工具,可以在NCBI 主页上使用该工具进行序列比对。
7. 基因表达数据库(Gene Expression Database):NCBI 提供了一款名为“GEO”的基因表达数据库,可以在NCBI 主页上查询基因表达数据。
8. 蛋白质结构数据库(Protein Structure Database):NCBI 提供了一款名为“RCSB PDB”的蛋白质结构数据库,可以在NCBI 主页上查询蛋白质结构信息。
生物信息学NCBI的使用

开始
按照工作要求,直接选择 Blast方法
蛋白质-蛋白质序列比对 也可以选择tblastn
序列输入方式
序列主体
选择搜索区域,这里我们 填入序列(copy+ 要搜索整个序列,不填 paste)Fasta格式, 第1个是”>”不能忘记! 序列信息描述 或者纯序列
选择搜索数据库,这 里我们选nr(非冗余的 设置搜索的范围,选择特定 蛋白序列库)。 物种,或者Entrez关键词 选择BLAST程序
空位罚分 对打分矩阵的调整过滤简单重复序列检索结果
图形示意结果
检索结果-匹配序列列表
目标序列描述部分
带有genbank的链接, 点击可以进入相应的 genbank序列
进入相应的genbank序列
物种来源
Graphics结果
检索结果
具体匹配情况
E值为0,不可能随机匹配 残基完全相同 空位为0
NCBI-BLAST的介绍
常用的Blast工具
在此进入蛋白质数据 库搜索P03958序列
核苷酸-核苷酸序列比对 蛋白质-蛋白质序列比对 蛋白质序列-核酸数据库翻译后的 核酸序列翻译成蛋白质序列-蛋白 核酸翻译成蛋白质序列-核酸数据 蛋白质序列比对 质数据库中的序列比对 库中的核酸译成的蛋白质序列比对
如果接受其他参数默认 设置,点击开始搜索
与核酸相关的数据库
与蛋白质相关的数据库
详细参数设置 最多显示100条序列
E值上限10如果联配的统计显著性值(E 值)小于该值
匹配要求更严格,结果报告中随机产生的匹配序列减少。
Word长度 (10),则该联配将被检出,换句话说,比较低的阀值将使搜索的
打分矩阵,取默认
谢谢
生物信息学
关于分子生物学中的同源性分析

在分子生物学的教学及研究中,经常对核苷酸或氨基酸序列进行比对以确定基因之间或蛋白质之间的同源关系,进而根据同源性来推测物种间的亲缘关系。
基因或蛋白质之间的同源关系包括直系同源和旁系同源,序列间的同源性可用相似性或一致性来进行量化,用相似性(一致性)来判断序列是否同源。
一、同源性的概念在生物学中,同源性(homology)是指在进化过程中源于同一祖先的分支之间的关系。
我们可以在生物学的不同层次(如形态性状、分子性状等)上进行同源性分析,形态性状由于进行上或个体发育上的共同来源而呈现出本质上的相似性,但其功能不一定相同,那么它们就是同源的,如马的前肢与鸟的翅就是同源器官。
在分子水平上同源性主要是指基因的核苷酸序列或蛋白质的氨基酸序列之间的相似程度。
同源基因或蛋白质(homolog)指遗传上从某一共同祖先经趋异进化而形成的具有不同序列的基因或蛋白质。
同源性是一个相对的概念,在一定水平和范围内对其研究才有意义[1]。
二、直系同源与旁系同源同源关系包括两种类型:直系同源(ortholog)和旁系同源(paralog)。
这里我们主要以同源基因为例来进行讨论,同源蛋白质是同样的情况。
同源基因是遗传上来自某一共同祖先DNA序列的基因,包括直系同源基因和旁系同源基因。
直系同源基因,又称直向或垂直同源基因,指的是这样一些基因,它们起源于这些基因所在物种的最近共同祖先的一个祖先基因。
这些基因通常具有相同的功能,但并不是绝对的,当我们比较直系同源基因时,可能会发现有的基因失去了原来的功能或者进化出了新的功能[2-5]。
因此,直系同源基因描述在不同物种中来自于共同祖先的基因。
旁系同源基因,又称横向或并行同源基因,指在一个特定的基因组中由于基因复制产生的同源基因。
当我们比较旁系同源基因时,发现它们可能彼此具有了新的功能,也可能成为假基因了[2-4]。
旁系同源基因描述在同一物种内由于基因复制而分离的同源基因。
如图1所示,祖先球蛋白基因(globin gene)经过复制后分离产生了α球蛋白和β球蛋白基因,这两类基因就是旁系同源基因。
NCBIblast使用教程[1]
![NCBIblast使用教程[1]](https://img.taocdn.com/s3/m/0f001b46910ef12d2bf9e73e.png)
E值范围
3.设置结果输出显示格式
选择需要显示的选项 以及显示的文件格式
显示数目
Alignment的显
筛选结果
示方式
点击开始搜索
其他一些显示格式参数
NCBIblast使用教程[1]
提交任务
返回查询号(request id) 修改完显示格式后点 击进入结果界面
可以修改显示结果格式
NCBIblast使用教程[1]
NCBIblast使用教程[1]
Blast程序评价序列相似性的两个数据
Score:使用打分矩阵对匹配的片段进行打分,这是
对各对氨基酸残基(或碱基)打分求和的结果,一般来 说,匹配片段越长、 相似性越高则Score值越大。
E value:在相同长度的情况下,两个氨基酸残基(或
碱基)随机排列的序列进行打分,得到上述Score值的 概率的大小。E值越小表示随机情况下得到该Score值的 可能性越低。
分析过程(一)
1.登陆ncbi的blast主页
2.选择程序,因为 查询序列是蛋白序 列可以选择blastp,
点击进入
也可以选择tblastn
作为演示, 我们这里选blastp
NCBIblast使用教程[1]
分析过程(二)
3.填入序列(copy+pa索整个序列,不填
w 其他问题:实际使用时选择哪种方式(网 络,本地化),参数的选择,结果的解 释…
NCBIblast使用教程[1]
Blast资源
1.NCBI主站点:
/BLAST/(网络版) ftp:///blast/ (单机版)
5.选择搜索数据库,这里我们 选nr(非冗余的蛋白序列库)。
是否搜索保守区域数据库 (cdd),蛋白序列搜索才有。
NCBI功能详介分解

GenBank Overview基本信息∙什么是GenBank?GenBank是一个有来自于70,000多种生物的核苷酸序列的数据库。
每条纪录都有编码区(CDS)特征的注释,还包括氨基酸的翻译。
GenBank属于一个序列数据库的国际合作组织,包括EMBL和DDBJ。
∙纪录样本 - 关于GenBank的各个字段的详细描述,以及同Entrez搜索字段的交叉索引。
∙访问GenBank - 通过Entrez Nucleotides来查询。
用accession number,作者姓名,物种,基因/蛋白名字,还有许多其他的文本术语来查询。
关于Entrez更多的信息请看下文。
用BLAST来在GenBank和其他数据库中进行序列相似搜索。
用E-mail来访问Entrez和BLAST可以通过Query 和BLAST服务器。
另外一种选择是可以用FTP下载整个的GenBank和更新数据。
∙增长统计 - 参见公布通知的2.2.6(每个分类的统计),2.2.7(每个物种的统计),2.2.8(GenBank 增长)小节。
∙公布通知,最新 - 最近和即将有的变化,GenBank的分类,数据增长统计,GenBank的引用。
∙公布通知,旧 - 同上相同,是过去公布的统计。
∙遗传密码 - 15个遗传密码的概要。
用来确保GenBank中纪录的编码序列被正确的翻译。
(向)GenBank提交(数据)∙关于提交序列数据,收到accession number,和对纪录作更新的一般信息。
∙BankIt - 用于一条或者少数条提交的基于WWW的提交工具软件。
(请在提交前用VecScreen去除载体)∙Sequin - 提交软件程序,用于一条或者很多条的提交,长序列,完整基因组,alignments,人群/种系/突变研究的提交。
可以独立使用,或者用基于TCP/IP的“network aware”模式,可以链接到其他NCBI的资源和软件比如Entrez和PowerBLAST。
网页方式下利用BLAST 程序进行基因/蛋白质序列比对

美国国家生物技术信息中心(National Center of Biotechology Information ,NCBI) 充分利用Internet ,为用户提供了丰富的生物信息资源。
NCBI 的BLAST 程序是进行核酸序列和蛋白质序列相似性比较的优秀工具。
1 BLAST简介NCBIBLAST(Basic Local Alignment Search Tool ,局部对比基本检索工具) 是将核酸序列或蛋白质序列与可用的序列数据库进行相似性比较的一系列程序。
其核心是程序BLAST210。
BLAST是一个寻找序列间具有相似性的区段,进而比较它们之间结构和功能的工具,而不是仅仅比较整个序列的同源性。
BLAST的应用范围相当广泛,适用于核酸或蛋白质序列与可用的序列数据库之间的比较,也可用于几个序列间的比较:核酸- 核酸、核酸- 蛋白质、蛋白质- 蛋白质之间。
NCBI 的BLAST 提供了网页、电子邮件以及FTP 三种方式进行序列分析,使用十分方便。
2 各种BLAST介绍BLAST经过不断发展完善,有以下几种类型:1 Nucleotide BLASTNucleotide BLAST是输入核酸序列,用这些序列与其它核酸序列比较。
2.1.1 Standard nucleotide - nucleotide BLAST(标准核酸- 核酸BLAST):以三种格式(FASTA 格式、GenBank Accession 编码或GI编码) 的核酸序列与NCBI 核酸序列数据库作比较。
2.1.2 MEGABLAST:该程序使用“模糊算法”加快了比较速度,可以用于快速比较两大系列序列。
2.1.3 Search for short , nearly exact sequences (近似的短序列检索) :该检索和带有默认参数的Standard nucleotide - nucleotideBLAST很相似,是以短序列进行检索。
NCBI在线BLAST使用方法与结果详解

NCBI在线BLAST使用方法与结果详解BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较的分析工具。
BLAST程序能迅速与公开数据库进行相似性序列比较。
BLAST结果中的得分是对一种对相似性的统计说明。
BLAST 采用一种局部的算法获得两个序列中具有相似性的序列。
Blast中常用的程序介绍:1、BLASTP是蛋白序列到蛋白库中的一种查询。
库中存在的每条已知序列将逐一地同每条所查序列作一对一的序列比对。
2、BLASTX是核酸序列到蛋白库中的一种查询。
先将核酸序列翻译成蛋白序列(一条核酸序列会被翻译成可能的六条蛋白),再对每一条作一对一的蛋白序列比对。
3、BLASTN是核酸序列到核酸库中的一种查询。
库中存在的每条已知序列都将同所查序列作一对一地核酸序列比对。
4、TBLASTN是蛋白序列到核酸库中的一种查询。
与BLASTX相反,它是将库中的核酸序列翻译成蛋白序列,再同所查序列作蛋白与蛋白的比对。
5、TBLASTX是核酸序列到核酸库中的一种查询。
此种查询将库中的核酸序列和所查的核酸序列都翻译成蛋白(每条核酸序列会产生6条可能的蛋白序列),这样每次比对会产生36种比对阵列。
下面是具体操作方法1,进入在线BLAST界面,可以选择blast特定的物种(如人,小鼠,水稻等),也可以选择blast所有的核酸或蛋白序列。
不同的blast程序上面已经有了介绍。
这里以常用的核酸库作为例子。
2,粘贴fasta格式的序列。
选择一个要比对的数据库。
关于数据库的说明请看NCBI在线blast数据库的简要说明。
一般的话参数默认。
3,blast参数的设置。
注意显示的最大的结果数跟E值,E值是比较重要的。
筛选的标准。
最后会说明一下。
4,注意一下你输入的序列长度。
注意一下比对的数据库的说明。
5,blast结果的图形显示。
没啥好说的。
6,blast结果的描述区域。
ncbi结构域

ncbi结构域NCBI(National Center for Biotechnology Information)结构域是一个用于存储和分析生物信息学数据的数据库。
它包含了一系列生物大分子(如蛋白质和核酸)的结构域信息,为生物学研究提供了重要的资源。
本文将介绍NCBI结构域的概念、应用和特点。
一、NCBI结构域的概念NCBI结构域是指生物大分子中具有特定功能和结构的区域。
它可以是蛋白质中的一个片段,也可以是整个蛋白质序列。
NCBI结构域数据库收集了大量已知的结构域信息,并通过分析和比对这些结构域,帮助研究人员理解生物大分子的结构和功能。
二、NCBI结构域的应用1. 蛋白质功能预测:通过比对未知蛋白质序列与已知结构域数据库中的结构域,可以预测该蛋白质的功能和结构。
这对于药物设计、疾病研究和基因工程等领域具有重要意义。
2. 进化研究:通过分析不同物种中相同结构域的差异,可以揭示生物进化过程中的变化和适应机制。
这有助于我们理解物种间的亲缘关系和共同祖先。
3. 蛋白质结构预测:通过寻找已知结构域数据库中与未知蛋白质序列相似的结构域,可以预测该蛋白质的三维结构。
这对于理解蛋白质的功能和相互作用具有重要作用。
三、NCBI结构域的特点1. 多样性:NCBI结构域数据库收集了来自各种生物物种的结构域信息,涵盖了广泛的生物多样性。
这使得研究人员可以对不同物种中的结构域进行比较和分析。
2. 可靠性:NCBI结构域数据库中的信息来自于大量的实验证据和研究成果,具有高度的可靠性。
研究人员可以放心地使用这些数据进行科学研究。
3. 更新性:NCBI结构域数据库定期更新,以收集最新的结构域信息。
这保证了研究人员可以及时获取到最新的数据。
4. 数据丰富性:NCBI结构域数据库不仅包含了结构域的序列信息,还提供了结构域的二级结构、功能注释和相互作用等详细信息。
这使得研究人员可以进行更深入的分析和研究。
5. 方便查询:NCBI结构域数据库提供了多种查询工具和方法,使得研究人员可以方便地搜索和筛选感兴趣的结构域信息。
ncbi blast的功能和种类

ncbi blast的功能和种类NCBI BLAST是一种广泛使用的生物信息学工具,用于比对生物序列。
BLAST是Basic Local Alignment Search Tool的缩写,它可以将一个给定的生物序列与数据库中的其他序列进行比对,以找到相似性和同源性。
NCBI BLAST提供了多种不同类型的BLAST程序,每个程序都针对不同类型的比对任务。
下面是NCBI BLAST常见的几种程序:1. BLASTN:用于比对核酸序列,如DNA和RNA。
BLASTN可以找到两个序列之间的相似性和同源性,并确定它们之间的区别。
这个程序通常用于基因组学和转录组学研究中。
2. BLASTP:用于比对蛋白质序列。
BLASTP可以找到两个蛋白质之间的相似性和同源性,并确定它们之间的区别。
这个程序通常用于蛋白质结构预测和功能注释研究中。
3. BLASTX:用于将未知核酸序列与已知蛋白质序列进行比对。
BLASTX将未知核酸序列翻译成蛋白质序列,然后与已知蛋白质序列进行比对。
这个程序通常用于寻找新基因或预测基因功能。
4. TBLASTN:用于将已知蛋白质序列与未知核酸序列进行比对。
TBLASTN将已知蛋白质序列翻译成核酸序列,然后与未知核酸序列进行比对。
这个程序通常用于在基因组中寻找新的蛋白质编码基因。
5. TBLASTX:用于将两个未知核酸序列进行比对。
TBLASTX将两个未知核酸序列翻译成蛋白质序列,然后进行比对。
这个程序通常用于寻找新基因或预测基因功能。
除了这些常见的程序之外,NCBI BLAST还提供了其他一些特殊的程序,如PSI-BLAST、RPS-BLAST和PHI-BLAST等。
PSI-BLAST是一个迭代的BLAST程序,可以在多次比对中改进结果,并生成一个更准确的蛋白质家族模型。
RPS-BLAST是一个与CDD(Conserved Domain Database)相关的程序,可以在已知域和未知蛋白质之间找到相似性和同源性。
序列相似性比较与同源性分析

序列相似性⽐较与同源性分析⾸先应该注意区分序列相似性与序列同源性的关系,序列相似不⼀定同源,但是判定同源性关系的时候有些算法(Maximum likelihood除外)要考虑到序列相似性。
序列相似性是将待研究序列与DNA或蛋⽩质序列库进⾏⽐较,⽤于确定该序列的⽣物属性,也就是找出与此序列相似的已知序列是什么,完成这⼀⼯作只需要⽤到两两序列⽐较算法,常⽤的程序包有BLAST,FASTA等。
同源性分析是将待研究序列加⼊到⼀组与之同源,但是来⾃不同物种的序列中进⾏多序列⽐对,以确定该序列与其它序列间的同源性⼤⼩。
多序列⽐较算法常⽤的程序包有CLUSTAL等。
1、序列⽐对,从数据库中寻找相似序列:⾸先打开NCBI的BLAST⽹站:,选择protein blast,然后将待⽐对序列粘贴进去,进⾏BLAST(⼀些参数的设置收藏夹或百度)。
等待⼀定时间后将会出现与所选数据库的⽐对结果,按照打分⾼低将top100(可以设置成其他数值)的序列显⽰出来,然后可以将该100条序列下载下来。
存成test.fasta⽂件。
这个⽂件就是在mega中进⾏多序列⽐对建树所⽤的⽂件。
2、多序列⽐对:打开mega,ALIGN-BUILDALIGNMENT-Create a new alignment-protein-open-retrieve sequences from file-no -test.fasta(或者直接拖动进去,或者双击打开test.fasta),然后点击Alignment——Align by ClustalW——OK——OK。
然后⽐对成功,选择Data——Export Alignment——MEGA format保存⽂件为test.meg,可以关闭Align会话框。
3、构建进化树:打开test.meg。
点击PHYLOGENY——选择最上⾯的ML⽅法,参数可以选择默认参数。
就出现了进化树。
当然⼀些参数最好还是⽤到,⽐如说可信度验证的次数设置最好要⼤于等于500次。
NCBI在线BLAST使用方法与结果详解

NCBI在线BLAST使用方法与结果详解NCBI(National Center for Biotechnology Information)是一个包含大量基因组学、生物信息学等相关数据和工具的数据库。
其中,BLAST (Basic Local Alignment Search Tool)是一种常用的序列比对工具,可用于在数据库中搜索相似序列。
一、BLAST简介BLAST是一种基于序列比对的方法,可用于确定一给定序列与数据库中序列的相似性。
其工作原理是将查询序列与数据库中的序列进行比对,并生成一个比对得分来衡量它们之间的相似程度。
通过BLAST的结果,可以获得序列的匹配位置、长度、相似性等信息,从而帮助研究人员进行更深入的生物学研究。
二、使用方法1. 打开NCBI网站首先,打开浏览器,输入NCBI的网址(https:///),进入NCBI的官方网站。
2. 进入BLAST页面在NCBI的主页上,找到“BLAST”或“BLAST and Alignments”选项,并点击进入BLAST页面。
3. 输入查询序列在BLAST页面上,找到“Enter Query Sequence”或“Enter accession number, gi, or FASTA sequence”等文本框,将需要查询的序列输入其中。
可以直接复制粘贴序列,或选择上传文件的方式输入。
4. 选择数据库在BLAST页面上,找到“Choose Search Set”或“Database”等选项,选择需要比对的数据库。
NCBI提供了多个数据库,如“nr”(非冗余蛋白数据库)、“nt”(非冗余核酸数据库)等,根据研究需要选择合适的数据库。
5. 设置参数根据需要,可以通过“Algorithm parameters”等选项来设置比对参数,如设置匹配的阈值、比对的方式等。
6. 运行BLAST设置完成后,点击“BLAST”或“Run BLAST”等按钮运行BLAST。
大豆转录因子WRI1基因的生物信息学分析

大豆转录因子WRI1基因的生物信息学分析作者:王巍杰吴丹王涛来源:《湖北农业科学》2016年第13期摘要:从NCBI查找大豆(Glycine max)基因组中转录因子WRI1基因,通过同源比对在大豆基因组中确定了31个同源基因。
利用在线分析工具和生物信息学方法对31个蛋白质进行了初步分析,发现蛋白质的一级结构存在较大差异,二级结构以无规则卷曲和α-螺旋为主要构成元件,亚细胞均定位于细胞核。
保守结构域分析发现,31个蛋白质的高保守区域由大约200个氨基酸残基组成;正选择位点分析发现Glyma08g24420.1和Glyma15g34770.1两个蛋白质序列的第381、382、383个氨基酸位点受到了正选择,进行了适应性进化。
关键词:大豆(Glycine max);WRI1;生物信息;正选择位点中图分类号:S565.1;Q78 文献标识码:A 文章编号:0439-8114(2016)13-3482-04DOI:10.14088/ki.issn0439-8114.2016.13.055植物油脂在人类日常生活中扮演着不可替代的角色,不仅可以作为食用油,还是重要的工业原料,是生物新能源开发的重要材料来源。
目前,随着植物油需求量的增加和消费者对膳食脂肪安全意识的提高,培育高油量、高质量的油料作物已经成为育种的主要任务之一。
植物油脂合成过程涉及许多关键酶,通过生物学方法,一些关键酶已经确定,如乙酰辅酶A羧化酶、丙酮酸激酶、脂肪酸延长酶等[1],抑制或提高这些关键酶的活性可以影响植物种子的含油量。
近年来,研究表明利用转录因子基因改造植物脂肪代谢过程,可以更好地提高油脂含量,改善油脂成分。
转录因子能与基因5′端上游特定序列专一结合,保证目的基因以特定的强度在特定的时间与空间表达蛋白质分子。
科研人员通过抑制或过表达手段已经研究了一些与油脂合成有关的转录因子的功能,例如WRINKLED1、LEAFYCOTYLEDON1、FUSCA3等[2],其中WRINKLED1研究的比较多。
NCBI使用方法

NCBI提供检索的服务包括:1.GenBank(NIH遗传序列数据库):一个可以公开获得所有的DNA序列的注释过的收集。
GenBank是由NCBI受过分子生物学高级训练的工作人员通过来自各个实验室递交的序列和同国际核酸序列数据库(EMBL和DDBJ)交换数据建立起数据库的。
它同日本和欧洲分子生物学实验室的DNA数据库共同构成了国际核酸序列数据库合作。
这三个组织每天交换数据。
其中的数据以指数形式增长,最近的数据为它已经有来自47000个物种的30亿个碱基。
2.Molecular Databases(分子数据库):Nucleotide Sequence(核酸序列库):从NCBI其他如Genbank数据库中收集整理核酸序列,提供直接的检索。
Protein Sequence (蛋白质序列库):与核酸类似,也是从NCBI多个不同资源中编译整理的,方便研究者的直接查询。
Structure(结构)——关于NCBI结构小组的一般信息和他们的研究计划,另外也可以访问三维蛋白质结构的分子模型数据库(MMDB)和用来搜索和显示结构的相关工具。
MMDB:分子模型数据库—一个关于三维生物分子结构的数据库,结构来自于X-ray晶体衍射和NMR色谱分析。
Taxonomy(分类学)——NCBI的分类数据库,包括大于7万余个物种的名字和种系,这些物种都至少在遗传数据库中有一条核酸或蛋白序列。
其目的是为序列数据库建立一个一致的种系发生分类学。
3.Literature Databases(文献数据库)(1)PubMed是NLM提供的一项服务,能够对MEDLINE上超过1200万条的上世纪六十年代中期至今的杂志引用和其他的生命科学期刊进行访问,并可以连接到参与的出版商网络站点的全文文章和其他相关资源。
(2)PMC/PubMed Center:也是NLM的生命科学期刊文献的数字化存储数据库,用户可以免费获取PMC的文章全文,除了部分期刊要求对近期的文章付费。
利用NCBI进行同源性一级结构分析

1首先把与比对的序列输入或导入下面的query sequence序列框中
2然后点勾选上所比对的项目与显示结果在新窗口中
3在新窗口中变出现比对的结果图,从上往下依次是Graphic Summary界面,Descriptions界面,Alignments内容界面中query是自己的序列,下面很多细红线条就是库里的同源序列了,根据此可以推测自己的编码序列的大小。
4新窗口中的比对图示下部是Descriptions界面,描述的同源序列
5如果勾选上比对出的几个序列前面的方框,然后点击Alignments Download GenPept Graphics Distance tree of results Multiple alignment等选项,分别会出
现两个序列比对结果,下载所勾选的序列,对应的蛋白序列,基因图示,进化树图像和所有勾选的序列的同源性比对结果图。
DNA序列比对同源性分析图解BLAST
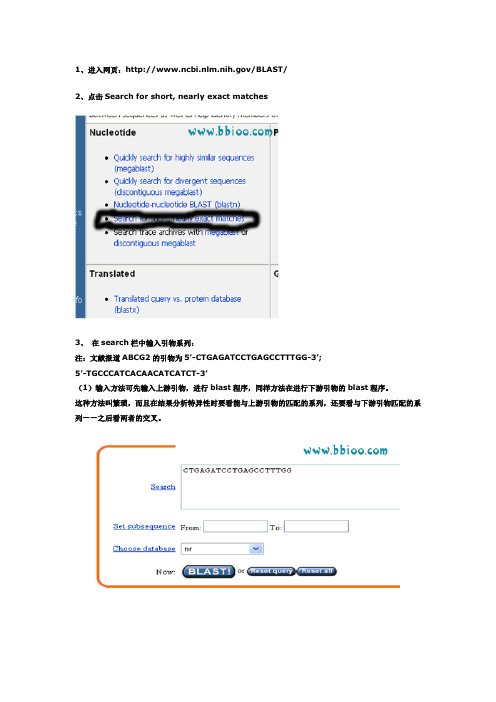
1、进入网页:/BLAST/2、点击Search for short, nearly exact matches3、在search栏中输入引物系列:注:文献报道ABCG2的引物为5’-CTGAGATCCTGAGCCTTTGG-3’;5’-TGCCCATCACAACATCATCT-3’(1)输入方法可先输入上游引物,进行blast程序,同样方法在进行下游引物的blast程序。
这种方法叫繁琐,而且在结果分析特异性时要看能与上游引物的匹配的系列,还要看与下游引物匹配的系列——之后看两者的交叉。
(2)简便的做法是同时输入上下游引物:有以下两种方法。
输入上下游引物系列都从5’——3’。
A、输入上游引物空格输入下游引物B、输入上游引物回车输入下游引物4、在options for advanced blasting中:select from 栏通过菜单选择Homo sapiensExpect后面的数字改为105、在format中:select from 栏通过菜单选择Homo sapiens Expect后面的数字填上0 106、点击网页中最下面的“BLAST!”7、出现新的网页,点击Format!8、等待若干秒之后,出现results of BLAST的网页。
该网页用三种形式来显示blast的结果。
(1)图形格式:图中①代表这些序列与上游引物匹配、并与下游引物互补的得分值都位于40~50分图中②代表这些序列与上游引物匹配的得分值位于40~50分,而与下游引物不互补图中③代表这些序列与下游引物互补的得分值小于40分,而与上游引物不匹配通过点击相应的bar可以得到匹配情况的详细信息。
(2)结果信息概要:从左到右分别为:A、数据库系列的身份证:点击之后可以获得该序列的信息B、系列的简单描述C、高比值片段对(high-scoring segment pairs, HSP)的字符得分。
按照得分的高低由大到小排列。
得分的计算公式=匹配的碱基×2+0.1。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
1首先把与比对的序列输入或导入下面的query sequence序列框中
2然后点勾选上所比对的项目与显示结果在新窗口中
3在新窗口中变出现比对的结果图,从上往下依次是Graphic Summary界面,Descriptions界面,Alignments内容界面中query是自己的序列,下面很多细红线条就是库里的同源序列了,根据此可以推测自己的编码序列的大小。
4新窗口中的比对图示下部是Descriptions界面,描述的同源序列
5如果勾选上比对出的几个序列前面的方框,然后点击Alignments Download GenPept Graphics Distance tree of results Multiple alignment等选项,分别会出
现两个序列比对结果,下载所勾选的序列,对应的蛋白序列,基因图示,进化树图像和所有勾选的序列的同源性比对结果图。
