引物设计大全
primer引物设计知识与技巧分析大全

PCR引物设计知识与技巧分析PCR引物设计知识与技巧分析Posted on 30 五月2009 by 柳城,阅读526 简洁版繁體自从1985年Mullis等发明了具有划时代意义的聚合酶链式反应(PCP) 以来,PCR已经成为了分子生物学领域最基本也是最重要的技术手段之一。
然而能否找到一对合适的核苷酸片段作为引物,使其有效地扩增模板DNA序列,无疑决定着PCR的成败。
现在动物遗传育种早已进入了分子时代,在基因水平寻求影响动物遗传表型的新基因突显重要,因此引物设计无疑又成为了寻找新基因的重中之重。
1 引物的设计以及初步筛选引物的设计与初步筛选基本上通过一些分子生物学软件和相关网站来完成的,目前运用软件Primer Premier 5 或美国whitehead 生物医学研究所基因组研究中心在因特网上提供的一款免费在线PCR引物设计程序Primer 3来设计引物,再用软件Oligo 6进行引物评估,就可以初步获得一组比较满意的引物。
但是对于初学者来说,运用软件和程序来设计引物好象无从着手,其实只要我们掌握了引物设计的基本原则和注意事项,所有问题便迎刃而解。
因为无论是软件还是程序,都是以这些基本原则和注意事项为默认标准来进行引物设计的。
所以,我们在进行引物设计的时候大可不必在软件和程序的参数上花费过多的时间来思考,如果没有特殊要求我们完全可以把一些参数设为默认值。
下面我们主要讨论一下引物设计的原则和注意事项。
①引物的长度一般为15-30 bp,最好在18~24 bp,因为太短易形成错配(False priming) 降低特异性,而太长也会降低特异性,并且降低产量。
②引物在模板内最好具有单一性,也就是说在模板内部没有错配。
特别是3’端,一定要避免连续4个以上的碱基互补错配。
③引物序列的GC 含量最好在40%一60%,且上下游引物序列GC含量的差异不要太大,3’端最后5个碱基最好不要富含GC,特别是连续3个的G或C。
引物设计知识点总结大全

引物设计知识点总结大全引物设计是分子生物学和遗传学研究中的重要环节,它在PCR扩增、基因测序、基因突变检测等实验中起着至关重要的作用。
引物设计的好坏会直接影响实验结果的准确性和可靠性。
下面是引物设计的相关知识点总结:一、引物设计的基本原则1. 引物长度:引物长度通常在18-30个碱基对之间,过短的引物容易产生非特异性扩增,过长的引物则会降低扩增效率。
2. 碱基组成:引物的碱基组成应尽量避免多聚腺嘌呤或多聚胸腺嘧啶的情况,以免引起扩增效率降低或引物间的二聚体形成。
3. Tm值匹配:引物的熔解温度(Tm值)应尽量匹配,以确保二聚体形成和特异性扩增。
4. 特异性:引物的特异性是引物设计的关键,必须确保引物只会与待扩增的目标DNA序列特异结合,而不与其他非目标DNA序列结合。
5. 避免互补二聚体:引物之间的互补二聚体会导致扩增效率降低或产生非特异性扩增,因此需要避免引物之间的互补结构。
二、引物设计的工具和方法1. 序列分析工具:常用的序列分析工具包括NCBI的BLAST、Primer-BLAST、OligoAnalyzer等,通过这些工具可以评估引物的特异性和二聚体形成潜力。
2. 引物设计软件:引物设计软件可以根据目标序列自动生成合适的引物序列,在设计引物过程中考虑多个因素如Tm值、长度、特异性等。
常用的软件有Primer3、Primer Premier等。
3. 引物设计策略:根据实验需求选择合适的引物设计策略,常用的策略包括温度梯度扩增引物设计、Asymmetrical PCR引物设计、引物修饰等。
三、引物设计中的注意事项1. 引物位置选择:引物应该选择在目标序列中的稳定区域,避免选择在重复序列、变异位点和剪切酶位点等容易引起问题的区域。
2. 引物长度控制:引物长度的选择需要平衡扩增效率和特异性,过短的引物可能导致扩增效率降低,过长的引物则可能增加非特异性扩增的风险。
3. 引物设计的平衡性:在设计引物时,需要平衡Tm值的匹配、特异性和二聚体形成的可能性,以达到最佳的扩增效果。
引物设计知识点汇总

引物设计知识点汇总引物设计在分子生物学和遗传学研究中起着至关重要的作用。
引物是用于特异性扩增DNA片段的短寡核苷酸序列,需要准确设计以保证扩增效率和特异性。
本文将汇总引物设计的相关知识点,包括引物选择、引物设计策略和引物设计工具等。
一、引物选择引物的选择是引物设计的第一步,关键点如下:1. 引物长度:一般选择18-22个碱基对的短寡核苷酸作为引物长度,过短的引物可能导致非特异性扩增,过长的引物则增加了扩增难度。
2. 引物的GC含量:引物的GC含量应在40%-60%之间,过高或过低的GC含量可能导致不稳定的引物结合和扩增效率下降。
3. 引物的翻译调谐能力:引物的序列应尽可能避免出现自身互补的二级结构,避免引物之间的相互结合,以保证扩增特异性。
4. 引物的特异性:引物应具有较高的特异性,即只特异性地扩增目标DNA片段,而不扩增非目标DNA。
二、引物设计策略在引物设计过程中,有以下几种常用的引物设计策略:1. 引物序列比对:将引物序列与目标序列比对,选择引物与目标序列高度互补的区域作为引物的设计区域。
2. 引物性能评估:使用引物设计工具对设计的引物进行性能评估,评估指标包括特异性、互补性、剪切位点等。
3. 引物序列调整:根据引物评估的结果,对引物序列进行调整,如调整引物的长度、GC含量等。
4. 引物结构优化:通过调整引物的二级结构和碱基组成,优化引物的稳定性和特异性。
三、引物设计工具引物设计工具可以帮助研究人员快速设计合适的引物,常见的引物设计工具有:1. Primer3:Primer3是一个广泛使用的引物设计软件,提供了丰富的设计选项和参数调整,可以根据用户需求生成高质量的引物序列。
2. NCBI Primer-BLAST:NCBI Primer-BLAST结合了引物设计和引物特异性评估的功能,能够为用户提供特异性较高的引物设计方案。
3. OligoAnalyzer:OligoAnalyzer是一款在线工具,可以评估引物的各项性质,如熔解温度、稳定性等。
pcr引物设计序列例题

PCR引物设计序列例题简介P C R(聚合酶链式反应)是一种常用的分子生物学技术,通过扩增DN A 分子可用于基因克隆、检测等多个领域。
而P CR引物则是PCR反应中的关键组成部分,它们的设计直接影响P CR反应的成功与否。
本文为您提供一些关于P CR引物设计序列的例题,希望对您在实验和研究中的引物设计有所帮助。
例题一:引物序列设计为了扩增目标基因的特定片段,我们需要设计一对引物,以下是目标基因的序列,请根据该序列设计两个引物,使其具有以下要求:-引物长度在18-25个碱基对之间-引物的3'端碱基G或C-引物的理论Tm在55-65℃之间目标基因序列:A G TC GA TC GA TT AG CGA T CG AG TC GA TC GA TCG G CT AC GA TC GA解答:根据目标基因的序列,我们可以设计如下两对引物:引物1:5'-A GT CG AT CG AT TAG C GA TC-3'引物1满足引物长度在18-25个碱基对之间的要求,同时3'端碱基为C,符合要求。
接下来我们计算一下该引物的理论T m。
根据以下两个规则计算引物的理论Tm:-A与T之间的配对带来的热能释放为-2.2k J/mo l-C与G之间的配对带来的热能释放为-3.8k J/mo l根据以上规则,我们可以计算每个碱基的贡献:-A-T配对:-2.2k J/m o l-C-G配对:-3.8k J/m o l-G-C配对:-3.8k J/m o l-T-A配对:-2.2k J/m o l引物1的理论T m计算如下:(-3.8×4)+(-2.2×10)=-15.2+-22=-37.2k J/mo l该引物的理论Tm为37.2℃,低于所需的要求。
引物2:5'-C GA TC GA TC GA TCG G CT AC G-3'引物2满足引物长度在18-25个碱基对之间的要求,同时3'端碱基为G,符合要求。
引物设计专题
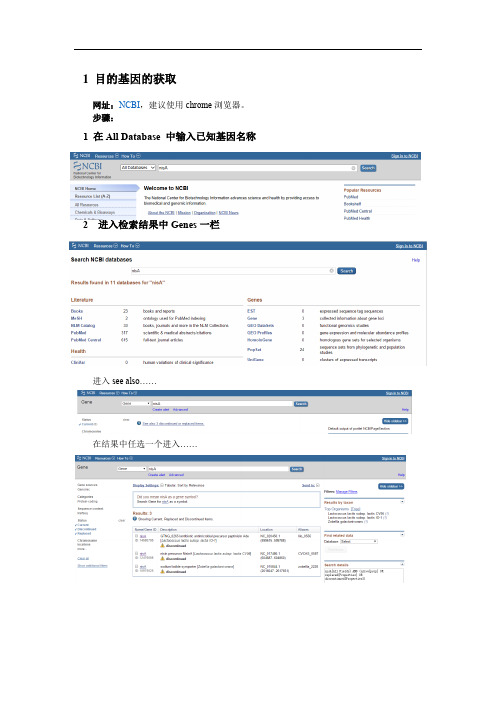
1 目的基因的获取网址:NCBI,建议使用chrome浏览器。
步骤:1 在All Database 中输入已知基因名称2 进入检索结果中Genes一栏进入see also……在结果中任选一个进入……3 找目的基因序列下拉至页面下端,进入genebank或fasta新页面下拉到底部出现目的基因序列4 若要查找该微生物中其他序列则将该生物体的名称复制粘贴,并在genome数据库中查找,得出如下结果:下拉至底部,在dendrogram中找到该微生物。
进入新页面,下拉到底部,进入如下Genome region即可得到该微生物的全基因序列。
Ctrl+F 可快速查找所需目的基因。
2 质粒上酶切位点的选择软件:Snapgene步骤:1.选定质粒载体,搜索得到质粒图谱,找出质粒的MCS(多克隆位点)区域及酶切位点。
2.用snapgene分析目的基因序列,剔除部分酶切位点。
3.结合图谱选择较短位点。
即酶切位点前端的转录翻译部分越短越好,以免翻译出的蛋白质过多影响目的蛋白的性质。
例:pET-28a中MCS酶切位点NcoI/NdeI/NheI/BamHI/EcoRI/SacI/SalI/HindIII/EagI/NotI/XhoI nisA 基因序列中经snapgene含有SacI酶切位点。
则酶切位点就应在除SacI以为的其他酶切位点中选择。
3 引物设计引物格式:5-保护碱基+ 酶切位点+ 上下游引物-3①保护碱基提高酶切时的活性,使得酶切酶更容易结合,酶切更易进行。
①酶切位点第2布中选择的酶切位点。
①上下游引物上下游引物需要用primer5来设计。
3.1 上下游引物设计软件:Primer premier 5 预读:PCR引物设计及软件使用前期:在NCBI找到自己所需的gene sequence,选择ORF操作:New→DNA sequence→复制序列→primer→S/A(正反引物) →Edit→复制粘贴所选序列(S-As is/A-Complement)→analysis →prime→分析参数(GC含量/Tm/Rating)→分析错误(Hairpin发夹结构/Diner二聚体/False错误引发)→OK保存。
引物设计(特别有用,良心推荐)

引物设计(特别有用,良心推荐)引物篇1.引物是如何合成的?目前引物合成基本采用固相亚磷酰胺三酯法。
DNA合成仪有很多种, 主要都是由ABI/PE 公司生产,无论采用什么机器合成,合成的原理都相同,主要差别在于合成产率的高低,试剂消耗量的不同和单个循环用时的多少。
亚磷酰胺三酯法合成DNA片段,具有高效、快速的偶联以及起始反应物比较稳定的特点。
亚磷酰胺三酯法是将DNA固定在固相载体上完成DNA链的合成的,合成的方向是由待合成引物的3'端向5'端合成的,相邻的核苷酸通过3'→5'磷酸二酯键连接。
第一步是将预先连接在固相载体CPG上的活性基团被保护的核苷酸与三氯乙酸反应,脱去其5'-羟基的保护基团DMT,获得游离的5'-羟基;第二步,合成DNA的原料,亚磷酰胺保护核苷酸单体,与活化剂四氮唑混合,得到核苷亚磷酸活化中间体,它的3'端被活化,5'-羟基仍然被DMT保护,与溶液中游离的5'-羟基发生缩合反应。
第三步,带帽(capping)反应,缩合反应中可能有极少数5'-羟基没有参加反应(少于2%),用乙酸酐和1-甲基咪唑终止其后继续发生反应,这种短片段可以在纯化时分离掉。
第四步,在氧化剂碘的作用下,亚磷酰形式转变为更稳定的磷酸三酯。
经过以上四个步骤,一个脱氧核苷酸被连接到固相载体的核苷酸上。
再以三氯乙酸脱去它的5'-羟基上的保护基团DMT,重复以上步骤,直到所有要求合成的碱基被接上去。
合成过程中可以观察TCA处理阶段的颜色判定合成效率。
通过氨水高温处理,连接在CPG上的引物被切下来,通过OPC, PAGE等手段纯化引物,成品引物用C18浓缩,脱盐,沉淀。
沉淀后的引物用水悬浮,测定OD260定量,根据定单要求分装。
2.引物纯化方式有哪些,如何选择?◆C18柱脱盐:有人称其为简易反相柱,它对DNA有特异性的吸附,可以被有机溶解洗脱,但不会被水洗脱,所以能有效地去除盐分。
引物设计知识点归纳图表

引物设计知识点归纳图表引物设计是一项重要的实验技术,在分子生物学、基因工程和遗传学等领域中具有广泛的应用。
合理设计和选择引物能够保证实验结果的准确性和可靠性。
本文将对引物设计的各个知识点进行归纳总结,以图表的形式展示,方便读者理解和应用。
1. 引物的长度长度是影响引物特异性和扩增效率的重要因素,合理选择引物长度能够提高PCR扩增的特异性和效率。
根据目标序列的特点,引物长度的选择范围一般为18-30个碱基对。
2. 引物的GC含量引物的GC含量对扩增的特异性和效率有重要影响,GC含量过高或过低都会影响PCR反应的结果。
通常可以选择引物的GC含量在40% - 60%之间,以提高PCR反应的成功率。
3. 引物的熔解温度(Tm值)引物的熔解温度是指引物在PCR反应中解链的温度,它与引物的碱基对组成、长度和浓度等因素相关。
合理选择引物的Tm值能够提高PCR的效率和特异性。
一般来说,两个引物的Tm值应该接近,以保证二者同时发挥作用。
4. 引物的互补性引物之间的互补性会导致二聚体的形成,影响扩增效果。
因此,设计引物时需要避免引物之间的互补性,以免产生非特异性扩增产物。
5. 引物的特异性引物的特异性是指引物只与目标序列互补而不与其他非目标序列互补。
特异性的引物设计可以通过使用比对软件进行序列比对来保证。
6. 引物的交叉反应引物的交叉反应指的是引物与非目标序列发生非特异性扩增。
为了避免引物的交叉反应,可以通过检查引物的互补性和特异性来进行预防。
7. 引物的杂交效率引物的杂交效率会影响PCR扩增的结果,因此合理设计引物的杂交效率可以提高PCR反应的特异性和效率。
杂交效率可以通过计算引物的形成结构和碱基对数目来预测。
总结:引物设计是PCR技术中至关重要的一步,合理的引物设计能够保证PCR扩增的有效性和特异性。
在设计引物时需要考虑引物长度、GC 含量、熔解温度、互补性、特异性、交叉反应和杂交效率等因素。
通过合理选择和设计引物,可以最大程度地提高PCR扩增的准确性和可靠性。
引物设计知识点归纳总结

引物设计知识点归纳总结引物设计是生物学和生物技术领域中非常重要的一个环节。
在分子生物学研究、基因工程、医学诊断、疾病预防等方面,引物设计都起着至关重要的作用。
引物设计的好坏直接影响到PCR扩增、序列特异性分析、RNA干扰、原位杂交、基因克隆等实验的效果和结果。
因此,深入理解引物设计的相关知识点对于提高实验效率和结果的可靠性非常重要。
本文将介绍引物设计的相关知识点,并对其进行归纳总结。
1. 引物设计的基本原则引物是在分子生物学实验中用于对特定DNA或RNA序列进行扩增、检测或特异性结合的人工合成的寡核苷酸序列。
设计好的引物对于实验的成功至关重要,而引物设计的基本原则包括:(1) 引物长度:引物长度一般在18-25碱基对之间,过短的引物可能导致扩增效率低,而过长的引物可能导致特异性差。
(2) GC含量:引物的GC含量应该在40%-60%之间,过高或过低的GC含量会影响引物的结合性能和特异性。
(3) 引物序列选择:引物的序列选择要尽量避免重复序列、近似重复序列和具有高度变异性的区域。
(4) 引物特异性:引物的特异性是指引物能够与目标序列特异性结合,而不结合其他非特异序列,引物特异性的好坏直接影响到实验结果的准确性和可靠性。
2. 引物设计的常见问题及解决方法在引物设计过程中,常常会遇到一些常见问题,例如引物特异性不足、引物二聚体形成、引物偏向性扩增等问题。
针对这些问题,有一些解决方法可以参考:(1) 引物特异性不足:可以通过生物信息软件对引物进行预测和评估,合理选择引物序列,避免与非特异序列发生交叉杂交。
(2) 引物二聚体形成:引物二聚体是指引物之间相互结合形成二聚体,导致引物的扩增效率降低。
可以通过调整引物长度和序列,以及优化PCR扩增条件来避免引物二聚体的形成。
(3) 引物偏向性扩增:引物偏向性扩增是指引物对特定序列的扩增效率高于其他序列,可以通过优化PCR扩增条件,如调整引物浓度、反应体系等来解决。
引物设计知识点总结图解

引物设计知识点总结图解引物设计是分子生物学研究中至关重要的一步,它涉及到DNA/RNA的扩增、测序和定量等诸多实验。
本文将通过图解的方式,对引物设计的相关知识点进行总结。
具体涉及引物设计的基本原则、引物长度、引物序列的选择、引物的特异性、引物的GC含量以及二聚体的形成。
希望通过本文的阐述,读者能够更好地理解引物设计的要点和注意事项。
1. 引物设计的基本原则引物设计的基本原则包括:引物长度适当、引物序列具有特异性、引物的理论特性符合要求、引物的二聚体形成能力低等。
2. 引物长度的选择引物长度一般在18-30个碱基对之间,过短的引物容易出现非特异性扩增产物,而过长的引物则可能导致扩增效率降低。
因此,在设计引物时需要注意选择适当的长度。
3. 引物序列的选择引物序列的选择是引物设计过程中的核心步骤。
引物的序列应在目标区域能够满足特异性,避免与非目标区域有较高的同源性。
此外,引物序列的碱基组合应尽量避免存在重复序列、多聚碱基或者易形成二聚体的碱基组合。
4. 引物的特异性引物的特异性是评价引物设计质量的重要指标之一。
合理设计的引物应能够特异性地与目标DNA/RNA序列配对,并能够排除与非目标序列的配对。
特异性的引物可以有效地避免非特异性扩增产物的生成。
5. 引物的GC含量引物的GC含量对扩增反应的效率和特异性具有重要影响。
过高或过低的GC含量均可能导致扩增效率降低或产生非特异性扩增产物。
因此,在设计引物时需要合理调整引物的GC含量,并确保GC含量在适宜的范围内。
6. 引物的二聚体形成引物的二聚体形成是指引物之间可能发生的相互结合。
合理设计的引物应该不能形成稳定的二聚体,以避免引物在扩增反应中发生错误的配对,导致产物的异常。
通过上述的图解,我们可以清晰地了解引物设计的主要知识点和注意事项。
在实际的引物设计过程中,需要根据具体的实验目的和条件选择合适的引物设计策略,并结合生物信息学工具进行引物序列的设计和评估。
引物设计实例分析(多图)
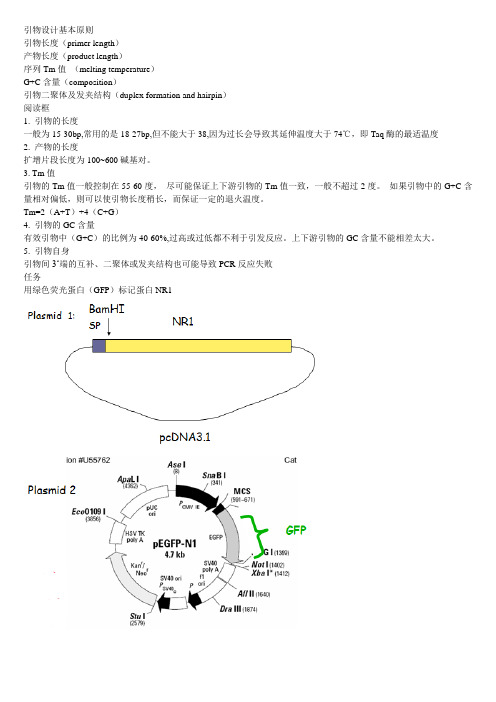
引物设计基本原则引物长度(primer length)产物长度(product length)序列Tm值(melting temperature)G+C含量(composition)引物二聚体及发夹结构(duplex formation and hairpin)阅读框1. 引物的长度一般为15-30bp,常用的是18-27bp,但不能大于38,因为过长会导致其延伸温度大于74℃,即Taq酶的最适温度2. 产物的长度扩增片段长度为100~600碱基对。
3. Tm值引物的Tm值一般控制在55-60度,尽可能保证上下游引物的Tm值一致,一般不超过2度。
如果引物中的G+C含量相对偏低,则可以使引物长度稍长,而保证一定的退火温度。
Tm=2(A+T)+4(C+G)4. 引物的GC含量有效引物中(G+C)的比例为40-60%,过高或过低都不利于引发反应。
上下游引物的GC含量不能相差太大。
5. 引物自身引物间3‘端的互补、二聚体或发夹结构也可能导致PCR反应失败任务用绿色荧光蛋白(GFP)标记蛋白NR1引物要求PCR扩增GFPGFP两边添加BamHI酶切位点保证NR1的阅读框不改变第一步:扩增GFP基本序列第二步:GC比值;Tm值第三步:酶切位点第四步:阅读框第五步保护序列Primer1: 5' GCGGggatccTATGGTGAGCAAGGGCGAGGA Primer2: 5' GCGCggatccctCTTGTACAGCTCGTCCA TGCC记得当初写本科论文,感到不知道讨论什么问题好。
愣是写了一大段的PCR条件摸索的讨论。
后来PCR成为实验最基本的一步了,但是发现在PCR中还是有许多需要注意的地方。
PCR的第一步就是引物设计了。
引物设计需要注意的地方很多,在大多数情况下,我们都是在知道已知模板序列时进行PCR扩增的。
在某些情况比如构建文库的时候也会在不知道模板序列的情况下进行设计。
这个时候随机核苷酸序列就与模板不是完全匹配。
引物设计知识点总结大全

引物设计知识点总结大全引物设计是分子生物学研究中的重要工具,主要用于PCR、测序、微阵列等分子生物学实验中。
引物设计的好坏直接关系到实验结果的准确性和可重复性。
因此,掌握引物设计的相关知识对于开展分子生物学研究具有重要意义。
下面就引物设计的相关知识点进行总结和归纳。
1. 引物的定义和作用引物是指在PCR、RT-PCR、测序等分子生物学实验中所使用的短链DNA或RNA分子,其作用是按照特定序列在模板核酸上引发聚合酶链反应,从而在实验中扩增目标DNA片段。
引物的设计需要遵循一定的规则,包括选择合适的长度、GC含量、Tm值等。
引物设计的好坏直接关系到PCR反应的效果和准确性。
2. 引物的选择在引物设计中,需要根据不同的实验目的和要扩增的目标DNA/RNA片段的知识选择合适的引物。
常见的引物包括前向引物、反向引物、探针引物等。
前向引物和反向引物通常用于PCR扩增,探针引物通常用于实时荧光定量PCR等实验。
3. 引物设计的要求在引物设计过程中,需要满足一定的要求,包括引物的长度、GC含量、Tm值、特异性和避免形成二聚体等。
引物的长度一般在15-25个碱基对之间,GC含量在40%-60%之间,Tm值在55-65摄氏度之间,具有特异性,避免形成二聚体。
4. 引物设计的工具目前,有许多在线工具可以帮助科研人员进行引物设计,如NCBI Primer-BLAST、IDT PrimerQuest、UCSC in silico PCR、Primer3等。
这些工具可以根据用户提供的信息自动设计出符合要求的引物。
5. 引物设计注意事项在引物设计过程中需要注意一些问题,如避免引物之间的二聚体形成、避免引物的自身二聚体等。
此外,还需要考虑引物在实验中可能遇到的特殊情况,如引物的结合位置、引物的特异性等。
6. 引物设计的优化为了得到更好的实验效果,有时候需要对引物进行优化。
这包括对引物序列的修饰、引物浓度的优化等。
通过对引物设计的优化,可以提高PCR扩增的特异性、准确性和效率。
引物的设计及修饰最全教程

引物的设计及修饰最全教程引物的设计及修饰1. 引物设计的基本原则是什么?引物设计的下列原则供您参考:1) 引物最好在模板cDNA的保守区内设计。
2) 引物长度⼀般在15-30碱基之间。
3) 引物GC含量在40%-60%之间,Tm值最好接近72℃。
4) 引物3′端要避开密码⼦的第3位。
5) 引物3′端不能选择A,最好选择T。
6) 碱基要随机分布。
7) 引物⾃⾝及引物之间不应存在互补序列。
8) 引物5′端和中间△G值应该相对较⾼,⽽3′端△G值较低。
9) 引物的5′端可以修饰,⽽3′端不可修饰。
10) 扩增产物的单链不能形成⼆级结构。
11) 引物应具有特异性。
2. 常⽤引物设计软件有哪些?常⽤的软件有Oligo 6和Primer Premier 5.0。
引物设计软件是根据引物设计的指导意见设计⽽成。
其实,PCR扩增的成败最关键的是反应模板的制备和反应条件的控制。
引物设计软件的缺点是,有时判断为该基因没有⼀段区域满⾜标准引物的要求。
⾦斯瑞为您提供以下引物设计相关软件:引物计算⼯具引物设计⼯具测序引物设计软件Real-time PCR 引物设计软件3. ⽂献上找到的引物和探针序列能否直接使⽤?通常国外的⽂献可信度⽐较⾼,可直接使⽤;但为了保险起见,最好⽤blast对引物探针的序列进⾏必要的验证;或者再进⼀步⽤引物设计软件对引物探针的⼆级结构和退⽕温度进⾏分析,这样更有利于您对整个实验的把握。
4. 如何计算引物的Tm值?Tm值的概念:DNA熔解温度,指把DNA的双螺旋结构降解⼀半时的温度,亦即DNA 变性过程中,紫外吸收值达到最⼤值的50%时的温度称为 DNA 的解链温度(Tm)。
⾦斯瑞采⽤以下⽅法计算Tm值:长度为20mer及以下的引物,Tm计算公式为:Tm = 4℃(G + C)+ 2℃(A + T)。
但这个公式只适⽤于14~20个碱基的引物,引物的TM值还与引物长度、碱基组成、引物使⽤缓冲溶液的离⼦强度等有关。
PCR引物设计汇总

PCR引物设计汇总PCR(聚合酶链反应)引物是PCR反应中的两个核酸序列,它们分别位于待扩增的DNA片段的两端。
合理设计的PCR引物是PCR反应成功的关键,它们决定了PCR扩增的特异性和效率。
1.引物长度:一般选择18-25个碱基的引物长度。
引物过短可能导致非特异性扩增,引物过长则降低扩增效率。
2.引物碱基组成:引物中尽量避免使用连续的同类碱基,如连续的A、T、C或G。
同时,引物设计中应尽量均衡使用四种碱基,避免GC含量过高或过低。
3.引物Tm值:引物的Tm值(解链温度)是很重要的参数,它决定了PCR反应的温度条件。
一般,引物的Tm应在50-60摄氏度之间,且相互之间的Tm值差别不应超过两度。
4.引物特异性:引物应具有足够的特异性,以确保只扩增目标DNA片段,避免扩增到非特异性产物。
5.引物末端:引物的3'末端不应含有碱基修饰物,以免影响引物的扩增效率。
下面是几种常见的PCR引物设计方法:1.传统引物设计方法:传统引物设计方法主要是基于DNA序列的特点进行设计。
根据待扩增DNA片段的序列信息,可以选择合适的引物位置,并确保引物的长度、碱基组成和Tm值满足设计原则。
2.引物设计软件:引物设计软件是根据一系列预先设定的算法和规则,自动设计合适的引物。
常用的引物设计软件有Primer3、Primer-BLAST等。
这些软件可以根据用户输入的目标序列信息,自动生成合适的引物序列,并提供引物的Tm值、特异性等信息。
3.引物库:引物库是包含大量已设计好的引物序列的数据库。
研究人员可以直接从引物库中选择合适的引物序列,以节省时间和精力。
常用的引物库有NCBI的PrimerBank和UCSC的Primer Database。
4.引物修饰:5.引物交互作用:引物交互作用是指多对引物之间的交叉杂交,形成二聚体或多聚体结构。
通过设计引物之间的相互作用,可以提高PCR的特异性和扩增效率。
常用的引物交互作用方法有引物交叉互补法、引物竞争法等。
引物设计原则最全汇总

引物设计原则最全汇总1.特异性:引物应与所需扩增的目标序列特异性结合,避免与非目标序列发生非特异性结合,以确保产生准确结果。
2.高GC含量:引物的GC含量应高于50%,以增加引物与目标序列的稳定性和特异性。
3.避免酶切位点:在引物设计过程中,应避免引物与目标序列中的酶切位点重叠,以防止扩增产物的酶切降解。
4.引物长度:引物的长度通常在18至30个核苷酸之间,过长的引物会降低特异性,而过短的引物则可能导致非特异性扩增。
5.引物的Tm值匹配:引物的熔解温度(Tm)应在同一PCR反应中保持一致,以确保引物能同时结合于目标序列并发挥作用。
6.避免互补性:在引物设计过程中,应避免引物之间存在互相互补的情况,以防止互补引物之间的杂交,从而导致错误的扩增结果。
7.引物末端修饰:常用的引物末端修饰包括磷酸化、末端标记和引物的截断,通过这些修饰可以提高引物的选择性和特异性。
8.引物的GC平衡:引物的GC含量应在一定范围内均衡,以避免在PCR反应中产生二聚体或无效的扩增。
9.引物序列的重复性:引物设计中应避免引物序列的重复性,以防止引物产生二聚体或与非目标序列互补结合。
10.引物的独特性:在引物设计中,应确保引物序列在目标基因组中的唯一性,避免与非目标序列存在相似区域。
11.引物的结合位点:引物的结合位点应尽可能位于目标序列的保守区域,以增加引物与目标序列的稳定性和特异性。
12.引物的交叉反应:在引物设计中,应避免引物之间存在交叉反应,即两个不同引物同时与同一目标序列结合。
13.引物与模板序列的一致性:在引物设计过程中,应将引物与目标序列进行比对,确保引物与目标序列的一致性,避免在扩增过程中形成不可扩增的结构。
14.避免自相互补性:在引物设计过程中,应避免引物序列存在自相互补性,防止引物自结合或形成不稳定的结构。
15.引物的GC间隔:在引物设计中,应使引物中的GC核苷酸尽可能均匀分布,以避免形成不稳定的结构。
16.引物的无副产物性:在引物设计过程中,应避免引物产生具有毒性或干扰扩增的副产物。
培训一 各种引物设计

2.GFP+基因蛋白融合。 GFP终止密码子去除。
EcoRI (2)
Terminator 1 Promoter P 2
AvaI (1 1 668 ) ApaLI (11 354)
NosT-S eqR Nos-E coR
BamHI (278 ) BamHI (28 6)
hptII
ApaLI (11 052) AvaI (1 0575)
基因定义
基因组DNA
RNA提取
cDNA
1.家族基因。 是指具有相同功能的,不同基因。
2.转录本。 是指一个基因有两个不同的转录结果。都来自于一个基因,由于选择性 剪切导致的。
基因扩增PCR
体系:酶,缓冲液Buffer, dNTP,MgCl2,引物F, R。Forward, Re
F引物
R引物
流程
YFP
Ps tI (8 06) Nco (1 00 8 ) I HindIII (1 047)
Misc Feature 5 Misc Feature 4
aadA
ApaLI (8 657)
pCXSN LysM2356-YFP
1 2696 bp
ApaLI (11 69)
LysM2356
HindIII (1 951 ) BamHI (21 52) EcoRI (2701 ) Nco (28 59) I BamHI (28 8 3) AvaI (28 91 )
1.模板准备,引物准备 2.PCR扩增 3.片段回收 4.连接TA克隆 5.转化大肠杆菌 6.测序,确定序列 7.提质粒,保存菌液(15%-30%甘油)
序列寻找
exocytosis
Cs3g16120
参考基因组网站:甜橙(Citrus sinensis)。 /cgi-bin/orange/search 柑橘菌根模式:枳壳(Poncirus trif.),做为砧木,使用范围最广,抗寒。 苜蓿基因组: /cgi-bin/medicago/overview.cgi。 NCBI:/ 文献搜索:/schhp?hl=zh-CN 基因研究情况。
引物设计原则最全汇总

引物设计原则(汇总)普通引物设计(适用于从载体上扩增模板):1.普通引物长度一般在20-30bp之间,常用24-28bp左右以保证基因特异性;2.下载基因序列到Vector NTI;3.找到所需安装载体序列;4,将基因序列的CDS高亮标记;5.寻找载体序列中常用酶切位点,一般为EcoRI、BamHI、Hindlll、XhoI等等,比对检测基因序列中是否有这些位点,有的话舍弃,最后选择两个酶切位点,最好离得远一点,并且最好buffer用一样的。
酶切位点一般是6bp的回文序列;6.从基因ATG开始往后选择10-20bp均可(我的习惯是27bp-6bp酶切位点-2bp保护碱基-xbp 补齐序列),但最好保证最后两个是G或者C,以减少错配率;7.将上游酶切位点序列补在ATG前方,并根据载体对框情况补足两者之间的空缺,再根据序列的GC含量和TM值在酶切位点前补足保护碱基,以保证GC和AT的含量不能过高。
注意,所有的补齐不能用到终止密码子;8.检测上游序列的结构情况,理论上不要太多二级结构以及3’端匹配即可;不过重复的序列也不能太多,以免移码;9.从下游终止密码子开始向前选择10-20bp均可,但最好保证最后两个是G或者C,以减少错配率;10.选择complementary sequence,在N端补齐下游酶切位点,如果tag在C端(即下游),则在第9点中应该从终止密码子前开始选择(即舍弃终止密码子),并且下游引物也要对框,如果tag在N端,则下游引物不需要对框,只要在N端加上下游酶切位点,再根据情况加上2个保护碱基,然后检测二级结构,原则上3’端部匹配即可。
不过重复的序列也不能太多,以免移码;11.将设计好的上下游引物放在一起检测二级结构,原则上3’端部匹配即可。
不过重复的序列也不能太多,以免移码;12.最后在NCBI的primer Blast网站上比对引物序列,看是否基因特异性的。
2011年10月18日左洁1.引物的长度一般为15-30 bp,常用的是18-27 bp,但不应大于38,因为过长会导致其延伸温度大于74℃,不适于Taq DNA聚合酶进行反应。
引物设计知识点归纳

引物设计知识点归纳引物设计是分子生物学研究中的重要环节,它直接影响到PCR扩增反应的效果和准确性。
对于进行引物设计的研究人员来说,掌握相关的知识点非常重要。
本文将对引物设计的相关知识进行归纳总结,以帮助读者更好地理解和应用。
一、引物的选择1. 引物长度:引物通常由18-25个碱基组成,长度不宜过短也不宜过长。
过短的引物可能导致非特异性扩增,过长的引物则可能影响扩增效率。
2. Tm值:引物的熔解温度(Tm)是指引物与目标序列杂交时熔解的温度。
Tm值的选择应使引物在PCR反应温度下能够与目标序列准确结合。
3. 引物碱基组成:引物的碱基组成应均衡分布,避免多个相邻碱基相同或相似的情况,以防止非特异性扩增的发生。
4. GC含量:引物中的GC含量影响其稳定性和特异性。
一般而言,GC含量在40-60%之间效果较好。
5. 避免互补:引物之间应避免出现互补的情况,以免形成二聚体或产生非特异性扩增。
二、引物设计工具1. Primer3:Primer3是一个常用的引物设计工具,提供了包括引物长度、Tm值和GC含量等参数的设定。
2. OligoAnalyzer:OligoAnalyzer是一款用于计算引物Tm值的工具,可以通过输入引物序列来预测其熔解温度。
3. BLAST:BLAST是一种用于在数据库中搜索引物靶点的工具,可以帮助判断引物的特异性。
4. UCSC in silico PCR:UCSC in silico PCR是一个在线工具,可以通过输入引物序列和目标序列来模拟PCR反应,判断引物的效果和特异性。
三、引物设计的注意事项1. 避免引物间二聚体:引物之间的互补性可能导致二聚体的形成,影响PCR反应的特异性和效果。
使用引物设计工具进行分析,避免引物间的互补性。
2. 引物特异性:引物应具有良好的特异性,即只与目标序列匹配,不与非目标序列杂交。
3. 引物位置选择:引物的选择应尽量位于目标序列的保守区域,以提高特异性和稳定性。
引物设计大全

primer premier为主,primer express为辅(针对探针),Oligo来验证,BLAST随时检验PCR引物设计原则PCR引物设计的目的是为了找到一对合适的核苷酸片段,使其能有效地扩增模板DNA序列。
因此,引物的优劣直接关系到PCR的特异性与成功与否。
要设计引物首先要找到DNA序列的保守区。
同时应预测将要扩增的片段单链是否形成二级结构。
如这个区域单链能形成二级结构,就要避开它。
如这一段不能形成二级结构,那就可以在这一区域设计引物。
现在可以在这一保守区域里设计一对引物。
一般引物长度为15~30碱基,扩增片段长度为100~600碱基对。
让我们先看看P1引物。
一般引物序列中G+C含量一般为40%~60%。
而且四种碱基的分布最好随机。
不要有聚嘌呤或聚嘧啶存在。
否则P1引物设计的就不合理。
应重新寻找区域设计引物。
同时引物之间也不能有互补性,一般一对引物间不应多于4个连续碱基的互补。
引物确定以后,可以对引物进行必要的修饰,例如可以在引物的5′端加酶切位点序列;标记生物素、荧光素、地高辛等,这对扩增的特异性影响不大。
但3′端绝对不能进行任何修饰,因为引物的延伸是从3′端开始的。
这里还需提醒的是3′端不要终止于密码子的第3位,因为密码子第3位易发生简并,会影响扩增的特异性与效率。
综上所述我们可以归纳十条PCR引物的设计原则:① 引物应用核酸系列保守区内设计并具有特异性。
② 产物不能形成二级结构。
③ 引物长度一般在15~30碱基之间。
④ G+C含量在40%~60%之间。
⑤ 碱基要随机分布。
⑥ 引物自身不能有连续4个碱基的互补。
⑦ 引物之间不能有连续4个碱基的互补。
⑧ 引物5′端可以修饰。
⑨ 引物3′端不可修饰。
⑩ 引物3′端要避开密码子的第3位。
PCR引物设计的目的是找到一对合适的核苷酸片段,使其能有效地扩增模板DNA序列。
如前述,引物的优劣直接关系到PCR的特异性与成功与否。
对引物的设计不可能有一种包罗万象的规则确保PCR的成功,但遵循某些原则,则有助于引物的设计。
引物设计大全

引物设计和Primer-BLAST的应用Lv Peng2015.11.18CONTENT1.PCR-引物设计目的2.引物设计原则3.设计引物软件4.在线设计工具5.probeBase 简介1.1PCR(Polymerase Chain Reaction)聚合酶链式反应1971 Khorana 提出设想1985Kary Mullis发明了PCR1986年5月Mullis在冷泉港实验室做专题报告冷泉港实验室(The Cold Spring Harbor Laboratory,缩写CSHL),又译为科尔德斯普林实验室。
几不同的PCR技术1.扩增已知序列两侧DNA的PCR:反向PCR(Inverse PCR,IPCR)、锚定PCR(anchored PCR)、RACE(Rapid Amplification of cDNA Ends)、连接介导的PCR(ligation-mediated PCR,LM-PCR);2.检测有限量稀有靶序列,即一对引物扩增产物不足以以通过凝胶电泳观察到的时:巢式PCR(nested PCR);3.快速、灵敏、特异而准确定量的PCR:实时荧光定量PCR (real-time quantitative PCR,RQ-PCR)。
特性优化碱基组成(G+C )含量应在40%-60%,4种碱基要分布均匀;长度一般为18-27个核苷酸长度。
上下游引物长度差别不能大于3bp ;重复和自身互补序列不能有大于3bp 的反向重复序列或自身互补序列存在;上下游引物互补性一个引物的3’末端序列不能结合到另一个引物的任何位点上;解链温度(Tm )两个引物的Tm 值相差不能大于5℃,扩增产物与引物的Tm 值相差不能大于10℃3’末端引物3’末端碱基尽量为G 或C ,不能使3’末端有NNGC 或NNCG 序列引物序列不要有局部的GC rich 或AT rich (特别是3’端),避开T/C 或A/G 的连续结构1.2引物设计原则引物特性及优化设计Primer Premier 6(P6)、Beacon Designer 8(DB )(自动搜索)Oligo 6(引物评价)Vector NTI Suit (综合分析)Primer Express (实时定量PCR 引物和探针设计)*Omiga *Dnastar1.3常用引物设计软件1.3.1 Primer 6和BD8设计引物参数•斑马鱼基因serpine1:抑制血管生成和新陈代谢•NCBI Reference Sequence: NM_001114559.1Primer Premier 6(PP6) 参数Tm:57℃+2℃Length:18+27 bpAmplicon Length:80—200 bp(G+C)%=40%—60%Beacon Designer 8(BD8)参数Tm:66℃+2℃Length:18+27 bpAmplicon Length:80—200 bp(G+C)%=40%—60%1.3.2 Beacon Designer 8(BD主界面)功能选择区序列碱基详细信息窗口序列基本信息引物特征其他信息所有引物和结构选择设计结构选项引物显示区(1)序列导入(2)参数选择(3)结果分析(4)引物检验序列名称序列长度1789mRNA 碱基序列(1)序列导入(2)参数选择(3)结果分析(4)引物检验基本参数高级参数引物Tm 值引物长度产物片段长度GC 含量ΔG 值:DNA 双链形成时所需的自由能,反映了双链结构内部碱基对的相对稳定性。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
引物设计和Primer-BLAST的应用Lv Peng2015.11.18CONTENT1.PCR-引物设计目的2.引物设计原则3.设计引物软件4.在线设计工具5.probeBase 简介1.1PCR(Polymerase Chain Reaction)聚合酶链式反应1971 Khorana 提出设想1985Kary Mullis发明了PCR1986年5月Mullis在冷泉港实验室做专题报告冷泉港实验室(The Cold Spring Harbor Laboratory,缩写CSHL),又译为科尔德斯普林实验室。
几不同的PCR技术1.扩增已知序列两侧DNA的PCR:反向PCR(Inverse PCR,IPCR)、锚定PCR(anchored PCR)、RACE(Rapid Amplification of cDNA Ends)、连接介导的PCR(ligation-mediated PCR,LM-PCR);2.检测有限量稀有靶序列,即一对引物扩增产物不足以以通过凝胶电泳观察到的时:巢式PCR(nested PCR);3.快速、灵敏、特异而准确定量的PCR:实时荧光定量PCR (real-time quantitative PCR,RQ-PCR)。
特性优化碱基组成(G+C )含量应在40%-60%,4种碱基要分布均匀;长度一般为18-27个核苷酸长度。
上下游引物长度差别不能大于3bp ;重复和自身互补序列不能有大于3bp 的反向重复序列或自身互补序列存在;上下游引物互补性一个引物的3’末端序列不能结合到另一个引物的任何位点上;解链温度(Tm )两个引物的Tm 值相差不能大于5℃,扩增产物与引物的Tm 值相差不能大于10℃3’末端引物3’末端碱基尽量为G 或C ,不能使3’末端有NNGC 或NNCG 序列引物序列不要有局部的GC rich 或AT rich (特别是3’端),避开T/C 或A/G 的连续结构1.2引物设计原则引物特性及优化设计Primer Premier 6(P6)、Beacon Designer 8(DB )(自动搜索)Oligo 6(引物评价)Vector NTI Suit (综合分析)Primer Express (实时定量PCR 引物和探针设计)*Omiga *Dnastar1.3常用引物设计软件1.3.1 Primer 6和BD8设计引物参数•斑马鱼基因serpine1:抑制血管生成和新陈代谢•NCBI Reference Sequence: NM_001114559.1Primer Premier 6(PP6) 参数Tm:57℃+2℃Length:18+27 bpAmplicon Length:80—200 bp(G+C)%=40%—60%Beacon Designer 8(BD8)参数Tm:66℃+2℃Length:18+27 bpAmplicon Length:80—200 bp(G+C)%=40%—60%1.3.2 Beacon Designer 8(BD主界面)功能选择区序列碱基详细信息窗口序列基本信息引物特征其他信息所有引物和结构选择设计结构选项引物显示区(1)序列导入(2)参数选择(3)结果分析(4)引物检验序列名称序列长度1789mRNA 碱基序列(1)序列导入(2)参数选择(3)结果分析(4)引物检验基本参数高级参数引物Tm 值引物长度产物片段长度GC 含量ΔG 值:DNA 双链形成时所需的自由能,反映了双链结构内部碱基对的相对稳定性。
应选择3’端ΔG 值较低(绝对值不超过9),而5’端和中间ΔG 值过高,容易在错配位点形成双链结构并引发DNA 聚合反应。
(能值越高越容易结合)(1)序列导入(2)参数选择(3)结果分析(4)引物检验外显子信息得分长度Tm值GC%(1)序列导入(2)参数选择(3)结果分析(4)引物检验引物二级结构所有引物FP :AGTGAAGATGGAGTGGAGGTAGRP :TCTGGCTGGCTGAAGTCTATC引物得分降序同向引物二聚体发夹结构交叉二聚体连续序列“—AA —”重复序列“—GCGC —”1.4常用在线设计引物工具•/site(Integrated DNA Technologies)•Web Primer:/cgi-bin/web-primer(酵母基因组数据库提供)•Primer Design:/GenWeb/Molecular/seq_anal/primer_design/primer_design.htm(较基础全面)•NCBI Primer-Blast/tools/primer-blast/(引物设计、验证)1.4.1NCBI Primer-Blast 简介Primer-BLAST ,能在线设计引物,并验证设计好的引物。
整合了目前流行的Primer3软件,同时进行NCBI 的Blast 进行引物特异性验证。
Primer-BLAST 免除了用另一个站点或工具设计引物的步骤,设计好的引物直接用Blast 进行引物特异性验证。
并且,Primer-BLAST 能设计出只扩增某一特定剪接变异体基因的引物—这是用来衡量基因在特定组织中特异性表达的重要特征。
Primer-Blast 引物设计引物验证BlastPrimer 31.4.2NCBI Primer-Blast引物设计模版链序列或登录号选择上下游引物的范围已设计引物PCR产物大小引物Tm值The Tm calculation is controlled by Table of thermodynamic parameters and Salt correction formula(under advanced parameters). The default Table of thermodynamic parameters is "SantaLucia1998" and the default Salt correction formula is "SantaLucia1998" as recommended by primer3 program.热力学参数表盐度修正公式外显子连接跨度检索数据库选择√√物种限定增加物种PCR 产物Tm 值引物长度引物GC 含量GC clamp :GC 夹,在一侧引物的5’端加上一个30-40bp 的GC 结构,这样在PCR 产物的一侧可产生一个高熔点区,使相应的感兴趣的序列处于低熔点区而便于分析。
能极大提高突变检出率。
最大单核苷酸聚合体最大3’端稳定性最大3’端GC 含量1.4.3NCBI Primer-Blast引物验证如果你已经设计好了引物,要验证引物的好坏。
可以在Primer-Blast中进行,在Primer Parameters 区填入你的一对引物。
选择好验证的目标据库(在specificity check区选择)。
根据需要可设置产物的大小,Tm值等。
特异性参数选择同前面引物设计1.5 Primer-Blast小结(1)Primer-Blast设计的引物软件设计的引物,经Primer-Blast验证(2)数据库是经过专家注释的数据,这样可以给出更准确的结果。
特别是Tm值、GC含量计算,引物二级结构、引物互补预测;(3)尽量使用没有冗除的数据库(如refseq_rna或genome database),nr数据库包括了太多的冗余的序列,会干扰引物的设计。
http://probebase.csb.univie.ac.at/node/8简介:probeBase is a curated database of rRNA-targeted oligonucleotide probes and primers.probeBase 是一个针对设计rRNA的寡聚核苷酸探针和引物的精选数据库。
主要用于设计rRNA探针和引物。
SearchSearch probeBase for target organisms, probe names,primers, target sites, references, etc.MatchMatch rRNA sequence(s) against probeBase and find all published probes targeting your sequence(s).ProxyMatch partial rRNA sequence(s) against full-lengthsequences in SILVA and find published probes potentiallytargeting your sequence(s).ListsView lists of probes (according to probe categories),primers, microarrays, references, etc. SubmissionSubmit new or missing probes to probeBase.LinksWebsites relevant to ribosomal RNA http://www.arb-silva.de搜索探针和引物匹配rRNA 序列找探针匹配部分rRNA 序列参考列表提交高质量rRNA 数据库1.6 probeBase应用实例(1)菌种鉴定:收集已培养或未培养微生物的16SrRNA序列信息,常用于鉴定新分离的菌株。
probeBase 可用来检索与16S rRNA有关的探针。
(2)原位杂交:FISH 技术成功应用的关键在于设计和获得具有高灵敏性和专一性的寡核苷酸探针以减少干扰,可使用ARB 软件包结合probeBase数据库等多种方法进行探针分析和设计。
1.7 参考文献1.Alexander Loy, Frank Maixner, Michael Wagner and Matthias Horn*(2006)probeBase—anonline resource for rRNA-targeted oligonucleotide probes: new features 2007 ,Nucleic Acids Research, Vol.35, D800–D804 Database issue2.Loy,A., Horn,M. and Wagner,M. (2003) probeBase: an online resourcefor rRNA-targeted oligonucleotideprobes. Nucleic Acids Res.,31, 514–516.3.Jian Ye1*, George Coulouris1 , Irena Zaretskaya1 , Ioana Cutcutache2 , Steve Rozen2 and Thomas LMadden1,Primer-BLAST: A tool to design target-specific primers for polymerase chain reaction, Ye et al.BMC Bioinformatics 2012, 13:1344.刘驰, 李家宝, 芮俊鹏, 安家兴, 李香真.16S rRNA基因在微生物生态学中的应用.生态学报, 2015,35(9):2769-2788附1:反向PCR(Inverse PCR, IPCR)反向PCR可用于研究与已知DNA区段相因此又可称为染色体缓移或染色体步移。
