数据搜索时有用的生物大分子数据库扫描
第二章 生物学数据库及其检索

二级数据库( Secondary database ):在 一级数据库的信息基础上进行计算机加工 处理并增加了许多的人为注释而构成的 (例如:NCBI的RefSeq数据库等)。
Primary vs. Secondary Databases
Curators
Sequencing Centers
Labs
➢ DDBJ的英文版网址: http://www.ddbj.nig.ac.jp/index-e.html/
国际上最权威的核酸序列数据库
日本国立遗传研究所的DDBJ http://www.ddbj.nig.ac.jp/searches-e.html
(二)基因组数据库GDB
• 基因组数据库(GDB)创建于1990年,是一个专门 汇集人类基因组数据的数据库,为人类基因组计 划(HGP)保存和处理基因组图谱数据。
计算机文档,是统一管理的相关数 据的集合,其储存形式有利于数据 信息的检索与调用。
二、生物学数据库
在生物信息学者们的努力下, 人类基因组序列数据连同其它多种 模式生物的序列数据及各自相应的 基因结构与功能信息皆可供众多生 物学家们免费接入与使用。
模式生物
Ureaplasma urealyticum
Chapter 2
第二节 常用数据库
常用数据库
类 序列
型 一次数据库
核 酸
基因组 序列
一次数据库
一次数据库
蛋
白
质
复合数据库
二次数据库
名称 Genebank EMBL DDBJ GDB SWISS-PROT PIR TrEMBL UniProt MIPS
GenPept NRL-3D
NRDB OWL SWISS-PROT+ TrEMBL PROSITE PRINTS BLOCKS Pfam IDENTIFY COGs ProDom
基于生物大数据技术的生物信息学分析工具介绍

基于生物大数据技术的生物信息学分析工具介绍生物信息学是一门综合应用生物学、计算机科学和统计学的交叉学科,旨在研究和理解生物体内的各种生物大分子(例如DNA、RNA和蛋白质)的结构、功能和相互作用。
随着高通量测序技术的发展,生物学实验产生的数据量呈指数级增长,从而催生了生物信息学领域的快速发展。
为了更好地处理和分析这些大规模的生物数据,生物信息学分析工具应运而生。
在本文中,我将介绍几个基于生物大数据技术的生物信息学分析工具。
1. BLAST(Basic Local Alignment Search Tool)BLAST是生物信息学中广泛使用的工具,用于在数据库中搜索生物序列的相似性。
它可以将一个给定的DNA或蛋白质序列与数据库中的其他序列进行比对,从而找到相似的序列。
BLAST可以用于比对已知序列和未知序列之间的相似性,从而帮助解析未知序列的功能和进化关系。
2. Clustal OmegaClustal Omega是一种用于进行多序列比对的工具。
多序列比对是生物信息学中常用的技术,旨在确定多个序列之间的共有保守区域和变异区域。
Clustal Omega使用改进的多序列比对算法,可以高效地处理大规模的序列数据,并生成准确的比对结果。
这些比对结果可以用于研究序列的演化关系、结构域的保守性和功能区域的变异性。
3. PEAKSPEAKS是一种用于蛋白质组学数据分析的软件工具。
它可以从质谱数据中识别和鉴定蛋白质,并预测蛋白质的修饰位点和结构域。
PEAKS提供了多种分析模式和算法,适用于不同类型的质谱数据和生物学问题。
它可以帮助研究人员更好地理解蛋白质的功能和相互作用,在疾病诊断和药物研发方面具有重要的应用价值。
4. DESeq2DESeq2是一种用于差异表达基因分析的统计学工具。
它可以从RNA测序数据中识别和比较不同条件下的差异表达基因。
DESeq2根据数学模型和统计方法,可以准确地判断哪些基因在不同条件下的表达水平存在显著差异。
生物信息学 实验三 数据库搜索-BLAST
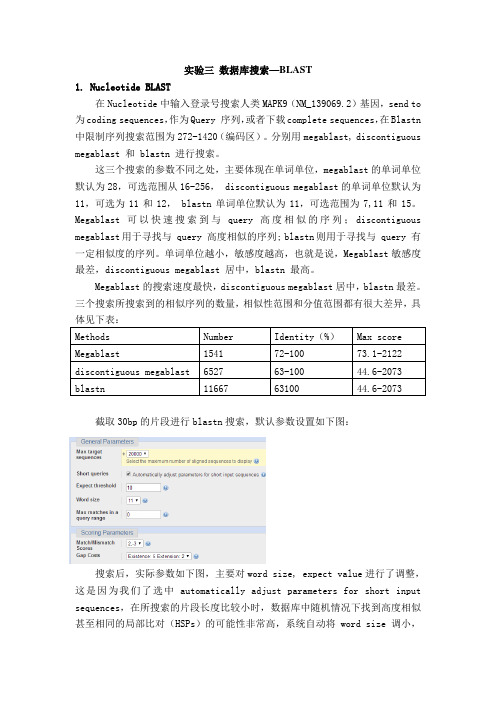
实验三数据库搜索—BLAST1. Nucleotide BLAST在Nucleotide中输入登录号搜索人类MAPK9(NM_139069.2)基因,send to 为coding sequences,作为Query 序列,或者下载complete sequences,在Blastn 中限制序列搜索范围为272-1420(编码区)。
分别用megablast, discontiguous megablast 和 blastn 进行搜索。
这三个搜索的参数不同之处,主要体现在单词单位,megablast的单词单位默认为28,可选范围从16-256, discontiguous megablast的单词单位默认为11,可选为11和12, blastn单词单位默认为11,可选范围为7,11和15。
Megablast 可以快速搜索到与query 高度相似的序列;discontiguous megablast用于寻找与 query 高度相似的序列; blastn则用于寻找与 query 有一定相似度的序列。
单词单位越小,敏感度越高,也就是说,Megablast敏感度最差,discontiguous megablast 居中,blastn 最高。
Megablast的搜索速度最快,discontiguous megablast居中,blastn最差。
三个搜索所搜索到的相似序列的数量,相似性范围和分值范围都有很大差异,具Methods Number Identity(%)Max score Megablast154172-10073.1-2122 discontiguous megablast652763-10044.6-2073 blastn116676310044.6-2073截取30bp的片段进行blastn搜索,默认参数设置如下图:搜索后,实际参数如下图,主要对word size, expect value进行了调整,这是因为我们了选中automatically adjust parameters for short input sequences,在所搜索的片段长度比较小时,数据库中随机情况下找到高度相似甚至相同的局部比对(HSPs)的可能性非常高,系统自动将 word size 调小,提高敏感度,而将 E-value 调大,确保有搜索结果出现。
生物信息学_复习题及答案(打印)(1)

生物信息学_复习题及答案(打印)(1)一、名词解释:1.生物信息学:研究大量生物数据复杂关系的学科,其特征是多学科交叉,以互联网为媒介,数据库为载体。
利用数学知识建立各种数学模型; 利用计算机为工具对实验所得大量生物学数据进行储存、检索、处理及分析,并以生物学知识对结果进行解释。
2.二级数据库:在一级数据库、实验数据和理论分析的基础上针对特定目标衍生而来,是对生物学知识和信息的进一步的整理。
3.FASTA序列格式:是将DNA或者蛋白质序列表示为一个带有一些标记的核苷酸或者氨基酸字符串,大于号(>)表示一个新文件的开始,其他无特殊要求。
4.genbank序列格式:是GenBank 数据库的基本信息单位,是最为广泛的生物信息学序列格式之一。
该文件格式按域划分为4个部分:第一部分包含整个记录的信息(描述符);第二部分包含注释;第三部分是引文区,提供了这个记录的科学依据;第四部分是核苷酸序列本身,以“//”结尾。
5.Entrez检索系统:是NCBI开发的核心检索系统,集成了NCBI 的各种数据库,具有链接的数据库多,使用方便,能够进行交叉索引等特点。
6.BLAST:基本局部比对搜索工具,用于相似性搜索的工具,对需要进行检索的序列与数据库中的每个序列做相似性比较。
P947.查询序列(query sequence):也称被检索序列,用来在数据库中检索并进行相似性比较的序列。
P988.打分矩阵(scoring matrix):在相似性检索中对序列两两比对的质量评估方法。
包括基于理论(如考虑核酸和氨基酸之间的类似性)和实际进化距离(如PAM)两类方法。
P299.空位(gap):在序列比对时,由于序列长度不同,需要插入一个或几个位点以取得最佳比对结果,这样在其中一序列上产生中断现象,这些中断的位点称为空位。
P2910.空位罚分:空位罚分是为了补偿插入和缺失对序列相似性的影响,序列中的空位的引入不代表真正的进化事件,所以要对其进行罚分,空位罚分的多少直接影响对比的结果。
pdb数据库使用方法

pdb数据库使用方法PDB数据库(Protein Data Bank)是一个著名的生物科学数据库,收录了各种生物大分子三维结构的信息。
本文将介绍PDB数据库的使用方法,帮助读者更好地利用这个有用的资源。
一、了解PDB数据库及其结构PDB数据库是由许多研究机构、大学和政府机构建立和维护的,收录了全球范围内各种生物大分子的三维结构数据,如蛋白质、核酸和复合物等。
PDB数据库中每一项数据都对应一个唯一的PDB ID号码,并且提供了该生物大分子的结构信息、序列信息、实验条件及解析方法等详细的数据。
为方便使用,PDB数据库的数据以PDB格式存储。
二、使用PDB数据库的搜索功能PDB数据库提供了一系列搜索选项,让用户按需查询数据。
用户可以按照PDB ID、蛋白质名字、结晶学条件等多种方式进行搜索。
在PDB数据库的主页页面,用户可以看到搜索选项,点击“Search”按钮即可进入搜索页面进一步操作。
三、使用PDB格式数据文件PDB格式数据文件是PDB数据库中唯一的数据类型,存储了生物大分子的结构信息、序列信息等所有数据。
这些数据可以通过下载PDB文件的方式进行获取。
用户可以在PDB数据库中找到对应的数据,进入数据详情页面后,点击“Download Files”按钮即可下载PDB格式的文件。
四、使用PDB格式数据文件的软件PDB格式的数据文件可以在很多软件上进行解析和编辑,包括众所周知的PyMol、Chimera等生物大分子分析软件,也有很多其他免费的软件可以用来查看或编辑PDB文件,如UCSF ChimeraX、MSM格式转换器等。
用户可以根据自己的需要选择合适的软件进行使用。
五、常用生物大分子分析软件介绍1. PyMolPyMol是一款非常流行的分子可视化软件,用于可视化、分析和编辑生物大分子的三维结构。
该软件具有强大的分子动画和交互式残基操作功能,可以进行序列对齐、氨基酸置换等功能,适合于研究生物分子结构和功能。
ncbi数据库检索解读

收集并储存大分子结构信息,部分来源于PDB
提供并及时更新后生生物的全基因组序列以及最为精确的注释. 是一个蛋白质信息最为准确的蛋白质数据库, 它所提供的蛋白质信息有着最详尽的注释和 最少的冗余..
5 UniProtKB\Swiss-prot
2.2.4 SRS 检索实例
已知BPMV的名字,查询其基因组的信息,核酸序 列信息,蛋白质序列信息和结构信息
第二章 数据库检索
2.1 综合性数据库 NCBI
2.1.1 NCBI简介
美国参议员Claude Pepper率先意识到信息计算机化过程 方法对指导生物医学研究的重要性,发起了在1988年11月4日 建立国立生物技术信息中心的立法. (National Center for Biotechnology Information , NCBI) . NCBI隶属于国立医学图书馆( National Library of Medicing, NLM)。NLM在创立和维护生物医学数据库方面有 丰富的经验。
包含用于群体进化或变异研究的比对序列
准确的基因表达谱数据和大规模的分子实验数据
公众医学信息中心,是NLM在生命科学领域 Central数据库 期刊文献的数字存档 医学主题5 Bookshelf 数据库
16 OMIM 数据库
主要着眼于可遗传或遗传性的基因疾病,包括文献, 序列记录,染色体定位图谱及相关的数据库的链接
7 uniSTS数据库 8 基因数据库 9 UniGene数据库
可通过基因名称,同义词,编号,出版物,染色体号等属性 寻找基因 GenBank 中基因序列的集合
10 SNP数据库 11 PopSet 12 GEO数据库 13 PubMed
用于存储包括单核苷酸替换,一两个碱基的插入 或缺失等多态性信息
生物信息学期末考试答案分析解析

一、名词Bioinformatics:生物信息学——是一门综合运用生物学、数学、物理学、信息科学以及计算机科学等诸多学科的理论方法,以互联网为媒介、数据库为载体、利用数学和计算机科学对生物学数据进行储存、检索和处理分析,并进一步挖掘和解读生物学数据。
Consensus sequence:共有序列——决定启动序列的转录活性大小。
各种原核启动序列特定区域内(通常在转录起始点上游-10及-35区域)存在共有序列,是在两个或多个同源序列的每一个位置上多数出现的核苷酸或氨基酸组成的序列。
Data mining:数据挖掘——数据挖掘一般是指从大量的数据中自动搜索隐藏于其中的有着特殊关系性的信息的过程。
数据挖掘通常是利用计算方法分析生物数据,即根据核酸序列预测蛋白质序列、结构、功能的算法等,实现对现有数据库中的数据进行发掘。
EST:(Expressed Sequence Tag)表达序列标签——是某个基因cDNA克隆测序所得的部分序列片段,长度大约为200~600bp。
Similarity:相似性——是直接的连续的数量关系,是指序列比对过程中用来描述检测序列和目标序列之间相同DNA碱基或氨基酸残基顺序所占比例的高低。
Homology:同源性——是两个对象间的肯定或者否定的关系。
如两个基因在进化上是否曾具有共同祖先。
从足够的相似性能够判定二者之间的同源性。
Alignment:比对——从核酸以及氨基酸的层次去分析序列的相同点和不同点,以期能够推测它们的结构、功能以及进化上的联系。
或是指为确定两个或多个序列之间的相似性以至于同源性,而将它们按照一定的规律排列。
BLOSUM:模块替换矩阵——是指在对蛋白质数据库搜索时,采用不同的相似性分数矩阵进行检索的相似性矩阵。
以序列片段为基础,从蛋白质模块数据库BLOCKS中找出一组替换矩阵,用于解决序列的远距离相关。
在构建矩阵过程中,通过设置最小相同残基数百分比将序列片段整合在一起,以避免由于同一个残基对被重复计数而引入的任何潜在的偏差。
生命科学中最常用的5个数据库介绍

生命科学中最常用的5个数据库介绍生命科学是一个庞大而复杂的学科,其中包含了关于生命现象的各种研究。
对于生命科学的研究,特别是在分子水平上进行的研究,需要大量的数据支持。
这些数据包括分子序列、蛋白质结构、代谢途径等等。
为了有效地管理这些数据,生命科学中广泛应用了各种数据库。
本文将介绍生命科学中最常用的5个数据库。
1. GenBankGenBank是全球最大的分子生物学数据库,包含了全球各地实验室提交的DNA和RNA序列。
它由美国国家生物技术信息中心(NCBI)维护。
GenBank包含了数十亿条序列记录,其中包括了不同物种的基因组、蛋白质序列、DNA和RNA序列等。
与DNA和RNA序列相关的信息包括序列长度、基序、带电的特殊域、结构域、转录因子结合位点以及其他数据。
GenBank还包含了元数据,如物种和菌株的信息、文献引用以及序列的提交日期。
2. PubMedPubMed是美国国家医学图书馆(NLM)维护的一个生命科学文献数据库,包括了生命科学、医学和健康相关的数百万篇论文。
PubMed提供了对文献的全文搜索和存储,使科学家在查找特定话题时更加方便。
除了搜索全文的功能,PubMed还提供了很多额外的服务,如翻译摘要、相关文章推荐、绘制图表等。
3. EnsemblEnsembl是一种数据库、搜索引擎和分析平台,专门用于处理各种生命科学的数据。
Ensembl已经成为了全球最大的基因组数据库之一,包含了人类、其他哺乳动物、鸟类、篮球、双子蝎、无脊椎动物等近700个物种的基因组信息。
Ensembl提供的数据包括生物序列、调控区域、基因家族、基因结构、基因组的变异和基因表达信息等。
4. Protein Data Bank (PDB)蛋白质数据银行(PDB)是一个三维蛋白结构数据库,由改华大学、美国罗格斯大学和欧洲生物信息研究所等机构共同维护。
PDB存储了全球各地实验室提交的蛋白质晶体结构和生化分析,包括了大多数已知的蛋白质家族和酶。
生物大数据分析的常用工具和软件介绍

生物大数据分析的常用工具和软件介绍生物大数据的快速发展和应用需求推动了生物信息学工具和软件的不断发展。
这些工具和软件提供了一系列功能,如序列分析、基因表达分析、蛋白质结构预测、功能注释等,帮助研究人员从大量的生物数据中提取有意义的信息。
下面将介绍一些常用的生物大数据分析工具和软件。
1. BLAST(Basic Local Alignment Search Tool)BLAST是最常用的序列比对工具之一,用于比对一条查询序列与已知序列数据库中的序列。
通过比对确定序列之间的相似性,从而推断其功能和结构。
BLAST具有快速、准确、用户友好的特点,适用于DNA、RNA和蛋白质序列的比对。
2. GalaxyGalaxy是一个基于Web的开源平台,提供了许多生物信息学工具和软件的集成。
它提供了一个易于使用的界面,使得用户可以通过拖放操作完成复杂的数据分析流程。
Galaxy支持不同类型的数据分析,包括序列比对、组装、注释、表达分析等。
3. R包R是一个功能强大的统计语言和环境,用于数据分析和可视化。
R包提供了许多用于生物数据分析的扩展功能。
例如,"Bioconductor"是一个R软件包,提供了丰富的生物数据分析方法和工具,包括基因表达分析、序列分析、蛋白质分析等。
4. GATK(Genome Analysis Toolkit)GATK是一个用于基因组数据分析的软件包,主要用于研究DNA变异。
它包含了各种工具和算法,用于SNP检测、基因型调用、变异注释等。
GATK还在处理复杂变异(如复杂多态位点)和群体遗传学分析方面具有独特的优势。
5. CytoscapeCytoscape是一个用于生物网络分析和可视化的开源平台。
它可以用于可视化和分析蛋白质-蛋白质相互作用网络、基因共表达网络、代谢网络等。
Cytoscape提供了丰富的插件,使得用户可以根据自己的需要进行网络分析和可视化。
6. DAVID(Database for Annotation, Visualization, and Integrated Discovery)DAVID是一个用于功能注释和富集分析的在线工具。
数据搜索时有用的生物大分子数据库扫描

生物大分子数据库扫描根据“Nucleic Acids Research”最新(指2007年)公布的数据,目前已有968个有关生物大分子数据库(参见文献Galperin M Y, The Molecular Biology Database Collection, 2007, 35: D3)。
有兴趣的读者可以参阅网站“/nar/database/a”。
我们这里将主要类型的数据库列于表4-2。
面这段是一个完整的SwissProt条目,现解释如下:ID 104K_THEPA STANDARD; PRT; 924 AA.AC P15711;DT 01-APR-1990 (Rel. 14, Created)DT 01-APR-1990 (Rel. 14, Last sequence update)DT 01-AUG-1992 (Rel. 23, Last annotation update)DE 104 kDa microneme-rhoptry antigen.OS Theileria parva.OC Eukaryota; Alveolata; Apicomplexa; Piroplasmida; Theileriidae;OC Theileria.OX NCBI_TaxID=5875;RN [1]RP NUCLEOTIDE SEQUENCE.RC STRAIN=Muguga;RX MEDLINE=90158697; PubMed=1689460; DOI=10.1016/0166-6851(90)90007-9;RA Iams K.P., Young J.R., Nene V., Desai J., Webster P., Ole-Moiyoi O.K.,RA Musoke A.J.;RT "Characterisation of the gene encoding a 104-kilodalton microneme-RT rhoptry protein of Theileria parva.";RL Mol. Biochem. Parasitol. 39:47-60(1990).CC -!- SUBCELLULAR LOCATION: In microneme/rhoptry complexes.CC -!- DEVELOPMENTAL STAGE: Sporozoite antigen.CC -------------------------------------------------------------------------- CC This Swiss-Prot entry is copyright. It is produced through a collaboration uniprot_sprot.datCC the European Bioinformatics Institute. There are no restrictions on its CC use as long as its content is in no way modified and this statement is not CC removed.CC -------------------------------------------------------------------------- DR EMBL; M29954; AAA18217.1; -.DR PIR; A44945; A44945.KW Antigen; Repeat; Sporozoite.FT DOMAIN 1 19 Hydrophobic.FT DOMAIN 905 924 Hydrophobic.SQ SEQUENCE 924 AA; 103626 MW; 289B4B554A61870E CRC64;MKFLILLFNI LCLFPVLAAD NHGVGPQGAS GVDPITFDIN SNQTGPAFLT AVEMAGVKYLQVQHGSNVNI HRLVEGNVVI WENASTPLYT GAIVTNNDGP YMAYVEVLGD PNLQFFIKSGDAWVTLSEHE YLAKLQEIRQ AVHIESVFSL NMAFQLENNK YEVETHAKNG ANMVTFIPRNGHICKMVYHK NVRIYKATGN DTVTSVVGFF RGLRLLLINV FSIDDNGMMS NRYFQHVDDKYVPISQKNYE TGIVKLKDYK HAYHPVDLDI KDIDYTMFHL ADATYHEPCF KIIPNTGFCITKLFDGDQVL YESFNPLIHC INEVHIYDRN NGSIICLHLN YSPPSYKAYL VLKDTGWEATTHPLLEEKIE ELQDQRACEL DVNFISDKDL YVAALTNADL NYTMVTPRPH RDVIRVSDGSEVLWYYEGLD NFLVCAWIYV SDGVASLVHL RIKDRIPANN DIYVLKGDLY WTRITKIQFTQEIKRLVKKS KKKLAPITEE DSDKHDEPPE GPGASGLPPK APGDKEGSEG HKGPSKGSDSSKEGKKPGSG KKPGPAREHK PSKIPTLSKK PSGPKDPKHP RDPKEPRKSK SPRTASPTRRPSPKLPQLSK LPKSTSPRSP PPPTRPSSPE RPEGTKIIKT SKPPSPKPPF DPSFKEKFYDDYSKAASRSK ETKTTVVLDE SFESILKETL PETPGTPFTT PRPVPPKRPR TPESPFEPPKDPDSPSTSPS EFFTPPESKR TRFHETPADT PLPDVTAELF KEPDVTAETK SPDEAMKRPRSPSEYEDTSP GDYPSLPMKR HRLERLRLTT TEMETDPGRM AKDASGKPVK LKRSKSFDDLTTVELAPEPK ASRIVVDDEG TEADDEETHP PEERQKTEVR RRRPPKKPSK SPRPSKPKKPKKPDSAYIPS ILAILVVSLI VGIL//ID 是指其身份号,924 AA是指有该序列有924个氨基酸残基AC 获取号;DT 序列测得的时间DE 对该序列必要的信息的说明,如该分子的分子量为104 kDa .OS 来源OX NCBI分类身份号RN [1]RP NUCLEOTIDE SEQUENCE.RC STRAIN=Muguga;RX 有关Medline的出版号RA 作者RT 引用文献题目RL 杂志名称,出版日期,卷期页CC 有关它的功能描述及其它相关信息方面的描述DR EMBL数据库中的获取号DR PIR数据库中的获取号KW 关键词FT 功能区的描述SQ 有关序列方面的信息,这部分是最主要的,因为该蛋白质的序列就列在下面。
生物信息数据库的查询和搜索

2.根据数据的获得方式又可以分为一级库 和二级库。一级数据库的数据都直接来源 于实验获得的原始数据,只经过简单的归 类整理和注释;二级数据库是在一级数据 库、实验数据和理论分析的基础上针对特 定目标衍生而来,是对生物学知识和信息 的进一步整理。
国际上著名的一级核酸数据库有Genbank 数据库、EMBL核酸库和DDBJ库等;蛋白 质序列数据库有SWISS-PROT、PIR等; 蛋白质结构库有PDB等。 国际上二级生物学数据库非常多,它们因 针对不同的研究内容和需要而各具特色, 如人类基因组图谱库GDB、转录因子和结 合位点库TRANSFAC、蛋白质结构家族分 类库SCOP等等。
5.1.7疾病数据库 疾病数据库主要收集与疾病相关的生物大分子的 信息,尤其是基因方面的情况。OMIM数据库是 一个收集人类基因与基因组中不正常现象的数据 库。SNP Consortium datahase是收集单核苷 酸多态性的数据库,根据这些数据可以与临床化 验检测结果相对应,从而找出致病基因。 OncoDB是收集用生物芯片研究癌症与基因表达 的数据库,其中有许多的资料中仍未确定癌症— 基因的对应关系。这类数据库是基础医学研究的 宝贵资源。
5.1.9分析与记录方式数据库 分析与记录方式数据库是指收集文献、图片、数 学分析方法、命名规则的数据库。PubMed数据 库是收录生物医学文献的摘要及引文的数据库, 在生物学与医学研究中有广泛的应用,在美国 NCBI网站可对PubMed数据库进行查询。 Bioimage数据库是收集生物学研究的专业图片的 数据库,由欧盟委员会资助建成,由牛津大学动 物系管理。BioModels数据库收录了已发表的用 于研究生物学与医学的数学模型。Genew数据库 专门收集人类基因的命名规则。
常用的生物数据库(二)

常用的生物数据库(二)引言概述:生物数据库是生物信息学领域的重要工具,可以帮助研究人员存储、管理和共享生物数据。
本文将介绍常用的生物数据库(二),以便研究人员更好地利用这些资源进行生物学研究。
正文内容:一、蛋白质相互作用数据库1. STRING数据库:提供蛋白质相互作用预测和注释功能。
2. IntAct数据库:收集整理蛋白质相互作用数据,提供数据检索和分析工具。
3. BioGRID数据库:整合多种物种的蛋白质相互作用数据,并提供丰富的功能注释。
二、基因组数据库1. GenBank数据库:包含大量的序列数据,包括基因组、转录本和蛋白质序列等。
2. ENSEMBL数据库:集成了各种生物信息学工具,提供全面的基因组注释信息。
3. UCSC数据库:基于人类基因组构建的浏览器,提供详细的基因组注释和可视化功能。
三、表达谱数据库1. GEO数据库:收集了大量的基因表达谱数据,可进行数据检索和分析。
2. ArrayExpress数据库:包含了来自各种高通量技术的表达谱数据,提供数据下载和分析工具。
3. TCGA数据库:整合了多种癌症的基因表达数据,可进行差异表达和生存分析等研究。
四、突变数据库1. dbSNP数据库:记录了常见的单核苷酸多态性(SNP)数据,是研究遗传变异的重要资源。
2. COSMIC数据库:专注于癌症相关的突变数据,包含了大量的突变谱系和功能注释信息。
3. ClinVar数据库:整合了与人类疾病相关的遗传变异数据,提供临床相关的注释信息。
五、药物数据库1. DrugBank数据库:收录了大量的药物信息,包括结构、作用机制和药理学数据等。
2. PubChem数据库:提供了大量的小分子化合物数据,可进行化学结构搜索和药物筛选等研究。
3. ChEMBL数据库:整合了化合物活性数据和药物靶点信息,可用于药物发现和优化。
总结:生物数据库为生物学研究提供了丰富的数据资源和分析工具。
蛋白质相互作用数据库、基因组数据库、表达谱数据库、突变数据库和药物数据库是常用的生物数据库之一。
生物数据库检索基本方法

生物数据库检索基本方法生物数据库是生物信息学研究的重要工具,可以存储和管理生物实验数据、基因组序列、蛋白质结构等丰富的生物信息资源。
生物数据库的检索方法多种多样,对于生物学研究者来说,熟练掌握生物数据库的检索技巧是进行生物学研究的基本要求之一、本文将探讨几种常用的生物数据库检索方法。
首先,关键字检索是最常用的数据库检索方法之一、用户可以通过输入关键字来相关的生物信息。
关键字可以是生物学的术语、基因名称、蛋白质名称等。
例如,在NCBI (National Center for Biotechnology Information)网站上,用户可以通过关键字数据库中的文章、序列、蛋白质等信息。
在关键字检索中要注意选择合适的关键字和结合逻辑运算符,如“与”、“或”、“非”等,以提高结果的准确性。
其次,序列相似性是生物数据库检索的重要方法。
序列相似性可以通过比对查询序列与数据库中的序列进行相似性计算,找到与查询序列具有高度相似性的序列。
常用的序列相似性工具包括BLAST (Basic Local Alignment Search Tool)、FASTA (Fast All)、Smith-Waterman等。
用户可以将待的序列输入到这些工具中,然后选择适当的数据库进行。
另外,数据库的交叉也是一种常用的检索方法。
交叉是指将一个数据库的结果与另一个数据库的结果进行对比和整合,在多个数据库中进行检索以获取更详细和全面的信息。
例如,在进行基因表达研究时,可以先在Gene Expression Omnibus (GEO)数据库中相关基因的表达数据,然后将结果与其他数据库中的信息进行整合,来进一步分析和解读实验结果。
最后,生物数据库的检索还可以借助于一些专门的数据库检索工具和软件。
这些工具和软件通常提供更高级、更专业的功能和功能,可以更有效地检索生物数据库中的信息。
例如,Ensembl、UniProt-GOA、Reactome 等数据库不仅提供了丰富的生物信息和数据,还提供了一系列分析工具和可视化工具,方便用户进行更深入的研究。
生物数据库介绍——NCBI

⽣物数据库介绍——NCBINCBI(National Center for Biotechnology Information,美国国家⽣物技术信息中⼼)除了维护GenBank核酸序列数据库外,还提供数据分析和检索资源。
NCBI资源包括Entrez、Entrez编程组件、MyNCBI、PubMed、PudMed Central、PubReader、Gene、the NCBI Taxonomy Browser、BLAST、Pimer-Blast、COBALT、RefSeq、UniGene、HomoloGene、ProtEST、dbMHC、dbSNP、dbVar、Epigenomics、the Genetic Testing Registry、Genome和相关⼯具、⽐对查看器、跟踪存档、Sequence Read Archive、BioProject、BioSample、ClinVar、MedGen、HIV-1/⼈类蛋⽩质相互作⽤数据库、Gene Expression Omnibus、Probe、Online Mendelian Inheritance in Animals、the Molecular Modeling Database、the Conserved Domain Database、the Conserved Domain Architecture Retrieval Tool、Biosystem、Protein Clusters and thePubChem suite of small molecule databases,所有这些资源可以在NCBI主页找到。
Databases⼀个提供有关基因组组装结构,装配名称和其他元数据,统计报告以及基因组序列数据链接等信息的数据库。
⼀个有关培养物、动植物样本和其他⾃然样本的精选元数据集。
记录显⽰样本状态,有关馆藏的机构的信息,以及NCBI中相关数据链接。
常用的生物数据库

转载)分子生物学相关数据库综合数据库:Entrez由NCBI开发的一个数据库检索系统,它综合了下述各大数据库的信息,包括核酸、蛋白以及Medline文摘数据库,在这三个数据库中建立了非常完善的联系。
因此,可以从一个序列查询到蛋白产物以及相关的结构、功能和文献信息,详见NCBI(美国国立生物技术信息中心) 简介。
EBI欧洲生物信息学研究所(European Bioinformatics Institute,EBI)是EMBL的分部,位于英国Hinxton的Wellcome Trust Genome Campus。
EBI维护和发布的数据库:üEMBL核酸数据库、欧洲原始核酸数据资源库üSwissProt蛋白质序列数据库[与瑞士生物信息学协会(Swiss Institute for Bioinformatics,SIB)的Amos Bairroch合作]üTrEMBL(SwissProt的附属数据库,由EMBL数据库编码序列翻译而来的蛋白质序列数据库)ü分子结构数据库(Molecular Structure Database,MSD)[与Brookhaven 国家实验室(纽约)的蛋白质三维结构数据库(Protein Data Bank,PDB)合作]ü放射杂交数据库(Radiation Hybrid database,RHdb)ü其他组织合作产生的分子生物学数据库:EBI还提供网络服务,通过互联网、其WEB界面和FTP服务器可以访问最新收集到的数据,同时也提供数据库和序列相似性的搜索工具。
核酸数据库:GenBankGenBank是NIH的基因序列数据库,由美国国立卫生研究院全国生物技术信息中心(NCBI)建立并维护,是所有公开的DNA序列的集合( Nucleic Acids Research 1998 Jan 1;26(1):1-7),GenBank包含所有已知的核苷酸及蛋白质序列、以及与之相关的生物学信息和参考文献,是世界上的权威序列数据库。
文献检索和浏览各大生物分子数据库

实验一文献检索和浏览各大生物分子数据库一、实验目的1、学习文献检索方法2、了解生物信息学常用数据库的结构二、实验内容文献检索是每个科研工作者必须具备的能力,这里主要以我校的资源为例,说明网络文献检索的一些基本方法。
国际上已经建立起许多分子公共数据库,包括基因组图谱数据库、核酸序列数据库、蛋白质序列数据库及生物大分子结构数据库等。
这些数据库由专门的机构建立和维护,他们负责收集、组织、管理和发布生物分子数据,并提供数据检索和分析工具,向生物学研究人员提供大量有用的信息,为他们的研究服务。
本实验通过登陆GenBank、EMBL、DDBJ三个国际上权威的核酸序列数据库、GDB基因组数据库、人类基因组数据库Ensembl、表达序列标记数据库dbEST、序列标记位点数据库dbSTS,以及PIR、SWISS-PROT、TrEMBL蛋白质序列数据库、蛋白质数据仓库UniProt、生物大分子数据库PDB等,了解各数据库的结构,。
三、实验仪器、设备及材料计算机(联网)四、实验原理建立生物分子数据库的动因是由于生物分子数据的高速增长,而另一方面也是为了满足分子生物学及相关领域研究人员迅速获得最新实验数据的要求。
生物分子信息分析已经成为分子生物学研究必备的一种方法。
数据库及其相关的分析软件是生物信息学研究和应用的重要基础,也是分子生物学研究必备的工具。
核酸序列是了解生物体结构、功能、发育和进化的出发点。
国际上权威的核酸序列数据库有三个,分别是美国生物技术信息中心(NCBI)的GenBank (/web/Genbank/index/html)、欧洲分子生物学实验室的EMBL-Bank (简称EMBL,/embl/index/html)及日本遗传研究所的DDBJ (http://www.ddbj.nig.ac.jp/)。
三个数据库中的数据基本一致,仅在数据格式上有所差别,对于特定的查询,三个数据库的响应结果一样。
以EMBL数据库为例,其每个序列,相关数据包括序列名称、序列、位点、关键字、来源、生物种类、参考文献、注释、序列中具有重要生物学意义的位点等。
常用的生物数据库

常用的生物数据库在当今的生命科学研究领域,生物数据库就如同一个个巨大的知识宝库,为科研人员提供了丰富的信息和宝贵的数据资源。
这些数据库涵盖了从基因序列到蛋白质结构,从疾病信息到生物进化等各个方面,对于推动生物科学的发展发挥着至关重要的作用。
接下来,让我们一起了解一些常用的生物数据库。
首先要提到的是 GenBank 数据库。
它是由美国国家生物技术信息中心(NCBI)建立和维护的,是全球最全面的核酸序列数据库之一。
GenBank 收录了来自各种生物的 DNA 和 RNA 序列,包括细菌、病毒、真菌、植物和动物等。
科研人员可以通过该数据库查询特定基因的序列信息,了解其结构和功能,为基因研究和基因工程提供了重要的基础。
另一个重要的数据库是 UniProt 。
它是整合了蛋白质序列、功能、分类和相互作用等信息的综合性蛋白质数据库。
UniProt 包含了大量经过人工注释和审核的数据,具有很高的准确性和可靠性。
对于研究蛋白质的结构与功能关系、蛋白质组学以及药物研发等领域来说,UniProt 是不可或缺的工具。
在疾病研究方面,OMIM(Online Mendelian Inheritance in Man)数据库是一个非常有价值的资源。
它主要聚焦于人类遗传疾病,提供了有关疾病的临床表现、遗传方式、基因定位和分子机制等详细信息。
对于医学研究人员和临床医生来说,OMIM 有助于诊断和治疗遗传疾病,以及深入了解疾病的发病机制。
PDB(Protein Data Bank)则是专门用于存储蛋白质和核酸等生物大分子三维结构的数据库。
通过 PDB ,科研人员可以直观地观察到生物大分子的空间结构,从而更好地理解其功能和作用机制。
这对于药物设计和开发具有重要的指导意义,因为药物的作用往往与靶点蛋白的结构密切相关。
KEGG(Kyoto Encyclopedia of Genes and Genomes)是一个综合性的生物通路数据库。
生物大分子的结构和功能扫描电镜和质谱分析的方法

生物大分子的结构和功能扫描电镜和质谱分析的方法生物体中的很多重要分子都属于大分子,例如蛋白质、核酸、多糖等,它们的结构和功能对于生命过程的正常进行至关重要。
因此,研究这些大分子的结构和功能,对于理解生命现象、探索生物学问题具有重要的意义。
而扫描电镜和质谱分析则成为了生物大分子结构和功能研究的重要手段。
一、扫描电镜扫描电镜(Scanning Electron Microscopy, SEM)是一种主要用于观察微小物体表面形态和结构的技术。
相比于传统光学显微镜,扫描电镜能够用高分辨率、三维的方法观察样本表面的形态和结构,因此非常适合用于生物大分子的结构研究。
使用扫描电镜观察生物大分子需要先将样品制备成适宜的形态和尺寸。
对于生物大分子如蛋白质,需要将样品分离出来并制备成可以被电镜观察的形态,通常通过冷冻过程来制备蛋白质样品,再通过高真空和电子束对样品进行观察。
通过扫描电镜,可以获得不同角度下的样品表面形态图像,从而还原出样品的三维形态。
扫描电镜的分辨率能够达到几纳米的级别,即可以观察纳米级别的物质。
因此,在生物大分子结构研究上,扫描电镜意义非凡。
扫描电镜可以揭示生物大分子的超微观结构,如蛋白质分子的折叠状态、表面拓扑结构等。
例如,扫描电镜研究指出,蛋白质的空洞可以承载金纳米颗粒,从而提供了一种用于制备三维结构的新方法。
二、质谱分析质谱分析(Mass Spectrometry, MS)是一种能够测量分子质量和碎片质量、分析分子构成和结构的技术。
对于生物大分子而言,质谱分析作为一种高灵敏度、高分辨率的生物大分子结构分析手段,得到了广泛应用。
对于蛋白质而言,质谱分析技术通常被用于两大研究领域:一是蛋白质的序列分析;二是蛋白质的结构研究。
蛋白质的序列分析是指通过对蛋白质分子中氨基酸序列的测定,揭示蛋白质的结构和功能。
质谱技术与现代分子生物学方法相结合,能够实现高通量的蛋白质组学分析,对于大规模测定蛋白质组的序列和定量等信息起到了重要作用。
分子生物学中常用数据库

分子生物学中常用数据库综合数据库:来源:/news/science/article/90048.html生物信息学网址链接:http://www.bioinformatics.ca/links_directory/Nucleic Acid Research Database Issue:/content/vol32/suppl_2/一、蛋白相关数据库蛋白质结构域预测工具Esignal:/esignal/信号传导系统蛋白的结构域预测工具,凡是涉及到信号传导系统的蛋白用这个预测效果最佳SignalP:http://www.cbs.dtu.dk/services/SignalP/信号肽预测工具,适合定位于非胞质位置的蛋白质Emotif:/emotif-search/结构域预测工具,由于其用motif电子学习的方法产生结构域模型,故预测效果比Prosite好Ematrix:/ematrix/是用Matrix的方法创建的结构域数据库,可与emotif互相印证。
其速度快,可快速搜索整个基因组InterPro:/InterProScan/EBI提供的服务,用图形的形式表示出搜索的结构域结果TRRD:http://wwwmgs.bionet.nsc.ru/mgs/gnw/trrd/转录因子结构域预测的最好数据库。
但不会用Protscale:/cgi-bin/protscale.pl可分析该序列的各种性状如活动度、亲水性(Kyte&Doolittle)、抗原性(Hopp&Woods)等通过寻找MOTIF和Domain来分析蛋白质的功能A. MOTIF是蛋白中较小的保守序列片断,其概念比Domain小PROSITE:/tools/scanprosite/是专门搜索蛋白质Motif的数据库,其中signature seqs是最重要的motif信息B. Domain:若干motif可形成一个Domain,每个Domain形成一个球形结构,Domain与Domain之间通常像串珠一样相连Pfam:可以搜索某段序列中的Domain,并以图形化表示出来。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
生物大分子数据库扫描根据“Nucleic Acids Research”最新(指2007年)公布的数据,目前已有968个有关生物大分子数据库(参见文献Galperin M Y, The Molecular Biology Database Collection, 2007, 35: D3)。
有兴趣的读者可以参阅网站“/nar/database/a”。
我们这里将主要类型的数据库列于表4-2。
面这段是一个完整的SwissProt条目,现解释如下:ID 104K_THEPA STANDARD; PRT; 924 AA.AC P15711;DT 01-APR-1990 (Rel. 14, Created)DT 01-APR-1990 (Rel. 14, Last sequence update)DT 01-AUG-1992 (Rel. 23, Last annotation update)DE 104 kDa microneme-rhoptry antigen.OS Theileria parva.OC Eukaryota; Alveolata; Apicomplexa; Piroplasmida; Theileriidae;OC Theileria.OX NCBI_TaxID=5875;RN [1]RP NUCLEOTIDE SEQUENCE.RC STRAIN=Muguga;RX MEDLINE=90158697; PubMed=1689460; DOI=10.1016/0166-6851(90)90007-9;RA Iams K.P., Young J.R., Nene V., Desai J., Webster P., Ole-Moiyoi O.K.,RA Musoke A.J.;RT "Characterisation of the gene encoding a 104-kilodalton microneme-RT rhoptry protein of Theileria parva.";RL Mol. Biochem. Parasitol. 39:47-60(1990).CC -!- SUBCELLULAR LOCATION: In microneme/rhoptry complexes.CC -!- DEVELOPMENTAL STAGE: Sporozoite antigen.CC -------------------------------------------------------------------------- CC This Swiss-Prot entry is copyright. It is produced through a collaboration uniprot_sprot.datCC the European Bioinformatics Institute. There are no restrictions on its CC use as long as its content is in no way modified and this statement is not CC removed.CC -------------------------------------------------------------------------- DR EMBL; M29954; AAA18217.1; -.DR PIR; A44945; A44945.KW Antigen; Repeat; Sporozoite.FT DOMAIN 1 19 Hydrophobic.FT DOMAIN 905 924 Hydrophobic.SQ SEQUENCE 924 AA; 103626 MW; 289B4B554A61870E CRC64;MKFLILLFNI LCLFPVLAAD NHGVGPQGAS GVDPITFDIN SNQTGPAFLT AVEMAGVKYLQVQHGSNVNI HRLVEGNVVI WENASTPLYT GAIVTNNDGP YMAYVEVLGD PNLQFFIKSGDAWVTLSEHE YLAKLQEIRQ AVHIESVFSL NMAFQLENNK YEVETHAKNG ANMVTFIPRNGHICKMVYHK NVRIYKATGN DTVTSVVGFF RGLRLLLINV FSIDDNGMMS NRYFQHVDDKYVPISQKNYE TGIVKLKDYK HAYHPVDLDI KDIDYTMFHL ADATYHEPCF KIIPNTGFCITKLFDGDQVL YESFNPLIHC INEVHIYDRN NGSIICLHLN YSPPSYKAYL VLKDTGWEATTHPLLEEKIE ELQDQRACEL DVNFISDKDL YVAALTNADL NYTMVTPRPH RDVIRVSDGSEVLWYYEGLD NFLVCAWIYV SDGVASLVHL RIKDRIPANN DIYVLKGDLY WTRITKIQFTQEIKRLVKKS KKKLAPITEE DSDKHDEPPE GPGASGLPPK APGDKEGSEG HKGPSKGSDSSKEGKKPGSG KKPGPAREHK PSKIPTLSKK PSGPKDPKHP RDPKEPRKSK SPRTASPTRRPSPKLPQLSK LPKSTSPRSP PPPTRPSSPE RPEGTKIIKT SKPPSPKPPF DPSFKEKFYDDYSKAASRSK ETKTTVVLDE SFESILKETL PETPGTPFTT PRPVPPKRPR TPESPFEPPKDPDSPSTSPS EFFTPPESKR TRFHETPADT PLPDVTAELF KEPDVTAETK SPDEAMKRPRSPSEYEDTSP GDYPSLPMKR HRLERLRLTT TEMETDPGRM AKDASGKPVK LKRSKSFDDLTTVELAPEPK ASRIVVDDEG TEADDEETHP PEERQKTEVR RRRPPKKPSK SPRPSKPKKPKKPDSAYIPS ILAILVVSLI VGIL//ID 是指其身份号,924 AA是指有该序列有924个氨基酸残基AC 获取号;DT 序列测得的时间DE 对该序列必要的信息的说明,如该分子的分子量为104 kDa .OS 来源OX NCBI分类身份号RN [1]RP NUCLEOTIDE SEQUENCE.RC STRAIN=Muguga;RX 有关Medline的出版号RA 作者RT 引用文献题目RL 杂志名称,出版日期,卷期页CC 有关它的功能描述及其它相关信息方面的描述DR EMBL数据库中的获取号DR PIR数据库中的获取号KW 关键词FT 功能区的描述SQ 有关序列方面的信息,这部分是最主要的,因为该蛋白质的序列就列在下面。
// 表明这个条目结束。
通过对上面这段的分析,读者可以知道Swiss-Prot数据库基本框架及所蕴含的生物信息学内容。
同时,人们也可在需要Swiss-Proto数据库时根据其格式编制出相应的计算机程序。
这一点其实对生物信息学工作者非常重要,因为一个生物信息学分析方法首先要得到正确的数据,而正确的数据则必须以相应数据库格式为基准。
有关该数据库用户可在“/”上获取。
二、PDB数据库的基本格式我们在前面曾经说过,当一个数据库的格式与内容都知道了后,说明对这个数据库的知识已有初步的掌握。
同前面一样,我们现在详细介绍PDB数据库的格式。
由于蛋白质结构涉及到蛋白质中每个原子的坐标,二级结构,一个蛋白质结构所占的容量是比较大的,因此它不可能象Swiss-Prot数据库那样将所有蛋白质序列放在一个文件中,而是一个蛋白质一个文件。
因此,到目前为止,PDB数据库应有41952个文件,这样就涉及到其文件名的问题,PDB数据库的文件如图4-10所示:图4-10 PDB数据库中的文件名格式下面是一个典型的PDB数据库格式的描述:PDB中所的分子空间结构信息文件的格式基本上都是一样的。
文件由若干记录组成,每一记录有80个字符(包括空格)。
开头的6个字符标明该记录的名称,现将各记录的意义分别叙述如下:HEADER------该记录列出分子所属功能类,正式收入PDB日期以及该分子的判别码OBSLTE------该记录列出已被新分子文件取代的一些旧的分子的有关信息。
COMPND------该记录出分子名SOURCE------该记录说明分子来源AUTHOR------该记录列出提供座标者的姓名REVDAT------该记录列出文件历次修改的日期等有关信息SPRSDE------该记录列说明此文件取代旧文件的有关信息JRNL--------该记录引用与确定该分子空间结构有关的主要文献REMARK------该记录为关于该分子文件的其它信息。
其中:REMARK1专用于列出与该结构有关的其它文献,REMARK2和REMARK3分别是关于晶体结构的分辨率及精华的信息SEQRES------列出蛋白质一级结构HET---------列出非标准基团或残基的信息(主要是指除标准20个氨基酸残基以外的基团信息。
具体格式如下:1-3列是“HET”;8-10列是非标准基团表示符;13列是链表示符号;14-17顺序号;18列是插入码;21-25列是非标准基团中的原子数目;31-70为注释HELIX--------列出分子中有关α螺旋的信息。
1-6列是“HELIX”; 8-10列是顺序号; 12-14螺旋表示符;16-18 残基名;20链表示符;22-25残基序号;26 插入码;28-30残基名;32链表示符;34-37残基序号;38插入码;39-40螺旋类别;41-70注释。
SHEET--------列出分子中有关β折叠的信息,其格式如下:1-5列为SHEET;8-10股号;12-14折叠表示符;15-16股数;18-20残基名;22链表示符;23-26残基序列号;27插入码;29-31 残基名;33 链表示符;34-37残基序号;38 插入码;39-40类型判别码;42-45 原子名;46-48 残基名;50链表示符;51-54 残基序号;55插入码;57-60 原子名;61-63 残基名;65 链表示符;66-69残基序号;70插入码。
TURN--------列出分子中有β转角(发夹结构)的信息;SSBOND------残出分子中有关二硫键的信息;SITE--------列出重要功能部位 1-4 SITE;8-10序号;12-14功能部位表示符;16-17组成功能部位的残基数;19-61 组成功能部位的四个残基的位置信息。
