如何找基因全序
如何查找基因的序列(全)

如何查找基因序列?(转载)(2010-08-01 11:47:41)如何查找基因序列?——在Genbank中寻找目的基因的实例——献给受类似问题困扰的广大酷友,以及给我动力和信心发表原创帖的基因酷的朋友们。
酷友感言:网络的世界很精彩,网络的查询很无奈。
为了我们的科学研究事业,为了我们能够顺利毕业,我们的广大酷友们在网络的海洋里遨游…遨游…咋就找不到彼岸呢?今天要设计这个基因的PCR引物,明天又要查那个基因的信息,那么大一张网,唉想起来就郁闷……鉴此,我们推出了利用Genbank查找基因序列的帖子,希望对大家有所帮助,并请大家多多指教!当然,如果您已经是此中高手,那就权当我是班门弄斧了,呵呵。
1. 根据文献搞reasearch肯定要读文献的,如果你曾经在文献中看到过你感兴趣的基因,而且文中还提到了该基因在Genbank中的ID号,那就好办了,直接打开,在Search后的下拉框中选择Nucleotide,把Genbank ID号输入GO前面的文本框中,点“GO”,就可以找到他了。
举例说明,例如:在2003年JBC的文章(Conditional Knock-out ofIntegrin-linked Kinase Demonstrates an Essential Role in Protein Kinase B/Akt Activation)中出现了“calreticulin (GenBank accession number gi 16151096)”,那么把“16151096”输入GO前面的文本框中,点“GO”,就可以找到该基因了(当然包括基因序列等相关信息)。
在出现了检索结果界面(下图)后,直接点击红箭头所指的 AY047586就可以看到基因的相关信息了...(呵呵,是不是有点太......easy 了)这里需要指出一下,在显示基因的页面右侧有一个Link,点击后出现一个小菜单,里面是与该基因相关的链接,很有用的,值得一个一个地去看看,这里我就不多说了。
一步一步教你使用NCBI查找DNA、mRNA、cDNA、Protein、promoter、引物设计、BLAST序列比对等

一步一步教你使用 NCBI 查找DNA、mRNA、cDNA、Protein、promoter、引物设计、BLAST序列比对等最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在 NCBI 上都可以方便的找到答案。
现在我就结合我自己使用 NCBI的一些经历(经验)跟大家交流一下 BCBI 的使用。
希望大家都能发表自己的使用心得,让我们共同进步!我分以下几个部分说一下 NCBI 的使用:Part one 如何查找基因序列、mRNA、PromoterPart two 如何查找连续的 mRNA、cDNA、蛋白序列Part three 运用 STS 查找已经公布的引物序列Part four 如何运用 BLAST 进行序列比对、检验引物特异性特别感本版版主,将这个帖子置顶!从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我投票却暂时不能投票的)各位战友表示真诚的感,各位战友!请大家对以下我发表的容提出自己的意见。
关于NCBI 其他方面的使用也请水平较高的战友给予补充First of all,还是让我们从查找基因序列开始。
第一部分利用Map viewer 查找基因序列、mRNA 序列、启动子(Promoter)下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:/mapview/index.html在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene 前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
设计引物原则以及如何查找基因序列

引物设计原则:1.找出这种细胞物种的PTN全长核苷酸序列2.采用primer premier 5.0软件设计引物设计应注意如下要点:● 1. 引物的长度一般为15-30 bp,常用的是18-27 bp,但不应大于38,因为过长会导致其延伸温度大于74℃,不适于Taq DNA聚合酶进行反应[2]。
● 2. 引物序列在模板内应当没有相似性较高,尤其是3’端相似性较高的序列,否则容易导致错配。
引物3’端出现3个以上的连续碱基,如GGG或CCC,也会使错误引发机率增加[2]。
● 3. 引物3’端的末位碱基对Taq酶的DNA合成效率有较大的影响。
不同的末位碱基在错配位置导致不同的扩增效率,末位碱基为A的错配效率明显高于其他3个碱基,因此应当避免在引物的3’端使用碱基A[3][4]。
另外,引物二聚体或发夹结构也可能导致PCR 反应失败。
5’端序列对PCR影响不太大,因此常用来引进修饰位点或标记物[2]。
● 4. 引物序列的GC含量一般为40-60%,过高或过低都不利于引发反应。
上下游引物的GC含量不能相差太大[2][5]。
● 5. 引物所对应模板位置序列的Tm值在72℃左右可使复性条件最佳。
Tm值的计算有多种方法,如按公式Tm=4(G+C)+2(A+T),在Oligo软件中使用的是最邻近法(the nearest neighbor method) [6][7]。
● 6. ΔG值是指DNA双链形成所需的自由能,该值反映了双链结构内部碱基对的相对稳定性。
应当选用3’端ΔG值较低(绝对值不超过9),而5’端和中间ΔG值相对较高的引物。
引物的3’端的ΔG值过高,容易在错配位点形成双链结构并引发DNA聚合反应[6]。
●7. 引物二聚体及发夹结构的能值过高(超过4.5kcal/mol)易导致产生引物二聚体带,并且降低引物有效浓度而使PCR反应不能正常进行[8]。
●8. 对引物的修饰一般是在5’端增加酶切位点,应根据下一步实验中要插入PCR产物的载体的相应序列而确定。
查找细胞系中基因序列的方法

查找细胞系中基因序列的方法文章标题:探索细胞系中基因序列的方法与挑战在当今科技发展迅猛的时代,生物学领域的研究也日新月异。
细胞系中基因序列的研究,对于理解生物学、疾病机制以及药物研发都具有重要意义。
然而,要想深入了解细胞系中基因序列,需要探索多种方法和技术,并面对众多挑战。
为了更好地了解细胞系中基因序列的方法和挑战,我们首先需要明确细胞系的概念。
细胞系是指源自同一个母细胞的一系列细胞,它们可以长时间稳定地传代培养,具有相似的形态、遗传特性和生物学行为。
在细胞系的研究中,寻找并验证基因序列是至关重要的一环。
一、细胞系中基因序列的方法1.1 PCR技术PCR(聚合酶链式反应)技术是目前应用最为广泛的一种方法,通过PCR 技术可以扩增和纯化细胞系中单个基因的DNA序列,为后续研究提供了重要的物质基础。
1.2 基因组测序技术随着高通量测序技术的发展,基因组测序技术也逐渐成熟。
研究者可以利用基因组测序技术,对整个细胞系的基因序列进行全面的测序和分析,从而获得更多的信息。
1.3 分子克隆技术分子克隆技术是一种常用的方法,可以将感兴趣的基因序列从细胞系中克隆出来,用于进一步的功能研究和表达。
以上这些方法,都可以帮助研究者在细胞系中寻找和验证基因序列,提供了丰富的实验手段。
二、细胞系中基因序列研究的挑战2.1 样本来源的复杂性细胞系来源的多样性和复杂性,是研究的首要挑战。
不同类型的细胞系可能存在着巨大的遗传变异,这需要研究者在样本来源的选择上格外谨慎。
2.2 基因重组的干扰在细胞系中进行基因序列研究时,可能会遇到基因重组等干扰因素,这些干扰会影响研究结果的准确性。
2.3 数据分析的复杂性对于大规模的基因组测序数据,数据分析将面临复杂多变的挑战。
研究者需要具备深厚的数据处理和分析能力,才能从海量数据中提取有效信息。
三、结语在细胞系中寻找和验证基因序列,需要综合运用多种方法和技术,并面对众多挑战。
对于研究者来说,需要不断更新知识、拓展视野,以更好地应对细胞系基因序列研究中的各种挑战。
一步一步教你使用NCBI查找DNA、mRNA、cDNA

一步一步教你使用 NCBI 查找DNA、mRNA、cDNA、...最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在 NCBI 上都可以方便的找到答案。
现在我就结合我自己使用 NCBI的一些经历(经验)跟大家交流一下 BCBI 的使用。
希望大家都能发表自己的使用心得,让我们共同进步!我分以下几个部分说一下 NCBI 的使用:Part one 如何查找基因序列、mRNA、PromoterPart two 如何查找连续的 mRNA、cDNA、蛋白序列Part three 运用 STS 查找已经公布的引物序列Part four 如何运用 BLAST 进行序列比对、检验引物特异性特别感谢本版版主,将这个帖子置顶!从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友!请大家对以下我发表的容提出自己的意见。
关于NCBI 其他方面的使用也请水平较高的战友给予补充First of all,还是让我们从查找基因序列开始。
第一部分利用Map viewer 查找基因序列、mRNA 序列、启动子(Promoter)下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:/mapview/index.html在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码、序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
应用NCBI的Map viewer查找基因序列

应用NCBI的Map viewer查找基因序列、mRNA序列、启动时间:2011-06-08 22:23来源:生物吧作者:刘浩点击: 796 次一步一步教你使用NCBI查找DNA、mRNA 、cDNA、Protein、引物设计、BLAST 序列比对等(作者:urbest) 关键词: NCBI 使用方法引物设计序列比对总共分为四个部分介绍一下NCBI的使用:Part one一步一步教你使用NCBI查找DNA、mRNA、cDNA、Protein、引物设计、BLAST序列比对等(作者:urbest)关键词: NCBI 使用方法引物设计序列比对总共分为四个部分介绍一下NCBI的使用:Part one 如何查找基因序列/mRNA/PromoterPart two 如何查找连续mRNA/cDNA/蛋白序列Part three 运用STS查找已经公布的引物序列Part four 如何运用BLAST进行序列比对、检验引物特异性Part one: 利用Map viewer查找基因序列、mRNA序列、启动子(Promoter)[同一个页面即可显示基因序列和启动子]下面以人的IL6(白细胞介素6)为例讲述一下具体的操作步骤一、打开Map viewer页面,网址为:/mapview/index.html在search的下拉菜单里选择物种,for后面填写你的目的基因。
操作完毕如图所示:二、点击“GO”出现如下页面:三、在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene前面的小方框里打勾,然后点击Filter,出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码、序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
现在普遍采用的是最上面的那个序列,这一条是世界范围的生物科学家用计算机合成的一个序列。
如何查找基因的序列(带图表)

e-133
gi|728993|sp|P40293|C79A_BOVIN B-cell antigen receptor comp...
312
3e-85
gi|126779|sp|P11911|C79A_MOUSE B-cell antigen receptor comp... 278
5e-75
gi|728994|sp|P40259|C79B_HUMAN B-cell antigen receptor comp... 55
1
2 3
比对结果会在10秒左右后出现, 注意结果很多,一直下拉右侧工具条,会看到很多不同意义的比对结果。
• Search BLAST (/BLAST/) for P11912
Database: All non-redundant(非冗余) sequences 6,507,231 sequences; 2,219,987,828 total letters
如果想找的基因是第一个序列即isoform a, 就可以点击NM_001025366.1, 得到如下界面:
If the gene sequence is known, and you want to find the corresponding GenBank ID, use /BLAST/ Click on the type of nucleotide search you want and enter the sequence. Results will be displayed with the GenBank ID included. To view information about a gene sequence with a given GenBank ID, go to /entrez/query.fcgi? db=Nucleotide
利用Pubmed Map viewer查找基因序列

利用Pubmed Map viewer查找基因序列、mRNA 序列、启动子(Promoter)来源:dxy/lovelifeangel 发布时间:2009-09-22 查看次数:2234下面以大鼠(rattus norvegicus)的结缔组织生长因子(C T G F)为例讲述一下具体的操作步骤1.打开Map viewer页面,网址/mapview/index.html在search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
现在普遍采用的是最上面的那个序列,这一条是世界范围的生物科学家用计算机合成的一个序列。
我也推荐大家使用这个序列。
4.点击上述三条序列第一条序列(即reference)对应的"Genes seq",出现新的页面,页面下方为:5.点击上图出现的“Download/View Sequence/Evidence ”,即下载查看序列等功能,结果如图所示:先对上面这张图做点简要的说明,在Sequence Format(序列输出格式)后面是一个下拉式选择菜单,默认的为FASTA 格式,还有一个是GenBank 格式。
我推荐大家选择GenBnak格式,因为这个格式提供了很多该基因的信息,而FASTA 格式只有基因序列。
6.在Sequence Format 后选择GenBank,然后点击下面的Display,目的基因的相关信息和序列就出现在眼前了。
步一步教你使用NCBI查找DNA、mRNA、cDNA

步一步教你使用N C B I查找D N A、m R N A、c D N A--------------------------------------------------------------------------作者: _____________--------------------------------------------------------------------------日期: _____________一步一步教你使用 NCBI 查找DNA、mRNA、cDNA、...最近看到很多战友在论坛上询问如何查询基因序列、如何进行引物设计、如何使用BLAST 进行序列比对……,这些问题在 NCBI 上都可以方便的找到答案。
现在我就结合我自己使用 NCBI的一些经历(经验)跟大家交流一下 BCBI 的使用。
希望大家都能发表自己的使用心得,让我们共同进步!我分以下几个部分说一下 NCBI 的使用:Part one 如何查找基因序列、mRNA、PromoterPart two 如何查找连续的 mRNA、cDNA、蛋白序列Part three 运用 STS 查找已经公布的引物序列Part four 如何运用 BLAST 进行序列比对、检验引物特异性特别感谢本版版主,将这个帖子置顶!从发帖到现在,很多战友对该帖给与了积极的关注,在此向给我投票的(以及想给我投票却暂时不能投票的)各位战友表示真诚的感谢,谢谢各位战友!请大家对以下我发表的内容提出自己的意见。
关于NCBI 其他方面的使用也请水平较高的战友给予补充First of all,还是让我们从查找基因序列开始。
第一部分利用Map viewer 查找基因序列、mRNA 序列、启动子(Promoter)下面以人的 IL6(白细胞介素 6)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址为:在 search 的下拉菜单里选择物种,for 后面填写你的目的基因。
如何查找基因的基本信息

如何查找基因的基本信息查找基因的基本信息,涉及到基因的编码区和引物设计等几个主要方面。
下面将详细介绍如何进行基因信息的查找。
第一步是确定所要查找的基因名称或基因序列。
基因名称通常使用基因符号或别名表示,例如TP53是TP53基因的基因符号,也可以通过其他名称如p53来。
如果已经有基因序列,可以直接在数据库中进行。
第二步是选择适当的数据库进行基因信息的。
常用的基因数据库包括:GenBank、EMBL、DDBJ、NCBI Gene、Ensembl等。
这些数据库都提供了基因的编码区和引物设计等基本信息。
第三步是进行基因信息的。
在选择数据库后,可以通过以下几种方式进行:1.基因名称:在数据库的栏中输入基因名称或基因符号,例如输入TP53或p53,以查找该基因的基本信息。
2.基因序列:如果已经有基因序列,可以将其复制粘贴到数据库的栏中进行。
数据库会自动匹配相似的序列并返回相应的基因信息。
3. Blast:如果只有部分基因序列或希望查找与已知序列相似的基因,可以使用基因比对工具如BLAST进行。
将序列输入到BLAST程序中,它会比对数据库中的序列并给出相似度高的匹配结果。
第四步是查找编码区信息。
在数据库中,可以找到基因的编码区序列、外显子和内含子的信息。
可以利用这些信息来了解基因的结构、序列特征和位置等。
第五步是引物设计。
根据所需引物的要求(如位点数量、长度、引物间的距离等),可以使用基因数据库提供的引物设计工具进行引物的设计。
常见的引物设计工具有Primer3、IDT PrimerQuest等。
这些工具可以根据基因序列自动设计合适的引物序列,并给出引物的物理化学参数和引物间的配对情况等信息。
第六步是验证基因信息。
在获得基因的基本信息、编码区和引物设计后,需要进行验证。
可以利用实验室技术如PCR、测序等对基因进行验证。
总结起来,查找基因的基本信息涉及到确定基因名称或序列、选择适当的数据库、进行基因信息的、查找编码区信息和引物设计等几个主要步骤。
已知目标基因名称如何在genbank里查找序列
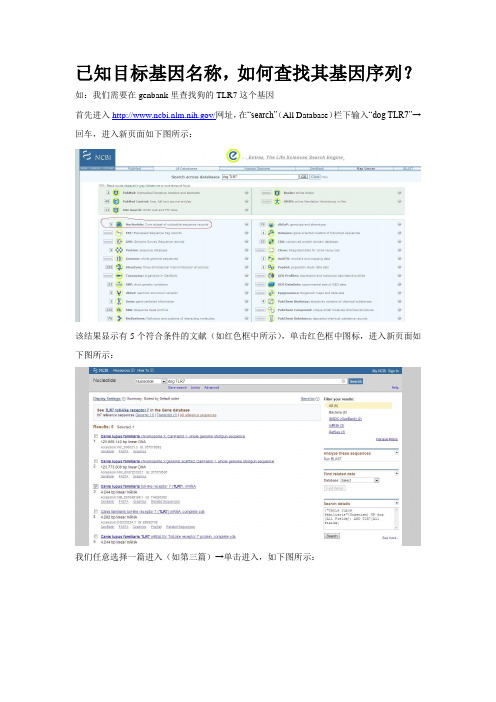
已知目标基因名称,如何查找其基因序列?如:我们需要在genbank里查找狗的TLR7这个基因
首先进入/网址,在“search”(All Database)栏下输入“dog TLR7”→回车,进入新页面如下图所示:
该结果显示有5个符合条件的文献(如红色框中所示),单击红色框中图标,进入新页面如下图所示:
我们任意选择一篇进入(如第三篇)→单击进入,如下图所示:
看起来非常之复杂,如果亲只需要这个序列用来设计PCR引物,那么这些都不用看,只需要单击文献下的“FASTA”(红色框内)即出现基因序列,如下图所示:
→然后就可以直接把这个序列复制下来发给试剂公司让他们设计引物→大功告成!!
PS:眼尖的亲们肯定看到了FASTA滴序列就是ORIGIN(上图黄色框所示)~~只是小写变大写~~格式更简明了!!
再PS:可能有亲会问明明你输入的是“dog TLR7”结果出来的是
差得有点远吧
正解“Canis lupus”=犬狼疮~~狗=犬也~~当初只怪我们太不专业啦~~所以这也提示亲们即使你不知道某个东东的具体学名是什么,按俗名搜也许会有意想不到滴结果哦!!祝各位亲们好运咯!!!。
NCBI数据库中搜索基因序列的私家套路三

NCBI数据库中搜索基因序列的私家套路三
作者:荷叶
转载请注明:解螺旋·临床医生科研成长平台
“荷叶师兄,教授让我研究新基因XXX的功能,我想问下…”
“小师妹,这个也是有套路的,首先你可以研究XXX在各器官、组织表达谱,其次……,你明白吧?”(师兄迫不及待的向小师妹传授经验之谈)
“哦!”(刚入科的小师妹好像明白怎么做了)
“你可以利用原核、真核表达系统将XXX表达出来研究其功能,再次……,你明白吧?”
“哦?”(小师妹感觉好多东西需要学)
“接下来你可以研究XXX的表达调控,……,你还可以……,你明白吧?师兄我硕士阶段就是这么做的。
”
“哦??”(小师妹感觉有点跟不上节奏)……
一个小时后师兄意犹未尽。
“师兄,我就是想问下在NCBI里如何快速、准确找到基因序列。
”(小师妹弱弱的说了一句)
“Easy!”荷叶师兄又开始传授经验。
套路一
进入NCBI网站,在Nucleotide(核苷酸)数据库中搜索,以人CCL2为例。
套路二
进入NCBI网站,在CCDS(共识编码序列)数据库中搜索,/projects/CCDS/CcdsBrowse.cgi
套路三(荷叶推荐)
进入NCBI网站,在Gene(基因)数据库中搜索。
找到T ools工具的下拉菜单中Sequence Text View,点击就可以查到基因序列,并且网站以不同颜色标注CCL2基因的不同部分,灰色的UTR,红色的外显子,绿色的内含子,以及一一对应的编码区。
如何查找基因的序列(带图表)

gi|125806|sp|P01658|KV3F_MOUSE IG KAPPA CHAIN V-III REGION ... 33
0.30
gi|125808|sp|P01659|KV3G_MOUSE IG KAPPA CHAIN V-III REGION ... 33
0.30
gi|1172451|sp|Q05793|PGBM_MOUSE Basement membrane-specific ... 33
Query: 181 Sbjct: 175
EYEDENLYEGLNLDDCSMYEDISRGLQGTYQDVGSLNIGDVQLEKP 226 +YEDENLYEGLNLDDCSMYEDISRGLQGTYQDVG+L+IGD QLEKP DYEDENLYEGLNLDDCSMYEDISRGLQGTYQDVGNLHIGDAQLEKP 220
如果想找的基因是第一个序列即isoform a, 就可以点击NM_001025366.1, 得到如下界面:
If the gene sequence is known, and you want to find the corresponding GenBank ID, use /BLAST/ Click on the type of nucleotide search you want and enter the sequence. Results will be displayed with the GenBank ID included. To view information about a gene sequence with a given GenBank ID, go to /entrez/query.fcgi? db=Nucleotide
在NCBI中查找基因并下载自己想要的前后序列

大功告成
• 现在你可以看到右边的序列了,但是很多时候都会有一些 N,那就说明这个序列在de novo过程中有一些GAP。这接 下来正是你发挥的好时候,可以通过PCR把这序列扩全。
大功告成?现在你可以看到右边的序列了但是很多时候都会有一些n那就说明这个序列在denovo过程中有一些gap
首先打开NCBI网址
/
• 选择Gene,后面填入基因名字和物种,如 GDF9 goat. • 点search
• 这是出现了你要的基因,点第一个就行。
• 进去后,你可以看到这个基因的所有信息,包括 所处的染色体、最新的文献、以及mRNA、蛋白、 DNA号。我们这里主要讲拉出该基因的前后两端 的序列,所以只关注以下部分。首先是位置号 40636994..40641698 ,其次是转录方向。
基因所处位置
序列
反向转录基因
ቤተ መጻሕፍቲ ባይዱ
• 网页往下走,可以看到Gnomic这一行。点击 GenBank.
• 这时,你看到这样一个界面,请注意红圈 那一块才是我们的重点。
您需要的基因区域,第4张 PPT里面的位置。你可以对区 域进行调整,如你需要该基 因的上游序列3000bp序列,由 于该基因反向转录,所以可 将后面的数字改成40644698. 然后点update view.
因为你需要得到序列,所以得把这 个勾上,如果你需要反向互补,也 可以勾上。记得一定要点update view.
在genbank中查找一基因的序列

如何在genbank中查找一基因的序列————————————————————————————————作者:————————————————————————————————日期:如何在genbank中查找一基因的序列1、在GeneBank 中查找基因序列只要输入accession号就可以了,下面网址就是一个基因的全部序列信息的例子,/Sitemap/samplerecord.html,在记录的末尾有各种记录的详细说明,如果你没有accession号,可以把你手头的编号用source等信息源转换成accession号,中文教程太古老了,如果你是初学者一定要养成看英文文献的习惯,要是特别想看中文翻译的话,书店里随便一本生物信息学书里都会介绍数据库的,不过有些翻译过来的东西真的很别扭,希望对你有帮助。
2、关于在GeneBank中查找序列我有几点体会:最直接、最简单的方法是手头有基因的accession号;如果没有就需要明确两个重要的内容,即基因名称及物种信息(如果有最好是拉丁全名),基因名称尽可能详细,避免搜出一些不相关的信息;搜索的时候建议先用NCBI的Gene数据库搜索,这样得到的accession号是属于NCBI工作人员重新整理过的Refseq的序列,这样会比较可靠;当然这个要看你的分析目的,如果你是要对该序列进行下游的分子生物学操作or分析,选这种序列我觉得会比较好,如果是要进行多序列的分析or其他目的需要全面分析该序列的,可能需要其他序列做补充,但是我觉得序列越多问题越说不清楚,因为毕竟不是自己的序列,如果Gene数据库里没有收录,那就只有在Nucleotide数据库里找了,但是还是建议采用Refseq的序列,Refseq序列特征如下:Accession prefix Molecule type CommentAC_ Genomic Complete genomic molecule, alternate assemblyNC_ Genomic Complete genomic molecule, reference assemblyNG_ Genomic Incomplete genomic regionNT_ Genomic Contig or scaffold, clone-based or WGSaNW_ Genomic Contig or scaffold, primarily WGSaNS_ Genomic Environmental sequenceNZ_b Genomic Unfinished WGSNM_ mRNANR_ RNAXM_c mRNA Predicted modelXR_c RNA Predicted modelAP_ Protein Annotated on AC_ alternate assemblyNP_ ProteinYP_c ProteinXP_c Protein Predicted modelZP_c Protein Predicted model, annotated on NZ_ genomic recordsa Whole Genome Shotgun sequence data.b An ordered collection of WGS for a genome.c Computed.其他值得考虑的是,对于真核生物最好找注释为全长的mRNA序列,原核生物最好有起始密码子和终止密码子;其他未尽事宜大家补充!3、如何在genbank查找某个细菌的基因序列?你输入这个细菌的名字直接查,一般会有的~~~~~而且一般第一个会是全基因组序列~~~进入ncbi的首页,database选nucleotide,输入你的关键词,如果库里收录里就会有的4、如何查找基因序列?——在Genbank中寻找目的基因的实例(1)根据文献搞reasearch肯定要读文献的,如果你曾经在文献中看到过你感兴趣的基因,而且文中还提到了该基因在Genbank中的ID号,那就好办了,直接打开 ,在Search后的下拉框中选择Nucleotide,把Genbank ID号输入GO前面的文本框中,点“GO”,就可以找到他了。
如何在NCBI查找基因序列

如何在NCBI查找基因序列在NCBI(美国国家生物技术信息中心)网站上查找基因序列是生物学和生物医学研究中常见的任务之一、NCBI提供了各种数据库和工具,可以轻松地和检索各种基因序列。
以下是一些可能有助于您查找基因序列的指南:1.登录NCBI网站:2.选择合适的数据库:- GenBank: GenBank是一个基因组、核酸序列和蛋白质序列的公共数据库。
您可以在GenBank上找到来自各个物种的多个基因序列。
- RefSeq: RefSeq是一个包含参考序列信息的数据库,包括基因组、转录本和蛋白质序列。
-GEO:GEO是一个基因表达谱数据库,可以提供基因序列在不同组织和条件下的表达情况。
- dbSNP: dbSNP是一个单核苷酸多态性(SNP)数据库,可以提供不同个体之间的基因序列差异信息。
3.使用工具:NCBI提供了多种工具和选项,可以帮助您查找特定基因序列。
-基础:在NCBI的主页上,您会看到一个栏。
在栏内输入基因名称、基因ID、关键词、物种等信息,然后按下按钮,系统会返回与您条件相匹配的结果。
-BLAST:BLAST(基本局部序列比对工具)是一个广泛使用的比对工具,可以用来查找特定序列。
您可以在主页上找到BLAST栏,将您的序列输入到栏内,选择相应的数据库,然后点击按钮,系统将返回与您的序列相似的其他序列。
4.进一步筛选和过滤结果:在结果页面,您可以使用不同的过滤选项来进一步筛选和过滤结果。
您可以根据物种、序列长度、相似性等进行筛选。
6.使用NCBIAPI和高级选项:如果您需要进一步定制化的和分析,您可以使用NCBI提供的API (应用程序编程接口)来自动化和批量处理。
此外,NCBI还提供了高级选项,可以通过高级菜单来设置更复杂的条件。
如何找基因全序列

利用Pubmed Map viewer查找基因序列、mRNA 序列、启动子(Promoter)遗传分子统计2010-03-24 16:11:24 阅读759 评论0字号:大中小下面以大鼠(rattus norvegicus)的结缔组织生长因子(C T G F)为例讲述一下具体的操作步骤1.打开Map viewer 页面,网址/mapview/index.html在search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:2.点击“GO”出现如下页面:3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene 前面的小方框里打勾,然后点击Filter. 出现下图:说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽管你分别点击后,序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
现在普遍采用的是最上面的那个序列,这一条是世界范围的生物科学家用计算机合成的一个序列。
我也推荐大家使用这个序列。
4.点击上述三条序列第一条序列(即reference)对应的"Genes seq",出现新的页面,页面下方为:5.点击上图出现的“Download/View Sequence/Evidence ”,即下载查看序列等功能,结果如图所示:先对上面这张图做点简要的说明,在Sequence Format(序列输出格式)后面是一个下拉式选择菜单,默认的为FASTA 格式,还有一个是GenBank 格式。
我推荐大家选择GenBnak格式,因为这个格式提供了很多该基因的信息,而FASTA 格式只有基因序列。
6.在Sequence Format 后选择GenBank,然后点击下面的Display,目的基因的相关信息和序列就出现在眼前了。
怎样使用NCBI搜索并下载基因序列

注意事项
NCBI的功能非常强大,熟练使用才能快速找到目标序列。
觉得实用投个票呗,共同学习!搜索键入“NCBI”,点击“搜索”,第一个就是官方主页,点击打开。
2
关键字查找
以H9亚型流感病毒HA基因下载为例,说明详细过程。
既然要下载基因序列,需要在栏目内找到“Nucleotide”,搜索栏内填入“Avian influenza virus H9 HA”,点击“search”。
3Leabharlann 添加限定词点击搜索后,我们看到页面中间显示了很多关于“Avian influenza virus H9 HA”的基因序列,如果其中的一个符合你的要求,就可以下载。如何不符合你的要求,可以继续在搜索条内添加限定,比如添加“2010”,即2010年分离到的病毒。
4
序列选定
找到目标基因序列后,我们要将其下载下来。以给出第一个序列为例,点击序列前面的“小方格”,将其选定。
怎样使用NCBI搜索并下载基因序列
分步阅读
NCBI是生物学科经常使用的搜索引擎,其收纳了大量的基因和蛋白序列,可以对这些序列进行比对、设计引物等,功能非常强大,下面是小编经常使用的一个功能,即基因序列信息的查找和下载,希望和大家一起学习。
工具/原料
NCBI,基因序列,查找,下载
方法/步骤
1
打开NCBI
5
下载序列
选定后,点击页面右上角的“Send to”,选择“file”,再将format选择为“FASTA”格式,点击“Create file”,将序列下载到自定的文件夹内。
6
用DNAStar打开
下载的序列为FASTA格式,需要使用相关的软件打开,小编经常使用的是DNAStar中的Editseq,这个功能还是很强大的。
如何查找基因序列?——在Genbank中寻找目的基因的实例

1. 根据文献搞reasearch肯定要读文献的,如果你曾经在文献中看到过你感兴趣的基因,而且文中还提到了该基因在Genbank中的ID号,那就好办了,直接打开 ,在Search后的下拉框中选择Nucleotide,把Genbank ID号输入GO前面的文本框中,点“GO”,就可以找到他了。
举例说明,例如:在2003年JBC的文章(Conditional Knock-out of Integrin-linked Kinase Demonstrates an Essential Role in Protein Kinase B/Akt Activation)中出现了“calreticulin (GenBank accession number gi 16151096)”,那么把“16151096”输入GO前面的文本框中,点“GO”,就可以找到该基因了(当然包括基因序列等相关信息)。
见附图在出现了检索结果界面(下图)后,直接点击红箭头所指的AY047586就可以看到基因的相关信息了...(呵呵,是不是有点太......easy了)这里需要指出一下,在显示基因的页面右侧有一个Link,点击后出现一个小菜单,里面是与该基因相关的链接,很有用的,值得一个一个地去看看,这里我就不多说了。
点击 AY047586后出现的界面如下:下面还有......最下面就是核酸序列(ORIGIN后面……)附件genbk005-2.JPG (143.55 KB)06-11-20 13:18样就得到了FASTA格式的序列文件,没有其他数字和格式的干扰。
附件06-11-20 13:272. 根据已经获得的基因的相关信息进行查找(待续......)正如路漫漫所说,如果只是知道基因的名字,怎么查序列呢?还是举例说明,比如我想做的基因名称是人的VEGF基因,那么怎么在Genbank中找到它呢?还是一步一步来...打开/在search后面的下拉框中选择Gene,然后在中间的文本框中输入基因名称“VEGF”,点击GO...搜索结果出来了,let me see... 啊,怎么这么多?689条,哪一条是我想要的基因呢?(作者注:这也许是大多数人对Genbank的第一印象,即东西太多了,不知道是哪个。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
如何找基因全序
————————————————————————————————作者:————————————————————————————————日期:
利用Pubmed Map viewer查找基因序列、mRNA 序列、启动子(Promoter)
遗传分子统计2010-03-24 16:11:24 阅读759 评论0字号:大中小
下面以大鼠(rattus norvegicus)的结缔组织生长因子(C T G F)为例讲述一下具体的操作步骤
1.打开Map viewer 页面,网址/mapview/index.html在search 的下拉菜单里选择物种,for 后面填写你的目的基因。
操作完毕如图所示:
2.点击“GO”出现如下页面:
3.在步骤二图示的右下角有一个Quick Filter,下面是让你选择的几个复选框,在Gene 前面的小方框里打勾,然后点击Filter. 出现下图:
说明一下:1、染色体的红色区域即为你的目的基因所处位置。
2、下面参考序列给出了三个,是不同的部门做出来的,经我验证,序列有微小的差异,但总体来说基本相同。
尽
管你分别点击后,序列代码等有所差异,但碱基基本一致,不影响大家研究分析序列。
现在普遍采用的是最上面的那个序列,这一条是世界范围的生物科学家用计算机合成的一个序列。
我也推荐大家使用这个序列。
4.点击上述三条序列第一条序列(即reference)对应的"Genes seq",出现新的页面,页面下方为:
5.点击上图出现的“Download/View Sequence/Evidence ”,即下载查看序列等功能,结果如图所示:
先对上面这张图做点简要的说明,在Sequence Format(序列输出格式)后面是一个下拉式选择菜单,默认的为FASTA 格式,还有一个是GenBank 格式。
我推荐大家选择GenBnak格式,因为这个格式提供了很多该基因的信息,而FASTA 格式只有基因序列。
6.在Sequence Format 后选择GenBank,然后点击下面的Display,目的基因的相关信息和序列就出现在眼前了。
点击后如图所示(网页较大,只抓取一小部分以作示范):
在上述打开的网页中,你可以看到基因长度,基因序列,以及这个基因是如何被报道出来的等各种信息。
你会看到: mRNA complement(join(2338..3702, 4082..4293, 4424..4675, 4861..5083, 5171..5454))这代表了从基因的2338 位开始就是转录区了,即我们常说的mRNA 片断,由于内含子的存在,所以mRNA 在DNA 序列上分成了几段。
CDS complement (join(3406..3702,4082..4293,4424..4675,4861..5083,5171.. 5230)) CDS 代表编码序列,即蛋白编码区是从3406开始的(ATG),由于剪接作用所以CDS 区也是不连续的。
说到这里,可能很多朋友都已经明白了promoter 即启动子区域在哪里了。
但我还是再唠叨几句:转录起始位点前面是基因的调控区,启动子区没有明显的位置定义,大家也只是猜测它的大体位置,如果你要研究promoter 区的话,建议你选择转录起始位点前的2000个碱基进行研究,一般默认的是这样。
当然你如果觉得长度太长不好研究的话,也可以只研究-1000 到0 这一千个碱基,因为一般情况下,启动子区的变异都在这个区域内。
这样大家就可以找到自己的目的基因序列和启动子了,这种方法可能使用的人不是很多,但我个人比较喜欢,因为它最大的优点是可以找到启动子区域和其他调控区域。
