遗传算法实验一
实验六:遗传算法求解TSP问题实验2篇

实验六:遗传算法求解TSP问题实验2篇第一篇:遗传算法的原理与实现1. 引言旅行商问题(TSP问题)是一个典型的组合优化问题,它要求在给定一组城市和每对城市之间的距离后,找到一条路径,使得旅行商能够在所有城市中恰好访问一次并回到起点,并且总旅行距离最短。
遗传算法作为一种生物启发式算法,在解决TSP问题中具有一定的优势。
本实验将运用遗传算法求解TSP问题,以此来探讨和研究遗传算法在优化问题上的应用。
2. 遗传算法的基本原理遗传算法是模拟自然界生物进化过程的一种优化算法。
其基本原理可以概括为:选择、交叉和变异。
(1)选择:根据问题的目标函数,以适应度函数来评估个体的优劣程度,并按照适应度值进行选择,优秀的个体被保留下来用于下一代。
(2)交叉:从选出的个体中随机选择两个个体,进行基因的交换,以产生新的个体。
交叉算子的选择及实现方式会对算法效果产生很大的影响。
(3)变异:对新生成的个体进行基因的变异操作,以保证算法的搜索能够足够广泛、全面。
通过选择、交叉和变异操作,不断迭代生成新一代的个体,遗传算法能够逐步优化解,并最终找到问题的全局最优解。
3. 实验设计与实施(1)问题定义:给定一组城市和每对城市之间的距离数据,要求找到一条路径,访问所有城市一次并回到起点,使得旅行距离最短。
(2)数据集准备:选择适当规模的城市数据集,包括城市坐标和每对城市之间的距离,用于验证遗传算法的性能。
(3)遗传算法的实现:根据遗传算法的基本原理,设计相应的选择、交叉和变异操作,确定适应度函数的定义,以及选择和优化参数的设置。
(4)实验流程:a. 初始化种群:随机生成初始种群,每个个体表示一种解(路径)。
b. 计算适应度:根据适应度函数,计算每个个体的适应度值。
c. 选择操作:根据适应度值选择一定数量的个体,作为下一代的父代。
d. 交叉操作:对父代进行交叉操作,生成新的个体。
e. 变异操作:对新生成的个体进行变异操作,以增加搜索的多样性。
遗传算法求函数最大值实验报告

遗传算法求函数最大值实验报告遗传算法是一种模拟自然界进化过程的优化算法,它通过模拟生物进化过程中的遗传、交叉和变异等机制,逐步优化解空间中的个体,以找到问题的最优解。
在本次实验中,我们利用遗传算法来求解一个函数的最大值。
下面我们将详细介绍实验的过程和结果。
首先,我们选择了一个简单的函数作为实验对象,即f(x) = x^2,在x的范围为[-10, 10]。
我们的目标是找到使函数值最大的x。
首先,我们需要定义遗传算法中的基本元素,包括编码方式、适应度函数、选择策略、交叉和变异操作等。
在本实验中,我们选择二进制编码方式,将x的范围[-10, 10]离散化为10位的二进制编码。
适应度函数即为f(x) = x^2,它表示个体的适应度。
选择策略采用轮盘赌选择算法,交叉操作采用单点交叉,变异操作采用随机位变异。
接下来,我们需要初始化种群,并迭代进行交叉和变异操作,直到满足终止条件。
在每一代中,我们根据适应度函数对种群中的个体进行评估,并根据选择策略选择父代个体进行交叉和变异操作。
通过交叉和变异操作,产生新的子代个体,并替代原有种群中的个体。
在本次实验中,我们设置了100个个体的种群,并进行了100代的迭代。
实验结果显示,经过多次迭代,算法逐渐优化到了最优解。
最终找到了使函数值最大的x,即x=10,对应的函数值为100。
总结起来,本次实验利用遗传算法求解函数的最大值,展示了遗传算法在优化问题中的应用。
通过适当选择编码方式、适应度函数和操作策略,我们可以有效地找到问题的最优解。
在后续的研究中,我们可以进一步探索遗传算法在更复杂问题上的应用,并通过改进算法的参数和操作策略来提高算法的性能。
人工智能导论实验(遗传算法)-参考模板

环境配置1.安装anaconda,并配置环境变量2.Win+R运行cmd打开命令行窗口,在命令行中创建并激活所需的Python环境,也可直接使用默认的base环境a)创建:conda create -n [新环境的名字] python=[Python版本号]比如:conda create -n myEnv python=3.7b)激活环境:conda activate [环境名]。
激活成功后命令行前面会有个括号显示当前使用的环境名:3.检查当前环境下是否已有需要用到的库,若没有,则需要安装a)查询命令:conda listb)安装新的库:conda install [库名]也可指定库的版本号:conda install [库名]=[版本号]4.执行指定的python文件:python [.py文件名]如果.py文件不在当前路径下,需要指定文件的完整路径完成下列实验1,2以及3、4、5任选其二。
实验1:产生式系统1.基本要求1.1掌握产生式系统的基本原理1.2运行产生式系统的示例代码1.3尝试向示例代码中添加新数据,并完成相应的推理2.实验报告2.1总结产生式系统的基本原理2.2产生式系统的源代码分析与实验记录2.3尝试向示例代码中添加新数据,并完成相应的推理3.作业无实验2:AStar求解八数码问题1.基本要求1.1掌握AStar算法的基本原理1.2编写并运行AStar算法求解八数码问题的示例代码。
给定矩阵初始状态,允许将0与相邻的4个数字之一交换,直到矩阵转变为目标状态。
输出每一步交换后的矩阵例12.实验报告2.1 总结AStar算法的基本原理2.2 如何描述八数码问题中两个状态间的距离?2.2 如何根据状态距离将八数码问题转换为AStar寻路问题?3.作业提交编写的AStar求解八数码问题代码实验3:AStar求解迷宫寻路问题1.基本要求1.1掌握AStar算法的基本原理1.2编写并运行AStar算法求解迷宫寻路问题的示例代码。
遗传算法实验报告

遗传算法实验报告遗传算法实验报告引言:遗传算法是一种模拟生物进化过程的优化算法,通过模拟自然选择、遗传变异和交叉等操作,逐步优化问题的解。
本实验旨在探究遗传算法在解决优化问题中的应用,并通过实验验证其效果。
一、实验背景遗传算法最早由美国科学家约翰·霍兰德于20世纪60年代提出,其灵感来源于达尔文的进化论。
遗传算法通过基因编码、适应度评估、选择、交叉和变异等操作,模拟了进化过程中的遗传和变异,从而找到问题的最优解。
二、实验目的本实验旨在通过遗传算法解决一个经典的优化问题,验证其在解决实际问题中的有效性。
同时,对遗传算法的参数设置和操作过程进行调整和优化,以提高算法的性能。
三、实验步骤1. 问题定义:选择一个经典的优化问题,例如旅行商问题(TSP)或背包问题。
2. 解空间建模:将问题的解表示为染色体,设计基因编码方式。
3. 适应度函数定义:根据问题的特点,设计一个能够评估染色体解的适应度函数。
4. 初始化种群:随机生成一组初始染色体,作为种群。
5. 选择操作:根据适应度函数,选择一部分较优秀的染色体作为父代。
6. 交叉操作:通过交叉操作,生成新的子代染色体。
7. 变异操作:对子代染色体进行变异操作,引入新的基因变异。
8. 适应度评估:计算新的子代染色体的适应度。
9. 父代替换:根据适应度函数,选择一部分较优秀的子代染色体替换掉父代染色体。
10. 终止条件判断:判断是否满足终止条件,若满足则结束算法,否则返回步骤5。
11. 输出结果:输出最优解及其适应度值。
四、实验结果与分析通过实验,我们得到了一组优化问题的最优解,并计算出其适应度值。
通过观察实验结果,我们可以发现遗传算法在解决优化问题中的有效性。
同时,我们还可以通过调整遗传算法的参数和操作过程,进一步提高算法的性能。
五、实验总结通过本次实验,我们深入了解了遗传算法的原理和应用。
遗传算法作为一种优化算法,具有较强的适应性和鲁棒性,在解决实际问题中具有广泛的应用前景。
实验五:遗传算法求解函数最值问题实验

EJdoublefitness(constCh^crrcscrcRc)
{
doublek,y;
decode(cx# y);
retiurnf(x, y)+5;
2•交叉操作
首先是根据交叉概率probCross选择要交叉的个体进行交叉
//根据交叉槪率进行交叉
for(inti=pre = -1;i<N; i++)
//根据变异概率迸行变异
for (inti=0;i<N; i++)
if(randQm61() < probMutation)
mutate(group[i]);
变异时先随机生成变异的位置,然后把改位的01值翻转。
//变异操作
Hvoidmutate(ChrcfnosomeS c)
{
//随机选行翻转
inti-rand()%len; c-g[i] =
//骑越
boolflag-令;
for(inti = j=0; i<1.亡n;i++)
swap(d.g[l]Jc2.g[i]);
if(i== crosspcint[j])
//妇杲若干个交叉点重合”则效果叠加 "偃数个交叉点效果瑁当于没有交叉点while(j<
flag = [flag;
3.变异操作
首先是根据变异概率probMutation选择要变异的个体
经过一定的进化之后得到最终种群,从中选择最优的个体即可得
到最终的结果。
//获取种群最优你
-intgetOptimal(Ch^crescr:-oup[mxn]doubled—double&』doubled\)
遗传算法实验指导书

实验二:遗传算法一、实验目的1.学会使用遗传算法matlab示例程序辅助学习2.理解遗传算法的原理及机制二、实验原理遗传算法(Genetic Algorithm)是模拟达尔文的遗传选择和自然淘汰的生物进化过程的计算模型,是一种通过模拟自然进化过程搜索最优解的方法,它是由美国Michigan大学J.Holland教授于1975年首先提出来的,并出版了颇有影响的专著《Adaptation in Natural and Artificial Systems》,GA这个名称才逐渐为人所知,J.Hilland教授所提出的GA通常为简单遗传算法(SGA)。
遗传算法是从代表问题可能潜在的解集的一个种群(population)开始的,而一个种群则由经过基因(gene)编码的一定数目的个体(individual)组成。
每个个体实际上是染色体(chromosome)带有特征的实体。
染色体作为遗传物质的主要载体,即多个基因的集合,其内部表现(即基因型)是某种基因组合,它决定了个体的形状的外部表现,如黑头发的特征是由染色体中控制这一特征的某种基因组合决定的。
因此,在一开始需要实现从表现型到基因型的映射即编码工作。
由于仿照基因编码的工作很复杂,我们往往进行简化,如二进制编码,初代种群产生之后,按照适者生存和优胜劣汰的原理,逐代(generation)演化产生出越来越好的近似解,在每一代,根据问题域中个体的适应度(fitness)大小挑选(selection)个体,并借助于自然遗传学的遗传算子(genetic operators)进行组合交叉(crossover)和变异(mutation),产生出代表新的解集的种群。
这个过程将导致种群像自然进化一样的后生代种群比前代更加适应于环境,末代种群中的最优个体经过解码(decoding),可以作为问题近似最优解。
三、实验条件1.Matlab 7.0以上版本遗传算法演示程序2.IE6.0,能通过INTERNET查阅相关资料四、实验内容1.启动GA示例程序1)启动Matlab 7.02)单击help菜单,在弹出的下拉菜单中选择“demos”3)在“demos”窗口左边的树形选择栏选择“toolbox”中“Genetic Algorithm and Direct Search Demos”分支,“demos”窗口右边即为遗传算法示例程序列表。
遗传算法实验报告

遗传算法实验报告遗传算法实验报告引言:遗传算法是一种模拟自然选择和遗传机制的优化算法,它通过模拟基因的变异和交叉来寻找问题的最优解。
本实验旨在通过编写遗传算法程序,探索其在求解优化问题中的应用。
一、实验背景遗传算法最初是为了模拟达尔文的进化论而提出的。
通过模拟自然界中的进化过程,遗传算法可以逐步优化解空间,找到问题的最优解。
遗传算法适用于各种优化问题,如旅行商问题、背包问题等。
二、实验目的本实验旨在通过编写遗传算法程序,研究其在求解优化问题中的效果。
具体目标如下:1. 熟悉遗传算法的基本原理和流程;2. 实现一个简单的遗传算法程序;3. 运用该程序求解一个具体的优化问题。
三、实验步骤1. 确定问题:选择一个具体的优化问题,如旅行商问题;2. 设计编码方式:将问题转化为遗传算法可以处理的编码形式,如二进制编码;3. 初始化种群:随机生成一定数量的个体作为初始种群;4. 评估适应度:根据问题的目标函数,计算每个个体的适应度;5. 选择操作:根据适应度选择优秀的个体作为父代,采用轮盘赌等方法进行选6. 交叉操作:对选出的父代进行交叉操作,生成新的个体;7. 变异操作:对新个体进行变异操作,引入随机性;8. 更新种群:将原种群和新生成的个体合并,更新种群;9. 判断终止条件:判断是否满足终止条件,如达到最大迭代次数或找到最优解;10. 输出结果:输出找到的最优解。
四、实验结果本实验选择旅行商问题作为优化问题进行求解。
旅行商问题是一个经典的组合优化问题,目标是找到一条最短路径,使得旅行商可以依次访问一系列城市并回到起点。
经过多次实验,得到了如下结果:- 初始种群大小为100个个体;- 迭代次数为1000次;- 交叉概率为0.8,变异概率为0.01;- 最优解为路径长度为1000的路径。
五、实验分析通过对遗传算法的实验结果进行分析,可以得出以下结论:1. 遗传算法能够在一定程度上寻找到问题的最优解,但并不能保证一定找到全局最优解;2. 实验中的参数设置对算法的性能有很大影响,不同的参数设置可能导致不同的结果;3. 遗传算法适用于解决各种优化问题,但对于复杂问题可能需要更多的优化和六、实验总结通过本次实验,我们深入了解了遗传算法的原理和应用。
TSP问题遗传算法求解实验报告

一、旅行商问题所谓旅行商问题(Travelling Salesman Problem , TSP),即最短路径问题,就是在给定的起始点S到终止点T的通路集合中,寻求距离最小的通路,这样的通路成为S点到T点的最短路径。
在寻找最短路径问题上,有时不仅要知道两个指定顶点间的最短路径,还需要知道某个顶点到其他任意顶点间的最短路径。
遗传算法方法的本质是处理复杂问题的一种鲁棒性强的启发性随机搜索算法,用遗传算法解决这类问题,没有太多的约束条件和有关解的限制,因而可以很快地求出任意两点间的最短路径以及一批次短路径。
假设平面上有n个点代表n个城市的位置, 寻找一条最短的闭合路径, 使得可以遍历每一个城市恰好一次。
这就是旅行商问题。
旅行商的路线可以看作是对n 个城市所设计的一个环形, 或者是对一列n个城市的排列。
由于对n个城市所有可能的遍历数目可达(n- 1)!个, 因此解决这个问题需要0(n!)的计算时间。
假设每个城市和其他任一城市之间都以欧氏距离直接相连。
也就是说, 城市间距可以满足三角不等式, 也就意味着任何两座城市之间的直接距离都小于两城市之间的间接距离。
二、遗传算法1 遗传算法介绍遗传算法是由美国J.Holland教授于1975年在他的专著《自然界和人工系统的适应性》中首先提出的,它是一类借鉴生物界自然选择和自然遗传机制的随机化搜索算法。
通过模拟自然选择和自然遗传过程中发生的繁殖、交叉和基因突变现象,在每次迭代中都保留一组候选解,并按某种指标从解群中选取较优的个体,利用遗传算子(选择、交叉和变异)对这些个体进行组合,产生新一代的候选解群,重复此过程,直到满足某种收敛指标为止。
遗传算法在本质上是一种不依赖具体问题的直接搜索方法,是一种求解问题的高效并行全局搜索方法。
其假设常描述为二进制位串,位串的含义依赖于具体应用。
搜索合适的假设从若干初始假设的群体集合开始。
当前种群成员通过模仿生物进化的方式来产生下一代群体,如随机变异和交叉。
遗传算法实验原理

遗传算法实验原理
嘿,朋友!今天咱来聊聊遗传算法实验原理。
这玩儿意啊,就像是大自然中生物进化的奇妙过程在计算机里的奇妙再现!想象一下,大自然如何让物种不断适应环境,变得越来越强大?遗传算法就是模仿这个过程的啦!
比如说,我们来假设要找一个最优的解决方案,就像是要找到最合适的出行路线。
遗传算法就像一群小精灵,它们不断尝试、组合和变异,一代一代地寻找更好的答案。
就好像你在一堆路线中不断摸索,尝试不同的走法,直到找到最快捷的那条路!
在这个过程中呢,每个小精灵都有自己的特点,就像每个人都有自己的性格一样。
然后通过遗传的操作,好的特点被保留下来,不好的被淘汰。
这不正像是现实生活中优秀的特质会被传承下去吗?而且你知道吗,这个过程是非常神奇的!有时候可能一开始那些小精灵看起来不咋地,但经过一代代的进化,哇塞,它们变得超级厉害!就好像原本很普通的一个人,经过不断努力和积累,最终变得非常出色!
那怎么实现这个过程呢?首先要有一个初始的种群,就像是一群种子选手。
然后通过各种计算和操作来模拟生物的遗传过程。
这个过程中充满了惊
喜和不确定性,就像你永远不知道下一秒会发生什么!有时候一个小小的改变,可能会带来巨大的不同,这是多么令人兴奋的事情啊!
我觉得遗传算法实验原理真的超级有趣,它让我们看到了在计算机世界里也能有如此神奇的进化和变化。
它就像是打开了一扇通往奇妙世界的大门,让我们可以尽情探索和尝试,去发现那些意想不到的惊喜!所以啊,大家快来一起感受遗传算法的魅力吧!。
《遗传算法》实验指导书

JC(b,as,c,N,p);
cout<<"交叉结果如下:"<<endl;
for(i=0;i<N;i++)
{
for(j=7;j>=0;j--)
cout<<b[i][j];
cout<<endl;
}
return 0;
}
实 验 六
辽宁鞍山科技大学理学院(系) 2010年 月 日
scanf("%d",&m);
sje(n,m);
printf("\n");
printf("请输入转换的二进制数长度:\n");
scanf("%d",&x);
printf("请输入转换的二进制数:\n");
for(i=x-1;i>=0;i--)
scanf("%d",&a[i]);
ejs(a,x);
}
实 验 三
{
b[i][j]=a[j];
a[j]=0;
}
}
cout<<endl<<"初始种群如下:"<<endl;
for(i=0;i<N;i++)
{
for(j=7;j>=0;j--)
cout<<b[i][j];
cout<<endl;
}
cout<<endl;
int *as=new int[N];
f(N,as);
人工智能化遗传算法实验报告

人工智能实验报告学号:姓名:实验名称:遗传算法实验日期:2016.1.5【实验名称】遗传算法【实验目的】掌握遗传算法的基本原理,熟悉遗传算法的运行机制,学会用遗传算法来求解问题。
【实验原理】遗传算法(Genetic Algorithm)是模拟达尔文生物进化论的自然选择和遗传学机理的生物进化过程的计算模型,是一种通过模拟自然进化过程搜索最优解的方法。
遗传算法是从代表问题可能潜在的解集的一个种群开始的,而一个种群则由经过基因编码的一定数目的个体组成。
每个个体实际上是染色体带有特征的实体。
在一开始需要实现从表现型到基因型的映射即编码工作。
由于仿照基因编码的工作很复杂,我们往往进行简化,如二进制编码,初代种群产生之后,按照适者生存和优胜劣汰的原理,逐代演化产生出越来越好的近似解,在每一代,根据问题域中个体的适应度大小选择个体,并借助于自然遗传学的遗传算子进行组合交叉和变异,产生出代表新的解集的种群。
这个过程将导致种群像自然进化一样的后生代种群比前代更加适应于环境,末代种群中的最优个体经过解码,可以作为问题近似最优解。
遗传算法程度流程图为:【实验内容】题目:已知f(x)=x*sin(x)+1,x∈[0,2π],求f(x)的最大值和最小值。
数据结构:struct poptype{double gene[length];//染色体double realnumber;//对应的实数xdouble fitness;//适应度double rfitness;//相对适应度double cfitness;//累计适应度};struct poptype population[popsize+1];//最后一位存放max/min struct poptype newpopulation[popsize+1];//染色体编码:[0,2]x π∈,变量长度为2 π,取小数点后6位,由于2262322*102;π<<因此,染色体由23位字节的二进制矢量表示,则X 与二进制串(<b 22 b 21…… b 0>)2之间的映射如下:()2222212010bb ......b 2'i i i b x =⎛⎫=∙= ⎪⎝⎭∑;232'21x x π=- 适应度函数:由于要求f(x)的最值,所以适应度函数即可为f(x)。
关于遗传算法的实验报告
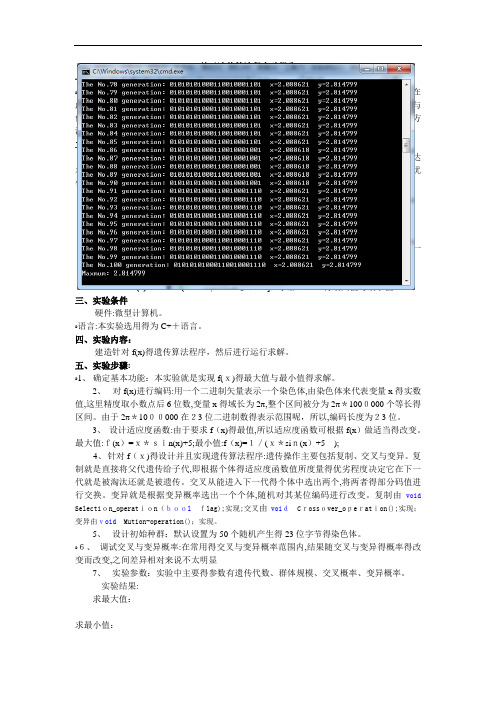
关于遗传算法得实验报告一、实验目得:ﻩ理解与掌握遗传算法得应用及意义,能用一门自己擅长得语言实现遗传算法得基本功能,在此基础上进一步理解与巩固对遗传算法得重要,以便在今后得学习与工作中能有效得运用与借鉴!需要指出得就是遗传算法并不就是能保证所得到得就就是最佳得答案但通过一定得方法可以将误差控制在一定得范围内!二、实验原理与题目:1、遗传算法就是一种基于空间搜索得算法,它通过自然选择、遗传、变异等操作以及达尔文得适者生存得理论,模拟自然进化过程来寻找所求问题得答案。
其求解过程就是个最优化得过程。
一般遗传算法得主要步骤如下:(1)随机产生一个确定长度得特征字符串组成得初始种群。
(2)对该字符串种群迭代地执行下面得步骤a与步骤b,直到满足停止准则为止:a计算种群中每个个体字符串得适应值;b应用复制、交叉与变异等遗传算子产生下一代种群。
(3)把在后代中表现得最好得个体字符串指定为遗传算法得执行结果,即为问题得一个解。
2、通过编码、设置种群、设置适应度函数、遗传操作、解码产生需要得解。
f(x)=x*sin(x)+1,x∈[0,2π],求解f(x)得最大值与最小值。
三、实验条件硬件:微型计算机。
ﻩ语言:本实验选用得为C++语言。
四、实验内容:建造针对f(x)得遗传算法程序,然后进行运行求解。
五、实验步骤:ﻩ1、确定基本功能:本实验就是实现f(x)得最大值与最小值得求解。
2、对f(x)进行编码:用一个二进制矢量表示一个染色体,由染色体来代表变量x得实数值,这里精度取小数点后6位数,变量x得域长为2π,整个区间被分为2π*1000000个等长得区间。
由于2π*1000000在23位二进制数得表示范围呢,所以,编码长度为23位。
3、设计适应度函数:由于要求f(x)得最值,所以适应度函数可根据f(x)做适当得改变。
最大值:f(x)=x*sin(x)+5;最小值:f(x)=1/(x*sin(x)+5);4、针对f(x)得设计并且实现遗传算法程序:遗传操作主要包括复制、交叉与变异。
遗传算法实验报告

遗传算法实验报告一、实验目的遗传算法是一种基于自然选择和遗传机制的优化算法,本次实验的主要目的是深入理解遗传算法的原理和工作机制,并通过实际编程实现来解决特定的优化问题,观察其性能和效果。
二、实验原理遗传算法模拟了生物进化的过程,通过对一组潜在的解决方案(称为个体或染色体)进行选择、交叉和变异操作,逐步迭代优化,以找到最优或近似最优的解。
在遗传算法中,每个个体都由一组基因表示,这些基因对应于问题的参数。
适应度函数用于评估每个个体的优劣程度,适应度高的个体更有可能被选择进行繁殖,产生下一代个体。
选择操作通常基于个体的适应度比例,适应度高的个体有更高的概率被选中。
交叉操作将两个父代个体的基因部分组合,生成新的子代个体。
变异操作则以一定的概率随机改变个体的某些基因,以增加种群的多样性。
三、实验环境本次实验使用 Python 编程语言,主要依赖的库有 numpy 用于数组操作,matplotlib 用于结果可视化。
四、实验步骤1、问题定义确定要优化的问题,例如求解函数的最大值或最小值,或者在给定约束条件下寻找最优的参数组合。
定义适应度函数,用于衡量每个个体的优劣。
2、编码方案确定如何将问题的解编码为染色体的形式。
常见的编码方式有二进制编码、实数编码等。
3、初始化种群随机生成一定数量的初始个体,组成初始种群。
4、选择操作根据个体的适应度计算选择概率,使用轮盘赌选择或其他选择方法选择父代个体。
5、交叉操作对选中的父代个体进行交叉,生成子代个体。
6、变异操作以一定的概率对个体的基因进行变异。
7、迭代更新重复进行选择、交叉和变异操作,生成新的种群,直到满足终止条件(如达到最大迭代次数或找到满意的解)。
8、结果分析对最终得到的最优个体进行解码,得到问题的解。
分析遗传算法的性能,如收敛速度、解的质量等。
五、实验结果与分析以求解函数 f(x) = x^2 在区间 0, 10 上的最大值为例,进行了遗传算法的实验。
1、适应度函数定义适应度函数直接采用目标函数 f(x) = x^2 ,即适应度越高,函数值越大。
正交实验 遗传算法

正交实验遗传算法正交实验是一种用于设计和优化复杂系统的实验方法,而遗传算法是一种基于生物进化原理的优化算法。
本文将介绍如何将正交实验与遗传算法相结合,以提高实验效果和优化结果。
我们来了解一下正交实验。
正交实验是一种通过设计独立变量的组合来探究因素对系统性能的影响的方法。
正交实验的核心思想是通过选择一组正交的试验条件,以尽量减少实验次数的同时保证实验结果的可靠性和可解释性。
正交实验的设计原则包括选择适当的因素水平、确定正交表和选择试验次数等。
而遗传算法是一种模拟生物进化过程的优化算法。
它通过模拟自然界的遗传、交叉和变异等过程,不断迭代地优化目标函数,以找到最优解。
遗传算法的基本流程包括初始化种群、评估适应度、选择操作、交叉和变异等步骤。
通过不断迭代优化,遗传算法能够在复杂的搜索空间中找到最优解。
将正交实验与遗传算法相结合,可以充分利用正交实验的设计原则和遗传算法的优化能力,有效地提高实验效果和优化结果。
具体而言,可以通过以下步骤来实现正交实验遗传算法:第一步是正交实验的设计。
根据实验目标和因素的种类与水平,选择合适的正交表,确定试验因素的水平。
正交表的选择应满足变量之间的正交性,以减少实验次数。
通过正交实验的设计,我们可以得到一组试验方案。
第二步是初始化种群。
将试验方案转化为遗传算法的染色体编码形式,并根据问题的具体情况,设置染色体的长度和编码方式。
根据正交实验的设计结果,生成初始种群,种群中的每个个体代表一个试验方案。
第三步是评估适应度。
根据问题的具体要求,设计适应度函数来评估每个个体的适应度。
适应度函数应能够准确地反映个体的性能指标,以便进行选择操作。
第四步是选择操作。
根据个体的适应度,利用选择算子选择优秀个体。
选择算子可以根据适应度大小进行比例选择、竞争选择等。
选择操作的目的是保留优秀个体,并为其提供繁殖的机会。
第五步是交叉操作。
通过交叉算子对选择的个体进行交叉,产生新的个体。
交叉操作的目的是模拟生物界的交配过程,增加种群的多样性,避免陷入局部最优解。
遗传算法解决函数优化问题

遗传算法解决函数优化问题实验⼀遗传算法解决函数优化问题XXXXXXXXXX⼀、实验⽬的1.掌握遗传算法的基本原理和步骤。
2.复习VB 、VC 的基本概念、基本语法和编程⽅法,并熟练使⽤VB 或VC 编写遗传算法程序。
⼆、实验设备微机三、实验原理遗传算法是⼀类随机优化算法,但它不是简单的随机⽐较搜索,⽽是通过对染⾊体的评价和对染⾊体中基因的作⽤,有效地利⽤已有信息来指导搜索有希望改善优化质量的状态。
标准遗传算法流程图如图1.1所⽰,主要步骤可描述如下:①随机产⽣⼀组初始个体构成初始种群。
②计算每⼀个体的适配值(fitness value ,也称为适应度)。
适应度值是对染⾊体(个体)进⾏评价的⼀种指标,是GA 进⾏优化所⽤的主要信息,它与个体的⽬标值存在⼀种对应关系。
③判断算法收敛准则是否满⾜,若满⾜,则输出搜索结果;否则执⾏以下步骤。
④根据适应度值⼤⼩以⼀定⽅式执⾏复制操作(也称为选择操作)。
⑤按交叉概率p c 执⾏交叉操作。
⑥按变异概率p m 执⾏变异操作。
⑦返回步骤②。
四、实验内容及步骤1.上机编写程序,解决以下函数优化问题:()221min 10i i i f x x =??=≤∑X2.调试程序。
3.根据实验结果,撰写实验报告。
图1.1 标准遗传算法流程图五、实验程序% % 清⼯作空间workspace,清屏幕显⽰% clear all; clc;%% tic; % 启动计时器%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%% % 参数赋值PopSize =30; % 种群规模Pc =0.65; % 交叉概率Pm =0.01; % 变异概率precision =22; % 根据精度要求,⼆进制字符串长度为22 iterative_thre =20; % 若连续iterative_thre次解⽆改进,则退出遗传算法%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%% % 初始化变量fitness = zeros(PopSize,1); % 存放所有染⾊体的适应度值SelectRate = zeros(PopSize,1); % 存放染⾊体的选择概率AccumulateRate = zeros(PopSize,1); % 存放染⾊体的累积概率num =0; % 结束遗传算法控制量bestfitness = 0; % 存放进化过程中最优的适应度值bestX =0; % 存放进化过程中最优解population = dec2bin(rand(PopSize,1)*(2^precision));% 产⽣初始群体,共PopSize个[0,2^precision]之间的⼗进制随机数,并将⼗进制数转换为⼆进制数newPop = population; % newPop存放新染⾊体的编码% 利⽤遗传算法求解最优解while num <= iterative_thretempX =bin2dec(population); % 将⼆进制串代表的⼆进制数转化为⼗进制数x’X = -1+tempX*3/(2^precision-1);% 按⽐例关系,将x'转化为区间[-1,2]内的实数x fitness = X.*sin(10*pi.*X)+2; % 计算适应度[thisbestfit,id] = max(fitness); % 找到当前最优适应度值thisbestX = X(id); % 找到与当前最优适应度值相对应的当前最优解if thisbestfit > bestfitness % 判断当前最优适应度值是否⽐上次进化结束后得到的最优适应度值要好bestX = thisbestX; % 修正最优解bestfitness =thisbestfit; % 修正最优适应度值num =0; % 清除算法结束控制量elsenum = num+1; % 算法结束控制量加1endSumFitness = sum(fitness); %计算适应度值总和SelectRate = fitness/SumFitness; %计算各个染⾊体的选择概率AccumulateRate(1)=SelectRate(1); % 计算各个染⾊体的累积概率for i=2:PopSize;AccumulateRate(i) = AccumulateRate(i-1)+SelectRate(i);endfor i =1:2:PopSize% 轮盘赌选出2个染⾊体,根据随机数与累积概率的⽐较进⾏选择pos = find(AccumulateRate>=rand);newPop(i,:) = population(pos(1),:);pos = find(AccumulateRate>=rand);newPop(i+1,:) = population(pos(1),:);% 交叉操作if oneorzero(Pc) ==1 % 判断这2个被选中的染⾊体是否需要进⾏交叉操作temp = randperm(21); % 产⽣1个随机数,以确定交叉位置position = temp(1);child1 = newPop(i,:); % 进⾏交叉child2 = newPop(i+1,:);tempchild = child1;child1(position:precision) = child2(position:precision);child2(position:precision) = tempchild(position:precision);newPop(i,:) =child1; % 将交叉后的⼦代存⼊新染⾊体矩阵中newPop(i+1,:) =child2;end% 变异操作newPop(i,:) = mutation(newPop(i,:),Pm);newPop(i+1,:) = mutation(newPop(i+1,:),Pm);endend% time =toc;%disp(['最优解为: ' num2str(bestX)]);% disp(['程序运⾏时间: ' num2str(time) 's']);六、结果分析1.利⽤实验数据,分析并解答以下问题,a)遗传算法中变异概率的设置对求解结果的影响。
遗传算法示例

实验一、过河问题一、问题描述有三个牧师和三个野人过河,只有一条能装下两个人的船,在河的任何一方或者船上,如果野人的人数大于牧师的认输,那么牧师就会有危险.找出一种按的渡河方法。
将该问题转变为:假如有n个牧师和n个野人准备渡河,但只有一条能容纳c个人的小船,为了防止野人侵犯牧师,要求无论在何处,牧师的人数不得少于野人的人数(除非牧师人数为0),且假定两种人都会划船,试设计一个算法,确定它们能否渡过河去,若能,则给出一只小船来回次数最少的最佳方案。
二、基本要求输入:牧师人数(即野人人数)n,小船一次至多载客人数c。
输出:若问题无解,则显示“渡河失败”信息,否则,输出一组最佳方案,用三组(X1,X2,X3)表示渡河过程中的状态。
并用箭头输出这些状态之间的迁移:目的状态<- <-中间状态<- <-初始状态。
例:当n=2,c=2时,输出000<-021<-211<-110<-221上述(X1,X1,X2)表示渡河过程中各个状态。
其中:X1表示始岸上牧师人数,X2表示始岸上野人人数,X3表示小船位置,(0-在目的岸,1-在起始岸)三、算法描述(1)算法基本思想的文字描述;从初始状态S(n,n,1)出发,形成的有合法且未达状态S11、S12、……、Sli。
再分别从S11、S12、……、Sli出发形成所有合法而未达状态S111、S112、……、Sli1、Sli2、Sli ……最终达到目标(0,0,0)(有解),或者找不到合法而未达状态(无解)。
若有解,则从目标返回找前趋状态,前趋状态的前趋状态……直到初始状态。
(2)判别(X1,X2,X3)为合法状态条件:X1=0或X1=n或X1=X2。
(3)数据结构:1 栈STACK ,记下“已达”状态及踪迹,并兼作队列。
2 STATE[X1][X2]=(4)算法基本思想的具体实现:1 初始化:置STATE[N+1][N+1][2]中的有状态为“未达”置队列STACK 空,cond 为当前是否已达到目标: cond= cond 置初值 2 以S (n,n,1)为始点,置STATE 为“已达”。
遗传算法实验

实验四遗传算法实验一、实验目的:熟悉和掌握遗传算法的原理、流程和编码策略,并利用遗传求解函数优化问题,理解求解TSP问题的流程并测试主要参数对结果的影响。
二、实验原理:旅行商问题,即TSP问题(TravelingSalesmanProblem)是数学领域中著名问题之一。
假设有一个旅行商人要拜访n个城市,他必须选择所要走的路径,路经的限制是每个城市只能拜访一次,而且最后要回到原来出发的城市。
路径的选择目标是要求得的路径路程为所有路径之中的最小值。
TSP问题是一个组合优化问题。
该问题可以被证明具有NPC计算复杂性。
因此,任何能使该问题的求解得以简化的方法,都将受到高度的评价和关注。
遗传算法的基本思想正是基于模仿生物界遗传学的遗传过程。
它把问题的参数用基因代表,把问题的解用染色体代表(在计算机里用二进制码表示),从而得到一个由具有不同染色体的个体组成的群体。
这个群体在问题特定的环境里生存竞争,适者有最好的机会生存和产生后代。
后代随机化地继承了父代的最好特征,并也在生存环境的控制支配下继续这一过程。
群体的染色体都将逐渐适应环境,不断进化,最后收敛到一族最适应环境的类似个体,即得到问题最优的解。
要求利用遗传算法求解TSP问题的最短路径。
三、实验内容及要求1、参考实验系统给出的遗传算法核心代码,用遗传算法求解TSP的优化问题,分析遗传算法求解不同规模TSP问题的算法性能。
2、对于同一个TSP问题,分析种群规模、交叉概率和变异概率对算法结果的影响。
3、增加1种变异策略和1种个体选择概率分配策略,比较求解同一TSP问题时不同变异策略及不同个体选择分配策略对算法结果的影响。
4、上交源代码。
四、实验结果(根据实验报告要求)1、画出遗传算法求解TSP问题的流程图。
2、分析遗传算法求解不同规模的TSP问题的算法性能。
(1)遗传算法执行方式说明:适应度值计算方法:当前路线的路径长度个体选择概率分配方法:适应度比例方法选择个体方法:轮盘赌选择交叉类型:PMX交叉变异类型:两点互换变异(2)实验模拟结果:城市个数历欢最好适应度历次最差适应度运行时间/血688.854388.3543379910146.009154.679352515210.027250.067429720278.942366.18L644025392.002■168,03630430513.155567.738704735627.6336S4.323855240745.577735,756927245727.03S25.06810434图2图1由图1和图2可知,遗传算法执行时间随着TSP问题规模的增大而增大,并且大致为线性增长。
遗传算法实验报告泽

遗传算法实验报告一、 问题描述已知函数10090060)(23++-=x x x x f , x[0,35 ],求函数的最大值。
05101520253035050010001500200025003000350040004500上图为函数10090060)(23++-=x x x x f 在x [0,35 ]区间上的曲线图像。
可以看出,该函数有多个极值点,如果使用其他的搜寻方法,很容易陷入局部最小点,而不能搜寻到真正的全局最小点,遗传算法具有通用、并行、稳健、简单与全局优化能力强等突出优点,适用于解决这种全局优化问题。
二、 方法原理遗传算法:遗传算法是借鉴生物界自然选择和群体进化机制形成的一种全局寻优算法。
与传统的优化算法相比,遗传算法具有如下优点:不是从单个点,而是从多个点构成的群体开始搜索;在搜索最优解过程中,只需要由目标函数值转换得来的适应值信息,而不需要导数等其它辅助信息;搜索过程不易陷入局部最优点。
目前,该算法已渗透到许多领域,并成为解决各领域复杂问题的有力工具。
在遗传算法中,将问题空间中的决策变量通过一定编码方法表示成遗传空间的一个个体,它是一个基因型串结构数据;同时,将目标函数值转换成适应值,它用来评价个体的优劣,并作为遗传操作的依据。
遗传操作包括三个算子:选择、交叉和变异。
选择用来实施适者生存的原则,即把当前群体中的个体按与适应值成比例的概率复制到新的群体中,构成交配池(当前代与下一代之间的中间群体)。
选择算子的作用效果是提高了群体的平均适应值。
由于选择算子没有产生新个体,所以群体中最好个体的适应值不会因选择操作而有所改进。
交叉算子可以产生新的个体,它首先使从交配池中的个体随机配对,然后将两两配对的个体按某种方式相互交换部分基因。
变异是对个体的某一个或某一些基因值按某一较小概率进行改变。
从产生新个体的能力方面来说,交叉算子是产生新个体的主要方法,它决定了遗传算法的全局搜索能力;而变异算子只是产生新个体的辅助方法,但也必不可少,因为它决定了遗传算法的局部搜索能力.交叉和变异相配合,共同完成对搜索空间的全局和局部搜索. 三、 实现过程(1)编码:使用二进制编码,随机产生一个初始种群。
遗传算法求解TSP问题

遗传算法求解TSP问题1、遗传算法前⼀篇遗传算法的基本内容在之前的博客已经应⽤过了之前遗传算法解决的是函数优化问题,即求解最⼤值或最⼩值问题;此次要解决的是组合优化问题中的TSP问题,即旅⾏商问题。
这边先介绍⼀下TSP问题TSP问题(Traveling Salesman Problem),即旅⾏商问题,⼜译为旅⾏推销员问题、货郎担问题,是数学领域中著名问题之⼀。
假设有⼀个旅⾏商⼈要拜访n个城市,他必须选择所要⾛的路径,路径的限制是每个城市只能拜访⼀次,⽽且最后要回到原来出发的城市。
路径的选择⽬标是要求得的路径路程为所有路径之中的最⼩值。
简单地说,TSP问题就是要找到图中的最短哈密尔顿回路,即全局最短路径。
然后遗传算法可以模仿⽣物进化,然后可以找到⼀个近似最优解,但其不⼀定是全局最优解。
2、实验原理1)产⽣初始种群;随机⽣成N个个体作为初始群体popm,随机选择⼀个种群;2)适应度函数;个体评价计算P(t)中各个个体的适应度,遗传算法在进化搜索中基本不利⽤外部信息,仅以适应度函数为依据,利⽤种群中每个个体的适应度值来进⾏搜索。
TSP的⽬标是路径总长度为最短3)选择运算;将使适应度较⼤(优良)个体有较⼤的存在机会,⽽适应度较⼩(低劣)的个体继续存在的机会也较⼩。
简单遗传算法采⽤赌轮选择机制4)交叉运算将交叉算⼦作⽤于群体;5)变异运算将变异算⼦作⽤于群体,并通过以上运算得到下⼀代群体P(t + 1);6)终⽌条件输出解。
3、代码实现1.city.m:随机⽣成N个城市的坐标并保存2.plot_route.m:实现连点画图3.染⾊体的路程代价函数 mylength.m4.适应度函数fit.m5.交叉操作函数 cross.m6.变异函数 Mutation.m7.main函数3、结果分析调整参数并分析运⾏结果(1)对于city_25.mat⽂件中的城市序列,参数ITER=2000,m=2,Pc=0.8,Pm=0.05保持不变,调整种群个数M的值,观察其结果变化:M=50M=100M=500由运⾏结果可知当M=100时得到TSP的最短路径长度均⼩于M=50和M=500运⾏得出的最短路径长度。
遗传算法实验

实验四遗传算法实验一、实验目的:熟悉和掌握遗传算法的原理、流程和编码策略,并利用遗传求解函数优化问题,理解求解TSP问题的流程并测试主要参数对结果的影响。
二、实验原理:旅行商问题,即TSP问题(Traveling Salesman Problem)是数学领域中著名问题之一。
假设有一个旅行商人要拜访n个城市,他必须选择所要走的路径,路经的限制是每个城市只能拜访一次,而且最后要回到原来出发的城市。
路径的选择目标是要求得的路径路程为所有路径之中的最小值。
TSP问题是一个组合优化问题。
该问题可以被证明具有NPC计算复杂性。
因此,任何能使该问题的求解得以简化的方法,都将受到高度的评价和关注。
遗传算法的基本思想正是基于模仿生物界遗传学的遗传过程。
它把问题的参数用基因代表,把问题的解用染色体代表(在计算机里用二进制码表示),从而得到一个由具有不同染色体的个体组成的群体。
这个群体在问题特定的环境里生存竞争,适者有最好的机会生存和产生后代。
后代随机化地继承了父代的最好特征,并也在生存环境的控制支配下继续这一过程。
群体的染色体都将逐渐适应环境,不断进化,最后收敛到一族最适应环境的类似个体,即得到问题最优的解。
要求利用遗传算法求解TSP问题的最短路径。
三、实验内容及要求1、参考实验系统给出的遗传算法核心代码,用遗传算法求解TSP的优化问题,分析遗传算法求解不同规模TSP问题的算法性能。
2、对于同一个TSP问题,分析种群规模、交叉概率和变异概率对算法结果的影响。
3、增加1种变异策略和1种个体选择概率分配策略,比较求解同一TSP问题时不同变异策略及不同个体选择分配策略对算法结果的影响。
4、上交源代码。
四、实验结果(根据实验报告要求)1、画出遗传算法求解TSP问题的流程图。
问题确定表示问题解的染色体(编码)初始化种群计算每个个体的适应值满足终止条件根据适应值选择输出最优解交叉变异2、分析遗传算法求解不同规模的TSP问题的算法性能。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
区域描述器FieldD中各参量的含义
code(i) 1表明子串i使用对数刻度; code(i) 0表明子串i使用算术刻度; lbin 和ubin是二进制行向量,表明由lb和ub所指定 的每个变量的范围中是否包含边界,取值为 0 表 明去掉边界;取值为 1 表明包含边界。
精品课件
二进制串到实值的转换函数——bs2rv
精品课件
单击Save按钮
精品课件
单击Close按钮
精品课件
§4.2 创建种群
1、创建基向量—— crtbase
精品课件
1、创建基向量——crtbase
调用格式 basev=crtbase(lind,base)
功能 利用基本字符集base产生由向量lind的元素确定
字符长度的串,以说明染色体中基因座的等位基因数量。 例1:在命令窗口中输入 >> basev=crtbase([4 6],[5 8]); basev =
§4.1 遗传算法工具箱的安装
第一步:将文件夹gatbx拷 贝至Matlab的工具箱目录 下或硬盘任一目录中;
精品课件
精品课件
第二步:启动Matlab, 并设置路径
精品课件
单击Add Floder…按钮或 Add With Subfloder…按 钮弹出对话框
精品课件
找到遗传算法工具箱放置的位置,单击确 定铵钮。
5555888888 再输入以下命令 >> [chrom,lind,basev]=crtbp(6,basev)
精品课件
[chrom,lind,basev]=crtbp(nind, basev);
chrom = 3410713003 2241446461 2433365075 3001443372 3402254267 3140316635
精品课件
§4.3 区域描述器
精品课件
区域描述器FieldD的结构如下:
len
lb
ub
FieldD
code
scale
lbin
ubin
精品课件
区域描述器FieldD中各参量的含义
len指明包含在矩阵 Chrom 的每个子串的长度; lb 和ub 为行向量,分别指明每个变量使用的下界和 上界; code 是二进制行向量,指明每个子串是怎样编码 的,code(i) 1表明子串i 按标准的二进制编码; code(i) 0表明子串i按灰度编码;scale是二进制行 向量,指明每个子串是否使用对数或算术刻度。
调用格式 Phen=bs2rv(Chrom,FieldD)
功能 根据区域描述器FieldD将二进制串矩阵 Chrom转换为实值矩阵,返回矩阵 Phen包含对应的种群表现型。
精品课件
例1:在命令窗口中输入
>> Chrom=crtbp(4,8) %创建一个初始种群,种群规模为4,编码长度为8 Chrom =
例2:在命令窗口中输入 >> [Chrom,lind,basev]=crtbp(3,5) Chrom =
01111 10110 00001 lind = 5 basev = 22222
精品课件
精品课件
[chrom,lind,basev]=crtbp(nind, basev);
例4:在命令窗口中输入 >> basev=crtbase([4 6],[5 8]) basev =
5555888888
精品课件
精品课件
2、创建二进制初始种群——crtbp
调用格式: ①[chrom,lind,basev]=crtbp(nind,lind); ②[chrom,lind,basev]=crtbp(nind, basev); ③[chrom,lind,basev]=crtbp(nind,lind,basev); 功能: ①创建一个以二进制编码的种群,nind指定种群规模,lind
精品课件
精品课件
将二进制串转化为实值
>> Phen=bs2rv(chrom,FieldD) Phen =
6.5490 3.7882 7.5843 1.1569
精品课件
精品课件
重新建立区域描述器
>> FieldD=[8;1;10;1;1;0;0] FieldD =
8 % 子串长度为8 1 % 每个子串的下边界为1 10 % 每个子串的上边界为10 1 % 用标准的二进制编码 1 % 每个子串使用对数刻度 0 % 每个变量的范围不包含下边界 0 % 每个变量的范围不包含上边界
精品课件
例3:在命令窗口中输入
>> FieldDR=[-2 0 4 1 -3;3 4 5 6 7] FieldDR =
-2 0 4 1 -3 34567 再输入以下命令 >> chrom=crtrp(3,FieldDR) chrom = 0.2571 1.2507 4.6831 4.0620 -2.8365 -1.7805 0.0515 4.0928 4.0427 -1.0993 -1.8641 1.5359 4.0353 1.0788 2.精8品6课9件2
lind = 10
basev = 5555888888
精品课件
精品课件
3、创建实值初始种群——crtrp
调用格式 chrom=crtrp(nind,FieldDR) 功能
创建一个大小为nind×nvar的随机实值矩阵, 其中nind指定了种群的规模,nvar确定了每个个 体的变量个数,FieldDR是一个大小2×nvar的矩 阵,包含每个个体变量的边界,第一行为上界, 第二行为下界。 需要注意的是在利用chrom=crtrp(nind,FieldDR) 之前先定义变量的边界。
11111000 01011000 10100100 00101011
精品课件
精品课件
建立区域描述器
&gD =
8 % 子串长度为8 -1 % 每个子串的下边界为-1 10 % 每个子串的上边界为10 1 % 用标准的二进制编码 0 % 每个子串使用算术刻度 1 % 每个变量的范围包含下边界 1 % 每个变量的范围包含上边界
确定染色体的长度。 ②返回一个长度为lind的染色体结构,染色体的等位基因的
基本字符由基本向量basev确定。 ③用于产生一个数量为nind的种群,染色体的长度为lind,
染色体等位基因的基本字符由基本向量basev确定。 精品课件
[chrom,lind,basev]=crtbp(nind,lind);
