测序结果分析教学文案
转录组测序结果解读

转录组测序结果解读
转录组测序是一种重要的基因组学技术,可以帮助科学家了解细胞内基因的表达模式和功能。
在转录组测序实验中,我们可以获得大量的RNA序列数据,通过对这些数据的解读,可以揭示基因在特定生理或病理条件下的表达水平和差异,从而进一步研究基因的调控机制和功能。
首先,转录组测序的结果可以帮助我们了解基因的表达水平。
通过对RNA序列数据进行比对和定量分析,可以计算出每个基因在样本中的表达水平。
这有助于我们确定哪些基因在特定条件下得到了上调或下调,并从中找出具有重要生物学意义的基因。
这些不同表达的基因可能与特定的生理过程、疾病发生和治疗效果等密切相关。
其次,转录组测序还可以帮助我们发现新的基因和转录本。
通过对RNA序列数据进行拼接和注释分析,我们可以发现一些未知的基因和转录本。
这些新的基因和转录本的发现可能会给我们带来对生物学过程的新的认识和思考。
此外,转录组测序结果的解读还包括富集分析和功能注释。
富集分析可以将不同表达的基因进行功能分类,进一步研究基因在特定途径和生物功能中的作用。
功能注释则可以帮助我们预测基因编码的蛋白质的功能和相互作用网络。
综上所述,转录组测序结果的解读是一个复杂但非常重要的工作。
通过对转录组测序数据的分析和解读,我们可以更好地理解基因的表达规律和功能,为生物学研究和医学应用提供有力的支持。
测序结果分析及序列拼接33页文档

பைடு நூலகம்
测序结果分析及序列拼接
46、法律有权打破平静。——马·格林 47、在一千磅法律里,没有一盎司仁 爱。— —英国
48、法律一多,公正就少。——托·富 勒 49、犯罪总是以惩罚相补偿;只有处 罚才能 使犯罪 得到偿 还。— —达雷 尔
50、弱者比强者更能得到法律的保护 。—— 威·厄尔
31、只有永远躺在泥坑里的人,才不会再掉进坑里。——黑格尔 32、希望的灯一旦熄灭,生活刹那间变成了一片黑暗。——普列姆昌德 33、希望是人生的乳母。——科策布 34、形成天才的决定因素应该是勤奋。——郭沫若 35、学到很多东西的诀窍,就是一下子不要学很多。——洛克
植物基因组测序完成结果初步分析报告

植物基因组测序完成结果初步分析报告简介:本报告基于对植物基因组测序完成结果的初步分析,旨在提供对测序数据的解读和分析,以及相关发现和未来研究的建议。
背景:随着高通量测序技术的迅速发展,植物基因组测序成为现代生物学的重要研究领域之一。
植物基因组测序的完成为我们理解植物基因组的结构、功能和进化提供了重要的工具和资源。
本次测序旨在获得某植物的完整基因组序列,为进一步研究该植物的功能基因提供参考。
结果分析:1. 基因组大小估计:通过对测序数据的初步分析,我们得出了该植物的基因组大小估计。
基因组大小是指一个生物体所有基因组成的总长,是评估基因组复杂性和特征的重要指标。
根据我们的分析,该植物预计的基因组大小为XX Mb。
2. 基因注释:我们利用已知的植物基因组数据库和基因预测软件对测序数据进行了基因注释。
通过比对已有的基因序列与我们测序结果的相似性,我们成功注释了一部分的基因,包括编码蛋白质的基因和非编码RNA基因。
同时,我们还发现了一些新的基因,这些新基因可能与该植物在特定环境中的适应性具有重要的联系。
3. 基因家族和表达谱研究:我们进一步对注释的基因进行了家族分析,发现了一些具有重要功能和进化意义的基因家族。
家族分析的结果有助于我们深入理解该植物基因组的起源和进化。
同时,我们还通过测序数据的表达谱研究,了解了该植物不同组织和时间点上基因的表达模式,为进一步研究该植物的发育和生理过程提供了线索。
4. 功能注释和通路分析:我们还对测序结果的基因进行了功能注释和通路分析。
通过比对已知的功能数据库,我们成功注释了一部分基因的功能。
进一步地,通过通路分析,我们发现了一些显著富集的通路以及基因在这些通路中的参与度,有助于我们深入了解该植物的生理和代谢过程。
未来研究建议:1. 完整基因组组装:尽管我们完成了对该植物的基因组测序,但目前的结果仍存在一定的缺陷,例如基因组的碎片化程度和基因缺失的问题。
因此,今后的研究可以通过进一步优化测序方法和使用高级的组装算法来实现完整基因组的测序和组装。
DNA测序结果分析
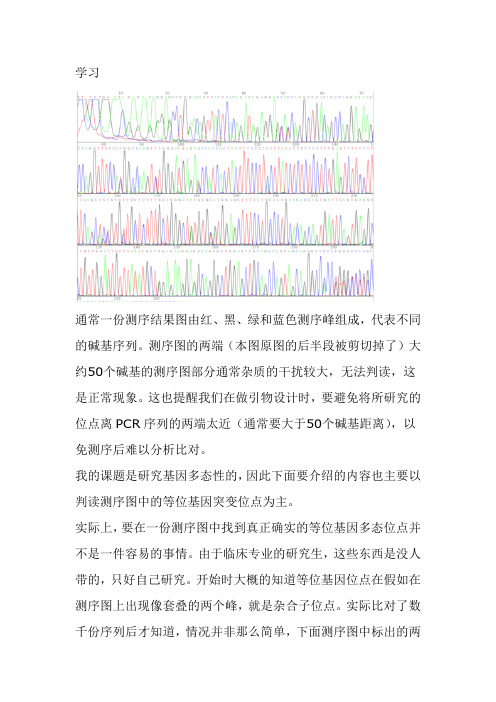
学习通常一份测序结果图由红、黑、绿和蓝色测序峰组成,代表不同的碱基序列。
测序图的两端(本图原图的后半段被剪切掉了)大约50个碱基的测序图部分通常杂质的干扰较大,无法判读,这是正常现象。
这也提醒我们在做引物设计时,要避免将所研究的位点离PCR序列的两端太近(通常要大于50个碱基距离),以免测序后难以分析比对。
我的课题是研究基因多态性的,因此下面要介绍的内容也主要以判读测序图中的等位基因突变位点为主。
实际上,要在一份测序图中找到真正确实的等位基因多态位点并不是一件容易的事情。
由于临床专业的研究生,这些东西是没人带的,只好自己研究。
开始时大概的知道等位基因位点在假如在测序图上出现像套叠的两个峰,就是杂合子位点。
实际比对了数千份序列后才知道,情况并非那么简单,下面测序图中标出的两个套峰均不是杂合子位点,如图并说明如下:说明:第一组套峰,两峰的轴线并不在同一位置,左侧的T峰是干扰峰;第二组套峰,虽两峰轴线位置相同,但两峰的位置太靠近了,不是杂合子峰,蓝色的C峰是干扰峰通常的杂合子峰由一高一略低的两个轴线相同的峰组成,此处的序列被机器误判为“C”,实际的序列应为“A”,通常一个高大碱基峰的前面1~2个位点很容易产生一个相同碱基的干扰峰,峰的高度大约是高大碱基峰的1/2,离得越近受干扰越大。
一个摸索出来的规律是:主峰通常在干扰峰的右侧,干扰峰并不一定比主峰低。
最关键的一点是一定要拿疑似为杂合子峰的测序图位点与测序结果的文本序列和基因库中的比对结果相比较;一个位点的多个样本相比较;你得出的该位点的突变率与权威文献或数据库中的突变率相比较。
通常,对于一个疑似突变位点来说,即使是国际上权威组织大样本的测序结果中都没有报道的话,那么单纯通过测序结果就判定它是突变点,是并不严谨的,因一份PCR产物中各个碱基的实际含量并不相同,很难避免不产生误差的。
对于一个未知突变位点的发现,通常还需要用到更精确的酶切技术。
基因测序报告解读

基因测序报告解读介绍基因测序是一种分析个体基因组的方法,通过对DNA序列进行测定和分析,可以揭示人类基因的组成和变异情况。
基因测序报告是基于个体基因组测序结果的解读分析,能够提供有关个体基因组的丰富信息。
本文将通过一步步的思考,解读基因测序报告。
第一步:基因组信息首先,我们需要了解基因测序报告中提供的基因组信息。
基因组信息通常包括个体的基因型、等位基因频率、基因的功能以及可能的疾病风险等。
通过查阅报告中的基因型信息,我们可以了解个体所携带的基因变异情况。
第二步:常见基因变异接下来,我们需要关注报告中提及的常见基因变异。
基因变异是指个体基因组中与常见基因型不同的碱基序列。
这些变异可能与个体的特定特征或疾病风险相关。
在报告中,常见的基因变异通常会被注明其对应的功能或与之相关的疾病。
第三步:疾病风险评估基于基因测序报告中的基因变异信息,我们可以进一步评估个体的疾病风险。
报告中可能提及一些与基因变异相关的疾病,这些疾病可能包括遗传性疾病、药物反应性等。
通过了解这些信息,我们可以更好地了解个体可能面临的健康风险,并采取相应的预防措施。
第四步:遗传性疾病在报告中,我们也可以找到与遗传性疾病相关的信息。
遗传性疾病是由基因突变引起的疾病,个体携带特定的基因变异可能具有遗传性疾病的风险。
通过了解基因测序报告中与遗传性疾病相关的信息,我们可以提前采取措施,例如进行定期检查、避免特定环境或生活方式等。
第五步:个性化健康管理基于基因测序报告,个体可以进行个性化的健康管理。
通过了解个体基因组中的变异情况,我们可以为个体提供针对其特定基因型的健康建议。
这些建议可能包括特定的饮食和运动指导,以及药物选择和剂量调整等。
结论基因测序报告的解读是了解个体基因组信息的关键步骤。
通过仔细阅读和分析报告中的基因型、基因变异、疾病风险等信息,我们可以更好地了解个体的基因组特征,并为其提供个性化的健康管理建议。
这有助于个体更好地了解自身的遗传特征,预防潜在的疾病,并改善生活质量。
测序结果分析

测序结果分析DNA测序技术已经成为了生物学研究的一项非常重要的工具,能够为我们提供大量的基因信息。
但是,得到大量的基因信息并不意味着我们就可以直接进行基因研究,我们还需要对测序结果进行深入的分析才能够更加准确地了解这些基因的特点和功能。
本文将介绍测序结果分析的基本流程和一些常见的方法。
测序结果分析的基本流程测序结果分析的基本流程可以分为以下几步:1.原始测序数据处理:对测序测序的原始数据进行过滤、去重、修剪等操作,得到高质量的序列数据。
2.序列比对:将样本读取序列与参考序列比对,确定SNP、INDEL、等变异信息。
3.基因注释:对比对结果进行注释,在数据库中查找相关基因的信息,如基因的长度、功能、结构、等等。
4.功能富集分析:对匹配到的基因进行功能富集分析,可以了解到哪些基因和功能在样本间被不同地表达。
常见的测序结果分析方法1. 原始数据处理原始数据处理主要包括数据过滤、去除低质量序列、去除接头等步骤。
之后,我们可以得到高质量的序列数据,用于后续的分析。
2. 序列比对序列比对是将样本测序序列与参考序列比对,比对结果用于寻找SNP、INDEL 等变异。
比对的方法包括全局比对和局部比对,全局比对速度慢但结果较准确,局部比对速度快但可能会产生错误结果。
一般常用的软件有Bowtie、BWA、STAR 等。
3. 基因注释基因注释是对比对结果定位到相应的基因序列上,然后通过与数据库进行比对进行注释。
常用的数据库包括NCBI GenBank、KEGG、UniProt等。
从中可以获得各种相关信息,例如功能、结构、长度等。
4. 功能富集分析功能富集分析是对匹配到的基因进行功能分析,可以了解哪些基因和功能在样本间被不同地表达。
常用的软件包括DAVID(Database for Annotation, Visualization and Integrated Discovery)、GO(Gene Ontology)等,这些数据库可以将富集到的功能直接表示为直线图或散点图等方式,分析结果比较清晰。
PCR引物设计及测序结果分析

PCR技术原理
以拟扩增的DNA分子为模板,以一对分别与 模板5′末端和3′末端互补的寡核苷酸片段为引 物,在DNA聚合酶的作用下,按照半保留复制的机 制沿着模板链延伸直至完成新的DNA合成,重复这 一过程,即可使目的DNA片段得到扩增。
基本原则:
引物与模板的序列要紧密互补 引物与引物之间避免形成稳定的二聚体
或发夹结构 引物不能在模板的非目的位点引发DNA
聚合反应(即错配)。
一般原则:
1. 引物的长度:配对引物的长度一般在1530bp之间比较合适。
尽可能使用两条引物的Tm值相同(最好相差不要超过 5℃)
Tm值的计算:一般的公式:Tm = 4 (G+C) + 2(A+T)
正义链
反义链
基本分子生物学研究手段
PCR PCR反应液体的成分:
PCR循环的程序: PCR原理:
基本分子生物学研究手段
RT-PCR
反转录PCR mRNA---cDNA-----PCR
基本分子生物学研究手段
Cutting by enzymes
基本分子生物学研究手段
Southern Blot
基本分子生物学研究手段
生物软件网下载 安装后,用文本编辑器打开WIN.INI,将vspace=DU
改为vspace=PU便可以使用全部功能。
Oligo primer 3 The Primer Generator NetPrimer
如何使用Primer Premier 5.0
测序结果分析

测序结果的判读测序结果为.abi格式,可用软件chrosmas打开,一种颜色的峰代表一个碱基,峰的高低表信号的强弱。
一个正常的N表示机器没法判读是哪种碱基,原因是:杂峰的信号高于机器默认的值,机器会认为该处有两个峰,因此不能判断确定是哪个峰,需要人工判读。
以下三种情况会出现N:有杂合子,有杂峰,反应已结束。
原因:测序产物纯化不够注意:染料峰位于序列的前100 碱基以内;酒精峰位于序列的220 ~ 320 碱基之间产生的原因是样品或毛细管内有灰尘等固体小颗粒原因:测序反应失败。
解决办法:改进条件,重做反应。
注意两个关键因素:引物与模板之间的比例:3.2 pmol: 200 ng。
模板DNA 的纯度和用量:1.6 ~ 2.0原因:残余的Dye 太多,纯化不够。
有测序反应,但效率低下信号太弱解决办法:纯化充分。
避开引物峰,确定新的分析起点1、PCR产物测序时出现重叠峰问题图1(模板中有碱基缺失,往往是单一位点(1-1)或两个位点(1-2)碱基缺失导致测序结果移码)解决方法:将PCR产物克隆到质粒(如T载体)中挑单克隆测序,或将PCR产物进行PAGE 纯化(至少琼脂糖充分电泳后切胶纯化)后再进行测序。
问题图2(PCR产物不纯,含部分序列一致的两种以上的片段,长度不一)解决方法:主要原因是PCR产物没有纯化,含有部分序列一致的两种以上长度不一的片段,将PCR产物进行PAGE纯化(至少琼脂糖充分电泳后切胶纯化)后再进行测序,便可解决。
问题图3(测序引物有碱基缺失)测序引物有碱基缺失(一般是引物的5'端缺失),和模板的碱基缺失即图1有些类似,所不同的是模板碱基缺失一般是在一段正常测序序列后才出现移码,而引物碱基缺失的话,则从测序一开始就出现移码,表面在图形上便是一开始就是严重的峰形重叠。
解决方法:重新合成引物,或将引物进行PAGE纯化2、克隆测序时出现峰形重叠原因:所挑选的重组子不是单克隆,所提供的测序用质粒中含有两种以上插入片段不同的质粒;或是是送测序的菌液污染解决方法:重新挑单克隆的菌落(划线分离单菌落),提质粒或送菌液再次测序。
基因测序数据分析的技术教程与实用方法分享

基因测序数据分析的技术教程与实用方法分享引言:随着科技的不断发展,基因测序已经成为现代生物学领域最为重要的研究手段之一。
基因测序数据是一种宝贵的资源,可以帮助科学家们探索基因组,了解生物的基因变异以及与疾病之间的关联。
由于基因测序数据的复杂性,全面了解基因测序数据分析的技术教程与实用方法变得至关重要。
本文将介绍一些常见的基因测序数据分析技术与实用方法,帮助读者更好地处理和解读基因测序数据。
一、基因测序数据分析的目标与步骤基因测序数据分析的目标是通过对测序数据进行处理和分析,在了解基因组的基础上,发现基因间的关联性以及与疾病之间的关系。
基因测序数据分析通常包括以下几个步骤:1. 数据预处理:数据预处理是基因测序数据分析的第一步,它包括数据质量控制、去除低质量序列和质量修复等操作。
数据预处理的目的是去除噪音和不可靠的数据,提高数据的准确性和可靠性。
2. 数据比对:数据比对是将测序数据与参考基因组进行对比的过程。
通过比对,可以确定测序reads的来源以及测序reads在参考基因组上的位置。
常用的比对工具有Bowtie、BWA等。
3. 变异检测:基因组的变异对于疾病的发生与发展具有重要影响。
通过对比测序数据和参考基因组的差异,可以检测出单核苷酸变异(SNP)、插入缺失(indel)等基因组变异。
常用的变异检测工具有GATK、samtools等。
4. 基因表达分析:基因表达分析是通过比对测序数据,计算基因在不同条件下的表达水平。
通过基因表达分析,可以了解基因在不同生物过程中的调控机制,识别关键调控基因。
常用的基因表达分析工具有DESeq、edgeR等。
二、基因测序数据分析的实用方法1. 基于UNIX操作系统的命令行工具在基因测序数据分析中,熟悉并掌握基于UNIX操作系统的命令行工具是非常重要的。
命令行工具可以提供更高效的数据处理和分析方法。
常用的命令行工具有Bash、Grep、Awk、sed等。
熟练使用这些工具可以帮助快速处理和分析测序数据。
甲基化测序结果分析

甲基化测序结果分析甲基化测序是当前广泛使用的基因表达调控分析技术。
甲基化测序通过检测DNA的甲基化水平,研究基因表达水平的变化,并确定DNA甲基化状态,以及它们如何影响表达水平。
甲基化测序结果分析是为了找出甲基化变化与表达水平变化之间的关联,以及利用这些变化来预测基因表达水平的变化。
甲基化测序结果可以用于研究癌症、免疫功能、再生医学和遗传病的遗传加工和表达调控机制,以及基因组学中基因组结构的变化。
甲基化测序结果的分析一般包括对比样本和对照样本之间的甲基化变化,以及确定参与甲基化变化的基因。
分析甲基化测序结果的主要方法包括首先确定不同样本的甲基化水平,然后利用统计学方法比较不同样本之间的甲基化变化。
此外,还可以通过研究样本之间相关甲基化位点的基因进行模式发现,找出导致甲基化变化的可能基因。
分析甲基化测序结果的另一个非常重要的步骤是确定参与表达水平变化的基因。
通常,当分析出的甲基化变化与表达水平变化显著相关时,就可以确定相应基因参与表达水平变化。
分析甲基化数据需要一系列统计学和生物学方法。
它需要利用合适的统计学方法探索不同样本之间甲基化变化的关联,使用生物信息学方法确定甲基化位点与基因之间的关联,并利用统计模型来预测甲基化水平与表达水平之间的关系。
此外,也可以使用计算机辅助的系统生物学分析方法来研究甲基化数据。
近年来,随着技术的进步,组学技术已被广泛地应用于甲基化测序结果分析中,从而可以更好地研究基因表达水平的变化及其原因,综合分析多个关联分析方法,有助于增强对甲基化数据的理解和发现。
甲基化测序结果分析是一项复杂的任务,对于科学家来说也是一项艰巨的挑战。
它可以帮助科学家从大量的数据中发现和预测真正的调控机制,并且可以帮助科学家以更直观的形式理解和探索生物学过程,从而提高研究效率,发掘新的发现。
基因组测序技术的数据分析与结果解释方法

基因组测序技术的数据分析与结果解释方法随着基因组测序技术的快速发展,数据产生的速度和规模也在不断增加。
如何对这些海量的基因组数据进行有效的分析和结果解释,成为了现代生物学研究的重要课题。
本文将介绍基因组测序技术的数据分析和结果解释方法,以帮助读者更好地理解和应用这一领域的知识。
第一部分:基因组测序数据分析方法基因组测序技术涉及到测序样本的DNA分子的测序读取。
首先,将测序样本中的DNA分子片段断裂,并将其转化为文库(library),然后通过PCR扩增和文库构建来放大和分离所需的DNA分子片段。
文库制备完成后,利用基因组测序仪对文库进行测序,产生大量的测序读取数据。
1. 数据质控和预处理基因组测序数据可能存在测序错误、噪声和低质量数据等,因此在进行数据分析之前,需要对数据进行质控和预处理。
可以使用质量评估工具对测序数据进行评估,剔除低质量的读取,并进行质量修剪和去除接头序列等预处理步骤。
2. 序列比对和拼接得到高质量的测序数据后,下一步是进行序列比对和拼接。
比对是将测序数据与参考基因组进行比较,以确定每个读取序列在参考基因组上的位置。
常用的比对工具包括Bowtie和BWA等。
拼接是将多个测序读取序列组装成较长的连续序列,常用的拼接工具有SOAPdenovo和SPAdes等。
3. 变异检测和突变注释基因组测序数据分析的重要任务是检测基因组中的变异和突变。
变异检测可以通过比对数据和参考基因组的差异来实现。
常用的变异检测工具有GATK和SAMtools等。
检测到的变异信息需要进行注释,以确定其可能的功能和疾病相关性。
第二部分:基因组测序结果解释方法基因组测序数据的分析结果需要进行解释,以揭示基因组的功能、变异的影响和相关的生物学机制。
1. 基因功能注释对检测到的变异和突变进行基因功能注释是结果解释的重要一环。
基因功能注释可以利用公共数据库、功能预测工具和生物学知识来确定变异的可能影响。
常用的功能注释工具有ANNOVAR和Variant Effect Predictor等。
实验2 引物设计与测序结果分析

学院:______ 班级:_______ 学号:_________ 姓名:__________ 成绩:______ 实验二引物设计及测序结果分析目的:1、掌握常规引物设计的原则及操作流程。
2、熟悉简并引物设计的原理及操作方法。
3、熟悉引物设计软件及在线引物设计工具的操作方法。
4、掌握使用相关软件及在线工具分析测序结果的方法。
内容:1、使用Primer Premier、Oligo、BLAST等软件及在线工具进行常规引物设计,并对引物扩增效率、特异性进行评价。
2、使用DNAMAN软件进行常规引物快速设计。
3、使用NCBI中的在线引物设计工具Primer-BLAST快速设计引物。
4、使用在线工具CODEHOP设计简并引物。
5、使用Chromas、BioEdit软件查阅测序结果峰图文件。
6、使用DNAMAN软件对测序序列进行编辑,进行序列拼接。
软硬件要求:联网计算机,预装Windows 7操作系统,预装IE或Chrome浏览器、英汉电子词典(有道词典或金山词霸),预装DNAMAN7、Primer Premier5、Oligo7、Chromas、BioEdit等生物信息学分析软件。
操作及问题:一、Primer Premier5、Oligo7、BLAST常规引物设计本部分操作将使用Primer Premier5、Oligo7、BLAST等软件及工具设计拟南芥AtBADH基因编码区全长特异引物。
(参考“第四章引物设计及测序结果分析”课件)(一)使用Primer Premier5搜索引物1、在NCBI数据中查找登录号为NM_001198470的序列记录,查阅相关信息,并下载序列将其保存为fasta格式文件。
问题1:该序列是什么类型的序列?该序列编码区在什么位置?2、打开Primer premier5软件,点击键ctrl+V将上一步中下载的序列粘贴入弹出的GeneTank窗口中(或者点击。
3、点击GeneTank窗口中左上角的Primer premier窗口中点击Search Criteria窗口中根据要求选择合适选项及参数,选定后,点击Search Progress窗口中有Search Results窗口;如没有出现数重新搜索引物。
转录组测序结果解读

转录组测序结果解读
(原创实用版)
目录
1.转录组测序结果概述
2.转录组测序结果的解读方法
3.转录组测序结果的实际应用
正文
【转录组测序结果概述】
转录组测序是一种研究细胞或组织中所有基因的表达情况的方法,它可以揭示基因的表达水平和表达模式。
通过转录组测序,我们可以得到大量的数据,包括基因的表达量、转录本信息等。
【转录组测序结果的解读方法】
对转录组测序结果的解读主要包括以下几个步骤:
首先,需要对原始测序数据进行质量控制,包括剔除低质量测序读段、去除接头序列等。
然后,通过比对测序数据和基因组参考序列,可以得到每个基因的表达量。
接着,对表达量进行统计分析,可以得到基因的表达模式和差异表达情况。
最后,根据差异表达情况,可以对基因功能进行推测,并对相关生物过程进行分析。
【转录组测序结果的实际应用】
转录组测序结果在生物学研究中有广泛的应用,包括基因功能研究、疾病诊断和治疗、植物育种等。
例如,通过转录组测序,我们可以研究某
个基因在特定生物过程中的作用,从而对相关疾病进行诊断和治疗。
测序报告总结与反思范文

测序报告总结与反思范文引言测序报告是进行DNA测序后所生成的结果展示和分析的文档,通过阅读报告可以获得关于DNA序列的各种信息。
本文将对测序报告进行总结与反思,总结测序过程中的经验和教训,反思测序结果的可靠性和局限性,以期提高测序的质量和效果。
总结测序过程测序过程中,我们首先进行DNA提取和纯化,然后使用特定的方法对DNA进行增幅,然后进行测序反应,得到原始序列数据,最后通过生物信息学分析得到测序结果。
整个过程需要严格控制实验操作、仪器设备和实验环境,并在每个步骤中进行质控。
在本次测序中,我们采用了高通量测序技术,通过测序仪器的高效率和高准确性,成功得到了高质量的测序结果。
测序结果分析通过对测序结果的分析,我们获取了DNA序列的信息,包括序列长度、碱基组成、SNP等。
这些信息对于后续的研究和应用具有重要的意义。
我们可以利用这些序列信息进行功能基因预测、物种鉴定等研究,揭示生物体的遗传信息和进化过程。
反思测序结果可靠性尽管我们在测序过程中严格依照操作流程进行,但仍存在一定的误差和偏差。
首先,DNA提取和纯化过程中可能存在污染和损伤,导致测序结果的不准确性。
其次,测序反应中可能会出现PCR扩增的偏差或随机错误,进一步影响结果的精确性。
此外,测序仪器的读取速度和准确性也会对结果产生影响。
因此,我们需要在测序过程中不断优化和改进,提高测序结果的可靠性。
测序结果局限性测序结果的局限性主要体现在两个方面。
首先,测序技术限制了我们在一定时间内获取的序列长度,从而无法测序特别长的DNA片段。
其次,由于测序结果只是DNA序列的信息,我们无法直接获得DNA的三维结构和功能信息。
因此,在研究中需要结合其他技术手段和方法进行进一步分析和验证,以获取更全面和准确的结果。
结束语通过测序报告的总结与反思,我们对测序过程和结果有了更深入的认识。
我们深刻认识到测序的重要性和复杂性,对测序技术的优化和提高有了更高的要求。
希望在今后的研究中,能够进一步完善测序流程和技术,提高测序结果的可靠性和全面性,为科学研究和应用提供更准确和可靠的数据基础。
转录组测序结果解读

转录组测序结果解读摘要:1.转录组测序简介2.转录组测序结果的解读流程3.转录组测序结果的生物学意义4.总结正文:1.转录组测序简介转录组测序是一种用于分析细胞或组织中所有基因的表达水平和转录本的方法。
通过对RNA 进行测序,可以获得大量的转录信息,从而深入了解基因表达的特征和调控机制。
转录组测序技术已成为生物学、医学等领域的重要研究手段。
2.转录组测序结果的解读流程转录组测序的结果通常包括大量的数据,需要进行专业的分析和解读。
解读流程一般包括以下几个步骤:(1)数据质量控制:通过筛选、去除低质量测序读段,提高数据质量。
(2)比对:将测序数据与基因组参考序列进行比对,得到每个基因和转录本的表达信息。
(3)表达量计算:根据比对结果,统计每个基因和转录本的表达量,常用的表达量计算方法有FPKM、TPM 等。
(4)差异表达分析:对不同样本或处理组之间的基因表达差异进行统计分析,找出具有生物学意义的差异表达基因。
(5)功能富集分析:对差异表达基因进行功能富集分析,如GO(Gene Ontology)富集分析和KEGG(Kyoto Encyclopedia of Genes and Genomes)通路富集分析,以了解差异表达基因在生物学功能和代谢通路上的分布。
3.转录组测序结果的生物学意义转录组测序结果可以为研究者提供丰富的生物学信息,如基因表达水平、转录本结构、调控机制等。
通过对转录组测序结果的解读,可以深入了解细胞或组织的生物学特征,为研究基因功能、疾病机制、药物研发等领域提供有力支持。
例如,在研究植物抗病性时,通过转录组测序可以发现与抗病性相关的基因,进而研究其功能和调控机制,为培育抗病性强的品种提供理论依据。
在医学领域,转录组测序可以用于研究肿瘤的发生机制、预后评估、个性化治疗等方面。
4.总结转录组测序结果的解读是一项复杂的工作,需要结合生物信息学方法和专业知识进行。
测序报告解读

测序报告解读随着科技的飞速发展,基因测序技术已经逐渐走进我们的日常生活中。
基因测序是通过测定DNA或RNA的核苷酸序列来获取个体或群体遗传信息的一种技术。
测序结果可以帮助我们了解个体的遗传特征、健康风险以及对特定药物的反应等重要信息。
而一份完整的测序报告解读则至关重要,因为它能够帮助个体及医疗专业人士更好地理解测序结果,从而做出更准确的健康管理决策。
报告概述一份测序报告通常包括患者基本信息、测序目的、样本信息、测序方法、结果解读、健康建议等内容。
患者基本信息包括姓名、性别、年龄、临床诊断等基本资料,有助于锁定特定个体或群体。
测序目的会明确说明此次测序的用途,是为了疾病诊断、基因检测还是其他研究目的。
然后,样本信息会介绍测序所采用的组织类型、采样时间和地点等相关信息。
随后,测序方法则会详细描述测序所采用的技术和平台,例如Illumina、PacBio等。
报告中的结果解读会根据测序数据提供相关的遗传信息和健康风险评估,从而引出健康建议和个性化治疗方案。
结果解读测序结果的解读是测序报告的核心内容。
结果解读一般包括以下几个方面的内容:遗传变异、健康风险、药物反应和其他相关遗传特征。
遗传变异是指在个体基因组中发现的突变或变异,可以分为致病变异、可能致病变异和良性变异等。
致病变异可能与遗传性疾病相关,需要引起重视;可能致病变异则可能对健康产生一定影响,需要结合具体情况进行评估;良性变异则通常不太会对健康造成影响。
健康风险评估是根据遗传变异的组合来评估个体可能患上某些疾病的概率,比如心血管疾病、癌症等。
然后,药物反应是根据遗传变异预测个体对特定药物的代谢情况,有助于制定个性化用药方案。
其他相关遗传特征可能包括血型、遗传感知能力等其他与健康相关的遗传信息。
健康建议基于测序结果,健康建议是测序报告中至关重要的一部分。
健康建议应针对个体的遗传特征和健康风险进行量身定制,包括健康生活方式指导、疾病预防策略、个性化治疗方案等。
RNA测序数据的整合与分析

RNA测序数据的整合与分析RNA 测序技术如今在生物学和医学研究中可是个大热门,而对于RNA 测序数据的整合与分析,那更是关键中的关键!咱先来说说为啥要整合和分析 RNA 测序数据。
就好比你有一堆拼图碎片,每一块都代表着一部分信息。
RNA 测序数据也是这样,单独看可能看不出啥名堂,但是把它们整合起来,仔细分析,就能拼出一幅完整的“生命画卷”。
我给您讲个事儿吧。
之前我跟着一个研究团队,他们正在研究一种罕见疾病。
一开始,大家对着零散的数据都有点摸不着头脑。
后来,通过对大量 RNA 测序数据的整合分析,发现了一些之前被忽略的关键基因表达变化。
就好像在黑暗中突然找到了一束光!那怎么进行整合呢?这可不是简单地把数据堆在一起。
首先得保证数据的质量,就像做菜得有新鲜的食材一样。
得检查数据有没有错误、偏差,把那些“坏果子”挑出去。
然后,根据研究目的和数据特点,选择合适的方法进行整合。
比如说,如果是研究不同组织中的基因表达,可能就得用特定的算法来校正差异。
分析 RNA 测序数据也有很多门道。
要看看基因的表达水平是高了还是低了,这就像观察市场上某种商品的供求关系一样。
还要分析基因之间的相互作用,是不是有“拉帮结派”或者“单打独斗”的情况。
有时候,一个小小的变化,可能会引发一系列的连锁反应。
比如说,在一次肿瘤研究中,我们发现某个基因的表达量突然升高。
进一步分析发现,它居然影响了周围好几个基因的表达,就像一个“带头大哥”带着小弟们一起“闹事”。
这一发现为治疗方案的制定提供了新的思路。
整合与分析 RNA 测序数据也不是一帆风顺的。
有时候会遇到数据缺失的情况,就像拼图少了几块,让人头疼。
还有的时候,不同批次的数据差异太大,整合起来就像把油和水混在一起,困难重重。
但正是这些挑战,让我们不断寻找更好的方法和工具。
在未来,随着技术的不断进步,RNA 测序数据的整合与分析肯定会越来越精准、高效。
说不定能解开更多生命的奥秘,为治疗各种疑难杂症带来新的希望。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
测序结果的判读
测序结果为.abi格式,可用软件chrosmas打开,一种颜色的峰代表一个碱基,峰的高低表信号的强弱。
一个正常的N表示机器没法判读是哪种碱基,原因是:杂峰的信号高于机器默认的值,机器会认为该处有两个峰,因此不能判断确定是哪个峰,需要人工判读。
以下三种情况会出现N:有杂合子,有杂峰,反应已结束。
原因:测序产物纯化不够
注意:染料峰位于序列的前100 碱基以内;酒精峰位于序列的220 ~ 320 碱基之间
产生的原因是样品或毛细管内有灰尘等固体小颗粒
原因:测序反应失败。
解决办法:改进条件,重做反应。
注意两个关键因素:引物与模板之间的比例:3.2 pmol: 200 ng。
模板DNA 的纯度和用量:1.6 ~ 2.0
原因:残余的Dye 太多,纯化不够。
有测序反应,但效率低下信号太弱
解决办法:纯化充分。
避开引物峰,确定新的分析起点
1、PCR产物测序时出现重叠峰
问题图1(模板中有碱基缺失,往往是单一位点(1-1)或两个位点(1-2)碱基缺失导致测序结果移码)
解决方法:将PCR产物克隆到质粒(如T载体)中挑单克隆测序,或将PCR产物进行PAGE 纯化(至少琼脂糖充分电泳后切胶纯化)后再进行测序。
问题图2(PCR产物不纯,含部分序列一致的两种以上的片段,长度不一)
解决方法:主要原因是PCR产物没有纯化,含有部分序列一致的两种以上长度不一的片段,将PCR产物进行PAGE纯化(至少琼脂糖充分电泳后切胶纯化)后再进行测序,便可解决。
问题图3(测序引物有碱基缺失)
测序引物有碱基缺失(一般是引物的5'端缺失),和模板的碱基缺失即图1有些类似,所不同的是模板碱基缺失一般是在一段正常测序序列后才出现移码,而引物碱基缺失的话,则从测序一开始就出现移码,表面在图形上便是一开始就是严重的峰形重叠。
解决方法:重新合成引物,或将引物进行PAGE纯化
2、克隆测序时出现峰形重叠
原因:所挑选的重组子不是单克隆,所提供的测序用质粒中含有两种以上插入片段不同的质粒;或是是送测序的菌液污染
解决方法:重新挑单克隆的菌落(划线分离单菌落),提质粒或送菌液再次测序。
3、样品有杂合/突变位点
模板中有杂合型突变,也就说模板本身在这个位点出现突变;或者是从基因组中扩增出来的杂合位点。
如果模板有杂合(突变或缺失),那么测序图形中其他的位點一般都是單一的峰形,然后突然在某一個位點出現重叠峰(如图中箭頭所示)。
解决方法:建议将DNA片段克隆到载体再测序。
4、polyA/T和C/Gcluster导致的套峰和测序信号衰减
RACE测序时经常遇到图4-1和图4-4的情形,解决方法:从另一端测序;或者构建载体进行测序。
5、基因中含有重复序列
可能的原因:样品中含有重復序列导致的测序结果和PolyA/T的结果一样,会导致Frame 滑动,较短的重復序列会导致测序结果出现移码;而较长的重復序列会使定序信号衰减。
解决办法:反向测序有时能够顺利的通过重復序列区域(但不是一定都能够),通过多次的测序结果比对,拼接可以得到全序列结果。
