chapter7-转录调控与基因功能注释
最新基因的转录和调节

①在DNA分子双链上,总是只有一股链用作模板指引 转录,另一股链不转录。能指引转录生成RNA的DNA 单链称为模板链,template strain,有时也称为有 意义链,sense strain或Watson链;相对于模板链 不指引转录的另外一股DNA单链称为编码链,coding strain,又称为反义链,antisense strain或Qick 链。
(四) DNA模板上启动子是控制转录的关键部位 基因转录的第一步就是RNA聚合酶结合到模板DNA
分子上,结合的部位称为启动子,promoter,它是结构
基因上游的调控序列,是控制转录的关键部位,该区域 含有较多的A—T配对。对多种原核生物基因转录起始 区的分析发现,如果以开始转录生成RNA 5’端第一个脱 氧核苷酸的位置为1,以负数表示上游的碱基序数,那 么不同基因的启动子之间存在着保守序列或一致性序列。
转录将基因信息从DNA传递到蛋白质
(一) 转录是基因信息从DNA传递到蛋白质的重要环节 DNA碱基排列顺序决定了编码蛋白质的氨基酸序列,
是蛋白质合成的原始模板。mRNA是蛋白质合成的直接模板, 其他几种RNA是参与翻译过程的重要因子。通过基因转录 遗传信息从细胞核转运到细胞质,从功能上衔接了DNA和 蛋白质这两种生物大分子。基因转录具有以下特点: 1.合成RNA的底物是5´-三磷酸核苷,包括ATP、GTP、CTP 和UTP。 2.在RNA聚合酶作用下一个NTP的3-OH和另一个NTP的5’P反应,形成磷酸酯键。 3.RNA碱基顺序由模板DNA碱基顺序决定,依靠NTP与DNA 碱基配对的亲和力被选择。
RNA聚合酶Ⅰ的转录产物是45S-rRNA,经剪接修饰 生成除5S-rRNA外的各种rRNA RNA聚合酶Ⅱ的转录产物是mRNA的前体hnRNA RNA聚合酶Ⅲ的转录产物是一些小分子量RNA,如 5S-rRNA、tRNA、snRNA等 真核细胞RNA聚合酶结构比较复杂,往往由多个亚基 组成,如图为酿酒酵母RNA聚合酶Ⅱ,由12个亚基组 成。
第l七章原核生物基因表达调控模式

一、基因表达(gene expression)
DNA
RNA
蛋白质
基因表达的调控:生物有机体对其基因表达的时 空程序、表达速率等所进行的调节和控制。
二、基因表达的调控(gene regulation) 1、转录水平上的调控:转录水平上的对基因的 调控决定于DNA的结构、RNA聚合酶的功能、 蛋白质因子及其他小分子物质的相互作用。 2、转录后水平上的调控:mRNA加工成熟水平 上的调控;翻译水平上的调控。
阻遏:在某些代谢物或化合物的作用下,基因由 原来的关闭状态转变为工作状态
阻遏物:能导致阻遏发生的分子 可阻遏基因:阻遏调节中被关闭的基因 可阻遏酶:可阻遏基因表达的酶
▪ 特殊代谢物调控的分子机理
特殊代谢物是调控蛋白的变构剂,与调控蛋白结合可 使调控蛋白的空间构像发生变化,从而改变其对基因 转录的影响
调节区
trpR RNA聚P合酶O
RNA聚合酶
Trp 低时
结构基因
mRNA
Trp 高时
Trp
色氨酸操纵子
调节区
trpR
PO
前导序列
前导mRNA
1
2
结构基因
衰减子区域
3
4
UUUU……
trp 密码子 终止密码子
第141a0a、前1导1密肽码编子码UU为区Ut:Ur包…p密含…U码序U子U列U1……衰减子结构 UUUU……
子lac mRNA/细胞。
• 在无葡萄糖有乳糖的培养基中,lac+细菌中将同时合成β-半乳糖
苷酶和透过酶。
图10.6 培养基添加诱导物(β-半乳糖苷)能迅速诱导Lac mRNA,稍
基因的转录、转录后调控

基因的转录、转录后调控基因是遗传信息的基本单位,而基因的转录和转录后调控是生命活动中至关重要的过程。
本文将简要介绍基因的转录和转录后调控的基本概念、重要的调控元件和机制。
基因的转录基因的转录是指DNA到RNA的过程,通过这个过程,基因的遗传信息将被转录为RNA。
在转录的过程中,RNA聚合酶与DNA的双螺旋结构结合,将DNA的碱基序列转化为RNA。
RNA按照DNA的序列从5’端向3’端合成,并且是单链结构。
这个过程在细胞质中进行,并且是一个复杂而精准的过程。
需要注意的是,基因的转录并非所有DNA都能被转录为RNA。
只有具有适当的启动子和主启动子的DNA序列才能在某些细胞类型中进行转录。
有时候还需要一些转录因子才能使启动子更加容易激活转录。
同时,基因的表达也是受到其他生理和环境因素的影响的。
基因的转录后调控转录后调控指的是对基因转录产物的调控,包括RNA的加工、修饰、稳定性及运输等过程。
转录后调控可以通过RNA的可变剪接、RNA的修饰、RNA干涉、RNA稳定性和RNA翻译等方式实现基因表达调控。
RNA的可变剪接RNA的可变剪接是指同一个基因的RNA前体分子(即前mRNA或者pre-mRNA)在不同的生理和生化状态下,会被不同的剪接因子剪切成不同的剪接变体。
这样,通过可变剪接就可以使具有同一基因信息的RNA表现出不同的性质。
例如,神经元特异性剪接因子的存在可以自然选择地使某些mRNA剪接成更具有神经元特异性的形式。
这样可变剪接不仅增加了RNA的多样性,而且还可以通过不同的剪接变体来实现基因的更加复杂的表达调控。
RNA序列的修饰RNA序列的修饰是指RNA分子中某些核苷酸上的化学修饰。
这些化学修饰可能影响RNA的稳定性、局部和全局的折叠以及RNA和其他分子之间的相互作用。
RNA序列修饰对生命活动的影响是多重的,它们可以通过影响转录、翻译和RNA间作用等多个层面来实现基因表达调控的效果。
RNA干涉RNA干涉是一种可以对RNA的表达和功能进行调控的机制。
基因转录的启动与调控

基因转录的启动与调控基因转录是生命活动中非常关键的一个过程,它确保了细胞内的合成蛋白质数量和种类的适当调节,同时也影响了细胞的多种功能。
在细胞内,基因的转录是由一系列过程驱动的,同时也受到许多因素的调节控制。
下面,我们将深入探讨基因转录的启动与调控。
一、转录起始位点的识别基因转录的起始位点是指RNA合成的起点,它是转录起始信号的重要组成部分。
起始位点的识别过程由两个主要因素驱动:第一个是DNA序列本身,第二个是RNA聚合酶 (RNAP) 与其它蛋白质的相互作用。
DNA序列中的启动子元件与其他水平调节元件共同参与了起始位点的识别。
启动子元件通常包括推动序列和启动子序列。
推动序列从1位点数到约-40位点数,通过与RNAP结合产生力学张力来引导其向下滑动。
起始位点通常位于推动序列的上游区,而启动子序列则位于起始位点的下游区。
RNA聚合酶结合到DNA的起始位点后,会依次进行复杂的动态结构变化,并且形成一个稳定的转录泡。
二、转录激活子复合物转录激活子是一种很重要的因子,它能够调节基因转录的速率及其表达的时期。
当DNA序列被特定的转录激活子激活时,这一基因的转录水平就会显著上升。
转录激活子复合物由多个蛋白质组成,它们可以与基因上的特定区域相互作用,从而识别特定的基因序列,激活DNA转录过程,调节基因表达。
三、染色质结构的重要性染色质的密度结构对基因转录具有极大的影响。
一般情况下,浓缩染色质的DNA序列较难转录。
因此,在启动基因转录前,染色质结构的松弛是极其关键的。
松弛染色质的最有效方式是通过其存在的酶催化代谢,在染色质上产生修饰标记,控制或激发转录因子的结合。
异构酶对N末端乙酰化的组蛋白H3和H4具有重要作用,这可以使染色质分子松弛并识别起始位点,可在进一步的过程中识别进一步的调节因子。
四、转录辅助因子在细胞内,转录因子是调控基因转录的一类蛋白质家族,它们可以促进RNA 聚合酶与DNA序列的结合,调节基因转录的速率,影响细胞的功能活性和特性。
转录调控名词解释

转录调控名词解释转录调控是一种技术,它可以调节基因表达。
它指的是影响基因表达的基因的组成、结构和互作的过程,从而影响细胞表型的方式。
简单来说,转录调控是对基因表达过程的控制和调整,使某些基因表达而其他基因不表达。
转录调控可以分为三个不同的步骤:增强子、增强子活动和转录因子结合。
增强子是DNA序列,在特定的位置形成编码蛋白质的基因上。
增强子可以激活和抑制其后面的基因,因此他们具有调节基因表达的能力。
增强子活动是指包括增强子在内的促使基因表达的过程中,DNA酸和蛋白质的相互作用。
转录因子结合是指转录因子和DNA的结合,它们可以识别和结合特定基因上的增强子,从而激活或抑制基因的表达。
一些转录因子在受到信号刺激时可以激活抑制的增强子,而其他的转录因子可以在增强子活动过程中发挥作用。
转录因子结合是调节基因表达的关键部位。
转录调控通常是一个复杂的过程,它可以以不同途径调节基因表达,比如RNA干扰、转录因子、结合位点等。
脂质和蛋白质也可以参与转录调控,它们可以变更增强子的活性,也可以抑制转录因子的结合。
在不同的细胞类型中,转录调控的作用会有所不同,比如类似细胞环境和内分泌系统。
在内分泌系统中,转录调控可以调节激素水平,或者在系统发生变化时,调节细胞的特殊功能。
转录调控是调节基因表达的一类重要技术,它可以调控细胞的功能和表型。
通过调节基因的表达,转录调控可以改变细胞的生理特征,从而影响细胞的行为。
通过调节基因的表达,转录调控可以控制和调整细胞的功能,从而改变细胞的表型和生命周期。
转录调控在多种研究领域扮演着重要的角色,比如发育生物学、病原学和药物发现等。
它可以用来解读基因表达的影响,从而对特定基因的功能和表达机理进行分析。
转录调控也可以用来鉴定特定基因的功能,并设计能够调节基因表达的药物。
转录调控是一种重要的技术,它可以调节基因表达,从而调节细胞的表型和生活周期。
它可以用于科学研究,用来揭示基因的功能,探讨基因的表达机理,确定新的药物靶标,及为药物研发提供有效治疗路径。
基因组学课件73基因表达的转录后调控
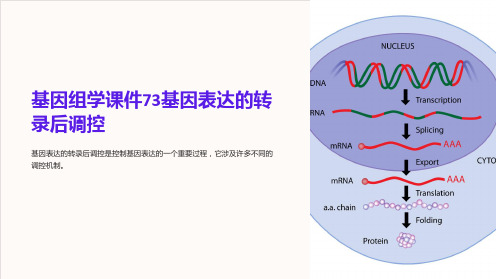
基因表达的转录后调控是控制基因表达的一个重要过程,它涉及许多不同的 调控机制。
转录后调控的定义和概述
定义
转录后调控是指 转录后 对 RNA 的 进一步调控作用,进而影响基因表达。
概述
它对格局和强度进行调整,以保证基因表达适应不同的细胞状态和环境的需要。
重要性
转录后调控在许多生物学过程中都起着至关重要的作用,尤其是在细胞分化和发育过程中。
转录后调控的机制和过程
1
mRNA降解
通过降解mRNA来调整基因表达水平,这是一个快速的机制。
2
RNA剪接
将RNA中的一个或多个内含子切割掉或不保留,产生不同的变异体。
3
RNA编辑
通过改变RNA序列中的碱基,以改变RNA翻译成蛋白质的方式,从而影响蛋白质 的功能。
miRNA的调节作用
miRNA的结构
未来转录后调控的研究方向
单细胞研究
发展单细胞技术,研究基因表 达在不同类型细胞中的转录后 调控机制。
技术革新
研究新的技术平台来更好地了 解转录后调控机制,例如: Riboseq,Proteomics
物种之间的比较
比较特定物种之间的转录后调 控差异,揭示物种进化的基础, 为生物学研究提供更多的细节。
3 影响因素
转录因子的表达水平和表 达模式可以在细胞分化和 特定环境下发生变化。
转录后修饰的调节作用
1
RNA剪接后修饰
包括带电氨基酸的碳酸酐化、磷Байду номын сангаас化、去甲基化等,它们可以增强或降低RNA的 稳定性。
2
蛋白质的翻译后修饰
一些修饰会影响蛋白质的配置或功能,从而影响基因表达。
转录调控与基因表达调控机制

转录调控与基因表达调控机制转录调控是指在DNA转录为RNA过程中,通过调节转录因子在DNA上的结合、启动子的开放和RNA聚合酶的招募等多个环节,来控制基因表达的过程。
基因表达是指表现出来的基因性状,它是由DNA的序列信息指导的过程,包含三个级别的调控机制:转录调控、RNA加工和蛋白翻译调控。
在转录调控中,核心的调控因子是转录因子,它们能够识别DNA上的特定序列,结合到启动子、增强子等位点上,调节染色质结构和RNA聚合酶的招募,从而影响转录的速率和效率。
转录因子通常包括DNA结合域和调控域,DNA结合域是掌握与DNA上特定序列结合的区域,而调控域则可以招募其他转录因子或配体、酶类等分子,形成调节复合物,并对基因表达发挥不同的调控作用。
除了转录因子以外,还存在一些非编码RNA能够通过抑制或增强转录因子的活性,或参与RNA的加工、剪接等环节,从而调控基因表达。
非编码RNA通常包括miRNA、lncRNA、siRNA等,其中miRNA能够识别底层靶基因mRNA并引导RNA蛋白复合体切割或抑制翻译,从而降低基因表达;而lncRNA则能够通过结合转录因子、DNA序列或其他蛋白,影响基因转录的起始、速率和空间定位等方面,发挥重要的调控作用。
在基因表达调控的过程中,还存在染色质构象和核小体等层次结构的影响。
染色质构象是指DNA、蛋白质和非编码RNA组合形成的三维结构,它可以隔离某些序列,阻拦或促进转录因子的结合,从而影响基因转录。
同时,染色质构象也可以受到某些特定修饰(如乙酰化、甲基化等)的调节,从而改变染色质的紧密程度和易位性。
除此之外,核小体也扮演着重要的调控角色。
核小体是由八个核心组蛋白(H2A、H2B、H3和H4)和一个连接组蛋白(H1)组成的核蛋白质复合物,它们协同作用形成近似“珠弹链”的结构,调节DNA的超级螺旋结构和染色质结构,并参与基因转录的起始和跑动等方面。
总体来看,转录调控与基因表达调控机制是复杂的、严密的多层次调控网络,它涉及到DNA、RNA、蛋白质和非编码RNA以及染色质、核小体等多个方面。
基因转录调控和功能的结构基础

基因转录调控和功能的结构基础基因转录是一个复杂的生物学过程,它涉及到DNA信号的获取、信号传递以及蛋白质的合成。
在分子生物学中,基因转录是生物体中基因的表达,它决定了某一物种的特征。
基因转录调控是指进一步控制基因转录的一系列调控机制,这些机制包括DNA编码和非编码区域的结构、启动子区域、调控元件以及转录因子等。
基因转录调控的调控方式主要是针对转录因子的调控,转录因子是在指定基因序列上能够相互作用并影响基因表达的体系。
调控元件是指转录因子识别的DNA 序列(哨兵基序)和蛋白质交互作用的区域。
转录因子与每个调控元件都有一些共性的某些蛋白质,这些蛋白质通过复合体的形式参与到基因转录的调节中。
例如,细胞中的调控元件往往包含了活性化和抑制元件,满足在不同物理环境和化学环境下的差异状态。
在真核生物中,这些调控元件一般是小片段(50到1000个碱基对)的DNA序列,这些DNA序列能够正或反调节靶基因的转录。
一些含有反式基序的DNA序列在结构上与转录因子连接,从而在基因转录调控系统的调控上起到了特定的作用。
除了调控元件的结构外,信号调控还涉及到其他一些结构方面的因素,例如表观遗传学标记、反转录途径、基因组重复以及蛋白质的修饰等。
表观遗传学标记是一些表观修饰因子的任意交互,包括化学变化的DNA、组蛋白标记、甲基化修饰和翻译后修饰等。
具体来说,这些表观遗传标记会随着时间的推移而改变,因此会在基因转录调控过程中产生不同的效果。
反转录途径是指对这种中性细胞或其他非主流细胞进行基因的转录,从而建立了另一组基因序列文件(RNA逆转录)。
基因组重复是一个更广泛的概念,它是指每个基因组内基因组元件的重复次数以及基因组内每个基因沿着序列的重复次数。
基因组重复会影响基因转录,从而影响细胞功能和行为。
蛋白质修饰是另一个重要的因素,它可以影响蛋白质的结构和稳定性,从而改变基因转录的速率和稳定性。
蛋白质修饰的主要形式包括磷酸化、乙酰化、丝氨酸/苏氨酸/酪氨酸磷酸化、葡萄糖磷酸化和泛素化等。
基因的转录、转录后调控

基因的转录、转录后调控基因的转录、转录后加工及逆转录转录(transcription)是以DNA单链为模板,NTP为原料,在DNA依赖的RNA聚合酶催化下合成RNA链的过程。
与DNA的复制相比,有很多相同或相似之处,亦有其特点,它们之间的异同可简要示于表13-1转录的模板是单链DNA,与复制的模板有较多的不同特点,引出了下列相关概念。
转录过程只以基因组DNA中编码RNA(mRNA、tRNA、rRNA及小RNA)的区段为模板。
把DNA分子中能转录出RNA的区段,称为结构基因(structure gene)。
结构基因的双链中,仅有一股链作为模板转录成RNA,称为模板链(template strand),也称作Watson(W)链(Watson strand)、负(-)链(minus strand)或反意义链(antisense strand)。
与模板链相对应的互补链,其编码区的碱基序列与mRNA的密码序列相同(仅T、U互换),称为编码链(coding strand),也称作Crick(C)链(Crick strand)、正(+)链(plus strand),或有意义链(sense strand)。
不同基因的模板链与编码链,在DNA分子上并不是固定在某一股链,这种现象称为不对称转录(asymmetric transcription)。
模板链在相同双链的不同单股时,由于转录方向都从5’→3’,表观上转录方向相反,如图13-1。
与DNA复制类似,转录过程在原核生物和真核生物中所需的酶和相关因子有所不同,转录过程及转录后的加工修饰亦有差异。
下面的讨论中将分别叙述。
参与转录的酶转录酶(transcriptase)是依赖DNA的RNA聚合酶(DNA dependent RNA polymerase,DDRP),亦称为DNA指导的RNA 聚合酶(DNA directed RNA polymerase),简称为RNA聚合酶(RNA pol)。
转录和转录水平的调控要点说明

转录和转录水平的调控要点说明SECTION 5转录和转录水平的调控重点:转录的反应体系,原核生物RNA聚合酶和真核生物中的RNA聚合酶的特点,RNA的转录过程大体可分为起始、延长、终止三个阶段。
真核RNA的转录后加工,包括各种RNA前体的加工过程。
基因表达调控的基本概念、特点、基本原理。
乳糖操纵子的结构、负性调控、正性调控、协调调节、转录衰减、SOS反应。
难点:转录模板的不对称性极其命名,原核生物及真核生物的转录起始,真核生物的转录终止,mRNA前体的剪接机制(套索的形成及剪接),第Ⅰ、Ⅱ类和第Ⅳ类含子的剪接过程,四膜虫rRNA前体的加工,核酶的作用机理。
真核基因及基因表达调控的特点、顺式作用元件和反式作用因子的概念、种类和特点. 以及它们在转录激活中的作用。
一.模板和酶:要点1.模板RNA的转录合成需要DNA做模板,DNA双链中只有一股链起模板作用,指导RNA合成的一股DNA链称为模板链(template strand),与之相对的另一股链为编码链(coding strand),不对称转录有两方面含义:一是DNA链上只有部分的区段作为转录模板(有意义链或模板链),二是模板链并非自始至终位于同一股DNA单链上。
2.RNA聚合酶转录需要RNA聚合酶。
原核生物的RNA聚合酶由多个亚基组成:α2ββ'称为核心酶,转录延长只需核心酶即可。
α2ββ'σ称为全酶,转录起始前需要σ亚基辨认起始点,所以全酶是转录起始必需的。
真核生物RNA聚合酶有RNA-polⅠ、Ⅱ、Ⅲ三种,分别转录45s-rRNA; mRNA(其前体是hnRNA);以及5s-rRNA、snRNA 和tRNA。
3.模板与酶的辨认结合转录模板上有被RNA聚合酶辨认和结合的位点。
在转录起始之前被RNA聚合酶结合的DNA部位称为启动子。
典型的原核生物启动子序列是-35区的TTGACA 序列和-10区的Pribnow盒即TATAAT序列。
第章基因表达和调控ppt课件

广泛表达
弱化子attenuator
▪ 大肠杆菌的色氨酸支配子 ▪ 在trp mRNA 5’端trp E基因的起始密码子
前有一个长162bp的DNA序列称为前导区, 其中第123~150位核苷酸假设缺失,trp基 因的表达程度可提高6倍
▪ 当mRNA开场所成后,除非培育基中完全不
含有色氨酸,否那么转录总是在这个区域 终止
原核生物:支配子 真核生物:“同表达基因群〞 (synexpression group),时间和空间上
2. 转录后的调控
▪ (1) 转录后RNA的切割调控 ▪ (2) mRNA的修饰和加工 ▪ ① 选择性剪接或可变剪接 ▪ ② 甲基化:6-甲基腺嘌呤(6mA),
5′Apm6ApC3′和 5′Gpm6ApC3′
一、调控元件
▪ 1. 启动子 ▪ 上游(Upstream):基因转录起点前面即5’
端的序列
▪ 下游(Downstream):基因转录起点后面即
3‘端的序列,把起点的位置记为+1
▪ 启动子区:RNA聚合酶同启动子结合的区
域
原核生物启动子
▪ Pribnow box (-10 sequence):
原核基因转录起始点的上游10碱基处(-10bp) 的序列,其根本构造是TATAATG
eukaryotes
DNA sequences involved in the control of transcription: enhancer
eukaryotes
eukaryotes
Structure of the enhancer of SV40
眼特异加强子 肠特异加强子
广泛加强子
只在眼 中表达
▪ ③ RNA编辑 ▪ 翻译扩增Translational amplification:在转录
转录调控和基因表达的调节

转录调控和基因表达的调节随着基因技术的不断发展,转录调控和基因表达的调节成为了分子生物学研究中的重要课题之一。
转录调控是指调控基因DNA转录成RNA的过程,是基因表达调节的关键环节之一。
基因表达调节是生物体内基因表达的动态调整,是维持生命正常运转的基础。
那么,转录调控和基因表达的调节是如何进行的呢?1. 转录调控的基本机制在基因表达过程中,存在着DNA、RNA和蛋白质三种生物大分子的相互作用。
其中RNA是承担转录、翻译功能的重要角色。
因此,在转录调控中起关键作用的分子之一就是RNA聚合酶,它是在DNA模板上合成mRNA的酶。
RNA聚合酶必须在DNA上准确识别启动子区域,同时在正确的条件下与一系列调节因子相互配合才能正常进行转录调控。
对DNA进行修饰(例如去甲基化、甲基化)或者是染色质结构的变化都会影响调控因子与启动子的相互作用,从而影响转录调控。
其次,一些转录因子可以直接与DNA结合来识别并绑定到启动子区域并调节RNA聚合酶的活性。
2. 基因表达调节的方式基因表达调节是通过启动子的活性、转录因子的稳定性、转录前体RNA的加工和稳定度以及MIRNA等多种方式进行的。
启动子是在基因调节过程中起关键作用的区域,它集中了一系列调控元件,包括各种转录因子结合位点和组蛋白修饰位点等。
因此,在基因表达调节中通过修饰启动子的状态来控制基因表达的活性是十分关键的。
其中转录因子是一类可以识别和结合到DNA上的调控分子,转录因子的数量和稳定性决定了要将某个基因表达到何种程度。
过程涉及到转录因子家族的扩张和缩小等复杂的调节机制。
此外,核糖体转录后的加工、修饰和分泌等形成的多种RNA分子也可以作为基因表达调控的重要手段。
其中MIRNA是一类短小的RNA分子,不仅可以直接与靶基因mRNA结合,而且还可以与转录因子相互作用,从而调节基因表达。
在不同的细胞环境中,不同的基因会表达不同的MIRNA,从而发挥不同的调控作用。
3. 化学品调控基因表达的策略现代分子生物学技术和生物化学技术的不断发展,许多新的手段也被用于探究转录调控和基因表达的调节。
转录调控与基因调节

转录调控与基因调节基因是生命的基本单位,有关基因的研究已经成为现代科学发展的重要方向之一。
实际上,基因的调控机制也是众多神秘现象的源头。
那么,什么是基因的调控机制呢?转录调控和基因调节是否有关呢?转录调控是指在生物细胞中,通过一系列复杂的调控机制来控制转录过程,从而调节基因表达的过程。
转录是指把DNA转录为RNA的过程,而这中间需要依赖一系列的转录因子、蛋白质以及其他调控成分的协同作用,才能进行顺利的转录。
基因调节则是指通过一系列的分子机制调节基因表达的过程,是基因调控的一个重要过程。
在细胞中,转录调控与基因调节是密不可分的关系。
实际上,转录调控对于基因调节的影响非常大,因为只有正常的转录过程才能产生出良好的基因表达,从而实现细胞正常的生长和分化。
为了实现有效的转录调控,细胞在转录因子的选择、mRNA的修饰和RNA降解等方面也开发了一系列的调控机制。
在转录调控的过程中,细胞内的蛋白质与核酸互相作用,通过某些分子机制来调控基因表达。
这一过程可以分为几个关键的步骤:1. 转录启动:这是转录调控中的最重要的一步,是转录起始的关键阶段。
在细胞内,转录因子会特定地与RNA聚合酶,与DNA结合并形成一个开放式的转录启动复合体。
2. 转录延伸:此时RNA聚合酶开始让模板DNA序列进行转录,生成一个长度为1000-2000个碱基的RNA前体。
3. 转录终止:RNA聚合酶通过终止信号序列终止转录,此时拷贝的前体RNA已经形成。
4. RNA后处理:前体RNA需经过后期加工,包括剪切、聚腺苷酸尾和RNA编辑,以生成成熟的mRNA。
在基因调节中,调节因子介导着表观遗传的调节机制。
表观遗传是指这些调节过程不会改变基因序列,而是通过其他一些分子机制来实现,比如DNA甲基化、组蛋白修饰等。
这些表观遗传机制已经被证明能够对许多生物学过程产生重要的影响,包括细胞周期、分化、发育和衰老等。
细胞内的基因调节与外部环境之间存在许多关联。
分子生物学基础第七章真核基因表达的调控第三节真核基因表达转录水平的调控

第七章 真核基因表达的调控
第三节 真核基因表达转录水平的调控
一、真核基因转录与染色质结构变化的关系 DNA绝大部分都在细胞核内与组蛋白等结合成染色质, 染色质的结构影响转录,至少有以下现象: 1.染色质结构影响基因转录 在真核细胞中以核小体为基本单位的染色质是真核基 因组DNA的主要存在方式。DNA盘绕组蛋白核心形成核小体, 妨碍了与转录因子及RNA聚合酶的靠近和结合,使基因的 活性受到抑制。 2.组蛋白的作用 组蛋白H1及核心组蛋白共同参与核小体的组装与凝聚。 在特殊氨基酸残基上的乙酰化、甲基化或磷酸化等修饰, 可改变蛋白质分子表面的电荷,影响核小体的结构,从而 调节基因的活性。
第三节 真核基因表达转录水平的调控
图7-6 碱性螺旋-环-螺旋结构图
第三节 真核基因表达转录水平的调控
螺旋-转角-螺旋结构域是最早发现于原核生物中的一个关键因子, 该结构域长约20个aa,主要是两个α-螺旋区和将其隔开的β转角。 其中的一个被称为识别螺旋区,因为它常常带有数个直接与DNA序列 相识别的氨基酸。其结构如图7-3所示。
图7-3 螺旋-转角-螺旋结构及其与 DNA的结合
第三节 真核基因表达转录水平的调控
2.增强子 增强子是指能使基因转录频率明显增加的DNA序列。增强子的作 用有以下特点。 ①增强效应十分明显。一般能使基因转录频率增加10~200倍,有 的可以增加上千倍, ②增强效应与其位置和取向无关。 ③大多为重复序列。 ④增强效应有严密的组织和细胞特异性。说明只有特定的蛋白质 (转录因子)参与才能发挥其功能。 ⑤没有基因专一性,可以在不同的基因组合上表现增强效应。 ⑥许多增强子还受外部信号的调控,如金属硫蛋白的基因启动区 上游所带的增强子,就可以对环境中的锌、镉浓度做出反应。 ⑦增强子要有启动子才能控
转录后修饰和基因调控的生物学功能

转录后修饰和基因调控的生物学功能生命的诞生和演化是一个复杂而精彩的过程,基因的调控和表达则是其中至关重要的环节。
生物体内的基因编码了蛋白质以及许多非编码RNA,这些RNA都需要经过调控才能发挥各自的生物学功能。
而转录后修饰和基因调控则是这个过程中最为重要的两个方面。
一、转录后修饰近年来,在研究基因的调控过程中,转录后修饰这个领域受到了越来越多的关注。
转录后修饰是指在RNA被合成后,在其他分子的作用下进行的化学修饰。
这些修饰会影响RNA的降解、本地化、翻译和互相作用,从而调控基因的表达。
RNA的修饰可以分为两类:核苷酸的修饰和磷酸酸酯的修饰。
其中,核苷酸的修饰包括:甲基化、脱胺基化、伪尿嘧啶修饰、2-羟甲基基团修饰等。
而磷酸酸酯的修饰则包括:丝氨酸磷酸化、酪氨酸磷酸化、苏氨酸磷酸化等。
这些修饰的存在使得RNA能够更为复杂地发挥作用。
例如,tRNA上的甲基化、脱胺基化和伪尿嘧啶修饰可以影响蛋氨酸的翻译,并调控细胞的代谢活性和分化。
此外,磷酸化也被发现可以影响RNA结构,从而调控RNA-蛋白质相互作用。
转录后修饰不仅影响基因表达,也影响了RNA丰富的功能,对生命的演化具有深刻的影响。
二、基因调控除了转录后修饰外,基因调控也是生物体内基因表达的重要手段。
基因调控是指在胞外或细胞内受到某些信号之后,通过各种分子和机制执行的一系列过程,从而调控基因的表达。
基因调控可以分为正向调控和负向调控两种。
例如,在转录因子的正向作用下,可以促进mRNA的合成,从而增强基因的表达。
而在转录因子的负向作用下,则可以抑制mRNA的合成,从而减弱或抑制基因的表达。
基因调控可以发生在DNA水平和RNA水平两个方面。
在DNA水平,基因的表达可以通过启动子、增强子和基因沉默等机制来调控。
而在RNA水平,则可以通过特定的miRNA和lncRNA来调控RNA的合成、定位和翻译。
基因调控的研究无疑将为解决许多疾病带来新的解决方案。
最近的研究表明,基因调控在某些疾病的发生和发展中起着至关重要的作用,这意味着掌握基因调控机制可以为治疗疾病提供新思路。
基因的转录和调节

碱基序列分析结果表明,启动子-10区的保守序列 为TATAAT,该区由Pribnow首先发现,称为Pribnow 盒。Pribnow盒能决定转录的方向,在Pribnow盒区 DNA双螺旋解开与RNA聚合酶形成复合物。
35区位于Pribnow盒的上游,是启动子中另外一个 重要区域,该区域也存在着类似于Pribnow盒的共同 序列TTGACAT。目前认为-35区是RNA聚合酶对转 录起始的辨认位点。 RNA聚合酶与-35区辨认结合后,能向下游移动,达 到-10区的Pribnow盒,在该区RNA聚合酶能和解开的 DNA双链形成稳定的酶-DNA开放启动子复合物,就 可以开始转录。
6
第一节基因转录和转录后加工
基因转录是RNA合成的主要方式和基因信息 表达的重要环节,是遗传信息从DNA向RNA传 递的过程。转录生成的RNA是初级转录产物, primary transcripts,必须经过不同方式的加工 和修饰才具有生物活性。
7
以DNA为模板合成RNA的过程称为转录, transcription,即把DNA的碱基序列转抄成 RNA。 在这个过程有很多因素参与其中,包括 1. DNA模板, template 2. RNA聚合酶, RNA polymerase 3. 三磷酸核糖核苷, NTP 4 .一些与转录相关的有同样的基 因,然而不同组织细胞的基因表达情况不同,有些基 因被启动进行表达,有些基因被抑制不表达或少表达。 即使在同一类型细胞的不同发育阶段,基因表达情况 也有不同。基因表达调控遵循一般的规则,即一个体 系在需要时被打开,不需要时就被关闭或抑制。这种 基因“开”和“关”的控制是通过对基因信息传递过 程的多个环节来实现的。
17
A 大肠杆菌RNA聚合酶全酶 B 酝酒酵母RNA聚合酶全酶
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
三、ChIP-seq技术
创立者: 2007年,Steven J.M. Jones等人率先提出的 特点:
染色质免疫沉淀后的DNA,直接进行高通量测序。 是一个“开放系统”。它可以检测更小的结合区段、未知的结 合位点、结合位点内的突变情况和蛋白亲合力较低的区段。
成本低,周期短,省去了标记和杂交等步骤,并且勿需多次重 复实验,极大提高了工作效率。
这种表示方法直观地给出模体各个位置上碱基出现的倾向
性和整个模体的序列的一致性。
二、转录因子结合位点的识别
基本概念:
通过收集可能被同一转录因子调控的基因启动子序列,在其中寻找
具有统计显著性的短片段,作为转录因子可能的结合位点,称之为转 录因子结合位点的识别
基本流程 :
收集可能被同一转录因子调控的多基因序列 通过多种计算方法从不同角度或不同层面去进行计算、评估和分析, 尽可能地屏蔽掉冗余序列和噪音序列,寻找出具有统计显著性的短 片段,作为转录因子可能的结合位点
BIOINFORMATIC ANALYSIS OF TRANSCRIPTIONAL REGULATION
Wednesday, December 03, 2014
第 一 节
引
言
一 、基因转录调节的基本模式
transcription factor
cis-regulatory element
转录调控可以控制转录何时发生以及产生多少RNA。
exes
Add proteinspecific antibody
Amplify DNA and Label ChIP- chip
Hybridize to arrays
Reverse crosslinks and purify DNA PCR
Input DNA IgG Sp1 c-Jun
No DNA
实验设计的关键
抗体质量:一个灵敏度高和特异性高的抗体可以得到富集的DNA片段,这有利于 探测结合位点。
样本量:Illumina,10-50ng DNA,需要用PCR进行扩增的轮数也比较少,因而由 PCR导致的偏差比较小
空白对照:空白对照是必要的,存在很多假阳性情况,举例:1. 开放的染色体区 域更容易被打断成片段,这样导致tag数在基因组上的分布是不均匀的;2.很多重复 序列会使做map的时候得到结果难以解释。空白对照的用途:可以判断由ChIP-Seq 得到的peak时候具有统计上的显著性。 三种类型的空白对照:1.部分进行免疫共沉淀前的DNA(input DNA),这是最常 用的;2.由免疫共沉淀得到而不含有抗体的DNA(mock IP DNA),使用这个的一个 问题是收集到的量可能不够;3.使用非特异免疫共沉淀方法得到的DNA. 测序深度:在发表的ChIP-Seq实验中,一般使用Illumina Genome Analyzer上一个 lane产生的数据作为一个基本单位,目前一个lane大概是8-15million reads(2009 数据)。判断足够的测序深度的标准是:当增加测序,得到更多的reads的时候不 能发现更多的东西。应该这一准则到结合位点的数量上就是:进行测序,增加 reads数而无法得到更多的结合位点。
结果分析的标准化尚待完善 分辨率较低,大于200 bp
基因芯片是 “封闭系统”, 只能检测已知序列
ChIP(染色质免疫沉淀)技术与芯片技术相结合后,产生了 染色质免疫共沉淀-芯片技术(Chromatin Immunoprecipitationchip, ChIP-chip)。免疫共沉淀结合微阵列技术可以在基因组范围 内筛选蛋白结合靶点,成为深入分析癌症、心血管疾病以及中央 神经系统紊乱等疾病的主要代谢通路的一种非常有效的工具。 一般来说,ChIP-chip分3步完成: ChIP:是在生理状态下把胞内蛋白质和DNA甲醛作用下交联在一起,用超
Motif discovery
logo→
frequency matrix →
(一)共有序列(consensus sequence)
将能与同一个转录因子结合的所有DNA 片段按照对应位
置进行排列,在每个位置上选择最可能出现的碱基,就
组成了该转录因子结合位点的共有序列。
共有序列中用A、C、G、T 之外的字母来表示结合位点
中各个位置上可能出现的碱基组合,这些字母称为 IUPAC 简并码。
Cross-link whole cells with formaldehyde Isolate genomic Sonicate DNA DNA to produce sheared, soluble chromtin
sequencing
Region sequenced
ChIP- seq
加入特异性抗体, Immunoprecip 沉淀蛋白质 - DNA itate and purify 复合物 imminocompl 去交联,纯化 DNA 应 用 PCR 技 术 , 特异性扩增目的 DNA片段
分辨率可提高到30-50bp。
ChIP-Seq的优势 1. 具有碱基层面的分辨率;
2. 不会有ChIP-chip中由DNA片段杂交导致的噪音,GC
含量、片段长度、片段浓度以及耳机结构都会对杂
交造成影响;
3. ChIP-chip中的微阵列信号不是线性增长的,其所测 量的范围有限。 4. 由于在设计array时,探针的数量、种类有限,当 coverage比较高的时候无法准确测量,也无法发现
二、 基因转录调节机制的研究方法
实验方法:
荧光素酶报告基因(luciferase report gene) 凝胶迁移(electrophoreticmobility shift assays) 染色质免疫沉淀(chromosome immunopreciation,
ChIP)
DNase 足迹法(DNase footprinting)
信息学分析
第 二 节 转录调控的高通量实验测定
一、ChIP(染色质免疫沉淀)技术
创立者:上世纪八十年代末,Alexander Varshavsky等人
ChIP是研究体内蛋白质与DNA相互作用的一种技术。它 利用抗原抗体反应的特异性,可以真实地反映体内蛋白因子与 基因组DNA结合的状况。
特点:
查询相关转录因子数据库,以确定转录因子
基本流程
cDNA chip ChIP-chip ChIP-seq 2-D PAGE-MS
>seq-1 TTAACCTCTTATCTCTCCCCAAGATCCCTGAAGCCAGGTACGAGCAAGATGAGAGTGGGTTATCTCTGGA >seq-2 TCCTGTAGTGGGCATTCCAGGAGCAGAATGGCGTCATAATTCATTTACTCTATAAGTCAGAGAGAAAAAT ∙∙∙∙ >seq-n TATGTGGTTATTAAATGTTAAGGAGATGCAGAGTAGGGTAAATTGTTTATCTGAGAGGCTGGGCTTAGGA
三 种 主 要 数 据 分 布 类 型
第三节 转录因子结合位点的信息学预测方法
一、转录因子结合位点的的表示方法
(一)共有序列(consensus sequence) (二)位置频率矩阵(position frequency matrix) (三)序列标识图(sequence logo)
consensus→
共有序列的表示方法简明易懂,却不能够反映每个位置
上不同碱基出现的概率。
IUPAC简并码
IUPAC code
W
Nucleotide
A or T
IUPAC code
B
Nucleotide
C, G or T
R
K S Y
A or G
G or T C or G C or T
D
H V N
A, G or T
q A,1 q A,2 ∙∙∙ q A,n
M= q
q C,1 q C,2 ∙∙∙ q C,n
G,1
q G,2 ∙∙∙ q G,n
q T,1 q T,2 ∙∙∙ q T,n
(三)序列标识图
序列标识图依次绘出模体中各个位置上出现的碱基,每个
位置上所有碱基的高度和反映了该位置上碱基的一致性,
每个碱基字母的大小与碱基在该位置上出现的频率成正比。
被RNA聚合酶转录的基因可被至少五种机制所调控:
特异性因子会改变RNA聚合酶对于特定启动子或一套启动子识别的特异性,使得 RNA聚合酶更多或更少地结合到这些启动子上。
阻遏因子会结合到DNA链上的靠近或覆盖启动子区域的那些非编码序列上,阻碍
RNA聚合酶顺利进入此链,故阻碍了基因的表达。 通用转录因子:这些转录因子将RNA聚合酶安放至编码蛋白序列的起始位置,继而 释放聚合酶以转录mRNA。 激活因子增强RNA聚合酶与特定启动子的相互作用,促进基因的表达。激活因子通 过增强RNA聚合酶对启动子的吸引而达到此作用,这一机制是通过与RNA聚合酶亚基的 相互作用或间接通过改变DNA结构而实现的。 增强子是位于DNA螺旋结构上的一些位点,它们通过与激活因子相结合以将DNA弯 曲使特定启动子朝向起始复合物。
声波打碎为0.2-2 kb的染色体小片段,然后通过目的蛋白质特异性抗体沉淀此 复合物,获得特异地作用于目的蛋白结合的DNA片段;
DNA处理:通过对目的片段的纯化与检测,从而获得蛋白质与DNA相互作
用的信息。其中共沉淀的 DNA和合适的对照用荧光标记,加在载玻片上,用 于芯片分析;
芯片分析:使用外源性DNA作为背景,免疫沉淀DNA与作为背景的对照进
针对某一特定候选转录因子,是否特异性结合于所调节的
靶基因某一预定区域内,如启动子区,进行检测。
对同一DNA底物, 可以运用多种不同的抗体 , 分别进行免疫
