Affymetrix 全基因组 SNP 芯片检测
SNP开发验证的研究方法和技术路线

SNP开发/验证的研究方法和技术路线1分子标记:分子标记,我想这部分是我们分子标记组最核心的任务。
现在,我们没有任何可用的标记检测我们的定位材料。
即使想要验证已经定位的QTLs,我们也需要相对应的区间内的分子标记,尤其是SNP 标记。
1.1 全基因组SNP—Affymetrix芯片:一套完整的全基因组的SNP芯片,相对于Douglas体系,其操作简单,高通量。
可以直接对定位群体进行初定位的扫描或是对育种材料的背景进行分析。
在国家玉米改良中心,有一套3k的Illumina芯片,就是用来对玉米材料进行高通量检测,基因型检测结果通常可以用来QTLs初定位,育种材料的群体划分与纯度鉴定以及低密度的关联分析等。
在此,我建议我们应该开发一套番茄基因型检测的芯片。
目前,只是查找到Illumina芯片有一套全基因SNP信息,包含7,720条探针。
而Affymetrix公司目前并没有相应的产品。
但是通过跟Affymetrix公司了解,可以利用Illumina芯片已有的结果进行开发。
番茄目前测序结果显示其全基因组大小为~760Mb,而玉米为~2,500Mb,但是他们包括的基因数目~30,000个,整体情况相近。
另外,番茄作为自交植物,其LD的衰减值应该更大,有效的历史重组会更少,遗传多样性低。
因此,综合考虑,我建议我们可以开发~3k芯片,应该可以满足大多数研究材料、育种材料的基因型检测需求。
虽然目前下一代测序技术蓬勃发展,但是对于用于基因型检测来讲,其数据分析与成本相对于芯片都要更复杂和更高。
总之,我们番茄处于刚刚发展阶段,我认为就基因型检测方面,芯片有其很高的应用价值。
即使像玉米,这样测序技术发展很多年的材料,芯片技术也在应用。
1.2全基因组SNP—Douglas:当用Affymetrix芯片检测鉴定完番茄基因型并完成基因型分析之后,1)对于优良的QTLs或是基因,页脚内容1我们可以直接选择覆盖整个区间的分子标记运行Douglas系统进行分子标记辅助育种,2)对于需要进一步验证的QTLs,我们也可以利用Douglas系统只检测材料覆盖定位区间的基因型,而不需要再一次利用Affymetrix芯片或是其他方法进行全基因检测(图1.1)。
全基因组关联分析(GWAS)解决方案

全基因组关联分析(GWAS)解决方案※ 概述全基因组关联研究(Genome-wide association study,GWAS)是用来检测全基因组范围的遗传变异与可观测的性状之间的遗传关联的一种策略。
2005年,Science杂志报道了第一篇GWAS研究——年龄相关性黄斑变性,之后陆续出现了有关冠心病、肥胖、2型糖尿病、甘油三酯、精神分裂症等的研究报道。
截至2010年底,单是在人类上就有1212篇GWAS文章被发表,涉及210个性状。
GWAS主要基于共变法的思想,该方法是人类进行科学思维和实践的最重要工具之一;统计学研究也表明,GWAS很长时期内都将处于蓬勃发展期(如下图所示)。
基因型数据和表型数据的获得,随着诸多新技术的发展变得日益海量、廉价、快捷、准确和全面:如Affymetrix和Illumina公司的SNP基因分型芯片已经可以达到2M的标记密度;便携式电子器械将产生海量的表型数据;新一代测序技术的迅猛发展,将催生更高通量、更多类别的基因型,以及不同类别的高通量表型。
基于此,我们推出GWAS的完整解决方案,协助您一起探索生物奥秘。
※ 实验技术流程※ 基于芯片的GWASAffymetrix公司针对人类全基因组SNP检测推出多个版本检测芯片,2007年5月份,Affymetrix公司发布了人全基因组SNP 6.0芯片,包含90多万个用于单核苷酸多态性(SNP)检测探针和更多数量的用于拷贝数变化(CNV)检测的非多态性探针。
因此这种芯片可检测超过180万个位点基因组序列变异,即可用于全基因组SNP分析,又可用于CNV分析,真正实现了一种芯片两种用途,方便研究者挖掘基因组序列变异信息。
Illumina激光共聚焦微珠芯片平台为全世界的科研用户提供了最为先进的SNP(单核苷酸多态性)研究平台。
Illumina的SNP芯片有两类,一类是基于infinium技术的全基因组SNP检测芯片(Infinium™ Whole Genome Genotyping),适用于全基因组SNP分型研究及基因拷贝数变化研究,一张芯片检测几十万标签SNP位点,提供大规模疾病基因扫描(Hap660,1M)。
基因芯片知名企业

基因芯片知名企业基因芯片是一种高通量的基因检测技术,可以同时检测数千个基因的表达水平、突变状态、拷贝数变化等信息。
这种技术在生物医学研究、临床诊断、药物研发等领域有着广泛的应用。
目前,全球有许多知名的基因芯片企业,其中包括Illumina、Affymetrix、Agilent、Thermo Fisher Scientific等。
Illumina是全球最大的基因芯片企业之一,总部位于美国加利福尼亚州圣地亚哥市。
该公司成立于1998年,最初是一家生物芯片企业,后来专注于基因测序和基因芯片技术的研发和生产。
Illumina的基因芯片产品包括全基因组芯片、转录组芯片、表观基因组芯片等,广泛应用于生物医学研究、临床诊断、药物研发等领域。
Illumina的技术优势在于高通量、高灵敏度、高精度和高可靠性,被广泛认可和应用。
Affymetrix是另一家知名的基因芯片企业,总部位于美国加利福尼亚州圣克拉拉市。
该公司成立于1992年,是全球第一家生产基因芯片的企业。
Affymetrix的基因芯片产品包括全基因组芯片、转录组芯片、SNP芯片等,应用于生物医学研究、临床诊断、药物研发等领域。
Affymetrix的技术优势在于高通量、高灵敏度、高特异性和高可靠性,被广泛认可和应用。
Agilent是一家全球性的科技企业,总部位于美国加利福尼亚州圣克拉拉市。
该公司成立于1999年,最初是惠普公司的电子测量与仪器部门,后来独立成为一家科技企业。
Agilent的基因芯片产品包括全基因组芯片、转录组芯片、miRNA芯片等,应用于生物医学研究、临床诊断、药物研发等领域。
Agilent的技术优势在于高通量、高灵敏度、高特异性和高可靠性,被广泛认可和应用。
Thermo Fisher Scientific是一家全球性的科技企业,总部位于美国马萨诸塞州威尔明顿市。
该公司成立于2006年,是由Thermo Electron Corporation和Fisher Scientific International合并而成。
SNP芯片数据分析

Affymetrix SNP芯片数据分析方案项目一、基本分析包括:芯片原始数据的处理和基因分型,我们给出有统计意义的SNP列表。
描述性统计,如minor allele frequency,Hardy-Weinberg equilibrium等。
显著性检验,实验组与对照组的差异,假阳性率(FDR)的计算等。
SNP的关联分析,建立线性模型或logistic回归模型等。
(所有的统计可以选择由SAS,SPSS,或S-Plus/R给出)项目二、Copy Number Variation(CNV)的计算。
CNV是目前的一个热点研究内容。
SNP芯片数据可以用于精确地计算CNV。
我们提供针对SNP芯片的基于CNAG(Copy Number Analyser for GeneChip), dChip(DNA-Chip Analyzer)和CNAT(Chromosome Copy Number Analysis Tool)等算法的CNV计算结果。
项目三、SNP注释通过SNP在染色体上的位置,利用寻找SNP可能影响的基因( or EST)。
我们也可以对相应基因进行功能的注释(gene ontology ,pathway和转录因子分析等),进而解释SNP可能的作用机理。
该部分可以参考常规表达谱芯片的分析。
项目四:基于模式识别的SNP挖掘传统的SNP挖掘使用统计学的方法来进行,往往在敏感性与特异性上有一定的限制。
利用一些模式识别/机器学习的方法可以更好解决SNP筛选问题。
我们提供基于决策树等SNP挖掘算法。
Hsiang-Yu Yuan et al. FASTSNP: an always up-to-date and extendable service for SNP function analysis and prioritization. Nucleic Acids Research 2006 34(Web Server issue):W635-W641项目五:诊断模型建立利用筛选到的SNP建立人工神经网络(ANN)、SVM、PAML等诊断模型,在临床上具有重要意义。
AFFYMETRIX基因芯片操作流程

AFFYMETRIX 基因芯片操作流程第一章真核靶片断制备<一> RNA 的抽提一、 哺乳动物细胞或组织RNA 的抽提1. 总 RNA 使用 QIAGEN ' 哺乳动物组织作为+2. Poly(A) mRNA 哺乳动物细胞使用 哺乳动物组织作为 离步骤或使用kit.二、 RNA 沉淀1. 总 RNA在用RNeasy Total RNA Isolation kit 分离或洗涤后没有必要沉淀总 RNA.调整洗脱体积以制备cDNA 合成接近希望的 RNA 浓度。
注:为获得足够量的标记 cRNA 用来评估和基因芯片表达探针杂交, AFFYMETRIX 建议开 始合成cDNA 的Poly(A) +mRNA 最小浓度为0.02卩g/卩l 时的最小量是0.2卩g,总RNA 最 小浓度为0.5卩g/卩l 时的最小量是5卩g.这样有两个好处: (1) 有足够量在各步检查样品浓度和质量 (2)制备足够的cRNA 用于杂交在TRIzol 分离和热酚提取后需要乙醇沉淀;见下面方法 +2. Poly(A) mRNA大多数Poly(A) +mRNA 分离过程都会导致得到较稀浓的 RNA ,所以需要在cDNA 合成前浓缩mRNA. 3. 沉淀步骤: (1) 力口 1/10体积3M NaOAc,PH5.2,和2.5倍体积乙醇. (2) 混匀,-20C 放置最少1小时. (3) 4C ,> 12000x g 离心 20 分钟. (4) 80%乙醇洗涤沉淀 2次.(5) 空气干燥沉淀.继续下面步骤前检查是否干燥 . (6)DEPC 处理水重新溶解沉淀.最合适的溶解体积由cDNA 合成中需要的 RNA 的浓度和量来决定.先阅读cDNA 合成的过程来决定这一步的适合溶解体积4. RNA 测定用分光光度计分析 RNA 浓度,在260nm1单位吸光度等于 40卩g/mlRNA.需要在260和280nm 测定吸光度来确定样品的浓度和纯度 A 260/A 280应接近2.0为较纯的RNA(比值在1.9-2.1也可)<二>由纯化的总RNA 合成双链cDNAAFFYMETRIX 强烈建议 HPLC 纯化 T7-(d7) 24 primerRNeasy Total RNA Isolation kit 成功抽提哺乳动物细胞总 RNA.RNA 的来源,建议使用 TRIzol 抽提总RNA.QIAGEN ' Oligotex Direct mRNA kit,从总 RNA 中抽提 mRNA . RNA 的来源,应首先使用 TRIzol 纯化,再进行一个 Poly(A)+mRNA 分一、第一链cDNA合成开始RNA的量:高质量RNA5.0卩g -40.0卩g纯化后RNA浓缩由260nm吸光度决定(1单位吸光度=40卩g/mlRNA ), A260/A280应接近2.0,在1.8-2.1的范围内。
全基因组扫描

遗传分析仍是当前对致病相关基因识别、鉴定的主要方法,分为连锁分析和关联研究两种。由于人类基因组多态性的研究以及SNP分型技术的发展,目前全基因组连锁分析和关联研究亦变得切实可行。根据研究规模的大小,可以将疾病遗传分析分为以下几类,即定位克隆、连锁不平衡基因定位、全基因组候选基因分析、候选基因关联研究和定位候选基因克隆,其中定位克隆、连锁不平衡基因定位和全基因组候选基因分析均属于全基因组扫描。
全基因组扫描所利用的是在人类基因组大量存在的微卫星或SNP,虽然当前使用较多的仍是微卫星,但由于芯片技术的发展,全基因组高分布密度的商品化SNP芯片相继面世(如Affymetrix公司的10k,100k和500k人基因组SNP芯片),越来越多的研究者使用SNP进行全基因组扫描。由于这些高密度的SNP芯片价格昂贵,不是一般的实验室所能承受。
不管是单基因疾病还是多基因疾病,通常是先行全基因组扫描(genome scanning);将疾病相关位点定位于染色体某个区域,然后再行候选基因策略或连锁不平衡分析,确定致病基因位点。如果利用家系进行连锁分析,即采用定位克隆;若是利用群体样本,则应用连锁不平衡分析进行基因定位。全基因组扫描已成功地应用在许多疾病的致病相关基因克隆上,并取得了一定的成果。
affymetrix-optima

• •
POC 样本的6号染色体整条为嵌合型缺失 通 14过 smooth signal 和allele peaks tracks可推测到嵌合比例为35%
14158: Mosaic monosomy 6 (~35%) on a POC sample
• •
Whole-genomeview aids in the identification of themosaic monosomy6 15 35% mosaic is clearly visible on the allele differencetrack and smooth signal
High density
Mendelian consistency checking
Aid in the confirmation of copy-number Events
SNPs
Triploidy
High-density SNPs allow for all of these performance attributes to be measured at gene-level resolution.
Relation
Family
CytoScan Optima有丰富的可 检测基因表型的SNP探针, 可运行亲源性分析 Chas 3.0 Trio Tool可检测亲缘 不一致
A-0015275 A-0012037 A-0012038 A-0015266 A-0012011 A-0012010 A-0015275 A-0012037 A-0012010
8 19 29
9 20
14 15 23 24 33 34 43 44 53 54 63 64 73 83 84 93 94 85 95 25 35 45 55 65
SNP检测方法汇总

现在SNP得常用检测方法主要有:Taqman法、质谱法、芯片法、测序法。
Taqman法:准确性高,适合于大样本、少位点,价格比较贵;质谱法:准确性高,适合于大样本、多位点(能检测25个位点);芯片法:准确性较低,适合于超多位点分析;测序法:非常准确,但就是价格也非常得高,但就是对于少样本、超多位点还就是非常好得选择。
SNP检测方法汇总分析SNP得方法有许多种,本文收集目前还在用得方法,按通量从高到低排列:全基因组测序这就是最贵得方法,但也就是瞧SNP最全得方法大概一个人样本,花2万元外显子组测序外显子组测序,也可以得到较全面得SNP信息大概一个人样本,花1、5万元随着人全基因组测序得价格降到2万元左右,外显子组测序会很快退出市场全基因组SNP芯片原理,核酸杂交,荧光扫描Illumina与Affymetrix都有很著名得全基因组SNP芯片,例如:Affymetrix: CytoScan,SNP 6、0,Illumina: 660,中华,450K等SNP芯片,在2000~5000元每样本,还就是比全基因组测序得2万元一个样本得价格要低质谱法原理,精确测量PCR产物得分子量,就可以知道SNP位点上就是A/C/G/T中得哪一个Sequenome MassArray法测中等通量得SNP位点就是十分准确得单个位点、单个样本得费用约2元人民币无需预制芯片、预订荧光探针,只要合成常规得PCR引物就可以做实验了如果测几十个点,到上百个点,就是很方便得方法SNPseq法此方法为天昊公司所创,一次测几百个位点原理:用Goldgate法做出针对某些位点得多重PCR片段高通量测序,数据分析得到SNP位点结果SNPlex中等偏高通量得方法,一次几十个位点原理:用末端特异得引物做多重PCR,把模板进行扩增基于毛细管电泳,把片段分离开,读颜色SNaPshot中等通量得方法设计3'位挨着目标位点得探针用双脱氧得荧光标记ddNTP做一个碱基得延伸毛细管电泳,瞧延伸得这个碱基就是什么颜色Taqman法Taqman原理,如果要找原理,请回复“荧光”两字Taqman方法,一次一管测一个位点通量最低,但就是结果可靠原理:设计与SNP位点互补得荧光探针,其中一个标VIC(红色荧光基团),另一个标FAM(绿色荧光基团),同时分别有淬来基团吸光Taq酶有5'-->3'得外切酶活性,如果探针粘有模板上,就被切碎探针被切碎后,荧光基团与淬灭基团分离,发出荧光。
SNP检测方法汇总

SNP检测方法汇总SNP(Single Nucleotide Polymorphism)是存在于基因组中的最小的遗传变异单位,是指基因组中单个核苷酸发生变化的现象。
SNP检测方法是针对这些变异进行分析和检测的工具或技术。
本文将对目前常用的SNP检测方法进行汇总和介绍。
1.基于PCR的SNP检测方法PCR是一种常用的DNA复制技术,在SNP检测中有多种变体,包括追踪标记PCR(TaqMan PCR)、Allele-Specific PCR(AS-PCR)、限制性片段长度多态性(RFLP)PCR等。
这些方法都利用PCR扩增目标DNA片段,并通过引入特定的引物或酶切位点来区分不同等位基因的差异。
2.基于测序的SNP检测方法测序是一种直接测定DNA序列的方法,可以通过测序检测SNP。
在基于测序的SNP检测中,有两种主要的方法:Sanger测序和大规模并行测序(Next-Generation Sequencing,NGS)。
Sanger测序是一种经典的测序方法,能够准确地确定单个核苷酸的序列,但是对于大规模SNP检测来说成本较高。
而NGS技术则可以同时测定多个样本的DNA序列,且速度和成本都更高效。
3.基于芯片的SNP检测方法芯片技术是通过固相法在芯片上固定已知的DNA片段,再与样本中的DNA进行杂交来实现SNP检测。
常用的芯片技术包括基于碱基延伸法(Primer Extension Assay)的Oligonucleotide Ligation Assay (OLA)、基于碱基延伸法的SNPstream和基于液相杂交法的GeneChip等。
这些方法在检测过程中通常采用荧光探针标记样本的SNP位点,通过荧光检测的方式进行分析和鉴定。
4.基于质谱的SNP检测方法质谱技术是通过检测质量-电荷比(m/z)来对样本中的DNA片段进行分析和检测的方法。
基于质谱的SNP检测主要采用基因分型质谱法(genotyping mass spectrometry),其中常用的方法有MALDI-TOF质谱(Matrix-Assisted Laser Desorption/Ionization Time-of-Flight Mass Spectrometry)和Sequenom质谱。
Affymetrix芯片质量评估

Affymetrix芯片质量评估在拿到(数据库下载或者自己实验得到)的芯片,最好先对芯片的质量做出评估,从而将有问题的芯片剔除。
在RobertGentleman的“Bioinformatics and Computational Biology Solutions UsingR and Bioconductor”书的第三章提到“Before any useis made of more complexmethods, an initial examination of the data can often show evidenceof possible quality problems.”。
以下关于Affymetrix芯片质控的图和脚本,也引用上述参考书。
首先是一些基础知识:1 Affymetrix芯片的数据格式主要有.dat和.cel两种。
DAT文件是原始芯片图像的扫描文件,需要用affy公司自己的软件打开。
CEL文件是DAT文件去除背景噪音后的文件,包括了每个探针的原始密度数值(rawintensityvalue)。
其中,我们最关注CEL文件,这也是我们后续载入Bioconductor中的原始数据类型。
后续需要对CEL文件进行“质量评估”、“归一化”、“注释”等一系列预处理。
2affy芯片的数据单元。
下图是一个“探针集(probeset)”,包括了11-20个长度位22nt的“探针(probe)”,图中每个亮格代表一个探针。
每个探针分为PM(PerfectMatch)和MM(Mis-Match)两种,区别就是MM探针故意将一个碱基设计错。
这样做的目的是为了控制芯片的非特异性杂交,从而获得更准确的信号值。
芯片质量控制:1. 对于“探针数据(probe-data level)”的三种图,使用"affy"packageboxplot():未处理的原始探针密度(以2为底取对数)的盒箱图。
affymetrix 基因表达谱芯片 差异基因 -回复

affymetrix 基因表达谱芯片差异基因-回复什么是Affymetrix基因表达谱芯片和差异基因?Affymetrix基因表达谱芯片是一种高通量基因分析工具,用于研究生物样本中基因的表达水平。
它由小型玻璃片或硅片制成,上面镶嵌着几十万个DNA探针,每个探针对应一个已知的基因序列。
通过检测样本中基因与这些探针的杂交态势,可以确定每个基因在样本中的表达水平。
差异基因是指在不同组织、时间点、环境或疾病状态下,表达水平有明显差异的基因。
通过对比不同组样本的基因表达谱,可以鉴定差异基因,从而了解其在不同生物学过程中的作用和调控机制。
接下来,我们将一步一步回答有关这两个主题的问题。
第一步:什么是Affymetrix基因表达谱芯片?Affymetrix基因表达谱芯片是一种高通量基因分析工具,采用探针杂交技术来测量基因在生物样本中的表达水平。
芯片上有成千上万个特定的DNA探针,每个探针与一个已知的基因序列相对应。
样本RNA通过逆转录生成cDNA,然后标记为探针上的亮度标记,并与芯片上的DNA探针进行杂交。
基于杂交的信号强度可以确定每个基因在样本中的表达水平高低。
第二步:Affymetrix基因表达谱芯片的工作原理是什么?Affymetrix基因表达谱芯片的工作原理基于探针与样本RNA之间的互补配对。
首先,将样本RNA转化为cDNA,并标记为亮度标记。
然后,将标记的cDNA与芯片上的DNA探针进行杂交。
杂交过程中,标记的cDNA与互补的DNA探针形成稳定的双链结构,杂交态势的强度与目标基因在样本中的表达水平成正比。
最后,通过检测杂交态势的强度,可以确定每个基因在样本中的表达水平。
第三步:为什么使用Affymetrix基因表达谱芯片?Affymetrix基因表达谱芯片具有以下优点:1. 高通量分析:Affymetrix芯片上的数千个探针允许一次性测量大量基因的表达水平,加快了实验进程。
2. 高度标准化:Affymetrix芯片的制造过程高度标准化,确保了数据的可靠性和可比性。
AFFYMETRIX基因芯片操作流程

AFFYMETRIX基因芯片操作流程1.设计芯片:根据研究需求,设计基因芯片的探针序列。
探针序列是一小段DNA或RNA序列,用于检测芯片上的特定基因。
通常,基因芯片上会有上万个探针,可以检测大量的基因。
2.提取RNA或DNA:从感兴趣的生物样本中提取总RNA或基因组DNA。
RNA或DNA提取的方法会根据具体的研究目的和样本类型而有所不同。
3.RNA/DNA标记:将提取的RNA或DNA样本进行标记。
在基因芯片研究中,常使用荧光标记的核苷酸来标记RNA或DNA。
标记的方法可以是直接标记或间接标记,具体选择取决于实验的设计和要求。
4.混合和杂交:将标记的RNA或DNA样本与设计好的探针序列混合,形成杂交溶液。
杂交时,样本中的标记的RNA或DNA会与芯片上的相应探针序列结合。
5.洗涤和扫描:对芯片进行洗涤去除杂质,然后使用芯片扫描仪对芯片进行扫描。
扫描会产生荧光图像,显示不同基因的表达水平或基因变异情况。
6.数据分析:使用专门的数据分析软件对扫描后的图像进行处理和分析。
这些软件可以提供丰富的数据分析工具,包括基因表达聚类、差异分析、通路分析等。
通过数据分析,可以得到关于基因表达的定量和质量信息。
7.结果解读:根据数据分析的结果,解读实验结果。
通过比较不同样本之间的基因表达差异,可以找到与研究目的相关的基因。
根据差异基因的功能注释和通路分析等,可以深入了解基因的作用和功能。
8. 结果验证:将一部分差异表达的基因进行验证实验,如RT-PCR、Northern blot等。
验证实验可以进一步确认基因表达的差异,并验证基因芯片分析的可靠性。
通过以上步骤,AFFYMETRIX基因芯片可以帮助研究人员高通量、高效率地研究基因的表达水平和基因变异等生物过程。
同时,数据分析和结果解读对于科研的深入和扎实也非常重要。
染色体微阵列技术检测父源性t(13;22)非平衡易位22q13缺失综合征一例及文献复习
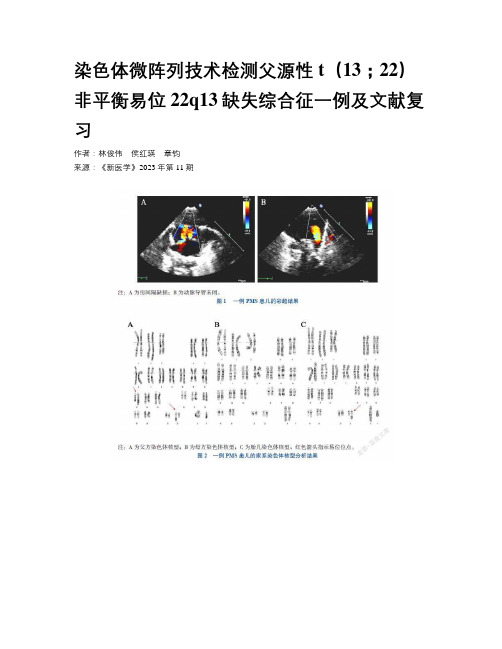
染色体微阵列技术检测父源性t(13;22)非平衡易位22q13缺失综合征一例及文献复习作者:林俊伟侯红瑛章钧来源:《新医学》2023年第11期【摘要】目的从遗传学角度分析22q13缺失综合征病因。
方法应用G显带的染色体核型分析技術及全基因组单核苷酸多态性微阵列分析技术(SNP-array)对一例22q13缺失综合征患儿进行遗传学检测,并对患儿父母进行外周血染色体核型分析,验证患儿染色体异常来源。
以“t(13;22)”为检索词,在生物医学文献外文数据库(PubMed)、中国知网全文数据库(CNKI)、万方数据库、重庆维普学术期刊数据库进行检索,收集亲代遗传t(13;22)非平衡易位病例进行分析。
结果该例患儿母亲染色体核型分析结果为46,XX,患儿父亲染色体核型分析结果为46,XY,t(13;22)(q31;q13.3),患儿SNP-array分析结果提示13号染色体长臂q31.1q34存在35.8 Mb片段重复,22号染色体长臂q13.3发生1.1 Mb片段缺失,染色体核型分析结果为46,XY,der(22)t(13;22)(q31;q13.3)pat。
文献复习3例患者病例表现为智力低下、癫痫、面部畸形、房间隔缺损等,其中一例出生11 d死亡。
结论患儿遗传父源性der(22)t(13;22)(q31;q13.3)片段,13号染色体存在35.8 Mb重复,为染色体异常,具有致病性;22号染色体存在1.1 Mb缺失,该变异片段包括已知的遗传综合征区域,即22q13缺失综合征。
【关键词】单核苷酸多态性微阵列技术;染色体非平衡易位;遗传学分析;染色体核型分析;22q13缺失综合征;费伦-麦克德米德综合征Single nucleotide polymorphisms array for detecting paternal t (13;22) unbalanced translocation 22q13 deletion syndrome: a case report and literature review Lin Junwei, Hou Hongying, Zhang Jun. Department of Obstetrics and Gynecology, the Third Affiliated Hospital of Sun Yat-sen University,Guangzhou 510630, ChinaCorresponding author, Zhang Jun, E-mail:****************【Abstract】 Objective To analyze the etiology of 22q13 deletion syndrome. Methods Genetic analysis was performed for a baby with 22q13 deletion syndrome using G-banded chromosome karyotype analysis and single nucleotide polymorphisms array (SNP-array) techniques. Chromosomal karyotype analysis of peripheral blood of parents was also conducted to identify the origin of chromosomal abnormality. Literature review was performed using the keywords of “t(13;22)” in PubMed, CNKI, Wanfang Data, Chongqing VIP databases. Cases of parental genetic t (13; 22)unbalanced translocation were analyzed. Results The mother’s karyotype was 46,XX,while the father’s karyotype was 46, XY, t(13; 22)(q31; q13.3). The results of SNP-array analysis showed 35.80 Mb fragment duplication in 13q31.1q34 and 1.10 Mb fragment deletion in 22q13.3 in the premature baby. The karyotype of the premature baby was 46, XY, der (22)t(13; 22)(q31; q13.3)pat. According to literature review, three patients presented with mental retardation, epilepsy, facial deformity and atrial septal defect, etc. One of them died 11 d after birth. Conclusions The child carries a paternally inherited der(22)t(13; 22)(q31;q13.3) translocation. Specifically, 35.80 Mb fragment duplication is observed in 13q31.1q34,which is abnormal and pathogenic. 1.10 Mb fragment deletion is noted in 22q13.3, which includes 22q13 deletion syndrome (PMS).【Key words】 Single nucleotide polymorphisms array; Unbalanced chromosomal translocation; Genetic analysis;Chromosome karyotype analysis;22q13 deletion syndrome; Phelan-McDermid syndrome染色体平衡易位是染色体结构变异常见的一种类型,正常人群平衡易位携带者发病率约为0.2%。
LOH检测
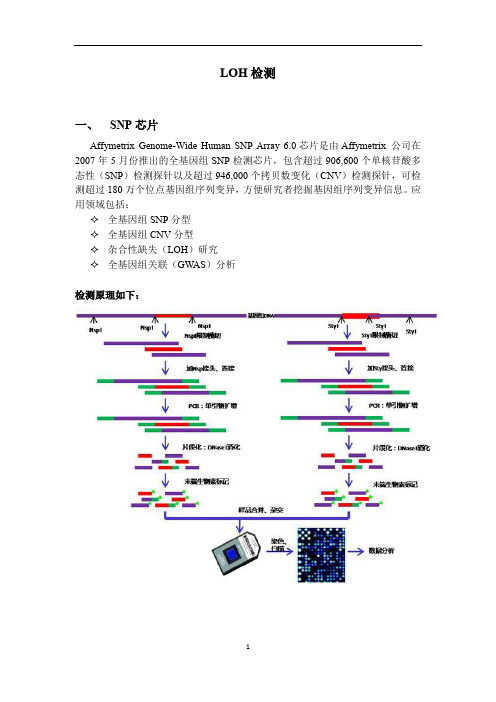
LOH检测一、SNP芯片Affymetrix Genome-Wide Human SNP Array 6.0芯片是由Affymetrix 公司在2007年5月份推出的全基因组SNP检测芯片,包含超过906,600个单核苷酸多态性(SNP)检测探针以及超过946,000个拷贝数变化(CNV)检测探针,可检测超过180万个位点基因组序列变异,方便研究者挖掘基因组序列变异信息。
应用领域包括:✧全基因组SNP分型✧全基因组CNV分型✧杂合性缺失(LOH)研究✧全基因组关联(GWAS)分析检测原理如下:1.样本要求基因组DNA:总量>1μg;浓度>50ng/μl;260/280在1.8-2.0之间,260/230>1.8;电泳检测无降解。
2.检测流程(1)基因组DNA抽提、质检与定量(2)酶切与连接反应(3)PCR扩增及纯化(4)片段化及标记(5)样品杂交与洗脱(6)图像扫描(7)数据处理与分析二、CGH芯片Affymetrix公司采用改进的长探针合成技术(约49mer),专门针对人类基因组序列变异检测推出了人类细胞遗传学全基因组2.7M芯片(Cytogenetics Whole-Genome 2.7M)和细胞遗传学310K芯片(Cytogenetics 310K)两种芯片产品,分别在高分辨率和低分辨率下实现对人类基因组的检测。
Affymetrix Cytogenetics Whole –Genome 2.7M芯片,是高密度的高分辨率CGH分析工具,覆盖了全基因组约270万个标记(探针间隔距离中位值为735bp),其中包括约40万个SNP位点。
该芯片不但可以实现拷贝数变异的高分辨率检测,发现微小缺失和扩增,还可以检测染色体中性杂合性缺失(copy neutral LOH)、单亲二体病(UPD)及嵌合现象。
其中,以均匀分布的SNP marker对各种类型的杂合性缺失LOH进行分类是Affemetrix拷贝数变异检测独有的特色。
基因芯片(Affymetrix)分析2:芯片数据预处理

基因芯片(Affymetrix)分析2:芯片数据预处理基因芯片技术的特点是使用寡聚核苷酸探针检测基因。
前一节使用ReadAffy函数读取CEL文件获得的数据是探针水平的(probe level),即杂交信号,而芯片数据预处理的目的是将杂交信号转成表达数据(即表达水平数据,expression level data)。
存储探针水平数据的是AffyBatch类对象,而表达水平数据为ExpressionSet类对象。
基因芯片探针水平数据处理的R软件包有affy, affyPLM, affycomp, gcrma等,这些软件包都很有用。
如果没有安装可以通过运行下面R语句安装:Affy芯片数据的预处理一般有三个步骤:•背景处理(background adjustment)•归一化处理(normalization,或称为“标准化处理”)•汇总(summarization)。
最后一步获取表达水平数据。
需要说明的是,每个步骤都有很多不同的处理方法(算法),选择不同的处理方法对最终结果有非常大的影响。
选择哪种方法是仁者见仁智者见智,不同档次的杂志或编辑可能有不同的偏好。
1 需要了解的一点Affy芯片基础知识Affy基因芯片的探针长度为25个碱基,每个mRNA用11~20个探针去检测,检测同一个mRNA的一组探针称为probe sets。
由于探针长度较短,为保证杂交的特异性,affy公司为每个基因设计了两类探针,一类探针的序列与基因完全匹配,称为perfect match(PM)probes,另一类为不匹配的探针,称为mismatch (MM)probes。
PM和MM探针序列除第13个碱基外完全一样,在MM中把PM的第13个碱基换成了互补碱基。
PM和MM探针成对出现。
我们先使用前一节的方法载入数据并修改芯片名称:用pm和mm函数可查看每个探针的检测情况:上面显示的列名称就是探针的名称。
而基因名称用probeset名称表示:名称映射时会看到。
三种常见SNP芯片的工作原理(illumina、Affymetrix和Agilent)

三种常见SNP芯片的工作原理(illumina、Affymetrix和Agilent)写在读前:此文较长,建议先收藏。
这篇文章是从我无意间在网上发现的,但是不清楚是谁整理的。
但是我通过插图的截图上的信息找到出处,这些内容都是陈巍在腾讯视频上发布的,有人讲他的课程内容整理下来。
我觉得不错,所以就搬到这里。
其实可以并不用看这个文字,直接找视频看也是行的,但是文字版本更利于收藏。
本来想把视频放进来的,但是网速限制了我。
SNP芯片的原理1.Illumina的SNP芯片原理Illumina的SNP生物芯片的优势在于:第1,它的检测通量很大,一次可以检测几十万到几百万个SNP 位点第2,它的检测准确性很高,它的准确性可以达到99.9%以上第3,它的检测的费用相对低廉,大约一个90万位点的芯片(每个样本的)检测费用在一、两千人民币Illumina的生物芯片系统,主要是由:芯片、扫描仪、和分析软件组成。
Illumina的生物芯片,由2部分组成:第1是玻璃基片,第2是微珠。
这个玻璃基片,它的大小和一张普通的载玻片差不多大小,它起到的作用,就是给微珠做容器。
在这个玻璃基片上,通过光蚀刻的方法,蚀刻出许多个排列整齐的小孔。
每个小孔的尺寸都在微米级,这些小孔是未来容纳微珠的地方。
小孔的大小与微珠正好相匹配,一个小孔正好容纳一个微珠。
微珠是芯片的核心部分,微珠的体积很小,只有微米级。
每个微珠的表面,都各偶联了一种序列的DNA片段。
每个微珠上,有几十万个片段,而一个珠子上的片段,都是同一种序列。
这些DNA片段的长度是73个碱基,而这73个碱基又分成2个功能区域。
靠近珠子的这一端的23个碱基的序列,被称为Address序列,它也是DNA片段的5'端。
它是标识微珠的标签序列。
标签序列,通过碱基的排列组合,得到许多可能,每种序列,就是相应微珠的身份证号码(ID号)。
DNA片段上离珠子远的那一端的50个碱基,也就是3'端的序列,被称作Probe序列,它的作用,是与目标DNA进行互补杂交。
affy芯片

affy芯片Affymetrix 芯片是一种基因表达分析工具,被广泛应用于分子生物学和生物医学研究领域。
它是一种鞘片式(Chip)的微阵列技术,利用光刻技术将成千上万个特定序列化的核苷酸探针固定在芯片上,从而能够同时检测和分析成千上万个基因的表达水平。
Affymetrix 芯片的主要工作原理是基于亲和力识别的,通过将目标的DNA或RNA样本在芯片上进行杂交反应,利用亲和力使目标序列与芯片上的探针序列结合。
然后利用激光器和光敏染料对芯片上的信号进行扫描和检测,以获得每一个探针的荧光强度。
通过对探针的荧光强度进行定量分析,可以推断出样本中每个基因的表达水平。
Affymetrix 芯片具有以下几个优点:1. 高通量分析:一张芯片上可以同时检测和分析成千上万个基因的表达水平,能够提供大规模的基因表达数据,从而更全面地了解基因的功能和调控网络。
2. 准确性高:芯片上的探针经过精确的设计和制造,能够对目标序列进行高度特异性的识别,从而使实验结果具有较高的准确性和重复性。
3. 快速和节省成本:相比传统方法,Affymetrix 芯片能够在较短的时间内完成大规模的基因表达分析,并且减少了实验的复杂性和所需的样本量,从而节省了成本和资源。
4. 大规模基因注释数据:Affymetrix 芯片的设计和分析过程中使用了大量的基因注释数据,包括基因功能、调控网络、疾病相关性等信息,能够提供更加全面和深入的数据解读。
Affymetrix 芯片在生物医学研究中有着广泛的应用,例如:1. 基因表达分析:通过比较不同条件下的基因表达水平,可以揭示基因的功能和调控网络,从而帮助我们更好地理解生物学机制和疾病发生的原因。
2. 药物研发和毒性评价:通过分析药物对基因表达的影响,可以评估药物的疗效和毒性,从而加速新药的研发过程。
3. 疾病诊断和预后评估:通过分析患者样本中基因的表达水平,可以为疾病的诊断和预后评估提供有力的依据,从而指导个性化的治疗策略。
affymetrix mrna芯片原理 -回复

affymetrix mrna芯片原理-回复什么是Affymetrix mRNA芯片?Affymetrix mRNA芯片是一种广泛应用于基因表达分析的高通量技术。
基因表达分析旨在研究特定组织或细胞中基因的活动水平,从而了解生物体的功能和调控机制。
Affymetrix mRNA芯片可以同时检测成千上万个基因的表达水平变化,进而帮助科学家了解这些基因的功能以及它们在不同生物过程中的作用。
那么,Affymetrix mRNA芯片是如何工作的呢?1. 提取mRNA:首先,从特定样本中提取出细胞总RNA,并选取其中只含有成熟的mRNA。
因为mRNA负责传递DNA信息并参与蛋白质合成,因此它被认为是基因表达水平的重要指标。
2. 反转录和合成cDNA:在此步骤中,将提取到的mRNA经过逆转录反应转化为cDNA。
逆转录反应利用百万寡聚dT引物和逆转录酶(reverse transcriptase)将mRNA转录为相应的cDNA分子。
3. 合成(in vitro transcription):cDNA会经过dATP、dCTP、dGTP 和dUTP标记酶法引入复制体中,为下一步骤做准备。
与此同时,附着四个标记dNTPs的RNA酶会将DNA模板进行复制,并在此过程中合成标记过的RNA。
4. 破碎和杂交:在芯片的制作中,Array探针被固定在玻璃芯片上。
为了便于杂交,标记的RNA应该处于单股形式,因此接下来需要将复制得到的RNA进行热碎化。
这样的碎片化过程是通过加热和酶消化来实现的。
随后,将碎片化RNA与芯片上的Array探针进行杂交。
Array探针是一种特异性寡核苷酸序列,与芯片上每一个目标基因对应。
5. 洗涤和染色:经过杂交后,非特异连接的标记RNA将被清洗掉,只留下与特定目标基因匹配的RNA。
随后,在芯片上加入一种特定染色剂,使得能够区分不同基因的表达水平。
6. 扫描和分析:用激光扫描仪测量芯片上不同位置的荧光密度,并将这些数据传输至计算机以生成图像。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
Affymetrix 全基因组SNP 芯片检测
单核苷酸多态性(single nucleotide polymorphism, SNP) 指基因组单个核苷酸的变异,它是最微小的变异单元,是由单个核苷酸对置换、颠换、插入或缺失所形成的变异形式。
单核苷酸多态性是基因组上高密度的遗传标志,在人类基因组中已发现的SNP数量超过3000万。
作为第三代遗传标记,SNP数量众多、分布密集、易于检测,因而是理想的基因分型目标。
SNP分型检测在疾病基因组(如疾病易感性),药物基因组(药效、药物代谢差异和不良反应)和群体进化等研究中具有重大意义。
在人研究方面,Affymetrix 公司有分别基于GeneChip和GeneTitan平台的SNP 6.0 芯片和针对中国人群设计的CHB1&2 Array,既可用于全基因组SNP分析,又可用于CNV分析,极大地方便了中国人类疾病GWAS研究。
Affymetrix公司针对多个农业物种也开发了多款商品化的基因分型芯片,如鸡、牛、水牛、鲑鱼、水稻、小麦、辣椒、草莓等,为农业育种研究、遗传图谱构建、群体基因组学研究提供研究手段。
此外,Affymetrix公司还支持定制芯片,最低起订量为480个样品。
检测原理| 技术优势| 产品列表| 定制芯片| 数据分析|
基于GeneChip平台的人SNP 6.0 芯片实验流程:
基于GeneTitan平台的Axiom基因分型芯片检测流程:
从SNP原理谈SNP分析技术之SNP芯片
日期:2012-05-21 来源:网络
标签:SNP原理SNP分析SNP芯片
摘要: SNP是近年来基因突变的热点研究之一。
它是指在单个的核苷酸上发生了变异,有四种不同的变异形式,而实际上只发生转换和颠换这两种。
当科学家弄清了SNP的突变原理以后,他们就着手对SNP进行分析,以求找到疾病相对应的突变位点或者是进行个性化药物治疗研究。
其中应用到的技术多达上百余种,其中包括有测序技术、质谱分析技术、HRM技术、Taqman技术以及SNP芯片技术。
恩必美生物新一轮2-5折生物试剂大促销!
Ibidi细胞灌流培养系统-模拟血管血液流动状态下的细胞培养系统
广州赛诚生物基因表达调控专题
SNP是近年来基因突变的热点研究之一。
它是指在单个的核苷酸上发生了变异,有四种不同的变异形式,而实际上只发生转换和颠换这两种。
当科学家弄清了SNP的突变原理以后,他们就着手对SNP进行分析,以求找到疾病相对应的突变位点或者是进行个性化药物治疗研究。
其中应用到的技术多达上百余种,其中包括有测序技术、质谱分析技术、HRM 技术、Taqman技术以及SNP芯片技术。
SNP 的分型技术可分为两个时代,一为凝胶时代,二为高通量时代。
凝胶时代的主要技术和方法包括限制性酶切片段长度多态性分析(RFLP)、寡核苷酸连接分析(OLA)、等位基因特异聚合酶链反应分析(AS2PCR)、单链构象多态性分析(SSCP)、变性梯度凝胶电泳分析(DGGE),虽然这些技术与高通量时代的技术原理大致一样,但是由于它不能进行自动化,只能进行小规模的SNP分型测试,所以必然会被淘汰。
高通量时代的SNP分型技术按其技术原理可分为:特异位点杂交(ASH)、特异位点引物延伸(ASPE)、单碱基延伸(SBCE)、特异位点切割(ASC)和特异位点连接(ASL)5 种方法。
此外,采用特殊的质谱法和高效液相层析法也可以大规模、快速检出SNP 或进行SNP 的初筛。
近年来已经在晶体上用“光刻法”实现原位合成,直接合成高密度的可控序列寡核苷酸,使DNA 芯片法显示出强大威力,对SNP 的检测可以自动化、批量化,并已在建立SNP 图谱方面投入实际应用。
DNA 芯片法有望在片刻之间评价整个人类基因组。
2007 年5月份,Affymetrix公司发布了Genome-wide SNP 6.0 芯片,除包括90多万个用于单核苷酸多态性(SNP)检测探针外,还有90多万个用于拷贝数变化(CNV)检测的探针,可使全基因组平均分辨率达3 kb,既可用于全基因组SNP分析,又可用于CNV分析,真正实现了一种芯片两种用途,方便研究者挖掘基因组序列变异信息。
通过基因分型信息还可以鉴别中性拷贝数的杂合性缺失(copy neutral LOH)、单亲二体病(UPD)及嵌合现象(可以精确检测到20% 嵌合体)。
近来Affymetrix 公司又陆续发布了多款针对东亚、中国、
欧洲、非洲等不同人群的SNP基因分型芯片,采用GeneTitan平台进行高通量检测,极大地方便了人类疾病GWAS研究。
另外,也推出了牛、水稻等物种的基因分型芯片。
基于GENE Chip平台的人SNP 6.0 芯片实验流程:
基于GeneTitan平台的Axiom基因分型芯片检测流程:
现已发现的单核苷酸多态性在人类基因组上就已经达到了三千万以上。
SNP分析无论是对于疾病的诊治、药物的开发还是物种群体的进化都具有十分重要的意义。
问:
大夫您好,我女儿是高龄产妇,36岁,现在孕周是29周+,因为高龄所以未做唐筛,直接羊水穿刺,FLSH结果一周后出来无异常,羊水核型分析是9号染色体臂间倒位,医生建议他们夫妻做了外周血染色体检测,现在结果未出。
为保险起见,医生还建议他们用羊穿剩余的细胞液继续做SNP Array基因芯片检测,结果两周后出。
现在刚拿到结果,非常不好,9号染色体没有问题,却查出X染色体上有7.44M的片段缺失,并且包含了33个致病基因,特别是有一个CDKL5基因的缺失。
医生建议放弃这个孩子,他们很不甘心,之前的几次排畸B超都显示胎儿无任何异常。
所以,我们还想再请教一下,这样的检测结果是否100%准确?有这些致病基因的缺失是否一定会出现相应的表型?他们还需要再做什么进一步的检查吗?北京贝康医学检验所资质如何?他们如果还想怀孕需要注意什么?30周引产是不是会非常危险?
胎儿基因芯片检测结果显示X染色体上存在7.44mb的基因片段缺失,内含33个致病基因,这样的胎儿是否一定会出现致病基因提示的那些表型?B超显示胎儿无问题,我们是否必须放弃这个胎儿?我女儿这是第二胎,头胎是剖腹产,已经过了三年半,现在要30周引产,是否只能顺产不能剖腹产,危险很大吗?
答:
建议问问羊水穿刺检查实验室,胎儿如果是男胎,最好查下母亲的基因芯片分析。
如果女胎,则查夫妇双方芯片,看是否遗传。
问:
实验室告知了是女胎,认为遗传可能性不大,因为如果有这么大片段的基因缺失,我们夫妻二人一定会有表型,但我们现在很健康,基本可以排除是遗传因素,应该是基因突变。
而且如果我们夫妻二人再做基因检查,好需要三周时间,这样胎儿月份就更大了,引产会更困难了吧?
答:
缺失这么大片段理论上会有表型,但只是理论上,最好需要验证夫妇芯片,还是建议查夫妇芯片,至少弄清这个问题。
引产在22-28周之间差别不大。
问:
谢谢何大夫,我女儿现在已经30周了,等做完夫妻芯片就该33周了,有点太迟了。
另外,如果证明是夫妻一方遗传给孩子的,那能保证孩子也会像父母一样没有表型,是健康的吗?再次感谢您的回复,我们一家人在得知检查结果后各种纠结痛苦难以名状,遗传专家的号又极为难挂,您的回复给了我们极大的帮助,不管最后结局如何,我们都对您感激不尽。
答:
如果遗传自夫妇双方之一,提示出生后理论上应该和夫妇之一表型类似,即没有多大影响,这个问题一直没得到证实,孕周一天天大,你考虑的问题可以理解,但没得到明确的答案,所以一直纠结。
应该拿到报告时就果断去检测。
使用电话咨询服务
提交时
间:2016-03-12 11:45:06 预约
时
间:
2016-03-12
服务
费:200元/次(最长15分钟) 订单
状
态:
已结束
病情信息:1,基因分析正常,父母核型正常,羊穿fish结果正常,羊穿核型异常,一条4号染色体为衍生染色体,短臂末端有遗传物质增加。
脐带血核型异常,嵌合体,46,xn,der(4)[3] /46,xn[42] ,异常核型细胞比例6%,正常核型细胞比例94%。
镜下分析45个细胞,核型配对15个细胞。
2,22周大排畸发现侧脑室双侧宽9mm,29周5天核磁共振左侧宽14.5右侧宽13.3,33周5天侧宽是15和17。
核磁共振除了侧宽,其他结构正常。
3,孩子可以生吗?嵌合体的异常核型对孩子有什么影响?孩子以后能正常生育的比例有多少?。
