引物设计实例分析
pcr引物设计序列例题

PCR引物设计序列例题简介P C R(聚合酶链式反应)是一种常用的分子生物学技术,通过扩增DN A 分子可用于基因克隆、检测等多个领域。
而P CR引物则是PCR反应中的关键组成部分,它们的设计直接影响P CR反应的成功与否。
本文为您提供一些关于P CR引物设计序列的例题,希望对您在实验和研究中的引物设计有所帮助。
例题一:引物序列设计为了扩增目标基因的特定片段,我们需要设计一对引物,以下是目标基因的序列,请根据该序列设计两个引物,使其具有以下要求:-引物长度在18-25个碱基对之间-引物的3'端碱基G或C-引物的理论Tm在55-65℃之间目标基因序列:A G TC GA TC GA TT AG CGA T CG AG TC GA TC GA TCG G CT AC GA TC GA解答:根据目标基因的序列,我们可以设计如下两对引物:引物1:5'-A GT CG AT CG AT TAG C GA TC-3'引物1满足引物长度在18-25个碱基对之间的要求,同时3'端碱基为C,符合要求。
接下来我们计算一下该引物的理论T m。
根据以下两个规则计算引物的理论Tm:-A与T之间的配对带来的热能释放为-2.2k J/mo l-C与G之间的配对带来的热能释放为-3.8k J/mo l根据以上规则,我们可以计算每个碱基的贡献:-A-T配对:-2.2k J/m o l-C-G配对:-3.8k J/m o l-G-C配对:-3.8k J/m o l-T-A配对:-2.2k J/m o l引物1的理论T m计算如下:(-3.8×4)+(-2.2×10)=-15.2+-22=-37.2k J/mo l该引物的理论Tm为37.2℃,低于所需的要求。
引物2:5'-C GA TC GA TC GA TCG G CT AC G-3'引物2满足引物长度在18-25个碱基对之间的要求,同时3'端碱基为G,符合要求。
《PCR引物设计》课件

04
pcr引物的应用与案例分 析
pcr引物在基因克隆中的应用
01
pcr引物用于基因克隆的目的是为了获得目的基因的序列信息, 进而进行后续的基因功能和表达研究。
02
设计特异性引物,通过pcr技术,从基因或基因组中筛选出目的基因。
引物设计需考虑基因序列的特异性、扩增效率和避免非特异性
03
扩增等因素。
引物特异性优化
避免引物间的互补
引物之间不应存在互补序列,以避免形成引物二聚体或发夹 结构。
避免引物与模板扩增 和产物。
引物扩增效率的优化
引物与模板的匹配度
引物的3'端应与模板完全匹配,以提 高引物的扩增效率。
引物之间的匹配度
两个引物之间应有良好的匹配度,以 保证PCR反应的顺利进行。
引导合成
引物作为合成子链的起点,通过与 DNA聚合酶的结合,引导合成与 模板互补的DNA链。
决定产物长度
引物的设计决定了PCR产物的长度 ,通过选择合适的引物,可以控制 产物的大小和特异性。
pcr引物设计的基本原则
特异性
长度和序列
引物应与模板DNA具有高度的特异性,避 免与其他非目标DNA序列发生非特异性结 合。
pcr引物的未来发展方向与挑战
引物设计的自动化
随着生物信息学的发展,未来引物设计 可能更加自动化,减少人工干预和误差
。
标准化和质量控制
建立引物设计的标准化流程,加强引 物设计的质量控制,确保实验结果的
可靠性和可重复性。
新型引物设计策略
针对特定需求,开发新型引物设计策 略,提高PCR反应的特异性和灵敏度 。
引物灵敏度测试
03
测试引物在不同模板浓度下的扩增效率,选择灵敏度较高的引
实例图解简并引物设计 共25页

碱M
基B
代 码
D N
碱基 代码 A, G Y G, T S A, C W G, C, T V A, G, T H A, G, C, T
碱基 C, T G, C A, T A, G, C A, C, T
3. 质量评估
•(1)参数评估
将初步得到的引物序列,粘贴入Oligo Calc 的文本框内, 按下“Calculate”按钮,得到引物的相关参数,如:长 度、GC 含量、Tm 值等信息。
马铃薯Y 病毒(Potato virus Y,PVY)是侵染马铃薯、
烟草、辣椒等茄科作物并造成严重危害的病毒之一,广 泛分布全球各马铃薯种植区。RT-PCR 技术具有高度的特 异性和灵敏性等特点,已经成为 PVY 检测最常见的方法。 但由于PVY 株系分化严重,不断有新的重组株系产生, 单一的特异性引物无法适应PVY 不同株系的检测需求, 需要设计一对简并引物以能够满足生产上的检测需求。
八、详细图解
1. 序列准备: •(1)GenBank 下载PVY 全基因组序列; •(2)由于基因序列比较大,且数量多,推荐用MAFFT 多重序列比对; •(3)扩增片段区域选择,CP 基因长度及位置如下图所示:
• 先将光标定位在第一条序列任意位置,然后在左下角"Site"处直接输 入 CP 基 因 上 游 分 界 点 位 置 ( 8391 ) 后 回 车 。 接 着 点 击 “Speicaes/Abbrv”和8391 那一列的交界点时按下键盘上“Shift” 不放,移动光标到“1”那一列,此时点击鼠标右键“Delete”删除 冗余序列。
• PS : 是 否 位 于 密
码子的第3 位,可
以
通
过
“ Tranlated
基因工程作业(引物设计)讲解

口腔鳞癌组织中肿瘤转移相关基因1(MTA1)一、选择原因及应用口腔鳞癌组织中肿瘤转移相关基因1(MTA1)在蛋白和mRNA的表达水平,揭示其与口腔鳞癌(OSCC)发生、发展的关系。
方法采用免疫组织化学法和原位杂交技术检测46例OSCC标本、15例口腔黏膜白斑与20例正常口腔黏膜标本中MTAl 基因的表达水平,并分析其与OSCC临床病理学参数的关系。
结果MTA1蛋白和MTA1mRNA在OSCC组织中的表达水平显著高于口腔黏膜白斑和正常口腔黏膜(P 〈0.05),口腔黏膜白斑中MTA1蛋白和MTA1mRNA表达水平显著高于口腔正常黏膜(P〈0.01),MTA1蛋白和MTA1mRNA表达与肿瘤浸润深度和淋巴结转移密切相关(P〈0.05)。
结论MTA1基因在蛋白和mRNA的表达水平在OSCC发生、发展及浸润转移过程中起一定促进作用,有望成为判断OSCC预后及选择治疗方案的一个新肿瘤标志物。
二、2查阅NCBI得到MTA1相关信息并获得目的基因PREDICTED: Gorilla gorilla gorilla metastasis associated 1 (MTA1), mRNANCBI Reference Sequence: XM_004055801.1FASTA GraphicsLOCUS XM_004055801 2872 bp mRNA linear PRI 03-DEC-2012DEFINITION PREDICTED: Gorilla gorilla gorilla metastasis associated 1 (MTA1),mRNA.ACCESSION XM_004055801VERSION XM_004055801.1 GI:426378238KEYWORDS .SOURCE Gorilla gorilla gorilla (western lowland gorilla) ORGANISM Gorilla gorilla gorillaEukaryota; Metazoa; Chordata; Craniata; Vertebrata; Euteleostomi;Mammalia; Eutheria; Euarchontoglires; Primates; Haplorrhini;Catarrhini; Hominidae; Gorilla.COMMENT MODEL REFSEQ: This record is predicted by automated computationalanalysis. This record is derived from a genomic sequence(NW_004005914) annotated using gene prediction method: GNOMON,supported by mRNA and EST evidence.Also see:Documentation of NCBI's Annotation Process##Genome-Annotation-Data-START##Annotation Provider :: NCBIAnnotation Status :: Full annotationAnnotation Version :: Gorilla gorilla Annotation Release 100Annotation Pipeline :: NCBI eukaryotic genome annotation pipelineAnnotation Method :: Best-placed RefSeq; Gnomon Features Annotated :: Gene; mRNA; CDS; ncRNA##Genome-Annotation-Data-END##FEATURES Location/Qualifierssource 1..2872/organism="Gorilla gorilla gorilla"/mol_type="mRNA"/sub_species="gorilla"/db_xref="taxon:9595"/chromosome="14"gene 1..2872/gene="MTA1"/note="Derived by automated computational analysis usinggene prediction method: GNOMON. Supporting evidence/codon_start=1/product="metastasis-associated protein MTA1"/protein_id="XP_004055849.1"/db_xref="GI:426378239"/db_xref="GeneID:101134898"/translation="MAANMYRVGDYVYFENSSSNPYLIRRIEELNKTANGNVEAKVVC FYRRRDISSTLIALADKHATLSVCYKAGPGADNGEEGEIEEEMENPEMVDLPEKLKHQ LRHRELFLSRQLESLPATHIRGKCSVTLLNETESLKSYLEREDFFFYSLVYDPQQKTL LADKGEIRVGNRYQADITDLLKEGEEDGRDQSKLETKVWEAHNPLTDKQIDQFLVVAR SVGTFARALDCSSSVRQPSLHMSAAAASRDITLFHAMDTLHKNIYDISKAISALVPQG GPVLCRDEMEEWSASEANLFEEALEKYGKDFTDIQQDFLPWKSLTSIIEYYYMWKTTD RYVQQKRLKAAEAESKLKQVYIPNYNKPNPNQISVNNVKAGVVNGTGAPGQSPGAGRA CESCYTTQSYQWYSWGPPNMQCRLCASCWTYWKKYGGLKMPTRLDGERPGPNRSNMSP HGLPARSSGSPKFAMKTRQAFYLHTTKLTRIARRLCREILRPWHAARHPYLPINSAAI KAECTARLPEASQSPLVLKQAVRKPLEAVLRYLETHPRPPKPDPVKSVSSVLSSLTPA KVAPVINNGSPTILGKRSYEQHNGVDGNMKKRLLMPSRGLANHGQTRHMGPSRNLLLN GKSYPTKVRLIRGGSLPPVKRRRMNWIDAPDDVFYMATEETRKIRKLLSSSETKRAAR RPYKPIALRQSQALPLRPPPPAPVNDEPIVIED" ORIGIN1 tcctcctctt cctctcccgc ccgcgccgcg gccctcccgt ccctgcgcgg cctcggcggc61 ctcggcggcg gcggcggcgg cggcggcagc agcgcggccc ctttaaacgc ctgcggcgccgcgccgagcg ccgcgcccgcaacatgtaca gggtcggaga241 ctacgtctac tttgagaact cctccagcaa cccatacctg atccggagga tcgaggagct301 caacaagacg gccaatggga acgtggaggc caaagtggtg tgcttctacc ggaggcggga361 catctccagc accctcatcg ccctggccga caagcacgca accctgtcag tctgctataa421 ggccggaccg ggggcggaca acggcgagga aggggaaata gaagaggaaa tggagaatcc481 ggaaatggtg gacctgcccg agaaactaaa gcaccagctg cggcatcggg agctgttcct541 ctcccggcag ctggagtctc tgcccgccac gcacatcagg ggcaagtgca gcgtcaccct601 gctcaacgag accgagtcgc tcaagtccta cctggagcgg gaggatttct tcttctattc661 tctagtctac gacccacagc agaagaccct gctggcagat aaaggagaga ttcgagtagg721 aaaccggtac caggcagaca tcaccgactt gttaaaagaa ggcgaggagg atggccgaga781 ccagtccaag ttggagacca aggtgtggga ggcgcacaac ccactcacag acaagcagat841 cgaccagttc ctggtggtgg cccgctctgt gggcaccttc gcacgggccc tggactgcag901 cagctccgtc cgacagccca gcctgcacat gagcgccgca gctgcctccc gagacatcac961 gctgttccac gccatggata ctctccacaa gaacatctat gacatctcca aggccatctc1021 ggcactggtg ccgcagggcg ggcccgtgct ctgcagggac gagatggagg agtggtctgc1081 atcagaggcc aaccttttcg aggaagccct ggaaaaatat gggaaggatt tcacggacat1141 tcagcaagat tttctcccgt ggaagtcgct gaccagcatc attgagtact actacatgtg1201 gaagaccacc gacagatacg tgcagcagaa acgcttgaaa gcagctgaag ctgagagcaa1261 gttaaagcaa gtttatattc ccaactataa caagccaaat ccgaaccaaa tcagtgtcaa1321 caacgtcaag gccggtgtgg tgaatggcac gggggcgccg ggccagagcc ctggggctgg1381 ccgggcctgc gagagctgtt acaccacaca gtcttaccag tggtattctt ggggtccccc1441 taacatgcag tgtcgtctct gcgcatcttg ttggacatat tggaagaaat atggtggctt1501 gaaaatgcca acccggttag atggagagag gccaggacca aaccgcagta acatgagtcc1561 ccacggcctc ccagcccgga gcagcgggag ccccaagttt gccatgaaga ccaggcaggc1621 tttctatctg cacacgacga agctgacgcg gatcgcccgg cgcctgtgcc gtgagatcct1681 gcgcccgtgg cacgctgcgc ggcaccccta cctgcccatc aacagtgcgg ccatcaaggc1741 cgagtgcacg gcgcggctgc ccgaagcctc ccagagcccg ctggtgctga agcaggcggt1801 acgcaagccg ctggaagccg tgcttcggta tcttgagacc cacccccgtc cccccaagcc1861 tgaccccgtg aaaagcgtgt ccagcgtgct cagcagcctg acgcccgcca aggtggcccc1921 cgtcatcaac aacggctccc ccaccatcct gggcaagcgc agctacgagc agcacaacgg1981 ggtggacggc aacatgaaga agcgcctctt gatgcccagt aggggtctgg caaaccacgg2041 acagaccagg cacatgggac caagccggaa cctcctgctc aacgggaagt cctaccccac2101 caaagtgcgc ctgatccggg ggggctccct gcccccagtc aagcggcggc ggatgaactg2161 gatcgacgcc ccggatgacg tgttctacat ggccacagag gagaccagga agatccgcaa2221 gctgctctca tcctcggaaa ccaagcgtgc tgcccgccgg ccctacaagc ccatcgccctgcgcccgtca acgacgagccgccccccgcc cctcgcccgc2401 ccacacggcc ccttcccagc cagcccgccg cccgcccctc agtttggtag tgccccacct2461 cccgccctca cctgcagaga aacgcgctcc ttggcggaca ctgagggagg agaggaagaa2521 gcgcggctaa cttattccga gaatgccgag gagttgtcgt ttttagcttt gtgtttactt2581 tttggctgga gcggagatga ggggccaccc cgtgcccctg tgctgcgggg ccttttgccc2641 ggaggccggg ccctaaggtt ttgttgtgtt ctgttgaagg tgccgtttta aattttattt2701 ttattacttt ttttgtagat gaacttgagc tctgtaactt acacctggaa tgttaggatc2761 gtgcggccgc ggccggccga gctgcctggc ggggttggcc cttgtctttt caagtaattt2821 tcatattaaa caaaaacaaa gaaaaaaatc ttataaaaag gaaaaaaacc aa//三、表达载体的选择我所选用的原核表达载体为质粒pBR322;pBR322 是一种常用的E. coli 克隆载体(1),为4,361 bp 的环状双链DNA(2)。
设计引物的实验报告

一、实验目的1. 掌握引物设计的原理和方法。
2. 学习利用生物信息学工具进行引物设计。
3. 了解引物在PCR实验中的应用。
二、实验原理引物是一段单链DNA或RNA,作为PCR反应的起始模板,与模板DNA链互补结合,从而在PCR反应中引导DNA的复制。
引物设计是PCR实验成功的关键因素之一。
三、实验材料1. 生物信息学工具:Primer Premier 5.0、Primer BLAST、OligoCalc等。
2. 实验样品:待扩增的DNA模板。
3. 其他:PCR试剂、DNA序列、引物合成等。
四、实验步骤1. 选择目标基因序列根据实验目的,选择合适的基因序列。
在本实验中,以某基因的cDNA序列为模板。
2. 利用生物信息学工具进行引物设计(1)打开Primer Premier 5.0软件,输入基因序列。
(2)设置引物设计参数,如:引物长度、Tm值、GC含量、引物间距离等。
(3)进行引物设计,得到多个引物序列。
(4)利用Primer BLAST和OligoCalc等工具对设计出的引物进行筛选,排除同源序列和二级结构。
3. 引物合成将筛选出的引物序列提交给引物合成公司,合成引物。
4. PCR实验(1)配制PCR反应体系,包括:引物、模板DNA、dNTPs、DNA聚合酶等。
(2)设置PCR反应程序,如:预变性、变性、退火、延伸等。
(3)进行PCR反应,观察扩增结果。
五、实验结果与分析1. 引物设计结果根据实验目的,设计出以下引物:上游引物:5'-ATCGTACGCTAGGCTG-3'下游引物:5'-CGTCTGACGACGTCAGT-3'2. PCR扩增结果通过PCR实验,成功扩增出目标基因片段。
六、实验结论1. 通过生物信息学工具进行引物设计,可提高引物设计的准确性和效率。
2. 合适的引物是PCR实验成功的关键,设计引物时需考虑多种因素。
3. 本实验成功设计并合成引物,为后续的PCR实验奠定了基础。
微生物 通用引物设计-概述说明以及解释

微生物通用引物设计-概述说明以及解释1.引言1.1 概述概述部分的内容可以包括对微生物和引物设计的简要介绍。
以下是一个例子:引言部分会对微生物和引物设计进行概述。
微生物是一类单细胞或多细胞的微小生物,包括细菌、真菌、病毒和藻类等。
这些微生物广泛存在于地球上的各个环境中,包括土壤、水体、大气等。
微生物在生态系统中起着至关重要的作用,如参与养分循环、有机物降解、生物防治等。
此外,微生物还对人类和动植物的健康具有重要影响,既可以致病也可以为人类提供有益的功能。
引物是在分子生物学研究中常用的工具,用于扩增特定基因片段或序列。
引物的设计是进行分子生物学实验的关键步骤之一。
引物必须具有与目标序列互补的碱基序列,以便能够特异性地结合并扩增目标序列。
引物设计的好坏直接关系到实验结果的准确性和可靠性。
在本文中,我们将探讨微生物的重要性以及引物设计的意义和原则。
深入了解这些内容将有助于我们更好地理解微生物世界,加强对微生物的研究和应用。
在接下来的章节中,我们将详细讨论微生物的重要性和引物设计的意义,以及引物设计的一些基本原则。
通过阅读本文,读者将能够更好地理解微生物的重要性和引物设计的关键作用。
1.2 文章结构文章结构部分的内容如下:文章结构部分旨在介绍本篇长文的整体架构和章节划分,以及每个章节的内容安排。
本文共包括引言、正文和结论三个部分。
引言部分将在开始时对微生物通用引物设计进行概述,介绍其背景和相关概念。
接着,将详细说明文章的结构,列举各个章节的标题和内容,以让读者了解全文的组织结构。
正文部分是本篇长文的核心部分,将涵盖微生物的重要性、引物设计的意义以及引物设计的原则三个小节。
在微生物的重要性一节中,将介绍微生物在自然界和人类生活中的重要作用,以引起读者对微生物的关注。
在引物设计的意义一节中,将阐述设计合适的引物对于微生物研究的重要性,以及有效引物设计的意义。
在引物设计的原则一节中,将详细解释引物设计中需要考虑的因素和原则,并介绍一些常用的引物设计方法。
PCR引物设计及测序结果分析

Sense strand or anti-sense strand
Useful information of the primer
引物类型 搜索模式
引物搜索选项设定
引物长度
5’引物位置范 围
3’引物位置范 围
产物大小范 围
搜索结果
28对引物 每对引物的信 息 引物分值 100分为满分
双击选中一对 引物
4、基因转录水平检测
RT-PCR可检测不同组织或细胞中基因的转录水平.
5、基因突变分析:
利用PCR与一些技术的结合可以大大提高基因突 变检测的敏感性。
二. PCR引物设计原理及相关软件的使用 ( Primer Premier 5.0 )
引物 引物的重要性 引物设计的原则 引物同源性分析 引物设计软件 如何使用Primer Premier 5.0 酶切位点及保护碱基的添加
2. 引物的GC%含量:一般为40%-60%。上 下游引物的GC含量不能相差太大。
3. 引物序列在模板内应当没有相似性较高, 尤其是3’端相似性较高的序列,否则容易 导致错配。 4. 引物3’端的末位碱基避开密码子的第3位, 且最好不选择A 5. 引物的5′端可以修饰,而3′端不可修饰。
6.
引物无回文对称结构,否则会形成发夹 结构;引物自身不能配对,否则易形成约 两个引物长度的引物二聚体;
8. 引物的保守性与特异性
保守性:通用引物——检测同一类病原微生物尽可
能多的型别
特异性:避免非特异性扩增
引物同源性分析
用Blastn软件进行同源性比较
尽可能选择与非目的基因同源性小的序列作为
引物
选择3’端与非目的基因不同源的序列作为引物
引物设计方法与分析

引物设计方法与分析查找基因相关信息在NCBI上搜索到目的基因,找到该基因的mRNA,在CDS选项中,找到编码区所在位置,在下面的origin中,Copy该编码序列作为软件查询序列的候选对象。
2.4.2 用Primer Premier5搜索引物(1) 打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。
点击Primer,进入引物窗口。
(2) 此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果”选项,点击Search按钮,进入引物搜索框,选择“PCR primers”,“Pairs”,设定搜索区域和引物长度和产物长度。
在Search Parameters里面,可以设定相应参数。
一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200bp的产物电泳跑得较散,所以可以选择 300~500bp。
(3) 点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。
82(4) 对于引物的序列,可以简单查看一下,避免出现下列情况:3’不要出现连续的3个碱基相连的情况,比如GGG或CCC,否则容易引起错配。
此窗口中需要着重查看的包括:Tm应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的Tm值最好不要相差太多,大概在2度以下较好。
该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。
若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。
最理想的引物,应该都不存在这些二级结构,即这几个按钮都显示为“None”为好。
引物设计实例分析(多图)
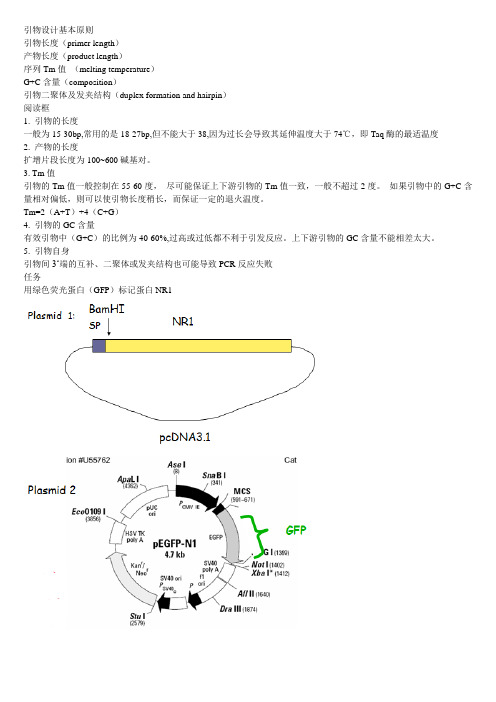
引物设计基本原则引物长度(primer length)产物长度(product length)序列Tm值(melting temperature)G+C含量(composition)引物二聚体及发夹结构(duplex formation and hairpin)阅读框1. 引物的长度一般为15-30bp,常用的是18-27bp,但不能大于38,因为过长会导致其延伸温度大于74℃,即Taq酶的最适温度2. 产物的长度扩增片段长度为100~600碱基对。
3. Tm值引物的Tm值一般控制在55-60度,尽可能保证上下游引物的Tm值一致,一般不超过2度。
如果引物中的G+C含量相对偏低,则可以使引物长度稍长,而保证一定的退火温度。
Tm=2(A+T)+4(C+G)4. 引物的GC含量有效引物中(G+C)的比例为40-60%,过高或过低都不利于引发反应。
上下游引物的GC含量不能相差太大。
5. 引物自身引物间3‘端的互补、二聚体或发夹结构也可能导致PCR反应失败任务用绿色荧光蛋白(GFP)标记蛋白NR1引物要求PCR扩增GFPGFP两边添加BamHI酶切位点保证NR1的阅读框不改变第一步:扩增GFP基本序列第二步:GC比值;Tm值第三步:酶切位点第四步:阅读框第五步保护序列Primer1: 5' GCGGggatccTATGGTGAGCAAGGGCGAGGA Primer2: 5' GCGCggatccctCTTGTACAGCTCGTCCA TGCC记得当初写本科论文,感到不知道讨论什么问题好。
愣是写了一大段的PCR条件摸索的讨论。
后来PCR成为实验最基本的一步了,但是发现在PCR中还是有许多需要注意的地方。
PCR的第一步就是引物设计了。
引物设计需要注意的地方很多,在大多数情况下,我们都是在知道已知模板序列时进行PCR扩增的。
在某些情况比如构建文库的时候也会在不知道模板序列的情况下进行设计。
这个时候随机核苷酸序列就与模板不是完全匹配。
3_引物设计及测序结果分析(2016教案)

28对引物
搜索结果
每对引物的信息
引物分值 100分为满分
双击选中一对引物
回到主窗口
引物信息
引物及产物信息
是否出现 hairpin,dimer,false priming and cross dimer
引物编辑
引物分析
• 首先检查引物二聚体尤其是3’端二聚体 形成的可能性。 • 二项检查是发夹结构(hairpin);与 二聚体相同,发夹结构的能值越低越 好。 • 第三项检查为GC 含量,以45-55%为 宜。 • 第四False priming 检查
• 发夹结构(引物自身连续互补碱基不能大于3bp ) • 发夹结构:DNA通过自身回折使得互补的碱基对相遇,也
就是引物有部分可以自身配对
– 尤其是要避免引物3’端形成发夹结构,否则将严重影响 DNA聚合酶的延伸。 – 两引物之间不应该存在互补性,尤应避免3′端的互补重 叠以防引物二聚体的形成。一般情况下,一对引物间
8.自由能分布
• ΔG值(自由能)反映了引物与模板结合的强弱 程度。一般3’端ΔG值不要超过9(ΔG值为负 值,这里取绝对值),如此则有利于正确引发 反应而可防止错误引发。3′末端双链的ΔG是 0~-2 kcal/mol时,PCR产量几乎达到百分 之百,随着其绝对值的增加产量逐渐下降,在 -6时只有40%、到-8时少于20%、而-10 时接近于0。 • 引物的3’端的∆G 值过高,容易在错配位点形 成双链结构并引发DNA 聚合反应。
7.引物的内部稳定性
• 过去认为,引物3’端应牢牢结合在模板上才能有 效地进行延伸,故3’端最好为G或C。 • 现在的观点认为,引物的5’端应是相对稳定结构, 而3’端在碱基配对的情况下最好为低稳定性结构, 即3’端尽可能选用A或T(有说不适宜用A),少 用G或C。 • 若模板不很清楚,引物3’端最后一个碱基最好为 T,其次是G或C,而不选A,国外资料表明,当 末位为T时,即使在错配的情况下也能引发链的 合成,而末位为A时,错配时引发大大降低,G或 C居中,可见,模板很清楚时选A可以提高特异性
LAMP引物设计实例
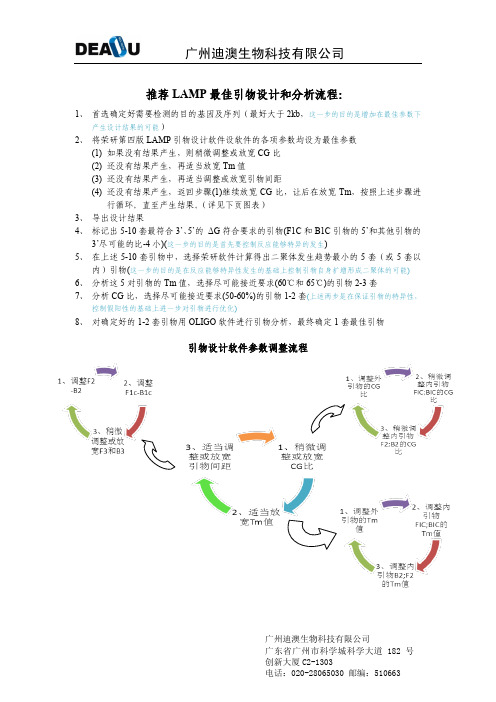
推荐LAMP最佳引物设计和分析流程:1、首选确定好需要检测的目的基因及序列(最好大于2kb,这一步的目的是增加在最佳参数下产生设计结果的可能)2、将荣研第四版LAMP引物设计软件设软件的各项参数均设为最佳参数(1)如果没有结果产生,则稍微调整或放宽CG比(2)还没有结果产生,再适当放宽Tm值(3)还没有结果产生,再适当调整或放宽引物间距(4)还没有结果产生,返回步骤(1)继续放宽CG比,让后在放宽Tm,按照上述步骤进行循环。
直至产生结果。
(详见下页图表)3、导出设计结果4、标记出5-10套最符合3’、5’的ΔG符合要求的引物(F1C和B1C引物的5’和其他引物的3’尽可能的比-4小)(这一步的目的是首先要控制反应能够特异的发生)5、在上述5-10套引物中,选择荣研软件计算得出二聚体发生趋势最小的5套(或5套以内)引物(这一步的目的是在反应能够特异性发生的基础上控制引物自身扩增形成二聚体的可能)6、分析这5对引物的Tm值,选择尽可能接近要求(60℃和65℃)的引物2-3套7、分析CG比,选择尽可能接近要求(50-60%)的引物1-2套(上述两步是在保证引物的特异性,控制假阳性的基础上进一步对引物进行优化)8、对确定好的1-2套引物用OLIGO软件进行引物分析,最终确定1套最佳引物引物设计软件参数调整流程6110第一套引物总体评价●从设计结果来看,引物3’和5’ ΔG是比较容易满足的●由于CG比例和目的序列有关,但可以通过输入较长的目的片段(>1Kb),而相对容易满足●该套引物的Tm值是根据PCR引物设计原则(不同引物之间Tm值最好能够接近)为标准从设计结果中选择的,所以和最佳Tm存在一定偏差,但是可以在设计结果选择中加以改善。
●引物二聚体和发夹结构是比较难避免的,需要通过引物设计软件primer 5和oligo进行两两匹配分析,由于需要针对3对,一共6条引物之间和引物本身进行错配分析,一套引物共需要进行21次分析,所以较为耗时,而且比较难以选择最佳结果。
过表达载体引物设计

过表达载体引物设计摘要:一、过表达载体引物设计概述1.过表达载体引物的定义2.过表达载体引物的应用背景3.过表达载体引物设计的重要性二、过表达载体引物设计方法1.基本设计原则2.引物长度与Tm 值选择3.引物序列优化4.避免引物间的互补性5.引物退火温度设定三、过表达载体引物设计工具与案例分析1.引物设计工具介绍2.设计实例及分析3.结果与讨论四、过表达载体引物设计注意事项1.防止引物二聚体形成2.避免引物与基因组DNA 结合3.考虑实验条件与需求正文:过表达载体引物设计是分子生物学实验中的一个重要环节,涉及到基因克隆、转染和表达等过程。
为了确保实验的顺利进行和结果的准确性,我们需要对过表达载体引物进行合理设计。
本文将简要介绍过表达载体引物设计的概述、方法、工具及注意事项。
一、过表达载体引物设计概述过表达载体引物,顾名思义,是用于将外源基因插入到表达载体中,实现基因过表达的引物。
过表达载体引物设计的质量直接影响到实验结果的可靠性和重复性。
在基因工程、蛋白质表达和功能研究等领域,引物的设计显得尤为重要。
二、过表达载体引物设计方法1.基本设计原则:引物设计应遵循5"→3"方向,严格遵循碱基互补配对原则。
此外,应尽量避免连续的相同碱基,以降低引物间的二聚体形成概率。
2.引物长度与Tm 值选择:引物长度通常为20-30 个碱基,Tm 值应根据实验条件进行合理选择,既要保证引物有效退火,又要避免引物间的配对。
3.引物序列优化:在满足基本原则的基础上,对引物序列进行进一步优化,如调整引物间的距离、引入GC 含量梯度等。
4.避免引物间的互补性:引物间的互补性容易导致引物二聚体形成,降低实验成功率。
因此,在设计过程中,应充分考虑引物间的相互作用。
5.引物退火温度设定:引物退火温度过高或过低都会影响实验效果。
一般而言,引物退火温度应略低于引物的Tm 值。
三、过表达载体引物设计工具与案例分析目前市面上有很多引物设计工具,如Primer Premier、Vector NTI 等。
实验2 引物设计与测序结果分析

学院:______ 班级:_______ 学号:_________ 姓名:__________ 成绩:______ 实验二引物设计及测序结果分析目的:1、掌握常规引物设计的原则及操作流程。
2、熟悉简并引物设计的原理及操作方法。
3、熟悉引物设计软件及在线引物设计工具的操作方法。
4、掌握使用相关软件及在线工具分析测序结果的方法。
内容:1、使用Primer Premier、Oligo、BLAST等软件及在线工具进行常规引物设计,并对引物扩增效率、特异性进行评价。
2、使用DNAMAN软件进行常规引物快速设计。
3、使用NCBI中的在线引物设计工具Primer-BLAST快速设计引物。
4、使用在线工具CODEHOP设计简并引物。
5、使用Chromas、BioEdit软件查阅测序结果峰图文件。
6、使用DNAMAN软件对测序序列进行编辑,进行序列拼接。
软硬件要求:联网计算机,预装Windows 7操作系统,预装IE或Chrome浏览器、英汉电子词典(有道词典或金山词霸),预装DNAMAN7、Primer Premier5、Oligo7、Chromas、BioEdit等生物信息学分析软件。
操作及问题:一、Primer Premier5、Oligo7、BLAST常规引物设计本部分操作将使用Primer Premier5、Oligo7、BLAST等软件及工具设计拟南芥AtBADH基因编码区全长特异引物。
(参考“第四章引物设计及测序结果分析”课件)(一)使用Primer Premier5搜索引物1、在NCBI数据中查找登录号为NM_001198470的序列记录,查阅相关信息,并下载序列将其保存为fasta格式文件。
问题1:该序列是什么类型的序列?该序列编码区在什么位置?2、打开Primer premier5软件,点击键ctrl+V将上一步中下载的序列粘贴入弹出的GeneTank窗口中(或者点击。
3、点击GeneTank窗口中左上角的Primer premier窗口中点击Search Criteria窗口中根据要求选择合适选项及参数,选定后,点击Search Progress窗口中有Search Results窗口;如没有出现数重新搜索引物。
生物信息学PCR引物设计

一般原则
4. 引物序列的GC 含量一般为40-60%,过高 或过低都不利于引发反应。上下游引物的 GC含量不能相差太大。 不同的算法推荐45-55%或50-60%
一般原则
5. ∆G 值是指DNA 双链形成所需的自由能, 该值反映了双链结构内部碱基对的相对稳 定性。应当选用3’端∆G 值较低(绝对值不 超过9),而5’端和中间∆G 值相对较高的 引物。引物的3’端的∆G 值过高,容易在错 配位点形成双链结构并引发DNA 聚合反应 。(能值越高越容易结合)
可以在5’端前添加G或C以提 高Tm值,或加入酶切位点, 然后再做Analyze。
决定后,将该引物选中,Ctrl+C、Ctrl+V粘贴: GCCAGTTCTGATATTTACACC
同理,编辑并保存下游引物
下游引物:AATGAAATCCAGTACGCTTC,方向默认是5’→3’
4.6 引物的二次筛选
3’ΔG的绝对值不要超过9,否则会在错配点形成双链
接下来进行引物具体分析。 点击Analyze
分析二聚体和发夹结构 点击Analyze – Duplex Formation – Forward Primer
上游引物间形成二聚体 要求ΔG小于4.5,配对碱基对不超过3
发夹结构 要求ΔG小于4.5,配对碱基对不超过3
会弹出一个页面有10秒钟的 更新提示
图形示意结果
带有genbank的链接,点击可以进入 相应的genbank序列 匹配序列描述
具体匹配情况
将四条序列保存在一个txt文档中 匹配情况,分值,e值
24
4.3 DNAMAN 同源序列比对获取保守区域
4.4 Primer Primier引物设计与筛选
三、引物设计流程
PCR引物设计实验

PCR引物设计实验PCR(聚合酶链式反应)是一种体外体温聚合酶链式反应,用于扩增DNA序列。
PCR引物是扩增特定DNA片段所需的短DNA序列,它们在PCR反应中与模板DNA序列特异性结合,并在DNA复制过程中提供扩增起始点。
因此,PCR引物设计的优劣直接影响PCR扩增的特异性和效率。
1.目标DNA序列选择和分析:首先,需要选择并分析目标DNA序列。
这可以通过参考已知序列数据库或使用DNA测序实验获得。
2.引物长度和理化性质选择:PCR引物的长度通常在18-30个碱基对之间,最好是20-25个碱基对。
引物长度的选择应考虑到特异性和扩增效率等因素。
此外,引物的理化性质也需要考虑,如GC含量、熔解温度和互补性等。
3.引物设计原则:引物一般分为前导引物和反导引物。
其设计应符合一定的原则,如:-引物长度相似:前导引物和反导引物的长度应相似,以提高扩增的特异性和效率。
-避免或最小化引物自身或引物间的互补性:互补性会导致非特异性扩增或导致自身产生二聚体。
-避免引物末端的非特异性:尽量避免或减少末端碱基对的非特异性,以提高特异性和扩增效率。
-避免引物末端的重复序列:重复序列容易导致非特异性扩增和有害的寡聚物形成。
4.引物序列分析和验证:设计好的引物序列需要进行一系列的分析和验证。
包括序列比对和互补性分析,以确定引物与目标DNA的特异性。
此外,还可以使用特定的软件工具进行引物性能和二聚体预测等分析。
5.引物合成和质量控制:设计好的引物需要通过化学合成获得。
合成后,需要进行质量控制以确保引物的纯度和质量。
6.引物应用实验:设计好和验证过的引物可用于PCR实验。
在PCR反应中,需要优化引物浓度、引物与模板DNA的比例、反应条件等因素,以获得最佳的PCR扩增效果。
总之,PCR引物设计是PCR实验的重要一步。
良好设计的引物具有特异性和高效性,可以提高PCR扩增的成功率和特异性。
因此,在设计PCR 引物时,需要考虑引物长度、互补性、特异性和理化性质等因素,并结合实验验证进行优化。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
• 为设计一条高效引物有几个参数必须考虑PRIMER PREMIER搜 索所考虑的参数依次如下 • Tm 融链温度:PRIMER PREMIER根据相邻二碱基对作用理论 nearest neighbor theory 来计算融链温度。PCR反应的合 适Tm范围为56到63°C。 • GC% GC含量:对于PCR反应来说GC含量在50%左右比较合适而 对于测序引物和杂交探针来说GC含量至少应为50%。 • Degeneracy 多义性:当设计多义引物时应尽量减少引物多 义性这样会带来更好的特异性应尽量避免3末端的多义性因 为这里即使一个碱基的错配都能阻止引物延伸。 • 3’ End Stability 3 末端稳定性:引物稳定性影响它的错配 效率。一条理想的引物应该有一个稳定性较强的5 末端和相 对稳定性较弱的3 末端,如果引物3 稳定性强有可能在即使 5 末端不配对的情况下造成错配形成非特异性扩secondary bands。 而3 末端稳定性低的引物较好的原因是在引物发生 错配时由于3 末端不太稳定引物结合不稳定而难以延伸。
引物设计实例分析
引物设计基本原则
• • • • • 引物长度(primer length) 产物长度(product length) 序列Tm值 (melting temperature) G+C含量(composition) 引物二聚体及发夹结构 (duplex formation and hairpin) • 阅读框
PRIMER PREMIER能即时分析引物二级结构。在左下的两排按钮可以 显示发现了何种二级结构,也显示二级结构的自由能数值 G 单位 kcal/mol。
• Edit Primer 引物编辑窗口可以用来设计 用于定点突变的引物或分析一条已有引物 序列。可以使用CTRL-X CTRL-C和CTRL-V快 捷键来实施剪切拷贝和粘贴删除当前引物 序列,并从剪贴板上粘贴是分析一条已有引 物的好方法。进行粘贴时Paste 粘贴窗口 会激活,用以将引物序列转化为反向互补 或反向互补形式,也可以手工键入引物序 列。一旦引物被编辑发点击Analyze 按钮 即可分析编辑后的引物。
第二步:利用Primer Premier 5设计引物
Primer1: 5’ Primer2: 5’
ATGGCATCCAAAAGAG
………….. …….
TCTAGTCTTTGAGAACA
• Primer Premier5.0是由加拿大的Premier公司开发的专业用于PCR或 测序引物以及杂交探针的设计、评估的软件,其主要界面分为序列编 辑窗口(Genetank),引物设计窗口(Primer Design),酶切分析 窗口(Restriction Sites)和纹基分析窗口(Motif),这里我们主 要介绍其引物设计功能 。
• Pair Rating 匹配度评分
• 匹配度低的引物对常常不太有效,是因为在 同样退火温度下,Tm低的引物决定扩增的 特异性,而Tm高的引物更易于形成非特异 性结合而造成错误的起始。PRIMER PREMIER会计算每一对引物的匹配度100分 为满分并按照分数按降序排列计。
4. 引物的GC含量
有效引物中(G+C)的比 例为40-60%,过高或过低都 不利于引发反应。上下游引 物的GC含量不能相差太大。
5.碱基础随机分布
• 引物中四种碱基的分布最好是随机的, 不要有聚嘌呤或聚嘧啶的存在。尤其3′ 端不应超过3个连续的G或C,因这样会 使引物在G+C富集序列区错误引发。
LOCUS NM_057143 1097 bp mRNA linear ROD 1 gtgccgagca cagttactgg aaggcttaac caaagttttg atgcctggga accgcgcagg 61 aaaaacacgc ggaacgtgct tcacagtggc ggctaactgc tctcgttcgc tgtgcagagc 121 cgtctggcag ggttgacctc ctaaagggat attccatctt tattaatcat tagtagtgtg 181 gtcagagact tagcaccatt ggtctccccc aacctggtcc agacatttca gcagtttatc 241 ggaacagcaa caacagcaac aaaaccttca aaatttacaa gtctttaaga aatagaaatg 301 gcatccaaaa gagctctggt catcctagcc aaaggagcag aggagatgga gacagtgatt 361 cctgtggaca tcatgcggcg agctgggatt aaagtcaccg ttgcaggctt ggctgggaag 421 gaccccgtgc agtgtagccg tgatgtagtg atttgtccgg ataccagtct ggaagaagca 481 aaaacacagg gaccatacga tgtggttgtt cttccaggag gaaatctggg tgcacagaac 541 ttatctgagt cggctttggt gaaggagatc ctcaaggagc aggagaacag gaagggcctc 601 atagctgcca tctgtgcggg tcctacggcc ctgctggctc acgaagtagg ctttggatgc 661 aaggttacat cgcacccatt ggctaaggac aaaatgatga acggcagtca ctacagctac 721 tcagagagcc gtgtggagaa ggacggcctc atcctcacca gccgtgggcc tgggaccagc 781 ttcgagtttg cgctggccat tgtggaggca ctcagtggca aggacatggc taaccaagtg 841 aaggccccgc ttgttctcaa agactagaga gcccaagccc tggaccctgg acccccaggc 901 tgagcaggca ttggaagccc actagtgtgt ccacagccca gtgaacctgg cattggaagc 961 ccactagtgt gtccacagcc cagtgaacct caggaactaa cgtgtgaagt agcccgctgc 1021 tcaggaatct cgccctggct ctgtactatt ctgagccttg ctagtagaat aaacagttcc 1081 ccaagctcct gacggct
• False Priming 错配
• 如果引物可以结合除目的位点外的其他区域 扩增效率将明显降低,目的产物带将减少或 出现涂布(smear ),PRIMER PREMIER 会检查每一条引物是否会与整个序列的其他 位点形成局部的错配,3 末端连续几个碱基 配对形成错配的倾向要高于引物上游区域同 样数量的碱基配对。可以分别设定确认为错 配的3 末端或引物全长形成连续碱基配对的 数量。
• GC Clamp GC钳:引物与目的位点的有效结合需 要有稳定的5 末端,这一段有较强稳定性的5 末 端称为GC钳。它保证引物与模板的稳定结合。
• Secondary Structures 二级结构
• 二级结构是引物设计中必须考虑的一个重要因素 二级结构能显著影响反应中能与模板正确结合的 引物数量。发卡结构的存在能限制引物与目的位 点的结合能力,从而降低扩增效率。形成发卡环 的引物则不能在PCR扩增中发挥作用。
1. 引物的长度
一般为15-30bp,常用的是1827bp,但不能大于38,因为过长 会导致其延伸温度大于74℃,即 Taq酶(Taq酶是从水生栖热菌 Thermus Aquaticus ( Taq )中
分离出的具有热稳定性的DNA 聚合酶。 对于PCR的应用有里程碑的意 义,该酶可以耐受90℃以上的高温而不失活,不需要每个循环加酶, 使PCR技术变得非常简捷、同时也大大降低了成本,PCR技术得以大量
6. 引物自身
引物间3’端的互补、二聚体或发 夹结构也可能导致PCR反应失败
引物自身不应存在互补序列,否则引物自 身会折叠成发夹状结构牙引物本身复性。 这种二级结构会因空间位阻而影响引物与 模板的复性结合。若用人工判断,引物自 身连续互补碱基不能大于3bp。
7. 引物之间
两引物之间不应不互补性,尤应避免3′ 端的互补重叠以防引物二聚体的形成。 的是序列编辑参看
这是根据模板序列寻找引物的界面,在该界面中可以设定所要搜索的引 物的类型,包括PCR引物,测序引物和杂交探针以及引物所在的链;另 外也能设定搜索引物的范围,以及最终PCR产物的长度和引物的长度等。
Automatic Search 自动搜索
在结果窗口中给出了程序给该对引物的打分(rating)和上下游引物的起 始位置和长度以及产物的长度。通过直接点击各对引物在相应引物搜寻 界面中相应的显示引物的各种信息。包括其各种参数和各种可能存在的 不利结构。
• Hairpin 发卡结构 • 发卡结构的形成是由于引物自身的互补碱 基分子内配对造成,引物折叠形成的二级结 构,并由于发卡结构的形成是分子内的反 应,仅仅需要三个连续碱基配对.发卡结构 的稳定性可以用自由能衡量,自由能大小取 决于碱基配对释放的能量以及折叠DNA形成 发卡环所需要的能量,如果自由能值大于0 则该结构不稳定,从而不会干扰反应.如果 自由能值小于0 则该结构可以干扰反应。 按下按钮All 可以显示其具体结构。
• Dimer 二聚体
• 引物之间的配对区域能形成引物二聚体,它是相同 或不同的两条引物之间形成的二级结构。它造成引 物二聚体扩增并减少目的扩增,产物二聚体可以在 序列相同的两条引物或正反向引物之间形成,如果 配对区域在3 末端问题会更为严重,3 末端配对很 容易引起引物二聚体扩增使用Dimer Report功能 可以预测二聚体的形成。 • 引物二聚体及发夹结构的能量一般不要超过4.5 , 二聚体和发夹结构形成的能值越高越稳定,越不符 合要求。
