分子对接
第十章分子对接

配体和受体分子之间的识别要比“锁和钥匙”模型复杂的多: 1.配体和受体分子的构象是变化的,而不是刚性的,配体和
受体在对接过程中互相适应对方,从而达到更完美的匹配; 2.其次,分子对接不但要满足空间形状的匹配,还要满足能
量的匹配。
配体和受体之间的通过底物分子与靶酶分子能否结合以及结合的强度最终是由形 成此复合物过程的结合自由能变化ΔGbind所决定的。
分子对接的目的:找到底物分子和受体分子的 最佳结合位置
找到最佳的结合位置
评价对接分子之间的结合强度
构象搜索方法
➢ 按照分子对接程序寻找配体与受体结合构 象方法的差异,可以分为三种:
1、局部优化法 2、深度搜寻法 3、全局优化法
➢ 局部优化法:不对配体和受体进行构象搜寻, 只是对初始构象进行优化,得到配体与受体结 合的一个局部最优构象;
➢ 通过分子对接确定复合物中两个分子正确的相对 位置和取向,研究两个分子的构象,特别是底物构象 在形成复合物过程中的变化,是确定酶激活剂、抑制 剂作用机制以及药物作用机制,设计新药的基础。
大肠杆菌乙酰辅酶A羧化酶
肉桂醛
➢ 在药物设计中,分子对接方法主要用来从小分子数据库中 搜寻与受体生物大分子有较好亲和力的小分子,进行药理 测试,从中发现新的先导化合物。
第十章 药物设计的基本方法 ——分子对接
内容
分子对接简介 分子对接法的基本原理
分子对接软件 Dock AutoDock
CADD基于结构方法分类
10.1 引言
分子对接就是两个或多个分子之间通过 几何匹配和能量匹配而相互识别的过程。
分子对接在酶学研究以及药物设计中具有 十分重要的意义。
➢ 在酶激活剂、酶抑制剂与酶相互作用以及药物分 子产生药理反应的过程中,小分子(通常意义上的 Ligand)与靶酶(通常意义上的Receptor)相互互结合, 首先就需要两个分子充分接近,采取合适的取向,使 两者在必要的部位相互契合,发生相互作用,继而通 过适当的构象调整,得到一个稳定的复合物构象。
分子对接全

蛋白质二级结构的主要形式
• -螺旋 ( -helix ) • -折叠 ( -pleated sheet ) • -转角 ( -turn ) • 无规卷曲 ( random coil )
-螺旋
-折叠
-转角和无规卷曲
-转角
无规卷曲是用来阐述没有确定规律性的那部 分肽链结构。
❖ 定量指标,需要结合分子动力学进一步评价
AutoGrid 格点中相关能量的计算
AutoDock 构象搜索及评价
❖ 免费软件/
AutoDock分子对接的流程:
1.围绕受体活性位点的氨基酸残基形成一个盒 子(box),并划分成格点; 2.用配体不同类型的原子作为探针(probe)进 行扫描,计算格点能量; 3.对配体在box范围内进行构象搜索; 4.根据配体的不同构象、方向、位置及能量进 行评分,最后对结果进行排序。
蛋白质分子中各亚基的空间排布及亚基接 触部位的布局和相互作用,称为蛋白质的四级 结构。
亚基之间的结合力主要是疏水作用,其次 是氢键和 结 构
从一级结构到四级结构
血红蛋白
二、酶及其抑制剂
酶是由活细胞产生的对其特异的底物 起高效催化作用的蛋白质。
酶的分子组成
❖ 单纯酶(simple enzyme):仅由氨基酸残基构成 ❖ 结合酶(conjugated enzyme)
苏氨酸 threonine Thr T 5.60
3. 酸性氨基酸 4. 碱性氨基酸
天冬氨酸 aspartic acid Asp D 2.97 谷氨酸 glutamic acid Glu E 3.22
赖氨酸
lysine
Lys K 9.74
精氨酸 arginine Arg R 10.76
分子对接
分子对接的基本原理
药物与受体的结合强度取决于结合的自由能变化
∆G结合 = ∆H结合 - T∆S结合 = -RT ln Ki
大部分的分子对接法忽略了全部的熵效应,而在焓 效应也只考虑配体与受体的相互作用能,即:
Einteraction= Evdw + Eelectrostatic + Eh-bond
(2)基于几何哈希技术“geometric hashing”的方法
第一部分中,几何哈希表从被对接的一个配体或一系 列配体中构建 。哈希矩阵含有配体名字和能调整配体在空 间方向的参考框架。
第二部分即识别阶段,蛋白质的特征用来识别哈希矩阵, 每一次匹配表示蛋白质的特征与哈希矩阵中已定义好方位的 配体相匹配,具有大量匹配信息的哈希矩阵代表着具有几个 吻合特征的配体和方位
分子对接的基本方法
(一) 刚性的分子对接方法
这种方法是最初的分子对接的方法,在对接中,小 分子和蛋白质两种都保持刚性。
(1)基于最大团搜索的方法 (Clique-Search Based Approaches)
对接两个刚性分子可以理解为分子在空间的匹配问 题,这种匹配可以是一种形状上的互补或相互作用。 如氢键受体与氢键给体的互补。搜索在三维空间中有 效的条件下的最大匹配
(3) 遗传算法和进化规划
遗传算法开始应用到分子对接技术,其特点为 :
第一步,一个称为染色体的线性表示符能够描述构型的所有 自由度,找到这个染色体描述符是算法中最困难的一步。第 二步,确定一个一个类似如打分函数的目标函数。
著名的GOLD软件包括了这种算法
(4)基于分子模拟的方法
模拟退火的方法,Autodock程序就采用了这种方法
(二)柔性对接的方法
分子对接的原理,方法及应用
三个氢键受体的作用表面
Pose clustering 算法中的作用点
17
(二)柔性对接的方法 (1)构象的系综方法
Flexibase用来储存小分子库中每个分子的一系列 不同构象,用距离几何和能量最小化的方法产生构象, 每个分子根据rmsd的差异选择25个系列构象。每个 构象采用FLOG刚性对接的方法进行对接。
性键的二面角,寻找小分子化合物与靶标大分子作 用的最佳构象,计算其相互作用及结合能 在库中所有分子均完成了对接计算之后,即可从中 找出与靶标分子结合的最佳分子
9
10
4 分子对接的分类
1、刚性对接:对接过程中,研究体系的构象不发生变化。 适合考察比较大的体系,如蛋白质和蛋白质间以及蛋白质 与核酸间的对接。
14
受体的活性位点
配体
有效匹配的距离图集
受体-配体的示意图,字母代表特征部分如氢键等, 相应的有效匹配的图集如右,三个环性顶点组织的三角形 为这个图集的一个最大团(clique)
15
(2)基于几何哈希技术“geometric hashing”的方 法
第一部分中,几何哈希表从被对接的一个配体或一 系列配体中构建 。哈希矩阵含有配体名字和能调整 配体在空间方向的参考框架。
9.涂瑶生,孙冬梅,陈玉兴,等.中药新药筛选新技术及实践.世界科
学技术一中医药现代化,2014,16(8):1696—1702.
10.白晓光,许乐幸,李神亮,等.基于靶蛋白结构的CDK2小分子抑制
剂研究进展.中国新药杂志,2011,20(17):1667—1672. .................
——涉及到底物分子和受体分子间结合能力的预测, 牵涉到结合自由能的计算。
23
5 代表性对接软件
分子对接简要介绍
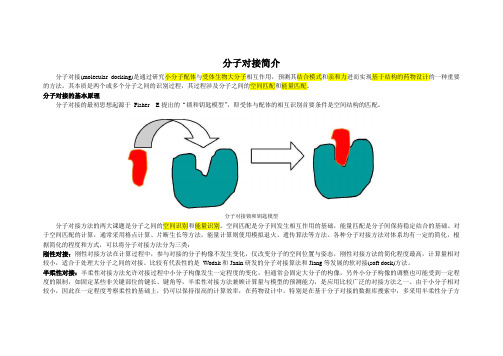
分子对接简介分子对接(molecular docking)是通过研究小分子配体与受体生物大分子相互作用,预测其结合模式和亲和力进而实现基于结构的药物设计的一种重要的方法。
其本质是两个或多个分子之间的识别过程,其过程涉及分子之间的空间匹配和能量匹配。
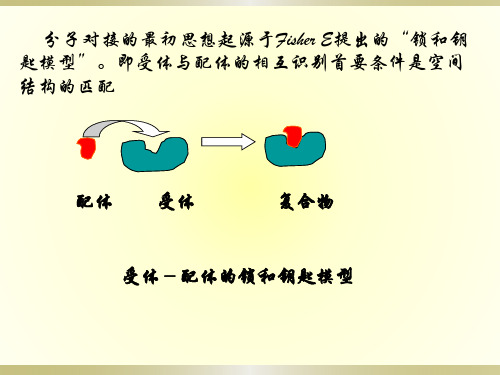
分子对接的基本原理分子对接的最初思想起源于Fisher E提出的“锁和钥匙模型”,即受体与配体的相互识别首要条件是空间结构的匹配。
分子对接锁和钥匙模型分子对接方法的两大课题是分子之间的空间识别和能量识别。
空间匹配是分子间发生相互作用的基础,能量匹配是分子间保持稳定结合的基础。
对于空间匹配的计算,通常采用格点计算、片断生长等方法,能量计算则使用模拟退火、遗传算法等方法。
各种分子对接方法对体系均有一定的简化,根据简化的程度和方式,可以将分子对接方法分为三类:刚性对接:刚性对接方法在计算过程中,参与对接的分子构像不发生变化,仅改变分子的空间位置与姿态,刚性对接方法的简化程度最高,计算量相对较小,适合于处理大分子之间的对接。
比较有代表性的是Wodak和Janin研发的分子对接算法和Jiang等发展的软对接(soft dock)方法。
半柔性对接:半柔性对接方法允许对接过程中小分子构像发生一定程度的变化,但通常会固定大分子的构像,另外小分子构像的调整也可能受到一定程度的限制,如固定某些非关键部位的键长、键角等,半柔性对接方法兼顾计算量与模型的预测能力,是应用比较广泛的对接方法之一。
由于小分子相对较小,因此在一定程度考察柔性的基础上,仍可以保持很高的计算效率,在药物设计中,特别是在基于分子对接的数据库搜索中,多采用半柔性分子方法。
其代表性软件是DOCK和AutoDock。
柔性对接:柔性对接方法在对接过程中允许研究体系的构像发生自由变化,由于变量随着体系的原子数呈几何级数增长,因此柔性对接方法的计算量非常大,消耗计算机时很多,适合精确考察分子间识别情况。
其中比较有代表性的方法有Accelrys 公司发展的基于分子力学和分子动力学的分子对接方法及Affinity 软件。
分子对接

分子对接(molecular docking)使依据配体与受体作用的“锁-钥原理”(lock and key principle),模拟小分子配体与受体生物大分子相互作用。
配体与受体相互作用是分子识别的过程,主要包括静电作用、氢键作用、疏水作用、范德华作用等。
通过计算,可以预测两者间的结合模式和亲和力,从而进行药物的虚拟筛选。
分子对接首先产生一个填充受体分子表面的口袋或凹槽的球集,然后生成一系列假定的结合位点。
依据受体表面的这些结合点与配体分子的距离匹配原则,将配体分子投映到受体分子表面,来计算其结合的模式和亲和力,并对计算结果进行打分,评判配体与受体的结合程度。
2 分子对接的原理2. 1 分子对接的一般原理分子对接是将已知三维结构数据库中的分子逐一放在靶标分子的活性位点处。
通过不断优化受体化合物的位置、构象、分子内部可旋转键的二面角和受体的氨基酸残基侧链和骨架, 寻找受体小分子化合物与靶标大分子作用的最佳构象, 并预测其结合模式、亲和力和通过打分函数挑选出接近天然构象的与受体亲和力最佳的配体的一种理论模拟分子间作用的方法。
2. 2 分子对接的互补性影响复合物分子稳定性的主要因素是疏水作用和键合力大小, Audie 等[ 2] 建立了一种新的经验自由能公式, 该方法能够预测蛋白质2蛋白质键合力大小, 准确率达到89% 。
而影响键合力的因素有作用位点空间位的互补、静电相互作用和氢键等, 且溶解熵对稳定受体2配体复合物起着重要的作用[ 3] , 所以分子对接的过程主要包括分子间的空间互补和电学性质互补。
空间互补是分子间发生相互作用的基础, 能量互补是分子间保持稳定结合的基础。
3 分子对接的种类分子对接的种类主要包括:(1)刚体对接:指在对接过程中,研究体系(受体和配体)的构象不发生变化。
适合考察比较大的体系,如蛋白质和蛋白质间以及蛋白质和核酸之间的对接。
(2)半柔性对接:指在对接过程中,研究体系尤其是配体的构象允许在一定的范围内变化。
分子对接
大分子准备
黄嘌呤氧化酶蛋白质晶体结构下载于 RCBS蛋白质数据库,PDB代码为 3NVY。 将3NVY晶体中的水分子保留,加上氢 原子后,再采用Tripos力场分子进行 结构优化。
分子对接
选用SYBYL-X1.3软件中的surflexDock程式。 采用柔性分子对接, 所有的参数都是Surflex-Dock的设定 值如探针分子户限为0.5,探针分子 膨胀为0.0。
结果与讨论
虎杖的阿斯特酚苷是 本研究中打分函数 (12.77)得第二高的 化合物 阿斯特酚苷的冲击效 应较低,能较容易进入 XO的活性部位中。 结构有7个羟基、2个 氧分子,有较强的极性, 能与XO产生多个相互 作用
结果与讨论
另一化合物虎杖甙亦是虎杖的成分, 打分函数(11.51) 是在本研究的63种 中草药成分中排第 三位。
分子对接的重要原则
互补性:包括空间结构的互补性和电学性 质的互补性。 ——决定识别过程的选择性 预组织性:受体和底物分子在识别之前将 受体中容纳底物的环境组织的愈好,其溶 剂化能力就越低,则它们的识别效果愈佳, 形成的复合物越稳定。 ——决定识别过程的结合能力
分子对接的种类
刚体对接:指在对接过程中,研究体 系的构象不发生变化。 半柔性对接:指在对接过程中,研究 体系尤其是配体的构象允许在一定的 范围内变化。 柔性对接:指在对接过程中,研究体 系的构象基本上可以自由变化的。
结论
花旗松素-3-O-a-L-鼠李糖苷、阿斯 特酚苷和虎杖甙有最好的潜在药物特 性,可做相关的生物活体和临床研究 以了解其确切功效,有机会成为治疗 高尿酸血症和痛风的药物。
谢谢!
实验过程
小分子准备 大分子准备 分子对接 预测ADMET
小分子准备
选取5种被证实能有效抑制黄嘌呤氧 化酶的中草药中的65个活性成份进 行研究,这5种草药是肉桂、野菊花、 花曲柳、土茯苓和虎杖。 65个已知成份的分子结构于台湾中 医药资料库下载,采用chemBioOffice 2008绘制并利用MM2力场分子力学 进行结构优化以确保结构的稳定性
分子对接

2.分子对接的原理
• 2.1 分子对接的一般原理
• 分子对接是将已知三维结构数据库中的分子逐一 放在靶标分子的活性位点处。通过不断优化受体 化合物的位置、构象、分子内部可旋转键的二面 角和受体的氨基酸残基侧链和骨架,寻找受体小分 子化合物与靶标大分子作用的最佳构象,并预测其 结合模式、亲和力和通过打分函数挑选出接近天 然构象的与受体亲和力最佳的配体的一种理论模 拟分子间作用的物分子稳定性的主要因素是疏水作用和 键合力大小。 • 而影响键合力的因素有作用位点空间位的互补、 静电相互作用和氢键等,且溶解熵对稳定受体-配体 复合物起着重要的作用。 • 所以分子对接的过程主要包括分子间的空间互补 和电学性质互补。 • 空间互补是分子间发生相互作用的基础,能量互补 是分子间保持稳定结合的基础。
• 3.3柔性对接
• 在对接过程中,配体和受体的构象是允许发生变化 的。一般用于精确考虑分子间的识别情况。 • 分子的柔性主要来自于可旋转健的旋转。这种变 化包括三个平动自由度、三个转动自由度以及配 体分子的部分二面角的变化。
4.主要分子对接软件
• 如果要进行分子对接试验,就必须先对所要对接的 分子进行结构模拟。当分子模型确立后,就可以利 用分子模拟软件在计算机上进行一系列实验。主 要软件有:
6.展望
• 分子对接方法的优势在于各种化合物数据库中的 分子均是已知化合物,且相当大一部份可以通过购 买得到或根据已知的合成路线合成,可以较快地进 行后续的药理测试。目前有许多的商用数据库,为 药物开发提供了一个比较好的工具。近年来,计算 机技术的发展、靶酶晶体结构数据和算法数量的 快速增长和商用小分子数据库的不断更新,使得分 子对接在药物设计中取得了巨大成功。
Stoddard等用二元对接方法成功地对麦芽糖和蛋白 质进行了对接。对接时,他们把配体和受体主链结 构当作刚性处理。
第十章分子对接

a. 锚优先搜寻
1) 对接锚片段 2) 加一个刚性片段,先内后外 先大后小 3)构象搜寻,得到NcNt个构象 4)根据构象能量差异按照一定 标准,选取Nc个构象 5)回到第2步,不断加锚,直到 所有的锚片段都对接过,跳过 5,得到对接构象。
b. 同时搜寻 先进行构象搜寻,然后将搜寻得到的构 象分别对接到受体的结合位点中。 根据参数设置不同,可以进行系统搜寻 或随机搜寻。
第十章 药物设计的基本方法 ——分子对接
内容
分子对接简介
分子对接法的基本原理
分子对接软件
Dock
AutoDock
CADD基于结构方法分类
10.1 引言
分子对接就是两个或多个分子之间通过 几何匹配和能量匹配而相互识别的过程。 分子对接在酶学研究以及药物设计中具有 十分重要的意义。
在酶激活剂、酶抑制剂与酶相互作用以及药物分 子产生药理反应的过程中,小分子(通常意义上的 Ligand)与靶酶(通常意义上的Receptor)相互互结合, 首先就需要两个分子充分接近,采取合适的取向,使 两者在必要的部位相互契合,发生相互作用,继而通 过适当的构象调整,得到一个稳定的复合物构象。 通过分子对接确定复合物中两个分子正确的相对 位置和取向,研究两个分子的构象,特别是底物构象 在形成复合物过程中的变化,是确定酶激活剂、抑制 剂作用机制以及药物作用机制,设计新药的基础。
分子对接方法根据不同的简化程度可以大致分 为以下三类: (1)刚性对接;研究体系的构象不发生变化 (2)半柔性对接;研究体系尤其是配体的构象 允许在一定的范围内变化 (3)柔性对接;研究体系的构象基本上是可以 自由变化的
刚性对接适合考察比较大的体系,比如蛋白质和蛋白质以 及蛋白质和核酸之间的相互作用,他计算较为简单,原理 也相对简单,主要是考虑构象之间的契合程度。 半柔性对接适合于处理小分子和大分子之间的对接。在对 接过程中,小分子的构象一般是可以变化的,但大分子则 是刚性的。由于小分子相对较小,因此在一定程度考察柔 性的基础上,还可以保持较高的计算效率,在药物设计尤 其在基于分子数据库的虚拟筛选过程中,一般采用半柔性 的分子对接方法。 柔性对接方法一般用于精确考察分子之间的识别情况,由 于在计算过程中体系的构象是可以变化的,因此柔性对接 在提高了对接准确性的同时却需要耗费较长的计算时间。
分子对接互作

分子对接互作分子对接互作是指两个分子之间的相互作用,通过相互结合形成稳定的复合物。
在生物学和药物研发领域,分子对接互作被广泛应用于药物设计、分子识别和酶催化等方面。
本文将从分子对接的原理、应用和发展前景等方面进行探讨。
一、分子对接的原理分子对接是基于分子间相互作用的原理进行的。
在分子对接过程中,通常有一个受体分子和一个配体分子,受体分子通常是蛋白质或其他生物大分子,而配体分子则是潜在的药物分子。
分子对接的目标是通过计算方法预测受体和配体之间的相互作用,以确定最佳的结合模式和结合能力。
分子对接的过程主要包括以下几个步骤:分子的几何构建、柔性对接、评分函数计算和结合模式优化。
在分子对接的过程中,需要考虑分子间的相互作用力,如静电作用、范德华力、氢键等。
通过计算和模拟,可以预测受体和配体之间的相互作用,从而为药物设计和分子识别提供理论依据。
二、分子对接的应用分子对接在药物研发中起着重要的作用。
通过分子对接技术,可以快速筛选出具有潜在药物活性的化合物,从而加速新药的研发过程。
同时,分子对接还可以帮助研究人员理解药物与受体之间的相互作用机制,为药物设计提供指导。
除了药物研发,分子对接在农药设计、材料科学和环境科学等领域也有广泛应用。
例如,在农药设计中,分子对接可以帮助研究人员预测化合物与靶标蛋白之间的相互作用,从而设计出具有高效杀虫活性的农药。
在材料科学中,分子对接可以帮助研究人员预测材料的性能和稳定性,为新材料的设计提供指导。
三、分子对接的发展前景随着计算机技术和生物信息学的不断发展,分子对接技术也在不断完善和发展。
目前,分子对接技术已经成为药物研发过程中不可或缺的一部分,能够为研究人员提供重要的信息和指导。
未来,随着计算机技术的进一步发展,分子对接技术将更加精确和高效。
分子对接技术还可以与其他技术相结合,如机器学习、人工智能等,从而提高预测的准确性和效率。
通过结合不同的方法和技术,可以更好地模拟和预测分子间的相互作用,为药物研发和分子识别提供更好的解决方案。
分子对接

网址:/s/blog_602a741d0100lw4g.html分子对接分类:AUTODOCK标签:杂谈一:概述分子对接是指两个或多个分子通过几何匹配和能量匹配相互识别的过程,在药物设计中有十分重要的意义。
药物分子在产生药效的过程中,需要与靶酶相互结合,这就要求两个分子要充分接近并采取合适的取向以使二者在必要的部位相互契合,发生相互作用,继而通过适当的构象调整,得到一个稳定的复合物构象。
通过分子对接确定复合物中两个分子正确的相对位置和取向,研究两个分子的构象特别是底物构象在形成复合物过程的变化是确定药物作用机制,设计新药的基础。
分子对接计算把配体分子放在受体活性位点的位置,然后按照几何互补、能量互补以及化学环境互补的原则来评价药物和受体相互作用的好坏,并找出两个分子之间最佳的结合模式。
由于分子对接考虑了受体结构的信息以及受体和药物分子之间的相互作用信息,因此从原理上讲,它比仅仅从配体结构出发的药物设计方法更加合理。
同时,分子对接筛选的化合物库往往采用的是商用数据库,比如可用化合物数据库(ACD)、剑桥晶体结构数据库(CSD)、世界药物索引(WDL)、药用化合物数据库(CMC)以及可用化合物搜索数据库(ACDSC)等等,因此筛选出来的化合物都为已知化合物,而且相当大一部份可以通过购买得到,这为科研提供了很大的方便,近年来,随着计算机技术的发展、靶酶晶体结构的快速增长以及商用小分子数据库的不断更新,分子对接在药物设计中取得了巨大成功,已经成为基于结构药物分子设计中最为重要的方法。
分子对接的最初思想源自于“锁和钥匙”的模型,即“一把钥匙开一把锁”。
不过分子对接,也就是药物分子和靶酶分子间的识别要比“钥匙和锁”的模型要复杂的多,首先表现在药物分子和靶酶分子是柔性的,这样就要求在对接过程中要相互适应以达到最佳匹配;再者,分子对接不仅要满足空间形状的匹配,还要满足能量的匹配,底物分子与靶酶分子能否结合以及结合的强度最终是由形成此复合物过程的结合自由能的变化值决定。
分子对接指南

分子对接指南分子对接是指两个或多个分子(通常是蛋白质和核酸)之间通过某些相互作用力(如氢键、静电相互作用、范德华力等)结合形成复合物的过程。
在药物设计、生物物理学、生物化学等领域中,分子对接是一种重要的研究方法。
下面是分子对接的一些基本步骤:1. 准备分子模型:确定参与对接的分子,并构建其三维模型。
2. 选择对接算法:常用的对接算法包括刚性对接和柔性对接。
3. 生成构象:为目标分子生成大量的可能构象。
4. 计算相互作用能:计算目标分子与受体分子之间的相互作用能,以评估其结合能力。
5. 筛选最佳构象:根据相互作用能等因素筛选出最佳的对接构象。
6. 分析结果:对最佳构象进行分析,以了解其结合模式和作用机制。
在进行分子对接时,需要注意以下几点:1. 选择合适的对接算法和参数:根据具体情况选择合适的对接算法和参数,以获得准确的对接结果。
2. 考虑分子的柔性:分子通常具有一定的柔性,因此在对接时需要考虑分子的柔性。
3. 处理大分子体系:对于较大的分子体系,需要使用合适的算法和硬件设备来加速计算。
4. 分析对接结果的可靠性:对接结果需要经过仔细的分析和验证,以确保其可靠性。
分子对接是一项复杂的任务,需要综合运用多种技术和方法。
分子对接的结果可以用于多种用途,以下是一些常见的应用:1. 药物设计:分子对接可以帮助研究人员发现新的药物靶点,并设计出能够与靶点结合的候选药物分子。
2. 生物分子相互作用研究:分子对接可以用来研究生物分子之间的相互作用,例如蛋白质-蛋白质相互作用、蛋白质-DNA 相互作用等。
3. 药物筛选:通过分子对接,可以筛选出能够与特定靶点结合的化合物,从而为药物研发提供候选化合物。
4. 蛋白质结构预测:分子对接可以用来预测蛋白质的三维结构,特别是在没有晶体结构的情况下。
5. 分子机制研究:分子对接可以帮助研究人员了解分子之间的相互作用机制,从而深入理解生物过程和疾病发生的机制。
总之,分子对接是一种重要的计算方法,在药物设计、生物分子相互作用研究、药物筛选、蛋白质结构预测和分子机制研究等领域都有广泛的应用。
分子对接 配位

分子对接配位分子对接配位是一种在药物设计和化学反应研究中常用的方法。
在这种方法中,通过计算机模拟和实验验证,确定两种分子之间的相互作用以及它们如何结合在一起。
这种方法可以帮助科学家设计更有效的药物分子或者理解分子之间的相互作用机制。
在分子对接配位中,通常会涉及到蛋白质和小分子之间的相互作用。
蛋白质是生物体内重要的分子,具有各种生物学功能,包括催化化学反应、传递信号和结构支持等。
而小分子则是化学药物或其他生物活性分子,通过与蛋白质结合来实现治疗或其他作用。
分子对接配位的过程通常包括以下几个步骤:1. 数据准备:首先,需要准备好蛋白质和小分子的结构数据。
蛋白质结构可以通过X射线晶体学或核磁共振等方法确定,而小分子的结构则可以通过分子模拟或实验手段得到。
2. 对接计算:接下来,通过计算机模拟的方法,对蛋白质和小分子之间的相互作用进行预测。
这一步通常使用分子对接软件,根据蛋白质的结构和小分子的结构,预测它们之间的结合方式和亲和力。
3. 结果分析:分子对接计算会生成多个可能的结合构象,科学家需要对这些结果进行分析,找到最有可能的结合方式。
这可能涉及到分子间的氢键、疏水作用、范德华力等相互作用。
4. 实验验证:最后,通过实验验证分子对接计算的结果。
这可以包括分子间的结合亲和力实验、生物活性实验等,来验证分子对接的有效性。
分子对接配位在药物设计中扮演着重要的角色。
通过分子对接配位,科学家可以预测药物分子和靶标蛋白质之间的结合方式,优化药物的生物活性和药效,减少研发的时间和成本。
同时,分子对接配位也可以帮助科学家理解分子之间的相互作用机制,促进生物医药领域的研究和发展。
分子对接

4.1 DOCK
DOCK是Kuntz研究小组发展的分子对接程序,可能是目前应用最为广泛的分子对接程序之一.它能自动地模拟配体分子在受体活性位点的作用情况,并把理论预测最佳的方式记录下来。而且该方法能够对配体的三维结构数据库进行自动搜索,因此被广泛应用于基于受体结构的数据库搜索的药物设计中,并取得了巨大的成功。用DOCK进行药物设计以及数据库的搜索基本上可以分为下面几个步骤:配体和受体相互作用位点的确定,评分系统的生成,DOCK计算及DOCK结果的处理与分析。活性位点的确定和表达是DOCK最重要的特点之一。活性位点特征的确定对于DOCK研究是非常重要的,因为配体分子和受体相互作用过程的模拟主要就是参考几何位点的几何特征进行的。在DOCK中,活性位点的确定通过sphgen程序来完成。DOCK软件包中sphgen程序生成受体表面所有的凹陷的负像,并对这些负像进行聚类分析。在DOCK程序中,表面点采用了Richards提出的模型。在这些表面点的基础上,采用sphgen程序生成了负像,它实际上由一些与分子表面点相切的圆球叠加而成。
在最早的AUTODOCK版本中,作者采用了模拟退火来优化配体和受体之间的结合。在
3.0版本中,Morris等发展了一种改良的遗传算法,即拉马克遗传算法(LGA)。测试结果表
明,LGA比传统的遗传算法比模拟退火具有更高的效率。在LGA方法中,作者把遗传算法和局部搜索(local search)结合在一起,遗传算法用于全局搜索,而局部搜索用于能量优化。在AUTODOCK中,局部搜索方法是自适应的,它可以根据当前的能量调节步长大小。LGA算法引入了拉马克的遗传理论,LGA最大的特点就是通过进化映射(developmental mapping)把基因型转化为表现型而实现局部搜索和遗传算法的结合。基因型空间通过遗传算子突变和交叉来定义;而表现型则通过问题的解来定义,这里表示体系的能量得分。
分子对接强结合6.5

分子对接强结合6.5
分子对接是一种计算化学方法,用于研究分子之间的相互作用。
强结合(或高亲和力)通常指的是分子之间的相互作用非常紧密,
结合力非常强。
在分子对接中,强结合可以表示为两个分子之间的
结合能非常高,通常在6.5千卡/摩尔以上。
分子对接强结合6.5的含义是指在分子对接研究中,两个分子
之间的结合能达到6.5千卡/摩尔以上。
这种强结合通常意味着这两
个分子之间存在着非常紧密的相互作用,可能会形成稳定的复合物
或结合物。
从理论角度来看,分子对接强结合6.5意味着这两个分子之间
的结合是比较牢固的,可能需要较大的能量才能够破坏它们之间的
相互作用。
这对于药物设计和生物化学研究来说具有重要意义,因
为强结合的分子对可能意味着更高的药物活性或者更稳定的蛋白质
结构。
在实际应用中,分子对接强结合6.5的结果可能会被用于筛选
药物候选化合物,设计蛋白质抑制剂或者研究生物分子的相互作用
机制。
这种强结合的相互作用可能会成为新药物设计和生物技术研
究的重要依据。
总的来说,分子对接强结合 6.5代表着在分子相互作用研究中,两个分子之间的结合非常牢固,这种强结合的结果可能会对药物设
计和生物化学研究产生重要影响。
分子对接方法

32
DOCK软件
自动地模拟配体分子在受体活性位点地作 用情况,并把理论预测最佳地相互作用方式 记录下来。 自动搜索配体的 三维结构数据库。
20
(3)基于pose clustering的方法 这种方法与几何哈希的方法相类似,也是一种 基于模式识别的方法。 在LUDI模型中,如图所示,对每一个作用基团,定 义作用中心和作用表面。受体的作用表面近似地用离 散的点表示,和对应的配体的中心目标点相匹配。
三个氢键受体的作用表面
Pose clustering 算法中的作用点
5
分子对接的最初思想起源于Fisher E提出的 “锁和钥匙模型”。即受体与配体的相互识别首 要条件是空间结构的匹配
配体
受体
复合物
受体-配体的锁和钥匙模型
6
这类方法首先要建立大量 化合物(例如几十至上百万个 化合物)的三维结构数据库, 然后将库中的分子逐一与靶标 分子进行“对接” (docking),通过不断优化 小分子化合物的位置(取向) 以及分子内部柔性键的二面角 (构象),寻找小分子化合物 与靶标大分子作用的最佳构象, 计算其相互作用及结合能。在 库中所有分子均完成了对接计 算之后,即可从中找出与靶标 分子结合的最佳分子(前50名 或前100名)
33
Dock软件
步骤 1.分子的准备工作 2.活性位点的确定 3.格点对接 4.柔性对接
34
分子的准备工作
在Chimera软件中进行 加氢原子 加电荷
得到的文件: 1)rec_charged.mol2 2)rec_noH.pdb 3)lig_charged.mol2
分子对接

2.4.2 分子对接的基本方法
(一) 刚性的分子对接方法
这种方法是最初的分子对接的方法,在对接中,小 分子和蛋白质两种都保持刚性。
(1)基于最大团搜索的方法 (Clique-Search Based Approaches)
靶标分类 Hydrolase
靶标结 构
X-ray
BCR-ABL Kinase
X-ray
Anthrax EF
IMPDH
Casein kinase II
K+ 通道
Thyroid homone receptor CDK2 TGFRK
cyclophilin
tRNA guanine transglycosla se
CDK2和CDK4的序列联配,序列同源性45.6%
通过分子对接得到CDK4-NU6102的作用模式
通过CDK4-NU6102复合物和CDK2-NU6102复合物结构对比, 可以很清楚地看到造成NU6102亲和力差别的主要原因是在CDK2- NU6102复合物中,Asp86位起了一个很重要的识别作用,它与配体 的磺胺基形成了两个稳定的氢键,并且通过能量分解的方法也可以 得到导致配体活性差异主要识别基团在磺胺基
Dock对接程序中刚性对接的算法就是基于这种思想 Dock利用球集来表示受体活性位点和配体的形状
一系列的球集填充在受体活性位点的表面,这些球
集代表能被配体占据的体积。配体可以用球集表示或者用 自己的原子表示,在Dock程序中,四个有效匹配的对应点 被考虑,先考虑配体中第一个球集与活性位点的球集的匹 配,第二个点则满足∆d ≤ ε,其中∆d为第二个匹配点中配 体和受体的球心与第一个点球心的距离,第三个点又必需 满足与前两个球心的距离限制,以上过程一直进行到找不 到更多匹配点为止。
分子对接简明教程

分子对接(Molecular Docking)理论所谓分子对接就是两个或多个分子之间通过几何匹配和能量匹配相互识别找到最佳匹配模式的过程。
分子对接对酶学研究和药物设计中有重要的应用意义。
分子对接计算是在受体活性位点区域通过空间结构互补和能量最小化原则来搜寻配体与受体是否能产生相互作用以及它们之间的最佳结合模式。
分子对接的思想起源于Fisher E的”钥匙和锁模型”,主要强调的是空间形状的匹配。
但配体和受体的识别要比这个模型更加复杂。
首先,配体和受体在对接过程中会由于相互适应而产生构象的变化。
其次,分子对接还要求能量匹配,对接过程中结合自由能的变化决定了两个分子是否能够结合以及结合的强度。
1958年D.E.Koshland提出分子识别过程中的诱导契合概念,受体分子活性中心的结构原本并非与底物完全吻合,但其是柔软和可塑的。
当配体与受体相遇时,可诱导受体构象发生相应的变化,从而便于他们的结合进而引起相应的反应。
分子对接方法根据不同的简化程度分为三类:刚性对接、半柔性对接和柔性对接。
刚性对接指在对接过程中,受体和配体的构象不发生变化,适合研究比较大的体系如蛋白-蛋白之间以及蛋白-核酸之间,计算简单,主要考虑对象之间的契合程度。
半柔性对接常用于小分子和大分子的对接,在对接过程中,小分子的构象可以在一定范围内变化,但大分子是刚性的。
这样既可以在一定程度上考察柔性的影响,又能保持较高的计算效率。
在药物设计和虚拟筛选过程中一般采用半柔性的分子对接方法。
柔性对接方法一般用于精确研究分子之间的识别情况,由于允许对接体系的构象变化,可以提高对接准确性但耗时较长。
分子对接的目的是找到底物分子和受体分子最佳结合位置及其结合强度,最终可以获得配体和受体的结合构象,但这样的构象可以有很多,一般认为自由能最小的构象存在的概率最高。
搜寻最佳构象就要用到构象搜索方法,常用的有系统搜索法和非系统搜索法。
系统搜索法通过改变每个扭转角评估所有可能的结合构象,进而选取能量最低的。
分子对接——精选推荐

分⼦对接分⼦对接是通过受体的特征以及受体和药物分⼦之间的相互作⽤⽅式来进⾏药物设计的⽅法。
主要研究分⼦间(如配体和受体)相互作⽤,并预测其结合模式和亲合⼒的⼀种理论模拟⽅法.近年来,分⼦对接⽅法已成为计算机辅助药物研究领域的⼀项重要技术。
分⼦对接⽅法分⼦对接⽅法的两⼤课题是分⼦之间的空间识别和能量识别。
空间匹配是分⼦间发⽣相互作⽤的基础,能量匹配是分⼦间保持稳定结合的基础。
对于⼏何匹配的计算,通常采⽤格点计算、⽚断⽣长等⽅法,能量计算则使⽤模拟退⽕、遗传算法等⽅法。
各种分⼦对接⽅法对体系均有⼀定的简化,根据简化的程度和⽅式,可以将分⼦对接⽅法分为三类。
刚性对接:刚性对接⽅法在计算过程中,参与对接的分⼦构像不发⽣变化,仅改变分⼦的空间位置与姿态,刚性对接⽅法的简化程度最⾼,计算量相对较⼩,适合于处理⼤分⼦之间的对接。
半柔性对接:半柔性对接⽅法允许对接过程中⼩分⼦构像发⽣⼀定程度的变化,但通常会固定⼤分⼦的构像,另外⼩分⼦构像的调整也可能受到⼀定程度的限制,如固定某些⾮关键部位的键长、键⾓等,半柔性对接⽅法兼顾计算量与模型的预测能⼒,是应⽤⽐较⼴泛的对接⽅法之⼀。
柔性对接:柔性对接⽅法在对接过程中允许研究体系的构像发⽣⾃由变化,由于变量随着体系的原⼦数呈⼏何级数增长,因此柔性对接⽅法的计算量⾮常⼤,消耗计算机时很多,适合精确考察分⼦间识别情况。
主要分⼦对接软件DOCKDock是应⽤最⼴泛的分⼦对接软件之⼀,由Kuntz课题组开发。
Dock应⽤半柔性对接⽅法,固定⼩分⼦的键长和键⾓,将⼩分⼦配体拆分成若⼲刚性⽚断,根据受体表⾯的⼏何性质,将⼩分⼦的刚性⽚断重新组合,进⾏构像搜索。
在能量计算⽅⾯,Dock考虑了静电相互作⽤、范德华⼒等⾮键相互作⽤,在进⾏构像搜索的过程中搜索体系势能⾯。
最终软件以能量评分和原⼦接触罚分之和作为对接结果的评价依据。
AutoDockAutodock是另外⼀个应⽤⼴泛的分⼦对接程序,由Olson科研组开发。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
AutoDock和AutoDock Tools 使用教程一、分子对接简介及软件介绍1.分子对接理论基础所谓分子对接就是两个或多个分子之间通过几何匹配和能量匹配而相互识别的过程。
分子对接在酶学研究以及药物设计中具有十分重要的意义。
在酶激活剂、酶抑制剂与酶相互作用以及药物分子产生药理反应的过程中,小分子(通常意义上的Ligand)与靶酶(通常意义上的Receptor)相互互结合,首先就需要两个分子充分接近,采取合适的取向,使两者在必要的部位相互契合,发生相互作用,继而通过适当的构象调整,得到一个稳定的复合物构象。
通过分子对接确定复合物中两个分子正确的相对位置和取向,研究两个分子的构象,特别是底物构象在形成复合物过程中的变化,是确定酶激活剂、抑制剂作用机制以及药物作用机制,设计新药的基础。
分子对接计算是把配体分子放在受体活性位点的位置,然后按照几何互补、能量互补化学环境互补的原则来实时评价配体与受体相互作用的好坏,并找到两个分子之间最佳的结合模式。
分子对接最初思想起源于Fisher E.的“锁和钥匙模型”(图),认为“锁”和“钥匙”的相识别的首要条件是他们在空间形状上要互相匹配。
然而,配体和受体分子之间的识别要比“锁和钥匙”模型复杂的多。
首先,配体和受体分子的构象是变化的,而不是刚性的,配体和受体在对接过程中互相适应对方,从而达到更完美的匹配。
其次,分子对接不但要满足空间形状的匹配,还要满足能量的匹配。
配体和受体之间的通过底物分子与靶酶分子能否结合以及结合的强度最终是由形成此复合物过程的结合自由能变化ΔG bind所决定的。
互补性(complementarity)和预组坦织(pre-organization)是决定分子对接过程的两个重要原则,前者决定识别过程的选择性,而后者决定识别过理的结合能力。
互补性包括空间结构的互补性和电学性质的互补性。
1958年Koshland提出了分子识别过程中的诱导契合(induced fit)概念,指出配体与受体相互结合时,受体将采取一个能同底物达到最佳结合的构象(图1)。
而受体与配体分子在识别之前将受体中容纳配体的环境组织的越好,其溶剂化能力越低,则它们的识别效果越佳,形成的复合物也就越稳定。
图1:“锁和钥模型(a)”和“诱导契合模型(b)”示意图2.分子对接常用方法分子对接方法根据不同的简化程度可以大致分为以下三类:(1)刚性对接;(2)半柔性对接;(3)柔性对接。
刚性对接指在对接的过程中,研究体系的构象不发生变化;半柔性对接是指在对接过程中,研究体系尤其是配体的构象允许在一定的范围内变化;而柔性对接则是指在对接过程中,研究体系的构象基本上是可以自由变化的。
当然,这只是一种简单的分类方法,而在很多分子对接程序中,实际上采取了多种处理方法。
在这些分子对接方法中,刚性对接适合考察比较大的体系,比如蛋白质和蛋白质以及蛋白质和核酸之间的相互作用,他计算较为简单,原理也相对简单,主要是考虑构象之间的契合程度。
半柔性对接适合于处理小分子和大分子之间的对接。
在对接过程中,小分子的构象一般是可以变化的,但大分子则是刚性的。
由于小分子相对较小,因此在一定程度考察柔性的基础上,还可以保持较高的计算效率,在药物设计尤其在基于分子数据库的虚拟筛选过程中,一般采用半柔性的分子对接方法。
柔性对接方法一般用于精确考察分子之间的识别情况,由于在计算过程中体系的构象是可以变化的,因此柔性对接在提高了对接准确性的同时却需要耗费较长的计算时间。
上述各种几何优化方法可以获得分子对应与初始态的优势构象,但实际上这样的构象可以有很多,一般认为自由能最小的构象存在的概率高,全局极小可能是比较重要的构象。
同时,配体和受体活性部位结合时的构象不一定是全局极小构象,而可能是某一低能构象(药效构象)。
不管是寻找分子的全局极小构象还是药效构象,均要使用构象搜索方法。
分子对接的目的是找到底物分子和受体分子的最佳结合位置。
因此,分子对接面临的最重要的问题就是如何找到最佳的结合位置以及如何评价对接分子之间的结合强度。
当然,这两个问题也是相互关联的。
如何找到最佳的结合位置就要牵涉到构象搜索方法。
常用的构象搜索方法有系统搜索法和非系统搜索法。
系统搜索法通过系统的改变每一个扭转角产生所有可能的构象,从中挑选出能量较低的构象,但计算量非常大。
所以通常使用非系统搜索法来寻找能量较低构象,常用方法有:(1)分子动力学方法(Molecular dynamics,MD);(2)随机搜索(Random search);(3)遗传算法(Genetic algorithm,GA);(4)距离几何算法(Distance geometry,DG)等。
随机搜索又包含:(1)完全随机算法;(2)蒙特卡罗法(Monte Carlo,MC);(3)模拟退火法(Simulated annealing,SA)。
分子对接的方法很多,下表(表1)列出了针对不同对接体系的常用对接方法:表1:常用的分子对接方法3.分子对接软件第一个分子对接程序是UCSF Kuntz小组于1982年开发的DOCK,早期的版本以刚性对接为主,从版开始考虑配体的柔性。
像这样的半柔性(刚性受体-柔性配体:rigid receptor-flexible ligand docking)对接程序还有AutoDock、FlexX等。
同时考虑配体和受体柔性的对接程序主要有FlexiDock,它采用遗传算法来对配体和受体的结合构像进行优化,在初始条件较好的情况下,FlexiDock可以比较精确的确定配体和受体的结合状态,但计算时间较长。
下表为一些有代表性的分子对接软件(表2)及软件的被引次数对比(图2)。
表2:有代表性的分子对接软件注:CAPRI,Critical Assessment of PRediction of Interactions,相互作用预测临界评价图2:分子对接软件——最常用的分子对接软件被引次数统计(ISI Web of Sciense,2005)4.AutoDock以及ADT(AutoDock Tools)是The Scripps Research Institute的OIson科研小组使用C语言开发的分子对接软件包,目前最新的版本为。
AutoDock其实是一个软件包,其中主要包含AutoGrid和AutoDock两个程序。
其中AutoGrid主要负责格点中相关能量的计算,而AutoDock则负责构象搜索及评价。
AutoDock在早期版本中使用的是模拟退火算法(Simulated Annealing Algorithm)来寻找配体与受体最佳的结合位置状态,而从版本开始使用一种改良的遗传算法,即拉马克遗传算法(Lamarckian Genetic Algorithm,LGA)。
测试结果表明,LGA比传统的遗传算法和模拟退火具有更高的效率。
在LGA方法中,作者把遗传算法和局部搜索(Local search)结合在一起,遗传算法用于全局搜索,而局部搜索用于能量优化。
LGA算法引入了拉马克的遗传理论,这个操作过程可下图(图3)。
图3:LGA算法操作过程图同时在AutoDock中配体和受体之间结合能力采用能量匹配来评价。
在和版本中,能量匹配得分采用简单的基于AMBER力场的非键相互作用能。
非键相互作用来自于三部分的贡献:范德华相互作用,氢键相互作用以及静电相互作用。
而在之后的版本中AutoDock提供了半经验的自由能计算方法来评价受体和配体之间的能量匹配。
为了加快计算速度,AutoDock采用格点对接的方法,但与DOCK中格点对接的处理方法有明显的区别。
DOCK中,格点上保存的不是能量,而是仅与受体有关的特征量。
而在AutoDock中,格点上保存的是探针原子和受体之间的相互作用能。
对于范德华相互作用的计算,每个格点上保存的范德华能量的值的数目与要对接的配体上的原子类型(表3)的数目一样。
如果一个配件中含有C、O和H三种原子类型,那么在每个格点上就需要用三个探针原子来计算探针原子与受体之间的范德华相互作用值。
当配体和受体进行分子对接时,配体中某个原子和受体之间的相互作用能通过周围8个格点上的这种原子类型为探针的格点值用内插法得到。
表3:AutoDock4中的原子类型(*为默认在gpf中存在的原子类型)静电相互作用的计算采用了一个静电势格点,在格点上储存受体分子的静电势。
当配体和受体分子对接时,某个原子和受体之间的静电相互作用能通过周围格点上静电势以及原子上的部分电荷就可以计算得到。
计算氢键相互作用时,格点的处理和范德华相互作用有点类似,每个格点上需要保存配体分子中所有氢键给体与氢键受体之间的相互作用能量,而且这些能量都是在氢键在最佳情况下的氢键能量值。
以上格点能量的计算都是由AutoDock中的AutoGrid程序计算得出的,AutoDock格点对接示意图如下图所示(图4)。
AutoDock格点对接的基本流程如下:首先,用围绕受体活性位点的氨基酸残基形成一个范围更大的Box,然后用不同类型的原子作为探针(probe)进行扫描,计算格点能量,此部分任务由AutoGrid程序完成。
然后AutoDock程序对配体在Box范围内进行构象搜索(conformational search),最后根据配体的不同构象(conformation),方向(orientation)、位置(position)及能量(energy)进行评分(scoring),最后对结果进行排序(ranking)。
图4:AutoDock格点对接示意图AutoDock目前的版本只能实现单个配体和受体分子之间的对接,程序本身还没有提供虚拟筛选功能(Virtual Screening),但是可以使用Linux/Unix中的Shell以及Python语言实现此功能。
同时AutoDock本省所包含的AutoDock以及AutoGrid程序是完全在命令附下操作的软件,没有图形界面,但是如果使用AutoDock Tools程序,就可以在几乎完全图形化的界面中完成分子对接以及结果分析等工作,下面我们就介绍一下AutoDock Tools。
ToolsAutoDock Tools(以下简称ADT)是The Scripps Research Institute,Molecular Graphics Laboratory (MGL)在Python Molecular Viewer(以下简称PMV,Python语言开发)基础上开发的针对AutoGrid和AutoDock程序开发的图形化的分子可视化及对接辅助软件,目前最新版本为1.5.2。
在这里我们使用的版本为,它的主界面主要包含以下几个部分(图5):图5:ADT1.5.1的主界面及窗口部件(1)PMV菜单:主要通过使用菜单命令对分子进行相关的操作,以及进行可视化设置;(2)PMV工具栏:PMV菜单中一些常用命令的快捷按钮;(3)ADT菜单:AutoGrid和AutoDock的图形化操作菜单;(4)分子显示窗口:3D模型分子的显示和操作窗口;(5)仪表板窗口部件:快速查看及设置分子的显示模型以及着色方式;(6)信息栏:显示相关操作信息。
