Affymetrix基因芯片产品
基因芯片知名企业

基因芯片知名企业基因芯片是一种高通量的基因检测技术,可以同时检测数千个基因的表达水平、突变状态、拷贝数变化等信息。
这种技术在生物医学研究、临床诊断、药物研发等领域有着广泛的应用。
目前,全球有许多知名的基因芯片企业,其中包括Illumina、Affymetrix、Agilent、Thermo Fisher Scientific等。
Illumina是全球最大的基因芯片企业之一,总部位于美国加利福尼亚州圣地亚哥市。
该公司成立于1998年,最初是一家生物芯片企业,后来专注于基因测序和基因芯片技术的研发和生产。
Illumina的基因芯片产品包括全基因组芯片、转录组芯片、表观基因组芯片等,广泛应用于生物医学研究、临床诊断、药物研发等领域。
Illumina的技术优势在于高通量、高灵敏度、高精度和高可靠性,被广泛认可和应用。
Affymetrix是另一家知名的基因芯片企业,总部位于美国加利福尼亚州圣克拉拉市。
该公司成立于1992年,是全球第一家生产基因芯片的企业。
Affymetrix的基因芯片产品包括全基因组芯片、转录组芯片、SNP芯片等,应用于生物医学研究、临床诊断、药物研发等领域。
Affymetrix的技术优势在于高通量、高灵敏度、高特异性和高可靠性,被广泛认可和应用。
Agilent是一家全球性的科技企业,总部位于美国加利福尼亚州圣克拉拉市。
该公司成立于1999年,最初是惠普公司的电子测量与仪器部门,后来独立成为一家科技企业。
Agilent的基因芯片产品包括全基因组芯片、转录组芯片、miRNA芯片等,应用于生物医学研究、临床诊断、药物研发等领域。
Agilent的技术优势在于高通量、高灵敏度、高特异性和高可靠性,被广泛认可和应用。
Thermo Fisher Scientific是一家全球性的科技企业,总部位于美国马萨诸塞州威尔明顿市。
该公司成立于2006年,是由Thermo Electron Corporation和Fisher Scientific International合并而成。
第十章基因芯片微阵列数据库

Agilent等芯片采用双荧光标记法检测和数据分 析。双荧光标记杂交技术中,两种不同样品的mRNA 被用不同的荧光标记。标记产物与芯片上的DNA探针
杂交后,在不同的激发波长和发射波长检测后,通过
激光共聚焦荧光扫描检测杂交信号。同一探针上的两
种不同荧光信号的相对强度被用于推算相应靶基因在
两种不同样品中的相对表达量。两个样品中通常一个 是对照样品,一个是待测样品。如果不同的芯片使用 相同对照样品,则不同芯片上的待测样品中基因表达 的水平也可被比较。
第十章 基因芯片微阵列数据库
基因芯片是所有生物芯片的佼佼者。其芯片制 作技术、数据分析方法及在各种生命科学领域内的 应用均遥遥领先于其他类型的生物芯片。
第一节 常用基因芯片及其数据库
一、Affymetrix芯片
Affymetrix基因芯片系同类产品的首创,
为最受欢迎的基因芯片之一,在生物各领域
应用广泛。
因芯片数据包括四项:
1、探针组代号。Affymetrix给每个探针组独特代号。
一般探针组代号与靶基因一一对应,但有例外。
2、表达值。经由MA55处理后得到的探针组表达值,
相当于靶基因表达值。
3、表达值预测。有三字母分别代表表达值是否真的存
在:P代表存在,A代表不存在,M代表介于两者之间。
基因表达的存在与否由统计学经分析探针组中每根探
芯片上的25核苷酸探针通过一种基于光刻合成 及组合化学的独特工艺直接在芯片上合成。芯 片设计的核心技术是探针对的使用:每一根匹 配探针(PM)均有一根相应的错误探针(MM) 与其相匹配。两个探针间的唯一区别在于第13 个核苷酸。PM的该位置核苷酸可同其靶基因完 全互补,MM则相反。这种设计利于对非特异杂 交作出修正。每一靶基因都有相应的多组探针 对。
AFFYMETRIX基因芯片操作流程

AFFYMETRIX 基因芯片操作流程第一章真核靶片断制备<一> RNA 的抽提一、 哺乳动物细胞或组织RNA 的抽提1. 总 RNA 使用 QIAGEN ' 哺乳动物组织作为+2. Poly(A) mRNA 哺乳动物细胞使用 哺乳动物组织作为 离步骤或使用kit.二、 RNA 沉淀1. 总 RNA在用RNeasy Total RNA Isolation kit 分离或洗涤后没有必要沉淀总 RNA.调整洗脱体积以制备cDNA 合成接近希望的 RNA 浓度。
注:为获得足够量的标记 cRNA 用来评估和基因芯片表达探针杂交, AFFYMETRIX 建议开 始合成cDNA 的Poly(A) +mRNA 最小浓度为0.02卩g/卩l 时的最小量是0.2卩g,总RNA 最 小浓度为0.5卩g/卩l 时的最小量是5卩g.这样有两个好处: (1) 有足够量在各步检查样品浓度和质量 (2)制备足够的cRNA 用于杂交在TRIzol 分离和热酚提取后需要乙醇沉淀;见下面方法 +2. Poly(A) mRNA大多数Poly(A) +mRNA 分离过程都会导致得到较稀浓的 RNA ,所以需要在cDNA 合成前浓缩mRNA. 3. 沉淀步骤: (1) 力口 1/10体积3M NaOAc,PH5.2,和2.5倍体积乙醇. (2) 混匀,-20C 放置最少1小时. (3) 4C ,> 12000x g 离心 20 分钟. (4) 80%乙醇洗涤沉淀 2次.(5) 空气干燥沉淀.继续下面步骤前检查是否干燥 . (6)DEPC 处理水重新溶解沉淀.最合适的溶解体积由cDNA 合成中需要的 RNA 的浓度和量来决定.先阅读cDNA 合成的过程来决定这一步的适合溶解体积4. RNA 测定用分光光度计分析 RNA 浓度,在260nm1单位吸光度等于 40卩g/mlRNA.需要在260和280nm 测定吸光度来确定样品的浓度和纯度 A 260/A 280应接近2.0为较纯的RNA(比值在1.9-2.1也可)<二>由纯化的总RNA 合成双链cDNAAFFYMETRIX 强烈建议 HPLC 纯化 T7-(d7) 24 primerRNeasy Total RNA Isolation kit 成功抽提哺乳动物细胞总 RNA.RNA 的来源,建议使用 TRIzol 抽提总RNA.QIAGEN ' Oligotex Direct mRNA kit,从总 RNA 中抽提 mRNA . RNA 的来源,应首先使用 TRIzol 纯化,再进行一个 Poly(A)+mRNA 分一、第一链cDNA合成开始RNA的量:高质量RNA5.0卩g -40.0卩g纯化后RNA浓缩由260nm吸光度决定(1单位吸光度=40卩g/mlRNA ), A260/A280应接近2.0,在1.8-2.1的范围内。
affymetrix 基因表达谱芯片 差异基因

Affymetrix基因表达谱芯片是一种常用的高通量基因检测技术,在生物医学领域得到了广泛的应用。
通过对细胞或组织中基因表达水平的全面分析,可以帮助科研人员发现不同生理状态或疾病状态下的差异基因。
本文将对Affymetrix基因表达谱芯片和差异基因进行深入探讨,希望可以为读者提供更全面的了解。
一、Affymetrix基因表达谱芯片概述Affymetrix基因表达谱芯片是一种利用基因芯片技术进行高通量基因表达分析的方法。
该芯片采用了高度平行的探针阵列,每个探针对应一个已知的基因序列,可以同时检测成千上万个基因的表达水平。
Affymetrix基因表达谱芯片的优点在于其高通量、高灵敏度和高特异性,可以快速、准确地获得大量的基因表达信息。
二、Affymetrix基因表达谱芯片的工作原理Affymetrix基因表达谱芯片的工作原理可以简要概括为以下几个步骤:1. RNA提取和标记:首先从待测样本中提取RNA,然后利用标记试剂将RNA转录成cDNA,并进行标记,通常使用生物素和荧光标记等方法。
2. 芯片杂交:标记好的cDNA与芯片上的探针阵列进行杂交,在杂交的过程中,标记的cDNA会特异性地结合到与其互补的探针上。
3. 芯片扫描:经过杂交后,芯片上的探针会产生荧光信号,然后使用芯片扫描仪对芯片进行扫描,测得每个探针的荧光强度。
4. 数据分析:最后对扫描得到的数据进行分析和解读,得到与样本中基因表达水平相关的信息。
三、差异基因的分析差异基因是指在不同生理状态或疾病状态下,其表达水平有明显差异的基因。
通过Affymetrix基因表达谱芯片的分析,可以筛选出在不同样本中表达水平有显著差异的基因,进而进行差异基因的进一步研究。
一般来说,差异基因分析主要包括以下几个步骤:1. 数据预处理:对Affymetrix芯片扫描得到的原始数据进行背景校正、归一化处理等,使得数据更加准确可靠。
2. 统计分析:利用统计学方法对处理后的数据进行差异分析,一般采用t检验、方差分析、贝叶斯统计等方法进行差异基因的筛选。
Affymetrix芯片质量评估

Affymetrix芯片质量评估在拿到(数据库下载或者自己实验得到)的芯片,最好先对芯片的质量做出评估,从而将有问题的芯片剔除。
在RobertGentleman的“Bioinformatics and Computational Biology Solutions UsingR and Bioconductor”书的第三章提到“Before any useis made of more complexmethods, an initial examination of the data can often show evidenceof possible quality problems.”。
以下关于Affymetrix芯片质控的图和脚本,也引用上述参考书。
首先是一些基础知识:1 Affymetrix芯片的数据格式主要有.dat和.cel两种。
DAT文件是原始芯片图像的扫描文件,需要用affy公司自己的软件打开。
CEL文件是DAT文件去除背景噪音后的文件,包括了每个探针的原始密度数值(rawintensityvalue)。
其中,我们最关注CEL文件,这也是我们后续载入Bioconductor中的原始数据类型。
后续需要对CEL文件进行“质量评估”、“归一化”、“注释”等一系列预处理。
2affy芯片的数据单元。
下图是一个“探针集(probeset)”,包括了11-20个长度位22nt的“探针(probe)”,图中每个亮格代表一个探针。
每个探针分为PM(PerfectMatch)和MM(Mis-Match)两种,区别就是MM探针故意将一个碱基设计错。
这样做的目的是为了控制芯片的非特异性杂交,从而获得更准确的信号值。
芯片质量控制:1. 对于“探针数据(probe-data level)”的三种图,使用"affy"packageboxplot():未处理的原始探针密度(以2为底取对数)的盒箱图。
基因芯片(Affymetrix)分析1:芯片质量分析

基因芯⽚(Affymetrix)分析1:芯⽚质量分析TAIR,NASCarray 和 EBI 都有⼀些公开的免费芯⽚数据可以下载。
本专题使⽤的数据来⾃NASCarray(Exp350),也可以⽤FTP直接下载。
下载其中的CEL⽂件即可(.CEL.gz),下载后解压缩到同⼀⽂件夹内。
该实验有1个对照和3个处理,各有2个重复,共8张芯⽚(8个CEL⽂件)。
为什么要进⾏芯⽚质量分析?不是每个⼈做了实验都会得到⾼质量的数据,花了钱不⼀定就有回报,这道理⼤家都懂。
芯⽚实验有可能失败,失败的原因可能是技术上的(包括⽚⼦本⾝的质量),也可能是实验设计⽅⾯的。
芯⽚质量分析主要检测前者。
1 R软件包安装使⽤到两个软件包:affy,simpleaffy:library(BiocInstaller)biocLite(c("affy", "simpleaffy"))另外还需要两个辅助软件包:tcltk和scales。
tcltk⼀般R基础安装包都已经装有。
install.packages(c("tcltk", "scales"))2 读取CEL⽂件载⼊affy软件包:library(affy)library(tcltk)选取CEL⽂件。
以下两种⽅法任选⼀种即可。
第⼀种⽅法是通过选取⽬录获得某个⽬录内(包括⼦⽬录)的所有cel⽂件:# ⽤choose.dir函数选择⽂件夹dir <- tk_choose.dir(caption = "Select folder")# 列出CEL⽂件,保存到变量cel.files <- list.files(path = dir, pattern = ".+\\.cel$", ignore.case = TRUE,s = TRUE, recursive = TRUE)# 查看⽂件名basename(cel.files)第⼆种⽅法是通过⽂件选取选择⽬录内部分或全部cel⽂件:# 建⽴⽂件过滤器filters <- matrix(c("CEL file", ".[Cc][Ee][Ll]", "All", ".*"), ncol = 2, byrow = T)# 使⽤tk_choose.files函数选择⽂件cel.files <- tk_choose.files(caption = "Select CELs", multi = TRUE, filters = filters,index = 1)# 注意:较⽼版本的tk函数有bug,列表的第⼀个⽂件名可能是错的basename(cel.files)## [1] "NRID9780_Zarka_2-1_MT-0HCA(SOIL)_Rep1_ATH1.CEL"## [2] "NRID9781_Zarka_2-2_MT-0HCB(SOIL)_Rep2_ATH1.CEL"## [3] "NRID9782_Zarka_2-3_MT-1HCA(SOIL)_Rep1_ATH1.CEL"## [4] "NRID9783_Zarka_2-4_MT-1HCB(SOIL)_Rep2_ATH1.CEL"## [5] "NRID9784_Zarka_2-5_MT-24HCA(SOIL)_Rep1_ATH1.CEL"## [6] "NRID9785_Zarka_2-6_MT-24HCB(SOIL)_Rep2_ATH1.CEL"## [7] "NRID9786_Zarka_2-7_MT-7DCA(SOIL)_Rep1_ATH1.CEL"## [8] "NRID9787_Zarka_2-8_MT-7DCB(SOIL)_Rep2_ATH1.CEL"读取CEL⽂件数据使⽤ReadAffy函数,它的参数为:# Not run. 函数说明,请不要运⾏下⾯代码ReadAffy(..., filenames = character(0), widget = getOption("BioC")$affy$use.widgets,compress = getOption("BioC")$affy$compress.cel, celfile.path = NULL, sampleNames = NULL,phenoData = NULL, description = NULL, notes = "", rm.mask = FALSE, rm.outliers = FALSE,rm.extra = FALSE, verbose = FALSE, sd = FALSE, cdfname = NULL)除⽂件名外我们使⽤函数的默认参数读取CEL⽂件:data.raw <- ReadAffy(filenames = cel.files)读⼊芯⽚的默认样品名称是⽂件名,⽤sampleNames函数查看或修改:sampleNames(data.raw)## [1] "NRID9780_Zarka_2-1_MT-0HCA(SOIL)_Rep1_ATH1.CEL"## [2] "NRID9781_Zarka_2-2_MT-0HCB(SOIL)_Rep2_ATH1.CEL"## [3] "NRID9782_Zarka_2-3_MT-1HCA(SOIL)_Rep1_ATH1.CEL"## [4] "NRID9783_Zarka_2-4_MT-1HCB(SOIL)_Rep2_ATH1.CEL"## [5] "NRID9784_Zarka_2-5_MT-24HCA(SOIL)_Rep1_ATH1.CEL"## [6] "NRID9785_Zarka_2-6_MT-24HCB(SOIL)_Rep2_ATH1.CEL"## [7] "NRID9786_Zarka_2-7_MT-7DCA(SOIL)_Rep1_ATH1.CEL"## [8] "NRID9787_Zarka_2-8_MT-7DCB(SOIL)_Rep2_ATH1.CEL"sampleNames(data.raw) <- paste("CHIP", 1:length(cel.files), sep = "-")sampleNames(data.raw)## [1] "CHIP-1" "CHIP-2" "CHIP-3" "CHIP-4" "CHIP-5" "CHIP-6" "CHIP-7" "CHIP-8"3 查看芯⽚的基本信息Phenotypic data数据可能有⽤,可以修改成你需要的内容,⽤pData函数查看和修改:pData(data.raw)## sample## CHIP-1 1## CHIP-2 2## CHIP-3 3## CHIP-4 4## CHIP-5 5## CHIP-6 6## CHIP-7 7## CHIP-8 8pData(data.raw)$Treatment <- gl(2, 1, length = length(cel.files), labels = c("CK","T"))pData(data.raw)## sample Treatment## CHIP-1 1 CK## CHIP-2 2 T## CHIP-3 3 CK## CHIP-4 4 T## CHIP-5 5 CK## CHIP-6 6 T## CHIP-7 7 CK## CHIP-8 8 TPM和MM查看:# Perfect-match probespm.data <- pm(data.raw)head(pm.data)## CHIP-1 CHIP-2 CHIP-3 CHIP-4 CHIP-5 CHIP-6 CHIP-7 CHIP-8 ## 501131 127.0 166.3 112.0 139.8 111.3 85.5 126.3 102.8## 251604 118.5 105.0 82.0 101.5 94.0 81.3 103.8 103.0## 261891 117.0 90.5 113.0 101.8 99.3 107.0 85.3 85.3## 230387 140.5 113.5 94.8 137.5 117.3 112.5 124.3 114.0## 217334 227.3 192.5 174.0 192.8 162.3 163.3 235.0 195.8## 451116 135.0 122.0 86.8 93.3 83.8 87.3 97.3 83.5# Mis-match probesmm.data <- mm(data.raw)head(mm.data)## CHIP-1 CHIP-2 CHIP-3 CHIP-4 CHIP-5 CHIP-6 CHIP-7 CHIP-8 ## 501843 89.0 88.0 80.5 91.0 77.0 75.0 79.0 72.0## 252316 134.3 77.3 77.0 107.8 98.5 75.0 99.5 71.3## 262603 119.3 90.5 82.0 86.3 93.0 89.3 94.5 83.8## 231099 123.5 94.5 76.5 95.0 89.3 87.8 95.5 91.5## 218046 110.3 93.0 74.8 100.5 86.0 89.5 104.5 102.3## 451828 127.5 77.0 80.3 94.5 72.3 79.0 86.3 67.84 显⽰芯⽚扫描图像(灰度)# 芯⽚数量n.cel <- length(cel.files)par(mfrow = c(ceiling(n.cel/2), 2))par(mar = c(0.5, 0.5, 2, 0.5))# 设置调⾊板颜⾊为灰度pallette.gray <- c(rep(gray(0:10/10), times = seq(1, 41, by = 4)))# 通过for循环逐个作图for (i in 1:n.cel) image(data.raw[, i], col = pallette.gray)如果芯⽚图像有斑块现象就很可能是坏⽚。
AFFYMETRIX基因芯片操作流程

A.生 B.旦 C.净 D.丑至200μl至300μl终体积100μl200μl300μl20×Eukaryotic Hybridization Controls冻存,使用前65℃,5分钟2.使用前室温平衡探针3.99℃,5分钟4.通过加样孔加入适量体积(见表 2.2)1×Hybridization Buffer湿润芯片5. 45℃,60rpm预杂交芯片10分钟6. 步骤3处理过的样品45℃,5分钟7. 最大速离心5分钟8. 从芯片中取出Buffer,加等体积处理好的杂交液9. 45℃,60rpm杂交芯片16小时表2.2Array 杂交体积总体积Standard 200μl250μlMidi 130μl160μlMini 80μl100μlMicro 80μl100μl第三章洗脱、染色、扫描芯片<一>实验和洗涤工作站设置一、进入Experiment Information洗脱、染色、扫描芯片之前都必须先定义一个Experiment二、准备洗涤工作站基因芯片洗涤工作站400用来洗脱和染色探针阵列。
<二>洗脱和染色1.杂交16小时后,从芯片中取出杂交液装入一个新的离心管,放置冰上或-80℃长时间保存。
2. Wash Buffer A充满芯片3.配制下列溶液表3.1 SAPE液(使用前配制,4℃储存)成分体积终浓度2×MES Stain Buffer 600.0μl1×50mg/ml acetyed BSA 48.0μl2mg/ml1mg/ml Streptavidin-12.0μl10μg/ml Phycoerythrin(SAPEDI 水540.0μl总体积1200μl表3.2抗体溶液成分体积终浓度2×MES Stain Buffer 300.0μl1×50mg/ml acetyed BSA 24.0μl2mg/ml 10mg/ml Normal Goat IgG 6.0μl0.1mg/ml 0.5mg/ml biotinylatedantibody3.6μl3μg/ml DI水266.4μl总体积600μl4.洗涤工作站按表 3.3工作表3.3标准格式EukGE-WS2 Midi格式Midi-euk2Micro/Mini格式Micro-1v1/Mini-euk2Post Hyb Wash#1 10 cycles of 2mixes/cycle withWash Buffer A at25℃10 cycles of 2mixes/cycle with WashBuffer A at 30℃10 cycles of 2mixes/cycle with WashBuffer A at 25℃Post Hyb Wash#2 4 cycles of 15 mixes/cycle withWash Buffer B at 50℃6 cycles of 15 mixes/cycle with Wash Buffer B at 50℃8 cycles of 15mixes/cycle with Wash Buffer B at 50℃StainStain the probe array for 10minutes in SAPE solution at 25℃Stain the probe array for 5minutes in SAPE solution at 35℃Stain the probe arrayfor 10minutes in SAPE solution at 25℃Post Stain Wash 10 cycles of 4 mixes/cycle withWash Buffer A at 25℃10 cycles of 4 mixes/cycle with Wash Buffer A at 30℃10 cycles of 4mixes/cycle with Wash Buffer A at 30℃2nd Stain Stain the probe array for 10minutes in antibody solution at 25℃Stain the probe arrayfor 5minutes inantibody solution at 35℃Stain the probe arrayfor 10minutes in antibody solution at 25℃3nd Stain Stain the probe array for 10minutes in SAPE solution at 25℃Stain the probe array for 5minutes in SAPE solution at 35℃Stain the probe arrayfor 10minutes in SAPE solution at 25℃Final Wash15 cycles of 4 mixes/cycle with Wash Buffer A at 30℃The holding temperature is 25℃15 cycles of 4 mixes/cycle with Wash Buffer A at 35℃The holdingtemperature is 25℃15 cycles of 4mixes/cycle with Wash Buffer A at 35℃The holdingtemperature is 25℃<三>扫描<四>关闭洗涤工作站附:溶液配制5×RNA Fragmentation Buffer:(200mM Tris-acetate,PH 8.1,500mM KOAc,150mM MgOAc 4.0ml 1M Tris-acetate,PH 8.1(冰醋酸调节PH0.64g MgOAc0.98g KOAcDEPC处理水至20ml强烈搅拌,混合均匀0.2μm过滤室温保存12×MES Stock:(1.22M MES,0.89M[Na+]1000ml:70.4g MES free acid monohydrate193.3g MES Sodium Salt800ml 分子生物级水混合,定溶至1000ml. PH在6.5-6.7之间0.2μm过滤不灭菌,2-8℃避光保存2×Hybridization Buffer:(1×:100mM MES, 1M [Na+],20mM EDTA,0.01%Tween2 50ml:8.3ml 12×M ES Stock17.7ml 5M NaCl4.0ml 0.5M EDTA0.1ml 10%Tween202-8℃避光保存Wash Buffer A:(6×SSPE,0.01%Tween20)1000ml:300ml 20×SSPE1.0ml 10%Tween20698ml 水0.2μm过滤Wash Buffer B:(100mM MES,0.1M [Na+],0.01%Tween20)1000ml:MES Stock Buffer83.3ml 12×5.2ml 5M NaCl1.0ml 10%Tween20910.5ml 水0.2μm过滤2-8℃避光保存2×Stain Buffer:(1×:100mM MES, 1M [Na+],0.05%Tween20 250ml:MES Stock Buffer41.7ml 12×92.5ml 5M NaCl2.5ml 10%Tween200.2μm过滤2-8℃避光保存10mg/ml Goat IgG Stock:溶解50mg在5ml PBS中4℃保存。
Affymetrix基因表达谱芯片操作指南(中文版)

7
地址:上海市张江高科技园区李冰路 151 号
电话:021-51320288 传真:021-51320266
杂交
终体积
体积 终浓度或量
91μl
30μl
1×
3μl 200μM each
1μl
10U
4μl
40U
1μl
2U
130μl
4
地址:上海市张江高科技园区李冰路 151 号
电话:021-51320288 传真:021-51320266
3. 混匀。稍微离心,16℃放置 2 小时。 4. 加 2μl 10U/μl T4 DNA 聚合酶。 5. 16℃放置 5 分钟。 6. 加 10μl 0.5M EDTA 终止反应。 7. 继续纯化 cDNA 步骤,或-20℃储存。
体积 4μl 2μl 1μl
c) 第一链合成 z 1-8 μg总RNA: 1 μL SuperScript II z 8.1-15 μg总RNA: 2 μL SuperScript II z 每1μg mRNA加1 μL SuperScript II. z 少于1μg mRNA 加1 μL SuperScript II. 在反应管中加入相应体积的 SuperScript II,混合均匀,42℃温浴 1 小时,反应结 束放置冰上至少 2 分钟。
反应试剂 T7-(d7)24 primer 50μM
RNA 稀释的 Poly-A Control
DEPC 水
体积(1-8 μg RNA) 2μl(100pmol)
1-8μg 2μl
到 12μl
体积(8.1-16 μgRNA) 2μl(100pmol) 8.1-16μg 2μl 到 11μl
3
Affymetrix表达谱芯片

Affymetrix表达谱芯片Affymetrix 人表达谱芯片服务芯片名称:Affymetrix GeneChip® Human Transcriptome Array 2.0 NEW!芯片介绍:Affymetrix 最新开发此款芯片为您提供支持所有转录本异构体的全转录组分析。
同时,针对超过40,000个非编码转录本的探针设计,还可帮助您对非编码RNA进行分析。
数据来源于RefSeq,En sembl,UCSC,MGC,nocode,lncRN db及Broad Institute, Human Body Map lincRNAs and TUCP catalog。
芯片名称:Affymetrix GeneChip® Human Genome U133 Plus 2.0 Array芯片介绍:此款芯片包含了47,000个转录本,代表了38,500个明确的人类基因。
数据库来源于GeneBank、dbEST、RefSeq、UniGene database(Build 159 January 25 2003)、Washington Universit y EST trace repository、NCBI human genome assembly(Build 31)。
芯片名称:GeneChip Human Gene 2.0 ST Array芯片介绍:Affymetrix 新一代全转录本芯片,一张芯片上可同时检测mRNA和lincRNA,在其上一代1.0 ST Array的基础上内容进一步丰富并更新了数据库。
全面覆盖转录组,可以同时检测>30,000个编码的转录本和>11,000个长链非编码转录本;探针设计最大程度覆盖所有外显子,可检测选择性拼接/转录本变体。
每个转录本覆盖特异探针的数目中值达到21种,每种探针的长度为25bp,这意味着每个转录本将被检测的碱基数中值高达525个,高重复性,高灵敏度。
基因芯片(Affymetrix)分析4:GO和KEGG分析

基因芯片(Affymetrix)分析4:GO和KEGG分析基因列表的分析一般都会涉及GO和KEGG分析,Bioconductor 提供了很多这方面的R工具包。
选择工作目录,读入上一次分析和保存的数据:1 获取AGI、GO和KEGG注释ath1121501GO为拟南芥基因的GO数据库,ath1121501PATH 为KEGG pathway数据库。
但不是每一个基因(probeset)都有GO 或KEGG注释,哪些基因有注释可以用mappedkeys函数获得:有PATH注释的probesets只有3018个,而有GO注释的有2万多个。
通过ath1121501XXXX获得的数据是AnnotationDbi软件包定义的ProbeAnnDbBimap类型数据,它们可以用as.list转成列表形式。
列表内每一个基因的注释内容也是列表形式:转换成列表类型的ProbeAnnDbBimap数据仍然是列表,但PATH和ACCNUM数据是二级列表(列表下只有一级列表),而GO 数据是三级列表(列表下还有两级的列表)。
所以得先编写get.GO函数,它把as.list产生的GO三级列表转成二级结构,和AGI和KEGG 的列表类似,方便后面的统一处理:使用这个函数和下列代码就可以获得AGI、GO和KEGG注释:上面代码有两点要注意:•switch()函数使用。
switch()是非常神奇的条件转向开关函数,它的参数(列表)可以是各种类型,变量、表达式、函数等都可以使用。
•列表到数据框类型数据的转换,我们使用了plyr软件包的llply 和ldply函数。
plyr是很著名的软件包,用于数据糅合。
这不属于本节的讨论范围,先不介绍,请自行学习使用。
由于探针id是唯一的,上面的代码用它作为关键字糅合数据。
得到的结果是数据框:这样每一个探针都得到了对应的AGI、GO和KEGG途径注释(如果有)。
其他类型数据如Pubmed ID可以使用类似方法获得,但编程之前得先了解它们的数据结构,最直接的方法就是使用head,summary和str等函数查看。
Affymetrix基因芯片产品培训课件(共30张PPT)

Human Gene 1.0 ST Array
36,079 32,020 2,967 579 513 466 21,014
Hu
40,
30,
5,6
996
3,4
11,
24,
EXON系列芯片
Design Statistics Suon Clusters Supported by Putative Full-Length mRNA Supported by Ensembl Transcripts Supported by EST Supported by Mouse or Rat mRNA Supported by Gene Prediction
microRNA芯片实验步骤
样本RNA抽提&质检
Poly (A) 加尾&FlashTag Biotin HSR 连接
生物素标记
芯片杂交-洗脱-扫描
芯片数据标准化
microRNA4.0特点
• 100%覆盖miRBase V20
• • • •
包括所有数据库记录的物种 人 2578 小鼠 1908 大鼠 728
芯片数据标准化
Gene系列芯片
Transcript coverage of the array
Total RefSeq transcripts covered NM ? RefSeq coding transcript, well-established annotation NR ? RefSeq non-coding transcript, well established annotation XM ? RefSeq coding transcript, provisional annotation XR ? RefSeq non- coding transcript, provisional annotation lincRNA transcripts RefSeq (Entrez) gene count
Affymetrix的农业基因组学解决方案

农业基因组学解决方案轻松、灵活、自动化的解决方案加速您的基因组选择和育种计划农业基因组学解决方案适合您所有研究阶段的平台2来自Affymetrix的农业基因组学解决方案为育种者和研究人员提供了一系列强大而灵活的基因分型工具,可经济高效地鉴定、验证并筛查植物和动物中的复杂遗传性状。
Affymetrix的遗传分析工具让您有能力:发现■通过遗传分析技术确定de novo遗传多样性■分析群体结构关联■鉴定与理想性状相关的遗传标记■确认标记-性状关联■了解对环境的遗传适应性管理■利用遗传信息来选取期望的结果■筛查植物和动物的理想性状基于芯片的基因分型的优点:经济■经济高效的基因分型工具简单■在单个技术平台上结合多个基因分型应用■轻松且简单的流程■几小时内即可获得准确的结果灵活■高通量的基因分型工具适合高密度或靶向基因分型应用■能基因分型所有您感兴趣的相关的标记■低样品量需求来自Affymetrix的基于芯片的基因分型产品为从全基因组分析到常规筛查的各种应用提供了完整的解决方案,且准确性和重复性最高、流程简单、成本最低。
3强大■对任何物种、任何基因组规模和任何倍性水平进行基因分型■Axiom ®分析可检测插入或缺失(indel)并保证包含所有候选SNP ,与相邻SNP 最近可达10 bp ,实现了更高效 的QTL 分析Axiom® 基因分型解决方案利用一种可靠的技术来加速表型性状关联性研究和基因筛选工作经过检测的基因型 或新发现的SNP定制设计咨询Axiom ®目录芯片和定制芯片每个样品1,500至260万个变异Axiom ®杂交样本制备方案手动和自动的选项可供选择Axiom ®试剂盒可靠的分析GeneTitan ®仪器无需动手的芯片处理和成像Genotyping Console™ 软件和Affymetrix ® PowerTools初步的基因分型 分析软件两个SNP 都能在Axiom 分析中基因分型Axiom ®基因分型解决方案为您提供多种芯片。
AFFYMETRIX基因芯片操作流程

AFFYMETRIX基因芯片操作流程1.设计芯片:根据研究需求,设计基因芯片的探针序列。
探针序列是一小段DNA或RNA序列,用于检测芯片上的特定基因。
通常,基因芯片上会有上万个探针,可以检测大量的基因。
2.提取RNA或DNA:从感兴趣的生物样本中提取总RNA或基因组DNA。
RNA或DNA提取的方法会根据具体的研究目的和样本类型而有所不同。
3.RNA/DNA标记:将提取的RNA或DNA样本进行标记。
在基因芯片研究中,常使用荧光标记的核苷酸来标记RNA或DNA。
标记的方法可以是直接标记或间接标记,具体选择取决于实验的设计和要求。
4.混合和杂交:将标记的RNA或DNA样本与设计好的探针序列混合,形成杂交溶液。
杂交时,样本中的标记的RNA或DNA会与芯片上的相应探针序列结合。
5.洗涤和扫描:对芯片进行洗涤去除杂质,然后使用芯片扫描仪对芯片进行扫描。
扫描会产生荧光图像,显示不同基因的表达水平或基因变异情况。
6.数据分析:使用专门的数据分析软件对扫描后的图像进行处理和分析。
这些软件可以提供丰富的数据分析工具,包括基因表达聚类、差异分析、通路分析等。
通过数据分析,可以得到关于基因表达的定量和质量信息。
7.结果解读:根据数据分析的结果,解读实验结果。
通过比较不同样本之间的基因表达差异,可以找到与研究目的相关的基因。
根据差异基因的功能注释和通路分析等,可以深入了解基因的作用和功能。
8. 结果验证:将一部分差异表达的基因进行验证实验,如RT-PCR、Northern blot等。
验证实验可以进一步确认基因表达的差异,并验证基因芯片分析的可靠性。
通过以上步骤,AFFYMETRIX基因芯片可以帮助研究人员高通量、高效率地研究基因的表达水平和基因变异等生物过程。
同时,数据分析和结果解读对于科研的深入和扎实也非常重要。
Affymetrix基因芯片在药物研发和个性化用药研究中的应用

·22·药物研发、个性化用药研究是基因芯片技术在药物开发中应用最为广泛的一个领域。
在药物靶点发现、药物作用机制、药物毒理评估研究中所使用的生物芯片主要是检测RNA水平的表达谱芯片;在个性化用药研究中,主要从人群的SNP入手,用SNP芯片或者再测序芯片评估个体的遗传背景是否适合使用某种药物,以及适合的剂量。
在当代药物开发过程中发现和选择合适的药物靶点是药物开发的第一步,也是药物筛选及药物定向合成的关键因素之一。
人体是一个复杂的网络系统,疾病的发生和发展必然牵涉到网络中的诸多环节。
当今严重威胁人类健康的心脑血管疾病、恶性肿瘤、老年性痴呆症和一些代谢紊乱疾病都是多因素作用的结果,往往不能简单归结于单一因素的变化。
DNA芯片可以从疾病及药物2个角度对生物体的多个参量同时进行研究以发掘药物靶点并同时获取大量其他相关信息。
因此可以说,在这种情况下,任何一元化的分析方法均不及 DNA芯片这种集成化的分析手段更具有优势。
1 药物靶点发现推荐产品:人类全基因组表达谱,外显子芯片,SNP芯片小鼠全基因组表达谱,外显子芯片大鼠全基因组表达谱,外显子芯片此外还有恒河猴,犬,猪,牛,果蝇,线虫,酵母等30余种模式生物的全基因组表达谱芯片。
1.1. 比较正常不同组织细胞中基因的表达模式基因的表达模式给它的功能提供了间接的信息。
例如只在肾脏中表达的基因就不大可能与精神分裂症有关。
一些药物的靶点是在整个身体中分布广泛的蛋白质,这类药物的不良反应往往比较大。
而选择只在特异组织中表达的蛋白作为药物筛选的靶点,可以减少药物对整体产生的不良反应,因而更引起人们的关注。
例如骨质疏松症(osteoporosis)与破骨细胞(osteoclast)的功能有关,破骨细胞可以破坏并吸收骨质,当骨质的形成与破坏出现不平衡的时候,就会导致骨质疏松症。
如果破骨细胞的功能得到抑制,那么就可以控制骨质疏松症的发生和发展。
利用已有的人类EST序列和DNAAffymetrix基因芯片在药物研发和个性化用药研究中的应用芯片技术,可以容易地得到只在破骨细胞中进行表达的基因,如编码半胱氨酸蛋白酶的cathepsink基因。
Affymetrix生物芯片简介
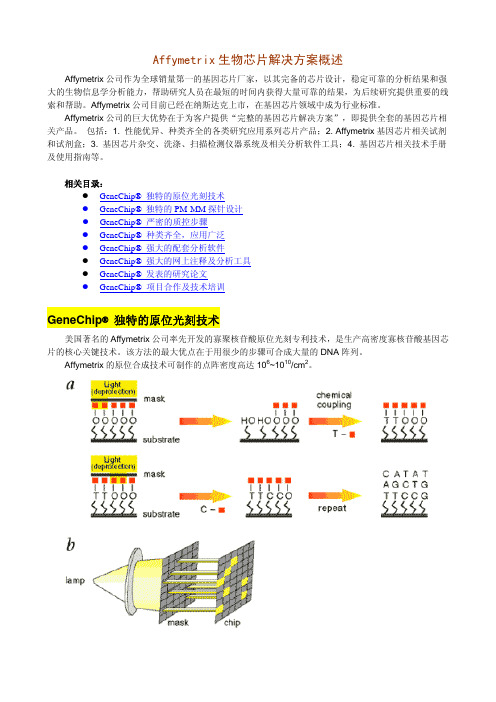
首先,使固相片基羟基化,并用光敏保护基团将其保护起来,然后选取适当的避光膜(mask)使需要 聚合的部位透光,其他部位不透光。这样,当光通过避光膜照射到支持物上时,受光部位的羟基就会发生 脱保护而活化,从而可以反应结合碱基。由于参与合成的碱基单体一端可以进行固相合成,另一端受光敏 基团的保护,所以原位合成后,可进行下一轮的光照、脱保护和固相合成。循环下去,不断改变避光膜的 透光位点,就可以实现在同一玻片上合成成千上万种预定序列的寡核苷酸探针。
GeneChip® 独特的原位光刻技术
美国著名的 Affymetrix 公司率先开发的寡聚核苷酸原位光刻专利技术,是生产高密度寡核苷酸基因芯 片的核心关键技术。该方法的最大优点在于用很少的步骤可合成大量的 DNA 阵列。
Affymetrix 的原位合成技术可制作的点阵密度高达 106~1010/cm2。
Affymetrix 公司的基因芯片产品在世界范围内已广泛应用于人类疾病的分子机理、肿瘤分型、药物筛 选、免疫学、信号转导、基因调控、细胞周期与凋亡、药物代谢与毒理、神经发育与分化、SNP 分析、细 菌基因组研究以及植物基因调控等各个方向,并取得了巨大的成功。
GeneChip® 强大的配套分析软件
GeneChip operating software version1.0 (简称 GCOS)
基因芯片(Affymetrix)分析2:芯片数据预处理

基因芯片(Affymetrix)分析2:芯片数据预处理基因芯片技术的特点是使用寡聚核苷酸探针检测基因。
前一节使用ReadAffy函数读取CEL文件获得的数据是探针水平的(probe level),即杂交信号,而芯片数据预处理的目的是将杂交信号转成表达数据(即表达水平数据,expression level data)。
存储探针水平数据的是AffyBatch类对象,而表达水平数据为ExpressionSet类对象。
基因芯片探针水平数据处理的R软件包有affy, affyPLM, affycomp, gcrma等,这些软件包都很有用。
如果没有安装可以通过运行下面R语句安装:Affy芯片数据的预处理一般有三个步骤:•背景处理(background adjustment)•归一化处理(normalization,或称为“标准化处理”)•汇总(summarization)。
最后一步获取表达水平数据。
需要说明的是,每个步骤都有很多不同的处理方法(算法),选择不同的处理方法对最终结果有非常大的影响。
选择哪种方法是仁者见仁智者见智,不同档次的杂志或编辑可能有不同的偏好。
1 需要了解的一点Affy芯片基础知识Affy基因芯片的探针长度为25个碱基,每个mRNA用11~20个探针去检测,检测同一个mRNA的一组探针称为probe sets。
由于探针长度较短,为保证杂交的特异性,affy公司为每个基因设计了两类探针,一类探针的序列与基因完全匹配,称为perfect match(PM)probes,另一类为不匹配的探针,称为mismatch (MM)probes。
PM和MM探针序列除第13个碱基外完全一样,在MM中把PM的第13个碱基换成了互补碱基。
PM和MM探针成对出现。
我们先使用前一节的方法载入数据并修改芯片名称:用pm和mm函数可查看每个探针的检测情况:上面显示的列名称就是探针的名称。
而基因名称用probeset名称表示:名称映射时会看到。
主要的基因芯片技术平台

PM探针
基因表达 开放
MM探针
Oligo arrays
20 µm
基因表达 关闭
多个检测结果可以参考
cDNA arrays
~ 150 µm
只有一个点的信号
Affymetrix 基因芯片的特点(6) 重复性高
同一样本, 不同批次的芯片
Signal Intensities: 13,400
Signal Intensities: 13,090
Sandwich Immunoassays
• highly parallel • high sensitivity • expensive equipment • labour intense • automation difficult
三、蛋白质芯片的制备
(一)、基质材料的选择和处理
➢ 聚丙烯酰胺凝胶 ➢ 聚偏二氟乙烯膜(PVDF) ➢ 硝酸纤维素膜 ➢ 载玻片 ➢ 硅片 ➢纸 ➢ 金等 ➢ 微珠
individual optical
fiber
fibers
• Bead diameter ~3 mm
• Bead center-to-center
distance ~5 mm—
highest feature density
• Bead surface is
derivatized with DNA
probes
第四部分,组织芯片
概
述
❖ 高通量
❖ 多组织标本
❖ 同时、平行分析
❖ 同时研究一个或多个基因或 蛋白的研究工具
❖ 高质、高效,省材,操作便 捷
❖ 应用广泛
• 成百甚至上千份 • 正常或处于疾病状态 • 疾病不同发展阶段 • FISH(荧光原位分子杂交) • mRNA ISH(原位杂交) • IHC(免疫组织化学)
affymetrix全基因组snp芯片检测

A f f y m e t r i x全基因组S N P芯片检测-CAL-FENGHAI-(2020YEAR-YICAI)_JINGBIANAffymetrix 全基因组 SNP 芯片检测单核苷酸多态性(single nucleotide polymorphism, SNP) 指基因组单个核苷酸的变异,它是最微小的变异单元,是由单个核苷酸对置换、颠换、插入或缺失所形成的变异形式。
单核苷酸多态性是基因组上高密度的遗传标志,在人类基因组中已发现的SNP数量超过3000万。
作为第三代遗传标记,SNP数量众多、分布密集、易于检测,因而是理想的基因分型目标。
SNP分型检测在疾病基因组(如疾病易感性),药物基因组(药效、药物代谢差异和不良反应)和群体进化等研究中具有重大意义。
在人研究方面,Affymetrix 公司有分别基于GeneChip和GeneTitan平台的 SNP 6.0 芯片和针对中国人群设计的CHB1&2 Array,既可用于全基因组SNP分析,又可用于CNV分析,极大地方便了中国人类疾病GWAS研究。
Affymetrix公司针对多个农业物种也开发了多款商品化的基因分型芯片,如鸡、牛、水牛、鲑鱼、水稻、小麦、辣椒、草莓等,为农业育种研究、遗传图谱构建、群体基因组学研究提供研究手段。
此外,Affymetrix公司还支持定制芯片,最低起订量为480个样品。
检测原理|?技术优势|?产品列表|?定制芯片|?数据分析|基于GeneChip平台的人SNP 6.0 芯片实验流程:?基于GeneTitan平台的Axiom基因分型芯片检测流程:从SNP原理谈SNP分析技术之SNP芯片日期:2012-05-21 ? ? 来源:网络标签:?SNP原理?SNP分析?SNP芯片摘要 :?SNP是近年来基因突变的热点研究之一。
它是指在单个的核苷酸上发生了变异,有四种不同的变异形式,而实际上只发生转换和颠换这两种。
当科学家弄清了SNP的突变原理以后,他们就着手对SNP进行分析,以求找到疾病相对应的突变位点或者是进行个性化药物治疗研究。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
MICRORNA4.0特点
• 100%覆盖miRBase V20
• 包括所有数据库记录的物种 • 人 2578 • 小鼠 1908 • 大鼠 728
• 重复性>0.95
• 技术重复 • 生物重复
LNCRNA芯片
• Humanቤተ መጻሕፍቲ ባይዱMouse/Rat Gene2.0 • HTA2.0(Human) • EG1.0-Human/Mouse
LNCRNA芯片实验流程
样本RNA抽提&质检 cDNA合成及片段化 生物素标记 芯片杂交-洗脱-扫描 芯片数据标准化
SNP/CNV芯片
• SNP6.0 • CytoScan 750K • CytoScan HD • OncoScan • 订制芯片
CYTO芯片适用样本
• 细胞 • 组织 • 血液 • 唾液 •骨
Total RefSeq transcripts covered
NM ? RefSeq coding transcript, well-established annotation
NR ? RefSeq non-coding transcript, well established annotation
HTA2
Array content Genes (transcript clusters) Transcripts Exons Exon clusters Splice junctions (probe sets)
Protein coding content 44,699 245,349 560,472 296,058 339,146
表达谱芯片探针设计
基因组DNA 初始转录本RNA
可变剪切转录本
Exon 1.0 ST Gene 1.0 ST 3’端表达谱
表达谱芯片实验步骤
样本RNA抽提&质检 cDNA合成及片段化 生物素标记 芯片杂交-洗脱-扫描 芯片数据标准化
GENE系列芯片
Transcript coverage of the array
XM ? RefSeq coding transcript, provisional annotation
XR ? RefSeq non- coding transcript, provisional annotation
lincRNA transcripts
RefSeq (Entrez) gene count
Background Subtraction Strategy
Total Features Per Array Interrogated Strand
1.4 million > 1 million 289,961 probe sets 306,583 probe sets 665,175 probe sets 220,262 probe sets 883,105 probe sets Along the entire length of the transcripts 4 Median intensity of up to 1,000 background probes with the same GC content >5,500,000 Sense²
表达谱芯片
• 3’IVT
• Human U133 Plus 2.0 • Mouse U430 2.0 • Rat U230 2.0 • PrimeView&Almac Xcel
• WT
• Human/Rat/Mouse Gene1.0 ST • Human/Rat/Mouse Exon1.0 ST • HTA2.0
AFFYMETRIX基因芯片产品
5”
5”
Up to ~7,000,000 features / chip
1.28 cm
Hybridized probe cell
RNA target
Oligonucleotide
probe
*
*
*
*
*
*
5-11µm
Raw data image with 1-6.9
million different complementary probes
MICRORNA芯片
• MicroRNA 4.0
• microRNA • Pre-microRNA • snoRNA&scaRNA
MICRORNA芯片实验步骤
样本RNA抽提&质检 Poly (A) 加尾&FlashTag Biotin HSR 连接 生物素标记 芯片杂交-洗脱-扫描 芯片数据标准化
Human Gene 1.0 ST Array 36,079 32,020
2,967
579
513 466 21,014
Human Gene 2.0 ST Array 40,716 30,654
5,638
996
3,428 11,086 24,838
EXON系列芯片
Design Statistics Summary Probe Sets Exon Clusters Supported by Putative Full-Length mRNA Supported by Ensembl Transcripts Supported by EST Supported by Mouse or Rat mRNA Supported by Gene Prediction Probe Selection Region Probes/Probe Selection Region
Thousands of copies of a specific oligonucleotide probe in each feature
芯片产品分类
• 表达谱芯片-mRNA
• 3’IVT • WT
• MicroRNA芯片 • lncRNA芯片 • SNP/CNV芯片 • 甲基化&转录因子芯片
Non-protein coding content 22,829 40,914 109,930 82,444 0
表达谱芯片应用
• U133——经典,相对旧,无价格优势,不做推荐 • Gene系列——基本,仅满足一般表达量检测需求 • Exon系列——全面,精确表达量检测 • HTA2——最佳,最全&最精确表达量 • EG1.0——最佳,唯一能够获得转录本表达量
