Thermo Scientific限制性内切酶一览表
常见限制性内切酶识别序列

常见限制性内切酶识别序列(酶切位点)(BamHI、EcoRI、HindII I、NdeI、XhoI等)Time:2009-10-22 PM 15:38Author:bioerHits: 7681 times在分子克隆实验中,限制性内切酶是必不可少的工具酶。
无论是构建克隆载体还是表达载体,要根据载体选择合适的内切酶(当然,使用T 载就不必考虑了)。
先将引物设计好,然后添加酶切识别序列到引物5' 端。
常用的内切酶比如Bam HI、EcoRI、HindII I、NdeI、XhoI等可能你都已经记住了它们的识别序列,不过为了保险起见,还是得查证一下。
下面是一些常用的II型内切酶的识别序列,仅供参考。
先介绍一下什么是II型内切酶吧。
The Type II restri ction system s typica lly contai n indivi dualrestri ction enzyme s and modifi catio n enzyme s encode d by separa te genes. The Type II restri ction enzyme s typica lly recogn ize specif ic DNA sequen ces and cleave at consta nt positi ons at or closeto that sequen ce to produc e 5-phosph atesand 3-hydrox yls. Usuall y they requir e Mg 2+ ions as a cofact or, althou gh some have more exotic requir ement s. The methyl trans feras es usuall y recogn ize the same sequen ce althou gh some are more promis cuous. Threetypesof DNA methyl trans feras es have been foundas part of Type II R-M system s formin g either C5-methyl cytos ine, N4-methyl cytos ine or N6-methyl adeni ne.酶类型识别序列ApaIType II restri ctionenzyme5'GGGCC^C 3'BamHIType II restri ctionenzyme5' G^GATCC3'BglIIType II restri ctionenzyme5' A^GATCT3'EcoRIType II restri ctionenzyme5' G^AATTC3'HindII IType II restri ctionenzyme5' A^AGCTT3'KpnIType II restri ctionenzyme5' GGTAC^C 3'NcoIType II restri ctionenzyme5' C^CATGG3' NdeIType II restri ctionenzyme5' CA^TATG 3'NheIType II restri ctionenzyme5' G^CTAGC3'NotIType II restri ctionenzyme5' GC^GGCCGC 3'SacIType II restri ctionenzyme5' GAGCT^C 3'SalIType II restri ctionenzyme5' G^TCGAC3' SphIType II restri ctionenzyme5' GCATG^C 3'XbaIType II restri ctionenzyme5' T^CTAGA3'XhoIType II restri ctionenzyme5' C^TCGAG3'要查找更多内切酶的识别序列,你还可以选择下面几种方法:1. 查你所使用的内切酶的公司的目录或者网站;2. 用软件如:Primer Premie r5.0或Bioe dit等,这些软件均提供了内切酶识别序列的信息;3. 推荐到NEB的REBA SE数据库去查(网址:http://rebas/rebase/rebase.html)当你设计好引物,添加上了内切酶识别序列,下一步或许是添加保护碱基了,可以参考:NEB公司网站提供的关于设计PC R引物保护碱基参考表下载(也可见图片)双酶切buf fer的选择(MBI、罗氏、NEB、Prom eg a、Takara)再给大家推荐一种新的不需要连接反应的分子克隆方法,优点包括:①设计引物不必考虑选择什么酶切位点;②不必考虑保护碱基的问题;③不必每次都选择合适的酶来酶切质粒制备载体;④而且不需要D NA连接酶;⑤假阳性几率低(因为没有连接反应这一步,载体自连的问题没有了)。
限制性核酸内切酶

限制性核酸内切酶限制性核酸内切酶:是识别DNA的特异序列,并在识别位点或其周围切割双链DNA的一类内切酶。
限制性核酸内切酶的分类:依照限制酶的结构,辅因子的需求切位与作用方式,可将限制酶分为三种类型,别离是第一型(Type I)、第二型(Type II)及第三型(Type III)。
第一型限制酶同时具有修饰(modification)及认知切割(restriction)的作用;还有认知(recognize)DNA 上特定碱基序列的能力,通常其切割位(cleavage site)距离认知位(recognition site)可达数千个碱基之远,并非能准确信位切割位点,因此并非经常使用。
例如:EcoB、EcoK。
第二型限制酶只具有认知切割的作用,修饰作用由其他酵素进行。
所认知的位置多为短的回文序列(palindrome sequence);所剪切的碱基序列通常即为所认知的序列。
是遗传工程上,有效性较高的限制酶种类。
例如:EcoRI、HindIII。
第三型限制酶与第一型限制酶类似,同时具有修饰及认知切割的作用。
可认知短的不对称序列,切割位与认知序列约距24-26个碱基对,并非能准确信位切割位点,因此并非经常使用。
例如:EcoPI、HinfIII。
限制酶在遗传学方面的应用:1、在甚因工程方面利用能产生“粘性结尾”的限制酶, 进行DNA的体外重组, 是较为方便的, 只要用同一限制酶处置不同来源的DNA, 由于所产生的水解片段具有相同的粘性结尾, 能够彼此“粘合”,再经连接酶处置, 就成为重组DNA分子了. 目前, 基因工程上, 限制酶要紧应用于以下两方面(1)目的基因与载体的重组细菌细胞中的限制酶能水解外源DNA , 因此必需通过适当的载体(质粒或噬菌体)的帮忙才能将外源DNA引人受体细胞并在其中增殖和表达。
将供体DNA与载体用一样的限制酶处置, 使载体带上各类各样的外源DNA片断, 然后引人受体细菌细胞增殖, 菌细胞增殖, 再挑选出所需的菌株, 便取得带有某一目的基因的繁衍系.用这种方式, 已成功地将酵母菌的咪哇甘油磷酸脱水酶基因、夕一异丙基苹果酸脱氢酶基因和色氨酸合成酶基因通过几噬菌体转人大肠杆菌,并表达了信息.(2)建造新的基因载体作为基因载体,在引人受体细胞后, 必需有较高的复制率, 以求取得大量的基因产物;必需具有一个选择性标志, 以便挑选;还要有一最多种限制酶的作用位点(每种酶只有一个切口);也要求利用平安。
限制性内切酶的说明

限制酶使用说明一、分类目前,已被发现的限制酶,根据其反应的必须因子和切断点等特性,被分为以下三大类:类别反应必须因子切点酶例 I 型 s-腺苷基蛋氨酸、ATP、Mg2+识别部位和切点不同,切断部位不定Eco B、Eco KII 型 Mg2+切断识别部位或其附近的特定部位Eco R I、Bam H I III 型 ATP、Mg2+识别部位和切点不同,但切断特定部位Eco P I、Hin f III 应用于基因工程研究用的限制酶,一般全是II型酶,现在市场上销售的酶都属于II型酶, 这些限制酶由于其反应条件和底物DNA种类的不同,其切断状况及出现Star活性的频率等各有不同,并且其程度也根据酶的不同而千差万别。
因而在使用限制酶时,必须对这些要素充分注意,确保目标序列的切断反应能顺利进行,下面具体介绍一下使用限制酶时的一些注意点。
二、注意事项1. 甲基化的影响从带有DNA甲基化酶基因的宿主菌中制备的DNA,其碱基的一部分已经被甲基化,因此即便使用能够识别、切断被甲基化部分的序列的限制酶,也几乎无法切断被甲基化的部分。
被甲基化的部位,根据底物DNA及宿主种类的不同而不同。
例如宿主菌为大肠杆菌的情况下,根据宿主的种类有以下两种情况:在进行转化时,通常使用的菌株为C600、HB101、JM109等,因为都带有dam、dcm甲基化酶,所以使用这些菌株制备的DNA时,必须注意。
另外,动物由来的DNA,CG序列多为5m CG;植物由来的DNA,CG及CNG序列多为5m CG和5m CNG。
2. Star活性限制酶在一些特定条件下使用时,对于底物DNA的特异性可能降低。
即可以把与原来识别的特定的DNA序列不同的碱基序列切断,这个现象叫Star活性。
Star活性出现的频率,根据酶、底物DNA、反应条件的不同而不同,可以说几乎所有的限制酶都具有Star活性。
并且,它们除了识别序列的范围增大之外,还发现了在DNA的一条链上加入切口的单链切口活性,所以为了极力抑制Star活性,一般情况下,即使会降低反应性能,我们也提倡在低甘油浓度、中性pH、高盐浓度条件下进行反应。
限制性内切酶

特征和种类
1.限制与修饰现象 早在 50 年代初,有许多学者发现了限制与修饰现象,当时称作寄主 控制的专一性(host controlled specificity)。 l 噬菌体表现的现 象便具有代表性和普遍性,其在不同宿主中的转染频率可明这一问题 (表 2-1)。 l 在感染某一宿主后,再去感染其它宿主时会受到限制。 E.coli 菌株 λ噬菌体感染率 lK lB lC E.coli K 1 10-4 10-4 E.coli B 10-4 1 10-4 E.coli C 1 1 1 说明 K 和 B 菌株中存在一种限制系统,可排除外来的 DNA 。 104 的存活率是由宿主修饰系统作用的结果,此时限制系统还未起作用。 而在 C 菌株不能限制来自 K 和 B 菌株的 DNA 。限制作用实际就是限 制酶降解外源 DNA ,维护宿主遗传稳定的保护机制。甲基化是常见的 修饰作用,可使腺嘌呤 A 成为 N6 甲基-腺膘呤,胞嘧啶 C 成为 5' 甲 基胞嘧啶。通过甲基化作用达到识别自身遗传物质和外来遗传物质的目 的。
Ⅰ 型(type Ⅰ)限制与修饰系统的种类很少,只占 1% ,能识别 专一的核苷酸顺序,并在识别点附近的一些核苷酸上切割DNA分子中的 双链,但是切割的核苷酸顺序没有专一性,是随机的。如 EcoK 和 EcoB。其限制酶和甲基化酶 (即 R 亚基和 M 亚基) 各作为一个亚基 存在于酶分子中,另外还有负责识别 DNA 序列的 S 亚基,分别由 hsdR、hsdM 和 hsdS 基因编码,属于同一操纵子(转录单位)。 EcoK 编码基因的结构为 R2M2S。 EcoB 编码基因的结构为 R2M4S2 。 EcoB 酶的识别位点如下,其中两条链中的 A 为甲基化位点, N 表示任意碱基。 TGA*(N)8TGCT EcoK 酶的识别位点如下,其中两条链中的 A 为可能的甲基化位点。 AA°C(N)6GTGC 但是 EcoB 酶和 EcoK 酶的切割位点在识别位点 1000bp 以外, 且无特异性。 Ⅲ 型(type Ⅲ)限制与修饰系统的种类更少,所占比例不到 1% , 也有专一的识别顺序,但不是对称的回文顺序。它在识别顺序旁边几个 核苷酸对的固定位置上切割双链。但这几个核苷酸对则是任意的,如 EcoP1 和 EcoP15 。它们的识别位点分别是 AGACC 和 CAGCAG , 切割位点则在下游 24-26bp 处。 在基因操作中,一般所说的限制酶或修饰酶,除非特指,均指 Ⅱ 型 系统中的种类。
限制性内切酶

II型限制性内切酶
首先由H.O. Smith和K.W. Wilcox在 1970年从流感嗜血菌Rd菌株中分离出来。
作用原理:识别双链DNA上未甲基化修饰的 一 小段明确的序列(多数是回文序列),然 后在识别位点之内的特定位置切割。
• 一般性状
对热不稳定,通常是溶于含有50%甘油的缓冲 液中贮存于–20℃环境下。取出使用时必须立即置 于冰浴中。NaCl有抑制作用,能被Mg2+激活,巯 基有保护作用,
I 型:切割位点不确定,不适用于基因工程。 Ⅱ型:基因工程的工具酶。 Ⅲ型:切割位点不在识别位点,对分子克隆操作
亦无实用意义。
I型限制性内切酶
首先由M.Meselson和R.Yuan在1968年从 大肠杆菌B株和K株分离得到。
作用原理:识别双链DNA未甲基化修饰的特 异序列,与该序列相互作用,然后延DNA分 子移动,在距离特异性识别位点约10001500bp处随机切开一条单链,造成大约75 个核苷酸的切口。
不同的限制性内切酶的最适反应温度不同。大 多数是37oC,少数要求40-65oC。
酶
Apa I Bcl I Mae II Taq I
最适 温度oC
30 50 50 65
酶
Apy I BstE II Mae III
最适 温度oC
30 60 55
酶
Ban I Mae I Sma I
最适温 度oC
50 45 25
ATP、 Mg2+和SAM
Mg2+
ATP、Mg2+ 和SAM
EcoB: 寄主特异性位 TGA(N)8TGCT
点识别序列 EcoK: AAC(N)6GTGC
回文序列 (IIs型除外)
限制性核酸内切酶

2. DNA的甲基化程度
大肠杆菌一般有两种甲基化酶修饰质粒 dam甲基化酶(修饰GATC中的A); dcm甲基化酶(修饰CCA/TGG的C)。
基因工程中必须使用甲基化酶失活突变 的菌株。
3. 温度
齐平末端:在识别序列内的对称轴上切割,其 切割产物具平头末端(不易重新连接)。
粘性末端:在识别序列2条链对应位上错位切割, 有5’端突出的粘性末端和3’端突出的粘性末端。
粘性末端的意义:粘性末端突出的单链部分可以与相同的酶或 同尾酶切割得到的粘性末端的单链部分互补配对。
粘性末端和平齐末端的动画对比
四 限制性内切酶的识别序列
• EcoRⅠ的识别序列 GAATTC
•
CTTAAG
• Hind Ⅲ的识别序列 AAGCTT
•
TTCGAA
• BamHⅠ的识别序列 GCATCC
•
CCTAGG
• 从以上举例的各种限制性酶切酶的识别序列看出, 它们具有共同的规律性,呈旋转对称或二重互补 对称。
• 有的限制性酶切酶可识别两种以上的核酸 序列。
不同的限制性内切酶的最适反应温度不同。大 多数是37oC,少数要求40-65oC。
酶
最适 温度oC
酶
最适 温度oC
酶
Apa I 30 Apy I 30 Ban I Bcl I 50 BstE II 60 Mae I Mae II 50 Mae III 55 Sma I Taq I 65
最适 温度 oC
特性
限制和修饰活 性
内切酶的蛋白 结构
I类内切酶 单一多功能的酶 3种不同的亚基
限制性内切酶酶切位点汇总

ApaI识别位点Acc65I识别位点ApaLI识别位点AccI识别位点ApeKI识别位点AciI识别位点ApoI识别位点AclI识别位点AscI识别位点AcuI识别位点AseI识别位点AfeI识别位点AsiSI识别位点AflII识别位点AvaI识别位点AflIII识别位点AvaII识别位点AgeI识别位点AvrII识别位点AhdI识别位点BaeI识别位点AleI识别位点BamHI识别位点AluI识别位点BanI识别位点AlwI识别位点BanII识别位点AlwNI识别位点BmrI识别位点BbvCI识别位点BmtI识别位点BbvI识别位点BpmI识别位点BccI识别位点Bpu10I识别位点BceAI识别位点BpuEI识别位点BcgI识别位点BsaAI识别位点BciVI识别位点BsaBI识别位点BclI识别位点BsaHI识别位点BfaI识别位点BsaI识别位点BfuAI识别位点BsaJI识别位点BglI识别位点BsaWI识别位点BglII识别位点BsaXI识别位点BlpI识别位点BseRI识别位点Bme1580I识别位点BseYI识别位点BmgBI识别位点BspMI识别位点BsiEI识别位点BspQI识别位点BsiHKAI识别位点BsrBI识别位点BsiWI识别位点BsrDI识别位点BslI识别位点BsrFI识别位点BsmAI识别位点BsrGI识别位点BsmBI识别位点BsrI识别位点BsmFI识别位点BssHII识别位点BsmI识别位点BssKI识别位点BsoBI识别位点BssSI识别位点Bsp1286I识别位点BstAPI识别位点BspCNI识别位点BstBI识别位点BspDI识别位点BstEII识别位点BspEI识别位点BstNI识别位点BspHI识别位点DdeI识别位点BstXI识别位点DpnI识别位点BstYI识别位点BstZ17I识别位点DpnII识别位点Bsu36I识别位点DraI识别位点BtgI识别位点DraIII识别位点BtgZI识别位点DrdI识别位点BtsCI识别位点EaeI识别位点BtsI识别位点EaeI识别位点Cac8I识别位点EagI识别位点ClaI识别位点EarI识别位点CspCI识别位点EciI识别位点CviAII识别位点EcoNI识别位点CviKI-1识别位点EcoO109I识别位点CviQI识别位点EcoP15I识别位点EcoRI识别位点EcoRV识别位点FatI识别位点FauI识别位点Fnu4HI识别位点FokI识别位点FseI识别位点FspI识别位点HaeII识别位点HaeIIIHgaI识别位点HhaI识别位点HincII识别位点HindIII识别位点HinfI识别位点HinP1I识别位点HpaI识别位点HpaII识别位点HphI识别位点Hpy188I识别位点Hpy188III识别位点Hpy99I识别位点HpyAV识别位点HpyCH4III识别位点HpyCH4IV HpyCH4V识别位点KasI识别位点KpnI识别位点MboI识别位点MboII识别位点MfeI识别位点MluI识别位点MlyI识别位点MmeI识别位点MnlI识别位点MscI识别位点MseI识别位点MslI识别位点MspA1I识别位点MspI识别位点MwoI识别位点NaeI识别位点NarI识别位点NciI识别位点NdeI识别位点NgoMIV识别位点NheI识别位点NlaIII识别位点NlaIV识别位点NmeAIII识别位点NotI识别位点NruI识别位点NsiI识别位点NspI识别位点PacI识别位点PaeR7I识别位点PciI识别位点PflFI识别位点PflMI识别位点PhoI识别位点PleI识别位点PmeI识别位点PmlI识别位点PpuMI识别位点PshAI识别位点PsiI识别位点PspGI识别位点PspOMI识别位点PspXI识别位点PstI识别位点PvuI识别位点PvuII识别位点RsrII识别位点SacI识别位点SacII识别位点SalI识别位点SapI识别位点Sau3AI识别位点Sau96I识别位点SbfI识别位点ScaI识别位点ScrFI识别位点SexAI识别位点SfaNI识别位点SfcI识别位点SfiI识别位点SfoI识别位点SgrAI识别位点SmaI识别位点SmlI识别位点SnaBI识别位点SpeI识别位点SphI识别位点SspI识别位点StuI识别位点StyD4I识别位点StyI识别位点SwaI识别位点TaqαI识别位点TfiI识别位点TseI识别位点Tsp45I识别位点Tsp509I识别位点TspMI识别位点TspRI识别位点Tth111I识别位点XbaI识别位点XcmI识别位点XhoI识别位点XmaI识别位点XmnI识别位点ZraI识别位点。
常用限制性内切酶和DNA聚合酶外文字符的规范编排

常用限制性内切酶和DNA聚合酶外文字符的规范编排一、限制性内切酶限制酶的命名,1973年由Smith和Nathans提出:用属名的头1个字母和种名的头2个字母,组成3个字母的略语,表示寄主菌的物种名称;如有菌株名,再加上一个字母,即第4个字母表示菌株;1种特殊的菌株,具有几个不同的限制与修饰体系,则以罗马数字表示在该菌株中发现某种酶的先后次序。
例如:Eco R I, 第1个大写字母E为大肠杆菌Escherichia coli的属名的第1个字母;第2、3两个小写字母co为其种名的2个字母;第4个字母大写R表示所用大肠杆菌的菌株编号。
再如,流感嗜血菌(Haemophilus influenzae)用Hin表示。
例如,从流感嗜血杆菌d株(Haemophilus influenzad d)中先后分离到3种限制酶,则分别命名为Hin d I、Hin d Ⅱ和Hin d Ⅲ。
限制性内切酶符号前3个字母为其来源菌株的拉丁名缩写形式,根据现有的外文字符使用规范,“生物学中属以下(含属)的拉丁文学名要用斜体字母”表示,所以,前3个字母用斜体形式编排。
第4个字母表示菌株,一般表示菌株的字母都用正体形式。
二、耐热DNA聚合酶DNA聚合酶是PCR反应的重要工具。
同限制性内切酶命名方法类似,耐热DNA聚合酶也与其来源菌名称有关。
例如:Taq DNA聚合酶、Pfu DNA聚合酶、Tth DNA聚合酶等,前3个字母都是表示来源于生物的,第1个字母是其来源生物的属名的首字母,第2、3个字母是其种名前的2个字母。
因此,这3个字母都应该用斜体,并且首字母要用大写;因为Taq DNA聚合酶是从一种水生栖热菌(Thermus aquaticus)yT1株分离提取的,Pfu DNA聚合酶是来自耐热菌Pyrococcus furiosus的一种DNA聚合酶,Tth DNA聚合酶是来自嗜热细菌Thermus thermophilus的一种DNA聚合酶。
常见限制性内切酶识别序列

常见限制性内切酶识别序列(酶切位点)(BamHI、EcoRI、HindIII、NdeI、XhoI等)Time:2009-10-22 PM 15:38Author:bioer Hits: 7681 times在分子克隆实验中,限制性内切酶是必不可少的工具酶。
无论是构建克隆载体还是表达载体,要根据载体选择合适的内切酶(当然,使用T 载就不必考虑了)。
先将引物设计好,然后添加酶切识别序列到引物5' 端。
常用的内切酶比如BamHI、EcoRI、HindIII、NdeI、XhoI等可能你都已经记住了它们的识别序列,不过为了保险起见,还是得查证一下。
下面是一些常用的II型内切酶的识别序列,仅供参考。
先介绍一下什么是II型内切酶吧。
The Type II restriction systems typically contain individual restriction enzymes and modification enzymes encoded by separate genes. The Type II restriction enzymes typically recognize specific DNA sequences and cleave at constant positions at or close to that sequence to produce 5-phosphates and 3-hydroxyls. Usually they require Mg 2+ ions as a cofactor, although some have more exotic requirements. The methyltransferases usually recognize the same sequence although some are more promiscuous. Three types of DNA methyltransferases have been found as part of Type II R-M systems forming either C5-methylcytosine, N4-methylcytosine or N6-methyladenine.酶类型识别序列ApaIType II restrictionenzyme5'GGGCC^C 3'BamHIType II restrictionenzyme5' G^GATCC 3'BglIIType II restrictionenzyme5' A^GATCT 3'EcoRIType II restrictionenzyme5' G^AATTC 3'HindIIIType II restrictionenzyme5' A^AGCTT 3'KpnIType II restrictionenzyme5' GGTAC^C 3'NcoIType II restrictionenzyme5' C^CATGG 3'NdeIType II restrictionenzyme5' CA^TATG 3'NheIType II restrictionenzyme5' G^CTAGC 3'NotIType II restrictionenzyme5' GC^GGCCGC 3'SacIType II restrictionenzyme5' GAGCT^C 3'SalIType II restrictionenzyme5' G^TCGAC 3'SphIType II restrictionenzyme5' GCATG^C 3'XbaIType II restrictionenzyme5' T^CTAGA 3'XhoIType II restrictionenzyme5' C^TCGAG 3'要查找更多内切酶的识别序列,你还可以选择下面几种方法:1. 查你所使用的内切酶的公司的目录或者网站;2. 用软件如:Primer Premier5.0或Bioedit等,这些软件均提供了内切酶识别序列的信息;3. 推荐到NEB的REBASE数据库去查(网址:/rebase/rebase.html)当你设计好引物,添加上了内切酶识别序列,下一步或许是添加保护碱基了,可以参考:NEB公司网站提供的关于设计PCR引物保护碱基参考表下载(也可见图片)双酶切buffer的选择(MBI、罗氏、NEB、Promega、Takara)再给大家推荐一种新的不需要连接反应的分子克隆方法,优点包括:①设计引物不必考虑选择什么酶切位点;②不必考虑保护碱基的问题;③不必每次都选择合适的酶来酶切质粒制备载体;④而且不需要DNA连接酶;⑤假阳性几率低(因为没有连接反应这一步,载体自连的问题没有了)。
常见分子试验限制性内切酶酶切位点大全

Alul 识别位点5: . AG T CT ...T 3:. TCJSA...^Aflll 识别位点Acil 识别位点孑...此GC (3)p J . ..GGG^G. ,.5JAcll 识别位点$…从七US杠…TTGC^AA …hAlwl 识别位点5 ..GljATC<Nl7 3 3 . ,. C C T A G (N),A 5'Acul 识别位点5. . CTGA AGfN), ▼…h3:」GACT TCfN)^.,. 5fAlwNl 识别位点e^XAGMN^TG,, 3 3:..GTC b MNNGAC (5)Afel 识别位点..AGCGCT .. .3P ..TCGCGA b ..5JApal 识别位点亍…GGGC 此.33\,CCCGGG...5JApaLl 识别位点3:..CAGGT i G...SAfllll 识别位点 片…M G R¥GT,・3b : - .TG^RQA …5"ApeKl 识别位点F …GtwGC …可 3\.CGWCJ5.._5]Aatll 识别位点Agel 识别位点员・ ^/T CCGGT ^.O -3\. TGGCC^A ...S^Acc65l 识别位点*:・.GT W KAC 」■丁 P :.. GA KMJG. ..5H Ahdl 识别位点5; ,G ACNN IvfNNGTC 3:..CTGNh!i NNNCAG (5)Accl 识别位点 *:・・ GTW KA C _・3 忆…GA KMJG. ..5Alel 识别位点5 ,TxACNffflNQTO- 7?3H 3 .GTGN\MNCAC 5"Bell识别位点Banl识别位点S^.CCRY^G.-.S^ Banll识别位点Bael识别位点Bcgl识别位点5:爲㈣CKgTGGW 3J 3:.屮㈣GUT(g2®N城” S BciVl识别位点5. ..GTATCCC帖3 . ,. C A 7 AGG(N}^. 5Apol识别位点&…P TA AT T J.y 3:.. YTTAA^R ..5JAscI识别位点S .^GG^GCGCC ・・・3 3J CCGCGCJ3G. ,.5JA Asel识别位点y…AfT AAT ■二住. T AATJA.,,5^AsiSI识别位点$: GRGGe -3”a^.c^ycGAG.-.s*Bbsl识别位点5. ,.GAAGAC(M).T.3" 3: ..CTTCTG{N}ti. . . 5^BbvCl识别位点亍…CtfTCAGC …IT 『…GGAG.CG…h『…GCGAT^GC..・3 p.. CGtyAGCG. . 5Aval识别位点Bbvl识别位点5 .GC AGC(M)D T. .3^ 3. . CGTCGiM^^-.S'CVCGRG…丁p;. G AGCY.C.,,5'Avail识别位点F…(SfeWGC二3" 『…ccwqp…hAvril识别位点忙,比TdGG ,一鼾..GGATGJD (5)Bccl识别位点5 .CCATC(N}f 3 3; .GGTAG(hl). 5ABceAl识别位点5. . . ACGGCIN)!* .3 3:…TGGCG(M)%f ^(fi>AC<N)J GTAYC㈣「3”P^(N)TG(N)4CATRG(叽圄BamHl 识别位点F…G%ATCC・・8 p:,.CCT AG^S.. .5'Bmtl 识别F …TG ATCA ・..3” P J ... ACTAGJ...5JBfal 识别位点&... <Tl AG ….3T 3:,.GAT ;C. ,.5JBfuAI 识别位点5 ACCTGC(N)7 3 :T ,.TGG ACG(N)… . . .5「ABgll 识别位点3 CGGI^NNNNCCG.. .5Bglll 识别位点罚 AT GT …丁 b …TCT*磬…甲Blpl 识别位点|5 …GC T TNAGC …IT 0- CG AN LCG .5'*Bme1580l 识别位点IS. ..GKGCM't , ..3_P^. . <^MCGKG .6BmgBl 识别位点|5\ ,.CAC T GTC.,.3J 忙…GTOf AO …hBmrl 识别位点. ACTGGG(N} T y3: .. TGACCC(N),^..^5;.,,GCTAGC,.・宙 3:. CJ J ATCG...5*Bpml 识别位点5:…CTGEAG(W …3" 3 . ..GACC1. .5'Bpu10l 识别位点酣…GC T TFJ AGC ■…丁3GG Ahl 匚8 …5,BpuEl 识别位点5. t CTTGAQ(M),7.3 3. .. G AACTC(M),^ hBsaAl 识别位点5; , YAC^GTR, ..3' 3: RT(\CAY. . 5^BsaBl 识别位点5; ,G^TNN>JNATC, -33J . CTANNJNNTAG .5'BsaHl 识别位点…GFTtG¥C 、・.3‘ :T.KYGQRG …5’Bsal 识别位点5r . .GGTCTCCM)/ 33. ^CCAGAGCM)^. . ,5HBsaJl 识别位点5:.任NNGG …了3 GGN忖Cf …5「Bmtl识别BsmA I 识别BsaXI 识别位点b J ..\(N)AC(N)^CTCC(«)|, 3y . /NjTGfN^GAGGCN). . 5rBseRI 识别位点|5\ .G AGGAG(N)r T .富 p j . ,.CTCCTC(N)lk ..5-BseYI 识别位点 厂氏G g C …3 卜…GGGT^p …£Bsgl 识别位点..GTGCAG(N}(7 3 3 . . C ACGTciN},^. , .5BsiEI 识别位点CGRv'fcG. .3 3 GCJfRGC. ,5JBsiHKAI 识别位点5:..GWGCWt ・・ + 3" 茁…妄WCGWG.…扩BsiWI 识别位点 耳讯 T R CG 773P'.. GC ATfi/:…h5: . GTCTC (谢一…才 3 . . .C AG AG Nh ■…亍BsmBI 识别位点5 ..CGTCTC(N)7 .3^ 3 . ..GCAGAG (NL ^ …5rBsmFI 识别位点亍…GGG AC^N),7 -3* 書…CCCTG^^. .,5rBsmI 识别位点k ;lh 0AATQCN T \3 T.. XTT ACGN■BsoBI 识别位点F …CGG4G …丁 3:..GRGC^C..5JBsp1286l 识别位点^TTGDGCrffc.rTr3: 一 ・ qHGGDG …5’BspCNI 识别位点5., .CTCAG(N)T 5 ,.y anf1 & ..CTCAGCNJ/7 3: . .G AG TC (N)^ _.53. . ,GAGTCfN>_ . 5_BspDI 识别位点t ;,T AftGAT (ip 3^TAGCJA.-.S^BslI识别位点P:, CC NN NWhT(4NGG. ..3^ 3d GGNN NNNMNCC . ..5F BspEI识别位点5;.. ffcCGGA …丁BssKI识别位点BstZ17l识别位点|5•…ft A T G A . h3^(3: . . AGTAGJ . . 5JBspMI识别位点MCCTGC(N)/. -T4TGG ACG(N\,^,.^ BspQI识别位点S\..GCTCTTC*^T..r S:.XS AGAAG(N^..&'BsrBI识别位点5 - cc(stfcr^33J. .. GGC^AG..・hBsrDI识别位点5 GC AATGHN T,,3^}…CGTTAqNN …hBsrFI识别位点fc;. R'tCGGV..^' 护…YGGcqp…yBsrGI识别位点|sT7 f^ifACA f +.3l p:-, AGATGJ ..5 ・BsrI识别位点S:. . AC TGG . 3*3:. TG ACJ3N …hBssHII识别位点p:. R CNGG…;r3:.i GGNCq t...5,BssSI识别位点S…WZGZ、、S3:_GTGG冬…5’BstAPI识别位点5;. r GCAMNNbTNlGC ,, 3J 3J,..CGTN L NNNNACG....5,JkBstBI识别位点F…TTtGAA …3" 卜…AAGCJT..6BstEII识别位点y…Gt TH AG G …丁扩……5’BstNI识别位点匚ftfwGG …丁3'. GG\\CC. 5^BstUI识别位点GGtG,…丁3:.,GqpCJ …5’BstXI识别位点6;, .CCANNMN^NTGG.T.a 3. .GGTN^NMNNNACC 5BstYI识别位点BssKI识别位点BstZ17l识别位点E:「F T GATCJ■了卜…¥0"初…亍BssKI识别位点BstZ17l识别位点p•…GT"T AC…科A…W冉TO…5"Bsu36l识别位点^.-CCfTNAGG...3rBtgl识别位点Is;.. (7c RY Ge...3" 卜…GG¥RQCBtgZI识别位点p:一GC(3 ATG(N】「33 ..CGC TAC(N)…k..5^BtsCI识别位点p•…GSGTG Wd p\.CGTC AC^hlN,. 5Btsl识别位点p•...G C A G TG 卜...CGTCghIN (5)Cac8l识别位点5•…GC砾IGC…丁『…CG卧X…割Clal识别位点5\ .. AT'feGAT , .,3TAGCJA (51)CspCl识别位点p- :ori(N)CAA(N^GTGG{N>i£l^ 3「(3\^.lJ(N)GTT(N):CACC(N)1frr;. 5*t: ~C^TG ,..3* 買…GT也p…h CviKl-1识别位点S...RC7CY ..*3J …5’CviQl识别位点5:. (frAC …:T3’…C仲…亍Ddel识别位点(7T N A G…3「…GANT卫….亍Dpnl识别位点CH,I;5 G A T C 3^3」CT AG . 5"rC%Dpnll识别位点F" t n T C…雷『…G T A £\一-亍Dral识别位点TT A …丁3 ■…AAAJTT …5’Dralll识别位点『…CACNMNtTG …丁扩…GTG*N 忖NCAG. hDrdl识别位点5\ GACNNNN T N NGTC ,3'3\.CTGNNNNNNCAG. ,.5JCviAll识别位点EcoRV 识别位点fc ;.. CCTNI^NNAGG,.,3^ p ;. r GG AN NNJN NTCC 5^EcoO109lHaell 识别位点F …RGCGE-丁 常…空GCGFt …时b ; , , AT TC ,-3 卜…GTTA Af …亍Eael 识别位点|5:…丹 GCCR,3 p\.RCCG<5^. .5'Eael 识别位点|5 ...丹 GCCR,S 卜...RCCGGy (5)Eagl 识别位点fc. V&GCCG 31 P : . GCGGGJC...5'Earl 识别位点5V T :GA TX TC 7731 3■…CT AJAG …品Fatl 识别位点F …tATG …3「GTAC A ...5JFaul 识别位点犷…CCCGC (N)7. . .3* ,GGGCGtN\^ ,5「Fnu4HI 识别位点6;.. GCTNGC ・・4 3 .. GGN£G (5)识别位点&…RG3CC¥・・:$Fspl 识别位点5;., TG<ft5CA,, 3EcoP15l 识别位点|5\ .C AGCAGfN^^ 3 p : ..GTCGIC(N)^. ,.5^HaelllEcoRl 识别位点Hgal 识别位点5 .GACGC (N )5T .3.CTCTTC(N)7 GAG AAG(N\Ecil 识别位点 B . GGC GG A(N)n T .3" 3 仁.CCGCCTeO 注…h Fokl 识别位点5 GG ATG(M)…T .3^ 3;. .CCT AC{M)nk ..5^Fsel 识别位点EcoNl 识别位点5 …GGCCGG 七 C …3一 3: . CCJ J GGCGG ..5*3. . ,CTGCG{M),k ..5-EcoRV识别位点Hhal识别位点$...GCG七 (3)3. .. QPGG ,.5MHin cll识别位点杖…GTYlH AC…3 歯…GA蔺TG…寸Hin dlll识别位点5;r. A>GCT T・、・3”『…TTCG《…5"Hinfl识别位点F…G鼻NTC…3"P' . C7 . 5"HinP1l识别位点航GG…了b:.tGqp …5*Hpal识别位点|5;.GTr^AC,-3'P:.. CA AJTG.-.5Hpall识别位点$...此GG・・S 卜...GGqp (5)Hphl识别位点6+<. GGTGA ㈣3 忙…GGACT 叫血*6Hpy188l识别位点TC N'GA…3”p j.., A C T …5”Hpy188lll识别位点亍…TCfNNGA…歹31.鼻GNN卫T..hHpy99l识别位点3'..1-h GCWGC. ,5・HpyAV识别位点5...,CCTTCtN)7,. 3* 3.r GGAAG(N)t..亍HpyCH4lll识别位点5;., ACN^T . .*3. , rt^NC A . . 5 HpyCH4lVHpyCH4\识别位点TGt A…3"3J AC fc GT...5JKasl识别位点亍…代CgC…丁3..4CCGCGp....5* Kpnl识别位点6;GGT AC T C f,■歹C£ATGG.…印Mboll识别位点Mbol识别位点SvT^MTrc …歹A…GTAG「HMboll识别位点MspAII 识别位点Nhel 识别位点 Mspl 识别位点3■…GGqC …5’Mlul 识别位点Nael 识别位点Mmel 识别位点Msel 识别位点賓…F T A A …科3:…―匸T …引NgoMIV 识别位点了…GtCGGC ■二歹3:「GGGUQT.Gfi* . G A AG A (N)J .3 P :, + CTTCT (N)7A . + ,5*Mfel 识别位点,…內ATTG …3”& ..GT TA A 卫…£Mwol 识别位点6;. . GCNNNNNT M NG C ・・.33J . . CGNN^NN NHNCG ..5Mlyl 识别位点G AGTC (rj ;7 了 :,CTCA G(N)「…5* h …GC 比GC …3「3:..CGG t CCG.…5JNarl 识别位点『,G 比GCG …丁3: . CCGqGG.^5*Mnll 识别位点5: .,CCTC 侔人二 3” $…GSG 讯厂$Ncil 识别位点&…CC T SGG .,3即…GGS^CC. ..5-Mscl 识别位点5: ,. TGGtCA^.S 3 ... ACCpGT ...5JNcol 识别位点5: ., Ct ATGG , , a^-GGTACp . . .5*Ndel 识别位点5 ..ATQ .3^3:..GT AlAC …5’Msll识别位点5 ...CMNNllNRTG (3)3 GTANN^NNY A C 5MspAII识别位点Nhel识别位点PshAI 识别位点 PaeR7l 识别位点Nlalll 识别位点 石GAT 贰+・玄 扩…少T AC "Pcil 识别位点 F U TGT …扩3 . ..TGTA^A.^5'NotI 识别位点TAGC, ..3p :・"CCGg •3眷…CGATQG 佔3\ GAGCI ;C. ..5*NmeAIII 识别位点5"…GC CG AG 呱二3PflMI 识别位点5;.,CCANNNN T NT&G <1,3 3:「GGT 轉NNNACC ・.,5'PhoI 识别位点5…GGtG …b$…GCGGCCGC …3 fa :.. CGGCGG_CG..,5^NruI 识别位点TCbfcG 昌…3 P :.. AGCpGT ..5PleI 识别位点S ;. . GAGTC IN)T 3 3:., CTCAG (N),A .,.5NsiI 识别位点$…ATGC 肝一团 卜….1;ACGTA .,5'PmeI 识别位点$…GTTT 工冉AG.S 3J ..CAAAJTTG..J 5^NspI 识别位点$…RCAI ■計…tr b ■…百GTMR一$PacI 识别位点b J ... TT AA'Tr AA・「3P J ... A A TJ AA TT (5)PmlI 识别位点CAC'GTG..,3 3: .GTC^CAC . 5JPpuMI 识别位点6;tl RG T GWC CY.^3^ 3: ..YCC W GjGR .. .5F GG 晶g …歹6:..GAGhTNN<5TC ,B …CC 轉GG …h S^.CTGNN^NGAG .NlalV 识别位点PflFI 识别位点P^.CTGNN A NNCAG...5*Psil识别位点胃…TTkTAA,■罔卜…从公T T…因PspGI识别位点卩…丸CWGG…y 卜…GGWCQ…5"PspOMI识别位点|5:・・bGGCCC:・h 忙…CGgcys…hPspXI识别位点6:.. V<fTCGAG6 ...3H 3 .BGAGCT;CV r5^Pstl识别位点$…CTGG帀…3". G^A0GT C .5・Pvul识别位点$…CGiATtG …丁P J...GCJAGC.,.5* Pvull识别位点5:CA^TG.,3-击...GTQJ3AG (5)Rsal识别位点b:.7GT\C...3l 卜…gjTd…戲Rsrll识别位点卧…C西WCCG…歹,…GCCWGjpg…扩Sacl识别位点时…GAGCe…丁扩…CJCGAG.^S^Sacll识别位点6;., GCQd^G,…y 3:.. GG^CGGC -.S^Sall识别位点<fT C G A C. *. 3^ 3J...CAGCT;G...5JSapl识别位点5:…GCTCTTC 啊人=*3「3「・CGAGAAG{Nhv5”Sau3AI识别位点b;..T GATC .313:.. GT AG^ ..5*Sau96l识别位点亍…品NCC…丁3\NG J p...5JSbfl识别位点3」GGf CGTGC …5”Seal识别位点5;, t AGT^CT < ,r3TCAJGA ...S^ ScrFI识别位点SnaBI 识别位点 Tfil 识别位点歛:・TAC'feTA .・.歹3: .. ATGJSAT .. S'Spel 识别位点5:二 Xt TAGT …丁 3J ... TGATC^A .-S^Sphl 识别位点GC ATGfc …3「 『…qpTACG …亍Sspl 识别位点6: ., AA T>TT , 3J ... TTAJAA...5JStul 识别位点AGC^CCT ••謁 3: . TCqSGA …hStyD4l 识别位点6:JCGNGG …孑 3’…GGNCq . SStyl 识别位点E :・,GPWWGG,・・:3”GGWWC&—WSwal 识别位点6*,.. A T T iTi A A T r ,. 3 3: ..T A A Aj T TA (5)Taqa I 识别位点■…fbGA …;r b : ■ • A Gb -CCfhGG . .3 p J . ..GGN^CC •.生SexAl 识别位点,…心GWGGT …了3\..TGGWCqA (5)SfaNI 识别位点fe : . . GC A JC (N)J. ,3' 忙 GGTAG (N);>1 + ,5*Sfcl 识别位点F …疔TRYAG …:T 卜…—ysSfil 识别位点b:. .GGCCNNNr7NGGCC. ..3H p.-CCGGN^NWUCCGG .5Sfol 识别位点S^.GGC'teCC,-^' 3 …CCI^pG 右•一一5"SgrAI 识别位点p •…CF?t CGG¥G …丁 p\.GYGGGC^RC .S^Smal 识别位点『…右右右gUC …hSmll 识别位点b, ,C T T Y RAG …3 P . r ,G AH YT^C .. 5Tlil 识别位点Xhol 识别位点(7TCGAG.-・33;.. G A G C T^C (5)Tsel 识别位点Xmal 识别位点賢…衣CGGG …丁3■…GGGgC …5’Xmnl 识别位点橫一上T£直匚…3",G AANN >IMTTC ,p ; ..CASTG^3:. XT TN N^N MA AG < …5"Zral 识别位点 $…X ATT …wfcC. GAC\STC ・.3rf(5... TT AA M ..5J 3:. CTGJCAG.. .5"TspMl 识别位点 $...比CGGG (3)$…GGGCC£…节与大家共享(非原创)6;ANNNNN^NNNTGG...3J 3; GGTNNNh^NNNNNACC-.STspRl 识别位点|5;-.NNCASTGNM T -.3d忸.&NNGTSACMN …5"Tthllll 识别位点6: , ,G AC hfNNGTC,-・旷 3\.CTGNN i NCAG...5^Xbal 识别位点ftT AGA (3)P ; .. AG A TCJ (5)Tsp45l 识别位点 Tsp509l 识别位点Xcml识别位点SnaBI识别位点Tfil识别位点。
限制性核酸内切酶详细资料大全

限制性核酸内切酶详细资料大全限制性核酸内切酶是可以识别并附着特定的脱氧核苷酸序列,并对在每条链中特定部位的两个脱氧核糖核苷酸之间的磷酸二酯键进行切割的一类酶,简称限制酶。
根据限制酶的结构,辅因子的需求切位与作用方式,可将限制酶分为三种类型,分别是第一型(Type I)、第二型(Type II)及第三型(Type III)。
Ⅰ型限制性内切酶既能催化宿主DNA的甲基化,又催化非甲基化的DNA的水解;而Ⅱ型限制性内切酶只催化非甲基化的DNA的水解。
III型限制性内切酶同时具有修饰及认知切割的作用。
基本介绍•中文名:限制性核酸内切酶•外文名:Restriction endonuclease•作用:切割DNA•别名:限制酶限制性内切酶定义,由来,分布区域,分类性质,用途,命名,类型,第一型限制酶,第二型限制酶,第三型限制酶,生理意义,定义是识别特定的核苷酸序列,并在每条链中特定部位的两个核苷酸之间的磷酸二酯键进行切割的一类酶。
由来一般是以微生物属名的第一个字母和种名的前两个字母组成,第四个字母表示菌株(品系)。
例如,从Bacillus amylolique faciens H 中提取的限制性内切酶称为Bam H,在同一品系细菌中得到的识别不同碱基顺序的几种不同特异性的酶,可以编成不同的号,如HindⅡ、HindⅢ,HpaI、HpaⅡ,MboI、MboⅡ等。
限制性核酸内切酶别名:Endodeoxyribonuclease简称限制酶酶反应限制性内切酶能分裂DNA分子在一限定数目的专一部位上。
它能识别外源DNA并将其降解。
单位定义:在指明pH与37℃,在0.05mL反应混合物中,1小时消化1μg的λDNA的酶量为1单位。
性状制品不含非专一的核酸水解酶(由10单位内切酶与1μg λDNA,保温16小时所得的凝胶电泳图谱的稳定性表示),这类酶主要是从原核生物中分离出来的,迄今已经从近300多种不同的微生物中分离出约4000种限制酶。
泰克科学FastDigest Eco31I (IIs类) 限制性内切酶说明书

FastDigest Eco31I (IIs class)Catalog Number FD0293, FD0294Pub. No. MAN0012440 Rev. B.005'...G G T C T C (N)1↓…3' 3'...C C A G A G (N)5↑...5'Contents and storageDescriptionFastDigest Eco31I (IIs class) belongs to specific class of restriction enzymes which recognize asymmetric DNA sequences and cleave outside of their recognition sequence. IIs class enzymes are useful for many applications, including their use in seamless cloning of multiple fragments in a predefined order (1, 2, 3).Thermo Scientific FastDigest enzymes are an advanced line of restriction enzymes for rapid DNA digestion. All FastDigest enzymes are 100 % active in the universal FastDigest and FastDigest Green buffers and are able to digest DNA in5-15 minutes. This enables any combination of restriction enzymes to work simultaneously in one reaction tube and eliminates the need for sequential digestions.∙ FastDigest enzymes are optimized to digest plasmid, genomic and viral DNA as well as PCR products and do not show star activity even in prolonged incubations∙ Enzymes used in common downstream applications such as ligation, blunting and dephosphorylation reactions also have100 % activity in FastDigest and FastDigest Green Buffer.∙ FastDigest Green Buffer includes a density reagent along with blue and yellow tracking dyes that allow for direct loading of the reaction mixtures on a gel.The blue dye of the FastDigest Green Buffer migrates with 3-5 kb DNA fragments in a 1 % agarose gel and has an excitation peak at 424 nm. The yellow dye of the FastDigest Green Buffer migrates faster than 10 bp DNA fragments in a 1 % agarose gel and has an excitation peak at 615 nm.For applications that require analysis by fluorescence excitation FastDigest Buffer is recommended, as the dyes of the FastDigest Green Buffer may interfere with some fluorescence measurements.For Research Use Only. Not for use in diagnostic procedures.WARNING! Read the Safety Data Sheets (SDSs) and follow the handling instructions. Wear appropriate protective eyewear, clothing, and gloves. Safety Data Sheets (SDSs) are available from /support.FastDigest Eco31I (IIs class) cleavage is impaired by overlapping dcm methylation. To avoid dcm methylation, use a dam–, dcm–strains.Protocol for fast digestion of different DNAbine the following reaction components at room temperature in the order indicated:2. Mix gently and spin down.3. Incubate at 37 °C in a heat block or water thermostat for 5 min. Optional: Inactivate the enzyme by heating for 5 min at 65 °C4.If the FastDigest Green Buffer was used in the reaction, load an aliquot of the reaction mixture directly on a gel.Note: The FastDigest Green Buffer can be used as an electrophoresis loading buffer for any DNA sample at a final 1X concentration. Higher concentrations of FastDigest Green Buffer in the sample supply excess salt concentration which may alter DNA mobility.Double and multiple digestion of DNA∙The combined volume of the enzymes in the reaction mixture should not exceed 1/10 of the total reaction volume.∙Use 1 µL of each enzyme and scale up the reaction conditions appropriately.∙If the enzymes require different reaction temperatures, start with the enzyme that requires a lower temperature, then add the second enzyme and incubate at the higher temperature.Note: Increase the incubation time by 3-5 min if the total reaction volume exceeds 20 µL. Use water thermostat, air thermostats are not recommended due to the slow transfer of heat to the reaction mixture.Recommendations for PCR product digestion∙When introducing restriction enzyme sites into primers for subsequent digestion and cloning of a PCR product, refer to/fd, Reaction Conditions Guide, to define the number of extra bases required for efficient cleavage.∙Use Thermo Scientific GeneJET PCR Purification Kit, #K0701 to purify PCR product prior digestion in following cases:-When PCR additives such as DMSO or glycerol where used, as they may affect the cleavage efficiency or cause star activity.-When PCR Product will be used for cloning. Active thermophilic DNA polymerase still present in PCR mixture may alter the ends of the cleaved DNA and reduce the ligation efficiency.* 0.5 mM ATP (#R0441) is required for T4 DNA Ligase activity.2 FastDigest Eco31I (IIs class)Reference1. Engler, C., Gruetzner, R., Kandzia, R., and Marillonnet, S. (2009) Golden gate shuffling: a one-pot DNA shuffling method based on type IIs restriction enzymes. PLoS One 4, e5553.2. Engler, C., Kandzia, R., and Marillonnet, S. (2008) A one pot, one step, precision cloning method with high throughput capability. PLoS One 3, e3647.3. Engler, C., and Marillonnet, S. (2013) Combinatorial DNA assembly using Golden Gate cloning. Methods Mol Biol 1073, 141-156.Limited product warrantyLife Technologies Corporation and/or it affiliate(s) warrant their products as set forth in the Life Technologies’ General Te rms and Conditions of Sale at /us/en/home/global/terms-and-conditions.html. If you have any questions, please contact Life Technologies at /support .The information in this guide is subject to change without notice.DISCLAIMER: TO THE EXTENT ALLOWED BY LAW, THERMO FISHER SCIENTIFIC INC. AND/OR ITS AFFILIATE(S) WILL NOT BE LIABLE FOR SPECIAL, INCIDENTAL, INDIRECT, PUNITIVE, MULTIPLE, OR CONSEQUENTIAL DAMAGES IN CONNECTION WITH OR ARISING FROM THIS DOCUMENT, INCLUDING YOUR USE OF IT. Important Licensing Information : These products may be covered by one or more Limited Use Label Licenses. By use of this product, you accept the terms and conditions of all applicable Limited Use Label Licenses.©2020 Thermo Fisher Scientific Inc. All rights reserved. All trademarks are the property of Thermo Fisher Scientific and its subsidiaries unless otherwise specified./support | /askaquestion Thermo Fisher Scientific Baltics UAB | V.A. Graiciuno 8, LT-02241 Vilnius, LithuaniaFor descriptions of symbols on product labels or product documents, go to /symbols-definition.02 April 2020。
限制性内切酶的热失活温度表
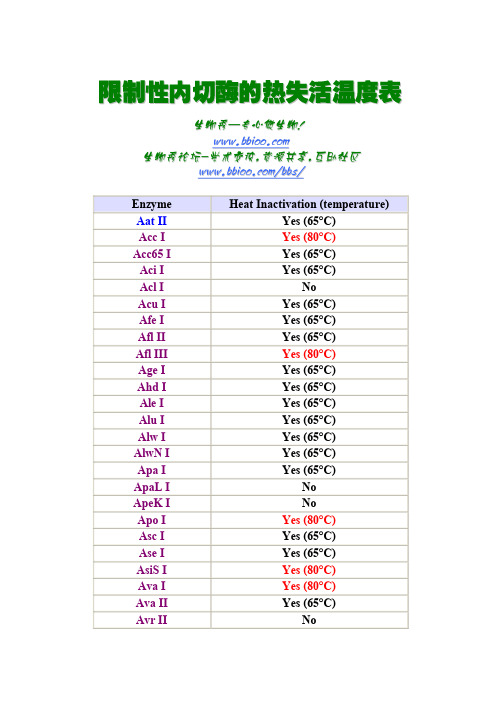
限 限制 制性 性内 内切 切酶 酶的 的热 热失 失活 活温 温度 度表表 生 生物 物秀 秀— —专 专心 心做 做生 生物物! w w w w w w . .b b b b i i o o o o . .c c oo m 生 生物 物秀 秀论 论坛 坛- -学 学术 术交 交流 流, ,资 资源 源共 共享 享, ,互 互助 助社社区 w w w w w w . .b b b b i i o o o o . .c c o o m m / /b b b b s s // EnzymeHeat Inactivation (temperature)Aat II Yes (65°C) Acc I Yes (80°C) Acc65 I Yes (65°C) Aci I Yes (65°C)Acl I No Acu I Yes (65°C) Afe I Yes (65°C) Afl II Yes (65°C) Afl III Yes (80°C) Age I Yes (65°C) Ahd I Yes (65°C) Ale I Yes (65°C) Alu I Yes (65°C) Alw I Yes (65°C) AlwN I Yes (65°C) Apa I Yes (65°C)ApaL I No ApeK I No Apo I Yes (80°C) Asc I Yes (65°C) Ase I Yes (65°C) AsiS I Yes (80°C) Ava I Yes (80°C) Ava II Yes (65°C)Avr IINoBae I Yes (65°C) BamH I NoBan I Yes (65°C) Ban II Yes (65°C) Bbs I Yes (65°C) Bbv I Yes (65°C) BbvC I Yes (80°C) Bcc I Yes (65°C) BceA I Yes (65°C) Bcg I Yes (65°C) BciV I Yes (65°C) Bcl I NoBfa I Yes (80°C) BfrB I Yes (80°C) BfuA I Yes (65°C) BfuC I Yes (80°C) Bgl I Yes (65°C) Bgl II NoBlp I No Bme1580 I Yes (80°C) BmgB I Yes (65°C) Bmr I Yes (65°C) Bmt I Yes (65°C) Bpm I Yes (65°C) Bpu10I Yes (80°C) BpuE I Yes (65°C) Bsa I Yes (65°C) BsaA I Yes (80°C) BsaB I Yes (80°C) BsaH I Yes (80°C) BsaJ I Yes (80°C) BsaW I Yes (80°C) BsaX I NoBseR I Yes (65°C)BseY I Yes (65°C) Bsg I Yes (65°C) BsiE I Yes (80°C) BsiHKA I Yes (80°C) BsiW I Yes (80°C) Bsl I Yes (80°C) Bsm I Yes (80°C) BsmA I Yes (80°C) BsmB I Yes (80°C) BsmF I Yes (80°C) BsoB I No Bsp1286 I Yes (65°C) BspCN I Yes (80°C) BspD I Yes (65°C) BspE I Yes (80°C) BspH I Yes (65°C) BspM I Yes (65°C) Bsr I Yes (80°C) BsrB I Yes (80°C) BsrD I Yes (80°C) BsrF I No BsrG I Yes (80°C) BssH II Yes (80°C) BssK I Yes (80°C) BssS I Yes (80°C) BstAP I Yes (80°C) BstB I No BstE II No BstF5 I No BstN I No BstU I No BstX I Yes (65°C) BstY I Yes (80°C) BstZ17 I NoBsu36 I Yes (80°C) Btg I Yes (80°C) BtgZ I Yes (80°C) Bts I Yes (80°C) Cac8 I Yes (65°C) Cla I Yes (65°C) CviA II Yes (65°C) Dde I Yes (65°C) Dpn I Yes (80°C) Dpn II Yes (65°C) Dra I Yes (65°C) Dra III Yes (65°C) Drd I Yes (65°C) Eae I Yes (65°C) Eag I Yes (65°C) Ear I Yes (65°C) Eci I Yes (65°C) EcoN I Yes (65°C) EcoO109 I Yes (65°C) EcoR I Yes (65°C) EcoR V Yes (80°C) Fat I Yes (65°C) Fau I Yes (65°C) Fnu4H I Yes (65°C) Fok I Yes (65°C) Fse I Yes (65°C) Fsp I Yes (65°C) Hae II Yes (80°C) Hae III Yes (80°C) Hga I Yes (65°C) Hha I Yes (65°C) HinP1 I Yes (65°C) Hinc II Yes (65°C) Hind III Yes (65°C)Hinf I Yes (80°C) Hpa I NoHpa II Yes (65°C) Hph I Yes (65°C) Hpy99 I Yes (65°C) Hpy188 I Yes (65°C) Hpy188 III Yes (65°C) HpyCH4 III Yes (80°C) HpyCH4 IV Yes (65°C) HpyCH4 V Yes (65°C) ICeu I Yes (65°C) ISce I Yes (65°C) Kas I Yes (65°C) Kpn I NoMbo I Yes (65°C) Mbo II Yes (65°C) McrBC Yes (65°C) Mfe I Yes (65°C) Mlu I Yes (65°C) Mly I Yes (65°C) Mme I Yes (80°C) Mnl I Yes (65°C) Msc I Yes (65°C) Mse I Yes (65°C) Msl I Yes (65°C) Msp I Yes (65°C) MspA1 I Yes (65°C) Mwo I NoNae I Yes (65°C) N.Alw I Yes (80°C) Nar I Yes (65°C) N.BbvC IA Yes (80°C) N.BbvC IB Yes (80°C) N.BstNB I Yes (80°C)Nci I No Nco I Yes (65°C) Nde I Yes (65°C) NgoM IV Yes (80°C) Nhe I Yes (65°C) Nla III Yes (65°C) Nla IV Yes (65°C) Not I Yes (65°C) Nru I Yes (65°C) Nsi I Yes (65°C) Nsp I Yes (65°C) Pac I Yes (65°C) PaeR7 I No Pci I Yes (65°C) PflF I Yes (65°C) PflM I Yes (65°C) Pho I NoPIPsp I NoPlSce I Yes (65°C) Ple I Yes (65°C) Pme I Yes (65°C) Pml I Yes (65°C) PpuM I No PshA I Yes (65°C) Psi I Yes (65°C) PspG I No PspOM I Yes (65°C) Pst I Yes (80°C) Pvu I Yes (80°C) Pvu II No Rsa I Yes (65°C) Rsr II Yes (65°C) Sac I Yes (65°C) Sac II Yes (65°C)Sal I Yes (65°C) Sap I Yes (65°C) Sau96 I Yes (80°C) Sau3A I Yes (65°C) Sbf I Yes (65°C) Sca I Yes (80°C) ScrF I Yes (65°C) SexA I Yes (65°C) SfaN I Yes (65°C) Sfc I Yes (65°C) Sfi I No Sfo I Yes (65°C) SgrA I Yes (65°C) Sma I Yes (65°C) Sml I No SnaB I Yes (80°C) Spe I Yes (65°C) Sph I Yes (65°C) Ssp I Yes (65°C) Stu I Yes (65°C) Sty I Yes (65°C) StyD4 I Yes (65°C) Swa I Yes (65°C) Taq I Yes (80°C) Tfi I NoTli I No Tse I No Tsp45 I No Tsp509 I No TspR I No Tth111 I No Xba I Yes (65°C) Xcm I Yes (65°C)Xho I Yes (65°C) Xma I Yes (65°C) Xmn I Yes (65°C) Zra I Yes (80°C)。
史上最全限制性内切酶酶切位点汇总

A系列AatII识别位点Acc65I识别位点AccI识别位点AciI识别位点AclI识别位点AcuI识别位点AfeI识别位点AflII识别位点AflIII识别位点AgeI识别位点AhdI识别位点AleI识别位点AluI识别位点AlwI识别位点AlwNI识别位点ApaI识别位点ApaLI识别位点ApeKI识别位点ApoI识别位点AscI识别位点AseI识别位点AsiSI识别位点AvaI识别位点AvaII识别位点AvrII识别位点BaeI识别位点BamHI识别位点BanI识别位点BanII识别位点BbvCI识别位点BbvI识别位点BccI识别位点BceAI识别位点BcgI识别位点BciVI识别位点BclI识别位点BfaI识别位点BfuAI识别位点BglI识别位点BglII识别位点BlpI识别位点Bme1580I识别位点BmgBI识别位点BmrI识别位点BmtI识别位点BpmI识别位点Bpu10I识别位点BpuEI识别位点BsaAI识别位点BsaBI识别位点BsaHI识别位点BsaI识别位点BsaJI识别位点BsaWI识别位点BsaXI识别位点BseRI识别位点BseYI识别位点BsiEI识别位点BsiHKAI识别位点BsiWI识别位点BslI识别位点BsmAI识别位点BsmBI识别位点BsmFI识别位点BsmI识别位点BsoBI识别位点Bsp1286I识别位点BspCNI识别位点BspDI识别位点BspEI识别位点BspHI识别位点BspMI识别位点BspQI识别位点BsrBI识别位点BsrDI识别位点BsrFI识别位点BsrGI识别位点BsrI识别位点BssHII识别位点BssKI识别位点BssSI识别位点BstAPI识别位点BstBI识别位点BstEII识别位点BstNI识别位点BstXI识别位点BstYI识别位点BstZ17I识别位点Bsu36I识别位点BtgI识别位点BtgZI识别位点BtsCI识别位点BtsI识别位点Cac8I识别位点ClaI识别位点CspCI识别位点CviAII识别位点CviKI-1识别位点CviQI识别位点DdeI识别位点DpnI识别位点DpnII识别位点DraI识别位点DraIII识别位点DrdI识别位点EaeI识别位点EaeI识别位点EagI识别位点EarI识别位点EciI识别位点EcoNI识别位点EcoO109I识别位点EcoP15I识别位点EcoRI识别位点EcoRV识别位点FatI识别位点FauI识别位点Fnu4HI识别位点FokI识别位点FseI识别位点FspI识别位点HaeII识别位点HaeIIIHgaI识别位点HhaI识别位点HincII识别位点HindIII识别位点HinfI识别位点HinP1I识别位点HpaI识别位点HpaII识别位点HphI识别位点Hpy188I识别位点Hpy188III识别位点Hpy99I识别位点HpyAV识别位点HpyCH4III识别位点HpyCH4IV HpyCH4V识别位点KasI识别位点KpnI识别位点MboI识别位点MboII识别位点MfeI识别位点MluI识别位点MlyI识别位点MmeI识别位点MnlI识别位点MscI识别位点MseI识别位点MslI识别位点MspA1I识别位点MspI识别位点MwoI识别位点NaeI识别位点NarI识别位点NciI识别位点NdeI识别位点NgoMIV识别位点NheI识别位点NlaIII识别位点NlaIV识别位点NmeAIII识别位点NotI识别位点NruI识别位点NsiI识别位点NspI识别位点PacI识别位点PaeR7I识别位点PciI识别位点PflFI识别位点PflMI识别位点PhoI识别位点PleI识别位点PmeI识别位点PmlI识别位点PpuMI识别位点PshAI识别位点PsiI识别位点PspGI识别位点PspOMI识别位点PspXI识别位点PstI识别位点PvuI识别位点PvuII识别位点RsrII识别位点SacI识别位点SacII识别位点SalI识别位点SapI识别位点Sau3AI识别位点Sau96I识别位点SbfI识别位点ScaI识别位点ScrFI识别位点SexAI识别位点SfaNI识别位点SfcI识别位点SfiI识别位点SfoI识别位点SgrAI识别位点SmaI识别位点SmlI识别位点SnaBI识别位点SpeI识别位点SphI识别位点SspI识别位点StuI识别位点StyD4I识别位点StyI识别位点SwaI识别位点TaqαI识别位点TfiI识别位点TseI识别位点Tsp45I识别位点Tsp509I识别位点TspMI识别位点TspRI识别位点Tth111I识别位点XbaI识别位点XcmI识别位点XhoI识别位点XmaI识别位点XmnI识别位点ZraI识别位点。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
FastDigest® BsaAI FastDigest® BsaBI FastDigest® BsaHI FastDigest® BsaJI
BseDI
CCTNAGC GCTNAGC YACGTR GATNNNNA
TC GRCGYC
CCNNGG CCNNGG
BseGI FastDigest® BseGI BseJI
37 °C 37 °C 37 °C 37 °C 37 °C 37 °C 37 °C 37 °C 37 °C 37 °C 37 °C 37 °C 37 °C 37 °C 37 °C 37 °C
T
50-100%
Buffer AarI + oligo
NR
B
100%
T
50-100%
F
F
O
0-20%
F
F
F
F + SAM
0-20% 100% 20-50%
0-20% 20-50%
100%
20-50%
50-100% 50-100% 50-100% 50-100%
0-20%
0-20%
100%
20-50%
F + SAM
F Buffer Bpu10I
F
T
F
0-20% 20-50% 50-100% 100% 50-100% 100%
BspPI
GGATC
BspTI FastDigest® BsrBI FastDigest® BsrDI FastDigest® BsrFI FastDigest® BssHII Bst1107I
BstXI
FastDigest® BstXI FastDigest® BstZ17I Bsu15I FastDigest® Bsu36I BsuRI
AGCT
Alw21I
GWGCWC
FastDigest®
Alw21I
GWGCWC
Alw26I
GTCTC
FastDigest® Alw26I
GTCTC
Alw44I
GTGCAC
FastDigest® CAGNNNCT
AlwNI
G
ApaI
GGGCCC
FastDigest® ApaI
GGGCCC
FastDigest® ApaLI
100% 100%
50-100% 0-20%
NR
100%
0-20% 20-50% 50-100% 50-100%
F T T Buffer BfuI
O
20-50% 0-20%
NR
0-20%
20-50% 50-100%
NR
50-100%
0-20% 0-20% 0-20%
100%
0-20% 0-20% 0-20%
内切酶
识别位点 孵育温度 推荐缓冲液 B
AanI
TTATAA
AarI
CACCTGC
AasI
AatII FastDigest® AatII FastDigest® AccI Acc65I FastDigest® Acc65I FastDigest® AciI FastDigest® AclI FastDigest® AcuI
0-20%
0-20% 0-20% 50-100% 0-20% 50-100% 50-100% 100%
BamHI
FastDigest® BamHI FastDigest® BanI BauI
GGATCC GGATCC GGYRCC CACGAG
37 °C Buffer BamHI 20-50%
37 °C
100% 100%
FastDigest® BbsI FastDigest® BbvI
BclI
FastDigest® BclI
BcnI
GAAGAC GCAGC TGATCA TGATCA CCSGG
BcuI
ACTAGT
BdaI
FastDigest® BfaI BfiI
TGANNNNN NTCA CTAG
F
37 °C
F
37 °C
T
0-20%
G 50-100%
NR 20-50% 20-50% 20-50%
100%
NR 50-100%
0-20% 0-20% 0-20% 20-50% 100% 100% 20-50%
100% 50-100%
在各种Fermentas缓冲液中的活性
O 0-20%
R 0-20%
50-100% 50-100% 20-50% 20-50%
100%
20-50%
F
F
F
T
50-100% 20-50% 0-20%
0-20%
100% 50-100%
T
20-50% 50-100% 20-50% 20-50%
100%
20-50%
F
O
NR
100%
100%
NR
NR
100%
T
20-50%
100% 50-100% 20-50%
50-100% 50-100%
0-20% 0-20% 0-20%
100% 100% 0-20%
0-20% 20-50%
100%
20-50% 100%
20-50%
100% 50-100% 20-50% 50-100%
0-20%
0-20%
100%
100%
0-20% 50-100% 100% 50-100%
50-100%
50-100% NR
50-100% 50-100% 20-50%
0-20% NR
F
R
0-20%
0-20% 50-100% 100%
20-50% 50-100%
100% 100%
100%
100% 100% 100% 100% 100%
100% 100% 100% 100% 100% 100% 100% 100%
T 100%
0-20%
0-20%
NR
2xT 20-50% 50-100%
0-20% 0-20%
0-20% 0-20%
50-100% 100%
0-20% 20-50%
100%
20-50% 20-50% 50-100%
20-50%
100%
100%
20-50%
20-50% 20-50%
NR 100%
NR
20-50%
37 °C
37 °C
37 °C 37 °C 37 °C 37 °C 37 °C 55 °C 37 °C 37 °C 55 °C 37 °C 37 °C 37 °C 55 °C
AdeI
FastDigest® AfeI FastDigest® AflII FastDigest® AgeI AjiI
GACNNNNN NGTC
GACGTC GACGTC
GTMKAC GGTACC GGTACC
CCGC
AACGTT
CTGAAG CACNNNGT
G AGCGCT
CTTAAG
ACCGGT CACGTC
GTGCAC
FastDigest® AscI
GGCGCGCC
FastDigest® AseI
ATTAAT
FastDigest®
AsiSI
GCGATCGC
FastDigest®
AvaI
CYCGRG
FastDigest® AvaII
GGWCC
FastDigest®
AvrII
CCTAGG
37 °C
37 °C
0-20%
0-20% 20-50% 0-20%
20-50% 50-100% 100% 50-100% 0-20% 50-100% 100% 50-100%
F
100% 100% 100% 100% 100% 100%
100% 100% 100%
100% 100%
100% 100% 100% 100% 100% 100% 100% 100% 100% 100% 100% 100%
37 °C 37 °C 55 °C 37 °C 37 °C 37 °C 30 °C 37 °C 37 °C 37 °C 37 °C 37 °C
37 °C 37 °C 37 °C 37 °C 37 °C
37 °C
37 °C 37 °C 37 °C 37 °C
37 °C 37 °C 37 °C 37 °C 37 °C 37 °C 65 °C 37 °C 37 °C 55 °C 55 °C 37 °C 65 °C
ACTGGG
BfmI
CTRYAG
BfuI
GTATCC
BglI
FastDigest® BglI
BglII FastDigest® BglII FastDigest® BlpI Bme1390I
GCCNNNNN GGC
GCCNNNNN GGC
AGATCT
AGATCT
GCTNAGC
CCNGG
FastDigest® Bme1580I
55 °C 55 °C 55 °C 65 °C 65 °C 55 °C 65 °C 65 °C 37 °C
F
2xT G F T T
G + SAM
20-50%
100%
20-50% 20-50%
100%
100% 100%
20-50% 50-100% 50-100% 50-100% 50-100% 50-100% 0-20% 20-50%
