高光谱数据处理工具软件使用手册
ENVI基本影像处理流程操作

ENVI基本影像处理流程操作ENVI(Environment for Visualizing Images)是一款基于科研的遥感数据处理软件,该软件可以进行多光谱、高光谱和雷达影像的处理、分析以及可视化等操作。
本文将介绍ENVI基本影像处理流程的操作步骤,并提供一些实用技巧。
1. ENVI软件的安装ENVI软件的安装包可以通过官方网站下载,安装过程需要注意以下几点:•系统要求:ENVI软件需要安装在一台配备了Windows操作系统的计算机上。
安装前请确认您的计算机符合软件的系统要求。
•授权方式:ENVI采用了USB加密狗的授权方式,安装时需要连入加密狗。
•安装路径:ENVI软件的安装路径需要选择到英文全路径,建议安装在较大的磁盘上。
2. ENVI软件的启动ENVI软件具有图形界面和命令行两种启动方式,这里只介绍图形界面启动方式。
操作步骤如下:1.在Windows桌面上找到ENVI的快捷方式,鼠标双击打开ENVI软件;2.程序启动后,会出现ENVI Main窗口,该窗口分为菜单栏和工具栏两部分;3.在菜单栏中选择基本操作>打开文件,弹出Open File窗口;4.选择需要打开的影像文件,点击打开按钮,ENVI软件会自动加载文件。
3. ENVI基本影像处理流程ENVI软件提供了多种基本影像处理流程,这里将介绍最常用的三种:图像增强、变换和分类。
下面的操作步骤均在ENVI软件的主界面中完成。
3.1 图像增强图像增强是指通过某些方法,使影像的细节更明显、对比度更强、噪声更小,从而使影像更易于解译。
下面介绍ENVI中的直方图均衡化增强方式。
1.打开需要增强的影像文件;2.在ENVI菜单栏中选择基本操作>图像增强>直方图均衡化;3.弹出直方图均衡化窗口,选择需要增强的波段,点击确定按钮;4.ENVI软件会自动完成直方图均衡化操作,并在新窗口中显示增强后的影像。
3.2 变换变换是指将原始影像进行变换,获得不同的波段组合,以便更好地提取地物信息。
ENVI基础功能教程

ENVI基础功能教程ENVI(Environment for Visualizing Images)是一款功能强大的遥感图像处理和分析软件。
它是由Exelis Visual Information Solutions 开发的,用于处理、分析和可视化各种遥感数据的工具。
本文将介绍ENVI的基础功能,并提供相应教程。
1.数据导入和显示ENVI可以导入多种遥感数据,包括多光谱、高光谱、雷达和激光雷达数据等。
用户可以选择打开单个文件或者批量导入数据集。
导入后,ENVI会将数据以图像形式显示在界面上。
教程:a.在ENVI中选择“文件”->“打开”来导入遥感图像。
b.选择要导入的图像文件,并点击“打开”按钮。
c.ENVI会将图像加载到显示窗口中。
2.图像增强ENVI提供了多种图像增强工具,用于改善图像质量和可视化效果。
这些工具包括亮度调整、对比度增强、直方图均衡化等。
教程:a.选择“处理”->“亮度/对比度调整”来打开亮度和对比度调整对话框。
b.在对话框中调整亮度和对比度滑块,直到达到理想的效果。
c.点击“应用”按钮来应用调整。
3.波段操作ENVI允许对图像的不同波段进行操作,包括波段合成、波段提取和波段重映射等。
这些功能有助于提取和分析图像中的特定信息。
教程:a.选择“处理”->“波段操作”来打开波段操作对话框。
b.在对话框中选择要进行的操作,如波段合成或波段提取。
c.根据对话框中的提示设置参数,并点击“应用”按钮来进行操作。
4.目标检测和分类ENVI提供了多种目标检测和分类算法,用于自动提取地物信息。
用户可以将图像分割成不同的类别,并根据需要进行后续分析。
教程:a.选择“分类”->“目标检测和分类”来打开目标检测和分类对话框。
b.选择合适的检测和分类算法,并设置参数。
c.点击“开始”按钮来进行目标检测和分类。
5.地形分析ENVI具有地形分析功能,可以对地表高程数据进行分析和可视化。
ENVI高光谱数据分析操作手册

感兴趣区和掩膜的选择和使用可具体情况具体分析,运行一项或两项均可。
北京卓立汉光仪器有限公司
4. 滤波
打开图像,FilterConvolutions and Morphology。在Convolutions and Morphology Tools 中,选择 Convolutions滤波类型(高通滤波 器、低通滤波 器、拉普拉斯算子、方向滤波器、高斯高通滤波器、高斯低通滤波器、中值滤波 器、Sobel、Roberts、自定义卷积核)。
2.3.2.3. 保存波谱库
北京卓立汉光仪器有限公司 在Spectral Libraries Resampling Parameters对话框中,为Resample Wavelength To选择匹配源,一般选择图像文件为参考。 输出重采样波谱库.sli
北京卓立汉光仪器有限公司
3. 感兴趣区和掩膜
3.1. 感兴趣区(ROI)
Display 窗 口 中 , Overlay → Region of Interest , 在 ROI 对 话 框 中 , 单 击 ROI_Type→Polygon. 绘制窗口中,选择Image,绘制一个多边形,右键结束,可根据需要多绘制 几个。
主菜单→Basic Tools→Subset Data via ROIs,选择裁剪图像。 在Saptial Subset via ROIs Parameters中,设置参数。 Select Input ROIs,选择绘制的ROI。 Mask Pixel Outside of ROIs选择yes。
4.1. 设置参数
Kernel Size(卷积核大小):奇数。 Image Add Back(加回值):将原始图像中的部分加回到卷积滤波结果图像中, Editable Kernel(卷积核中各项的值)。
高光谱数据分析ENVI操作手册

高光谱数据分析ENVI操作手册1.常见参数选择主菜单→File→Preferences●用户自定义文件(User Defined Files)图形颜色文件,颜色表文件,ENVI的菜单文件,地图投影文件等。
需重启ENVI ●默认文件目录(Default Directories)默认数据目录,临时文件目录,默认输出文件目录,ENVI补丁文件、光谱库文件、备用头文件目录等,需重启ENVI。
●显示设置(Display Default)可以设置三窗口中各个分窗口的显示大小,窗口显示式样等。
其中可以设置数据显示拉伸方式(Display Default Stretch),默认为2%线性拉伸。
●其他设置(Miscollaneous)制图单位(Page Unit),默认为英寸(Inches),可设置为厘米(Centimeters)还有缓冲大小(cache size),可以设置为物理内存的50-75%左右。
Image Tile Size不能超过4M。
2.显示图像及其波谱2.1.打开文件●主菜单,Open Image File→文件名.raw。
●或Window→Available Bands List→File →Open Image File→文件名.raw。
2.2.显示图像●显示单波段灰度级图像:Gray color,选择的波段一般是图像显示最清晰的波段。
●显示伪彩色图像:RGB color,选择具有明显吸收谷、强烈反射作用和所含信息量较大的波段作为彩色合成RGB波段。
●显示真彩色图像:波段列表(Available Bands List)中,右键→Load TrueColor 。
●图像保存:Display窗口,File→Save Image As→Image File,选择输出格式、路径和名称,OK。
●动画显示:Display窗口,Tools→Animation,动态显示各波段图像,能很快的分辨出包含信息量较多的波段。
ENVI高光谱数据处理流程
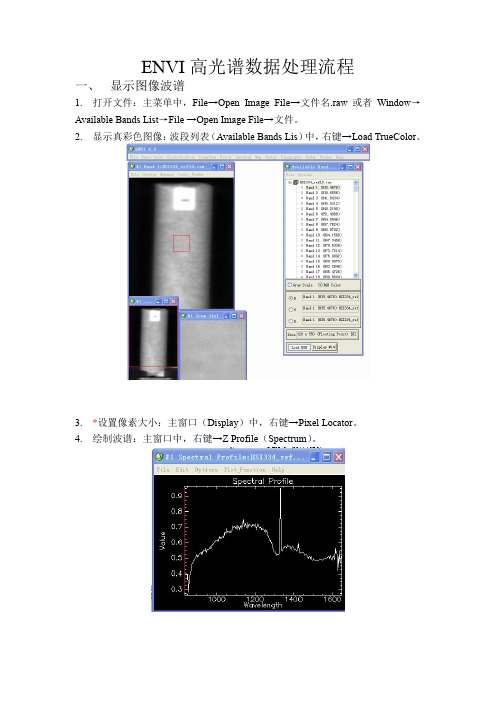
ENVI高光谱数据处理流程一、显示图像波谱1.打开文件:主菜单中,File→Open Image File→文件名.raw或者Window→Available Bands List→File →Open Image File→文件。
2.显示真彩色图像:波段列表(Available Bands Lis)中,右键→Load TrueColor。
3.*设置像素大小:主窗口(Display)中,右键→Pixel Locator。
4.绘制波谱:主窗口中,右键→Z Profile(Spectrum)。
5.收集任意点波谱:Spectral Profile中,Options→Collect Spectra,点击图像任6.光谱平滑:Spectral Profile中,Options→Set Z Profile Avg Window,将window7.部分光谱:主菜单→Basic Tools→Resize Data(Spatial/Spectral)→Spectral Subset,选择需要的光谱波段。
生成新的文件,右键→Load True Color to<new>。
显示新图像。
8.关闭所有文件:File→Close All Files。
二、标准波谱库主菜单→Spectral→Spectral Libraries→Spectral Library Viewer→安装文件夹下,ITT\IDL\IDL80\products\envi48\spec_lib。
共有usgs_min、veg _lib、jpl_lib、jhu_lib四个标准波谱库。
在Spectral Library Viewer中,单击波谱名称,自动显示波谱。
三、自定义波谱库1.输入波长范围:在菜单中,Spectral Spectral Library→Spectral Library Builder2.波谱收集:以从影像数据中收集波谱为例:a)打开高光谱图像,收集任意点波谱。
《使用ENVI的高光谱工具处理多光谱数据》

专题二十四 使用ENVI的高光谱工具处理多光谱数据(节选)1.1.专题概述本专题的目的是向用户展示如何使用ENVI先进的高光谱工具对多光谱数据进行分析。
要更好地理解高光谱处理的概念及其工具,请参见ENVI高光谱辅导指南。
要获取额外的详细信息,请参见《ENVI遥感影像处理实用手册》(ENVI User’s Guide)或者ENVI的在线帮助。
♦本专题中使用的文件光盘:《ENVI遥感影像处理专题与实践》附带光盘 #1♦背景知识ENVI并非仅设计成高光谱影像处理系统。
在1992年,ENVI的开发者就决定开发出一个通用的影像处理软件,它包含一整套的基本处理工具,弥补了商业软件缺乏强大灵活处理功能的不足,使得它能够处理各种科学格式的影像数据。
它对全色、多光谱、高光谱以及基本和改进雷达影像数据都提供了支持。
当前,ENVI包含了与其它主要影像处理系统(例如:ERDAS,ERMapper和PCI)相同的基本处理功能。
其中,ENVI在前沿遥感研究中采用了许多不同的先进算法。
虽然这些算法都是在处理成像光谱仪数据或者多达上百个波谱波段的高光谱数据基础之上发展而来,但是它们也可以应用到多光谱数据和其它标准数据类型的处理上。
本专题将对某些分析Landsat Thematic Mapper(TM)数据的方法进行介绍。
本专题分为两个独立的部分:1)使用标准或者经典多光谱分析技术,对TM影像数据进行典型的多光谱分析,2)使用ENVI高光谱工具对相同的数据集进行分析。
1.2.使用ENVI的高光谱工具分析多光谱数据♦读取TM影像数据z要从磁带中读取数据,可以在ENVI主菜单中选择File → Tape Utilities → Read Known Tape Formats → Landsat TM(或者对于新的EDC-格式的磁带选择NLAPS)。
z要从光盘中读取数据,可以选择File →Open External File → Landsat → Fast,或者选择File → Open External File → Landsat → NLAPS(对于NLAPS数据)。
光谱分析软件说明书

光譜影像分析軟體操作1. 載入檔案, 副檔名為 raw. 載入後檔案路徑會顯示在Path內.
2. 選取多點資料, 先選擇ROI的工具. 然後在有興趣的位置按下滑鼠左鍵, 要再新增時先按下Ctrl 鍵再按滑鼠左鍵即可. 在右下角的畫面則會出現光譜的波形.
3. 改變其他波長的影像可移動Profile上的RGB直線或是直接改變波長數值然後按SET鍵.
4. 顯示單色影像先將選項改為Mono然後按下SET. 顯示不同波長影像可直接輸入波長或是移動紅色游標後按SET.
5. 若要讓光譜波形較平滑可先選擇Analyzer改變Smoothing 的值在點選要觀察的位置.
6. 資料輸出請先選擇Report Setting 然後選擇要輸出的項目, 選擇Mono Image 中的Export 需要輸入波長. 想連同光譜輸出請先選擇特定點.。
ENVI高光谱数据处理流程

ENVI高光谱数据处理流程ENVI(Environment for Visualizing Images)是一款功能强大的遥感数据处理软件,用于高光谱数据的处理和分析。
它提供了许多功能模块,可以进行数据导入、预处理、特征提取、分类和可视化等操作。
下面是ENVI高光谱数据处理流程的详细介绍。
1.数据导入首先,我们需要将高光谱数据导入ENVI软件。
ENVI支持导入多种高光谱数据格式,如Hyperion、AVIRIS等。
可以通过ENVI的文件菜单选择导入数据或者使用ENVI API导入数据。
2.数据预处理在数据导入之后,我们需要对高光谱数据进行预处理,以减少噪声和增强图像的质量。
ENVI提供了多种数据预处理方法,包括大气校正、大气校正和去除噪声。
可以根据数据的需求选择适当的预处理方法。
3.特征提取特征提取是高光谱数据分析的关键步骤。
在这一步骤中,我们可以利用ENVI提供的各种特征提取算法来提取数据中的有用信息。
ENVI提供了许多特征提取算法,包括主成分分析(PCA)、线性判别分析(LDA)、最大似然分类(MLC)等。
4.分类分类是高光谱数据处理的一个重要环节。
ENVI提供了多种分类算法,用于将数据分成不同的类别。
可以使用ENVI的分类工具对特征提取后的数据进行分类,根据分类结果进行应用。
5.可视化可视化是高光谱数据处理的最后一步。
ENVI提供了丰富的可视化工具,可以对数据进行可视化和可视化分析。
可以通过ENVI的图像菜单选择适当的可视化工具,并根据需要生成图像。
以上是ENVI高光谱数据处理的基本流程。
当然,根据具体的应用和需求,还可以根据需要选择其他的处理方法和工具。
此外,ENVI还支持自定义算法和脚本编程,以满足更高级的数据处理需求。
总结起来,ENVI高光谱数据处理流程包括数据导入、数据预处理、特征提取、分类和可视化等步骤。
通过这些步骤,我们可以对高光谱数据进行全面的处理和分析,从而获取有用的信息并进行进一步的应用。
ENVI高光谱分析

ENVI高光谱分析ENVI高光谱分析是一种用于图像处理和数据分析的软件平台,主要用于处理和分析在大气、地球表面和水体等领域获取的高光谱数据。
高光谱数据是指在较窄波段范围内获取的光谱信息,通常包含数百个波段。
ENVI高光谱分析利用这些波段信息,可以提供更详细、更精确的数据结果,有助于理解地球表面的复杂变化和环境过程。
1.数据预处理:ENVI高光谱分析可以对高光谱数据进行预处理,包括大气校正、辐射校正、几何纠正等。
这些预处理步骤可以消除由于大气、仪器和环境等因素引起的杂乱噪声,并提高数据的质量和可靠性。
2.特征提取:ENVI高光谱分析可以通过使用不同的数学和统计算法,从高光谱数据中提取目标的特征信息。
这些特征可以用于分类、目标检测、遥感变化检测等应用。
3.数据可视化:ENVI高光谱分析可将高光谱数据以多种方式进行可视化,包括光谱曲线、散点图、等高线、伪彩色图等。
这些可视化方法有助于用户直观地理解数据的内在规律和潜在关系。
4.数学建模和分析:ENVI高光谱分析提供了多种数学建模和分析工具,包括主成分分析、线性回归、非线性回归、聚类分析等。
这些工具可以帮助用户识别数据中的模式和趋势,从而进行进一步的数据分析和解释。
5.地物分类:ENVI高光谱分析可进行高光谱图像的地物分类,包括监督分类和非监督分类。
监督分类需要用户提供一些参考样本,用于训练分类器;非监督分类则通过统计分析和像元聚类等方法,自动划分不同地物类型。
6.数据挖掘:ENVI高光谱分析可以挖掘高光谱数据中的隐藏信息和趋势,帮助用户发现新的知识和洞见。
数据挖掘算法包括关联规则挖掘、聚类分析、分类分析等。
ENVI高光谱分析在许多领域具有广泛的应用,包括地球科学、环境监测、农业、气象、地质勘探等。
例如,在农业领域,ENVI高光谱分析可以帮助农民分析土壤和植被的光谱特征,以优化施肥、灌溉和作物管理等决策。
在环境监测领域,ENVI高光谱分析可以检测和监测大气污染、水体污染、土壤侵蚀等环境问题。
和乌林哈(Andurinha)——光谱数据处理工具包说明书

Package‘andurinha’October12,2022Type PackageTitle Make Spectroscopic Data Processing EasierVersion0.0.2Description The goal of'andurinha'is provide a fast and friendly way to process spectroscopic data.It is intended for processing several spectra ofsamples with similar composition(tens to hundreds of spectra).It compilesspectroscopy datafiles,produces standardized and second derivative spectra,finds peaks and allows to select the most significant ones based on the second derivative/absorbance sum spectrum.It also provides functions for graphicevaluation of the outputs.Depends R(>=3.5.0)License GPL-2|file LICENSEURL https:///noemiallefs/andurinhaBugReports https:///noemiallefs/andurinha/issues Encoding UTF-8LazyData trueImports signal,tidyr,ggplot2,cowplot,rlang,utils,plyrSuggests extrafont,dplyr,knitr,rmarkdown,testthat,MASSRoxygenNote7.1.1VignetteBuilder knitrNeedsCompilation noAuthor Noemi Alvarez Fernandez[aut,cre](<https:///0000-0003-2690-4051>),Antonio Martinez Cortizas[aut](<https:///0000-0003-0430-5760>)Maintainer Noemi Alvarez Fernandez<*********************>Repository CRANDate/Publication2020-08-1308:40:02UTC12andurinhaData R topics documented:andurinha (2)andurinhaData (2)findPeaks (3)gOverview (4)importSpectra (5)plotPeaks (6)Index8 andurinha andurinha:Tools to make spectroscopic data processing easierDescriptionThis package contains a set of functions that makes spectroscopic data processing easier and faster.It is intended for processing several spectra(tens to hundreds)of samples with similar composi-tion.It compiles spectroscopy datafiles,produces standardised and second derivative spectra,finds peaks and allows to select the most significant ones based on the second derivative/absorbance sum spectrum.It also provides functions for graphic evaluation of the outputs.andurinha functions1.importSpectra:in case you have your spectra in separatedfiles(.csv)this function importsand bind them in a single data frame.2.findPeaks:finds peaks and allows to select the most relevant based on the second derivativesum spectrum.erview:generates a graphic overview of the spectroscopic data.4.plotPeaks:makes a graphic representation of the peaks over the second derivative/absorbancesum spectrum.andurinhaData Andurinha data setDescriptionA collection of FTIR-ATR mid-infrared spectra of peat samples with increasing degree of peathumification.UsageandurinhaDatafindPeaks3FormatA data frame with1736observations of3peat samples.•WN:wave numbers of the spectra.•A-C:absorbances of a FTIR spectra of three peat samples.SourceEcoPast research group,Universidade de Santiago de Compostela(Spain)https://ecopast.es See AlsoimportSpectra,findPeaks,plotPeaks and gOverviewfindPeaksfindPeaksDescriptionThis functionfinds peaks and allows to the most relevant based on the second derivative/absorbance sum spectrum.UsagefindPeaks(data,resolution=4,minAbs=0.1,cutOff=NULL,scale=TRUE,ndd=TRUE)Argumentsdata A data frame object,which contains in thefirst column the wave numbers and in the following columns the samples absorbances.resolution The equipment measurement resolution;by default4cm-1.minAbs The cut off value to check spectra quality;by default0.1.cutOff The second derivative/absorbance sum spectrum cut off to reduce the raw peaks table;by default NULL.scale By default(TRUE)the data is scaled by e FALSE in case you do not want to scale it.ndd By default(TRUE)the peaks are searched based on the second derivative sum e FALSE in case you want to search them based on the absorbancesum spectrum.4gOverview ValueA list with a collection of data frames which contains:1.dataZ:the standardised data by Z-scores.2.secondDerivative:the second derivative values of the data.3.sumSpectrum_peaksTable:the peaks wave numbers and their second derivative/absorbancesum spectrum values.4.peaksTable:the selected peaks wave numbers and their absorbance for each spectrum.See AlsoimportSpectra,gOverview and plotPeaksExamples#Find Peaks based on the absorbance sum spectrumfp.abs<-findPeaks(andurinhaData,ndd=FALSE)#See the peaks table of the absorbance sum spectrumfp.abs$sumSpectrum_peaksTable#Find Peaks based on the second derivative sum spectrumfp.ndd<-findPeaks(andurinhaData)#See the peaks table of the second derivative sum spectrumfp.ndd$sumSpectrum_peaksTable#Select a cutOff to reduce the number of peaks in the table#(i.e.select the most relevant)#fp.ndd$sumSpectrum_peaksTable%>%#arrange(desc(sumSpectrum))#Run findPeaks()with the new cutOfffp.ndd2<-findPeaks(andurinhaData,cutOff=0.25)gOverview gOverviewDescriptionThis function generates a graphic overview of the spectroscopic data.UsagegOverview(data_abs,data_ndd,fontFamily=NULL)importSpectra5 Argumentsdata_abs A data frame,which contains in thefirst column the wave numbers and in the following columns the samples absorbances.data_ndd A data frame,which contains in thefirst column the wave numbers and in the following columns the samples second derivative values.fontFamily The desired graphic font family.ValueIf data_ndd is provided:It returns a grid with three plots:•The ensemble of all samples spectra.•The ensemble of the second derivative spectra of all samples.•The average and standard deviation spectra.If data_ndd is omitted:It returns a grid with two plots:•The ensemble of all samples spectra.•The average and standard deviation spectra.See AlsoimportSpectra,findPeaks and plotPeaksExamples#Grapic overview of your raw datagOverview(andurinhaData)#Graphic overview of your processed data by findPeaks()fp<-findPeaks(andurinhaData)gOverview(fp$dataZ,fp$secondDerivative)importSpectra importSpectraDescriptionIn case you have your spectra in separatedfiles(.csv)this function imports and binds them in a single data frame.Thefiles directory must contain only the samplesfiles.UsageimportSpectra(path,sep=";")Argumentspath A character vector with the full path to the data directory;by default corresponds to the working directory,getwd.sep Thefield separator character;by default sep=";".ValueA data frame with the structure:•First column(WN):wave numbers of the spectra.•1-n:samples spectra(the column names correspond to thefiles names).See AlsofindPeaks,gOverview and plotPeaksExamples#Create an empty directory#Now create some spectra separate filesA<-andurinhaData[,1:2]B<-andurinhaData[,c(1,3)]C<-andurinhaData[,c(1,4)]MASS::write.matrix(A,file=tempfile(pattern="A.csv"),sep=";")MASS::write.matrix(A,file=tempfile(pattern="B.csv"),sep=";")MASS::write.matrix(A,file=tempfile(pattern="C.csv"),sep=";")#TryimportSpectra(path=paste0(tempdir(),"/"),";")plotPeaks plotPeaksDescriptionThis function makes a graphic representation of the peaks over the second derivative and/or ab-sorbance sum spectra.UsageplotPeaks(peaksWN,data_abs,data_ndd,fontFamily=NULL)ArgumentspeaksWN A vector with the peaks wave numbers.data_abs A data frame,which contains in thefirst column the wave numbers and in the following columns the samples absorbances.data_ndd A data frame,which contains in thefirst column the wave numbers and in the following columns the samples second derivative values.fontFamily The desired graphic font family.See AlsoimportSpectra,findPeaks and gOverviewExamples#Plot the peaks found by findPeaks()#1.Based on absorbance sum spectrumfp.abs<-findPeaks(andurinhaData,ndd=FALSE)plotPeaks(fp.abs[[3]]$WN,fp.abs$dataZ)#2.Based on second derivative spectrumfp.ndd<-findPeaks(andurinhaData,cutOff=0.25)plotPeaks(fp.ndd[[4]]$WN,fp.ndd$dataZ,fp.ndd$secondDerivative)Index∗datasetsandurinhaData,2andurinha,2andurinhaData,2findPeaks,2,3,3,5–7getwd,6gOverview,2–4,4,6,7importSpectra,2–5,5,7plotPeaks,2–6,68。
ENVI高光谱数据处理流程

ENVI高光谱数据处理流程一、显示图像波谱1.打开文件:主菜单中,File→Open Image File→文件名.raw或者Window→Available Bands List→File →Open Image File→文件。
2.显示真彩色图像:波段列表(Available Bands Lis)中,右键→Load TrueColor。
3.*设置像素大小:主窗口(Display)中,右键→Pixel Locator。
4.绘制波谱:主窗口中,右键→Z Profile(Spectrum)。
5.收集任意点波谱:Spectral Profile中,Options→Collect Spectra,点击图像任6.光谱平滑:Spectral Profile中,Options→Set Z Profile Avg Window,将window7.部分光谱:主菜单→Basic Tools→Resize Data(Spatial/Spectral)→Spectral Subset,选择需要的光谱波段。
生成新的文件,右键→Load True Color to<new>。
显示新图像。
8.关闭所有文件:File→Close All Files。
二、标准波谱库主菜单→Spectral→Spectral Libraries→Spectral Library Viewer→安装文件夹下,ITT\IDL\IDL80\products\envi48\spec_lib。
共有usgs_min、veg _lib、jpl_lib、jhu_lib四个标准波谱库。
在Spectral Library Viewer中,单击波谱名称,自动显示波谱。
三、自定义波谱库1.输入波长范围:在菜单中,Spectral Spectral Library→Spectral Library Builder2.波谱收集:以从影像数据中收集波谱为例:a)打开高光谱图像,收集任意点波谱。
高光谱数据分析ENVI操作手册

4.1. 设置参数
Kernel Size(卷积核大小):奇数。 Image Add Back(加回值):将原始图像中的部分加回到卷积滤波结果图像中, Editable Kernel(卷积核中各项的值)。
有助于保持图像的空间连续性。
滤波前
滤波后
北京卓立汉光仪器有限公司
5. 主成分分析列出各波段以及相应的百分比,可自主选择主成分波段。“No”系统会计 算特征值和显示供选择的输出波段。
5.2. 协方差矩阵、特征向量矩阵的统计
主菜单,Basic ToolsStatisticsView Statistics File,打开主成分分析中得到 的统计文件,可以得到各个波段的基本统计值、协方差矩阵、相关系数矩阵和特 征向量矩阵。 当协方差矩阵数据量较大时,不能直接在统计文件中显示,这时可通过输出 ASCII文件并导入到excel中来查看协方差矩阵和特征向量矩阵。 波长、 反射率和协方差矩阵、特征向量矩阵的数据分析可采用其他数值统计 分析软件进行。
2.2. 添加注记
在Spectral Library Plots窗口中,Option→Annotate Plot,手动添加注记,如文 Annotation窗口中,Object选择注记类型后,在Spectral Library Plots窗口中左 在Spectral Library Plots窗口中,右键→Plot Key,添加注记,名称和颜色在
选择Memory或在Enter Output Filename输入文件名生成新的文件。 右键→Load True Color to<new>,显示新图像。
北京卓立汉光仪器有限公司
1.6. 光谱数据输出
光谱曲线窗口中,File→Save Plot As→ASCII,在Output Plots to ASCII File文 件中,Selsct Plot To Output选中需要输出曲线的点,输出路径和名称,OK。
ENVI基本操作

ENVI基本操作ENVI(Environment for Visualizing Images)是一款功能强大的遥感数据处理和分析软件。
它提供了许多基本操作,可以帮助用户处理和分析遥感图像。
下面将介绍一些ENVI的基本操作。
1. 数据加载:打开ENVI软件后,可以通过点击"File",然后选择"Open Data File"来加载遥感图像数据。
用户可以选择打开各种格式的遥感数据,如多光谱数据、高光谱数据、雷达数据等。
2.图像显示:在加载数据后,ENVI会自动显示图像。
可以使用鼠标在图像上点击并拖动来滚动图像。
用户还可以使用放大和缩小工具来调整图像显示的比例。
3.图像增强:ENVI提供了多种图像增强的工具,如直方图均衡化、对比度拉伸、滤波等。
用户可以根据自己的需求选择不同的增强方法,以改善图像的可视化效果。
4. 像元值提取:ENVI允许用户在图像上选择一个或多个像元,并提取其像元值。
用户可以通过点击"View",然后选择"Pixel Inspector"来使用像元值提取工具。
5.特征提取:ENVI提供了多种特征提取的工具,如主成分分析(PCA)、线性判别分析(LDA)、像元不相关特征转换(ICASSP)等。
这些工具可以帮助用户从遥感图像中提取有用的特征信息。
6.分类:ENVI提供了多种分类算法,如最大似然分类、支持向量机(SVM)、随机森林等。
用户可以根据自己的需求选择不同的分类算法,并根据已知的地物类别进行训练和分类。
7.可视化和分析:ENVI提供了丰富的可视化和分析工具,如波段组合、变化检测、空间统计分析等。
用户可以使用这些工具来进一步分析和理解遥感图像。
8. 数据输出:ENVI允许用户将处理和分析后的结果以不同的格式输出,如图像文件、矢量文件、栅格数据等。
用户可以通过点击"File",然后选择"Save As"来保存结果。
ERDAS软件操作指南
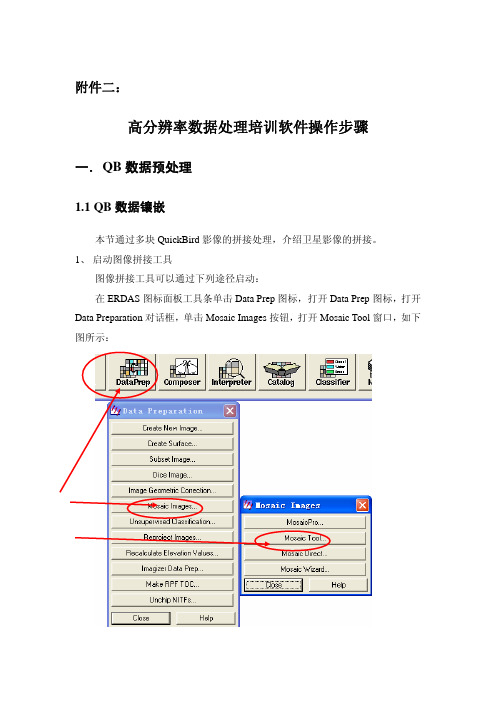
附件二:高分辨率数据处理培训软件操作步骤一.Q B数据预处理1.1 QB数据镶嵌本节通过多块QuickBird影像的拼接处理,介绍卫星影像的拼接。
1、启动图像拼接工具图像拼接工具可以通过下列途径启动:在ERDAS图标面板工具条单击Data Prep图标,打开Data Prep 图标,打开Data Preparation对话框,单击Mosaic Images按钮,打开Mosaic Tool窗口,如下图所示:2、加载Mosaic图像在Mosaic Tool工具条单击Add Images,打开Add Images for Masaic对话框。
在Add Images for Masaic对话框中,需要设置以下参数:(1) 选择拼接图像。
(2) 单击Add按钮,加入图像。
(3) 重复前3步骤(也可同时一次加入多幅图像)。
(4) 单击Close按钮。
3、运行Mosaic工具在Mosaic Tool菜单条单击Process|Run Mosaic命令(或直接点击),打开Run Mosaic对话框,在Run Masaic对话框中设置如下参数:(1) 确定输出文件名。
(2) 单击OK。
4、退出Mosaic工具1.2 多文件单命令批处理点击菜单栏Session | Open Batch Command File 输入*.bcf批处理文件的路径,点击OK。
弹出Batch Command对话框点击Next,弹出如下对话框:点击按钮,添加需要处理的文件或者文件路径。
(路径设置在InputDir 对话框中删除所选择的文件名称,即默认为选中文件所在的文件夹,对文件夹下所有文件做相同处理),设置完成点击Finish即可。
1.3 photoshop影像拼接本节将介绍利用Photoshop进行无人机影像的拼接处理。
二.数据拉伸步骤如下图所示:(1)打开数据拉伸对话框(2)对拉伸对话框中的各项参数进行设置拉伸的目的是把16位的QB数据,转换为无符号8位数据。
高光谱数据处理基本操作规范

高光谱分辨率遥感用很窄(10-2l)而连续的光谱通道对地物持续遥感成像的技术。
在可见光到短波红外波段其光谱分辨率高达纳米(nm)数量级,通常具有波段多的特点,光谱通道数多达数十甚至数百个以上,而且各光谱通道间往往是连续的,每个像元均可提取一条连续的光谱曲线,因此高光谱遥感又通常被称为成像光谱(ImagingSpectrometry)遥感。
行输出。
三、波谱库1、标准波谱库软件自带多种标准波谱库,单击波谱名称可以显示波谱信息。
2、自定义波谱库ENVI提供自定义波谱库功能,允许基于不同的波谱来源创建波谱库,波谱来源包括收集任意点波谱、ASCII文件、由ASD波谱仪获取的波谱文件、感兴趣区均值、波谱破面和曲线等等。
3、波谱库交互浏览波谱库浏览器提供很多的交互功能,包括设置波谱曲线的显示样式、添加注记、优化显示曲线等四、端元波谱提取端元的物理意义是指图像中具有相对固定光谱的特征地物类型,它实际上代(1)MNF变换了主成分分析在高光谱数据处理中的不足。
(2)计算纯净像元指数PPIPPI生成的结果是一副灰度的影像,DN值越大表明像元越纯。
作用及原理:纯净像元指数法对图像中的像素点进行反复迭代,可以在多光谱或者高光谱影像中寻找最“纯”的像元。
(通常基于MNF变换结果来进行)纯净像元指数可以将N维散点图映射为一个随机单位向量来计算,每次映射的极值像元被记录下来,并且被标为极值的总数也被记录下来。
按照多次映射每个像元被记录为极值像元的次数来决定该像元是否为纯净像元。
(3)端元波谱收集n维可视化工具-选取样本像元-生成地物平均波谱五、波谱识别和图像分类4、高光谱和多光谱实质上的差别就是,高光谱的波段较多,谱带较窄(比如hyperion有242个波段,带宽10nm);5、多光谱相对波段较少;6、高光谱遥感就是比多光谱遥感的光谱分辨率更高,但是光谱分辨率高的同时空间分辨率会降低。
高光谱数据处理流程

高光谱数据处理流程第一篇嘿,亲爱的小伙伴们!今天咱们来聊聊高光谱数据处理流程这个超有趣的话题。
一开始呀,拿到高光谱数据就像是拿到了一个神秘的宝藏盒子。
得先进行数据的预处理,这就好比给宝藏盒子打扫打扫卫生,把那些不干净的、乱糟糟的东西清理掉。
比如说,要去除噪声,就像把耳边的杂音去掉,让数据更清晰、更纯净。
然后呀,是特征提取。
这就像是把宝藏的独特之处找出来,让它们更加显眼。
通过一些巧妙的方法,把数据里隐藏的重要特征给揪出来。
再之后呢,就是分类和识别啦。
这就像是给不同的宝藏贴上标签,让咱们清楚地知道它们是什么,有什么特别的地方。
可别忘了对处理结果进行评估哦。
就好像检查一下咱们挑出来的宝藏是不是真的宝贝,处理得好不好。
怎么样,高光谱数据处理流程是不是很有意思呀?第二篇嗨哟,亲爱的朋友们!今天来和大家讲讲高光谱数据处理流程哟!拿到高光谱数据的时候,感觉就像拿到了一本神秘的魔法书。
第一步,咱们得给它来个“梳洗打扮”,也就是做数据的校准。
把那些不准确的、歪歪扭扭的数据给纠正过来,让它们乖乖排好队。
然后呢,要做辐射定标。
这就好像给魔法书里的文字加上特别的标记,让它们的意思更明确。
接着哟,是进行几何校正。
就像是把魔法书里歪掉的图片摆正,让一切看起来都整整齐齐的。
再往下走,是大气校正。
这就像是给魔法书吹走那些挡住视线的迷雾,让里面的内容清晰可见。
做完这些,就是光谱分析啦。
这时候就像是在魔法书里寻找神奇的线索,看看能发现什么奇妙的东西。
还有哦,图像分类也不能少。
把相似的内容归到一起,就像给魔法书里的故事分类一样。
最后呀,还要反复检查,看看咱们的处理是不是完美,有没有遗漏的小细节。
高光谱数据处理流程就像一场奇妙的冒险,充满了惊喜和挑战!。
gdal读取高光谱

**使用GDAL读取高光谱数据**高光谱数据是遥感领域中常见的一种数据类型,它提供了丰富的地物光谱信息。
为了有效地处理和分析这些数据,我们需要使用专门的工具来读取和操作它们。
GDAL(Geospatial Data Abstraction Library)是一个开源的地理空间数据转换库,它能够读取、写入和处理多种栅格数据格式,使得处理高光谱数据变得相对简单。
**1. 安装GDAL**首先,确保你的系统中已经安装了GDAL。
你可以通过包管理器或直接从GDAL的官方网站下载并安装。
**2. 导入必要的库**在Python中使用GDAL,首先需要导入相关的库:```pythonfrom osgeo import gdal, gdalconst```**3. 读取高光谱数据**假设我们有一个ENVI格式的高光谱数据文件(通常包括一个头文件和一个或多个二进制文件),我们可以使用以下代码读取它:```python# 指定文件路径file_path = "path_to_your_hyperspectral_data"# 打开数据dataset = gdal.Open(file_path, gdalconst.GA_ReadOnly)# 获取数据的宽度、高度和波段数width = dataset.RasterXSizeheight = dataset.RasterYSizebands = dataset.RasterCount# 遍历每个波段并读取数据for band in range(1, bands + 1):band_data = dataset.GetRasterBand(band).ReadAsArray()# 此处可以对每个波段的数据进行进一步处理```**4. 数据处理和分析**一旦你读取了数据,你可以进行各种处理和分析,例如计算统计信息、应用滤波器、提取特征或执行分类。
**5. 关闭数据集**数据处理完成后,记得关闭数据集以释放资源:```pythondataset = None # 关闭数据集```**总结**:通过GDAL,我们可以轻松地读取和处理高光谱数据。
高光谱ENVI使用方法简介

高光谱制图—FLAASH大气校正
FLAASH是目前精度最高的大气辐射校正模型, 使用了 MODTRAN 4+ 辐射传输模型的代码,基 于像素级的校正 FLAASH可对Landsat, SPOT, AVHRR, ASTER, MODIS, MERIS, AATSR, IRS等多光谱、高光谱 数据、航空影像及自定义格式的高光谱影像进行 快速大气校正分析。能有效消除大气和光照等因 素对地物反射的影响,获得地物较为准确的反射 率和辐射率、地表温度等真实物理模型参数
高光谱制图—FLAASH大气校正(5)
如果要自动保存前面所输入的FLAASH参 数 如果需要生成相关诊断文件(如通道定义 文件等)
高光谱影像地理坐标定位
空间遥感平台在传感器采集数据的同时也精确地 记录了自身的几何信息,使用这些几何信息如星 历、姿态数据以及传感器探元与成像数据上像元 间的几何关系等,可以计算出影像上每一个像元 所对应的经纬度,其结果将作为影像数据的辅助 地理信息一并打包发布给用户。利用这些详细的 输入几何信息(Input Geometry)使得影像不需 要选择大量地面控制点就可以进行几何精纠正, 即ENVI所谓的地理坐标定位Georeference)。
比较N维散点图和二维散点图 利用N维散点图进行端元选取,理解使用菜 单Class Controls的使用 N维可视化仪同光谱剖面的链接,使用鼠 标中键来进行光谱曲线的绘制 光谱分析与N维可视化仪连接起来
高光谱影像分析-光谱切面
光谱切面包括水平切面、垂直切面和任意 方向切面。 切面是一幅ENVI影像,沿水平方向的切面, 样本数等于光谱波段数,行数等于采样数; 沿垂直方向的切面,样本数等于行数;对 于任意方向的切面,样本数等于沿ROI折 线的像元总数
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
高光谱数据处理工具软件使用手册
一数据读取操作
1 读取南京中地仪器公司的光谱数据(单个文件)
[sampleName,lambda,spectrum ]=hyperReadZD(fname)
2 读取北师大波谱库的数据(单条记录)
[metaInfo,lambda,spectrum]=hyperReadSPL(fname,iStart,iEnd,i Step,desPathStr)
参数:如果输入参数iStart,iEnd,iStep则将读取的波谱数据按照以上三个参数进行重采样。
三参数的含义分别是:起始波段(nm),终止波段(nm),采样间隔(nm);如果输入desPathStr,则将采样后的结果作为zip文件保存在目录desPathStr内。
3 读取Envi波谱库数据(单个文件,多条记录)
[samplename,lambda,spectral]=hyperReadEnvi(fname)
4 读取ASD波谱仪数据
[measured, lambda, reference] = hyperReadAsd(filename)
5 读取高光谱影像数据(AVIRIS格式)
[M, wavelengths_nm] = hyperReadAvirisRfl(filename, height, width, bands)
二数据转换
1 高光谱立方体数据转换为二维数组
[M] = hyperConvert2d(M)
输入:
M - 高光谱立方体数据(m x n x p)
输出:
M –二维矩阵形式(p x N)
2 将二维数组转换为数据立方体
[img] = hyperConvert3d(img, h, w, numBands)
输入:
M –二维数据矩阵 (p x N)
输出:
M –三维数据立方体 (m x n x p)
3 光谱重采样
[ output ] = hyperResample( M, currentWaveLengths, desiredWaveLengths )
输入:
M –二维高光谱数据矩阵 (p x N)
currentWavelengths –数据M的当前波长.(p x 1)
desiredWavelengths –将要转换的目标波长
输出:
M_resampled –将M按照目标波长重新采样的结果。
三光谱变换
1 主成份变换
[M_pct] = hyperPct(M, q)
输入:
M –二维数据矩阵(p x N)
q –保留主成份个数
输出:
M_pct –变换后的二维数据矩阵(q x N) which is result of transform V –转换系数.
2 最小噪声分离技术
M = hyperMnf(M, h, w)
输入
M –二维数据矩阵 (p x N)
h –行数
w –列数
Outputs
M –变换后的二维数据矩阵
H –转换系数
四光谱解混(端元提取)
1 高光谱自动目标提取
[ U, indices ] = hyperAtgp( M, q, Maug )
输入
M -二维数据矩阵(p x N)
q –端元个数
Maug –端元先验光谱(I可选),(p x (# targets))
输出
U –端元光谱矩阵 (p x q)
indices –端元在矩阵M中的位置
2 像元纯度指数法
[U] = hyperPpi(M, q, numSkewers)
输入
M –二维 (p x N)
q –提取端元个数
numSkewers - Number of "skewer" vectors to project data onto. 输出
U - Recovered endmembers (p x N)
五相对含量计算(丰度成图)
[ X ] = hyperFcls( M, U )
输入
M –高光谱数据矩阵(p x N)
U –端元光谱(p x q)
输出
X –含量计算结果 (q x N)
六光谱相似性测量
1 光谱角计算(SAM)
[errRadians] = hyperSam(a, b)
输入:
a –目标光谱.
b –参考光谱.
输出:
errRadians –光谱角(弧度)。
