GeneBank的使用资料
genbank数据库格式注释

GenBank(Gene Bank)数据库是一个包含大量基因序列和相关注释信息的公共数据库。
该数据库由美国国家生物技术信息中心(NCBI)维护管理,是全球最大的基因组序列数据库之一。
GenBank数据库中包含了来自不同生物种类的数百万条DNA序列和蛋白质序列,为生物学研究提供了重要的数据资源。
1. GenBank数据库的格式GenBank数据库中的每一条记录都按照一定的格式进行注释,以便用户能够快速准确地获取所需的信息。
其格式一般包括以下几个部分:(1)LOCUS:该部分包括了记录的名称、长度、分子类型等基本信息。
(2)DEFINITION:该部分包括了对记录的简要描述和功能注释。
(3)ACCESSION:该部分包括了该记录在数据库中的唯一编号。
(4)VERSION:该部分包括了记录的版本信息和更新日期。
(5)KEYWORDS:该部分包括了与记录相关的关键词和主题词。
(6)SOURCE:该部分包括了记录所对应的生物学来源和生物信息学来源。
(7)ORIGIN:该部分包括了记录的序列数据。
(8)FEATURES:该部分包括了记录的特征、基因结构和编码蛋白质等信息。
(9)REFERENCE:该部分包括了记录的文献来源和引用信息。
2. GenBank数据库的使用GenBank数据库的注释格式为用户提供了方便快捷地获取所需基因序列和注释信息的方法。
用户可以通过关键词、编号、序列相似性等方式来检索数据库中的记录,并获取所需的数据。
用户还可以利用数据库提供的分析工具和数据可视化工具对基因序列进行进一步的研究和分析。
3. GenBank数据库的意义GenBank数据库作为全球最大的基因组序列数据库之一,为生物学研究提供了重要的数据资源和工具。
研究人员可以通过该数据库获取各种生物物种的基因序列和相关注释信息,从而加快基因功能研究的进程。
GenBank数据库中的数据也为生物信息学研究和生物医学领域的发展提供了重要支持。
NCBI及GeneBank介绍(CHENGWEI)-XXXX0327

3. 检索事例
检索号:JX984951 Norovirus Hu/GII.4/GZ2010-
L88/Guangzhou/CHN/2011 capsid protein (VP1) gene, complete cds
cds:Coding sequence.
电子显微镜下 诺如病毒形态
诺如病毒三维结构
2.2. PubMed
PubMed comprises more than 22 million
citations for biomedical literature from MEDLINE((美)联机医学文献分析和检索 系统), life science journals, and online books. Citations may include links to fPuulbl-Mteexdt由c源on自ten(t 美fro)m联P机ub医Me学d文C献en分tra析l 和a检nd索pu系bl统ish、er生w命eb科sit学es杂. 志和网上图书的 超过22,000,000篇生物医学引文组成。引 文也可能链接自PubMed Central 和出版 商网站的全文。
GenBank识别 标志
意义
LOCUS ACCESSION
标识字符串及短描述字 唯一的提取号
DEFINITION VERSION KEYWORDS SOURCE ORGANISM REFERENCE
简单的描述 可更新的序列版本号 关键字 来源生物体 生物体分类谱系 引文编号
AUTHORS TITLE JOURNAL
STSs(Sequenced tagged site):短的在 基因组上可以被唯一操作的序列,用于产 生作图位点。
在操作中,STS是用于辨别PCR引物对并生 成作图试剂的唯一的序列,每个STS序列位 点对应于基因组中一个单独的位置。
gene bank
限定词
/codon_start= /country= /db_xref= /direction=
含 义
相对于序列第一个碱基, 相对于序列第一个碱基, 编码序列密码子的偏移量 DNA样本的来源国 DNA样本的来源国 其他数据库信息的交叉索 引号 DNA复制方向 DNA复制方向
/environmental 序列直接从环境材料中获 _sample= 得而没有指明来源物种
无法用重组特性关键 词描述的重组事件 通过重组所消除的 DNA
repeat_region
基因组中所包含的重复序列 iDNA
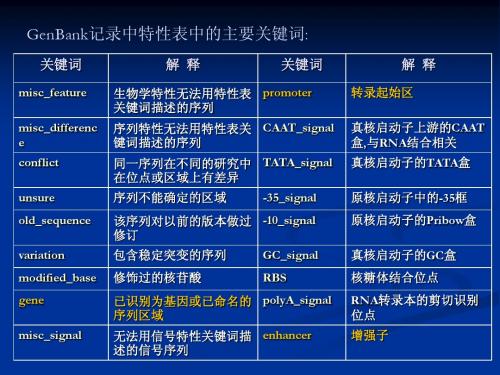
关键词
misc_structure stem_loop D_loop
解 释
无法用结构关键词描述的核 酸序列高级结构或构型 发夹结构 线粒体中DNA中的取代环 线粒体中DNA中的取代环
生物学特性无法用特性表 promoter 关键词描述的序列
misc_difference 序列特性无法用特性表关 CAAT_signal 键词描述的序列 conflict unsure old_sequence variation modified_base gene misc_signal 同一序列在不同的研究中 TATA_signal 在位点或区域上有差异 序列不能确定的区域 -35_signal 该序列对以前的版本做过 -10_signal 修订 包含稳定突变的序列 修饰过的核苷酸 GC_signal RBS
/transposon= 转座子
含 义
获得序列的生物变种 假基因 表明特性间的间隔序 列已被替换 重复序列的组织方式 获得序列的分子类型 同一原核生物的血清 学特征 获得序列的天然宿主 特性的通用名称 获得序列的亚克隆
指明重复区域的重复元件构 /variety= 成 同一物种的不同血清学特征 /pseudo 获得序列的物种性别 /replace=
GeneBank的使用

GenBank数据库
n 物种:GenBank 库里的数据按来源于大约100,000个 物种,其中56%是人类的基因组序列(所有序列中的 34%是人类的EST序列)
n 记录:每条GenBank数据记录包含对序列的简要描 述,它的科学命名,物种分类名称,参考文献,序 列特征表,及序列本身
GenBank数据库
ACCESSION
n ACCESSION (编号):具有唯一性和永久性,在文 献中引用这个序列时,应该以此编号为准。
KEYWORDS
n KEYWORDS (关键词)字段:由该序列的提交者提 供,包括
– 该序列的基因产物 – 其它相关信息
SOURCE
n SOURCE (数据来源)字段:说明该序列是从什么生 物体、什么组织得到的
n 序列特征表:包含对序列生物学特征注释如:编码 区、转录单元、重复区域、突变位点或修饰位点等
n 分类:所有数据记录被划分为如细菌类、病毒类、 灵长类、啮齿类,以及EST数据、基因组测序数据 、大规模基因组序列数据等16类,其中EST数据等 又被分成若干文件
注释内容
n 序列条目关键字:
– LOCUS (代码), – DEFINITION (说明), – ACCESSION(编号), – NID符(核酸标识), – KEYWORDS (关键词), – SOURCE (数据来源), – REFERENCE (文献), – FEATURES (特性表), – BASE COUNT (碱基组成) – ORIGIN (碱基排列顺序)。
n 次关键字ORGANISM (种属):指出该生物体的分类 学地位
REFERENCE
n REFERENCE(文献)字段:说明该序列中的相关文献 ,包括
[知识]如何在genbank中查找一基因的序列
![[知识]如何在genbank中查找一基因的序列](https://img.taocdn.com/s3/m/03c94e7dae1ffc4ffe4733687e21af45b207fe59.png)
如何在genbank中查找一基因的序列1、在GeneBank 中查找基因序列只要输入accession号就可以了,下面网址就是一个基因的全部序列信息的例子,/Sitemap/samplerecord.html,在记录的末尾有各种记录的详细说明,如果你没有accession号,可以把你手头的编号用source等信息源转换成accession号,中文教程太古老了,如果你是初学者一定要养成看英文文献的习惯,要是特别想看中文翻译的话,书店里随便一本生物信息学书里都会介绍数据库的,不过有些翻译过来的东西真的很别扭,希望对你有帮助。
2、关于在GeneBank中查找序列我有几点体会:最直接、最简单的方法是手头有基因的accession号;如果没有就需要明确两个重要的内容,即基因名称及物种信息(如果有最好是拉丁全名),基因名称尽可能详细,避免搜出一些不相关的信息;搜索的时候建议先用NCBI的Gene数据库搜索,这样得到的accession号是属于NCBI工作人员重新整理过的Refseq的序列,这样会比较可靠;当然这个要看你的分析目的,如果你是要对该序列进行下游的分子生物学操作or分析,选这种序列我觉得会比较好,如果是要进行多序列的分析or其他目的需要全面分析该序列的,可能需要其他序列做补充,但是我觉得序列越多问题越说不清楚,因为毕竟不是自己的序列,如果Gene数据库里没有收录,那就只有在Nucleotide数据库里找了,但是还是建议采用Refseq的序列,Refseq序列特征如下:Accession prefix Molecule type CommentAC_ Genomic Complete genomic molecule, alternate assemblyNC_ Genomic Complete genomic molecule, reference assemblyNG_ Genomic Incomplete genomic regionNT_ Genomic Contig or scaffold, clone-based or WGSaNW_ Genomic Contig or scaffold, primarily WGSaNS_ Genomic Environmental sequenceNZ_b Genomic Unfinished WGSNM_ mRNANR_ RNAXM_c mRNA Predicted modelXR_c RNA Predicted modelAP_ Protein Annotated on AC_ alternate assemblyNP_ ProteinYP_c ProteinXP_c Protein Predicted modelZP_c Protein Predicted model, annotated on NZ_ genomic recordsa Whole Genome Shotgun sequence data.b An ordered collection of WGS for a genome.c Computed.其他值得考虑的是,对于真核生物最好找注释为全长的mRNA序列,原核生物最好有起始密码子和终止密码子;其他未尽事宜大家补充!3、如何在genbank查找某个细菌的基因序列?你输入这个细菌的名字直接查,一般会有的~~~~~而且一般第一个会是全基因组序列~~~进入ncbi的首页,database选nucleotide,输入你的关键词,如果库里收录里就会有的4、如何查找基因序列?——在Genbank中寻找目的基因的实例(1)根据文献搞reasearch肯定要读文献的,如果你曾经在文献中看到过你感兴趣的基因,而且文中还提到了该基因在Genbank中的ID号,那就好办了,直接打开 ,在Search后的下拉框中选择Nucleotide,把Genbank ID号输入GO前面的文本框中,点“GO”,就可以找到他了。
如何在genbank中查找一基因的序列

如何在genbank中查找一基因的序列如何在genbank中查找一基因的序列1、在GeneBank 中查找基因序列只要输入accession号就可以了,下面网址就是一个基因的全部序列信息的例子,,在记录的末尾有各种记录的详细说明,如果你没有accession号,可以把你手头的编号用source 等信息源转换成accession号,中文教程太古老了,如果你是初学者一定要养成看英文文献的习惯,要是特别想看中文翻译的话,书店里随便一本生物信息学书里都会介绍数据库的,不过有些翻译过来的东西真的很别扭,希望对你有帮助。
2、关于在GeneBank中查找序列我有几点体会:最直接、最简单的方法是手头有基因的accession号;如果没有就需要明确两个重要的内容,即基因名称及物种信息(如果有最好是拉丁全名),基因名称尽可能详细,避免搜出一些不相关的信息;搜索的时候建议先用NCBI的Gene数据库搜索,这样得到的accession号是属于NCBI工作人员重新整理过的Refseq的序列,这样会比较可靠;当然这个要看你的分析目的,如果你是要对该序列进行下游的分子生物学操作or分析,选这种序列我觉得会比较好,如果是要进行多序列的分析or其他目的需要全面分析该序列的,可能需要其他序列做补充,但是我觉得序列越多问题越说不清楚,因为毕竟不是自己的序列,如果Gene数据库里没有收录,那就只有在Nucleotide数据库里找了,但是还是建议采用Refseq的序列,Refseq序列特征如下:Accession prefix Molecule type CommentAC_ Genomic Complete genomic molecule, alternate assemblyNC_ Genomic Complete genomic molecule, reference assemblyNG_ Genomic Incomplete genomic regionNT_ Genomic Contig or scaffold, clone-based or WGSaNW_ Genomic Contig or scaffold, primarily WGSaNS_ Genomic Environmental sequenceNZ_b Genomic Unfinished WGSNM_ mRNANR_ RNAXM_c mRNA Predicted modelXR_c RNA Predicted modelAP_ Protein Annotated on AC_ alternate assemblyNP_ ProteinYP_c ProteinXP_c Protein Predicted modelZP_c Protein Predicted model, annotated on NZ_ genomic recordsa Whole Genome Shotgun sequence data.b An ordered collection of WGS for a genome.c Computed.其他值得考虑的是,对于真核生物最好找注释为全长的mRNA序列,原核生物最好有起始密码子和终止密码子;其他未尽事宜大家补充!3、如何在genbank查找某个细菌的基因序列你输入这个细菌的名字直接查,一般会有的~~~~~而且一般第一个会是全基因组序列~~~进入ncbi的首页,database选nucleotide,输入你的关键词,如果库里收录里就会有的4、如何查找基因序列——在Genbank中寻找目的基因的实例(1)根据文献搞reasearch肯定要读文献的,如果你曾经在文献中看到过你感兴趣的基因,而且文中还提到了该基因在Genbank中的ID号,那就好办了,直接打开,在Search后的下拉框中选择Nucleotide,把Genbank ID 号输入GO前面的文本框中,点“GO”,就可以找到他了。
GeneBank

GenBank数据库简介1. GenBank属于一个序列数据库的国际合作组织,包括EMBL和DDBJ。
是NIH 遗传序列数据库,一个所有可以公开获得的DNA序列的注释过的收集。
GenBank 同日本和欧洲分子生物学实验室的DNA数据库共同构成了国际核酸序列数据库合作。
唯一人类基因序列集合(UniGene),人类基因组基因图谱,分类学浏览器,同国立癌症研究所合作的癌症基因组剖析计划(CGAP)等数据库。
GenBank以指数形式增长,核酸碱基数目大概每14个月就翻一个倍。
2. 纪录样本- 关于GenBank的各个字段的详细描述,以及同Entrez搜索字段的交叉索引。
3. 访问GenBank - 通过Entrez Nucleotides来查询。
用accession number,作者姓名,物种,基因/蛋白名字,还有许多其他的文本术语来查询。
关于Entrez 更多的信息请看下文。
用BLAST来在GenBank和其他数据库中进行序列相似搜索。
用E-mail来访问Entrez和BLAST可以通过Query和BLAST服务器。
另外一种选择是可以用FTP下载整个的GenBank和更新数据。
4. 增长统计- 参见公布通知的 2.2.6(每个分类的统计),2.2.7(每个物种的统计),2.2.8(GenBank增长)小节。
5. 公布通知,最新- 最近和即将有的变化,GenBank的分类,数据增长统计,GenBank的引用。
6. 公布通知,旧- 同上相同,是过去公布的统计。
7. 遗传密码- 15个遗传密码的概要。
用来确保GenBank中纪录的编码序列被正确的翻译。
向GenBank提交数据:1. 关于提交序列数据,收到accession number,和对纪录作更新的一般信息。
2. BankIt - 用于一条或者少数条提交的基于WWW的提交工具软件。
(请在提交前用VecScreen去除载体)3. Sequin - 提交软件程序,用于一条或者很多条的提交,长序列,完整基因组,alignments,人群/种系/突变研究的提交。
genbank序列条目的主要内容

一、GenBank序列条目的概述GenBank是一个公共数据库,收录了全球范围内大量的生物学序列信息。
这些序列包括了DNA序列、RNA序列以及蛋白质序列等。
GenBank数据库的建立旨在为科研人员提供一个信息共享的评台,以便更好地开展生物信息学研究和基因组学研究。
二、GenBank序列条目的结构GenBank序列条目通常由多个部分组成,主要包括以下几个方面的信息:1. LOCUS部分:该部分包括了序列的名称、长度、分子类型以及其他相关的信息。
2. DEFINITION部分:该部分包括了对序列的简要描述。
3. ACCESSION部分:该部分包括了序列的访问编号,用于标识该序列在数据库中的唯一性。
4. VERSION部分:该部分包括了序列的版本信息,用于标识同一序列的不同版本。
5. KEYWORDS部分:该部分包括了序列的关键词信息,便于用户进行检索和分类。
6. SOURCE部分:该部分包括了序列的来源信息,例如该序列来自哪种生物体。
7. ORGANISM部分:该部分包括了序列的生物学分类信息,例如属、种、亚种等信息。
8. REFERENCE部分:该部分包括了与该序列相关的文献引用信息。
9. FEATURES部分:该部分包括了序列的特征信息,例如基因的编码区域、启动子区域等。
10. BASE COUNT部分:该部分包括了序列中各种碱基的数量统计信息。
11. ORIGIN部分:该部分包括了序列的具体碱基序列信息。
三、GenBank序列条目的应用GenBank数据库中的序列信息对于生物学领域的研究具有重要意义。
科研人员可以通过GenBank数据库快速获取到所需的生物学序列信息,从而开展基因功能研究、进化分析、生物信息学分析等工作。
GenBank数据库也为生物学领域的大数据分析提供了重要的数据支持,有利于推动生物学研究的发展。
四、GenBank序列条目的质量保证为了确保GenBank数据库中的序列信息的准确性和可靠性,数据库管理人员会对提交的序列信息进行严格的筛选和审核。
如何在genbank中查找一基因的序列

如何在genbank中查找一基因的序列如何在genbank中查找一基因的序列1、在GeneBank 中查找基因序列只要输入accession号就可以了,下面网址就是一个基因的全部序列信息的例子,,在记录的末尾有各种记录的详细说明,如果你没有accession号,可以把你手头的编号用source 等信息源转换成accession号,中文教程太古老了,如果你是初学者一定要养成看英文文献的习惯,要是特别想看中文翻译的话,书店里随便一本生物信息学书里都会介绍数据库的,不过有些翻译过来的东西真的很别扭,希望对你有帮助。
2、关于在GeneBank中查找序列我有几点体会:最直接、最简单的方法是手头有基因的accession号;如果没有就需要明确两个重要的内容,即基因名称及物种信息(如果有最好是拉丁全名),基因名称尽可能详细,避免搜出一些不相关的信息;搜索的时候建议先用NCBI的Gene数据库搜索,这样得到的accession号是属于NCBI工作人员重新整理过的Refseq的序列,这样会比较可靠;当然这个要看你的分析目的,如果你是要对该序列进行下游的分子生物学操作or分析,选这种序列我觉得会比较好,如果是要进行多序列的分析or其他目的需要全面分析该序列的,可能需要其他序列做补充,但是我觉得序列越多问题越说不清楚,因为毕竟不是自己的序列,如果Gene数据库里没有收录,那就只有在Nucleotide数据库里找了,但是还是建议采用Refseq的序列,Refseq序列特征如下:Accession prefix Molecule type CommentAC_ Genomic Complete genomic molecule, alternate assemblyNC_ Genomic Complete genomic molecule, reference assemblyNG_ Genomic Incomplete genomic regionNT_ Genomic Contig or scaffold, clone-based or WGSaNW_ Genomic Contig or scaffold, primarily WGSaNS_ Genomic Environmental sequenceNZ_b Genomic Unfinished WGSNM_ mRNANR_ RNAXM_c mRNA Predicted modelXR_c RNA Predicted modelAP_ Protein Annotated on AC_ alternate assemblyNP_ ProteinYP_c ProteinXP_c Protein Predicted modelZP_c Protein Predicted model, annotated on NZ_ genomic recordsa Whole Genome Shotgun sequence data.b An ordered collection of WGS for a genome.c Computed.其他值得考虑的是,对于真核生物最好找注释为全长的mRNA序列,原核生物最好有起始密码子和终止密码子;其他未尽事宜大家补充!3、如何在genbank查找某个细菌的基因序列你输入这个细菌的名字直接查,一般会有的~~~~~而且一般第一个会是全基因组序列~~~进入ncbi的首页,database选nucleotide,输入你的关键词,如果库里收录里就会有的4、如何查找基因序列——在Genbank中寻找目的基因的实例(1)根据文献搞reasearch肯定要读文献的,如果你曾经在文献中看到过你感兴趣的基因,而且文中还提到了该基因在Genbank中的ID号,那就好办了,直接打开,在Search后的下拉框中选择Nucleotide,把Genbank ID 号输入GO前面的文本框中,点“GO”,就可以找到他了。
NCBI及GeneBank介绍(CHENGWEI)-XXXX0327

sGeeqnueBnacneks.是Ge美nB国an国k 立is 卫par生t o研f 究the院I维nte护rna的tio基nal 因N序uc列leo数tid据e 库Se,qu汇en集ce并D注ata释ba了se所C有oll公abo开ra的tio核n , 酸w序hi列ch。comprises the DNA DataBank of Japan 生以(LtGh物D及aerbeDn技oe欧BBroaa术rJ洲tgon)ar,k信y分n由it(zh息E子a美etMi中o生B国EnsL心u物国)er,xo建学acp立nhe立da实a卫nGng,验ee生nMd与B室a研ota日anl核究eok本cna苷u院atDlNda酸N下arCAil数B属数yBIbi.据国据oaTslhio库立库seg.sye 一起,都是国际核苷酸序列数据库集团的成 员。
2.5 核苷酸序列数据库 ——基本检索功能
(三)序列长度检索([SLEN])
2.5 核苷酸序列数据库 ——基本检索功能
(四)范围检索
1、序列接受号范围检索:
AF114696:AF114714[ACCN]
序列接受号的检索限定词为[ACCN]or[ACCESSION] 2、序列长度范围检索:
OMIM强调表型和基因型关系。每天更新,词目包 含大量的其他遗传资源。
/omim/
5.NCBI热门资源——ESTs
Expressed sequence tag
• ESTs表达序列标签,是一些短的(300~500bp)、单次 (测序)阅读的cDNA序列。它们代表了特定组织或发育 阶段表达的基因。也包括来自于差异显示和RACE实验的 cDNA序列。
genebank 序列格式 -回复

genebank 序列格式-回复什么是基因库(genebank)序列格式?基因库序列格式是一种用于存储和共享生物学资源的标准化数据格式。
基因库(genebank)是一个重要的生物信息资源,它收集并存储了大量的基因序列和相关的遗传信息。
基因库的建立旨在为科学家、研究人员和决策者提供可靠、准确和全面的基因数据,以促进生命科学研究和发展。
基因库序列格式采用一种结构化的方式将基因序列和相关信息进行编码和存储。
这种格式通常是基于文本文件,使用特定的语法和标记,使得生物学家和研究人员能够方便地读取、理解和解析这些序列数据。
基因库序列格式通常由国际标准组织或机构制定,并在全球范围内广泛应用和接受。
基因库序列格式的主要目的是确保基因数据的一致性、可比性和可重复性。
通过统一的格式和标准,科学家和研究人员可以轻松地比较和分析不同的基因序列,进而加快对生物遗传特征和进化关系的研究。
此外,基因库序列格式还可以为基因工程、农业改良和医药研发等领域提供重要的参考。
在基因库序列格式中,每个基因序列通常由一系列字母和数字的符号表示,代表不同的碱基组合。
这些序列通常以FASTA(一种常用的标准文件格式)或GenBank格式(一种基因库标准)的方式进行编码。
序列数据的文件通常包括多个字段,例如基因名、序列来源、序列长度、注释信息等。
此外,基因库序列格式还可以包括其他附加信息,如基因序列的剪接变异、启动子区域、开放阅读框(ORFs)等。
这些信息可以帮助科学家更全面地理解和研究基因序列的功能和特征。
在基因库建设和更新过程中,科学家和研究人员会对已知的基因序列进行注释和描述,并添加到相应的序列格式中,以便其他人可以使用和共享。
基因库序列格式的重要性不言而喻。
它为科学家和研究人员提供了一个标准化的平台,使他们能够共享、比较和分析基因序列数据。
通过基因库序列格式,科学家和研究人员可以更好地了解生物的进化历史、形态特征和功能特性,为解决各种生物学问题和应对全球性挑战提供有效的支持。
NCBI及GeneBank介绍(CHENGWEI)-20130327解读

(2)基因组DNA重叠群(NT_*):e.g.:NT_000347
(3)完整的基因组或染色体(NC_*):e.g.:NC_000907 (4)基因组的局部区域(NG_*):e.g.:NG_000019 (5)从人类基因组序列注释、加工得到的序列模型记 录 (XM,XP,or XR_*):e.g.:XM_000483
NCBI及GenBank数据库的使用
动物科学学院 程伟 2013年3月27日
内容提要
1.NCBI的介绍 2.GeneBank及PubMed的介绍 3.检索事例 4.BLAST (Basic Local Alignments Tool) 序列相似性比较工具介绍 5.NCBI热门资源介绍
1.WHAT IS NCBI?
2.4 GenBank数据库界面
点击进入核酸数据库 检索界面
2.5 核苷酸序列数据库
2.5 核苷酸序列数据库 ——基本检索功能
(一)限定词检索(基因名、物种名、作者等) (二)特殊标志符检索(AY123456、AF123456等) (三)序列长度检索([SLEN])
(四)范围检索
(五) 限制检索
2.5 核苷酸序列数据库 ——基本检索功能
(三)序列长度检索([SLEN])
2.5 核苷酸序列数据库 ——基本检索功能
(四)范围检索
1、序列接受号范围检索:
AF114696:AF114714[ACCN]
序列接受号的检索限定词为[ACCN]or[ACCESSION] 2、序列长度范围检索:
3、PDB序列接受号(Protein Data Bank ): 1个阿拉伯数字+3个字母。e.g.:1TUP
2.5 核苷酸序列数据库 ——基本检索功能
(二)特殊标志符的格式(核酸序列)
引物设计GeneBank数据库和软件的使用

切位点,若有则直接利用该酶切位点进行扩增;若无 可寻找与其基本相似的位点进行扩增。 (2)扩增片段里自身无启动子 在5’端增加的酶切位点(所选的酶切位点与启动子的 酶切位点相同)中必须含起始密码子
PCR引物设计基本思路
B:若扩增的DNA用于阻断
Melting temperature graph
Per 25-mer
GC% graph
Per 25-mer
Stability
Per 5-mer
第一步:在NCBI中搜索目标片段的核酸序列
选用短小芽孢杆菌菌株KS12的角蛋白酶序列
第二步:输入所要分析的核酸序列
载入待分析序列
Primer Premier 的启动界面
引物设计原理
引物设计的目的是为了找到一对合适的 核苷酸片段,使其能有效地扩增模板 DNA序列。引物设计总体上包含三个程 序:序列下载,同源性比较,引物设计 筛选 。
引物设计需要考虑的因素
引物长度(primer length) 产物长度 product length) 序列Tm值 (melting temperature) 引物与模板形成双链的内部稳定性(internal stability,
But …
引物编辑
引物编辑
Edit primer here
Analysis the edit result
Accept the edit result Return to the main window
Some other useful function of PP5
Enzyme
中间四个钮分 ADD: 从所有 DELETE:从 EDIT: 编辑酶 FILTER: 如果 筛选所需酶, 可 的接头Overha 接头为那几个 到酶切结果, 有 Table: 酶切位 SEQ; 整段序列
如何在genbank中查找一基因的序列

如何在genbank中查找一基因的序列GenBank是美国国立卫生研究院维护的基因序列数据库,汇集并注释了所有公开的核酸以及蛋白质序列。
每个记录代表了一个单独的、连续的、带有注释的DNA或RNA片段。
这些文件按类别分为几组:有些按照系统发生学划分,另外一些则按照生成这些序列数据的技术方法划分。
目前GenBank中所有的记录均来自于最初作者向DNA序列数据库的直接提交。
这些作者将序列数据作为论文的一部分来发表,或将数据直接公开。
GenBank由位于马里兰州Bethesda的美国国立卫生研究院下属国立生物技术信息中心建立,与日本DNA数据库(DDBJ)以及欧洲生物信息研究院的欧洲分子生物学实验室核苷酸数据库(EMBL)一起,都是国际核苷酸序列数据库合作的成员。
所有这三个中心都可以独立地接受数据提交,而三个中心之间则逐日交换信息,并制作相同的充分详细的数据库向公众开放(虽然格式上有细微的差别,并且所使用的信息系统也略有不同)。
GenBank数据库格式的详细说明/Sitemap/samplerecord.html1、在GeneBank中查找基因序列只要输入accession号就可以了,如果你没有accession号,可以把你手头的编号用source等信息源转换成accession号。
2、关于在GeneBank中查找序列我有几点体会:最直接、最简单的方法是手头有基因的accession号;如果没有就需要明确两个重要的内容,即基因名称及物种信息(如果有最好是拉丁全名),基因名称尽可能详细,避免搜出一些不相关的信息;搜索的时候建议先用NCBI的Gene数据库搜索,这样得到的accession号是属于NCBI工作人员重新整理过的Refseq的序列,这样会比较可靠;.其他值得考虑的是,对于真核生物最好找注释为全长的mRNA序列,原核生物最好有起始密码子和终止密码子;3、如何在genbank查找某个细菌的基因序列?输入这个细菌的名字直接查,一般就会找到,而且一般第一个会是全基因组序列。
如何在genbank中查找一基因的序列.docx

如何在genbank中查找一基因的序列.docx如何在 genbank 中查找一基因的序列1、在GeneBank 中查找基因序列只要输入accession号就可以了,下面网址就是一个基因的全部序列信息的例子,,在记录的末尾有各种记录的详细说明,如果你没有accession 号,可以把你手头的编号用source 等信息源转换成accession号,中文教程太古老了,如果你是初学者一定要养成看英文文献的习惯,要是特别想看中文翻译的话,书店里随便一本生物信息学书里都会介绍数据库的,不过有些翻译过来的东西真的很别扭,希望对你有帮助。
2、关于在GeneBank中查找序列我有几点体会:最直接、最简单的方法是手头有基因的accession号;如果没有就需要明确两个重要的内容,即基因名称及物种信息(如果有最好是拉丁全名),基因名称尽可能详细,避免搜出一些不相关的信息;搜索的时候建议先用NCBI 的Gene数据库搜索,这样得到的accession 号是属于 NCBI工作人员重新整理过的 Refseq 的序列,这样会比较可靠;当然这个要看你的分析目的,如果你是要对该序列进行下游的分子生物学操作 or 分析,选这种序列我觉得会比较好,如果是要进行多序列的分析or 其他目的需要全面分析该序列的,可能需要其他序列做补充,但是我觉得序列越多问题越说不清楚,因为毕竟不是自己的序列,如果 Gene 数据库里没有收录,那就只有在Nucleotide数据库里找了,但是还是建议采用Refseq 的序列,Refseq 序列特征如下:Accession prefix Molecule type CommentAC_Genomic Complete genomic molecule, alternate assemblyNC_Genomic Complete genomic molecule, reference assemblyNG_Genomic Incomplete genomic regionNT_Genomic Contig or scaffold, clone-based or WGSaNW_Genomic Contig or scaffold, primarily WGSaNS_Genomic Environmental sequenceNZ_b Genomic Unfinished WGSNM_mRNANR_RNAXM_c mRNA Predicted modelXR_c RNA Predicted modelAP_Protein Annotated on AC_ alternate assemblyNP_ProteinYP_c ProteinXP_c Protein Predicted modelZP_c Protein Predicted model, annotated on NZ_ genomic recordsa Whole Genome Shotgun sequence data.b An ordered collection of WGS for a genome.c Computed.其他值得考虑的是,对于真核生物最好找注释为全长的mRNA序列,原核生物最好有起始密码子和终止密码子;其他未尽事宜大家补充!3、如何在genbank查找某个细菌的基因序列你输入这个细菌的名字直接查,一般会有的~~~~~而且一般第一个会是全基因组序列~~~进入ncbi 的首页,database 选nucleotide ,输入你的关键词 , 如果库里收录里就会有的4、如何查找基因序列——在Genbank中寻找目的基因的实例(1)根据文献搞reasearch肯定要读文献的,如果你曾经在文献中看到过你感兴趣的基因,而且文中还提到了该基因在Genbank中的ID 号,那就好办了,直接打开,在Search后的下拉框中选择Nucleotide,把 Genbank ID 号输入GO前面的文本框中,点“GO”,就可以找到他了。
genebank 序列格式 -回复

genebank 序列格式-回复GeneBank 序列格式是一种常用的基因序列存储和共享格式。
它允许科学家们以统一的规范将基因序列数据上传到数据库中,并可供其他研究人员在全球范围内访问和使用。
GeneBank 序列格式不仅为基因组学和生物信息学研究提供了便捷的工具,还促进了基因组学领域的合作和交流。
GeneBank 序列格式的核心是使用一种特定的文本格式来描述基因序列。
这种格式以头部信息开始,包含有关该序列的重要元数据。
头部信息通常包括序列的名称、起始和终止位点、源细胞或生物体的详细信息以及其他注释信息。
通过头部信息,科学家可以了解序列来源、物种信息以及任何与该基因序列相关的实验条件或研究背景。
在头部信息之后,GeneBank 序列格式将基因序列本身以一种标准的方式进行编码和存储。
序列通常以一条完整的碱基序列的形式呈现,使用A、T、C 和G 表示腺嘌呤、胸腺嘧啶、胞嘧啶和鸟嘌呤四种碱基。
此外,序列中还可能包含一些特殊的标记和符号,用于表示突变、插入或删除等遗传变异信息。
在GeneBank 序列格式中,每个基因序列都有一个唯一的标识符号,通常称为“Accession Number”。
这个标识符号在全球范围内是唯一的,以便科学家们能够准确地引用和引用特定的基因序列。
此外,GeneBank 还为每个序列分配了一个版本号,以允许不断更新和改进序列数据。
这种机制确保了基因序列数据的可靠性和可追溯性。
GeneBank 序列格式还支持多种类型的注释信息。
与基因序列相关的注释可以包括编码蛋白质的氨基酸序列、启动子区域的定位、转录因子结合位点的预测以及可能存在的RNA或蛋白质结构域的标记。
这些注释信息使科学家们能够更全面地了解基因序列的功能和结构,从而指导进一步的基因组学研究和生物信息学分析。
利用GeneBank 序列格式,科学家们能够通过公共数据库共享和访问基因序列数据。
这种开放的合作模式极大地促进了基因组学领域的进步和创新。
genebank 序列格式 -回复

genebank 序列格式-回复Genebank序列格式是一种用于存储和共享生物学序列信息的标准格式。
它提供了一个规范的方式来记录DNA、RNA和蛋白质序列的数据,以便科学家和研究人员能够方便地访问和分析这些数据。
Genebank序列格式的主要目的是促进科学研究和生物学信息的共享和合作。
首先,Genebank序列文件通常以FASTA格式开始。
FASTA格式是一种文本格式,其中包含一个标识符行和一个或多个序列行。
标识符行以符号“>”开头,并包含有关序列的信息,通常是序列的名称或其他描述。
序列行是由包含碱基或氨基酸代码的字符组成的行。
每行通常限制为特定的字符数(例如80个字符),以便在打印或显示时保持可读性。
Genebank序列文件中的每个序列通常都有一个唯一的标识符。
这些标识符有助于区分不同的序列,并使其能够在Genebank数据库或其他生物信息学工具中进行检索。
标识符通常包括物种名称、序列源(例如细胞核、粒线体或叶绿体等)以及一些编号或版本信息。
除了FASTA格式,Genebank序列文件还包含一些元数据,如序列的长度、修饰信息和相关文献引用等。
这些信息在序列文件的开头以类似于注释的形式提供。
元数据部分以“LOCUS”行开始,紧跟着一些关键词和值对,用于描述序列的特征和属性。
这些关键词和值对提供了序列的位置、长度、分子类型和修饰信息等。
Genebank序列文件还包含有关序列来源和制备过程的信息。
这些信息被称为特征表,以“FEATURES”和“ORIGIN”关键词分隔。
特征表中的每一行描述了序列的一个属性,例如基因、转录起始位点或蛋白质结构域等。
每个属性由关键词和值对表示,关键词通常描述属性的类型,而值表示属性的具体信息。
特征表还可以包含一些修饰信息,如突变或插入序列等。
最后,Genebank序列文件以“”符号结尾,表示文件的结束。
该符号用于区分不同的序列或数据库记录,以便在解析和处理序列文件时能够正确识别序列的边界。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
D31716 特性表
关键字
CDs are recurring units in polypeptide chains
序列本身
D31716
序列本身
序列结束
4859 bp
D31716
GenBank数据记录
GenBank数据记录
GenBank数据库结构
GenBank序列文件由单个的序列条目组成。 序列条目由字段组成,每个字段由关键字起始,后面为该 字段的具体说明。
生物信息资源中心通过计算机网络提供该数据库文件。
注释条目:文章的格式
(/genbank/
Genbank
Genbank 查找页面
D31716
描述部分
CDs are recurring units in polypeptide chains (sequence and structure motifs), the extents of which can be determined by comparative analysis. Molecular evolution uses such domains as building blocks and these may be recombined in different arrangements to make different proteins with different functions. CD s编码序列,含终止密码子 polyA_signal 多聚A信号
KEYWORDS
KEYWORDS (关键词)字段:由该序列的提交者提供,包
括
• 该序列的基因产物
•
其它相关信息
SOURCE
SOURCE (数据来源)字段:说明该序列是从什么生物体、 什么组织得到的
次关键字ORGANISM (种属):指出该生物体的分类学地位
REFERENCE
REFERENCE(文献)字段:说明该序列中的相关文献,包括
录单元、重复区域、突变位点或修饰位点等
分类:所有数据记录被划分为如细菌类、病毒类、灵长类
、啮齿类,以及EST数据、基因组测序数据、大规模基因
组序列数据等16类,其中EST数据等又被分成若干文件
注释内容
序列条目关键字:
• LOCUS (代码), • DEFINITION (说明), • ACCESSION(编号), • NID符(核酸标识), • KEYWORDS (关键词), • SOURCE (数据来源), • REFERENCE (文献), • FEATURES (特性表), • BASE COUNT (碱基组成) • ORIGIN (碱基排列顺序)。 新版的核酸序列数据库将引入新的关键词SV (序列版本号),用“编 号.版本号”表示,并取代关键词NID
• AUTHORS (作者), • TITLE (题目)及 • JOURNAL(杂志名)等, 以次关键词列出。
MEDLINE的代码:该代码实际上是个超文本链接,点击它
可以直接调用上述文献摘要。
一个序列可有多篇文献,以不同序号表示,并给出该序列中
哪一部分与文献有关。
FEATURES
FEATURES (特性表):具有特定的格式,用来详细描述序
GenBank库包含所有已知的核酸序列和蛋白质序列, 以及与 它们相关的文献著作和生物学注释。
NCBI可提供广泛的数据查询、序列相似性搜索以及其它分
析服务。
数据库
• •
序列文件:注释内容——文章 索引文件:检索目录——文摘
GenBank数据库结构
完整的 GenBank数据库包括序列文件,索引文件以及其它
有关文件。
索引文件是根据数据库中作者、参考文献等建立的,用于
数据库查询。
GenPept是由GenBank中的核酸序列翻译而得到的蛋白质
序列数据库
数据格式为FastA。
GenBank数据库结构
GenBank中最常用的是序列文件。 序列文件的基本单位:是序列条目,包括核苷酸碱基排列 顺序和注释两部分。
生物信息学数据库
核酸序列数据库 蛋白质序列数据库 蛋白质结构数据库 基因组数据库
生物信息学数据库的分类
生物信息学数据库
欧洲分子生物学实验室的EMBL http://www.embl-heidelberg.de 美国生物技术信息中心的GenBank /Genbank/ 日本国立遗传研究所的DDBJ http://www.ddbj.nig.ac.jp/searches-e.html 核酸序列数据库
LOCUS
LOCUS (代码):是该序列条目的标记,或者说标识符,
• • • •
蕴涵这个序列的功能:如HUMCYCLOX表示人的环氧化酶。 序列长度 类型 种属来源
• 录入日期等
说明字段是有关这一序列的简单描述
ACCESSION
ACCESSION (编号):具有唯一性和永久性,在文献中引 用这个序列时,应该以此编号为准。
字段分若干次子字段,以次关键字或特性表说明符开始。 每个序列条目以双斜杠“//”作结束标记
GenBank数据库结构
序列条目的格式非常重要,关键字从第一列开始,次关键
字从第三列开始,特性表说明符从第五列开始。
每个字段可占一行,也可以占若干行。
若一行中写不下时,继续行以空格开始
GenBank数据库
GenBank数据库结构
作用:了解序列数据库的格式,有助于更好地提高数ห้องสมุดไป่ตู้库
检索的效率和准确性。
DDBJ数据库的内容和格式与GenBank相同,此处不作详细
介绍。
分别介绍EMBL和GenBank的数据库结构
GenBank数据库数据注释
(/genbank/ )
物种:GenBank 库里的数据按来源于大约100,000个物种, 其中56%是人类的基因组序列(所有序列中的34%是人类的 EST序列)
记录:每条GenBank数据记录包含对序列的简要描述,它 的科学命名,物种分类名称,参考文献,序列特征表,及 序列本身
GenBank数据库
序列特征表:包含对序列生物学特征注释如:编码区、转
