高通量测序:第二代测序技术详细介绍
二三代测序技术的介绍和比较

二三代测序技术的介绍和比较二代测序技术(也称为高通量测序技术)和三代测序技术是目前最常用的两种DNA测序技术。
下面将对这两种技术进行详细介绍和比较。
1.二代测序技术:二代测序技术的代表性平台包括Illumina HiSeq、Ion Torrent PGM 等。
其工作原理是将DNA样本切割为较短的片段,并通过PCR扩增产生大量的拷贝。
然后,这些片段被连接在测序芯片上,每个片段都被反复地鸟嘌呤(A)、胸腺嘧啶(T)、胞嘧啶(C)、鸟嘧啶(G)四种碱基中的一种互补的碱基识读,并记录下与之相对应的碱基序列。
这些碱基序列最后被计算机软件组装为完整的DNA序列,进而获取样本的遗传信息。
优点:(1)高通量:可以同时测序数百万个DNA片段,获得庞大数量的数据。
(2)成本低廉:通过并行测序的方式,可以大大减少测序成本。
(3)高精度:二代测序技术的错误率较低,可以达到0.1%以下。
(4)测序速度快:每天可获得几百GB的数据。
缺点:(1)仅适用于短序列:由于二代测序技术的局限性,只能测序相对较短的DNA片段,对于长序列的测序存在困难。
(2)高度依赖参考序列:在组装过程中,需要有可靠的参考序列作为基础,否则可能出现组装错误。
(3)无法解析复杂的基因组结构:由于只能产生相对较短的序列片段,二代测序技术无法很好地解析复杂的基因结构,例如重复序列。
2.三代测序技术:三代测序技术的代表性平台包括PacBio SMRT、Oxford Nanopore等。
三代测序技术的特点是可以直接测量DNA单分子的临床序列。
该技术中的样本DNA被引入到小孔中,随后测序设备会通过测量DNA分子在小孔中的电信号变化来捕捉和记录碱基序列。
这种技术可以完整地获取较长的DNA片段,从而提供了更全面和准确的基因组信息。
优点:(1)长读长:能够测序较长的DNA片段,可以获得更全面和准确的基因组信息。
(2)无需参考序列:三代测序技术不需要依赖已知的参考序列,可以直接解析未知基因组。
第二代测序技术介绍

第二代测序技术介绍第二代测序技术,也被称为高通量测序技术,是指在测序过程中同时进行多个DNA分子的测序,从而大大提高了测序的速度和效率。
相对于第一代测序技术,第二代测序技术具有更高的通量、更低的成本和更快的速度,在基因组学、生物信息学、医学和生物学等领域有着广泛的应用。
Illumina(Solexa)测序是目前应用最广泛的第二代测序技术。
它基于细胞自组装技术,通过将DNA片段固定在玻璃基质上,并利用化学物质来控制DNA的扩增和添加荧光标记的核苷酸,实现对DNA片段的扩增和测序。
Illumina测序技术具有高通量、高准确性和低成本的特点,适用于基因组、转录组和表观组测序。
Ion Torrent测序是一种基于半导体技术的第二代测序技术。
它利用DNA聚合酶酶活性引发的质子释放来检测DNA的序列,并通过电信号的变化来记录测序结果。
相较于其他技术,Ion Torrent测序具有简单、快速和低成本的优点,适用于小型测序项目和临床应用。
454测序是第二代测序技术中的一种经典方法。
它基于乳酸菌酶(Luciferase)酶活性,将测序反应中的核苷酸加入到DNA链的末端,在光信号的测量下实现测序。
由于454测序采用的是无法扩增的方法,因此其通量较低,但在研究复杂序列、病毒学和微生物学等领域仍有一定的应用。
与第一代测序技术相比,第二代测序技术具有几个重要的优点。
首先,第二代测序技术可以同时测序多个DNA分子,大大提高了测序的通量和效率。
其次,第二代测序技术的成本更低,可以用于大规模的测序项目。
第三,第二代测序技术的速度更快,可以在较短的时间内完成测序。
最后,第二代测序技术对样本的要求更低,可以从少量样本中获取足够的DNA序列信息。
总之,第二代测序技术的出现和发展为生物信息学和基因组学领域的研究提供了巨大的机会和挑战。
通过不断的技术创新和优化,第二代测序技术将进一步推动基因组学和生物学等领域的发展,为人类健康和疾病研究提供更多的解决方案。
二代测序技术简介

二代测序技术简介一、什么是二代测序技术?二代测序技术,也被称为高通量测序技术,是一种快速、高效的DNA 或RNA序列测定方法。
相比传统的Sanger测序技术,二代测序技术具有较高的测序效率和容量,能够同时测序数百万到数十亿个碱基对,大大提高了测序的速度和数据产量。
常用的二代测序技术包括Illumina 测序技术、Ion Torrent PGM 测序技术等。
二、Illumina二代测序技术的原理与过程1. 原理Illumina二代测序技术基于桥式扩增和碱基扩增的原理。
DNA样本经过打断、连接和PCR扩增等处理后,将单链DNA固定于特定表面上,并在每个DNA分子之间形成成千上万个桥式扩增复合物。
在模板DNA的存在下,通过逐个反复封闭、复制和荧光标记的方式,进行碱基的逐渐扩增,并利用荧光信号记录测序结果。
2. 过程(1)样本制备:包括DNA或RNA的提取、打断、连接和PCR扩增等步骤,以获得特定长度的DNA片段。
(2)文库构建:将DNA片段连接到Illumina测序芯片上的适配器上,并进行PCR扩增,形成DNA桥式扩增复合物。
(3)测序芯片加载:将DNA桥式扩增复合物置于测序芯片上,使得每个DNA分子都与芯片上的特定区域相结合。
(4)桥式扩增:通过逐个反复封闭、复制和荧光标记的方式进行碱基的逐步扩增,形成簇团。
(5)图像获取:利用高分辨率成像系统拍摄簇团的荧光信号。
(6)数据分析:将图像数据转化为碱基序列,通过比对和组装等算法,得到原始测序数据。
三、Illumina二代测序技术的优势和应用领域1. 优势(1)高通量:能够在较短时间内产生大规模的测序数据。
(2)高准确性:其错误率低于其他二代测序技术,能够提供高质量的测序结果。
(3)可扩展性:适用于不同规模的测序项目,从几个目标区域到整个基因组的测序,具有较高的灵活性。
(4)低成本:相对于传统的Sanger测序技术,具有更低的测序成本。
2. 应用领域(1)基因组学研究:能够对物种的基因组进行全面测序和变异分析,有助于揭示基因组结构和功能。
高通量测序技术及其在基因研究中的应用

高通量测序技术及其在基因研究中的应用随着科技的不断发展,生命科学领域也在不断涌现出新的技术和方法。
其中,高通量测序技术是最重要的一种技术之一。
通过高通量测序技术,不仅可以快速准确地测定DNA序列,还可以对基因表达、DNA甲基化、蛋白质互作等多个方面进行深入研究,为生物学领域的研究提供了有力的工具。
下面将对高通量测序技术及其应用进行详细介绍。
一、什么是高通量测序技术高通量测序技术又称为第二代测序技术,它是指一种通过并行测序的方式,对样本中的DNA进行高速测量并获取其序列信息的技术。
高通量测序技术的原理非常简单,它将DNA样本进行随机的分离、扩增、分离、读取等多个步骤,最终生成数百万条DNA片段的测序产物。
这些产物可以通过计算机软件进行处理和分析,获得整个DNA序列的信息。
二、高通量测序技术的类型高通量测序技术的发展已经经历了多个阶段。
目前,市面上已经存在多个高通量测序技术平台。
其中最常用的是Illumina公司和Ion Torrent公司的高通量测序技术。
Illumina公司的高通量测序技术基于测序-合成(sequencing-by-synthesis,SBS)原理,并采用双端30bp或100bp定向测序或PE150bp或PE250bp的测序方式,单个测序通量可达到数百Gb-数Tb。
而Ion Torrent公司的高通量测序技术则采用了基于半导体学的测序原理,并采用了无筛分子筛分子筛分子筛分子筛分子筛分子筛分子筛分子筛分子筛分子筛分子筛分子筛分子筛分子筛分子筛分子筛分子筛分子简单的操作流程,可以对小型基因组进行有效的测序。
三、高通量测序技术在基因研究中的应用高通量测序技术在基因研究中应用广泛,其中最常用的是全基因组测序、RNA测序、甲基化测序等。
1、全基因组测序全基因组测序是指通过高通量测序技术,对生物的整个基因组进行测序。
通过全基因组测序,可以获取整个基因组的序列信息,并对基因组结构、基因型等方面进行研究。
二代测序技术原理

二代测序技术原理二代测序技术,又称高通量测序技术,是指在同一时间内对多个DNA片段进行测序的技术。
它是第二代测序技术的代表,相比于传统的Sanger测序技术,具有高通量、高速度和低成本的特点。
本文将对二代测序技术的原理进行详细介绍。
首先,二代测序技术的原理基于DNA合成和荧光标记。
在测序过程中,DNA样品会被切割成小片段,然后这些小片段会被连接到载体上,形成文库。
接下来,文库中的DNA片段会被放大成簇,然后通过化学方法进行测序。
在测序过程中,每个碱基会被荧光标记,当碱基被读取时,荧光信号会被记录下来,从而确定DNA序列。
其次,二代测序技术的原理还包括高通量测序仪器和生物信息学分析。
高通量测序仪器能够同时对数百万个DNA片段进行测序,大大提高了测序的速度和效率。
而生物信息学分析则是对测序数据进行处理和解读,包括序列拼接、基因组比对和变异分析等步骤,从而得到最终的测序结果。
此外,二代测序技术的原理还涉及到测序质量和数据处理。
测序质量是指测序结果的准确性和可靠性,而数据处理则是对测序数据进行清洗和过滤,去除噪音和错误,保证数据的准确性和可信度。
总的来说,二代测序技术的原理是基于高通量测序仪器和生物信息学分析,通过DNA合成和荧光标记的方法对DNA进行测序,最终得到DNA序列。
这项技术的出现,彻底改变了传统测序技术的局限性,大大提高了测序的速度和效率,为基因组学研究和临床诊断提供了强大的工具。
综上所述,二代测序技术的原理是一项复杂而精密的技术,它的出现极大地推动了基因组学和生物医学领域的发展,为人类健康和疾病治疗提供了重要的支持和保障。
随着技术的不断进步和完善,相信二代测序技术将会在未来发挥更加重要的作用。
第二代测序数据分析原理

第二代测序数据分析原理第二代测序技术是近年来迅速发展起来的高通量测序技术,能够产生大量的DNA序列数据。
与第一代测序技术相比,第二代测序技术具有更高的产量、更快的速度和更低的成本,成为当前基因组学研究和医学诊断的重要工具之一第二代测序数据分析原理是指对产生的高通量测序数据进行处理和解读的过程。
该过程涉及到数据的质控、序列比对、变异检测和功能注释等多个步骤,以获取对生物学问题回答所需的信息。
下面将详细介绍第二代测序数据分析的原理。
1.数据质控数据质控是第二代测序数据分析的第一步,其目的是剔除低质量的序列,保证后续分析得到的结果的准确性。
主要的质控步骤包括去除低质量碱基、去除接头序列和过滤冗余数据。
这些步骤可以通过使用不同的软件工具来实现,如Trimmomatic、FastQC等。
2.序列比对序列比对是将测序数据与参考基因组进行比对的过程。
参考基因组可以是已知的基因组序列,也可以是人工合成的探针序列。
序列比对主要采用两种方法:短序列比对和长序列比对。
短序列比对常用的算法有Bowtie、BWA等,长序列比对常用的算法有BLAST、GSNAP等。
3.变异检测变异检测是根据测序数据中的变异信息来鉴定样本中存在的单核苷酸多态性(SNP)、插入缺失(indel)等变异类型。
变异检测的过程主要包括变异鉴定、变异筛选和变异注释。
变异鉴定的方法包括泛素缺失、泛素纯化和下一代序列法。
变异筛选使用一系列的过滤条件来减少假阳性的产生,如频率过滤、质量过滤和功能过滤等。
变异注释是将检测到的变异与已有的数据库进行比对,以获取变异的生物学功能信息,如GEMINI、ANNOVAR等。
4.功能注释功能注释是将检测到的变异与基因、通路等功能元件进行关联,从而了解变异对生物学功能的影响。
功能注释的方法包括基因本体论(GO)、通路分析、蛋白质相互作用网络分析等。
这些方法可以帮助研究者理解变异的生物学意义以及变异在特定疾病中的作用机制。
综上所述,第二代测序数据分析原理包括数据质控、序列比对、变异检测和功能注释等多个步骤。
第二代测序原理

第二代测序原理第二代测序技术是一种高通量测序技术,它的原理是基于DNA合成和光学信号检测。
在第二代测序技术中,DNA样本首先被打断成较小的片段,然后这些片段被连接到载体上,形成一个DNA文库。
接下来,文库中的DNA片段会通过PCR扩增,产生大量的同一片段序列。
然后,这些DNA片段会被固定在固相载体表面,并进行测序反应。
在测序反应中,DNA片段会被逐一合成,每次合成一个碱基。
在每次合成过程中,会释放出荧光信号,这个信号会被检测器捕获并记录下来。
通过记录下的荧光信号,就可以确定DNA片段的序列。
这种高通量的测序技术可以同时测序成千上万个DNA片段,大大提高了测序效率。
除了高通量之外,第二代测序技术还具有快速、低成本、高灵敏度等优点。
由于其快速高效的特点,第二代测序技术被广泛应用于基因组学、转录组学、表观基因组学等领域。
它为科学家们提供了一个强大的工具,帮助他们更好地理解基因组的结构和功能。
然而,第二代测序技术也存在一些局限性。
例如,由于测序反应中使用的荧光标记物会随着时间的推移而褪色,导致测序结果的准确性下降。
此外,第二代测序技术在测序过程中会产生大量的数据,需要强大的计算和存储设备来处理和存储这些数据。
为了克服这些局限性,科学家们不断改进第二代测序技术,提高其测序准确性和效率。
例如,引入了新的荧光标记物,提高了测序反应的稳定性;开发了新的数据分析算法,加快了数据处理的速度。
这些改进不断推动着第二代测序技术的发展,使其在基因组学研究中发挥着越来越重要的作用。
综上所述,第二代测序技术是一种高通量、快速、低成本的测序技术,具有广泛的应用前景。
随着技术的不断改进和完善,相信第二代测序技术将在基因组学研究中发挥越来越重要的作用,为人类健康和生命科学的发展做出更大的贡献。
高通量测序的原理及应用

高通量测序的原理及应用1. 概述高通量测序(High-throughput sequencing),也被称为第二代测序技术,是一种用于快速、准确且具有高通量的DNA测序方法。
相比于传统的测序方法,高通量测序技术在测序速度、准确度和成本上有明显的优势。
本文将介绍高通量测序的原理及其在生物医学、生态学和农业等领域的应用。
2. 原理高通量测序的原理基于DNA的复制和测序。
下面列举高通量测序的几种常见方法:•Sanger测序法–Sanger测序法是最早被广泛应用的测序方法之一。
它基于DNA合成中的酶法延伸原理进行测序。
通过控制核苷酸的浓度,可以在DNA合成中引入荧光标记。
随着合成的扩增,核苷酸会停留在特定位置,之后通过电泳分析荧光标记的顺序来测定目标DNA序列。
•454测序法–454测序法是一种基于密集插入测序技术的高通量测序方法。
通过将待测DNA样本切割成较小的片段,并与特定合子序列连接,形成序列文库。
之后,这些片段将在流动细胞中进行多轮酶法扩增,并通过荧光探针进行检测,从而实现对目标DNA序列的测定。
•Illumina测序法–Illumina测序法是目前最广泛应用的高通量测序技术之一。
该方法通过将DNA样本分离成独立的DNA片段,并连接到流动细胞矩阵中。
接下来,在不同的扩增循环中,特定的核苷酸会被逐步加入,并通过荧光探针的检测来确定DNA的序列。
最终,可以通过计算机软件将这些测定的片段合并成完整的目标DNA序列。
3. 应用高通量测序技术在各个领域有广泛的应用,包括:•生物医学研究–在生物医学领域,高通量测序技术可以帮助研究人员对人类遗传病的发生机制进行深入研究。
通过对大规模的基因组数据进行测序和分析,可以寻找与特定遗传病相关的基因变异并探索潜在的治疗方法。
此外,高通量测序还可以用于肿瘤学研究,帮助研究人员了解肿瘤发展、进展和治疗的分子机制。
•生态学研究–高通量测序技术可以应用于生态学研究中,帮助研究人员分析和识别不同环境下的微生物群落组成。
第二代测序技术

SO、油包水PCR
SOLiD流程
3、含DNA模板P1磁珠的固定
SOLiD流程
4、SOLiD双碱基编码原理及测序流程
SOLiD流程
4、SOLiD双碱基编码原理及测序流程
SOLiD流程
5. 数据分析原理
Polonator
第二代高通量测序技术简介
四大高通量测序平台
Solexa,454 (GS-FLX),
SOLiD和Polonator
测序原理
合成法测序(Sequencing by Synthesis) 连接法测序(
Roche:(2005,2007,2008) 原理:在DNA聚合酶、ATP硫酸化酶、荧光 素酶和双磷酸酶的作用下,将每一个dNTP的 聚合与一次化学发光信号的释放偶联起来, 通过检测化学发光信号的有无和强度,达到 实时检测DNA序列的目的。
至300-800bp间,经末端修复与特异性接头 连接等修饰后变性处理回收单链的DNA 。
454 (GS-FLX)流程
包水的混合 物,每个独特的片断在自己的微反应器里进 行独立的扩增,回收纯化;
454 (GS-FLX)流程
3、测序反应:携带DNA片段的磁珠被放入 PTP板中供测序反应使用。
454 (GS-FLX)流程
4、数据分析:GS FLX系统在10小时的运行 当中可获得100余万个读长,读取超过4-6亿 个碱基信息
Solexa-Illumina Genome Analyzer
测序原理: Polonator系统高质量测序是一种以连接 反应进行DNA序列分析的技术,该系统采用 结合在磁珠上单分子DNA片段簇为测序模板, 以CY5、Texas Red、CY3、6-FAM四色荧 光标记的9碱基单链荧光探针混合物进行连续 的连接反应为基础,对扩增的DNA片段进行 大规模高通量测序。
二代测序技术-illumina测序原理

二代测序技术-illumina测序原理
Illumina测序技术是一种常用的二代测序技术,也被称为高通量测序技术。
其原理主要包括以下几个步骤:
1. DNA片段制备:首先,将待测的DNA样本进行特定处理,如剪切、连接接头等,生成适合测序的DNA片段。
2.聚合酶链反应(PCR)扩增:将DNA片段进行PCR扩增,以产生大量的DNA模板,供后续测序反应使用。
3.测序芯片制备:将PCR扩增得到的DNA模板固定在测序芯片的表面上,使得每个DNA模板都与芯片上的一个特定位置对应。
4.引物结合与扩增:在测序芯片上,加入带有特定序列的引物,并进行碱基扩增反应。
这种扩增反应是逐个碱基进行的,每次只加入一种碱基。
5.碱基荧光标记:每种碱基都与特定的荧光染料结合,不同的碱基配对会产生不同的荧光信号。
6.成像和信号检测:使用激光或其他光源对测序芯片上的DNA模板进行扫描,并检测每个位置的荧光信号。
7.数据处理和碱基识别:通过分析得到的荧光信号,识别每个位置的碱基。
8.重复扩增和成像:重复以上步骤,直到获得足够的测序数据。
通过以上步骤,Illumina测序技术可以高效地获得大量的测序数据,具有高通量、高准确性和较低的成本等优点,被广泛应用于基因组学、转录组学和表观遗传学等领域的研究。
第二代测序技术的发展及应用

第二代测序技术的发展及应用第二代测序技术的发展及应用随着科学技术的迅猛发展,基因测序技术也得到了极大的改进与突破。
第二代测序技术的出现,不仅在基因组学、生物学和医学领域取得了巨大的突破,也给人类社会带来了深远的影响。
本文将详细介绍第二代测序技术的发展历程以及其在各个领域的应用。
第二代测序技术的发展历程第二代测序技术,也称为高通量测序技术,是指相对于第一代测序技术(即Sanger测序技术)而言的新一代测序方法。
第一代测序技术虽然准确可靠,但是过程复杂,耗时长,测序成本高昂,限制了测序的应用范围。
因此,人们急需开发一种更高效、更经济、更快速的测序技术。
第二代测序技术的发展可以追溯到2005年,当时Illumina公司(前身为Solexa公司)首次提出了一种基于“桥式扩增”(bridge amplification)的高通量测序方法。
该方法利用DNA模板的扩增以及荧光标记的核苷酸,通过多次循环的扩增过程和荧光信号的检测,实现了高效、高通量的DNA测序。
此后,Illumina公司推出了一系列基于该原理的测序平台,如MiSeq、HiSeq和NovaSeq等,成为了第二代测序技术的代表。
在与Illumina公司几乎同时,Roche公司也推出了一种全新的测序方法,称为454测序技术。
该技术基于聚合酶链反应(PCR)和荧光探测,通过在四个玻片上同时进行测序反应,实现了高通量的DNA测序。
尽管Roche公司在此之后退出了测序市场,但他们的贡献促进了第二代测序技术的发展。
此外,Ion Torrent公司还开发了一种基于离子探测的第二代测序技术。
该技术消除了传统测序方法中的荧光检测步骤,直接通过离子检测测量DNA链的合成过程。
因为离子检测的原理简单,该技术成本低廉,操作简单,具有非常广阔的应用前景。
第二代测序技术在各个领域的应用1. 基因组学研究:第二代测序技术使得人类可以更加深入地研究基因组的组成和功能。
通过对大规模DNA样本的测序,可以获得各种生物的完整基因组序列,并深入研究基因组的组织结构、重复序列和非编码RNA等。
简述第一二三代测序技术原理

简述第一二三代测序技术原理
第一代测序技术原理:
第一代测序技术又称为Sanger测序技术,是由Frederick Sanger在1977年首次提出并开发的。
这种方法依靠DNA链
延伸的DNA聚合酶做模板并进行荧光标记,使用一种称为链终止的化学方法,会使DNA链延伸终止在特定核苷酸,生成所有长度的DNA片段,然后使用聚丙烯酰胺凝胶电泳分离各个片段。
随后,通过电泳图谱能够分辨出不同长度的DNA片段,从而得到DNA序列。
第二代测序技术原理:
第二代测序技术是基于测序-by-synthesis原理,是通过将DNA 组装到表面上,并添加能够照亮每个核苷酸的化学试剂进行测序。
这些试剂可以逐个核苷酸累加,并用相应的光信号发送给计算机进行分析。
第二代测序技术包括Illumina, 454, Ion Torrent,和SOLiD。
Illumina使用激光照亮DNA序列中的核苷酸,并记录生成的荧光信号。
此技术具有高通量、低成本和快速的优点。
第三代测序技术原理:
第三代测序技术是一种实时单分子测序技术,采用单个自然DNA分子,并通过流速调节使DNA通过膜孔,然后测定膜孔中的电学性质来识别核苷酸(如Ion Torrent,Oxford Nanopore)。
这些技术还包括基于纳米技术和单分子DNA氧
化的PacBio技术。
这些技术具有不同的优点,包括高精确度、高通量和更真实的序列。
高通量测序:第二代测序技术详细介绍
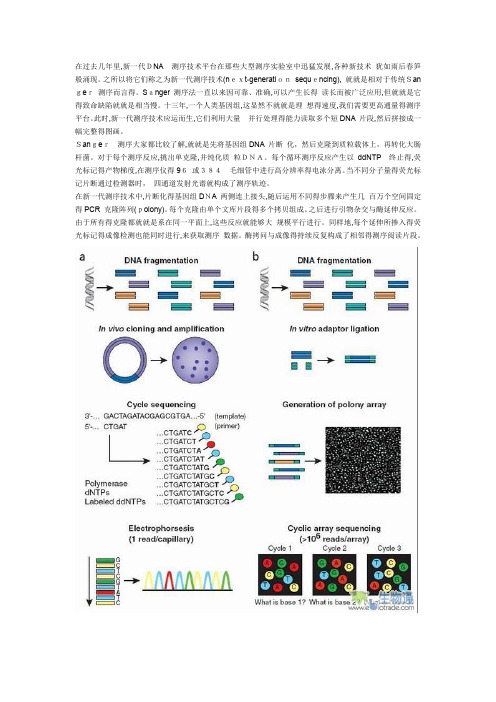
在过去几年里,新一代DNA测序技术平台在那些大型测序实验室中迅猛发展,各种新技术犹如雨后春笋般涌现。
之所以将它们称之为新一代测序技术(next-generationsequencing), 就就是相对于传统San ger测序而言得。
Sanger 测序法一直以来因可靠、准确,可以产生长得读长而被广泛应用,但就就是它得致命缺陷就就是相当慢。
十三年,一个人类基因组,这显然不就就是理想得速度,我们需要更高通量得测序平台。
此时,新一代测序技术应运而生,它们利用大量并行处理得能力读取多个短DNA 片段,然后拼接成一幅完整得图画。
Sanger测序大家都比较了解,就就是先将基因组DNA 片断化,然后克隆到质粒载体上,再转化大肠杆菌。
对于每个测序反应,挑出单克隆,并纯化质粒DNA。
每个循环测序反应产生以ddNTP终止得,荧光标记得产物梯度,在测序仪得96或384毛细管中进行高分辨率得电泳分离。
当不同分子量得荧光标记片断通过检测器时,四通道发射光谱就构成了测序轨迹。
在新一代测序技术中,片断化得基因组DNA 两侧连上接头,随后运用不同得步骤来产生几百万个空间固定得PCR 克隆阵列(polony)。
每个克隆由单个文库片段得多个拷贝组成。
之后进行引物杂交与酶延伸反应。
由于所有得克隆都就就是系在同一平面上,这些反应就能够大规模平行进行。
同样地,每个延伸所掺入得荧光标记得成像检测也能同时进行,来获取测序数据。
酶拷问与成像得持续反复构成了相邻得测序阅读片段。
Solexa高通量测序原理--采用大规模并行合成测序法(SBS,Sequencing-By-Synthesis)与可逆性末端终结技术(Reversible TerminatorChemistry)--可减少因二级结构造成得一段区域得缺失。
--具有高精确度、高通量、高灵敏度与低成本等突出优势--可以同时完成传统基因组学研究(测序与注释)以及功能基因组学(基因表达及调控,基因功能,蛋白/核酸相互作用)研究----将接头连接到片段上,经PCR扩增后制成Library。
第三代测序技术(单分子实时DNA测序)与第二代测序技术(高通量测序技术)简介

第三代测序技术(单分子实时DNA测序)与第二代测序技术(高通量测序技术)简介第三代测序技术(单分子实时DNA测序)与第二代测序技术(高通量测序技术)简介第三代测序技术简介如果有人告诉你用显微镜实时观测单分子DNA聚合酶复制DNA,并用它来测序,你一定会认为他异想天开,没有一点生物的sense。
我最初就是这样认为的,然而它不仅可以实现,而且已经实现了~这个就是被称为第三代的测序技术,Pacific Biosciences公司推出的“Single Molecule Real Time(SMRT) DNA Sequencing”(单分子实时DNA测序)。
我有幸在NIH听到了这个技术发明人Stephen Turner博士的讲座,根据自己粗浅的理解记录整理一下。
要实现单分子实时测序,有三个关键的技术。
第一个是荧光标记的脱氧核苷酸。
显微镜现在再厉害,也不可能真的实时看到“单分子”。
但是它可以实时记录荧光的强度变化。
当荧光标记的脱氧核苷酸被掺入DNA链的时候,它的荧光就同时能在DNA链上探测到。
当它与DNA链形成化学键的时候,它的荧光基团就被DNA聚合酶切除,荧光消失。
这种荧光标记的脱氧核苷酸不会影响DNA聚合酶的活性,并且在荧光被切除之后,合成的DNA链和天然的DNA链完全一样。
第二个是纳米微孔。
因为在显微镜实时记录DNA链上的荧光的时候,DNA链周围的众多的荧光标记的脱氧核苷酸形成了非常强大的荧光背景。
这种强大的荧光背景使单分子的荧光探测成为不可能。
Pacific Biosciences公司发明了一种直径只有几十纳米的纳米孔[zero-mode waveguides (ZMWs)],单分子的DNA聚合酶被固定在这个孔内。
在这么小的孔内,DNA链周围的荧光标记的脱氧核苷酸有限,而且由于A,T,C,G这四种荧光标记的脱氧核苷酸非常快速地从外面进入到孔内又出去,它们形成了非常稳定的背景荧光信号。
而当某一种荧光标记的脱氧核苷酸被掺入到DNA链时,这种特定颜色的荧光会持续一小段时间,直到新的化学键形成,荧光基团被DNA聚合酶切除为止(见图)。
高通量测序技术--第二代测序技术

高通量测序技术--第二代测序技术高通测序技术—量第二—测序代术技高量通序技测术是对统传测一序次命性的革变改一,次几对万十到百万几DN条分A子行进列序定,测此因在些有文献中称其下为代一测技术(nex序 gteenrtiano sequnecing足)见其时代的改变,同划时高通测量使得序一对个种物转的组和基录因进组行细致全貌的析分成为能,可以所又称为被度测深(d序eepseq uneincg)。
从2019年自44 5Lif ecSenies公司c2(07年0该司被公Rcoe正式收购h推)了454出 FXL磷酸焦序测台平4(4 F5L Xyrpsoeuqencng piatlorfm)以来曾,推过3出70xl D3AN测仪序(330xl D7N AAalynze)的Appliedr BoiySste(AmIB这家一直)占着测序据场最市份大额公司的的先地位领就始动摇开,因为了们他的头产拳毛品细管列电泳测阵序系列(series ca仪ilplayra rayr elcetrohopesirsse uencing qmahcnei)s遇了两到强有力的竞个对手,争个一就是氏公罗司(oRhce的4)4 5测仪(序ocRh GSF X Lequesncre,)另一,就是2个00年美国6lluIinma司公出推的olSxe基a因分组析平台G(neoem nalyzeArp lafotmr,)为,此020年7BA公司推I了自主出发研S的LODi 测仪序A(I SBOLD siqueencre。
)这三个测平台序即目为前通量高测平台的代序。
表(见一表)公司名称技术原技理开术发商者业式模Aplyp BosistemysABI()基于珠磁大的模规并克行连隆DNA接序法测美国 gencAourt私基因组人学公司(AP)上G市司公:售设备和试销获取利剂Il润umilan合成测法序英国 Sloxae司公席科首家D学avdiB entlye上市司:公销售设和备试获取利润剂Rche 大规o模并行焦磷合成测序法酸国美44 5iLf Sceiecnse公司的创始人onJtahn Raohbtreg上市公司:销售设和试剂获取利润备Heilcos大模规行并单子合成分测序法美斯坦福国学大生物程学家工Stehpen Quake上公市司2:070年月首5公开次股(募PO) IoCmplee tGneomicsDNA纳阵米列组合与探锚针连接测序定法国Comp美lteeGe nomci公司首席科s家ra学oje drmdnaa 私人公c司投:资额4650万美为元表一:流主测序平一览台这些平台共的同点特是极高的序测通量相对于,统传测序的96毛道管测序,高通细测序一量次实可以验取4读0到万40万条0列。
高通量测序:第二代测序技术详细介绍

在过去几年里,新一代DNA 测序技术平台在那些大型测序实验室中迅猛发展,各种新技术犹如雨后春笋般涌现。
之所以将它们称之为新一代测序技术(next-generation sequencing),是相对于传统Sanger 测序而言的。
Sanger 测序法一直以来因可靠、准确,可以产生长的读长而被广泛应用,但是它的致命缺陷是相当慢。
十三年,一个人类基因组,这显然不是理想的速度,我们需要更高通量的测序平台。
此时,新一代测序技术应运而生,它们利用大量并行处理的能力读取多个短DNA 片段,然后拼接成一幅完整的图画。
Sanger 测序大家都比较了解,是先将基因组DNA 片断化,然后克隆到质粒载体上,再转化大肠杆菌。
对于每个测序反应,挑出单克隆,并纯化质粒DNA。
每个循环测序反应产生以ddNTP 终止的,荧光标记的产物梯度,在测序仪的96 或384 毛细管中进行高分辨率的电泳分离。
当不同分子量的荧光标记片断通过检测器时,四通道发射光谱就构成了测序轨迹。
在新一代测序技术中,片断化的基因组DNA 两侧连上接头,随后运用不同的步骤来产生几百万个空间固定的PCR 克隆阵列(polony)。
每个克隆由单个文库片段的多个拷贝组成。
之后进行引物杂交和酶延伸反应。
由于所有的克隆都是系在同一平面上,这些反应就能够大规模平行进行。
同样地,每个延伸所掺入的荧光标记的成像检测也能同时进行,来获取测序数据。
酶拷问和成像的持续反复构成了相邻的测序阅读片段。
Solexa 高通量测序原理--采用大规模并行合成测序法(SBS, Sequencing-By-Synthesis)和可逆性末端终结技术(Reversible Terminator Chemistry)--可减少因二级结构造成的一段区域的缺失。
--具有高精确度、高通量、高灵敏度和低成本等突出优势--可以同时完成传统基因组学研究(测序和注释)以及功能基因组学(基因表达及调控,基因功能,蛋白/核酸相互作用)研究----将接头连接到片段上,经PCR 扩增后制成Library 。
二代测序法

二代测序法介绍二代测序法(second generation sequencing),也称为高通量测序,是一种用于测定DNA或RNA序列的方法。
相比于传统的Sanger测序方法,二代测序法具有更高的通量和更快的测序速度,因此被广泛应用于基因组学研究、生物医学研究和临床应用等领域。
二代测序技术原理二代测序技术通过将DNA片段进行大规模并行测序,来实现高通量测序。
整个测序过程可以分为DNA片段制备、文库构建、芯片上测序、图像分析和数据处理等步骤。
DNA片段制备首先,从待测样品的DNA中提取所需片段。
常用的DNA片段制备方法有PCR扩增、酶切和构建文库等。
文库构建将DNA片段连接到适当的文库载体上。
文库是DNA片段的集合,用于在后续步骤中进行测序。
构建文库的方法包括PCR扩增文库、切割文库和合成文库等。
芯片上测序将文库中的DNA样品倒置到芯片上,每个DNA片段会与芯片上的固定DNA序列匹配。
然后,使用荧光染料或其他方法来标记每个DNA片段的序列。
通过读取芯片上的荧光信号,可以获得DNA片段的序列信息。
图像分析和数据处理将芯片上的图像转换为原始数据,然后对数据进行处理和分析。
这包括配对序列的拼接、错误校正和序列比对等步骤。
最终,可以根据处理后的数据获得DNA片段的准确序列信息。
二代测序技术的优势相比传统的Sanger测序方法,二代测序技术具有以下几个优势:1.高通量:二代测序技术可以并行测序大量的DNA片段,从而大大提高了测序效率。
2.速度快:二代测序技术的测序速度很快,可以在较短的时间内完成大量的测序工作。
3.低成本:由于高通量和快速测序速度,二代测序技术的测序成本相对较低。
4.应用广泛:二代测序技术可以应用于基因组学研究、转录组学研究、表观遗传学研究和临床应用等各个领域。
二代测序技术的应用二代测序技术在科学研究和临床应用中有着广泛的应用。
基因组学研究二代测序技术在基因组学研究中发挥了重要作用。
通过对不同生物体的基因组进行测序,可以揭示其基因组的组成和结构。
二代测序原理范文

二代测序原理范文二代测序是指第二代高通量测序技术,也称为下一代测序技术。
它是相对于第一代测序技术的一种新一代测序技术。
二代测序技术以其高通量、高准确性和低成本的特点,对生命科学领域的研究和应用产生了革命性的影响。
二代测序技术有多种不同的原理,其中最常见的是Illumina公司的技术,也被称为SBS(Sequencing by Synthesis)技术。
下面将以Illumina技术为例,详细介绍二代测序的原理。
Illumina二代测序技术的主要步骤包括文库构建、DNA片段连接、PCR扩增、DNA片段固定到测序芯片上、质控、测序和数据分析等。
具体流程如下:首先,将DNA样本加工成文库。
文库的构建过程包括将DNA剪切成适当大小的片段,然后加上适配体(由引物和适配物组成,用于连接DNA片段和测序引物),并进行片段连接。
然后,将DNA片段连接到测序芯片上。
测序芯片是一块玻片,上面有数百万个96孔小孔,每个小孔下面有数百万个聚合物支持点。
通过较长的捕获序列将DNA片段固定在聚合物支持点上。
接下来是PCR扩增。
扩增的目的是将固定在测序芯片上的DNA片段复制成数百万个相同的片段,以便进行测序。
PCR扩增需要引物和适配体的配对,引物用于定向扩增目标DNA片段。
之后是测序。
测序分为两个步骤:首先是引物结合和延伸,其次是信号检测和图像化。
在引物结合和延伸步骤中,测序芯片被浸入含有测序试剂的流动细胞,并且通过合成DNA链中每个位置的碱基来进行序列测定。
在每个碱基的加入过程中,会释放出荧光信号。
接下来,这些信号被检测到,并转化为电信号,然后被图像化。
最后,进行数据分析。
测序仪将生成原始数据,然后需要进行数据分析以获取最终的测序结果。
数据分析的过程包括原始数据的去噪、序列拼接和质量控制等。
总而言之,二代测序技术通过将DNA样本分解为数百万个小片段,并对它们进行多次重复的扩增和测序,最终得到完整的DNA序列信息。
这一技术的高通量、高准确性和低成本的特点,使其在基因组学、遗传学、生物医学研究和临床诊断中有着广泛的应用。
ngs二代测序方法描述

ngs二代测序方法描述NGS(Next Generation Sequencing)是一种高通量二代测序技术,也被称为第二代测序技术。
它是在传统的Sanger测序技术基础上发展而来的,通过并行测序的方式,大大提高了测序效率和产出。
本文将详细介绍NGS二代测序方法的原理和应用。
一、原理NGS二代测序方法的核心原理是通过将DNA或RNA样本分离成小片段,并在微纳米级平台上进行扩增、定点合成和测序。
具体的步骤如下:1. 文库构建:将DNA或RNA样本进行加工处理,包括断裂、末端修复、连接接头等步骤,使其适用于测序。
2. 扩增:将文库中的DNA或RNA片段扩增,使其在微纳米级平台上充分复制。
3. 定点合成:将扩增的DNA或RNA片段定点固定在微纳米级平台上,并进行模板的制备,以便进行后续的测序步骤。
4. 测序:通过荧光标记的碱基,使用碱基的互补配对原则进行测序。
测序过程中,通过摄像机记录荧光信号,并将其转化为碱基序列。
5. 数据分析:将测序得到的碱基序列进行数据处理和分析,包括序列比对、SNP检测、基因组拼装等步骤。
二、应用NGS二代测序方法在生物学和医学领域有着广泛的应用,包括以下几个方面:1. 基因组学研究:NGS可以对整个基因组进行高通量测序,从而揭示基因组的结构和功能。
通过测序,可以快速、准确地获得大量的基因组数据,并用于研究基因组变异、基因表达调控等方面。
2. 转录组学研究:通过对RNA样本的测序,可以获得转录组的信息,包括基因表达水平、剪接变异等。
NGS可以帮助科研人员更全面地了解基因的表达调控机制,发现新的基因和转录本。
3. 表观遗传学研究:NGS可以用于研究DNA甲基化和组蛋白修饰等表观遗传学调控机制。
通过对DNA或染色质的测序,可以获得高分辨率的表观遗传学数据,揭示表观遗传学对基因表达和细胞功能的影响。
4. 癌症基因组学研究:NGS可以帮助科研人员揭示癌症的发生机制、驱动基因和潜在的治疗靶点。
第二组--第二代测序技术的原理及应用

第二代测序技术的核心思想是边合成边测序( Sequencing by Synthesis),即通过捕捉新合成的末端的标记来确定 DNA的序列,现有的技术平台主要包括Roche(罗氏公司)/454 FLX、 Illuminate公司/Solexa Genome Analyzer和(ABI公司) Applied Biosystems SOLID system。 这三个技术平台各有优点,454 FLX的测序片段比较长,高质量的读长 (read)能达到400bp;Solexa测序性价比最高,不仅机器的售价比其 他两种低,而且运行成本也低,在数据量相同的情况下,成本只有454 测序的1/10;SOLID测序的准确度高,原始碱基数据的准确度大于 99.94%,而在15X覆盖率时的准确度可以达到99.999%,是目前第二代 测序技术中准确度最转录组学的研 究中得到了广泛应用,得到高度好评。但是像其他新生技 术一样,RNA测序技术也面临一些挑战,比如RNA产生的海 量数据的生物信息学处理,获得高质量转录图谱最佳测序 量的确定等。
(3)、smallRNA测序 小RNA测序技术采用胶分离技术,收集样品中18-30nt的RNA 片段,利用高通量测序技术,一次性获得单碱基分辨率的 数百万条小RNA序列信息,依托强大的生物信息分析平台, 鉴定已知小RNA,并预测新的小RNA及其靶标基因。 基于Illumina HiSeqTM 2000高通量测序技术的小RNA数字 化分析,采用边合成边测序的方法,可减少二级结构造成 的区域缺失。该技术可以对高质量序列进行序列长度分布 的统计及样品间公共序列统计,将筛选后的高质量序列分 类注释,从而获得样品中包含的各组分及表达量信息,并 对所有小RNA片段进行注释,对新的miRNA则进行靶基因预 测。
??4单碱基延伸测序singlebaseextensionandsequencing?在测序的flowcell中加入四种荧光标记的dntpdna聚合酶以及接头引物进行扩增在每一个测序簇延伸互补链时每加入一个被荧光标记的dntp就能释放出相对应的荧光测序仪通过捕获荧光信号并通过计算机软件将光荧光测序仪通过捕获荧光信号并通过计算机软件将光信号转化为测序峰从而获得待测片段的序列信息
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
高通量测序:第二代测序技术详细介绍-标准化文件发布号:(9456-EUATWK-MWUB-WUNN-INNUL-DDQTY-KII在过去几年里,新一代DNA 测序技术平台在那些大型测序实验室中迅猛发展,各种新技术犹如雨后春笋般涌现。
之所以将它们称之为新一代测序技术(next-generation sequencing),是相对于传统Sanger 测序而言的。
Sanger 测序法一直以来因可靠、准确,可以产生长的读长而被广泛应用,但是它的致命缺陷是相当慢。
十三年,一个人类基因组,这显然不是理想的速度,我们需要更高通量的测序平台。
此时,新一代测序技术应运而生,它们利用大量并行处理的能力读取多个短DNA 片段,然后拼接成一幅完整的图画。
Sanger 测序大家都比较了解,是先将基因组DNA 片断化,然后克隆到质粒载体上,再转化大肠杆菌。
对于每个测序反应,挑出单克隆,并纯化质粒DNA。
每个循环测序反应产生以ddNTP 终止的,荧光标记的产物梯度,在测序仪的96 或384 毛细管中进行高分辨率的电泳分离。
当不同分子量的荧光标记片断通过检测器时,四通道发射光谱就构成了测序轨迹。
在新一代测序技术中,片断化的基因组DNA 两侧连上接头,随后运用不同的步骤来产生几百万个空间固定的PCR 克隆阵列(polony)。
每个克隆由单个文库片段的多个拷贝组成。
之后进行引物杂交和酶延伸反应。
由于所有的克隆都是系在同一平面上,这些反应就能够大规模平行进行。
同样地,每个延伸所掺入的荧光标记的成像检测也能同时进行,来获取测序数据。
酶拷问和成像的持续反复构成了相邻的测序阅读片段。
Solexa 高通量测序原理--采用大规模并行合成测序法(SBS, Sequencing-By-Synthesis)和可逆性末端终结技术(Reversible Terminator Chemistry)--可减少因二级结构造成的一段区域的缺失。
--具有高精确度、高通量、高灵敏度和低成本等突出优势--可以同时完成传统基因组学研究(测序和注释)以及功能基因组学(基因表达及调控,基因功能,蛋白/核酸相互作用)研究----将接头连接到片段上,经 PCR 扩增后制成 Library 。
----随后在含有接头(单链引物)的芯片( flow cell )上将已加入接头的 DNA 片段变成单链后通过与单链引物互补配对绑定在芯片上,另一端和附近的另外一个引物互补也被固定,形成“桥”----经30伦扩增反应,形成单克隆DNA簇----边合成边测序(Sequencing By Synthesis)的原理,加入改造过的DNA 聚合酶和带有4 种荧光标记的dNTP。
这些dNTP是“可逆终止子”,其3’羟基末端带有可化学切割的基团,使得每个循环只能掺入单个碱基。
此时,用激光扫描反应板表面,读取每条模板序列第一轮反应所聚合上去的核苷酸种类。
之后,将这些基团化学切割,恢复3'端粘性,继续聚合第二个核苷酸。
如此继续下去,直到每条模板序列都完全被聚合为双链。
这样,统计每轮收集到的荧光信号结果,就可以得知每个模板DNA 片段的序列。
目前的配对末端读长可达到2×50 bp,更长的读长也能实现,但错误率会增高。
读长会受到多个引起信号衰减的因素所影响,如荧光标记的不完全切割。
Roche 454 测序技术“一个片段 = 一个磁珠 = 一条读长(One fragment =One bead = One read)”1)样品输入并片段化:GS FLX 系统支持各种不同来源的样品,包括基因组DNA、PCR产物、BAC、cDNA、小分子RNA 等等。
大的样品例如基因组DNA 或者BAC 等被打断成300-800 bp 的片段;对于小分子的非编码RNA 或者PCR 扩增产物,这一步则不需要。
短的PCR 产物则可以直接跳到步骤3)。
2)文库制备:借助一系列标准的分子生物学技术,将A 和B 接头(3’和5’端具有特异性)连接到DNA 片段上。
接头也将用于后续的纯化,扩增和测序步骤。
具有A、B 接头的单链DNA 片段组成了样品文库。
3)一个DNA 片段=一个磁珠:单链DNA 文库被固定在特别设计的DNA 捕获磁珠上。
每一个磁珠携带了一个独特的单链DNA 片段。
磁珠结合的文库被扩增试剂乳化,形成油包水的混合物,这样就形成了只包含一个磁珠和一个独特片段的微反应器。
4)乳液PCR 扩增:每个独特的片段在自己的微反应器里进行独立的扩增,而没有其他的竞争性或者污染性序列的影响。
整个片段文库的扩增平行进行。
对于每一个片段而言,扩增后产生了几百万个相同的拷贝。
随后,乳液混合物被打破,扩增的片段仍然结合在磁珠上。
5)一个磁珠=一条读长:携带DNA 的捕获磁珠随后放入PTP 板中进行后继的测序。
PTP 孔的直径(29um)只能容纳一个磁珠(20um)。
然后将PTP 板放置在GS FLX 中,测序开始。
放置在四个单独的试剂瓶里的四种碱基,依照T、A、C、G 的顺序依次循环进入PTP 板,每次只进入一个碱基。
如果发生碱基配对,就会释放一个焦磷酸。
这个焦磷酸在ATP 硫酸化酶和萤光素酶的作用下,经过一个合成反应和一个化学发光反应,最终将萤光素氧化成氧化萤光素,同时释放出光信号。
此反应释放出的光信号实时被仪器配置的高灵敏度CCD 捕获到。
有一个碱基和测序模板进行配对,就会捕获到一分子的光信号;由此一一对应,就可以准确、快速地确定待测模板的碱基序列。
这也就是大名鼎鼎的焦磷酸测序。
6)数据分析:GS FLX 系统在10 小时的运行当中可获得100 多万个读长,读取超过4-6 亿个碱基信息。
GS FLX 系统提供两种不同的生物信息学工具对测序数据进行分析,适用于不同的应用:达400 MB 的从头拼接和任何大小基因组的重测序。
GS FLX 系统的准确率在99%以上。
其主要限制来自同聚物,也就是相同碱基的连续掺入,如AAA 或GGG。
由于没有终止元件来阻止单个循环的连续掺入,同聚物的长度就需要从信号强度中推断出来。
这个过程就可能产生误差。
因此,454 测序平台的主要错误类型是插入-缺失,而不是替换。
ABI SOLID 测序技术a. 文库制备 SOLiD 系统能支持两种测序模板:片段文库(fragment library)或配对末端文库(mate-paired library)。
使用哪一种文库取决于你的应用及需要的信息。
片段文库就是将基因组DNA 打断,两头加上接头,制成文库。
如果你想要做转录组测序、RNA 定量、miRNA 探索、重测序、 3’, 5’-RACE、甲基化分析、ChIP 测序等,就可以用它。
如果你的应用是全基因组测序、SNP 分析、结构重排/拷贝数,则需要用配对末端文库。
配对末端文库是将基因组DNA 打断后,与中间接头连接,再环化,然后用EcoP15 酶切,使中间接头两端各有27bp 的碱基,再加上两端的接头,形成文库。
b. 乳液PCR/微珠富集在微反应器中加入测序模板、PCR 反应元件、微珠和引物,进行乳液PCR (Emulsion PCR)。
PCR 完成之后,变性模板,富集带有延伸模板的微珠,去除多余的微珠。
微珠上的模板经过3’修饰,可以与玻片共价结合。
看到这里,是不是有一种似曾相识的感觉呢?那就对了,此步骤与454 的GS FLX 基本相同。
不过SOLiD 系统的微珠要小得多,只有1 um。
乳液PCR 最大的特点是可以形成数目庞大的独立反应空间以进行DNA 扩增。
其关键技术是“注水到油”,基本过程是在PCR 反应前,将包含PCR 所有反应成分的水溶液注入到高速旋转的矿物油表面,水溶液瞬间形成无数个被矿物油包裹的小水滴。
这些小水滴就构成了独立的PCR 反应空间。
理想状态下,每个小水滴只含一个DNA 模板和一个P1 磁珠,由于水相中的P2 引物和磁珠表面的P1 引物所介导的PCR 反应,这个DNA 模板的拷贝数量呈指数级增加,PCR 反应结束后,P1 磁珠表面就固定有拷贝数目巨大的同来源DNA 模板扩增产物。
c. 微珠沉积 3’修饰的微珠沉积在一块玻片上。
在微珠上样的过程中,沉积小室将每张玻片分成1 个、4 个或8 个测序区域。
SOLiD 系统最大的优点就是每张玻片能容纳更高密度的微珠,在同一系统中轻松实现更高的通量。
d. 连接测序这一步可就是SOLiD 的独门秘笈了。
它的独特之处在于没有采用惯常的聚合酶,而用了连接酶。
SOLiD 连接反应的底物是8 碱基单链荧光探针混合物。
连接反应中,这些探针按照碱基互补规则与单链DNA 模板链配对。
探针的5’末端分别标记了CY5、Texas Red、CY3、 6-FAM 这4 种颜色的荧光染料。
探针3’端1~5 位为随机碱基,可以是ATCG四种碱基中的任何一种碱基,其中第1、2 位构成的碱基对是表征探针染料类型的编码区,下图的双碱基编码矩阵规定了该编码区16 种碱基对和4 种探针颜色的对应关系,而3~5位的“n”表示随机碱基,6~8位的“z”指的是可以和任何碱基配对的特殊碱基。
单向SOLiD 测序包括五轮测序反应,每轮测序反应含有多次连接反应。
第一轮测序的第一次连接反应由连接引物“n”介导,由于每个磁珠只含有均质单链DNA 模板,所以这次连接反应掺入一种8 碱基荧光探针,SOLiD 测序仪记录下探针第1、2 位编码区颜色信息,随后的化学处理断裂探针3’端第5、6 位碱基间的化学键,并除去6~8 位碱基及5’末端荧光基团,暴露探针第5 位碱基5’磷酸,为下一次连接反应作准备。
因为第一次连接反应使合成链多了5 个碱基,所以第二次连接反应得到模板上第6、7 位碱基序列的颜色信息,而第三次连接反应得到的是第11、12 位碱基序列的颜色信息……几个循环之后,引物重置,开始第二轮的测序。
由于第二轮连接引物n-1 比第一轮错开一位,所以第二轮得到以0,1 位起始的若干碱基对的颜色信息。
五轮测序反应反应后,按照第0、 1 位,第1、2 位... …的顺序把对应于模板序列的颜色信息连起来,就得到由“0,1,2,3…”组成的SOLiD 原始颜色序列。
e. 数据分析SOLiD 测序完成后,获得了由颜色编码组成的SOLiD 原始序列。
理论上来说,按照“双碱基编码矩阵”,只要知道所测DNA 序列中任何一个位置的碱基类型,就可以将SOLiD 原始颜色序列“解码”成碱基序列。
但由于双碱基编码规则中双碱基与颜色信息的简并特性(一种颜色对应4 种碱基对),前面碱基的颜色编码直接影响紧跟其后碱基的解码,所以一个错误颜色编码就会引起“连锁解码错误”,改变错误颜色编码之后的所有碱基。
和其它所有测序仪一样,测序错误在所难免,关键是对测序错误的评价和后续处理。
由于 SOLiD 系统采用了双碱基编码技术,在测序过程中对每个碱基判读两遍,从而减少原始数据错误,提供内在的校对功能。
