真核生物启动子预测相关数据库资源概述
生物信息-名词解释

逐个克隆法:对连续克隆系中排定的BAC克隆逐个进行亚克隆测序并进行组装(公共领域测序计划)。
全基因组鸟枪法:在一定作图信息基础上,绕过大片段连续克隆系的构建而直接将基因组分解成小片段随机测序,利用超级计算机进行组装。
单核苷酸多态性(SNP),主要是指在基因组水平上由单个核苷酸的变异所引起的DNA序列多态性。
遗传图谱又称连锁图谱,它是以具有遗传多态性(在一个遗传位点上具有一个以上的等位基因,在群体中的出现频率皆高于1%)的遗传标记为“路标”,以遗传学距离(在减数分裂事件中两个位点之间进行交换、重组的百分率,1%的重组率称为1cM)为图距的基因组图。
遗传图谱的建立为基因识别和完成基因定位创造了条件。
物理图谱是指有关构成基因组的全部基因的排列和间距的信息,它是通过对构成基因组的DNA分子进行测定而绘制的。
绘制物理图谱的目的是把有关基因的遗传信息及其在每条染色体上的相对位置线性而系统地排列出来。
转录图谱是在识别基因组所包含的蛋白质编码序列的基础上绘制的结合有关基因序列、位置及表达模式等信息的图谱。
比较基因组学:全基因组核苷酸序列的整体比较的研究。
特点是在整个基因组的层次上比较基因组的大小及基因数目、位置、顺序、特定基因的缺失等。
环境基因组学:研究基因多态性与环境之间的关系,建立环境反应基因多态性的目录,确定引起人类疾病的环境因素的科学。
宏基因组是特定环境全部生物遗传物质总和,决定生物群体生命现象。
转录组即一个活细胞所能转录出来的所有mRNA。
研究转录组的一个重要方法就是利用DNA芯片技术检测有机体基因组中基因的表达。
而研究生物细胞中转录组的发生和变化规律的科学就称为转录组学。
蛋白质组学:研究不同时相细胞内蛋白质的变化,揭示正常和疾病状态下,蛋白质表达的规律,从而研究疾病发生机理并发现新药。
蛋白组:基因组表达的全部蛋白质,是一个动态的概念,指的是某种细胞或组织中,基因组表达的所有蛋白质。
代谢组是指是指某个时间点上一个细胞所有代谢物的集合,尤其指在不同代谢过程中充当底物和产物的小分子物质,如脂质,糖,氨基酸等,可以揭示取样时该细胞的生理状态。
真核生物的启动子

真核生物的启动子由于真核生物中有三种不同的RNA聚合酶,因此也有三种不同的启动子,其中以启动子Ⅱ最为复杂,它和原核的启动子有很多不同:(1)有多种元件:TATA 框,GC框,CATT框,OCT等;(2)结构不恒定。
有的有多种框盒如组蛋白H2B;有的只有TATA框和GC框,如SV40早期转录蛋白,(3)它们的位置、序列、距离和方向都不完全相同,(4)有的有远距离的调控元件存在,如增强子;(5)这些元件常常起到控制转录效率和选择起始位点的作用;(6)不直接和RNA pol 结合。
转录时先和其它转录激活因子相结合,再和聚合酶结合。
(一)Ⅱ类基因的启动子和调控区Ⅱ类基因的启动子由核心元件和上游元件组成。
核心元件包括TATA框和转录起始位点附近的启始子(initiator,Inr)。
在起始点一般没有同源序列,但mRNA的第一个碱基倾向A,另一侧翼由Py 组成(在原核启动子的CAT起始序列也有这种情况),称为起始子(initiator),一般由PY2CAPY5构成,位于-3~+5,可能提供RNA pol Ⅱ识别。
无论TATA是否存在,Inr对于启动子的强度和起始位点的选择都是十分重要的。
现已分离纯化了与Inr特异结合的蛋白质因子。
1.核心元件TATA框合又称Hogness框,Goldberg-Hogness框,俚语称为金砖(Goldbrick),其一致序列是:T85A97T93A85A63A83A50,常在起始位点的上游-25左右,相当于原核的-10序列。
但-10是不可缺少的,而真核启动中也有的缺乏TATA 框。
其作用是:(1) 选择正确的转录起始位点,保证精确起始,故也称为选择子(selector),当有的基因缺少TATA框时,可能由Inr来替代它的这一作用,如鼠的脱氨核苷转移酶(Tdt)基因就没有TATA框,但有17bp的Inr;(2) 影响转录的速率。
TATA框的8bp的保守序列一般都是由A.T对组成,少数情况在其中的两个位点上由G.C对取代了A.T,可见它是较容易打开。
常见真核启动子及原核启动子特点
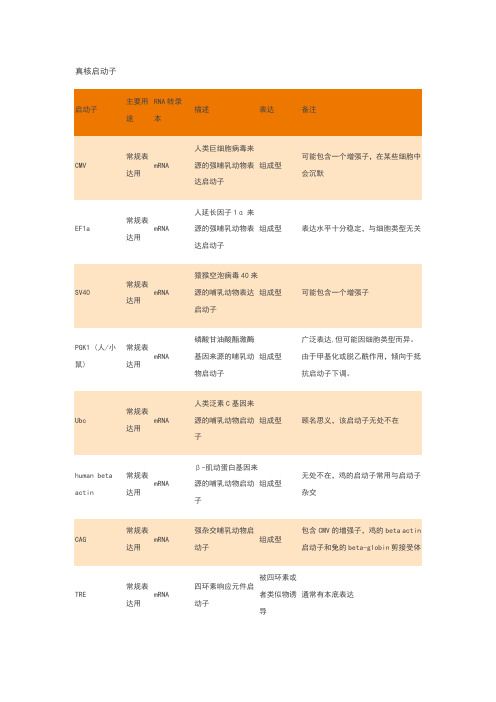
真核启动子EF1a常规表达用mRNA人延长因子1α来源的强哺乳动物表达启动子组成型表达水平十分稳定,与细胞类型无关PGK1 (人/小鼠)常规表达用mRNA磷酸甘油酸酯激酶基因来源的哺乳动物启动子组成型广泛表达,但可能因细胞类型而异。
由于甲基化或脱乙酰作用,倾向于抵抗启动子下调。
human beta actin常规表达用mRNAβ-肌动蛋白基因来源的哺乳动物启动子组成型无处不在,鸡的启动子常用与启动子杂交TRE常规表达用mRNA四环素响应元件启动子被四环素或者类似物诱导通常有本底表达Ac5常规表达用mRNA果蝇Actin 5c 基因来源的强昆虫启动子组成型果蝇表达系统的常用启动子CaMKIIa 光遗传学基因表达mRNACa2+/钙调蛋白依赖的蛋白激酶 II 启动子特异的用于中枢神经系统/神经元表达。
受到钙和钙调蛋白调节。
TEF1常规表达用mRNA酵母转录延伸因子启动子组成型与哺乳动物的EF1a 启动子类似ADH1常规表达用mRNA乙醇脱氢酶I的酵母启动子被乙醇抑制全长版本很强,促进高表达。
截短启动子是组成型的,表达较低。
Ubi常规表达用mRNA玉米泛素基因的植物启动子组成型在植物中促进高表达U6小RNA表达shRNA来源于人U6小核启动子组成型小鼠U6也使用,但效率略差。
常用的原核表达系统启动子T7lac高水平基因表达T7噬菌体来源的启动子加上lac 操纵子 几乎没有本底表达,需要T7 RNA 聚合酶,受到lac 操纵子的控制,可以被IPTG 诱导。
常用与pET 载体,受到lac 操纵子的严格调控Sp6体外转录/常规表达Sp6噬菌体来源的启动子 组成型, 需要SP6 RNA 聚合酶当用于体外转录的时候,专路方向有可能是正向的也可能是反向的,取决于启动子相对于目的基因的方向araBAD常规表达用阿拉伯糖代谢操纵子的启动子阿拉伯糖诱导 弱,常用与pBAD 载体。
适合于快速调控和低的本底表达lac 常规表达用Lac操纵子来源的启动子可以被IPTG或者乳糖诱导在常规的大肠杆菌中,lacI阻遏蛋白表达量不高,仅能满足细胞自身的lac操纵子,无法应付多拷贝的质粒的需求,导致非诱导条件下较高地表达,为了让表达系统严谨调控产物表达,能过量表达lacI阻遏蛋白的lacIq 突变菌株常被选为Lac/Tac/trc表达系统的表达菌株。
(工具篇):如何查找基因的启动子及预测转录因子?

(工具篇):如何查找基因的启动子及预测转录因子?最近长链非编码RNA(lncRNA)很火热,好不容易找到了一个心仪的lncRNA(关于怎么找,我们之前也聊过:自己做测序、芯片;从别人的数据里挖据;或移植研究从其他疾病里扯一个过来验证),那么问题来了:分子有了,机制部分我该往哪个方向扯呢?很多人可能都会仔细寻找下游靶分子,以证明该lncRNA参与了xx调控,具有某个功能,表明该lncRNA分子在疾病发生发展过程中起到了很重要的作用。
其实,我们还可以往上游做,以丰富机制研究的深度。
今天我们就聊一聊,预测一下参与调控lncRNA表达转录因子的方法。
今天我们通过2个方式进行预测:1、需要用到UCSC、PROMO数据库首先,我们需要找到lncRNA的启动子序列。
打开UCSC数据库:举例:HOTAIR输入:HOTAIR点击GO点击红色的那个序列得到这么一个图,点击红色框,继续点击,得到这个界面,我们需要修改一些参数:转录起始位点上游2000nt和下游100nt区域为我们所选的启动子区。
SubmitOK,启动子序列有了。
拷贝下来。
接下来,我们打开PROMO数据库:http://alggen.lsi.upc.es/cgi-bin/promo_v3/promo/promoinit.cgi?dirDB=TF_8.3在SelectSpecies进行部分设置,Submit另外,如果对转录因子有选择的话,也可以在SelectFactors中进行设置。
最后,我们点击SearchSites将刚刚得到的启动子序列粘贴进行。
另外,默认容错率15%,如果得到的转录因子过多,我们可以进行调整,设置成5%或0%。
Submithttp://alggen.lsi.upc.es/cgi-bin/promo_v3/promo/promo.cgi?dirDB=TF_8.3&idCon=148056 381600&getFile=resumSearchRes.html我最终设置了容错率为0,一共得到了120个预测的转录因子。
真核生物启动子TATA_box__省略__box和CAAT_box的分析_张小辉

真核生物启动子TATA2box・GC2box和CAAT2box的分析张小辉,祁艳霞 (河南科技大学动物科技学院,河南洛阳471003)摘要 下载了已经通过试验证实的2541条真核生物启动子序列,运用生物信息学方法分析了T A T A2b ox、G C2b ox和C AA T2b ox的数量及其在启动子中的分布情况。
结果表明,有23.85%真核生物启动子序列中至少有1个T A T A2b ox,且T A T A2b ox主要分布在转录起始位点前24~36bp的区域内;有47.30%真核生物启动子序列中至少有1个G C2b ox,且G C2b ox在转录起始位点前23~128bp的区域内分布比较集中;有42.35%真核生物启动子序列中至少有1个C AA T2b ox,且C AA T2b ox在转录起始位点前51~159bp的区域内分布比较集中。
这说明T A T A2b ox在真核生物启动子中的位置比较固定,对基因转录的正确起始可能起着重要的作用;而G C2b ox和C AA T2b ox分布区域较广,数量也明显多于T A T A2b ox。
关键词 真核生物;启动子;T A T A2b ox;G C2b ox;C AA T2b ox中图分类号 Q789 文献标识码 A 文章编号 0517-6611(2008)04-01380-02A nalysis on T A T A2box,G C2box and CAA T2box in Euk aryotic P romotersZH ANG X iao2hui et al (C ollege of Anim al Science and T echn ology,H enan University of Science and T echn ology,Lu oyang,H enan471003) Abstract 2541eukary otic prom oter sequences that had been validated by test were d ownloaded and the number of T A T A2b ox,G C2b ox and C AA T2b ox and its distribution conditions in the prom oters were analyzed by bioin form atics m eth od.T he results sh owed that23.85%eukary otic prom oter sequences had at least one T A T A2b ox and T A T A2b ox distributed m ainly in the area23~128bp ahead of transcription initiation site.47.30%eukary otic prom oter sequences had at least one G C2b ox and G C2b oxes were concentrated in the area23~128bp ahead of transcription initiation site.42.35%eukary otic pro2 m oter sequences had at least one C AA T2b ox and C AA T2b oxes were concentrated in the area51~159bp ahead of transcription initiation site.T he results indicated that the position of T A T A2b ox in eukary otic prom oters was relatively fixed and it m ight play an im portant role in the correct initiation of gene transcription.But G C2b ox and C AA T2b ox were w idely distributed and their number was obviously m ore than that of T A T A-b ox.K ey w ords Eukary ote;Prom oter;T A T A2b ox;G C2b ox;C AA T2b ox 启动子是位于结构基因5’端上游的1段DNA序列,能够指导全酶同模板正确结合,活化RNA聚合酶,启动基因转录。
常见真核启动子及原核启动子特点

For personal use only in study and research; not for commercial use真核启动子EF1a常规表达用mRNA人延长因子1α来源的强哺乳动物表达启动子组成型? 表达水平十分稳定,与细胞类型无关PGK1 (人/小鼠)常规表达用mRNA磷酸甘油酸酯激酶基因来源的哺乳动物启动子组成型? 广泛表达,但可能因细胞类型而异。
由于甲基化或脱乙酰作用,倾向于抵抗启动子下调。
human beta actin常规表达用mRNAβ-肌动蛋白基因来源的哺乳动物启动子组成型? 无处不在,鸡的启动子常用与启动子杂交TRE常规表达用mRNA四环素响应元件启动子被四环素或者类似物诱导通常有本底表达Ac5常规表达用mRNA果蝇Actin 5c 基因来源的强昆虫启动子组成型? 果蝇表达系统的常用启动子CaMKIIa光遗传学基因表达mRNA?Ca2+/钙调蛋白依赖的蛋白激酶 II 启动子特异的 用于中枢神经系统/神经元表达。
受到钙和钙调蛋白调节。
TEF1常规表达用mRNA酵母转录延伸因子启动子组成型? 与哺乳动物的EF1a 启动子类似ADH1常规表达用mRNA乙醇脱氢酶I 的酵母启动子被乙醇抑制全长版本很强,促进高表达。
截短启动子是组成型的,表达较低。
Ubi常规表达用mRNA玉米泛素基因的植物启动子组成型? 在植物中促进高表达U6小RNA 表达shRNA 来源于人U6小核启动子组成型? 小鼠U6也使用,但效率略差。
常用的原核表达系统启动子T7lac 高水平基因表达T7噬菌体来源的启动子加上lac操纵子几乎没有本底表达,需要T7 RNA聚合酶,受到lac操纵子的控制,可以被IPTG诱导。
常用与pET载体,受到lac操纵子的严格调控araBAD 常规表达用阿拉伯糖代谢操纵子的启动子阿拉伯糖诱导弱,常用与pBAD载体。
适合于快速调控和低的本底表达lac 常规表达用Lac操纵子来源的启动子可以被IPTG或者乳糖诱导在常规的大肠杆菌中,lacI阻遏蛋白表达量不高,仅能满足细胞自身的lac操纵子,无法应付多拷贝的质粒的需求,导致非诱导条件下较高地表达,为了让表达系统严谨调控产物表达,能过量表达lacI阻遏蛋白的lacIq 突变菌株常被选为Lac/Tac/trc表达系统的表达菌株。
原核真核启动子及BL21相关知识

感受态BL21、BL21(DE3)及BL21(DE3)pLysSBL21没有T7 RNA聚合酶基因, 因此不能表达T7启动子控制的目的基因, 但是可以表达大肠杆菌启动子lac、tac、trc及trp控制的基因。
BL21(DE3)是λDE3溶原菌,带有T7 RNA聚合酶,可用于表达T7启动子控制的目的基因,也可以表达大肠杆菌启动子lac、tac、trc及trp控制的基因。
BL21(DE3)pLysS含有pLysS质粒,一种与pET共存的表达T7裂解酶的质粒,可以减少目的基因的本底表达,提供更严紧的控制。
真核原核启动子原核:几个常用的启动子和诱导调控表达系统1.最早应用于的表达系统的是Lac乳糖操纵子,由启动子lacP + 操纵基因lacO + 结构基因组成。
其转录受CAP正调控和lacI负调控。
cUV5突变能够在没有CAP的存在下更有效地起始转录,该启动子在转录水平上只受lacI 的调控,因而随后得到了更广泛采用。
lacI产物是一种阻遏蛋白,能结合在操纵基因lacO 上从而阻遏转录起始。
乳糖的类似物IPTG可以和lacI产物结合,使其构象改变离开lacO,从而激活转录。
这种可诱导的转录调控成为了大肠杆菌表达系统载体构建的常用元件。
3.tac启动子是trp启动子和lacUV5的拼接杂合启动子,且转录水平更高,比lacUV5更优越。
4.trc启动子是trp启动子和lac启动子的拼合启动子,同样具有比trp更高的转录效率和受lacI阻遏蛋白调控的强启动子特性。
5.在常规的大肠杆菌中,lacI阻遏蛋白表达量不高,仅能满足细胞自身的lac操纵子,无法应付多拷贝的质粒的需求,导致非诱导条件下较高地表达,为了让表达系统严谨调控产物表达,能过量表达lacI阻遏蛋白的lacIq 突变菌株常被选为Lac/Tac/trc表达系统的表达菌株。
现在的Lac/Tac/trc载体上通常还带有lacIq 基因,以表达更多lacI阻遏蛋白实现严谨的诱导调控。
真核生物启动子预测相关数据库资源概述

真核生物启动子预测相关数据库资源概述刘玉瑛1,张江丽2(1.首都师范大学生命科学学院,北京100037;2.廊坊师范学院生命科学学院,河北廊坊065000)摘要启动子是基因表达调控的重要元件,深入研究启动子的结构和功能,是理解基因转录调控机制和表达模式的关键。
随着生物技术和计算机技术的高速发展,应用生物信息学技术对启动子进行预测和分析的方法得到了很大发展。
对目前常用的真核生物启动子预测相关数据库和软件资源作了简单介绍。
关键词真核生物;启动子;数据库;预测中图分类号Q24文献标识码A文章编号0517-6611(2007)24-07418-02The Databases o f Eukaryo tic Promoters and Related So ftw are ResourcesLIU Yu2ying et al(Co lleg e of Life Science,Capital N ormal U niv ersity,B eijin g100037)Abstract Eu kary o tic pro mo ters are i mp ortan t elemen ts in reg ulatio n o f the e xpres si on.T o stud y the structu re and functio n o f a p ro m oter deeply,i t is the key to kno w ho w the gene reg ulates its transcri pti on an d starts its exp ression.With the fast d evelo pmen t o f bio log ical and co m puter techno lo gy,sig nifican t ac hiev ements h av e been made in co mp utatio nal predictio n o n Eu kary o tic pro mo ters.In thi s paper mai nly in tro duces the pro g ress made in the datab ases o f predictin g E ukaryo tic p ro mo ters as w ell as the related so ftw are reso urces w as in tro duced.Key w ords Wikipedia;Pro m oter;D atab ase;Predicti on作为基因表达所必需的重要序列信号和基因转录水平上一种重要的调控元件,真核生物的启动子一直是现代分子生物学的研究热点。
启动子-文档资料

2012.11.22
.
1
一.启动子相关介绍
1.1 启动子的概念
启动子:一段能被RNA聚合酶特异性识别和结合,能正 确有效的起始转录的一段DNA调控序列,一般位于基因5端 上游区域。真核生物具有三种RNA聚合酶,而RNA 聚合酶Ⅱ 型启动子在真核生物中最为常见,也最为复杂。有多复杂? 比如,有包括TATA框、GC框等多种元件;比如存在远端调 控序列;比如通过多种转录因子间接与RNA聚合酶结合。
.
9
图1:核心启动子机制图
弥散型核心启动子往往只含有集中型启动子部分的核心元件, 没有固定的核心元件及核心调控模式,其调控模式比集中型要复 杂得多。
.
10
图2.分散型核心启动子机制
.
11
1.4 研究启动子的意义
研究启动子的意义:研究启动子的功能序列对于基 因表达调控机制的研究具有十分重要的意义,能使外源 基因高效,准确,长期稳定地在目的部位表达的启动子 序列是基因治疗和基因工程研究的热点之一。
.
2
启动子是在DNA转录为RNA这一步过程中发挥作用的, 在此要与DNA自身复制起始点(称作复制子)和由mRNA翻译 为蛋白质时的翻译起始点(以起始密码子ATG为标志)区别 开来。.3. Nhomakorabea4
1.2 启动子的认识
传统的启动子理论认为人类每个基因平均拥有约一 个启动子且启动子上供读取信息的起始点只有一个、一 个启动子只能转录出一种 RNA 。然而,来自日本理化 研究所等机构的研究人员近日在《自然·遗传学》杂志 上发表论文说,他们分析了约 1400 万条 RNA 链,并 以此为线索研究这些 RNA 链是从 DNA 的哪个部位开始 转录而成的。研究人员在此基础上确定,人类 DNA 拥 有的启动子数量超过 19 万个,平均每个基因至少有 5 个启动子。
一文教会你查找基因的启动子、UTR、TSS等区域以及预测转录因子结合位点

一文教会你查找基因的启动子、UTR、TSS等区域以及预测转录因子结合位点基础知识首先我们了解一些基础知识(注:文中图片皆可点击放大查看!):启动子(promoter):与RNA聚合酶结合并能起始mRNA合成的序列。
做生信分析时,一般选择上游1 kb,下游 500 nt,也有选上下游各1 kb的。
如果关注核心启动子,可见生信宝典之前发布的Jaspar数据库介绍。
获取正链或负链的启动子序列时要注意方向。
之前awk的教程中有些提及。
转录起始点(TSS):转录时,mRNA链第一个核苷酸相对应DNA链上的碱基,通常为一个嘌呤。
UTR(Untranslated Regions):即非翻译区,是信使RNA (mRNA)分子编码区(CDS)两端的非编码片段。
5’-UTR从mRNA起点的甲基化鸟嘌呤核苷酸帽延伸至AUG起始密码子,3’-UTR从编码区末端的终止密码子延伸至多聚A尾巴(Poly-A)的末端。
生信老司机以中心法则为主线讲解组学技术的应用和生信分析心得- 限时免费中讲述了如何基于高通量数据对这些区域的调节变化进行分析,可配合此文观看。
1. 查找基因的启动子区域-NCBI1. 打开PubMed:/pubmed2. 选择Gene,输入IL17A,点击search,结果如下图,点击第一个:3. 下拉到下图位置,可以看到该基因的以下信息:点击Tools,选择Sequence Text View:还可以看到如下序列信息:4. 以上只是该基因的一些信息,可以用于查找相应的UTR等区域,下面进入正题,寻找promoter区域。
还是拉到如下图位置,点击FASTA:5. 基因位置信息如下图:6. 一般认为基因上游2 kb区域为该基因的promoter区域,所以将基因上游2 kb序列调出来:7. 复制上述序列就是基因的启动子序列了。
2. 查找基因的启动子区域-UCSC1. 打开UCSC:/,点击Table Browser:2. 按照下图所示填好基因相关信息,点击get output:3.选择genomic:4. 勾选Promoter/Upstream by选项,并将其改为2000 bases,然后点击get sequence:5. 得到下面的序列信息,开头直到第一个大写字母前面的所有小写字母序列即为该基因的promoter序列,你可以跟NCBI上得到的序列比对一下,看看是不是一样的呢?3. 转录因子结合位点的预测1.后面的预测步骤是改版前的Jaspar,可见上一篇介绍Jaspar的文章学习在新版Jaspar中怎么预测启动子区域的转录因子结合位点。
真核生物启动子研究概述

真核生物启动子研究概述李圣彦;郎志宏;黄大昉【期刊名称】《生物技术进展》【年(卷),期】2014(000)003【摘要】启动子是调控基因表达的重要基因元件,它的调控作用是多层次多因素共同作用的结果。
通过启动子的调控能够控制基因表达的水平、部位及方式。
深入研究启动子对于了解生物的生长发育、防御系统、疾病等都有非常重要的意义。
本文综述了启动子克隆、生物信息学分析和预测的方法,比较了两种启动子分析方法,介绍了启动子甲基化、多态性和合成启动子的研究进展,以期能够为启动子的研究提供参考。
%Promoter is an important element in regulation of gene expression. The regulation role of promoter is the result of a multi-level interaction of multiple factors. The level, location and manner of gene expression are regulated by the promoter, so deeply study of promoter isof great importance for understanding biological growth, defense system and disease. This paper reviews the methods of promoter cloning, bioinformatics analysis and forecasting of promoter, compares two methods of promoter analysis, introduces the progress of promoter methylation and polymorphism, so as to provide a reference for further study of promoters.【总页数】7页(P158-164)【作者】李圣彦;郎志宏;黄大昉【作者单位】中国农业科学院生物技术研究所,农业部农业基因组学重点实验室北京,北京100081;中国农业科学院生物技术研究所,农业部农业基因组学重点实验室北京,北京100081;中国农业科学院生物技术研究所,农业部农业基因组学重点实验室北京,北京100081【正文语种】中文【相关文献】1.真核生物启动子TATA-box·GC-box和CAAT-box的分析 [J], 张小辉;祁艳霞2.几个真核生物启动子计算机预测数据库资源概述 [J], 刘玉瑛;张江丽3.真核生物启动子的预测技术 [J], 孙吉贵;韩霄松;卢欣华;行荣;仲洋4.真核生物启动子的研究及应用 [J], 黄玉;杨波;迟小华;刘丽宏;李薇;卢学春5.真核生物启动子的鉴定方法 [J], 石慧;李建远因版权原因,仅展示原文概要,查看原文内容请购买。
DNA启动子概述

DNA启动子概述DNA启动子是一种在真核生物基因表达过程中起关键作用的DNA区域。
它位于基因的上游区域,通常由一系列特定的序列组成,能够与转录因子结合并调控基因的转录活性。
DNA启动子的功能是启动基因的转录过程,从而使DNA上的编码信息转化为RNA分子,为蛋白质的合成奠定基础。
本文将详细探讨DNA启动子的结构和功能。
DNA启动子的结构一般包括两个主要部分:核心启动子和调控元件。
核心启动子位于基因序列的最近上游区域,通常由一段约50到100碱基对长的DNA序列组成。
这个区域包括转录起始位点(TSS)和与RNA聚合酶结合的序列。
核心启动子是转录起始的基本位置,是RNA聚合酶与DNA结合的特定区域。
除了核心启动子,DNA启动子周围还存在其他调控元件,比如启动子增强子和抑制子。
启动子增强子是一类能够增强基因启动子活性的DNA序列,它通常位于核心启动子之上游区域。
增强子可与转录因子及其他调控蛋白结合,提高转录因子与启动子核心序列的亲和力,从而促进RNA聚合酶的结合和基因的高水平转录。
相反,启动子抑制子则是一类能够抑制启动子活性的DNA序列,通常位于基因序列的上游或下游区域。
抑制子与转录因子结合后,能够阻止RNA聚合酶的结合,从而降低基因的转录活性。
DNA启动子的核心序列通常包含一些高度保守的序列单元。
其中一个重要的序列单元是转录起始位点(TSS),它是RNA聚合酶开始转录的位置。
TSS位点通常在基因的上游区域,与RNA聚合酶结合后,起始转录活性,开始将DNA序列翻译成RNA。
除了TSS,其他常见的核心序列单元包括TATA盒、CCAAT盒和GC盒。
TATA盒是最为典型的核心序列,通常位于TSS之前约25到35碱基对处,它的序列为TATAAA。
TATA盒在转录起始过程中起关键作用,能够吸引和定位RNA聚合酶与启动因子的结合。
CCAAT盒和GC盒是进一步调控基因转录的其他序列单元。
它们位于TSS 之上游和核心序列之外,能够与转录因子结合,调节基因的转录活性。
3PlantCARE数据库htt...

摘要启动子是基因表达调控重要的元件,启动子的活性间接反映了它所控制的基因表达。
组织特异性启动子作为启动子的一种,可以启动外源基因在受体植物的特定组织器官中高效表达,减少不必要的浪费。
WRKY基因家族是在植物中起特异作用的一类转录调控因子,它被证明了参与植物的抗病反应,还影响植物的衰老、抗胁迫以及生长和发育。
本实验克隆了一个玉米WRKY基因的启动子,采用GUS报告基因对WRKY 基因的启动子功能进行了分析,通过启动子的删减实验,水稻的遗传转化及组织的GUS染色,取得如下结果:1、根据网上预测的WRKY基因设计引物,从玉米(玉米品种B73)中扩增了该基因的启动子部分,命名为P2880,在启动子顺式作用元件预测网站上分析该启动子序列,预测到存在TATA盒、CAAT盒和GATA盒等多个作用元件。
2、克隆出WRKY基因上游的启动子并且克隆了4个5’端缺失启动子,5个启动子的长度依次为2880bp、1812bp、1254bp、680bp和355bp。
将5个启动子与含有GUS报告基因的质粒pCAMBIA1301连接并构建了pCAM2880GUS和4个5’端缺失启动子载体,将缺失载体分别命名为pCAM1812GUS、pCAM1254GUS、pCAM680GUS和pCAM355GUS。
3、用农杆菌介导法将所有植物表达载体转化水稻,获得了37棵转基因植株。
由实验结果可知,pCAM2880GUS载体对应的转基因植株愈伤组织染色结果为蓝色,而pCAM1812GUS、pCAM1254GUS、pCAM680GUS、pCAM355GUS载体对应的转基因植株的愈伤组织均未染上蓝色,由此可知P2880启动子的核心区域位于转录起始位点前2880bp至1812bp之间。
综上所述,本研究克隆了一个玉米WRKY基因启动子,并发现该启动子是一个愈伤特异性启动子,而目前WRKY基因未有愈伤组织表达特异性的报道,该启动子的克隆对玉米相关基因功能研究具有重要的意义。
真核生物启动子数据库

302–303Nucleic Acids Research,2000,Vol.28,No.1©2000Oxford University PressThe Eukaryotic Promoter Database(EPD)Rouaïda Cavin Périer,Viviane Praz,Thomas Junier,Claude Bonnard and Philipp Bucher* Swiss Institute of Bioinformatics and Swiss Institute for Experimental Cancer Research,Ch.des Boveresses155, 1066-Epalinges s/Lausanne,SwitzerlandReceived October6,1999;Accepted October8,1999ABSTRACTThe Eukaryotic Promoter Database(EPD)is an anno-tated non-redundant collection of eukaryotic POL II promoters for which the transcription start site has been determined experimentally.Access to promoter sequences is provided by pointers to positions in nucleotide sequence entries.The annotation part of an entry includes a description of the initiation site mapping data,exhaustive cross-references to the EMBL nucleotide sequence database,SWISS-PROT, TRANSFAC and other databases,as well as biblio-graphic references.EPD is structured in a way that facilitates dynamic extraction of biologically mean-ingful promoter subsets for comparative sequence analysis.WWW-based interfaces have been devel-oped that enable the user to view EPD entries in different formats,to select and extract promoter sequences according to a variety of criteria,and to navigate to related databases exploiting different cross-references.The EPD web site also features yearly updated base frequency matrices for major eukaryotic promoter elements.EPD can be accessed at http://www.epd.isb-sib.chDATABASE DESCRIPTIONThe term promoter has two different meanings in biology:(i)a gene region immediately upstream of a transcription initiation site,and(ii)a cis-acting genetic element controlling the rate of transcription initiation of a gene.The Eukaryotic Promoter Database(EPD)is a database of promoters in the former sense. Information about promoters in the latter sense can be found in other databases such as TRANSFAC(1),ooTFD(2),TRRD (3),PlantCARE(4)and PLACE(5).EPD was originally designed as a resource for comparative sequence analysis and,as such,has played an instrumental role in the characterization of eukaryotic transcription control elements(6,7),as well as in the development of eukaryotic promoter prediction algorithms(8).The main purpose of the database is to keep track of experimental data that define transcription initiation sites of eukaryotic genes.This type of functional information is linked to promoter sequences via machine-readable pointers to positions within sequences of the EMBL nucleotide sequence database(9).EPD is a rigorously selected,curated and quality-controlled database.In order to be included,a promoter must fulfill a number of conditions laid down in the user manual.Most importantly,the transcription start site must be mapped experimentally with an estimated precision of 5bp or higher. All information in EPD originates from a critical examination and independent interpretation of the experimental data presented in the cited research publications.Published conclusions and feature table annotations in EMBL entries are never blindly relied upon.At present,EPD is confined to promoters recognized by the RNA POL II system of higher eukaryotes(multicellular plants and animals).Note that this restriction does not a priori exclude viral promoters.EPD is also a strictly non-redundant database.The general rule is that one entry corresponds to one transcription initiation site in a anisms are distinguished at the taxonomic level of the species.According to this policy,data from different literature sources pertaining to the same transcription initiation sites are represented by the same entry.Likewise, promoters belonging to different alleles of the same gene,or to the same gene in different subspecies,are covered by the same entry regardless of whether they differ in sequence.The user manual provides more details about how certain non-trivial cases such as promoters of tandemly repeated genes or retro-transposable elements,are handled.A comprehensive description of the contents and format of EPD has been published earlier(10).User interfaces and software support for local installations have been previously described(11).RECENT DEVELOPMENTSDatabaseThe objective of exhaustive cross-referencing between EPD promoters and EMBL sequences is being given high priority at the moment,especially with regard to genomes that are complete(Caenorhabditis elegans)or at an advanced stage of sequencing(Arabidopsis,Drosophila,human).As a consequence, the number of EMBL cross-references has increased by>1000 since last year(Table1).Moreover,the internal EPD cross-references have been revised.Until now,such links were only used to connect alternative promoters of the same gene.In future releases,promoters of different genes occurring at a short distance from each other(<1000bp),will also be cross-referenced.Such pairs of promoters usually promote transcription in opposite directions and often share upstream regulatory elements.As a new format feature,a keyword(KW)line type*To whom correspondence should be addressed.Tel:+41216925892;Fax:+41216925945;Email:philipp.bucher@isrec.unil.chNucleic Acids Research,2000,Vol.28,No.1303has been introduced and so far been populated with keywords imported from SWISS-PROT(12).This feature is intended to enhance the query capabilities of various access tools.Additional keywords relating to properties of the promoter rather than to properties of the corresponding gene product will be added in the near future.DocumentationThe user manual has been extensively revised.Bibliographic references have been added to the section explaining the representation of transcript mapping data.Some of them are accompanied by direct hyperlinks to figures in online journals exemplifying a particular technique.Several additional documents have recently been made available over the web.One contains a list of all‘homology groups’defined in EPD.Such groups consist of homologous promoters exhibiting significant sequence similarity in the–79to+20region among themselves. Another document presents the hierarchical promoter classifi-cation system of EPD.Promoter element descriptionsWeight matrix descriptions of four major eukaryotic promoter elements(TATA-box,initiator,GC-box and CCAAT-box) have previously been derived from EPD release17(7).We have now decided to make updated versions of such matrices available on a yearly basis from the EPD web pages.The latest versions were produced from EPD release60using a Baum–Welch hidden Markov model training algorithm(program buildmodel of SAM release1.3.3,Hughey&Krogh1998,http://www. /research/compbio/sam.html).ACCESSFTPThe following files are available from ftp.epd.isb-sib.ch/pub/ databases/epd•Flat-files containing the EPD database in the new and in the old format.•EPD user manual.•Sequence libraries in EMBL and FASTA format containing promoter sequences from–499to+100relative to the tran-scription start site.•A slightly reduced version of EPD in ASN.1format designed for import into the GenBank–Entrez data environment(13), including a formal data description in ASN1.•Icarus scripts for indexing EPD by SRS(14).WWWThe following services are offered at http://www.epd.isb-sib.ch •Access to EPD entries by ID or accession number.The following formats are available:text only,HTML and HTML combined with a graphic representation of sequence objects by a Java applet(15).•A page for downloading promoter sequence subsets defined in EPD.•Access to EPD entries and corresponding promoter sequences via a query form.Access to EPD via SRS is provided by the Swiss EMBNet node at /SUPPLEMENTARY MATERIALRelevant URL links are available at NAR Online.ACKNOWLEDGEMENTEPD is funded in part by grant31-54782.98from the Swiss National Science Foundation.REFERENCES1.Heinemeyer,T.,Chen,X.,Karas,H.,Kel,A.E.,Kel,O.V.,Liebich,I.,Meinhardt,T.,Reuter,I.,Schacherer,F.and Wingender,E.(1999)Nucleic Acids Res.,27,318–322.Updated article in this issue:Nucleic Acids Res.(2000),28,316–319.2.Ghosh,D.(1999)Nucleic Acids Res.,27,315–317.Updated article in thisissue:Nucleic Acids Res.(2000),28,308–310.3.Kolchanov,N.A.,Ananko,E.A.,Podkolodnaya,O.A.,Ignatieva,E.V.,Stepanenko,I.L.,Kel-Margoulis,O.V.,Kel,A.E.,Merkulova,T.I.,Goryachkovskaya,T.N.,Busygina,T.V.,Kolpakov,F.A.,Podkolodny,N.L.,Naumochkin,A.N.and Romashchenko,A.G.(1999)Nucleic Acids Res.,27,303–306.Updated article in this issue:Nucleic Acids Res.(2000),28,298–301.4.Rombauts,S.,Dehais,P.,Van Montagu,M.and Rouze,P.(1999)Nucleic Acids Res.,27,295–296.5.Higo,K.,Ugawa,Y.,Iwamoto,M.and Korenaga,T.(1999)Nucleic Acids Res.,27,297–300.6.Bucher,P.and Trifonov,E.N.(1986)Nucleic Acids Res.,22,10009–10026.7.Bucher,P.(1990)J.Mol.Biol.,212,563–578.8.Fickett,J.W.and Hatzigeorgiou,A.G.(1997)Genome Res.,7,861–878.9.Stoesser,G.,Tuli,M.A.,Lopez,R.and Sterk,P.(1999)Nucleic Acids Res.,27,18–24.Updated article in this issue:Nucleic Acids Res.(2000),28, 19–23.10.Cavin Périer,R.,Junier,T.and Bucher,P.(1998)Nucleic Acids Res.,26,353–357.11.Cavin Périer,R.,Junier,T.,Bonnard,C.and Bucher,P.(1999)Nucleic Acids Res.,27,307–309.12.Bairoch,A.and Apweiler,R.(1999)Nucleic Acids Res.,27,49–54.Updated article in this issue:Nucleic Acids Res.(2000),28,45–48.13.Benson,D.A.,Boguski,M.,Lipman,D.J.and Ostell,J.(1994)NucleicAcids Res.,22,3441–3444.14.Etzold,T.,Ulyanov,A.and Argos,P.(1996)Methods Enzymol.,266,114–128.15.Junier,T.and Bucher,P.(1998)In Silico Biol.,1,13–20.16.The Flybase Consortium(1999)Nucleic Acids Res.,27,85–88.17.Pearson,P.,Francomano,C.,Foster,P.,Bocchini,C.,Li,P.andMcKusick,V.(1994)Nucleic Acids Res.,22,3470–3473.18.Blake,J.A.,Richardson,J.E.,Davisson,M.T.and Eppig,J.T.(1999)Nucleic Acids Res.,27,95–98.Updated article in this issue:Nucleic Acids Res.(2000),28,108–111.Table1.Database cross-references in EPD release60Database Number of links EPD internal188EMBL(9)2978 TRANSFAC(1)1700SWISS-PROT(12)1058FlyBase(16)116MIM(17)234MGD(18)126 MEDLINE2393。
生物学数据库大全

生物学数据库大全★INSD,国际核酸序列数据库(International Nucleotide Sequence Databank)。
由日本的DDBJ、欧洲的EMBL和美国的GenBank三家各自建立和共同维护。
★EMBL库,欧洲分子生物学实验室的DNA和RNA 序列库。
/embl.html★GenBank,美国国家生物技术信息中心(NCBI)所维护的供公众自由读取的、带注释的DNA序列的总数据库。
/Web/Genbank/★ DNA Databank of Japan (DDBJ) ,日本核酸数据库。
http://www.ddbj.nig.ac.jp/★GSDB是由美国国家基因组资源中心(NCGR)维护的DNA序列关系数据库(Genome Sequence DataBase)。
/gsdb/★TIGR DATAbase,是世界上最大的cDNA数据库,还有大量的EST序列和人类基因索引(HGI)。
/tdb/hcd/overview.htmlDNA序列数据库包括与DNA的复制、转录、修复等有密切关系的蛋白质因子。
★BioSino是中国自主开发的核酸序列公共数据库。
/★CUTG,MM子使用频度表。
http://www.dna.affrc.go.jp/~nakamura/CUTG.htmlhttp://www.kazusa.or.jp/codon/http://www.dna.affrc.go.jp/~nakamura/CUTG.html★EPD,真核生物启动子数据库(Eukaryotic Promotor Database)。
http://www.epd.isb-sib.ch/★TRANSFAC,真核生物基因表达调控因子的数据库。
http://transfac.gbf.de/TRANSFAC★ TRRD.真核生物基因组转录调控区数据库。
http://www.mgs.bionet.nsc.ru/mgs/dbases/trrd4/★OOTFD,转录因子和基因表达数据库。
真核生物基因结构的预测分析

翻译
编码区预测
基因结构分析
蛋白质序列
蛋白质理化性质 二级结构预测 结构域分析 重要信号位点分析 三级结构预测
Codon bias 选择性剪切 GC Content 转录调控因子 限制性酶切位点
序列比对 功能注释 KEGG GO 系统发育树
3
真核生物基因的主要结构
4
基因结构分析常用软件
密码子表的选择
计算所有指数 27
CodonW结果界面
各项指数输出结果
密码子使用频率
28
• CAI (Codon Adaptation Index)密码子适应指数
目标基因与高表达基因的密码子偏好性的相似程度 (1完全相同,0完全不相同,本例为0.173)
• CBI (Condon Bias Index)密码子偏好指标
24
基因密码子偏好性
25
1.研究蛋白质结 构功能中的作用 2.在表达外源基 因方面的作用 3.在生物信息学 研究中的作用
26
基因密码子偏好性: CodonW
http://mobyle.pasteur.fr/cgi-bin/portal.py?form=codonw#forms::codonw
粘帖目的序列
BLAST比对到的三条mRNA序列
36
输入基因组序列或序列数据库号
输入相似性序列
判断用于分析的序列间的 差异,并调整比对参数 比对阈值
不受默认内含子长度限 制, 默认长度:内部内含子 为35kb, 末端内含子为 100kb 输出格式选择
选择物种
37
Spidey输出结果
第一条蓝色序列 为基因组序列, 橘黄色为外显子 外显子对应于 外显子对应于 基因组上的 mRNA/cDNA上的 起始/结束位置 起始/结束位置 供体、受体位点
《基于序列能量和结构信息的原核生物与真核生物启动子预测》范文

《基于序列能量和结构信息的原核生物与真核生物启动子预测》篇一一、引言原核生物和真核生物作为生命世界中的两大生物类型,各自在进化、发育、代谢等许多生物学方面具有显著的差异。
特别是在基因调控领域,这两种生物类型的启动子(promoter)结构与功能存在显著的差异。
随着生物信息学和计算生物学的发展,利用序列能量和结构信息来预测启动子已成为一种有效的方法。
本文将详细介绍基于序列能量和结构信息的原核生物与真核生物启动子预测的原理和方法。
二、序列能量与启动子预测(一)序列能量分析启动子是基因转录调控的重要区域,通常由DNA序列组成,含有特定模式的信息以指导转录因子的结合。
通过计算序列的能量变化,我们可以了解序列的稳定性和转录因子的结合能力。
在启动子预测中,我们主要关注的是序列的能量分布和变化规律。
(二)启动子预测基于序列能量的分析,我们可以对启动子进行预测。
通常,具有较低能量的序列更稳定,更有可能成为启动子的一部分。
通过比较已知的启动子序列的能量分布模式,我们可以对未知序列进行预测。
此外,还可以利用机器学习等方法建立预测模型,提高预测的准确度。
三、结构信息与启动子预测(一)结构信息分析除了序列能量外,DNA的结构信息也是启动子预测的重要依据。
DNA的结构包括双螺旋结构、碱基堆积、超螺旋等,这些结构可能影响转录因子的结合和基因的表达。
通过分析DNA的结构信息,我们可以更好地理解启动子的功能和作用机制。
(二)结构信息在启动子预测中的应用结合DNA的结构信息,我们可以更准确地预测启动子的位置和功能。
例如,通过分析DNA的弯曲程度、碱基堆积等结构特征,我们可以确定转录因子结合的位点,从而预测出可能的启动子区域。
此外,还可以利用三维结构模型等手段,进一步验证和优化预测结果。
四、原核生物与真核生物启动子的预测比较(一)原核生物与真核生物启动子的差异原核生物和真核生物的启动子在结构和功能上存在显著的差异。
原核生物的启动子通常较短,含有特定的转录因子结合位点;而真核生物的启动子则较为复杂,包含多个调控元件和辅助元件。
《2024年基于序列能量和结构信息的原核生物与真核生物启动子预测》范文

《基于序列能量和结构信息的原核生物与真核生物启动子预测》篇一一、引言基因调控在原核生物和真核生物中发挥着核心作用,其中启动子作为基因表达的首要调控元件,其识别与预测对于理解基因表达机制、疾病诊断和治疗等具有重要意义。
随着生物信息学和计算生物学的发展,基于序列能量和结构信息的启动子预测方法逐渐成为研究热点。
本文旨在探讨基于序列能量和结构信息的原核生物与真核生物启动子预测的方法及其应用。
二、启动子概述启动子是位于基因5'端的一类特殊DNA序列,能够识别、结合RNA聚合酶,从而启动基因的转录过程。
原核生物与真核生物的启动子在结构与功能上存在差异。
原核生物启动子通常较短,结构简单;而真核生物启动子结构复杂,包含多种调控元件。
启动子的准确预测对于理解基因表达模式、疾病发生机制及新药研发具有重要作用。
三、序列能量在启动子预测中的应用序列能量是指DNA序列中各碱基的能量分布情况,反映了序列的稳定性和信息含量。
在启动子预测中,可以通过分析序列能量分布,识别出潜在的启动子区域。
利用生物信息学软件和算法,可以计算DNA序列的能量分布,进而预测启动子的位置和类型。
这种方法在原核生物和真核生物的启动子预测中均有所应用。
四、结构信息在启动子预测中的作用除了序列能量外,DNA序列的结构信息也是启动子预测的重要依据。
通过分析DNA序列的二级结构和三级结构,可以更准确地识别潜在的启动子区域。
例如,某些特定的二级结构如发夹结构、茎环结构等可能在启动子区域形成,这些结构信息对于启动子的识别和预测具有重要价值。
此外,三维空间结构信息也可以为启动子预测提供有力支持。
五、原核生物与真核生物启动子预测的比较与分析原核生物与真核生物的启动子在结构和功能上存在差异,因此在预测方法上也有所不同。
对于原核生物,由于启动子结构相对简单,主要依靠序列能量和简单的结构信息进行预测。
而对于真核生物,由于启动子结构复杂,需要结合多种调控元件和高级结构信息进行预测。
真核生物的启动子

真核生物的启动子由于真核生物中有三种不同的RNA聚合酶,因此也有三种不同的启动子,其中以启动子Ⅱ最为复杂,它和原核的启动子有很多不同:(1)有多种元件:TATA 框,GC框,CATT框,OCT等;(2)结构不恒定。
有的有多种框盒如组蛋白H2B;有的只有TATA框和GC框,如SV40早期转录蛋白,(3)它们的位置、序列、距离和方向都不完全相同,(4)有的有远距离的调控元件存在,如增强子;(5)这些元件常常起到控制转录效率和选择起始位点的作用;(6)不直接和RNA pol 结合。
转录时先和其它转录激活因子相结合,再和聚合酶结合。
(一)Ⅱ类基因的启动子和调控区Ⅱ类基因的启动子由核心元件和上游元件组成。
核心元件包括TATA框和转录起始位点附近的启始子(initiator,Inr)。
在起始点一般没有同源序列,但mRNA的第一个碱基倾向A,另一侧翼由Py 组成(在原核启动子的CAT起始序列也有这种情况),称为起始子(initiator),一般由PY2CAPY5构成,位于-3~+5,可能提供RNA pol Ⅱ识别。
无论TATA是否存在,Inr对于启动子的强度和起始位点的选择都是十分重要的。
现已分离纯化了与Inr特异结合的蛋白质因子。
1.核心元件TATA框合又称Hogness框,Goldberg-Hogness框,俚语称为金砖(Goldbrick),其一致序列是:T85A97T93A85A63A83A50,常在起始位点的上游-25左右,相当于原核的-10序列。
但-10是不可缺少的,而真核启动中也有的缺乏TATA 框。
其作用是:(1) 选择正确的转录起始位点,保证精确起始,故也称为选择子(selector),当有的基因缺少TATA框时,可能由Inr来替代它的这一作用,如鼠的脱氨核苷转移酶(Tdt)基因就没有TATA框,但有17bp的Inr;(2) 影响转录的速率。
TATA框的8bp的保守序列一般都是由A.T对组成,少数情况在其中的两个位点上由G.C对取代了A.T,可见它是较容易打开。
那些转录因子相关数据库及如何预测转录因子结合位点?

那些转录因子相关数据库及如何预测转录因子结合位点?我们知道,大多数真核生物的基因都是不连续的,即基因的编码序列在DNA分子上是不连续的,被非编码序列隔开。
但真核生物基因在无转录因子时处于不表达状态,因为RNA聚合酶自身无法启动基因的转录,只有当转录因子(transcriptionfactor, TF, 一种蛋白质)结合到其识别的DNA序列上后,基因才开始表达。
首先我们要看,啥是转录因子呢?转录因子就是能结合在基因的启动子区域,可以启动基因表达,或者操纵基因表达,或者,应该有的可能会堵在前面阻碍基因表达的。
当然,在序列上转录因子不光会结合启动子区,还会结合在Enhancer,也就是促进子之类的元件上。
说简单点,就是转录因子,就是一种能结合在DNA上的蛋白。
于是就产生了这样的技术,用抗体固定在大珠子上,然后把转录因子的蛋白质沉淀下来。
沉淀的过程中,把染色质的DNA消化成片段,这样就能顺带着转录因子,把它结合的DNA序列拉下来了,这个就是ChIP,染色质免疫共沉淀。
而这里就涉及到另外一个概念,即转录因子结合位点(transcription factor binding site, TFBS), 它是转录因子调控基因表达时,与DNA上一段特殊的核苷酸序列相结合的区域,这段区域就称为启动子(Promoters),位于基因的调控区。
华丽丽的分割线在上一篇文章中,我们分享了如何寻找目的基因启动子序列(点我查看上篇帖子),那么接下来我们就要介绍一些转录因子相关的数据库及预测转录因子结合位点的问题了。
TRANSFAC数据库TRANSFAC数据库是基于真核生物转录调控所建立的数据库,由BIOBASE公司负责日常更新和维护工作,其中收集了大量与基因转录水平有关的数据,如转录因子及其DNA结合位点和相应的靶基因等信息。
该数据库分为公开版本和专业版本两个部分,用户只需登陆该网站,按照要求完成相应的注册,利用所获得的账号可以免费查询公开版本中所有的信息,而专业版本则需要用户付费使用。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
真核生物启动子预测相关数据库资源概述刘玉瑛1,张江丽2(1.首都师范大学生命科学学院,北京100037;2.廊坊师范学院生命科学学院,河北廊坊065000)摘要启动子是基因表达调控的重要元件,深入研究启动子的结构和功能,是理解基因转录调控机制和表达模式的关键。
随着生物技术和计算机技术的高速发展,应用生物信息学技术对启动子进行预测和分析的方法得到了很大发展。
对目前常用的真核生物启动子预测相关数据库和软件资源作了简单介绍。
关键词真核生物;启动子;数据库;预测中图分类号Q24文献标识码A文章编号0517-6611(2007)24-07418-02The Databases o f Eukaryo tic Promoters and Related So ftw are ResourcesLIU Yu2ying et al(Co lleg e of Life Science,Capital N ormal U niv ersity,B eijin g100037)Abstract Eu kary o tic pro mo ters are i mp ortan t elemen ts in reg ulatio n o f the e xpres si on.T o stud y the structu re and functio n o f a p ro m oter deeply,i t is the key to kno w ho w the gene reg ulates its transcri pti on an d starts its exp ression.With the fast d evelo pmen t o f bio log ical and co m puter techno lo gy,sig nifican t ac hiev ements h av e been made in co mp utatio nal predictio n o n Eu kary o tic pro mo ters.In thi s paper mai nly in tro duces the pro g ress made in the datab ases o f predictin g E ukaryo tic p ro mo ters as w ell as the related so ftw are reso urces w as in tro duced.Key w ords Wikipedia;Pro m oter;D atab ase;Predicti on作为基因表达所必需的重要序列信号和基因转录水平上一种重要的调控元件,真核生物的启动子一直是现代分子生物学的研究热点。
用实验的方法分析和鉴定启动子是多年以来进行启动子研究的主要途径。
近年来,随着人类基因组测序的完成和根据实验获得的对启动子的序列特征与结构功能的认识,利用生物信息学的方法,通过计算机模拟和计算来预测基因启动子的相关信息获得越来越多的应用。
笔者对目前常用的几个启动子预测数据库和相关软件资源作一简单介绍。
1真核生物启动子的基本结构真核生物的启动子有3种类型,分别由R NA聚合酶Ñ、Ò和Ó进行转录。
典型的真核生物启动子由核心启动子、上游元件和应答元件构成。
核心启动子包括起始子和基本启动子。
其中起始子是DN A解链并起始转录的位点。
基本启动子序列为中心在-25~-30的7bp保守区,其碱基频率为:T85A97T93A85A2 63A83A50,通常被称为T A T A框或Goldberg2Ho gne ss框,具有选择正确的起始位点,保证精确起始的功能。
同时,T A T A框还能影响转录速率。
如兔的珠蛋白基因中T A T A框的保守序列A T AAA A人工突变为A TG T AA时,转录效率会下降80%。
上游元件主要包括C AA T框和GC框两种,均具有增强转录活性的功能。
其中,C AA T框的保守序列是GGC T2 C A A TC T,一般位于上游-75紧靠-80,与其相互作用的因子有C TF家族的成员C P1、C P2和核因子NF21等;GC框的保守序列是G TGGGC G GG GC AA T,常以多拷贝形式存在-90处,识别该序列的转录激活因子为Sp1。
两种上游元件同时存在或者只存在其中之一,但并非所有真核基因的启动子都存在上游启动子元件,有些植物细胞中几乎不存在C AA T框。
应答元件通常位于基因上游,能被转录因子识别和结合,从而调控基因的专一性表达。
如热激应答元件、激素应答元件、c A M P应答元件、金属应答元件、糖皮质激素应答元作者简介刘玉瑛(1982-),女,北京人,硕士研究生,研究方向:生物化学与分子生物学。
收稿日期2007204223件和血清应答元件等。
应答元件含有短重复序列,不同基因中应答元件的拷贝数相近。
2真核生物启动子预测相关数据库资源2.1EPD(Eukaryotic promote r database)[1]EP D数据库(http://w w w.e pd.isb2sib.ch/或者f tp://f tp.e pd.isb2sib.c h/ pub/da taba se s/epd)是一个针对真核R NA聚合酶II型启动子的非冗余数据库。
现有启动子序列数据1500多个,按层次组织。
关于启动子的描述信息直接摘自科学文献。
该数据库中所有的启动子均经过一系列实验证实,如:是否为真核R NA聚合酶Ò型启动子、是否在高等真核生物中有生物学活性、是否与数据库中的其他启动子有同源性等。
同时,EPD 与其他的相关数据库如EM B L、S WI S S2P RO T、TRA NS FAC等,实现了数据的交叉链接。
在其最新版本(第76版)中,EPD 将收集的启动子分为6大类:植物启动子、线虫启动子、拟南芥启动子、软体动物启动子、棘皮类动物启动子和脊椎动物启动子,共2997个条目,其中人类启动子有1871个,约占总数的62%。
EPD数据库是目前唯一一个源自实验数据的真核生物启动子数据库,是常用的预测软件测评的手段之一。
2.2PLAC E(Plant cis2ac ting regulatory DNA elements)[2]P LAC E数据库(http://w ww.dna.af frc.go.jp/htdoc s/PL AC E/, F TP服务器为ftp://ftp.dna.a ff rc.go.jp/)是从已发表文献中搜集植物顺式作用元件资料而建立的模体数据库(mo tif data base),始于1991年。
目前服务器位于日本农林渔业部。
P LAC E数据库中只囊括维管植物的信息,其他与植物顺式作用元件同源的非植物模体也同时被收录。
并且所收录信息根据实验最新进展随时得到更新。
同时,PL AC E数据库中还包括了对每个模体的描述和在PubM ed中的相关文献编号,以及在DDB J/E MB L/GenB ank的核酸序列数据库的登录号,点击后可阅读相关文献摘要等信息。
登陆PL AC E数据库界面,用户可通过关键词、S RS关键词或者同源序列查询顺式作用元件的信息。
关键词可以是模体名称、涉及的诱导子或者植物激素、胁迫类型、该基因表达的组织或者器官、原始文献的作者、模体序列、植物种属等。
查询结果显示位点(模体)名称、位置、序列和PL ACE登录号,同时,也可以用F AS T A 格式批量上传序列信息。
安徽农业科学,J ou rnal o f Anh ui Ag ri.Sci.2007,35(24):7418-7419责任编辑孙红忠责任校对李洪2.3TRRD(T ranscription regulatory regions database)[3] TRR D数据库(http://w w w.bione t.nsc.ru/trrd/),即转录调控区数据库。
其数据来源于已发表的科学论文,包含特定基因各种结构与功能特性,包括转录因子结合位点、启动子、增强子、沉默子的位置以及基因表达调控模式等。
2001年的6.0版本综合了3898篇科学文献中的1167个基因,5537个转录因子结合位点,1714个调控区域,14个座位控制区和5335个表达模式。
在TR RD数据库中,所有信息被分列于5个相关的数据表中:TR RD GENES(包含所有TR RD库基因的基本信息和调控单元信息);TRRD SI TES(包括调控因子结合位点的具体信息);TRR DF AC TOR S(包括TRRD中与各个位点结合的调控因子的具体信息);TRR DEXP(包括对基因表达模式的具体描述);TR RDB IB(包括所有注释涉及的参考文献)。
TR2 RD的主页提供了对这几个数据表的检索服务。
除此之外,数据库还提供了另外2个工具:¹序列获得系统(S RS),用于搜索TRR D和与外部信息和软件资源进行整合;ºTRR D Vie w er,以基因图谱的形式提供相关信息的描述。
2.4TRANS FAC(T ranscriptional re gulation,from patte rns to profiles)[4]TR ANS F AC数据库(http://w ww.ge ne2regulation.c o m/或者http://transfac.gbf.de/TRA NS FAC/)是一个真核基因顺式调控元件和反式作用因子数据库,数据搜集的对象从酵母到人类。
TRA NS FAC数据库中的数据资源被分为6大类别:S I TE类数据是关于真核基因的不同调控位点信息,GENE 类数据描述具有多个调控位点的基因信息,FAC TOR类数据描述结合于这些位点的蛋白质因子信息,C EL L类数据则说明蛋白质因子的细胞来源,C LAS S类数据包含转录因子分类的基本信息,M A TR IX数据以矩阵的形式定量描述结合位点核苷酸的统计分布。
此外,还有几个与TRA NS FAC密切相关的扩展库:P A THODB库收集了转录区域中可能导致病态的突变数据;S/M AR T DB收集了蛋白质结合位点的特征信息及作用于这些位点的蛋白质信息;TR ANS P A T H库用于描述与转录因子调控相关的信号传递的网络;C YTO M ER库表现了人类转录因子在各个器官、细胞类型、生理系统和发育时期的表达状况。
3前景与展望对真核生物启动子进行计算机预测和鉴定是一项具有挑战性的研究工作。
到目前为止,尽管相关数据库和软件资源得到了很大的丰富和发展,但仍存在着明显不足,如:¹大多数数据库对于数据的创新、精确性和准确性没有权威评价,数据过多、重复,分类较粗等;º人类公共数据库中,只有极少数被实验证实的顺式作用元件,绝大多数基因的启动子仍然未知;»采用人类基因组信息来预测植物、真菌等远缘物种的基因结构时,数据准确性不高,但目前针对植物、真菌等的生物信息学数据库远没有人类的全面和完善;¼数据库中c D NA和ES T簇经常是不完整序列,特别是5c端,故无法确定转录起始位点的确切位置,从而影响启动子的预测;½真核生物的顺式作用元件比原核生物复杂,需要考虑多种因素[5]。
