用软件设计引物(Primer Premier & Oligo)
如何使用Primer Premier 5.0

生物秀-专心做生物w w w .b b i o o .c o m生物秀-专心做生物w w w .b b i o o .c o m生物秀-专心做生w w w .b b i o o .c o m生物秀-专心做生物w w .b b i o o .c o m物秀-专心做生物.b b i o o .c o m生物秀-专心做生物w w w .b b i o o .c o m生物秀-专心做生w w w .b b i o o .c o m生物秀-专心做生w w w .b b i o o .c o m生物秀-专心做生w w w .b b i o o .c o m生物秀-专心做生w w w .b b i o o .c o m生物秀-专心做生物w w w .b b i o o .c o m生物秀-专心做生物w w w .b b i o o .c o m生物秀-专心做生物w w w .b b i o o .c o m生物秀-专心做生w w w .b b i o o .c o m生物秀-专心做生w w w .b b i o o .c o m生物秀-专心做生w w w .b b i o o .c o m生物秀-专心做生物w w w .b b i o o .c o m生物秀-专心做生物w w w .b b i o o .c o m生物秀-专心做生物w w w .b b i o o .c o m生物秀-专心做生w w w .b b i o o .c o m生物秀-专心做w w w .b b i o o .c o m生物秀-专心w w w .b b i o o .c生物秀-专心做w w w .b b i o o .c o m生物秀-专心做w w w .b b i o o .c o m①代表这些序列与引物匹配的得分值小于40分②代表这些序列与引物匹配的得分值位于40~50分③代表这些序列与引物匹配的得分值位于50-80分,④代表这些序列与引物匹配的得分值位于80-120分生物秀-专心做生物w w .b b i o o .c o m秀-专心做生物b b i o o .c o m。
利用Primer Premier5.0来验证引物

利用Primer Premier5.0来验证引物
我们做实验,有时会用到别人已经用过的引物,但如何才能知道这个引物是否正确呢?可利用Primer Premier5.0来验证引物。
下面以常用内参GAPDH为例将验证过程介绍如下:
GAPDH的引物序列来自一片外文文献:
上游:AATGCATCCTGCACCACCAA
下游:GTAGCCATATTCATTGTCATA
所得片断为516bps。
首先在NCBI中查到GAPDH的mRNA序列。
然后打开Primer Premier5.0,点File→New→DNA Sequence,将上面的mRNA 序列复制到NewSequence 框内,注意选择as is。
先加上游引物:点Primer→点左上角s(sense)→点Edit Primers, 将上游引物复制到编辑框(用Ctrl+V快捷键),注意选择as is→点Analyze→点Prime,即可见输入的上游引物与模板匹配上了→点Ok。
再加下游引物:点左上角A(antisense)→点Edit Primers, 将下游引物复制到编辑框(用Ctrl+V快捷键),注意选择reversed→点Analyze→点Prime,即可见输入的下游引物与模板匹配上了→点Ok。
此时即可看到上游和下游引物的相关参数。
注:在Primer Premier5.0中,上游引物的显示方向为5'→3',下游引物为3'→5',均为从左往右看。
《2024年利用软件PrimerPremier5.0进行PCR引物设计的研究》范文

《利用软件PrimerPremier5.0进行PCR引物设计的研究》篇一一、引言随着分子生物学技术的飞速发展,聚合酶链式反应(PCR)已成为实验室研究中不可或缺的技术之一。
PCR引物设计是PCR实验成功的关键步骤之一,其准确性直接影响到PCR的扩增效率和特异性。
因此,选择合适的引物设计软件对于PCR实验的成功至关重要。
本文将介绍如何利用PrimerPremier5.0软件进行PCR引物设计的研究。
二、PrimerPremier5.0软件简介PrimerPremier5.0是一款功能强大的引物设计软件,具有直观的用户界面和丰富的功能,可帮助研究人员快速设计高质量的PCR引物。
该软件支持多种PCR类型,包括常规PCR、实时荧光定量PCR等,可满足不同实验需求。
三、PCR引物设计流程1. 输入目标序列:打开PrimerPremier5.0软件,输入需要设计引物的目标序列。
目标序列可以是基因片段、cDNA等。
2. 设置引物参数:根据实验需求,设置引物的长度、GC含量、退火温度等参数。
PrimerPremier5.0软件会根据这些参数,自动筛选出符合要求的引物序列。
3. 设计引物:在软件中,通过设置不同的引物组合,生成多个潜在的引物序列。
这些引物序列的特异性和扩增效率可通过软件进行评估和预测。
4. 筛选引物:根据实验需求和引物评估结果,选择最合适的引物序列。
在选择过程中,需要考虑引物的特异性、扩增效率、引物间及引物与模板间的互补性等因素。
5. 验证引物:将设计的引物序列导入PCR实验中,进行验证。
通过PCR实验结果,评估引物的特异性和扩增效率。
四、PrimerPremier5.0软件的优势1. 直观的用户界面:PrimerPremier5.0软件具有直观的用户界面,使得用户可以轻松地完成引物设计过程。
2. 丰富的功能:该软件支持多种PCR类型,可满足不同实验需求。
同时,该软件还具有引物评估和预测功能,可帮助用户选择最合适的引物序列。
引物设计软件使用

一、引物设计step by step1、在NCBI上搜索到目的基因,找到该基因的mRNA,在CDS选项中,找到编码区所在位置,在下面的origin中,Copy该编码序列作为软件查询序列的候选对象。
2、用Primer Premier5搜索引物①打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。
点击Primer,进入引物窗口。
②此窗口可以链接到“引物搜索”、“引物编辑”以及“搜索结果”选项,点击Search按钮,进入引物搜索框,选择“PCR primers”,“Pairs”,设定搜索区域和引物长度和产物长度。
在Search Parameters里面,可以设定相应参数。
一般若无特殊需要,参数选择默认即可,但产物长度可以适当变化,因为100~200b p的产物电泳跑得较散,所以可以选择300~500bp.③点击OK,软件即开始自动搜索引物,搜索完成后,会自动跳出结果窗口,搜索结果默认按照评分(Rating)排序,点击其中任一个搜索结果,可以在“引物窗口”中,显示出该引物的综合情况,包括上游引物和下游引物的序列和位置,引物的各种信息等。
④对于引物的序列,可以简单查看一下,避免出现下列情况:3’不要出现连续的3个碱基相连的情况,比如GGG或CCC,否则容易引起错配。
此窗口中需要着重查看的包括:T m应该在55~70度之间,GC%应该在45%~55%间,上游引物和下游引物的T m值最好不要相差太多,大概在2度以下较好。
该窗口的最下面列出了两条引物的二级结构信息,包括,发卡,二聚体,引物间交叉二聚体和错误引发位置。
若按钮显示为红色,表示存在该二级结构,点击该红色按钮,即可看到相应二级结构位置图示。
最理想的引物,应该都不存在这些二级结构,即这几个按钮都显示为“None”为好。
但有时很难找到各个条件都满足的引物,所以要求可以适当放宽,比如引物存在错配的话,可以就具体情况考察该错配的效率如何,是否会明显影响产物。
primer premier5引物设计步骤

primer premier5引物设计步骤
引物是在PCR反应中用于扩增目标DNA片段的短链寡核苷酸序列。
Primer Premier 5是一款常用的引物设计软件,以下是引物设计的一般步骤:
1. 目标序列准备:将目标DNA序列输入到Primer Premier 5软件中。
确保序列准确无误,并确定要扩增的目标区域。
2. 引物设计参数设置:根据实验需求和PCR反应条件,设置引物设计的相关参数。
包括引物长度、引物的GC含量、引物间的碱基对数目等。
3. 引物设计策略选择:根据需求选择合适的引物设计策略,如末端限制酶切位点、避免引物间的二聚体形成等。
4. 引物设计和评估:软件将自动生成多个候选引物序列,并根据一定的评估标准对其进行评估,包括引物的互补性、引物之间的二聚体形成、GC含量等。
5. 引物的选择和优化:根据评估结果选择一个或多个最合适的引物序列。
根据需要,可以对引物进行进一步优化,如修改引物长度、GC含量、碱基组成等。
6. 引物合成和实验验证:根据设计的引物序列进行引物合成,并进行PCR 实验验证引物的扩增效果和特异性。
引物设计是PCR实验中至关重要的步骤,引物的质量和合适性直接影响PCR 反应的效果。
Primer Premier 5软件提供了便捷的引物设计工具和功能,可以帮助研究人员高效地设计和评估引物序列。
设计引物的实验报告

一、实验目的1. 掌握引物设计的原理和方法。
2. 学习利用生物信息学工具进行引物设计。
3. 了解引物在PCR实验中的应用。
二、实验原理引物是一段单链DNA或RNA,作为PCR反应的起始模板,与模板DNA链互补结合,从而在PCR反应中引导DNA的复制。
引物设计是PCR实验成功的关键因素之一。
三、实验材料1. 生物信息学工具:Primer Premier 5.0、Primer BLAST、OligoCalc等。
2. 实验样品:待扩增的DNA模板。
3. 其他:PCR试剂、DNA序列、引物合成等。
四、实验步骤1. 选择目标基因序列根据实验目的,选择合适的基因序列。
在本实验中,以某基因的cDNA序列为模板。
2. 利用生物信息学工具进行引物设计(1)打开Primer Premier 5.0软件,输入基因序列。
(2)设置引物设计参数,如:引物长度、Tm值、GC含量、引物间距离等。
(3)进行引物设计,得到多个引物序列。
(4)利用Primer BLAST和OligoCalc等工具对设计出的引物进行筛选,排除同源序列和二级结构。
3. 引物合成将筛选出的引物序列提交给引物合成公司,合成引物。
4. PCR实验(1)配制PCR反应体系,包括:引物、模板DNA、dNTPs、DNA聚合酶等。
(2)设置PCR反应程序,如:预变性、变性、退火、延伸等。
(3)进行PCR反应,观察扩增结果。
五、实验结果与分析1. 引物设计结果根据实验目的,设计出以下引物:上游引物:5'-ATCGTACGCTAGGCTG-3'下游引物:5'-CGTCTGACGACGTCAGT-3'2. PCR扩增结果通过PCR实验,成功扩增出目标基因片段。
六、实验结论1. 通过生物信息学工具进行引物设计,可提高引物设计的准确性和效率。
2. 合适的引物是PCR实验成功的关键,设计引物时需考虑多种因素。
3. 本实验成功设计并合成引物,为后续的PCR实验奠定了基础。
利用软件PrimerPremier5.0进行PCR引物设计的研究

利用软件PrimerPremier5.0进行PCR引物设计的研究利用软件PrimerPremier5.0进行PCR引物设计的研究引言:PCR (Polymerase Chain Reaction) 是一种广泛应用于分子生物学和基因工程领域的技术,通过引物(primers)的选择和设计,能够扩增特定的DNA片段。
PCR引物设计的质量和合理性对PCR扩增的成功与否起着至关重要的作用。
PrimerPremier5.0 是一款被广泛使用的引物设计软件,具有许多功能,可以辅助研究人员选择高质量的引物。
本文旨在研究和探讨利用PrimerPremier5.0软件进行PCR引物设计的方法和优势。
方法:1. 引物设计目标:在PCR实验中,为了获得准确和高效的扩增结果,引物设计时需要考虑以下几个因素:- 引物长度:通常情况下,引物的长度应在18到30个碱基对之间。
- GC含量:GC含量应尽量控制在40%到60%之间,过高或过低都会影响引物的特异性和扩增效率。
- 引物间的互补性:引物之间应避免互补性或三联体结构的形成,以避免非特异性扩增。
- 引物的熔解温度(Tm):引物的Tm值应接近,以确保特异性扩增。
2. PrimerPremier5.0软件的使用:- 安装并启动PrimerPremier5.0软件。
- 导入所需扩增区域的目标序列。
- 在软件中设置引物的参数,如长度、GC含量和Tm值等。
- 执行引物设计程序,软件会根据设置的参数,自动在目标序列中搜索合适的引物。
结果与讨论:通过PrimerPremier5.0软件的使用,可以高效地选择到符合设计要求的引物。
软件会根据设定的参数,自动搜索并筛选出多个候选引物。
1. 引物长度:为了确保引物能够与模板DNA进行特异性结合,通常将引物长度设定在18到30个碱基对之间。
经过软件筛选后,可得到一组长度适当的引物。
2. GC含量:合适的GC含量可以提高引物的稳定性和特异性。
软件会自动根据设定的GC含量范围,筛选出具有适当GC含量的引物。
引物设计及Primer Premier 使用介绍

Sequence name
Choose a function
Use these two button to translate the DNA seq to a protein seq or a
protein seq to a DAN seq 8种密码子偏好
引物设计界面 First you can design the primer manually
引物设计的原则
3、引物Tm
引物所对应模板序列的Tm 值最好72℃ 左右。至少要在55-80℃之间。 Tm=2(A+T)+4(C+G)
引物设计的原则
4、引物3’端
• 引物3’末端和模板的碱基完全配对 • 防止一对引物内的同源性 • 考虑末端5个碱基的ΔG • 引物3′端要避开密码子的第3位
引物设计的原则
121 ggggctccta gagaacccgg gggcgcttga ccgcgcgcgg gcggcccgcg ggtcgtacat 181 cgcgaggtcg tcgcactcgc gcaacccaga gccaggcccg ctgtgcccgg agctcatgag 241 caccatgcac ctgctgacat tcgccctgct tttttcctgc tccttcgcc c gcgcggatcc 301 cgaccccaag atcgtcaaca tcggcgcggt gctgagcacg cgcaagcatg aacagatgtt 361 ccgcgaggca gtaaaccagg ccaataagcg acacggctct tggaagatac agctcaacgc 421 cacttctgtc acccacaagc ccaacgccat acagatggcc ctgtcagtgt gtgaggacct 481 catctctagc caggtctacg ctatcctagt tagccacccg cctactccca acgaccactt 541 cactcccacc cctgtctcct acacagctgg cttctacaga atccctgtcc tgggactgac 601 tacccgaatg tccatctact ctgacaagag tatccacctg agtttccttc gcacggtgcc 661 gccctactcc caccagtcca gcgtctggtt tgagatgatg cgagtctaca actggaacca 721 catcatcctg ctggtcagcg acgaccacga gggacgggca gcgcagaagc gcttggagac 781 gttgctggag gaacgggagt ccaaggcaga gaaggtgctg cagtttgacc caggaaccaa
Primer Premier使用手册中文版--教你如何使用软件设计引物

PrimerPREMIERVersion 5.0 for Windows and PowerMacintosh使用说明书PREMIERBiosoft International3786 Corina way,Palo Alto, CA 94303-4504电话650 856-2703传真650 843-1250电子邮件************************目录目录 (2)版权声明 (5)简介 (6)新版更新 (6)基础指南 (6)GENETANK 序列编辑 (7)GeneTank Window 序列编辑窗口 (7)查看序列 (7)打开已有序列 (7)创建新序列 (7)保存序列 (8)编辑序列 (8)查找 (8)查看不同链 (8)语音校读 (8)编辑序列比较结果 (9)序列粘贴窗口 (9)参数设置 (9)Rating Parameters 评分参数 (10)序列比较 (10)Alignment Parameters 序列比较参数设置 (10)序列翻译 (12)Edit Codon Table 窗口 (12)PRIMER 引物设计 (14)Primer Premier 引物设计窗口 (14)Direct Select 即点即选框 (14)在序列比较中应用即点即选功能 (14)性状列表 (15)二级结构. . . . . . .. . . . .. . . . . .. . .. . . . . . . .. . . . . . . . .. . . . . . . . . . .. . . . . .. . .. .. . . . . .. . . . . (15)Edit Primer 引物编辑窗口 (16)Search Criteria 搜索标准窗口 (16)Search Parameter 搜索参数窗口 (17)Automatic Search 自动搜索 (17)Manual Search 手动搜索及引物设计原理 (18)Search Results 搜索结果窗口 (20)Multiplex/Nested Primer 复式及巢式PCR反应引物窗口 (21)Database 引物数据库窗口 (21)Reports 分析报告 (22)Synthesis Order Form 合成订单 (22)Optical Activity 比吸光度 (22)Degeneracy 多义性 (23)Graphs 图表 (23)Restriction Enzyme Analysis 限制性内切酶分析 (24)Restriction Enzyme Analysis 限制性内切酶分析窗口 (24)Restriction Sites 酶切位点窗口 (24)Edit Enzyme 限制内切酶编辑窗口 (24)Filter 特征筛选窗口 (25)Motif Analysis 基序分析 (26)Motif Analysis 基序分析窗口 (26)Motif Sites 基序位点窗口 (26)Edit Motif 基序编辑窗口 (26)Menu Commands 菜单命令 (27)GeneTank 主菜单 (27)File (27)Edit (27)View (27)Search (27)Function (27)Translate (28)Window (28)Help (28)Primer 主菜单 (28)File.....................................................................................................Edit .................................………………........................................... Function.......................…………………..................................................... Report.................……………................................................................................ Graph..............………….................................................................................. Window...........……………….....................................................................................Help ...........………….......................................................................................Enzyme Results Menu 主菜单..........………………..................................... File...........................…………........................................................................... Function..............……………................................................................................ Window.........................…………….....................................................................Help ............................…………........................................................................Motif Menu 主菜单.................................................................................. File...........................……………........................................................................... Function..................………………......................................................................... Window.....................……………….........................................................................Help ........................................………………..........................................................附录 (30)附录A 公式法则…………….…………………………………………..........附录B 多义碱基表.………………..…………………………......................附录C 氨基酸缩写代码……….….………………………………..................附录D 软件上限...…………….…………………………….......................附录E 参考文献 (36)附录F 系统要求 (37)务与技术支持.............…………………………….............................................他信息...................…………………….....................................................者后记...................……………………….....................................................版权声明Copyright © 2000 by PREMIER Biosoft International. All rights reserved.Information in this manual may change without notice and does not represent a commitment on the part of PREMIER Biosoft International.The software described in this manual is provided by PREMIER Biosoft International under a License Agreement. The software may be used only in accordance with the terms of the agreement.PREMIER Biosoft International "Premier" claims copyright to this program and documentation as an unpublished work. Claim of copyright does not imply waiver of Premier's other rights.This program and documentation are confidential and the property of Premier. Use, examination, reproduction, copying, decompilation, transfer, and/or disclosure to others are strictly prohibited except by express written agreement with Premier.PREMIER Biosoft International 简称Premier 版权所有本说明书中的内容可以由Premier单方面更改而无需另行通知本说明中涉及的软件由Premier提供并应有书面授权软件的使用必须在授权范围之内该软件及相关文档作为一项未公开发表的工作其版权归Premier所有对版权的声明并非意味放弃其他权利该软件及相关文档是Premier的机密与财产除非有Premier的书面授权任何的使用检查再生产复制分割传递或/和解密行为均被禁止以上版权声明仅是原英文声明的翻译如有歧义应以原英文声明为准译者声明本软件说明书著作权归PREMIER Biosoft International 所有译者则保留中文翻译的相关版权并授权生物软件网 共享发布该说明书的中文翻译本中文翻译仅限于软件使用者参考其内容如有歧义应以原英文说明书为准译者及生物软件网不承担由翻译打印网页等错误带来的直接和间接损失译者Wenkelly电子邮件*******************简介PRIMER PREMIER 是一种用来帮助研究人员设计最适合引物的应用软件利用它的高级引物搜索引物数据库巢式引物设计引物编辑和分析等功能可以设计出有高效扩增能力的理想引物也可以设计出用于扩增长达50kb以上的PCR产物的引物序列该软件主要由一下四个主要功能板块组成GeneTank 序列编辑Primer 引物设计Align 序列比较Enzyme 酶切分析Motif 基序分析新版更新种间交叉引物设计利用来自多个物种的序列设计扩增引物病理检测引物设计可用来在高保守区域设计引物等位基因特效引物设计专用于扩增某一类相关序列中特定成员的引物基础指南这段指南并非详细的说明仅为使用者对PRIMER PREMIER的菜单选项有一个直观的了解四个功能板块都有各自的窗口和菜单正在使用中的窗口就是您所见到的窗口以下列出了各功能板块的主要菜单选项GeneTank File, Edit, View, Search, Function, Translate, Window,Help.Primer File, Edit, Function, Report, Graph, Window, Help.Enzyme File, Function, Window, Help.Motif File, Function, Window, Help.使用功能菜单时该菜单所属的窗口就会被激活利用各菜单中的window选项记录的所有打开的窗口可以方便的在各个窗口之间进行切换所有的列表都有复选功能对于windows或Power Mac系统而言在点击同时按住CTRL键即可将当前的选择加入选单按住SHIFT键同时点击可以选择两次点击之间范围内的所有选项GeneTank利用GeneTank您可以打开DNA或者蛋白质序列也可以选择不同的阅读框架共三种将DNA翻译成蛋白质或利用蛋白序列反推出DNA序列为方便使用软件已经提供了普通密码子列表和几种线粒体密码子列表您也可以输入您自己的其他线粒体密码子列表序列编辑器提供了包括多媒体语言校读在内的完整的序列编辑功能这一功能板块包括以下部分GeneTank 打开浏览编辑及翻译序列Translation 将打开的序列翻译或反推成其他序列Edit Codon Table 编辑翻译所依据的密码子列表GeneTank窗口GeneTank的窗口用来显示序列及其文件题头信息序列可以3碱基10碱基一组显示您可以将打开的序列翻译或反推成其他序列您也可以使用通常的拷贝剪切和粘贴命令编辑当前序列GeneTank的窗口上也有启用Primer Align Enzyme Motif功能板块的快捷键查看序列点击相应按钮或从View菜单中选择您可以查看文件题头改变5 末端序列起始位置选择3碱基或10碱基一组的显示方式文件题头是指存在于序列文件中位于序列信息上游的文字内容点击header按钮或从View菜单中选择可以显示文件题头信息改变5 末端序列起始位置默认为1 序列将从您指定的5 位置开始显示序列可选操作View >Show HeaderView >GroupingView > 5’ Seq No 某些版本没有这一项打开已有序列使用菜单File >Open 可以激活文件选择对话框用来打开您希望处理的包含特定序列的文件如果序列文件是GenBank的标准格式PRIMER PREMIER会自动识别打开文件中的序列起始位置或是您曾经使用本软件菜单中File > Save命令以PRIMER PREMIER默认格式保存过该文件序列将自动打开如果打开其他格式的序列文件将出现一个可卷动的Import Sequence 窗口并显示全部序列在序列第一行的起始处双击则双击处以前的所有信息将被认为是文件题头然后PRIMER PREMIER会自动剔除非序列字符并将序列在GeneTank里打开创建新序列该项功能可以让您手动键入一个新的序列您可以键入任何DNA或蛋白序列并存在您指定的文件夹内并可以象其他如GenBank格式的文件一样进行操作Windows用户请注意默认设置的DNA序列文件后缀为SEQ 蛋白序列文件后缀为PRO 当然您可以使用其它的任何序列文件类型使用提醒如果您选错文件发现并没有您所希望的序列只需要关闭Import Sequence 窗口重来一次即可保存序列使用本软件保存的序列可以被软件自动识别而无需让您指出序列起始位置建议您在诸如以下情况保存序列您需要经常使用同样的序列您已经编辑完一段序列并希望保存结果您已经输入一段新序列编辑序列只需在GeneTank窗口内您就可以方便的编辑DNA或蛋白质序列而编辑翻译或原始序列十分方便在任一序列上的改变将会正确的反应到与之相关的其他序列例如如果你在一段蛋白序列上无论该序列是被翻译的或是用来反推DNA的原始序列删除一个氨基酸残基在相应DNA上的对应三位密码子将同时被删除但如果您改变一段翻译出的序列这一改变将仅仅在另一段由此翻译出的序列而不会出现在原始序列上编辑序列可以使用直接键入或利用剪贴板通常的剪切拷贝粘贴同样可以方便的在此使用其快捷键分别是CTRL-X, CTRL-C和CTRL-V软件提供三碱基一组的显示方式以利于蛋白相关的工作在键入DNA序列时可以使用A C G T或者附录B所列出的多义碱基在键入蛋白序列时可以使用附录C中所列出的氨基酸单字母记号可以从任何文本编辑器如记事本中的剪贴板中拷贝一段序列并将其粘贴进来当您完成编辑后请注意保存序列查找利用GeneTank窗口上的Find 按钮您可以从指针起始位置开始查找序列中一段特定字符查找到第一个后您也可以使用Find Next 按钮在余下的序列中继续查找使用Search 菜单选择Go To Position可以将指针移到您指定的位点可选操作某些版本在Edit菜单下Search >FindSearch >Find NextSearch >Go To Position查看不同链要查看该序列的互补链您只需要点击GeneTank窗口上的A按钮就会显示该序列的互补链此时互补而点击S按钮就能恢复成原来的序列您亦可点击dsDNA按钮以双链形式显示可选操作View > Sense StrandView > Anti-sense StrandView > ds DNA语音校读在GeneTank打开一段序列后您可以按下语言按钮启用语音校读功能序列将从指针所在的位置开始被依次朗读序列再次按下语音按钮将停止朗读按下键入朗读键将在键入的同时朗读输入的序列再次按下该按钮可以停止键入朗读可选操作Function > Speaker编辑序列比较结果当一项多序列比较完成后比较结果可以通过编辑来获得更准确的配对通常ClustalW法则能够给出一个准确的比较结果因而手工编辑并非必要但对于个别情况或出于特殊要求可能有这种需要所以本软件也提供这一功能将点击指针指向的碱基可以实施改动按下空格键可以插入一个空隙按下Delete键则可以删除一个空隙一旦比较结果被改动最大和最小同源性参数也会自动改变粘贴序列窗口这一窗口使得一段核酸序列通过选择以四种不同的方式粘贴上去9分别为正向As is 反向reversed 互补complemented 以及反向互补reverse complemented 这样便于从其他程序中粘贴序列而无需考虑其序列保存的方向参数设置Preferences 参数设置窗口可以改变本软件所使用的多种默认参数也可以允许您查看及更改引物评分参数设定二级结构在引物评分中所占的权重系数默认引物长度默认值为25默认引物长度在Primer Premier窗口使用即点即选功能时得到的引物长度该项参数也是Primer Premier窗口启动时的初始默认值这项数值可以设定为1到70个碱基引物对搜索最大值默认值为100在自动搜索引物时得到的结果数量可以很多由于该软件按照引物评分来衡量引物效率有些引物虽然合乎标准但评分较低可能没有全部列出的必要尽管我们设置了100对引物这一较大的数值您仍然可以改变该项参数为10到1000之间的任意值核酸浓度默认值为250 pM根据文献7 Freier等提供的计算公式这是没有自身配对时退火的寡聚核苷酸的总浓度它即不是引物浓度也不是起始模板浓度而是PCR产物的浓度由于在PCR过程模板的浓度变化很大很难确定具体数值根据文献11的建议Rychlik等一般经验公式在普通PCR 反应中将其当作250 pM 这项数值被用来计算引物的退火温度10单价离子浓度默认值为50 mM这项数值是指反应混合液中所有单价离子的总浓度游离Mg2+离子浓度默认值为1.5 mM这项数值是指反应混合液中充当辅基的Mg2+离子浓度总Na+当量这项数值是根据单价离子浓度和游离Mg2+离子浓度计算得来计算公式如下Sprt即取平方根总Na+当量单价离子浓度4 Sqrt 游离Mg2+离子浓度1000这项数值用来计算引物的退火温度自由能计算设置温度默认值为25 °C这项数值用来计算自由能G 自由能计算公式为G= H T S.评分参数Rating parameters 评分参数窗口是用来设定二级结构在引物评分中的权重系数二级结构越稳定引物评分就会越低相对于在引物中间形成的二级结构来说3 末端的二级结构对PCR扩增的影响会更大为了将这一效应计算在内软件将3 末端的二级结构的自由能数值G减去1 这一修正会使3 末端的二级结构显得更加稳定这一修正值是根据未公开实验结果制定的您可以根据自己的需要来更改软件只标注是引物二级结构中最稳定的一个自由能数值G 如果没有检测到二级结构则该数值为0序列比较这一部分仅在Primer Premier的Windows版本中使用对于Macintosh系统序列比较是提交到网上完成并下载比较结果在Primer的教程中有关于这方面的完整信息Align 序列比较窗口让您将已经打开的序列进行比较序列选择框显示所有将要比较的序列使用Add 和Delete 按钮可以增加一个序列或移除一个序列使用Options 按钮可以打开序列比较的参数设置窗口序列比较参数设置您可以利用Alignment Parameters 序列比较参数设置窗口来修正ClustalW法则用来优化比较结果的各项参数序列比较可以用两种模式完成一种准确但较慢slow/accurate一种快速但较粗略fast/approximate 选择不同的模式将会得到不同的结果SLOW/ACCURATE模式的序列比较参数这些参数无法改变序列比较的运行速度它们被用来对序列比较评分然后换算为百分比数值即最后显示的百分数并转化为进化树上的进化距离1 Gap Open Penalty 比较结果中一个空隙的罚分2 Gap extension penalty 比较结果中一个空隙每延长一碱基的罚分3 Protein weight matrix 这是描述氨基酸之间的相似性的评分标准4 DNA weight matrix 为核酸配对制定的评分标准包括IUB多义密码子11FAST/APPROXIMATE模式的序列比较参数这种同源性评分是由一种快速粗略的通用比较方法计算得来的它包括4项参数有两种方法用以快速获得序列比较结果1 只计算准确配对的部分k-tuples2 只计算最佳配对的情况K-TUPLE SIZE 这是默认的准确配对的片段长度增加这一数值可以提高运行速度对于蛋白序列最大值为2 对于DNA最大值为4 减少这一数值则可以增加灵敏度对于较长的序列例如大于1000字符您可能需要增加默认值GAP PENALTY 这是快速比较种每个空隙的罚分若非设定成很大的值对比较的速度影响不大TOP DIAGONALS 在一个虚拟的点划分分析法中需要计算的在每个对角线上使用的ktuple 配对数量在比较中只有最佳配对的情况才会被使用由这项参数指定其数量增加这一数值可以提高运行速度减少这一数值则可以增加灵敏度多序列比较参数这些参数控制最终的多序列比较每个多序列比较包括多个双序列比较的过程按照预定顺序依次进行相应的基本控制参数有两种空隙罚分gap penalty 以及位置同源或替代的权重系数scores for various identical/nonidentical residues1 和2 GAP PENALTY由菜单选项1和2控制它们设定空隙以及空隙长度造成的罚分增加空隙罚分可以减少比较结果中空隙的数量增加空隙长度的罚分可以使空隙更短末端的空隙不会被罚分3 DELAY DIVERGENT SEQUENCES 打开这一选项将会先比较同源性高的序列后比较同源性较低的序列该项数值即将特定同源性百分数设定为阀值低于该同源性的序列将会推迟比较4 The TRANSITION WEIGHT 为碱基转换A< >G或C < >T即嘌呤之间和嘧啶之间的替换设定从0到1的权重系数系数0意味将碱基转换看作一个完全的错配系数1意味将其看作一个完全的配对对于远源序列建议将其设置为0 而对于同源性较高的序列将这一系数适当增加会有好处5 PROTEIN WEIGHT MATRIX 该选项可以打开一个让你选择权重矩阵weightmatrices 的菜单对于蛋白质分析默认的矩阵是由Jorja和Steven Henikoff 创建的BLOSUM 系列矩阵请注意是使用一系列矩阵因为到底使用哪一个矩阵取决于序列之间的同源程度进化差异不同所使用的矩阵也不同6 DNA WEIGHT MATRIX 该选项打开一个菜单选择一个而不像蛋白分析那样是一系列核酸权重矩阵默认的一个是BESTFIT用来比较核酸序列的矩阵7 在权重矩阵中如果您愿意可以使用正值也可以使用负值如果不使用NEGATIVE MATRIX 负值权重矩阵选项系统将默认使用正值8 PROTEIN GAP PARAMETERS 蛋白序列空隙参数选项可以打开一个菜单让您可以设置蛋白序列比较结果的空隙罚分参数该选项仅在蛋白序列比较中可用更多信息如果您想得到关于设置参数和ClustalW法则的更多信息请从以下网址获取由原作者提供的完整文件www-igbmc.u-strasbg.fr/BioInfo/ClustalW/clustalw.html序列翻译激活序列在GeneTank窗口的一个列表框内显示了原始序列以及由它翻译出的其他序列12产生翻译序列的机制如下Translated DNA 从原始的蛋白序列推导DNA序列或由原始DNA翻译出蛋白序列再由蛋白序列反推出的DNA序列Translated Protein 从原始DNA翻译而成蛋白序列或原始蛋白序列翻译出DNA序列再由DNA序列反推出蛋白序列推导DNA按钮当前序列为原始或推导DNA时该按钮不可用当前序列为蛋白序列时点击该按钮将会激活一个Codon Table Selection窗口您可以选择密码子列表和阅读框架翻译结果中的多义碱基请参照说明书的附录可选操作Translate > To DNA翻译蛋白质按钮当前序列为原始或推导蛋白序列时该按钮不可用当前序列为DNA序列时点击该按钮同样将会激活Codon Table Selection窗口您可以选择密码子列表和阅读框架如果原序列中存在的多义碱基使密码子无法翻译成为蛋白残基该残基位点会用*号代替可选操作Translate > To ProteinEdit Codon Table窗口使用这一窗口您可以创造新的密码子列表删除或编辑已有的密码子列表但PRIMER PREMIER 提供的标准密码子列表是不能编辑的使用ADD 按钮创建新的密码子列表会使用标准列表并初始命名为<New Organism 某一数字> 使用Edit Codon Table Name对注意如果您多次翻译一段序列只有最后一次的翻译结果才能被保留而以前的翻译结果将被忽略13话框可以编辑列表名称使用DELETE 按钮则删除当前列表要改变某一密码子对应的氨基酸只需要用鼠标将其拖放到相应位置就可以点击密码子时鼠标指针会发生变化提示您正对其操作任何拖放操作会改变两行内容一行即某种氨基酸得到一个密码子的同时另一行失去这个密码子14Primer 引物设计Primer功能板块包括了设计引物的搜索引擎软件包含了强大的自动搜索法则只需要简单的操作就可以得到合适的引物PRIMER PREMIER也提供了人工控制搜索引擎的方法便于根据您的特殊要求制定标准输出的引物通过全面的即时分析工具计算出引物的多种参数和二级结构关键参数如T m值GC含量和自由能G还能以图形显示还有另一个窗口来显示引物的比吸光度activity 单位nmoles/OD和ug/OD 及引物的分子量还可以将当前引物输入数据库打印或填写引物合成定购单为设计用于定点突变的引物该项功能板块也提供包括即使分析和限制性酶切位点分析的引物编辑工具您可以通过输入碱基或氨基酸残基来手动修改引物为分析多对反应引物如在复式及巢式PCR中使用function菜单下Multiplex/Nested选项您可以分析所有想用到的引物可以选择事先搜索的引物或手动添加引物软件提供将引物储存到数据库的功能也提供填写引物合成定购单的功能定购单上会自动填入引物序列和其他重要信息该项功能板块由以下部分组成Preferences 参数设置窗口Primer Premier 引物设计窗口Edit Primer 引物编辑窗口Search Criteria 搜索标准窗口Search Parameter 搜索参数窗口Search Results 搜索结果窗口Multiplex/Nested Primer 复式及巢式PCR引物设计窗口Database 引物数据库窗口Synthesis Order Form 生成引物合成订单Reports 结果报告Graphs 图表Primer Premier 引物设计窗口Primer Premier 引物设计窗口是分析引物的关键该窗口包括以下功能即点即选Direct Select 引物性状列表和二级结构显示具体介绍如下Direct Select 即点即选框当您将鼠标指针移至该框中指针将变为铅笔型点击框中任何一处会产生一条起始位置最接近点选位置并与原序列互补的引物所有引物性状将即时更新引物5 和3 末端也会标记上以区分引物的扩增方向如果点选一组序列比较结果引物序列将依据保守序列来确定这使得您很容易知道引物与某一特定序列有哪些错配位点在序列比较中应用即点即选功能在利用高保守序列区域设计引物时Primer Premier会在引物窗口显示序列比较这样您很容易知道引物与每一特定序列有哪些错配位点点击序列任一地方将根据保守序列产生一条引物并加以分析引物也可以被编辑以便同某一特定序列匹配这些可以用来设计针对等位基因的引物通过View 菜单选项可以切换显示多数倾向和少数分歧碱基Majority or Minority Consensus 切换到显示少数分歧碱基可以看出哪些核苷酸不能很好的与所有序列匹配性状列表这一列表显示了当前引物对的各种性状您可以通过以下三种方式选择一对引物作为当前引物对Direct Select框根据上述使用Direct Select的方法选择一条引物切换到另一条互补链选择反向引物得到一对引物对在Search Results 搜索结果窗口里选择一对引物对使用输出命令在数据库窗口里选择一对引物对.性状列表能自动更新正反引物的长度起始位置融链温度T m GC含量GC% 多义性比吸光度Optical Activity 单位ug/OD 对于引物对来说长度条显示了PCR产物的长度而T a Opt项显示了PCR反应适宜的退火温度各种引物性状计算公式请参阅附录A二级结构PRIMER PREMIER能即时分析引物二级结构在左下的两排按钮可以想您显示发现了哪些二级结构而按下的按钮表明相邻的图框里显示的是何种二级结构除了二级结构图框里也显示二级结构的自由能数值G 单位kcal/mol在PRIMER PREMIER窗口内的图框里显示的是最稳定的二级结构连续碱基配对用实线显示而不连续碱基配对用虚线显示按下按钮All 就会给出当前二级结构的报告您选择正向引物的发卡结构并点击All 就会显示以下内容注意如果您想使用上述方法选择一条例如如正向引物而不是一对引物您需要手动回复原来的反向引物来和新引物配对注意如果有至少连续三个碱基配对就会被认为能形成发卡二聚体或错配之类的二级结构15可能出现的二级结构按照稳定性大小自动排序最稳定的一种排在最前使用卷动条可以看到未显示的部分除了上述功能这一板块还具有激活以下功能的快捷按钮Search Criteria 搜索参数窗口Search Results 搜索结果如果已经实施过搜索窗口以及Edit Primer 引物编辑窗口点击按钮可以使相应部分伴随引物对以图形显示16Edit Primer 引物编辑窗口Edit Primer 引物编辑窗口可以用来设计用于定点突变的引物或分析一条已有引物序列在windows系统或Power Mac的Command-X Command-C 及Command-V系统中您可以使用CTRL-X CTRL-C和CTRL-V快捷键来实施剪切拷贝和粘贴删除当前引物序列并从剪贴板上粘贴是分析一条已有引物的好方法进行粘贴时Paste 粘贴窗口会激活用以将引物序列转化为反向互补或反向互补形式您也可以手工键入引物序列一旦引物被编辑发生变化Analyze 按钮就可以使用点击Analyze 按钮即可分析编辑后的引物您可以修改实际序列或是翻译后的氨基酸残基如果您修改氨基酸残基相应位置可能出现多义密码子除了上述的引物性状和二级结构资料具体参阅PRIMER PREMIER窗口的章节该窗口也提供了限制性酶分析功能您可以通过点击Enzyme 按钮通过手动或软件提供的筛选方案来选择一组限制性酶您可以使用Prime 按钮来将引物移动到更合适的结合位点通常这项功能可以让您确认是否有其它比当前位点更稳定更适合的结合位点存在其他信息如果您希望改变阅读框或使用其它密码子列表请关闭本窗口从主菜单上选择Function >GeneTank激活GeneTank窗口再选择Translate > Change Parameters打开Select Codon Table窗口来改变阅读框或选择其它密码子列表然后重新打开本窗口可选操作Function > Edit PrimerSearch Criteria 搜索标准窗口以下是基本搜索标准的详细信息根据PCR引物测序引物或杂交探针的不同要求选择不同的搜索标准是很有必要的。
Primer premier 5.0设计pcr引物

1.安装软件Primer premier5.0。
程序下载:Primer Premier 5.0 /Soft/2005/114.htm 2.程序中双击打开3.点FILE---NEW—DNA SEQUENCE如图所示。
4.输入目的基因片段,可以复制后用ctrl+V键拷贝到栏内,后应加数个N以备后续设计时加酶切位点及保护碱基,如图所示。
5.选中enzyme图标,将所选质粒上的多克隆酶切位点加入左栏6.选中OK键,分析目的基因中所含的酶切位点,选插入位点时就应排除这些酶7.选中primer图标,点S图标,edit primer,开始设计正义链。
8.软件默认引物为25个碱基9.可将鼠标点在设计框的3端从右向左删除7-9个碱基,保留16-18个配对即可10.在引物的5端加入选好的酶切位点并在其左侧加3个保护碱基,入该图加入HIND III酶切位点及TTA保护碱基,完成后点analyze,认为可以后点OK。
11.选中左上角A图标,用鼠标拉动滑块将待选引物放至目的基因末端。
如图,选中EDIT PRIMERS图标,开始设计反义链。
12.从3端删除7-9个碱基同正义链。
13.将酶切位点加在5端,应将产品目录所示的酶切位点序列从右至左加入(注意不要加反)如图加入BamH I酶切位点及CGC3个保护碱基。
完成后点analyze,认为可以后点OK。
14.最后分析结果如图,反义链的FALSE PRIMING可以不考虑,RATING表示引物评分也可以不考虑,主要看Tm值正义链和反义链相差不应超过3度。
GC含量不应超过60%15.该软件有个缺点,不能保存分析结果,只能选择打印16.如果设计RT-PCR检测的引物就如下所示。
同上输入目的基因片段,选SEARCH 图标,选择参数,一般选PCR primers---both—100至250个碱基,引物长短20+/-2,search parametere中的参数可以不选,为默认设置。
点OK。
primer premier5设计引物的原理

primer premier5设计引物的原理
Primer Premier 软件设计引物的原理主要基于以下原则:
1. 引物长度:以15-30bp为合适,最常用的是18-24bp。
2. 引物GC含量:应在40-60%之间。
3. 引物碱基分布:应遵守随机性,避免连续出现4个以上的单一碱基,尤其在引物的3端不应该出现连续的G/C。
4. 避免自身互补:引物自身不能含有互补序列,否则会形成发夹一样的二级结构,影响PCR作用效果。
5. 避免引物二聚体:两个引物之间不应该有多于4个的互补/同源的碱基。
6. 引物3'端:是G/C。
7. 简并引物的出现:出于遗传密码的煎饼性。
有时需要根据一段氨基酸的保守序列反推到DNA水平设计引物。
由于大多数氨基酸(二十种常见氨基酸中的十八种)的遗传密码不只一种,由氨基酸序列反推DNA序列时,就遇到部分碱基的不确定性。
这种不确定性因物种或者细胞亚结构的不同而异。
Primer可以针对各种不同的遗传密码规律设定不同DNA和氨基酸的相互转换,这取决于被分析的序列出处。
这样设计出来的引物实际上是多种序列的混合物在某些位点有所变化,但大部分还是相同的,称之为简并引物。
遵循这些原则和技巧,可以帮助设计出更有效和可靠的PCR引物。
引物设计Premier Primer 5

生物软件应用论文引物设计软件Premier Primer 5姓名宋瑞雪__学号20107610833引物设计软件Premier Primer 5生物信息处理班宋瑞雪20107610833PCR引物设计引物设计的目的是为了找到一对合适的核苷酸片段,使其能有效地扩增模板DNA序列。
PCR反应中有两条引物,即5′端引物和3′引物。
设计引物时以一条DNA 单链为基准(常以信息链为基准),5′端引物与位于待扩增片段5′端上的一小段DNA序列相同;3′端引物与位于待扩增片段3′端的一小段DNA序列互补。
引物设计的基本原则:1、引物Tm:引物所对应模板序列的Tm 值最好72℃左右。
至少要在55-80℃之间。
Tm=2(A+T)+4(C+G)。
2、引物长度:大多数应用的最短引物长度为18个核苷酸。
如果期待的产物长度等于或小于500 bp,选用短的(16~18)的引物;若产物长5 kb,则用24个核苷酸的引物。
有人用20~23个核苷酸引物得到40 kb的产物。
3、引物碱基:G+C含量以40-60%为宜,G+C太少扩增效果不佳,G+C 过多易出现非特异条带。
ATGC最好随机分布,避免5个以上的嘌呤或嘧啶核苷酸的成串排列。
4、ΔG(自由能)值:ΔG值反映了引物与模板结合的强弱程度。
引物的ΔG值最好呈正弦曲线形状,即5’端和中间ΔG值较高,而3’端ΔG值相对较低,且不要超过9(ΔG值为负值,这里取绝对值),如此则有利于正确引发反应而可防止错误引发。
5、两个引物之间不应存在互补序列,尤其是避免3 ′端的互补重叠,引物内部不应出现互补序列。
6、引物与非特异扩增区的序列的同源性不要超过70%,引物3′末端连续8个碱基在待扩增以外不能有完全互补序列,否则易导致非特异性扩增。
7、引物3‘端的碱基,特别是最末及倒数第二个碱基,应严格要求配对,最佳选择是G和C。
8、引物的5 ′端可以修饰。
如附加限制酶位点,引入突变位点,用生物素、荧光物质、地高辛标记,加入其它短序列,包括起始密码子、终止密码子等。
PCR引物设计及Primer_Premier_5.0使用介绍

Use these two button to translate the DNA seq to a protein seq or a protein seq to a DAN seq 8种密码子偏好
引物设计界面 First you can design the primer
manually
Sense strand or anti-sense strand
引物设计的原则
2.引物GC含量在40%~60%之间,Tm值最 好接近72℃。 GC含量(composition)过高或过低都 不利于引发反应。上下游引物的GC含量 不能相差太大。若按公式Tm=4(G+C) +2(A+T)估计引物的Tm值,则有效引 物的Tm为55~80℃,其Tm值最好接近 72℃以使复性条件最佳。
引物设计的原则
3.引物3′端要避开密码子的第3位。 如扩增编码区域,引物3′端不要终止于 密码子的第3位,因密码子的第3位易发 生简并,会影响扩增的特异性与效率。
引物设计的原则
4.引物3′端不能选择A,最好选择T。 引物3′端错配时,不同碱基引发效率存 在着很大的差异,当末位的碱基为A时, 即使在错配的情况下,也能有引发链的合 成,而当末位链为T时,错配的引发效率 大大降低,G、C错配的引发效率介于A、T 之间,所以3′端最好选择T。
PCR引物设计及相关软件使用
主要内容
• • • • • PCR介绍 引物设计原理 引物设计原则 Primer Premier 5.0 介绍 举例说明引物的设计
1、什么是PCR 聚合酶链反应(Polymerase Chain Reaction ,PCR)是80年代中期发展起来的 体外核酸扩增技术。它具有特异、敏感、 产率高、快速、简便、重复性好、易自动 化等突出优点;能在一个试管内将所要研 究的目的基因或某一DNA片段于数小时内扩 增至十万乃至百万倍;可从一根毛发、一滴 血、甚至一个细胞中扩增出足量的DNA供分 析研究和检测鉴定。
Primer Premier 5软件设计引物
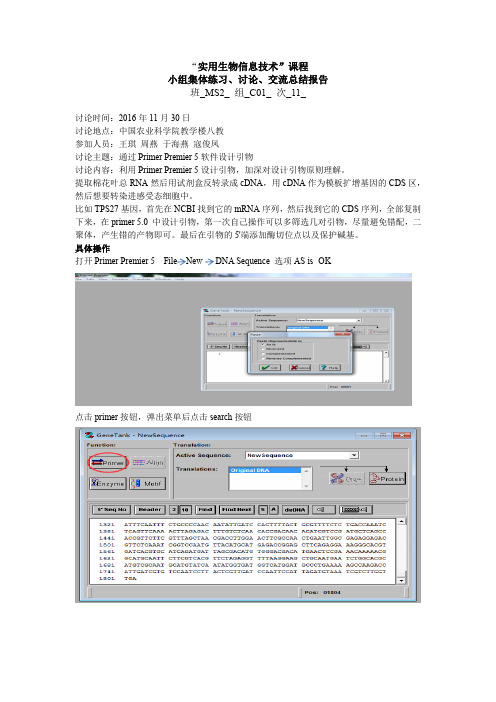
“实用生物信息技术”课程小组集体练习、讨论、交流总结报告班_MS2_ 组_C01_ 次_11_讨论时间:2016年11月30日讨论地点:中国农业科学院教学楼八教参加人员:王琪周燕于海燕寇俊凤讨论主题:通过Primer Premier 5软件设计引物讨论内容:利用Primer Premier 5设计引物,加深对设计引物原则理解。
提取棉花叶总RNA然后用试剂盒反转录成cDNA,用cDNA作为模板扩增基因的CDS区,然后想要转染进感受态细胞中。
比如TPS27基因,首先在NCBI找到它的mRNA序列,然后找到它的CDS序列,全部复制下来,在primer 5.0 中设计引物,第一次自己操作可以多筛选几对引物,尽量避免错配,二聚体,产生错的产物即可。
最后在引物的5'端添加酶切位点以及保护碱基。
具体操作打开Primer Premier 5 File New DNA Sequence 选项AS is- OK点击primer按钮,弹出菜单后点击search按钮选择PCR primer,Pairs 参数,并选择所需PCR产物长度,OK会弹出搜索结果菜单,点击OK在搜索出的结果列表里选择TM合适、GC含量高、无各种BUG的引物,点击edit primer,将所得引物复制而后点击S/A按钮,并继续点击edit primer,复制反向引物,引物设计达成。
得到3' 端GAAGTTCCAAGGTCGTTA 5' 端TCGTGGGCAGA TTTA TGT然后添加酶切识别序列到引物5' 端。
常用的内切酶比如BamHI、EcoRI、HindIII、NdeI、XhoI,这里我选择XhoI(一般在构建质粒上有且目标基因本身不被识别就可以)插入酶切位点后点击Analyz.发现Tm为64.5 有Dimer 和False Priming因此该对引物不可以。
经过分析酶切位点后没有完全没有BUG的引物,因此选择几个与设计原则背离较小的引物。
Primer_5基本使用

Primer_5基本使⽤Primer 5.0使⽤⽅法Primer Premier 5.0软件⽤途Primer Premier是⼀种⽤来帮助研究⼈员设计最适合引物的应⽤软件,利⽤它的⾼级引物搜索引物数据库,巢式引物设计,引物编辑和分析等功能,可以设计出有⾼效扩增能⼒的理想引物,也可以设计出⽤于扩增长达50kb以上的PCR产物的引物序列。
其主要功能分四⼤块,其中有三种功能⽐较常⽤,即引物设计、限制性内切酶位点分析,DNA基元(motif)查找和同源性分析功能。
Primer 5.0软件基础指南⼀. Primer 引物设计Primer功能板块包括了设计引物的搜索引擎软件,包含了强⼤的⾃动搜索法则只需要简单的操作就可以得到合适的引物。
Primer Premier也提供了⼈⼯控制搜索引擎的⽅法便于根据您的特殊要求制定标准输出的引物,通过全⾯的即时分析⼯具计算出引物的多种参数和⼆级结构关键参数,如Tm值GC含量和⾃由能G,还能以图形显⽰。
为设计⽤于定点突变的引物,该项功能板块也提供包括即使分析和限制性酶切位点分析的引物编辑⼯具,您可以通过输⼊碱基或氨基酸残基来⼿动修改引物。
您可以分析所有想⽤到的引物,可以选择事先搜索的引物,或⼿动添加引物软件。
⼆. Manual Search ⼿动搜索及引物设计原理如下:在过去⼗年中,为获得⾼效扩增PCR⽅法得到的很⼤发展,我们对引物稳定性⼆级结构和适宜长度的了解,使得我们在保证引物特异性的前体下,⼤⼤提⾼了引物的扩增效率⽽Primer Premier 软件的搜索法则很好的维持了⾼效性和特异性之间的平衡。
1.引物长度引物的长度控制着引物的特异性和在PCR反应中的退⽕温度,对多数实验适宜引物长度为18到24个碱基,等于或少于15碱基的短引物有时⽤于简单的基因作图或特殊的⽂库构建。
library protocol 28到35碱基的长引物对于从⼀系列⾼度相关的分⼦中扩增序列和特殊样本的克隆能起到重要作⽤,长PCR引物能给扩增带来更好的特异性长引物,也允许您通过降退⽕温度来能提⾼反应的灵敏度,不过长引物通常更易于形成包括发卡结构,⼆聚体⾃⾝互补等⼆级结构。
bioedit 和Primer 使用软件

实验四使用Bioedit 和Primer 软件一、实验目的1、掌握Bioedit 一个核苷酸和蛋白质序列分析软件的使用,完成如序列比对、序列检索等内容。
2、掌握primer一个引物设计软件的使用,包括单双向引物、探针的设计与酶切位点的分析等。
二、实验器材计算机,Bioedit 和primer软件,核苷酸和蛋白质序列。
三、实验内容应用我们预先准备好的序列,然后在bioedit里先打开,然后进行序列的分析;将需要设计引物的序列在primer里打开,然后设置相关参数进行引物设计。
四、实验步骤1、在电脑里将以前下载好的FASTE格式的序列,全部复制到一个新的TXT格式文本中,保存到桌面方便使用。
2.在Bioedit里打开桌面上新建的TXT格式文本;进行序列分析Accessory application选择Clustalw Multiple alignment弹出的对话框点击Run clustalw等待分析结果;并根据需要保存实验结果。
3、打开NCBI主页在搜索框中,分别将相对的序列GI值输入,打开相关文章,将文章转换成genbank格式,在上面查找相关信息,填写相关表格对应信息。
4、在ncbi中选择5个氨基酸序列,下载成FASTE格式,在电脑上新建一个TXT格式文本,将下载的5个序列全部整理在一起,保存在我的桌面上,方便使用。
5、在bioedit中进行氨基酸序列分析,先打开保存的氨基酸序列文件,进行序列分析Accessory application选择Clustalw Multiple alignment弹出的对话框点击Run clustalw等待分析结果,保存结果图片,再根据相关表格要求填写相关的结果数据。
6、打开NCBI的主页,进入BIASTE选择BIASTE P 将下载在桌面上的氨基酸序列进行比对,在显示结果中找到显示的保守序列。
将相应名称填入表格中。
7、在bioedit进行序列分析的结果上,应用primer软件进行引物和探针的设计。
Primer Premier中文使用说明

Primer Premier 4.10Primer Premier4.0是由加拿大的Premier公司开发的专业用于PCR或测序引物以及杂交探针的设计,评估的软件,和Plasmid Premier2.02一起是该公司推出的最新的软件产品。
其主要界面同样也是分为序列编辑窗口(Genetank),引物设计窗口(Primer Design),酶切分析窗口(Restriction Sites)和纹基分析窗口(Motif)。
这里我们主要介绍其引物设计功能,其他功能的介绍请参看Plasmid Premier2.02。
打开程序首先进入的是序列编辑参看,与Plasmid Premier相比,其多了一个语音校正的功能,即在输入序列的时候,程序自动将碱基读出,以便用户进行校正,保证输入的正确和快速。
点击该界面的按钮即可进入到程序的引物设计窗口。
该界面共分为四层,最上面一层左面是5个控制按钮,用于实现引物设计中的各种功能,包括引物自动寻找,寻找结果查看和引物编辑;右边是观察两个引物在模板上结合位置的直观图以及对正链还是负链引物进行选择;第二层是显示模板和引物序列及二者间的配对情况的显示;第三层是显示两个引物的各种参数,包括给引物的打分,引物以及产物的起始位置、长度、Tm值、GC%消光系数、简并性;最后一层是给出的有关于引物的二聚体结构、发卡结构、错配情况和引物间二聚体结构的预测,左边是显示是否存在以上各种对PCR扩增有影响的结构,右边显示的是这些结构的位置,结构细节和稳定能,利用这些参数可以对引物作出可靠的评价。
下面是根据模板序列寻找引物的界面,在该界面中可以设定所要搜索的引物的类型,包括PCR引物,测序引物和杂交探针以及引物所在的链;另外也能设定搜索引物的范围,以及最终PCR产物的长度和引物的长度等。
并通过来点击按钮来设置一些搜寻参数:这些参数包括引物的Tm值,GC比,有简并性碱基,3’端稳定性,引物的稳定性,重复序列,二聚体/发卡结构和与模板及可能的杂质DNA(需要从另外的序列文件中读入)之间的错配情况,这些参数的设定可以根据要求变化,程序本身根据一定的标准分成从极高严谨性到极低严谨性5个档次。
《2024年利用软件PrimerPremier5.0进行PCR引物设计的研究》范文

《利用软件PrimerPremier5.0进行PCR引物设计的研究》篇一一、引言PCR(聚合酶链式反应)是分子生物学和遗传学中常用的一种实验技术,它对于特定基因序列的克隆、分析、鉴定和扩增具有重要意义。
在PCR技术中,引物设计是一个关键的步骤,其成功与否直接影响着PCR反应的效果和准确度。
随着生物信息学的发展,引物设计软件为研究人员提供了更加方便快捷的途径。
本篇文章旨在研究并阐述如何利用软件PrimerPremier5.0进行PCR引物设计。
二、PrimerPremier5.0简介PrimerPremier5.0是一款专门用于设计PCR引物的软件,具有友好的界面和强大的功能。
它可以针对特定基因序列进行引物设计,同时还可以根据用户的需求进行参数调整,如引物的长度、GC含量、退火温度等。
此外,该软件还具有自动筛选和优化引物的功能,大大提高了引物设计的效率和准确性。
三、利用PrimerPremier5.0进行PCR引物设计的步骤1. 打开PrimerPremier5.0软件,输入需要设计的基因序列。
2. 根据实验需求设置引物设计的参数,如引物长度、GC含量、退火温度等。
3. 软件将自动分析基因序列,并在可能的位置设计引物。
用户可以根据需要调整引物的位置和参数。
4. 软件将给出所有可能的引物组合,用户可以根据引物的质量、特异性等指标进行筛选。
5. 对筛选出的引物进行优化,如调整引物的长度、GC含量等,以提高PCR反应的效率和准确性。
6. 保存并导出设计的引物序列,用于后续的PCR实验。
四、注意事项1. 在进行引物设计时,应尽量选择特异性较高的区域进行设计,以避免非特异性扩增。
2. 引物的长度和GC含量应适中,以保证PCR反应的效率和准确性。
3. 退火温度是PCR反应中一个重要的参数,应根据引物的具体情况进行调整。
4. 在使用PrimerPremier5.0进行引物设计时,应遵循软件的操作指南,以保证设计的准确性和效率。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
用软件设计引物(Primer Premier & Oligo)使用不合适的PCR 引物容易导致实验失败:表现为扩增出目的带之外的多条带(如形成引物二聚体带),不出带或出带很弱,等等。
现在PCR 引物设计大都通过计算机软件进行,软件的立足点也是按照引物设计的基本原则,只是更加快速和自动化而已。
我们可以直接提交模板序列到特定网页,如著名的primer3(/),得到设计好的引物,也可以在本地计算机上运行引物设计专业软件。
一般来说,专门进行PCR引物设计的专业软件功能更为强大,但使用起来却不太容易。
软件的引物设计功能主要体现在两个方面:首先是引物分析评价功能,该功能只有少数商业版软件能够做到,其中以“Oligo 6”最优秀;其次是引物的自动搜索功能,各种软件在这方面的侧重点不同,因此自动搜索的结果也不尽相同。
据笔者的经验,自动搜索功能以“Premier Primer”为最强且方便使用,“Oligo 6”其次,其他软件如“Vector NTI Suit”、“DNAsis”、“Omiga”和“Dnastar”都带有引物自动搜索功能,但搜索结果不是十分理想。
要想得到效果很好的引物,在自动搜索的基础上还要辅以人工分析。
笔者认为引物设计软件的最佳搭配是“Oligo”和“Premier”软件合并使用,以“Premier”进行自动搜索,“Oligo”进行分析评价,如此可快速设计出成功率很高的引物。
Primer Premier 5.0 的使用技巧简介1. 功能“Premier”的主要功能分四大块,其中有三种功能比较常用,即引物设计、限制性内切酶位点分析和DNA 基元(motif)查找。
“Premier”还具有同源性分析功能,但并非其特长,在此略过。
此外,该软件还有一些特殊功能,其中最重要的是设计简并引物,另外还有序列“朗读”、DNA 与蛋白序列的互换、语音提示键盘输入等等。
有时需要根据一段氨基酸序列反推到DNA 来设计引物,由于大多数氨基酸(20 种常见结构氨基酸中的18 种)的遗传密码不只一种,因此,由氨基酸序列反推DNA 序列时,会遇到部分碱基的不确定性。
这样设计并合成的引物实际上是多个序列的混和物,它们的序列组成大部分相同,但在某些位点有所变化,称之为简并引物。
遗传密码规则因物种或细胞亚结构的不同而异,比如在线粒体内的遗传密码与细胞核是不一样的。
“Premier”可以针对模板DNA 的来源以相应的遗传密码规则转换DNA 和氨基酸序列。
软件共给出八种生物亚结构的不同遗传密码规则供用户选择,有纤毛虫大核(Ciliate Macronuclear)、无脊椎动物线粒体(Invertebrate Mitochondrion)、支原体(Mycoplasma)、植物线粒体(Plant Mitochondrion)、原生动物线粒体(Protozoan Mitochondrion)、一般标准(Standard)、脊椎动物线粒体(Vertebrate Mitochondrion)和酵母线粒体(Yeast Mitochondrion)。
2. 使用步骤及技巧其主要功能在主界面上一目了然。
限制性酶切点分析及基元查找功能比较简单,点击该功能按钮后,选择相应的限制性内切酶或基元(如-10 序列,-35 序列等),按确定即可。
常见的限制性内切酶和基元一般都可以找到。
你还可以编辑或者添加新限制性内切酶或基元。
进行引物设计时,打开DNA序列后点击按钮primer,出现“search criteria”窗口,有多种参数可以调整。
搜索目的(Seach For)有三种选项,PCR 引物(PCR Primers),测序引物(Sequencing Primers),杂交探针(Hybridization Probes)。
搜索类型(Search Type)可选择分别或同时查找上、下游引物(Sense/Anti-sense Primer,或Both),或者成对查找(Pairs),或者分别以适合上、下游引物为主(Compatible with Sense/Anti-sense Primer)。
另外还可改变选择区域(Search Ranges),引物长度(Primer Length),选择方式(Search Mode),参数选择(Search Parameters)等等。
使用者可根据自己的需要设定各项参数。
如果没有特殊要求,建议使用默认设置。
然后按search,随之出现的Search Progress 窗口中显示Search Completed 时,再按OK,这时搜索结果以表格的形式出现,有三种显示方式,上游引物(Sense),下游引物(Anti-sense),成对显示(Pairs)。
默认显示为成对方式,并按优劣次序(Rating)排列,满分为100,即各指标基本都能达标。
点击其中一对引物,如第1#引物,并把上述窗口挪开或退出,显示“Peimer Premier”主窗口,所得结果分三部分,最上面是图示PCR 模板及产物位置,中间是所选的上下游引物的一些性质,最下面是四种重要指标的分析,包括发夹结构(Hairpin),二聚体(Dimer),错误引发情况(False Priming),及上下游引物之间二聚体形成情况(Cross Dimer)。
当所分析的引物有这四种结构的形成可能时,按钮由变成,点击该按钮,在左下角的窗口中就会出现该结构的形成情况。
一对理想的引物应当不存在任何一种上述结构,因此最好的情况是最下面的分析栏没有,只有。
值得注意的是中间一栏的末尾给出该引物的最佳退火温度,可参考应用。
在需要对引物进行修饰编辑时,如在5’端加入酶切位点,可点击,然后修改引物序列。
若要回到搜索结果中,则点击按钮。
如果要设计简并引物,只需根据源氨基酸序列的物种来源选择前述的八种遗传密码规则,反推至DNA 序列即可。
对简并引物的分析不需像一般引物那样严格。
总之,“Premier”有优秀的引物自动搜索功能,同时可进行部分指标的分析,而且容易使用,是一个相当不错的软件。
Oligo 6.22 使用技巧简介1. 功能在专门的引物设计软件中,“Oligo”是最著名的。
它的使用并不十分复杂,但初学者容易被其复杂的图表吓倒。
Oligo 5.0 的初始界面是两个图:Tm 图和ΔG 图;Oligo 6.22 的界面更复杂,出现三个图,加了个Frq 图。
“Oligo”的功能比“Premier”还要单一,就是引物设计。
但它的引物分析功能如此强大以至于能风靡全世界。
2. 使用(以Oligo 6.22 为例)Oligo 6.22 的启动界面显示的三个指标分别为Tm、ΔG 和Frq,其中Frq 是6.22 版本的新功能,为邻近6至7 个碱基组成的亚单位在一个指定数据库文件中的出现频率。
该频率高则可增加错误引发的可能性。
因为分析要涉及多个指标,起动窗口的cascade 排列方式不太方便,可从windows菜单改为tili 方式。
如果觉得太拥挤,可去掉一个指标,如Frq,这样界面的结构同于Oligo5.0,只是显示更清楚了。
在设计时,可依据图上三种指标的信息选取序列,如果觉得合适,可点击Tm 图块上左下角的Upper 按钮,选好上游引物,此时该按钮改变,表示上游引物已选取好。
下游引物的选取步骤基本同上,只是按钮变成Lower。
G 值反映了序列与模板的结合强度,最好引物的G 值在5’端和中间值比较高,而在3’端相对低。
Tm 值曲线以选取72℃附近为佳,5’到3’的下降形状也有利于引物引发聚合反应。
Frq 曲线为“Oligo 6”新引进的一个指标,揭示了序列片段存在的重复机率大小。
选取引物时,宜选用3’端Frq 值相对较低的片段。
当上下游引物全选好以后,需要对引物进行评价并根据评价对引物进行修改。
首先检查引物二聚体尤其是3’端二聚体形成的可能性。
需要注意的是,引物二聚体有可能是上游或下游引物自身形成,也有可能是在上下游引物之间形成(cross dimer)。
二聚体形成的能值越高,越不符合要求。
一般的检测(非克隆)性PCR,对引物位置、产物大小要求较低,因而应尽可能选取不形成二聚体或其能值较低的引物。
第二项检查是发夹结构(hairpin);与二聚体相同,发夹结构的能值越低越好。
一般来说,这两项结构的能值以不超过4.5 为好。
当然,在设计克隆目的的PCR 引物时,引物两端一般都添加酶切位点,必然存在发夹结构,而且能值不会太低。
这种PCR 需要通过灵活调控退火温度以达到最好效果,对引物的发夹结构的检测就不应要求太高。
第三项检查为GC 含量,以45-55%为宜。
有一些模板本身的GC 含量偏低或偏高,导致引物的GC 含量不能被控制在上述范围内,这时应尽量使上下游引物的GC 含量以及Tm 值保持接近,以有利于退火温度的选择。
如果PCR 的模板不是基因组DNA,而是一个特定模板序列,那么最好还进行False priming site 的检测。
这项检查可以看出引物在非目的位点引发PCR 反应的可能性。
如果引物在错配位点的引发效率比较高,就可能出假阳性的PCR 结果。
一般在错配引发效率以不超过100 为好,但对于特定的模板序列,还应结合比较其在正确位点的引发效率。
如果两者相差很大,比如在正确位点的引发效率为450 以上,而在错误位点的引发效率为130,那么这对引物也是可以接受的。
当我们结束以上四项检测,按Alt+P 键弹出PCR 窗口,其中总结性地显示该引物的位置、产物大小、Tm 值等参数,最有用的是还给出了推荐的最佳退火温度和简单的评价。
由于“Oligo”软件的引物自动搜索功能与“Primer Premier 5”的相类似,并且似乎并不比后者更好用,在此不再赘述。
其实,使用软件自动搜索引物就是让计算机按照人的要求去寻找最佳引物,如果参数设置得当将大大提高工作效率。
除了本地引物设计软件之外,现在还有一些网上引物设计软件,如由Whitehead Institute开发的“Primer 3”等。
该软件的独特之处在于,对全基因组PCR 的引物设计;可以将设计好的引物对后台核酸数据库进行比对,发现并排除可引发错配的引物。
因此建议经常做全基因组PCR 的用户试用。
用软件设计引物(Primer Premier & Oligo)细节补充在NCBI上搜索到我们感兴趣的基因,找到该基因的mRNA,在CDS选项中,找到编码区所在位置,在下面的origin 中,Copy该编码序列作为软件查询序列的候选对象。
一:使用Primer Premier 5打开Primer Premier5,点击File-New-DNA sequence, 出现输入序列窗口,Copy目的序列在输入框内(选择As),此窗口内,序列也可以直接翻译成蛋白。
