原核表达步骤总结
原核表达步骤

Chi l 原核表达基本试验步骤将克隆化基因插入合适载体后导入大肠杆菌用于表达大量蛋白质的方法一般称为原核表达。
这种方法在蛋白纯化、定位与功能分析等方面都有应用。
大肠杆菌用于表达重组蛋白有以下特点:易于生长和控制;用于细菌培养的材料不与哺乳动物细胞系统的材料昂贵;有各种各样的大肠杆菌菌株与与之匹配的具各种特性的质粒可供选择。
但是,在大肠杆菌中表达的蛋白由于缺少修饰和糖基化、磷酸化等翻译后加工,常形成包涵体而影响表达蛋白的生物学活性与构象。
表达载体在基因工程中具有十分重要的作用,原核表达载体通常为质粒,典型的表达载体应具有以下几种元件:(1)选择标志的编码序列;(2)可控转录的启动子;(3)转录调控序列(转录终止子,核糖体结合位点);(4)一个多限制酶切位点接头;(5)宿主体内自主复制的序列。
原核表达一般程序如下:获得目的基因-准备表达载体-将目的基因插入表达载体中(测序验证)-转化表达宿主菌-诱导靶蛋白的表达-表达蛋白的分析-扩增、纯化、进一步检测,其中包括:一、试剂准备(1)LB培养基。
(2)1M IPTG(异丙基硫代-β-D-半乳糖苷):2.38g IPTG溶于10ml ddH2O中,0.22μm滤膜抽滤,-20℃保存。
CCY的IPTG是1M的,用时进行1000倍稀释。
二、操作步骤(一)获得目的基因1、通过PCR方法:以含目的基因的克隆质粒为模板,按基因序列设计一对引物(在上游和下游引物分别引入不同的酶切位点),PCR循环获得所需基因片段。
2、通过RT-PCR方法:用TRIzol法从细胞或组织中提取总RNA,以mRNA为模板,逆转录形成cDNA第一链,以逆转录产物为模板进行PCR循环获得产物。
(二)构建重组表达载体1、载体酶切:将表达质粒用限制性内切酶(同引物的酶切位点)进行双酶切,酶切产物行琼脂糖电泳后,用胶回收Kit或冻融法回收载体大片段。
2、PCR产物双酶切后回收,在T4DNA连接酶作用下连接入载体。
原核蛋白表达流程

原核蛋白表达是一种常用的蛋白质生产方法,可以通过大肠杆菌等原核细菌表达目标蛋白。
以下是一个典型的原核蛋白表达流程:1. 选择表达系统和载体:选择适合的原核表达系统和载体。
常用的原核表达系统包括大肠杆菌系统(如E.coli),常用的载体包括pET、pGEX等。
2. 构建重组质粒:将目标基因克隆到选定的表达载体上,通常采用限制性内切酶切割和连接方法,确保目标基因正确插入载体。
3. 转化宿主菌:将构建好的重组质粒转化入宿主菌中,一般选择适当的大肠杆菌菌株,如BL21(DE3)等。
4. 培养菌液:将含有重组质粒的宿主菌接种到适当的培养基中,进行菌液的培养。
培养条件可根据所选的菌株和载体进行优化,包括温度、培养时间、培养基成分等。
5. 诱导表达:在适当的生长阶段,向培养基中加入适量的诱导剂,常用的诱导剂包括异丙基-β-D-硫代半乳糖苷(IPTG)。
6. 细胞破碎:经过一定时间的表达后,收集培养菌液并将细菌进行破碎,释放目标蛋白。
破碎方法可以选择超声波破碎、高压破碎等。
同时添加适量的蛋白酶抑制剂,避免蛋白质的降解。
7. 蛋白纯化:通过一系列的蛋白纯化步骤,如亲和层析、离子交换层析、凝胶过滤层析等,分离纯化目标蛋白。
此步骤可以根据目标蛋白的特性和需求进行优化。
8. 鉴定和确认:对纯化得到的蛋白进行鉴定和确认,如SDS-PAGE凝胶电泳、Western blot等。
确保表达的目标蛋白符合预期。
9. 储存和应用:将纯化好的目标蛋白进行适当的保存和储存,确保其稳定性和活性。
根据需要,可以进行后续的功能研究、结构分析、制备抗体等应用。
需要注意的是,原核蛋白表达流程可以根据实验目的和具体要求进行调整和优化。
不同的表达系统和载体可能需要适应性调整。
此外,对特定蛋白的表达可能需要进一步优化培养条件和蛋白纯化步骤。
大肠杆菌的原核表达实验过程结果

大肠杆菌的原核表达实验过程结果
大肠杆菌的原核表达实验一般分为以下几个步骤:
1. 构建表达载体:将待表达的基因克隆到适当的表达载体上,如常用的pET、pGEX等载体中。
2. 转化大肠杆菌:将表达载体转化到大肠杆菌细胞中。
可以通过化学方法、电转化、冷冻复苏、热激转化等方法进行。
3. 诱导表达:在大肠杆菌细胞进行生长至适当时期后,添加适宜浓度的诱导剂,如IPTG等,诱导待表达基因蛋白的合成。
4. 细胞收获和破碎:诱导一定时间后,收获大肠杆菌细胞并进行破碎,以获得待表达蛋白。
5. 蛋白提取:对细胞破碎物进行离心、超滤等步骤,去除残余细胞构成和杂质,得到含有待表达蛋白的上清液。
6. 纯化和分析:将上清液进行分离、纯化、鉴定,以确定表达蛋白相应的分子量、酶活性等。
在以上步骤中,实验者需要进行质控和评估,确认实验步骤是否正确和表达蛋白是否达到预期,以确保实验结果的可靠性和准确性。
原核表达步骤总结
原核表达步骤原核表达先要将基因克隆到原核表达载体上,然后通过转化到JM109或BL21等菌株中,诱导表达蛋白,然后进行蛋白纯化。
本实验方案的前提是,目的基因已克隆到载体,并已转进入JM109菌株中。
一.鉴定目的蛋白是否在大肠杆菌JM109或BL21中大量表达(一)制样1 . 挑取经过双酶切鉴定的单克隆菌落于700ul LB培养基,加入0.7ul Amp (100mg/mL),37o C200r/min摇床培养,过夜活化。
2. 以1:50比例(200ul),将活化的过夜培养物加入10mL LB液体培养基中,加入10uLAmp(100mg/ml),37o C200r/min摇床扩大培养2h-3h,期间取样监控菌液的OD值,控制菌液OD600在0.6-1.0之间,以使大肠杆菌处于最适合表达外源蛋白的生长状态。
(一般3h时,菌液浓度及达到标准,但是不同的基因对菌的影响不同,所以第一次实验时需要确定这个最佳时间)3. 从10ml扩大培养物中取3ml菌液作为不加IPTG的空白对照(CK),其余7ml菌液加入7ul IPTG(储存浓度为0.5mol/l),使IPTG终浓度达到0.5mmol/l。
以200r/min的转速,37o C摇床培养3h。
4. 以5000r/min离心2min收集菌体,倾倒上清,每个离心管收集3ml培养物。
5. 加入1ml dH2O,将管底沉淀用振荡器打散以充分洗涤,8000r/min离心2min,倾倒上清。
6. 重复步骤5。
将离心管中的水倒干净。
(二)菌落SDS-PAGE1. 在收集的菌体中加入200ul 1×SDS PAGE loading buffer(可根据沉淀的量增加或减少loading buffer的量,一般200ul比较合适)。
用漩涡器剧烈震荡,确保将管底沉淀震散。
2. 将样品于100℃恒温加热器上开盖加热10min(Marker也要加热)。
样品凉后,12000r/min离心3min,取每管的上清点样。
原核表达步骤

原核表达步骤原核表达是指在原核生物体内将基因转录成RNA,再将RNA翻译成蛋白质的过程。
本文将详细介绍原核表达的步骤。
1. 转录DNA的双链结构被酶RNA聚合酶解开,从而形成mRNA链。
RNA聚合酶沿着DNA模板链移动,将mRNA链合成在一起。
在这个过程中,RNA聚合酶根据DNA模板链上的碱基序列,选择正确的核苷酸,将其加入到正在合成的mRNA链上。
2. 剪接在细胞核内,mRNA链是在原核生物上转录的。
这些mRNA链可能包含顺式调节区域(UTR)和内含子区域。
在剪接过程中,内含子被剪除,UTR被保留下来。
这个过程由小核RNA(snRNA)和蛋白质共同完成。
3. 翻译翻译是将mRNA链转化为氨基酸序列的过程。
翻译是在核糖体中完成的。
核糖体是由rRNA和蛋白质组成的复合体。
核糖体通过识别mRNA上的起始密码子来开始翻译过程。
起始密码子是AUG。
核糖体将氨基酸连接在一起,直到遇到终止密码子。
终止密码子分别是UAA,UAG和UGA。
翻译完成后,成品蛋白被释放出来。
4. 后翻译修饰在翻译完成后,蛋白质可能需要进行后翻译修饰。
这些修饰可以包括磷酸化,甲基化,硫化,酰化和糖基化等。
这些修饰可以改变蛋白质的结构和功能,从而影响其生物学活性。
5. 折叠蛋白质被合成后,需要进一步折叠成其最终形态。
这个过程由分子伴侣和蛋白酶等分子机器完成。
分子伴侣可以协助蛋白质正确地折叠。
蛋白酶可以降解不正确折叠的蛋白质,防止它们对细胞造成损害。
6. 定位在折叠完成后,蛋白质需要被定位到其最终的位置。
这个过程由信号肽和其他分子机器完成。
信号肽是一段氨基酸序列,可以将蛋白质定位到细胞膜,内质网,线粒体等亚细胞结构中。
原核表达是一个复杂的过程,包括转录,剪接,翻译,后翻译修饰,折叠和定位。
这些步骤需要各种不同的分子机器和分子信号来协同完成。
理解原核表达的步骤可以帮助我们更好地理解生物学过程,从而为生命科学的研究和应用提供基础。
原核表达步骤

5、 pET重组子鉴定
如果亚克隆成功,阳性菌落数远大于阴性菌落。
检验转化子的方法很多,包括PCR、质粒抽提及酶切分析、测序,体外转录和翻译。
若用PCR方法,可选用载体上的T7promoter (5‘载体特异性引物)and termintor(3‘特异性引物)引物中的一个和基因中的一个序列引物进行PCR验证,并可以判断插入方向。
6、DE3溶原菌的诱导表达
(1)从新鲜的划线平板中挑取单克隆到50ml含50ug/ul 卡那霉素的液体培养基中,37℃培养至OD600为0.4-1。
(2)培养基中加入100mMIPTG至终浓度为0.4mM(T7启动子)或1 mM(T7lac 启动子),继续培养2-3小时。
(3)将摇瓶置于冰上5min,5000g 4℃离心5min收集菌体。
(4)重悬细胞于0.25倍体积预冷的20mMTris-HCl (pH8.0)中,离心。
(5)除去上清,菌体保存于-70℃或继续纯化。
7、 SDS-PAGE进行目标蛋白质分析
(1)机械破碎细胞。
一般用弗氏压碎法或超声波处理。
(2)裂解液14000g离心10min,分离可溶和不溶部分。
(3)100ul可溶上清中加入100ul 4×SDS上样buffer和水。
85℃迅速加热3min 使蛋白变性,然后上样进行SDS-PAGE分析,观察蛋白表达。
(4)若目标蛋白在不溶部分中,需进行包涵体纯化。
750ul20mMTris-HCl, pH7.5重悬沉淀,离心10000g5min, 除去上清重复洗涤。
然后1.5ml 1%SDS上样buffer中重悬沉淀,重复(3)的步骤进行SDS-PAGE分析。
原核表达的详细步骤

原核表达详细步骤PartⅠ选择表达的目的基因一、基因序列1. 得到靶基因DNA(cDNA)序列,有几种方式寻找正确的读码顺序:①利用生物信息学在NCBI上blast同源基因,找到同源蛋白,再在DNA的ORF中找到正确的读码。
②实验方法,即得到蛋白,进行测序,然后在DNA上找到正确的读码。
③利用mRNA的特征,找到启动子,编码区,终止子。
在编码区中找到翻译起始密码子与终止密码子(cDNA)。
2. 注意事项:①区别ORF和CDS→ORF一般在DNA上的定义,寻找原则是翻译起始密码子和终止密码子;CDS可以是DNA上的定义,也可以是mRNA上的定义,分为complete CDS 和partial CDS,是从第一个核酸开始读,连续读下去,complete CDS读码是“M、、、、、、、、、*”,partial CDS的读码是相应的AA②在进行试验设计时,充分利用生物信息学的信息后,在进行试验设计。
二、抗原决定簇的预测1、原理:蛋白质表面部分可以使免疫系统产生抗体的区域叫抗原决定簇。
一般抗原决定簇是由6-12 氨基酸或碳水基团组成,它可以是由连续序列(蛋白质一级结构)组成或由不连续的蛋白质三维结构组成。
变性蛋白只是天然蛋白伸直的了产物,用来免疫动物具有更强的抗原性。
只是天然蛋白中被包在内部的抗原决定簇也会暴露出来,如果用该变性抗原制备的抗体来检测变性抗原是可以的,如果用来检测天然蛋白,可能会有假阳性。
做单抗也可以,同样道理,筛选出的单抗可能对抗的抗原决定簇处于天然抗原的内部,是否能用还要看将来该单抗用来干什么。
2、选择原则:(1)、亲水性:大部分抗原决定簇是亲水性的。
(2)、处于结构表面:大部分抗体只与蛋白质表面部分结合。
(3)、有弹性:许多已知的抗原决定簇是在自由活动区域。
所以一般来说蛋白质的N 端及C 端是很好的抗原决定簇区域。
3、选定抗原决定簇的步骤:(1)预测:如软件预测DNAstar(Protean)预测,Dnaman。
原核表达步骤

3、重组蛋白所处的环境:发酵温度高或胞内pH接近蛋白的等电点时容易形成包涵体。
CCY的IPTG是1M的,用时进行1000倍稀释。ﻫ二、操作步骤ﻫ(一)获得目的基因
1、通过PCR方法:以含目的基因的克隆质粒为模板,按基因序列设计一对引物(在上游和下游引物分别引入不同的酶切位点),PCR循环获得所需基因片段。ﻫ2、通过RT-PCR方法:用TRIzol法从细胞或组织中提取总RNA,以mRNA为模板,逆转录形成cDNA第一链,以逆转录产物为模板进行PCR循环获得产物。
12、用3ml 含400mM咪唑洗脱目的蛋白。
13、镍柱用20%的乙醇适量,4℃保存。
14、SDS-PAGE电泳检测蛋白纯化效果(上柱前样本,上柱后样本,杂蛋白,目的蛋白,未诱导蛋白)
附相关试剂配制:
50mM PBS
NaH2PO4.2H2O 1.48g
Na2HPO4.12H2O 14.5046g
NaCL 29.3g
表达载体在基因工程中具有十分重要的作用,原核表达载体通常为质粒,典型的表达载体应具有以下几种元件:ﻫ(1)选择标志的编码序列;
(2)可控转录的启动子;ﻫ(3)转录调控序列(转录终止子,核糖体结合位点);
(4)一个多限制酶切位点接头;ﻫ(5)宿主体内自主复制的序列。
原核表达一般程序如下:获得目的基因-准备表达载体-将目的基因插入表达载体中(测序验证)-转化表达宿主菌-诱导靶蛋白的表达-表达蛋白的分析-扩增、纯化、进一步检测,其中包括:ﻫ一、试剂准备ﻫ(1)LB培养基。ﻫ(2)1M IPTG(异丙基硫代-β-D-半乳糖苷):2.38g IPTG溶于10ml ddH2O中,0.22μm滤膜抽滤,-20℃保存。
原核表达和真核表达
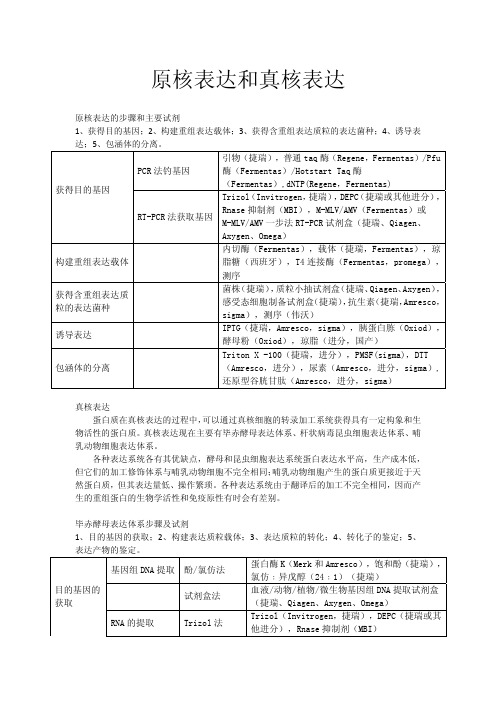
原核表达和真核表达原核表达的步骤和主要试剂1、获得目的基因;2、构建重组表达载体;3、获得含重组表达质粒的表达菌种;4、诱导表达;5、包涵体的分离。
获得目的基因 PCR法钓基因引物(捷瑞),普通taq酶(Regene,Fermentas)/Pfu酶(Fermentas)/Hotstart Taq酶(Fermentas),dNTP(Regene,Fermentas)RT-PCR法获取基因Trizol(Invitrogen,捷瑞),DEPC(捷瑞或其他进分),Rnase抑制剂(MBI),M-MLV/AMV(Fermentas)或M-MLV/AMV一步法RT-PCR试剂盒(捷瑞、Qiagen、Axygen、Omega)构建重组表达载体 内切酶(Fermentas),载体(捷瑞,Fermentas),琼脂糖(西班牙),T4连接酶(Fermentas,promega),测序获得含重组表达质粒的表达菌种 菌株(捷瑞),质粒小抽试剂盒(捷瑞、Qiagen、Axygen),感受态细胞制备试剂盒(捷瑞),抗生素(捷瑞,Amresco,sigma),测序(伟沃)诱导表达 IPTG(捷瑞,Amresco,sigma),胰蛋白胨(Oxiod),酵母粉(Oxiod),琼脂(进分,国产)包涵体的分离 Triton X -100(捷瑞,进分),PMSF(sigma),DTT (Amresco,进分),尿素(Amresco,进分,sigma),还原型谷胱甘肽(Amresco,进分,sigma)真核表达蛋白质在真核表达的过程中,可以通过真核细胞的转录加工系统获得具有一定构象和生物活性的蛋白质。
真核表达现在主要有毕赤酵母表达体系、杆状病毒昆虫细胞表达体系、哺乳动物细胞表达体系。
各种表达系统各有其优缺点,酵母和昆虫细胞表达系统蛋白表达水平高,生产成本低,但它们的加工修饰体系与哺乳动物细胞不完全相同;哺乳动物细胞产生的蛋白质更接近于天然蛋白质,但其表达量低、操作繁琐。
原核表达步骤总结

原核表达步骤原核表达先要将基因克隆到原核表达载体上,然后通过转化到JM109或BL21等菌株中,诱导表达蛋白,然后进行蛋白纯化。
本实验方案的前提是,目的基因已克隆到载体,并已转进入JM109菌株中。
一.鉴定目的蛋白是否在大肠杆菌JM109或BL21中大量表达(一)制样1 . 挑取经过双酶切鉴定的单克隆菌落于700ul LB培养基,加入0.7ul Amp (100mg/mL),37o C200r/min摇床培养,过夜活化。
2. 以1:50比例(200ul),将活化的过夜培养物加入10mL LB液体培养基中,加入10uLAmp(100mg/ml),37o C200r/min摇床扩大培养2h-3h,期间取样监控菌液的OD值,控制菌液OD600在0.6-1.0之间,以使大肠杆菌处于最适合表达外源蛋白的生长状态。
(一般3h时,菌液浓度及达到标准,但是不同的基因对菌的影响不同,所以第一次实验时需要确定这个最佳时间)3. 从10ml扩大培养物中取3ml菌液作为不加IPTG的空白对照(CK),其余7ml菌液加入7ul IPTG(储存浓度为0.5mol/l),使IPTG终浓度达到0.5mmol/l。
以200r/min的转速,37o C摇床培养3h。
4. 以5000r/min离心2min收集菌体,倾倒上清,每个离心管收集3ml培养物。
5. 加入1ml dH2O,将管底沉淀用振荡器打散以充分洗涤,8000r/min离心2min,倾倒上清。
6. 重复步骤5。
将离心管中的水倒干净。
(二)菌落SDS-PAGE1. 在收集的菌体中加入200ul 1×SDS PAGE loading buffer(可根据沉淀的量增加或减少loading buffer的量,一般200ul比较合适)。
用漩涡器剧烈震荡,确保将管底沉淀震散。
2. 将样品于100℃恒温加热器上开盖加热10min(Marker也要加热)。
样品凉后,12000r/min离心3min,取每管的上清点样。
原核表达步骤

原核表达步骤一、转录(Transcription)转录是指DNA转录为RNA的过程,即DNA的信息通过RNA的合成而表达出来。
转录是生物体中基因表达的第一步,也是生物体合成蛋白质的关键步骤之一。
在原核生物中,转录过程分为三个阶段:启动、延伸和终止。
启动阶段是指RNA聚合酶(RNA polymerase)与DNA的结合,并在DNA上找到起始转录位点。
延伸阶段是指RNA聚合酶沿DNA模板链向下滑动,并合成RNA链。
终止阶段是指RNA聚合酶在遇到终止信号时停止转录,释放出合成的RNA链。
二、剪接(Splicing)剪接是指在转录过程中,将RNA前体分子中的内含子(intron)剪切除去,将外显子(exon)连接起来的过程。
原核生物中的剪接方式相对简单,通常是直接将外显子连接起来,形成成熟的RNA分子。
剪接的主要作用是消除内含子的干扰,使RNA分子能够直接参与翻译过程。
通过剪接,原核生物能够快速生成成熟的RNA分子,提高基因表达效率。
三、翻译(Translation)翻译是指将RNA的信息翻译成蛋白质的过程。
在原核生物中,翻译过程发生在核糖体(ribosome)中,涉及到mRNA、tRNA和rRNA等多种分子。
翻译分为三个主要步骤:启动、延伸和终止。
启动阶段是指核糖体与mRNA的结合,并识别起始密码子(AUG)位置。
延伸阶段是指核糖体沿mRNA滑动,将氨基酸依次加入正在合成的多肽链中。
终止阶段是指核糖体在遇到终止密码子(UAA、UAG、UGA)时停止翻译,释放合成的多肽链。
四、调控(Regulation)调控是指控制基因表达水平的过程,包括转录调控和翻译调控两个方面。
原核生物通过调控转录和翻译过程中的各个环节来实现基因表达的精确调控。
转录调控主要通过转录因子(transcription factor)和启动子(promoter)之间的相互作用来实现。
转录因子能够结合到启动子上,促进或抑制RNA聚合酶的结合,从而调控基因的转录水平。
原核表达操作步骤及注意事项

原核表达操作步骤及注意事项时间:2010-03-03 14:05:01 来源:作者:点击:1046次将克隆化基因插入合适载体后导入大肠杆菌用于表达大量蛋白质的方法一般称为原核表达。
这种方法在蛋白纯化、定位及功能分析等方面都有应用。
大肠杆菌用于表达重组蛋白有以下特点:易于生长和控制;用于细菌培养的材料不及哺乳动物细胞系统的材料昂贵;有各种各样的大肠杆菌菌株及与之匹配的具各种特性的质粒可供选择。
但是,在大肠杆菌中表达的蛋白由于缺少修饰和糖基化、磷酸化等翻译后加工,常形成包涵体而影响表达蛋白的生物学活性及构象。
表达载体在基因工程中具有十分重要的作用,原核表达载体通常为质粒,典型的表达载体应具有以下几种元件:(1)选择标志的编码序列;(2)可控转录的启动子;(3)转录调控序列(转录终止子,核糖体结合位点);(4)一个多限制酶切位点接头;(5)宿主体内自主复制的序列。
原核表达一般程序如下:获得目的基因-准备表达载体-将目的基因插入表达载体中(测序验证)-转化表达宿主菌-诱导靶蛋白的表达-表达蛋白的分析-扩增、纯化、进一步检测一、试剂准备1、LB培养基。
2、100mM IPTG(异丙基硫代-β-D-半乳糖苷):2.38g IPTG溶于100ml ddH2O中,0.22μm滤膜抽滤,-20℃保存。
二、操作步骤(一)获得目的基因1、通过PCR方法:以含目的基因的克隆质粒为模板,按基因序列设计一对引物(在上游和下游引物分别引入不同的酶切位点),PCR循环获得所需基因片段。
2、通过RT-PCR方法:用TRIzol法从细胞或组织中提取总RNA,以mRNA为模板,逆转录形成cDNA 第一链,以逆转录产物为模板进行PCR循环获得产物。
(二)构建重组表达载体1、载体酶切:将表达质粒用限制性内切酶(同引物的酶切位点)进行双酶切,酶切产物行琼脂糖电泳后,用胶回收Kit或冻融法回收载体大片段。
2、PCR产物双酶切后回收,在T4DNA连接酶作用下连接入载体。
原核表达步骤

悬细菌。超声破碎(300W4 S/4S,99次)。超声后液体变清澈。超声时 探头离管底一定距离,防止管破裂。
&12000g, 10mi n,取上清作为待纯化的样品。
9、 混匀镍柱,根据培养物及表达水平装柱2ml(加膜防镍柱漏),用三 个柱床体积50mM PBS(不可溶细菌加8M尿素50mM PBS平衡柱子
原剂使二硫键正常形成。一般在尿素浓度4M左右时复性过程开始,到2M左右时结束。对于
盐酸胍而言,可以从4M开始,到1.5M时复性过程已经结束。
复性中常采用的方法有: 稀释复性:直接加入水或缓冲液,放置过夜,缺点是体积增加较大,变性剂稀释速度太快, 不易控制。
透析复性: 好处是不增加体积, 通过逐渐降低外透液浓度来控制变性剂去除速度, 有人称易 形成沉淀,且不适合大规模操作,无法应用到生产规模。
1可溶性蛋白在细胞容易受到蛋白酶的攻击,包涵体表达可以避免蛋白酶对外源蛋白的降 解。
2、降低了胞外源蛋白的浓度,有利于表达量的提高。
3、包涵体中杂蛋白含量较低,且只需要简单的低速离心就可以与可溶性蛋白分离,有利于 分离纯化。
4、对机械搅拌和超声破碎不敏感,易于破壁,并与细胞膜碎片分离。
分离:
1离心:5000-20000g15min离心,可使大多数包涵体沉淀,与可溶性蛋白分离。
杆菌中表达的蛋白由于缺少修饰和糖基化、磷酸化等翻译后加工,常形
成包涵体而影响表达蛋白的生物学活性及构象。
表达载体在基因工程中具有十分重要的作用, 原核表达载体通常为质粒, 典型的表达载体应具有以下几种元件:
(1)选择标志的编码序列;
(2)可控转录的启动子;
原核表达步骤

1.引物的设计及合成2.基因的克隆: PCR反应PCR产物的回收和纯化PCR产物的酶切3.质粒pET-32a的扩增、提取及酶切接种含有pET-32a空质粒的DH5α的菌株至LB (含有Amp l00ug/mL)中,37℃培养过夜。
取5mL菌液,按质粒提取试剂盒说明书提取质粒,酶切提取出的质粒,-20℃保存。
4.重组原核表达质粒的构建:4.1重组质粒的连接4.2感受态细胞的制备和转化:参照《分子克隆实验指南》的方法进行BL21感受态细胞的制备[16]。
在平板上挑取新鲜的BL21菌落,接种到5mLLB培养基中,37℃振荡培养2~5h,OD600达到0.35~0.5之间,取l~2mL菌液于无菌离心管中,冰上放置至0℃,4℃、5000 rpm离心5min,弃上清,用200uL CaCl2(0.1M)重悬,冰上放置30min,4℃、5000 rpm离心5min,弃上清,0.1M CaC12100uL重悬,4℃放置备用。
4.3重组质粒转化感受态细胞:将10uL重组原核表达质粒加入到100uL的BL21感受态细胞中,轻轻混匀,冰上放置30min,将离心管放入42℃水浴中热休克90s,立即将离心管冰浴5~10min,每管加入800uL的LB培养基,然后放入37℃摇床中100rpm培养45min,取出离心管5000rpm离心5min,弃去600uL上清,将沉淀的细菌混匀并涂布氨卞抗性的LB平板,涂布后正置30min后倒置于37℃培养箱中,培养12~16h。
4.4 重组质粒的鉴定4.4.1 PCR鉴定:挑取抗性LB平板上长出的BL21重组菌,并接种LB培养基中(含Amp100ug/mL),37℃、200rpm摇振培养3~5h,PCR鉴定。
4.4.2 酶切鉴定:应用限制性内切酶对提取的质粒进行双酶切鉴定,通过双酶切鉴定目的基因有无插入及大小是否正确。
酶切结束后进行1%琼脂糖凝胶电泳,凝胶成像系统下观察并拍照记录。
4.4.3重组质粒基因序列测定:挑选PCR检测条带亮,酶切鉴定正确的菌株测序。
原核表达操作流程

(1)PCR扩增及产物回收双引物扩增,25µL体系如下:上游加酶切位点引物1µL 、下游加酶切位点引物1uL、ddH2O 10µL、PrimeSTAR Mix12.5 µL、cDNA 0.5µL 、。
PCR反应程序为:①98℃2min;② 98℃15s、55℃15s、72℃30s、36个循环;③72℃5min;④保持在4℃。
取所有PCR产物电泳检查扩增结果,并进行大量胶回收。
(2)载体构建及质粒转化将PCR产物进行BamHI和NotI双酶切,琼脂糖凝胶电泳分离目的DNA 片段,用胶回收试剂盒回收该片段;同样采用BamHI和NotI对载体质粒pET-28a 双酶切,琼脂糖凝胶电泳分离纯化,胶回收试剂盒回收载体片段。
将PCR目的片段连接到载体上,连接体系为:回收片段XμL(50ng)、回收载体YμL(50-100ng)、Ligation mix X+Y uL、16℃连接过夜。
取连接产物10μL转化入E.coli Trans5α 感受态细胞中,冰上放置30min,42℃水浴90s,冰上放置1~2min,加250μL 的LB 液体培养基于37℃摇床缓慢摇45min,涂含卡纳霉素的LB 平板,37℃培养过夜。
在平板上挑取单菌落,PCR 筛选阳性克隆。
(3)原核诱导表达及表达条件优化将重组质粒转化入E.coli BL21(DE3)的感受态细胞内,涂布于含卡纳青霉素30μg/mL 的LB 平板中,于37℃培养过夜。
次日,挑单菌落于50mL培养基中过夜培养。
将50mL扩大培养之后的菌液分成两份,分别加入含有500mL培养基的1L锥形瓶中继续扩大培养,大约经过2-3个小时之后,当OD600接近0.6-1.0时,取2mL菌液离心,去上清液,得到菌体沉淀后放-20℃保存,作为SDS-PAGE对照样品。
然后,加入100mM IPTG 至终浓度1mM(IPTG/培养基体积=1/100),37℃培养4h后离心得沉淀菌体(4℃4000g,20min),-20℃。
原核表达的详细步骤

原核表达详细步骤PartⅠ选择表达的目的基因一、基因序列1. 得到靶基因DNA(cDNA)序列,有几种方式寻找正确的读码顺序:①利用生物信息学在NCBI上blast同源基因,找到同源蛋白,再在DNA的ORF中找到正确的读码。
②实验方法,即得到蛋白,进行测序,然后在DNA上找到正确的读码。
③利用mRNA的特征,找到启动子,编码区,终止子。
在编码区中找到翻译起始密码子与终止密码子(cDNA).2。
注意事项:①区别ORF和CDS→ORF一般在DNA上的定义,寻找原则是翻译起始密码子和终止密码子;CDS可以是DNA上的定义,也可以是mRNA上的定义,分为complete CDS 和partial CDS,是从第一个核酸开始读,连续读下去,complete CDS读码是“M、、、、、、、、、*”,partial CDS的读码是相应的AA②在进行试验设计时,充分利用生物信息学的信息后,在进行试验设计.二、抗原决定簇的预测1、原理:蛋白质表面部分可以使免疫系统产生抗体的区域叫抗原决定簇.一般抗原决定簇是由6-12 氨基酸或碳水基团组成,它可以是由连续序列(蛋白质一级结构)组成或由不连续的蛋白质三维结构组成.变性蛋白只是天然蛋白伸直的了产物,用来免疫动物具有更强的抗原性.只是天然蛋白中被包在内部的抗原决定簇也会暴露出来,如果用该变性抗原制备的抗体来检测变性抗原是可以的,如果用来检测天然蛋白,可能会有假阳性。
做单抗也可以,同样道理,筛选出的单抗可能对抗的抗原决定簇处于天然抗原的内部,是否能用还要看将来该单抗用来干什么。
2、选择原则:(1)、亲水性:大部分抗原决定簇是亲水性的。
(2)、处于结构表面:大部分抗体只与蛋白质表面部分结合.(3)、有弹性:许多已知的抗原决定簇是在自由活动区域。
所以一般来说蛋白质的N 端及C 端是很好的抗原决定簇区域.3、选定抗原决定簇的步骤:(1)预测:如软件预测DNAstar(Protean)预测,Dnaman.在线网站预测(http://www-bimas.cit。
原核表达操作步骤及注意事项

原核表达操作步骤及注意事项原核表达操作步骤及注意事项发布时间: 2019-06-03 新闻来源:将克隆化基因插入合适载体后导入大肠杆菌用于表达大量蛋白质的方法一般称为原核表达。
这种方法在蛋白纯化、定位及功能分析等方面都有应用。
大肠杆菌用于表达重组蛋白有以下特点:易于生长和控制;用于细菌培养的材料不及哺乳动物细胞系统的材料昂贵;有各种各样的大肠杆菌菌株及与之匹配的具各种特性的质粒可供选择。
但是,在大肠杆菌中表达的蛋白由于缺少修饰和糖基化、磷酸化等翻译后加工,常形成包涵体而影响表达蛋白的生物学活性及构象。
表达载体在基因工程中具有十分重要的作用,原核表达载体通常为质粒,典型的表达载体应具有以下几种元件:(1)选择标志的编码序列;(2)可控转录的启动子;(3)转录调控序列(转录终止子,核糖体结合位点) ;(4)一个多限制酶切位点接头;(5)宿主体内自主复制的序列。
原核表达一般程序如下:获得目的基因-准备表达载体-将目的基因插入表达载体中(测序验证)-转化表达宿主菌-诱导靶蛋白的表达-表达蛋白的分析-扩增、纯化、进一步检测一、试剂准备1、LB 培养基。
2、100mM IPTG(异丙基硫代-β-D-半乳糖苷):2.38g IPTG溶于100ml ddH2O 中,0.22μm滤膜抽滤,-20℃保存。
二、操作步骤(一)获得目的基因1、通过PCR 方法:以含目的基因的克隆质粒为模板,按基因序列设计一对引物(在上游和下游引物分别引入不同的酶切位点),PCR 循环获得所需基因片段。
2、通过RT-PCR 方法:用TRIzol 法从细胞或组织中提取总RNA ,以mRNA 为模板,逆转录形成cDNA 第一链,以逆转录产物为模板进行PCR 循环获得产物。
(二)构建重组表达载体1、载体酶切:将表达质粒用限制性内切酶(同引物的酶切位点)进行双酶切,酶切产物行琼脂糖电泳后,用胶回收Kit 或冻融法回收载体大片段。
2、PCR 产物双酶切后回收,在T4DNA 连接酶作用下连接入载体。
原核表达实验报告

原核表达实验报告
一、实验目的
本实验旨在通过原核表达系统将目标蛋白在大肠杆菌中进行表达,并对其进行纯化和鉴定。
二、实验材料
1. 目标蛋白基因克隆质粒;
2. 大肠杆菌感受态细胞;
3. 抗生素(如氨苄青霉素);
4. IPTG诱导剂;
5. SDS-PAGE凝胶电泳试剂盒;
6. 蛋白质纯化试剂盒。
三、实验步骤
1. 大肠杆菌转化:将目标蛋白基因克隆质粒转化到大肠杆菌感受态细胞中,加入抗生素(如氨苄青霉素)筛选阳性克隆。
2. 诱导表达:将阳性克隆的大肠杆菌接种到含有IPTG诱导剂的培养基中,使其表达目标蛋白。
3. 收集菌体:培养一定时间后,收集大肠杆菌菌体。
4. 裂解菌体:将收集到的大肠杆菌菌体进行裂解,释放目标蛋白。
5. 纯化目标蛋白:使用蛋白质纯化试剂盒对目标蛋白进行纯化。
6. 鉴定目标蛋白:通过SDS-PAGE凝胶电泳分析目标蛋白的表达情况和纯度。
四、实验结果与分析
1. 大肠杆菌转化结果:通过抗生素筛选,获得阳性克隆。
2. 诱导表达结果:在含有IPTG诱导剂的培养基中,目标蛋白得到了有效表达。
3. 菌体收集结果:成功收集到大肠杆菌菌体。
4. 裂解菌体结果:菌体裂解后,释放出目标蛋白。
5. 纯化目标蛋白结果:通过蛋白质纯化试剂盒,成功纯化了目标蛋白。
6. 鉴定目标蛋白结果:通过SDS-PAGE凝胶电泳分析,目标蛋白得到了高纯度的表达。
五、实验结论
本实验通过原核表达系统成功表达了目标蛋白,并通过纯化和鉴定获得了高纯度的目标蛋白。
这为进一步研究该蛋白的功能和应用提供了基础。
原核表达操作步骤及注意事项

原核表达操作步骤及注意事项将克隆化基因插入合适载体后导入大肠杆菌用于表达大量蛋白质的方法一般称为原核表达。
这种方法在蛋白纯化、定位及功能分析等方面都有应用。
大肠杆菌用于表达重组蛋白有以下特点:易于生长和控制;用于细菌培养的材料不及哺乳动物细胞系统的材料昂贵;有各种各样的大肠杆菌菌株及与之匹配的具各种特性的质粒可供选择。
但是,在大肠杆菌中表达的蛋白由于缺少修饰和糖基化、磷酸化等翻译后加工,常形成包涵体而影响表达蛋白的生物学活性及构象。
表达载体在基因工程中具有十分重要的作用,原核表达载体通常为质粒,典型的表达载体应具有以下几种元件:(1)选择标志的编码序列;(2)可控转录的启动子;(3)转录调控序列(转录终止子,核糖体结合位点);(4)一个多限制酶切位点接头;(5)宿主体内自主复制的序列。
原核表达一般程序如下:获得目的基因-准备表达载体-将目的基因插入表达载体中(测序验证)-转化表达宿主菌-诱导靶蛋白的表达-表达蛋白的分析-扩增、纯化、进一步检测一、试剂准备1、LB培养基。
2、100mM IPTG(异丙基硫代-β-D-半乳糖苷):2.38g IPTG溶于100ml ddH2O中,0.22μm滤膜抽滤,-20℃保存。
二、操作步骤(一)获得目的基因1、通过PCR方法:以含目的基因的克隆质粒为模板,按基因序列设计一对引物(在上游和下游引物分别引入不同的酶切位点),PCR循环获得所需基因片段。
2、通过RT-PCR方法:用TRIzol法从细胞或组织中提取总RNA,以mRNA为模板,逆转录形成cDNA 第一链,以逆转录产物为模板进行PCR循环获得产物。
(二)构建重组表达载体1、载体酶切:将表达质粒用限制性内切酶(同引物的酶切位点)进行双酶切,酶切产物行琼脂糖电泳后,用胶回收Kit或冻融法回收载体大片段。
2、PCR产物双酶切后回收,在T4DNA连接酶作用下连接入载体。
(三)获得含重组表达质粒的表达菌种1、将连接产物转化大肠杆菌DH5α,根据重组载体的标志(抗Amp或蓝白斑)作筛选,挑取单斑,碱裂解法小量抽提质粒,双酶切初步鉴定。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
原核表达步骤原核表达先要将基因克隆到原核表达载体上,然后通过转化到JM109或BL21等菌株中,诱导表达蛋白,然后进行蛋白纯化。
本实验方案的前提是,目的基因已克隆到载体,并已转进入JM109菌株中。
1.鉴定目的蛋白是否在大肠杆菌JM109或BL21中大量表达(1)制样1 . 挑取经过双酶切鉴定的单克隆菌落于700ul LB培养基,加入0.7ul Amp(100mg/mL),37o C200r/min摇床培养,过夜活化。
2. 以1:50比例(200ul),将活化的过夜培养物加入10mL LB液体培养基中,加入10uLAmp(100mg/ml),37o C200r/min摇床扩大培养2h-3h,期间取样监控菌液的OD值,控制菌液OD600在0.6-1.0之间,以使大肠杆菌处于最适合表达外源蛋白的生长状态。
(一般3h时,菌液浓度及达到标准,但是不同的基因对菌的影响不同,所以第一次实验时需要确定这个最佳时间)3. 从10ml扩大培养物中取3ml菌液作为不加IPTG的空白对照(CK),其余7ml菌液加入7ul IPTG(储存浓度为0.5mol/l),使IPTG 终浓度达到0.5mmol/l。
以200r/min的转速,37o C摇床培养3h。
4. 以5000r/min离心2min收集菌体,倾倒上清,每个离心管收集3ml培养物。
5. 加入1ml dH2O,将管底沉淀用振荡器打散以充分洗涤,8000r/min 离心2min,倾倒上清。
6. 重复步骤5。
将离心管中的水倒干净。
(二)菌落SDS-PAGE1. 在收集的菌体中加入200ul 1×SDS PAGE loading buffer(可根据沉淀的量增加或减少loading buffer的量,一般200ul比较合适)。
用漩涡器剧烈震荡,确保将管底沉淀震散。
2. 将样品于100℃恒温加热器上开盖加热10min(Marker也要加热)。
样品凉后,12000r/min离心3min,取每管的上清点样。
上样量一般30ul—40ul,marker 20ul。
(3)SDS-PAGE分析1. 根据目的片段的大小,制作不同浓度的分离胶蛋白分子量 (kDa)凝胶浓度 (%)4-402012-451510-701215-1001025-2008分离胶(两块)配方15%12%8% ddH2O 3.4ml 4.9ml 6.9ml 30% Acr-Bis (29:1)7.5ml 6.0ml 4.0ml1.5M Tris-HCl( pH8.8) 3.8ml 3.8ml 3.8ml10%SDS150ul150ul150ul10%过硫酸铵150ul150ul150ulTEMED20ul20ul20ul总体积15ml15ml15ml按表中所列顺序从上到下加试剂,加完TEMED后,轻轻摇匀液体,一块胶加7ml溶液。
加完后, 用1mLddH2O封住分离胶液面,在室温(37℃)凝结30min,至分离胶与水之间出现一条清亮的分界线,倾倒水层,用滤纸条将残余的水吸干。
2.制作浓缩胶浓缩胶(两块)配方5%ddH2O 3.4 mL30% Acr-Bis (29:1)0.85 mL1M Tris-HCl( pH6.8)0.63 mL10%SDS50 uL10%过硫酸铵50 uLTEMED20 uL总体积5ml胶配好后混匀,灌胶2ml,插梳子时根据点样量和浓缩胶长短调整梳子插入的深度,于室温(37℃)凝胶30min。
3.胶凝好后,拔梳子,将胶放入电泳槽中,加入1×Tris-Gly电泳缓冲液没过点样孔,胶外侧缓冲液界面应完全没过电极4.Marker点5uL,用10uL小枪点样,以防样品冲出点样孔与旁边样品混杂。
电压打到70V,保证电流在20mA-30mA之间,当样品跑入浓缩胶时可适当调高电压,但不宜多次调整电压,否则条带会跑歪。
5.当溴酚蓝电泳至胶槽底部时,一般需要2.5h,停止电泳,剥胶,将浓缩胶部分切除后,加入考马斯亮蓝染色液,40r/min染色3h,可以过夜染色。
6.用移液器回收考马斯亮蓝染色液,再用蒸馏水将浮色冲洗干净后,倒入考马斯亮蓝脱色液,40r/min脱色至胶背景透明,期间可每2h更换一次脱色夜。
根据菌落SDS-PAGE的结果,可以看到目的蛋白是否大量表达。
2. 鉴定目的蛋白形成的是包涵体还是可溶性蛋白1. 从冻存的菌液中取10ul,接种于5ml的LB培养基中,加入5ulAmp(100 mg/ml),过夜活化菌株。
2. 按照1:50的比例,从活化的菌株中,取1ml菌液接种于50mlLB培养基中,加入50ul Amp(100 mg/ml),扩大培养3 h。
3. 扩大培养后的菌液,取出10ml,不加IPTG,作为空白对照。
剩余的40ml菌液中加入40ul IPTG(0.5mol/l),诱导蛋白表达。
两者在37℃,200rpm的条件下培养3h。
4. 用50ml 离心管收集菌体,8000rpm,离心3min。
注意配平。
5. 倒掉上清,加入40ml左右的dH2O洗菌体一遍,12000 rpm,离心2min。
注意要将菌体充分打散。
6. 倒掉上清,用枪头将上清吸干净。
根据菌体的浓度,加入适量的PBS buffe r 悬浮菌体。
7. 用大功率的超声波将菌体破碎,其间超声波每工作2min,停止1min。
菌体要始终保持在冰水混合物上,以保证低温,蛋白不易变性。
如此反复6-10遍,直至菌液清澈。
8. 12000rpm, 离心3min,分离上清和沉淀。
9. 在沉淀中加入250ul 1×SDS Loading buffer,取200u l 上清,加入50ul 5×SDS Loading buffer,在恒温加热器上100℃加热10min。
10.加热后的样品冷却到室温后,12000rpm,离心2min。
11.取上清40u l 点样,按照一中的方法进行SDS-PAGE检测。
点样顺序是:对照的上清,沉淀,样品的上清,沉淀。
12.根据SDS-PAGE结果,判断目的蛋白是以包涵体还是以可溶性蛋白的形式产生。
如果蛋白在沉淀中大量表达,则形成包涵体,需要改变诱导表达条件,比如将扩大培养与诱导培养的温度改为25℃,并延长相应培养时间。
如果蛋白在上清中表达,则形成可溶性蛋白,可继续进行下一步实验。
3. 纯化目的蛋白(整个过程要尽量保持在4℃进行)1. 大量诱导目的蛋白(一般需要2-4 L的菌量),用超声波破碎细胞,收集上清。
2. GST纯化柱的柱床保存在20%的乙醇中,使用之前,先用吸管将柱床轻轻加入到柱子中,打开柱子,流出乙醇,使柱床沉积下来。
3. 加入10ml PBS buffer,洗柱床3遍,使得柱床重新平衡。
4. 将准备好的样品加入到柱子中,每次加入5-10ml样品,使样品结合在柱子上(每次上样量不能太多,因为柱子的结合能力有限)。
5. 用PBS buffer洗柱子3遍,每次用10ml,去除柱子中非特异性结合的杂蛋白。
6.7.8. 用分光光度计测每一管的OD值,记录下读数,将读数大于200的样品留下(一般是第2至第4管),冻于-20℃冰箱中。
9. 重复步骤4-8,直至所有样品均纯化完毕。
10.可以用SDS-PAGE检测纯化出来的样品浓度以及纯度。
重生柱子的方法:(柱子使用三四次之后,需要重生,即让柱子上的GST结合基团重新暴露出来)1.用还原型谷胱甘肽洗脱液洗柱子3遍,每次加入10ml。
2. 用 PBS缓冲液洗柱子4遍,每次加入10ml。
3. 用50 mmol Tris HCl(pH 8.0)洗柱子3遍,每次加入10ml。
4.用0.5M NaCl(pH 8.0)洗柱子3遍,每次加入10ml。
5. 用100 mM醋酸钠(pH 4.5)洗柱子3遍,每次加入10ml。
6. 用0.5M NaCL洗柱子3遍,每次加入10ml。
柱子长时间不用,需要按照以下方式清洗及保存:1. 按照重生的1-6步骤先将柱子重生一遍。
2. PBS洗柱子2遍,每次加入10ml。
3. 将柱床用吸管吸出,分别用5%,10%,15%,20%的乙醇洗2遍。
4. 用单蒸水轻轻冲洗柱子底部的膜,洗出杂蛋白。
5. 柱床保存在含20%乙醇的PA瓶中。
柱子和柱床置于4度冰箱保存。
如果柱子用于纯化不同的蛋白, 只需要按照重生方法的1,2步骤操作即可,然后直接纯化另外一个蛋白。
如果柱子流速很慢,可能是柱子底部的膜被杂蛋白堵住了。
柱子重生之后,将柱床用吸管吸出,然后用单蒸水将柱子底部的膜轻轻冲洗,再将柱床加入到柱子中,重新上样。
如果柱子需要重生,请用完柱子的同学自觉完成这份工作,以免影响下一位同学的使用。
4. 根据不同的用途浓缩蛋白1. 若蛋白用于制作抗体如果洗脱液用的是tris-bufferr配制的,需要用1/4的PBS buffer进行透析,然后在冷冻干燥机中冻干浓缩,因为公司最终需要将目的蛋白溶于PBS buffer中。
如果洗脱液是用PBS buffer配制的,则直接在冷冻干燥机中冻干浓缩。
GST 纯化柱手册上建议的是用tris-bufferr配制洗脱液,但用PBS buffer配制的洗脱液可以将目的蛋白洗脱下来。
可自己选用。
2. 若蛋白用于活性检测由于蛋白在tris-bufferr和PBS buffer中,均能保持活性,所以不论用哪种,都不需要透析,直接浓缩干燥即可。
