发酵来源的化学药物的菌种鉴别
发酵工程菌种的来源及选育

细菌分离:以乳酸菌为例
放线菌的分离
(一)采样
菜园土或林地土
土壤中放线菌最丰富,品种齐全。 可以筛选新的放线菌。
从堆肥或过热的材料中如干 草或蔗渣中可分离到大量的嗜热 放线菌,从淡水和海洋环境中分 离到嗜碱性的和嗜酸性的菌种。
自然风干,备用
气生孢子能耐干燥,耐湿热能力 高于营养体,室温保存20天, 放线菌的数量和组成变化不大, 细菌大部分死亡。
(二)生态学参数及培养基的组成原则
1、加入培养基中的天然提取物种类和用量、环境生物物 理学参数以及用于平板涂布分离样本的溶剂都会影响 实验中所要分离的细菌的数量和种类。
2、就分离培养基的组成而言,部分培养基中必须含有 10-50%的天然提取物。加入培养基中的天然提取物, 部分培养基中则应含有多种碳、氮源,如几丁质、纤 维素或果胶。
随机分离原理与技术
从产物入手,通过设计高产培养基和建立快速 灵敏专一的筛选方法,从随机分离的菌落中筛 选出所需的目的菌。
技术关键:产物合成条件的选择与控制及相应 筛选方法的确定。
随机分离技术举例
药理活性化合物产生菌的筛选
药理活性化合物是指能抑制人类代谢中某一 个关键酶的微生物产物即酶抑制剂,从而达到 治疗的目的。
样本采集时所需的工具通常有无菌刮铲、土样采集器、 镊子、解剖刀、手套、无菌小塑料袋和塑料瓶等。
采样的注意事项
1、采样时应尽可能保持相对无菌; 2、所采集的样本必须具有某种代表性; 3、采好的样必须完整地标上样本的种类及采集日期、
地点以及采集地点的地理、生态参数等; 4、应充分考虑采样的季节性和时间因素,因为真正的
(2)化学法
通过培养基中添加某些化学成分来增加微生物的数量。 如:用添加CaCO3稳定培养基的PH来分离嗜碱性的放线菌。
酸奶菌种的分离及鉴定(5篇)

酸奶菌种的分离及鉴定(5篇)第一篇:酸奶菌种的分离及鉴定酸奶菌种的分离及鉴定乳酸菌是指一群通过发酵糖类,产生大量乳酸的细菌总称。
乳酸从形态上可分为球菌和杆菌,并且均为革兰氏染色阳性、在缺少氧气的环境中生长良好的兼性厌氧性或厌氧性细菌。
目前,对乳酸菌的应用研究,着重于食品(如发酵乳制品、发酵肉制品和泡菜)和医药工业等人类生活密切相关的领域。
目前市售的各种酸奶制品中, 作为发酵剂的乳酸菌, 通常为保加利亚乳杆菌和嗜热链球菌这两株菌。
用嗜热链球菌和保加利亚乳酸杆菌混合培养发酵的乳酸饮品能补充人体肠道内的有益菌,维持肠道的微生态平衡,且含有易于吸收的营养素,具有抑制腐败菌、提高消化率、防癌及预防一些传染病等功效,并能为食品提供芳香风味,使食品拥有良好的质地。
保加利亚乳杆菌(L.Bulgarius):长杆形,直径1-3mm左右,能产生大量的乳酸。
酸碱度方面,为耐酸或嗜酸性,因低pH能防止一些微生物的生长;温度方面,为嗜温至少许嗜热,最适生长温度在37-45℃之间,对低温非常敏感。
嗜热链球菌(S.thermophilus):卵圆形,直径0.7-0.9微米,呈对或链状排列,无运动性。
为健康人肠道正常菌群,可在人体肠道中生长、繁殖。
可直接补充人体正常生理细菌,调整肠道菌群平衡,抑制并清除肠道中对人具有潜在危害的细菌。
本研究对市售主要品牌酸奶中(河南花花乳业生产的酸奶)乳酸菌进行了分离鉴定,并进一步探讨制备酸奶条件(温度、时间等),以达到最佳的天然酸奶质量效果。
一、实验内容(1)乳酸菌的分离纯化1.无菌操作倒平板、十倍稀释、划线分离,恒温培养2.菌落观察与镜检 3.筛选生产用菌株(2)优化酸奶制作条件1.制备发酵液2.不同条件下,接种发酵菌剂并发酵生产 3.观察发酵情况4.品尝发酵产品,进行质量评价 5.记录结果二、实验器材1)菌种:新鲜乳酸饮料(标记只含有保加利亚乳杆菌和嗜热链球菌)2)试剂:脱脂奶粉、蔗糖、1.6%溴甲酚绿乙醇溶液(溴甲酚绿、无水乙醇)、酵母膏、琼脂、革兰氏染液(结晶紫染液、卢戈氏碘液、95%乙醇、沙黄)、75%乙醇、香柏油、1mol/L NaOH、1mol/L HCl、碳酸钙;0.4gNaOH固体、4.2ml浓HCL(分析纯)、20gCaCO3固体、酵母膏20g、琼脂30g香柏油、脱脂奶粉100g、蔗糖10g;3)仪器:高压蒸汽灭菌锅、恒压干热灭菌箱、超净工作台、光学显微镜、培养箱、pH试纸、酸乳瓶、培养皿(φ9或φ12)、试管、300ml三角瓶(带玻珠)、移液管、天平、牛角匙、电炉、量筒、漏斗、漏斗架、玻璃棒、棉塞、吸管、线绳、标签、500ml锥形瓶、250ml锥形瓶、250ml烧杯、酒精灯、石棉网、接种针(环)、擦镜纸四、实验方法4.1乳酸菌的分离纯化 4.1.1分离(1)配制BCG牛乳培养基,分装三角瓶,包扎,灭菌备用。
菌种鉴定方法及步骤

菌种鉴定方法及步骤菌种鉴定是指通过对菌株样本进行系统学、形态学、生理学和生化学等各方面的观察与检测,以确定其属、种分类地位的过程。
菌种鉴定方法及步骤如下:2.制备纯培养物:分离并得到纯培养菌株,避免混杂的现象。
3.形态学观察:观察菌株的形态特征,包括菌落形态、菌落颜色、菌丝生长特征等。
4.微观形态学观察:观察菌株的细胞形态、孢子形态、菌丝形态等。
5.分类地位确定:通过对菌株的形态特征进行比对和研究,确定其属、种分类的地位。
1.生长条件观察:观察菌株在不同的生理因素(如温度、pH值、氧气含量等)下的生长情况。
2.营养需求观察:观察菌株在不同营养物质(如碳源、氮源等)的利用情况,以及对不同抗生素和化学物质的敏感性。
3.生化特征检测:进行生化试验,如氧化酶试验、氧化发酵试验、内酯酶试验等,以检测菌株的生化特征。
4.产物检测:检测菌株的代谢产物,如抗生素、酶等,以判断其代谢途径和功能。
1.提取菌株基因组DNA:通过细菌培养物提取菌株的基因组DNA。
2.PCR扩增:使用特异性引物进行PCR扩增,选择适当的靶基因进行扩增。
3.DNA测序及分析:对扩增的DNA产物进行测序,利用生物信息学方法对测序结果进行分析和比对。
4.基因序列比对:将测序结果与数据库中已知菌种的基因序列进行比对,以确定菌株的分类地位。
四、传统鉴定方法的优缺点1.优点:传统鉴定方法操作简单,成本低廉;不需要复杂设备,适用于一般实验室进行菌株鉴定。
2.缺点:传统鉴定方法对菌株鉴定仅限于形态学、生理学和生化学等方面的观察,鉴定结果存在一定的主观性和局限性;需要较长的时间才能得到鉴定结果。
五、分子生物学鉴定方法的优势1.高精确性:分子生物学鉴定方法通过对菌株的基因序列进行比对,可得到较高的精确性,减少了主观性和局限性。
2.快速性:分子生物学鉴定方法在提取DNA和PCR扩增等步骤后,可以迅速得到鉴定结果,节省了时间。
3.可靠性:分子生物学鉴定方法的结果具有较高的可靠性,有助于解决传统鉴定方法在分类地位判断方面的困难。
菌种鉴定标准操作规程

菌种鉴定标准操作规程目的:建立菌种鉴定标准操作规程范围:适用于******鉴定标准操作职责:内容:1整体操作步骤1.1纯化、分离待鉴定菌用接种环挑取待鉴定菌落或少许含菌溶液到相应的培养基上(如TSA),划线分离,以出现微生物单菌落为止。
1.2挑取纯化后的单菌落,进行革兰染色、镜检,以确定待鉴定菌的革兰属性。
1.3如镜检结果为革兰阴性杆菌,则先进行发酵实验。
如发酵实验结果为阴性,则选用API20NE试剂条进行鉴定;如发酵实验结果为阳性,则在经过细胞色素氧化酶实验后选用API20E试剂条进行鉴定。
1.4如镜检结果为革兰阳性球菌,则先进行过氧化氢酶实验。
如实验结果为阴性则选用API Strep试剂条进行鉴定,如实验结果为阳性则选用API Staph试剂条进行鉴定。
1.5假如镜检结果为革兰阳性杆菌并且有内生芽胞,则选用API 50CHB试剂条进行鉴定;否则,根据运动性实验和过氧化氢酶实验结果选用API Coryne或其他试剂条进行鉴定。
1.6选定正确的试剂条以后,根据不同的API试剂条的标准操作规程准备接种物,进行接种、培养等操作并将结果形成数码,用API鉴定软件鉴定或查询待检菌的名称均可。
鉴定结果应记录,必要时,给出相应解释。
2革兰氏染色2.1染色剂的配制2.1.1结晶紫染色液:甲液:结晶紫 1.0g95%的酒精20ml乙液:草酸铵0.8g水80ml将甲液和乙液混合均匀,静置48h使用,置密闭棕色瓶中储存。
2.1.2碘液:碘 1.0g碘化钾 2.0g水300ml配制时先用3~5ml水将碘化钾溶解,再加入碘,用力摇匀,使之全部溶解后,再加水稀释至300ml,摇匀,分装在棕色瓶中储存。
2.1.3稀石碳酸红溶液:碱性品红 1.0g石碳酸 5.0ml95%乙醇10.0ml配制时用乙醇溶解碱性品红,然后加入石碳酸溶液,使用时加水稀释至100ml,摇匀。
2.2操作:2.2.1涂片:用接种环挑取液体培养物中菌体在载玻片上涂成薄层,固体培养物则先在载玻片上滴一滴蒸馏水或生理盐水,用接种环挑取少量菌体在载玻片上涂成薄层。
发酵大肠杆菌染杂菌鉴定方法

发酵大肠杆菌染杂菌鉴定方法(原创实用版3篇)《发酵大肠杆菌染杂菌鉴定方法》篇1发酵大肠杆菌染杂菌的鉴定方法通常包括以下步骤:1. 采集样本:采集发酵大肠杆菌染杂菌的样本,例如发酵液或菌落,进行检测。
2. 分离和纯化:将样本中的杂菌分离出来,并进行纯化培养,得到单个菌落。
3. 鉴定菌种:通过菌落形态、生长特性、生化反应等方法,对分离出来的单个菌落进行鉴定,确定其菌种。
4. 检测发酵大肠杆菌的活性:通过检测发酵大肠杆菌的代谢产物、酶活性等指标,确定其活性是否受到杂菌的影响。
常用的检测方法包括微生物学检测方法、分子生物学检测方法等。
《发酵大肠杆菌染杂菌鉴定方法》篇2发酵大肠杆菌染杂菌的鉴定方法通常包括以下步骤:1. 菌落形态观察:将发酵大肠杆菌染杂菌样本涂布在含有伊红美蓝指示剂的培养基上,菌落呈深紫色,并有金属光泽。
2. 涂片镜检:将发酵大肠杆菌染杂菌样本涂片,用荧光显微镜观察,可以看到菌体周围有弯曲的菌毛和鞭毛。
3. 培养特性检测:将发酵大肠杆菌染杂菌样本分别接种在含不同糖类的培养基上,观察其生长情况。
发酵大肠杆菌能在含有乳糖、蔗糖、麦芽糖等糖类的培养基上生长,而其他细菌则不能。
4. 生化反应测试:将发酵大肠杆菌染杂菌样本分别进行糖类发酵试验、靛基质试验、脲酶试验等生化反应测试,根据反应结果进行鉴定。
5. 16S rRNA 基因序列分析:对于难以通过上述方法鉴定的菌株,可以进行16S rRNA 基因序列分析,通过比对序列确定其属于哪一类菌株。
《发酵大肠杆菌染杂菌鉴定方法》篇3发酵大肠杆菌染杂菌的鉴定方法通常包括以下步骤:1. 菌落形态观察:将发酵大肠杆菌样品涂布在含有伊红美蓝指示剂的培养基上,菌落呈深紫色,并有金属光泽。
而杂菌菌落则呈淡紫色或无色,无金属光泽。
2. 涂片镜检:将发酵大肠杆菌样品涂布在载玻片上,加入适量的生理盐水,盖上盖玻片,在显微镜下观察。
发酵大肠杆菌的细胞呈短杆状,两端钝圆,单个存在。
发酵食品中乳酸菌的菌种鉴定与优化

发酵食品中乳酸菌的菌种鉴定与优化发酵食品已经成为人们餐桌上不可缺少的一部分,而乳酸菌作为发酵的关键成分,在其中发挥着重要的作用。
乳酸菌不仅可以增加食品的口感和风味,还能提供人体所需的益生菌,对于维持肠道健康和促进免疫系统功能有益处。
因此,对发酵食品中乳酸菌的菌种鉴定与优化显得尤为重要。
菌种鉴定是发酵食品生产中必不可少的一环。
通过准确鉴定乳酸菌的菌种,可以确保产品的质量和安全性。
传统的菌种鉴定方法主要依赖于形态学和生理学特征,如形状、色素、生长温度等。
然而,这些方法存在一定的局限性,不仅需要耗费大量时间和人力,而且对技术要求较高。
因此,现代的分子生物学方法逐渐被应用到菌种鉴定中。
PCR(聚合酶链式反应)是一种常用的分子生物学技术,可以准确鉴定乳酸菌的菌种。
通过设计特异性引物,可以从菌落或食品样品中扩增目标DNA片段,并利用电泳技术测定DNA的迁移速率,从而确定乳酸菌的菌种。
此外,最近兴起的高通量测序技术也为菌种鉴定带来了革命性的突破。
通过对乳酸菌样品进行DNA提取、测序,再与已知菌种的DNA序列进行比对,就可以快速准确地鉴定乳酸菌的菌种。
除了鉴定菌种,优化乳酸菌的生长条件也是提高发酵食品品质的重要环节。
乳酸菌对温度、pH值等环境因素敏感,不同菌种对环境的适应性也不同,因此合理的生长条件可以促进乳酸菌的生长和代谢产物的形成。
目前,常用的优化方法主要包括传统的单因素实验和统计学方法。
传统的单因素实验通过改变一个因素,如温度或酸碱度,来观察其对乳酸菌生长的影响。
这种方法简单直观,容易掌握,但繁琐且耗时。
与之相对的是统计学方法,如响应面法和正交试验设计。
这种方法能够同时考虑多个因素之间的相互作用和影响程度,从而找到最优条件。
通过这些方法,可以优化乳酸菌的生长条件,提高其存活率和代谢产物的质量。
发酵食品中乳酸菌的菌种鉴定与优化是一个综合性的过程,需要借助于多种技术和方法。
通过科学合理的菌种鉴定和生长条件优化,可以提高发酵食品的质量和产品的市场竞争力。
发酵工程设计菌种筛选方案

发酵工程设计菌种筛选方案第一步:确定目标产物在菌种筛选之前,首先需要确定目标产物是什么。
这个产物可以是食品添加剂、药物、化学品等。
确定目标产物后,可以确定需要的菌种类型,例如革兰氏阳性菌、革兰氏阴性菌、真菌等。
此外,还需要确定产物的生产条件,例如温度、pH值、氧气需求等。
第二步:收集菌种资源在进行菌种筛选之前,需要先收集各种潜在的菌种资源。
这些资源可以通过从自然环境中进行采集、从已有的菌种库中获取或者通过其他方式获取。
一般来说,我们需要收集大量的菌株资源,以便进行后续的筛选工作。
第三步:菌种初步筛选在收集到菌株资源后,需要进行初步的筛选。
这一阶段的目标是快速地排除那些不太可能产生目标产物的菌株。
菌株的筛选可以通过观察菌落形态、生长速度、色素产生等方法进行。
第四步:菌种培养条件的优化在进行初步筛选后,我们需要对筛选出来的菌株进行培养条件的优化。
这包括温度、pH 值、培养基成分等。
通过优化培养条件,可以进一步筛选出对目标产物生产更为适合的菌种。
第五步:菌种产量的筛选在确定了合适的菌种后,需要进行产量的筛选工作。
这一阶段的目标是确定哪些菌株在相同的培养条件下可以产生最高的目标产物产量。
这一步通常需要进行大规模的培养实验。
第六步:菌种稳定性的筛选最后,需要对筛选出来的菌株进行稳定性的评估。
确定哪个菌株在不同培养条件下都能稳定地产生目标产物。
这一步通常需要进行长期的培养实验。
综上所述,菌种的筛选是一个复杂而又关键的工作。
通过以上的步骤,可以有效地筛选出适合生产目标产物的菌株,为发酵工程的成功提供保障。
臭豆腐发酵菌种的筛选与鉴定

246 2007, Vol. 28, No. 06 食品科学※生物工程臭豆腐发酵菌种的筛选与鉴定卢义伯,潘超*,祝义亮(南京雨润集团技术中心,江苏南京 210041)摘要:采用定性培养和定量培养的方法,从臭豆腐发酵乳液中分离筛选出优势菌种,并采用菌种鉴别试纸条进行鉴定。
分析表明,荧光假单孢菌是臭豆腐发酵过程中的优势菌种,霉菌在臭豆腐发酵乳液中数量远小于细菌,实验结果为臭豆腐的工业化生产提供了理论依据。
关键词:臭豆腐;菌群筛选;鉴定Bacterium Screening and Identifying from Stinky TofuLU Yi-bo,PAN Chao*,ZHU Yi-liang(Technical Center of Nanjing Yurun Group, Nanjing 210041, China)Abstract:Dominant bacterium is separated from stinky tofu qualitatively and quantitatively, identified with indicator paper.It was concluded that pseudomonas (PS.fluo./putida) is the dominant bacterium but amount of mould is relatively small. This assayaffords the theoretical basics for industrial production of stinky tofu.Key words:stinky tofu;bacterium screening;identification中图分类号:Q93-331 文献标识码:A 文章编号:1002-6630(2007)06-0246-04臭豆腐是南京秦淮特色风味小吃,同时也是中国人臭豆腐发酵乳液取自农家制作的臭豆腐发酵乳民的传统美食,受到广大青少年的青睐。
乳酸菌的鉴定
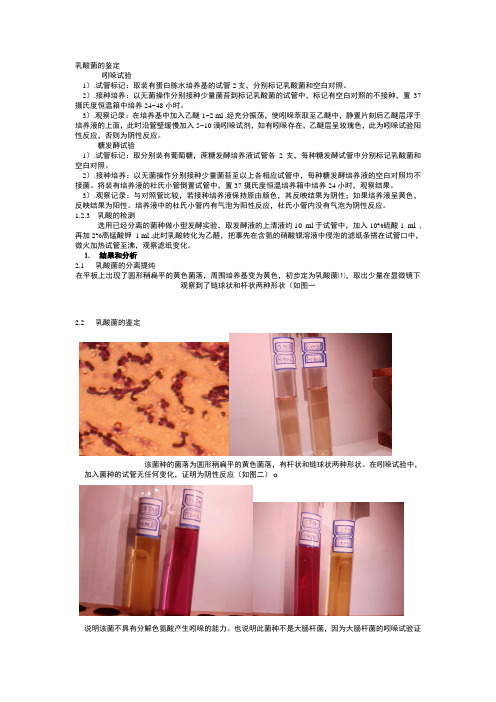
乳酸菌的鉴定吲哚试验1).试管标记:取装有蛋白胨水培养基的试管2支,分别标记乳酸菌和空白对照。
2).接种培养:以无菌操作分别接种少量菌苔到标记乳酸菌的试管中,标记有空白对照的不接种,置37摄氏度恒温箱中培养24~48小时。
3).观察记录:在培养基中加入乙醚1~2 ml ,经充分振荡,使吲哚萃取至乙醚中,静置片刻后乙醚层浮于培养液的上面,此时沿管壁缓慢加入5~10滴吲哚试剂,如有吲哚存在,乙醚层呈玫瑰色,此为吲哚试验阳性反应,否则为阴性反应。
糖发酵试验1).试管标记:取分别装有葡萄糖,蔗糖发酵培养液试管各2支,每种糖发酵试管中分别标记乳酸菌和空白对照。
2).接种培养:以无菌操作分别接种少量菌苔至以上各相应试管中,每种糖发酵培养液的空白对照均不接菌。
将装有培养液的杜氏小管倒置试管中,置37摄氏度恒温培养箱中培养24小时,观察结果。
3).观察记录:与对照管比较,若接种培养液保持原由颜色,其反映结果为阴性;如果培养液呈黄色,反映结果为阳性。
培养液中的杜氏小管内有气泡为阳性反应,杜氏小管内没有气泡为阴性反应。
1.2.3 乳酸的检测选用已经分离的菌种做小型发酵实验,取发酵液的上清液约10 ml于试管中,加入10%硫酸1 ml ,再加2%高锰酸钾 1 ml ,此时乳酸转化为乙醛,把事先在含氨的硝酸银溶液中侵泡的滤纸条搭在试管口中,微火加热试管至沸,观察滤纸变化。
1.结果和分析2.1 乳酸菌的分离提纯在平板上出现了圆形稍扁平的黄色菌落,周围培养基变为黄色,初步定为乳酸菌⑴,取出少量在显微镜下观察到了链球状和杆状两种形状(如图一2.2乳酸菌的鉴定该菌种的菌落为圆形稍扁平的黄色菌落,有杆状和链球状两种形状。
在吲哚试验中,加入菌种的试管无任何变化,证明为阴性反应(如图二)。
说明该菌不具有分解色氨酸产生吲哚的能力。
也说明此菌种不是大肠杆菌,因为大肠杆菌的吲哚试验证明为阳性。
在糖发酵试验中,在加入葡萄糖的发酵培养液中加入菌种后,培养液变为黄色,反应结果为阳性,且杜氏小管内无气泡(如图三);加入蔗糖的发酵培养液中加入菌种后的反应结果也为阳性且杜氏小管内无气泡(如图四)。
发酵工程(菌种选育)重点

尽管通过增殖培养效果显著,但还是处于微生物的 混杂生长状态。因此还必须分离,纯化,在固体培 养基平板上获得单菌落。 纯种分离的方法有划线分离法、稀释分离法。
分离的效率取决于培养基养分、pH、温度和 加入的选择性抑制剂。 嗜中性放线菌分离培养基pH常在6.7~7.5, 嗜酸性放线菌pH降至4.5~5.0。 放线菌筛选时,可加入真菌抗生素,对放线 菌无作用。 分离放线菌分离平板通常在25~30℃培养。 嗜热菌45~55 ℃ ,嗜冷菌4~10 ℃ 。
当代并不表现出来, 在筛选时就会被淘汰; 若突变性状是显
性的, 那么, 在当代就表现出来, 但在进一步传代后, 就会
出现分离现象, 造成生产性状衰退, 因此,应尽可能选择孢
子或单倍体的细胞作为诱变对象。
即使如此, 有时也会出现表型延迟现象, 这是因为诱变
剂往往只作用于DNA分子的一条单链,DNA 进一步复制后,
实例:碱性纤维素酶产生菌的筛选 文献:产生菌为中性牙孢杆菌,嗜碱牙孢杆菌、放线菌及霉菌 →80℃、30min处理 ↓ 0.0075%曲利本蓝+1%CMC(羧甲基纤维素),pH10.5、 培养3~4天,选择有凹陷圈的菌落 采样(造纸 厂) 26株为组成型 从285个土样中获得62株 36株为诱导型
菌种鉴定方法及手段 真菌细菌检测

菌种鉴定方法及手段真菌检测/细菌检测菌种鉴定一般就只要提取基因组DNA,然后PCR扩增16srDNA片段,上GeneBank 或者Eztaxon对比即可。
一般序列相似度在97%以上就可以认为是同种细菌。
常规鉴定常规鉴定内容有形态特征和理化特性。
形态特征包括显微形态和培养特征;理化特性包括营养类型、碳氮源利用能力、各种代谢反应、酶反应和血清学反应等。
自动鉴定BIOLOG鉴定系统以微生物对不同碳源的利用情况为基础,检测微生物的特征指纹图谱,建立与微生物种类相对应的数据库。
通过软件将待测微生物与数据库参比,得出鉴定结果。
该系统已获美国FDA认可,已逐步应用于食品和饮品企业、环保、海洋生物/水产品、制药、农业微生物、生物治理、化妆品、临床等领域的微生物鉴定试验中。
拥有国内最全的BIOLOG数据库,涉及革兰氏阴性菌、革兰氏阳性菌、厌氧菌、酵母、丝状真菌在内近2000种微生物。
分子生物学鉴定应用分子生物学方法从遗传进化角度阐明微生物种群之间的分类学关系,是微生物分类学研究普遍采用的鉴定方法。
科标生物检测中心拥有微生物菌种分类鉴定的分子生物学实验室,配有PCR仪、高速冷冻离心机、电泳仪、HPLC、凝胶成像系统、紫外控温分析系统等先进仪器设备,以及DNAMAN、BIOEDIT、CLUSTALX、TREEVIEW等序列分析软件。
可采用核酸序列分析法分析细菌16S rDNA/16S-23S rDNA区间序列、酵母18S rDNA/26S rDNA(D1/D2)序列及丝状真菌的18S rDNA/ITS1-5.8S-ITS2序列,提供科学的鉴定结果。
API细菌鉴定API鉴定系统涵盖15个鉴定系列,约有1000种生化反应,已可鉴定超过600种的细菌。
鉴定过程中,可根据细菌所属类群选择适当的生理生化鉴定系列,通过软件将待测细菌与数据库参比,得出鉴定结果。
可应用API50CH系列、API20E系列、API Staph系列对乳酸杆菌(Lactobacillus sp.)和相关细菌、芽孢杆菌(Bacillus sp.)、葡萄球菌属(Staphylococcus sp.)、微球菌属(Micrococcus sp.)和库克菌属(Locuria sp.)进行鉴定。
乳酸菌生化鉴定条

乳酸菌生化鉴定条一、糖发酵实验糖发酵实验是鉴定乳酸菌的重要生化反应之一。
不同的乳酸菌对糖的发酵能力不同,通过观察糖发酵过程中的产酸和产气情况,可以鉴别乳酸菌的种类。
常用的糖类包括葡萄糖、乳糖、果糖等。
二、氧化发酵实验氧化发酵实验是鉴定乳酸菌的另一种生化反应。
在缺氧条件下,乳酸菌能够利用丙酮酸氧化产能,生成乳酸和其他代谢产物。
通过观察氧化发酵过程中的产酸和产气情况,可以鉴别乳酸菌的种类。
三、乙酰甲基甲醇实验乙酰甲基甲醇实验是鉴定乳酸菌的一种特殊生化反应。
在酸性条件下,某些乳酸菌能够将乙酰甲基甲醇氧化成乙酸,同时产生氢气。
通过观察反应过程中的颜色变化和气泡产生情况,可以鉴别乳酸菌的种类。
四、丙二酸实验丙二酸实验是鉴定乳酸菌的另一种特殊生化反应。
某些乳酸菌能够利用丙二酸作为碳源进行生长,同时产生二氧化碳。
通过观察反应过程中的二氧化碳产生情况,可以鉴别乳酸菌的种类。
五、产氨实验产氨实验是鉴定乳酸菌的一种生化反应。
某些乳酸菌能够利用氨基酸产生氨,通过观察反应过程中的颜色变化和气味变化,可以鉴别乳酸菌的种类。
六、磷酸酯酶实验磷酸酯酶实验是鉴定乳酸菌的一种生化反应。
某些乳酸菌能够产生磷酸酯酶,分解磷酸酯类物质,产生磷酸盐和有机酸。
通过观察反应过程中的颜色变化和沉淀产生情况,可以鉴别乳酸菌的种类。
七、吲哚实验吲哚实验是鉴定乳酸菌的一种生化反应。
某些乳酸菌能够产生吲哚酶,分解吲哚类物质,产生靛蓝色素。
通过观察反应过程中的颜色变化,可以鉴别乳酸菌的种类。
八、甲基红实验甲基红实验是鉴定乳酸菌的一种生化反应。
某些乳酸菌能够将葡萄糖转化为乳酸,在酸性条件下,葡萄糖分解成乙醇和二氧化碳。
通过观察反应过程中的颜色变化和气泡产生情况,可以鉴别乳酸菌的种类。
九、伏-普实验伏-普实验是鉴定乳酸菌的一种特殊生化反应。
在葡萄糖和乳糖的存在下,某些乳酸菌能够将乳糖分解成葡萄糖和半乳糖,通过观察反应过程中的颜色变化和沉淀产生情况,可以鉴别乳酸菌的种类。
发酵工程与设备第二章、第二讲发酵工业菌种的分离筛选

透明圈法
在平板培养基中加入溶解性较差的底物,使培 养基混浊; 能分解底物的微生物便会在菌落周围产生透明 圈,圈的大小初步反应菌株利用底物的能力。 分离水解酶产生菌时较多采用,如蛋白酶、淀粉 酶、脂肪酶、核酸酶等;
例如用此法分离产生碱性蛋白酶的芽孢杆菌
土壤经巴氏消毒,以减少不产芽孢的微生物;然后铺在 pH8-9的琼脂培养基(含有均匀的不溶性蛋白质)表面; 碱性蛋白酶产生菌能消化平板上的不溶性蛋白质,产生 一透明圈。
若被检菌能分泌某些抑制菌生长的物质,如抗生素等, 便会在该菌落周围形成工具菌不能生长的抑菌圈
6、随机分离方法
有些微生物的产物对产生菌的筛选没有任何选择性好处, 因此常随机地分离所需菌种,为此发展了一些快速筛选方法 并归纳出高产培养基成分的选择准则如下:
1)制备一系列的培养基,其中有各种类型的养分成为 生长限制因素。
第三次平板分离
第三次原种斜面
复筛 不纯 第四次平板分离
第三次菌种保藏
第四次原种斜面
初步工艺条件摸索
再复筛
种子培养
1株3-5瓶 较优菌株
保藏及进一步做生产性能试 验或作为育种的出发菌株
某些必要试验和毒性试验
2、分离与筛选的设计要求
在筛选所需菌株时应考虑以下一些重要指标:
1、菌的营养特征 2、菌的生长温度
但作为初筛的手段是有意义的
变色圈法
对于一些不易产生透明圈产物的产生菌,可在底 物平板中加入指示剂或显色剂,使目的微生物菌落 周围呈现变色圈,从而能被快速鉴别出来;
如 筛选果胶酶产生菌
用含0.2%果胶为唯一碳源的培养基平板,对含微 生物样品进行分离,待菌落长成后,加入0.2%刚果 红溶液染色4h,具有分解果胶能力的菌落周围便会 出现绛红色水解圈。
风味水豆豉发酵菌种的筛选与鉴定

风味水豆豉发酵菌种的筛选与鉴定陈兴林;俞露;黄建敏【摘要】为给贵州省风味水豆豉工业化生产提供可选的生产菌株,从传统发酵的细菌型水豆豉中分离筛选适于豆豉生产的菌种,并对分离出的优势菌株进行产气筛选,优选出不产气的3株优势菌株,经鉴定发现3株菌株均为枯草芽孢杆菌.【期刊名称】《浙江农业科学》【年(卷),期】2017(058)002【总页数】3页(P275-276,279)【关键词】水豆豉;发酵;菌种;PCR;鉴定【作者】陈兴林;俞露;黄建敏【作者单位】贵州省质量技术监督局,贵州贵阳550012;贵州大学酿酒与食品工程学院,贵州贵阳550025;贵州大学酿酒与食品工程学院,贵州贵阳550025【正文语种】中文【中图分类】TS214.9豆豉古名“幽菽”,古代称大豆为“菽”,“幽”是指把大豆煮熟后幽闭发酵的意思[1],至秦朝始更名为豆豉。
豆豉是以黄豆为原料,经过微生物发酵制得的传统发酵食品,味道鲜美,既能调味又能入药,长期食用可开胃增食、祛风散寒、消积化滞[2]。
豆豉发酵中微生物起着至关重要的作用,孙森等[3]对天然制曲豆豉发酵过程中菌相的变化进行了研究,结果表明,优势菌群为乳酸菌、芽孢杆菌和酵母菌。
水豆豉是细菌型豆豉,是由黄豆经过细菌作用发酵而成的酿造调味食品,因含水量大,故产品呈湿态,在堆积发酵过程中发生水解作用产生一些特殊气味[4]。
水豆豉营养丰富,含有丰富的蛋白质,维生素和矿物质[4-7]。
日本的“纳豆”与我国的水豆豉是相似的发酵食品,它最早是在唐朝由鉴真和尚带到日本的。
豆豉起源于民间,植根于民间,发展于民间,它以特殊的工艺、丰富的营养、独特的风味深受广大消费者的喜爱,并越来越受到人们的重视。
鄯晋晓等[2]研究表明,细菌型豆豉中的主要菌种为枯草芽孢杆菌,它能形成大量淀粉酶和蛋白酶,是细菌型豆豉发酵中必不可少的菌种。
张建华[8]对自然发酵曲霉型豆豉曲中微生物的分布进行了研究,结果表明,自然发酵豆豉中主要的微生物菌群是细菌和霉菌。
药厂菌种鉴定

药厂菌种鉴定
药厂菌种鉴定是指在药品生产过程中对菌种进行鉴定的过程。
药厂常常需要使用菌种来制备药品,因此菌种的质量和纯度对药品的质量和效果具有重要影响。
药厂菌种鉴定通常包括以下几个步骤:
1. 菌种的保存:药厂会将常用的菌种保存在冰箱或液氮罐中,确保菌种的存活和保持纯度。
2. 菌种的分离与纯化:从保存的菌种中选取单一的菌落,通过连续传代培养,分离出纯种。
3. 微生物特性鉴定:通过观察菌落形态、生长特性、产酶能力等特征,结合显微镜观察菌体形态和结构,初步判断菌种。
4. 生理生化特性鉴定:通过对菌种进行生理和生化试验,如代谢产物分析、酶活性测定、抗生素敏感性测试等,进一步确定菌种。
5. 分子生物学鉴定:利用分子生物学方法,如PCR、DNA测序等,分析菌种的基因序列和遗传特征,准确鉴定菌种。
通过以上步骤的鉴定,药厂可以确保所使用的菌种符合质量标准,并且能够生产出高质量的药品。
同时,定期对保存的菌种进行鉴定和更新,也能保证菌种的活
力和纯度。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
发酵来源的化学药物的菌种鉴别
审评四部审评七室 张明平
通过发酵获得的化学药物同其他化学合成的药物一样,审评时应适用同样的技术要求。
但通过发酵获得的化学药物同纯化学合成的药物也存在不一样的特点,在应用以上原则时会有不同的具体要求。
在发酵的过程中,原本在化学合成中的一步或多步化学反应在菌体内,通过菌体的初级或次级代谢完成。
而目前尚无有效手段,可对这些代谢过程如调控化学反应一样进行精细的监控。
但可以通过对菌种,发酵条件和工艺过程的确认,间接的判断生产工艺的可行性。
为了降低这种间接的判断带来的药品质量方面的安全性风险。
因此研发单位应对菌种鉴别和发酵工艺进行深入研究;寻找药品质量与二者之间的直接联系,判断和控制影响药品质量的关键参数。
本文将根据文献调研的结果,结合审评工作的实践,对发酵来源的化学药物的菌种鉴别的技术要求谈一点自己的看法。
通过发酵得到的药物很多,大体可分为以下三类。
第一类是由生物整体或部分实体的药物;如菌苗、疫苗、类毒素等自动免疫制品;抗毒素、抗血清等被动免疫制品;诊断用菌液、血清、毒素、抗原、抗体等诊断制剂以及治疗用抗体制剂等,统称为生物制品。
该类制品不在本文讨论的范畴。
第二类为微生物初级代谢产物;如构成机体大分子骨架的氨基酸、核苷酸,和辅酶、酶的辅基、维生素等非机体构成物以及与物质代谢、能量代调有关的有机酸、醇类等。
第三类为来源于微生物次级代谢产物的药物;其中最著名的一类药物就是抗生素;其他还包括酶抑制剂与诱导剂,免疫调节剂与细胞功能调节剂、受体拮抗剂与激动剂以及具有其他药理活性的物质。
初级代谢产物和次级代谢产物的生产依赖于其生产菌种特异性的积累目标产物,因此原则上要求菌种鉴别的专属性越高越好;一般应当将生产菌株与同种不同株的其他菌株区分开。
对菌株进行鉴别提供形态特征、培养特征、生理生化特征、细胞壁化学成分四方面的资料。
此外还
可采用DNA 杂交法,16SrRNA 基因序列分析,RELP,RAPD等手段对对同一种属的不同菌株进行鉴别。
但因初级代谢产物主要是生长所必须的小分子化合物,代谢路径较明确,在各种生物中差异较小;因此对菌种鉴别可适当放宽技术要求。
如氨基酸生产菌的菌株多为某些营养缺陷型的菌株,因此结合形态特征资料和生理生化特征资料就可以较好的保证菌株的专属性。
次级代谢产物通常与微生物的基本生命活动无关,其合成过程要比初级代谢复杂得多;但从次级代谢产物中分离得到的有用的药物也要多得多。
次级代谢过程按照起始分子和反应模式可进行如下划分:I级反应,初级代谢产物被转化为次级代谢产物的中间体。
II级反应,主要是缩合,相近的小分子单元被聚合成大分子,为次级代谢的关键步骤。
III级反应,对次级代谢产物的基本结构进行各种修饰,常常导致产生一族的大量类似物。
由此可见,要保证得到正确的可作为药物的次级代谢产物,就应当保证在生产中使用了正确的菌种。
且由于次级代谢的复杂性,同种不同株的生产菌可能产生很多物理化学性质(但药效未必相同)非常相似的类似物,分离难度很大,因此必须在申报资料中提供详细的生产菌株鉴别资料。
否则产品的质量可控性无法保证。
值得一提的是,发酵来源的化学药物,尤其是分子结构较复杂的次级代谢产物,应该通过过程控制保证产品质量;因此在生产工艺的技术要求上,仿制药和创新药并无区别。
同样应当提交详细的菌种鉴别和生产工艺研究资料。
目前的菌种的分类检定主要依据细菌的形态学、代谢产物、酶活性和表面抗原等表型特征,这些方法能对菌种进行初步分类。
随着分子生物学的发展,出现了多种对细菌分类、鉴定的DNA 技术,它们的分类适用范围、辨别力、重复性、解释难易度和标准化各不相同。
以下参照文献,对各种菌种鉴别方法做简单介绍。
【1】G+ Cmo1 %含量的测定;不同细菌DNA 有不同G+ C 含量的平均值,同种细菌菌株间的差异不大于5 % ,同属间种的差异不超过15 %。
测定( G + C) mo1 %的方法主要为热变性法,其原理为:天然DNA 在一定
的离子浓度和pH 介质中不断加热变性时,随着碱基之间氢键的不断打开,互补双螺旋不断变成单链,导致核酸碱基在260 nm 紫外吸收明显增加,当双链完全变成单链后,紫外吸收停止增加,在热变性过程中,紫外吸收增加的中点值所对应的温度为热变性温度( Tm) 。
Tm 值就是通过升高温度使吸光度增加来测定的,细菌DNA 中G+ C 含量越高,其Tm 值也较高。
用此方法能将不同种的细菌区别开,操作较简单,但分辨力不高,不能对菌株进行区分。
【2】16SRNA 基因序列分析其原理为:以16SrRNA 模板,以一个或多个寡核苷酸链作引物,用反转录酶合成cDNA。
反应体系由4 支反应管组成,分别测定A、T、C、G 四种碱基。
每个反应管中都加有dNTP 和一种ddNTP ,ddNTP 随机加入到DNA 链中,通过改变dNTP 的浓度比例即可控制ddNTP 的掺入机会,即控制链的延长反应。
反应到预定时间后,4 支管的反应物同时在聚丙烯酰胺凝胶上进行电泳分离,电泳结果可以用放射自显影显示,再从自显影图谱上直接读出16SrRNA 序列。
特异性和灵敏度较好,可用于检定生产菌的种属,但不一定适用于同种不同株的生产菌鉴别。
【3】PFGE( Pulsed - field gel elect rophoresis) 脉冲电场凝胶电泳。
所有超过一定大小的线状DNA 分子在琼脂糖凝胶中的迁移速率相同。
当线状DNA 双链体的螺旋半径超过凝胶孔径时,即达到分辨力的极限,此时凝胶不能再按分子大小筛分DNA ,DNA象通过弯管一样,以其一端指向电场一级而通过凝胶。
这种迁移模式谓之“爬行”。
即使低浓度的琼脂糖也不能分辨大于750kb 的线状DNA 分子。
PFGE在凝胶上外加正交的交变脉冲电场。
每当电场方向改变后,大DNA 分子便滞留在爬行管里,直至沿新的电场轴向重新定向后,才能继续向前移动,DNA 分子越大,这种重排所需要的时间就越长。
若DNA 分子变换方向的时间小于电脉冲周期时. DNA 分子就可以按大小分开。
现在已可分辨大于5 000kb 的DNA 分子。
PFGE 被认为是分子生物学分类方法的“金标准”,可用于比较不同的菌株。
但PFGE耗时较长, 需2 ~ 3天。
【4】RFLP ( Rest riction Fragment Length Polymorphisml) 限制性片段长度多态性。
不同细菌有其独特的核苷酸序列,用同一种限制性内切酶切割不同细菌基因组DNA 后会产生不同长度的限制性片段,这种限制性片段长度多态性可通过电泳分析表现出来。
RFL P 作为检测细菌种内或种间DNA水平的最敏感的方法之一,可作为种以下菌株间的鉴定方法。
【5】RAPD (Random Amplified Polymorphic DNA)随机扩增多态性技术,其原理为:采用核苷酸序列的随机引物进行基因组DNA 扩增,非特异性引物可以结合在模板DNA 的多个位点,产生多个PCR产物,采用琼脂糖凝胶电泳将产物分离开,将每株菌在每条引物下扩增后产生的图谱合成为针对一株菌的单一图谱, 再通过软件进行分析。
此方法的敏感性和特异性在于细菌的整个基因组作为基础产生DNA 图谱。
细菌种、株间基因特异性可通过获得DNA 片段大小及数量来确定。
它对于区分亲缘关系近的细菌非常有效。
与其它方法相比较,本方法快速,区别程度高,不需要对基因组DNA 有更深入详细的了解。
【6】AFLP(扩增片段长度多态性分析) 为选择性限制片段扩增技术,先用一中等切割频率(如EcoRI)和一较高切割频率(如Mse I) 限制性酶消化少量纯化DNA ,再将二个接头(含各自的限制性位点,但在接头序列中掺入点突变,在连接后,原限制性位点不恢复) 与基因组限制片段两端连接,然后用接头特异引物在严格条件下进行PCR 扩增,在引物3′端有1~3个选择性核苷酸伸入未知染色体限制片段内。
本法要求DNA 量少,且不须杂交,故避免了限制性片段长度多态性(RFLP) 基因分型不可重复的原因———DNA 部分消化和不清晰图谱,而且AFLP 在实验室内和实验室间重复性均好,可把图谱储存在数据库中,用于先前或今后以及实验室间图谱比较和鉴定新菌株。
AFLP分析可用作菌种鉴定和菌株区分(90 %~100 %同源性为相同株,60 %~90 %同源性为同种不同株,40 %~60 %同源性为同属不同种, <40 %为不同属) 。
AFLP分析具有与RFLP,RAPD 等相同的分类学范围,并兼有这些技术的优点,在多数情况下有最
高的分辨力。
申报单位应根据生产的实际,选择适当的菌种鉴别手段对生产菌株进行鉴别,提供详细研究资料,以证明产品质量可控。
【参考文献】
1、微生态细菌的遗传学分类方法研究进展;幺山山; 曾明;微生物学免疫学进展, 2003年 02期。
2、《药物微生物技术》;李越中;化学工业出版社;2004年4月第一版。
3、《微生物药物学》;张致平;化学工业出版社;2003年3月第一版。
4、《生物化学》;沈同,王镜岩;高等教育出版社;1991年10月第二版。
