噬菌体抗体淘筛方法
噬菌体抗体库筛选技术研究进展 - 文章编号1007-8738(2005)S-0058

[ 1 ] W illats W G. Phage disp lay: p racticalities and p rospects [ J ]. P lant M ol B iol, 2002, 50 ( 6) : 837 - 854.
[ 2 ] Ladner RC. Phage disp lay and pharmacogenom ics [ J ]. Pharm acog2 enom ics, 2000, 1 ( 2) : 199 - 202.
收稿日期 : 2004 - 03 - 22; 修回日期 : 2004 - 05 - 08 基金项目 : 国家自然科学基金资助项目 (No. 30371399) 作者简介 : 薛国柱 (1966 - ) , 男 , 河南新野人 , 副主任医师 , 博士生.
Tel: ( 29) 83375259; Email: xgzh2003@ yahoo. com. cn
噬菌体 抗 体 库 的 筛 选 包 括 两 个 主 要 步 骤 : 淘 筛 和 鉴 定 [5 ] 。淘筛 (panning)是将噬菌体抗体库与选择用的抗原共 同孵育 , 通过几轮洗脱 , 收集结合的噬菌体 。将获得的噬菌 体感染细菌并扩增 , 再进行下一轮的淘筛 。经几轮淘筛后 , 便可富集到与抗原特异性结合的噬菌体感染的多克隆菌株 。 鉴定过程是从噬菌体感染的多克隆菌株中挑选出单克隆菌 株 。即将淘筛出的噬菌体感染细菌 、铺板 、挑选 , 即可得到 高特异性单克隆菌株 。
由于膜蛋白密度的差异及膜分子暴露程度的不同 , 对未 知抗原的分离鉴定有很大困难 , 因而在很长一段时间内抗体 库没有被用于对肿瘤特异性抗体的筛选 [11 ] 。最近 , 直接用肿 瘤细胞从单链噬菌体抗体库中筛选肿瘤特异性抗体已有报 道 。如 Kup sch等 [12 ]筛选出与黑色素瘤细胞特异性结合的抗 体 。R idgway等 [13 ]采用先将正常支气管上皮细胞系与非特异 性的噬菌体抗体清除后 , 再用肺腺癌细胞系进行筛选的方法 , 得到抗 CD55的单链抗体 。但即使这样 , 其筛选效率仍较低 , 而且容易丢失亲和性高的噬菌体 。 Siegel等报道了磁性细胞 分离法 (magnetically2activated cell sorting, MACS) 。即用抗原 阳性的细胞包裹磁珠 , 然后与大量抗原阴性的细胞混合 , 加 入待筛选的噬菌体作用后 , 再通过磁柱快速分离结合特异性 噬菌体的抗原阳性细胞 。他们采用 MACS法 , 以人血红细胞 Rh (D ) +细胞为靶细胞 , Rh (D ) - 细胞吸收非特异性噬菌体 , 成功地从 Fab噬菌体抗体库中筛选出一系列抗 Rh (D)的抗体。
抗体筛选鉴定推荐流程

抗体筛选鉴定推荐流程抗体筛选鉴定推荐流程:1.包被免疫管:将100ug 抗体加入到2mL PBS中并加入到免疫管中,4度过夜孵育。
2.封闭:将扩增和纯化噬菌体文库后的噬菌体500ul 加入到1mL 3% BSA中,室温旋转孵育2h。
同时往包被好的免疫管中加入2-3mL 3% BSA,室温旋转孵育2h。
3.抗原和噬菌体孵育:将封闭后的免疫管用含有0.01%吐温的PBS洗3次,每次5分钟。
将封闭后的噬菌体文库加入到封闭后的免疫管中,添加PBS直至2-3mL,室温旋转孵育1h。
4.清洗:将抗原和噬菌体孵育后的免疫管用含有0.01%吐温的PBS洗20次,每次5分钟。
5.洗脱:往免疫管中加入1mL 100mM Trimethymime,室温孵育10分钟,加入1M Tris-HCl中和Trimethymime,将最后1.5mL 的洗脱噬菌体转移到新的离心管中。
将洗脱的噬菌体按照扩增和纯化噬菌体文库扩增后再重复筛选过程2次,逐次减半包被免疫管的抗体量,得到3次筛选后的洗脱噬菌体。
6.ELISA鉴定:将上一步筛选得到的噬菌体稀释10^6倍后,取100ul加入到OD600为0.5的SS320菌液中,37度培养30分钟后涂布含有四环素和氨苄霉素的2X YT培养板上,37度过夜培养第二天得到单克隆菌落。
挑选96个单克隆菌落到含有四环素和氨苄霉素的2X YT培养液的96孔细胞培养板上,37度培养3-4小时后往培养孔中加入卡那霉素和20:1的辅助噬菌体,30度过夜培养。
第二天将过夜培养后的细胞液离心,获得上清液。
将过夜包被抗原和用3%BSA封闭过后的96孔ELISA板中加入上一步获得的噬菌体上清液,室温孵育1h。
用含有0.05%吐温的PBS清洗3次后,用噬菌体抗体抗体作为一抗,用相应的二抗TMB显色后在波长450读取每个孔的吸光值。
选取吸光值读数最高的SS320菌落送去测序,得到抗体的基因序列。
抗体筛选技巧:1. 合适量的抗原包被和抗原的分子量大小、疏水亲水性质、结构有关,也和包被缓冲液和包被介质的选择有关,合理的包被是成功筛选的基础,如果有必要可以进行预实验确定包被的条件。
噬菌体抗体淘筛方法

这两个文库来自于单个人的V H和Vκ的基本结构,具有和那些已知结构的抗原结合位点的侧链多样性,并且在成熟的体系中中具有很高的多样性。
由这个框架来编码的标准结构是目前人抗体技术体系中最普通的。
在能够形成抗原结合表面的前提下重链的CDR3设计的尽可能短。
这两个文库都可以在未知筛选的克隆的序列的情况下进行筛选和免疫亲和力成熟。
这两个文库是噬菌粒/单链片段可变区格式的,并且都经过与蛋白A和蛋白L的结合筛选,所以未筛选的克隆的大多数都是具有功能的。
TomlinsonI构建在plT2载体((HIS myc 标签),多样化的侧链主要是来自于原始体系中多样性的位点(一共18个残基,H50, H52, H52a, H53, H55, H56, H58, H95, H96, H97, H98, L50, L53, L91, L92, L93, L94 and L96)此文库经过筛选后与体细胞突变的多样性的共同作用使其成熟。
我们保证上述信息在没有另外通知之前是严格可信的。
使用此文库之前请认真阅读以下材料确认收到以上材料2. 确认收到的文库未溶解,且使用之前-70摄氏度保存。
3. 参照方案G制备KM134. 在使用文库前清仔细阅读这个方案:在含有100 μg/ml氨苄青霉素和1 %葡萄糖的TYE平板上将对照划线培养,置于培养箱37℃过夜培养,挑单菌落置于摇床,接种在5ml含有100 μg/ml氨苄青霉素和1 %葡萄糖的2倍TY培养基内,37℃过夜培养。
分别制备阳性和阴性的噬菌体对照(第一天到第十天过夜用500微升)。
用阳性对照和阴性对照产生的噬菌体按照1:100的比例混合,进行一轮的筛选,用100 μg/ml的遍在蛋白在PBS中包被。
通过单克隆噬菌体ELISA检验遍在蛋白的富集程度(经过第一轮筛选后应该超过50%)5. 尽可能的用专用的移液管和一次性的手套,由于噬菌体会非特异的吸附到其他的塑料上,建议使用聚丙烯管。
6. 为提高感染效率,大肠杆菌应该在37℃培养至对数生长期(600nm的OD值应该在0.4)(1)、从一个小型的培养板上转移一个细菌克隆至5ml的2倍TY培养基(不加抗生素和葡萄糖),37℃震荡过夜培养(2)、次日稀释100倍至新鲜的2倍TY培养基内,震荡培养至培养至对数生长期600nm的OD值应该在0.4。
抗噬菌体菌株筛选

抗噬菌体菌株筛选噬菌体的分离取疑似噬菌体感染的生产菌株斜面或平板,用无菌蒸馏水将菌苔洗下,无菌过滤或离心,收集滤液或上清液。
在摇瓶培养基中接种1%生产菌株的孢子或细胞悬浮液,适温下培养24 h,加入1%的上述滤液或56离心上清液,继续培养24 h,无菌离心或过滤,收集上清液或滤液。
取上述上清液或滤液和生产菌株的孢子或细胞悬浮液各0.25ml,混匀后加在生产菌株的平板培养基上,用刮棒涂布均匀,适温下培养48~96h,确认是否生成噬菌斑。
噬菌体的纯化与增殖将上述平板上长出的噬菌斑用接种针移入生产菌株培养12~24 h的摇瓶培养液中,继续培养24 h左右,无菌过滤或离心收集滤液或上清液。
取上述滤液或上清液和生产菌株的孢子或细胞悬浮液各0.25 ml,混匀后加在生产菌株的平57板培养基上,用刮棒涂布均匀,适温下培养48~96h,确认是否生成噬菌斑。
重复以上两个步骤,使噬菌体达到纯化和增殖的目的。
最终所得滤液或上清液作为纯化噬菌体原液置冰箱保存。
噬菌体效价的测定取6支灭菌并烘干的小试管,分别标注10-1、10-2、10-3、10-4、10-5和10-6,各加入0.9 ml无菌蒸馏水。
在10-1小试管中加入纯化噬菌体原液0.1ml,混匀后取出0.1ml移入10-2小试管中,如此类推,分别获得6个稀释度的噬菌体稀释液。
在5支灭菌并烘干且分别标注10-3、10-4、10-5、10-6和10-7的小试管中,分别加入0.1 ml上述10-2、10-3、10-4、10-5和10-6的噬菌体稀释液和0.9 ml生产菌株的孢子或细胞悬浮液,摇匀后再加入50℃的生产菌株琼脂培养基,迅速摇匀后分别倾入标注10-3、10-4、10-5、10-6和10-7的灭菌培养皿中,凝固后置适温下培养48~96 h,对所形成的噬菌斑计数。
抗噬菌体菌株选育步骤噬菌体抗性的检测将上述经过诱变和加热处理的生产菌株孢子或细胞悬浮液0.5 ml涂布到生产菌株的平板培养基上,表面干燥后加入0.1 ml保存的噬菌体原液,涂布均匀后置适温下培养48~72 h,观察噬菌斑的形成并计数(如无噬菌体原液,也可用噬菌斑制成悬浮液)。
噬菌体展示筛选抗体技术介绍

探索生物医学中的创新应用
目录
01 噬菌体展示筛选抗 体概述
06 噬菌体展示抗体的 发展前景
02 噬菌体展示技术原 理
03 噬菌体展示抗体筛 选流程
0Hale Waihona Puke 噬菌体展示抗体的 应用案例05 噬菌体展示抗体的 优点与缺点
01 噬菌体展示筛选抗体 概述
噬菌体展示筛选抗体概述
1 噬菌体展示筛选抗体技术介绍
疗效果并降低副作用。
05 噬菌体展示抗体的优 点与缺点
噬菌体展示抗体的优点与缺点
噬菌体展示抗体的优 势
噬菌体展示技术能够快速、 高效地筛选出特异性抗体, 大大缩短了实验周期,提高 了研究效率。
噬菌体展示抗体的局 限性
噬菌体展示技术虽然筛选速 度快,但存在假阳性率高的 问题,需要进一步的验证和 优化。
噬菌体展示筛选抗体技术是一种利用噬菌体表面
噬菌体展示筛选抗体的优势 2 展示特定蛋白质,通过生物淘选方法寻找与目标
噬菌体展示筛选抗体技术具有高度灵活性和多样
抗原特异性结合的抗体的方法。
性,能够快速、高效地筛选到具有高亲和力和特
异性的抗体,为免疫学研究和药物开发提供了重
要工具。 3 噬菌体展示筛选抗体的应用前景
噬菌体展示抗体的优 势
噬菌体展示技术具有高度灵 活性和多样性,可以快速、 大量地筛选出特异性强、亲 和力高的抗体,为生物医学 研究和药物开发提供了重要 工具。
噬菌体展示抗体的应 用前景
噬菌体展示抗体技术在肿瘤 治疗、免疫诊断、疫苗研发 等领域具有广泛应用前景, 有望为人类健康事业做出重 要贡献。
谢谢大家
噬菌体展示筛选抗体技术在疾病诊断、治疗和预
防等方面具有广泛的应用前景,包括癌症治疗、
噬菌体抗体文库淘选

噬菌体抗体文库淘选1.主要实验仪器表1实验所用主要仪器仪器名称型号/厂家高速冷冻离心机Neofuge15R生物安全柜Heal Force电热恒温水浴锅HHW21.600AⅡ恒温培养箱Heal force(3)5ml5%脱脂牛奶/PBST30℃封闭1h。
(4)5ml PBS洗涤1次。
(5)每管中加入500ul,10^11-10^12pfu的文库噬菌体(或者上一轮的扩增噬菌体),30℃孵育2h。
(6)5ml PBST洗涤4-6次(后几轮可根据富集程度增加洗涤次数)。
(7)每管中加入500ul的Gly-HCl(pH=2.2)洗脱噬菌体,室温振荡孵育6-8min左右。
加入120-130ul的Tris-HCl(pH=9.6)中和溶液至pH=7.0-8.0。
(8)将洗脱后的噬菌体稀释后,侵染对数期的大肠杆菌TG1,铺板测定滴度。
3.2洗脱噬菌体的扩增(1)吸取洗脱后的噬菌体,加入到对数期的大肠杆菌TG1菌液中,37℃静置30min后,220rpm培养30min-1h。
(2)培养基中加入抗生素Amp,37℃,220rpm培养至菌液OD=0.4-0.6左右。
(3)在菌液中加入辅助噬菌体。
37℃静置30min后,220rpm培养45min-1h。
(4)3000-5000rpm离心菌液,弃上清。
用同等体积2YT-Amp-Kan培养基重悬菌体。
30℃,220rpm培养过夜。
(5)次日,8000rpm,4℃,20min离心菌液,将上清转入新的离心管中。
加入1/4体积的(4)用PBS稀释每一轮扩增后的噬菌体,稀释倍数3倍递增。
初始浓度为10^12pfu/ml。
每孔中加入100ul稀释后的扩增噬菌体。
30℃孵育1h,300ul PBST洗涤4-6次。
(5)加入100ul二抗(抗噬菌体M13)稀释液,30℃孵育1h,300ul PBST洗涤4-6次。
(6)加入100ul显色液TMB避光显色3-8min,加入100ul2M HCl终止反应,酶标仪读数(450nm-620nm)。
噬菌体展示文库的筛选技术 2005

噬菌体展示文库的筛选技术李丽芳 张映(山西农业大学动物科技学院,太谷 030801)摘 要: 噬菌体展示技术(Phage Display Techniques ,PD T )是一种用于筛选和改造功能性多肽的生物技术。
该技术作为筛选与多种靶分子(如抗体、酶类、细胞表面受体等)具有特异性亲和力或活性的肽的一个有效方法,自问世以来,已取得了很大的发展,并被广泛地应用于基因治疗、基因疫苗研究、抗原表位研究、药物设计、研究细胞信号传导等领域[1]。
但该技术在两方面仍需进一步完善:(1)寻求更为有效的表达载体;(2)进一步完善筛选方法。
关键词: 噬菌体 肽库 抗体库 筛选The Selection T echniques of Phage Display LibrariesLi Lifang Zhang Y ing(A ni mal S cience and Technolog y Depart ment of S hanx i A g ricult ure Universit y ,S hanx i Tai gu 030801)Abstract : Phage Display Techniques (PD T )is a biotechnique used for screening and reconstructing functingal polypeptide.It ,s a effective method of select the peptide which has high affinities to many given targets (such as an 2tibody ,Enzyme ,surface receptor of cell ).It has been made much progress and are widely used in many fields ,such as genetic treatment ,the study of genetic vaccine ,epitope mapping ,drug discovery after its appearance.But it has to get more perfact in two sides :(1)Seek more effective expression vector ;(2)G et more perfect selection methods.Many selection methods are reviewed in this article.K ey words : Phage Peptide library Antibody library Selection 噬菌体表面展示技术是一种将外源多肽或蛋白质的DNA 序列插入到噬菌体外壳蛋白的一个结构基因的适当位置上,外源基因将随着外壳蛋白的表达而表达,从而使多肽或蛋白以与外壳蛋白融合的形式展示在噬菌体表面。
噬菌体抗体淘筛方法

噬菌体抗体淘筛方法一、噬菌体展示技术原理噬菌体展示技术基于噬菌体颗粒表面的基因插入法。
通过将抗体片段的基因插入到噬菌体基因组中,使噬菌体能够在其表面展示抗体。
随后,将含有目标抗原的库经过一系列的筛选步骤,如淘洗、洗涤和分离,以筛选出与目标抗原特异性结合的抗体。
噬菌体核心蛋白质pIII等表面蛋白链通过基因插入的方法与外源基因连接,实现抗原展示。
二、噬菌体抗体淘筛方法的流程1.抗原制备:首先,需要制备目标抗原。
可以通过多种方法制备抗原,如重组蛋白表达系统、细胞和细胞溶解物、组织和分离物等。
2.抗体库构建:构建包含大量抗体片段的抗体库。
一般使用转录组或基因组DNA作为起始材料,使用PCR扩增抗体基因片段,并将其插入合适的载体中。
3.噬菌体包装:将构建好的抗体库与噬菌体粒子一起包装成噬菌体颗粒。
4.抗原吸附:将噬菌体抗体库与目标抗原进行反应,使抗体与抗原结合。
5.淘洗分离:用洗涤缓冲液对混合物进行混洗,以除去非特异性结合的噬菌体。
6.噬菌体放大:将经过淘洗分离的噬菌体在培养基中进行放大培养。
7.ELISA筛选:使用ELISA检测噬菌体是否与目标抗原的特异结合。
将阴性和阳性对照样品和待测样品加入到蛋白质包被的酶标板孔中,通过检测酶标物质的生成或反应物颜色的变化,判断噬菌体是否与目标抗原结合。
8.质粒DNA提取和测序:选择特异性结合抗原的噬菌体进行质粒DNA 提取和测序,以获取抗体的DNA序列。
9.后续鉴定和分析:鉴定筛选出的抗体的亲和力、特异性、敏感性等性质,以及进行进一步的功能分析。
三、噬菌体抗体淘筛方法的注意事项1.抗原的选择:选择合适的抗原非常关键,抗原应具有特异性且容易从培养基或生物样品中提取。
2.抗体库的构建:构建抗体库时,要确保插入的抗体片段多样性和覆盖性。
3.抗原吸附条件的优化:抗原吸附条件的优化对淘洗分离步骤的效果具有重要影响。
4.筛选条件的优化:在ELISA筛选过程中,需要对反应温度、时间、缓冲液浓度等条件进行优化,以提高筛选效果。
噬菌体抗体库筛选操作流程tomlinsonij

噬菌体抗体库筛选操作流程tomlinsonijtheLibrariesGeneral Introduction toOver the past 10 years Greg Winter’s lab at the MRC Laboratory of Molecular Biology and the MRC Centre for Protein Engineering (Cambridge, UK) has created a number of artificial libraries of antibodies that can be used to derive binders to almost any target molecule using phage display and selection. These binders can be used for all the same applications as conventional monoclonal antibodies (ELISA, Western blotting, FACS, immunohistochemistry etc) but can be isolated in a fraction of the time and without the need for animal immunisation. To date these so called “na?ve” or “single pot” phage-antibody libraries have been used successfully in hundreds of molecular biology labs world-wide to derive highly specific antibody reagents to a wide range of different proteins, peptides or small molecule compounds.The latest libraries (Tomlinson I and J) that are being distributed by the MRC HGMP Resource Centre each comprise over 100 million different scFv fragments cloned in an ampicillin resistant phagemid vector and transformed into TG1 E. Coli cells (scFv fragments comprise a single polypeptide with the VH and VL domains attached to one another by a flexible Glycine-Serine linker). By carefully following the protocol provided, large numbers of phagemids can be produced and used to select specific binders to target molecules that are attached to the surface of a tube or biotinylated and captured by streptavidin coated beads (so called “panning”). After each round of panning, the non-binders are washed away and the phagemids bound to the target molecule/s are eluted and amplified by infection into fresh TG1 cells. After producing new phagemids from the previous round of panning, the process can be repeated. Typically two or three rounds of panning are required to ensure that more than half the different scFvs in the selected population bind to the target molecule. The monoclonal scFvs can then be screened for binding (using a simple ELISA based protocol) and then used for further analysis of the target molecule. Since all the functional scFvs in the Tomlinson I and J libraries bind Proteins A and L, either of these secondary reagents can be used for detection, purification or immobilisation. Alternatively, secondary reagents that bind the attached myc or HIS6 tags can be used, although in our experience it is better to use the Protein A or L reagents.Finally, we would like to emphasise that these libraries represent a valuable resource. Whether you are familiar with phage display or not we recommend that you perform test selections and subsequent ELISA screening using the anti-bovine serum albumin and anti-bovine ubiquitin controls provided. Only when these experiments have been successfully carried out should you defrost the libraries and start preparing library phage.Derivation of librariesBoth libraries are based on a single human framework for V H (V3-23/DP-47 and J H 4b) and V κ (O12/O2/DPK9 and J κ1) with side chain diversity incorporated at positions in the antigen binding site that make contacts to antigen in known structures and are highly diverse in the mature repertoire. The canonical structure (V H : 1-3, V κ: 2-1-1) encoded by this framework is by far the most common in the human antibody repertoire. The CDR3 of the heavy chain was designed to be as short as possible yet still able to form an antigen binding surface. Both libraries can be selected and affinity matured without knowing the sequences of selected clones. Libraries are in phagemid/scFv format and have been pre-screened for binding to Protein A and Protein L so that the majority of clones in the unselected libraries are functional.Constructed in pIT2 (HIS myc tag). Diversified (DVT) side chains based mainly on those positions which are diverse in the primary repertoire (total of 18 residues - H50, H52, H52a, H53, H55, H56, H58, H95, H96, H97, H98, L50, L53, L91, L92, L93, L94 and L96). After selection, can be matured by incorporating additional diversity based on somatic mutation. Library size (with insert): 1.47 x 108 Percentage insert: 96%As above but with NNK side chains. Library size (with insert): 1.37 x 108 Percentage insert: 88%1. Check that you have received:a tube of Library I (~500 µl)a tube of Library J (~500 µl)a glycerol stock of a positive control anti-ubiquitin ScFv in bacterial strain TG1 (labeled TG1-antiubi)a glycerol stock of a positive control anti-BSA ScFv in bacterial strain TG1 (labeled 13CG2)a glycerol stock of T-phage resistant E. Coli. TG1 for propagation of phage (labeled TG1Tr)(lac-proAB) supE thi hsdD5/F' traD36 proA+B lacI q lacZM15)(K12a glycerol stock of E. Coli. HB2151 for expression of antibody fragmentsara ?(lac-proAB) thi/F' proA+B lacI q lacZ?M15)(K12KM133 (~100 µl with 107 pfu/ml)Phage2. Check the library is still frozen and make sure you keep it frozen at -70°C until needed.3. Make stock of KM13 according to Protocol G.4. Run through the protocols using the control clones before you use the library: Streak the controlson TYE plates containing 100 µg/ml ampicillin and 1 % glucose. After overnight growth at 37°C in an incubator pick a single colony from each and grow these overnight (shaking at 37°C) in 5 ml 2xTY1 containing 100 µg/ml ampicillin and 1 % glucose. Make phage for the positive and negative controls separately (use 500 µl of overnight in D1-D10). Use a 1:100 mixture of phage produced from the positive and the negative controls and perform one round of selection (C1-C11) using 100 µg/ml of ubiquitin2 in PBS for coating. Check for enrichment of ubiquitin binders (should be over 50% after one round of selection) by monoclonal phage ELISA (E9-E14).5. Wherever possible use devoted pipettes and disposable plastic ware. The use of polypropylenetubes is recommended as phage may adsorb non-specifically to other plastics.6. For efficient infection of phage, E. coli must be grown at 37°C and be in log phase (OD at 600 nmof 0.4). To prepare this:i. Transfer a bacterial colony from a minimal media plate into 5 ml of 2xTY medium (no antibioticsor glucose). Grow shaking overnight at 37°C.ii. Next day dilute overnight 1:100 into fresh 2xTY medium. Grow shaking at 37°C until OD 600 is0.4 (1.5-2 hrs)7. All centrifugations, except those performed in a micro centrifuge, are performed at 4°C.8.Libraries I and J must be used separately and preferably in parallel. This will ensureselecting the most antigen binding clones.In advanceGather all equipment and reagents (product details are given in the notes at the end of all the protocols). Make sure you have all the necessary media and plates for bacterial growth (the large Bio-Assay plates need to be air-dried in a sterile environment for 2 hrs before use).Plan your time - most of the daily procedures can be performed simultaneously, so read through each protocol carefully before starting.StepsProcedureDay 1 (5 hrs) A1-A6 Grow libraries I and J and make phageB1-B3 Make secondary stock of librariesDay 2 (6 hrs) A7-A12 Grow libraries I and J and make phage (cont.)C1 Coat immunotubes for 1st round of selectionDay 3 (6.5 hrs) C2-C11 1st round of selectionDay 4 (3 hrs) D1-D6 Make phage from 1st round of selectionC1 Coat immunotubes for 2nd round of selectionDay 5 (6.5 hrs) D7-D11 Make phage from 1st round of selection (cont.)C2-C11 2nd round of selectionDay 6 (3 hrs) D1-D6 Make phage from 2nd round of selectionC1 Coat immunotubes for 3nd round of selectionDay 7 (6.5 hrs) D7-D11 Make phage from 2nd round of selection (cont.)C2-C11 3rd round of selectionDay 8 (3 hrs) D1-D6 Make phage from 3rd round of selectionE1 Coat 96 well plate for polyclonal phage ELISADay 9 (6.5 hrs) D7-D11 Make phage from 3rd round of selection (cont.)E2-E8 Polyclonal phage ELISAFurther characterisation of individual clones can be performed by monoclonal phage ELISA (protocol E), monoclonal ELISA using soluble scFv fragments (protocol F), PCR screening (to check for insert, protocol H) and sequencing (protocol I).1. Add the library stock to 200 ml pre-warmed 2xTY containing 100 µg/ml ampicillin and 1 %glucose.2. Grow shaking at 37°C until the OD 600 is 0.4 (1-2 hrs).3. Take 50 ml of this and add 2x1011 KM13 helper phage3. (Use the remaining 150 ml to make asecondary bacterial stock of the library by following protocol B).4. Incubate without shaking in a 37°C water bath for 30 min.5. Spin at 3,000 g for 10 min (3,600 rpm in Centra 8 or equivalent). Resuspend in 100 ml of 2xTYcontaining 100 µg/ml ampicillin, 50 µg/ml kanamycin and 0.1% glucose.6. Incubate shaking at 30°C overnight.7. Spin the overnight culture at 3,300 g for 30 min (4,000 rpm in Centra 8 or equivalent).8. Add 20 ml PEG/NaCl (20 % Polyethylene glycol 6000, 2.5 M NaCl)to 80 ml supernatant. Mix welland leave for 1 hr on ice.9. Spin 3,300 g for 30 min (4,000 rpm in Centra 8 or equivalent). Pour away PEG/NaCl. Respinbriefly and aspirate any remaining dregs of PEG/NaCl.10 Resuspend the pellet in 4 ml PBS and spin at 11,600 g for 10 min in a micro centrifuge to removeany remaining bacterial debris.11. Store the phage at 4°C for short term storage or in PBS with 15 % glycerol for longer termstorage at -70°C.12. To titre the phage stock dilute 1µl phage in 100µl PBS, 1µl of this in 100µl PBS and so on untilthere are 6 dilutions in total. Add 900µl of TG1 at an OD 600 of 0.4 to each tube and incubate at 37°C in a waterbath for 30 mins. Spot 10 µl of each dilution on a TYE5 plate containing 100 µg/ml ampicillin and 1 % glucose and grow overnight at 37°C. Phage stock should be 1012-1013/ml, enough for at least 10 selections.1. Grow the remaining 150 ml from A3 for a further 2 hr shaking at 37°C.2. Spin down the cells at 10,800 g for 10 min. Resuspend in 10 ml of 2xTY containing 15 % glycerol.3. Store this secondary stock in 20x 500 µl aliquots at -70°C. Use one aliquot for each phagepreparation according to protocol A - this will only be necessary if you wish to do more than 10 selections.(Alternatively, phage can be selected using biotinylated antigen in solution or affinity chromatography. For details see Winter et al. (1994) Annu. Rev. Immunol.12, 433)immunotube6 overnight with 4 ml of the required antigen. The efficiency of coating can1. Coatdepend on the antigen concentration, the buffer and the temperature. Usually 10-100 µg/mlantigen in PBS is used.2. Next day wash tube 3 times with PBS (pour into the tube and then pour it immediately out again).3. Fill tube to brim with 2 % MPBS (2 % Marvel milk powder7 in PBS). Incubate at rt. Standing onthe bench for 2 hr to block.4. Wash tube 3 times with PBS.1012 to 1013 phage from A11 in 4 ml of 2 % MPBS. Incubate for 60 min at rt. rotating using 5. Addan under-and-over turntable and then stand for a further 60 min at rt. Throw away supernatant.6. Wash tubes 10 (round 1)-20 (rounds 2 and 3) times with PBS containing 0.1 % Tween 20.7. Shake out the excess PBS and elute phage by adding 500 µl of trypsin-PBS (50 µl of 10mg/mltrypsin stock solution8 added to 450 µl PBS) and rotating for 10 min at rt using an under-and-over turntable.8. Take 1.75 ml of TG1 at an OD 600 of 0.4 and add 250 µl of the eluted phage (the remaining 250µl should be stored at 4°C). Incubate for 30 min at 37°C in a water bath without shaking.µl, 10 µl of a 1:102 dilution and 10 µl of a 1:104 dilution on TYE plates containing 1009. Spot10µg/ml ampicillin and 1 % glucose and grow overnight at 37°C to titre the phage.10. In round 1 if using a complex antigen (eg cells, cell lysates etc): take the remaining TG1 cultureand spin at 11,600 g in a micro centrifuge for 5 min. Resuspend the pelleted bacteria in 1 ml of 2xTY and plate on a large square Bio-Assay dish9 containing TYE, 100 µg/ml ampicillin and 1 % glucose.In round 1 if using a single hapten, carbohydrate or protein antigen and in subsequent rounds for all antigens: take the remaining TG1 culture and spin at 11,600 g in a micro centrifuge for 5 min.Resuspend the pelleted bacteria in 50 µl of 2xTY and plate on a regular TYE plate containing 100 µg/ml ampicillin and 1 % glucose.11. Grow plates at 37°C overnight.The first round of selection is the most important. Any errors made at this point will be amplified in subsequent rounds of selection. After each round you should get back at least 100 infective phage. If you get less than this it is probable that a mistake has occurred. If so, try repeating the infection with a freshly grown TG1 culture (from a new overnight) at an OD 600 of 0.4 using the remaining 250 µl of eluted phage from C7. If this still yields less than 100 phage, repeat the selection starting at C1.1. After overnight growth add 7 ml of 2xTY 15 % glycerol to the large square Bio-Assay dish or 2mlsto the regular plates and loosen the cells with a glass spreader, mixing them thoroughly. After inoculating 50 µl of the scraped bacteria to 50 ml of 2xTY containing 100 µg/ml ampicillin and 1 % glucose, store 1 ml of the remaining bacteria at -70°C in 15% glycerol.2. Grow shaking at 37°C until the OD 600 is 0.4 (1-2 hrs).3. Take 10 ml of this culture and add 5x1010 helper phage.4 Incubate without shaking in a 37°C water bath for 30 min.5. Spin at 3,000 g for 10 min. Resuspend in 50 ml of 2xTY containing 100 µg/ml ampicillin, 50 µg/mlkanamycin and 0.1% glucose.6. Incubate shaking at 30°C overnight.7. Spin the overnight culture at 3,300 g for 15 min.8. Add 10 ml PEG/NaCl (20 % Polyethylene glycol 6000, 2.5 M NaCl)to 40 ml supernatant. Mix welland leave for 1 hr on ice.9. Spin 3,300 g for 30 min. Pour away PEG/NaCl. Respin briefly and aspirate any remaining dregsof PEG/NaCl.10. Resuspend the pellet in 2 ml PBS and spin at 11,600 g for 10 min in a micro centrifuge to removethe remaining bacterial debris.11. Use 1 ml of this phage for the next round of selection (protocols C and D) and store 1 ml at 4°C.12. Repeat selection for another 2 rounds.Populations of phage produced at each round of selection can be screened for binding by ELISA to identify "polyclonal" phage antibodies. Phage from single colonies can then be screened by ELISA to identify "monoclonal" phage antibodies. Alternatively, after a polyclonal phage ELISA you could proceed directly to making monoclonal soluble antibody fragments, see protocol F. In general, we have found that 2% Marvel in PBS is best for blocking during phage ELISA whereas 3% BSA in PBS is best for blocking during scFv ELISA.1. Coat a 96 well flexible assay plate10 overnight with 100 µl per well of antigen in the same bufferand at the same concentration as used for selection.2. Wash wells 3 times with PBS. Plates can be immersed in a shallow bath containing PBS but youshould check that all wells fill with wash solution (if they do not you may create false positivesduring later washes). Discard liquid by flipping plate over and then shaking it. Add 200 µl per well of 2 % MPBS (2 % Marvel in PBS) or 3% BSA-PBS (3% bovine serum albumin in PBS) to block and incubate for 2 hr at rt.3. Wash wells 3 times with PBS. Add 10 µl PEG precipitated phage from the end of each round ofselection in 100 µl 2 % MPBS (or 3 % BSA-PBS).4. Incubate for 1 hr at rt. Discard phage solution and wash 3 times with PBS-0.1 % Tween 20.5. Add 1 in 5000 dilution of HRP-anti-M1311 in 2 % MPBS (or 3 % BSA-PBS). Incubate for 1 hr atrt., wash 3 times with PBS-0.1 % Tween 20.µl of substrate solution (100 µg/ml TMB12 in 100 mM sodium acetate, pH 6.0. with10 µl 1006. Addof 30 % hydrogen peroxide added to 50 ml of this solution directly before use) to each well and leave at rt. for 2-15 min. A blue colour should develop.7. Stop the reaction by adding 50 µl 1 M sulphuric acid. The blue colour should turn yellow.9. Inoculate individual colonies from the titration plates from each round of selection (see C11) into100 µl 2xTY containing 100 µg/ml ampicillin and 1 % glucose in 96 cell-well plates13. Growshaking (250 rpm) overnight at 37°C.10. Use a 96 well transfer device14 to transfer a small inoculum (about 2 µl) from this plate to asecond 96 cell-well plate containing 200 µl of 2xTY with 100 µg/ml ampicillin and 1 % glucose per well. Grow shaking (250 rpm) at 37°C for 2 hr. (Make glycerol stocks of the original 96-well plate, by adding glycerol to a final concentration of 15 %, and then storing the plates at -70°C).11. After 2 hr incubation (of the second plate) add 25 µl 2xTY containing 100 µg/ml ampicillin, 1 %glucose and 109 helper phage.12. Shake (250 rpm) at 37°C for 1 hr. Spin 1,800 g for 10 min, then aspirate off the supernatant.13. Resuspend pellet in 200 µl 2xTY containing 100 µg/ml ampicillin and 50 µg/ml kanamycin. Growshaking (250 rpm) overnight at 30°C.14. Spin at 1,800 g for 10 min and use 50 µl of the supernatant in phage ELISA as detailed above.Individual colonies picked from the various rounds of selection (as plated on the dilution series) can be induced in TG-1 to produce soluble scFv (F2-F6). This will ensure the expression of all selected clones including those in which the scFvs contain TAG stop codons (TG-1 as able to suppress termination and introduce a glutamate residue at these positions). Unfortunately, since the TAG stop codon between the scFv and the gIII gene is also suppressed this leads to co-expression of the scFv-pIII fusion which tends to lower the overall levels of scFv expression, even in clones where there are no TAG stop codons in the scFv itself. To circumvent this problem, the selected phage can be used to infect HB2151 (a non-suppresor strain) which is then induced to give soluble expression of antibody fragments (scFv genes that do not contain TAG stop codons will now yield higher levels of soluble scFv than in TG-1, but those that contain TAG stop codons will not produce any soluble scFv) (F1-F6). The expressed scFvs can then be used in ELISA. Detection of bound scFv can be performed using either Protein A-HRP15 or Protein L-HRP16 conjugates.1. From each selection take 10 µl of eluted phage and infect 200 µl exponentially growing HB2151bacteria (OD 600 of 0.4) for 30 min at 37°C in a water bath. Plate 50 µl, 50 µl of a 1:102 dilution,50 µl of a 1:104 dilution and 50 µl of a 1:106 dilution on TYE plates containing 100 µg/mlampicillin and 1 % glucose and grow overnight at 37°C.2. Pick individual colonies into 100 µl 2xTY 100 µg/ml ampicillin and 1 % glucose in 96 cell-wellplates and grow shaking (250 rpm) overnight at 37°C./doc/d1ca22bb960590c69ec376eb.html e a 96 well transfer device14 to transfer a small inocula (about 2 µl) from this plate to a second96 cell-well plate containing 200 µl 2xTY containing 100 µg/ml ampicillin and 0.1 % glucose perwell. Grow shaking (250 rpm) at 37°C until the OD 600 is approximately 0.9 (about 3 hr). (A stock can be made of the first plate, by adding glycerol to a final concentration of 15 % and storing at -70°C).4. Once OD 0.9 is reached (wells look quite cloudy) add 25 µl 2xTY containing 100 µg/ml ampicillinand 9 mM IPTG (isopropyl β-D-thiogalactoside, final concentration 1 mM IPTG). Continueshaking (250 rpm) at 30°C overnight.5. Coat a 96 well flexible assay plate overnight with 100 µl per well of antigen in the same buffer andat the same concentration as used for selection.6. Spin the plate from step F4 at 1,800 g for 10 min and use 50 µl of the supernatant (take care notto transfer any bacteria) for ELISA in 3% BSA-PBS (final concentration) (protocol E) except usinga 1:5000 dilution of Protein A-HRP15 or Protein L-HRP16 to detect binding in step E5.µl TG1 at an OD 600 of 0.4 with 10 µl of 100-fold serial dilutions of KM13 helper 2001. Infectphage3 (in order to get well separated plaques) in a 37°C water bath (without shaking) for 30 min.Add to 3 ml molten H-top agar (42°C) and pour onto warm TYE plates (no antibiotics). Allow to set and incubate overnight at 37°C.2. Pick a small plaque into 5 ml of fresh TG1 at an OD 600 of 0.4. Grow for about 2 hr shaking at37°C.3. Add to 500 ml 2xTY in a 2 litre flask and grow shaking at 37°C for 1 hr. Add kanamycin to a finalconcentration of 50 µg/ml (no glucose). Grow overnight shaking at 30°C.4. Spin overnight culture at 10,800 g for 15 min. Add 100 ml PEG/NaCl (20 % polyethylene glycol6000, 2.5 M NaCl) to 400 ml supernatent and leave for 1 hr on ice.5. Spin 10,800 g for 30 min. Pour away PEG/NaCl.6. Resuspend the pellet in 8 ml PBS and add 2 ml PEG/NaCl. Mix well and leave for 20 minutes onice.7. Spin 3,300 g for 30 min. Respin briefly and aspirate any remaining dregs of PEG/NaCl.8. Resuspend pellet in 5 ml PBS and spin at 11,600 g for 10 min in a micro centrifuge to remove anyremaining bacterial debris.9. Store the helper phage at 4°C for short term storage or in PBS with 15 % glycerol for longer termstorage at -70°C.10. To titre the helper phage take 45µl phage and add 5µl trypsin stock solution. Incubate for 30 minsat 37 °C. Dilute 1µl of trypsin treated phage in 1ml PBS and make five 100 fold serial dilutions of this in 1ml aliquots of PBS. Add 50µl of the six dilutions to six separate tube containing 1ml of TG1 at an OD 600 of 0.4. Mix, add 3 ml molten H-Top and pour evenly onto TYE plates.Perform the same dilution series using 1µl of non-trypsin treated phage. The titre of the trypsin treated phage should be 105-108 lower than for the non-trypsin treated phage. If not, pickanother plaque and repeat helper phage preparation.Once the libraries have been selected you may wish to check individual clones for the presence of full length VH and Vκinsert. All PCRs are at annealing temperature of 55°C. 1 min extension for V H or Vκon their own, 2 min extension for V H and Vκ together.For V H only use LMB3: CAG GAA ACA GCT ATG AClink seq new: CGA CCC GCC ACC GCC GCT Gwith insert = 527 bpwithout insert = 227 bpFor Vκ only use DPK9 FR1 seq: CAT CTG TAG GAG ACA GAG TCpHEN seq: CTA TGC GGC CCC ATT CAwith insert = 368 bpwithout insert = no bandFor V H and Vκ use LMB3: CAG GAA ACA GCT ATG ACpHEN seq: CTA TGC GGC CCC ATT CAwith insert = 935 bpwithout insert = 329 bpFor sequencing of selected clones the following primers are recommended.For V H use link seq new CGA CCC GCC ACC GCC GCT GFor Vκ use pHEN seq CTA TGC GGC CCC ATT CAXhoI NotIRBS CAGGAAACAGCTATGACCATGATTACGCCAAGCTTGCATGCAAATTCTATTTCAAGGAGACAGTCATA ATG AAA TAC CTA ----------------> M K Y L LMB3SfiI __NcoI__TTG CCT ACG GCA GCC GCT GGA TTG TTA TTA CTC GCG GCC CAG CCG GCC ATG GCC GAG GTG TTT L P T A A A G L L L L A A Q P A M A E V F XhoI linkerGAC TAC TGG GGC CAG GGA ACC CTG GTC ACC GTC TCG AGC GGT GGA GGC GGT TCA GGC GGA GGTD Y W G Q G T L V T V S S G G G G S G G GSalI NotI HIS-tag GGC AGC GGC GGT GGC GGG TCG ACG GAC ATC CAG ATG ACC CAG GCG GCC GCA CAT CAT CAT CACG S G G G G S T D I Q M T Q A A A H H H H<------------------------link seq newmyc-tagCAT CAC GGG GCC GCA GAA CAA AAA CTC ATC TCA GAA GAG GAT CTG AAT GGG GCC GCA TAG ACTH H G A A E Q K L I S E E D L N G A A * T<---------------------pHEN seq1. 2xTY is 16g Tryptone, 10g Yeast Extract and 5g NaCl in 1 litre.2. Bovine ubiquitin (5 mg) is available from Fluka, Chemika-BioChemika, Industriestrasse 25, CH-9470 Buchs, Switzerland, Tel +41 81 755 2511, Fax +41 81 756 5449.3. KM13 is the protease cleavable helper phage described i n Kristensen and Winter, Folding andDesign3, 321-328 (1998).4. PBS is5.84 g NaCl, 4.72 g Na2HPO4 and 2.64 g NaH2PO4.2H20, pH 7.2, in 1 litre.5. TYE is 15g Bacto-Agar, 8g NaCl, 10g Tryptone, 5g Yeast Extract in 1 litre.6. Nunc Maxisorp immuno test tubes (Cat. No. 4-44202) are available from Gibco BRL, LifeTechnologies Ltd., P. O. Box 35, Trident House, Washington Road, Paisley, PA3 4EF, Scotland, U.K, Tel +44 141 814 6100, Fax +44 141 887 1167.7. 'Marvel' is dried skimmed milk powder.8. Trypsin (T-1426 Type XIII from Bovine Pancreas - Sigma Chemical Company Ltd., Fancy Rd.,Dorset, BH17 7NH, U.K, Tel +44 1202 733114; Fax +44 1202 715460) made up in 50mM Tris-HCl pH7.4, 1mM CaCl2 and stored at -20°C9. Nunc Bio-Assay dish is available from Gibco BRL (see note 6).10. Falcon MicroTest III flexible 96 well flat bottomed assay plates are available from BectonDickinson Labware, Becton Dickinson and Co., 2 Bridgewater Lane, Lincoln Park, NJ 07035,USA.11. HRP-anti-M13 is available from Amersham International plc, Amersham Place, Little Chalfont,Buckinghamshire, HP7 9NA, UK. Tel: +44 01494 544000; Fax: +44 01494 542929.12. TMB is 3,3',5,5'-tetramethylbenzidine and is available from Sigma (see Note 8). A 10 mg/ml stocksolution can be made by dissolving the TMB in DMSO.13. Corning 'Cell Wells' flat-bottomed multiple well tissue culture treated plates are available fromCorning Glass Works, Corning N.Y. 14831. USA.14. The 96 well transfer device is a piece of wood the size of a microtitre plate with a handle on oneside and 96 metal pins (each 7 cm long with a concave end) on the other. This can be sterilised between bacterial transfer by immersion in a bath of ethanol and then by flaming (hold well away from your body and any flamable objects). If you haven't got one of these (or something similar) you will have to make one yourself or alternatively use a multichannel pipette for bacterialtransfer.15. Horse Radish Peroxidase conjugated Protein A is available from Amersham International plc (seenote 11)14. Horse Radish Peroxidase conjugated Protein L is available from Actigen Ltd, 5 Signet Court,Swanns Road, Cambridge, CB5 8LA, UK. Tel: +44 01223 319101; Fax: +44 01233 316443.。
噬菌体抗体库筛选技术及在肿瘤研究中的应用

综述#讲座噬菌体抗体库筛选技术及在肿瘤研究中的应用薛国柱,吕勇刚,窦科峰第四军医大学西京医院肝胆外科,陕西西安710032Selection technology of phage antibody library and its application in tumor researchX UE Guo-z hu,L U##Yong-gang,DO U K e-f engDep artment of H ep ato-biliar y Surgery,X ij ing H osp ital,Four th M ilitar y Medical University,X i.an710032,P.R.China=摘要>噬菌体抗体是指表达在噬菌体表面的抗体分子Fab或scF v,这些分子的群体称为抗体库。
经特定抗原或细胞筛选后可以获得针对该抗原或细胞表面标志物的特异性抗体。
经典的筛选方法为固相或液相抗原识别,前提条件是能得到抗原纯品,对于抗原无法提纯或抗原性质不确定的情况则不能适用,所以又出现全细胞筛选,直接利用肿瘤细胞或组织细胞进行筛选,无需纯抗原,但目标抗原一定要有较高的表达水平,对于表达较低的情况,一种策略是先利用正常细胞筛选,去掉无关抗原后,再利用肿瘤细胞筛选;另一种策略是细胞内化筛选,特异性抗体被内化入细胞,然后用酸性洗脱液洗脱掉细胞膜上非特异结合的噬菌体抗体后裂解细胞,获得特异性抗体。
其余筛选方法还有切片组织筛选、体内筛选等,适用面较窄,未获推广。
筛选效率主要通过以下几项的检测:转化数,即被感染的噬菌体数,它随筛选轮数的增加而增加;抗体基因插入载体的频率,高比例的丢失意味着筛选的低效率;抗体亲和性。
抗体库技术避免了杂交瘤技术中的许多难点,如细胞融合、动物免疫等,目前该技术已逐渐在肿瘤诊断和肿瘤治疗中发挥重要作用。
肿瘤防治杂志,2005,12(23):1829-1832[ABSTRAC T]P hag e antibo dy means tho se F ab o r scF v, w hich display in the membrane of phag es.T he reper toires o f these antibo dies ar e antibo dy librar y.Bio pinned by antig en or cell,r elevant antibo dy can be g ot.Classical bio panning tech-nique is antig en(so lidif ied o r liquefied)recog nitio n by pur ified antig en,when the purif ied antig en can not be obtained,the classical technique can not be applied.So full cell biopanning has appeared,w hich needs not pur ify antig en,but the t arg et antig en must be highly ex pressed,o therw ise,some str ategy can be used.O ne is wiping off the irrespect ive ant igen by no r-mal cells,then tumor cell antig en is r elatively high,another str ateg y is inter nalizing biopanning:special antibody is inter-nalized by certain technique,then the ir relat ive ant ibo dy is w ashed off,special antibo dy is g ot by splitting the cells.Oth-er techniques such as in tissue sect ions selection and in viv o selectio n w ere na rro wly applied.T he bio panning efficiency is detected by fo llo w ing aspects:tr ansfo rming unit,enhanced w ith the r ound of bio panning;the frequency of g ene fusing, hig h delet ion of antibody g ene meaning lo w efficiency;the af-finity o f antibody.T he antibody librar y technique can avo id many pr oblems of hy bridoma such as cell fusio n and animal immunity.It has been used in tumo r diagnosis and tr eat ment.Chin J Cance r Pr ev T r eat,2005,12(23):1829-1832=关键词>肿瘤/免疫学;噬菌体/免疫学;抗体,细菌;综述文献[KEYWORDS]neoplasms/immunolog y;bacter iophages/immunolog y;ant ibo dies,bacter ial;r ev iew lit erature =中图分类号>R730.3=文献标识码>A=文章编号>1009-4571(2005)23-1829-04噬菌体抗体是指表达在噬菌体表面的抗体分子Fas或单链抗体ScFv。
噬菌体筛选过程

噬菌体筛选过程
噬菌体筛选过程是指通过一系列实验方法和结果的分析,找出具有高效寄生能力和选择性的噬菌体。
该过程通常包括以下几个步骤: 1. 菌株的选择:首先需要选择一个目标菌株,并确定其生长条件和培养基。
这些条件应该能够满足噬菌体的生长和寄生需求。
2. 噬菌体的初步筛选:在满足菌株生长条件的基础上,使用不同的噬菌体悬液进行初步筛选。
评价指标通常包括噬菌效率、寄生速度和选择性等。
3. 噬菌体的进一步筛选:根据初步筛选结果,选择效果较好的噬菌体进行进一步筛选。
该阶段通常包括寄生速度、选择性、感染范围等指标的评估。
4. 噬菌体的鉴定和纯化:在得到优良噬菌体后,需要进行鉴定和纯化工作,以保证筛选得到的噬菌体纯度和有效性。
5. 噬菌体的应用:最后,筛选得到的噬菌体可以应用在医疗、农业等领域,例如治疗细菌感染、控制农业有害菌等。
- 1 -。
第 4 章 GB2随机突变噬菌体文库的构建及淘选
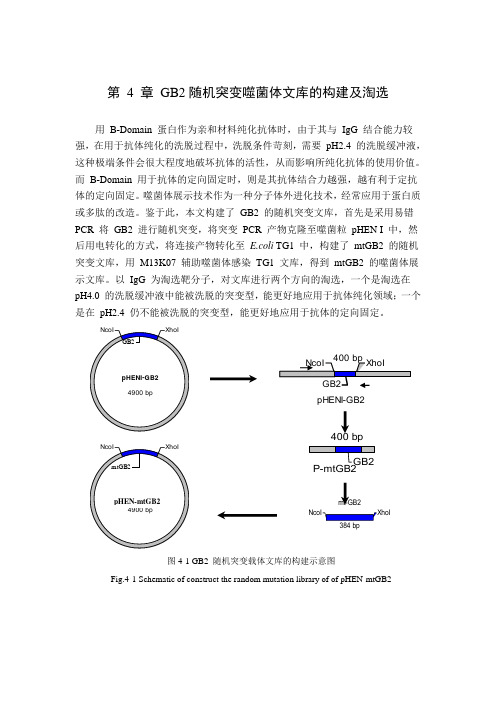
第 4 章 GB2随机突变噬菌体文库的构建及淘选 用 B-Domain 蛋白作为亲和材料纯化抗体时,由于其与 IgG 结合能力较强,在用于抗体纯化的洗脱过程中,洗脱条件苛刻,需要 pH2.4 的洗脱缓冲液,这种极端条件会很大程度地破坏抗体的活性,从而影响所纯化抗体的使用价值。
而 B-Domain 用于抗体的定向固定时,则是其抗体结合力越强,越有利于定抗体的定向固定。
噬菌体展示技术作为一种分子体外进化技术,经常应用于蛋白质或多肽的改造。
鉴于此,本文构建了 GB2 的随机突变文库,首先是采用易错 PCR 将 GB2 进行随机突变,将突变 PCR 产物克隆至噬菌粒 pHEN I 中,然后用电转化的方式,将连接产物转化至 E.coli TG1 中,构建了 mtGB2 的随机突变文库,用 M13K07 辅助噬菌体感染 TG1 文库,得到 mtGB2 的噬菌体展示文库。
以 IgG 为淘选靶分子,对文库进行两个方向的淘选,一个是淘选在 pH4.0 的洗脱缓冲液中能被洗脱的突变型,能更好地应用于抗体纯化领域;一个是在 pH2.4 仍不能被洗脱的突变型,能更好地应用于抗体的定向固定。
pHENI-GB2400 bp mt-GB2图4-1 GB2 随机突变载体文库的构建示意图 Fig.4-1 Schematic of construct the random mutation library of of pHEN-mtGB2pH6.0洗脱缓冲液洗脱淘选多轮淘选多轮图4-2亲和力减弱文库 A 淘选过程示意图 Fig. 4-2 Schematic of the panning of library A图4-3 亲和力增强文库 B 淘选过程示意图Fig. 4-3 Schematic of the panning of library B4.1 材料、仪器与试剂4.1.1材料E.coli TG1 购自Invitorgen、噬菌粒pHEN I、Help phage M13K07 由英国剑桥医学研究委员会(MRC) 分子生物学实验室Greg Winter 博士惠赠;小鼠腹水由本实验室制备。
噬菌体筛选

噬菌斑分析1、将合适的宿主菌接种到M9TB培养基上,37℃摇至0D600=1.0;2、使用前,可将宿主菌保存在4℃,保存超过48h的菌不可使用;3、熔化足够量的顶层培养基,每个平板需5ml,将熔化后的培养基放在45-55℃的水浴锅中;4、使用无菌LB培养基制备一系列稀释度的样品。
通常选择103-106的重组噬菌体稀释度。
空白对照则选择107;10-210-310-710μL样品100μL …………990μL 900μL5、准备4mL的无菌管,加入250μL的宿主菌。
从最高稀释度开始加起,每管加入100μL噬菌体稀释液。
注意换枪头;6、在每管中加入3mL的顶层培养基,混合后倒在无抗LB/LB carb+/LB carb+amp+平板上,轻摇使其迅速铺平;7、平板静置几分钟后,翻转,37℃培养3-4h或室温过夜;8、数噬菌斑数并计算噬菌体滴度。
如:稀释度为10-6的平板上有200个噬菌斑,则滴度为200×106×10=2×109pfu/mL样品中噬菌体总数由滴度×样品总量决定。
如:1μg的载体DNA被包装,最终的包装反应体积为0.3mL(25μL包装提取物+5μL连接物+270μL无菌培养基)。
在包装反应中pfu总数为2×109pfu/mL×0,3mL=6×108pfu。
另外,因为使用了1μg的载体,效率是6×108pfu。
文库扩增当原始重组体总数少于5×106,选择平板裂解法;当需要大量扩增时,选择液体裂解法。
平板裂解扩增1、接种1mL过夜培养的宿主菌到50mL TB培养基,37℃摇至OD600=0.6-1.0,如果使用5615菌,当OD600=0.5时,添加IPTG至终浓度1mM,然后继续摇30min;2、如果包装反应保存在氯仿里,轻微离心分离出氯仿,将噬菌体吸出;3、计算需要的细胞和噬菌体量。
每10mL细胞含1×106,将噬菌体和细胞混合,加入50mL培养基,这些混合物需在20min内倒入平板。
噬菌体抗体淘筛方法

这两个文库来自于单个人的V H和Vκ的基本结构,具有和那些已知结构的抗原结合位点的侧链多样性,并且在成熟的体系中中具有很高的多样性。
由这个框架来编码的标准结构是目前人抗体技术体系中最普通的。
在能够形成抗原结合表面的前提下重链的CDR3设计的尽可能短。
这两个文库都可以在未知筛选的克隆的序列的情况下进行筛选和免疫亲和力成熟。
这两个文库是噬菌粒/单链片段可变区格式的,并且都经过与蛋白A和蛋白L的结合筛选,所以未筛选的克隆的大多数都是具有功能的。
TomlinsonI构建在plT2载体((HIS myc 标签),多样化的侧链主要是来自于原始体系中多样性的位点(一共18个残基,H50, H52, H52a, H53, H55, H56, H58, H95, H96, H97, H98, L50, L53, L91, L92, L93, L94 and L96)此文库经过筛选后与体细胞突变的多样性的共同作用使其成熟。
我们保证上述信息在没有另外通知之前是严格可信的。
使用此文库之前请认真阅读以下材料确认收到以上材料2. 确认收到的文库未溶解,且使用之前-70摄氏度保存。
3. 参照方案G制备KM134. 在使用文库前清仔细阅读这个方案:在含有100 μg/ml氨苄青霉素和1 %葡萄糖的TYE平板上将对照划线培养,置于培养箱37℃过夜培养,挑单菌落置于摇床,接种在5ml含有100 μg/ml氨苄青霉素和1 %葡萄糖的2倍TY培养基内,37℃过夜培养。
分别制备阳性和阴性的噬菌体对照(第一天到第十天过夜用500微升)。
用阳性对照和阴性对照产生的噬菌体按照1:100的比例混合,进行一轮的筛选,用100 μg/ml的遍在蛋白在PBS中包被。
通过单克隆噬菌体ELISA检验遍在蛋白的富集程度(经过第一轮筛选后应该超过50%)5. 尽可能的用专用的移液管和一次性的手套,由于噬菌体会非特异的吸附到其他的塑料上,建议使用聚丙烯管。
6. 为提高感染效率,大肠杆菌应该在37℃培养至对数生长期(600nm的OD值应该在0.4)(1)、从一个小型的培养板上转移一个细菌克隆至5ml的2倍TY培养基(不加抗生素和葡萄糖),37℃震荡过夜培养(2)、次日稀释100倍至新鲜的2倍TY培养基内,震荡培养至培养至对数生长期600nm的OD值应该在0.4。
用不同方法从大容量噬菌体抗体库中筛选抗完整食管癌细胞的单链抗

用不同方法从大容量噬菌体抗体库中筛选抗完整食管癌细胞的
单链抗
噬菌体抗体库是一种非常强大的生物技术工具,可以用于筛选出针对特定疾病、特定抗原的单链抗体。
在搜索抗完整食管癌细胞的单链抗体时,可以采用以下几种方法。
第一种方法是基于表面展示技术的筛选。
此方法利用噬菌体表面的展示蛋白,将特定单链抗体的DNA序列插入其中,并让
噬菌体体表面展示这种单链抗体分子。
接下来,将经过筛选的噬菌体与完整食管癌细胞接触,将其在体外培养。
通过重复筛选和挑选,可以得到经过筛选的却对完整食管癌细胞具有很强抗体效果的单链抗体。
第二种方法是利用串联抗体的筛选。
此方法先将多个单链抗体连接在一起,形成一个可变区域长的串联单链抗体,再将其插入噬菌体表面展示蛋白中。
同样的,将经过重复筛选和挑选,可以得到抗完整食管癌细胞效果很好的串联单链抗体。
第三种方法是利用功能筛选法。
此方法首先需要将完整食管癌细胞切成小碎片,然后使用这些碎片投放到噬菌体抗体库中,让这些碎片吸附在噬菌体表面,以模拟完整食管癌细胞在人体内的生存环境。
接下来,通过加入某些化合物或特定条件,可以筛选出对完整食管癌细胞有特异性的单链抗体。
综上所述,虽然每种筛选方法都各有优缺点,但我们可以选择最适合的筛选方法,以此从大容量的噬菌体抗体库中筛选出一种高效抗完整食管癌细胞的单链抗体,以提高食管癌治疗效果。
亲和层析法用于噬菌体抗体库的筛选

亲和层析法用于噬菌体抗体库的筛选陈瑛炜,罗文新,王明桥,王晋,李利峰,袁权,张军,夏宁邵(福建省医学分子病毒学研究中心,厦门大学细胞生物学与肿瘤细胞工程教育部重点实验室,厦门 361005)摘要:本研究报道一种基于固定化金属亲和层析(IMAC )的噬菌体抗体库液相筛选方法。
将纯化的带有His 标签的抗原与噬菌体抗体库混合,噬菌体抗体与抗原充分结合后再加入亲和介质,使噬菌体抗体抗原复合物通过His 标签与介质结合,然后通过充分洗涤去除非特异性噬菌体抗体,最后将特异性噬菌体抗体洗脱下来,感染TG 1,进行下一轮筛选。
整个筛选过程中抗原与抗体的结合在液相中完成,不仅消除了固相介质对抗原表位的影响,也更有利于噬菌体抗体与抗原的充分作用。
将此方法应用于HEV N E2蛋白特异性人源噬菌体抗体的筛选,抗原竞争EL ISA ,阳性血清阻断,可溶性单链抗体表达检测及测序结果表明,最终获得2个特异性人源抗体。
关键词:噬菌体抗体库;固定化金属亲和层析;液相筛选中图分类号:R373;Q78 文献标识码:A 文章编号:1000-8721(2006)01-0044-06收稿日期:2005-04-27;修回日期:2005-07-12基金项目:福建省科技重大项目(编号:2002F013);国家十五创新药物博士基金(编号:2003AA2Z3539);福建省自然科学基金(编号:C0310005);厦门市科技计划项目(编号:3502Z20055002)。
作者简介:陈瑛炜(1982-),女,硕士,从事基因工程抗体研究。
通讯作者:罗文新,副教授,Tel 186259222184113;Fax :86259222181258;E 2mail :wxluo @jingxian 1xmu 1edu 1cn 噬菌体抗体库技术已经成为目前获得人源抗体的主要途径之一。
从大容量天然人源噬菌体抗体库中,理论上可以获得针对任何抗原的人源单抗。
噬菌体抗体库筛选方法包括纯抗原筛选,非纯抗原筛选,功能性筛选,选择性感染筛选等[1]。
噬菌体抗体库筛选技术研究进展

噬 菌 体 展 示 技 术 ( hg i l eh o g ) P aeds a tcnl y 是 py o 近年 来新 兴起 的一 项 分 子 生 物学 技 术 , 基 本 原 理 其
是应 用 基 因工 程 手 段把 外 源 D A片 段 克 隆到 适 当 N
体 抗体库 分 天然库 、 合成 库 和特免 库 , 特免抗 体库 来 源 于经 特异抗 原免 疫 过 的动 物 , 较 容 易从 中筛 选 可 获 得高 亲和力 的特 异性 抗体 。天 然库 与合成 库则 需 要 抗 体 库 容 量 足 够 大 , 有 达 到 l ~1m以 上 只 0 0
来筛 选 的靶 分 子更是 多种 多样 , 有单 一靶 抗 原分 子 ,
部 分 可通过 噬菌 体 D A序 列测 定得 到 , 样就 建 N 这
立 了基 因型 和表 现型 之 间的对应 关 系 。如 果将 编码 抗 体分 子 的基 因与噬 菌体外 壳 蛋 白基 因融 合 ,使表 达 的抗 体附着 在 噬菌体 颗粒 表 面 , 形 成 了噬 菌体 就
・
7 ・ 0
微生物学免疫学进展 2 1 年第 4 02 0卷第 1 期
Po i M c b lm u o Fb 2 1 , o 4 , o 1 r i o i m nl e . 0 2 V 1 0 N . gn r oI .
・
综
述 ・ Βιβλιοθήκη 噬 菌体 抗 体 库 筛 选 技 术 研究 进展
毕 司英 综述 ; 晓燕 审校 毛
( 州 生 物制 品研 究 所 , 州 7 0 4 ) 兰 兰 30 6
摘要 :噬菌体抗体库 技术 是一项新 兴的基因工程抗体技术 , 应用这项 技术获得 高特异性抗体 的关键 之一就是筛选 环节 。根据抗原性质 以及筛选 目的的不 同 , 筛选方法的选择 也不相 同 , 种筛选 策略 的优化对 中和抗体 的获得 有 各
噬菌体展示肽库的筛选方法及其应用

噬菌体展示肽库的筛选方法及其应用1985年,SmithGP利用基因工程手段将一段外源肽序列展示在丝状噬菌体的表面[1]。
1988年[2]他们又将合成的随机序列的寡核苷酸片段克隆到丝状噬菌体,表达后每个噬菌体粒子的表面展示一种肽段,所有这些展示不同肽段的噬菌体构成了噬菌体展示肽库。
1990年,他们通过亲合筛选,得到了与特定蛋白结合的结合肽,并由于噬菌体表达的肽与编码基因直接相关,扩增和分离目的克隆后,很容易得到其DNA序列[3]。
这样就建立了噬菌体表面展示的随机肽库技术,这项技术一经产生就显示其无与伦比的生命力,被广泛用于生命科学的各个领域,并带来广泛而深远的影响。
传统的药物筛选大多数是从自然界的动、植物及微生物中分离天然的具有特定药理作用的化学物质,然后直接应用或再以此作为药物化学的先导化合物,再进一步设计、加工、合成,筛选有效的功能药物。
此方法具有一定的盲目性,筛选周期长。
而采用分子进化工程技术则会大大加速这一过程。
根据所需要的药物特性,选用适当的方法构建含有大量异质性分子的组合库,用靶分子进行筛选,先筛选药物先导化合物,然后进一步优化设计,最终确定候选的药物结构。
近年来,引入组合策略和模拟进化思想,建立了一种从噬菌体随机肽库中筛选药物先导化合物的新方法[4],即用库容量极大的随机肽库去快速筛选具有较高特异性和亲和力的理想目的肽。
通过此种方法可以快速筛选生物活性肽、蛋白质、受体及其他化合物等新型药物或先导化合物。
这一方法具有传统的药物筛选无法比拟的优越性,将药物开发带入了一个崭新的时代。
1噬菌体展示系统的建立早在1986年Geysen就认为含有关键残基的短肽能够模拟蛋白质上的决定族。
在多数情况下,几个关键残基与它的结合分子所形成的非共价键构成了全部结合的主要部分,即蛋白质之间的相互作用或识别是通过局部残基肽段间的相互作用来实现的。
1982年,Dulbecco提出将病原体的免疫原与λ噬菌体和其他病毒的衣壳蛋白融合,便可产生能够用作疫苗的表面展示外来多肽的病毒颗粒。
噬菌体库固相-液相筛选的标准操作规程

噬菌体库固相-液相筛选的标准操作规程(编号:063)
1、目的及适用范围
为了避免筛选到非特异性结合的噬菌体克隆,采用轮换淘选法,主要用于噬菌体展示库的初步筛选。
2、主要仪器
酶联条、微量移液器、摇床、垂直混匀仪、三角烧瓶、37℃摇床
3、试剂及配制方法
3.1 1×TBS配方:
Tris-HCI缓冲液(0.5M pH7.6)100mL
NaCl 8.5-9g
双蒸水加至1000mL
先以少量双蒸水溶解NaCl,再加入Tris-HCl缓冲液,最后加双蒸水至1000mL,充分摇匀。
3.2 1×TBST(含0.05%Tween20):
1000mL1×TBS中加入0.5mL Tween20
3.3 包被液:(pH 9.6 0.05 M碳酸盐缓冲液)
Na2CO3 1.59g
NaHCO3 2.93g
加蒸馏水至 1000mL
溶解后每瓶50mL分装,高压灭菌备用。
3.4 5%[W/V]BSA:100mL1×TBS中加入5g BSA。
3.5 2 M Tris·Cl(pH8.0):242g Tris置于800mL超纯水中,溶解后,HCl调pH值至8.0,再用超纯水定容至1000mL。
3.6 0.2 M Gly-HCl(pH2.2):15g glycine 置于800mL超纯水中,溶解后,HCl调pH值至2.2,再用超纯水定容至1000mL。
3.7 2×YT:
胰蛋白胨16g/L
酵母提取物10g/L
氯化钠5g/L
根据经验值用NaOH调节该培养基的pH,使其达到7.4(该pH适合目前使用最广的原核表达
128。
噬菌体库的筛选原理

噬菌体库的筛选原理
噬菌体库(phage library)是一种用于筛选特定目标的噬菌体(phage)的方法。
其筛选原理如下:
1. 构建噬菌体库:从已知的DNA或RNA样品中,随机将目标序列片段插入噬菌体的基因组中,并将这些基因组片段包裹在噬菌体的外壳中。
构建好的噬菌体库含有许多噬菌体,每个噬菌体携带着不同的目标序列片段。
2. 筛选目标:将噬菌体库与特定靶点进行反应,例如与靶蛋白质结合。
未结合的噬菌体可以被洗去,而与靶点结合的噬菌体则保留下来。
3. 提取目标噬菌体:将与靶点结合的噬菌体进行扩增,使其数量增加。
通常使用大肠杆菌等宿主细胞进行扩增。
4. 再次筛选:将扩增后的目标噬菌体再次与靶点进行反应和洗脱,以进一步筛选出特异性更高的噬菌体。
5. 验证筛选结果:对所得的单个目标噬菌体进行验证,确认其与靶点的结合能力和特异性。
噬菌体库筛选原理的核心是利用噬菌体随机插入外源基因的特点,通过与目标分子结合的能力将其筛选出来。
这种方法被广泛应用于蛋白质相互作用、抗体产生
和蛋白质工程等领域。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
这两个文库来自于单个人的V H和Vκ的基本结构,具有和那些已知结构的抗原结合位点的侧链多样性,并且在成熟的体系中中具有很高的多样性。
由这个框架来编码的标准结构是目前人抗体技术体系中最普通的。
在能够形成抗原结合表面的前提下重链的CDR3设计的尽可能短。
这两个文库都可以在未知筛选的克隆的序列的情况下进行筛选和免疫亲和力成熟。
这两个文库是噬菌粒/单链片段可变区格式的,并且都经过与蛋白A和蛋白L的结合筛选,所以未筛选的克隆的大多数都是具有功能的。
TomlinsonI构建在plT2载体((HIS myc 标签),多样化的侧链主要是来自于原始体系中多样性的位点(一共18个残基,H50, H52, H52a, H53, H55, H56, H58, H95, H96, H97, H98, L50, L53, L91, L92, L93, L94 and L96)此文库经过筛选后与体细胞突变的多样性的共同作用使其成熟。
我们保证上述信息在没有另外通知之前是严格可信的。
使用此文库之前请认真阅读以下材料确认收到以上材料2. 确认收到的文库未溶解,且使用之前-70摄氏度保存。
3. 参照方案G制备KM134. 在使用文库前清仔细阅读这个方案:在含有100 μg/ml氨苄青霉素和1 %葡萄糖的TYE平板上将对照划线培养,置于培养箱37℃过夜培养,挑单菌落置于摇床,接种在5ml含有100 μg/ml氨苄青霉素和1 %葡萄糖的2倍TY培养基内,37℃过夜培养。
分别制备阳性和阴性的噬菌体对照(第一天到第十天过夜用500微升)。
用阳性对照和阴性对照产生的噬菌体按照1:100的比例混合,进行一轮的筛选,用100 μg/ml的遍在蛋白在PBS中包被。
通过单克隆噬菌体ELISA检验遍在蛋白的富集程度(经过第一轮筛选后应该超过50%)5. 尽可能的用专用的移液管和一次性的手套,由于噬菌体会非特异的吸附到其他的塑料上,建议使用聚丙烯管。
6. 为提高感染效率,大肠杆菌应该在37℃培养至对数生长期(600nm的OD值应该在0.4)(1)、从一个小型的培养板上转移一个细菌克隆至5ml的2倍TY培养基(不加抗生素和葡萄糖),37℃震荡过夜培养(2)、次日稀释100倍至新鲜的2倍TY培养基内,震荡培养至培养至对数生长期600nm的OD值应该在0.4。
(1.5-2小时)7. 所有的离心,除了在微型离心机中操作的之外,都是在4℃下进行。
8.两个文库最好是同时使用,以保证筛选得到最多的抗原结合克隆。
In advance准备好用到的仪器和试剂(详见方案后注解),确保有足够的用于细菌增殖的培养基(大的生物分析的培养皿在使用前进行灭菌和干燥处理)在实验之前仔细阅读这个方案,以便安排你的时间同时进行操作Steps ProcedureDay 1 (5 hrs) A1-A6 Grow libraries I and J and make phageB1-B3 Make secondary stock of libraries第一天A1-A6 增殖文库I和J,并且制备噬菌体B1和B3 制作文库的第二原种Day 2 (6 hrs) A7-A12 Grow libraries I and J and make phage (cont.)C1 Coat immunotubes for 1st round of selection第二天A7-A12 增殖文库I和J,并且制备噬菌体C1 包被第一轮筛选用免疫管Day 3 (6.5 hrs) C2-C11 1st round of selection第三天C2-C11 第一轮筛选Day 4 (3 hrs) D1-D6 Make phage from 1st round of selectionC1 Coat immunotubes for 2nd round of selection第四天D1-D6 从第一轮筛选的结果中制备噬菌体C1 包被第二轮筛选用免疫管Day 5 (6.5 hrs) D7-D11 Make phage from 1st round of selection (cont.)C2-C11 2nd round of selection第五天D7-D11 从第一轮筛选的结果中制备噬菌体(继续)C2-C11 第二轮筛选Day 6 (3 hrs) D1-D6 Make phage from 2nd round of selectionC1 Coat immunotubes for 3nd round of selection第六天D1-D6 从第二轮筛选的结果中制备噬菌体(继续)C1 包被第二轮筛选用免疫管Day 7 (6.5 hrs) D7-D11 Make phage from 2nd round of selection (cont.)C2-C11 3rd round of selection第七天D7-D11 从第二轮筛选的结果中制备噬菌体(继续)C2-C11 第三轮筛选Day 8 (3 hrs) D1-D6 Make phage from 3rd round of selectionE1 Coat 96 well plate for polyclonal phage ELISA第八天D1-D6 从第三轮筛选的结果中制备噬菌体E1 包被多克隆噬菌体ELISA用的96孔板Day 9 (6.5 hrs) D7-D11 Make phage from 3rd round of selection (cont.)E2-E8 Polyclonal phage ELISA第九天D7-D11 从第三轮筛选的结果中制备噬菌体(继续)E2-E8 多克隆噬菌体ELISA每个克隆的进一步的描述可以通过单克隆噬菌体ELISA(方案E),单克隆噬菌体ELISA用水溶性的scFv 片段(方案F)。
PCR检测(检测插入,方案H)和测序(方案I)1. 将文库的加入到200ml预热的含有100 μg/ml氨苄青霉素和1 %葡萄糖的2xTY培养基上2. 37°C振荡培养至OD 600为0.43. 从中取50ml,加入2x1011 KM13辅助噬菌体(剩余的150ml在方案B中用来制备文库的第二细菌储存液)4. 37°C水中温浴30分钟5. 3000g,离心10min,用100ml含有100 μg/ml氨苄青霉素、50 μg/ml的卡那霉素和1 %葡萄糖的2xTY培养基6. 30度振荡过夜培养7. 将过夜产物在3300g离心30分钟8. 取上层液体80ml,加入20mlPEG/NACL(含20 % PEG,2.5 M NaCl)溶液9. 3300g离心30分钟,倒掉PEG/NACL溶液,重新离心,吸去剩余的PEG/NACL溶液10 用4mlPBS缓冲液悬浮沉淀,11600g离心10分钟,去掉任何剩余的细菌残渣11. 短期使用的话可以保存在4℃,长期保存的话置于含有15%甘油的PBS中,-70℃保存。
12. 将噬菌体稀释100倍,然后继续稀释至6个浓度,加入900μl OD 600 为0.4的TG1至每一个管中,37°C水浴30min,取每个稀释浓度的10μl加入到TYE培养基上,其中包含100 μg/ml氨苄青霉素和1 %葡萄糖,37摄氏度过夜培养。
浓度在1012-1013/ml,至少可以进行10次筛选1.将A3步骤中剩余的150ml液体,37摄氏度下振荡培养2小时2、10800g离心10分钟,用10ml含有15%甘油的2xTY悬浮3、每管500微升,分装20管,保存于-70℃。
每次制备的时候用其中一管。
这个仅限于你打算做超过10次筛选的时候。
或者,噬菌体可以用生物素酰化的抗原或者亲和层析来完成1、用目的抗原4ml过夜来包被免疫管。
包被的效率取决于抗原的浓度,温度和缓冲液,通常用抗原浓度10-100 μg/ml的PBS缓冲液。
2. 第二天用PBS缓冲液洗三次(倒入管内,然后马上倒出来)3. 用含有2%的脱脂奶粉的磷酸缓冲液注满管子,室温下孵化,置于工作台上两个小时候停止4. 用PBS缓冲液洗管3次5. 将1012- 1013噬菌体键入到4ml的含有2%的脱脂奶粉的磷酸缓冲液。
室温下旋转培养孵育60分钟,然后静止培养60分钟,弃掉上清液6. 用含有0.1%的吐温20的磷酸缓冲液洗管子10-20遍7. 甩干多余的PBS缓冲液,用含有50 μl of 10mg/ml胰蛋白酶的磷酸缓冲液500微升稀释噬菌体,用可翻滚的摇床室温培养10min.8. 取1.5mlOD值为0.4的大肠杆菌TG1,加入250 μl稀释后的噬菌体(剩余的4°C保存),37℃水浴30min..9. 吸取液体,及稀释100倍的,和稀释10000倍的溶液各10微升,加到含有100 μg/ml氨苄青霉素、和1 %葡萄糖的TYE培养基上,37度过夜培养来测定噬菌体10、第一轮筛选时如果用复合抗原,取剩余的TG1培养物,11600g离心5min。
将底部的细菌用1ml的2xTY,涂于大的圆形的Bio-Assay dish,包含100 μg/ml氨苄青霉素、和1 %葡萄糖的TYE培养基上。
如果用的是单个的半抗原,糖类或者蛋白质抗原及所有抗原的下面的步骤:取剩余的TG1培养物,11600g离心5min。
将底部的细菌用1ml的2xTY,涂于大的圆形的Bio-Assay dish,包含100 μg/ml 氨苄青霉素、和1 %葡萄糖的TYE培养基上。
11. 37°C过夜培养第一轮的筛选是最重要的。
任何的错误都会导致以后的错误。
每轮结束后最少能够得到100个火星的噬菌体,如果少于这个数量,说明可能出现了错误。
所以,试着用新鲜的TG1感染剩余的250ml噬菌体,如果这一步仍然少于100噬菌体,从C1开始重复筛选。
1. 过夜增殖以后,加7ml含有15%甘油的2 xTY到large square Bio-Assay dish或者加2ml到常规的培养皿,然后用玻璃涂布器松散细胞,彻底混匀。
将得到的50 μl细菌接种到50 ml包含100 μg/ml 氨苄青霉素、和1 %葡萄糖的2xTY中。
剩余的1ml保存在15%的甘油中,-70°C保存。
2. 振荡培养至OD600为0.43. 取10ml培养物,加入5x1010辅助噬菌体4 37°C水浴30min5. 3000g离心10分钟。
用含有50 ml包含100 μg/ml氨苄青霉素、和0.1 %葡萄糖的2xTY悬浮6. 30°C振荡培养过夜7. 将过夜产物3300g离心10分钟8. 加入10ml PEG/NaCl (20 % PEG 6000, 2.5 M NaCl)至40ml上清液。
