GeneTools 操作指南(中文)
GENEXAssistant3.5使用指导书

GENEX Assistant 3.5 使用指导书1、新建工程:打开GENEX Assistant 3.5在弹出新建工程窗口中选择LTE对工程进行命名并选择保存路径:2、导入工参:工程模板详见附件:3、导入LOG:4、解压LOG:解压后弹出窗口:5、K PI指标导出:显示KPI列表:6、阈值设定:修改后:SINR阈值设置:其他指标阈值调整如RSRP SINR可根据DT指导文档进行调整;7、调整图层:调整后:&异常事件分析:将异常事件拖入图层中,右侧图层标示Event Layer中会显示异常事件及对应的出现次数:双击某个异常事件,在弹出的对话框中点击确定,即弹出分析窗口:可根据个人分析习惯对分析窗口进行调整:调整后如下:9、GENEX Assistant后台KPI统计自定义设置操作:1)打开GENEX Assistan新建工程后,点击<Tools>,下拉菜单中选择<custom KPI>如下图2)进入<custom KPI>界面,点击左下角<Add>,选择点击<counting KPI>,如下图:3 )通过设置公式:覆盖率=分子/分母;其中分子={-110dbmvRSRP<0dbm}a nd{-3db<SINR<50db}的个数;分母={-150dbm<RSRP<0dbm}and{-20db<SINR<50db}的个数设置如下图:分子设置:进行vcounting KPI>界面后,在name里命名,然后点击<Add >,进行指标定义,选择下拉菜单<IE>点击<IE>®行里面进行指标Serving RSRP选择设置,设置好后点击0K。
在Con diti on里面会显示刚才RSRP设置范围,然后点击<and>键,再点击<IE>,在里面选择PCC SINR设置,设置后点击ok在Counting Rule界面,可看到刚刚设置Serving RSRP和PCC SINR的阀值范围情况,然后点击<Edit Action>进入<Counting Operation Expression>界面,设置如下,然后点击OK在回到Counting Rule界面,点击OK在回到<counting KPI>界面后,点击OK分子设置完成。
基因编辑工具及其使用教程

基因编辑工具及其使用教程基因编辑是一种先进的生物技术,它可以对生物体的基因组进行精确的修改和调整,从而改变其遗传特征和生物功能。
基因编辑工具是实现基因编辑的关键工具,其中最常用的工具是CRISPR-Cas9系统、TALEN和ZFN。
本文将分别介绍这些基因编辑工具的原理和使用方法,以帮助读者对其有更好的理解。
1. CRISPR-Cas9系统CRISPR-Cas9系统是一种基于细菌免疫系统的基因编辑技术。
它包括两部分:CRISPR序列和Cas9蛋白。
CRISPR序列是一段DNA片段,其中包含着特定的序列重复和间隔,这些序列能与目标基因的序列互补配对。
Cas9蛋白是一种酶,它可以识别CRISPR序列与目标基因序列之间的匹配,并在目标基因的特定位置上剪切DNA链。
使用CRISPR-Cas9进行基因编辑的步骤如下:1) 设计并合成目标基因的CRISPR序列和Cas9蛋白。
2) 将CRISPR序列和Cas9蛋白导入到目标细胞中。
可以使用载体来将CRISPR-Cas9系统导入到细胞中。
3) Cas9蛋白识别并与目标基因序列结合,形成Cas9-gRNA复合物。
gRNA是指导RNA,它可以与CRISPR序列配对,指导Cas9蛋白在目标基因的特定位置上进行剪切。
4) Cas9蛋白在目标基因的特定位置上剪切DNA链,从而导致基因组的突变。
这种突变可以是基因组上的缺失、插入或替换。
5) 细胞会利用自身修复机制修复被剪切的DNA链,从而实现基因编辑的目标。
2. TALENTALEN是一种由转录激活因子(TF)结合域和核酸酶结合域组成的基因编辑工具。
与CRISPR-Cas9系统相比,TALEN具有更高的特异性和更低的离靶效应。
使用TALEN进行基因编辑的步骤如下:1) 设计并合成目标基因的TALEN,其中每个TF结合域与基因的特定DNA序列相互作用。
2) 将TALEN导入到目标细胞中,通常是通过转染或电穿孔方法。
3) TALEN识别并与目标基因序列结合,形成TALEN-DNA复合物。
Genemapper-微卫星分析中文操作指南

Genemapper-微卫星分析中文操作指南GeneMapper 软件 微卫星分析中文简易操作手册微卫星分析流程:软件术语介绍:术语 定义Analysis Parameters 软件中分析样品所使用的特定设置;用于计算片段大小和基因 型,这些特定设置包括analysis method, size standard 和panel bin在marker 内设定等位基因的片段大小和颜色 bin set 等位基因特定实验条件下一组binmarker 微卫星标记,通过位点名称、片段大小、荧光标记和重复长度 进行设定 panel Marker 组合 kitpanel 组合设定微卫星分析方法:1. 登录软件2. 创建分析所需kit 、panel 和marker 等参数;3. 创建新文件,导入数据;4. 设置分析参数;5. 执行初始分析;5. 创建Bin 以及Bin Set ;分析并检查结果: 1. 编辑分析方法; 2. 分析数据; 3. 查看结果;打印并导出数据(可选): 1. 打印结果; 2. 导出结果。
一.设定微卫星分析方法:1.登录GeneMapper软件:1.1选择开始 -- All Programs -- Applied Biosystems --GeneMapper--GeneMapper ;或双击桌面快捷方式打开软件;1.2登录GeneMapper软件:用户名默认为gm,在Passwprd中输入密码,点击OK进入GeneMapper软件2创建分析所需kit、panel和marker等参数:2.1 点击软件上方工具栏Tools,选择Panel Manager;2.4 选中1 STR panel,点击2新建Marker,在3位置依次输入Marker Name(Marker名称) ,Dye Color (荧光标记种类) ,Min Size,Max Size (片段最小值和最大值) ,Marker Repeat (重复次数) ;2.5重复2.4步骤,完成所有Marker设置,点击OK保存;3. 创建新文件,导入数据:3 .1 点击1 新建文件夹,在2 Project Type中文件夹类型选择Microsatellite ,点击OK;3.2 导入数据:点击1 Add Samples To Project 添加数据,在弹出的对话框中选择待分析数据,通过3Add To List 添加到右边的面板中,点击4 Add 完成数据添加;4. 设置分析参数:4.1 完成数据添加后,在1 位置,Analysis Method中选择New Analysis Method新建分析方法;在弹出的对话框中选择分析类型2Microsatellite,点击OK确认;4.2 设置分析参数:在Analysis Method Editor中,点击1General,填写2Name(分析方法名称);之后点击3 Allele,在将4 Bin Set选择none;其它参数为软件默认,不需要修改;点击OK确认;4.3 选定好之前设定的分析方法,在后续的栏目中设定Panel 和SizeStandard(分子量内标);4.4 完成分析参数设定后,选中Analysis Method,Panel和Size Standard栏目,点击1File,选择2Fill Down;此时,Analysis Method,Panel 和Size Standard栏目会使用之前选定的分析参数;也可以使用鼠标分别选择;4.5 保存分析文件:点击File,选择Save Project,输入文件夹名称:5. 执行初始分析:5.1 点击分析按钮1,分析数据:5.2 在1 Genotypes中查看初步分析结果,或通过2 Display Plots选项查看样品分型图:5.3 在样品分型图界面中,Plot Setting选择 Microsatellite Default;通过软件上方的快捷按钮编辑数据显示方式:6 创建Bin Set:6.1 打开Tools—Panel Manager,选中1 STR Kit,点击2 新建Bin Set,在弹出的New Bin Set 对话框3中输入Bin Set名称:6.2 添加参考数据:6.2.1 确保1 Bin Set已选择新建的Bin Set,选中2 STR Panel,此时会显示出之前新建的每个Marker,点击3 Add Reference Data 添加参考数据;6.2.3 在Add Microsatellite Reference Data对话框中,选择1 参考数据,通过2 Add To List 添加至右面的面板中,点击3 Add,完成添加:6.3 生成Bin Set:完成参考数据添加后,点击1 Auto Bin,之后使用软件默认参数,点击2 OK,自动生成Bin Set:6.4 编辑Bin Set:分别选中每个Marker 1 查看其对应的Bin,选中2 Bin,右键进行Bin的编辑或者删除,也可通过软件3处的快捷图标进行Bin 的编辑;完成后点击OK生成Bin Set;二.分析并检查结果:1.编辑分析方法:点击软件上方快捷图标1 Analysis Method Editor,将Allele中的2 Bin Set修改为之前新建的Bin Set,其他参数保持默认,点击OK;2.点击分析按钮再次分析数据,通过Genotypes查看分型结果,或选中数据通过Display Plots查看样品分型图;选中需要查看的数据,否则无法查看Samples Plot:查看样品分型图:三.打印并导出数据(可选):1.在View中选择不同的界面(Samples,Genotypes等),再通过File—Export/Print导出或者打印相应的数据:2.在Analysis 中选择不同的界面(Display Plots,Report Manager等),再通过File—Export/Print 导出或者打印相应的数据:。
DNAStar使用手册

DNAStar使⽤⼿册DNAStar 中⽂使⽤说明书编者:宋晨⼀、EditSeq (2)三、MapDraw (23)四、MegAlign (32)五、PrimerSelect (42)六、Protean (54)七、SeqMan II开始 (64)⼀、EditSeq打开已有序列我们从⽤苹果计算机打开“TETHIS21MA ”和⽤Windows 打开“tethis21.seq”开始。
假设序列的末尾有载体序列污染。
我们在⽤EditSeq 打开序列的同时,⽤Set Ends 命令去除5’和3’污染序列。
从⽂件菜单(FILE MENU),选择Open。
打开⽂件夹“Demo Sequences”单击选定单击位于对话框右下⾓的序列“TETHIS21”。
Set Ends 按,点钮。
Set Ends 被打开(如右)。
在5’框和3’框中键⼊50和850击OK。
单击Open 打开序列。
当EditSeq 窗⼝打开时,序列长度显⽰在右上⾓。
通过“setting ends,”现在你只有最初序列中的801 bp 的⽚段。
Set Ends 选择在全部Lasergene 应⽤程序中都可以使⽤。
寻找开放读框在这⼊门的⼀部分中,我们将确定序列中最⼤的ORF,并翻译它。
从SEARCH MENU 找到ORF,点击打开会出现右边的对话框。
单击Find Next 寻找第⼀个ORF 的位置。
继续点击Find Next 直到你把ORF 的位置选定在位置183-455。
ORF的坐标会出现在EditSeq 窗⼝的顶端附近。
DNA 序列翻译这⼀节中我们介绍如何翻译我们的ORF,不过任何序列中的读框内部分翻译。
如果你的选择是在三联码的读框内,三联码指⽰棒显⽰为实⼼⿊线(如左图)。
如果都可以⽤下⾯的⽅法进⾏你的选择是不在三联码的读框内,左边的箭头和右⾯的箭头显⽰向左或向右移动⼀个bp,以使所选序列成为三的倍数。
选定ORF,从GOODIES MENU ranslate)。
GENEX Probe 3.5软件及GENEX_Assistant 3.5软件使用及报告输出指导书
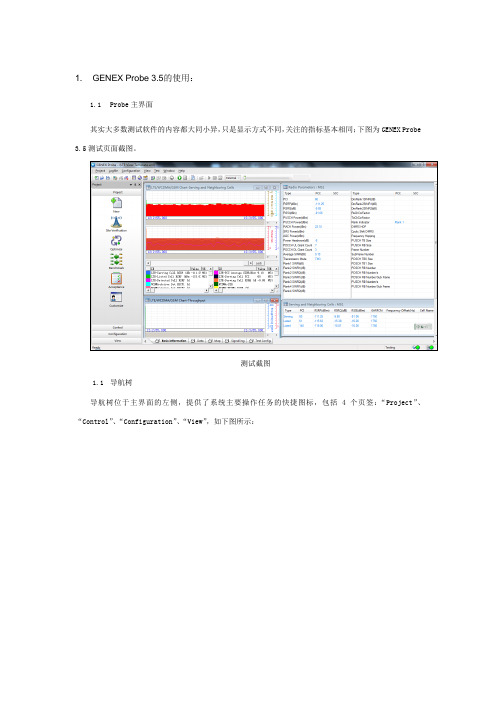
1. GENEX Probe 3.5的使用:1.1Probe主界面其实大多数测试软件的内容都大同小异,只是显示方式不同,关注的指标基本相同;下图为GENEX Probe 3.5测试页面截图。
测试截图1.1导航树导航树位于主界面的左侧,提供了系统主要操作任务的快捷图标,包括4个页签:“Project”、“Control”、“Configuration”、“View”,如下图所示:导航树截图1.1.1“Project”页签“Project”页签主要为工程相关项,主要用于新建及打开历史保存工程等,如下图所示:“Project”页签截图1.1.2“Control”页签“Control”页签为测试控制项内容“Control”页签截图1.1.3“Configuration”页签“Configuration”页签为设备配置窗口,用于连接配置外接测试设备端口等;“Configuration”页签截图1.1.4“View”试图“View”试图截图1.2新建工程新建工程需要根据测试网络的制式选择工程,对应会自动弹出相应的窗口,也可以新建空工程,根据需要自己选择关注窗口;新建工程截图1新建工程截图2新建工程截图3操作界面用于显示各种视图窗口及配置窗口,可以由多个工作表组成。
您可以根据需要,添加多个工作表。
右键单击图中的操作界面状态栏,在弹出的右键菜单中可以添加、删除、重命名工作表,如下图所示。
新建工程截图41.3外接测试设备连接3.1在Probe主界面,单击图标,打开“Device Configure”对话框。
3.2选择一个已添加的外部设备。
3.3单击图标,检查该外部设备的端口是否可用。
设备连接端口截图1设备连接端口截图2设备连接端口截图31.4测试模板制定4.1新建模板模板截图1 4.2历史模板导入模板截图2测试模板可以同时新建或导入多个,启动测试模板可以通过Enable选项中的YES或NO来控制;在“Disconnection Mode”中选择“Disconnected by Time”或“Disconnected after transfer completed”时,可以设置该属性。
GeneTools 操作指南(中文)

描述栏
描述栏显示当前泳道的一些相关信息,您可以在“Tracks”菜单使用使用“Description” 来编辑当前泳道的描述。
剖面图栏
剖面图栏显示当先选中泳道的峰值剖面图。 如果您选择其中一个泳道作为条带匹配标准,其剖面图会一直显示在该栏,以于其他的 泳道进行对比分析。 在“Extras”菜单 “Configuration”里可以对各个项目的颜色进行更改。 您也可以在剖面图栏对各个条带进行编辑,通过鼠标右键。
5. 使用双向箭头选择于分子量标注的第一峰
GeneTools
入门指南
基因有限公司售后服务部编译
编者言 本手册系原版英文说明书编译而来,希望它能为您操作该软件时 带来帮助。由于译者的水平限制,手册中可能存在错误和遗漏,如与 英文说明书发生冲突,请英文原版说明书为准。本手册只作为您操作 的参考依据,不能作为其他评定标准。
基因公司售后部应用工程师:张传峰 2004-3-23
分子量标准库
当前使用的分子量标准
4. 在分子量标准库里选择所需的分子量标准
运行 GeneTools
1 点击开始按钮、选择程序菜单,进入 Syngene 子菜单,选择 GeneTools,双击运行。 根据程序的设置,用户名称对话框可能会出现在界面,如下图:
2 如果您先前已经输入用户名,您就可以从下拉框中选择您的用户名,否则输入您的用 户名。 用户名将被打印在报告中,保存在文件的历史记录中,以便于您追踪是谁创建和修改 了该文件。 如果您不想显示用户名提示对话框,您可以不选择“Show at program”,这样以来, 最后注册使用用户名就会被默认为当前用户,自动加载。 3 从 Extras 菜单中选择 Configuration,弹出 GeneTools configuration (软件属性 设置)对话框:
gptool使用手册

GPTool使用手册一、概述GPTool是一个功能强大的自然语言处理工具,可以帮助您处理和分析大量的文本数据。
它支持多种语言,包括英语、中文、法语和西班牙语等,可以完成的任务包括文本分类、情感分析、命名实体识别等。
二、安装要安装GPTool,您需要先安装Python和pip。
安装完成后,您可以通过以下命令在终端或命令提示符中安装GPTool:1. 打开终端或命令提示符2. 输入以下命令并回车:```pip install gptool```3. 等待安装完成。
安装完成后,您就可以在Python中使用GPTool 了。
三、基本用法1. 导入模块:在Python脚本中,您需要导入GPTool模块才能使用其功能。
您可以使用以下代码导入模块:```pythonimport gptool```2. 创建模型:使用GPTool之前,您需要创建一个模型。
可以使用以下代码创建一个简单的模型:```pythonmodel = gptool.Model()```3. 训练模型:使用训练数据对模型进行训练。
您可以使用以下代码训练模型:```pythonmodel.train(train_data)```其中,`train_data`是一个包含训练数据的列表或数组。
每个数据样本应该是一个字符串或文本序列。
4. 预测:使用训练好的模型进行预测。
您可以使用以下代码进行预测:```pythonpredictions = model.predict(test_data)```其中,`test_data`是一个包含测试数据的列表或数组。
每个数据样本应该是一个字符串或文本序列。
`predictions`是一个包含预测结果的列表或数组。
每个预测结果是一个分类标签或概率分布等。
5. 保存和加载模型:您可以使用以下代码将训练好的模型保存到磁盘上,并在需要时加载模型:```pythonmodel.save("model.h5") # 保存模型到文件loaded_model = gptool.load("model.h5") # 从文件加载模型```6. 其他功能:GPTool还提供了许多其他功能,如文本清洗、特征提取、语言特性和分析等。
GenomeStudio分析软件流程
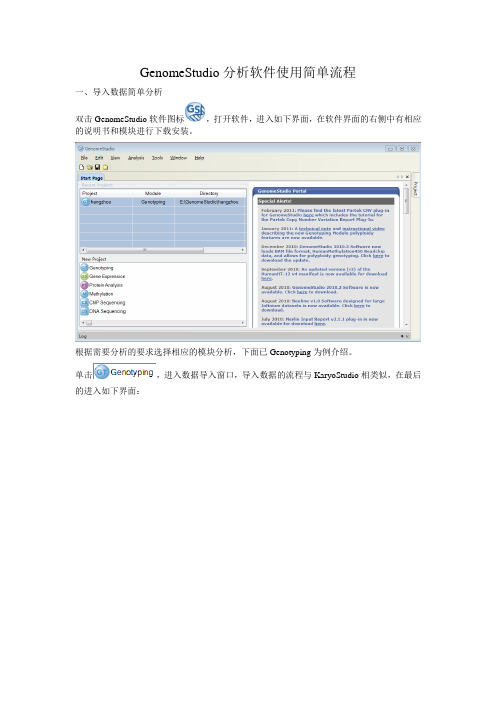
GenomeStudio分析软件使用简单流程一、导入数据简单分析双击GenomeStudio软件图标,打开软件,进入如下界面,在软件界面的右侧中有相应的说明书和模块进行下载安装。
根据需要分析的要求选择相应的模块分析,下面已Genotyping为例介绍。
单击,进入数据导入窗口,导入数据的流程与KaryoStudio相类似,在最后的进入如下界面:选择相应的栏目,如下图所示:点击“Finish”,完成数据导入流程,软件自动运算,运算完成之后出现如下界面:选择:tool菜单下的Show Genome Viewer…等待运算后,软件自动跳出相应的选择界面:选择需要分析的标本和需要看到相关的参数,点击ADD 将所选择的栏目添加到右侧的空白栏中,界面如下:点击OK ,进入如下界面:选择updata ,软件自动运算显示染色体相应信息 可以逐个样本进行分析:选择相应的样本后,点击updata ,右侧的对话框中会显示全部染色体的相关信息,右侧的对话框的可以显示全部染色体的信息,点击可以选择需要显示染色体的信息,也可以对显示信息进行编辑。
分析数据时可以逐个染色体分析,发现有异常区域时,可以双击异常区域在染色体上相应的位置,进行进一步的放大观察分析,具体步骤如下面图解:图中放大的区域部分为缺失,在B allele Freq.图中缺少了中间杂合的信息。
下图显示的是重复的情况:二、输出报告点击下面界面上的analysis菜单下的reports 下面的report wizard选择final report 点击NEXT,进入一级菜单,根据实验需要选择,在如下对话框中,可以根据需要输出的数据选择hide 或者show编辑完成之后,点击next ,最后选择输出的路径和文件名,完成报告输出。
生物化学与分子生物学实验教程教学设计

生物化学与分子生物学实验教程教学设计一、前言生物化学与分子生物学实验是生物学专业重要的实验之一,也是培养学生实验操作技能和科研能力的重要途径。
本文档旨在为生物化学与分子生物学实验课程的教师提供一份教学设计,以便帮助教师们更好地开展实验教学工作,提高实验课教学质量。
二、实验设计思路本实验旨在帮助学生了解生物化学反应及分子生物学技术的基本原理,培养学生实验操作技能,提高学生实验设计、数据处理与分析及科学文献阅读等能力。
实验首先介绍PCR、蛋白质电泳、Western Blotting等基本技术原理,然后让学生利用这些技术进行数据收集、数据分析以及文献阅读分析等操作,以便加深学生对生物化学实验的实质理解。
三、实验流程1.实验一:DNA提取 * 软件操作:GeneTools软件操作,学生处理自己的数据,可自由选择实验材料; * 实验目的:了解DNA提取技术原理,学习基本的PCR技术,实践DNA扩增操作; * 实验操作:基于DNA提取,利用PCR扩增目标序列,通过基因克隆技术或元素分析进行确认。
2.实验二:蛋白质分析 * 软件操作:根据实验目的不同,选择不同的实验材料,如酵母细胞、细胞生物学等; * 实验目的:了解蛋白质分析技术原理,学习基本的电泳方案的设计、样品处理、文献阅读等技能; * 实验操作:离心样品,利用SDS-PAGE技术进行分离,然后利用Western blotting技术进行成像,获得目标蛋白质信息。
3.实验三:酶动力学实验 * 软件操作:根据实验目的不同,选择不同的实验材料; * 实验目的:学习常用酶中的蛋白质特性,了解酶的基本性质; * 实验操作:利用比色法测定酶的活性、酶的浓度、反应温度及pH等因素对酶活力的影响。
四、实验设备和材料•常规实验室设备及仪器:pH计、分光光度计、电泳仪、洗净浓液泵、高速离心计;•主要实验材料:酵母cell、酵母基因、细胞总RNA、大肠杆菌总蛋白、SDS-PAGE gel、antibody、DNA纯化试剂盒等。
genedoc使用方法

genedoc使用方法
Genedoc是一个用于序列分析和编辑的软件。
以下是使用Genedoc进行
序列比对和编辑的基本步骤:
1. 打开两个需要进行比对的序列文件。
2. 选择比对算法,Genedoc支持多种常见的比对算法,如Smith-Waterman算法和Needleman-Wunsch算法。
3. 查看比对结果,结果将以可视化方式呈现,方便用户分析和理解。
4. 如果需要对序列进行编辑,可以在Genedoc中打开序列文件,进行添加、删除、替换碱基等操作。
5. 编辑完成后,可以保存修改后的序列文件。
以上步骤仅供参考,建议查阅Genedoc软件使用说明或咨询专业人士,获取更准确的信息。
基因数据的使用流程

基因数据的使用流程1. 导入基因数据•确保已经获取到所需的基因数据,数据格式通常为FASTA、FASTQ 等。
•使用适当的生物信息学工具,如BioPython、SeqIO等,将基因数据导入到程序中进行处理和分析。
2. 数据预处理•查看基因数据的质量和完整性,检查是否存在噪音、缺失值和重复序列等问题。
•使用合适的方法进行数据清洗、去除不必要的信息和修复错误。
•对数据进行标准化或转换,使其符合进一步分析所需的格式和要求。
3. 基因序列分析3.1 序列比对•使用适当的比对工具(如BLAST、Bowtie、BWA等)将基因序列与已知的参考序列或其他数据库中的序列进行比对。
•根据比对结果,评估序列的相似性、匹配程度和变异情况。
3.2 序列注释•使用生物信息学工具(如Geneious、NCBI等)对基因序列进行注释,从而获得关键的生物学信息。
•注释内容包括基因功能、蛋白质结构、进化信息等。
可以通过比对已知的注释数据库(如GeneBank、UniProt等)进行注释。
3.3 基因表达分析•使用转录组分析工具(如DESeq2、edgeR等)对基因表达进行定量分析。
•通过对不同样本(如对照组和实验组)的基因表达量进行比较,发现差异表达基因并进行统计学分析。
•利用PCA(Principal Component Analysis)和聚类分析等方法对表达模式进行可视化分析。
3.4 功能富集分析•对不同ially富集分度的差异表达基因进行功能富集分析。
•使用生物信息学工具(如DAVID、GOseq等)对差异表达基因进行GO(Gene Ontology)富集和KEGG(Kyoto Encyclopedia of Genes andGenomes)通路分析。
•从而揭示差异表达基因的潜在生物学功能和通路。
4. 结果解释和可视化•结合上述分析结果,解释基因数据中的生物学意义和发现。
•使用适当的可视化工具(如R、Python的Matplotlib、Seaborn等)将分析结果可视化,以便更直观地展示和解释数据。
gene_tool操作指南

一 进入 GeneTools 在桌面上双击软件图标 这时出现选择用户 的对话框 如图
选择用户或输入新的用户名 点击 OK 即可进入 GeneTools 软件环 境 二 确定所分析图片的属性
打开你所分析的图片 这时出现确定该图片属性的对话框 如图
从 Document 中选择图片的种类 Colony Gel PCR Spot blot 从 Electrophoresis direction 中选择电泳的方向 从 Image type 中 选 择 图 片 的 类 型 Fluorescence ( 荧 光 ) Absorption(吸收光) 如果想手动调节选带 则可在 Number of 中输入泳道的数目 若想让软件自动寻道 可设为默认值 0 设置完后 点击 OK 即可进入分析环境
若使用该 Marker 时 返回 From standard 选中 Marker1 再点击 Assign from standard 即可
五 浓度的定量分析 1 选择基线去背景 点击工具栏上的综合参数图标
出现综合参数设定对话框
如图
在 Baseline correction 的 Method 中选择适合的类型 推荐 lowest lope , 在 Offset 中选择 YES 在 Peak detection 中设置最小条带的宽度 高 度及误差范围等 然后点击 OK 注意 在进行浓度的分析时一定要去背景 否则所计算的浓度值不 正确 2 量和浓度的定量计算
用鼠标点击作为参考的条带 点击量的校准与计算图标
会出现 Assign calibrated 对话框 如图
Raw volume 中的值是计算机本身所计算的一个光密度值 在 Calibrated quantitved 中输入一个参考值或一个绝对值 然后点击 OK 即可得出其它条带的相对于该条带的一个值 如果输入的是一 个相对浓度值 则计算出来的就是相对浓度值 在右下框图的表格 中查看所需要的数值 比如输入一个量为 100 其它的结果为如图
GeneScan_文操作手册

GeneScan㆗文操作手冊1. 打開GeneScan software, 在File㆘選擇New2. 會看到以㆘的視窗, 選擇project的選項3. 此時已經打開㆒個新的project, 將要分析的data加到project.在project㆘選擇Add sample Files4. 點選你要分析的data , 按Add or Add all把待分析的data加到㆘面的視窗.完成後按㆘Finish5. 待分析的data就會被加到Analysis control window如㆘圖6. 重設Parameter, 先找出primer peak. (A) 單獨點選sample File(左鍵點㆓㆘) , 點㆘ Raw data 的 tool bar將游標移到primer peak之後,並記㆘左㆘方X軸的數值※只需觀察其㆗幾個sample即可(在同㆒個Run,通常primer peak 出現的位置會很相近)6. (B) 回到analysis control window, 在Parameter column 點選右邊的小箭頭, 會出現以㆘畫面, 點選Define New會看到Parameter setting window , 選擇This Range, 把analysis range改在primer peak之後. 另存新檔名※在Peak Detection選項㆗可依照sample螢光強弱調整設定7. 回到analysis control window選擇新設定的parameter.8. 選擇正確的size standard※使用ROX 500 or LIZ 500 , 選擇GS500 -2509. 點選左㆖方區域, 全選所有的sample, 按㆘Analyze開始分析10. 當分析失敗時有可能是(a)訊號太弱 (b)size standard分析不通過 . (可以從raw data 判別出來).11.當size standard分析不通過,必須重設size standard, 點選sizestandard column ㆗點選右邊的小箭頭, 會出現以㆘畫面, 點選Define New用游標點選每㆒支peak,並打入正確的 size於㆘面的空格㆗.設定完後另存新檔, 再回到analysis control window重新分析.12. 單獨點選sample File (左鍵點㆓㆘),進入sample result window在左㆘方有tool bar可切換window如㆘13. 切換到Result control window(1)可選擇要顯示的顏色(2)Panel, 可將要顯示的data加在同㆒個panel(3)顯示電泳圖(4) 選擇㆒次顯示的panel數 (1 – 8)(5) 顯示圖表(6) 原始data的sample name(7) 顏色可更改成任意想表示的顏色(8) 原本螢光標定的顏色及強度(9) 自動換行的功能(10) 顯示(11) 指定panel㆗將先前data清除(12) 列印(13) 將先前所選擇的data全數清除14. 在main window㆗,Setting / preference / Result display將Align By Size選起來(將peak依片段大小排列)15. 在Result control window 把所有的size standard放在㆒起比對,並確定其㆗size都正確(此時將Quick Tile切換到On,再㆒㆒點選所有的size standard)16. 如何將兩個帶相同顏色的片段放在㆒起比對?先將兩個data加在同㆒個panel㆗點選兩個方塊㆗右邊的那㆒個,選擇不同的顏色。
GENios Plus 操作手册(中译版)

操作手册文件号:1 112 9042002年6月文件版本:1.1软件版本:4.4xTecan服务网点AustriaTecan Austria GmbH Untersbergstrasse 1aA-5082 Grödig / Salzburg AustriaTel.: +43 62 46 89 33 Fax: +43 62 46 72 770 AsiaTecan Asia (Pte) Ltd. 80, Marine Parade #13-04 Singapore 449269 SingaporeTel.: +65 44 41 886 Fax: +65 44 41 836SwitzerlandTecan Schweiz AG Seestrasse 103CH-8708 Männedorf SwitzerlandTel.: + 41 1 922 81 11 Fax. : +41 1 922 81 12 BelgiumTecan Benelux B.V.B.A. Vaartdijk 55B-2800 Mechelen BelgiumTel.: +32 15 42 13 19 Fax. +32 15 42 16 12FranceTecan France S.A.Parc d'Activités de Pissaloup Batiment Hermes IIRue Edouard BranlyF-78190 TrappesFranceTel.: +33 1 30 68 81 50 Fax: +33 1 30 68 98 13 ItalyTecan Italia S.r.l.Via F.lli CerviPalazzo BerniniCentro Direzionale Milano2 I-20090 Segrate (Mi)ItalyTel.: +39 02 215 21 28 Fax: +39 02 215 97 441JapanTecan Japan Co. LtdMeiji Seimei Fuchu Building 10F 1-40 MiyamachiFuchu City, TokyoJapanTel.: +81 42 334 88 55Fax: +81 42 334 04 01 USATecan USP.O. Box 13953 Research Triangle Park, NC 27709USATel.: +1 919 361 5200 Fax: +1 919 361 5201SpainTecan SpainSabino de Arana, 32 E-08028 Barcelona SpainTel.: +34 93 490 01 74 Fax: +34 93 411 24 07 GermanyTecan Deutschland GmbH Theodor-Strom-Straße 17 D-74564 Crailsheim GermanyTel.: +49 79 51 94 170 Fax: +49 79 51 50 38USATecan Boston200 Boston Avenue Suite 3000 Medford, MA 02155 USATel.: +1 781 306 08 27 Fax: +1 781 306 0837 NetherlandsTecan Benelux B.V.B.A. Industrieweg 30,NL-4283 Giessen, Netherlands;Tel.: +31 018 34 48 17 4 Fax: +31 018 34 48 06 7United Kingdom Tecan UKTheale Court11-13 High Street ThealeUK-Reading RG7 5AH United Kingdom Tel.: +44 11 89 300 300 Fax: +44 11 89 305 671注意本手册在制作过程中已尽力避免文字错误,但Tecan 公司不保证没有相关错误的出现,并不予承担由此产生的后果。
说明书翻译

第一部分说明及初始安装说明:该手册为SYNGENE 生物图像系统的安装和使用作了充分介绍:●GeneGenius●MultiGenius●ChemiGenius●GeneGnome●GeneWizard该手册含有关GeneSnap 软件使用的全部内容。
该手册分为三部分:说明与安装(本章)、操作及参考内容。
本章前三部分分别对每个系统进行简要的介绍以及系统模块间关联描述。
最后一个部分是关于该软件总的介绍,这在各系统中是一致的。
该手册依次对每个系统的操作进行描述:GeneGenius、MultiGenius、ChemiGenius系统的说明在同一章中,因为这三个系统的说明是相同的。
而各用一章独立介绍GeneGnome和GeneWizard 的操作。
每一章都将教您如何运用基本的操作来采集图像并使用基因分析工具。
在本手册参考内容部分的第一章(即:第五章、GeneSnap的使用),介绍了一些采集图像的其他方法以及如何控制波形图的显示。
接着介绍了如何采集扫描的图像,包括使用各种工具增强图像效果以及添加注解等。
第六章、GeneSnap操作界面,介绍了该程序界面的编排、如何使用图像采集工具箱、图像窗口、直方图窗口及浏览窗口。
第七章、工具条,介绍了工具条常用的一些功能,以及每个工具的使用方法。
第八章、菜单,依次列出每个菜单及涉及到的相关操作命令。
最后附有一个附件,介绍如何安装桢接受器板以及计算机内所有必须的软件。
GeneGenius概述:GeneGenius是个全自动的综合性的凝胶文档分析系统,它在图像采集及处理方面具有显著的优势,是实验室中处理各种凝胶载体,包括DNA凝胶、蛋白凝胶和放射自显影等的理想工具。
完整的GeneGenius系统包括以下部分:配有8比特计算机控制数码相机的SYNGENE暗室;●带过滤器的自动对焦镜头;●照射面积为20cm×20cm的紫外透射光源;●白光板●Epi 白光照明●Epi 紫外光照明●紫外/红外过滤器●自动紫外过滤环(其他过滤器也可以使用)●紫外防护屏●带有桢接受器、显示器,键盘和鼠标的计算机●打印机●GeneSnap采集软件●Genetools 数学分析软件●微软Office软件—小型企业版(可选)如果您已向SYNGENE购买了完整版GeneGenius系统,那么桢接受器和所有必须的软件都已经装进计算机,所以您要做的就是把各系统模块整合起来。
SynGene GeneGenius系统操作方式及方法
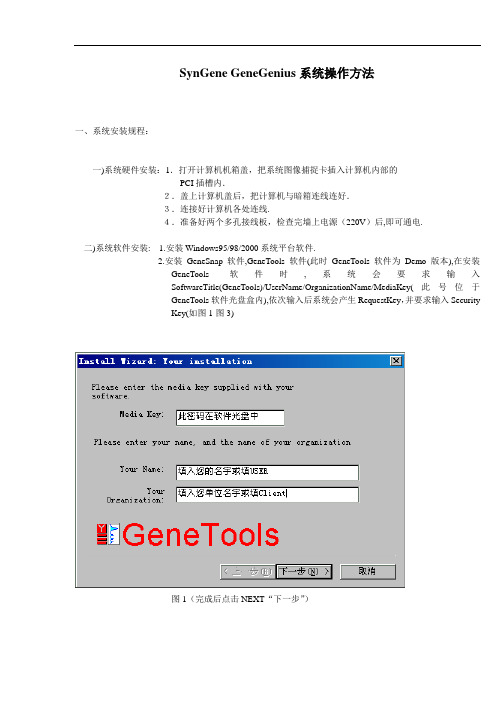
SynGene GeneGenius系统操作方法一、系统安装规程:一)系统硬件安装:1.打开计算机机箱盖,把系统图像捕捉卡插入计算机内部的PCI插槽内.2.盖上计算机盖后,把计算机与暗箱连线连好.3.连接好计算机各处连线.4.准备好两个多孔接线板,检查完墙上电源(220V)后,即可通电.二)系统软件安装: 1.安装Windows95/98/2000系统平台软件.2.安装GeneSnap软件,GeneTools软件(此时GeneTools软件为Demo版本),在安装GeneTools软件时,系统会要求输入SoftwareTitle(GeneTools)/UserName/OrganizationName/MediaKey(此号位于GeneTools软件光盘盒内),依次输入后系统会产生RequestKey,并要求输入SecurityKey(如图1-图3)图1(完成后点击NEXT“下一步”)图2(完成后点击NEXT“下一步”)图3(在从厂家获取密码前可先点击CANCEN“取消”,以完成别的任务)3.安装打印机驱动软件.4.重新启动计算机,进入BIOS/CMOS系统设置(对于DELL计算机应按F2进入)5.把Periphal mode(外部插件模式)下的Parallel Mode 设置为ECP或Bi-direct方式;I/OAdress设置为378H;DMA Mode设置为36.如打印机为三菱P91DW,则还应把后台打印方式改为EMF;及打印机面板上的Gamma值设为3以上7.通过E-mail(内容为SoftwareTitle/UserName/OrganizationName/MediaKey等各项数据)向厂家(SynGene)申请Security Code,8.获取Security Code后,打开计算机”开始/程序/SynGene/Install Wizard”并输入Security Code.此时GeneTools软件可正式使用,系统安装也正式完成.如图4、图3图4(点击打开Install Wizard)9.(设置GeneSnap软件)打开GeneSnap软件进入菜单Extras/Initial后选择Don’t open adocument on startup后点击OK然后退出GeneSnap软件并保存Configuration设置.10.再一次进入GeneSnap软件选择上一次保存的Configuration文件名后,退出二、系统开机及关机方法:一)开机方法:依次打开暗箱电源开关,打印机电源开关,显示器电源开关及计算机电源开关,数秒后即可二)关机方法:1.选择Windows的”开始/关闭系统/选择关机后点击OK”即可关闭计算机2. 依次关闭暗箱电源开关,打印机电源开关,显示器电源开关即可三、系统操作方法:一)图像捕获:1.开机进入Windows后,选择打开GeneSnap软件,软件界面如下图(图一)图5,GeneSnap软件界面(如软件安装中第9步未作设定,则图一中图像窗口将不可见。
GeneTools Product FAQ

GeneTools Product FAQFAQ 1:Can I export the results tables to Excel from GeneTools?Yes, you can. The package directly links to Excel, automatically launching the program and entering the results on a worksheet. It is also possible to export GeneTools data as csv (comma separated variable) files which can be utilised with many software packages.FAQ 2:Can I get a background subtraction that copes with a non-uniform background on the gel?If you select Track Borders it takes a single pixel line down the side of the track and subtracts this pixel for pixel. You do, however, have to be careful that you do not have the edges of the track touching any bands as this can affect the result. Another possibility, avoiding problems caused by the track touching the bands, is to perform a combination of the pixel for pixel subtraction on the borders and the Lowest Slope. This is called Track Borders & Slope subtraction.A new feature of GeneTools is the Rolling Disk background subtraction. The user selects a size of d isc (defaults to 30 pixels) which rolls along the track histogram and very closely follows the natural background of the gel.FAQ 3:Can I analyse a gel that is distorted and the tracks have run unevenly down the gel?It is possible to analyse distorted gels. However, to get the most accurate results, you need more than one molecular weight standard. The software will automatically interpolate between the standards as you enter them, trying to follow any distortions within the gel. The more standard lanes you incorporate into the gel, the more accurate the analysis.FAQ 4:Can I analyse smiling tracks and bands?The software will automatically detect and compensate for distorted bands. This is achieved by drawing the band marker line to the same angle as the band, thereby correcting the grimace. There is no need for any time-consuming manualadjustments. The software even detects that grimacing of bands often increases in severity with increasing distance from the well position.FAQ 5:Can I use more than one standard on the same gel?Yes. The software will interpolate between them ensuring accurate results even on very distorted or smiley gels.FAQ 6:Can I count both blue and white colonies, and is this possible on the same plate?Yes, you can. Just set the relevant parameters in the sample properties dialog box.FAQ 7:When counting colonies there are often groups which are close together. Can the software separate these?The software runs a separation technique when analysing a colony image. It counts the colonies initially found, then runs a separation algorithm before recounting them for the final result.FAQ 8:How can I compare tracks from different gels, and are they background corrected?There is a facility for overlaying track profiles from different gels for comparison. There is no limit to the number of tracks and gels you can compare at any one time. You can also zoom in and print specific areas of interest within the comparison.FAQ 9:Is it possible to import images captured using other methods such as a scanner?You can import TIFF and BMP images into GeneTools for analysis. However, to maintain Good Laboratory Practice, the results table will include the line 'GLP Warning: This image was created from a non GLP source?FAQ 10:My gel has run partially diagonally which means that the marker on the left side of the gel is vertical but the one on the right has started to smile. How can I analyse this?The analysis software will allow more than one standard to be set on a gel. By selecting the propagation technique, you can accurately analyse even very unevenly run gels. In this case, it is better to use two standards and then select 揢se Nearest Standard?option. This will allow for distortions in the local area of the standard. An even more accurate way would be to use three standards and interpolate between them.FAQ 11:How do I get the best results from my GeneTools software with the minimum of manual input?Fundamentally, by obtaining the best image prior to analysis. It is most important to frame your sample properly, adjusting the zoom so that it fills as much of the screen as possible. Also the use of integration, either by Auto-exposure or manually, will brighten the image, making analysis easier. When using Auto-exposure do not worry about saturation of bands, the software will prevent this. The bigger and better the image you grab directly from the camera, the easier it is for GeneTools to perform an analysis quickly and automatically. If you obtain a good image, analysis can be carried out in as little as 10 seconds, with the touch of a single button.。
GeneSnap简要操作说明
GeneSnap简要操作说明
1、打开暗箱电源开关,电源在暗箱的后面。
2、启动电脑,GeneSnap自动运行,如果您已经取消了自动运行,双击桌面快捷方式即可。
3、GeneSnap第一次启动,会弹出对话框,要求您输入登陆信息。
输入您的登陆名,如果您不希望每次都显示该对话框,勾上下面的单选框。
4、单击按钮,显示图像捕捉窗口。
5、在捕捉按钮为绿色(曝光时间小于5s)的状态下,您可以调整曝光时间、选择光源、选
择虑光片。
单击,进入图像实时预览状态。
6、选择光源为白光,在白光下调整凝胶的位置,使图像显示在图像框的中间,调整缩放
使图像充满窗口的70~90%,调整聚焦,使图像达到最清晰,关闭暗箱门。
7、选择光源为UV,虑光片选择为EtBr/UV,调整光圈,使图像明暗合适,对比明显,
单击捕捉按钮,冻结图像。
8、也可以使用自动曝光按钮,进行自动曝光。
9、使用联拍按钮,可以自动联拍一系列的图片。
使用上下箭头选择联拍图片数,选择第一个单选框,进行图像叠加,否则不进行叠加。
选择第二个单选框,使用相同的时间联拍,选择曝光时间。
10、图片捕捉完成后,单击按钮,保存图片为.sgd 格式。
也可以使用
File→Export image 保存为其它格式。
11、使用按钮,发送到GeneTools进行分析。
本手册为GeneSnap的操作步骤简介,具体操作请参考原版英文说明书,并以英文说明书为准!
基因公司售后服务部。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
描述栏显示当前泳道的一些相关信息,您可以在“Tracks”菜单使用使用“Description” 来编辑当前泳道的描述。
剖面图栏
剖面图栏显示当先选中泳道的峰值剖面图。 如果您选择其中一个泳道作为条带匹配标准,其剖面图会一直显示在该栏,以于其他的 泳道进行对比分析。 在“Extras”菜单 “Configuration”里可以对各个项目的颜色进行更改。 您也可以在剖面图栏对各个条带进行编辑,通过鼠标右键。
运行 GeneTools
1 点击开始按钮、选择程序菜单,进入 Syngene 子菜单,选择 GeneTools,双击运行。 根据程序的设置,用户名称对话框可能会出现在界面,如下图:
2 如果您先前已经输入用户名,您就可以从下拉框中选择您的用户名,否则输入您的用 户名。 用户名将被打印在报告中,保存在文件的历史记录中,以便于您追踪是谁创建和修改 了该文件。 如果您不想显示用户名提示对话框,您可以不选择“Show at program”,这样以来, 最后注册使用用户名就会被默认为当前用户,自动加载。 3 从 Extras 菜单中选择 Configuration,弹出 GeneTools configuration (软件属性 设置)对话框:
4 确保选择 Auto match peaks,Auto locate tracks 和 Auto locate peaks 5 不要选择 Auto import methods。
打开一个安全格式文件
注意:如果你是从 GeneSnap 中直接打开 GeneTools,样本属性对话框会立即弹出,你可以直
果在常用工具栏里出现,均可以在常用工具栏里直接双击运行。常用工具栏的命令基 本上可以满足您大部分分析需要。
标准的打开文件对话框
注意:如果您想分析一个并非有 GeneSnap 捕捉的图像,对于其他格式的图片(tif、bmp), 可以通过“File”菜单选择“NEW(import)”来打开。GeneTools 将创建一个安全格式文件, 弹出标准的 Windows 打开对话框,您可以选择您要打开的图片类型,更多信息请参阅实时 帮助。 2 使用该对话框选择所需要的安全格式文件 3 点击“Open” 注意:如果您打开一个已经分析过的图像,图象会立即弹出在图像窗口。 如果您已经设置“Auto import methods”,并且选择了一个图像输入,“import method” 对话框会立即弹出。更多信息,请参阅实时帮助。
文件格式
GeneTools 以一种安全格式保存文件数据,SynGene Gel document 或者 .sgd 格式文件。 这些文件格式和图像捕捉软件 GeneSnap 完全兼容。
这种文件格式确保您的数据不会被随意破坏,同时您还可以对数据进行追踪和复制。 您也可以打开其他非安全格式文件( tiff、bmp 等)进行分析。当您这样操作时, GeneTools 会自动拷贝一份原图像并以安全格式保存。您对原图像所做的任何改变都不会影 响已保存的安全格式文件。
简 介
本手册是 GeneTools 快速入门指南,向您简单的介绍如何使用 GeneTools 来完成您的实
验结果分析。虽然只是对 GeneTools 进行简单的描述,但是您可以发现,本手册所描述的功 能和步骤基本上能覆盖您实验分析中所有的需要,如果您想详细了解 GeneTools 的各个功 能,请您参阅随仪器附送的 GeneTools_Reference_Manual。
接跳到第四步进行操作。
打开一个已存在的安全格式文家 1 从常用工具栏中选择“Open”按钮:
或者 从“File”菜单选择“Open”来打开文件 注意:从常用工具栏选择点击相应的按钮,是比价简洁明快的方式。本手册介绍的工具,如
GeneTools 提供了很多配置参数选项,您可以通过修改这些参数,使软件来按照您的需 要来进行工作。但是,您并不需要经常修改这些参数,因为默认的参数设置在大多数情况下 都是适合的,您修改的参数可以保存为默认值,在下一次打开图像时会生效。
GeneTools 也提供了一系列的工具,可以然您更好的手动控制处理过程和编辑分析结 果。但是您并不需要经常使用这些工具,因为 GeneTools 提供的自动分析工具是如此强大和 灵敏,它们会很好的处理绝大多数的图片,极少数特殊图像可能需要您进行手动编辑、分析。
l 设置综合参数
secure sample file 保存
一旦创建了 secure sample file, 如果您想再次调用该文件,您必须先保存该图像 为 .sgd 格式。分析后的图像,分析数据将随图像一起保存。
另存为其他文件名: 1. “File”菜单,选择“Save as”弹出另存对话框。 2. 选择或创建新的保存文件夹 3. 输入文件名 4. 保存文件
样本属性对话框会立即弹出
4 选择 Analysis Type 为 Gel(Genetools 默认的分析类型是 Gel,您也可以通过 Extra - configuration-Analysis type 改变默认的设置)。 5 选择电泳的方向,默认方向是 Down。 您不必要修改其它的设置,尤其是: l GeneTools 自动检测图像类型 l 您应该设置“Number of tracks”为 0,这样 GeneTools 会自动进行泳道定 义。 6 点击“OK”键 短暂的停顿以后,GeneTools 会自动进行泳道定位和条带定位。 结果会显示在凝胶窗口。 关于该命令和相关主题的介绍请参阅在线帮助的相关章节 Reference → Gel analysis → How to → Create and work with secure sample files : l 打开一个 secure sample file l 创建一个新的 secure sample file l 使用查询功能 l 获取图像创建 secure sample file
5. 使用双向箭头选择于分子量标注的第一峰
峰值栏
该栏显示剖面图栏条带的编号和其他信息。显示信息与您在图表栏的选择项目有关。 l 如果选择分子量标准曲线,就是显示分子量 l 如果选择质量校正标准曲线,就显示条带的质量 l 如果选择系统树图,就显示条带的位置
图表栏 图标栏显示:
l 所选泳道的分子量校正曲线 l 所选泳道的质量校正曲线 l 匹配泳道之间的系统关系树图
图表栏 结果栏
亮度重置 亮度调节 对比重置 对比调节
中间色调重置
中间色调调节
下面简单介绍图像窗口的各个部位,详细信息您可以参阅在线帮助。
图像区
图像区显示图片以及条带和峰所在的位置,以蓝色方框显示泳道,黄色表示峰值,为更 好的吻合数据,条带的边缘以曲线表示。 如果您在不同的泳道之间使用条带匹配,在匹配的条带之间会以红线连接表示。 您可以在“View”菜单里设置需要显示的项目,如泳道标识、峰值标识、条带边缘、匹 配线、背景线等。在“Extras”菜单 “Configuration”里可以对各个项目的颜色进行 更改。 鼠标点击所需的条带即可选中,同样方法选中泳道。右键单击条带,弹出条带快捷菜单, 可以对条带进行相应的处理。
泳道标识栏
该栏显示泳道的标识以及该泳道的目前所处的状态: l 非激活状态(泳道标识以灰色显示) l 分子量条带标准 ( MWS ) l 条带匹配标准 (MS) l 当前条带匹配标准 (MS*) 您可以在泳道标识栏单击选中所需泳道,也可以在图像区使用右键单击所需的泳道,弹 出泳道对话框,对泳道进行相应的编辑。
GeneTools
入门指南
基因有限公司售后服务部编译
编者言 本手册系原版英文说明书编译而来,希望它能为您操作该软件时 带来帮助。由于译者的水平限制,手册中可能存在错误和遗漏,如与 英文说明书发生冲突,请英文原版说明书为准。本手册只作为您操作 的参考依据,不能作为其他评定标准。
基因公司售后部应用工程师:张传峰 2004-3-23
分子量分配。
GeneTools 软件自己内置了一个分子量标准库,您可以对分子量标准库进行编辑。如果
其中没有符合您要的分子量标准,您也可以根据自己的需求,创建适合自己的分子量标
准。
确定分子量标准条带,分配分子量:
1. 选择分子量标准泳道(Marker 条带)
2. 选择对应分子量标准的第一条带
凝胶分析
本章主要介绍如何使用 GeneTools 进行凝胶分析,欲了解详细的功能,您可以参阅实时 帮助。 本章开始介绍了如可运行 GeneTools,如何进行参数设置制,以方便您以后的章节使中。
打开 GeneTools
本章节假设 GeneTools 已经安装到您的计算机上,并且设置为开机自动运行。如果您刚 刚安装好 GeneTools,就刚好是这种情况,所有的参数均处于默认状态。当然如果您的软件 是以前安装的,其中有些参数已经改变,在继续本章节前您可以把他们全部恢复到默认状态。
使用相同的文件名保存先前的图像可以直接使用保存快捷图标: “File”菜单选择“Save”命令,如果图像以前没有被保存过,将弹出另存为
对话框,提示您输入文件名:参考上文。
使用图像窗口
您打开的图像将显示在图像窗口: 图像区 泳道标识栏 描述栏 剖面图栏 峰值栏
分子量标准库
当前使用的分子量标准
4. 在分子量标准库里选择所需的分子量标准
3. “Track”菜单选择“MW standard”定义当前选中泳道为分子量标准泳道,在弹出
的对话框里,选择相应命令,分配分子量:
使用双向箭头选择当前条带
在使用软件的任何时候,如果您对某一特征感兴趣,可以直接查看实时帮助,本软件提 供功能强大的帮助软件,详细介绍了如何操作 GeneTools。
分析技术
GeneTools 支持凝胶分析、克隆计数、手动条带定量和斑点分析。每一种方法都在下列 章节中独立介绍。
