第六章分子生物学研究法
第六章:常用分子生物学技术——RNA干扰技术

识别并切割(qiēgē)与siRNA反义链互补的靶mRNA
(5)dsRNA的再生成
第十ānrǎo)
第十七页,共四十页。
RNA干扰(gānrǎo)
(RNA interference, RNAi)
siRNA(小干扰RNA):21-23nt,由dsRNA裂 解而成的小片段,可诱导mRNA降解。siRNA主要
药学 分子生物 (yào xué)
学
第一页,共四十页。
生物芯片 (biochip) (shēnɡ wù xīn piàn)
以生物、电子、机械和信息技术为基础,在 固相支持物表面建立集成、连续、微型分析系 统(xìtǒng),形成芯片,实现对生物大分子的准确、 快速、高通量、自动化检测。
第二页,共四十页。
第三十一页,共四十页。
REGγ基因敲除鼠(下图)出现脊椎(jǐ zhuī)弯曲等早衰现象
第三十二页,共四十页。
Gab1基因(jīyīn)敲除导致小鼠缺血性血管新生、侧枝循环建立出现 缺陷(图示上半部分),主要是由于血管内皮细胞管状结构形成的 信号调控通路出现障碍而引起的(图示下半部分)。
第三十三页,共四十页。
2、 RNA干扰技术缺点
“脱靶效应”(off-target effects)
第二十四页,共四十页。
RNA干扰技术(jìshù)的应用
1、基因功能研究(功能失活策略) “基因敲减(knockdown)”
2、基因治疗(基因(jīyīn)失活性治疗) (1)病毒感染性疾病
通过RNAi抑制 RNA病毒(bìngdú)复制 (2)基因过表达引起的疾病(如肿瘤)
特定功能基因的部分片段被同源的外源DNA片 段替代,从而使靶基因失活。
分子生物学-课件-分子生物第六章可编辑全文

• 使双链DNA解链度达到50%所需的温度称为解链温度 (Tm)、变性温 度、熔点
• DNA的解链温度一般在82-95℃ ,与DNA的分子大小和碱基组成、 溶液的pH值和离子强度(+)等有关
二、复性
• DNA复性:缓慢降温可以使热变性DNA重新 形成互补双链结构
且RNA的完整性和纯度都很高
3、氯化锂-尿素法
• 利用高浓度尿素变性蛋白质同时抑制RNA 酶,氯化锂选择性沉淀RNA
• 缺点:存在DNA污染,氯化锂沉淀RNA会 丢失一些小分子RNA,如5sRNA等
• 优点:快速、简便、产量高,尤其适用于大 量样品少量组织细胞的RNA提取
4·热酚法
• 将异硫氰酸胍、巯基乙醇和SDS等联合使用,可以快速裂 解细胞,解离核蛋白复合物,释放RNA,并有效抑制 RNase的活性
• 在某些理化因素的作用下,维 系核酸二级结构的氢键和碱基 堆积力受到破坏,DNA双螺旋 结构松散,变成单链的过程称 为核酸的变性
• 核酸双螺旋区的氢键断裂,变 成单链,但并不涉及共价键的 断裂
DNA解链曲线
DNA变性的本质是氢键的断裂
核酸的变性因素
• 变性方法
–热变性、酸碱变性、化学变性剂(乙醇、 尿素和甲酰胺 )
(3)煮沸裂解法
• 以溶菌酶、Triton裂解细菌,然后以沸水浴加热,不 仅促进细菌裂解,还可以使蛋白质和染色体DNA、质 粒DNA变性
• 当降低温度时,闭环质粒DNA复性留在上清液中,而 染色体DNA保持与细胞膜碎片结合、沉淀,可以通过 离心除去
• 在离心上清液中加入有机溶剂 (例如异丙醇)便得到质 粒DNA粗品沉淀
《分子生物学》课程思政教学设计
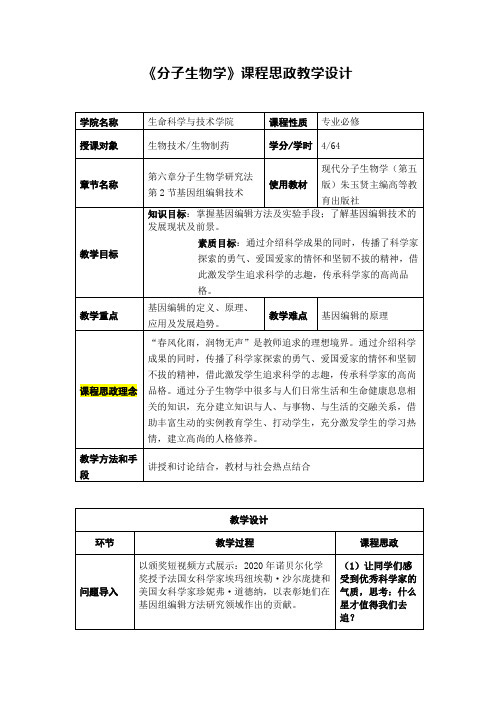
以颁奖短视频方式展示:2020年诺贝尔化学奖授予法国女科学家埃玛纽埃勒·沙尔庞捷和美国女科学家珍妮弗·道德纳,以表彰她们在基因组编辑方法研究领域作出的贡献。
•基因编辑定义:是一种可以在基因组水平上对DNA序列进行改造的遗传操作技术。
•基因编辑原理:通过特异的内切酶复合体,精确识别细胞内DNA片段中靶点的核苷酸序列,在预定的位置切割D NA,切断的DNA被细胞内的DNA修复系统的精确编辑(基因编辑---修正,删除,插入)从而达到定点改造基因
的目的。
•基因编辑三大主要技术:第一代:ZF N(锌指核酸酶,1996年);第二
代:TALEN(转录激活样效应因子核
酸酶,2011年);第三代:CRISPR/C as(成簇的规律性间隔的短回文重复序列,2013年)。
•CRISPR/Cas9基因编辑原理:CRISPR-CAS系统的组成主要包括:由不连续的重复序列R( repeat)与长度相似的间隔序列S( spacers)间隔排列而
成的CRISPR簇,前导序列L( lead er)以及一系列CRISPR相关蛋白基因cas。
•。
分子生物学的研究方法例题和知识点总结

分子生物学的研究方法例题和知识点总结分子生物学是一门从分子水平研究生命现象、生命本质、生命活动及其规律的科学。
它的研究方法多种多样,下面我们将通过一些例题来深入理解这些方法,并对相关知识点进行总结。
一、核酸提取与纯化核酸包括 DNA 和 RNA,是分子生物学研究的重要对象。
提取和纯化高质量的核酸是后续实验的基础。
例题:从植物叶片中提取总 DNA,需要经过哪些步骤?知识点:1、破碎细胞:使用机械研磨、酶解法等破坏细胞壁和细胞膜,释放出核酸。
2、去除杂质:通过加入蛋白酶 K 去除蛋白质,用酚/氯仿抽提去除酚类、多糖等杂质。
3、沉淀核酸:常用乙醇或异丙醇沉淀 DNA,离心后获得核酸沉淀。
4、洗涤和溶解:用 70%乙醇洗涤沉淀去除盐分,干燥后用适当的缓冲液溶解。
二、PCR 技术(聚合酶链式反应)PCR 是一种用于扩增特定 DNA 片段的技术。
例题:设计一对引物用于扩增某基因的特定片段,需要考虑哪些因素?知识点:1、引物长度:通常为 18 25 个核苷酸。
2、碱基组成:G + C 含量在 40% 60%之间,避免形成稳定的二级结构。
3、特异性:引物要与目的基因特异性结合,避免与其他序列有过多的同源性。
4、退火温度:根据引物的碱基组成计算退火温度,以保证扩增的特异性和效率。
PCR 的基本步骤包括:1、变性:高温使双链 DNA 解离为单链。
2、退火:降低温度,引物与单链 DNA 结合。
3、延伸:在 DNA 聚合酶的作用下,从引物 3'端开始合成新的DNA 链。
三、基因克隆基因克隆是将目的基因插入到载体中,导入宿主细胞进行复制和表达。
例题:简述构建重组质粒的过程。
知识点:1、目的基因获取:可以通过 PCR 扩增、从基因文库中筛选等方法获得。
2、载体选择:常见的载体有质粒、噬菌体等,要根据实验需求选择合适的载体。
3、酶切和连接:用相同的限制性内切酶分别切割目的基因和载体,然后用 DNA 连接酶将它们连接起来。
2013-2014分子生物学章节练习题第5-6章练习题

第五、六章分子生物学研究方法练习题1一、【单项选择题】1.一般的限制性核酸内切酶II作用的特点不包括A.在对称序列处切开DNAB.DNA两链的切点常不在同一位点C.酶切后产生的DNA片段多半具有粘性末端D. DNA两链的切点常在同一位点,E.酶辨认的碱基一般为4-6个4.下列关于建立cDNA文库的叙述哪项是错误的A.从特定组织或细胞中提取mRNAB.将特定细胞的DNA用限制性核酸内切酶切割后,克隆到噬菌体或质粒中,C.用逆转录酶合成mRNA的对应单股DNAD.用DNA聚合酶,以单股DNA为模板合成双链DNA5.限制性核酸内切酶的通常识别序列是A.粘性末端B.RNA聚合酶附着点C.回文对称序列,D.多聚腺苷酸E.甲基化的“帽”结构9.基因工程的操作程序可简单地概括为A.载体和目的基因的分离、提纯与鉴定B.分、切、连、转、筛,C.将重组体导入宿主细胞,筛选出含目的基因的菌株D.将载体和目的基因接合成重组体E.限制性核酸内切酶的应用11.常用质粒有以下特征A.是线性双链DNAB.插入片段的容量比λ噬菌体DNA大C.含有抗生素抗性基因,D.含有同一限制性核酸内切酶的多个切口E.不随细菌繁殖而进行自我复制14.表达人类蛋白质的最理想的细胞体系是A.大肠杆菌表达体系B.原核表达体系C.酵母表达体系D.昆虫E.哺乳类细胞表达体系,18.在分子生物学上“重组DNA技术”又称为A.酶工程B.蛋白质工程C.细胞工程D.发酵工程E.分子克隆技术22.基因组代表一个细胞或生物的A.部分遗传信息B.整套遗传信息,C.可转录基因D.非转录基因E.可表达基因25.在已知序列信息的情况下,获取目的基因的最方便方法是A.化学合成法B.基因组文库法C.cDNA文库法D.PCRE.差异显示法27.PCR主要的酶是A.DNA连接酶B.反转录酶C.末端转移酶D.碱性磷酸酶E. Taq DNA聚合酶28.重组DNA技术中实现目的基因与载体DNA拼接的酶A.DNA聚合酶B.RNA聚合酶C.DNA连接酶D.RNA连接酶E.限制性核酸内切酶30.最常用的筛选转化细菌是否含重组质粒的方法是A.营养互补筛选B.抗药性筛选,C.免疫化学筛选D.PCR筛选E.分子杂交筛选33.用于重组DNA的限制性核酸内切酶,识别核苷酸序列的A.正超螺旋结构B.负超螺旋结构C.α-螺旋结构D.回文结构,E.锌指结构58、基因治疗是指A.对有基因缺陷的细胞进行修复,从而使其恢复正常,达到治疗疾病的目的B.把健康的外源基因导入到有基因缺陷的细胞中,达到治疗疾病的目的C.运用人工诱变的方法,使有基因缺陷的细胞发生基因突变恢复正常D.运用基因工程技术,把有缺陷的基因切除,达到治疗疾病的目的93.PCR实验的特异性主要取决于A.DNA聚合酶的种类B.反应体系中模板DNA的量C.引物序列的结构和长度D.四种dNTP的浓度E.循环周期的次数94.基因剔除(knock out)的方法主要被用来研究A.基因的结构B.基因的功能C.基因的表达D.基因的调控E.基因的突变95.反义核酸作用主要是A.封闭DNA B.封闭RNAC.降解DNA D.降解DNA E.封闭核糖体的功能105. 酵母单杂交技术是分析【】的实验系统。
分子生物学考点整理1

分子生物学考点整理符广勇朱兰第一章.绪论一、分子生物学概念分子生物学是从分子水平研究生命本质为目的的一门新兴边缘学科,是研究核酸、蛋白质等所有生物大分子结构与功能相互关系的科学,是人类从分子水平上真正揭开生物世界奥秘、由被动地适应自然界转向主动地改造和重组自然界的基础学科。
二、重组DNA技术又称基因技术,是20世纪70年代初兴起的技术科学,目的是将不同的DNA片段按照人们的设计定向连接起来,在特定的受体细胞中与载体同时复制并得到表达,产生影响受体细胞的新的遗传性状。
三、基因表达的调控基因表达的调控主要表现在信号传导研究、转录因子研究及RNA剪辑三个方面。
四、转录因子转录因子是能与基因5`端上游特定序列专一结合,从而保证目的基因以特定的强度在特定的时间与空间表达的蛋白质分子。
第二章.染色体与DNA一、染色体上的蛋白质染色体上的蛋白质主要包括组蛋白和非组蛋白。
根据凝胶电泳性质可以把组蛋白分为H1、H2A、H2B、H3、H4。
这些组蛋白都含有大量的赖氨酸和精氨酸。
二、组蛋白的特性1.进化上的极端保守性不同种生物组蛋白的氨基酸组成是十分相似的,特别是H3、H4。
2.无组织特异性到目前为止,仅发现鸟类、鱼类及两栖类红细胞不含H1而带有H5,精细胞染色体的组蛋白是鱼精蛋白这两个例外。
3.肽链上氨基酸分布的不对称性碱性氨基酸集中分布在N端的半条链上。
4.组蛋白的修饰作用包括甲基化、乙酰化、磷酸化、泛素化及ADP核糖基化。
5.富含赖氨酸的组蛋白H5三、HMG蛋白叫高迁移率蛋白四、真核细胞DNA序列的分类1.不重复序列2.中度重复序列3.高度重复序列重复序列的意义:若某一重复序列出现错误,对基因的影响不大,稳定性较高;在短时间内可同时产生大量的基因产物。
重复序列的应用:应用于分子标记的作用:卫星DNA(便于分子标记)和微卫星DNA五、真核生物基因组与原核生物基因组的区别1.真核基因组庞大,原核生物基因组小2.真核基因组存在大量的重复序列,原核基因组没有重复序列3.真核基因组大部分是非编码序列,原核基因组大多是编码序列4.真核基因组的转录产物为单顺反子,原核基因组转录产物多为多顺反子5.真核基因是断裂基因,有内含子结构,原核基因为连续基因,几乎没有内含子结构6.真核基因组存在大量的顺式作用原元件,包括启动子、增强子和沉默子等,原核基因组基本没有增强子和沉默子7.真核基因组存在大量的DNA多态性,原核基因组很少有8.真核基因组具有端粒结构,原核基因组没有端粒结构六、重叠基因(Overlapping gene)指两个或两个以上的基因共有一段DNA序列,或是指一段DNA序列成为两个或两个以上的基因的组成部分。
分子生物学 分子生物学研究法

5‘ 供者
探针
3‘ 受者
Taqman法 分子信标(molecular beacon)法
高效液相色谱 MALDI-TOF质谱分析法 DNA芯片技术(DNA chip)
SNP数据库
国立生物技术信息中心 德国的HGBAS网站
JST的数据库
2.5基因打靶(gene targeting)
通过DNA定点同源重组,改变基因组中 的某一特定基因,在生物活体内研究该 基因的功能。(反向遗传学)
抗原-抗体 特异性结合
SDS-PAGE 后转膜
基因表达产 物――蛋白
的检测
ELISA 原理和用途类似于Western blot,但在酶标板中操作,无需SDSPAGE转膜,操作简单,可批量检测,并可半定量测定。
Southern blot
Northern blot
Western blot
双脱氧法测序
gradient gel electrophoresis,DGGE) 原理:当双链DNA在变性梯度凝胶中进行 到与DNA变性温度一致的凝胶位置时, DNA发生部分解链,电泳迁移率下降, DNA链中有一个碱基改变时,会在不同 的时间发生解链,因影响电泳速度变化 的程度而被分离。
荧光共振能量传递 (fluorescent resonance energy transfer, FRET)
基因敲除(gene knockout):定向敲除 基因敲入(gene knockin):定向替代
基因打靶的必备条件
胚胎干细胞(ES细胞)
能在体外培养,保留发育的全能性
打靶载体
Neo(新霉素)阳性筛选标志 HSV-tk阴性筛选标志:单纯疱疹病毒
(herpes simplex virus) 胸腺嘧啶激酶 (thymidine kinase)
分子生物学研究方法

分子生物学研究方法
首先,核酸提取与分析是分子生物学的基础。
通过从生物体细胞中提
取核酸,可以获得DNA和RNA的样本,为后续的实验提供原料。
核酸的分
析方法包括凝胶电泳和聚合酶链式反应(PCR)等。
凝胶电泳可以根据核
酸分子的大小和电荷差异进行分离,从而获得目标核酸的纯度和浓度信息。
PCR是一种体外扩增DNA的方法,可以在短时间内从少量的DNA模板扩增
到大量的DNA产物,为后续实验提供更多的材料。
其次,蛋白质的表达与纯化是分子生物学的另一个重要研究方法。
蛋
白质是生物体内功能的执行者,因此研究蛋白质的表达与功能对于揭示生
命活动的机理具有重要意义。
蛋白质的表达通常利用重组DNA技术,将目
标基因克隆到可表达载体中,并转化到细菌或其他表达系统中。
随后,通
过诱导表达、细胞破碎、蛋白质纯化等步骤,最终获得目标蛋白质的产物。
蛋白质的纯化方法包括亲和层析、离子交换层析、凝胶过滤等多种方法,
可以根据蛋白质的性质和特点选择合适的方法进行纯化。
另外,生物信息学和计算生物学方法也是分子生物学研究的重要手段。
随着DNA测序技术的快速发展,大规模的基因组、转录组和蛋白质组数据
被不断积累,因此需要开发合适的计算方法进行存储、管理和分析这些数据。
生物信息学和计算生物学的研究方法涉及到生物数据库的建立和维护、序列比对、基因和蛋白质结构预测、分子动力学模拟等等。
这些方法可以
帮助研究人员更好地理解和分析分子生物学数据,从而揭示生命活动和疾
病发生发展的机理。
(整理)分子生物学研究方法

一.名词解释IP(免疫沉淀、免疫印迹):是以抗体和抗原之间的专一性作用为基础的用于研究蛋白质相互作用的经典方法。
这个实验是用特异性抗体结合细胞裂解液中的目的蛋白,洗涤后解离特异性抗体和目的蛋白质,经聚丙烯酰胺凝胶电泳(SDS-PAGE),最后用Western blot分析目的蛋白质。
这种方法可以分析溶液中的、细胞内的、结合在细胞膜上的蛋白质或者受体,但只能是定性或者半定量的实验。
NLS(核定位序列):核定位信号是另一种形式的信号肽, 可位于多肽序列的任何部分。
一般含有4~8个氨基酸, 且没有专一性, 作用是帮助亲核蛋白进入细胞核。
可分为单一型NLS和双分型NLS。
单一型NLS,由一段连续的碱性氨基酸序列排列而成(RKKKRKV),双分型NLS,有两段碱性氨基酸被中间10~12GE 非特异性氨基酸所分割而形成,(KRPAA TKKAGQAKKKKLDK)。
SUMO(small ubiquitin-related modifier):是一种小的泛素相关修饰蛋白,它与泛素在序列上虽只有18%相同,但在二级结构和三级结构上有惊人的相似。
SUMO家族成员都有独特的N端氨基酸序列和C端外延序列。
SUMO 化(sumoylation)的功能与泛素化(ubiquitination)不同,它并不是蛋白降解,反而加强它们的稳定性或调解它们在细胞内的定位和分布,甚至与凋亡相关。
二.问答题1.什么是近交系小鼠,它们有什么特征?有哪些常见的近交系小鼠?答:近交系小鼠:是经连续20代(或以上)的全同胞兄妹交配(或亲代与子代交配)培育而成的小鼠,品系内所有个体都可追溯到起源于第20代或以后代数的一对共同祖先。
特性:①基因位点的纯合性(Homozygosity)近交系小鼠中任何一个基因位点上纯合子的概率高达99%,因而也能繁殖出完全一致的纯合子后代。
②遗传组成的同源性(Isogenicity)品系内所有动物个体都可追溯到一对共同祖先,个体在遗传上是同源的,基因型完全一致。
分子生物学考试复习题及答案

分⼦⽣物学考试复习题及答案第⼀章绪论⼀.简述分⼦⽣物学的主要内容。
1.DNA重组技术(⼜称基因⼯程)2.基因表达调控研究3.⽣物⼤分⼦的结构功能的研究——结构分⼦⽣物学4.基因组、功能基因组与⽣物信息学研究⼆.什么是遗传学的中⼼法则和反中⼼法则?遗传学中⼼法则:描述从⼀个基因到相应蛋⽩质的信息流的途径。
遗传信息贮存在DNA中,DNA被复制传给⼦代细胞,信息被拷贝或由DNA转录成RNA,然后RNA翻译成多肽。
反中⼼法则:由RNA逆转录到DNA,再由DNA转录到RNA,然后RNA翻译成多肽、蛋⽩质。
第⼆章染⾊体与DNA⼀、名词解释半保留复制:DNA复制时,以亲代DNA的每⼀条链作为模版,合成完全相同的两个双链⾃带DNA分⼦,每个⼦代DNA分⼦中都含有⼀条亲代DNA链的复制⽅式。
冈崎⽚段:在DNA复制过程中,前导链能连续合成,⽽滞后链只能是断续的合成5 3 的多个短⽚段,这些不连续的⼩⽚段称为冈崎⽚段。
复制⼦:从复制原点到终点,组成⼀个复制单位,叫复制⼦。
插⼊序列:最简单的转座⼦,不含有任何宿主基因,它们是细菌染⾊体或质粒DNA的正常组成部分。
DNA的变性和复性:变性:指DNA双链的氢键断裂,完全变成单链。
复性:指热变性的DNA缓慢冷却,单链恢复成双链。
双向复制:复制从⼀个固定的起始点开始,同时向两个⽅向等速进⾏。
引物酶:⼀种特殊的RNA聚合酶,在DNA模版上合成⼀段RNA链,这段RNA链是DNA复制起始时必需的引物。
转座⼦:存在与染⾊体DNA上可⾃主复制和位移的基本单位。
碱基切除修复:⾸先由糖苷⽔解酶识别特定受损核苷酸上的N-β糖苷键,在DNA上形成AP位点,由AP核酸内切酶把受损的核苷酸的糖苷-磷酸键切开,移去包括AP位点在内的⼩⽚段DNA,由聚合酶Ⅰ合成新⽚段,DNA连接酶最终把两者连接成新的DNA链。
DNA重组修复:即“复制后修复”。
机体细胞对在复制起始时尚未修复的DNA部位可先复制再修复。
在复制时,先跳过损伤部位,在和成链中留下⼀个缺⼝。
第6章 分子生物学研究方法(下)

④具有良好的机械性能
非特异吸附少
4.2 常用的固相支持物 ①硝酸纤维素膜:优点是吸附能力强,杂交信号本 底低。缺点是DNA分子结合不牢固 ②尼龙膜:优点是结合单链,双链DNA的能力比硝酸 纤维素膜强;缺点:杂交信号本底高 ③化学活化膜:优点:DNA与膜共价结合;对不同 大小的DNA片段有同等结合能力;缺点:结合能 力较上述两种膜低
(2)电转法 利用电场的电泳作用将凝胶中的DNA转移到 固相支持物上。
(3)真空转移法
此法原理与毛细管虹吸法相同,只是以滤膜在下,凝
胶在上的方式将其放臵在一个真空室上,利用真空作用
将转膜缓冲液从上层容器中通过凝胶和滤膜抽到下层真 空室中,同时带动核酸片段转移到凝胶下面的尼龙膜或
硝酸纤维素膜上。
各种转移方法的比较:
6.1.2 RNA选择性剪接技术
RNA的选择性剪接是指用不同的剪接方式从 一个mRNA前 体产生不同的mRNA剪接异构 体的过程。可分为:平衡剪 切、 5’ 选择性剪切、 3’ 选择性剪切、外显子遗漏型剪切及 相互排斥性剪切。常用RT-PCR法研究某个基 因是否存在 选择性剪切。 果蝇 Dscam 基 因可 以通过可变 剪接产生38000多 种 可 能 的 mRNA 异构体
Illumina Solexa 测序 Workflow
Illumina Sequencing Technology
有参考基因组序列生物信息分析
• 基因结构优化
• 鉴定基因可变剪接 • 预测新转录本 • SNP 分析 • 基因融合鉴定
5.4.1 RACE 法克隆基因全长 (Rapid Amplification of cDNA Ends)
4.3 Southern印迹的常用方法 (1)毛细管虹吸印迹法
6分子生物学研究法(下)——基因功能研究技术

目前,主要采用两种PCR方法,重叠延伸技术(左图)和 大引物诱变法(右图),在基因序列中进行定点突变。
6.2 基因敲除技术
1、基本原理
经典遗传学(Forward genetics)是从一个突变体的表型出 发,研究其基因型,进而找出该基因的编码序列。 现代遗传学(Reverse genetics,反向遗传学)首先从基因 序列出发,推测其表现型,进而推导出该基因的功能。 基因敲除(gene knock-out)又称基因打靶,通过外源DNA 与染色体DNA之间的同源重组,进行精确的定点修饰和基因 改造,具有专一性强、染色体DNA可与目的片段共同稳定遗 传等特点。 基因敲除分为完全基因敲除和条件型基因敲除(又称不完全 基因敲除)两种。 完全基因敲除是指通过同源重组法完全消除细胞或者动植物 个体中的靶基因活性,条件型基因敲除是指通过定位重组系 统实现特定时间和空间的基因敲除。 噬菌体的Cre/Loxp系统、Gin/Gix系统、酵母细胞的 FLP/FRT系统和R/RS系统是现阶段常用的四种定位重组系 统,尤以Cre/Loxp系统应用最为广泛。
ChIP不仅可以检测体内转录因子与 DNA的动态作用,还可以用来研究 组蛋白的各种共价修饰与基因表达 的关系。 定性或定量检测体内转录因子与 DNA的动态作用。 ChIP-chip and ChIP-seq:在基因 组水平研究DNA结合蛋白、组蛋白 修饰以及核小体分布。 ChIP-seq相对于ChIP-chip来说,具 有更高的分辨率、更小的噪音以及 更广的基因组覆盖范围。 Nucleosome Occupancy study: 一 些转录因子本身并不含有DNA结合 结构域,它是通过参与形成蛋白复 合物从而改变染色质结构来发挥作 用的;因此我们可以通过研究基因 组上核小体的分布变化来揭示转录 因子作用的过程。
分子生物学 第六章

摆动性
• 反密码子与密码子之间的配对并不完全遵照 碱基互补规律,称为摆动配对。
二、tRNA
(一)结构特点 1.二级结构:三叶草结构
四环: 二氢尿嘧啶环 反密码子环 额外环 胸腺嘧啶假尿嘧啶胞嘧啶环 一臂: 氨基酸接受臂
2.三级结构——“倒L型”
(二)起始tRNA
密码子 氨基酸 表示方法
(二)延伸
1.进位 • 氨酰-tRNA 按照mRNA 分子的编码 信息进入并 结合到核糖 体A位。
(二)延伸
2.成肽
• 转肽酶催化 肽酰-tRNA 上的肽酰基 转移到A位 氨酰-tRNA 上的氨基酸 α-氨基上。
(二)延伸
3.转位
• 转位酶催化核 糖体沿mRNA 的3‘方向移动 一个密码子的 距离,使 mRNA上的下 一个密码子进 入A位,肽酰tRNA由A位移 入P位。
三、修饰
(一)磷酸化 是指在蛋白激酶的催化作用下,ATP的γ-磷酸 基被转移到蛋白质特定位点上的过程。 通常蛋白质的丝氨酸、苏氨酸和在糖基转移酶的作用下,蛋白质的特定 氨基酸残基被共价连接上寡糖链的过程。 • 糖链与氨基酸的连接主要有O型连接和N型 连接两种方式。
终止密码子: 琥珀石(UAG) 赭石(UAA) 卵白石(UGA)
起始密码子: AUG(甲硫氨酸)
2.特性
(1)完整性:有始有终 (2)方向性:5’到3’ (3)连续性:不中断、无重叠 (4)简并性:多对一 (5)统一性:万物统一 (6)摆动性::3’位可变 (7)偏爱性:使用频率各异
简并性
• 一种氨基酸具有 两个或两个以上 的密码子为其编 码,这一特性称 为遗传密码的简 并性。
一、mRNA (一)结构特点
原核 生物
真核 生物
第六章:分子生物学研究方法(下全文编辑修改

人类染色体端粒DNA的荧光原位杂交
4. 基因定点突变技术
• 通过改变基因特定位点核苷酸序列来改变所编码 的氨基酸序列,用于研究某个(些)氨 基酸残基 对蛋白质结构功能的影响。
二、基因敲除技术
• 基因敲除(gene knock-out)又称基因打靶,通 过外源DNA与染色体DNA之间的同源重组,进行 精确的定点修饰和基因改造。
方法: • 以9~11bp作为标签(tag)=49=262144组合 • 串联tag并通过两端接头PCR扩增 • 扩增产物进行测序 • 每个tag通过Genebank或EST数据库进行比对 • 确认tag代表的基因表达情况
2. RNA的选择性剪接从一个 mRNA前体产生不同的mRNA剪接异构体的过程。
2.凝胶阻滞试验 ➢ 是用于研究DNA与蛋白质相互作用的一种
特殊的凝胶电泳技术。
➢ 当DNA与蛋白质结合时,在电泳中会受到阻滞, 说明可能与某种特殊蛋白结合了。
3. DNAase足迹试验 ➢ 蛋白质结合在DNA片段上,能保护结合部位不被
DNAase水解,电泳中对应于结合部位没有条带。
• 常用RT-PCR法研究某个基因是否存在选择性剪切。
3.原位杂交技术( In Situ Hybridization,ISH)
• 用标记的核酸探针,在组织、细胞上对核酸进行 定位和相对定量研究的一种手 段。
• 探针标记用同位素或地高辛、生物素荧光标记等。
地
同
高
位
辛
素
标
标
记
记
染色体原位杂交
三、蛋白质与RNA、DNA相互作用
1. 酵母单杂交系统 • 将待测转录因子cDNA与表达载体导入酵母细胞,
该基因产物如 果能够与顺式作用元件相结合,就 能激活启动子,使报告基因得到表达。
分子生物学的研究方法例题和知识点总结

分子生物学的研究方法例题和知识点总结分子生物学作为一门研究生物大分子结构与功能的学科,对于理解生命现象的本质具有至关重要的意义。
其研究方法多种多样,涵盖了从分子水平到细胞水平,再到整体生物个体水平的多个层次。
下面我们将通过一些具体的例题来深入探讨分子生物学的研究方法,并对相关知识点进行总结。
一、分子克隆技术分子克隆技术是分子生物学研究中最常用的方法之一,它能够实现特定 DNA 片段的复制和扩增。
例题:假设我们需要从一个复杂的基因组中克隆出编码某种特定蛋白质的基因。
首先,我们通过设计特异性引物,利用 PCR(聚合酶链式反应)技术扩增出目标基因片段。
然后,将这个片段插入到合适的载体(如质粒)中,转化到宿主细胞(如大肠杆菌)中进行扩增和表达。
知识点:1、 PCR 技术的原理:基于 DNA 半保留复制的原理,通过高温变性、低温退火和适温延伸的循环过程,实现 DNA 片段的指数级扩增。
2、载体的选择:常见的载体有质粒、噬菌体和病毒等,需要根据实验目的和宿主细胞的特性来选择。
3、转化方法:包括化学转化法、电穿孔法等,目的是将重组载体导入宿主细胞。
二、核酸杂交技术核酸杂交技术是基于核酸分子的碱基互补配对原则,用于检测特定核酸序列的存在。
例题:为了检测某一细胞样本中是否存在特定的 mRNA 分子,我们可以设计与之互补的 DNA 探针,进行 Northern 杂交实验。
知识点:1、 Southern 杂交:用于检测 DNA 分子。
2、 Northern 杂交:用于检测 RNA 分子。
3、探针的标记:可以采用放射性同位素标记或非放射性标记(如荧光标记、生物素标记等)。
4、杂交条件的优化:包括温度、离子强度等,以保证特异性杂交的发生。
三、基因测序技术基因测序技术能够确定 DNA 或 RNA 分子的核苷酸序列。
例题:对一个未知基因进行测序,以确定其碱基组成和排列顺序。
知识点:1、 Sanger 测序法:通过双脱氧核苷酸终止法进行测序。
分子生物学研究方法

分子生物学研究方法
分子生物学研究方法是研究生物分子结构、功能和相互作用的一系列实验方法和技术。
这些方法帮助科学家了解细胞的基本结构和功能,研究生物分子在疾病发展、遗传变异和进化中的作用。
以下是一些常用的分子生物学研究方法:
1. DNA提取:从细胞或组织中提取DNA,以用于后续实验。
2. 聚合酶链式反应(PCR):用于扩增DNA片段,以便进行分析和检测。
3. 凝胶电泳:用电场将DNA、RNA或蛋白质分离成不同大小的片段,以便研究其结构和功能。
4. 蛋白质纯化:通过一系列步骤将目标蛋白质从混合物中纯化出来,以获得足够的纯度用于研究。
5. 克隆:将DNA序列插入到载体中,以产生大量目标DNA 分子,用于进一步的分析和实验。
6. 基因测序:确定DNA序列的顺序,以研究基因功能、分析遗传变异或进行进化研究。
7. 基因表达:将目标基因转录成mRNA,并翻译成蛋白质,以研究基因功能和调控机制。
8. 蛋白质相互作用:使用技术如亲和层析、酵母双杂交等研究蛋白质之间的相互作用关系,以探索细胞信号传导和代谢途径。
9. 基因编辑:利用技术如CRISPR/Cas9,对细胞或生物体的基因进行精确的编辑,以研究基因功能或治疗遗传疾病。
分子生物学研究方法的不断发展和创新使得科学家可以更深入地了解生物分子的结构、功能和相互作用,为疾病治疗和生物技术的发展提供了基础。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
5
第一节 基因表达研究技术
2 RNA的选择性剪接技术
❖ RNA的选择性剪接是指用不同的剪接方式从 一个mRNA前 体产生不同的mRNA剪接异构 体的过程。可分为:平衡剪 切、5’选择性剪 切、3’选择性剪切、外显子遗漏型剪切及 相 互排斥性剪切。常用RT-PCR法研究某个基 因是否存在 选择性剪切。
负对照, mRNA的 同义 链, 无杂交 信号
2020/8/14
9
正对照, UBQ10杂交信 号遍布于 整个 胚珠
第一节 基因表达研究技术
3. 原位杂交技术
❖ 荧光原位杂交(FISH)
❖ 首先对寡核苷酸探针做特殊的修饰和标记, 然后用原位杂 交法与靶染色体或DNA上特定 的序列结合,再通过与荧光 素分子相耦联的 单克隆抗体来确定该DNA序列在染色体上 的 位置。
2020/8/14
11
寡核苷酸 介导的 DNA 突变技 术
2020/8/14
12
重叠延伸介导的定点诱变
2020/8/14
13
大引物诱变法
第二节 基因敲除技术
1. 基本原理
❖ 经典遗传学(Forward genetics)是从一 个突变体的表型 出发,研究其基因型,进而 找出该基因的编码序列。
❖ 现代遗传学(Reverse genetics,反向遗传 学)首先从基 因序列出发,推测其表现
❖ T-DNA插入失活技术是目前在植物中使用最 为广泛的基 因敲除手段。
❖ 利用根癌农杆菌T-DNA介导转化,将带有报 告基因的 DNA序列整合到基因组DNA上,如 果这段DNA插入到目 的基因内部或附近,就 会影响该基因的表达,从而使该 基因“失 活”。
❖ LongSAGE技术。标签来自转录物3’端一段21bp的序列, 可 以进行快速分析并与基因组序列数据相匹 配。其原理与 ShortSAGE方法类似,只是 用了不同的IIS类标签酶( MmeI),并将程 序做了相应修改。
2020/8/14
4
常规SAGE方法 (short SAGE) 基本 流程
2020/8/14
2020/8/14
10
第一节 基因表达研究技术
4. 基因定点突变技术
❖ 通过改变基因特定位点核苷酸序列来改变所 编码的氨基酸 序列,用于研究某个(些)氨 基酸残基对蛋白质的结构、 催化活性以及结 合配体能力的影响,也可用于改造DNA调 控 元件特征序列、修饰表达载体、引入新的酶切位点等。
❖ 目前,主要采用两种PCR方法,重叠延伸技 术和大引物诱 变法,在基因序列中进行定点 突变。
果 蝇 Dscam 基因可 以通过可 变剪接产生 38000 多 种 可 能 的mRNA异构体
2020/8/14
6
选择性剪切的不同类型
RT-PCR检测5个拟南芥转录 调控因子基因的选择性剪切
2020/8/14
7
第一节 基因表达研究技术
3. 原位杂交技术
❖ 原位杂交(ISH)是 用标记的核酸探针,经放射自显影或 非放射 检测体系,在组织、细胞、间期核及染色体 上对核 酸进行定位和相对定量研究的一种手 段,分为RNA和染色 体原位杂交两大类。
❖ 显微注射命中率较高,技术难度相对大些。 电穿孔法命 中率比显微注射低,操作使用方 便。
❖ 胚胎干细胞(ES细胞)分离和体外培养的 成功奠定了哺 乳动物基因敲除的技术基础。
2020/8/14
18
模式动物小鼠 中完全基因敲除的 主要技术 策略与 应用。
2020/8/14
19
第二节 基因敲除技术
3. 植物基因敲除技术
❖ 基因敲除(gene knock-out)又称基因打 靶,通过外源 DNA与染色体DNA之间的同源重 组,进行精确的定点修 饰和基因改造,具有 专一性强、染色体DNA可与目的片段 共同稳 定遗传等特点。型, 进而推导出该基因的功能。
2020/8/14
14
1. 基本原理
❖ 基因敲除分为完全基因敲除和条件型基因敲除(又称不完 全基因敲除)两种。
完全基因敲除是指通过同源重组法完全消除细胞或者 动物个体中的靶基因活性
条件型基因敲除是指通过定位重组系统实现特定时 间 和空间的基因敲除。
❖ 噬 菌 体 的 Cre/Loxp 系 统 、 Gin/Gix 系 统 、 酵 母 细 胞 的 FLP/FRT系统和R/RS系统是现阶段常 用的四种定位重组 系统,尤以Cre/Loxp系统 应用最为广泛。
2020/8/14
2
第六章 分子生物学研究法(下)
❖内容: 1. 基因表达研究技术 2. 基因敲除技术 3. 蛋白质及RNA相互作用技术 4. 基因芯片及数据分析 5. 其他分子生物学技术
2020/8/14
3
第一节 基因表达研究技术
1. 基因表达系列分析技术(SAGE)
❖ SAGE是以DNA序列测定为基础分析全基因组表达 模式的 技术。任何长度超过9-10个碱基的核 苷酸片段都可能代表 一种特异性的转录产 物,因此,根据某个序列占总标签数 的比例 可分析所对应基因的表达频率。
第六章 分子生物学研究法(下)
——基因功能研究技术
2020/8/14
1
第六章 分子生物学研究法(下)
从20世纪中叶开始,分子生物学研究得到高速 发展,主要原因之一是研究方法,特别是基因操作 和基因工程技术的进步。
基因操作主要包括DNA分子的切割与连接、核 酸分子杂交、凝胶电泳、细胞转化、核酸序列分析 以及基因人工合成、定点突变和PCR扩增等,是分 子生物学研究的核心技术。
2020/8/14
15
用取代型(a)或插入型(b)载体进行完全基因敲除实验
2020/8/14
16
正负筛选法(PNS法)筛选已发生同源重组的细胞
2020/8/14
17
第二节 基因敲除技术
2. 高等动物基因敲除技术
❖ 真核生物基因敲除的技术路线主要包括构建 重组基因载 体,用电穿孔、显微注射等方法 把重组DNA导入胚胎干 细胞纯系中,使外 源DNA与胚胎干细胞基因组中相应部 分发 生同源重组,将重组载体中的DNA序列整 合到内源 基因组中并得以表达。
❖ RNA原位杂交用放射性或非放射性(如地高 辛、生物素等 )标记的特异性探针与被固定 的组织切片反应,若细胞中 存在与探针互补 的mRNA分子,两者杂交产生双链RNA, 可 通过放射性标记或经酶促免疫显色,对该基 因的表达产 物做出定性定量分析。
2020/8/14
8
mRNA的互补链, 杂交信号集中于 纤 维细胞中。
