spm8详细教程
SPM中文教程 汇总(已整理)

一、SPM的安装与启动先安装matlab,然后将SPM复制到matlab下的一个文件夹(SPM2需要matlab6.0或以上版本)。
启动matlab,首先set path,然后在matlab命令窗口中输入SPM即可启动,然后选择fMRI,也可以直接输入SPM fMRI二、SPM数据处理概要先将所得数据进行空间预处理(对齐,平滑,标准化等),然后进行模型估计(将刺激的时间、间隔与血流动力函数进行卷积,所得结果与全脑象素信号进行相关分析),最后察看结果。
三、SPM8数据处理的一般步骤为方便后续的数据处理,如果数据分散处理后整合,建议所有处理数据路径保持一致,要统一路径。
处理前首先要采用数据转换软件将dicom数据转换成SPM解析格式,然后进行数据预处理,预处理结束后到matlab安装目录中备份spm*.ps文件,其中包含了空间校正和标准化的信息,然后进行建模分析。
运行命令:spm fmri,打开spm8的操作界面我们称左上侧的窗口为按钮窗口(button window),左下侧的窗口为输入窗口(input window),右侧大窗口为树形结构窗口或图形窗口(Tree Building Window or the graphics window)。
在spm8和spm5中,每一步处理都采用了直观的“树形结构”的面板,如果一个分支项左面有“+”号,你可以双击显示子分支项,如果一个分支项右面有“<-X”号,你必须为之指定选项(否则不能运行该tree),分支项的选项在其右侧面板指定,而帮助信息则在下面的面板中显示。
如果我们处理数据没有特殊需求,我们只关心带有“<-X”项目并完成输入即可,其余均可采用默认设置。
另外注意在Tree Building Window的顶部菜单,新增了一个菜单项“TASKS”,在使用批处理分析时非常重要。
以下内容,还可以参考E:\《汇总》中“静息态fMRI的数据预处理流程”这部分的讲述。
SPM8数据预处理
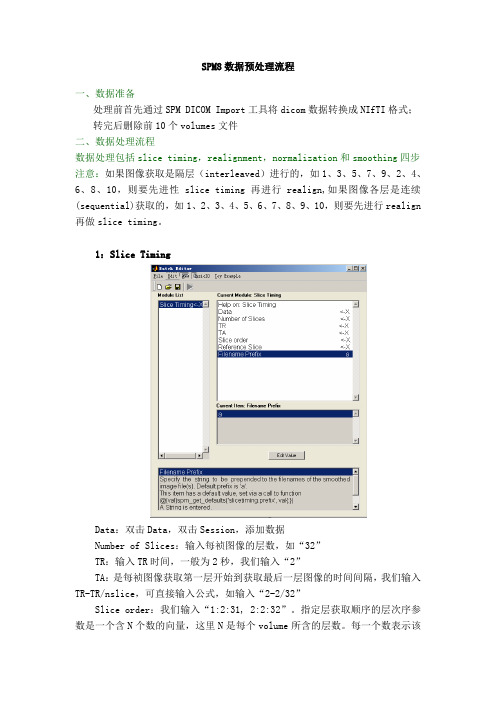
SPM8数据预处理流程一、数据准备处理前首先通过SPM DICOM Import工具将dicom数据转换成NIfTI格式;转完后删除前10个volumes文件二、数据处理流程数据处理包括slice timing,realignment,normalization和smoothing四步注意:如果图像获取是隔层(interleaved)进行的,如1、3、5、7、9、2、4、6、8、10,则要先进性slice timing 再进行realign,如果图像各层是连续(sequential)获取的,如1、2、3、4、5、6、7、8、9、10,则要先进行realign 再做slice timing。
1:Slice TimingData:双击Data,双击Session,添加数据Number of Slices:输入每祯图像的层数,如“32”TR:输入TR时间,一般为2秒,我们输入“2”TA:是每祯图像获取第一层开始到获取最后一层图像的时间间隔,我们输入TR-TR/nslice,可直接输入公式,如输入“2-2/32”Slice order:我们输入“1:2:31, 2:2:32”。
指定层获取顺序的层次序参数是一个含N个数的向量,这里N是每个volume所含的层数。
每一个数表示该层在图像(volume)中的位置。
向量内的数字排列顺序是这些层的获取时间顺序。
如行向量 [1 3 5 7 9 11 13 15 17 19 21 23 25 2 4 6 8 10 12 14 16 18 20 22 24](在Matlab中可表示为[1:2:25,2:2,25])各种扫描类型和输入的层顺序如下:ascending 升序扫描(first slice=bottom): [1:1:nslices];descending 降序扫描(first slice=top): [nslices:-1:1];interleaved 间隔扫描(middle-top):for k = 1:nslices,round((nslices-k)/2 + (rem((nslices-k),2) * (nslices - 1)/2)) + 1,end; interleaved (bottom -> up): [1:2:nslices 2:2:nslices],如[1:2:25,2:2,25];interleaved (top -> down): [nslices:-2:1, nslices-1:-2:1]Reference Slice:我们输入“31”。
磁共振实验数据SPM8处理流程

磁共振实验数据SPM8处理流程磁共振实验数据的处理流程主要包括原始数据预处理、功能图像处理和统计分析三个步骤。
其中,SPM8是一款常用的功能磁共振成像软件,可以用于实现磁共振实验数据的处理与分析。
以下是磁共振实验数据SPM8处理流程的详细步骤:1.原始数据预处理:(1)数据导入:将原始数据导入SPM8软件,通常数据的格式为DICOM 或NIfTI格式。
(2)切片时间校正:根据数据采集过程中的切片顺序和时间信息,对数据进行切片时间校正,以获得各个时间点上的切片数据。
(3)运动校正:校正数据中由于受试者运动引起的图像运动,通常使用刚体变换或流形形变方法进行运动校正,以消除不同时间点之间的运动偏移。
(4)标准空间转换:将运动校正后的数据转换到参考空间中,通常使用统一分割方法,将每个受试者的数据映射到一个标准的空间模板上。
(5)降噪处理:对数据进行降噪处理,一般使用高斯平滑方法,平滑数据以减小噪声干扰。
2.功能图像处理:(1)激活检测:通过对原始数据进行激活检测,识别可能激活区域。
常用的方法包括广义线性模型(GLM)和独立成分分析(ICA)等。
(2) 功能图像标准化:将激活检测得到的功能图像进行标准化处理,以便进行后续的统计分析。
常用的方法包括z-score标准化和百分位标准化等。
(4)时间序列提取:从功能图像中提取感兴趣脑区的时间序列数据,以便进行统计分析。
3.统计分析:(1)组间比较:使用统计学方法对不同组别的数据进行比较,常用的方法包括单样本t检验、双样本t检验和方差分析等,可以得到激活区域和激活程度的差异信息。
(2) 相关性分析:分析不同脑区之间的功能连接关系,通常使用Pearson相关系数或互信息等方法进行分析。
(3)多变量分析:使用多变量模式识别方法(如支持向量机、随机森林等)对脑图像数据进行分类或预测,以探索脑区之间的潜在模式。
以上是磁共振实验数据SPM8处理流程的主要步骤,这些步骤将原始数据进行预处理,提取功能图像,以及进行统计分析,从而得出与研究目的相关的结论。
ASL数据处理教程

选择MASK,根据图像 dimension大小选择MASK 输出目录 Let`s go!
谢谢观赏 !
标准化
进入标准化界面
Normalize
T1分割后“seg-sn” 文件 ASL功能像图像
双击,按下图所示修改数值
平滑
进入平滑界面
Smooth
标准化后的功能像
双击,修改数值如图所示
统计分析
预处理已经完成
进行统计分析,本文选用配对T检验
打开rest,进入统计分析界面
Paired T-test
3D-ASL数据预处理
所需软件PM fmri
配准
进入配准界面
Coregister
ASL CBF图像(功能像)
T1图像(结构像)
T1分割
进入分割界面 分割结构像
Segment
配准后的T1结构像
两项均选择 “Native + Modulate Normalize”
spmfMRI数据处理

目录一、数据准备(先设置数据输入和输出目录,再转换数据格式) ............ 二、数据预处理流程 .................................................. 0、预处理的 workflow................................................. 1、SliceTiming 时间层校正............................................ 2、Realignment 头动校正.............................................. 3、Coregister 配准................................................... 4、Segment 分割...................................................... 5、Normalize 空间标准化.............................................. 6、Smooth 平滑....................................................... 三、GLM 模型和 Specify1st-level ...................................... 四、实例:任务态数据预处理和一阶分析的批处理 ........................ SPM 简介和安装SPM,即统计参数图,也是这个软件的最终输出,它是由英国伦敦大学的 Friston 教授 等人在通用数学软件包 Matlab 基础上开发的软件系统,其统计功能非常强大,设计这个软 件包的初衷是采用统计的方法来处理 fMRI,PET 和 SPECT 的数据。
VBM8使用手册

VBM8 处理流程1.下载和安装➢SPM8:从SPM官方网站( )下载SPM8及最新的update包。
把SPM8解压到要安装的目录,同时把update包解压,并直接覆盖SPM8相关内容,完成更新。
然后打开matlab,把spm8文件夹加入matlab 路径目录,即完成安装。
➢VBM8:从VBM官方网站(()下载压缩包,解压后放入spm8/toolbox下,即完成安装。
➢运行VBM8:1.启动matlab2.>> spm fmri3.Spm8→toolbox→VBM82.VBM8分析流程简要➢预处理1.把T1W 标准化到MNI space,.并分割出灰质(GM),白质(WM),脑积液(CSF).相关参数可以通“Estimate and write”模块来调整。
2.通过“VBM8 Check data quality” 菜单中的“Display One slice for all images”和“Checksample homogeneity using covariance”两个选项检查分割和标准化的质量。
3.采用spm自带的spm →smooth选项对预处理好的组织图像进行平滑。
➢统计分析1.通过SPM→ Specify 2nd Level 模块指定统计模型。
2.采用SPM→ Estimate 模块估计模型3.采用SPM→ Results 模块定义contrast,观测结果。
3.VBM 分析流程详细描述➢组织分割与标准化VBM8→ Estimate and writeVolumes ←X :输入解剖图像,一般为T1W图像。
由于在后续分割中,需要和MNI先验模板对齐,所以这里输入数据最好能和先验MNI 模板方向大致相同。
若图像和模板方向差异较大,可以使用SPM的Display 和Check Reg按钮进行手动调整。
Estimation Options:使用默认参数即可。
这里若不采用SPM自带的组织先验模板TPM,则可选择自己定制的模板。
SPM使用简介

SPM使用简介一、Spm的安装与启动:先安装matlab,然后将spm复制到matlab下的一个文件夹(Spm2需要和matlab6.0或以上版本配合使用)。
启动matlab,首先set path,然后在matlab命令窗口中输入spm即可启动,然后选择fmri,也可以直接输入spm fmri二、Spm数据处理概要先将所得数据进行空间预处理(对齐,平滑,标准化等),然后进行模型估计(将刺激的时间、间隔与血流动力函数进行卷积,所得结果与全脑象素信号进行相关分析),最后察看结果。
三、Spm数据处理一般步骤1、转换数据dicom格式转换为img文件,将以层为单位的数据转换成以全脑为单位的数据。
2、Slice timing校正系列成像中层与层获得时间的不同,使一个TR中的各层获得时间一致(如都在一个TR的开始),相当于afni中tshift所做的工作。
3、Realign(相当于afni中的registration)分两步:1)coregister,将每个session的第一个scan与第一个session的第一个scan进行比较,然后将每个sessioni中的其他scan与本session中的第一个scan进行比较,得到每个filename.img 文件的转换参数,生成filename.mat文件,同时为每个session生成一个对齐参数(realignment parameters),文件名为realignment_params_*.txt2)reslice,用filename.mat文件对filename.img重新切片,生成rfilename.img文件。
并可依选择生成一个平均象,名为meanfilename.img。
4、Normailze选用realign步骤中得到的平均象与模板进行比较,获得进行标准化的参数,参数文件命名为filename_sn3d.mat,然后依据此参数文件对每个img文件进行标准化,生成文件nfilename.img.5、Smooth推荐为象素大小的两至三倍。
SPM中文教程汇总已修订版

S P M中文教程汇总已 Document number:PBGCG-0857-BTDO-0089-PTT1998一、SPM的安装与启动先安装matlab,然后将SPM复制到matlab下的一个文件夹(SPM2需要或以上版本)。
启动matlab,首先set path,然后在matlab命令窗口中输入SPM即可启动,然后选择fMRI,也可以直接输入SPM fMRI二、SPM数据处理概要先将所得数据进行空间预处理(对齐,平滑,标准化等),然后进行模型估计(将刺激的时间、间隔与血流动力函数进行卷积,所得结果与全脑象素信号进行相关分析),最后察看结果。
三、SPM8数据处理的一般步骤为方便后续的数据处理,如果数据分散处理后整合,建议所有处理数据路径保持一致,要统一路径。
处理前首先要采用数据转换软件将dicom数据转换成SPM解析格式,然后进行数据预处理,预处理结束后到matlab安装目录中备份spm*.ps文件,其中包含了空间校正和标准化的信息,然后进行建模分析。
运行命令:spm fmri,打开spm8的操作界面我们称左上侧的窗口为按钮窗口(button window),左下侧的窗口为输入窗口(input window),右侧大窗口为树形结构窗口或图形窗口(Tree Building Window or the graphics window)。
在spm8和spm5中,每一步处理都采用了直观的“树形结构”的面板,如果一个分支项左面有“+”号,你可以双击显示子分支项,如果一个分支项右面有“<-X”号,你必须为之指定选项(否则不能运行该tree),分支项的选项在其右侧面板指定,而帮助信息则在下面的面板中显示。
如果我们处理数据没有特殊需求,我们只关心带有“<-X”项目并完成输入即可,其余均可采用默认设置。
另外注意在Tree Building Window的顶部菜单,新增了一个菜单项“TASKS”,在使用批处理分析时非常重要。
spmfMRI数据处理

目录SPM 简介和安装..............................................................................................................................1 一、数据准备(先设置数据输入和输出目录,再转换数据格式) ...........................................2 二、数据预处理流程 ....................................................................................................................... 2 0、预处理的 workflow ....................................................................................................................2 1、Slice Timing 时间层校正 ...........................................................................................................3 2、Realignment 头动校正 ..............................................................................................................3 3、Coregister 配准..........................................................................................................................5 4、Segment 分割.............................................................................................................................6 5、Normalize 空间标准化..............................................................................................................6 6、Smooth 平滑 ..............................................................................................................................8 三、GLM 模型和 Specify 1st-level.................................................................................................9 四、实例:任务态数据预处理和一阶分析的批处理 ................................................................. 13SPM 简介和安装SPM,即统计参数图,也是这个软件的最终输出,它是由英国伦敦大学的 Friston 教 授等人在通用数学软件包 Matlab 基础上开发的软件系统,其统计功能非常强大,设计这个 软件包的初衷是采用统计的方法来处理 fMRI,PET 和 SPECT 的数据。
SPM数据处理

Realign
Coregistrer
r
r
rr*
Normalization
将实验数据从被试自己的脑空间配准至标准脑空间 SPM中使用MNI脑图谱 标准化时,部分选项的选择与前期是否经过coregister
会有所不同。 本教程假设没有经过coregister,即不使用被试的T1结
构相信息,而直接完成EPI --- EPI的配准过程。
Normalize
为方便与其它程序 结合,这里建议改 为 -90 -126 -72; 90 90 108. 这时得到的结果是 61 * 73 * 61的。
体素大小改为3,3,3
注:默认的boudingbox 对应的是53 * 63 * 46
其它参数不变,然后就可以点RUN了 ,将得到一堆wr*的文件
数据导入
选择文件夹保存输出文件
数据导入
选择好相 应的文件 夹后,单 击“.\”
单击“Done”确认
数据导入
选择输出文件格式(可以根据习惯选择,都可用, spm5也支持4d的数据)
数据导入
开始转换
数据导入
数据导入
这一步会生成一个.mat文件,会在解剖像文件名 加“s”的前缀,在功能像文件名加“f”的前缀,(s 和f的前缀取决于dicom的头信息)。
Cautious: 特殊原因,此示例图来自SPM8
Estimate and Write
选择realign得到的 mean*文件 在文件选择框中选择所 有经过realign得到的功 能相文件,如r*.img
选择SPM安装 路径下 templates目录 中EPI.nii文件
注:如果前期经过coregister,这里的source image将为被试 的T1结构相,而Template Image将改为同目录下T1.nii
ASL数据处理教程PPT资料

标准化
进入标准化界面
Normalize
T1分割后“seg-sn” 文件 ASL功能像图像
双击,按下图所示修改数值
平滑
进入matlab,打开SPM fmri 进行统计分析,本文选用配对T检验 Ps:默认的检验方式为 进行统计分析,本文选用配对T检验 “Native + Modulate Normalize” 进入matlab,打开SPM fmri 点击红框,将两组数据所在文件夹添加,主要每个人前后数据命名应对应。 “Native + Modulate Normalize” 进行统计分析,本文选用配对T检验 T1分割后“seg-sn”文件 所需软件:matlab spm8 rest 3D-ASL数据预处理 所需软件:matlab spm8 rest
matlabspm8restmatlabspm8restmatlabmatlabspmfmrispmfmri进入配准界面coregistercoregisteraslcbf图像功能像t1图像结构像t1t1进入分割界面分割结构像进入分割界面分割结构像segmentsegment配准后的t1结构像两项均选择nativemodulatenormalize进入标准化界面normalizenormalize双击按下图所示修改数值asl功能像图像t1分割后segsn文件进入平滑界面smoothsmooth标准化后的功能像双击修改数值如图所示预处理已经完成预处理已经完成进行统计分析本文选用配对进行统计分析本文选用配对tt检验检验restrestpairedpairedttesttest点击红框将两组数据所在文件夹添加主要每个人前后数据命名应对应
3D-ASL数据预处理
所需软件:matlab spm8 rest
进入matlab,打开SPM fmri
spm8详细教程

spm8详细教程SPM8数据处理教程Lab 1. 数据的预处理一、概述:数据的预处理(preprocess):(1)Convert dicom files to hdr files and img images;(2)Slice Timing;(3)Realign: Estimate & Reslice;(4)Coreg: Estimate;(5)Segment;(6)两次Normalise: Write;(7)Smooth;(8)Fmri model specification;(8)Model Estimation。
二、DICOM Import:说明:转换完后会在解剖像文件名前加s,在功能像文件名前加f。
可以用“Display”来查看刚转换的图像。
(Display还有一个重要的作用是定义原点——前联合和AC-PC连线,这在做Normalise中很重要。
定义后reorient一下即可写入头信息)。
三、Slice timing1、目的:使用数学的方法使不同时间扫面的层校正为同一时间获得层。
2、过程:Date.session---[选中所有要处理的f*.文件]Number of slices---[扫描层数]TRTA---[可以用公式表示:TR-(TR/nslices)]Slice order---[扫描顺序:例如1:2:31 2:2:32分别表示从1或2开始,每间隔2个数扫描一张,即1、3、5… 2、4、6…]Reference slice---[参考层,一般选择中间的一张,对于各层扫描,一般选择即是时间的中点,又是大脑的中点。
如果是层数为偶,可以选择中点处2层中的任一层]Filename Prefix—a [默认头文件名为a]3、说明:Block Design的实验数据这一步可以跳过;event related design的实验数据必须做这一步。
四、Realign: Estimate & Reslice1、目的:如果在容许的头动范围内,可以使用一定的算法校正信号,使其靠近真实值,如果超过了这个规定的范围,则必须剔除这组数据。
SPM8MEG处理流程
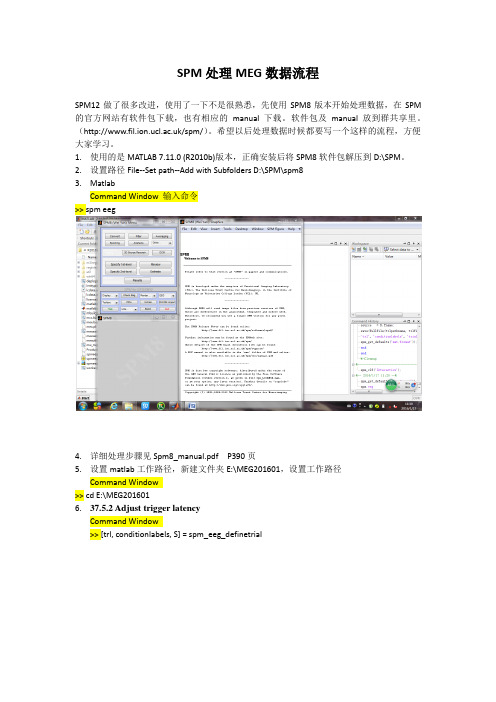
SPM处理MEG数据流程SPM12做了很多改进,使用了一下不是很熟悉,先使用SPM8版本开始处理数据,在SPM 的官方网站有软件包下载,也有相应的manual下载。
软件包及manual放到群共享里。
(/spm/)。
希望以后处理数据时候都要写一个这样的流程,方便大家学习。
1.使用的是MATLAB 7.11.0 (R2010b)版本,正确安装后将SPM8软件包解压到D:\SPM。
2.设置路径File--Set path--Add with Subfolders D:\SPM\spm83.MatlabCommand Window 输入命令>> spm eeg4.详细处理步骤见Spm8_manual.pdf P390页5.设置matlab工作路径,新建文件夹E:\MEG201601,设置工作路径Command Window>> cd E:\MEG2016016.37.5.2 Adjust trigger latencyCommand Window>> [trl, conditionlabels, S] = spm_eeg_definetrial选择数据EM008\LOW\文件夹中EM008CHENZHIBING_YAOZHIJIAN_20090415_03.ds.meg4 输入如下选择如下(暂时选择这个)•Answer “no” to the question about reviewing trials.•Answer “yes” to the prompt to save the trial definition.•Enter a filename like trials run1.mat and save in the MEG directory.7.37.5.3 Convert 数据转换Press the Convert button, and in the file selection window again select EM008CHENZHIBING_YAOZHIJIAN_20090415_03.ds.meg4 file. At the prompt“Define settings?” select “yes”. Here we will use the option to de fine more precisely the part of data that should be read during conversion. Answer “trials” to “How to read?”, and “file”to “Where to look for trials?”. Then in the file selector window, select the new trials run1.mat file. Press “no” to “Read across trials?” and select “meg” for “What channels?”. Press “no” to avoid saving the channel selection. Enter the name of the output dataset(espm8_EM008_LOW,这一步处理完会在文件前加字母“e”). One files will be generated espm8_EM008_LOW.dat. After the conversion is complete the data file will be automatically opened in the SPM8 reviewing tool. If you click on the “MEG” tab you will see the MEG data which is already epoched. By pressing t he “intensity rescaling” button (with arrows pointing up and down) several times you will start seeing MEG activity.点击上图MEG等可以初步查看数据8.37.5.4 Baseline correction 基线校正We need to perform baseline correction as it is not done automatically during conversion.This will prevent excessive edge artefacts from appearing after subsequent filtering and downsampling. Select Baseline correction from the “Other” drop-down menu and select the espm8_EM008_LOW.mat file. Enter [−200 0] for “Start and stop of baseline [ms]”. The progress bar will appear and the resulting data will be saved to dataset bespm8_EM008_LOW.{mat,dat}.9.37.5.5 Downsample降频Select Downsample from the “Other” drop-down menu and select the bespm8_EM008_LOW.mat file. Choose a new sampling rate of 200 (Hz). The progress bar will appear and the resulting data will be saved to dataset d bespm8_EM008_LOW.{mat,dat}.到目前为止,共生成如图文件10.以上是预处理步骤,后面是3D重建点击3D Source Reconstruct---Load---E:\MEG201601\dbespm8_EM008_LOW.mat Done输入low111.新建E:\MEG201601\MRI文件夹;点击主界面DICOM Import,选择\EM008\MRI\EM008\DICOM\PA1\ST1\SE1文件夹中所有文件,右键select all(IM1、IM10、IM100等所有文件),Done。
如何做spm8第二阶段

Group Analysis (Random Effects Analysis)In order to run the group statistical analysis, all images must be the same size, havethe same voxel sizes and the same origins.If .mat files are present (note that they often are not for the swr images), then they must all contain identical information. This means not only that they should begin as images of equivalent size and dimensions, but that they should be normalized and smoothed to the same sizes. Presumably, you should work with a statistically significant number of subjects (perhaps 6?)What you should do: Group analysis is considerably easier if all data is preprocessed and analyzed the same way and contrasts were all entered in the same order for each subject.Make a directory for the group analysis, so you don’t overwrite files (SPM.mat and all the images resulting from Estimation) in your subject directories. Make sure you are in your new group directory in Matlab, then start SPM.<Basic Models>Select Design Type-> One Sample t-testOne Sample T-Test: Is the mean signal value different than 0?Two Sample T-Test: Is the mean signal value of Group1 different than Group2?Paired T-Test: Is the mean signal value of Condition1 different than Condition2 (for one group)?One Way Anova: Is the mean signal value of 3 or more groups/conditions different?Simple Regression (Correlation): Does the variable 'a' in a linear regression equal 0?y=ax+b (y is the value of the contrast and x the predictive factor) Multiple Regression: Does the variable 'a' in a linear regression equal 0 for each predictor? y=a1x1+anxn+b (y is the value of the contrast and x1…n the predictive factors)Ancova: Does the mean signal value of a group/condition 1 differ from one or more other groups/conditions when the effect of a predictive factor x is controlled?”Select Images”(Navigate through each subject directory and choose the correct con*.img by clicking it. Do NOT hit “Done” until you have selected all the individual images that willcontribute to that group analysis.GMsca: Grand Mean Scaling: <no grand mean scaling> explicitly mask images: NoGlobal Calculations: OmitA new SPM.mat is createdEstimate: (Select the newly created SPM.mat) All of the files that estimation usually creates are created for the group analysis: beta img and hdr; mask img and hdr, ResMS img and hdr, RPV img and hdr)When it runs (which should be quick) you’ll have images that show you significant activation across the group as a whole. (Although a contrast window appears at this point, it may give you trouble. Close it and then hit results, select the newly created spm.mat and follow the steps below.)Contrasts: You will still need to define a contrast: Clickanalysis, click it).Define the contrast in the contrast manager: Probably 1 will do, assuming the contrast was already defined for individuals and at this second order level you just want to see all of the data that survives the second order grou p analysis. Click “Done”.You can look at these images just as you would look at individual images of activation results: Click <Results>, navigate to the SPM.mat file of interest, click it, choose (or define) the contrast from the contrast manager, and click “Done”).Troubleshooting Images with Different CharacteristicsIf your subjects have slightly different sized images (different origin and dimensions), this problem can be fixed after the fact by coregistering the images you want to change to some target image. Here’s an example:Coregister your “wrong” smoothed images to a good one, like so:<Coregister># of subjects: 1Which option: Coregister and resliceTarget Image: swargood_em1.imgSource image: swarwrong_em1.img (This one will be registered and resliced to resemble the Target image.)Other images DoneCheck your image dimensions, origins etc. by displaying the images with SPM <display>.Troubleshooting Contrasts: If you enter the contrasts into the contrast manager in the same order for each subject, this should assure that con* files are named the same way for each subject. If you have not done this, you can display your contrast names in the contrast manager and get the correspondence of numbered con* files to the named contrasts (<Results>, navigate to and select your SPM mat file, hit done, your contrast names and their corresponding con images will be displayed).作为一个规范的原理,贝氏定理对于所有机率的解释是有效的;然而,频率主义者和贝叶斯主义者对于在应用中机率如何被赋值有着不同的看法:频率主义者根据随机事件发生的频率,或者总体样本里面的个数来赋值机率;贝叶斯主义者要根据未知的命题来赋值机率。
磁共振实验数据SPM8处理流程

磁共振实验数据SPM8处理说明一、数据准备为方便后续的数据处理,如果数据分散处理后整合,建议所有处理数据路径保持一致,要统一路径。
处理前首先要采用数据转换软件将dicom数据转换成SPM 解析格式,转换时格式请选择NIfTI,可用SPM输入面板中的DiCOM Import模块转换,也可以采用专门的转换软件,如MRIcovert。
然后进行数据预处理,预处理结束后到matlab安装目录中备份spm*.ps文件,其中包含了空间校正和标准化的信息,然后进行建模分析。
二、数据处理流程1:Slice TimingSpm8和以往版本一样,先安装matlab7.1以上版本,然后将spm8文件夹放在matlab安装目录中,我们一般放在toolbox文件夹中,然后启动matlab用菜单中设置路径命令增加spm8的路径并保存。
之后我们运行命令:spm fmri,这样将打开spm8的操作界面,我们称左上侧的窗口为按钮窗口(button window),左下侧的窗口为输入窗口(input window),右侧大窗口为树形结构窗口或图形窗口(Tree Building Window or the graphics window)。
Slice Timimg用来校正1个volume中层与层之间获取(采集)时间的差异,对事件相关设计的实验尤为重要。
我们在按钮窗口中的预处理面板中点击“Slice Timimg”,将出现如下对话框:在spm8和spm5中,每一步处理都采用了直观的“树形结构”的面板,如果一个分支项左面有“+”号,你可以双击显示子分支项,如果一个分支项右面有“<—X”号,你必须为之指定选项(否则不能运行该tree),分支项的选项在其右侧面板指定,而帮助信息则在下面的面板中显示。
如果我们处理数据没有特殊需求,我们只关心带有“<—X”项目并完成输入即可,其余均可采用默认设置。
另外注意在Tree Building Window的顶部菜单,新增了一个菜单项“TASKS”,在使用批处理分析时非常重要。
Samplitude 8 讲义

补充讲义资料:Samplitude 8.0音频制作软件Samplitude 是德国Magix公司设计生产的一款融合录音、混音、MIDI音乐制作的专业级音频工作站,现最新版本为10,该教程以8.0版本介绍。
Magix是一家专业设计制作音频、视频.图形图像的公司,音频软件还有更高级的Music Studio,适合普通使用者的Music Maker等.Samplitude通常简称为Sam。
Sam是一个庞大的软件系列,08年前共有二大系列,Sequoia是最高级的版本,具有广电级别的专业音频工作站。
由于各款之间的统一性,只要掌握其一款使用方法其他的版本也能顺利使用。
一.Samplitude 8.0 主界面布局1.打开Sam 8双击Sam图标,第一次运行弹出启动选择视窗.该视窗分两部分:启动选择、帮助。
(1)左边“启动选择”部分“新建多轨项目”按钮,可建立一个新建的虚拟专案工程; “载入多轨项目”按钮,可以打开一个已经建立的虚拟专案工程;在下面的“最近项目”部分,可以浏览并打开最近开启的专案工程。
什么是“虚拟专案工程”? “专案工程”在录音和混音领域称为一个专案工程; “虚拟”是在电脑中模拟实际的过程。
“录制波形文件”按钮,可以开始进行非建立专案工程的音频录制; “载入波形文件”可以使用波形编辑器打开硬盘中已经存在的音频文件并进行破坏性编辑处理。
(2)右边的“帮助”视窗部分可以执行各种帮助操作, “载入演示项目”按钮,可以开启Sam 8为你准备的Demo Audio 演示曲目. “打开帮助系统”按钮,可以开启Magix为你准备的几段有关的Sam 8使用方法的视频教程(如非正版Sam 8,此功能无效)。
每一次开启Sam 8,在下面的“每次一条”部分都会随机的出现一段有关的Sam 8的介绍文字.右下角按钮可以关闭启动选择视窗。
如果每一次开启Sam 8时都不需要自动弹出这个启动选择视窗,可在左下角的“总是在启动时显示”取消.如果再次想使用此视窗,可在帮助选单(菜单栏)中选择“启动向导/每一次”,就可以再次开启这个视窗。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
SPM8数据处理教程Lab 1. 数据的预处理一、概述:数据的预处理(preprocess):(1)Convert dicom files to hdr files and img images;(2)Slice Timing;(3)Realign: Estimate & Reslice;(4)Coreg: Estimate;(5)Segment;(6)两次Normalise: Write;(7)Smooth;(8)Fmri model specification;(8)Model Estimation。
二、DICOM Import:说明:转换完后会在解剖像文件名前加s,在功能像文件名前加f。
可以用“Display”来查看刚转换的图像。
(Display还有一个重要的作用是定义原点——前联合和AC-PC连线,这在做Normalise中很重要。
定义后reorient一下即可写入头信息)。
三、Slice timing1、目的:使用数学的方法使不同时间扫面的层校正为同一时间获得层。
2、过程:Date.session---[选中所有要处理的f*.文件]Number of slices---[扫描层数]TRTA---[可以用公式表示:TR-(TR/nslices)]Slice order---[扫描顺序:例如1:2:31 2:2:32分别表示从1或2开始,每间隔2个数扫描一张,即1、3、5… 2、4、6…]Reference slice---[参考层,一般选择中间的一张,对于各层扫描,一般选择即是时间的中点,又是大脑的中点。
如果是层数为偶,可以选择中点处2层中的任一层]Filename Prefix—a [默认头文件名为a]3、说明:Block Design的实验数据这一步可以跳过;event related design的实验数据必须做这一步。
四、Realign: Estimate & Reslice1、目的:如果在容许的头动范围内,可以使用一定的算法校正信号,使其靠近真实值,如果超过了这个规定的范围,则必须剔除这组数据。
2、头动范围(Check Realign):平动≤2.0mm and 旋转≤2.0degree[严老师观点]3、过程:Realign: Estimate & ResliceDate.session---[选中所有经过层间校正的文件,以af*开头] Filename Prefix—r [默认头文件名为r]其余参数默认即可。
4、matlab中如何查看头动范围打开rp_af*.txt文件,前3列为平动数据、后3列为旋转数据;命令:b=load(‘rp_af*.txt’)----[载入头动数据文件] c=max(abs(b))---[取b值的绝对值的最大值,表示找出每列的最大值]c(4:6)=c(4:6)*180/pi---[4-6列为转动,将以弧度为单位的数值转化为以角度为单位的值,pi表示π]5、说明:运行结束后将生成一对mean*文件(平均脑)、一个rp*.txt文件(头动参数文件)及若干对r*文件。
五、Coregister1、目的:是将所有的图像同一个volume对齐,对功能像与结构像做一个信息的变换。
我们相信对于被试,功能像与结构像是线性相关的平动与转动,而不是扭曲的。
由功能像向结构像去配,对于结构像中的hdr文件存有一个矩阵,而这个矩阵就包含了功能像的信息。
2、过程:Coreg Estimate:---[只需要将旋转的矩阵写入到hdr文件中,不需要生成新的文件,也就是对3D文件做一个刚体的变换,变换到功能像空间里] Reference Image---[选择头动校正生成的mean*文件]Source Image---[选择3D文件]其余项目默认即可。
3、说明:Source image与Reference image的关系,可以认为是将结构像向以mean开头的功能像里估计,估计结束后就可以将旋转矩阵写入到精度更高的3D文件当中,最后做出的图像的分辨率就会很高。
六、Segment1、目的:要将被试的结构像配到功能像里,就需要将结构像进行分割。
一般分割为灰质、白质和脑髓液三部分。
2、过程:Date---[选择配准后的3D图像]Affine Regularisation---[选择东亚人大脑模板]其余参数默认即可。
七、Normalise1、目的:将不同容积及形状的被试的大脑放到一个标准空间里,用一个公用的坐标系去描述具体的一个位置。
2、方法:A. Normalize by using EPI templates;B. Normalize by using T1 image unified segmentation3、第二种方法的过程:用3D像文件做分割,用分割的信息去做空间标准化,分割要做三个小步骤,被试既有结构像,又有功能像,我们要用结构像分割所得到的信息来做功能像的空间标准化。
首先,要保证功能像与结构像在同一个位置。
所以,需要做一次coregister,即配准。
先把被试的结构像变换到被试的功能像空间里,然后将变换到功能像里的结构像分割所得到的相应信息运用到功能像里。
结构像在功能像空间里被分割后,会得到一个矩阵。
这个矩阵就会告诉我们如何从被试的功能空间去往标准空间。
也就是mni空间。
我们可以根据这些信息应用到功能像里,写进去以后就会自动配准到标准空间里去。
Normalise: WriteData: new subjectParameter File---[参数文件,选择3D文件夹下segment后的文件,有2个文件,分别是seg_sn.mat和seg_inv_sn.mat,前者表示由功能像到标准化空间去配,而后者正相反,股选择前者。
]Image to Write---[要写入的文件,选择3D文件下的ms*.img 文件]Bounding box---[默认的偏小,可以改为-90 -126 -72 90 90 108] Voxel sizes—[改为1 1 1]其余参数默认即可。
4、再次Normalise,做完头动校正后,以平均脑文件做一次标准化。
Normalise: WriteData: new subjectParameter File---[参数文件,选择3D文件夹下segment 后的seg_sn.mat文件]Image to Write---[要写入的文件,选择头动校正后以r开头的文件]Bounding box---[默认的偏小,可以改为-90 -126 -72 90 90 108] Voxel sizes—[改为3 3 3]其余参数默认即可。
5、说明:运行完毕会生成若干对waf*打头的img/hdr文件,同时还会生成一个mean*_sn.mat文件(存放变换参数)。
八、Smooth1、过程:Image to smooth---[选择做完标准化后的以w开头的功能像文件]FWHM改为6 6 6---[半宽全高,通常取体素的2倍];其余默认。
2、说明:高斯平滑后会生成若干对swaf*打头的img/hdr文件。
Lab 2. 参数估计一、Specify 1st-lever(很容易出错奥!)Directory---[选择一个存放数据的输出目录]Timing parametersUnits for design---[如选scans,其后durations的时间按TR 的倍数计算、如选seconds,则以秒为单位计算]Interscan interval---[TR值]Microtime resolution---[通常默认为16,除非TR很长]Microtime onset---[在ER设计中,slice timing时,reference slice的扫描次序,例如reference slice为第25层,是第13个扫描的,这里就填13]Data & DesignSubject/sessionScans----[选择做完Smooth的所有文件]ConditionNameOnsets---[输入任务条件的启动向量,代表任务刺激启动的扫面数,如1:14:70,代表任务从第1个TR开始,每14个TR为一个周期,共70个TR]Durations---[ER设计填0、block设计填刺激任务的持续时间,注意前面Units for design的选择!!!]Factorial designName和levels其余默认即可。
二、EstimateSelect SPM.mat其余默认三、Results1、Select SPM.mat其余默认。
2、说明:(1)定义对比,一般0不用输,要看超过基线或control的部分,contrast选1,反之可以选-1;(2)extent threshold:范围的阈值定义多少个连在一起的有意义的体素数目才不认为有可能是噪声。
这个数值的选择一般要结合选定的P值和smooth中FWHM值来定。
(3)Overlays中选择slice查看2D激活图、选择sectors查看3D激活图,文件选择经标准化后的3D文件,以wms开头;也可选Render,在spm8工具箱中的render中的3个模板,用以查看在玻璃脑中的激活图。
(4)标化后的图像的结果还可以用MSU插件(toolbox)来获得每一个激活区的大小(体素个数)和相应的坐标以及对应的脑功能区或解剖结构。
