NTSYS 使用说明
NTSYS 使用说明

NTSYS 使用说明最近在做AFLP,因为是一切从头开始,真正体会到其中的艰辛,现在将我的一些经验给大家分亨,共勉.至于详细的步骤我就不写了,在这里只讲一下数据处理的问题.很多文献上只说将条带转换为”0”, ”1”矩阵,讲的很不具体, 其实分子标记只是将条带看作遗传位点, 它认为出现条带的为显性(AA,Aa),无条带为隐性(aa).统计时将位于同一迁移位置的条带认为是同一遗传位点.如下图所示,图中有五个泳道,A,B,C,D,E.根据条带的有无统计数据,第一个箭头处标记为,(1,0,1,0,0);第二个箭头处标记为,(1,1,1,1,1),其它统计方法一样.还有一个分析软件可用于AFLP数据分析--Cross checker.有兴趣的可以用一下,就是需要将胶板扫描为电子版,然后处理,可以自动生成01矩阵,但是也需要人工修正一下.具体使用我就不讲了.得到的01矩阵既可以用excel输入到ntsys,也可以用ntsys自带的ntedit输入.使用ntedit 的方法就不讲了,重点讲一下用excel输入数据,因为刚开始不懂就把数据输入到excel中了,可是导入时发现导不进去,于是在网上一阵狂找,终于找到了解决方法,拿出来与大家分享.(下面的大部分内容来自网络,至于从那里下的还真不知道了,我将里面的内容稍作修改,未得原作者同意就引用,莫怪!!!!!!!!!!!!!)1.先建立一个0,1构成的矩阵:在excel中,按如下规则输入数据,A1=1表示有带记为1,B1=9表示AFLP有9个带型(就是你得到的位点总数,图示有9个条带,所以为9), C1=35表示样本数(你所分析的个体数),D1=0表示无带记为0。
A1,B1,C1,D1输入的内容必须与所示一样,否则不识别.第二行表示的是样本名称。
从第三行开始的A列表示带型名称。
见下图:2. 保存时要保存为Microsoft Excel 5.0/95工作薄(*.xls)格式,一定要保存为这个格式,否则ntsys不识别的.3.假设数据保存文件名为a,打开ntsys,点击file—edit file, 打开文件a,得到一个矩阵图.将这个文件另存为nts格式.下面就可以用得到的矩阵进行遗传多样性分析并构建聚类图.下面步骤为引用打开NTSYS-PC程序,点击similarity出现如下界面:再点击Qualitative data,出现如下界面:点击input file,打开刚才生成的NTS矩阵文件,点击out file起一个文件名(假设叫111),然后点击compute按钮。
ISSR分析 NTSYS软件使用步骤

6 建树:打开 NTSYSpc 软件,在“Graphics”文件,然后点击 computer 运行,
7 上一步运行后的结果如下
带叶兜兰 长瓣兜兰 杏黄兜兰 亨利兜兰
硬叶兜兰
紫毛兜兰 文山兜兰 巨瓣兜兰 麻栗坡兜兰 同色兜兰 紫纹兜兰 天伦兜兰
2 用 ntedit 软件打开刚才的 excel 文件,将生成的文件保存成 .NTS 格式。文 件名为“11111”,
3 用 NTSYSpc 软件的 Similarty 菜单下的“genetic distance”项插入刚才保存的 “11111.NTS” 文件, 测出遗传距离 genetic distance” 并继续保存文件名为 5555, “ . 格式为.NTS (展示如下图)
用 ntedit 软件打开上一步保存的文件名为“5555”的文件,观察基因距离 genetic distance 的结果如下:
4
5 集群: 打开 NTSYSpc 软件,在“Clustering”菜单下的“SAHN”项插入刚才保存的 “5555.NTS” 文件,点击 computer 运行,并将运行后的结果保存为文件“6666” , 格式为.NTS
交给上课老师
介绍:
主要实验仪器: (包括名称、型号或者规格)
PCR 扩增后的电泳结果
(包括原始图片、数据)
图 1 1-16 号部分样品基因组 DNA 的 电 泳 图 谱
实验结果分析步骤
1
将数条带的结果输入到 excel 里面,将文件保存成 50/95 格式。
其中 1 表示有带,0 表示无带,B 字母下的 29 表示带型总数,C 字母下的 12 表示样本数。
生物科学大实验报告
成绩:
实验题目:植物 ISSR 分子标记技术的应 用(四)——ISSR—PCR 扩增结果的数据 统计和分析
NTsys-pc2.02图解使用说明

NTsys-pc2.02图解使用说明Edit by mycobio2004.111 数据的录入方法:1.1 利用Ntedit直接录入数据0、1二元数据中的数据缺失记为2。
其中列标可以写为样品编号,在No.rows 栏中写入0、1数据总数,No.cols 栏中写入样品总数。
文件另存为*.nts格式。
1.2 从excel表中直接读入数据Excel表中输入数据格式如下图。
A1必须为1,B1为0、1数据总数,C1为样品总数。
打开Ntedit程序,选择从Excel表输入,结果见上图。
文件另存为*.Nts格式1.3 Ntsys-pc可以直接运行*.phy格式的文件(由phylip和phytool产生)1.4 DNA序列数据Ntsys-PC也可以分析,但好像用的人较少。
建议大家使用phylip或者其他的软件。
DNA序列数据在Excel中输入格式如下:1.5 其他数据的Excel输入如下2 聚类分析Ntsys-pc2.02界面如下以下以图中数据为例介绍聚类过程:2.1 首先用similarity程序组中的SimQual计算形似系数矩阵。
Coefficient通常选用SM 或DICE,结果输出到另一文件。
2.2 以上步的结果作为input file利用Clustering程序组中的SHAN或者Njoin进行计算,聚类分法选用UPGMA,ties选用FIND,Maximum no. tied trees至少大于样品数。
Njoin程序组界面如下,rooting method可以选用Outgroup,但需输入外元。
2.3 将SHAN或NJoin方法得到的tree file文件输入到Graphics程序组中的tree plot程序中计算得到树图如下利用options可以对树图进行描述与处理.在此略去.2.4 一致性分析:可以用Clustering中的consens程序进行,两个不同文件分别输入;同一文件中不同的进化树之间的分析,则只输入到input tree1 file即可。
NTSYS软件进行聚类分析——UPGMA实例

NTSYS软件进行聚类分析——UPGMA实例第一部分说明文档Cluster analysis 聚合分析NTSYSpc最常见的使用是对某些相似或相异矩阵进行各种聚类分析。
以下是一个批处理例子;首先,标准化数据矩阵,其次,计算各列之间的距离系数,第三,采用单链路聚类方法,第四,计算表面值(超度量)矩阵和相关系数,第五,以散点图形式显示结果并同时输出距离矩阵。
" Standardize the variables*stand o=data.nts r=sdata.nts" Compute a distance matrix*simint o=sdata.nts r=dist.nts c=dist" Do a single-link cluster analysis of the distance matrix*sahn o=dist.nts r=tree.nts cm=single" Compute cophenetic values*coph o=tree.nts r=coph.nts" Compute the cophenetic correlation*mxcomp x=coph.nts y=dist.nts" Display phenogram*tree o=tree.nts" Display distance matrix*output o=dist.nts第二部分实例解析如果你的数据集包含量纲不一致的变量,则必须要先经过标准化处理,可以用STAND 组件完成。
如下图指明了标准化窗口。
Test.nts文件将被按行(意味着行为变量)标准化,并输出标准化文件名为teststand.nts。
如果你的变量量纲一致(如,基因序列)或者是定性数据则不需要标准化处理。
输出结果如下(5个变量的简单统计)下一步,相似或非相似矩阵数据集必须要在标准化后的数据集上构建,用来衡量各OTUS(列)两两之间的相似/非相似程度。
NTSYS软件使用详细说明

软件使用详细说明一. 数据处理方法:excel5/95格式数据1)首先得到0/1数据,输入excel中,格式如图所示:其中1表示数据格式为rectangular data matrix,12表示数据共12行(本例中表示12个个体),30表示数据共30列(本例中表示30个位点),0表示无缺失数据(若有缺失,则用1表示,缺失值可用-999或其它数字代替)。
2)格式及数据输入正确后,点击另存为excel5/95格式,命名为。
3)采用NTedit数据编辑器打开所保存的文件file>open file in a grid,在文件类型中选择excel格式,找到要分析的文件并打开,查看是否有错误,或需要修改的地方,没有问题后,保存为.nts格式。
:txt格式数据1)另一种数据处理方法,首先在excel中得到数据,如下图(注意:第一行与第一种方法不同,1表示数据格式为rectangular data matrix;12B表示共12行(本例中表示12个个体,行标签位于数据主体的开始,B表示Beginning of each row),30L表示共30列(本例中表示30个位点,L:label表示列标签),0表示无缺失)。
或者如下图格式(其中第一行为1 12L 30L 0,解释略;第二行为每行的行标签;第三行为每列的列标签;第四行起为数据主体。
):2)格式及数据都处理好之后,点文件另存为,保存为文本文件.txt格式。
3)得到txt格式文件后,即可直接用ntsys进行分析(只要格式正确,ntsys可以对txt文件进行分析,而不用再转换或保存成.nts格式)。
:直接采用NTedit进行数据的输入和保存1)对于数据量不大的数据,可以直接采用NTedit进行数据的输入,如图所示:2)数据输入好后,点击file>save file将数据保存.nts格式。
二. 计算遗传距离矩阵或相似性矩阵(distance matrix or similarity matrix)对于0/1数据和定性数据:打开ntsys软件,在similarity模块中选择simqual,input file中输入要分析的文件名称,如,计算方法中矩阵系数coefficient选择dice,output file命名输出文件名称如aflp01-dice。
如何用NTsys构建0、1数据(AFLP)进化树

NTSYS-PC使用说明NTSYS是一个聚类分析的软件,可以用来分析AFLP,RAPD等电泳带型,也可用于微生物群落多样性的相似性分析。
下面简单介绍一下其用法:1.先建立一个0,1构成的矩阵:在excel中,按如下规则输入数据,A1=1表示有带记为1,B1=535表示AFLP样本数, C1=19表示有19个带型,D1=0表示无带记为0。
第二行表示的是样本名称。
从第三行开始的A列表示带型名称。
见下图:2.选择另存为,在其中的保存类型中选择“文本文件(制表符分隔)”然后点保存,确认。
3.打开NTSYS软件点“Similarity”下拉选择”“Qualitative date”在“input file”中选择刚才保存的.txt文件,在“output file”中输入保存文件名。
“Byrows”一项不选×,“coefficient”中选择J,点compute进行运算。
4.点软件左边第二项选择“SAHN”在“input file”中选择上一步运算出来的文件在“output tree file”中输入保存文件名。
点compute进行运算。
5.选择左边第二项中的“Cophenetic Values”在“input tree file”中选择刚才计算的tree文件,输入output的文件名,点compute进行计算。
6.作Mat检测:点击左面第三项,选择“Matrix comparison plot”在“input file(1)”中选择“Qualitative date”计算出的结果,在“input file(2)”中选择“Cophenetic Valuess”计算出的结果。
点击compute进行计算,r值在0.7以上为可信。
NTSYS软件使用说明

NTSYS软件使用说明NTSYS软件使用说明1、软件简介NTSYS软件是一款专为生物学研究设计的统计分析工具。
它能处理大规模数据集,提供多种数据分析方法和可视化功能,帮助研究人员快速准确地分析数据。
2、安装与启动2.1 安装NTSYS软件在官方网站上NTSYS软件的安装包,然后按照安装向导的指示进行安装。
安装完成后,将在电脑桌面上NTSYS软件的快捷方式。
2.2 启动NTSYS软件双击NTSYS软件的快捷方式图标,即可启动NTSYS软件。
在启动过程中可能需要输入许可证信息,根据实际情况填写。
3、数据导入3.1 导入文本数据在NTSYS软件中,可以导入以文本格式保存的数据。
首先菜单栏的“文件”,然后选择“导入”-“文本文件”。
接下来,选择需要导入的文本文件,并按照指示完成导入过程。
3.2 导入Excel数据NTSYS软件也支持导入Excel数据。
在菜单栏的“文件”中选择“导入”-“Excel文件”,然后选择需要导入的Excel文件,并按照指示完成导入过程。
4、数据预处理4.1 数据过滤在NTSYS软件中,可以根据特定的条件对数据进行过滤。
菜单栏的“数据”-“过滤”,选择需要过滤的数据集和条件,并按照指示完成过滤设置。
4.2 数据清洗NTSYS软件提供了数据清洗的功能,可以删除重复数据、空值数据等。
菜单栏的“数据”-“清洗”,选择需要清洗的数据集,并按照指示完成清洗过程。
5、数据分析5.1 描述性统计NTSYS软件可以对数据进行描述性统计分析,包括均值、标准差、最大值、最小值等。
菜单栏的“统计”-“描述性统计”,选择需要分析的数据集,并按照指示完成分析过程。
5.2 方差分析NTSYS软件支持方差分析,可以分析一组或多组数据之间的差异。
菜单栏的“统计”-“方差分析”,选择需要分析的数据集和方差分析方法,并按照指示完成分析过程。
5.3 相关分析通过NTSYS软件进行相关分析,可以研究两个或多个变量之间的相关性。
ntsys-pc遗传多样性分析软件使用说明

ntsys-pc遗传多样性分析软件使用说明ntsys-pc遗传多样性分析软件使用说明一、软件简介ntsys-pc遗传多样性分析软件是一款专门用于遗传多样性研究的软件。
它提供了丰富的功能和工具,可以对遗传数据进行分析、计算和可视化展示。
本文档将详细介绍ntsys-pc软件的安装、配置和使用方法,帮助用户快速上手并充分发挥软件的优势。
二、安装和配置2.1 安装步骤a) ntsys-pc安装程序。
b) 运行安装程序,按照向导提示完成安装。
2.2 软件配置a) 运行ntsys-pc软件。
b) 确认软件配置,如存储路径、默认数据格式等。
c) 根据需要,进行个性化配置,如语言选择、主题设置等。
三、数据导入和格式转换3.1 数据导入a) 支持导入多种格式的遗传数据,如GENEPOP、FASTA、PHYLIP等。
b) 在软件界面中选择导入数据,选择相应的文件格式并加载数据。
3.2 数据格式转换a) 支持将导入的数据格式转换成其他格式,以满足不同分析需求。
b) 在软件界面中选择数据格式转换工具,设置输入和输出的数据格式以及其他参数。
四、遗传多样性分析4.1 群体遗传结构分析a) 使用多样性指数计算工具,计算群体遗传多样性指数,如He、Ho、FST等。
b) 使用主坐标分析(PCoA)工具,将群体间的遗传关系可视化。
4.2 种群遗传结构分析a) 使用聚类分析工具,根据遗传相关性将样本进行分类。
b) 使用结构分析工具,根据模型和参数对种群进行分群和成分分析。
五、结果展示和导出5.1 结果展示a) 结果以图表和表格形式展示,便于直观理解和分析。
b) 可对结果进行自定义排版和格式设置,以满足个性化需求。
5.2 结果导出a) 支持将结果导出为多种格式,如图像(PNG、JPEG)、表格(Excel、CSV)等。
b) 在软件界面中选择导出功能,设置输出格式和目标路径。
六、附件附件1:ntsys-pc安装程序附件2:样例数据文件注:本文所涉及的法律名词及注释1、版权(Copyright):指作品的创作权,即著作权。
ntsys说明书
我当时看到的说明是英文的,本文不是那个英文说明的汉语版。
我只是把我掌握的简单介绍一下。
这个软件可以分析的东西很多,我只会这么多。
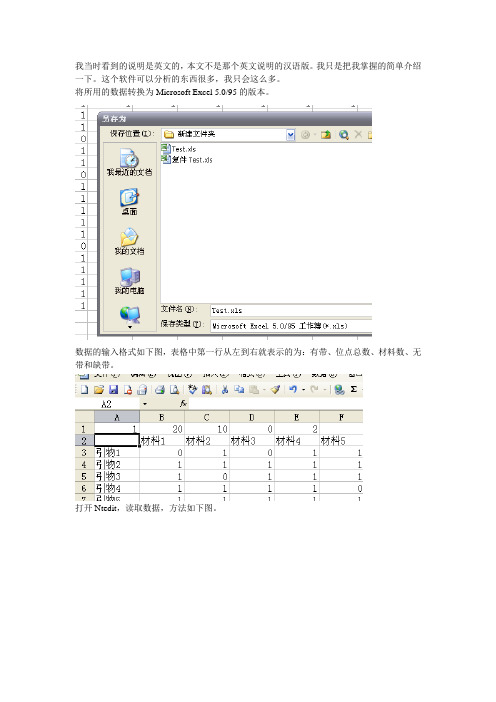
将所用的数据转换为Microsoft Excel 5.0/95的版本。
数据的输入格式如下图,表格中第一行从左到右就表示的为:有带、位点总数、材料数、无带和缺带。
打开Ntedit,读取数据,方法如下图。
数据读取后将Missing改为表示缺带的值。
如下图将数据转化为.nts的格式打开ntsys界面如下选择similarity选项下的Qualitative data。
打开刚才保存的nts格式的文件,在input file 处只需双击就可以打开如下图所示的窗口,另为output file处也是一样的。
需要注意的是操作过程中生成的新文件很多,需要命名的文件很多,因此一定要守记每一步操作生成了什么文件,给了它什么样的名字。
请大家认真看图中我给新文件的命名。
各界面中会出现不同的可选项,请大家按图中所示进行操作。
细节问题我就不再提醒了。
这上一步可以选择所要计算的相似系数种类。
一般常用的为SM系数和J系数。
选择好了以后点compute就可以了,下面的操作也是一样的。
计算过相似系数后可以如下图的操作对相似系数进行编辑打开后的界面如下图计算过相似系数就可能对各材料进行聚类分析了,先选择clusting,然后再点Shan按钮。
出现如下图的界面,在下面这个界面中可以选择聚类的种类。
一般常用的为图中所选择的,也就是默认的。
输入刚才计算的相似系数文件,会得到一个新文件(在这里我给它命名为shan J.nts)。
上一步完了之后就可以做出聚类图了,选择Graphics下面的Tree plot就可以出现下面的界面,然后把刚才得到的shan J.nts 文件输入,就可以得到聚类图了。
会出现下面两图。
输入相应的文件,并保存新生成的文件。
下面的操作为,选择Ordination栏,并选择Eigen。
同样按图中所示输入相应的文件,并保存好生成的文件。
NTsys-pc使用 进行RAPD等分子标记数据分析,需要用一些专 …

NTsys-pc使用进行RAPD等分子标记数据分析,需要用一些专用软件,国内早些时候有人用POPGENE软件,该软件特点是数据处理简单,但功能有限!目前国外常用的软件是NTSYS,该软件虽然不大,但五脏俱全,只可惜它的说明书里关于数据的处理没有详细的样板,给不太熟悉计算机的科学研究者带来了很大的麻烦!最近关于该软件发表的题目较多,但是全面系统的介绍该软件使用的未见!基于此,我呼吁咱们开设一个专题,专门来讨论这一软件!祝大家新年快乐!我目前正要研究该软件!有新的体会会与大家来分享的,呵呵!!ntsys软件使用方法集锦!ntsys软件在遗传多样性分析中有很好的用途,本帖的目的是把其应用方法集中一下,供大家使用,希望大家也能把自己知道的各种使用方法贴上来交流。
文献1:是关于数据的输入,做聚类分析。
文献2:求遗传距离(有的文献上是先求DICE系数即相似度,然后用1-DICE,即为遗传距离,这样求的结果跟文献二的结果不同,有明白的谷友吗?)文献3:做Principal Coordinate Analysis几个分子生物学分析软件NTSYS,POPGENE,WINAMOVA,AMOVAPREP、DCFA等哪位有需要的可回帖或加我QQ50012762另外,关于AMOVA的使用,在网上能搜索到的教程几乎没有,我也是摸索了一下才明白,由于AMOVAPREP的数据有限制,因此推荐张富民和葛颂一文介绍的DCFA,这个软件没有数据输入限制,对数据量大的建议使用。
但是DCFA,WINAMOVA在使用过程中都会出现奇怪的错误,对于这个主要是存在空格回车等问题,自己将文件稍做修改一下即可。
因为上传好像下载要金币是吧,这样不方便了,有些人就下不到了当然了,在这要感谢下中科院植物所的葛颂老师,因为DCFA软件我也是发了邮件向他要的,所以也很感谢他,否则也不能顺利运算!我这里也没有中文的操作,其实整个操作过程并不复杂,数据按照给的软件本身的例题输入就可以了,主要问题在于有时候运行会出错,特别是用WINAMOVA的时候会出现群体数量不够的错误,我做的时候有出现,所以我只是自己摸索到了解决的方法,就是空格的问题,如果有需要我找个时间做一个中文的上来,可是关于空格的问题不好写就是了,而且AMOVAPRE本身对数据量有限制,所以数据过多的话需要用DCFA辅助解决,就不用AMOVAPRE了关于主成份分析的一些问题一些关于遗传多样性分析的文章中常常会用到主成份分析或者主坐标分析,请问各位,主成份分析和主坐标分析有什么区别?本人用NTSYS生成主成份分析图后,发现图中不同地点或不同居群样品不能用不同的符号相区分,不知各位高人是用什么软件做主成份分析的呢?最好介绍一种在主成份分析图中能将不同居群样品用不同符号相区分的方法。
NTSYSpc使用方法
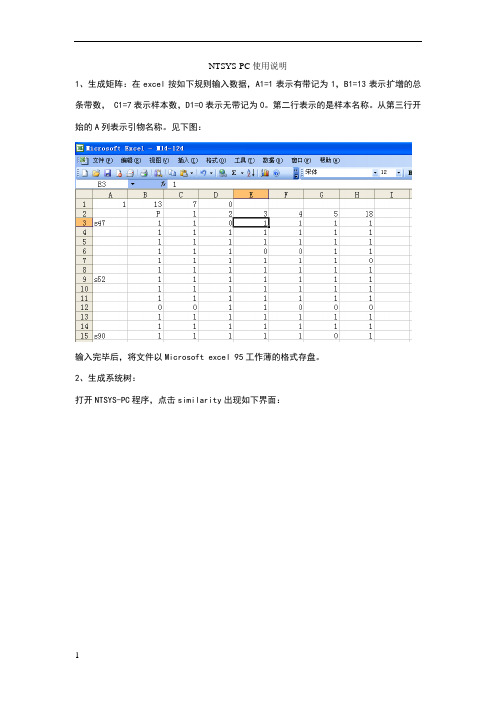
NTSYS-PC使用说明1、生成矩阵:在excel按如下规则输入数据,A1=1表示有带记为1,B1=13表示扩增的总条带数, C1=7表示样本数,D1=0表示无带记为0。
第二行表示的是样本名称。
从第三行开始的A列表示引物名称。
见下图:输入完毕后,将文件以Microsoft excel 95工作薄的格式存盘。
2、生成系统树:打开NTSYS-PC程序,点击similarity出现如下界面:再点击Qualitative data,出现下面的界面:点击input file,打开生成的excel文件,点击out file起一个文件名(假设叫A),然后点击compute按钮。
继续点击clustering,出现下面的界面,再点SHAN,出现如下界面:点击input file,打开文件刚才保存的文件111,点击out file起一个文件名(假设叫222),把In case of ties改为Find,然后点击compute按钮。
计算完成!点击input file,打开文件A,点击out file起一个文件名(假设叫B),然后点击compute 按钮。
然后点Graphics再点tree plot,出现下面的界面:点击input file,打开文件B,然后点击compute按钮,就会出现聚类图。
3、生成遗传相似度表:点File中的Edit file,打开文件A,再点compute按钮,就会得到遗传相似度表。
这个软件的功能很强大,但我也只会做聚类图和遗传相似度,如果其他朋友还会使用其它功能,请多交流。
缺失数据可用999代替,写在列的后面,软件就可以识别了TREE plotDisplays a tree matrix (such as produced by the SAHN module) or the NJOIN module. The algorithm used to convert a tree matrix into a dendrogram display is given in Rohlf (1975a). A generalization of this procedure is used to plot a tree with different heights for each OTU.The tree is displayed only a page at a time. The number of OTUs per page can be changedby either opening the plot options window or by pressing the + or - keys. The current page to be displayed can be selected either from the plot options window or by pressing the z and x keys.<Parameters> Program parameters and batch codes.2000 by Applied Biostatistics, Inc.。
NTSYS软件使用详细说明上课讲义

Ntsys2.1软件使用详细说明一. 数据处理方法1.1:excel5/95格式数据1)首先得到0/1数据,输入excel中,格式如图所示:其中1表示数据格式为rectangular data matrix,12表示数据共12行(本例中表示12个个体),30表示数据共30列(本例中表示30个位点),0表示无缺失数据(若有缺失,则用1表示,缺失值可用-999或其它数字代替)。
2)格式及数据输入正确后,点击另存为excel5/95格式,命名为aflp01.xls。
3)采用NTedit数据编辑器打开所保存的文件file>open file in a grid,在文件类型中选择excel格式,找到要分析的文件并打开,查看是否有错误,或需要修改的地方,没有问题后,保存为.nts格式。
1.2:txt格式数据1)另一种数据处理方法,首先在excel中得到数据,如下图(注意:第一行与第一种方法不同,1表示数据格式为rectangular data matrix;12B表示共12行(本例中表示12个个体,行标签位于数据主体的开始,B表示Beginning of each row),30L表示共30列(本例中表示30个位点,L:label表示列标签),0表示无缺失)。
或者如下图格式(其中第一行为1 12L 30L 0,解释略;第二行为每行的行标签;第三行为每列的列标签;第四行起为数据主体。
):2)格式及数据都处理好之后,点文件另存为,保存为文本文件.txt格式。
3)得到txt格式文件后,即可直接用ntsys进行分析(只要格式正确,ntsys可以对txt文件进行分析,而不用再转换或保存成.nts格式)。
1.3:直接采用NTedit进行数据的输入和保存1)对于数据量不大的数据,可以直接采用NTedit进行数据的输入,如图所示:2)数据输入好后,点击file>save file将数据保存.nts格式。
二. 计算遗传距离矩阵或相似性矩阵(distance matrix or similarity matrix)对于0/1数据和定性数据:打开ntsys软件,在similarity模块中选择simqual,input file 中输入要分析的文件名称,如aflp01.nts,计算方法中矩阵系数coefficient选择dice,output file 命名输出文件名称如aflp01-dice。
ntsys使用说明

ntsys使用说明NTSYS-PC使用说明1、生成矩阵:在excel按如下规则输入数据,A1=1表示有带记为1,B1=13表示扩增的总条带数,C1=7表示样本数,D1=0表示无带记为0。
第二行表示的是样本名称。
从第三行开始的A列表示引物名称。
见下图:输入完毕后,将文件以Microsoft excel 5.0/95工作薄的格式存盘。
2、生成系统树:打开NTSYS-PC程序,点击similarity出现如下界面:再点击Qualitative data,出现下面的界面:点击input file,打开生成的excel文件,点击out file起一个文件名(假设叫A),然后点击compute按钮。
继续点击clustering,出现下面的界面,再点SHAN,出现如下界面:点击input file,打开文件刚才保存的文件111,点击out file起一个文件名(假设叫222),把In case of ties改为Find,然后点击compute按钮。
计算完成!点击input file,打开文件A,点击out file起一个文件名(假设叫B),然后点击compute 按钮。
然后点Graphics再点tree plot,出现下面的界面:点击input file,打开文件B,然后点击compute按钮,就会出现聚类图。
3、生成遗传相似度表:点File中的Edit file,打开文件A,再点compute按钮,就会得到遗传相似度表。
这个软件的功能很强大,但我也只会做聚类图和遗传相似度,如果其他朋友还会使用其它功能,请多交流。
缺失数据可用999代替,写在列的后面,软件就可以识别了TREE plotDisplays a tree matrix (such as produced by the SAHN module) or the NJOIN module. The algorithm used to convert a tree matrix into a dendrogram display is given in Rohlf (1975a).A generalization of this procedure is used to plot a tree with different heights for each OTU.The tree is displayed only a page at a time. The number of OTUs per page can be changed by either opening the plot options window or by pressing the + or - keys. The current page to be displayed can be selected either from the plot options window or by pressing the z and x keys.Program parameters and batch codes.2000 by Applied Biostatistics, Inc.在clustering中单击SAHN后,右侧的对话框中有一个选项:Maximum no. tied trees,在这个选项中输入大于等于样本数。
[精华]ntsys矩阵的制作
![[精华]ntsys矩阵的制作](https://img.taocdn.com/s3/m/83e892ceff00bed5b8f31d0d.png)
[精华]ntsys 矩阵的制作1 矩阵的制作操作:在Excel里,按照NTsys的要求面做好矩阵。
第1行第1个数字一般填1,代表要处理的数据为数字矩阵。
第1行第2个数字填行数,第3个数字填列数。
第1行第4个数字,如果有缺失值填1,没有缺失值填0.当然最好不要有缺失值。
第2行按照顺序填写每一列的名称。
第2行的定格要空出来。
第3行开始才是正式的数据矩阵。
从第3行开始,第1列作为要操作的单元名称。
数据准备好后,一般要存为Excel97格式。
再用ntedit打开,查看是否有错误,如果没有错误,建议另存为“.nts”格式的文件,如data.nts.2 矩阵的标准化,数据标准化的是为了便于数据的横向比较。
操作:采用左侧output&Transf. 栏下的Standardization模块. 一般是将数据减去算术平均值,并除以标准差,转换为N(0,1)分布。
3 计算距离矩阵操作:采用左侧Similarity 栏下的 Interval data模块(又称simint),默认为平均分类距离(Average taxonomic distance)。
其公式可以参考帮助文件。
当然,有多种距离可供选择:如: Bray-Curtis distance.DIST Average taxonomic distance.DISTSQ Squared average distances.EUCLID Euclidean distances.EUCLIDSQ Euclidean distances squared. MANHAT Average Manhattan distances (city block). PSHAPE Penrose's shape coefficient. PSIZE Penrose's size coefficient.CORR Pearson product-moment correlation. 4 依据第3步计算出的距离矩阵,进行聚类分析,计算聚类树矩阵。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
NTSYS 使用说明
2007-12-16 10:52
标签:桌面样本 yspc ntsy 分析分类:流水账
/200712/354/LZkriVFb.exe
/200712/354/LckriVFb.id
如何用Ntsyspc2.1 计算遗传距离(Genetic Distance)?
可以用NTsys-pc2.1来鉴定亲缘关系的远近
1.假设你的数据文件名为A(Excel 文件),存放于C:/桌面
2.打开Ntsyspc2.1,click "File", click "Edit file",打开文件A
3.Edit 完毕之后,click "Save file as",将文件存放于桌面,命名为A-1(NTS 文件)
4.click Ntsyspc2.1 面板上的"Similarity",click "Genetic distance"
5.双击Input data file 右边的空白,打开A-1 文件
6.双击Output file 右边的空白,将文件存放于桌面,命名为A-2 (NTS 文件)
7.click Ntsyspc2.1 面板上的"Compute"按钮
8.OK,你的遗传距离就存放在文件A-2 里了
NTSYS-PC使用说明
NTSYS是一个聚类分析的软件,可以用来分析RFLP,RAPD等电泳带型,也可用于微生物群落多样性的相似性分析。
下面简单介绍一下其用法:
1.先建立一个0,1构成的矩阵:在excel中,按如下规则输入数据,A1=1表示有带记为1,B1=9表示RFLP有9个带型, C1=35表示样本数,D1=0表示无带记为0。
第二行表示的是样本名称。
从第三行开始的A列表示带型名称。
见下图:
2. 打开NTSYS-PC程序,点击similarity出现如下界面:
再点击Qualitative data,出现如下界面:
点击input file,打开刚才生成的excel矩阵文件,点击out file起一个文件名(假设叫111),然后点击compute按钮。
文件111是一个相似度表。
3. 继续点击clustering,出现如下界面:
再点SHAN,出现如下界面:
点击input file,打开文件刚才保存的文件111,点击out file起一个文件名(假设叫222),把In case of ties改为Find,然后点击compute按钮。
计算完成!
4. 然后点Graphics, 再点tree plot,出现下面的界面:
点击input file,打开文件222,然后点击compute按钮,就会出现聚类图。
OK了!
Ntsys-pc软件进行聚类分析
1 数据的录入方法:
1.1 利用Ntedit直接录入数据
0、1二元数据中的数据缺失记为2。
其中列标可以写为样品编号,在No.rows 栏中写入0、1数据总数,No.cols 栏中写入样品总数。
文件另存为*.nts格式。
1.2 从excel表中直接读入数据
Excel表中输入数据格式如下图。
A1必须为1,B1为0、1数据总数,C1为样品总数。
打开Ntedit程序,选择从Excel表输入,结果见上图。
文件另存为*.Nts格式
1.3 Ntsys-pc可以直接运行*.phy格式的文件(由phylip和phytool产生)
1.4 DNA序列数据Ntsys-PC也可以分析,但好像用的人较少。
建议大家使用phylip或者其他的软件。
DNA 序列数据在Excel中输入格式如下:
1.5 其他数据的Excel输入如下
2 聚类分析
Ntsys-pc2.02界面如下
以下以图中数据为例介绍聚类过程:
2.1 首先用similarity程序组中的SimQual计算形似系数矩阵。
Coefficient通常选用SM 或DICE,结果输出到另一文件。
2.2 以上步的结果作为input file利用Clustering程序组中的SHAN或者Njoin进行计算,聚类分法选用UPGMA,ties选用FIND,Maximum no. tied trees至少大于样品数。
Njoin程序组界面如下,rooting method可以选用Outgroup,但需输入外元。
2.3 将SHAN或NJoin方法得到的tree file文件输入到Graphics程序组中的tree plot程序中计算
得到树图如下
利用options可以对树图进行描述与处理.在此略去.
2.4 一致性分析:
可以用Clustering中的consens程序进行,两个不同文件分别输入;同一文件中不同的进化树之间的分析,则只输入到input tree1 file即可。
通常多选用MAJRUL方法
(原文地址:/blog/qingyong3366/18505258)。
