mega软件延时程序
mega的使用

mega的使⽤MEGA的使⽤产⽣背景及简介随着不同物种基因组测序的快速发展,产⽣了⼤量的DNA序列信息,这时就需要⼀种简便⽽快速的统计分析⼯具来对这些数据进⾏有效的分析,以提取其中包含的⼤量信息。
MEGA就是基于这种需求开发的。
MEGA 软件的⽬的就是提供⼀个以进化的⾓度从DNA和蛋⽩序列中提取有⽤的信息的⼯具,并且,此软件可以免费下载使⽤。
现在我们使⽤的是MEGA4的版本。
它主要集中于进化分析获得的综合的序列信息。
使⽤它我们可以编辑序列数据、序列⽐对、构建系统发育树、推测物种间的进化距离等。
此软件的输出结果资源管理器允许⽤户浏览、编辑、打印输⼊所得到的结果⽽且所得到的结果具有不同形式的可视化效果。
此外,该软件还能够得出不同序列间的距离矩阵,这是他不同与其他分析软件的地⽅。
在计算矩阵⽅⾯有⼀些⾃⼰的特点:1.推测序列或者物种间的进化距离2.根据MCL(Maximum Composite Likeliood method)的⽅法构建系统发育树3.考虑到了不同碱基替换的不同的⽐率,考虑到了碱基转换和颠换的差别。
4.随时可以使⽤标注:所以的结果输⼊都可以使⽤标注,⽽且标注的内容可以被保存,复制。
具体使⽤我们以分析20个物种的⾎红蛋⽩为例来具体说明此软件的具体使⽤情况。
⼀.启动程序1.运⾏环境:在Windows 95/98, NT, ME, 2000, XP, vista等操作系统下均可使⽤。
2.下载安装:可以直接登陆/doc/d5e9bde30722192e4436f6c6.html 进⾏下载安装,另外还可以从/doc/d5e9bde30722192e4436f6c6.html /tools/phylogeny.php中的链接进去。
3.双击桌⾯快捷⽅式图标,进⼊主界⾯;或者从开始菜单,单击图标启动。
⼆.序列分析。
1.启动单击后,会出现如下界⾯:这⾥有三个选项,分别对应三种不同的情况:以下分别予以介绍:Create a new alignment :是在你没有任何⽐对的时候使⽤,⽐如你只有⼀个fasta 格式的序列就可以选择这个选项。
MEGA使用说明书

MEGA软件构建系统发育树摘要:以白色念珠菌属下面的十个种的18s RNA 为例,构建系统发育树来说明MEGA 软件的使用方法。
1背景简介1.1 MEGA(分子进化遗传分析)MEGA 的全称是Molecular Evolutionary Genetics Analysis。
MEGA is an integrated tool for automatic and manual sequence alignment, inferring phylogenetic trees, mining web-based databases, estimating rates of molecular evolution, and testing evolutionary hypotheses. MEGA 可用于序列比对、进化树的推断、估计分子进化速度、验证进化假说等。
MEGA 还可以通过网络(NCBI)进行序列的比对和数据的搜索。
最新版本:MEGA 5.1 Beta (软件开发者建议其结果不用于发表文章)建议下载版本:MEGA 5.05 for Windows and Mac OS。
MEGA 5 has been tested on the following Microsoft Windows® operating systems: Windows 95/98, NT, 2000, XP, Vista, version 7, Linux and Mac OS [1].MEGA 5.05 可免费下载,只需输入名字及有效邮箱,下载链接会发送至邮箱,点击可下载。
1.2 系统发育树定义系统发育树(英文:Phylogenetic tree)又称为演化树(evolutionary tree),是表明被认为具有共同祖先的各物种间演化关系的树。
是一种亲缘分支分类方法(cladogram)。
在树中,每个节点代表其各分支的最近共同祖先,而节点间的线段长度对应演化距离(如估计的演化时间)1.3 系统发育树的分类根据有根和无根来区分:树可分为有根树和无根树两类。
MEGA软件的使用

MEGA软件的使用Mega是一款操作十分简便的遗传学分析软件,其界面十分友好,即使初学者也很易上手。
1、数据的录入及编辑Mega软件能够接受多种数据格式,如FASTA格式、Phylip格式、PAUP数据格式等等。
而且Mega软件专门提供了把其他格式的数据转换位Mega数据格式的程序。
首先,打开Mega程序,有如下图所示的操作界面:单击工具栏中的“File”按钮,会出现如下图所示的菜单:从上图可以看出,下拉菜单有“Open Data”(打开数据)、“Reopen Data”(打开曾经打开的数据,一般会保留新近打开的几个数据)、“Close Data”(关闭数据)、“Export Data”(导出数据)、“Conver To MEGA Format”(将数据转化为MEGA格式)、“Text Editor”(数据文本编辑)、“Printer Setup”(启动打印)、“Exit”(退出MEGA程序)。
单击“Open Data”选项,会弹出如下菜单:浏览文件,选择要分析的数据打开,单击“打开”按钮,会弹出如下操作界面:此程序操作界面,提供了三种选择数据选择:Nucleotide Sequences(核苷酸序列)、Protein Sequences(蛋白质序列)、Pairwise Distance(遗传距离矩阵)。
根据输入数据的类型,选择一种,点击“OK”即可。
如果选择“Pairwise Distance”,则操作界面有所不同;如下图所示:根据遗传距离矩阵的类型,如果是下三角矩阵,选择“Lower Left Matrix”即可;如果是上三角矩阵,选择“Upper Right Matrix”即可。
点击“OK”按钮,即可导入数据。
如果是核苷酸数据,则读完之后,会弹出如下对话框:如上图,如果是编码蛋白质的核苷酸序列,则选择“Yes”按钮;如果是不编码蛋白质的核苷酸序列,则点击“No”按钮。
之后,会弹出如下操作窗口:此作界面的名称是“Sequence Data Explorer”,在其最上方是工具栏“Data”、“Display”、“Highlight”等,然后是一些数据处理方式的快捷按钮,在操作界面的左下方是每个序列的名称。
用ICCAVR6编写的mega1max504程序
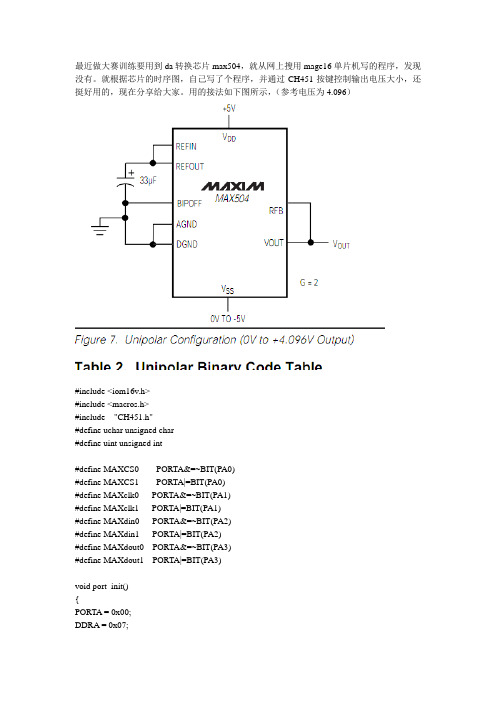
最近做大赛训练要用到da转换芯片max504,就从网上搜用mage16单片机写的程序,发现没有。
就根据芯片的时序图,自己写了个程序,并通过CH451按键控制输出电压大小,还挺好用的,现在分享给大家。
用的接法如下图所示,(参考电压为4.096)#include <iom16v.h>#include <macros.h>#include "CH451.h"#define uchar unsigned char#define uint unsigned int#define MAXCS0 PORTA&=~BIT(PA0)#define MAXCS1 PORTA|=BIT(PA0)#define MAXclk0 PORTA&=~BIT(PA1)#define MAXclk1 PORTA|=BIT(PA1)#define MAXdin0 PORTA&=~BIT(PA2)#define MAXdin1 PORTA|=BIT(PA2)#define MAXdout0 PORTA&=~BIT(PA3)#define MAXdout1 PORTA|=BIT(PA3)void port_init(){PORTA = 0x00;DDRA = 0x07;PORTB = 0x07;DDRB = 0x07;PORTC = 0x00;DDRC = 0x00;PORTD = 0x00;DDRD = 0x00;}void delay(uint a){uint x,y;for(x=0;x<1140;x++){for(y=0;y<a;y++);}}void max504_write(uint data){uchar i;MAXCS1;MAXclk1 ;MAXclk0;MAXCS0 ;for(i=0;i<16;i++){if(data&0X8000) //先发送4位哑位,msb-lsb,0,0 {MAXdin1;}else{MAXdin0;}data<<=1;MAXclk1 ;MAXclk0 ;}NOP();MAXCS1;NOP();}void main(){uint da;uchar key;port_init();ch451_init();ch451_write(CH451_RESET); //ch451初始化ch451_write(CH451_SYSON2);ch451_write(CH451_BCD);LightenAllLed(); //全亮一秒后灭,以检验ch451能否正常工作delay(1000);ClearLed();max504_write(0); //启动max504,可有可无NOP();NOP();max504_write(0x07d0);while(1){key=ch451_read();if(key!=0){switch(key){case 0x45: //通过按键控制产生不同的电压值da=0x0000;break;case 0x4d:da=0x007c;break;case 0x55:da=0x00fc;break;case 0x5d:da=0x0178;break;case 0x65 :da=0x01f4;break;case 0x4e :da=0x03e8;break;case 0x56 :da=0x05dc;break;case 0x5e :da=0x07d0;break;case 0x66 :da=0x01f4;break;case 0x6e :da=0x01f4;break;case 0x76 :da=0x01f4;break;}max504_write(da);}key=0;}}Ch451头文件,编译时只要将其包含在headers就行了。
MEGA4的中文使用说明

MEGA4的中文使用说明产生背景及简介随着不同物种基因组测序的快速发展,产生了大量的DNA 序列信息,这时就需要一种简便而快速的统计分析工具来对这些数据进行有效的分析,以提取其中包含的大量信息。
MEGA 就是基于这种需求开发的。
MEGA 软件的目的就是提供一个以进化的角度从DNA 和蛋白序列中提取有用的信息的工具,并且,此软件可以免费下载使用。
现在我们使用的是 MEGA4 的版本。
它主要集中于进化分析获得的综合的序列信息。
使用它我们可以编辑序列数据、序列比对、构建系统发育树、推测物种间的进化距离等。
此软件的输出结果资源管理器允许用户浏览、编辑、打印输入所得到的结果而且所得到的结果具有不同形式的可视化效果。
此外,该软件还能够得出不同序列间的距离矩阵,这是他不同与其他分析软件的地方。
在计算矩阵方面有一些自己的特点:1. 推测序列或者物种间的进化距离2. 根据MCL(Maximum Composite Likeliood method)的方法构建系统发育树3. 考虑到了不同碱基替换的不同的比率,考虑到了碱基转换和颠换的差别。
4. 随时可以使用标注:所以的结果输入都可以使用标注,而且标注的内容可以被保存,复制。
具体使用我们以分析 20 个物种的血红蛋白为例来具体说明此软件的具体使用情况。
启动程序1. 运行环境:在Windows 95/98, NT, ME, 2000, XP, vista 等操作系统下均可使用。
2. 下载安装:可以直接登陆 进行下载安装,另外还可以从/tools/phylogeny.php 中的链接进去。
3. 双击桌面快捷方式图标, 进入主界面;或者从开始菜单,单击图标启动。
序列分析1.启动单击后,会出现如下界面:这里有三个选项,分别对应三种不同的情况:以下分别予以介绍:Create a new alignment :是在你没有任何比对的时候使用,比如你只有一个fasta 格式的序列就可以选择这个选项。
XMEGA初步使用

//
//
} //=========================================================向、定时器、外部中断、AD 转换的初始化函数,很重要的哦 //例子能正确运行全靠它了============================================ void HardwareInit(void) { PORTA_DIR = 0x00;//PA 口做 AD 输入,所以要设置为输入口啦 PORTA_OUT = 0x00; PORTB_DIR=0x00;//PB 口也设置成输入 PORTB_OUT=0x00; PORTC_DIR=0xFF;//PC 口设置成输出 PORTC_OUT=0x00;//输出 0 PORTD_DIR=(PD3TXD) ; //USART 的输出和输入设置 PORTD_OUT&= ~(PD2RXD); PORTE_DIR=PE3OUT;//PE3 为输出,其它为输入 PORTE_OUT=0x00; PORTF_DIR=0x00;//PF 为输入 PORTF_OUT=PF1KEY; //==time/count C 0 初始化为 PWM ================================= TCC0_CTRLA=0x01;//DIV 0 TCC0_CTRLB=0xF3;//PWM TCC0_CTRLC=0x00; TCC0_CTRLD=0x00; TCC0_CTRLE=0x01;//8 bit mode //============================================= //ADC 输入初始化=========================================== //注意 ADC 可以同时转化 4 路信号,这里只用到 1 路 ADCA_CTRLA=0x01;//enble ADC input ADCA_CTRLB=0x04;//8bit //ADCA_REFCTRL=0x00;//1.0V ADCA_REFCTRL=0x01;//Vcc/1.6 ADCA_PRESCALER=0x02;//DIV by 4 ADCA_CH0_CTRL=0x01;//singal ended//只用到这一路 ADCA_CH1_CTRL=0x01;//singal ended//这路 ADC 没用 ADCA_CH2_CTRL=0x01;//singal ended//这路 ADC 没用 ADCA_CH3_CTRL=0x01;//singal ended//这路 ADC 没用 //=================================================*/ //interrupt system=======================================
MEGA使用说明书

MEGA使用说明书MEGA是一款提供云存储服务的应用,它可以帮助用户上传、存储和共享文件。
本文将介绍如何在不同平台上使用MEGA。
第一步:注册帐号在使用MEGA之前,首先需要注册一个帐号。
用户可以通过MEGA官网或者应用程序进行注册。
注册流程1.打开MEGA官网或者应用程序;2.点击“注册”;3.输入电子邮件地址和密码;4.通过电子邮件验证注册帐号。
注意事项1.MEGA仅支持使用电子邮件进行注册,不支持使用手机号注册;2.为了保障帐号的安全,建议使用不易被猜测的密码,并开启双重身份验证。
第二步:下载MEGA应用在注册完帐号后,用户可以下载MEGA应用。
目前MEGA提供了桌面客户端和移动端应用。
用户可以根据自己的需求选择下载。
下载流程下面将介绍如何在不同平台上下载MEGA应用。
Windows1.打开MEGA官网;2.点击“立即下载”;3.在弹出的下载页面中,选择Windows版本;4.下载完后,双击MEGA安装程序,按照提示完成安装。
macOS1.打开MEGA官网;2.点击“立即下载”;3.在弹出的下载页面中,选择macOS版本;4.下载完后,双击DMG文件,将MEGA拖动到应用程序中。
Android1.打开Google Play商店;2.搜索“MEGA”应用;3.点击“安装”;4.下载、安装完成后,打开应用程序。
iOS1.打开App Store;2.搜索“MEGA”应用;3.点击“获取”,输入指纹或密码;4.下载、安装完成后,打开应用程序。
注意事项1.在下载安装应用程序时,需要保障网络通畅,同时保障设备空间充足;2.应用程序可能会因地区、设备、系统等因素而有所不同。
用户在下载安装时需要根据自己的实际情况进行选择。
第三步:上传文件在安装完成MEGA应用后,用户可以开始使用了。
下面将介绍如何上传文件。
上传流程1.打开MEGA应用程序;2.点击“上传”按钮;3.选择要上传的文件;4.点击“上传”。
注意事项1.上传的文件格式应符合MEGA规定,不支持上传非法内容;2.上传的文件大小应符合MEGA规定,如果超过限制,建议将文件分割成较小的部分。
MEGA软件的使用

MEGA软件的使用Mega是一款操作十分简便的遗传学分析软件,其界面十分友好,即使初学者也很易上手。
1、数据的录入及编辑Mega软件能够接受多种数据格式,如FASTA格式、Phylip格式、PAUP数据格式等等。
而且Mega软件专门提供了把其他格式的数据转换位Mega数据格式的程序。
首先,打开Mega程序,有如下图所示的操作界面:单击工具栏中的“File”按钮,会出现如下图所示的菜单:从上图可以看出,下拉菜单有“Open Data”(打开数据)、“Reopen Data”(打开曾经打开的数据,一般会保留新近打开的几个数据)、“Close Data”(关闭数据)、“Export Data”(导出数据)、“Conver To MEGA Format”(将数据转化为MEGA格式)、“Text Editor”(数据文本编辑)、“Printer Setup”(启动打印)、“Exit”(退出MEGA程序)。
单击“Open Data”选项,会弹出如下菜单:浏览文件,选择要分析的数据打开,单击“打开”按钮,会弹出如下操作界面:此程序操作界面,提供了三种选择数据选择:Nucleotide Sequences(核苷酸序列)、Protein Sequences(蛋白质序列)、Pairwise Distance(遗传距离矩阵)。
根据输入数据的类型,选择一种,点击“OK”即可。
如果选择“Pairwise Distance”,则操作界面有所不同;如下图所示:根据遗传距离矩阵的类型,如果是下三角矩阵,选择“Lower Left Matrix”即可;如果是上三角矩阵,选择“Upper Right Matrix”即可。
点击“OK”按钮,即可导入数据。
如果是核苷酸数据,则读完之后,会弹出如下对话框:如上图,如果是编码蛋白质的核苷酸序列,则选择“Yes”按钮;如果是不编码蛋白质的核苷酸序列,则点击“No”按钮。
之后,会弹出如下操作窗口:此作界面的名称是“Sequence Data Explorer”,在其最上方是工具栏“Data”、“Display”、“Highlight”等,然后是一些数据处理方式的快捷按钮,在操作界面的左下方是每个序列的名称。
系统发育树mega使用流程

系统发育树mega使用流程下载温馨提示:该文档是我店铺精心编制而成,希望大家下载以后,能够帮助大家解决实际的问题。
文档下载后可定制随意修改,请根据实际需要进行相应的调整和使用,谢谢!并且,本店铺为大家提供各种各样类型的实用资料,如教育随笔、日记赏析、句子摘抄、古诗大全、经典美文、话题作文、工作总结、词语解析、文案摘录、其他资料等等,如想了解不同资料格式和写法,敬请关注!Download tips: This document is carefully compiled by theeditor. I hope that after you download them,they can help yousolve practical problems. The document can be customized andmodified after downloading,please adjust and use it according toactual needs, thank you!In addition, our shop provides you with various types ofpractical materials,such as educational essays, diaryappreciation,sentence excerpts,ancient poems,classic articles,topic composition,work summary,word parsing,copy excerpts,other materials and so on,want to know different data formats andwriting methods,please pay attention!1. 数据准备:收集要分析的序列数据,可以是 DNA、RNA 或蛋白质序列。
怎样使用MEGA建立进化树

怎样使用MEGA建立进化树在进行生物信息学研究中,建立进化树是一项非常重要的任务。
MEGA (分子进化遗传学分析)是一款常用的软件,专门用于进行进化树和多序列分析。
下面将详细介绍如何使用MEGA建立进化树。
安装完成后,打开MEGA软件。
在MEGA的主界面上,有几个常用的功能选项,包括「File」、「Edit」、「View」、「Tools」、「Align」、「Phylogeny」和「Help」。
我们主要关注「Phylogeny」(进化树)选项。
在新窗口中,我们需要选择构建进化树的方法。
MEGA支持多种构建进化树的方法,包括Neighbor Joining、Maximum Parsimony、Maximum Likelihood和Bayesian等。
在这里,我们以Neighbor Joining方法为例进行演示。
在Neighbor Joining方法中,我们需要先选择计算进化距离的方法。
MEGA支持许多计算进化距离的方法,如P-distance、Kimura 2-parameter、Tamura 3-parameter等。
在这里,我们选择P-distance方法。
在选择了计算进化距离的方法后,我们还需要选择树的标准。
MEGA支持Bootstrap(Bootstrap方法是统计学中一种用于评估统计性信号和树的可靠性的方法)和Nearest-Neighbor Interchange等标准。
在这里,我们选择Bootstrap标准。
在选择了进化距离的方法和树的标准后,我们需要选择输入序列数据的文件格式。
MEGA支持多种格式的序列文件,如FASTA、PHYLIP和MEGA 等。
选择相应的格式后,我们需要导入序列数据。
可以通过从文件中导入或从剪贴板中粘贴来导入序列数据。
MEGA是一款非常强大的进化树分析软件,但对于初学者来说,可能需要一些时间去了解其中的各种选项和功能。
因此,建议在使用MEGA之前,先阅读相关文档和教程,以便更好地使用MEGA进行进化树的构建和分析。
mega的使用

MEGA的使用产生背景及简介随着不同物种基因组测序的快速发展,产生了大量的DNA序列信息,这时就需要一种简便而快速的统计分析工具来对这些数据进行有效的分析,以提取其中包含的大量信息。
MEGA就是基于这种需求开发的。
MEGA 软件的目的就是提供一个以进化的角度从DNA和蛋白序列中提取有用的信息的工具,并且,此软件可以免费下载使用。
现在我们使用的是MEGA4的版本。
它主要集中于进化分析获得的综合的序列信息。
使用它我们可以编辑序列数据、序列比对、构建系统发育树、推测物种间的进化距离等。
此软件的输出结果资源管理器允许用户浏览、编辑、打印输入所得到的结果而且所得到的结果具有不同形式的可视化效果。
此外,该软件还能够得出不同序列间的距离矩阵,这是他不同与其他分析软件的地方。
在计算矩阵方面有一些自己的特点:1.推测序列或者物种间的进化距离2.根据MCL(Maximum Composite Likeliood method)的方法构建系统发育树3.考虑到了不同碱基替换的不同的比率,考虑到了碱基转换和颠换的差别。
4.随时可以使用标注:所以的结果输入都可以使用标注,而且标注的内容可以被保存,复制。
具体使用我们以分析20个物种的血红蛋白为例来具体说明此软件的具体使用情况。
一.启动程序1.运行环境:在Windows 95/98, NT, ME, 2000, XP, vista等操作系统下均可使用。
2.下载安装:可以直接登陆进行下载安装,另外还可以从/tools/phylogeny.php中的链接进去。
3.双击桌面快捷方式图标,进入主界面;或者从开始菜单,单击图标启动。
二.序列分析。
1.启动单击后,会出现如下界面:这里有三个选项,分别对应三种不同的情况:以下分别予以介绍:Create a new alignment :是在你没有任何比对的时候使用,比如你只有一个fasta格式的序列就可以选择这个选项。
Open a saved alignment session:使用它可以打开一个我们已经比对好的序列文件;Retieve a sequence from a file :这种情况同第一种情况相似,只是不用选择是DNA 还是蛋白质序列比对,选择的也是fasta格式的文件,打开后的界面都是一样的。
mega328的例子程序

mega328的例子程序标题:Mega328微控制器的例子程序1. 介绍Mega328微控制器Mega328微控制器是一款功能强大的单片机芯片,广泛应用于各种电子设备中。
它具有高性能、低功耗和丰富的接口资源,适用于各种应用场景。
2. 简要介绍Mega328的基本特性Mega328微控制器具有32KB的闪存、2KB的SRAM和1KB的EEPROM。
它支持多种通信接口,包括UART、SPI和I2C,并且拥有23个可编程IO引脚。
3. 使用Mega328实现LED闪烁通过编写简单的程序,可以利用Mega328的GPIO功能实现LED的闪烁。
我们可以通过设置引脚的输入输出状态和延时来控制LED的亮灭,从而实现闪烁效果。
4. 使用Mega328实现按键控制LED开关除了控制LED的闪烁,Mega328还可以通过外部按键来控制LED 的开关。
通过读取按键的状态,我们可以判断用户是否按下按键,然后根据按键状态来控制LED的亮灭。
5. 使用Mega328实现ADC功能Mega328还具有模数转换器(ADC)功能,可以将模拟信号转换为数字信号。
通过编写程序,我们可以设置ADC的输入引脚和分辨率,并读取模拟信号的数值。
6. 使用Mega328实现PWM输出Mega328支持脉冲宽度调制(PWM)输出,可以用来控制电机的转速、LED的亮度等。
通过设置PWM的频率和占空比,我们可以实现精确的输出控制。
7. 使用Mega328实现定时器功能Mega328内置了多个定时器,可以用来实现精确的定时功能。
通过编写程序,我们可以设置定时器的计数值和工作模式,从而实现各种定时任务。
8. 使用Mega328实现串口通信Mega328支持串口通信,可以与其他设备进行数据交换。
通过编写程序,我们可以设置串口的波特率、数据位数、停止位数等参数,实现可靠的数据传输。
9. 使用Mega328实现温度传感器的读取Mega328可以与温度传感器进行连接,通过读取传感器的输出信号来获取当前温度。
mega16程序典例

}
}
void delay_nms(unsigned int n)
{
unsigned int i;
for(i=0;i<n;i++)
delay_1ms();
}
/************************循迹程序*********************************************/
void white(void);//盲区循迹辅助函数
void led(char);//一般循迹程序,输入行走格数
void led1(char);//一般循迹程序,输入行走格数
void led2(char);//后退循迹,输入行走格数
void turnleft(void);//左转
void turnright(void);//右转
case 0X02:PORTA=0X11;break;
case 0X0C:PORTA=0X24;break;//右偏
case 0X08:PORTA=0X24;break;
case 0X04:PORTA=0X24;break;
}
}
else if(key>=0X20)
{
PORTA=0X30;
delay_nms(10);
PORTA&=~BIT(PA0);
ADMUX=0X27;//使用外部参考电压,右对齐,ADC0选择7通道
ADCSR=0X80;//控制寄存器,adc使能,取消中断,2分频
ADCSR|=BIT(6);// ADCR代表数字6,启动ADE
while(!(ADCSR&(BIT(4))));//ADIF对应4,将第四位置位,其余为0
MEGA 5.05使用教程

MEGA常用工Biblioteka 及使用方法超通俗版本将拼好后的序 列和下载的序 列填在一个txt 文档中
保存后,将文件后缀由 .txt 改成 .fas
打开MEGA 5.05软件,点击菜单栏File,选择open a file,打开刚才的 .fas文件。弹出窗口后选择Align。弹出副窗口。
关于系统 发育树
点击主窗口中Phylogeny下选择想用的算法(最大似然法ML或者是邻 接法NJ),一般选用ML。
各参数设置如图,点击 Computr进行计算,构建 出一个系统发育树。
左侧工具栏可对 树的拓扑结构进 行调整。点击保 存,将文件保存 成mts格式(仅 可以用MEGA打 开)。也可以点 击上方工具栏中 的Image,save as 为其他格式 。若想更改字体 ,可将文件保存 成PDF格式后, 用Adobe Illustrator CS5 对其进行修改。
点击TA图标,可弹出小窗口,由次窗口可知简约信息位点,不变位点 等信息。点击工具栏statistics,选择第一项进行核酸比较,可在excel 表中导出碱基组成信息。
关于遗传 距离
点击主窗口中Distance中的第一项,可对物种之间遗传距离进行计算 。注意Bootstrap参数设置(一般为1000)。模型选用Kimura双参数模 型进行计算。点击Computr进行计算
在主窗口中,点击上方工具栏W标志,将序列比对对齐。
序列对齐,修饰后,点击工具栏 保存图标,将文件进行保存。格式为 mas格式
保存后关闭上一个副窗口,回到主窗口,重新在File中打开刚保存的 .mas格式的文件。弹出新的副窗口(序列已对齐)
mega重点解读

MEGA 的使用产生背景及简介随着不同物种基因组测序的快速发展,产生了大量的DNA序列信息,这时就需要一种简便而快速的统计分析工具来对这些数据进行有效的分析,以提取其中包含的大量信息。
MEGA就是基于这种需求开发的。
MEGA软件的目的就是提供一个以进化的角度从DNA和蛋白序列中提取有用的信息的工具,并且,此软件可以免费下载使用。
现在我们使用的是MEGA4的版本。
它主要集中于进化分析获得的综合的序列信息。
使用它我们可以编辑序列数据、序列比对、构建系统发育树、推测物种间的进化距离等。
此软件的输出结果资源管理器允许用户浏览、编辑、打印输入所得到的结果而且所得到的结果具有不同形式的可视化效果。
此外,该软件还能够得出不同序列间的距离矩阵,这是他不同与其他分析软件的地方。
在计算矩阵方面有一些自己的特点:1.推测序列或者物种间的进化距离2.根据MCL(Maximum Composite Likeliood method)的方法构建系统发育树3.考虑到了不同碱基替换的不同的比率,考虑到了碱基转换和颠换的差别。
4.随时可以使用标注:所以的结果输入都可以使用标注,而且标注的内容可以被保存,复制。
具体使用我们以分析20个物种的血红蛋白为例来具体说明此软件的具体使用情况。
一.启动程序1.运行环境:在Windows 95/98, NT, ME, 2000, XP, vista等操作系统下均可使用。
2.下载安装:可以直接登陆进行下载安装,另外还可以从/tools/phylogeny.php中的链接进去。
3.双击桌面快捷方式图标,进入主界面;或者从开始菜单,单击图标启动。
二.序列分析。
1.启动单击后,会出现如下界面:这里有三个选项,分别对应三种不同的情况:以下分别予以介绍:Create a new alignment :是在你没有任何比对的时候使用,比如你只有一个fasta格式的序列就可以选择这个选项。
Open a saved alignment session:使用它可以打开一个我们已经比对好的序列文件;Retieve a sequence from a file :这种情况同第一种情况相似,只是不用选择是DNA还是蛋白质序列比对,选择的也是fasta格式的文件,打开后的界面都是一样的。
mega使用教程

m e g a使用教程(总9页) -CAL-FENGHAI.-(YICAI)-Company One1-CAL-本页仅作为文档封面,使用请直接删除MEGA的主界面:使用步骤1. 序列导入可以通过Data导入数据也可以通过file导入数据。
如果打开的文件是比对结果,选择Analyze;如果打开的文件是序列文件,选择Align。
另外双击这些后缀名文件即可自动导入序列,导入后会弹出MEGA比对界面。
如果fasta 序列导入报错,多是因为序列长度不同导致:如果序列长度不同,可以采用新建文件,将序列文件导入的方法。
步骤:Align → Edit/Build Alignment → create a new alignment → Data → open → Retrieve sequences from File将复制输入的序列另存输出看看。
步骤:data → Export alignment → fasta format序列长度都被用横线补齐了。
2. 多序列比对选择muscle或者clustalw进行比对:clustalw 一般用于DNA ,muscle多用于蛋白。
在比对之前需先选中要进行比对的序列(Shift),还可以对序列或者序列名进行编辑(双击)。
比对参数选择:保存比对文件,进化树分析提供数据。
一般导出的比对结果保存为fasta格式,或者直接点击保存按钮将结果,保存为二进制的mas或meg文件。
3. 构建进化树导入数据:将刚刚另存的meg 文件重新导入到mega程序中(直接拖入工作界面),并选择构建进化树。
参数选择:参数设置,Bootstrap method一般选择1000~1500;第一次绘图时建议选择500,这样运行速度会比价快,结果合适再调至1000重新进行进化分析。
描述:进化树可视化:View→Tree/Branch style选择树的模式,也可以通过右图菜单进行选择。
发散树环状树进化树简单美化:可视化文件的保存:?Newick——标准树文件,用于下游可视化软件的导入?png——压缩图像文件?pdf——矢量图像文件保存为png/pdf,显示不全怎么办——将图和图注复制到WORD中保存即可(Image Copy to Clipboard 粘贴到WORD)。
进化树构建之Mega篇,试了好半天才弄明白,赶紧写出来

进化树构建之Mega篇,试了好半天才弄明白,赶紧写出来1. 安装从Mega网站下载安装Mega 4版本按默认安装。
/2. Mega文件生成2.1. 已有Mega文件打开Mega程序的主界面, click me to activate a data file这个操作就打开了mega文件,然后执行相关的操作。
Mega是一个free 软件,所以请大家一定要在参考文献中列出Mega的出处,具体的文本在查看进化树的窗口菜单中点caption就可以得到。
2.2. 无Mega,仅有fasta文件双击准备好的fasta格式的序列文件,默认使用mega打开。
打开后Ctrl - A选定全部,执行Alignment > clustal W(默认参数)转换成meg格式:data > export to > Mega,选择地点和名称,输一个标题,不用蛋白数据了。
关闭这个窗口,程序会询问“open the data file in Mega”,确定,即可打开刚才保存的文件。
3. 建树在标题为Mega 4.的这个窗口中执行建树:建树:phylogeny > construct phylogeny > NJ > Method(NJ) > Gap ( pairwise deletion) > method(nucleotide > jukes-cantor > test > bootstrap > replication(1000) > computer。
此中参数选择是否正确我未知,请各位自行选择。
得到两张图,Original tree / bootstrap consensus tree,用后面那张图,接下来对图形进行修改。
选择outGroup,subTree > root,然后就可以用鼠标去选择外群了,如果是做物种的系统发育关系,则外群应该选择远缘关系的,如果是做群体即一个物种下的,则外群要选择近缘的;子树的翻转和颠倒等等操作,subTree > flip 或者是subTree > roll,选定后用鼠标去点节点就是了。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
/******************************************************************************
12M 延时计算公式 = 4.17+(n-1)*0.5 us
8M 延时计算公式 = 6.25+(n-1)*0.75 us
8M 延时计算公式 = 2.88+(n-1)*0.75 us
7.3728M 延时计算公式 = 6.24+(n-1)*0.81 us 或者 6.24+(n-1)*0.82 us
4M 延时计算公式 = 5.75+(n-1)*1.5 us
3.6864M 延时计算公式 = 6.24+(n-1)*1.63 us 或者 6.24+(n-1)*1.62 us
2M 延时计算公式 = 11.5 +(n-1)*3.0 us
1M 延时计算公式 = 23.0+(n-1)*6.0 us
*******************************************************************************/
3.6864M 延时计算公式 = 13.02+(n-1)*1.63 us
2M 延时计算公式 = 24.00+(n-1)*3.0 us
1M 延时计算公式 = 48.00+(n-1)*6.0 us
*******************************************************************************/
void delay (unsigned int n)
{
unsigned int i;
i = n;
while (i--)
{
;
}
}
/******************************************************************************
12M 0.083us
8M 0.125us
7.3728M 0.14us 0.27us
4M 0.25us
3.6864M 0.27us
2M 0.5us
1M 1.0us
8M 大约延时1000.25us
4M 大约延时999.5us
******************************************************************************/
void delay_1ms(void)
{
unsigned int i;
void delay_1us (void)
{
NOP();
}
/******************************************************************************
12M 延时计算公式 = 1.92+(n-1)*0.5 us
3.6864M 延时计算公式 = 13.29+(n-1)*1.63 us
2M 延时计算公式 = 24.5+(n-1)*3.0 us
1M 延时计算公式 = 49.0+(n-1)*6.0 us
*******************************************************************************/
for(i=1;i<(unsigned int)(xtal*143-2);i++);//xtal为晶振大小,单位为MHz
}
7.3728M 延时计算公式 = 6.78+(n-1)*0.81 us 或者 6.51+(n-1)*0.82 us
4M 延时计算公式 = 12.5+(n-1)*1.5 us
3.6864M 延时计算公式 = 13.56+(n-1)*1.63 us
void delay (unsigned int n)
{
unsigned int i;
for (i=n;i>0;i--)
{
;
}
}
/******************************************************************************
void delay (unsigned int n)
{
while (n--);
}
/*******************************************************************************
NOP();延时时间,这个就不用多说了吧
2M 延时计算公式 = 25.00+(n-1)*3.0 us
1M 延时计算公式 = 50.00+(n-1)*6.0 us
*******************************************************************************/
*******************************************************************************/
/******************************************************************************
12M 0.75us
8M 1.13us
7.3728M 1.22us
4M 2.25us
3.6864M 2.44us
2M 4.5us
1M 9.0us
*******************************************************************************/
12M 延时计算公式 = 4.08+(n-1)*0.5 us
8M 延时计算公式 = 6.13+(n-1)*0.75 us
7.3728M 延时计算公式 = 6.65+(n-1)*0.81 us 或者 6.51+(n-1)*0.82 us
4M 延时计算公式 = 12.25+(n-1)*1.5 us
void delay (unsigned int n)
{ Biblioteka unsigned int i; for (i=0;i<n;i++);
}
/******************************************************************************
12M 延时计算公式 = 4.0+(n-1)*0.5 us
8M 延时计算公式 = 6.0+(n-1)*0.75 us
7.3728M 延时计算公式 = 6.51+(n-1)*0.81 us 或者 6.51+(n-1)*0.82 us
4M 延时计算公式 = 12.0+(n-1)*1.5 us
