Icimapping 连锁图中文操作说明
INPHO DTMaster中文操作手册

北京超图软件股份有限公司 2012 年 2 月
教程 ..................................................................................................................................................4 案例学习:...............................................................................................................................5 A 浮动的窗口界面 ..........................................................................................................5 B 处理较大数据-稀疏模式 .............................................................5 C 捕获点/线 .....................................................................................................................6 D 用“Create Point List” 捕捉数据.................................................................................7 E 用“Add Point List” 捕捉数据........................................................................................9 F 用侧视图编辑雷达数据...........................................................................................10 G 编辑 MATCH-T数据 ................................................................................................10 H 导入矢量数据——DXF例子 ......................................................................................12 I 导出矢量数据——分开导出DXF................................................................................13 J 过滤器 .........................................................................................................................14 K 检查工程-线交叉 .......................................................................................................18
QTL IciMapping3.0 简单教程

QTL IciMapping3.0 定位简单应用教程张茜中国农科院2012.6.14主要步骤•数据准备•新建project•导入数据•构建图谱•QTL定位准备数据•.map格式将txt格式后缀名改成.map即可(表头信息不能动),一个map文件中包括General Information、Marker Types 、Information for Chromosomes and Markers三部分信息主要更改数据:7为F2群体;1一般不动;Marker space type 选1或2均可,只要保持数据对应Maker Types带型统计方法这些数据是标记在第几条染色体(group)上,未构建图谱侧全为0点File 选New Project新建一个工作项命名保存路径点File 选*map导入构建准备好的map格式图谱的数据打开,完成数据导入点击分组,在此处出现group群点可以看到一个group下所含标记,右键点击一个标记可以对其位置调动或者删除完成分组后,点击ordering,转换成染色体组再点此按钮完成沟通准备工作,工具栏上的map图标变蓝可以点击构图了点击map 按钮出现图谱(右)点击即可出现下一个染色体图谱点击出现整体图谱Save 可以保存各种格式的图QTL定位数据准备将构图所得结果F2bip(在project-map-result文件下)先复制一份,再用txt打开方式打开所复制文件。
Bip文件中包含5部分General Information、Information for Chromosomes andMarkers、Linkage map (Marker namefollowed by position or the interval length)、 Marker Type 、Phenotypic Data更改数据:0改成1选File-open file-*bip打开更改保存好的bip格式文件选ICIM-ADD添加的下框(一般都默认),此时start按钮从灰色变黑色,单击即可进行定位Start 完了点ADD即出现下图加性效应图显性效应图总染色体添加lod值线下一个染色体在Graph 下可以选择连锁图和lod (上)或者连锁图和QTL(下)图谱结果信息在map目录下QTL结果信息在BIP目录下信息栏补充•QTL ICIMapping是在*map(oppen file子菜单下)下完成构件图谱,在*bip(oppen file子菜单下)下完成QTL定位。
云南连锁业务简易操作手册

常用功能介绍1、网格操作平台提供了功能强大的数据展现控件——网格,在网格中您可以方便的进行数据查看、维护、统计以及简单的分析等操作。
平台中提供了三种基本的网格(一般网格、分页网格、树型网格)。
一般网格是最基础的一种数据展现和数据编辑网格。
分页网格是在一般网格的基础上增加了分页获取数据,分页显示数据的功能。
树型网格是种独立的网格,在树型网格中数据的展现可以按照数据的上下级关系进行分层展现,使得数据查看起来结构非常清晰。
2、设置网格样式网格为多样化的数据展现提供了样式管理器,用户可以根据日常工作的需要,将要查看的数据列拖拽到合适的位置,把不重要的数据列放到列定制中,调整好各列的最佳宽度,如果有分组的必要可以将分组列拖拽到网格顶部的分组面板中,甚至可以将某些列进行汇总统计等,操作完毕后点击鼠标右键,选择【网格样式…】,在弹出的如下管理器中保存设置好的样式在上面的网格显示样式管理器中,您还可以将某一样式设定为默认显示风格,方法是在样式名前的画勾,默认的显示风格是指第一次进入此网格所在的界面,网格就以您设定的默认风格显示数据。
在网格显示样式管理器中点击鼠标右键,还可以打开或删除选定的显示风格。
退出网格显示样式管理器,在网格中点击鼠标右键,选择【加载样式】,选择你所要加载的一种显示样式,可以灵活方便的切换数据显示,每个网格可以定义数个需要的样式。
3、网格数据获取针对不同的网格,数据的获取也不一样。
一般网格和树型网格数据的获取很简单,它展现的数据永远是满足条件的所有数据。
分页网格则不同于以上两种网格,分页网格提供了数据分页显示的机制,比较适宜于大量数据的展现上面是分页网格的工具条,从第三个按钮开始依次是刷新数据:保持当前数据查询的条件不变,直接向应用服务器发送数据请求,然后将满足条件的数据显示在网格中。
首页:如果当前网格数据不是首页数据,可以点击此按钮,重新获取首页数据。
前一页:如果当前网格数据不是首页数据,可以点击此按钮,重新当前页的前一页数据。
QTLIciMapping3.0简单教学教程

QTL IciMapping3.0 定位简单应用教程张茜中国农科院2012.6.14主要步骤•数据准备•新建project•导入数据•构建图谱•QTL定位准备数据•.map格式将txt格式后缀名改成.map即可(表头信息不能动),一个map文件中包括General Information、Marker Types 、Information for Chromosomes and Markers三部分信息主要更改数据:7为F2群体;1一般不动;Marker space type 选1或2均可,只要保持数据对应Maker Types带型统计方法这些数据是标记在第几条染色体(group)上,未构建图谱侧全为0点File 选New Project新建一个工作项命名保存路径点File 选*map导入构建准备好的map格式图谱的数据打开,完成数据导入点击分组,在此处出现group群点可以看到一个group下所含标记,右键点击一个标记可以对其位置调动或者删除完成分组后,点击ordering,转换成染色体组再点此按钮完成沟通准备工作,工具栏上的map图标变蓝可以点击构图了点击map 按钮出现图谱(右)点击即可出现下一个染色体图谱点击出现整体图谱Save 可以保存各种格式的图QTL定位数据准备将构图所得结果F2bip(在project-map-result文件下)先复制一份,再用txt打开方式打开所复制文件。
Bip文件中包含5部分General Information、Information for Chromosomes andMarkers、Linkage map (Marker namefollowed by position or the interval length)、 Marker Type 、Phenotypic Data更改数据:0改成1选File-open file-*bip打开更改保存好的bip格式文件选ICIM-ADD添加的下框(一般都默认),此时start按钮从灰色变黑色,单击即可进行定位Start 完了点ADD即出现下图加性效应图显性效应图总染色体添加lod值线下一个染色体在Graph 下可以选择连锁图和lod (上)或者连锁图和QTL(下)图谱结果信息在map目录下QTL结果信息在BIP目录下信息栏补充•QTL ICIMapping是在*map(oppen file子菜单下)下完成构件图谱,在*bip(oppen file子菜单下)下完成QTL定位。
IQsignal中文操作手册

发射测试结果(1)
单次测 试结果 信号波形 信号平均功率 平均测 试结果
红色线为 均值波形
底噪
(重点关注指标)试结果(2)
频谱模板
蓝色谱线不 能超出红色 限制线
功率幅度 统计图 判定产品功放 是工作在饱和 区还是线性区 (读数如箭头所 示) 蓝线紧贴红 线代表产品 工作在线性 区,两线分 离较大代表 产品功放在 饱和区工作
测试数据显示区
选择中间图 表显示内容
选择左下方图 表显示内容
选择右下方图 表显示内容
IQsignal软件界面——VSG界面
选择Tools->Vector Signal Generator即可进入 信号单次 发射按键 信号连续 发射开关 选择连续发射 或一次性发射 数据包数量 选择射频信号或 基带信号输出 设定频道和调制 方式(速率)
电脑IP设定步骤
本地连接---Internet协议---写入IP及子网掩码---(高级---添加---写入 IP及子网掩码--)确定---确定(若只需设定一个IP则不需进入高级选项)
IQsignal软件中的仪器IP地址设定
打开IQsignal软件,若提示错误如右图所示, 则可能是软件中的仪器IP地址设定不正确,应 按以下步骤写入正确的仪器IP地址:
接收测试
产品控制软件选择测试项目为Rx
设定要测试的频道 和速率
IQsignal的VSG界面设定频道与上面一样
设定VSA的调制方式 性发射数据包数量
、功率
和一次
点击产品控制软件的 ,点击VSA的 之后观察产品控制软件的接收数据包统计数据
接收测试结果
接收到的良好 数据包数量
本例中仪器VSG发射了1000个信号强度为-50dBm的数据包,接收到良好的 数据包数量为 912 ,故PER为 (1000-912)/1000 = 8.8% <10% 因此可证明,在-50dBm的信号强度下产品接收能力良好。(对于11b调制方 式,PER界限为8%) 然后降低仪器VSG信号功率,数据包统计数量复位清零 ,重复上页第 5 项操作,直至接收到良好数据包数量小于900,即 PER>10% ,则可初步 判定产品的接收灵敏度界限值(是否达到要求)。 更改频道和速率,重复上述操作,测试产品在不同频道速率的接收能力情况。 可绘制出PER与信号功率的关系曲线图,便于全面了解产品接收能力情况。
IMC智能管理中心操作手册v1

目录1 前提说明 (3)2 IMC系统配置 (3)2.1 系统登陆 (3)2。
2 SNMP模板配置 (4)2。
3 T ELNET模板配置 (5)2.4 报警服务器配置 (7)2。
4.1 Mail报警配置 (7)2。
5 设备分组配置 (8)2。
6 系统用户配置 (9)2。
6。
1 访问控制模板配置 (9)2。
6.2 操作员配置 (10)2.6。
3 在线操作员操作 (15)3 IMC资源配置 (15)3.1 新增设备配置 (16)3。
2 IMC性能配置及监控 (18)3.3 设备导入导出配置 (19)3.4 拓扑导入导出配置 (21)4 IMC业务配置 (22)4。
1 接入设备配置 (22)4。
2 服务配置管理配置 (25)4.3 接入时段策略管理配置 (28)4。
4 P ORTAL服务管理配置 (30)4。
4。
1 服务器配置 (31)4.4。
2 设备配置 (32)4.4.3 IP地址组配置 (34)4.4.4 启用Portal服务管理配置 (36)4。
5 设备配置管理 (36)4。
5.1 自动备份计划操作配置 (36)4。
5。
1.1 ............................................................................................................. 自动备份计划配置操作364.5。
1.2 自动备份计划立即备份操作 (39)4。
5.1。
3 .....................................................................................................................查看设备配置信息414。
5。
1。
4 ..................................................................................................................查看设备配置校验444.5。
质谱联用仪使用方法说明书

质谱联用仪使用方法说明书使用前的准备:在开始使用质谱联用仪之前,请确保您已经仔细阅读并理解了本使用方法说明书,并且熟悉了质谱联用仪的基本原理和操作流程。
在使用之前,请确保质谱联用仪处于正常工作状态,并进行必要的校准和准备工作。
1. 仪器连接和设置1.1 连接电源:将质谱联用仪的电源线插入适配器的插座,并将适配器插入电源插座。
确保电源电压与质谱联用仪的额定电压相符。
1.2 连接气源:使用适当的连接器将气源管线连接到质谱联用仪上的气源接口。
1.3 连接数据线:根据需要,选择合适的数据连接线(如USB或LAN线),将质谱联用仪连接到计算机或数据采集系统上。
2. 仪器校准和准备2.1 校准质谱联用仪:按照质谱联用仪的校准操作流程,对仪器进行校准。
确保校准参数的准确性和稳定性。
2.2 准备进样样品:根据实验需求,准备好需要进行分析的样品。
注意样品的准备和保存应符合实验要求。
2.3 准备色谱柱:根据实验需求,选择适当的色谱柱,并进行预处理和准备。
3. 开机和初始化3.1 打开电源开关:轻按质谱联用仪上的电源开关,将仪器的电源打开。
确保仪器显示屏正常亮起。
3.2 初始化仪器:根据质谱联用仪的操作指南,进行仪器的初始化操作。
确保系统软件和硬件处于正常工作状态。
4. 设置实验参数4.1 打开仪器控制软件:根据实验要求,在计算机上打开质谱联用仪的控制软件,并确认与仪器连接的通讯正常。
4.2 设置离子源参数:根据实验需求,设置离子源的工作模式、离子化方式、离子源温度等参数。
4.3 设置色谱参数:根据实验需求,设置色谱流体的流速、温度、梯度程序等参数。
4.4 设置质谱参数:根据实验需求,设置质谱仪的离子扫描范围、离子选择器和离子检测器的参数等。
5. 运行实验5.1 进样样品:按照仪器操作指南,将准备好的样品通过进样系统输入到质谱联用仪中。
5.2 开始实时监测:在控制软件中点击“开始监测”按钮,开启实时监测模式,观察样品的质谱图和色谱图的生成情况。
中联his系统院感管理系统操作手册

前言随着计算机的普及及互联网技术的广泛应用,各行各业都具有了划时代性质的变革,人们越来越多地利用网络来为自己服务。
医院作为人类文明与进步的重要象征的载体,并且也同其他行业一样受到了这股网络浪潮的冲击。
医院的管理和经营模式也发生了显著的变化,同时也为社会提供了更新更完备的各项医疗服务。
医院这一与人类自身利益息息相关的职能机构正朝着信息化的方向发展。
国内医院信息化起步较晚,系统建设总体水平偏低,但是90年代中后期特别是近几年得到了快速的发展。
为了加强中联公司用户的医院信息化管理水平,保证各业务环节的正常运行和软件使用,中联重庆公司项目部组织编写了《院感管理系统操作手册》,供医院院感科在实际工作中使用。
本书包含了常用操作流程、主界面介绍、主点功能讲解以及常见问题分析,希望能给医院管理人员带来帮助。
编者2011年11月30日目录一、疾病申报 (4)1.1操作流程 (4)1.2主界面介绍 (4)1.3 重点功能讲解 (5)1.3.1参数设置 (5)1.3.2修订 (5)1.3.3接收 (6)1.3.4发送 (6)1.3.5回退 (7)1.4 常见问题分析 (7)1.3.5.1找不到可供书写的疾病报告 (7)1.3.5.2找不到疾病报告 (7)二、病例监测 (8)2.1操作流程 (8)2.2主界面介绍 (8)2.3重点功能讲解 (9)2.3.1参数设置 (9)2.3.2报表 (10)2.3.3过滤条件设置 (10)2.4常见问题分析 (11)三、病例日报 (11)主界面介绍 (11)四、环境监测 (12)4.1操作流程 (12)4.2主界面介绍 (13)4.3重点功能讲解 (13)4.3.1新增 (13)4.3.2过滤条件设置 (14)五、人员监测 (14)5.1操作流程 (14)5.2主界面介绍 (15)5.3重点功能讲解 (16)5.3.1暴露源信息 (17)5.3.2血清学检查 (17)5.3.3评估 (17)一、疾病申报功能介绍:院感科审核临床科室上报的各类报告卡,或申报表。
QTL_IciMapping3.0_简单教程

QTL IciMapping3.0 定位简单应用教程张茜中国农科院2012.6.14主要步骤•数据准备•新建project•导入数据•构建图谱•QTL定位准备数据•.map格式将txt格式后缀名改成.map即可(表头信息不能动),一个map文件中包括General Information、Marker Types 、Information for Chromosomes and Markers三部分信息主要更改数据:7为F2群体;1一般不动;Marker space type 选1或2均可,只要保持数据对应Maker Types带型统计方法这些数据是标记在第几条染色体(group)上,未构建图谱侧全为0点File 选New Project新建一个工作项命名保存路径点File 选*map导入构建准备好的map格式图谱的数据打开,完成数据导入点击分组,在此处出现group群点可以看到一个group下所含标记,右键点击一个标记可以对其位置调动或者删除完成分组后,点击ordering,转换成染色体组再点此按钮完成沟通准备工作,工具栏上的map图标变蓝可以点击构图了点击map 按钮出现图谱(右)点击即可出现下一个染色体图谱点击出现整体图谱Save 可以保存各种格式的图QTL定位数据准备将构图所得结果F2bip(在project-map-result文件下)先复制一份,再用txt打开方式打开所复制文件。
Bip文件中包含5部分General Information、Information for Chromosomes andMarkers、Linkage map (Marker namefollowed by position or the interval length)、 Marker Type 、Phenotypic Data更改数据:0改成1选File-open file-*bip打开更改保存好的bip格式文件选ICIM-ADD添加的下框(一般都默认),此时start按钮从灰色变黑色,单击即可进行定位Start 完了点ADD即出现下图加性效应图显性效应图总染色体添加lod值线下一个染色体在Graph 下可以选择连锁图和lod (上)或者连锁图和QTL(下)图谱结果信息在map目录下QTL结果信息在BIP目录下信息栏补充•QTL ICIMapping是在*map(oppen file子菜单下)下完成构件图谱,在*bip(oppen file子菜单下)下完成QTL定位。
RationalDMIS快速使用手册

RationalDMIS 快速使用手册爱科腾瑞科技(北京)有限公司目录第一章:图形区和鼠标操作的介绍 (1)1.1 用户界面介绍 (1)1.2 鼠标操作 (1)第二章:测头构建/机器模型构建 (2)2.1 如何构建测头? (2)2.2 测头构建时需要注意什么? (2)第三章:标准球定义及测头校准 (5)3.1 定义标准球 (5)3.2 首次校验 (5)3.3 校验测头设置 (6)3.4 CNC 校验 (6)3.5 快捷校验 (7)第四章:元素测量 (8)4.1 手动测量各元素 (8)4.2 CNC 测量各元素 (9)4.3 生成DMIS 程序,使用DMIS 程序测量 (10)第五章:公差操作 (12)5.1 传统的公差计算操作 (12)5.2 一个元素同时计算多种公差的快捷操作 (12)5.3 多个元素求同一个公差 (12)第六章:输出报告常用设置 (14)6.1 输出元素定义 (14)6.2 报告头 (14)第七章:输出报告制作 (15)7.1 输出报告右键修改菜单 (15)7.3 输出报告上下公差修改 (16)7.4 输出报告删除多个元素 (16)7.5 在某元素前插入元素 (16)7.6 如何用程序控制输出报告中的参数 (17)7.8 运行程序时如何自动输出到EXCEL 摸板 (18)7.9 输出报告自动保存到外部指定目录下 (18)第八章:图形报告制作问题 (21)8.1 图形报告设置 (21)8.2 创建元素图形报告 (22)8.3 创建公差图形报告 (23)8.4 创建测量点图形报告 (24)8.5 图形报告的尺寸标注 (26)8.6 图形报告的自动排列 (27)8.7 制作图形报告时调整CAMERA (27)8.8 图形报告小窗口右键菜单介绍 (28)第一章:图形区和鼠标操作的介绍1.1 用户界面介绍1.2 鼠标操作图形区移动/旋转功能:平移图形:按下Ctrl 键+鼠标左键移动或者按下鼠标中键移动旋转图形:按下鼠标左键放大缩小图形:按下鼠标中间滚动或者按下Ctrl+shift+鼠标左键将图形中某个元素设置为图形照相机的中点:键盘上按住S 键不放,然后在某个元素上按下鼠标左键鼠标中间滚轮在图形区,前后滚动鼠标中间滚轮,这样可以方便地对图形进行放大缩小的操作.如果这时用户按下shift 键的话,则放大缩小的程度就会变小.这样适合用户对图形进行微调.数据区操作拖放目标:按下鼠标左键显示属性区:双击鼠标左键右键菜单:在目标上点击鼠标右键多选目标:Ctrl/Shift+鼠标左键第二章:测头构建/机器模型构建2.1 如何构建测头?1〉在操作工作区选择“测头”(快捷键 Ctrl+F2)—〉构建测头2〉根据实际测头的参数,在测头构建界面中选择合适的测头3〉最后,点击“添加/激活”,完成测头的构建4〉如果需要查看定义的测头,请到“双数据区”中的“测头数据区”查看,测头名称前面有测头图标的为当前使用的测头。
爱色丽Color iMatch操作手册

QC(品质控制)应用
一、语言选择 在关闭程序的情况下, 打开 “开始”—“ 所有程序”—“ GretagMacbeth”—“ Color iControl” 下的“iConfigure”:
显示如图: 语言选项:选择显示的语言 选择桌面:设置默认的应用 行业测量模式,在软件内可以更 改,建议为空白。 配色软件功能配置: iQC--只有测色,没有配色 Textile--纺织行业应用 Paints---涂料、塑料工业
二、仪器校正 1。打开程序,在出现 Please Wait...画面后,显示如下:
第 7 页
iQC 软件以工作(.job)的形式展开,类似于微软的 WORD 文档;每个 job 相 对 独 立 , 可以储存不同的设置和色样。 � Open Last e-job--------- 开启之前最后储存的一个 job 工作 � 打开新工作------------------ 开启一个全新的工作,工作内设置为系统默认的设置 � 只开程序,不开工作-----只打开 iQC 程序,不开启任何工作 � 取消-------------------------- 关闭程序 2。选择“打开新工作”,显示如:
其它画面直接按下一步, 直至安 装完成。
仪 器 安 装
软件安装完成后,在桌面上会出现一个右图所示的 Color iControl 图标, 双击此图标启动软件,显示 Please wait 画面(注意:软件初始化需要一定时 间,根据电脑的配置情况,等候时间长短不一,请耐心等待) 。 1.初始化完成后,出现右图所 示对话框: 操作员 ID--- 在设置每个操作员 的权限和密码后有效,暂可不输。 取消---------退出程序。 Setup--------进入设置画面。
X-rite Color iQC/iMatch 电脑测配色系统 操作手册
GC-PLACE操作手册
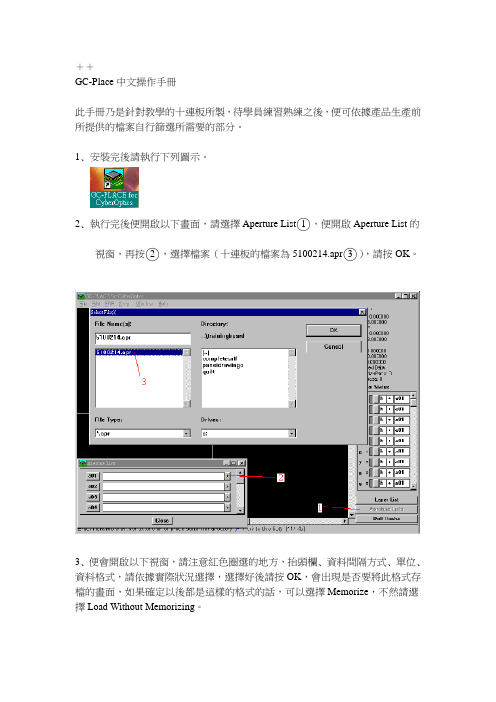
++GC-Place中文操作手冊此手冊乃是針對教學的十連板所製,待學員練習熟練之後,便可依據產品生產前所提供的檔案自行篩選所需要的部分。
1﹑安裝完後請執行下列圖示。
2﹑執行完後便開啟以下畫面,請選擇Aperture List○1,便開啟Aperture List的視窗,再按○2,選擇檔案(十連板的檔案為5100214.apr○3),請按OK。
3﹑便會開啟以下視窗,請注意紅色圈選的地方,抬頭欄﹑資料間隔方式﹑單位﹑資料格式,請依據實際狀況選擇,選擇好後請按OK,會出現是否要將此格式存檔的畫面,如果確定以後都是這樣的格式的話,可以選擇Memorize,不然請選擇Load Without Memorizing。
的檔案的位置與路徑。
5﹑待檔案選取完畢之後,請選擇執行File→Load Layers【Ctrl+L】,會出現Data type的視窗,請選擇Auto(如下圖),按OK。
6﹑之後會出現下面視窗,請在Layers的欄位輸入ALL或點進入裡面選擇所要載入的檔案,請按OK。
7﹑上述動作完成後,請依序將個個層級打開觀看(參考8),並依據需求與層級的內容給予適當的名稱,保留或刪除(刪除時請至File→Unload Layers【Ctrl+Shift+L】,並按去選擇要刪除的層級,並將Clear Layer names的選項打勾(將名稱去掉)),此時所剩下的層級如果沒有排列整齊,可使用File→Reorder Layers【F8】,先選擇要移動的層級(Layers),再選擇要移動至那一層的層級(Above),再按OK即可。
8﹑此時將Solder Paste(3)那一層打開並使用Edit模式○6(請參考下面圖示,此欄位可用H—隱藏,V—觀看,E—修改),將Solder Paste全選Mark起來,便會發現包含有Pads和Traces○7,此時選CAM→Auto Convert Sketched Pads 【Ctrl+K】(請注意下圖紅色圈選的地方,請在Top Sold Paste處輸入層級),執行後,便會將所有的Traces轉變成Pads(全選後可由○7得知)。
飞易特区域HIS系统管理操作手册实用资料

飞易特区域HIS系统管理操作手册实用资料(可以直接使用,可编辑优秀版资料,欢迎下载)飞易特区域HIS系统管理操作手册浙江飞易特软件2021年5月目录目录 (2)第一章系统概述 (3)1.1功能概述 (3)1.2系统特点 (3)第二章系统登录与切换 (4)2.1系统登录 (4)2.2系统切换 (5)第三章数据字典 (6)3.1区域HIS (6)3.1.1出院类型 (6)3.1.2门诊分类 (7)3.1.3住院分类 (8)3.1.4财务分类 (8)3.1.5药品剂型 (9)3.1.6剂量单位 (9)3.1.7用法设置 (10)3.1.8途径设置 (11)3.1.9结算方式 (12)3.1.10药品类别 (12)3.1.11科室类型 (13)第四章基础数据 (15)4.1区域HIS (15)4.1.1项目调价 (15)4.1.2病区设置 (17)4.1.3挂号费用设置 (17)4.1.4套餐设置 (18)4.1.5医生设置 (19)4.1.6科室设置 (20)4.1.7床位设置 (20)4.1.8配伍禁忌 (21)4.1.9配套使用 (21)4.1.10体检设置 (22)4.2公用数据 (23)4.2.1药房设置 (23)4.2.2医疗机构 (23)4.2.3病种设置 (24)4.2.4项目设置 (25)4.2.5医保药品 (26)4.2.6行政区域 (27)第五章系统管理 (28)5.1用户 (28)5.2发票打印格式 (29)5.3用户组 (31)5.4修改密码 (32)5.5清空数据 (33)5.6医院组 (34)5.7公告管理 (34)第一章系统概述1.1功能概述药库管理功能采用一体化设计,界面友好,实用性强,与药房管理模版相关联。
主要功能有:药库入库:包括采购入库、期初入库、销售退库等业务类型。
药库出库:包括采购退货、科室领药、药房调拨等业务类型。
药房调拨:对中心和站点药房的请领单进行审核、保存。
爱普灵操作指南

软件一
• 点开索引图示,如 右图共分三部分 1、病人信息和 ApneaLink准备或 下载时输入病人信 息一样 2、当大量数据 时,可以输入姓、 名或生日搜索你需 要的信息
病人信息 报告 趋势图
‹#› ApneaLink Plus © ResMed 2013
软件三---趋势图
1、上界面(窗口)
2、下界面时间段标记 3、时间标记线 4、信号 5、通道 6、下界面(窗口) 7、滚动条或方向指示
信号太小 未定义暂停
•
1、添加或修改事件,选择事件按鼠标右键会弹出如上窗口,点击所选事件即可
•
2、删除事件,点击所选事件,按键盘Delete即可
‹#› ApneaLink Plus © ResMed 2013
报告设置
添加Logo在报告 左上角(map格 式)
添加治疗处方报告
报告打印份数
报告提示整晚血氧 饱和度低于88%或 89%的时间 添加病人问诊病史报 告(在AHI〉5时显示 或总是显示
• 三个传感器与主记录 仪连接
‹#› ApneaLink Plus © ResMed 2013
导联连接示意图
电源指示灯
开关按键
鼻导管连接
下载线连接电脑 呼吸努力度连接
血氧饱和度连接
‹#› ApneaLink Plus © ResMed 2013
开始记录
• 按住开关按钮持续约3秒,直 至绿灯发光; • 约10分钟后绿灯变淡,但持续 发光,证明记录仪工作正常; • 如果红灯亮,检查血氧传感器 或电池电量; • 如果绿灯闪烁,检查气流传感 器
8、事件
9、事件类型选择
10、从一个事件到另一个事 件的转换
11、选择每屏时间段 12、信号基线调整 13、信号波幅调整
JIUYIN CEMP分店操作手册

九垠真金JIUYIN CEMP分店管理流程目录第一章分店管理概述 (4)1.1九垠真金商业自动化管理系统简介 (4)1.2九垠真金商业自动化管理系统特点 (4)第二章采购补货物价管理 (5)2.1手工定制补货单 (5)2.2自动生成补货单 (8)2.3补货作废清查 (11)2.4商品调价处理 (13)2.5商品促销管理 (16)第三章配送调拨管理 (16)3.1配送调拨处理 (16)3.2配送收货确认 (24)3.3直配定单录入和确认 (27)第四章帐务报表管理 (31)4.1业务日结汇总 (31)4.2日终数据初始化 (38)第五章卖场管理 (41)5.1收银员缴款 (41)5.2电子称数据转换 (46)5.3POS机数据下传 (48)5.4接收POS机数据 (51)第六章库房管理 (51)6.1商品报溢处理 (51)6.2商品报损处理 (55)6.3商品盘点处理 (59)第七章远程数据传输 (59)7.1数据上传 (59)7.2数据下载 (61)第一章分店管理概述1.1九垠真金商业自动化管理系统简介九垠真金商业管理系统认识到,要在瞬息万变的市场中实现盈利的增长,需要一个能够更可靠地满足商业经营者需求,且整个系统的库存又比传统方式更少的经营基础。
为此,九垠真金软件根据中国商业企业规范化管理的特征与要求,吸取发达国家的先进管理经验,充分利用计算机网络技术、数据库技术与条码技术等现代高科技手段实现商业管理流程的自动化。
它不紧是高科技术在商业流通领域中的大规模应用,更是一种科学,高效的管理理念的革新。
提高了管理者对市场反映速度和预测的准确度,降低了市场需求的不稳定性,准确掌握各品类的贡献度,为企业建立一套基于业务流控制管理的方法,从而为企业建立起长期发展的基础。
1.2九垠真金商业自动化管理系统特点九垠真金商业自动化管理软件在国家商贸委有关领导的大力支持下,在众多大型商家的帮助下,依靠自身的高科技优势,构成了商业自动化领域的完美结合,形成九垠真金软件独到的管理精髓:✧支持赠品管理及连带商品管理✧供货商家促销管理,商家赞助管理,商家清仓管理✧设定商家结转级别,支持商品不同折扣率下的商家抽成✧单品批次管理✧设立退换货库✧自动分货以及分场(店)直配管理✧支持多级配送管理✧支持一码多品和一品多码管理✧商品上柜、下柜管理✧商情管理✧自动产生新分店初始数据✧各种促销方式,不同分店可不同方式促销✧配送中心、分店双向访问,数据自动化控制✧设计有操作安全系数✧记录操作员的日志第二章采购补货物价管理2.1手工定制补货单手工定制补货单补货单效验申报上传数据手工定制补货单的一般步骤:1、双击分店后台管理系统打开程序运行界面,并双击采购补货系统模块:(如图1)图12、进入采购补货模块后,点击补货管理菜单中的“定制生成补货单”选项(如图2)图23、系统弹出的定制生成补货单窗口时,点击菜单中的“新单”键(如图3)这时会在右边的单据编号中自动生成编号,即补货单编号。
(仅供参考)Icimapping 连锁图中文操作说明
遗传连锁图谱构建
12. 高级用户 – 锚定信息的管理 • 清空作图结果: 鼠标指向遗传群体文件名称”Arabidopsis.map”, 然后右击, 从弹
出的快捷菜单中, 选择”Clear Results”, 然后在弹出的对话框中选择”OK”
• 建立新的锚定信息群: 鼠标指向”锚定信息/分组信息/染色体信息展示窗口”, 然后右击, 从弹出的快捷菜单中, 选择”New Anchor”. 锚定信息群“Anchor1[0]” 出现在这个窗口内.
• 连锁图谱可以保存 成多种文件格式
8
遗传连锁图谱构建
11. 在”结果展示窗口”中浏览结果文件 • 在”工程窗口”的文件列表中, 双击待浏览的结果文件, 这个结果文件将显示在”
结果浏览窗口”, 图例中, 打开/显示的文件是”Aradbidopsis.sum”
• 所有输出结果文件保存在”D:\Tutorial\MAP\Arabidopsis\Results\...”中 • “Tutorial”是工程的名称 • “MAP”指示软件的功能. 目前有7大功能 • “ArabidopsisRIL”是导入遗传群体文件的前缀 • “Results”是保存结果文件夹的名称
• 从标记群中删除标记 (例如”Group4”中的”FRI”和”SNP254”): 鼠标指向待删除 标记” FRI”, 然后右击, 从弹出的快捷菜单中, 选择”Delete” 将”FRI” 从”Group4[*]”中删除; 对”SNP254”重复上述过程.
11
遗传连锁图谱构建
14. 高级用户 – 排序标记(即连锁群)信息的管理 • 连锁群间移动标记 (例如, 把 “Chromosome5”和”Deleted Markers”中的一些标
请注意: 开始之前, 请确认您的计算机已安装了QTL IciMapping集成软件!
网康ICG操作手册
用户管理用户是互联网访问的标识主体(即谁在访问),是NS-ICG互联网访问管理的目标对象。
出身份认证信息外,一个完整的用户定义还包含其组织信息和访问策略。
每一个互联网访问都对应唯一的用户(或用户组),这是NS—ICG实现基于用户和用户组的访问控制的基础。
而建立完整和准确的用户信息更是保证NS-ICG进行有效互联网访问审计(监控、查询、报表等)的关键。
用户管理模块提供全面的用户管理功能,主要有如下几个功能模块:用户导入-——--—--—————-—可通过扫描当地局域网内的IP导入用户、导入LDAP用户或者自定义导入用户。
组织管理—-—-—-—--—--—--手动构建企业组织架构和用户信息.认证管理———--—-—-———-—-配置用户上网的识别和认证管理方式,可针对不同用户采用不同的识别认证方式,支持本地人证和多多种第三方认证;支持用户绑定设置。
用户导入用户导入主要支持三种方式:IP导入、LDAP导入和网康自定义导入。
IP 导入主要通过扫描设备IP来进行用户导入,LDAP导入时导入LDAP服务器上的用户,而网康自定义导入则是通过TXT或者CSV文件导入用户信息。
IP导入NS—ICG提供两种用户信息的建立方式.其一,可以通过已存在的用户信息数据源,导入到NS-ICG系统中,如依据IP地址导入用户、从已建立的LDAP 服务器中导入用户、根据网康自定义格式导入用户;其二,可以通过管理界面手工管理。
第1步:通过【用户管理】-—【用户导入】-—【IP导入】进入如下图所示页面:第2步:点击“扫描”或者“浏览"本地文件后点击“导入”,进入“导入用户列表"画面,如下图:用户名:手动输入用户名;导入选项:导入IP与MAC:保存用户名、IP地址和MAC地址导入MAC:保存用户名、MAC地址导入IP:保存用户名、IP地址填充用户名:如果选择该项,ICG 会将相应的IP 地址作为用户名进行自动填充;选择导入位置:根据选择,导入到组织管理的指定位置(注:需提前配置好组织架构)第 3 步:管理员根据需要选择导入的数据和填充画面各项信息后,点击右上角的“导入"按钮,进行导入.或者选择“返回”按钮,返回到前一画面。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
• 输入”工程名称” (例如, 图 示中的工程名称为Tutorial) 和选择路径 (例如, 图示中 的路径为D:\)
• 单击”OK” 完成”新工程”创 建
新建工程显示在”工程窗 口”中
所有与此工程相关的各种 输入/输出, 按特定的组织
形式自动保存在路 径”D:\Tutorial\...”下的各
种文件夹中
• 单击”打开(O)” 完成
由此选择打开EXCE3L 2003/2007文件
遗传连锁图谱构建
5. MAP功能浏览: MAP功能把窗口分为4部分 • 工程窗口 • 标记整理信息和结果展示窗口 • 锚定信息/分组信息/染色体信息展示窗口 • 参数选择/设置窗口
工程窗口
标记整理信息和 结果展示窗口
锚定信息/ 分组信息/ 染色体信息 展示窗口
• 连锁图谱可以保存 成多种文件格式
8
遗传连锁图谱构建
11. 在”结果展示窗口”中浏览结果文件 • 在”工程窗口”的文件列表中, 双击待浏览的结果文件, 这个结果文件将显示在”
结果浏览窗口”, 图例中, 打开/显示的文件是”Aradbidopsis.sum”
• 所有输出结果文件保存在”D:\Tutorial\MAP\Arabidopsis\Results\...”中 • “Tutorial”是工程的名称 • “MAP”指示软件的功能. 目前有7大功能 • “ArabidopsisRIL”是导入遗传群体文件的前缀 • “Results”是保存结果文件夹的名称
• 对连锁群重命名: 鼠标指向连锁群”Chromsome4”, 然后右击, 从弹出的快捷菜 单中选择”Rename”实现对”Chromosome4”的重命名, 或者…
• 将连锁群上移或下移
• 删除连锁群内的所有标记
• 改变连锁群首尾标记的循序
12
遗传连锁图谱构建
15. 高级用户 – 在EXCEL中管理遗传群体的信息 • 工作表”GeneralInfo”定义遗传群体的一些基本信息, 每项信息占1行.
6
10. 绘制连锁图谱 • 单击”MAP”图标
遗传连锁图谱构建
• 或者从”Figures”菜单 中选择”Linkage Map”
• 通过参数设置改变连 锁图谱的格式
7
10. 绘制连锁图谱 (续) • 在图形区域右击鼠
标弹出快捷菜单, 通过快捷菜单改变 图形的格式
遗传连锁图谱构建
• 在作图区域右击鼠 标, 以复制/打印/保 存连锁图谱
参数选择/设置窗口
4
6. 标记分群
遗传连锁图谱构建
• 单击Grouping下的 箭头, 软件将利用默 认/设定参数对标记 进行分群, 例如图例 中, 按LOD临界值 3.00 的标准分群
7. 标记排序
• 单击Ordering下的箭 头, 软件将利用默认 /设定参数对标记进 行排序, 例如图例中, 利用nnTwoOpt算法 排序
右击, 从弹出的快捷菜单中, 选择”New Group”. 一个新群”Group5[0]”出现在 这个窗口内.
• 向新建标记群中添加标记 (例如”Group4”中的”F6L9.78”和”SNP53”): 鼠标指向 待移动标记”F6L9.78”, 然后右击, 从弹出的快捷菜单中, 选择”Move to -> Group5” 将”F6L9.78” 添加到”Group5[*]”中; 对”SNP53”重复上述过程.
5
遗传连锁图谱构建
8. 标记排序调整 (此选 项可跳过, 调整的目 的是获得更短的图 谱)
• 单击Rippling下的箭 头, 软件将利用默认 /设定参数对标记顺 序进行调整, 例如图 例中, 利用SARF做标 准, 窗口大小为5
9. 结果输出
这个图标只有在执行 “Outputting”后才可用
• 单击Outputting下 的箭头, 软件将输 出作图结果, 例如 图例中, 输出成对 标记间的LOD值, 重 组率, 图距, 以及 QTL分析中用到的 输入文件
13
如何构建整合图谱?
• 选择软件的IMP功能 • 向工程中导入待整合的
图谱. 例如, 把软件中附 带的”Arab_1.imp”打开. • 依次执行”Grouping”, “Ordering”, “Rippling (可 选项)”, 和”Outputting”建 立整合图谱.
14
如果想要计算F2群体中, 2个显性标记间的重组率, 怎么办?
• 向锚定信息群中添加标记 (例如”SNP71”和”SNP251”): 鼠标指向待添加标 记”SNP71”, 然后右击, 从弹出的快捷菜单中, 选择”Anchor to -> Anchor1” 将”SNP71” 添加到Anchor1中; 对”SNP251”重复上述过程.
10
遗传连锁图谱构建
13. 高级用户 – 分组标记信息的管理 • 建立新的标记群: 鼠标指向”锚定信息/分组信息/染色体信息展示窗口”, 然后
2. 软件浏览. 单击StartPage中的”左箭头”或 ”右箭头”, 可以看到: • 软件所能处理的遗传群体类型 • 共显性标记是如何编码的 • 显性标记是如何编码的 • 隐性标记是如何编码的
1
3. 建立一个”新工程” • 单击”File”菜单 • 选择”New Project”
遗传连锁图谱构建
9
遗传连锁图谱构建
12. 高级用户 – 锚定信息的管理 • 清空作图结果: 鼠标指向遗传群体文件名称”Arabidopsis.map”, 然后右击, 从弹
出的快捷菜单中, 选择”Clear Results”, 然后在弹出的对话框中选择”OK”
• 建立新的锚定信息群: 鼠标指向”锚定信息/分组信息/染色体信息展示窗口”, 然后右击, 从弹出的快捷菜单中, 选择”New Anchor”. 锚定信息群“Anchor1[0]” 出现在这个窗口内.
2
遗传连锁图谱构建
4. 向新建工程中导入遗传群体 • 单击”File”菜单, 然后选择
File -> OpeΒιβλιοθήκη File -> *.map
• 或者单击”Open”, 然后选择 *.map (Linkage map construction) 文件类型
• 在文件列表中选择要打开的 群体 (例如: “C:\CAAS\QTL IciMapping\Examples\MAP\ ArabidopsisRIL.map”)
• 选择”Tool -> 2pointREC”
• 选择群体类型 • 选择标记类型 • 输入不同标记型
下的观测样本量 • 单击”RUN”
• 浏览重组率的估 计值, 检验遗传连 锁的LOD值, 以及 位点间的图距等
15
请注意: 开始之前, 请确认您的计算机已安装了QTL IciMapping集成软件!
遗传连锁图谱构建 (MAP)
1. 打开软件: QTL IciMapping 软件可通 过两种方式打开 • 双击QTL IciMapping 软件在桌 面上的快捷图标 • 从”所有程序”菜单中选择QTL IciMapping 软件
记移动到”Chromosome4”): 鼠标指向所要移动的标记”F6L9.78”, 然后右击, 从 弹出的快捷菜单中选择”Move to -> Chromosome4”将 ”F6L9.78”移动 到”Chromosome4”; 对”SNP53”, “FRI”和”CNP254”重复上述过程.
• 对连锁群”Chromsome4”再排序: 鼠标指向连锁群”Chromsome4”, 然后右击, 从弹出的快捷菜单中选择”Ordering”实现对”Chromosome4”的重排序
• 从标记群中删除标记 (例如”Group4”中的”FRI”和”SNP254”): 鼠标指向待删除 标记” FRI”, 然后右击, 从弹出的快捷菜单中, 选择”Delete” 将”FRI” 从”Group4[*]”中删除; 对”SNP254”重复上述过程.
11
遗传连锁图谱构建
14. 高级用户 – 排序标记(即连锁群)信息的管理 • 连锁群间移动标记 (例如, 把 “Chromosome5”和”Deleted Markers”中的一些标
• 工作表”Genotype”定义遗传群体中每个个体的标记型, 每个标记占1行. 每行 第1列为标记名称, 其他列为标记型.
• 工作表”Anchor”定义标记的锚定信息, 每个标记占1行. 每行第1列为标记名称, 第2列为锚定信息, 用正整数表示, 0表示未知锚定信息.
• EXCEL中管理遗传群体的注意事项 • 从软件附带的EXCEL群体实例开始, 用这些实例做模板 • 不要改变EXCEL工作表的名称 • 确认信息的完整性, 不同工作表间无冲突. 例如, 工作表”GeneralInfo”定 义群体大小为120, 那么工作表”Genotype”要包含121列. 每行第1列为标 记名称, 其他120列为标记型. • 文件名称, 标记名称, 性状名称中不包含空格.
