ThermoScientificMaxima第一链cDNA合成试剂盒适用于RT-qPCR
总RNA提取,cDNA第一条链合成,cDNA纯化,3‘端加尾实验,荧光定量PCRprotocol

RNA Extraction Protocol1.实验原理异硫氰酸胍/苯酚法-----目前实验室使用的方法!✓细胞在变性剂异硫氰酸胍的作用下被裂解,同时核蛋白体上的蛋白变性,核酸释放✓释放出来的DNA和RNA由于在特定pH下溶解度的不同而分别位于整个体系中的中间相和水相,从而得以分离细胞或组织1. 样品裂解2. 分离总RNA2.实验试剂与器材2.1实验试剂RNA提取试剂Trizol(Invitrogen)、氯仿、异丙醇、75%乙醇、无水乙醇、DEPC 水,反转录试剂盒(Fermentas),cDNA纯化试剂盒(Promega),末端加尾酶TdT (Thermo)。
2.2实验器材微量移液器(Eppendorf),电泳仪,电泳槽,超净工作台,4℃低温离心机(Eppendorf),常温离心机(Eppendorf),匀浆机(IKA),高压蒸汽灭菌锅,通风橱,紫外分光光度计(Eppendorf),PCR仪(Takala),剪刀,镊子,0.2mL、1.5mL离心管,枪头。
3.实验前准备工作实验前将要用到的试剂配好(75%乙醇:75mL的无水乙醇+25mL的去离子水;DEPC水:在1000ml双蒸水中加入DEPC原液1ml,然后摇匀过夜),与离心管、枪头、剪刀、镊子(用DEPC水处理过的,浸泡过夜)一起灭菌(126℃,90min),之后剪刀和镊子最好在紫外灯下照射半小时。
提前20min打开低温离心机预冷。
准备两幅手套,一个口罩,塑胶手套不能离开通风橱,整个实验过程尽量降低外源RNA酶对样品中RNA的降解。
4.实验步骤4.1取样(1)组织样品:将组织在RNA保护液中用剪刀剪碎,每50~100mg组织加入1ml TRIzol,用匀浆仪进行匀浆处理。
样品体积不应超过TRIzol体积10℅。
(2)贴壁培养细胞:不须消化,直接在培养板中加入TRIzol裂解细胞,每10cm2面积(即3.5cm直径的培养板)加1ml,用移液器吸打几次。
第一链cDNA合成试剂盒Transcriptor First Strand cDNA Synthesis Kit 中文说明书

第一链cDNA合成试剂盒Transcriptor First Strand cDNA Synthesis Kit中文说明书2013-12-07 13:20:16Transcriptor First Strand cDNA Synthesis Kit试剂盒包装与含量小瓶/瓶盖标签适用于a) 04 379012001b)04896866001c************1红色TranscriptorReverseTranscriptase(逆转录酶)a) 1瓶,25 μl (20 U/μl)b) 1瓶,50 μl (20 U/μl)c) 2瓶,各50 μl (20 U/ μl)•储存缓冲液:200 mM 磷酸钾,2 mM 二硫苏糖醇,0.2% Triton X-100(v/v),50% 甘油(v/v),pH 约为7.22无色Transcriptor RTReaction Buffer(5×)(逆转录缓冲液)a) 1瓶,1 mlb) 1瓶,1 mlc) 2瓶,各1 ml• 5×浓度:250 mM Tris/HCl,150 mM KCl,40 mM MgCl2,pH约为8.5(25°C)3无色ProtectorRNase Inhibitor(RNase抑制剂)a) 1瓶,50 μl(40 U/μl)b) 1瓶,100 μl(40 U/μl)c) 2瓶,各100 μl(40 U/μl)•储存缓冲液:20 mM Hepes-KOH,50 mM KCl,8 mM 二硫苏糖醇,50 % 甘油(v/v),pH 约为7.6 (4°C)4黄色/紫色Deoxynuc-leo-tideMix(dNTP)a) 1瓶,100 μl(黄色瓶盖)b) 1瓶,200 μl(紫色瓶盖)c) 2瓶,各200 μl(紫色瓶盖)• dATP, dCTP, dGTP, dTTP各10 mM5蓝色Anchored-oligo(dT)18Primer(锚定oligo(dT)18引物)a) 1瓶,100 μl(50 μM)b) 1瓶,200 μl(50 μM)c) 2瓶,各200 μl(50 μM)6Random Hexamera) 1瓶,100 μl(600 μM)蓝色Primer(随机引物)b) 1瓶,200 μl(600 μM)c) 2瓶,各200 μl(600 μM)7绿色Control RNA(对照RNA)a) 1瓶,20 μl(50 ng/μl)•包含提取于永生细胞系(K562)的总RNA片段稳定溶液8绿色Control Primer Mix PBGD(对照基因引物)a) 1瓶,40 μl•5 μM 人类PBGD特异性正向与反向引物9(b和c为瓶7)无色Water, PCR-gradea) 1瓶,1 mlb) 2瓶,各1 mlc) 3瓶,各1 ml注意:货号为***********和***********的产品不含有control试剂(瓶7和瓶8),因此瓶7即为Water, PCR-grade。
ThermoScientificRevertAidFirstStrandcDNASynthesisKitK说明书第一链cDNA合成试剂盒

RevertAid第一链cDNA Synthesis试剂盒#K1621, #K1622分析证明书质量控制#K1621Lot采用100 fg对照GAPDH RNA和对照引物进行RT-PCR反应,通过在1%琼脂糖上进行凝胶电泳和溴化乙锭染色显示得到足够量的496 bp的产物质量认证人:Jurgita Zilinskiene目录页码试剂盒组成 (2)存储条件 (2)产品说明 (2)注意事项 (3)操作步骤 (6)RT-PCR (6)合成cDNA用于克隆………..………………………………………7 实验对照……………………………………………………………….8 问题分析与解决 (10)** 一个单位的RiboLockRNase酶抑制剂抑制5ng RNA酶A 50%的活性。
存储条件试剂盒中所有组分应存储在-20°C。
对照用RNA可存储于-70°C以便长期使用。
产品说明RevertAid第一链cDNA合成试剂盒以mRNA或者总RNA为模板,高效合成第一链cDNA。
本试剂盒使用RevertAid M-MuLV反转录酶,它的RNA酶H的活性与AMV反转录酶相比较低。
该反转录酶可耐受42-50°C温度,合成的cDNA片段长度达13kb。
试剂盒中含有RiboLock 重组RNA酶抑制剂,防止RNA降解,可耐受55°C高温。
试剂盒同时含有oligo(dT)18和随机六聚体引物。
随机六聚体引物与模板非特异性地结合,以总RNA中任何RNA为模板合成cDNA。
oligo(dT)18选择性和RNA 3’po ly(A)配对结合,只以有poly(A)尾巴的mRNA为模板合成cDNA。
使用本试剂盒也可采用序列特异性引物。
合成的第一链cDNA能直接用作PCR或荧光定量PCR的模板,第二链cDNA的合成或线性RNA扩增,也可用于需要用带有放射性或非放射性核苷酸标记第一链cDNA的实验,比如将标记好的第一链cDNA 作为杂交实验中的探针或者用于微阵列分析。
原发性高血压患者血清miRNA-663水平与RAAS生化标志物的相关性分析

原发性高血压患者血清miRNA-663水平与RAAS生化标志物的相关性分析刘文果;陈石磊;万志佳;魏星【摘要】目的探讨原发性高血压患者血清miRNA-663水平与肾素-血管紧张素-醛固酮系统(RAAS)生化标志物的相关性.方法受试者分为原发性高血压组、健康对照组,每组50例.分别采用荧光实时定量聚合酶链反应、化学比色法及放射免疫法检测外周血血清miRNA-663、血管紧张素转换酶(ACE)活性,以及肾素(RA)、血管紧张素(Ang)Ⅱ 、醛固酮(ALD)的表达.结果相对于健康对照组,原发性高血压组患者外周血血清miRNA-663表达显著降低,ACE活性及RA、AngⅡ 、ALD表达均显著升高(P<0.01);原发性高血压患者血清miRNA-663水平与血清ACE活性及RA、AngⅡ 、ALD水平均呈显著负相关.结论原发性高血压患者外周血血清miRNA-663表达显著下调,RASS生化标志物显著升高,外周血血清miRNA-663表达与RASS生化标志物呈显著负相关关系.【期刊名称】《检验医学与临床》【年(卷),期】2018(015)019【总页数】4页(P2899-2901,2905)【关键词】miRNA-663;高血压;肾素;血管紧张素转换酶;血管紧张素Ⅱ【作者】刘文果;陈石磊;万志佳;魏星【作者单位】重庆市结核病防治所检验科 400050;陆军军医大学军事预防医学院防原医学教研室,重庆400038;重庆市沙坪坝区妇幼保健计划生育服务中心检验科400030;重庆市奉节县人民医院检验科 404600【正文语种】中文【中图分类】R446.11原发性高血压是以血压升高为主要临床表现的综合征,是多种心、脑血管疾病的重要病因和危险因素。
原发性高血压会严重影响心、脑、肾等重要脏器的结构与功能,最终导致这些器官的功能衰竭,迄今其仍是心血管疾病死亡的主要原因之一[1-2]。
原发性高血压的发病机制十分复杂且不明确,目前研究表明原发性高血压是多因素、多层次共同作用的结果。
ThermoScientificMaxima第一链cDNA合成试剂盒适用于RT-qPCR

产品信息Thermo ScientificMaxima第一链cDNA合成试剂盒(适用于RT-qPCR)#K1641,#K1642/onebio分析证书经验证,在两步法RT-qPCR中,对于不同起始量(5 ng-0.5 fg)的RNA逆转录产物,使用Thermo Scientific Maxima SYBR Green/ROX qPCR 预混液进行qPCR扩增。
其反应效率在90-110%之间,曲线斜率在-3.09和-3.58之间,且相关系数>0.99。
质量授权人:Jurgita Zilinskiene目录页码试剂盒组分 (4)贮存条件 (4)产品描述 (4)重要提示 (5)RT-qPCR实验方案 (6)对照反应 (6)疑难解答 (7)参考文献 (8)试剂盒组分贮存条件所有试剂盒成分都应贮存于-20 °C。
产品描述Thermo Scientific Maxima第一链cDNA合成试剂盒是合成cDNA第一链的方便系统,适用于两步法实时定量RT-PCR(RT-qPCR)中的cDNA合成。
试剂盒采用先进的Maxima 反转录酶(RT),该酶源自M-MuL V 逆转录酶的体外进化。
这种酶相比M-MuL V逆转录酶热稳定性高,扩增效果强劲且具有更高的cDNA合成效率。
Maxima第一链cDNA合成试剂盒能够在较高温度下(55-65℃),在很宽的总RNA量范围内(从1 pg到5 μg)合成cDNA。
合成反应能在15-30分钟内完成。
Maxima第一链cDNA合成试剂盒的各组分经预混合,以节约时间和减少移液错误。
试剂盒由Maxima Enzyme Mix、5×反应混合液、无核酸酶的水三部分组成。
Maxima Enzyme Mix包含Maxima反转录酶和Thermo Scientific Ribolock RNase抑制剂。
重组的Ribolock RNase抑制剂能有效保护RNA模板在高于55℃时不被RNase A、B和C降解。
天根 一步法逆转录PCR试剂盒说明书

通用型一步法RT-PCR试剂盒说明书前言本试剂盒适用于对各种动、植物、病毒RNA进行PCR 检测。
用户可根据被分析基因,配合一对引物,或同时采用Taqman 荧光探针,先将提取的RNA反转录(reverse transcription).成cDNA,再经过聚合酶链式反应(Polymerase Chain Reaction)技术对特异性片段进行扩增,进行电泳或实时荧光分析。
一步法RT-PCR可使RT及PCR过程在单管中完成,比分开进行RT及PCR更方便。
由于不需要在RT完成后打开反应管,尽可能地避免了样品间的相互污染。
规格20人份适用仪器适用于各种普通PCR仪器和实时荧光PCR仪器。
试剂盒组成试剂准备根据下表配制反应液:振荡混匀后,按每管 40 μl(可根据需要调整)分装,备用。
PCR扩增将均一化后的RNA提取样本10 μl加入各反应管,混匀、离心后,置普通PCR仪器或实时荧光PCR仪器上进行扩增及实时检测。
结果判断扩增产物可直接进行电泳检测;实时荧光检测可根据相应仪器的配套软件进行结果分析。
保存及有效期-20℃保存,有效期为6个月。
注意事项1. 开始检测前请仔细阅读本说明书全文。
2. 整个检测过程中,反应体系的配制、样本处理及加样、PCR扩增(荧光检测)应分区进行以避免污染。
3. 操作人员应戴口罩,经常更换一次性手套,以避免RNA酶的污染;实验中所用器具均应经过除RNA酶处理。
4. 试剂盒组成中的试剂使用前应充分融化并混匀。
5. 进行实时荧光分析时,应使用透光性能较好的一次性薄壁离心管;.荧光探针应避光保存,加入缓冲液中后,应尽快进行扩增。
6. 注意适当处理检测中遗留的样品、扩增产物及可能被污染的试剂。
生产企业:上海蓝创生物科技发展有限公司。
cDNA第一链合成试剂盒

cDNA 第一链合成试剂盒操作方法1、逆转录反应( 1)在无菌薄壁管中加入以下成份:Total RNA 0.5-1.0 μg(dN) 9 or oligo(dT) 18 100pmol( 2)混匀后, 70℃变性 5min,将薄壁管迅速置于冰上冷却,然后依次加入以下成份:5×M-MLV Buffer 4μldNTPs( 10mM each)RNasin 1μl 10U100mM DTTM-MLV Reverse Transcriptase 2μl 100UDEPC-treated water up to 20 μl( 3)混匀后短暂离心,使用oligo(dT) 18 引物时在42℃,使用随机引物(dN) 9 时在37℃下温育 1 小时。
(4) 94 ℃ 5min 终止反应后,冰上冷却。
(5)合成的 cDNA第一链可直接用于 PCR扩增或进行其它下游操作。
2、 PCR扩增( 1)在 PCR薄壁管中加入以下试剂Synthesized cDNA 2-5 μl10×PCR Buffer 2μlUpper primer 10pmolLower primer 10pmoldNTPs(10mM each)0.5 μlTaq DNA Polymerase 1.5UddH2O up to 20 μl( 2)混匀后短暂离心,然后进行PCR扩增。
94℃预变性 2.5min 。
进入下列30~40 个循环(此扩增条件仅供参考):94℃30s, 60℃ 45s ,72℃ 1min ~3min。
最后 72℃延伸 7min。
注意事项1、确保 RNA完整性及纯度。
RNA质量是决定合成cDNA第一链成功的关键因素,其纯度及完整性可由 OD260/OD280比值和进行琼脂糖凝胶电泳检测。
较纯的 RNA 样品 OD260/OD280比值通常为 1.8-2.0 ,若该比值偏低,应进一步纯化。
真核生物RNA样品电泳图谱 28s 和 18s 的比值约为 2:1 ,否则,表明有 RNA降解。
ThermoScientificRevertAidFirstStrandcDNASynthesisKitK说明书第一链cDNA合成试剂盒

RevertAid第一链cDNA Synthesis试剂盒#K1621, #K1622分析证明书质量控制#K1621Lot采用100 fg对照GAPDH RNA和对照引物进行RT-PCR反应,通过在1%琼脂糖上进行凝胶电泳和溴化乙锭染色显示得到足够量的496 bp的产物质量认证人:Jurgita Zilinskiene目录页码试剂盒组成 (2)存储条件 (2)产品说明 (2)注意事项 (3)操作步骤 (6)RT-PCR (6)合成cDNA用于克隆………..………………………………………7 实验对照……………………………………………………………….8 问题分析与解决 (10)** 一个单位的RiboLockRNase酶抑制剂抑制5ng RNA酶A 50%的活性。
存储条件试剂盒中所有组分应存储在-20°C。
对照用RNA可存储于-70°C以便长期使用。
产品说明RevertAid第一链cDNA合成试剂盒以mRNA或者总RNA为模板,高效合成第一链cDNA。
本试剂盒使用RevertAid M-MuLV反转录酶,它的RNA酶H的活性与AMV反转录酶相比较低。
该反转录酶可耐受42-50°C温度,合成的cDNA片段长度达13kb。
试剂盒中含有RiboLock 重组RNA酶抑制剂,防止RNA降解,可耐受55°C高温。
试剂盒同时含有oligo(dT)18和随机六聚体引物。
随机六聚体引物与模板非特异性地结合,以总RNA中任何RNA为模板合成cDNA。
oligo(dT)18选择性和RNA 3’po ly(A)配对结合,只以有poly(A)尾巴的mRNA为模板合成cDNA。
使用本试剂盒也可采用序列特异性引物。
合成的第一链cDNA能直接用作PCR或荧光定量PCR的模板,第二链cDNA的合成或线性RNA扩增,也可用于需要用带有放射性或非放射性核苷酸标记第一链cDNA的实验,比如将标记好的第一链cDNA 作为杂交实验中的探针或者用于微阵列分析。
诺唯赞反转录试剂盒 说明书

•
•Thermo 反转录试剂盒K1622使用说明
•Thermo Scientific RevertAid First Strand cDNA Synthesis Kit是一套用于以RNA为模板高效合成第一链cDNA的完整系统,可合成长达13kb 的cDNA。
试剂盒配套提供的RiboLockRNase Inhibitor,可有效防止RNA模板的降解。
试剂盒配套提供oligo(dT)18引物和随机六聚体引物。
oligo(dT)18引物可以选择性地与mRNA中的poly (A)尾结合。
随机六聚体引物无需poly (A)尾,因此可转录mRNA 5’-端区域,也可以无poly
(A)尾的RNA (如micoRNA)为模板合成cDNA。
该试剂盒也可使用基因
特异性引物。
•Thermo 反转录试剂盒K1622使用说明优点:
•合成全长的第一链cDNA,最长可合成13kb的cDNA
•最佳反应温度为42℃
•完全一提供RT反应所需的所有组分
•应用:
•第一链cDNA合成,作为RT-PCR和实时RT-qPCR的模板
•构建全长cDNA文库
•反义RNA合成
•Thermo 反转录试剂盒K1622使用说明
•RevertAid First Strand cDNA Synthesis Kit合成全长cDNA
• 1 μg小鼠心脏总RNA作为模板,用oligo(dT)18 引物进行逆转录反应。
由此合成的cDNA,再作为Taq DNA Polymerase后续PCR扩增的模板。
末端2024 bp片段的成功扩增,表明长度为13.8 kb的全长RN***段得到成功的逆转录。
•。
通脉大生片对肾虚排卵障碍型不孕大鼠卵巢指数和 FSHR mRNA 表达的影响

通脉大生片对肾虚排卵障碍型不孕大鼠卵巢指数和 FSHRmRNA 表达的影响温莹莹;傅春华;弓丽华;黄金珠;周昕【摘要】目的:探讨以“补肾调冲法”立法的通脉大生片对排卵障碍型不孕( ASR)大鼠卵巢FSHR mRNA表达的调节作用。
方法:选取9日龄SD大鼠,给予颈背部皮下注射丙酸睾酮,建立肾虚ASR大鼠模型,根据大鼠阴道涂片结果筛选造模成功大鼠,随机分组。
灌胃5周后处死,取卵巢,计算卵巢指数。
荧光定量RT-PCR检测计算各组大鼠卵巢FSHR mRNA表达量。
结果:与正常组FSHR mRNA表达量比较,模型组降低且差异有统计学意义(P<0.05);与模型组FSHR mRNA表达量比较,通高组、右归丸组上调,差异有统计学意义(P<0.05),通中组、来曲唑组均上调,差异有显著统计学意义(P<0.01),通低组升高,差异无明显统计学意义(P>0.05);通高、中组FSHR mRNA表达量上调幅度明显高于通低组,尤以通中组最显著。
结论:通脉大生片可治疗肾虚排卵障碍型不孕,其机制与调节FSHR mRNA的表达水平,提高卵巢反应性有关。
【期刊名称】《江西中医药》【年(卷),期】2014(000)012【总页数】4页(P23-26)【关键词】通脉大生片;排卵障碍;卵巢指数;FSHR mRNA【作者】温莹莹;傅春华;弓丽华;黄金珠;周昕【作者单位】成都中医药大学四川成都610075;成都中医药大学四川成都610075;成都中医药大学四川成都610075;成都中医药大学四川成都610075;成都中医药大学四川成都610075【正文语种】中文【中图分类】R285.5不孕症是妇科临床的常见病,正成为全球生殖医学领域一个紧迫和日益引起关注的热点与难点问题。
据统计不孕症在我国的发病率约为10%[1],而排卵功能障碍型不孕约占女性不孕症的25%~30%[2]。
以“补肾调冲”立法的通脉大生片是成都中医药大学附属医院的院内制剂,应用于临床50多年,其促排卵作用已被大量临床病例所证实,具有确切疗效[3]。
使用快速反转录试剂盒合成第一链cDNA

RT延伸。
注意事项
1. 下列操作步骤适用于模板量为50 ng-2 μg的总RNA,如果总RNA量大于2 μg,请按比例扩 大反应体系。
2. 在冰上进行操作,防止RNA发生降解。 3. 不需分开变性和退火两个步骤。但是对于二级结构很复杂的RNA模板,推荐使用变性步
KR116-0μl 25 μl 50 μl 1 ml
KR116-02 (100 rxn)
200 μl 200 μl 100 μl 200 μl 2×1 ml
KR116-03 (1000 rxn) 10×200 μl 10×200 μl 10×100 μl 10×200 μl 10×2×1 ml
操作步骤
使用快速反转录试剂盒合成第一链cDNA
50 ng-2 μg总RNA可建立20 μl反应体系。
1. 将模板RNA在冰上解冻;5×gDNA Buffer、FQ-RT Primer Mix、10×King RT Buffer、 RNase-Free ddH2O在室温(15-25℃)解冻,解冻后迅速置于冰上。使用前将每种溶液 涡旋振荡混匀,简短离心以收集残留在管壁的液体。
以下操作步骤请在冰上进行。为了保证反应液配制的准确性,进行各项反应时,应先配 制成Mix,然后再分装到每个反应管中。
2. 按照表1的基因组DNA的去除体系配制混合液,彻底混匀。简短离心,并置于42℃,孵育 3 min。然后置于冰上放置。
表1 gDNA去除反应体系
组成成分
使用量
5×gDNA Buffer
产品特点
反转录效率高:反转录效率可达95%以上; 操作简单快速:反应体系配制简单,21 min完成cDNA第一链的合成; 通读复杂模板:能够作用于GC含量高,二级结构复杂的RNA模板; 样品普适性高:对不同物种来源及杂质较多的RNA模板的适用性高; 后续兼容性好:后续配合荧光定量检测产品,灵敏度高、稳定性好。
cdna_synthesis_kit

0.2-2 μg 1 μl 1 μl 4在PCR仪上按以下条件进行反转录反应:
30°C
5min
42°C 30-40min
95°C
5min
4°C
产品组分
组分名称 M-MuLV 反转录酶 (200 U/μl) 5XRT PCR Buffer dNTP Mixture (10 mM each) Rnase Inhibitor (40 U/μl) Oligo (dT)15 Primer (20 μM)
cDNA 第一链合成试剂盒 1st cDNA Synthesis Kit
使用说明书
Cat. No.: D0401 包装量:50次
产品说明
本试剂盒采用莫洛尼氏鼠白血病病毒反转录酶M-MuLV ,该酶通过点突变使RNase H活性缺失,而保持完全的 mRNA-directed DNA多聚合活性,有效增强了cDNA的 合成效率、增加了该酶与模板/引物的亲和力, 从而增 加了酶的活性和持续合成能力。本试剂盒优化了反应 Buffer,能使反应的模板量(Total RNA)可在0.2-2 μg 范围之间进行逆转录反应。cDNA合成后进行PCR扩增 既可检测到长cDNA(>10 kb)片段,也可检测到低丰 度cDNA样品。本试剂盒提供了由Total RNA或mRNA合 成cDNA第一链所需要的全部试剂,并提供两种cDNA合 成引物。合成的第一链cDNA可广泛用于2nd Strand的合 成、杂交、PCR扩增、Real-Time PCR反应等。
Rnase Free H2O
储存条件
-20°C保存,避免反复冻融。
数量 50 μl 250 μl 50 μl 25 μl 50 μl
1 ml
操作方法
1.按以下组分配制反转录反应液
Thermo Scientific Rever id First Strand cDNA Synthesis Kit K 说明书 第一链cDNA合成试剂盒

#K1621, #K1622
分析证明书 #K1621 Lot
质量控制
采用100 fg对照GAPDH RNA和对照引物进行RT-PCR反应,通过在1%琼脂糖上进行凝胶电泳和溴 化乙锭染色显示得到足够量的496 bp的产物
质量认证人:Jurgita Zilinskiene
RT-PCR…………………………………………………………….6 合成cDNA用于克隆………..………………………………………7 实验对照……………………………………………………………….8 问题分析与解决……………………………………………………...10
Page 1
试剂盒成分
RevertAid™第一链cDNA合成试剂盒
2. 37°C 孵育 30 分钟。
3. 加入1 μl 50 mM EDTA,65°C 孵育10分钟。在含有二价阳离子(1)而缺乏螯合剂的环境中,RNA
产品说明
RevertAid™第一链cDNA合成试剂盒以mRNA或者总RNA为模板,高效合成第一链cDNA。本试剂 盒使用RevertAid™ M-MuLV反转录酶,它的RNA酶H的活性与AMV反转录酶相比较低。该反转录酶可 耐受42-50°C温度,合成的cDNA片段长度达13kb。
试剂盒中含有RiboLock™ 重组RNA酶抑制剂,防止RNA降解,可耐受55°C高温。 试剂盒同时含有oligo(dT)18和随机六聚体引物。随机六聚体引物与模板非特异性地结合,以总RNA 中任何RNA为模板合成cDNA。oligo(dT)18选择性和RNA 3’poly(A)配对结合,只以有poly(A)尾巴的 mRNA为模板合成cDNA。使用本试剂盒也可采用序列特异性引物。 合成的第一链cDNA能直接用作PCR或荧光定量PCR的模板,第二链cDNA的合成或线性RNA扩增, 也可用于需要用带有放射性或非放射性核苷酸标记第一链cDNA的实验,比如将标记好的第一链cDNA 作为杂交实验中的探针或者用于微阵列分析。
PCR - 操作

RT-qPCR步骤组织取材:各组大鼠分别于造模后6d、12d、18d断头处死,冰上取大鼠脊髓L4-6,PBS液洗净,置液氮内冻存1个小时以上,-80℃保存。
总RNA提取准备工作1)试剂和材料:无水乙醇、氯仿、Glycogen(可能需要)、1.5ml Eppendorf管(RNase-free)、Tips(RNase-free)(管盖与管壁都需标记样品名称)2)75%乙醇、Trizol、氯仿、异丙醇预冷。
3)手套:取RNase-free的物品时必须戴手套。
4)擦洗取液器:DEPC配制的70%乙醇擦洗取液器的内部和外部。
5)试验前将生物安全柜UV及标本处理间UV打开照射30分钟。
操作步骤(所有操作均在冰上进行)1)加Trizol和样本到1.5ml EP管:●组织:(剪取米粒大小)加Trizol后,室温放置5min,使其充分裂解。
注:此时可放入-70℃长期保持。
●贴壁细胞:不须消化,可直接用Trizol进行消化、裂解,Trizol体积按10cm2/ml。
将培养皿中的培养液彻底洗干净,将1 ml Trizo直接加在培养皿中的细胞上。
●悬浮细胞:可直接收集、裂解,每1ml Trizol可裂解5×106动物。
800 g离心10 min后彻底弃上清。
加入1 ml Trizol Reagent,立即用移液器抽打5 次。
2)离心:12,000rpm 离心5min,弃沉淀。
3)氯仿:按200ul氯仿/ml Trizol加入氯仿,振荡混匀后室温放置15min。
注:禁用漩涡振荡器,以免基因组DNA断裂。
4)离心:4℃12,000g离心15min。
5)取上清:吸取上层水相,至另一离心管中。
注:千万不要吸取中间界面;若同时提取DNA和蛋白质,则保留下层酚相存于4℃冰箱,若只提RNA,则弃下层酚相。
6)异丙醇:按0.5ml异丙醇/ml Trizol 加入异丙醇混匀,室温放置5-10min。
7)离心:4℃12,000g离心10min。
一管式双链cDNA合成试剂盒说明书
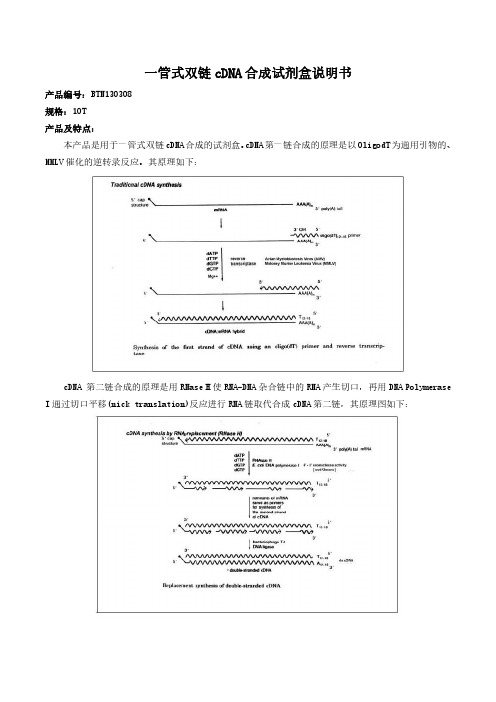
一管式双链cDNA合成试剂盒说明书产品编号:BTN130308规格:10T产品及特点:本产品是用于一管式双链cDNA合成的试剂盒。
cDNA第一链合成的原理是以OligodT为通用引物的、MMLV催化的逆转录反应。
其原理如下:cDNA第二链合成的原理是用RNase H使RNA-DNA杂合链中的RNA产生切口,再用DNA Polymerase I通过切口平移(nick translation)反应进行RNA链取代合成cDNA第二链,其原理图如下:本产品具有下列特点:1.即开即用,含有合成两条cDNA链的所有试剂。
2.适用于含poly rA的RNA,对不含poly rA的RNA(如细菌RNA),不能使用本试剂盒制备ds cDNA。
3.一管式进行,第一链cDNA合成后不需要任何处理,直接进入第二链的合成。
4.所得双链cDNA可以直接用于后续的常规PCR、real-time PCR、cDNA文库构建、抑制性差减杂交等。
5.本试剂盒足够10次80μL体系的双链cDNA合成反应。
6.如果需要合成全长cDNA,最好选用SMART全长cDNA双链合成试剂盒。
规格及成分:成分规格Oligo dT引物10μlcDNA第一链合成缓冲液40μlMMLV逆转录酶(200U/μL)10μl5×cDNA第二链合成缓冲液160μl20×cDNA第二链合成酶混合液40μlT4DNA聚合酶20μl0.2M EDTA溶液40μl超纯水1ml说明书1份保存条件:低温运输,-20℃保存,有效期一年。
自备试剂:mRNA提取试剂盒使用方法:一:mRNA的制备本试剂盒不提供mRNA提取试剂,用户需自备相关试剂。
二:双链cDNA的合成1.在一个干净的塑料离心管中,按下表设置cDNA合成反应:成分用量自备mRNA样品4μL(0.5-2μg)Oligo dT引物1μL70℃2分钟后室温放置2分钟,短暂离心cDNA第一链合成缓冲液4μLMMLV逆转录酶1μL轻柔吹打混匀后42℃保温90分钟合成第一链cDNA超纯水48μLcDNA第二链合成缓冲液16μLcDNA第二链合成酶混合液4μL轻柔吹打混匀后,短暂离心,16℃保温2小时自备的T4DNA聚合酶2μL轻柔吹打混匀后,16℃继续保温30分钟0.2M EDTA溶液4μL注:如果不需要将双链cDNA平末端化,则可以跳过T4DNA聚合酶处理步骤。
MagMAX预分装病毒RNA DNA提取试剂盒说明书

MagMAX 预分装病毒 RNA/DNA 提取试剂盒说明书储存条件及有效期试剂于15°C - 30°C 储存。
生产日期及使用期限见试剂盒外包装。
适用仪器适用于生命科技公司(Life Technologies)生产的KingFisher™ Flex 全自动核酸提取仪(货号:A5400630、C5400630)。
样本要求适用样本类型:全血、血清、血浆、口腔液、组织/器官、环境拭子等样本。
样本采集:按照各样本类型常规采集方法进行采集。
样本保存和运输:采集后的样本可立即用于核酸提取,或2°C- 8°C 保存(不超过24小时),长期保存应置于-20°C 以下,避免反复冻融。
样本运送可采用加冰密封进行运输。
检验方法1. 需自备的仪器、试剂、耗材产品名称MagMAX™预分装病毒RNA/DNA 提取试剂盒包装规格包装规格:预分装2×96次反应/盒货号:A52990预期用途用于核酸提取和纯化步骤。
本试剂盒仅供科研使用(包括动物和环境用途)。
提取产物类型病毒核酸(RNA 和DNA)检验原理采用磁珠法核酸分离技术,携带正电荷的磁珠易于吸附负电荷的核酸,主要用于从样本中提取DNA 和RNA 。
将液体样本与结合缓冲液、蛋白酶K 和磁珠混合。
核酸与磁珠结合后,经过数次洗涤与磁场捕获,洗脱下来的核酸即可通过PCR 或其它特定方法来检测特定的RNA 或DNA 。
磁分离法是广泛应用于动物健康等行业且能实现高通量、自动化的核酸提取方法。
主要组成成分2.3.3 在程序运行完之后(约25分钟),立刻取出洗脱液板,提取完成。
注意:为了防止蒸发,尽快将洗脱液板里的上清液转移到无核酸酶的板或者管中,立刻用于下一步实验。
或者用一张新封膜封好洗脱液板,放入-80°C冰箱保存。
所有使用完毕的深孔板用封膜封好丢弃。
5. 制作样本板5.1 撕去预分装结合缓冲液深孔板的封膜,向其每个孔中加入蛋白酶K/IPC 混合物(在步骤4制备)。
Thermo Scientific Maxima H Minus First Strand cDNA

PRODUCT INFORMATIONThermo ScientificMaxima H Minus First Strand cDNA Synthesis Kit with dsDNase#K1682 100 rxns Lot __ Expiry Date __Store at -20°C/onebioCOMPONENTS OF THE KITMaxima H Minus FirstStrand cDNA Synthesis Kit with dsDNase#K1681 20 rxns #K1682 100 rxns Maxima H Minus Enzyme Mix 25 µL 120 µL 5X RT Buffer250 mM Tris-HCl (pH 8.3 at 25°C), 375 mM KCl, 15 mM MgCl 2, 50 mM DTT150 µL500 µL10 mM dNTP Mix 50 µL 250 µL Oligo(dT)18 Primer, 100 µM 25 µL 120 µL Random Hexamer Primer,100 µM25 µL 120 µLdsDNase 20 µL 2 x 50 µL 10X dsDNase Buffer 100 µL 100 µL Water, nuclease-free 1.25 mL 2 ⨯ 1.25 mLCERTIFICATE OF ANALYSISFunctional testsRT-PCR using 100 fg of control GAPDH RNA and GAPDH control primers generated a prominent 496 bp product on 1% agarose gel after ethidium bromide staining.dsDNase is functionally tested in removal of genomic DNA contamination from RNA sample and subsequent RT-qPCR amplification. Efficiency of genomic DNA removal is ≥99.85%. RNA quantity is not affected by dsDNase treatment. Quality authorized by:Jurgita ZilinskieneRev.1DESCRIPTIONThermo Scientific™ Maxima™ H Minus First Strand cDNA Synthesis Kit with dsDNase provides a dramatically simplified workflow that combines genomic DNAelimination and cDNA synthesis into one-tube procedure. The kit contains a novel double-strand specific DNase (dsDNase) engineered to remove contaminating genomic DNA from RNA preps in 2 minutes without damage to quality or quantity of RNA. Highly specific dsDNase activity towards double-stranded DNA ensures that RNA and single-stranded DNA (such as cDNA and primers) are not cleaved and dsDNase treated RNA can be directly added to reverse transcription.The kit uses the Maxima H Minus Reverse Transcriptase (RT), an advanced enzyme derived by in vitro evolution of M-MuLV RT. The enzyme features the highestthermostability among the derivatives of M-MuLV RT and lacks the RNase H activity. The Maxima H Minus First Strand cDNA Synthesis Kit is capable of synthesizing cDNA up to 20 kb from a wide range of total RNA amounts (1 pg to 5 µg) at elevated temperatures (50-65 °C)superceeding other systems in ability to prepare full length cDNA. Due to increased synthesis rate reaction can be completed in 30 min. Maxima H Minus Enzyme Mix contains Maxima H Minus Reverse Transcriptase and Thermo Scientific™ RiboLock™ RNase Inhibitor. The recombinant RiboLock RNase Inhibitor effectively protects RNA from degradation by RNases A, B and C at temperatures up to 55 °C.The kit is supplied with both oligo(dT)18 and random hexamer primers. Random hexamer primers bind non-specifically and are used to synthesize cDNA from all RNAs in total RNA population. The oligo(dT)18 primer selectively anneals to the 3’-end of poly(A) RNA,synthesizing cDNA only from poly(A) tailed mRNA. Gene-specific primers may also be used with the kit to prime synthesis from a specified sequence.The synthesized cDNA can be used directly in PCR with a variety of thermostabile DNA polymerases, in qPCR with Thermo Scientific Maxima qPCR Master Mixes or in second strand cDNA synthesis.IMPORTANT NOTESAvoiding ribonuclease contaminationRNA purity and integrity are essential for synthesis of full-length cDNA. RNA can be degraded by RNase A, which is a highly stable contaminant found in any laboratoryenvironment. To prevent contamination both the laboratory environment and all prepared solutions must be free of RNases.General recommendations to avoid RNase contamination: ∙ Use certified nuclease-free labware or DEPC-treat all tubes and pipette tips to be used in cDNA synthesis. ∙ Wear gloves when handling RNA and all reagents, as skin is a common source of RNases. Change gloves frequently.∙ Use RNase-free reagents, including high quality water (e.g., Water, nuclease-free, #R0581).∙ Keep the kit components tightly sealed when not in use. Keep all tubes tightly closed during the reverse transcription reaction.Template RNATotal cellular RNA isolated by standard methods issuitable for use with the kit. Purified RNA must be free of salts, metal ions, ethanol and phenol to avoid inhibiting the cDNA synthesis reaction.RNA sample qualityAssess RNA integrity prior to cDNA synthesis. Total eukaryotic RNA can be analyzed by agarose gelelectrophoresis followed by ethidium bromide staining. The RNA is considered to be intact, if both 18S and 28S rRNA appear as sharp bands after electrophoresis of total RNA. The 28S rRNA band should be approximately twice as intense as the 18S rRNA. Any smearing of rRNA bands is an indication of degraded mRNA. If this occurs, a new sample of total RNA should be prepared. Alternatively, total RNA can be analyzed using a bioanalyzer (e.g., Agilent 2100) which provides quantitative information about the general state of the RNA sample, the RNA integrity number (2). A reference gene/target gene 3’:5’ integrity assay (3) can also be used to determine the integrity of the RNA sample.RNA quantity∙ Use 1 pg - 5 µg of total RNA, 0.1 pg - 500 ng of poly(A) mRNA or 0.01 pg – 500 ng of specific RNA transcript to generate first strand cDNA as the initial step of a two-step RT-PCR protocol.∙ Use 1 µg of isolated mRNA to generate first strand cDNA for second-strand synthesis and subsequent cloning reactions.PrimersSynthesis of first strand cDNA can be primed with either oligo(dT)18 primer, random primers or gene-specific primers.Oligo(dT)18 primes cDNA synthesis from the poly(A) tail present at the 3’-end of eukaryotic mRNA. Random primers initiate cDNA synthesis from the total RNA population (rRNA and mRNA). Therefore, using random primers for first strand synthesis results in a greater complexity of the generated cDNA compared with the oligo(dT)18 primer. As a consequence, the sensitivity and specificity of subsequent PCR reactions may be reduced. However, there are several applications where it isbeneficial to use random primers, such as cDNA synthesis using mRNAs without a poly(A) tail, or cDNA synthesis using poly(A)-enriched RNA samples.Gene-specific primers are used to synthesize specific cDNA from a pool of total RNA or mRNA and must be obtained by the user.CONTROL REACTIONS for RT-PCR / RT-qPCRThe following negative control reactions should be used to verify the results of the first strand cDNA synthesis.∙ Reverse transcriptase minus (RT-) negative control is important in RT-PCR and RT-qPCR reactions to assess for genomic DNA contamination of the RNA sample. The control RT- reaction should contain allreagents for the reverse transcription reaction except the Maxima H Minus Enzyme Mix.∙ No template control (NTC) is important to assess for reagent contamination. The NTC reaction should contain all reagents necessary for the reverse transcription reaction except the RNA template.(continued on reverse page)PROTOCOLSPlease read the IMPORTANT NOTES section before starting. After thawing, mix and briefly centrifuge the components of the kit. Store on ice.Genomic DNA elimination1.Add the following reagents into a sterile, RNase-freetube on ice:10X dsDNase Buffer 1 µLdsDNase 1µLTemplate RNA total RNAor poly(A) mRNAor specific RNA1 pg - 5 µg0.1 pg – 500 ng0.01 pg - 500 ngWater, nuclease-free To 10 µL2.Mix gently and spin down.3.Incubate for 2 min at 37°C in the preheated thermomixer or water bath.4.Optional. If If RNA sample is to be used for RT-PCR amplification of long fragments (≥ 3 kb), perform dsDNase inactivation by incubating the sample at 55°C for 5 min in the presence of 10 mM DTT.5.Chill on ice, briefly centrifuge again and place on ice.6.Add first strand cDNA synthesis reagent to the same tube according the following RT-PCR or RT-qPCR protocols.RT-PCRI.First Strand cDNA Synthesis.The following protocol is optimized to generate first-strand cDNA for use in two-step RT-qPCR.1.After genomic DNA elimination, add the following reagents into the same tube in the indicated order:Primer oligo (dT)18 primeror random hexamerprimeror gene-specificprimer1 µL (100 pmol)1 µL (100 pmol)2-20 pmol10 mM dNTP Mix 1 µL (0.5 mM final concentration)Water, nuclease-free to 15 µL2.Optional. If the RNA template is GC-rich or is known to contain secondary structures, mix gently, centrifuge briefly and incubate at 65 °C for 5 min. Chill on ice, briefly centrifuge again and place on ice.3.Add the following components in the indicated order: 5X RT Buffer 4 µLMaxima H Minus Enzyme Mix 1 µLTotal volume 20 µL Mix gently and centrifuge. Incubate:– if an oligo(dT)18 primer or gene-specific primer is used,incubate for 30 min at 50 °C.– if a random hexamer primer is used, incubate for 10 minat 25 °C followed by 30 min at 50 °C.– for transcription of GC-rich RNA, the reactiontemperature can be increased to 65 °C.4.Terminate the reaction by heating at 85°C for5 minutes.The reaction product of the first strand cDNA synthesiscan be used directly in PCR, or stored at -20 °C for up toone week. For longer storage, -70 °C is recommended.Avoid freeze/thaw cycles of the cDNA.II.PCRThe product of the first strand cDNA synthesis reactioncan be used directly in PCR. The volume of the first strandcDNA synthesis reaction mixture should not comprisemore than 1/10 of the total PCR reaction volume.Normally, 2 µL of the first strand cDNA synthesis reactionmixture is used as template for subsequent PCR in a 25µL total volume. Taq DNA polymerase, PCR Master Mix(2X) or Maxima Hot Start PCR Master Mix (2X) can beused to amplify fragments less than 3 kb. ThermoScientific™ DreamTaq™ DNA polymerase is suitable foramplification of longer fragments up to 6 kb. ThermoScientific™ Phusion™ High-Fidelity DNA Polymerases arerecommended to generate amplicons up to 20 kb.RT-qPCRI.First Strand cDNA SynthesisThe following protocol is optimized to generate first-strandcDNA for use in two-step RT-qPCR.1.After genomic DNA elimination, add the followingreagents into the same tube in the indicated order:Primers oligo (dT)18 primerand random hexamerprimer orgene-specific primer0.25 µL (25 pmol)0.25 µL (25 pmol)2-20 pmol10 mM dNTP Mix 1 µLWater, nuclease-free to 15 µL2.Optional. If the RNA template is GC-rich or is known tocontain secondary structures, mix gently, centrifugebriefly and incubate at 65 °C for 5 min. Chill on ice,briefly centrifuge again and place on ice.3.Add the following components to the reaction tube in theindicated order:5X RT Buffer 4 µLMaxima H MinusEnzyme Mix1 µLTotal volume 20 µLMix gently and centrifuge.4.Incubate for 10 min at 25°C followed by 15 min at 50 °C.5.Terminate the reaction by heating at 85 °C for5 minutes.Note. For RNA template quantities greater than 1 µg,prolong the reaction time to 30 min. For RNA templatesthat are GC-rich or have a large amount of secondarystructure, the reaction temperature can be increased to 65°C.The reverse transcription reaction product can be useddirectly in qPCR, or stored at -20 °C for up to one week.For longer storage, -70 °C is recommended. Avoidfreeze/thaw cycles of the cDNA.II.qPCRThe product of the first strand cDNA synthesis reactioncan be used directly in qPCR. The volume of first strandcDNA synthesis reaction mixture should not comprisemore than 1/10 of the total PCR reaction volume. Normally,2 µL of the RT mixture is used as template for subsequentqPCR in 25 µL total volume. Maxima H Minus First StrandcDNA Synthesis Kit is optimized for use with ThermoScientific™Luminaris™ qPCR master mixes.References1.Wiame, I., et al., Irreversible heat inactivation of DNaseIwithout RNA degradation, BioTechniques, 29, 252-256,2000.2.Fleige, S., Pfaffl, M.W., RNA integrity and the effect on thereal-time qRT-PCR performance, Mol. Aspects Med., 27,126-139, 2006.3.Nolan, T., et al., Quantification of mRNA using real-time RT-PCR, Nat. Protoc., 1, 1559-1582, 2006.Note:This product or the use of this product is covered by US patentapplication US20110065606A1 and correspondingcounterparts. The purchase of this product includes a non-transferable license to use this product for the purchaser'sinternal research. All other commercial uses of this product,including without limitation product use for diagnosticpurposes, resale of product in the original or any modified formor product use in providing commercial services require aseparate license. For further information on obtaining licenses****************************************PRODUCT USE LIMITATIONThis product is developed, designed and sold exclusively forresearch purposes and in vitro use only. The product was nottested for use in diagnostics or for drug development, nor is itsuitable for administration to humans or animals.Please refer to /onebio for MaterialSafety Data Sheet of the product.© 2013 Thermo Fisher Scientific Inc. All rights reserved. Alltrademarks are the property of Thermo Fisher Scientific Inc.and its subsidiaries.TROUBLESHOOTINGLow yield or no product in RT-PCR or RT-qPCRPoor integrity of RNA template.RNA purity and integrity is essential for synthesis andquantification of cDNA. Always assess the integrity ofRNA prior to cDNA synthesis Use freshly preparedRNA. Multiple freeze/thaw cycles of the RNA sampleand synthesized cDNA is not recommended.Follow general recommendations to avoid RNasecontamination and discard low quality RNA.Low template purity (inhibitors in RNA sample).Trace amounts of reagents used in RNA purificationprotocols may remain in solution and inhibit first strandsynthesis, e.g., SDS, EDTA, guanidine salts,phosphate, pyrophosphate, polyamines, spermidine. Toremove trace contaminants, re-precipitate the RNA withethanol and wash the pellet with 75% ethanol.Insufficient template quantity.Increase the amount of RNA template in the first strandreaction to the recommended level. Following DNase Itreatment, terminate the reaction by heat inactivation ofthe enzyme in the presence of EDTA (to bind Mg2+).GC rich template.If the RNA template is GC rich or is known to containsecondary structures, the temperature of the reversetranscription reaction can be increased up to 65 °C.Excess amount of cDNA in qPCR/PCR.Decrease amount of cDNA synthesis reaction inqPCR/PCR. The volume of the cDNA synthesis reactionmixture should not exceed 10% of the final PCRreaction mixture.No product in long RT-PCR (>5 kb)Suboptimal priming.Use oligo (dT)18 primer or gene specific primer.If random primers are used, reduce the amount ofrandom primers to 25 pmol per 20 µL reaction mixture.RT-PCR product in RT-minus controlRNA template is contaminated with DNA.The appearance of a PCR product in the negativecontrol reaction (RT-) indicates that the reaction is stillcontaminated with DNA. To completely remove residualDNA, prolong RNA incubation with dsDNase step up to5 minutes.Note that trace amounts of reagents used in RNApurification protocols (e.g., SDS, EDTA, guanidinesalts, phosphate, pyrophosphate, polyamines,spermidine) may remain in solution and inhibit HL-dsDNase activity. To remove trace contaminants, re-precipitate the RNA with ethanol, wash the pellet with75% ethanol and dissolve in nuclease-free water.。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
产品信息
Thermo Scientific
Maxima第一链cDNA合成试剂盒(适用于RT-qPCR)#K1641,#K1642
/onebio
分析证书
经验证,在两步法RT-qPCR中,对于不同起始量(5 ng-0.5 fg)的RNA逆转录产物,使用Thermo Scientific Maxima SYBR Green/ROX qPCR 预混液进行qPCR扩增。
其反应效率在90-110%之间,曲线斜率在-3.09和-3.58之间,且相关系数>0.99。
质量授权人:Jurgita Zilinskiene
目录页码
试剂盒组分 (4)
贮存条件 (4)
产品描述 (4)
重要提示 (5)
RT-qPCR实验方案 (6)
对照反应 (6)
疑难解答 (7)
参考文献 (8)
试剂盒组分
贮存条件
所有试剂盒成分都应贮存于-20 °C。
产品描述
Thermo Scientific Maxima第一链cDNA合成试剂盒是合成cDNA第一链的方便系统,适用于两步法实时定量RT-PCR(RT-qPCR)中的cDNA合成。
试剂盒采用先进的Maxima 反转录酶(RT),该酶源自M-MuL V 逆转录酶的体外进化。
这种酶相比M-MuL V逆转录酶热稳定性高,扩增效果强劲且具有更高的cDNA合成效率。
Maxima第一链cDNA合成试剂盒能够在较高温度下(55-65℃),在很宽的总RNA量范围内(从1 pg到5 μg)合成cDNA。
合成反应能在15-30分钟内完成。
Maxima第一链cDNA合成试剂盒的各组分经预混合,以节约时间和减少移液错误。
试剂盒由Maxima Enzyme Mix、5×反应混合液、无核酸酶的水三部分组成。
Maxima Enzyme Mix包含Maxima反转录酶和Thermo Scientific Ribolock RNase抑制剂。
重组的Ribolock RNase抑制剂能有效保护RNA模板在高于55℃时不被RNase A、B和C降解。
5×反应混合液包含剩余的反应组分:反应缓冲液、dNTPs、Oligo(dT)18和随机引物。
无核酸酶的水用于反应体系的配制和RNA样本的溶解。
已通过相关检验证明其中不含脱氧核糖核酸内切酶、脱氧核糖核酸外切酶、核糖核酸酶和磷酸酶。
重要提示
避免核糖核酸酶污染
RNA的纯度和完整性对于合成全长cDNA至为重要。
RNA能够被RNase A降解,而在任何实验环境中都能发现这种稳定的污染物。
本试剂盒中所有成分都经过严格检测,确保其不含RNase。
为了避免污染,实验环境以及所准备的试剂溶液都必须不含RNase。
避免RNase污染的一般建议:
∙使用确保无核酸酶的实验器具,或者用DEPC水处理cDNA合成中所用到的反应管和枪头等。
∙整个实验过程中请戴手套,因为皮肤是RNA酶的一个常见来源。
同时实验过程中注意经常更换手套。
∙使用无核酸酶的试剂,包括高纯度的水(如无核酸酶的水,#R0581)。
∙未使用试剂盒时请严格密封保存。
在反转录过程中,所有反应管要确保扣严。
模板RNA
通过标准方法分离的总细胞RNA适用于该试剂盒。
纯化的RNA需确保不含盐分、金属离子、乙醇和苯酚,以防其抑制cDNA合成反应。
对于RT-PCR,模板RNA需确保没有DNA污染。
在cDNA合成之前,可先用无核酸酶的DNase I(#EN0521)去除痕量DNA。
实验中需要逆转录酶负对照反应。
该反应含有除Maxima Enzyme Mix以外的所有其他RT反应组分。
RNA样本质量
在合成cDNA之前需对RNA的完整性进行评估。
真核生物总RNA能够通过琼脂糖凝胶电泳后进行EB染色来分析检测。
如果总RNA电泳后有18S和28S rRNA的锐利条带,可认为RNA是完整的。
28S RNA条带的亮度大约为18S rRNA条带的两倍。
rRNA条带出现任何拖尾现象都表示mRNA存在降解。
如果这种情况发生,需重新制备总RNA样品。
另外,也可用生物分析仪器(如Agilent 2100)分析总RNA,该仪器能对RNA样本的大致状况,RNA的完整数(RIN)(2)进行定量。
内参基因/目的基因3’:5’完整性分析(3)也能用于检测RNA样本的完整性。
引物
Maxima第一链cDNA合成试剂盒包含Oligo(dT)18引物和随机引物,以引导第一链cDNA的合成。
该引物混合物可确保检测低拷贝转录物时的高灵敏性。
RT-qPCR实验方案
开始前请阅读本手册的重要提示部分(第5页)。
第一链cDNA合成
第一链cDNA合成反应可以以单个反应进行,而对于不同的RNA模板,也可以进行一系列的平行反应。
因此,反应混合液既可以通过将各组分溶液单独混合,也可以将RT反应中除了RNA模板外的所有组分制成预混液。
解冻后,轻轻涡旋并短暂离心试剂盒所有组分。
冰上放置。
1.的反应管中。
2.
3.25℃孵育10分钟,之后再50℃孵育15分钟。
注:若RNA模板量大于1 μg,则延长反应时间至30分钟。
若RNA模板富含GC或含有大量的二级结构,可将反应温度提高至65℃。
4.85℃加热5分钟以终止反应。
该反转录反应产物能够直接用于qPCR,或者在-20℃条件下存放一周。
如需长时间贮存,推荐贮存于-70℃。
另外应避免cDNA的反复冻融。
qPCR
第一链cDNA合成的产物能够直接用于qPCR。
加入的合成的cDNA第一链产物体积不应超过PCR反应总体积的1/10。
通常情况下,在25 μl体系的qPCR反应中加入2 μl合成的cDNA第一链作为模板。
Maxima第一链cDNA合成试剂盒适于和Maxima qPCR master mixes (#K0221、#K0231、#K0241、#K0251、#K0261)搭配使用。
对照反应
实验中应利用如下负对照来确认第一链cDNA合成的结果。
Reverse transcriptase minus(RT-)负对照:RT-PCR和RT-qPCR中应利用此负对照来检验RNA样本中基因组DNA的污染情况。
此负对照RT-反应含有除Maxima Enzyme Mix 以外的所有组分。
无模板负对照(NTC):此负对照用来检验试剂污染情况。
NTC反应含有除模板以外的所有组分。
疑难解答
参考文献
1.Wiame, I., et al., Irreversible heat inactivation of DNaseI without RNA degradation, BioTechniques, 29, 252-256, 2000.
2.Fleige, S., Pfaffl, M.W., RNA integrity and the effect on the real-time qRT-PCR performance, Mol. Aspects Med., 27, 126-139, 2006.
3.Nolan, T., et al., Quantification of mRNA using real-time RT-PCR, Nat. Protoc., 1,
1559-1582, 2006.
注:
本产品或使用本产品都必须遵守美国专利申请US20110251080A1及相应的副本。
购买本产品包括购买非转让许可,以便买方可将本产品用于其内部研究。
本产品的所有其他商业用途,包括但不限于诊断用途、原装或改装转售或利用产品提供商业服务,均需要一份单独的许可证。
欲了解更多关于许可申请的信息,请联系@。
产品使用限制
本产品的开发、设计及销售专供研究和体外试验使用。
本产品不得用于诊断试验或药物开发,也不适合向人或动物注射。
了解本产品的材料安全数据表,请浏览/onebio。
©2012 Thermo Fisher Scientific公司,版权所有。
所有其他商标均归Thermo Fisher Scientific公司或其子公司所有。
