10种观赏植物查尔酮合成酶基因生物信息学分析
不同植物查尔酮合成酶CHS基因的生物信息学分析(1)

Bioinformatics Analysis of Chalcone Synzyme ( CHS) Genes in Different Plants
ZHANG Tao ( College of Life Science,Qingdao Agricultural University,Qingdao 266109,China) Abstract: In this paper,the nucleic acid sequences and the conjectural amino acid sequences of chalcone synthase ( CHS) genes registered in GenBank in 10 plant species were analyzed by using bioinformatics method. Several parameters of these sequences,including physicochemical property,signal peptide,transmembrane regions,hydrophobicity or hydrophilicity,functional domains and secondary structures,were detailedly analyzed,and the phylogenetic tree was constructed for CHS amino acid sequences. The results showed that the full - length of ORF of 10 CHS genes was about 1. 2 kb and encoded 399 amino acids. All amino acid sequences of CHS in different plant species contained 1 potential N - glycosylation site ( 32NMSS) ,4 protein kinase c phosphorylation sites ( 69TIR,158SVK,202TFR,359SAK) and 1 chalcone synthase active position ( 161RLMMYQQGCFAGGTVLR) . All the amino acid sequences were hydrophobicity protein,without signal peptide and transmembrane regions. Key words: Chalcone synzyme; Bioinformatics; Function analysis; Structure prediction
樱桃查尔酮合成酶
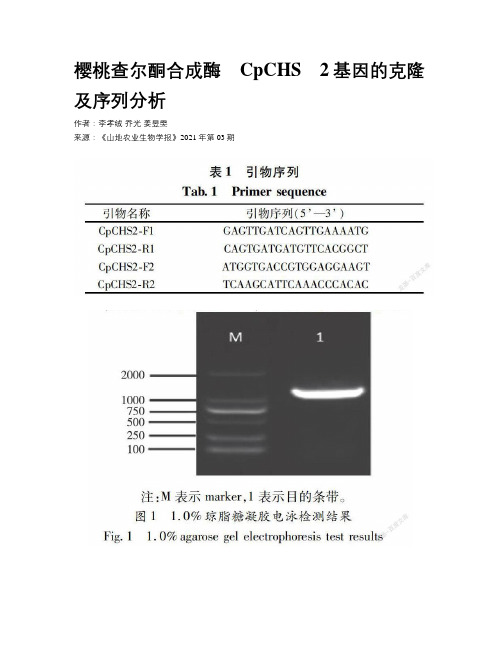
樱桃查尔酮合成酶CpCHS2基因的克隆及序列分析作者:李孝绒乔光姜昱雯来源:《山地农业生物学报》2021年第03期摘要:查尔酮合成酶基因是类黄酮生物合成的关键基因,本文通过巢氏PCR,从‘玛瑙红’樱桃的根系组织中克隆获得一个1176 bp的查尔酮合成酶基因的cDNA序列,编码391个氨基酸,命名为CpCHS2 (基因登录号:MT112906)。
该基因编码的蛋白分子量为42.68 kD,等电点为5.76,属疏水性蛋白且不具有跨膜区,主要定位在细胞质中,二级结构中α-螺旋、β-折叠为蛋白质最大量的结构原件。
系统进化树分析表明,CpCHS2和同属蔷薇科的红叶李及碧桃的亲缘关系最近,并在进化上高度保守。
关键词:樱桃;CpCHS2;基因克隆;生物信息学分析中图分类号:S662.5文献标识码:A文章编号:1008-0457(2021)03-0001-06国际DOI编码:10.15958/ki.sdnyswxb.2021.03.001Cloning and Sequence Analysis of CpCHS2 Gene in Cerasus PseudocerasusLI Xiaorong,QIAO Guang*,JIANG Yuwen(College of Life Science,Guizhou University/Institute of Agro-bioengineering/Key Laboratory of Plant Resources Conservation and Germplasm Innovation in Mountainous Region Ministry of Education,Guiyang,Guizhou 550025,China)Abstract:Chalcone synthetase gene is the key gene for flavonoid biosynthesis.In this paper,a full-length cDNA of the CHS gene was obtained by Nested PCR from the root tissue of Cerasus pseudocerasus ‘Manaohong’,whose complete cDNA was 1,176 bp,encoding 319 amino acids,and being designated as CpCHS2 (Gene accession number is MT112906).The protein encoded by the gene has a molecular weight of 42.68 kD and an isoelectric point of 5.76.It was a hydrophobic protein and did not have a transmembrane region,which was mainly located in the cytoplasm.In the secondary structure,α -helix and β -sheet were the largest structural elements of the protein.The phylogenetic tree analysis showed that the CpCHS2 and Prunus cerasifera and Amygdalus persica of the same genus Rosaceae were closely related and highly conserved in evolution.Keywords:Cerasus pseudocerasus;CpCHS2;gene cloning;bioinformatic analysis類黄酮是一类具有强烈生物活性的多酚类次生代谢产物,它广泛存在于植物中,除影响植物的生长发育,还在与环境相互作用方面起非常重要的作用[1- 2]。
植物查尔酮合成酶分子生物学研究进展

植物查尔酮合成酶分子生物学研究进展河南农业科学植物查尔酮合成酶分子生物学研究进展王燕,许锋,程水源(1,长江大学园艺园林学院,湖北荆州434025;2,黄冈师范学院生命科学与工程学院,湖北黄冈438000)摘要:查尔酮合成酶(chalconesynthase,CHS,EC2.3.1.74),是植物类黄酮物质合成途径中的第一个酶,也是植物次生代谢途径中的关键酶之一,对植物具有非常重要的生理意义.为此,综述了查尔酮合成酶基因结构,基因进化,表达调控机理以及诱导因子,概述了查尔酮合成酶基因工程在植物生理方面的研究,并进一步对查尔酮合成酶的分子生物学研究做了展望.关键词:植物;查尔酮合成酶;分子生物学;基因工程中图分类号:943.2文献标识码:A文章编号:1004—3268(2007)08—0005—05查尔酮合成酶(chalconesynthase,CHS,EC2.3.1.74)是植物类黄酮物质合成途径中的第一个酶.它催化该途径的第一步,即3个分子的丙二酰一CoA和1个分子的对香豆酰一CoA结合形成第一个具有C15架的黄酮类化合物一查尔酮.该产物进一步衍生转化构成了各类黄酮化合物l1].此中间物的异构化和功能基团的进一步取代都能导致黄酮,异黄酮和花色素苷的合成.这些化合物为自然界提供了颜色,并参与了植物的多种生理过程,包括防紫外线辐射,抗病,生长素运输,花粉的育性等,.现已对许多植物查尔酮合成酶基因进行了分离克隆和测序,很多学者通过转基因方法成功地将外源查尔酮合成酶基因导入植物,提高了转基因植物查尔酮合成酶活性,并且这些植物都表现出了目的生理性状.而且查尔酮合成酶在植物中是普遍存在的,它的分子进化存在多种途径,也有许多查尔酮合.成酶基因的分子进化途径的研究报道.在此基础上对国内外植物查尔酮合成酶分子生物学及基因工程的研究进展进行了综述.1查尔酮合成酶基因结构自从第一个荷兰芹的chs序列在l983年发表以来,到目前为止,已从多种双子叶,单子叶和裸子植物中克隆了s基因,例如,玉米,高粱,兰花,矮牵牛引,拟南芥,金鱼草l.],豆类~.和松树|l等.所有报道的chs基因都属于多基因家族,chs基因在结构上非常保守,除金鱼草的1个chs基因AMCHS含有2个内含子外[1o2.其余的chs 均只包含1个内含子和2个外显子.而且这个内含子的位置在已发现的序列中均相同,即位于第65位(以欧洲赤松PinusSylvestris的PSCHS为标准)的半胱氨酸密码子内第一和第二位碱基之间,其长度从几十碱基对到几千碱基对不等.外显子l较短,只编码约60个氨基酸,且长度变异较大;外显子2编码约340个氨基酸,在进化中较保守,易于排序,提供的进化信息较多|1.王金玲等的研究也表明,可用CHS基因外显子2代表全基因进行研究口. Koes等研究矮牵牛的CHS基因家族包括8~l0个成员,在植物正常发育中仅CHs—A和CHs—J在花中表达,前者转录mRNA占CHS总mRNA的90[8l2查尔酮合成酶基因进化CHS在植物中是普遍存在的,现认为最早在陆生植物中出现,例如,轮藻纲和苔藓植物『2.有资料显示,CHS是从脂肪酸代谢途径中的一个酶进化而来.越来越多的证据显示,在进化的过程中,CHS的功能有过多次转变,例如,不断重复地转变成芪合成酶(stilbenesynthase,STS).Tropf等收稿日期:2007—03—14基金项目:湖北省自然科学基金(2002AB094);湖北省青年杰出人才基金(2003AB014);教育部新世纪优秀人才计划(NCET一04—0746);湖北省教育厅重大科技项目(Z200627002)作者简介:王燕(1967一),女,江苏南通人,吾0教授,主要从事银杏次生代谢分子生物学方面的研究.通讯作者:程水源(1965一).男,湖北天门人,教授,博士生导师,主要从事银杏次生代谢方面的研究.5?2007年第8期通过CHS和STS的进化指出,在进化的历史中,STS曾经几次从CHS独立地进化出来.Lanz等指出,CHS在植物的不同类群中是很保守的,已有数据表明,CHS基因是一个较大的基因家族,其编码区比较保守,长约1.2kb,科之间的氨基酸同源性在7O~90[243.Ursula等首次尝试用CHS基因编码区的DNA序列来研究物种的进化关系引,当时他们只分析了7个种8个序列;王金玲等于2000年共分析了19个科的83个序列_1川.基于最筒约法对所研究的CHS基因外显子2部分DNA序列构建的系统进行自展分析的结果表明:各科序列在分支图上的分布情况不同,对于大部分分科,同一科的序列都形成一组,只有少数科的序列分布在相距很远的分支中,在CHS基因的进化中,经常发生基因重复一分歧(duplication—divergence). 攀枝花苏铁的2个克隆CPA1和CPA5分布在相距很远的分支中,说明CHS基因的重复在裸子植物就已经发生.杨俊波等的研究表明l2:山茶属CHS基因家族在进化过程中已分化为A,B,C3个家族,包括A1,A2,A3,B1,B2,C等6类不同的基因成员.其中只有A2类成员为全部被研究的5种植物所共有,而其他类成员只在部分被研究的植物中发现.所有这些CHS成员具有很高的同源性,在核苷酸水平上同一亚家族内基本上高于9O,不同亚家族间也在78%以上.从推测的氨基酸组成看,山茶属内CHS基因的功能一旦发生了分化,各类成员的碱基替代率会有较大差异.进一步分析认为,该属CHS基因的分化直到近期还在活跃地进行.Ferrerd等也指出不同种的进化式样有一定的差别,这种不同的进化式样可能是物种形成后受不同环境因素影响而形成的.近几年来的研究结果表明,CHS只是植物聚酮化合物合成酶家族中的一个成员.通过功能和序列鉴定这个家族的其他成员包括:STS,ACS,2PS,这些蛋白与CHS的序列同源性在65~75之间. Durbin等指出,CHS非常适合于基因复制的研究和基因家族起源的调查引.但是,目前还没有从假定的早期陆生植物中或者其可能的祖先植物中分离得到CHS及其相关的基因,关于这些植物中CHS 相关蛋白合成的次生代谢产物也没有研究.3CHS基因表达的调控研究3.1CHS表达的调控机理CHS是类黄酮生物合成过程中第一个关键酶,6CHS基因的表达受多种内外因素的调控.将CHS基因的启动子片段与GU5报告基因连接,导入植物细胞,通过转基因,原生质体瞬时表达,定点专一突变等手段对CHS基因的启动子进行研究,在CHS 基因启动子区找到了一些作用元件.正是这些元件以及他们与转录因子之间的相互作用,决定了CHS 基因的表达方式受发育和内外因素的复杂调控【2. 与查尔酮基因表达相关的顺式作用元件与反式作用因子已陆续被发现川,如ACE元件(ACGele—ment)『3l,.,H区(H—box)_33],富含AT元件(A T —richelement)_34I.,沉默子(Silencer)[.,P区(BoxP)[37J,工区和Ⅱ区(BoxI和BoxⅡ)_3.3.2CHS表达的诱导因子查尔酮合成酶往往受不同的发育调控和组织特异性调控,对不同外界刺激的敏感程度也不同. Senebier在18世纪末最早发现类黄酮的生物合成受光的调控,在一些植物中,花色素苷只有在光照下才会产生.Arthur也认为,刺激花色素苷合成最有效的光是蓝光/UV—A和UV—B.CHS的表达产生mRNA受到蓝光,紫外光的调节.在香菜细胞培养中,CHS表达产生最大量mRNA同时需要蓝光和紫外光.在自芥和香菜中,暗生长的幼苗CHS表达产生mRNA受光敏色素调节,而成熟叶片中, UV—B和uV—A/蓝光受体介导CHS表达产生mRNA.拟南芥中,紫外光和蓝光控制幼苗CHS表达产生mRNA和成熟叶组织的CHS表达.光敏色素对幼苗的CHS表达起作用[3.除了受光诱导外,CHS的表达还受病原微生物侵染和机械损伤等各种外界因子所诱导c4¨.4CHS基因工程与植物生理代谢4.1CHS转基因在植物花色的影响方面研究截至目前,大多数花卉新品种都是通过传统育种方法来获得的[4.而传统的方法存在着很多的局限性.遗传工程技术的发展给观赏植物产业的发展开辟了一条新的途径,在大多数植物种类中,类黄酮化合物是最重要的花色素.类黄酮生物合成基因的克隆,为遗传工程手段改变花色奠定了基础.目前,遗传工程技术可以从两个方面来改变花的颜色. 第一:抑制类黄酮生物合成基因的活性,导致中间产物的积累和花色的改变;第二,引入新基因来补充某些品种缺乏合成某些颜色的能力【l4.抑制类黄酮生物合成基因的活性有2种方法:一是通过反义RNA技术将目的基因的反义链连接河南农业科学在启动子后面,并转化植物,使目的基因的表达受到抑制;二是通过向植物中引入额外数量的目的基因拷贝,以共抑制技术方式,使目的基因的表达受到抑制[4.CHS基因的反义抑制和共抑制技术已经在牵牛,天竺葵,菊花和玫瑰中取得了成功].用遗传工程技术改变花色的另一条途径是通过转基因技术.例如,牵牛中的二氢黄酮醇4~还原酶不能把二氢黄酮醇(dihydrokaempfero1)转化为合成花葵素糖苷的中间产物,用传统的方法很难培育出橘红色的牵牛花.Meyer等利用遗传工程技术,将玉米的dfr基因转化进入开白花的牵牛中,使该DFR基因在牵牛中表达,产生的dIr酶能使二氢黄酮醇转化为相应的中间产物,进一步合成花葵素糖苷,培育出了橘红色的牵牛花[4.4.2CHS基因工程在植物育性方面的研究类黄酮与植物的育性有密切的关系.它在花粉中主要的合成部位是绒毡层,然后运输到子囊腔并最后进入花粉粒的外壁,成为组成外壁的一个重要的成分.因此,类黄酮在花粉粒的形成中起着非常重要的作用.研究认为,chs—a转基因植物雄性不育现象的产生可能是由于chs在花药中的转录.邵莉等人将正向chs—a基因转入矮牵牛中,在成功地改变了花的颜色的同时还发现了转基因植物也出现了雄性不育的现象.在玉米中,人们早就发现由于CHS突变(C2, Whp)而产生的不育的白色花粉粒.与此同时,水稻中类CHS基因的异常表达也可能导致花粉粒败育l_4.张毅等研究证明了在水稻花药中特异表达的类查尔酮合成酶基因D5mRNA的积累在四分体时期达到高峰并持续到小孢子时期,而这一时期与外壁的形成密切相关,而且有报道表明,拟南芥的雄性不育突变体msl2即为花粉外壁形成的缺陷型.4.3CHS基因工程在植物防御反应中的研究对于chs在植物防御反应中的调控方式的研究正在两个方向上进行:一是观察植物受到病原微生物侵染后的各种生理变化;另一个是研究与CHS表达相关的调控因子及其基因的调控方式.在病原微生物与植物相互作用的过程中,各种激发因子和抑制因子大都存在于细胞之外,而CHS 及其他与真菌有关的基因的表达均发生在细胞内, 细胞外的信号如何传递到细胞内,是一个引人注目的问题.目前已确证与CHS表达有关的信号传递过程是磷酸肌醇(PI)途径.不少研究结果显示.植物在遭受病原微生物侵染后,CHS活性显着增强,chs活跃转录,这些结果表明,由CHS调控的苯丙烷代谢反应很可能是植物抵抗病原物侵染的重要防卫反应之一[5.齐放军等用水稻抗白叶枯病近等基因系材料及转基因系材料,研究了水稻白叶枯病菌互作中,水稻防卫基因chs的转录特征[5.Northern检测结果显示,在与白叶枯病菌非亲和性互作中,水稻chs基因的转录均不受到诱导或只是受到很微蜀弓的诱导. 表明由chs基因调控的苯丙烷核心反应的启动和产物的合成,有助于增强水稻对白叶桔病菌的抗性.试验结果显示:互作中水稻chs基因是否受到白叶枯病菌的诱导,不仅与水稻中抗病基因有关,而且还与抗病基因的种类有关.表明水稻chs基因在互作中是否受到诱导转录,取决于其水稻功能性抗病基因产物对白叶枯病菌的专化性识别[5.从植物受到病原微生物侵染开始,到chS被诱导表达一系列调控过程至今还缺乏一个完整的认识.相信随着chs这一模式基因在植物防御机制中的作用研究的不断深入,人类不但将进一步认清植物基因调控的机理,而且可以通过利用它们的调控途径来改变某些基因的表达效应,从而为改良作物品种,提高作物抗病能力开辟一条崭新的途径.5展望迄今,有关学者至少对1o余种植物查尔酮合成酶基因进行了转化研究,如烟草,核桃,芸香,水稻,玫瑰,百合,矮牵牛,黄瓜,玉米等,尤其是在观赏植物花色的转基因研究方面最为深入.在未来农业中,开展CHS基因的克隆,结构特点,表达部位和时空表达模式的研究.以便有目的的用之于转化植物,使之在转基因植物中大量,持久地表达,提高目的次生代谢产物的含量,使植物查尔酮合成酶转基因应用进入产业化具有重要意义.参考文献:[1]程水源,颐曼如,束怀瑞.银杏叶黄酮研究进展[J].林业科学,2000,36(6):110一l15.[2]KoesRE,FrancescaQ,JosephNM.Theflavonoid biosyntheticpathwayinplants:functionandevolution 口].Bioessays,l994,16:123—132.[3]MartinCR.Structure,function,andregulationofthe chalconesynthase[C]//.InternationReviewofCytolo gy,l993,147:233—284.[4]ReimoldU,KroegerM,KreuzalerF,eta1.Coding72007年第8期E5J[6]E7]E8][9][io3[11][12][13][14][15]and3noncodingnucleotide thasemessengerRNAandsequenceoftheenzyme[J].18O6.sequenceofchalconesyn—assignmentofaminoacidEMBOJ,1983,2:1801一FrankenP,NiesbachKU.WeydemannU,eta1.The duplicatedchalconesynthasegenesC2andWhp(white pollen)ofZeamaysareindependentlyregulated;evi—dencefortranslationalcontrolofWhpexpressionby theanthocyaninintensifyinggene[J].EMBOJ,1991,10:2605—2612.LoC,CoolbaughRC.NicholsonRI.Molecular characterizationandinsilicoexpressionanalysisof chalconesynthasegenefamilyinsorghumbicolor[J]. PhysiolMolPlantPathol,2002,61:179—188. LiewCF,GohCJ,LohCS,eta1.Cloningandchar—acterizationoffull——lengthcDNAclonesencodingchal—conesynthasefromtheorchidBromheadiafinlaysoni—ana[J].PlantPhysiolBiochem.1998,9:647—655. HohonTA,BruglleraF,TanakaY.Cloningandex—pressionofehalconesynthasefrompetuniahybrida[J]. PlantJ,1993,4:1003—1010.SaslowskyDE,DanaCD,WinkelSB.Anallelicse—riesforthechalconesynthaselocusinArabidopsis[J]. Gene,2000,225:127—138.SommerH,SaedlerH.Structureofthechalcone synthasegeneofAntirrhinummajus[J].MolGen Genet,1986,202:429—434.AkadaS,KungS,DubeSK.Nueleotidesequenceof asoybeanchalconesynthasegenewithapossiblerole inultraviolet--Bsensitivity,gmcbs6[J].PlantPhysi~ol,1993,102:699—702.ArioliT,HowlesPA,WeinmanJJ,eta1.Trifoli- umsubterraneumchalconesynthaseisencodedbya multigenefamily[J].Gene,1994,138:79—86. MckhammHI,HirschAM.Isolationofchalcone synthaseandchalconeisomerasecDNAfromalfalfa (MedicagosativaL.):highesttranscriptlevelsoc~curinyoungrootsandroottips[J2.PlantMolBiol, 1994,24:767—777.SchroderJ,RaiberS,BergerT,eta1.Plant polyketidesynthases:achalconesynthase-typeen~zymewhichperformsacondensationreactionwith methylmalonyl—CoAinthebiosynthesisofC—meth~ylatedchalcones[J].Biochemistry,1998,23:8417—8425.FliegmannJ,SchroderG,SchanzS,eta1.Molecularanalysisofchalconeanddihydropinosylvinsynthase fromScotspine(pinussylvestris),anddifferential regulationoftheseandrelatedenzymeactivitiesin stressedplants[J].PlantMolBiol,1992,3:489—5O3.8?[16][17][18][19][2O][21][22][23][24][25][26][27][283Y angJ,HuangJ,GuH,eta1.Duplicationanda—daptiveevolutionofthechalconesynthasegenesof Dendranthema(Asteraceae)[J].MolBiolEvol, 2002,19:1752—1759.王金玲,瞿礼嘉,陈军,等.cHS基因外显子2的进化规律及其用于植物分子系统学研究的可行性[J].科学通报,2000,45(9):942—950.KoesRE,SpeltceG,V anderE.Clonigandmolecu—larcharacterizationofthechalconesynthasemulti—genefamilyofRetuniohybrida[J].Gene,1989,81:245—157.StaffordHA.Flavonoidevolution:anenzymicap—proaeh[J].PlantPhysiol,1991,96:680—685. KubitzkiK.Phenylpropanoidmetabolisminrelation tolandplantoriginanddiversification[J].Plant Physiol,1987,131:17—24.MarkhamKR.Distributionofflavonoidsinthelower plantsanditsevolutionarysignificance.intheFla—vonoids(JBHarborne,ed)[M].ChapmanandHall, London.1988:427—468.MarkhamKR.Bryophyteflavonoids,theirstruc—tures,distribution,andevolutionarysignificance IN]∥.BrophytesTheirChemistryandChemical Taxonomy(H.D.ZinsmeisterandR.Mues,eds.) OxfordClarendonPress,1990:143—161. TropfS,KarcherB,SchroderG,etaZ.Reaction mechanismsofhomodimericplantpolyketidesyn—thase(stilbenceandchalconesynthase).asingleac—tivesiteforthecondensingreactionissufficientfor synthesisofstilbenes,chalconee,and6~de—oxychalcones[J].JBiolChem,1995,270:7922—7928.LanzT,TropfS,MarnerFJ,eta1.Theroleofcys—teinesinpolyketidesynthasesitedirectetedmutagen—esisofresveratrolandchalconesynthase,twoen—zymesindifferentplant—specificpathways[J].JBiol Chem,1991,266:9971—9976.UrsulaNK,BarzenEB.Chalconesynthasegenein plants:atooltostudyevolutionaryrelationships[J].JMolEvol,1987,26:213—225.Y angJB,TianX,LiDZ,eta1.Molecularcomposi—tionandevolutionofthechalconesynthase(CHS) genefamilyinfivespeciesofcamellia(Theaceae)[J]. ActaBotanicaSinica,2003,45(6):659~666.FerrerJI,JezJM,BowmanME,eta1.Structure ofchalconesynthaseandthemolecularbasisofplant polyketidebiosynthesis[J].Nature,1999,6(8):775—840.DurbinML,McCaigB,CleggMT.Molecularevo—lutionofchaleonesynthasemultigenefamilyinthe morningglorygenome[J2.PlantMolBiol,2000,42:河南农业科学79—92.r29]AkiraNakatsuka,Y okoI,MasumiY.Spatialand temporalexpressionofchalconesynthaseanddi—hydroflavonol4~reductasegenesintheasiatichy—bridlily[J].PlantScience,2003,165(4):759—767.[3O]廖靖军,安成才,吴思,等.查尔酮合成酶基因在植物防御反应中的调控作用[J].北京大学,2000,36 (4):569—571.r3门WeisshaarB,ArmstrongGA,BlockA.Lightinduc—ibleandconstituivelyexpressedDNAbindingproteins recognizingaplantpromoterelementwithfunctional relevanceinlightresponsiveness[J].EMBOJ,1991,10(7):1777—1786.r32]AustinM,NoelJ.Thechalconesythasesuperfamily oftype,polyketidesynthases[J].NatProdRep,2003,20:79—110.[33]YuLM,LambCJ,DixonRA.Purificationand biochemcalcharacterizationofproteinswhichbindto theHboxciselementimplicatedintranscriptionalac—tivationofplantdefensegenes[J].PlantJ,1993,3(6):805—810.[34]KibaA,ToyodaK,IchinoseY.Specificinhibitionof cellwallboundATPasebyfungalsuppressiorfrom mycosphaerellapiodes[J].PlantCellPhysiology, 1995,36:809—817.r35]LawtonMA,DeanSM,DronM,eta1.Silencerre—gionofachalconesynthasepromotercontainsmulti—piebindingsitesforfactor,SBF1,closelyrelatedtoGT[J].PlantMolBiol,1991,16(2):235—249.[36]DaCE,KleinL,SchmelzerE.BPF1,apathogenin—ducedDNAbindingproteininvolvedintheplantde—fenseresponse[J].PlantJ,1993,4:125—135.[37]Y oshiyukiI,HikaruS,KazuhiroT,eta1.Contrary operationsofBox-Ielementofpeaphenylalanineam—monia-lyasegene1promoterfororgan-specificex'' pression[J].PlantPhysiologyandBiochemistry,2001,39(5):355—362.[38]Y amadaT,SriprasertsakP,KatoH.Functionalas—nalysiosofthepromotersofphenylalanineammonial—yasegenesinpeaEJ].PlantCellPhysiol,1994,35:93——104.[39]王曼,王小菁.蓝光,紫外光的受体及其对CHS表达诱导的研究[J].植物学通报,2002,19(3):265一[4O][42][43][44][45][46][473[48][49][5O][51][52]271.DanglJI,HahlborockK,SchellJ.Regulationand StructureofChalconeSynthaseGenes[M]//.NewY ork:AcademicPress,1989:155—173.V anderM,StuitjeAR,MolNJ.Regulationof GeneralPhenylpropanoidandFlavonoidGeneEx—pression[M].CRCPress,BocaRaton,1993:125—155.赵昶灵,郭传明,陈俊愉.植物花色呈现的生物化学, 分子生物学机制及其基因工程改良[J].西北植物学报,2003,23(6):1024—1035.邵莉,李毅,杨美珠,等.查尔酮合酶基因对转基因植物花色和育性的影响[J].植物,1996,38(7): 517—524.V anderM,StamME,TunenA,eta1.Antisense inhibitionofflavonoidbiosynthesisinpetuniaanthers resultsinmalesterility[J].PlantCell,1992,4:253262.ElidaG,FadiC,HamdanF.Cloningofthechapero—nint—complexpolypeptide1genefromSchistosoma mansoniandstudiesofitsexpressionlevels[J].Para—sitolRes,2000,86:253—258.赵云鹏,陈发棣,郭维明.观赏植物花色基因工程研究进展[J].植物通报,2003,20(】):51—58. MeyerP,HeidmannI,ForkmannG.Anewpetunia flowercolourgeneratedbytransformationofamutant withamaizegeneEJ].Nature,1987,330:677~688.郑宏红,瞿礼嘉,刘美华.花药特异性表达的类查尔酮合酶基因D5与水稻花粉发育相关[J].科学通报, 2000,45(11):1132—1138.张毅,瞿礼嘉,刘美华,等.对一个在水稻雄蕊中大量表达的cNDA的结构和表达分析[J].科学通报, 1998,43(4):607~611.TaylarPE,GloverJA,LavithisM.Geneticcontrol ofmalefertilityinArabidopsisthaliana:structuralan—alysesofpostmeioticdevelopmentmutants[J].Plan—ta,1998,2O5:492—505.WannerIA,IiGQ,ThePhenylalanineammonia—lyasegenefamilyinArabidoposisthaliana[J].Plant MolBio,1995,27:328—335.齐放军,高学文,王金生,等.携带不同抗白叶枯病基因的水稻防卫基因pal和chs的转录特征[J].农业生物技术,2000,8(4):337—340.9'。
番茄查尔酮合成酶基因的鉴定及生物信息学分析

Ab s t r a c t F l a v o n o i d s a r e a k i nd o f i mp o r t a n t s e c o n d a r y me t a b o l i t e s i n p l a n t s . Us u a l l y , i t wa s f o u n d i n ru f i t s .
Y a n g Y u e j i a n
I n s t i t u t e o f Ve g e t a b l e s , Z h e j i a n g Ac a d e my o f Ag i r c u l t u r a l S c i e n c e s , Ha n g z h o u , 3 1 0 0 2 1 Co r r e s p o n d i n g a u t h o r , h z y y j @y a h o o . c o m. c n
To ma t o
R u a n Me i y i n g Wa n H o n g j i a n Ye Qi n g j i n g Wa n g R o n g j i n g Ya o Z i L i u Y u n f e i
v e g e t a b l e s , b e a n s , t e a a n d ma ny o t h e r p l a n t s a s c o mb i n a t i o n( l f a v o n o i d g l y c o s i d e S ) o r f r e e s t a t e s ( f l a v o n o i d g l y c o — s i d e s ) or f m. I t h a s i mp o r t nt a r o l e i n r e g u l a t i n g p l a n t g r o w t h nd a d e v e l o p me n t . C h a l c o n e s y n t h a s e , t h e i f r s t k e y
矮牵牛的查尔酮合酶(CHS)基因的生物信息学分析

农学学报
JOURNAL OF AGRICULTURE
· 35 ·
导 法 将 CHS 基 因 反 义 片 断 导 入 非 洲 菊 (Gerbera jamesonii, Bolus.)中,内源 CHS 合成受到抑制,花色素 合成大大受阻,从而使花色发生改变。因此,利用基因 工程技术对 CHS 基因进行调控具有重要意义。近年 来很多物种的 CHS 基因被相继克隆,但利用生物信息 学的方法系统研究这些基因间的进化关系尚未见相关
2.4 氨基酸序列之间的多重比对分析 从图 2 可以看出,CHS 基因的同源性较高,达到
80.55% 。 这 说 明 该 基 因 比 较 保 守 ,有 多 个 同 源 性 达 到 100%的区段,这些区段序列信息将对其他物种中 该 基 因 的 克 隆 具 有 很 重 要 的 参 考 作 用 ,也 为 后 续 利 用 RNA 干扰的转基因方法改变花色提供了保守区 段。此外,在序列信息中还可发现,一些 CHS 基因
(School of Life Sciences, Hunan University of Science and Technology, Xiangtan 411201, Hunan, China)
Abstract: To study the homology and evolutionary relationships among CHS (chalcone synthase) genes, and using the CHS gene in Petunia hybrida as studied object, its base distribution, amino acids composition and hydrophobicity or hydrophilicity were analyzed by the software and bioinformatics web site, as well as multiple comparisons and phylogenetic analysis with other 10 species. The mRNA single sequence contained 1170 bp base, the number of amino acids encoding protein was 389 and its relative molecular mass was 42580.02, leucine gave the highest content of 10.80% , hydrophobic maximum value was 2.038, hydrophobic minimum value was -2.208, and no obvious hydrophilic or hydrophobic area was found. The homology of multiple comparisons of the amino acid sequence for CHS genes in 11 species achieved 80.55%. The genes were in a stable state, encoded protein was hydrophilic protein, and the evolutionary process was conservative. The conservative sequence information achieved in these CHS genes laid a foundation for other new CHS gene cloning. Key words: Chalcone Synthetase Gene; Flower Colour; Bioinformatics; Sequence Analysis
植物查尔酮异构酶的生物信息学分析

摘 要 : 用生物信息学方法和 工具对 G n ak中的洋葱 、 豆 、 采 eB n 豌 番茄和茶 等植物 的黄酮类 化合物合成关键 酶查 尔酮异构酶( HI的核 酸和氨基 酸序 列进行 了比对、 C ) 分析 和建模 , 而对其 进
分子结构 、 理化性质 、 亚细胞 定位 、 白转运肽 、 蛋 跨膜结构域 、 疏水性 、 分子 系统进化 、 白质二级和 蛋
基查 尔酮生成异黄酮类化合物 和黄酮类化合 物 ; 另一种
主要存在 于非豆 科植物 中 , 化 6- 氧查尔酮 生成 5 催 , 脱 羟基黄烷酮[ 。目前 C I 因 已先后 从豌 豆、 】 H 基 矮牵牛 、 苜蓿等多种植物 和人类粪便厌氧细菌中被克隆出来 引, 通过代谢工程的研 究证明 改变 C 活力 可 以有效 调控 HI
关键词 : 查尔酮异构酶 ; 生物信息学 ; 黄酮类化合物
中图分类号 : 4. ; 58 文献标识码 : 文章编号 : 0 -00 (08 0 —0 9 —0 Q 9 65Q 5 A 1 1 09 20 )2 l3 5 0
黄酮类 化合 物是 由苯丙 烷类化 合物衍 生得 到的一
大类植物次生代谓 产物 , } 其特点是具有 C -3C 的基本 6 一6 C 骨架 , 并可根据中间吡喃环 的不 同氧化水 平和两侧 A、 B
核酸及氨基酸序列的分子结构 和理 化性质 分析 、 开放阅
读框的查找 和翻译使用 Vetr T c I o N 8软件完成 ; 核酸及
氨基酸序 列的 同源性 比对使 用 Cut ls lX软 件 和 N B a CI 上的 Bat l 在线工具完成 ; s 分子系统发生树使 用 ME A G 3 的 P y gn h l ey中的 N ih o- i n o e br on g方法构建 ; g J i 亚细胞定 位情况的考察 使用 T re . evr 线 完成 , 对 agt 1 1S re 在 P 并
石榴查尔酮合成酶蛋白的生物信息学分析

石榴查尔酮合成酶蛋白的生物信息学分析作者:冯立娟尹燕雷杨雪梅唐海霞李英朋鹿英来源:《山东农业科学》2019年第02期摘要:本研究用生物信息学方法对石榴及其他19个物种CHS蛋白的系统进化和motif元件进行分析,重点预测分析石榴、巨桉和拟南芥CHS蛋白的理化性质、氨基酸组成、磷酸化位点和二级结构。
结果表明,石榴与巨桉亲缘关系最近,与拟南芥最远。
石榴、巨桉和拟南芥CHS蛋白由20种氨基酸组成,数量和比例存在差异,石榴和拟南芥中亮氨酸数量最多,巨桉中丙氨酸最多;存在丝氨酸、苏氨酸和酪氨酸3个磷酸化结合位点,多数发生在苏氨酸和丝氨酸位置上;二级结构由α-螺旋、延伸链、无规则卷曲和β-转角构成,α-螺旋和无规则卷曲占比较高。
在20个物种CHS蛋白的motif元件中,石榴CHS蛋白motif数量和位置与巨桉完全一致,与大多数物种相似,与拟南芥差异较大。
本研究结果可为深入研究CHS生物学功能及其调控石榴果实呈色的机理提供理论依据。
关键词:石榴; 花青苷;查尔酮合成酶;生物信息学中图分类号:S665.403文献标识号:A文章编号:1001-4942(2019)02-0007-06Bioinformatics Analysis of CHS Protein in PomegranateFeng Lijuan;;Yin Yanlei;;Yang Xuemei;;Tang Haixia;;Li Yingpeng;;Lu Ying3(1.Shandong Institute of Pomology, Taian 271000, China;2. Guan County Dianzi Forestry Station, Guanxian ;252525, China;3.Shiheng Town Forestry and Fruit Station of Feicheng City, Feicheng 27161;China)AbstractThe phylogenetic and motif elements of pomegranate and other 19 cultivars of CHS proteins were analyzed by bioinformatics methods in the study. The physicochemical properties,amino acid composition, phosphorylation sites and second structure of CHS proteins were emphatically analyzed in Punica granatum, Eucalyptus grandis and Arabidopsis thaliana. The results showed that Punica granatum had the closest relationship with Eucalyptus grandis and the farthest relationship with Arabidopsis thaliana. There were twenty amino acids in the CHS proteins of Punica granatum, Eucalyptus grandis and Arabidopsis thaliana, and the number and proportion were different. The leucine number was the highest in Punica granatum and Eucalyptus grandis, and alanine number was the highest in Arabidopsis thaliana. There were phosphorylation binding sites of serine, threonine and tyrosine, respectively, most of them existed in threonine and serine. The secondary structure was consist of alpha helix, extended strand, random coil and beta-turn, in which,the proportion of alpha helix and random coil were higher. In the motif elements of CHS protein of twenty cultivars, the number and location of CHS protein motifs in Punica granatum were identical to Eucalyptus grandis, similar to most cultivars, and significantly different from Arabidopsis thaliana. The results could provide theoretical bases for the further study on biological functions of CHS and mechanism of regulating the coloration of pomegranate fruit.KeywordsPunica granatum; Anthocyanin; Chalcone synthase; Bioinformatics查爾酮合成酶(chalcone synthase,CHS)是植物类黄酮代谢途径中关键限速酶之一,催化香豆酰辅酶A和丙二酰辅酶A合成柚皮素查尔酮,最终代谢成花青苷、黄酮醇和黄烷酮等类黄酮类物质[1-3]。
白木香查尔酮合酶(AsCHS1)基因的克隆和生物信息学分析

香有具行气止痛 、 温 中止呕、 纳气平 喘的功效 , 用于 胸腹 胀 闷 疼 痛 、 胃寒 呕 吐 呃 逆 、 肾 虚 气 逆 喘 急 。 沉香为极珍贵芳香类药材 , 因其产量低 、 价格高 , 远 远 不 能满 足市 场 的需 求 。沉 香 叶 的资 源 丰 富 , 探 讨
沉 香 叶代 替沉 香药 材 的可行性 是 沉香研 究 开发 中 的
一
酮合成和累积等方面 , 有助于明确 白木香沉香中黄
酮合 成 的分 子 机制 , 探 索 利用 沉香 中 C H S基 因提 高 沉香 中 的黄酮 含量 的可 能性 。本研究 通过 高通 量测
序 获得 C H S基 因的 c D N A全 长序 列 , 对其 序 列特 征
进 行 了生 物信 息学分 析 , 通过 荧光 定 量 P C R技术 研
国产 沉香 为瑞 香科 植物 白木 香 A q u i l a r i a s i n e n s i s ( L o u t . ) G i l g含树脂 的木材 。作 为我 国传 统 中药 , 沉
物 的关 键 酶之 一是查 尔酮 合成 酶 ( C H S ) 。通过 研究 查尔 酮合 成酶 基 因的结 构 , 表 达方 式 , 在 沉香 中的黄
2 0 1 0年 5月 于 海 南 省 海 口市演 丰镇 采 集 三 年 生 栽 培 白木 香 A .s i n e n s t s 茎( 次 生木 质部 已完 全 分 化) , 经 中 国 医学 科 学 院药 用 植 物 研 究 所 魏 建 和研
究员鉴定 , 凭证标本保存于 中国医学科学院药用植 物 研究 所海 南分 所 ( 凭 证号 2 0 1 0 0 0 3 ) 。酒精 灯 火焰
白木香 A s C H S 1的基 因编码 区序 列 , 并对 A s C HS 1 蛋 白进 行理 化性质 、 蛋 白二级 结构 及 三维结构 预测 分析 。另 外 , 采用 q R T —
豆科植物查尔酮合成酶基因密码子偏好性分析

Th e r e s e a r c h a b o u t c o d o n b i a s f r o m CHS g e n e s o f l e g u mi n o u s p l a n t s h a s g r e a t s i g n i ic f a n c e t o i n c r e a s e t h e e x p r e s s i o n
C H S基 因 密 码 子 的 偏 好 性 , 对提 高 豆 科植 物 C H S基 因 的表 达 水 平 具 有 重 要 意 义 。 为 此 , 运用 C H I P S 、 C U S P和 C o d o n W
程序分析豆科植物 C H S基 因密 码 子 的偏 好 性 , 并用决 明 C H S基 因对 分 析 结 果 的 可 信 度 进 行 验 证 。结 果 表 明 , 在 豆 科 植物 C H S基 因 的密 码 子 中 , 以 A或 u结 尾 的 密 码 子 偏 好 性 较 强 。基 于 C H S基 因 R S C U值 的 聚 类 结 果 显 示 , 豆 科 植 物
与茄科植物 ( 包括烟 草) 聚为一类 , 表 明 烟草 作 为研 究 豆 科 植 物 C H S基 因功 能 的模 式 植 物 较 为 合 适 。通 过 对 决 明 C H S
基 因 密 码 子 偏 好 性 与 大 肠 杆 菌 和 酵 母 基 因组 密 码 子 偏 好 性 的 比 较 , 发现两 者均存在 差异 , 但 酵 母 的 差 异 低 于 大 肠 杆 菌, 表 明 酵母 表 达 系 统 更 加 适 合 作 为 决 明 C H S基 因 的外 源 表 达 系统 。 然 而 , 若 要使决 明 C H S基 因 能 够 在 酵 母 表 达 系 统 中高 效 表 达 , 仍 需对其密码子进行优化。
百合查尔酮合成酶(chs)基因的cDNA克隆与分析

Ab ta t sr c :The s e ii 5 i e ss n he ie c o d n O c s r e e on e ore p c fc 1rm r y t sz d a c r i g t on e v d r gi s ofr p t d CH S e e g n we e u e O a qu r r s d t c ie ̄H S DNA c by RT— PCR. fa A r gme t o bo 0bp wa bt i d a h n fa ut87 s o a ne nd t e
文 章 编 号 : 0 4 2 8 2 1 ) 1 0 2 —0 1 0 —3 6 (0 1 0 — 1 4 3
Cln n n q e cn fCh lo e S n h s n DNA n lu o i g a d Se u n i g o ac n y t a e Ge ec iLii m
河 南农 业 科 学 , 0 1 4 ( ) 1 4 1 6 2 1 ,0 1 :2 —2
J u n l fHe a rc l r l ce c s o r a n n Ag iut a in e o u S
百 合 查 尔酮 合成 酶 (h ) 因的 c NA克 隆与 分析 c s基 D
s q ncn e uls s ow e h t he f a m e t l n h w a 7 p a d is s qu n e w a v r 91 e ue i g r s t h d t a :t r g n e gt s 8 3b n t e e c s o e
统 育 种 的 重 要 补 充 。RNA 于 扰 技 术 目 前 是 转 基 因
育种 的研究热 点 , 本研 究 根据 百合 C 基 因的保 守 HS
三种蕨类植物查尔酮合成酶(CHS)基因的克隆与分析的开题报告

三种蕨类植物查尔酮合成酶(CHS)基因的克隆与分析的开题报告摘要:蕨类植物是一类古老的植物,具有良好的药用和食用价值。
查尔酮合成酶(CHS)是蕨类植物合成次生代谢物的重要酶,在蕨类植物的生物合成过程中起着关键作用。
本研究旨在克隆三种不同蕨类植物的CHS基因,并对其进行序列分析和表达模式研究,以期深入探究蕨类植物的生物合成机制。
1. 研究背景蕨类植物是一类古老的植物,分布广泛,包括了蕨、石松、蘑苔等多种,具有重要的药用和食用价值。
蕨类植物是地球上最早的裸子植物之一,其生存时间可追溯到4.6亿年前,对环境的适应能力极强。
查尔酮合成酶(CHS)是蕨类植物合成次生代谢物的重要酶,在化学防御、异色花色和抗氧化等方面起着关键作用。
随着基因工程和生物技术的发展,研究CHS基因及其调控机制已经成为国内外学者的热门研究课题。
2. 研究目的本研究旨在克隆三种不同蕨类植物(蕨、石松、蘑苔)的CHS基因,并对其进行序列分析和表达模式研究,以期深入探究蕨类植物的生物合成机制。
3. 研究方法(1)样本采集:分别采集蕨、石松和蘑苔的新鲜叶子样本。
(2)总RNA提取:采用TRIzol法提取样本总RNA。
(3)cDNA合成:将提取的总RNA进行逆转录反应,制备出cDNA模板。
(4)CHS基因克隆:采用PCR扩增方法,使用通用引物扩增CHS 基因的全长序列。
(5)克隆序列分析:将PCR产物进行酶切、测序和分析,获得CHS基因的全长序列,并进行多序列比对和物种系统发育树构建。
(6)实时荧光定量PCR:采用实时荧光定量PCR技术对三种蕨类植物中CHS基因的表达模式进行研究。
4. 预期结果预计可以从三种不同的蕨类植物中成功克隆到CHS基因,并获得其全长序列。
通过多序列比对和系统发育树构建,可以对三种蕨类植物的CHS基因进行进化分析和比较。
通过实时荧光定量PCR技术,可以研究三种蕨类植物中CHS基因的表达模式,揭示CHS基因在蕨类植物中的生物合成机制。
不同植物查尔酮合成酶CHS基因的生物信息学分析

d n h s o h mia rp ry in e t e r s mb a e r go s y r p o ii rh d o h l i ig p y i c e c l o e t ,sg a p p i ,ta me r n e in ,h d o h b ct o y r p i ct u cin o i s a d s e c p l d n y i y,fn t a d man e — ol n
ZHANG o Ta
)Ge e n Di e e tP a t n si f r n ln s
( oeeo i cec , iga g cl rl nvrt, i do26 0 , hn ) Cl g f f Si e QndoA r u ua U i sy Qn a 6 19 C ia l Le n i t ei g A src: ntippr tence c qecs n e ojc r mn c eune f hl n nhs C S ee b t t I s ae,h ul c i s une dt net a a ioai sqecs a oes tae( H )gn s a h ia d e a hc ul d oc c y
o d r tu t r s e e d t i dy a ay e n ay sr cu e ,w r eal l l z d,a d te p yo e ei r e w s c n tu td f rCHS a n cd s q e c s h e u t e n n h h l g n t t a o sr ce o e e mi o a i e u n e .T e r s l s s o e h t h 1 一ln t h w d t a e f 1 e gh o ORF o 0 C e e a b u . b a d e c d d 3 9 a n cd . A la i o a i e e c so t u f 1 HS g n s w s a o t1 2 k n o e 9 mio a i s l m n c d s q n e f n u f
植物查尔酮异构酶的生物信息学分析

植物查尔酮异构酶的生物信息学分析雷桅;邹祥;向阳;汤绍虎;孙敏【期刊名称】《北方园艺》【年(卷),期】2008(000)002【摘要】采用生物信息学方法和工具对GenBank中的洋葱、豌豆、番茄和茶等植物的黄酮类化合物合成关键酶查尔酮异构酶(CHI)的核酸和氨基酸序列进行了比对、分析和建模,进而对其分子结构、理化性质、亚细胞定位、蛋白转运肽、跨膜结构域、疏水性、分子系统进化、蛋白质二级和三级结构等重要参数进行了预测和推理.结果表明:该类酶基因的全长包括5'、3'非翻译区和一个开放阅读框,无蛋白转运肽,且定位于细胞质基质,是一个疏水性蛋白,二级结构均以随机卷曲和α-螺旋为主要构件,洋葱和豌豆的CHI三维建模成功.【总页数】5页(P193-197)【作者】雷桅;邹祥;向阳;汤绍虎;孙敏【作者单位】西南大学,生命科学学院,三峡库区生态环境教育部重点实验室,重庆,400715;西南大学药学院,重庆,400715;西南大学,生命科学学院,三峡库区生态环境教育部重点实验室,重庆,400715;西南大学,生命科学学院,三峡库区生态环境教育部重点实验室,重庆,400715;西南大学,生命科学学院,三峡库区生态环境教育部重点实验室,重庆,400715【正文语种】中文【中图分类】Q946.5;Q558【相关文献】1.油橄榄查尔酮合酶与查尔酮异构酶基因全长的克隆及序列分析 [J], 陈文拴;黄乾明;陈华萍;杨泽身;王安逸;苏光灿2.鸳鸯茉莉查尔酮异构酶基因(CHI)cDNA的克隆与生物信息学分析 [J], 曹玉婷;邱栋梁3.植物查耳酮异构酶生物信息学分析 [J], 陈克克;武雪4.狗枣猕猴桃查尔酮异构酶1(AkCHI1)的生物信息学分析 [J], 刘丹; 李然红; 陈鑫; 王立凤5.石榴查尔酮异构酶生物信息学分析 [J], 陈川;凯迪日耶·玉苏普因版权原因,仅展示原文概要,查看原文内容请购买。
大白菜查尔酮合成酶基因BrCHS1的克隆与特征分析

大白菜查尔酮合成酶基因BrCHS1的克隆与特征分析王小霞;刘志勇;李承彧;辛喜凤;冯辉【期刊名称】《沈阳农业大学学报》【年(卷),期】2012(043)002【摘要】查尔酮合成酶(chalcone synthase,CHS)是类黄酮合成途径前期的1个关键酶.本研究从大白菜花瓣中克隆到1个查尔酮合成酶家族基因,命名为BrCHS1.序列比对发现BrCHS1氨基酸序列与其他物种CHS氨基酸序列的同源性多数在80%以上,利用实时荧光定量PCR技术分析BrCHS1在大白菜黄色和白色花各种花器官中的表达水平,结果表明:BrCHS1在黄色花瓣中的表达量远高于其他花器官.以FT50×H1 的F2代分离群体为试材,根据BrCHS1周围序列设计SSR1引物,寻找与大白菜白色花基因紧密连锁的分子标记,结果显示,BrCHS1基因与白花性状基因并不连锁.【总页数】6页(P137-142)【作者】王小霞;刘志勇;李承彧;辛喜凤;冯辉【作者单位】沈阳农业大学园艺学院,沈阳110161;沈阳农业大学园艺学院,沈阳110161;沈阳农业大学园艺学院,沈阳110161;沈阳农业大学园艺学院,沈阳110161;沈阳农业大学园艺学院,沈阳110161【正文语种】中文【中图分类】S634.1【相关文献】1.嫁接陆地棉查尔酮合成酶与查尔酮异构基因的克隆及r表达分析 [J], 宋成攀;夏松波;王孝刚;张教海;秦鸿德;张友昌;冯常辉;别墅2.菊芋查尔酮合成酶基因的克隆与表达分析 [J], 李文静; 孙艳香; 付亚娟; 苏彦苹; 王聪艳; 侯晓强; 张新业3.藤茶查尔酮合成酶基因AgCHS1的克隆及功能鉴定 [J], 许明;林世强;倪冬昕;伊恒杰;刘江洪;杨志坚;郑金贵4.辣木查尔酮合成酶(CHS)基因的克隆及其序列分析 [J], 林玲;方健超;陈观水;艾育芳5.樱桃查尔酮合成酶CpCHS2基因的克隆及序列分析 [J], 李孝绒;乔光;姜昱雯因版权原因,仅展示原文概要,查看原文内容请购买。
罗汉果查尔酮合成酶基因的生物信息学分析

罗汉 果查 尔酮合 成 酶基 因的生物信 息学分 析
王 志强 蒙姣荣 邹 承武 z 黄 锦 宽 林 纬 黎起 秦 陈保善
1 西 亚 热 带 生 物 资源 保护 利 用 重 点 实验 窒 , 宁 , 3 0 5 2广 西 大 学 生命 科 学 与 技 术 学 院 , 宁 , 30 5 3广 西 大 学农 学 院, 宁 ,3 0 5 广 南 50 0 ; 南 5 00 ; 南 50 0 通 讯 作 者 ,hn aj x uc cey o@g ue n d
关键词 罗汉果, 查尔 酮合 成酶 基 因, 进化 分析
Bi i o m a isAn l sso e Ch lo e S n ha eGe efo o H a o o nf r tc ay i ft ac n y t s n r m Lu n Gu h
( ・・  ̄r s no R , r o o ve r ‘ 5iaii 一・ ta ●、 J
W a qa ngZhi ing M e i o o ngJa r ng Zo e g u Ch n wu Hu n i ua a g Jnk n Li i n we Liq q n ii Che o h n t r Ba s a
1 Gu n x y La o a o y o u r pia o e o r e Co s r a i n a d Utl a i n a g iKe b r t r f S bto c lBi r s u c n e v t n i z t ,Na n n ,5 ห้องสมุดไป่ตู้ 0 ;2 Co lg fLie S i n e a d Te h o o y o i o n i g 3 0 5 le e o f ce c n c n l g ,
金钗石斛查尔酮合酶基因的生物信息学分析

第38卷第4期2020年7月 贵州师范大学学报(自然科学版)JournalofGuizhouNormalUniversity(NaturalSciences)Vol.38.No.4Jul.2020引用格式:徐 ,高方平,郑远静,等.金钗石斛查尔酮合酶基因的生物信息学分析[J].贵州师范大学学报(自然科学版),2020,38(4):64 68.[XUH,GAOFP,ZHENGYJ,etal.BioinformaticsanalysisofchalconesynthasegenefromDendrobiumnobile[J].JournalofGuizhouNormalUniversity(NaturalSciences),2020,38(4):64 68.]金钗石斛查尔酮合酶基因的生物信息学分析徐 1,高方平1,郑远静1,周娜娜1,孙 威1,2(1.贵州师范大学生命科学学院/植物生理与发育调控重点实验室,贵州贵阳 550025;2.西南喀斯特山地生物多样性保护重点实验室,贵州贵阳 550025)摘要:利用生物信息学工具及方法对金钗石斛查尔酮合成酶基因进行分析。
结果显示,金钗石斛查尔酮合酶基因(DnCHS)完整的CDS为1188bp,共编码395个氨基酸,蛋白分子量为43 10kD,理论等电点为6 22,且该蛋白为亲水性蛋白,不存在信号肽,很可能定位在细胞质中。
同时,蛋白质二级和三级结构的分析表明,DnCHS蛋白由33 92%的α螺旋,8 35%的延伸链以及57 72%的无规则卷曲组成。
系统进化分析显示,DnCHS与红掌的亲缘关系最近。
关键词:金钗石斛;查尔酮合酶;生物信息学中图分类号:S567 文献标识码:A 文章编号:1004—5570(2020)04-0064-05DOI:10.16614/j.gznuj.zrb.2020.04.010BioinformaticsanalysisofchalconesynthasegenefromDendrobiumnobileXUHui1,GAOFangping1,ZHENGYuanjing1,ZHOUNana1,SUNWei1,2(1.SchoolofLifeScience/KeyLaboratoryofPlantPhysiologyandDevelopmentRegulation,GuizhouNormalUniversity,Guiyang,Guizhou550025,China;2.KeyLaboratoryofStateForestryAdministrationonBiodiversityConservationinKarstMountainAreaofSouthwestofChina,Guiyang,Guizhou550025,China)Abstract:Inthispaper,weusedbioinformaticstoolsandmethodstoanalyzechalconesynthasegeneofDendrobiumnobile,analysisresultsshowedthatthecompleteCDSofDnCHSwas1188bp,enco dingatotalof395aminoacids.Themolecularweightofthisproteinwas43.10kD,anditstheoreticalisoelectricpointwas6.22.ThentheresultsalsoindicatedthatDnCHSproteinwashydrophobicwhichdidnotcontainsignalpeptideandwaslikelylocatedinthecytoplasm.Meanwhile,theresultsofsec ondaryandtertiarystructureanalysesshowedthatDnCHSproteinwascomposedof33.92%αhelix,8.35%extendedstrandand57.72%randomcoil.Atlast,phylogeneticanalysisrevealedthatDnCHSwascloselyrelatedtotheCHSofAnthuriumandraeanum.Keywords:Dendrobiumnobile;chalconesynthase;bioinformaticsanalysis46收稿日期:2020-02-28基金项目:贵州省联合基金项目(黔科合LH字[2016]7211号,黔科合LH字[2017]7358);贵州师范大学资助博士科研项目(0516006);贵州省重点实验室建设项目(黔科合计Z字[2011]4005)通讯作者:孙 威(1985-),女,博士,副教授,研究方向:植物次生代谢与调控,E mail:sunwei889@163.com.0 引言金钗石斛(Dendrobiumnobile)简称金钗,其“色泽堪比金钗、茎似钗股、无歧、侧枝如钗、股短而中实”,是一种传统的名贵中药[1]。
银杏查尔酮合成酶基因启动子_GbCHSP_调控元件及功能分析

园艺学报 2010,37(12):1919–1928Acta Horticulturae Sinica银杏查尔酮合成酶基因启动子(GbCHSP)调控元件及功能分析李琳玲1,*,程华1,*,程水源1,**,许锋2,王燕2,姜德志2(1黄冈师范学院生命科学与工程学院,湖北黄冈 438000;2长江大学园艺园林学院,湖北荆州 434025)摘 要:通过染色体步移方法从银杏(Ginkgo biloba L.)基因组中克隆到查尔酮合成酶基因(CHS)翻译起始位点上游1 711 bp的启动子序列。
生物信息学分析表明,该启动子片段中存在多个顺式作用元件,包括紫外/蓝光响应单元、植物激素响应单元、真菌诱导元件、MYB结合位点、TATA-box和CAAT-box等。
亚克隆了CHS转录起始位点上游1 402 bp序列,将其与GUS基因构建融合表达载体pBI121 + CHSP,以pBI121-35S作为负对照,通过农杆菌(LBA4404)介导法分别转入烟草。
结果表明,银杏CHS启动子序列能驱动GUS基因在烟草中的表达,表达具有组织特异性。
GbCHSP的功能研究将有助于揭示银杏叶黄酮的积累与GbCHS基因表达的分子机理。
关键词:银杏;查尔酮合成酶基因启动子;GUS;调控元件分析中图分类号:S 664.3 文献标识码:A 文章编号:0513-353X(2010)12-1919-10 Regulatory Elements and Functional Analysis of Chalcone Synthase Gene Promoter from Ginkgo biloba L.LI Lin-ling1,*,CHENG Hua1,*,CHENG Shui-yuan1,**,XU Feng2,WANG Yan2,and JIANG De-zhi2(1College of Life Science and Engineering,Huanggang Normal University,Huanggang,Hubei 438000,China;2College of Horticulture and Gardening,Yangtze University,Jingzhou,Hubei 434025,China)Abstract:The regulative sequence(1 711 bp)of chalcone synthase gene promoter(CHSP)from Ginkgo biloba L. was cloned by genomic walking. In silico analysis suggested that the sequence contained several typical cis-acting elements,including UV/blue light responsive elements,phytohormone responsive elements,fungal elicitor responsive elements,MYB binding site,TATA-box and CAAT-box. A 1 402 bp promoter sequence upstream 5′ of translation start site of GbCHS was cloned and designated as GbCHSP,respectively. pBI121 + CHSP and pBI121-35S were constructed and transformed into tobaccos by LBA4404. These results showed that pBI121 and pBI121 + CHSP both could drive the transient expression of GUS in tobaccos and pBI121 + CHSP expressed differentially in root,stem and leaf tissues of tobacco. These results will be help to understand the transcriptional regulatory mechanism on GbCHS expression and flavonoids accumulation.Key words:Ginkgo biloba L.;chalcone synthase gene promoter;GUS;the analysis of cis-element收稿日期:2010–09–07;修回日期:2010–12–06基金项目:国家自然科学基金项目(30971974);湖北省自然科学基金重点项目(2008CDA061)* 为同等贡献作者** 通信作者Author for correspondence(E-mail:s_y_cheng@)1920 园艺学报37卷银杏(Ginkgo biloba L.)叶片具有重要的药用价值,其中主要的活性成分是银杏叶黄酮(van Beek,2002)。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
( 西南林业大学 西南地区生物多样性保育 国家林业局重点实验室 ,云南 昆 明6 5 0 2 2 4)
摘要 :运用分子生物学软件对金花茶 、菊花 、杜 鹃、荷 花 、金银花 、贯 叶金丝桃 、牡丹 、龙 胆 、桃树 、兜兰 1 0 种观赏植物 的查尔酮合成酶基因 ( C H S )进行 核酸 序列 预测 和分析 ,采 用邻 接距 离矩 阵法构 建 了系统发 育树 , 并进行系统发育分析 ,结果表明 ,( 1 )观赏植 物查尔酮合 成酶基 因所编码 蛋 白质 为稳定 的疏水性 单链蛋 白质 ,
q u e n c e s a n a l y s i s aห้องสมุดไป่ตู้n d pr o t e i n pr e di c t i o n we r e c o n d u c t e d. Th e r e s u l t s o f t h i s s t u d y i n d i c a t e d t h a t c h a l c o n e s y n z y me
杜 鹃 与金 花茶 、牡 丹 与 菊 花 的 关 系 都 较 近 。
关键词 :观赏植物 ;查尔酮合成酶 ;基 因 ;蛋 白质 ;生物信息学 中图分类号 :Q 7 8 文献标识码 :A 文章编号 :1 6 7 2— 8 2 4 6( 2 0 1 3 )0 5— 0 0 6 2— 0 7
r o l e i n c a t a l y z i n g i n c y t o p l a s mi c ma t r i x wi t h o u t t r a n s p o r t i n g b y p r o t e i n, a te f r t h e y we r e s y n t h e s i z e d o n f r e e r i b o —
Ab s t r a c t :T h e c h a l c o n e s y n z y me g e n e s( C H S )o f 1 0 o r n a m e n t a l p l a n t s i n c l u d i n g C h r y s a n t h e mu m m o r f i o l i u m, C a m e l l i a n i t i d i s s i ma ,H y p e r i c u m p e r f o r a t u m,P r u n u s p e r s i c a ,N e l u m b o n u c f i e r a , P a p h i o p e d i l u m c o n c o l o r ,L o n i c e — r a j a p o n i c a ,R h o d o d e n d r o n s i m s i i ,P a e o n i a s u f f r u t i c o s a a n d G e n t i a n a t r i lo f r a ,w e r e s t u d i e d .A m i n o a c i d s s e —
以 一 螺旋和无规则卷曲为主;( 2 )可能在游离核糖体上合成后不经 蛋 白转运 ,直接在细胞 质基质的特定部位 中 行使催化功能 ;( 3 )查 尔酮合成酶的作用机制可能与组织特异性 和发育阶段 以及对外界不同刺激敏感 程度相关 ;
( 4 )查尔酮合成酶基因具有超基 因家族 ,C H S基因在进化 中存在 基因重复 与分歧 ,其保守 性好 ,除桃树有 2个 外显 子 ,其余植物 只有 1 个外显子 ,且几种植物的 C H S外显子剪切位点不 同;( 5 )龙胆与其他植 物的关 系较远 ,
g e n e s e n c o d e d s t a b l e s i n g l e h y d r o p h o b i c pr o t e i n s .c o mp o s e d o f r a n d o m c o i l a n d o l — h e l i x.Th e y ma y p l a y a s p e c i a l
第4 2卷
第 5期
西
部
林
业
科
学
V0 1 . 42 No . 5
2 0 1 3年 1 0月
J o u r n a l o f We s t C h i n a F o r e s t r y S c i e n c e
Oc t . 2 01 3
1 0种 观赏 植 物查 尔 酮 合成 酶基 因生 物信 息 学分 析
Z H ANG Ta i — k u i ,LI U Zh e n g,ZHU F a n g — mi n g, Z H ANG Ha n— y a o
( Ke y L a b o r a t o r y o f B i o d i v e r s i t y Co n s e r v a t i o n i n S o u t h we s t C h i n a, S t a t e F o r e s t r y Ad mi n i s t r a t i o n S o u t h we s t F o r e s t r y Un i v e r s i t y. Ku n mi n g Yu n n a n 6 5 0 2 2 4, P . R. Ch i n a )
Bi o i n f o r ma t i c s An a l y s i s o f Ch a l c o ne S y n z y me Ge n e s f r o m
Te n Ki n d s o f Or na me n t a l Pl a nt s
