图位克隆原理 PPT课件
图位克隆原理
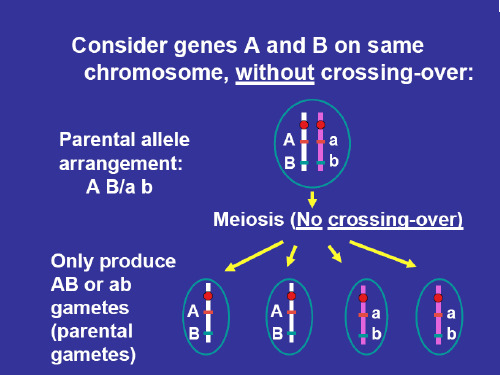
注意后两种情 况反映的是来 自于两个配子 的染色体区段 的情况
包含来自于 两个亲本的 染色体片段
包含来自于 Japonica 两 条 染色体区段
包含来自于 Indica 两 条 染 色体区段
上图中M是指含有隐性突变位点的亲本A某个分子标记的PCR 上图中M是指含有隐性突变位点的亲本A某个分子标记的PCR 扩增产物, 是指一个与M有遗传差异的野生型亲本B 扩增产物,m是指一个与M有遗传差异的野生型亲本B相对分 子标记的PCR扩增产物, 19是指 PCR扩增产物 是指M 杂交后的F 子标记的PCR扩增产物,1-19是指M与m杂交后的F1代的自交 后代( 这些植株都是有突变表型的, 后代 ( 即 F2) , 这些植株都是有突变表型的 , 说明突变位点 是纯合的。 19个定位群体植株中 个定位群体植株中, 10、11、13、 是纯合的。这19个定位群体植株中,1、4、8、10、11、13、 15、18、19仅包含来自于亲本 的染色体区段, 仅包含来自于亲本A 15、18、19仅包含来自于亲本A的染色体区段,没有发生交 12、16、17包含 个来自于亲本A 包含1 换; 2 、 3 、 7 、 9、 12、 16、 17 包含1 个来自于亲本A 的染色 体区段以及1条来自于亲本B的染色体区段, 体区段以及 1 条来自于亲本 B 的染色体区段 , 即发生了一次 交换; 14仅包含来自于亲本 的染色体区段, 仅包含来自于亲本B 交换;5、6、14仅包含来自于亲本B的染色体区段,也就是 发生了两次交换,因此总交换数是7 13次 发生了两次交换,因此总交换数是7+3×2=13次,总配子数 19× 38个 为19×2=38个
F1 plant
自交
F2 plants
只有左侧三种植株会选入定位群体
图位克隆-李金伟.

李金伟
2012215423
无论头上是怎样的天空,我准备承受任何风暴。
内容简要
1
图位克隆技术的基本原理 图位克隆的一般步骤(考研)
图位克隆在植物基因分离中的应用 图位克隆的现状和前景
2
3
4
图位克隆技术的基本原理
图位克隆(Map - based cloning) 又称定位克隆(positional cloning) , 1986 年 首先由剑桥大学的Alancoulson 提出,是近几年 来随着各种植物的分子标记图谱相继建立而发展 起来的一种行的基因克隆的一种方法
图位克隆的一般步骤
最为常用的分子 标记
SSLPs
SSLP是基于PCR的分 子标记,检测比较直接 ,只需要通过设计引物 便可检测鉴定的SSLP标 记
SNPs
SNPs标记的检测也比 较直接,它是不同生态 型之间基因组中的单个 核苷酸的差别,这些差 别的核苷酸通常位于不 编码区域,常见的检测 SNPs标记的方法主要是 剪切扩增多态性序பைடு நூலகம்( CAPS),它是基于PCR的
侧翼分子 标记
混合样 品作图
指在准确鉴定目的基因表型(单基因控制的隐性突变 )的基础上,把大群体中单株突变体分成若干组,以 组为单位提取DNA,形成一个混合的DNA池进行分析, 根据所有池中分子标记与目的基因发生的重组数来确 定与目的基因连锁最紧密的分子标记及目的基因附近 所有分子标记的顺序
图位克隆的一般步骤
图位克隆的一般步骤
2.4目的基因的筛选和鉴定 2.4.1目的基因的筛选
筛选和鉴定目的基因是图位克隆技术的最后一个关键环 节。当一旦找到与目的基因紧密连锁的分子标记并鉴定出分 子标记所在的大片段克隆后,就可以利用该克隆为探针进行 染色体步移,逐渐靠近目的基因。
图位克隆的基本原理和方法

r/r
R/R
P1 纯合突变体
×
P2 ZS97
F1
R/r
F2
R/R
R/r
r/r
primary mapping method
Bulked Segregment Analysis:群组分离分析法。原理是将分 离群体(F2、BC1等)中的个体依据研究的目标性状(如抗病、 感病)分成两组,每一组取样8-15份,在每一组群体中将各个 体DNA等量(等浓度等体积)混合,形成两个DNA池(如抗 病池和感病池)。同时需要构建两个亲本(ZH11/ZS97)的 BSA池。由于分组时仅对目标性状进行选择,因此两个池间 理论上就应主要在目标基因区段存在差异。
精细定位
• 扩大群体,进行精细定位
Complementation test
找到候选基因后可对候选基因进行分析以排除非目标基因, 可以通过比较扩增,测序,表达量检测及目标性状相关的 生化和生理途径等方法来排除。当然最后要说明问题的话 就是构建互补载体做一个互补验证。
将构建的BSA池分别用本室的255对在亲本ZH11/ZS97间存在多态性的SSR标 记进行PCR扩增,经过跑PAGE胶找到与目标性状紧密连锁的标记。
HL15(M )
HL15(W ) ZS97
ZH11
HL15(M ) HL15(W ) ZS97
ZH11
HL15(M) HL15(W) HY5(M) HY5(W) HY3(M) HY3(W) HY2(M) HY2(W) HY1(M) HY1(W) ZS97 ZH11
图位克隆的基本原 理和方法
何宗顺 2011.12.7
图位克隆的基本原理
其基本的原理是: 功能基因在基因组中都有相对较稳定的基因座,通过找到 与目标性状紧密连锁的分子标记,在利用分子标记技术对 目的基因精细定位的基础上,由于水稻全基因组序列的发 布,我们可以得到已定位区段的候选基因,通过对候选基 因进行分析,确定目标基因,最后经遗传转化试验证实目 的基因功能。
图位克隆原理知识讲解

SNP
= single nucleotide polymorphism
• 单核苷酸多态性 • 主要是指在基因组水平上由单个核苷酸的变异
所引起的DNA序列多态性。SNP所表现的多态性 只涉及到单个碱基的变异,这种变异可由单个 碱基的转换或颠换所引起,也可由碱基的插入 或缺失所致。但通常所说的SNP并不包括后两种 情况。(后面两种情况的多态性一般归为InDel)
F1 plant
假设: 只存在单交换
自交
F2 plants
只有左侧三种植株会选入定位群体
ቤተ መጻሕፍቲ ባይዱ
分子标记M5与突变位点的遗传距离:
M5处发生交换的 植株包括三种情况
10/100
6/100
4/100
(20/(100×2 )) ×100%=10cM
假设: 统计100个个体
分子标记M4与突变位点的遗传距离:
比较产物长度
• CAPs
= cleaved amplified polymorphic sequences 酶切扩增多态性
利用酶切位点
Marker: MIG5-B
HC C
L
• dCAPs = derived CAPS 设计引物引入错配碱基,从而引入酶切位点
其它标记
InDel
= insertion-deletion 插入缺失标记,指的是两 种亲本中在全基因组中的差异,相对另一个亲 本而言,其中一个亲本的基因组中有一定数量 的核苷酸插入或缺失。根据基因组中插入缺失 位点,设计一些扩增这些插入缺失位点的PCR 引物,这就是InDel标记。 部分SSLP就是由 InDel转化而来
在拟南芥的众多生态型中最常用的三种是 • Landsberg erecta(Ler) • Columbia(Col) • Wassilewskija(Ws)
图位克隆的基本原理和方法-PPT课件

将构建的BSA池分别用本室的255对在亲本ZH11/ZS97间存在多态性的SSR标 记进行PCR扩增,经过跑PAGE胶找到与目标性状紧密连锁的标记。
8
HL15(M )
HL15(W ) ZS97
ZH11
HL15(M ) HL15(W ) ZS97
ZH11
HL15(M) HL15(W) HY5(M) HY5(W) HY3(M) HY3(W) HY2(M) HY2(W) HY1(M) HY1(W) ZS97 ZH11
找到紧密连锁的分子标记之后我们就要进行一个“阳性”检测: 即检测这个找到的分子标记是不是真正的与目标性状连锁。 我们对F2代群体中随机选取一个200左右的小群体,将找到的分 子标记分别扩增这些样,看表型与基因型是否对应。 同时我们可以用这个小群体进行一个初步的基因定位。
9
一. 表型观察
Fine mapping
4
作图群体
将纯合突变体与ZS97进行杂交,对杂交的种子进行基 因型鉴定,找到杂合型种子(即种子能够发生分离), 将杂合F1代种子种下去后就能得到F2代群体。
5
r/r
R/R
P1 纯合突变体
×
P2 ZS97
F1
R/r
F2
R/R
R/r
r/r
6
primary mapping method
Bulked Segregment Analysis:群组分离分析法。原理是将分 离群体(F2、BC1等)中的个体依据研究的目标性状(如抗病、 感病)分成两组,每一组取样8-15份,在每一组群体中将各个 体DNA等量(等浓度等体积)混合,形成两个DNA池(如抗 病池和感病池)。同时需要构建两个亲本(ZH11/ZS97)的 BSA池。由于分组时仅对目标性状进行选择,因此两个池间 理论上就应主要在目标基因区段存在差异。
图位克隆的基本原理和方法1

交换有两种带型:H和B
单株 1 M1 M2 M3 M4 M5
2
3
4
5
6
7
8
9
10
H H
A A A
A A
A H H
A A
A A H
B BA A AH AA A AA A
A B B
A A
A H B
A A
A H H
A A
A A B
B A
A A A
基因和表型相对应!
开发新的标记:
新的SSR标记: /modules/redbtools/ssrscan.php, 运行SSRScan就会得到该段序列的简单重复序列。
中输入定位区间的物理位置,即可显示出候选基因。
精细定位
• 扩大群体,进行精细定位
Complementation test
找到候选基因后可对候选基因进行分析以排除非目标基因, 可以通过比较扩增,测序,表达量检测及目标性状相关的
生化和生理途径等方法来排除。当然最后要说明问题的话
就是构建互补载体做一个互补验证。
Trait
Qualitative traits Quantitative traits
method
bulked segregment analysis whole-genome QTL screen
Mapping population
F2、BC1 RIL、 DH 、NIL
作图群体
将纯合突变体与ZS97进行杂交,对杂交的种子进行基 因型鉴定,找到杂合型种子(即种子能够发生分离), 将杂合F1代种子种下去后就能得到F2代群体。
InDel/Caps标记:将要开发标记的日本晴区段序列与93-11做一个 blast,选取插入或者缺失比较大的位点设计引物。
基因的图位克隆

要点
1. 克隆基因的意义
2. 基因克隆的常用策略及比较 3. 图位克隆的基本程序和以 xa13为例介绍图位克隆 的详细过程 5. 总结及展望
1.为什么要克隆基因?
克隆基因有助于我们解释基因的生物学
功能、调控模式、进化关系等。
在植物中,大部分基因的功能未知。
Tabata, S. et al. 2000, Nature 408, 796 - 815
功能进行干扰,从而证明特定序列所控制的表型。
正向遗传学:从已有的相对表型的差异开始,去分离引起表型变化的对应核苷酸序列改变。
Janny L. P. at el.2003.TRENDS in Plant Science l.8:484-491
• 基因克隆的主要方法:
• T-DNA标签法:当T-DAN导入植物基因组中,并插入基因的编码区或重要的调控区,
• 基因的抑制表达:通过降低目的基因在细胞的表达水平,
使基因的功能下降甚至消失,来研究基因的功能。
• 基因的超量表达:通过人为的重组DNA手段,在生物体细
胞中大量地、持续的表达目的基因,使目标基因的功能得到超 常发挥。 优点:目的明确,对由基因家族控制的相关形状,抑制表达能同 时降低此基因家族成员的功能;抑制表达和超表达是研究时空表 达的有力工具。
• 原理:在减数分裂︱时期,同源染色体之间配对,非姊妹染色单体发生交换,与目
标基因距离越远,发生交换的频率就越大,距离越近,发生交换的频率就越小(遗传 学第三定律)。(王亚馥,戴灼华. 遗传学.1999,第三章:60-76)与基因发生最小交 换频率的两侧分子标记,就是与基因最紧密连锁的分子标记,这两个分子标记之间的 区段就是包含这个基因的目标区段。
基因的图位克隆ppt课件

• G. Zhang et al等利用IR24和IRBB13杂交,得到的F2分离群体,利用BSA法,找 到了一个RAPD标记OPAC05,在抗病池和感病池之间有多态性。从而将 xa13定 位于OPAC05附近。
•
G. Zhang et al. 1996 , Theor Appl Genet 93:65 70
常发挥。
优点:目的明确,对由基因家族控制的相关形状,抑制表达能同 时降低此基因家族成员的功能;抑制表达和超表达是研究时空表 达的有力工具。
缺点:抑制表达和超表达会使相关基因的功能紊乱,常常很难判 断相关表型的改变是由于目标基因还是由于被紊乱的基因引起的。
葛莘. 高级植物分子生物学, 2004
•另外还有TILLING技术、芯片技术、比较基因组学、生物信息学……都被广泛的应 用于基因的克隆。
如果在xa13两侧都检测到有来自IR24的标记基因型, 则这个两个标记就是基因xa13的边界。
后来储朝晖博士,为了验证以上的结果,利用极端隐性分组分析法对来自IRBB13 和IR24杂交的250株极端抗病F2单株把 xa13 重新定位于S14003和RG163之间,同时 一个新发现的CAPS 标记E6a定位于标记RG136和xa13基因之间,从而以Ea6和 S14003作为构建物理图谱的起点。
• 3. 图位克隆和以 xa13为例介绍图位克隆的详细过程
• 原理:在减数分裂︱时期,同源染色体之间配对,非姊妹染色单体发生交换,与目
标基因距离越远,发生交换的频率就越大,距离越近,发生交换的频率就越小(遗传 学第三定律)。(王亚馥,戴灼华. 遗传学.1999,第三章:60-76)与基因发生最小交 换频率的两侧分子标记,就是与基因最紧密连锁的分子标记,这两个分子标记之间的 区段就是包含这个基因的目标区段。
图位克隆

序列(CAPS),它也是基于PCR 的。
另外,一种更为有效的方法衍生的CAPS (dCAPS) (Michaels and Amasino,1998; Nam et al., 1989)可把任何已知的点突变 作为分子标记,只要在PCR 时引入不配对 的引物,使扩增的序列在一个生态型中具 有限制性酶切位点,而在另一生态型中没 有,以形成多态性。
2.4 目的基因的筛选和鉴定
筛选和鉴定目的基因是图位克隆技术的最后一个 关键环节。当一旦找到与目的基因紧密连锁的分 子标记并鉴定出分子标记所在的大片段克隆后, 就可以利用该克隆为探针进行染色体步移,逐渐 靠近目的基因。
染色体步移是指利用已知的阳性克隆称为 “二次克隆”,如此反复即可取到所需要的克隆,获 得目的基因。染色体步移的主要局限在于当必须经过 一个无法克隆的片段时,步移的过程会被打断;当克 隆的一端是重复序列时,步移的方向会发生偏差。为 克服这些弊端,Tanksley 等(1995)又发展了染色体登 陆的方法。
2.粒是一种含有噬菌体的Cos 位点的质粒载 体, 大小在5~7Kb 左右, 可高效地克隆25~35Kb 的DNA 片段。人工染色体的插入片段最长可达 2Mb 左右,能够覆盖完整的真核基因,最早发展 的人工染色体是YAC。所以当要克隆大片段DNA 时,需要YAC 作载体。以YAC 为载体,可将 300~1 000Kb 的DNA 片段克隆。因此,以YAAC、PAC 等几 种以细菌为寄主的载体系统。
1 图位克隆技术的原理
功能基因在基因组中都有相对较稳定的基因座, 在利用分子标记技术对目的基因精细定位的基础 上,用id 等),从 而构建基因区域的物理图谱,再利用此物理图谱 通过染色体步移(chromosome walking) 逼近目的 基因或通过染色体登陆(chromosome landing) (Tanksley et al., 1995)方法最后找到包含有该目 的基因的克隆,最后经遗传转化试验证实目的基 因功能。
图位克隆原理

(2)不完全显性:F1表现为双亲性状的中间型。
(3)共显性:F1同时表现双亲性状,而不是表 现单一的中间型。
RR ↓ rr
•由于根长还受影响培
Rr
养环境等影响,因为
↓
不完全显性的情况比
1RR : 2Rr : 1rr 较难判断
孟德尔规律
►二、非等位基因间的相互作用
无互作: 有互作:
9:3:3:1
9:7 互补作用 15:1 重叠作用 13:3 抑制作用 9:6:1 累加作用 9:3:4 隐性上位作用 12:3:1 显性上位作用
什么是重组子
►在突变位点附近区域内绝大部分 的样品跑样结果都应该为只有非 重组的Col条带,即记录Rf为0, 只有少数的样品存在交换,Rf记 为1。
►图中的6、12号样品即为重组子
筛选重组子的目的
► 在筛选出6、12号两个重组子以后,假设 在目标区域内寻找到新的标记引物,就 不需要把所有的个体跑样,只需要跑重 组子个体
►END
交换次数 配子总数
×100%
3×2 + 7
= 19 × 2
×100% ≈34 cM
SSLPs
SSLPs
►40.48 %
►4.76%
►20.00 %
4.76%
12.5% 12.5% 36%
筛选 F2 代 短根 移栽
3周左右
提基因组
各对引物PCR
统计计算Rf
步骤总结
缩小范围 寻找新Marker 扩大群体 (筛选重组子)
►密度: 每 6.6kb 存在一个
SNP
= single nucleotide polymorphism
►单核苷酸多态性
►主要是指在基因组水平上由单个核苷酸的变异 所引起的DNA序列多态性。SNP所表现的多态性 只涉及到单个碱基的变异,这种变异可由单个 碱基的转换或颠换所引起,也可由碱基的插入 或缺失所致。但通常所说的SNP并不包括后两种 情况。(后面两种情况的多态性一般归为InDel)
图位克隆原理

图位克隆原理图位克隆(Positional Cloning)是一种重要的分子生物学技术,它是通过对特定基因的物理位置进行定位,从而实现对该基因的克隆和研究。
图位克隆技术的原理是基于遗传连锁和物理定位相结合的方法,可以帮助科学家们找到与特定性状或疾病相关的基因。
在图位克隆的过程中,首先需要通过遗传连锁分析确定目标基因的大致位置,然后利用物理定位技术进一步缩小目标基因的范围,最终实现对目标基因的克隆。
下面将详细介绍图位克隆的原理和步骤。
1. 遗传连锁分析。
遗传连锁分析是通过观察不同基因之间的遗传连锁关系,确定目标基因在染色体上的大致位置。
这一步骤通常利用家系分析和连锁图谱构建等方法,确定目标基因与已知标记基因之间的遗传距离和连锁关系。
通过这一步骤,可以初步确定目标基因所在的染色体和染色体区域。
2. 物理定位技术。
物理定位技术是利用分子标记和染色体显微操作等方法,对目标基因进行更精确的定位。
这一步骤通常利用分子标记的特异性杂交和染色体行为的特征等,进一步缩小目标基因的范围,最终确定目标基因的具体位置。
物理定位技术的发展使得科学家们能够更加精确地定位和克隆目标基因。
3. 克隆目标基因。
通过遗传连锁分析和物理定位技术,科学家们可以确定目标基因的大致位置和具体范围,从而利用克隆技术对目标基因进行克隆。
克隆技术通常包括构建基因文库、筛选目标基因、进行测序和功能分析等步骤,最终实现对目标基因的克隆和研究。
总结。
图位克隆技术是一种重要的分子生物学技术,它通过遗传连锁分析和物理定位技术,实现对目标基因的精确定位和克隆。
图位克隆技术的发展为科学家们研究特定性状和疾病相关基因提供了重要的工具和方法。
随着分子生物学和基因工程技术的不断发展,图位克隆技术将在基因定位和克隆研究中发挥越来越重要的作用。
图位克隆的基本原理和方法1

找到紧密连锁的分子标记之后我们就要进行一个“阳性”检测: 即检测这个找到的分子标记是不是真正的与目标性状连锁。 我们对F2代群体中随机选取一个200左右的小群体,将找到的分 子标记分别扩增这些样,看表型与基因型是否对应。
同时我们可以用这个小群体进行一个初步的基因定位。
一. 表型观察
Fine mapping
精细定位
• 扩大群体,进行精细定位
Complementation test
找到候选基因后可对候选基因进行分析以排除非目标基因, 可以通过比较扩增,测序,表达量检测及目标性状相关的 生化和生理途径等方法来排除。当然最后要说明问题的话 就是构建互补载体做一个互补验证。
谢谢大家
将构建的BSA池分别用本室的255对在亲本ZH11/ZS97间存在多态性的SSR标 记进行PCR扩增,经过跑PAGE胶找到与目标性状紧密连锁的标记。
HL15(M )
HL15(W ) ZS97
ZH11
HL15(M ) HL15(W ) ZS97
ZH11
HL15(M) HL15(W) HY5(M) HY5(W) HY3(M) HY3(W) HY2(M) HY2(W) HY1(M) HY1(W) ZS97 ZH11
67 AA AA AA BH BB
8 9 10 AA B AA A AA A HA A HB A
基因和表型相对应!
开发新的标记:
新的SSR标记: /modules/redbtools/ssrscan.php, 运行SSRScan就会得到该段序列的简单重复序列。
二. 筛选交换单株
三. 开发新的分子标记
r/r
R/R
P1 纯合突变体
×
P2 ZS97
F1
图位克隆

1.1 构建遗传作图群体 用于作图群体的类型可有多种。
一般说来,在这类群体中,异花授粉植物分子标记的检出率较自花授粉植物的高。
但是对于基因的图位克隆而言,培育特殊的遗传群体是筛选与目标基因紧密连锁的分子标记的关键环节。
这些遗传材料应该满足这样的条件,即除了目标基因所在座位的局部区城外,基因组DNA序列的其余部分都是相同的,在这样的材料间找到的多态性标记才可能与目标基因紧密连锁。
目标基因的近等基因系(NILs)是符合条件的一类群体。
近等基因系是指几乎仅在目标性状上存在差异的两种基因型个体,这可通过连续回交的途径获得。
由于NILs的遗传组成特点,一般凡是能在近等基因系间揭示多态性的分子标记就极有可能位于目标基因的两翼附近。
Martin等(1993)就是用RAPD技术分析番茄PTO基因的近等基因系而获得了与该基因表现共分离的分子标记,以该分子标记为探针筛选基因组文库而实现了染色体登陆。
又如在玉米上,利用AFLP技术分析玉米CMS-S型雄性不育的恢复基因及近等基因系,已获得与目标基因紧密连锁的分子标记并以其用作分离RF3基因的探针。
一般说来,当标记为显性遗传时,欲获得最大遗传信息量的F2群体,须借助于进一步的子代测验,以分辨F2中的杂合体。
为此,Michelmore等(1991)发明了分离群体分组分析法(bulked segregant analysis,BSA)以筛选目标基因所在局部区域的分子标记。
其原理是将目标基因的F2(或BC1)代分离体的各个体仅以目标基因所控制的性状按双亲的表型分为两群,每一群中的各个体DNA等量混合,形成两个DNA混合池(如抗病和感病、不育和可育)。
由于分组时仅对目标性状进行选择,因此两个DNA混合池之间理论上主要在目标基因所在局部区域的差异,这非常类似于NILs,故也称作近等基因池法。
研究表明,近等基因系法、分离群体分组分析法再结合AFLP等强有力的分子标记技术使人们能够在短时间内从数量众多的分子标记中筛选出与目标基因紧密连锁的标记。
- 1、下载文档前请自行甄别文档内容的完整性,平台不提供额外的编辑、内容补充、找答案等附加服务。
- 2、"仅部分预览"的文档,不可在线预览部分如存在完整性等问题,可反馈申请退款(可完整预览的文档不适用该条件!)。
- 3、如文档侵犯您的权益,请联系客服反馈,我们会尽快为您处理(人工客服工作时间:9:00-18:30)。
包含来自于 Ler两条染色 体区段
**
Col
* ** * * **
1、4、8、10、11、13、15、18、19共9个 样品,只包含来自于亲本Col的染色体区段, 没有发生交换
**
*
Ler
5、6、14共3个样品,只包含来自于亲本Ler的 染色体区段,也就是发生了两次交换
** * * * **
Col Ler
2、3、7、9、12、16、17共7个样品,包含1个 来自于亲本Col的染色体区段以及1条来自于亲 本Ler的染色体区段,即发生了一次交换
• 突变位点与分子标记M的遗传距离:
交换次数 配子总数
×100%
3×2 + 7
= 19 × 2
×100% ≈34 cM
SSLPs
SSLPs
• 40.48% • 4.76%
有 照果时也隐不长并性是得不突,不能变造好准成的确短情判根况断突表。,变型例体杂可如交能出F是现x1代2中种根情L、e况r或:者对 •单 基 因建 中• (为代议根母长1、处)短本短理突分突根方变离变等法为;体,:隐自在分a性a交盆类(突所上后短变得标全根,A(明部)杂aa,移(a交待栽)长不苗,,根成A长分表)功A大成现(,后长为长种(根F根子12、) 突 变至 对 真• (((粗假3周短 短2定。显)后根根位性突)))用突变编,,的变为号杂表S显后SA交现LA性单突不为P(突引株变成F短变1物提体功代根,做取的全)杂P基母部C交x因本R为F组,自短1,验a代交L根a用证e为(种r任杂A长为a意交根AA一苗) • 真的F1杂交苗为双带,假A的a(为短单根带)(col带)
•• •
注 某注突些意意变隐事株事,性项系项因突如2:此:变果一理刚般论好出上是现F2双的代A突杂a突都(变交变为长,F大短2根则代多根)分数(a离为a比) 隐应会性为根
据播不种同数情的况1/而4。偏但离实3:验1上,,例分如离1是5:随1机等的情,况 单 基 因 •• 如 苗 体实长某未自,果际根些必则交在情占株能可种F况大系全1能(代未多,部A造a中必数萌萌aA成)混是 , 发 发,使A收有短 率 ,3a短:集(非根 特 就根1到长杂占 别 会,杂大F根交少 低 使但2交于3)苗代数 , 成总1F:, 种:2因 苗。/体代4子即1此的a上,a中混短a也(但a混有根应的短由有突个该种根于突变体是子)这变体
比较产物长度
• CAPs
= cleaved amplified polymorphic sequences 酶切扩增多态性
利用酶切位点
Marker: MIG5-B
HC C
L
• dCAPs = derived CAPS 设计引物引入错配碱基,从而引入酶切位点
其它标记
InDel
= insertion-deletion 插入缺失标记,指的是两 种亲本中在全基因组中的差异,相对另一个亲 本而言,其中一个亲本的基因组中有一定数量 的核苷酸插入或缺失。根据基因组中插入缺失 位点,设计一些扩增这些插入缺失位点的PCR 引物,这就是InDel标记。 部分SSLP就是由 InDel转化而来
在拟南芥的众多生态型中最常用的三种是 • Landsberg erecta(Ler) • Columbia(Col) • Wassilewskija(Ws)
• 其中Col生态型用于拟南芥的全基因组测序。
Marker
SSLPs CAPs dCAPs InDel SNP
• SSLP
= simple sequence length polymorphisms 简单序列长度多态性 又称 SSR= simple sequence repeats
孟德尔规律
• 二、非等位基因间的相互作用
无互作: 有互作:
9:3:3:1
9:7 互补作用 15:1 重叠作用 13:3 抑制作用 9:6:1 累加作用 9:3:4 隐性上位作用 12:3:1 显性上位作用
•END
Thanks for attention.
突
杂交F1代
变
突变体 x Ler
显性突变 AA(短根)
aa(长根)
Aa(短根)
隐性突变
杂交F2代 Aa(长根)
AA Aa(长根): aa(短根)
单
3 :1
基 因
杂交F2代
突
Aa(短根)
变 显性突变
AA Aa(短根): aa(长根) 3 :1
• 注意事项:一般出现的突变大多数为隐性 • 注突意变事,项因2:此由理于论根上长F1还代会都受为环长境根因(A素a影) ;响如,
• STEP3: 把目标区域内的BAC名字分次键入 到AMP的Marker搜索栏
• 注意:
• 每个株系到精确定位后期可能用到数十上 百的MARKER,请将自己使用、订制过的 MARKER的相关资料清晰记录下来,便于 日后查看。
目标区域内基因搜索
附加
• 由于我们以短根为筛选突变体的指标,因 此下面提到的突变的基因都应该会导致幼
重组子筛选
1. 建议使用一对位于相对确信的区域内的 次低重组率标记引用来筛选 2. 可以根据情况调整用来筛选 重组子的Marker 3. 要选择好用、绝大部分一次 就能扩出来的标记引物
引物寻找
• 1.山大丁老师 提供的引物 • 2.TAIR网上提供的常用Marker • 3.AMP上提供的常用Marker • 4.TAIR网上公布的所有多态性位点资料
于亲本Col中m4染色体区
段,在该差异位点两侧
设计引物经PCR扩增后得
到的PCR产物就会有不同
长度,电泳后M4泳动速
度慢,m4泳动速度快,
因此通过电泳就可以分
辨来自于两个亲本的染
色体区段
注意后两种情 况反映的是来 自于两个配子 的染色体区段 的情况
包含来自于 两个亲本的 染色体片段
包含来自于 Col 两 条 染 色体区段
• 图中的6、12号样品即为重组子
筛选重组子的目的
• 在筛选出6、12号两个重组子以后,假设 在目标区域内寻找到新的标记引物,就 不需要把所有的个体跑样,只需要跑重 组子个体
• 例: Rf Marker1 Marker2 Marker3
6号个体
1
0
1
12号个体 1
0
0
表格的跑样结果显示Marker2最接近突 变位点,Marker3次之,下一步可以 在Marker2和Marker3附近寻找新标 记引物
• 其他位点可能存在交换,利用带型差异判断交 换次数
Ler
Col
wild type
假设: Aa 突变位点 有5个Marker
mutant 杂交
图中的两个亲本不但在 A/a位点有差异,在其他 位点也存在大量的遗传 差异,可以利用这些差 异设计分子标记,对染 色体的具体区段进行标 定。在该例子中,该染 色体上总共确定了5个分 子标记,标定了5个不同 区段。
F1 plant
假设: 只存在单交换
自交
F2 plants
只有左侧三种植株会选入定位群体
分子标记M5与突变位点的遗传距离:
M5处发生交换的植 株包括三种情况
10/100
6/100
4/100
(20/(100×2 )) ×100%=10cM
假设: 统计100个个体
分子标记M4与突变位点的遗传距离:
苗短根。
隐性突变 突变基因: a(短根)
单 基
正常基因: A (长根)
因
突 变 显性突变 突变基因: A(短根)
正常基因: a (长根)
• 由于我们以短根为筛选突变体的指标,因
此下面提到的突变的基因都应该会导致幼
苗短根。
杂r
单
aa(短根) AA(长根)
基
因
Aa(长根)
• 密度: 每 6.6kb 存在一个
SNP
= single nucleotide polymorphism
• 单核苷酸多态性
• 主要是指在基因组水平上由单个核苷酸的变异 所引起的DNA序列多态性。SNP所表现的多态性 只涉及到单个碱基的变异,这种变异可由单个 碱基的转换或颠换所引起,也可由碱基的插入 或缺失所致。但通常所说的SNP并不包括后两种 情况。(后面两种情况的多态性一般归为InDel)
突各低情于况1/造4。成因的此偏,离统程计度AF无a2(表法短型估根时计),要短统根计大播于 变1法种便/分4总分显也辨数析性有,、。突可因萌变A能为发A只F数1A是的、a随(筛长机短选根分根和、离)收短造:种根成十a等的a分项(,重目长两要,根者,以)无要
谨防种子污染。 3 :1
补充
孟德尔规律
STEP1: 把目标区域内的Marker序列贴进TAIR 网上的Seqviewer上搜索目标区域
MPK6 FP:
AATCTTGTAATCTGGTGCGTG
• 点击目标区域
• STEP2: 把目标区域内的BAC名字记录下来 • 例如:K14A17至MRC8之间的BAC名字有
• K14A17→MCE21 →MGD8 →MTO12 →MKP6 →MIG5 →MEB5 →MBG14 →MRC8
• 20.00%
4.76%
12.5% 12.5% 36%
筛选 F2 代 短根 移栽
3周左右
提基因组
各对引物PCR
统计计算Rf
步骤总结
缩小范围 寻找新Marker 扩大群体 (筛选重组子)
什么是重组子
• 在突变位点附近区域内绝大部分 的样品跑样结果都应该为只有非 重组的Col条带,即记录Rf为0, 只有少数的样品存在交换,Rf记 为1。
• 一、显隐性关系的相对性
(1)完全显性:F1表现与亲本之一完全一样, 而非双亲的中间型或同时表现双亲的性状;
(2)不完全显性:F1表现为双亲性状的中间型。 (3)共显性:F1同时表现双亲性状,而不是表 现单一的中间型。
