教程说明(中)
INCA使用说明

INCA使⽤说明INCA6.2 使⽤说明天津同步动⼒有限公司尚震1.介绍本教程适⽤于使⽤INCA 的新⼿,INCA 主要特点:测量和标定。
使⽤此教程需要最基本的WINDOWS 操作能⼒。
1.1准备开始本教程必须保证系统装有INCA 软件。
本教程中的任务可以在演⽰模式下进⾏,这意味着你不需要任何实际硬件。
硬件模拟在ETK 测试设备和V A DI 测试设备。
该装置会随着INCA 软件安装。
确认0400.hex 和0400.a2l 这两个⽂件在\ETA SData\INCA6.2\Data\Demo 中。
最后请确认您使⽤本教程的计算机有⼀个互联⽹浏览器。
1.2建⽴⼀个DA TABA SE点击Database 选择new,然后输⼊⼯程的名字点击OK1.3 创建⼀个顶层⽂件夹点击Edi t->A dd->A dd to fol der输⼊⽂件夹名称并按回车键1.4 创建⼀个⼯作区单击Edi t->A dd->A dd fol der 并把⽂件夹命名为w orkspace选中w orkspac e 的⽂件夹,单击Edi t->A dd->Workspac e 并命名为OneETK1.5 添加⼀个⼯程先新建⼀Fol der 命名为004 并将其选中,单击Edi t->A dd->ECU proj ec t(A2L)然后添加\i nc a6.2\data\04001.6把⼯程添加到w orkspac e 中选中⼯作区OneETK,单击4 proj ec t/devi c e 下⾯打开按钮右边的选中 0400 单击OK单击 4 proj ec t/devi c e会出现许多设备,选中 ETKtestdevi c e 单击 OKINCA 界⾯会变成1.7添加V A DI 硬件组件下⾯的单击5 H ardw are选中ETK test devi c e 单击右键选中Insert选中V A DI test devi c e 单击OK然后会出现下⾯的界⾯关闭上⾯对话框返回硬件连接会显⽰然后 H ardw are->H ardware status会显⽰2.建⽴⼀个实验下⾯将学习如何将变量和窗⼝添加到实验中,并如何配置功能。
MICROCHIP 公用仪表基础教程 说明书

公用仪表基础教程 2006 Microchip Technology Inc.DS39757A_CNDS39757A_CN 第 ii 页 2006 Microchip Technology Inc.提供本文档的中文版本仅为了便于理解。
MicrochipTechnology Inc.及其分公司和相关公司、各级主管与员工及事务代理机构对译文中可能存在的任何差错不承担任何责任。
建议参考Microchip Technology Inc.的英文原版文档。
本出版物中所述的器件应用信息及其他类似内容仅为您提供便利,它们可能由更新之信息所替代。
确保应用符合技术规范,是您自身应负的责任。
Microchip 对这些信息不作任何明示或暗示、书面或口头、法定或其他形式的声明或担保,包括但不限于针对其使用情况、质量、性能、适销性或特定用途的适用性的声明或担保。
Microchip 对因这些信息及使用这些信息而引起的后果不承担任何责任。
未经Microchip 书面批准,不得将Microchip 的产品用作生命维持系统中的关键组件。
在Microchip 知识产权保护下,不得暗中或以其他方式转让任何许可证。
商标Microchip 的名称和徽标组合、Microchip 徽标、Accuron 、dsPIC 、K EE L OQ 、micro ID 、MPLAB 、PIC 、PICmicro 、PICSTART 、PRO MATE 、PowerSmart 、rfPIC 和SmartShunt 均为Microchip Technology Inc .在美国和其他国家或地区的注册商标。
AmpLab 、FilterLab 、Migratable Memory 、MXDEV 、MXLAB 、PICMASTER 、SEEVAL 、SmartSensor 和The Embedded Control Solutions Company 均为Microchip Technology Inc .在美国的注册商标。
剪映电脑版使用说明教程
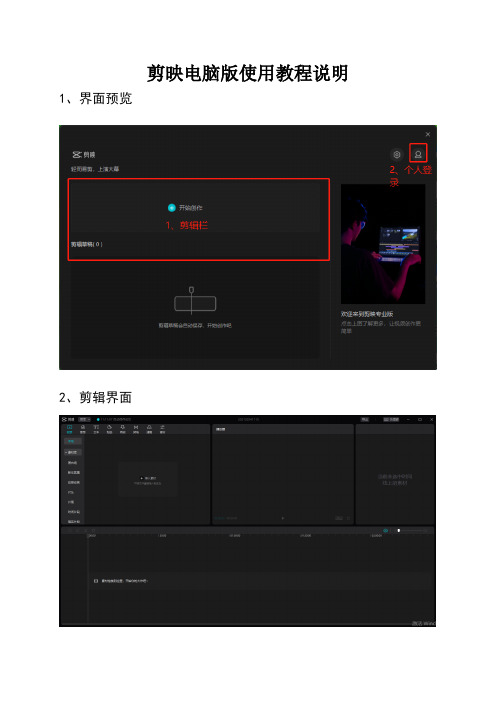
剪映电脑版使用教程说明1、界面预览2、剪辑界面菜单栏主要有视频、音频、文本、贴纸、特效、转场、滤镜、调节8大部分组成。
3.1.1素材栏:此次主要是你导入的视频和照片,以及剪映自身随提供的素材(在素材库中)。
3.1.2预览栏:播放可以查看你选择的素材内容。
3.1.3剪辑栏:又称为工作区或者轨道栏,剪映支持多轨道剪辑,可以把导入的素材拖拽到上面进行剪辑加工处理。
3.1.4特效栏:主要是对你导入的素材进行处理,包括画面、声音、速度的调整。
3.2特效栏讲解3.2.1 画面3.2.1.1 透明度通过调节透明度,两层轨道可以实现叠化的效果。
如图所示。
通过调节第一上层的透明度,实现了2层画面叠加的效果。
3.2.1.2 磨皮瘦脸此项功能主要是针对美颜效果的,人像使用。
3.2.1.3 混合模式主要功效是可以用不同的方法将对象颜色(上层)与底层对象(下一层)的颜色混合。
当您将一种混合模式应用于某一对象时,在此对象的图层或组下方的任何对象上都可看到混合模式的效果。
混合模式包括:滤色、变暗、变亮、叠加、强光、柔光、线性加深、颜色变暗、颜色加深、正片叠底。
这几种混合使用的时候,可以得到不同的效果。
我经常使用的是滤色模式。
3.2.2 音频3.2.2.1 音量主要是对画面的声音高低进行调节。
可以在红框地方点击下,直接输入数值,也可在右侧蓝框处点击上下的小箭头进行详细的微调整,确保得到更好的效果。
3.2.2.2变声变声模式主要有:大叔、萝莉、女士、男生、怪物,根据制作效果自行选择。
可以把原有的视频的声音变成不同的声音。
3.2.3 变速此项功能是对视频的速度进行调整,通过调整倍数实现快镜头和慢镜头的效果。
倍数越大,视频速度越快;倍数越小,视频速度越慢。
当视频的速度加快时,视频时长减少,当视频速度变慢时,视频时长增加。
也可以自定义视频的时长,实现快慢镜头的效果。
例如你选择的视频时长是30秒,你可以自定义时长为5秒。
声音变调可选择是否开启,开启后,原来视频的声音音调会随着视频速度变化进行变化。
flow3d官方培训教程中的实例中文说明

3D学习——3 算例1新建一个项目, & :\3D\\\,使用缺省选项,因为将引入其它形状作为固体, 1中坐标范围()为:X: 5.0~15.0, Y: 5.0~15.0, Z: 0.0~15.0的单位对3D来说是未知的,可能是英寸、英尺、毫米等,现在假设模型是(国际单位),那么流体或固体的属性都应该是的。
(这里有些糊涂,3D会使用文件中的单位么?)模拟的情况为从圆柱形底部入口向球形水箱内充水,计算域应该和此形状范围相近,略大一点但不能紧贴着形状边界。
底边界的位置和边界条件类型有关,如果入口处流速已知那么模拟多少入口长度没有关系,因为断面形状是固定的,但是如果特定位置的压力是已知的,那么要把边界放在该位置处因为压力会受入口长度的重力和粘性效应影响而变化。
建议计算域要大于最大几何尺寸的5%,底边界除外,可以小于5%,这样计算域底部和入口交叉,不会挡住水流,因此计算域定义为X: 4.95~15.05 Y: 4.95~15.05 Z: 0.05~15.05在的 1中按上面参数修改计算域尺寸,然后在 1上右键选择更新显示。
= 数= = ν= 数= = gΔρL^2/σ= 数= = ^2ρ/σU是特征流速,L是特征长度,g是重力加速度,ρ是密度,σ是表面张力系数。
这个问题中大约用100s充满水,冲水体积540立米,入口直径2m,入口流速为540/(100*π*1^2)=1.7,数为1.7*2/(1.06)=3.4e6由于数远大于1,因此惯性力比粘性力更重要,即不需要很密的网格考虑粘性力,也不需要指定粘性特性。
考虑表面张力影响的系数为= gΔρL2/σ = 9.8^2 * 1000 ^3 * (2m)^2/(0.073^2) = 5.4e5= 2ρ/σ = 2m * (1.7 )^2 * 1000 ^3 / (0.073^2) = 7.9e4可以看出表面张力也不需要考虑。
缺省网格在X、Z方向为10,Y方向为1。
儿童文学教程教案中编

儿童文学教程教案——中编中编教学内容教学思路基本说明中国儿童文学部分教材以中国现代儿童文学为主,当代儿童为学为辅。
基本依据史的线索来进行。
中国现代儿童文学更多的是理论的探索,实践略少,优秀作品不多,但里程碑作品值得提及。
这些作品无论是思想上还是艺术上都带上了比较明显的时代烙印。
鉴于实际情况和教学的需要,本编计划时间不多。
以线索和作家的形式带过,让学生对这段儿童文学史有个大概的了解就可以了。
但当代儿童文学这块要加强,多为学生推荐当代优秀的儿童文学作家和作品。
第一章中国儿童文学概况第一节中国儿童文学的萌发儿歌(童谣):我国第一部个人搜集整理的儿歌集——明代吕坤编著的《演小儿语》。
神话:集中保存在《山海经》《楚辞》《穆天子传》等古籍中。
寓言、童话:《战国策》等古书中有所记载。
这些具有民间印记的作品,对儿童文学的萌发更有较为直接的作用。
古代小说中也蕴藏这一定的儿童文学因素。
《世说新语》《搜神记》《三国志》《镜花缘》《西游记》《聊斋志异》等。
古代文学与启蒙读物等的有关章节、篇什,在漫长的岁月中,不仅成为儿童的精神食粮,而且作为中国儿童文学之萌芽的种种初级形态,与口传的儿童文学一道,为以后儿童文学的发展提供了很多宝贵的经验。
清末以来,萌发状态中的儿童文学逐渐出现了自成一宗的趋向。
这与相关的艺术传统有关,也于现实发展及其需要有关。
第二节中国现代儿童文学中国现代儿童文学产生于五四时期。
就五四时期的创作情况看,各种体裁均有上乘之作:童话方面:茅盾(《寻快乐》)、郑振铎(《朝露》)、赵景深(《一片槐叶》)陈衡哲的《小雨点》等,尤其是叶圣陶的《稻草人》。
儿童小说方面:鲁迅(《故乡》)、叶圣陶(《一课》)、冰心(《三儿》)、王统照《雪后》等儿童散文方面:鲁迅的《风筝》、冰心的《寄小读者》、刘半农《饿》、许地山《落花生》、丰子恺《华瞻的日记》等儿童剧方面:郭沫若的《黎明》,黎景晖的《葡萄仙子》等儿童诗方面:郭沫若的《天上的市街》、沈从文的《忆》等五四时期的儿童文学,虽然处于发轫阶段,但却呈现出繁茂的景象,较之以往的儿童文学与儿童读物,在主题、题材、乃至于格调等方面都有重大的变化。
Analyst中文操作手册

Analyst 软件基本操作培训教程 美国应用生物系统中国公司关于本教程的说明:本教程为应用生物系统中国公司在中国使用的客户培训的辅助材料。
本教程仅提供给经过应用生物系统中国公司培训合格的操作人员使用。
本教程内容仅限于Analyst软件基本操作部分。
本教程解释权在美国应用生物系统中国公司。
版本号:A目录I. PPG质量校准1手动质量校准1-1 开软件,连机,进入手动调谐状态,调用己有的质量校准方法1-2 进质量校准溶液,调离子源喷雾针位置1-3 采集PPG标准品质谱数据1-4 质量校准数据计算1-5 手动调整分辨率1-6 质量校准2自动质量校准2-1 自动质量校准的设定2-2 自动优化结果II A.针泵进样ESI源MS-MS 方法手动优化1.确定母离子:Q1 单级质谱实验(Q1全扫描)2.Product Ion Scan(碎片离子扫描)3.碎片离子扫描之CE单参数优化4.Precursor Ion Scan(母离子扫描)5.Neutral Loss Experiment(中性丢失扫描)II B. 针泵进样ESI源MRM定量方法手动优化1.先确定母离子:Q1 单级质谱实验(Q1全扫描)2.检查信号稳定性3.确定子离子Product Ion Scan(碎片离子扫描)4.优化MRM 定量分析质谱参数4-1 优化Q1参数使灵敏度最高及稳定性良好4-2 继续优化MRM离子对的CE 与 CXPIII. 针泵进样ESI源MRM定量方法参数自动优化1.先确定母离子:Q1 单级质谱实验(Q1全扫描)2.检查信号稳定性3.定量参数自动优化3-1 MRM自动优化的设置3-2 MRM 参数自动优化的过程3-3 MRM 电压参数自动优化的例子IV. 离子源TIS连液相用流动注射手动优化V. 离子源TIS连液相用流动注射自动优化VI. 带液相条件质谱采集方法和批文件的建立和使用1-1 调节离子源位置1-2 建立Acquisition Method采集方法1-3 建立 Acquisition Batch采集批文件VII. 定量分析方法的建立和数据分析1A:用 Wizard导航器建立一个定量数据分析方法 (手动方式)1B:用已建立的定量数据分析方法分析数据2 查看和编辑定量结果表2-1查看和编辑结果列表中的各列,如何取消Audit trail(稽核跟踪)原因设定2-2查看校准曲线。
诺基亚QD教程

QD详细教程与说明大全(大家最好看看,可能可以帮你解决你所遇到的问题)【格卡】方法一(手机格卡):在手机上进入1功能表→2工具→3存储卡→4(左软键选择“格式化存储卡”)→5完成退出。
注:在进行到第4步时,有很多选项可供选择,大家依字面意思就可理解作用(都是对MMC卡进行操作的)。
方法二(电脑格卡):1按一下关机键→2选择“取出存储卡”→3在屏幕上提示可以取卡时将MMC卡从手机中取出→4将卡插入读卡器→5连接到电脑USB接口上→6在电脑中多出来的“可移动磁盘”上点击…右键‟7→点击“格式化”→8在弹出的面版上将“FAT32”改为“FAT”如图(MMC卡不是第一次格卡的话,可以选择快速格式化)→9单击确定开始格卡→10格卡完毕后,在MMC卡上新建五个文件包,即:“Images-Others-Sounds-System -Videos”。
注:方法一很慢,而且不太安全,在卡上文件较多或有错的情况下,格卡常常会失败,并提示卡已损坏。
方法二很快,并且不会出错,除非MMC卡真损坏。
但是需要有电脑和读卡器。
【格机】在待机情况下输入*#7370#,手机会重起并自动进行格式化C盘和将系统备份还原到C 盘的操作,当中会出现输入锁码的提示,如果你没有更改过锁码,那么默认是12345,输入即可继续。
格机大概会在数分钟以内完成,没有具体测试过时间,总之是用不了多久啦!我在格机时是将MMC卡取出后再格的,这样格机后,卡上绝大多数程序是可以正常运行的。
格机后C盘就只剩下系统了,以前在菜单中的前边8个选项会保留,因为那是属于系统内的组件,除自己安装之外的各类输入法也会保留,初次格机的朋友不用但心会少了什么东西。
格机后,所有注册信息、个人设置、名片夹内的信息等等,将被全部删除,还有线路更改的快捷键功能会被取消(个人觉得这个功能对于绝大多数朋友来说是一点用也没有,包括本人也觉得毫无用处)。
总之,失去的一切程序都是可以再次安装的,这些程序在网上也都容易找到。
SACS中文教程

SACS中⽂教程SACS 中⽂教程建模流程简叙.1启动程序启动SACS 5.2 Executive程序,出现如下主界⾯:点击左下⾓的“Directory”选项卡,在“CURRENT DRIVE”中选择⽂件所在的硬盘盘符;在CURRENT DIRECTORY 窗⼝中选择⽂件存储⽬录。
CURRENT DIRECTORY窗⼝CURRENT DRIVE 选项框双击“INTERACTIVE”窗⼝中的“MOEL”按纽,出现如下界⾯:选择“Create new model”,点击“OK”按纽确认。
出现如下界⾯:中选择“JACKET “(导管架)类型,使⽤向导建模。
根据向导出现的界⾯,依次输⼊以下数据:根据以上步骤,已建⽴了导管架的主框架,见下图,我们可以根据设计图纸或设计思路,接下来建更详细的模型。
灵活的运⽤向导可以节省建模的时间。
尤其是对于有斜度的导管架、塔等采⽤向导建模会相对简单些,且不容易出错。
通⽤的建模规则.1点的建⽴2.1.1点坐标系的定义⼀般以平台轴线围成的四边形的中⼼作为原点;X轴:平台北向为X轴正向;Y轴:平台东向为Y轴正向;Z轴:垂直⽔⾯向上为Z轴正向,零点为海图⾯;2.1.2 点的命名⼀个平台整个模型包括有很多模块,⼤概有成千上万个点构成,为⽅便建模(模型的导⼊等)及校对,有序的点编号将使模型变得有条理,便于管理。
根据以往设计的经验对整个平台每个模块结构上的点的命名进⾏了规范。
z导管架点的命名规则以下我们以四条腿的导管架举例来说明导管架点的命名⽅法:1、导管架腿上的点命名以xxxL(L代表leg),第⼀个x为其导管架的层数。
后两个根据实际需要编号;2、每层平⾯内点的命名以Hxxx(H代表HORIZONTAL),第⼀个x为层数。
后两个xx根据实际需要编号;3、对⽴⾯上x⽀撑的交点的命名以Xxxx(x代表x-brace)第⼀个x跟第⼆个x代表上下两层的层数,第三个x根据实际情况编号;z上部组块点的命名规则以下我们以四条腿的上部组块举例来说明上部组块点的命名⽅法:1、上部组块上的点命名以A(B/C/D..)xxx(L代表leg),第⼀个字母表⽰层数,第⼀层为A开头,第⼆层为B开头依次类推,第⼆、三不⽤字母,均使⽤数字编号,如果表⽰的点是在腿上,则最后⼀个数字⽤L表⽰。
SRIM软件使用说明教程(中文)

教程#1——离子在固体中的射程、剂量及辐照损伤简述该教程将介绍如何确定离子的能量和剂量,使其注入靶后能达到我们所要求的浓度和深度。
为了说明这一点,我们以在CMOS 半导体器件中注入n 型井为例。
注入硅中的离子(即注入原子)应为n 型元素,并在表面以下约为250 nm(2500Ǻ)深处达到浓度峰值(以投影射程计)。
掺杂原子的浓度峰值为每平方厘米5×1018个离子。
尽管这看起来有些复杂(特别是如果你不是一个电气工程师的话),但它只要求磷(P )或砷(As )或锑(Sb )元素的离子被直接注入到样品的一定深度并形成一定的浓度(磷、砷和锑原子都是硅中的n 型掺杂剂)。
作为一个附加条件,我们假定注入离子(即加速器加速的粒子)的能量不超过200keV 。
【注意:TRIM 很多情况下使用Ǻ(埃)作为单位是因为其大约是固体中单层原子的厚度。
这常用于估计靶的微观损伤。
】这一系列问题将是该教程的主旨。
在阅读完本教程之后,你将能够回答将任意离子注入到任意靶材料情况下的这些问题。
确定入射离子的种类和能量● 点击桌面上的SRIM 图标● 点击Stopping and Range Tables (S&R Tables ) 首先输入离子。
开始可以点击在“ION ”旁边上的帮助按钮。
阅读后点击键关闭窗口。
为了在硅中注入形成一个n 型井,你需要从元素周期表的第五列中选择一种元素来作为杂质元素注入。
典型的掺杂元素是磷(P )、砷(As )或锑(Sb )。
我们选项居中的砷(As )开始。
要键入一种离子,点击窗口中Ion 边上的键打开元素周期表并选择As 作为入射离子。
程序将会自动填充描述入射离子性质的各种选项框。
注意到其使用的离子质量并不是砷问题: ●注入何种元素? ●需要注入多大的剂量(ions/cm 2)? ●靶在注入后是否会产生非晶化? CLOSEPT的平均原子质量,而是丰度最大的同位素(MAI )的质量。
LTspice-一简介(中文教程)

免费电路图仿真软件LTspice 一简介(中文教程)欢迎转载,转载请说明出处-DPJ关键字:PSpice 仿真,电路图,LTspice仿真,pspice模型,spice,电路仿真,功放电路图仿真,信号放大仿真1. LTspice 电路仿真软件简介LTspice 电路图仿真软件简介(支持PSpice和Spice库的导入)LTspiceIV 是一款高性能Spice III 仿真器、电路图捕获和波形观测器,并为简化开关稳压器的仿真提供了改进和模型。
我们对Spice 所做的改进使得开关稳压器的仿真速度极快,较之标准的Spice 仿真器有了大幅度的提高,从而令用户只需区区几分钟便可完成大多数开关稳压器的波形观测。
这里可下载的内容包括用于80% 的凌力尔特开关稳压器的Spice 和Macro Model,200 多种运算放大器模型以及电阻器、晶体管和MOSFET 模型。
在电路图仿真过程中,其自带的模型往往不能满足需求,而大的芯片供应商都会提供免费的SPICE模型或者PSpice模型供下载,LTspice可以把这些模型导入LTSPICE中进行仿真。
甚至一些厂商已经开始提供LTspice模型,直接支持LTspice的仿真。
这是其免费SPICE 电路仿真软件LTspice/SwitcherCADIII所做的一次重大更新。
这也是LTspice 电路图仿真软件在欧洲,美国和澳大利亚,中国广为流传的根本原因。
LTspice IV 具有专为提升现有多内核处理器的利用率而设计的多线程求解器。
另外,该软件还内置了新型SPARSE 矩阵求解器,这种求解器采用汇编语言,旨在接近现用FPU (浮点处理单元) 的理论浮点计算限值。
当采用四核处理器时,LTspice IV 可将大中型电路的仿真速度提高3 倍,同等设置的精度,电路仿真时间远远小于PSpice的计算时间(本来你要等待3个小时,现在一个小时就结束了)。
功能强大而且免费使用仿真工具,何乐而不为呢?这里不是贬低pspice软件,cadence的Pspice软件具有更加丰富的配置和应用,可以进行更加繁多的电路仿真和设置,因为大多数工程师不需要非常复杂的应用,所以,免费的LTspice可以满足基本的应用。
PowerDesigner使用教程中文
使用PowerDesigner以下说明介绍本实验用PowerDesigner中的部分功能,由于版本不同与实验实际环境略有差异。
●打开PowerDesigner软件开始菜单->程序->Sybase->PowerDesigner 12●画UML图1.新建Object-oriented ModelFile->new…选择Object-oriented Model在上图中选择use-case。
选择画板(Palette)中的不同图形画use case图。
3.修改属性双击角色、用例或联系,打开属性对话框,修改其属性。
注意: (任务2)对每个Use-case详细描述:双击打开use case属性对话框,选择specification页。
4. 检查模型在workspace选中要检查的模型,主菜单tools->check model或快捷键F4,检查模型是否有错误,改正错误5.(任务3)新建其他UML模型在workspace中选中所在的object-oriented model,单击鼠标右键弹出菜单,选择new 新建其他模型。
对于活动图(activity diagram)和时序图(sequence diagram),请自行学习作图方法(参见帮助)。
(任务4)画E-R图一、新建概念数据模型1)选择File-->New,弹出如图所示对话框,选择CDM模型(即概念数据模型)建立模型。
2)完成概念数据模型的创建。
以下图示,对当前的工作空间进行简单介绍。
3)选择新增的CDM模型,右击,在弹出的菜单中选择“Properties”属性项,弹出如图所示对话框。
在“General”标签里可以输入所建模型的名称、代码、描述、创建者、版本以及默认的图表等等信息。
在“Notes”标签里可以输入相关描述及说明信息。
当然再有更多的标签,可以点击"More>>"按钮,这里就不再进行详细解释。
flow3d官方培训教程中的实例中文说明(可编辑修改word版)

Flow3D学习——3 算例1 Aerospace TutorialAerospace Tutorial新建一个项目,Model Setup Tab-Meshing & Geometry Tab-SubcomponentTab-Geometry Files-c:\Flow3D\gui\stl_lib\tank.stl,Type and Potential使用缺省选项,因为将引入其它形状作为固体,Subcomponent 1中坐标范围(Min/Max)为:X: 5.0~15.0, Y: 5.0~15.0, Z: 0.0~15.0tank.stl的单位对FLOW-3D来说是未知的,可能是英寸、英尺、毫米等,现在假设模型是SI(国际单位),那么流体或固体的属性都应该是SI的。
(这里有些糊涂,FLOW-3D会使用STL文件中的单位么?)模拟的情况为从圆柱形底部入口向球形水箱内充水,计算域应该和此形状范围相近,略大一点但不能紧贴着形状边界。
底边界的位置和边界条件类型有关,如果入口处流速已知那么模拟多少入口长度没有关系,因为断面形状是固定的,但是如果特定位置的压力是已知的,那么要把边界放在该位置处因为压力会受入口长度的重力和粘性效应影响而变化。
建议计算域要大于最大几何尺寸的5%,底边界除外,可以小于5%,这样计算域底部和入口交叉,不会挡住水流,因此计算域定义为X: 4.95~15.05 Y: 4.95~15.05 Z: 0.05~15.05在Mesh-Cartesian的Block 1中按上面参数修改计算域尺寸,然后在Block 1上右键选择Update Mesh更新显示。
Re = Reynold数 = Inertial Force/Viscous Force = UL/νBo = Bond数 = Gravitational Force/Surface Tension Force = gΔρL^2/σWe = Weber数 = Inertial Force/Surface Tension Force = LU^2ρ/σU是特征流速,L是特征长度,g是重力加速度,ρ是密度,σ是表面张力系数。
stata16中文入门教程.pdf说明书

Stata软件入门教程李昂然浙江大学社会学系Email: ********************版本:2020/02/051. 导论本教程将快速介绍Stata软件(版本16)的一些基本操作技巧和知识。
对于详细的Stata介绍和入门,小伙伴们可以参考Stata官方的英文手册以及教程所提供的学习资料。
跟其他大多数统计软件一样,Stata可以同时通过下拉菜单以及命令语句来操作。
初学者可以通过菜单选项来逐步熟悉Stata,但是命令语句的使用是Stata用户的最佳选择。
因此,本教程将着重介绍命令语句的使用。
对于中文用户来讲,在打开Stata之后,可以通过下拉菜单选项中的用户界面语言选择将中文设置为默认语言。
同时,也可以在命令窗口中输入set locale ui zh_CN来设置中文显示。
在选择完语言后,记得重新启动Stata。
需要提醒大家,虽然Stata用户界面可以显示中文,但是统计分析的结果仍然将以英文显示。
本教程中使用的案列数据源自中国家庭追踪调查(China Family Panel Studies)。
具体数据出自本人于2019年发表于Chinese Sociological Review上“Unfulfilled Promise of Educational Meritocracy? Academic Ability and China’s Urban-Rural Gap in Access to Higher Education”一文中使用的数据。
关于数据的具体问题,请联系本人。
同时,本教程提供相应的do file和数据文件给同学们下载,同学们可以根据do file复制本教程的全部内容。
下载地址为我个人网站:https://angranli.me/teaching/温馨提示:关于Stata操作的大多数疑问,都可以在官方手册上找到答案。
同时,在Stata中输入help [command]便可以查看关于命令使用的详细信息。
HPDS2017教程程序使用说明

公路路面设计程序系统(HPDS2017)使用说明本说明由十一个部分和四个附件组成,它们是:一、系统总说明 ----------------------------------------------------------------------- 1二、系统主菜单窗口使用说明 ----------------------------------------------------------- 5三、改建路段留用路面结构顶面当量回弹模量计算程序(HOC)使用说明----------------------- 6四、沥青路面设计与验算程序(HAPDS)使用说明 ------------------------------------------ 8五、路基验收时路段内实测路基顶面弯沉代表值计算程序(HOCG)使用说明-------------------- 15六、路面交工验收时路段内实测路表弯沉代表值计算程序(HOCA)使用说明-------------------- 17七、改建路段原路面当量回弹模量计算程序(HOC1)使用说明-------------------------------- 19八、新建单层水泥混凝土路面设计程序(HCPD1)使用说明 ---------------------------------- 21九、新建复合式水泥混凝土路面设计程序(HCPD2)使用说明 -------------------------------- 28十、旧混凝土路面上加铺层设计程序(HCPD3)使用说明 ------------------------------------ 33十一、基(垫)层或加铺层及新建路基交工验收弯沉值计算程序(HCPC)使用说明 ------------- 38附件一、沥青路面材料代码与材料名称对照表 --------------------------------------------- 40 附件二、水泥混凝土路面基(垫)层材料代码与材料名称对照表 ----------------------------- 43 附件三、版权声明 --------------------------------------------------------------------- 44 附件四、作者简介 --------------------------------------------------------------------- 44现分别叙述如下:一、系统总说明1.本系统是根据新发行的《公路沥青路面设计规范》JTG D50-2017和已发行的《公路水泥混凝土路面设计规范》JTG D40-2011的有关内容编制的,共包括如下九个程序:(1)改建路段留用路面结构顶面当量回弹模量计算程序HOC(2)沥青路面设计与验算程序HAPDS(3)路基验收时路段内实测路基顶面弯沉代表值计算程序HOCG(4)路面交工验收时路段内实测路表弯沉代表值计算程序HOCA(5)改建路段原路面当量回弹模量计算程序HOC1(6)新建单层水泥混凝土路面设计程序HCPD1(7)新建复合式水泥混凝土路面设计程序HCPD2(8)旧混凝土路面上加铺层设计程序HCPD3(9)基(垫)层或加铺层及新建路基交工验收弯沉值计算程序HCPC2.系统的特点(1)采用Visual Basic 6.0 for Windows 语言编程,在Windows系统下运行,有良好的用户界面;(2)功能齐全,凡公路路面设计与计算所需的程序应有尽有;(3)计算速度快,精度高;(4)数据输入采用可视化、全屏幕的窗口输入方式,操作简单方便,一目了然。
Victoria 4.2 中文图文使用说明教程
Victoria 4.2 中文图文使用说明教程介绍:一款具备硬盘表面检测/硬盘坏道修复/SMART信息察看保存/Cache缓存控制等多功能的工具;工具支持众多型号硬盘解密;支持全系列检测和修复;程序版本:4.2(当前最新测试版)运行平台:windows系列平台版本说明:在4.x版本以前的3.x都是用于DOS平台,并且改进了PRO接口和众多以前在DOS版本无法处理的功能,支持IDE/SATA硬盘,在硬盘密码操作方面;可以备份BIN文件获得察看和修改;支持最新标准ATA-8协议;可以对处理速度做出对比;那么我们首先了解这个程序吧 ^_^下载:Victoria 4.2下载图一:Victoria 4.1程序文件程序中文使用说明:打开程序后显示图一窗口:图二:程序主界面注意: 进入程序后,首先查看状态指示灯DRSC与DRDY是否为蓝色状态;当出现BUSY或者IDNF/ABRT等状况,请重新启动硬盘,硬盘没有就绪则除电源控制等综合通用通能外;修复执行等命令无法进行;图三:Standard界面窗口Standard面板说明:1)红色标注框为主菜单选项按钮;2)蓝色部分为Standard设置窗口3)粉红色为Standard子设置窗口4)黄色部分为公用控制面板5)橘红色部分为硬盘状态指示灯信息面板Standard窗口关键按钮解释:端口/资源选择图四:端口扫描/资源选择(红色框)PORT 端口Custom 自定义Primary 主盘Secondary 从盘端口资源扫描结果显示图五:端口资源扫描结果显示(蓝色框,这里可以直接点击选择)PCI SCAN:扫描SATA/ATA端口;Host protected area ^|hV FM 2RHPA:取得原始LBA;HPA:设置新的LBA;Security manager:Lock:SET PASSWORD 设置密码 ] l +<-Unlock:解锁Erase:清除Master:主控制密码High:高级密码User:用户密码Max:最大长度一:硬盘检测与坏道修复:进入程序如下图:上图中DRSC与DRDY指示灯可以看出,硬盘未准备就绪或者端口没有选择;Standard----port内选择端口;选择待修硬盘端口,选择后如下图显示;DRSC与DRDY指示灯准备就绪;port显示为我们待修硬盘端口;正确选择后,进入TESTS功能窗口;如下图:上图绿色部分设置测试方式;然后选择红色标注部分;测试将会开始;绿色部分设置名词,可以参考本站MHDD说明内术语;使用ERASE/REMAP/Defectoscop这几个功能使用后都可以起到修复缺陷的功能;Trackback: /TrackBack.aspx?PostId=1165238Victoria 4.3硬盘检测软件的使用方法在网上都可以找到,但是都是抄来抄去。
CTL映射在R中的教程说明书

CTL mapping in RDanny Arends,Pjotr Prins,and Ritsert C.JansenUniversity of GroningenGroningen Bioinformatics Centre&GCCRevision#1First written:Oct2011Last modified:Jan2018Abstract:Tutorial for the Correlated Trait Loci(CTL)mapping package to reconstruct genetic regulatory networks using the R package’ctl’and the mapctl commandline tool.This tutorial is targetted at people with a basic understand-ing of genetics and would like to analyse there inbred cross(RIL,F2,DH,BC)or any outbred population(e.g.Human GW AS)using SNP markers.The main focus will be explaining the functionality of the software,and a how-to format your data for R when using datasets currently stored in Excel format,tab delimitedfiles(.csv)or use read.cross from R/qtl.In the methodology and background only a short introduction in CTL mapping theory is given.Software:/download.htmlManual:/manual.htmlSource-code:/DannyArends/CTLmappingE-mail:**********************Some words in advanceWelcome reader to this tutorial,you’ll be thrown into the world of network reconstruction using differential correla-tions.The CTL mapping methodology is not the easiest but the results can be of great help unraveling trait to trait interactions in all types of crosses.In this tutorial I’ll try to use as few difficult concepts as possible.But some always sneak in.Enjoy this tutorial,and good luck hunting that missing heritability.Danny Arendse a good text editore a repository/backup systemBackground&MethodologyDifferences in correlations between traits within an inbred population are determined at each genetic marker.Pheno-types are assigned to genotype groups and a single phenotype is used to scan all other phenotypes for a loss or gain of correlation.The likelihood profiles(similar to QTL profiles)of this’loss of correlation’measurement shows a very high degree of overlap with classical QTL profiles.However additional information is available from the phenotype x phenotype interactions.With the right dataset(ideally a combination of:classical phenotypes,protein abundance and gene expression)CTL shows the genetic wiring of the classical phenotypes and identify key players in the genetic/ protein network underlying classical phenotypes using QTL and CTL information.Format and load your dataLoading your Excel/CSV dataUse the example excelfile and simply replace the example data by your own then:F ile−>SaveAs−>T ODOAfter you have stored the data as plain text open thefiles in a text editor(do not format data using MS WORD,for windows Notepad++is a good option)and check to see if your data is formatted correctly.If this is not the case,please reformat your data to match the example format shown below.The genotypes.csvfile containing the genotype matrix is stored individuals(rows)x genetic marker(columns): Marker1Marker2Marker3...MarkerNInd1111 (2)Ind2122 (1)Ind3221 (1)..................IndN111 (2)The phenotypes.csvfile containing individuals(rows)x traits(columns)measurements:Trait1Trait2Trait3...TraitNInd1 5.811.09.0... 1.6Ind2 6.312.2NA... 1.3Ind3 5.111.112.3... 2.0..................IndN9.815.823.0... 3.4NOTE:Individual order must match between the genotype and the phenotypefile...After verifying yourfiles are formatted correctly(this is the source of most errors).Start R and load in the data,check that the output looks like below:>setwd("/Path/To/Data/")>genotypes<-read.csv("genotypes.csv",s=1,header=FALSE,sep="\t")>traits<-read.csv("phenotypes.csv",s=1,header=FALSE,sep="\t")>genotypes[1:5,1:10]#Show5individuals,10markers>traits[1:5,1:10]#Show5individuals,10traitsUse an R/qtl formatted datasetProvided is the main interface functions to R/qtl:CT Lscan.cross()this functions accepts and R/qtl formatted cross object as input and will scan CTL,perform permutations and transform the detected differential correlation to LOD score matrices.As an example we show the code to load the internal multitrait dataset provided in R/qtl,load your own by using the read.cross()function:>require(qtl)#Loads the R/qtl package>data(multitrait)#Loads the dataset>multitrait#Print basic dataset information>?read.cross#List of formats supported by R/qtlAdding genetic map informationThis step is optional,but plots and generated networks look much nicer when chromosome locations of genetic mark-ers are supplied.The’mapinfo’object is matrix with3columns:"Chr"-the chromosome number,"cM"-the location of the marker in centiMorgans and the3rd column"Mbp"-The location of the marker in Mega basepairs.NOTE:Marker order must match with the genotypefile...The structure of the mapinfo.csvfile:Chr cM MbpInd11 1.00.01Ind21 1.20.34Ind31 3.1 1.3............IndN24120.820.3We load the genetic map data into R by using the following command:>mapinfo<-read.csv("mapinfo.csv",s=1,s=TRUE)>mapinfo[1:5,1:3]#Show the first5marker recordsFinally...after all this we can start scanning QTL and CTL...Scanning for CTL/CT LscanWe start explaining how to map CTL using the basic options of the CT Lscan function.This function scan CTL and produces an output"CTLobject".>require(ctl)#Loads the R/ctl package>data(ath.metabolites)#Loads the example dataset>geno<-ath.metab$genotypes#Short name>traits<-ath.metab$phenotypes#Short name>#Scan all phenotypes against each other for CTLs,using the default options>ctls<-CTLscan(geno,traits)If you have loaded an R/qtl"cross"object,use the CT Lscan.cross interface,this will automagically extract the cross, check if the type of"cross"is supported,and deal with the geno.enc parameter:>library(qtl)>data(multitrait)>ctls<-CTLscan.cross(multitrait)Now you might feel tempted to just dive into the results,But...first lets take a look at some options that might apply to your experimental setup like treatments applied or growth conditions,and which(if set incorrectly)could seriously reduce mapping power.Also you might want to add your own/previous QTL results.CTLscan optionsCTL mapping can be performed using three different strategies("Exact","Full",and"Pairwise")to calculate signif-icance.The default strategy"Exact"is the fastest one,but the statistical power of this stategy is poor and it doesn’t handle data distributions with a lot of outliers properly.For publication it is better to use one of the other strategies "Full"or"Pairwise"since these strategies perform permutations,which deals with outliers and non-normal distribu-tions in a correct way.When the startegy is not set to the default"Exact",the important parameter to consider is the n.perms parameter, which should be set to as many as possible.When you just want to have a quick estimation if there are any effects (1)use the default strategy,or(2)use only a limited a mount of permutations.However,when publishing any results, make sure to increase the number of permutations to e.g.15000,for a quick look at any effects the default of setting of doing100permutations should be sufficient:>ctls_quick_scan1<-CTLscan(geno,traits)>ctls_quick_scan2<-CTLscan(geno,traits,strategy="Full",n.perm=100)>ctls_for_publication<-CTLscan(geno,traits,strategy="Full",n.perm=15000)To make the analysis fast,use the nthreads parameter,this will use multiple logical cores when the pc has a multi-core CPU,and can reduce the required total analysis by quite a bit.The algorithm will not allow you to specify more cores then logical processors,so when setting a value too large the algorithm will use only the maximum amount of logical cores detected.>ctls_quicker_scan<-CTLscan(geno,traits,nthreads=4)Non-parametric analysis(using ranked data)is done by default,to minimize the effect of outliers on the analysis. However if your trait distributions are normally distributed you should/can enable paramteric analysis,to obtain more statistical power.To perform parametric analysis set the parameter parametric to TRUE,this use the original phenotype values for the analysis.>ctls_parametric_scan<-CTLscan(geno,traits,parametric=TRUE)A new feature added to allow computation of CTLs on a trait by trait basis is to disable multiple testing correction. By default it is enable to prevent users from overestimating CTLs in their data.Since CTL mapping is doing a lot of statistical testing during a single analysis run,proper multiple testing should always be performed.However when running CTL mapping on a cluster(e.g.on a trait by trait basis)it might be needed to disable the automated multiple testing correction,since each node of the cluster will only see a subset of the data.Disable multiple testing adjustment, byu setting the parameter adjust to FALSE.>ctls_uncorrected_scan<-CTLscan(geno,traits,adjust=FALSE)When the analysisfinishes with an error,you can turn on verbose output,but setting the parameter verbose to TRUE. This will produce a lot of textual output in the R window,which can sometimes help to identify an issue with the dataset.turning on verbose output has no influence on the analysis,and will only make the algorithm produce more text output during the analysis.>ctls_scan<-CTLscan(geno,traits,verbose=TRUE)RESULTS-Plots and CTL networksSo now that we have our ctls calculated we can visualize the results in multiple ways.The easiest way isfirst to plot the two overview CTL heatmaps.Thefirst heatmap shows the summarized LOD scores for the Trait X Marker CTLs. This plot normally looks very similar to a QTL heatmap:>image(ctls,against="markers")The second heatmap shows the summarized LOD scores for the Trait*Trait CTLs,this shows the summarized amount of evidence wefind for each Trait*Trait interaction:>image(ctls,against="phenotypes")After the overview plots it is time to go more in depth,and look at some individual CTL profiles the CTLobject’ctls’contains multiple CTLscans.To print a summary of thefirst scan use:>ctls[[1]]To plot other summaries just replace the1by the trait number.We can also use the plot function on the individual trait CTL scans and show a more detailed view of the(selected)Trait x Marker X Traits interactions detected in the dataset. >plot(ctls[[1]])Get a list of significant CTLsGet significant CTL interactions by using CTLsignificant.>sign<-CTLsignificant(ctl_scan)CTLnetwork and cytoscapeThe CTLnetwork function outputs CTL and(optional)QTL data from the CTLobject into a network format,if mapinfo is available it is used.The network format is relatively easy and can also be parsed by many other programs.As an example we use cytoscape,butfirst lets generate some network inputfiles:>ctl_network<-CTLnetwork(ctls,lod.threshold=5,mapinfo)The CTL edges in the network_full.siffile are annotated as:Trait CTL Trait LOD ScoreThe CTL edges in the network_summary.siffile look like this:Trait CTL Trait avgLOD Score sumLOD Score nTraitsAdditional QTLs are added to bothfiles in the folowing structure:Trait QTL Marker LOD ScoreThis allows thefirst additional infromation column(Nr4)to act as an distance measurement,the closer two traits or two markers are together in the network the stronger a detected QTL or a CTL.Big data and DctlBig datsets require special care,R isn’t the best platform to deal with large scale genomics data from micro-or tilling arrays(Just to mention RNAseq and full DNA strand sequencing).Thus we also provide a commandline version of our CTL mapping routine:mapctl.exe,for more information on all the command line parameters can be found at /mapctl.html The mapctl commandline tool uses the same inputfiles as the R version,and supports qtab the new genotype and phenotype encoding scheme used by qtlHD.It provides only QTL and CTL scanning and permutation features,the output is analyzed in R as outlined above.To load the results from a mapctl scan into R by specifying the inputfiles and the output directory created by the tool using:>ctls<-read.dctl("genotypes.csv","phenotypes.csv",results="<PATH>/output/")Some famous last wordsPackage CTL function overviewCTLscan Main function for Genome wide scan for Correlated Trait Loci work Scan only a subset of traits for CTL using start gene(s) CTLpermute Significance thresholds for CTL by permutationplot Plot CTL profiles at increasing cut-offsimage Image a multiple phenotype scan。
Godot中文教程

场景和节点seraph526 在星期三, 10/14/2015 - 15:10 提交介绍想像一个,你不再是一个游戏开发者,而是一名大厨!将时髦的衣服换成绒边帽和双排扣夹克.现在,你不做游戏,而是为你的客人烹饪新鲜美味的食物. 现在,一位大厨如何烹饪食物?分为两道工序,首先是材料,其次是准备工序的介绍.这样,每个人都可以按照食谱制作,并品尝你伟大的料理.用Godot做游戏和做菜的感觉非常像.使用引擎就像置身于厨房中.在这间厨房里,"节点"就像是装满了要烹饪的原材料的冰箱.有许多类型的节点,一些显示图片,另外一些播放声音,还有一些显示3D模型等等.有许多这样的节点节点让我们开始学习基础知识.节点是创建游戏的基本元素,它有以下几个特点: *要定义一个名字*有可编辑的属性*逐帧运行以接收callback调用*可以被继承(拥有更多的函数) *可以作为其他节点的子节点最后一点很重要.节点可以拥有其他节点作为子节点.当我们按这种方式组织节点,节点便成了"tree".在Godot中,按这种方式管理节点的方式成就了一个伟大的管理项目的工具.因为不同的节点有不同的函数,组合他们会创建更复杂的节函数. 这些可能你还不太清楚,没什么太深的理解.但是所有这些将来都会有所说明.现在最重要的是要记住,节点的存在,而且可以按上述的方式组织节点.场景既然已经定义了节点的存在,下一步便是说明什么是场景.场景由一组节点构成,这些节点被分级管理(以树的形式).它有以下的特征: 一个场景通常只有一个root节点*场景可以被保存到磁盘并加载加来*场景可以被instanced*(后面会详细说明) *运行一个游戏也就是运行了一个场景*一个项目中可以有几个场景,但是要开始游戏,一定要先加载其中一个. 基本上,Godot编辑器就是一个"场景编辑器".它有许多工具用来编辑2D和3D 场景以及用户界面,但所有的编辑器都是都是围绕编辑场景和节点展开的.创建新项目理论是枯燥的.让我们改变主题,实践一下.跟随一个传统的教程,第一个工程将是"helloworld".为此,将使用编辑器. 当Godot在项目外运行时,工程管理器将会出现.它帮助开发者管理他们的项目.要创建一个新项目.要使用"new Project"选项.选择并创建项目路径和定义项目名称.编辑器新项目创建后,下一步就是打开它.这将打开Godot编辑器.下图是我们新打开时编辑器的样子.如前文所述,在Godot中制作游戏,就像置身于厨房.所以,让我们打开冰箱并填加一些新节点到场景中去.我们将以"HelloWorld"项目开始.按下"新节点"按扭:这将打开节点创建对话框,它显示了所能创建的节点的长度列表:这里,我们选择"Label",搜索节点通常更快捷一些.最后,创建Label!.当创建按扭被按下时,发生了很多事:首先,场景变为2D编辑器(因为Label是2D节点类型),Label显示,被选中,在viewport 的左上角. 节点出现在场景树编辑器中(在右上角),label属性出现在Inspector面板中(在右下角). 下一步,我们将改变label的"Text"属性,将其值改为"Hello World":OK,所有的事都准备好了,可以运行场景了!点击顶部的"PLAY SCENE"按扭(或者按F6):哎呀运行前场景要先保存,选择Scene->Save,保存场景为"helllo.scn"一类的名字这里有一些有趣的事情.文件对话框比较特别,它允许保存场景到工程内部.工程的根目录是"res://",它就是资源路径.也就是说,文件只能保存到项目内部.将来,当在Godot中做文件操作时,记住"res://"是资源路径,不管是什么平台或安装路径,这是从游戏内部定位资源的方法. 在保存了场景并按下run之后,"Hello World!"最终正常运行了:成功了!!配置项目好了,是时候对项目做一些配置了.现在,运行场景是唯一执行程序的方法.项目通常都有向个场景,所以要将它们中的一个设置为主场景.主场景将在项目运行时被第一个加载. 这些设置都被存储在engine.cfg文件中,这是一个纯文本的win.ini格式的文件,以便可以方便的编辑.还有很多选项可以设置以声明项目如何运行,为了让设置变得简单,可以通过项目设置对话框设置engine.cfg.要访问此对话框,可通过Scene->Project Settings打开. 窗口打开后,任务是设置一个主场景.我们可以很容易的改变application/main_scene的属性,选择"hello.scn"为主场景修改后,点击"regular Play"按扭(或F5)会运行项目,无论当前是哪个场景被编辑.返回项目设置对话框.这个对话框提供了很多可以填加到engine.cfg的选项,这里显示了它们的默认值.如果默认值是正确的,我们不需要云改变它们.当值改变时,名字左侧的选择框会变为选中状态.这意味着这个值会被保存到engine.cfg文件中,会被项目记住所做的更改. 这里是边注,为将来提供参考,有一些断章取义(毕竟,这只是第一篇教程).通过使用Globals singleton可以填加自定义的设置选项,并且可以实时读取.实例化seraph526 在星期三, 10/14/2015 - 15:27 提交基本原理有一个小型的场景,有些节点在场景中,可能有很多小的项目都可以正常工作,但当项目成长时,越来越多的节点被使用,它很快就会变得难以管理.为了解决这个问题,Godot允许项目被划分为不同的场景.这和其他的一些游戏引擎的工作方式不一样.事实上,差别很大.所以请不要跳过这篇教程.重申一下:场景是节点的集合,按照trew的结构组织,只能有一个单独的节点做为这个树的根结点.在Godot中,场景可以创建并保存到磁盘上.任意场景都可以创建和并按你希望的形式保存.之后,当编辑一个存在的场景或新场景时,其他场景可以被初始化为此场景的一部分.上图中,场景B被加载并初始化为场景A中的一个实例,开始可能会觉得奇怪,但当看完这篇教程时会完全了解是什么回事.初始化, Step by Step要学习如何初始化,让我们从下载一个pre-made souene. 解压下载的场景到任意我们喜欢的路径下.然后,使用import选项加载到项目管理器定位到你的项目,打开"engine.cfg"文件.新的项目会出现在项目列表中.要管理这个项目,可以点击"Edit"选项. 这个项目包含两个场景"ball.scn"和"container.scn".ball场景只是一个带有物体属性的球,container场景有一个很好的碰撞形状.球会被限制指定的区域中.打开container场景,选择根节点:之后,点击"+"按扭,这就是初始化按扭!选择ball场景(ball.scn),球会出现在origin(0,0)位置,围绕场景中心移动ball,如下:点击"Play"按扭,瞧!初始化的小球都落到底部的陷阱中多一些场景中可以有任意多个初始化实例,试下创建更多的球,或者复制(ctrl-D或复制按扭):再一次运行场景:是不是很酷? 这就是实例如何工作的.编辑实例选中实例化的小球中的一个,打开属性编辑器.我们让它的弹力再大些,查看bounce参数,将其值设置为1.0:接下来,会出现一个绿色的"revert"按扭,当这个按扭出现时,意味着我们编辑一个实例场景的属性,将重写这个实例为特别的值.甚至属性在原场景中编辑过.自定义的值将会重写它们.按下"revert"按扭将恢复场景中原来的值.结论实例化看起来很简单,实际上它还有很多功能,下一节实例化教程将会说明剩下的部分...实例化(继续)seraph526 在星期三, 10/14/2015 - 16:06 提交概括实例化有很多方便的用法.一目了然,使用实例,你可以: *细分场景,使之易于管理. *更加灵活的方法替代prefabs(可以工作在更多层级的功能更加强大的实例化). *设计更加复杂的游戏流程甚至是UIs的方法(UI元素在Godot中也是节点).设计语言但是,实例化场景真正强大的点在于它像一门优秀的设计语言一样工作.这使得Godot完全不同于其他的游戏引擎.整个引擎从底层都是围绕这一概念设计的. 用Godot制作游戏时,建议的方式是将其他的设计模式如MVC或Entity-Relationship diagrams放到一边.以更加自然的方式开始制作游戏.开始设想游戏中的可视化元素,那些不光可以被程序员命名,也可以被任何人命名的游戏元素.如,这里是一个简单的射击类游戏的创意思路:任何类型的游戏都可以十分简单的想像出这样的图表.只要写下想法中的元素,然后用箭头标注所有权关系. 这个图表存在后,制作游戏基本就成了为这些节点创建场景,使用实例化(通过代码,之后会介绍,或者通过编辑器)表示所有权关系编写游戏的大部分时间(或者一般的软件)都花在设计游戏模式并让游戏组件适应这一模式上.基于场景的设计取代了这些,使得开发更快更直接,允许直接关注游戏本身.基于场景/实例化的设计节省了大部分上述的工作,使工作变得十分有效率,因为大部分组件被直接映射成一个场景.这样,不再需要或只需很少的模式代码.下面是一个复杂点的例子,一个开放沙盒类型的游戏,其中有很多资源或部分是交互的:用家具搭建一些屋子,之后连接它们.制作一间房子,用这些房间作为这间房子的内部.这间房子可能是堡垒的一部分,堡垒有许多房子组成.最后,堡垒可以被放置到世界地图上.也要填加一些守卫和一些NPCsg到先前创建的场景中. 使用Godot,可以快速的按你所想实现游戏,因为只是多个场景的制作和初始化.编辑器UI也设计成无需程序员操作的模式,所以,通常一个游戏开发小组包括3D或2D艺术家,关卡设计师,游戏设计师,动画师等等,都是通过编辑器界面工作.脚本seraph526 在星期三, 10/14/2015 - 16:08 提交介绍讲了许多关于不编辑就可以让用户制作游戏的工具.这是许多独立开发者的梦想,不学编程就可以制作游戏.这一需求已经存在了很长时间了,甚至是在公司内部,游戏设计者也希望更多的控制游戏的流程.许多产品被制作出来承诺无编辑环境,但是往往结果都是不完美的,太复杂或跟传统的代码比没有效率.结果,编辑在这里停滞了很长一段时间.事实上,游戏引擎中通常的做法是增加一些工具以减少为特殊任务重新编码的数量,加快开发的速度.在这个意义下,Godot为了这个目标做了一些有用的决定策略.首先也是最重要的场景系统.开始,它的目的还不是很明确,但是后来工作的很好.那就是让程序员从架构设计的代码中解放出来.当使用场景系统设计游戏时,整个项目是碎片化的补充场景(不是各自独立的).各场景间相互补充,而不是各自独立.之后将会有大量的例子,但是要记住这非常重要.为此有大量的专业编辑知识,这意味着一个不同于MVC的设计模式.Gosot允许放弃MVC模式的一些效率损失,取而代之的是场景填充(scenes as a complement)设计模式Godot脚本也使用extend模式,也就是说脚本可以继承自有可能的引擎类.GDScriptGDScript (click link for reference)是动态类型脚本语言,以适应Godot引擎,设计它主要有以下几个目标: *第一个,也是最重要的,让它变得简单,熟悉,尽可能的易学. *使代码易读,让错误更安全.语法大部分是借鉴Python.程序员通常要花几天去学习它,不超过两周就会适应它. 像其他动态类型语言一样,高产出(代码易学,可快速编写,没有编译等等)是以平衡效率为代价的,但是引擎中大部分核心代码是用C++编写的(Vector ops,phsics,math,indexing等等),对大部分类型的游戏来说保证足够的效率.任何情况下,如果要求更高的运行效率,重要部分的代码可以用C++重写并且暴露给脚本.允许用C++的类替换GDScript类,而不用改变游戏的其他部分.脚本化场景在继续之前,确保已经阅读了GDScript相关说明.它是一个简单的语言,说明非常短,看一下不用花太长时间.场景设置教程以脚本化一个简单的GUI场景开始.使用增加节点对话框用下面提到的节点创建下面的结构:Panelo Labelo Button 它在场景树中的样子如图所示:同时确保它看起来像是在2D编辑器中,所以它是有意义的:最后,保存场景,一个合适的名字可以是"sayhello.scn"填加脚本选择Panel节点,然后点击"Add Script"图标:会弹出脚本创建对话.这个对话框允许选择语言,类名等等. GDScript在脚本文件中不使用类名.所以该区域不可编辑. 脚本要继承自"Panel"(因为这意味着继承的节点类型为Pannel,这里是自动由编辑器填充的). 选择脚本名称(如果你之前保存过场景,那么脚本会被自动命名为sayhello.gd),之后点击"Create"按扭.这些完成后,脚本就创建了,并且被填加到节点上.你可以在extra图标看到它,也可以在脚本属性找到:要编辑脚本,点击上面的按扭(虽然,UI可以让你直接到脚本编辑器界面),以下是script的模板:它里面没有很多东西.当节点(和它的子节点)进入激活的场景时会调用"_ready()"函数.(记住,这不是构造函数,构造函数是"_init()")信号处理Signals大多是使用在GUI节点中,(虽然其他节点也有这些).当一些特别的动作发生时会发送Signals,可以连接到任意脚本实例的任何程序.在这个步骤中,按扭上的点击(pressed)信号将被链接到一个自定义的函数. 有个GUI用来连接signals,只要选中节点并点击"Signals"按扭:这将显示按扭能发射的信号的列表.但是这个示例不会使用它.我们不想制作一个太简单的东西.所以,请关闭那个屏幕!任何情况下,在这一点上,非常清楚,我们对点击信号有兴趣.我们不通过可视化界面创建它,而是通过代码的方式. 对此,有一个Godot程序员经常会用到的函数,这就是get_node().此函数通过路径得到当前树结构下的节点,或场景上的其它元素.路径是指向挂载脚本的节点.为了得到button,要使用如下语句:```pythonget_node("Button")``` 然后,下一步,挡button点击时,返回调用callback会被填加到脚本.这将会改变label 上文本的内容:```pythonfunc _on_button_pressed():get_node("Label").set_text("HELLO!")``` 最终,按扭"pressed"信号会被连接到_ready()中的callback,使用connect().```pythonfunc _ready(): get_node("Button").connect("pressed",self,"_on_button_pressed") ``` 最终的脚本看起来像这个样子:```python扩展Panelmember variables here, example:var a=2var b="textvar"func _on_button_pressed(): get_node("Label").set_text("HELLO!")func _ready(): get_node("Button").connect("pressed",self,"_on_button_pressed") ``` 运行场景当你按下按扭时,会得到我们预期的效果.脚本(继续)seraph526 在星期三, 10/14/2015 - 16:09 提交处理Godot中的一些行为是通过callbacks或虚函数触发的,所以不需要写一直监测运行的代码.另外,许多事情可以由动画师来完成.但是,通常还是要在每一帧运行一个脚本的情况.有两种运行模式,空闲处理和固定处理.空闲处理通过Node.set_process() 函数激活.激活后, Node.set_process() 每帧都会执行.```pythonfunc _ready(): set_process(true)func _process(delta): [dosomething..]``` delta参数描述从上次调用_process()到现在过去的时间(以秒为单位的浮点类型) 固定处理类似,唯一的要求是要和物理引擎同步. 简单的测试这点的方法是创建一个只有一个Lable节点的场景,代码如下:```python extends Labelvar accum=0func _ready(): set_process(true)func _process(delta): accum+=delta set_text(str(accum))``` 这会显示一个按秒增加的计算器.组节点可以被添加到组中(每个节点下,你想要多少都可以).这对于管理大场景来说是简单而且有效的功能.有两种方法做这个,第一种方法是通过UI,从组按扭实现:第二种方法是通过代码.这是一个有用的例子,如,标记场景,谁是敌人.```pythonfunc _ready(): add_to_group("enemies")``` 这种方式,如果玩家潜行到秘密基地,如果被发现,所有的组中的敌人都会发出警报.通过调用SceneMainLoop.call_group():```pythonfunc _on_discovered():get_scene().call_group(0,"guards","player_was_discovered")``` 上述方法调用组中"guards"的每一位成员的函数"player_was_discovered".或者,通过调用SceneMainLoop.get_nodes_in_group()得到"guards"节点全部列表:```pythonvar guards = get_scene().get_nodes_in_group("guards")``` 关于SceneMainLoop的更多知识点将在后面讲述。
cadence中文详细教程

第一章. Cadence cdsSPICE 的使用说明Cadence cdsSPICE 也是众多使用SPICE 内核的电路模拟软件之一。
因此他在使用上会有部分同我们平时所用到的PSPICE 相同。
这里我将侧重讲一下它的一些特殊用法。
§ 1-1 进入Cadence 软件包一.在工作站上使用在命令行中(提示符后,如:ZUEDA22>)键入以下命令icfb&↙(回车键),其中& 表示后台工作。
Icfb 调出Cadence 软件。
出现的主窗口如图1-1-1所示:图 1-1-1Candence 主窗口 二.在PC 机上使用1)将PC 机的颜色属性改为256色(这一步必须);2)打开Exceed 软件,一般选用xstart 软件,以下是使用步骤:start method 选择REXEC (TCP-IP ) ,Programm 选择Xwindow 。
Host 选择10.13.71.32 或10.13.71.33。
host type 选择sun 。
并点击后面的按钮,在弹出菜单中选择command tool 。
确认选择完毕后,点击run !3)在提示符ZDASIC22> 下键入:setenv DISPLAY 本机ip:0.0(回车)4)在命令行中(提示符后,如:ZUEDA22>)键入以下命令icfb&↙(回车键)即进入cadence 中。
出现的主窗口如图1-1-1所示。
以上是使用xstart 登陆cadance 的方法。
在使用其他软件登陆cadance 时,可能在登录前要修改文件.cshrc ,方法如下:在提示符下输入如下命令:vi .cshrc ↙ (进入全屏幕编辑程序vi )将光标移至setevn DISPLAY ZDASIC22:0.0 处,将“ZDASIC22”改为PC 机的IP ,其它不变(重新回到服务器上运行时,还需按原样改回)。
改完后存盘退出。
然后输入如下命令: source .cshrc ↙ (重新载入该文件)以下介绍一下全屏幕编辑程序vi 的一些使用方法:vi 使用了两种状态,一是指令态(Command Mode ),另一是插入态(Insert Mode )。
ICEM教程

ICEM教程预览说明:预览图片所展示的格式为文档的源格式展示,下载源文件没有水印,内容可编辑和复制根据自己的体会写的操作说明。
一.非结构化网格的一般步骤:1,导入几何体(Ug中定义family,输出tin文件)2,检查体:Repair Geometry (有时需要补面),给边界面取名。
检查体时,如果出现黄线,就说明几何体有问题,红色、蓝色线为正常的。
3,生成body,(非结构化网格必须依据body生成,流通区域建立body,如果要算热态的,固体区域也要生成body;有几个封闭区域生成几个body,且其名称必须不同。
)4, 设置全局网格(global mesh setup< global mesh size>,< set up periodicity>)。
在Global Mesh Setup 设置参数。
为了加密孔上的网格,要用Curvature/Proximity Based Refinement。
Refinement为近似圆时的多边形的边数。
5,设置周期边界网格,周期面上的网格必须一致,所以必须在设置周期面之后才能计算网格(compute mesh)。
使用mesh sizes for parts命令。
周期面必须要定义base(回转轴的基点),Angle (扇形面的角度),在这里旋转轴与ug中的模型有关,如果ug中不是以三个基准轴的话,就要自己找点(用Geometry的做点法来定)。
6,计算网格Compute Mesh。
7,display mesh quality,如果网格质量不行,可以在局部区域使用creat mesh density 命令加密网格。
8,smooth Elements Globaly,Smoothing iterations一般选择25次,Up to quality一般为0.49,choose slovr10.边界条件可以选择在fluent中设置(设置边界条件BoundaryConditions),直接输入网格二.一些操作技巧:要查看内部网格,可以点中mesh再单击右键,选择cut planes;creat mesh density,如果设置的尺寸不对,需要修改,点中Geometry下拉菜单中的density 再单击右键,选择modify density。
